摘要
背景
松材线虫病(Pine wilt disease, PWN)是由松材线虫(Pine wood线虫)引起的一种病害。松材线虫目前,在欧洲和包括日本在内的东亚国家松树林面临的最大威胁。构建详细的DNA标记连锁图谱,鉴定抗水痘病基因/位点,提高抗病性松果体thunbergii,以及其他松果体也容易感染PWD的物种。
结果
总F1188个个体的作图群体从PWD抗性之间的杂交衍生黑鲷品种“田边54”(耐秩2到PWD)和“土佐清水63”(耐秩4到PWD)与PWN接种,以及用于疾病症状进行评价。要PWN电阻进行关联分析中,一组三个地图的构建;使用集成的双向伪测交方法,和共有地图人口型的异花授粉产生的两个亲本的地图。的linkage map of ‘Tanabe 54’ consisted of 167 loci, and covered 14 linkage groups (LGs), with a total genetic distance of 1214.6 cM. The linkage map of ‘Tosashimizu 63’ consisted of 252 loci, and covered 14 LGs, with a total genetic distance of 1422.1 cM. The integrated consensus map comprised 12 LGs with the basic chromosome number of黑鲷,总遗传距离1403.6 cM。利用表型数据和连锁图谱进行的QTL分析表明,PWN抗性由一个显性等位基因控制,该显性等位基因来源于‘Tanabe 54’母本。该主要QTL位于连锁群3上,并被标记为QTLPWD1对于松材线虫病1.
结论
的PWD1位点为一个主要抗性QTL松果体共识LG03,以显性方式作用,赋予松木线虫抗性。从目前的研究中获得的信息将对黑鲷育种计划,以提高对PWD的抗性,也帮助鉴定易感基因松果体物种。
背景
松材线虫(PWN)引起的松材线虫病(PWD)松材线虫[1],当发生导致萎蔫并最终死亡PWN的松树的增加与通过受感染的树水运被损害的程度内的数目,[2那3.].目前,残疾人士是全球松林最大的威胁[2].PWD的致病因子是PWN,而松叶甲虫(Monochamus作为一个载体。PWN被认为起源于北美[4并且在美国广泛分布;然而,并没有发现任何PWD流行病,而且这种疾病只在少数外来松树种发生[5那6].松材线虫是一种人为引进的侵入性病原体,1968年在日本,1982年在中国,1988年在韩国,1999年在欧洲造成了广泛的危害[2].
该PWD是在松林一个长期存在的问题(松果体thunbergiiParl。和赤松摘要。在日本,大约有4000万人口3.松树林自1978年以来一直摧残由PWN [7].作为应对日本PWD的一种手段,日本西部于1978年开始了一项培育抗PWD松树品种的育种项目[8],除北海道外,日本全国各地都在推进相关工程。[7那9].在第一个养殖项目1978年至1984年,16 PWD耐黑鲷克隆(选择效率;从15000株候选树中筛选出抗pwd的92株赤松克隆(选择效率;0.8%)从11,000棵候选树中选出[8].然后将选定的无性系培育成抗pwd的无性系松果体品种。
根据接种PWN后开放授粉后代的存活率对这些品种进行评估;抗性品种开放授粉后代的平均存活率为51%黑鲷和65%赤松,分别比未选择人口多35%及18% [10.].16个品种中15个开放授粉家系的狭义遗传力最大值为0.486 in黑鲷,这表明抗(耐)被继承在添加剂方式[11.].此外,还表明遗传因子的数量黑鲷采用完全双列杂交设计,采用16个品种中的3个品种(包括‘Tanabe 54’)进行双列分析,抗性为1.96 [12.]研究结果表明,大约有两种遗传因素参与其中,并且抗性是加性的;然而,尚未使用分子遗传学方法确定PWD抗性的位点。
识别的遗传决定为PWD电阻不仅对PWD抗性克隆在发展的关键黑鲷,同时也将提供有价值的信息在其他的抗性基因座/基因的筛选和育种策略松果体物种。在东亚国家(中国、韩国、台湾),PWD已经蔓延松果体物种如马尾松羊肉。和p . koraiensis摘要。&调查(13.那14.].在欧洲,PWD也在迅速蔓延,造成了严重的损失p .松树艾特。森林[15.那16.那17.那18那19].此外,松果体欧洲和东亚的森林被认为面临严重破坏的风险,因为栖息地的转移和PWD的蔓延,由于气候变化[20.].另外,各种因素如在PWN,国际贸易木材的状态的昆虫媒介的变化,和不完整的植物检疫处理也可引起进一步的损害[21].为了应对PWD,我们对每个候选个体或家族进行了抗性或耐受性的筛选、选择和遗传评估松果体物种,目的是识别具有抗性的个体和获得遗传资源[18那22那23]。如果可用基因辅助选择(GAS)或标记辅助选择(MAS)工具,抗药性亲本/个体可快速用于促进遗传改良和更具成本效益的育种。
遗传连锁图谱在遗传分析和分子育种计划中起主要作用,并已广泛应用于遗传位点的识别使用农艺性状,如生物或非生物胁迫抗性和产量,它可以促进遗传改良和更具成本效益的养殖.在某些人松果体利用基因组简单序列重复(SSR)、表达序列标签(EST)衍生的SSR或单核苷酸多态性(SNP)标记构建了高密度遗传连锁图谱[24那25那26那27],以及农艺性状(如生长、木材特性和非生物/生物胁迫抗性)的连锁分析和全基因组关联研究(GWAS)已经进行[27那28].之前的遗传连锁图谱黑鲷主要依靠优势标记,如随机扩增多态性DNA (RAPD)标记,以鉴定对松针瘿蚊(Thecodiplosis japonensis),由17个连锁群组成,跨越1469.8 cM, 98个标记[29那30.].然而,利用共显性标记来提高遗传连锁图谱的分辨率和密度是确定目标性状的有效位点和开发MAS标记的关键。
据我们所知,这是PWD阻力的第一QTL定位研究。本研究的目的是确定基因位点与PWD抵抗性黑鲷品种。具体目标为:(1)构建高可信度的遗传连锁图谱黑鲷;和(ii)识别位点为在PWD电阻黑鲷利用构建的遗传连锁图谱。
结果
人群对PWD的抗性评价
我们在一个F1接种后56天(dpi),根据表中所示的评分表1.外部症状在21 dpi时出现,并迅速进展到35 dpi,而疾病症状稳定在56 dpi(附加文件)1).在56 dpi时,PWN抗性的表型分数显示出明显的双峰分布,抗性组(分数1-2)占所有受试个体的50%,而易感组(分数3-4)占所有受试个体的50%(表2)2).表型分离率与1:1 (x2= 0.00,p = 1.00), and were different from 3:1 (x2 = 62.67,p = 0.00). 这些结果表明,在F1人口是由一个主效基因控制。我们使用F的表型得分1人口在56 DPI为QTL分析。
标记多态性
87个基因组SSR标记的六十一(70.11%)和28个(13.59%)的206 EST-SSR标记物中的至少一个映射父母是信息。除了SSR数据外,使用FluidiGM 96.96动态阵列芯片的138(17.97%)和Goldengate测定中的768个SNP中的268个(30.99%)对至少一个映射父母提供信息量;因此,用于连杆映射的标记总数为465.其中,其中98(21.08%)在父母两者中进行偏析:17等位基因(AB×CD);16具有三个等位基因(EF×例如例如);65两位等位基因(HK×HK);140(30.11%)仅在“坦蓬54”(LM×L1)中杂合,而254(48.82%)仅为“Tosashimizu 63”(NN×NP)杂合(表3.).单核苷酸多态性为双等位基因,分别为lm × ll、nn × np和hk × hk。
构建遗传连锁图谱
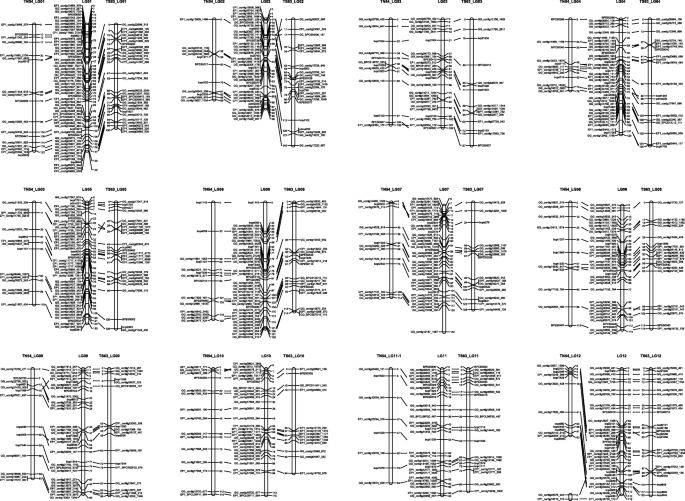
绘制了三张地图;使用集成的双向伪测试交叉方法生成的两个亲本图,以及使用JoinMap 4.1生成的集成共识图(图4)。1).‘Tanabe 54’连锁图谱包含167个位点,覆盖14个位点(TN54_LG01 ~ TN54_LG13),总遗传距离为1214.6 cM。‘Tosashimizu 63’的连锁图谱包含252个位点,覆盖14个位点(TS63_LG01 ~ TS63_LG14),总遗传距离为1422.1 cM。该图谱由12个LGs组成,总遗传距离为1403.6 cM。‘Tanabe 54’和‘Tosashimizu 63’地图标记间的平均距离分别为7.9 cM和6.2 cM。‘Tanabe 54’的5个株系(TN54_LG01, 02, 03, 05, 07)和‘Tosashimizu 63’的5个株系(TS63_LG05, 06, 07, 08, 10)的缝隙宽度均大于30 cM4).
我们将本研究中构建的连锁图谱与p . taeda最近报道的共识地图[24].397条est序列在黑鲷连锁图谱来鉴定连锁群的编号和DNA标记的顺序黑鲷基于p . taeda连锁图。在397个基因中黑鲷连锁图谱中,109条与p . taeda基因,BLASTn e-值从10E-14到0(附加文件2).映射的基因在另一连杆的相对顺序映射有两个松果体物种之间高度相关,R2= 0.898-0.999(附加文件3.),虽然在一些连锁群(LG04)中没有足够的标记来比较定位基因的相对顺序,而在另一个连锁群(LG12)中有少量标记存在明显的位置差异。
一种新的抗PWD基因位点的鉴定
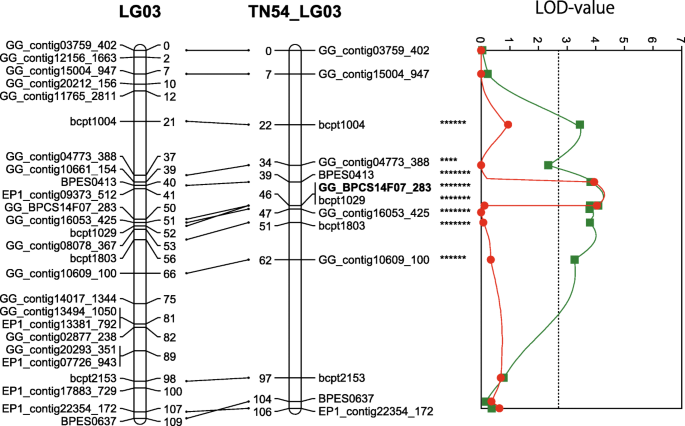
利用构建的遗传连锁图谱和PWN接种试验的表型数据进行QTL分析,发现LG03上有一个抗PWD的位点(图3)。2和表5).该QTL仅在母系‘Tanabe 54’连锁群3中被鉴定,LOD评分最高(LOD = 4.27)。通过1000种排列试验,在α = 0.05的显著性水平下,PWD抗性的LOD评分阈值为2.6,以此作为检测PWD抗性的QTL的阈值。利用区间作图(IM)和多重QTL作图(MQM)在LG03上定位了一个主要QTL,该QTL解释了高达9.9%的PWD抗性表型变异,LOD在38.7 ~ 45.8 cM范围内最大为4.27。另一方面,利用整合图谱对QTL数据进行重新分析,再次确认亲本连锁图谱上检测到的QTL,发现LG03上有1个主效QTL, LOD值最高(LOD = 5.51)。通过1000种排列试验,在α = 0.05的显著性水平下,PWD抗性的LOD评分阈值为4.3,以此作为检测PWD抗性的QTL的阈值。区间作图和MQM作图证实了一个主要QTL在LG03上的定位,该QTL对PWD抗性表型变异的贡献率高达12.6%,LOD在41.8 ~ 50.8 cM范围内最高为5.51。MQM mapping中的辅因子选择在所有得分中选择标记GG_BPCS14F07_283作为该区域的辅因子。
使用非参数Kruskal-沃利斯(KW)测试,PWD阻力有三个基因组SSR标记相关联;一个EST来源的SSR标记,并在2 LG03 EST来源的SNP标记(表6).LG03上的所有qtl均用加性母本效应解释(表2)6).
讨论
在日本,抗病育种项目已经培育出183个抗病无性系黑鲷和246个的抗性克隆赤松自1978年以来虽然抗性性状遗传一直采用耐几个家庭的开放苗[已核实11.,涉及的基因座尚未确定。
本研究是首次尝试检测PWN抗性区域。F1利用抗性品种组合‘Tanabe 54’בTosashimizu 63’的定位群体,对PWN抗性位点进行了定位。对F1个人表明PWN阻力是由单个显性等位基因,这是从“田边54”母本衍生控制。对于性这个主效QTL被定PWD1对于松材线虫病1.
遗传连锁图谱的构建
遗传连锁图谱在遗传研究和育种中具有重要意义,可用于定位所需性状、鉴定构建连锁图谱所需的QTL和众多遗传标记。在以前的研究中,17个连锁群跨越1469.8条 cM,平均标记密度为15.0 cM的构建主要依靠RAPD等显性标记来鉴定松针瘿蚊的抗性(T.丹顶鹤)黑鲷.本发明的联系地图会聚到1203.6cm的1203.6cm的1203.6cm的距离为3.3cm的距离,这限定了59个多晶型SSR标记和369个SNP标记的位置,并且接近先前构造的连杆图的长度。
此外,在本研究中开发和利用EST-SSR衍生和SNP标记使我们能够构建的更翔实的遗传连锁图谱黑鲷通过与从信息比较p . taeda,是迄今为止所研究的松科植物中遗传信息最密集、最准确的[24].在一些连锁组中,标记数量不足以比较定位基因(LG04)的相对顺序,但在连锁组(LG12)中标记数量较少,存在明显的位置差异。为了研究同染色体的保存和染色体之间的重排p . taeda和黑鲷对于所有的连接基团,包括连锁群4和12(LG04和LG12)基因组,多个标记物需要在将来被使用。然而,本研究构建的遗传连锁图谱提供了足够的信息来估计电阻和一些候选基因的主效QTL在将来有针对性的位置。
Paternal and maternal linkage maps constructed using a double pseudo-test cross strategy revealed shorter linkage groups for the maternal map (1214.6 cM) than those for the paternal map (1422.1 cM). The difference in length might be due to differences in the number of informative SNP markers between the overall linkage groups on the maternal (113 SNPs) and paternal (192 SNPs) maps. In addition, the length of LG04 and LG12 showed a noteworthy inconsistency between the maternal and paternal maps. To explore whether this is related to sex, population size or sequencing errors, additional markers need to be developed and more research is required.
PWD抗性的QTL分析
在本研究中,疾病严重程度采用4个严重程度进行评估,并转化为死亡率等二元标准,为QTL的计算提供了可靠的数据。表型评价,评估外部枯萎症状后人工接种是最常用的方法对于评估敏感性和耐药性主机松树,并透露,PWD阻力是一个遗传性状基因家族的基因分析基于死亡率评估(活着的和死去的)(9那11.那32].在本研究中,F中的PWD电阻1群体由一个主基因控制,表明疾病严重导致的死亡率,这一发现得到了实际QTL分析的支持。
利用表型值对F1人口成功检测出一个主要PWD1在构建的LG03基因图谱上发现PWD抗性位点(图3)。2和表5),对应于‘Tanabe 54’母系隔离(图。2,桌子5和6).虽然最初的假设不包括主要的检测PWD1基因仅在“田边54”与PWD阻力,这是一个非常有趣的结果表明PWD性的遗传。In a previous study, the heritability of resistance traits in resistant varieties was evaluated based on survival rates of open seedlings, and it was shown that the survival rate of open-pollinated progeny in ‘Tanabe 54’ and ‘Tosashimizu 63’ was 47.5 and 61.1%, respectively, while that of susceptible pines was 12.5% [33].基于16个抗性品种中15个开放授粉家系存活数据的狭义遗传力估计最大值为0.486,表明抗性(耐受性)将以加性方式遗传[11.,以及遗传因素的数量黑鲷阻力为1.96 [12.].从以往的研究和这项研究的结果,所获得的知识证实,“田边54”具有参与性的PWD杂合等位基因,这种基因是一个主要优势表现出累加效应。因此,我们认为有关PWD性两个遗传因素之一在先前的研究报道12.已经在这项研究中被确认。
在'tanabe 54'中检测到PWD电阻的基因座,但不是'tosashimizu 63'。一个可能的解释是'Tosashimizu 63'是参与PWD电阻的另一个基因座的隐性纯合,但在当前交叉中不能检测到效果。事实上,在'坦蓬54'中检测到的轨迹对表型特性有很少有助于表型特征(由标记的性状解释的表型方差的百分比)。此外,“Tosashimizu 63”中的PWD抗性等位基因也可能受到“TanaBe 54”的等位基因的基本相互作用的影响,以及涉及多个基因的QTL而不是主要基因的可能性。要识别“Tosashimizu 63”的电阻座,有必要再次与易感F交叉'Tosashimizu 63'1通过杂交“田边54”和“土佐清水63”获得的个体。然而,这些易感个体需要被克隆,因为他们将在接种试验丢失,将需要大量时间,到达该配对是可能的年龄。这将是必要培育新材料,以确定未来的“土佐清水63”的抗性基因座进行接种试验。
电阻轨迹(rhg1-b)大豆包囊线虫(异皮线虫属甘氨酸)被认为是一个加性和不完全显性基因,该位点由三个基因在31 kb跨度内的拷贝数变化介导[34那35那36].的轨迹PWD1,对表型性状贡献不大,可能表现出类似于大豆包囊线虫抗性(rhg-1b).
与最近更新的相比p . taeda共识遗传图谱[24,我们能够确定PWD1轨迹位于松果体LG03共识。相对高度保守的基因顺序(共线性)黑鲷和p . taeda联系地图有助于鉴定候选基因或基因组区域PWD1在黑鲷及相关松果体物种。的白松疱锈病的抗性基因和基因组学研究中p . flexilis和p . lambertiana已经鉴定出几个主要的抗性基因(Cr1,Cr2,Cr4)锚定在松果体共识遗传图谱通过与比较p . taeda基因地图(37那38那39那40].通过对其高密度连锁图谱和基因组信息的比较,也可能鉴定出抗PWD的候选基因p . taeda[25那40那41那42那43].At the present stage, the QTL region (41.8–50.8 cM) detected in黑鲷corresponds to the region from 52.1 cM to 117.37 cM on the linkage map ofp . taeda它含有几十个基因。基于来自p . taeda,有可能通过搜索同源基因和DNA标记的发现进一步候选区和基因黑鲷.
造成松材线虫的松树萎蔫病造成以松林构成严重威胁[20.那44].在亚洲(日本、韩国、中国大陆和台湾)和欧洲(葡萄牙和西班牙),一些松果体种类(黑鲷那赤松那p . koraiensis和马尾松来自亚洲,p的抗旱性和p .松树来自欧洲)显示出对PWN的易感性,已经受到PWD的严重影响。其他几个松果体品种也处于严重损害的风险,由于未来气候变化[20.].此外,不仅气候变化,但因素,如在PWN,经由国际贸易受侵染的木质材料的进口,和不完整的植物检疫处理的昆虫媒介的变化可进一步增加损害[21].作为解决PWD问题的长期方法之一,目前正在进行育种计划,以建立抗PWD品种。本研究所获得的连锁图谱和PWD抗性基因/位点,为进一步开展PWD抗性的遗传研究奠定了基础黑鲷和别的松果体易受PWD影响的物种。
在本研究中,我们利用多基因分型系统,构建了一个基础图黑鲷这可以作为解决易感性的遗传方法的基础松果体PWD物种。此外,这是第一个发现PWN抗性的主要位点之一的报告松果体物种。将要进行进一步的研究需要使用DNA微阵列或下一代测序(NGS)来开发多种标志物并构建用于高密度图谱黑鲷.另一方面,需要改进对PWD抗性的表型评价方法,以发现更多对PWD抗性有贡献的QTL。到目前为止,使用DNA微阵列或NGS的基因分型系统和下一代测序仪已经可以用于针叶树物种和高密度连锁图已经构建[24那25那45那46].此外,在表型方面,叶绿素荧光和油树脂流动已经被用来评价对PWN侵染的耐受性p .松树,并对松树对PWN的生理条件进行了评估[18].通过将这些技术和方法应用于其他抗性家庭和抗性黑鲷在今后的研究中,将有可能进一步提高对PWD抗性性状贡献位点的鉴定,实现GAS和MAS在PWD抗性育种中的应用。
结论
在本研究中,我们更新了黑鲷利用SSR标记和来自est的共显性标记,构建了单优势DNA标记的连锁图谱。通过与先前构造的链接图信息进行比较,可以方便地实现p . taeda标记密度在松科中最高。此外,对其进行了QTL分析1利用所构建的遗传连锁图和PWN接种试验的表型数据进行群体定位,在LG03上发现了一个抗PWD的位点。该QTL仅在母系‘Tanabe 54’连锁群3中发现,是抗性的主要QTL。
方法
植物材料
16个抗性品种黑鲷选择为第一个育种计划,并根据接种PWN后开放授粉后代的存活率进行抗性等级(1-5级)排序;更高的存活率被认为意味着更强的抵抗力[33].F1在日本熊本市森林树木育种中心(FFPRI-FTBC)的林产研究所九州地区育种办公室(Kyushu Regional Breeding Office), 188个个体由两个抗性品种(母本‘Tanabe 54’和父本‘Tosashimizu 63’)的单杂交获得。母本的田边54较低阻力水平(要求等级2),和父本Tosashimizu 63的阻力水平较高(四级)。后代在2013年3月被播种,种植场林产品研究所,林木育种中心(FFPRI-FTBC)在茨城县,日本在2014年4月。秧苗以30厘米× 30厘米的间距种植。全基因组DNA1利用DNeasy Plant Mini Kit (Qiagen, Hilden, Germany)从叶片中提取种群,并进行多态性分析,如下所示。
人工接种和分型
2014年7月23日进行了PWN接种。本研究中使用的PWN是Ka-4分离物,自2003年以来一直用于PWD抗性育种项目。接种时,在5℃时用刀剃掉主茎 离地面1厘米处露出形成层细胞,3000条线虫悬浮在50厘米处 将μl无菌水注入剃毛区域。每7周评估一次疾病症状 根据表中所示的评分量表,从14 dpi到56 dpi的天数1.
基因组基因分型SSR
共87个基因组dna衍生的SSR标记(登录号:LC416800-LC416869),其中已有17个标记[47那48那49那50]中,使用从“54田边”和“土佐清水63”,和信息SSR标记,其表明无论是在雌性或雄性亲本杂合模式的基因组DNA被用于基因分型的个体188的F分析1映射人口(附加文件4).Multiplex PCR with three or four SSR primer pairs was performed using a Multiplex PCR Kit (Qiagen), with 2× QIAGEN multiplex PCR master mix, 0.25 μM of each primer pair, and 40 ng of genomic DNA in a total volume of 10 μl. Amplification was performed on a Veriti thermal cycler (Thermo Fisher Scientific, Waltham, MA, USA) using an initial denaturation step of 95 °C for 15 min, followed by 30 cycles of denaturation at 94 °C for 30 s, annealing at 57 °C for 1.5 min, and extension at 72 °C for 1 min, with a final extension step of 60 °C for 30 min. The PCR products (1 μl) were mixed with 0.2 μl GeneScan 500 LIZ size standard (Thermo Fisher Scientific) and 9.8 μl of Hi-Di formamide (Thermo Fisher Scientific) prior to electrophoresis. The length of amplified fragments was analyzed on an ABI 3130xl sequencer (Thermo Fisher Scientific) and alleles were scored with GeneMapper v5.0 software (Thermo Fisher Scientific).
est - SSR标记的开发及基因分型
采用PureLink植物RNA试剂(Thermo Fisher Scientific)从植物针叶和茎中提取总RNA黑鲷“Namikata 37”。cDNA文库构建为每个器官和如先前所述进行测序,然后分析和数据处理[51].以> 50 bp的连续序列为基础,搜索微卫星或SSR基序(最小长度为15 bp,最小重复为二、三、四核苷酸),获得756个无重复的候选SSR标记(登录号:HX995015-HX995770)。针对每个SSR的侧翼序列设计引物对,如前所述[51].16个基因组DNA黑鲷对756个SSR标记进行分析。利用206个具有多态性的SSR标记对188 F进行了基因型分析1后代(附加文件5).基因组SSR分型的PCR和分型分析方法如上述所述。
的EST衍生的SNP标记和基因分型发展
为了构建参考转录组,我们从抗性品种的茎中提取了总RNA,黑鲷' Namikata 73 '和一个易感品种(加树),黑鲷PWN Ka-4分离株在接种后1、3、6、12 h和1、2、3、7 dpi的8个时间点接种‘Kataura 1’。使用RNeasy Plant Mini试剂盒(Qiagen)分离总RNA,使用安捷伦生物分析仪2100系统(安捷伦技术,Palo Alto, CA)评估总RNA的质量。将混合RNA样品通过喷雾、接头连接和乳浊液PCR合成cDNA,并使用Roche 454- flx生命科学测序仪(Roche/454 Life Sciences, Branford, CT)对1/2板进行测序。从454-FLX产生的所有序列数据存储在DDBJ sequence Read Archive [DDBJ: DRA000531]。
Roche 454-FLX系统中的序列使用cutadapt工具修剪适配器序列和poly(A/T)序列,并删除低质量序列和短序列(< 100 bp)。将用于开发EST-SSR标记的Roche 454-FLX reads和Sanger reads用Newbler version 2.6进行组装,得到一个包含22066个contigs和1286个单基因的单基因集。利用NCBI-nr、PGI数据库和KEGG GENES数据库,BLASTX分析共注释了23352个unigenes(附加文件)5).454-FLX和Sanger测序产生的所有组装数据均存放在转录组鸟枪组装部的DDBJ中[DDBJ:IADK01000001-IADK01023202];与EST-SSR标记重叠的序列优先于已经给出的登录号(见附加文件)6).
对于SNP发现,使用Illumina Hiseq 2500平台(Illumina,Branford,CT)对参考序列进行重新排序。使用五种耐药克隆和三个易感克隆。在Nematode接种之前从针和茎中提取RNA,并在与线虫接种后3天,并为每个克隆混合。RNA提取和质量检查遵循相同用于EST图书馆建设的方法。使用Truseq RNA样品制备试剂盒Ver.2(Illumina),对来自每个器官,雾化,适配器连接(包括个体识别指标的指标),桥接PCR和配对结束的cDNA合成,对Illumina Hiseq进行2500平台。在Illumina HiSeq系统上测序的读数也通过Cafadapt修剪适配器序列和Poly(A / T)。然后,通过BWA映射每个库的读取以参考序列[31]和SNPs使用SAMtools软件进行鉴定[52]的默认设置。
对于SNP基因分型,我们在两个独立的SNP测定平台上使用:Illumina的Goldengate平台和KBiosciences Kaspar Assay平台的768个SNPS阵列。通过在抗性和易感克隆(Hirao等人未发表)之间的基因表达分析中选择了在每个平台上提供的768个候选基因和SNPs,并且特别注意每个差异表达的转录物只选择一个SNP。对于Illumina的Goldengate平台,768个选定的SNPS设计与定制寡核苷酸合并的测定(OPA; Illumina Inc.,San Diego,CA)设计,含有等位基因特异性和特异性寡核苷酸用于Illumina Goldengate测定。根据标准制造商的协议,使用768-OPA和Illumina的Beadarray Express读卡进行SNP标记的基因分型。使用GenomeStudio软件(Illumina)推断出每个轨迹的自动等位基因。构成opa的SNP列表及其侧翼区域都在附加文件中呈现7.
对于另一个SNP检测平台,SNPs基因分型采用竞争性等位基因特异性聚合酶链反应KASPar chemistry (KBiosciences Ltd., Hoddesdon, UK),使用Fluidigm (Fluidigm Corp., San Francisco, CA) 96.96动态阵列,根据标准制造商的协议。使用EP1芯片(Fluidigm公司)测量芯片的荧光端点图像,并绘制在两个轴上,并使用Fluidigm SNP基因分型分析软件(Fluidigm, 2011)评估基于EP1测量的基因型。共768个SNP标记(8个标记面板,包含96个SNP)的SNP及其侧翼区域列表见附加文件8.
连锁图施工
采用JoinMap 4.1软件进行连锁分析[53]与种群类型异花授粉(CP) [54].为了构建‘Tanabe 54’和‘Tosashimizu 63’的整合图谱,将在至少一个亲本中显示多态性的所有分离标记(ab × cd、lm × ll、nn × np、ef × eg和hk × hk)应用于JoinMap配置的CP模式中。采用卡方检验计算标记分离率。显示严重扭曲偏析的标记(P-value < 0.001)被排除在地图构建之外。标记物的最小对数比值(LOD)评分为5.0,重组频率为0.45。采用回归映射算法构建联动地图,根据Kosambi映射函数计算地图距离[55].所有其他计算条件设定为默认设置使用。连锁群的数量和在这项研究中标记的顺序进行测定。
双重伪测试交叉策略[54]涂敷了用于遗传连锁图谱的构建。用于映射的LOD阈值设置为4.0和0.45的重组频率。的marker configurations ab × cd, lm × ll, and ef × eg were used for the maternal maps ‘Tanabe 54’, and configurations ab × cd, nn × np, and ef × eg were used for the paternal maps ‘Tosashimizu 63’. The marker configuration hk × hk refers to a bi-parental marker with genotypes hh, hk and kk, and hk was coded as the non-informative marker (−-). The genetic linkage map was drawn with MapChart 2.2 software [56]和同源连锁组用软件进行比较并绘图,使每个整合图谱组位于亲本图谱之间。
链图编号和从EST中提取的DNA标记的顺序黑鲷通过鉴定高度保守的松果体基因或直系基因的本地BLAST分析(Ë值< 10平台以及)p . taeda[24].根据BLAST分析结果,确定连锁群的数目和方向,或定位基因的相对顺序和位置黑鲷物的基础上确定从信息p . taeda连锁图。
QTL分析
使用MapQTL 6.0软件[进行QTL分析57].分别用‘Tanabe 54’和‘Tosashimizu 63’亲本进行QTL初步鉴定;然后利用整合图谱对亲本连锁图谱上检测到的qtl进行重新分析。在本QTL分析中采用二元性状方法,将抗病和感病表型分别转化为0和1(表2)2),分别,并且使用两种不同的方法。First, interval mapping (IM) at 1 cM intervals was carried out to detect QTLs. The genome-wide and LG-specific LOD (logarithm of the odds) thresholds for each QTL were calculated using a permutation test with 1000 repetitions atP< 0.05(5%)。以近侧翼标记为辅助因子,进行多位点QTL定位。其次,对性状进行非参数KW测试模块,以确定最接近被检测QTL的标记的显著性。根据KW检测到的显著SSR基因型计算MapQTL6中QTL的等位效应,采用Knott等人提出的公式[58].
数据和材料的可用性
本研究期间产生或分析的所有数据均包含在本文及其附加信息文件中。
缩写
- 美东时间:
-
表达序列标签
- LG:
-
连锁群
- LOD:
-
概率的对数
- MAS:
-
分子标记辅助选择
- QTL:
-
数量性状位点
- SNP:
-
单核苷酸多态性
- SSR:
-
简单的序列重复
参考文献
- 1。
清原T,线虫德重的接种Y.实验,Bursaphelenchus在松树上。J Japanese For Soc. 1971; 53:210-8。
- 2.
富泰K.松材线虫,松材线虫.植物病理学杂志。2013;51:61-83。
- 3.
Jones Jt,Moens M,Mota M,Li H,Kikuchi T.松材线虫:比较基因组学和宿主-寄生虫分子相互作用的机会。植物病虫害学报2008;9:357-68。
- 4.
Tarès S, Lemontey J-M, de Guiran G, Abad P.物种特异性卫星DNA的使用松材线虫作为诊断探针。植物病理学。1994;84:294-8。
- 5.
松林线虫:对美国森林的威胁?工厂说。1981;65:1022-7。
- 6.
加拿大和美国松材线虫和松材枯萎病的简要概述。见:松树枯萎病。东京:施普林格日本;2008.13页。
- 7.
日本农林水产省林业厅。森林和林业趋势的年度报告- 2009财政年度。东京:日本农林水产省林业厅;2010.(在日本)。
- 8.
Fujimoto K, Toda T, Nishimura K, Yamate H, Fuyuno S.松材线虫抗性育种项目。-该项目十年的研究和成果概要。树种公牛研究所1989;7:1-84。
- 9。
日本松材品种对松材枯萎病抗性选育现状。林业科学技术。2008;4:51-7。
- 10。
Toda T,Kurinobu S.实现了在所选红松后代耐受性中观察到的遗传增益(赤松)及黑松(p . thunbergii)松树枯萎病。Silvae麝猫。2002;51:42-4。
- 11.
户田T,在松树萎蔫病抗性Kurinobu S.遗传改良松果体thunbergii:在苗圃进行人工接种的预先甄别的有效性。J For Res. 2001; 6:197-201。
- 12.
Kuramoto N, Ohira M, Hiraoka Y, Taniguchi T, Kashiwagi M, Inoue Y, et al.;日本黑松和红松全双等位系对松材线虫病抗性的遗传分析。日本社议会。2007;118:393(日文)。
- 13.
赵BG。中国松材线虫病。见:赵bg, Futai K, Sutherland JR, Yuko T,编辑。松树枯萎病东京:施普林格日本;2008.p。年龄在18岁至25岁之间。
- 14.
Shin S-C。韩国松树枯萎病。见:赵bg, Futai K, Sutherland JR, Yuko T,编辑。松树枯萎病东京:施普林格日本;2008.26 - 32页。
- 15.
Burgermeister W、Braasch、Sousa E、Penas AC、Mota M、Metge K等。首次报告松材线虫在葡萄牙和欧洲。线虫。1999; 1:727-34。
- 16.
等。松木线虫的发病率Bursaphelenchus xylophlius斯坦纳与布勒,1934(尼克尔,1970),西班牙。线虫学。2011;13:755-7。
- 17.
索利曼T,Mourits MCM,范德华WERF W,Hengeveld GM,罗比内特C,兰辛克AGJMO。框架造型入侵物种的经济影响,适用于松材线虫在欧洲。Plos一个。2012; 7:e45505。
- 18.
Menéndez-Gutiérrez M, Alonso M, Toval G, Díaz rPinus Pinaster.半同胞家系的耐受松材线虫(松材线虫).牛津大学出版社。2018;91:38-48
- 19。
黄志强,王志强,王志强,等。松材线虫病对欧洲森林的威胁。acta botanica yunica(云南植物研究),2012;
- 20.
平田A、中村K、中高K、小米南Y、田中N、大林H等。未来气候变化情景下松材线虫病的潜在分布。PLoS One。2017;12:e0182837。
- 21.
d’errico G, Carletti B, Schröder T, Mota M, Vieira P, Roversi PF.属线虫发生的最新进展Bursaphelenchus在地中海地区林业。2015;88:509-20。
- 22。
关键词:松材线虫,抗病性,选育见:BG Z, Futai K, Shutherland J, Takeuchi Y,编辑。松树枯萎病东京:施普林格日本;2008.334 - 50页。
- 23。
松材线虫在松材线虫病中的应用(松材线虫)为松树育种计划的工具。对于病理学。2012; 42:521-5。
- 24.
威斯布鲁克JW,Chhatre VE,吴L-S,Chamala S,内维斯LG,穆尼奥斯P,等人。一个共识遗传图谱Pinus Taeda.和Pinus elliottii.两个基因型 - 表型发现群体中联动不平衡的程度Pinus Taeda..G3。2015; 5:1685年至1694年。
- 25.
作者:Neves LG, Davis JM, Barbazuk WB, Kirst mPinus Taeda.L。)基于外显子组序列捕获基因分型。G3。2014; 4:29-37。
- 26.
莫拉加-苏亚索P,Orellana的L,Quiroga的P,Balocchi C,Sanfuentes E,惠滕RW等。一个遗传连锁图谱的发展松果体放射虫纲和抗沥青溃疡病相关qtl的检测。树结构功能。2014;28:1823-35。
- 27.
Bartholomé J, Bink MC, Van Heerwaarden J, Chancerel E, Boury C, Lesur I, et al.;海松育种计划中两个主要性状:高度生长和茎直度的连锁和关联作图。《公共科学图书馆•综合》。2016;11:1-21。
- 28.
熊JS,McKeand SE,Isik的楼WegrzynĴ,尼尔DB,曾ZB,等。数量性状基因座在火炬松的远交系谱影响分叉缺陷。BMC遗传学。2016; 17:1-11。
- 29.
日本黑松松针瘿蚊抗性基因的RAPD标记松果体thunbergii).Theor Appl Genet. 2000; 100:391-5。
- 30.
林毅夫,林志强,王志强,等。松针瘿蚊抗性基因的AFLP标记。Theor Appl Genet 2004; 108:1177-81。
- 31.
李洪波,李洪波。基于盲轮变换的短读快速定位。生物信息学。2009;25:1754-60。
- 32.
Menéndez-Gutiérrez M, Alonso M, Toval G, Díaz R.松林线虫敏感性的变异Pinus Pinaster.艾特。从伊比利亚半岛和法国的种源。安脊髓损伤。2017; 74:76年刊森林科学。
- 33.
田家田。松材线虫病抗性选育研究赤松和黑鲷.树木品种的公牛。2004;20:83-217(日文英文摘要)。
- 34。
金男,HYTEN DL,特AF,DIERS BW。的SCN抗性基因座的精细作图rhg1从PI 88788.植物基因组J. 2010 -b; 3:81-9。
- 35。
郭X,李泰杰,王克,王克,贝利斯,等。基因拷贝数变异在基因工程中的应用Rhg1介导大豆的线虫抗性。科学。2012;338:1206-9。
- 36.
刘胜军,张国栋,张国栋,等。大豆GmSNAP18大豆包囊线虫的抗性有两种类型。Nat Commun。2017;8:1-11。
- 37.
Liu JJ, Schoettle AW, Sniezko RA, Sturrock RN, Zamany A, Williams H, et al.;遗传的映射松果体flexilis主要基因(Cr4),利用基于转录组的SNP基因分型研究白松泡锈病的抗性。BMC Genomics. 2016; 17:1-12 BMC Genomics. 2016;
- 38.
刘JJ,Sniezko RA,Zamany A,威廉姆斯^ h,王N,Kegley A,等人。饱和核不SNP映射确定了功能候选人和选择工具松果体monticola Cr2白松泡锈病抗性的控制位点。acta botanica yunnanica(云南植物研究),2017;
- 39.
KD, Eckert AJ, Wegrzyn JL, Delfino-Mix A, Davis DA, Burton DC等。松类比较作图:糖松(Pinus lambertiana Dougl.)和火炬松(Pinus lambertiana Dougl.)Pinus Taeda.L.)。树遗传学基因组织。2011; 7:457-68。
- 40.
尼尔DB,Wegrzyn JL,史蒂文斯KA,子敏AV,Puiu d,Crepeau MW等。解码火炬松使用单倍体DNA和新颖组件的策略的大规模基因组中。基因组Biol。2014; 15:1-13。
- 41.
Zimin A,Stevens KA,Crepeau MW,Holtz Morris A,Koriabine M,Marçais G等。22 Gb火炬松基因组的测序和组装。遗传学。2014;196:875–90。
- 42.
Zimin Av,Stevens Ka,Crexeau MW,Puiu D,Wegrzyn JL,Yorke Ja,等。错误:使用长读单分子测序的Loblolly Pine Mega-基因组的改进组装。傻瓜。2017; 6:1-4。
- 43.
Wegrzyn JL, Liechty JD, Stevens KA, Wu LS, Loopstra CA, Vasquez-Gross HA, et al.;火炬松的独特特征(Pinus Taeda.通过序列注释揭示的巨基因组。遗传学。2014;196:891 - 909。
- 44.
pukkara T, Möykkynen T, Robinet C.松木线虫潜在传播的比较(松材线虫)在芬兰和伊比利亚模拟与胞自动机模型。对于病理学。2014; 44:341-52。
- 45.
引用本文:刘志强,刘志强,刘志强,等。海松遗传分析的高密度SNP分析(Pinus Pinaster.).摩尔生态资源。2016;16:574-87。
- 46。
三岛K,平尾T,Tsubomura男,田村男,栗田男,鼻M等人。的新颖的推定的致病基因和遗传标记用于柳杉雄性不育识别(日本柳杉粳稻《BMC基因组学》2018;19(1):277。
- 47。
黄志刚,李志刚,李志刚,等。红松微卫星基因座的分离与鉴定。赤松.摩尔生态。2000;9:1186-7。
- 48.
黄志强,王志强,王志强,等。植物微卫星标记的分离与鉴定赤松摘要。调查。使用双重PCR技术。Mol Ecol Notes. 2006; 6:80-2。
- 49.
Iwaizumi MG、Tsuda Y、Ohtani M、Tsumura Y、Takahashi M.最近的分布变化影响了物种遗传多样性和结构的地理梯度赤松自然种群在日本。对于ECOL管理。2013; 304:407-16爱思唯尔。
- 50
宫田,田村,田卫东,等。历史种子利用和转移对老种遗传多样性和结构的地理特异性的影响松果体thunbergii人口。Ecol管理。2018;408:211–9。
- 51
Shirasawa K, Oyama M, Hirakawa H, Sato S, Tabata S, Fujioka T, et al.;的EST-SSR连锁图谱萝卜铜霉素的比较基因组学。DNA Res。2011; 18:221-32牛津大学出版社。
- 52.
李H,手机B,Wysoker A,Fennell T,Ruan J,Homer N等人。序列对齐/地图格式和samtools。生物信息学。2009; 25:2078-9。
- 53.
Van Ooijen JW.JoinMap 4:实验群体遗传连锁图计算软件。荷兰:Kyazma BV;2006。
- 54.
张志强,张志强。巨桉和桉树的遗传连锁图谱桉树urophylla使用伪测交:映射策略和RAPD标记。遗传学。1994; 137:1121至1137年。
- 55.
从重组值估算地图距离。安优生学。1943;12:172-5。
- 56.
Voorrips RE. MapChart:图形化表达连锁图和qtl的软件。J在这里。2002;93:77-8。
- 57.
范Ooijen JW。MapQTL 6.0版,用于绘制二倍体物种实验居群数量性状位点的软件。瓦赫宁根:Kyazma BV;2009.
- 58。
诺特,马隆德,海利,安德森,戴维斯,埃勒格伦等。野猪与大白猪杂交数量性状位点的多标记定位。遗传学。1998;149:1069 - 80。
确认
我们感谢S.服部博士和K.大岛博士在中心组学和生物信息学,东京大学利用罗氏454 FLX生命科学序排序。我们也感谢M.矢野博士和农业生物为GoldenGate的SNP基因分型研究所的Y.片寄博士和给我们使用他们的珠子阵列读写权限。我们感谢M.国寿博士果树科学与秋田县立大学的H.高桥博士的NARO研究所建议在连锁图谱和QTL分析的建设。我们感谢M.高桥博士和FTBC的H.星博士,FFPRI为他们的研究项目以及协调。我们还要感谢我们的同事在FTBC,FFPRI的植物材料的生产和种植田间管理部分。
资金
EST-derived SNP标记和基因分型的开发由“日本黑松和红松抗松材线虫品种技术开发项目”(日本农林水产省林业厅)资助。
作者信息
从属关系
贡献
TH -制备DNA/RNA,测序EST,分析EST数据,对每个DNA标记进行基因分型,构建连锁图谱,进行QTL分析,撰写手稿,修改手稿;KMa -贡献材料,撰写手稿,并修改手稿;HH -分析测序数据,编写DDBJ数据提交;KS - est测序及EST-SSR标记开发KI -开发了基因组SSR标记;KMi -使用Fluidigm EP1系统进行基因分型和取样;MT - est衍生SNP标记;提出研究项目,指导研究过程,撰写论文。所有作者协助稿件准备,阅读并批准最终稿件。
相应的作者
道德声明
伦理批准和同意参与
本研究要求无伦理批准。
同意出版
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1。
的phenotypes in an F1 population before inoculation and from 14 days post-inoculation (dpi) of PWN until 56 dpi. Full view of individual numbers 1 to 96.The phenotypes in an F1 population before inoculation and from 14 days post-inoculation (dpi) of PWN until 56 dpi. Full view of individual numbers 97 to 198.
附加文件2:表S1。
同源标记的对应映射黑鲷和p . taeda连杆组。
附加文件3:表S2。
Syntenic之间的关系松果体thunbergii连锁群和p . taeda连锁群。p . taeda遗传连锁图谱数据基于之前的一份报告[25].
附加文件4:表S3。
87个基因组SSR标记的详细资料松果体thunbergii.
附加文件5:表S4。
来自Roche 454-FLX和Sanger reads assembly的EST BLAST搜索结果摘要。454和Sanger数据的登录ID与DDBJ数据库转录组鸟枪组装(TSA)部分相同。
附加文件6:表S5。
206个EST-SSR标记的详细信息来源于756个非冗余est松果体thunbergii.
附加文件7:表S6。
768个单核苷酸多态性特征松果体thunbergii在Illumina的GoldenGate平台上,描述了位点名称、LG位置、目标SNP和所查询SNP两侧的序列(括号中)。
附加文件8:表S7。
底漆信息为768个SNP松果体thunbergii生物标志物96.96动态阵列(Fluidigm)使用KASPar检测。描述了位点名称、LG位置、目标SNP和所查询SNP的侧翼序列(括号中)。此外,还描述了等位基因特异性引物ASP1和ASP2、位点特异性引物LSP和特异性靶标扩增引物STA的序列。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Hirao, T., Matsunaga, K., Hirakawa, H。等一种新颖的主要基因座为在日本黑松松木线虫抗性遗传图谱和识别的构建(松果体thunbergii).BMC植物杂志19,424(2019)。https://doi.org/10.1186/s12870-019-2045-y
收到了:
接受:
发表:
关键词
- 松果体thunbergii
- 松木线虫
- 松木病
- 电阻
- 遗传连锁图