抽象的
背景
常见的bean (菜豆L.)是世界各地直接人类消费的最重要的食物豆类,因为它代表了营养和健康益处的宝贵组件来源。
结果
我们进行了一项研究,以定义和解释普通bean (菜豆沿着包括波兰在内的南欧国家的东西线从葡萄牙到乌克兰的种质。这是基于p .寻常的并被设计来更好地描述其在欧洲的分布和驯化途径。利用包括地理来源和不同phaseolin类型(与中美洲和安第斯的基因库相对应)的多作物护照描述符,共获得了来自9个基因库和12个地理来源的782份材料。我们选择了33个基因组/基因相关/基因库相关的核简单序列重复标记,覆盖了整个基因组的遗传多样性p .寻常的基因组。总体多态性信息含量为0.800。在没有指明地理来源的情况下,全球结构聚类分析产生了10个遗传聚类。在光伏SHP1标记对欧洲人的基因库分配最有帮助p .寻常的种质资源是光伏shp1-b。Amova的结果表明,89%的分子变异性在782张的过程中共用,其中南欧西部到东部线(包括波兰)的不同地理起源中的4%分子变异性。
结论
本研究表明,欧洲的多样化线p .寻常的种质从南欧(葡萄牙,西班牙,意大利,斯洛文尼亚)的西部地区到南欧的东部地区。这一进展将三个地理上分离的亚组定义为北部(波兰,乌克兰,罗马尼亚),南部(阿尔巴尼亚,保加利亚)和中央(波斯尼亚和黑塞哥维那,塞尔维亚,匈牙利)地区。
背景
普通菜豆(菜豆L.)是世界上最重要的食用豆类直接使用(回顾,见[1])。p .寻常的也是多酚化合物的丰富来源,多酚化合物有许多促进健康的特性[2].
在过去的十年里,有几千份phopeolus.这些基因在欧洲不同地区收集得越来越多,并储存在国家基因库中。目前,phopeolus.基于网络的欧洲植物遗传资源搜索目录(EURISCO)数据库包含超过50,000条记录,其中包括40,000多条特征和评价记录,以及585张照片[3.].由于野生形态的分布,许多研究表明p .寻常的已经经历了至少两个隔离和独立的驯化过程。因此,已记录了两种美洲独立事件(即,在Mesoamerica和Andes中)[1,4,5],来自两大驯养的基因池起源于(审查,见[6])。
Phaseolin是普通豆类主要的种子贮藏蛋白,是一种重要的分子标记物[7].对于中间酵母来源,特定类型的Phaploolin是Sanilac,SAN类型,中美洲,M型和BOYACÁ,如B型[7,8].安第斯基因库的主要phaseolin类型是Tendergreen,作为T型,其次是竞争者,作为C型,这也可以表明与中美洲形式的混合起源。Huevo de Huanchaco或H型phaseolin也属于安第斯族。使用三个indel跨越标记SHP1-A, SHP1-B和SHP1-C进行起源基因库的鉴定,如Nanni et al. [991 .答案:cp .寻常的结果表明,在中美洲和安第斯基因库中均未发现Ib单倍型。
基于形态学变异和微卫星多样性评价,Rendón-Anaya等[10等[11,12,13,14[]报道了伴随驯化的过程,如驯化品种遗传多样性的提高,是由于驯化瓶颈和野生群体与驯化群体之间的杂交事件。普通豆子的野生祖先分布非常广泛,从墨西哥北部到阿根廷西北部,这在当今作物的野生祖先中是不寻常的[15].这些最近的研究还报道了野生和栽培普通豆的分子生态学、干旱相关基因/多态性的选择和适应[16,17,18].此外,最近的两项研究报告称,野生普通豆的全基因组关联预测了对干旱的广泛不同适应[19,20.,物种分化在phopeolus.豆类导致适应和驯化的平行签名[21].遗传多样性评估与结构菜豆coccineusL.清楚地区分欧洲和中美洲基因池,其患有中度至强细胞质瓶颈,随之而来的膨胀p . coccineus通过多次驯化活动传入欧洲[22].
对于欧洲p .寻常的种质资源Carović-Stanko等[8]的研究发现,欧洲物种主要来自安第斯山脉(约68%),中美洲物种较少(约27%)。其余的欧洲品种代表了这两个基因库之间假定的杂交品种。此外,Maras等人[23指出安第斯基因型在构成西巴尔干半岛的五个前南斯拉夫共和国(马其顿除外)的国家中比中美洲更为普遍。这种趋势可能是过去这些国家内部政治监管的结果。这两项研究都使用了分子和种子蛋白标记的组合。然而,简单序列重复(simple sequence repeat, SSR)标记也被证明是不同重要农艺物种群体遗传学研究中信息最丰富、最有效和最具成本效益的工具之一[23,24,25,26,27,28,29,30.,31,32].最近,Campa等人[33发表了一项关于西班牙语的研究p .寻常的多样性(308个本地品系,主要用于快速消费),其中70%的品系与安第斯基因库相关。该面板由3099个单核苷酸多态性(SNP)标记特征,这些标记是通过基因分型测序获得的,揭示了面板内广泛的遗传多样性和低水平的冗余物质[33].此外,利用多样性阵列技术(DArT)对土耳其普通菜豆的遗传多样性和群体结构进行了分析,结果表明,土耳其普通菜豆的主要群体为A(优势)和B(优势),5个未分类基因型。这些代表了三个有意义的杂种优势群体用于育种[34].因此,分子技术的当前进步可以提供人类选择的证据,该迹象是作物驯化期间和之后的众多基因座[35].此外,凭借其不同的适应能力,共同豆类可能适用于有机农业系统[36].
在本研究中,我们在葡萄牙到乌克兰的12(基于国家)地区的基因分为782种,沿着南欧国家(包括波兰)的西部到东部,使用33个地点:24个用于多样性水平的标记;三个与基因池分配相关的标记;和六种基因相关标记。这项研究的主要目标是:(i)识别和收集最理性的不同p .寻常的来自这些欧洲地区的加入;(ii)获得全球多样化水平和遗传结构p .寻常的来自这些欧洲地区,但未说明其地理来源;(iii)界定遗传上的亲缘关系p .寻常的从他们的地理起源方面从这些欧洲区域的进入;(iv)检测等位基因多样性光伏这些欧洲地区种质资源基因库定位的SHP1标记;和(v)确定来自这些欧洲地区的种质资源的遗传潜力,以便进行关联图研究,以帮助确定遗传多样性的新来源。本研究的数据主要用于调查欧洲12个地区普通菜豆的遗传多样性水平和遗传结构,并对其在欧洲的扩展过程和相关多样性有新的认识。这些知识现在可以用于普通豆子的育种,特别是在它对不同环境的适应方面。
结果
基因型遗传的地理来源
在使用多作物护照描述符的初始筛查的基础上,全套样本由12个地理起源(23),波斯尼亚和黑塞哥维那(57),匈牙利(277),意大利(20),波兰(18),葡萄牙(8),罗马尼亚(14),塞尔维亚(218),斯洛文尼亚(97),西班牙(11)和乌克兰(20)(附加档案4:表S1;每个国家/地区的评估中的评估中的数量包括在图4中。4b).来自斯洛文尼亚、意大利、葡萄牙和西班牙的63份材料中,phaseolin类型信息包括C型(23份)、b型(1份)、H型(1份)、S型(18份)和T型(20份),作为基因库分配的方向(附加文件)4:表S1)。
欧洲南部西北线的多元化水平和遗传结构p .寻常的种质资源
在782份供试材料中,频率≥5%的等位基因总数为5.39个,私人等位基因总数为27.52个。所有SSR标记均具有多态性,782份材料的多态性均达到显著水平HWE所有位点的偏差(p< 0.001)。此外,结合位点间的遗传变异参数和其他群体遗传学指标,未发现任何可能是由于显著的零等位基因频率造成的偏差。由期望杂合度(He),为0.822。总的来说,图片平均每个位点有效等位基因为6.842个,平均为22.767个基于“增大化现实”技术(表1).SSR标记(如图片> 0.9)为GATS91 (图片 = 0.923;He = 0.928;不= 13.749), BMd001 (图片= 0.917;He = 0.923;不= 12.801, ata006 (图片 = 0.905;He= 0.912;不= 11.346)和SSR-IAC62 (图片 = 0.925;He = 0.930;不= 14.137),它定义了最高的He(> 0.9)及最高不(> 11)。对于这些位点,基于“增大化现实”技术高(> 23)。Locus BMD001总体上最高基于“增大化现实”技术(43.451)和我(2.944)。最高的F为0.663,其中Ffor locus BM210(−0.794)。SSR-IAC167和BM210的多态性位点最少He(0.585; 0.540;分别为0.540),图片(0.542;0.434),不(2.405;2.172)和我(1.215;0.867)(表1).
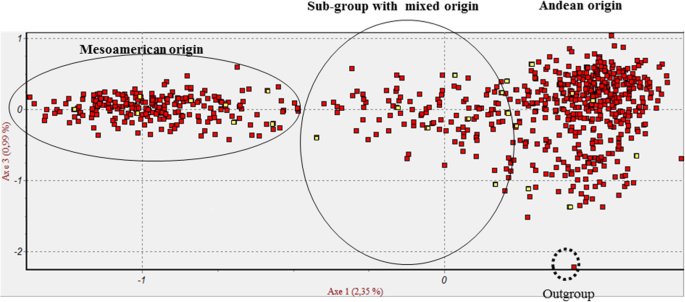
这些从西欧到东欧的南欧国家在FCA的全球分布定义了四个因素,完整性水平为5.35%。总体上,确定了3个主要类群,分别对应于中美洲和安第斯亚类群、混合起源的亚类群和只包含中美洲和安第斯的外类群p . coccineus加入(无花果。1).为了将每个组的成员资格联系起来,每个具有已知类型的phableolin的标准摘要各自用于取向。因此,将具有phoplin T型的含量分配给典型的Andean基因库,将具有Phaploolin S型的含量分配给典型的MesoAmerican基因库,以及具有Pha样力C型的含量是亚组的成员,其中来自Andean提取的混合起源亚组并导致中间群组亚组(图。1,每个集群内的黄色符号)。基于整个系列的资源之间的系统发育关系(附加文件2:图S2),根据NEI的标准遗传距离进行三个主要组的分析[45]和UPGMA聚类方法。
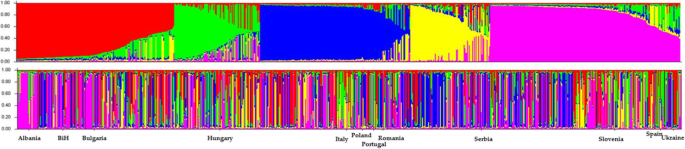
当采用贝叶斯/埃文诺方法而不指定起源种群时,782份种质的混合模型生成了10个遗传聚类(附加文件)1:图S1)。10个聚类间的计算遗传距离均值为0.681 ~ 0.812,对应F统计量(置)从0.013到0.150。
不同地理起源之间的遗传相关性
在33个位点数据的基础上,对12个地理来源的遗传亲缘关系进行了测定。聚类分析结果表明,该种质仅有3个遗传聚类,遗传多样性(全基因组多样性)分别为0.749、0.738和0.772。此外,分子的变异性p .寻常的九个基因库中的含量为3%,而另外97%p .寻常的基因库的分子变异性没有解释显著的结构(AMOVA,p> 0.010)(附加文件6:表S3)。
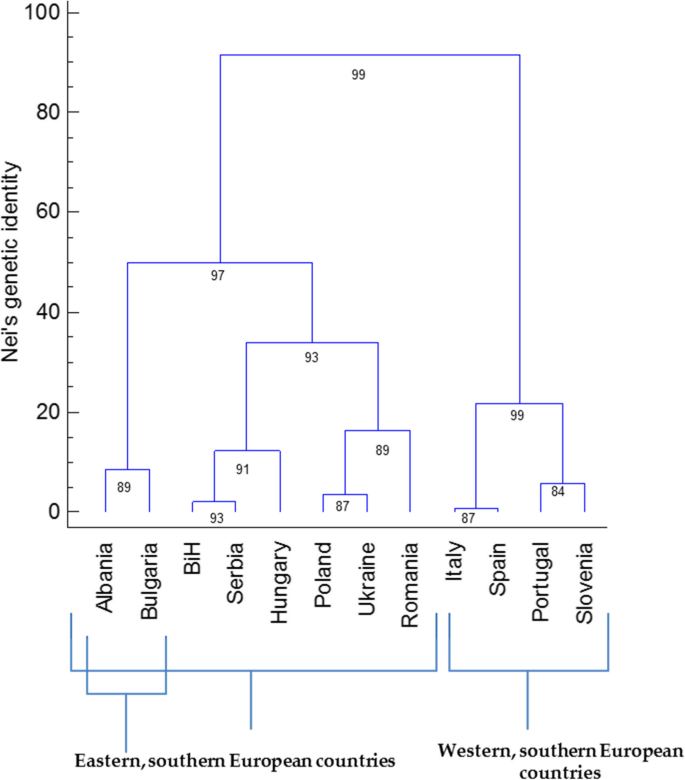
考虑到地理来源(即12个国家)而不是基因库来源的信息,使用贝叶斯/埃文诺算法生成了7个遗传聚类(附加文件3.:图S3)。为了澄清这些分析,我们使用了相同的算法和选择最佳聚类数量的相同标准,但在输入矩阵中给出了不同的原始信息(即,没有指定地理来源的人口[只有一个/欧洲来源];利用基因库来源信息[8个基因库来源];并指定地理起源[12地理起源]);因此,这些结果是不同的(即,不同数量的遗传簇)。在遗传结构的基础上生成的遗传簇的数量从3个(匈牙利)到11个(意大利,波兰)(表)3.).同一聚类间的平均距离在0.669 ~ 0.808之间。AMOVA结果显示,在782份材料中,89%的分子变异是共享的,而在不同地理来源之间只有4%的分子变异,这并没有定义从西欧到东欧的这些南欧地区的显著结构(表)2).
对于每个地理来源内的两两比较,Nei的遗传特性在邻国之间最高,如波兰和乌克兰(0.882),波斯尼亚和黑塞哥维那和塞尔维亚(0.892),以及非邻国之间,如意大利和西班牙(0.810)(图2)。2).利用Slatkin私等位基因法估算地理起源间的基因流[46]为0.541,Barton和Slatkin [47]是0.401。
来自匈牙利、塞尔维亚和斯洛文尼亚的加入没有显示任何偏离HWE对于任何一个基因座。匈牙利、意大利、波兰和斯洛文尼亚的遗传变异水平最高(呃e> 0.8)。考虑基于“增大化现实”技术,来自塞尔维亚的遗传变异达到了适合保存的较高水平(基于“增大化现实”技术 = 1.678). Correspondingly, the highest proportion of molecular variability explained for PCoA (with the first three axes) was for the Serbia accessions (73.5%), followed by the accessions from Bosnia and Herzegovina (71.0%). Moreover, PCoA was performed within each country, and the proportions of explained genetic variability with the first three axes are given in Table3..无论为每个地理原点定义的遗传簇数,平均值He跨越每个来源的集群因阿尔巴尼亚的粮食而异,达到意大利和塞尔维亚加入的0.809(表3.).没有统计学上与hwe差异偏差的基因座(p> 0.05)7:表S4。
考虑到的意思是图片,所应用的33个SSR标记是匈牙利加入(0.815)的最佳信息,以及阿尔巴尼亚加入的最少信息(0.569)(表3.).
与基因池分配相关的PVSPHP1标记的等位基因
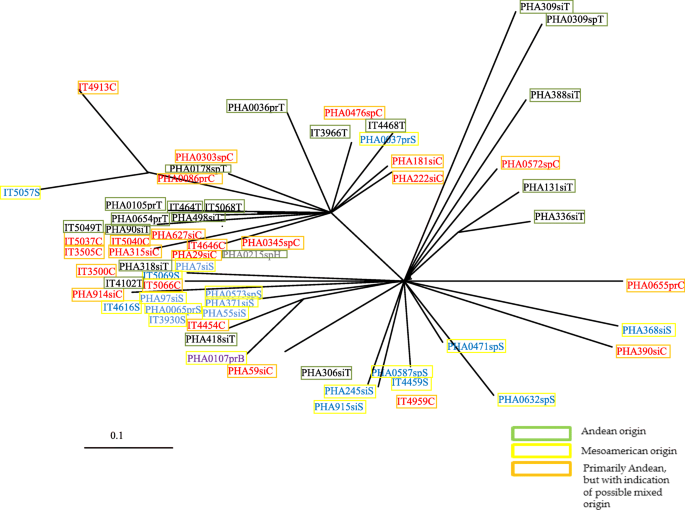
基于有关Pha样力类型的已知信息,在筛选63种附加方案光伏SHP1-A,光伏SHP1-B和光伏SHP1-C位点,以确定是否存在与phaseolin类型或基因库分配相关的显著模式。总的来说,这三个位点偏离了HWE三种主要phaseolin类型(即C、S、T)组的差异有统计学意义(p < 0.01),除了phaseolin T和S基团中的PvSH1-C.在这里,这些标记可用于区分phoplool T和S类型。通常,具有Phopololin C或S类型和P phapOolin C或T类型的含量聚集在一起,除了acce4102t,pHa0107prb,pha418sit和pha318sit除外(图。3.).
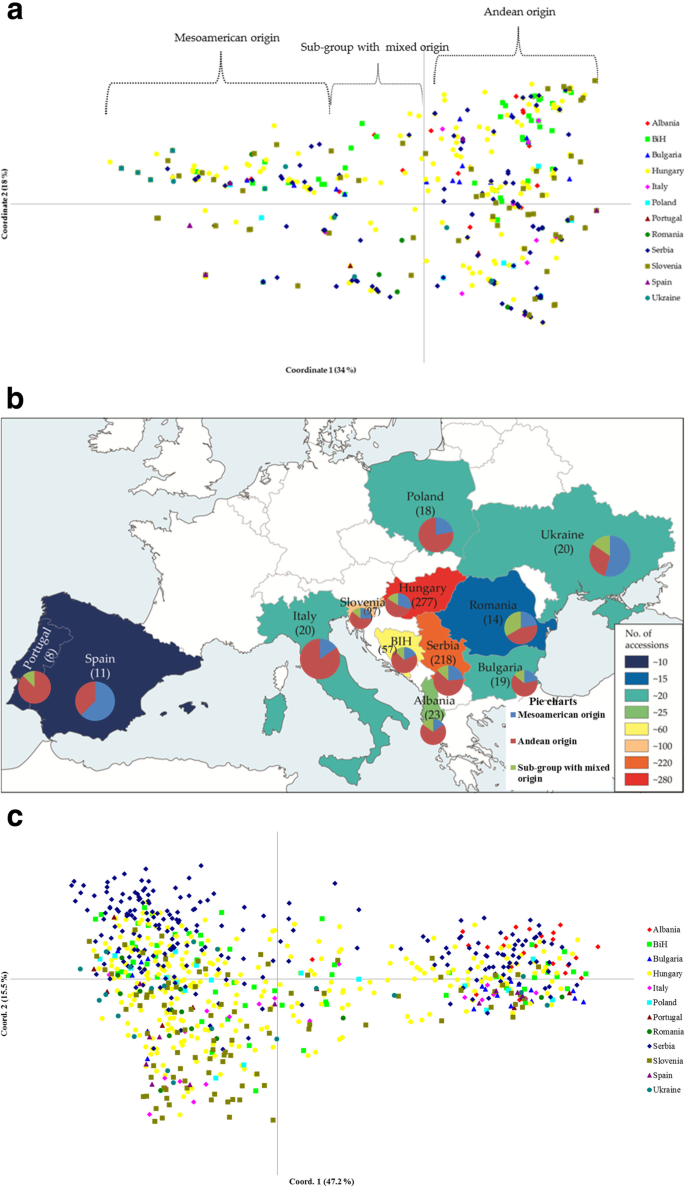
对于地理起源,没有明显的偏离HWE被看见了标记光伏SHP1-C为保加利亚,阿尔巴尼亚和葡萄牙的加入,并为标记光伏西班牙入队的成绩是Sh1-A。分布模式沿着光伏SHP1标记产生两组;来自安第斯起源的主要类群(图。4a,右)通过混合起源的亚群,到中美洲起源的主要群体(图。4一个,左),从而组合来自所有屏蔽地理起源的依次(图。4c)。
一个主坐标分析分布为光伏对12个地理来源的材料进行了SHP1标记。b用饼图映射,显示每个国家的三个子组的细分光伏SHP1标记包括每个地理来源内评估的品种数量(括号中)。地图来源:https://www.morevectors.com/administrative-europe-map-vector/.c来自12个地理起源的所有33个基因座的全局主坐标分析分布
从图中可以看出。4a,所有地理起源的依次沿着前两个主坐标分布,共享相同p .寻常的从安第斯到中美洲的种质资源,并与混合基因库亚群。在详细资料的基础上光伏SHP1标记及PCoA分布如图所示。4B, 12个饼状图叠加在地理地图上,显示了每个国家三个亚组的细分情况。图的背景。4B保留关于物种数量的信息(括号中),以便更好地呈现来自每个地理来源的分析物种。从图中可以看出。4B,Andean Origin是10个欧洲国家的主要。Andean基因型的最低水平是西班牙(37.5%)和乌克兰(30.8%),意大利(84.6%)和葡萄牙(87.5%)最高。平均而言,欧洲人p .寻常的来自12个地理来源的种质占安第斯起源的62%,中美洲起源的25.8%,混合起源的12.2%。4b)。
为了便于比较,图中显示了来自12个地理来源的所有33个位点的PCoA的全球分布。4c. PCoA的前三个轴累计解释了73.5%的遗传变异(图。4c;仅显示前两个轴的数据)。
全球结构分析为地理上的起源光伏Sh1基因座形成5个遗传簇(0.623≤He≥0.8028)(根据结构图中最高的Q值归属于最可能的起源遗传组)。5).
关联图谱研究的遗传潜力
四个基因座适用于关联映射(光伏M04,光伏M21、SSR-IAC62、SSR-IAC66)和两个与角斑/炭疽病抗性相关的位点(光伏M95、SSR-IAC167)为研究对象。共形成10个遗传聚类,782个种质(0.7808≤)被细分为地理类群He≥0.7951)或不(0.8308≤He ≥ 0.8437). No statistically significant deviations fromHWE(p > 0.05)为位点光伏M04,光伏M21和SSR-IAC167为意大利成员,光伏M21、SSR-IAC62和光伏波兰加入M95,光伏M04,光伏M21、光伏M95和SSR-IAC66的葡萄牙加入,和光伏M21、光伏M95为西班牙队效力。
葡萄牙(6.500),波兰(6.500),意大利(6.333)和保加利亚(6.167)加入中检测到频率≥5%的最高等位基因。为波兰获得最高数量的有效等位基因(不= 6.227)及匈牙利(不 = 6.155) accessions. The lowest numbers of both allele frequency ≥ 5% (4.000) and不(3.382)为阿尔巴尼亚入盟。在乌克兰、罗马尼亚和葡萄牙的移民中没有检测到私人等位基因。相比之下,私人等位基因(Np),匈牙利(3.667)、塞尔维亚(2.500)和斯洛文尼亚(1.667)的入学率最高。斯洛文尼亚(4.167)、匈牙利(4.000)、塞尔维亚(3.500)、波兰(3.167)和波斯尼亚和黑塞哥维那(3.167)地区的等位基因数量最高。根据性状相关标记,来自阿尔巴尼亚(He= 0.648;呃e= 0.663),罗马尼亚(He = 0.709;呃e= 0.736)和波斯尼亚-黑塞哥维那(He = 0.712;呃e= 0.719)。关联图谱研究中多样性潜力最大的物种来自意大利(He= 0.812;呃e = 0.834), Spain (He= 0.771;呃e = 0.809) and Slovenia (He= 0.793;呃e = 0.809) (Table4).这些统计数据和表中的数据4计算出来自每个国家/地理来源的摘录,并且与基于所有起源的遗传结构一起计算的遗传集群不兼容。在这里,我们计算了每个国家内的等位基因模式,以比较这些欧洲国家之间的人口统计参数,而不是比较从这些欧洲国家进行的遗传集群。因此,这些分析在所提出的数据方面遵循不同的观点:在没有先前信息的全球范围内(图。1)或与地理起源的先前信息(图。2),或分别对每个国家的细分/国家水平上的等位基因模式进行分析(表4).此外,表3.总结了具有相关的基因座的数据,以显示所有33个洛尼的完整数据和地理详细信息,单独为每个国家/地区计算。
讨论
本研究的第一个主要成果是获得了代表核心的大量高度多样化的资源p .寻常的来自南欧西部到东部地区的种质。根据选择标准(地理来源、生物状态、祖先资料、表型种子特征、phaseolin类型),共收集了来自12个地理来源的782份材料。这代表了适应欧洲生长条件的重要农艺性状的丰富来源。
我们的数据显示高水平的p .寻常的遗传多样性在欧洲基因库中得以维持。三个遗传簇在p .寻常的在九个基因库中保存,与考虑主要的三个亚组p .寻常的起源:中美洲,安第斯和一个混合起源的亚群。为了证实这一点,每个亚类内的平均多样性水平仅在0.738到0.772之间变化。研究中包括的9个欧洲基因库实际上保存了p .寻常的种质根据中间酵母和Andean基因库的相对平等。相比之下,当基因库或地理起源的信息都不用于细分梳理时,产生了10个遗传簇。对来自不同观察结果的群集数量的结果的解释的关键点是遗传结构分析的不同现有输入信息和数据处理。这些集群分析的数据在整个整个782欧洲加入过程中揭示了高水平的遗传多样性。He各基因群的遗传多样性在0.6814 ~ 0.8116之间,包括中美洲和安第斯亚群,以及可能来自这两个基因库的杂种的亚群。这一分配得到了FCA的确认。FCA的重点是通过在没有任何先验信息(如phaseolin类型、系统发育关系)的情况下展示782份材料的分布格局,定义利用多元方法总结出的个体间的总体/一般遗传多样性。该FCA生成了三个主要的类群,分别是中美洲类群和安第斯类群,并在已知phaseolin类型的标准类群(63个类群)的基础上混合,作为区分哪个类群是哪个类群的定位点。本FCA与附加文件中的系统图没有直接联系2:图S2是基于遗传关系(即,NEI的标准遗传距离)与UPGMA聚类方法组合进行的,从而具有与FCA相比使用的不同算法。在这里,我们尤其认为这两个不同的算法生成了三个可以定义为Mesoamerican,Andean和混合的主要组(未在附加文件中分配2:图S2,目的是说明整个782个种质的系统发育)。为了将这些数据与之前的研究进行比较,Maras等人[23]描述了与来自西方巴尔干半岛的两个基因池相对应的两个大簇。本研究中的比较表明,欧洲西部和东部地区的换乘引入了不同的等位基因,从而增加了p .寻常的多样性。这也反映在目前研究的分子数据的第三个亚组中,它显示了中美洲和安第斯的混合起源。
标记多样性及其在西部至东欧的适用性p .寻常的集合
本研究中使用的33个SSR标记已被证明是通过Mesoamerican和Andean种质区分多态性的多态性,物种特异性和信息性,[9,37,38,39,40,41,42,43,44].这些标记具有不同的重复基序,覆盖了所有的连锁基团p .寻常的基因组。总体而言,所选的SSR标记具有较高的适用性,可用于检测这些欧洲种质资源的高度多样化(图片 = 0.800;He= 0.822),确定基因库结构。事实上,从以前的研究数据来看,总体而言图片(作为标记多样性的一般衡量标准)p .寻常的利用36个位点(作为基因组和基于基因的标记的组合,如本研究)的606个栽培基因型的供试材料为0.64(根据野生和栽培材料计算)[48].相比之下,当使用13个基因座时,来自西方巴尔干半岛的种质与西欧洲欧洲线相关的种质更加遗传均匀,所以通过基因座之间的分集参数的较低值(图片= 0.72;He = 0.76) [16].此外,基于26个SSR基因座,He克罗地亚地方种族的发病率为0.572 [7].然后对一套123个SSR标记(SSR- iac系列)进行SSR标记筛选图片当筛选20时,从0.05到0.83变化p .寻常的栽培基因型[40].高基于“增大化现实”技术(平均值22.767)作为遗传变异的基本测度,反映了欧洲普通豆种质的异质性。因此,F可能表明未检测到的空等位基因(例如,在ATA016, BM157)导致了过度杂合度(例如,在BM210)。该SSR标记对匈牙利和斯洛文尼亚种质资源的贡献率最高。同样,对于匈牙利、葡萄牙、塞尔维亚和斯洛文尼亚的入学率,所有位点都显示出显著的偏离HWE(p < 0.05).这种情况表明,这33个基因座可以有效地研究p .寻常的种质。
在西欧到东欧之间的遗传结构和多样化水平p .寻常的种质资源
尽管有高水平的遗传变异p .寻常的种质资源in the present study (i.e., 10 genetic clusters), only 4% of the molecular variability was found among the 12 geographic origins from these western-to-eastern European areas, with 8% among the accessions and 89% within the accessions, and with 97% of the molecular variability maintained within the European gene banks.
本研究表明,基于Nei 's遗传身份矩阵(两两比较)[454],欧洲品种的多样化系p .寻常的种质从南欧(葡萄牙,西班牙,意大利,斯洛文尼亚)的西部地区遵循东部零件,其中包括三个地理上分离的亚组:北部亚组(波兰,乌克兰,罗马尼亚),南部亚群(阿尔巴尼亚,保加利亚)和中央亚组(波斯尼亚和黑塞哥维那,塞尔维亚,匈牙利)。Mesoamerican普通豆立群地位可能在1506年的西班牙和葡萄牙抵达欧洲,并在1528年以类似的方式在秘鲁探索秘鲁[7,49].豇豆分布在欧洲、亚洲和非洲,与中美洲和安第斯的基因库相似,并且在这两个基因库之间形成了杂交品种[7,23,50,51].Maras等人。[23]报告说,对于西巴尔干国家来说,他们的马其顿血统均匀地分布在这两个基因库中,这可能表明p .寻常的主要来自地中海盆地被引入西巴尔干。考虑到这一点,没有参考每种地理起源的征收数量,在匈牙利,意大利,波兰和斯洛文尼亚州内检测到更高水平的遗传多样性呃e > 0.800 and基于“增大化现实”技术 > 4.000. These accessions might therefore be identified for conservation purposes. The highest numbers of genetic clusters (K ≥ 9) were seen for Portugal, Serbia, Ukraine, Italy and Poland, which revealed the highest levels of genetic diversity among the accessions that originated from these geographic areas.
从遗传结构上看,匈牙利的遗传混合品种最多,只有3个遗传簇。对于罗马尼亚品种的PCoA,协方差距离矩阵显示的分子变异比例较低(62.8%)是由前三个轴解释的,这反映出与其他地理来源相比,这些品种分布的重要性较小。
为全局分析使用三个光伏SHP1标记,5个遗传簇,揭示了每个基因库(如中美洲和安第斯)的一个亚群和一个共同的亚群,包括来自混合起源(可能假定的杂种)的资源。这一发现可以通过已知的phaseolin类型的亲缘关系得到简短的证实,其中的系统图还定义了5个类群。典型的安第斯phaseolin T型与中美洲S型(IT4102T、PHA0107prB、PHA418siT)有所区别。当指定人口的地理来源,光伏SHP1标记区分了中美洲和安第斯的全部基因库。此外,这表明在某些特定的地理起源范围内,安第斯物种比中美洲物种更普遍。在没有使用事先资料的情况下,这些附注的全球分布显示出三个主要群体,它们再次代表了整个西欧至东欧的中美洲、安第斯和混合群体p .寻常的种质。结合光伏因此,SHP1标记是唯一有效的分析,以显示这些欧洲种质的详细基因库从属关系光伏SHP-1B是最有区别的轨迹),特别是对于欧洲中央东部的地区。相比之下,光伏SHP1-C基因座对保加利亚、阿尔巴尼亚和葡萄牙的遗传贡献率不高光伏SHP1-A轨迹并不提供西班牙的进入信息。从这些数据看,哪个光伏SHP1适用于地理原产地在欧洲的地区p .寻常的种质很重要。因此,少数选择的SSR标记对于辨别基因池附属物而言优于许多SSR。地理来源和信息性/有用性光伏本研究应用的SHP1标记反映的数据如图所示。4a和图中的饼图。4结合欧洲普通豆种质的所有三个标记可以根据中美洲、安第斯和混合群体来区分它们的起源。目前的研究表明,这种欧洲普通豆的种质大部分是安第斯山脉,除了这里的两个边境国家:西班牙(西欧)和乌克兰(南欧)。正如Nanni等人提出的[9),这三个光伏SHP1内跨标记可用于种质鉴定,特别是用于追踪驯化的中美洲和安第斯基因库的分布。这3个标记是突出不同普通菜豆基因型间多态性的分子标记光伏SHP1基因序列。这三个人的数据光伏SHP1标记成功地表明了欧洲种质的基因库起源(见图)。4作为对比,当使用全部33个标记时,详细提取了全球PCoA分布,考虑到每个加入国家的地理来源(如图所示)。4c)。因此,提出了附加信息以显示这些欧洲国家的换乘的可变性。
如今,技术允许通过许多基因组对数千个SNP标记进行基因分型[52,53,54].最近,Blair等人发表了一项研究,描述了重组和连锁不平衡p .寻常的基因组基于跨豆腿正交基因的SNP阵列以及先进的重组近交线参考映射群(BAT93 X JALO EEP558)和国际可用多样性面板[55].基于基因基因(SSR)标记对于基因组关联研究很重要[56].尽管SSRS从候选基因方法中仍然很重要,但它们不在基因组 - 范式范围内。在本研究中,在整个33个SSR标记中,选择了六个SSR标记作为适合关联映射研究的特征/基因相关标记。这些涉及对Perseguini等人的研究。[57他开发了一套500个普通豆芯的收藏p .寻常的登记入册。对于这些(58个ssr),平均值图片只有0.29。在本研究中,西班牙、斯洛文尼亚和意大利的遗传多样性水平最高呃e和He值。关联图谱研究的最高遗传潜力是在匈牙利、塞尔维亚和斯洛文尼亚的遗传中计算出来的,考虑到来自这些地理来源的遗传的特定等位基因Np值)。基于叶绿体标记和两个不相连的核位点(光伏SHP1), Angioi等[58]估计,欧洲大豆种质中相对较高的比例(约44%)来自中美洲和安第斯山脉基因库之间的杂交。在核评估的基础上进行的本次研究的数据表明,安第斯起源的物种所占比例最高。另一方面,有迹象表明,中美洲和安第斯基因库的基础上的亚群光伏SHP1标记,其中5个遗传簇被定义为这一西欧到东欧的种质。作为比较,Sinkovič等[59]报道,仅根据种子的14种形态特征p .寻常的和p . coccineus来自斯洛文尼亚一个基因库(也反映了它的起源)的种质包括三个群体。
结论
来自本研究的数据显示,这一沿欧洲南部的西对东部地区的782个摘要集中了遗传变异性的宝贵来源。这些具有已知的背景,其通常用于进一步的遗传研究和形成包括来自这些不同地理起源的最有希望的核心的核心集合。此外,我们已经表明,使用的33个基因组和基因特异性SSR标记对于欧洲的多样化研究非常适用p .寻常的并有足够的能力区分中美洲和安第斯的种质资源。的三个跨索引标记光伏SHP1基因在确定这些欧洲物种的基因库从属关系方面是有效的和信息丰富的。我们也证明了这些欧洲基因库已经成功地维持并共享了这种高度多样化的欧洲基因p .寻常的科学和应用的种质资源。
我们在此表明,Mesoamerican和Andean的加入都在通过南欧的西部到东线来到欧洲。在这种蔓延期间,它们适应欧洲农业气候环境,这反映在增加的多样化水平上,这些水平包括MesoAmerican和Andean的混合亚组和对每个地理起源特异的等位基因。然而,鉴于研究问题和正在进行的分析,需要小心地进行与适应有关的任何结论,从而应来自这方面的唯一线索应该来自新分析。作为本研究的透视图,可以建议使用近似贝叶斯计算建模来检查本研究的估计。另一种观点将是使用新方法。cortes等。[54与MesoAmerican基因库中,鉴定了基于84个基因的SNP标记,并且在Andean基因库中检测到略高于群体内部多种多样性。然而,他们强调,SSR标记仍然必须确定分层,亲本多态性和在每个基因库中发生的进化过程[54].此外,Galeano等人[55[,]报道了在候选基因或转录本序列(即表达序列标签)中发现SNP,这在植物遗传学中一直是一种反复出现的策略,主要是因为基于基因的SNP标记本身可能是主要为安第斯多样性面板形成的性状的致病SNPp .寻常的.这两项研究中基于基因的SNP标记可能对欧洲人的分析特别有用p .寻常的种质资源,其中安第斯基因型占优势。
方法
基于多作物护照描述符的初步筛选
我们使用基于网络的EURISCOphopeolus.该数据库鉴定了> 800份样本,涵盖了欧洲大陆不同地区的不同环境,从西班牙到乌克兰,沿着南欧国家的东西线,波兰也包括在样本中。与多种作物护照基本描述符和种子特征相关的现有数据来自不同国家基因库管理员,包括地理来源、生物学状态、祖先数据、种子表型特征、phaseolin类型(对应中美洲/安第斯起源)和种子材料。
这种植物材料的可得性是有限的,根据基因库的可得性,每次入库可获得9至32个种子。在这些鉴定的> 800份材料中,我们成功地获得了782份材料,这是基于某些基因库的可用性和要求的加入的可行性。每一个“增收”代表一个群体或基因型,包括品种、商业品种和地方品种。植物材料的原始来源有:AIS(斯洛文尼亚和阿尔巴尼亚种质)、IBGR-UNI-BL(波斯尼亚和黑塞哥维那)、CPD(匈牙利)、CSIC(西班牙)、DAB(意大利)、IHAR(波兰)、IPK(德国)和IRP-NS(塞尔维亚)。本研究的所有资料都列在附加文件中4S1:表。种子材料通过种子材料转让协议转让,用于L4-7520项目的研究目的。这些文件可从作者处获得。
植物材料和DNA提取
在斯洛文尼亚农业学院(纬度,46°06';经度,14°51';高度,320米A.S.L.)的青年植物在一个温室里生长。在播种之前,用5%次氯酸钠进行化学消毒六个种子。在第一真叶的阶段,采样60mg至100毫克新鲜和健康的植物组织。如Pipan等人所述进行DNA的提取过程。[27,30.和Maras等人[23].使用荧光仪(Qubit 3.0;将DNA稀释至最终均匀浓度为5.6 ng/μL。一个p . coccineus加入被作为外群体加入(即来自斯洛文尼亚的PHA220)。将具有phaseolin型数据的材料作为内部标准或锚点,以确保基因库的正确分配。其中包括中美洲的S型、M型和B型phaseolin,安第斯的T型、C型和H型phaseolin。我们使用基于phaseolin的对照,而不是众所周知的基因库对照,如安第斯基因型Calima (G4494)和Chaucha Chuga (G19833),以及中美洲基因型ICA Pijao (G5773)和Dorado (DOR364)。有必要包括和分析西欧和东欧国家(例如斯洛文尼亚、意大利、西班牙、葡萄牙;附件见图。3.)来自本研究中的其他分析的探讨(即,他们的本土地理环境和起源)。
基因分型和片段分析
以确定植物的多样性和遗传结构p .寻常的利用了由9个研究小组开发的33个基因组特异性标记[9,37,38,39,40,41,42,43,44].选择的标准是分布在所有连锁群(即24个SSR标记)。利用三个indel跨越标记SHP1-A、SHP1-B和SHP1-C进行起源基因库的鉴定[9].来自Benchimol等人的六个标记。,Campos等人。和哈伊等人。用过[40,41,43],因为这些与特定基因相关联(有关详细数据,请参阅附加文件5:表S2)。
PCR反应进行的最后一卷11.5μL包含8.4 ng基因组DNA和以下试剂,与初始浓度:1μL(10×PCR缓冲(Biotools、西班牙),从每个10毫米0.2μL的混合核苷酸(Sigma-Aldrich ZDA), 0.5μL MgCl 50毫米2(Biotools, Spain), 0.1 μL 10 μM正向引物(Sigma-Aldrich, ZDA), 0.25 μL 10 μM反向引物(Sigma-Aldrich, ZDA), 0.183 μL 10 μM 5 '荧光标记通用引物(6-FAM, NED或HEX;和0.5 μL 5 U Taq DNA聚合酶(Biotools,西班牙)。每个SSR的正向引物都添加了一个18 bp的尾序列5 ' -TGTAAAACGACGGCCAGT-3 ' (M13(−21)),由Schuelke [60].
使用热循环仪(Veriti; Thermofisher Scientific)在以下“触控”条件下进行PCR分析,这依赖于每个引物对:94°C 4分钟;在94℃下循环1分钟;每循环在0.7℃下降低60(62)℃至49.5(51.5)℃的温度下降30秒;72°C 1分钟;然后在94℃下进行23个循环30秒;53°C 30秒;72°C 1分钟;和最终延伸5分钟,72°C。在遗传分析仪(3130XL;应用生物系统)上进行片段分析,并且通过与内部尺寸标准(Genescan-350 ROX;应用生物系统)进行等级长度使用Genemapper 4.0软件(应用生物系统)来确定等位基因长度。
数据分析
以下参数使用Identity 1.0计算[61]及“微卫星工具包”[62软件:变异性,包括观察到的等位基因数量(没有);预期的杂合性(He);和多态信息内容(图片).使用GenAlEx 6.1软件对12个地理类群中的每个类群计算了以下进一步的参数[63]:有效数量(不)及私人(Np等位基因;香农的信息指数(我);固定指数(F);频率为> 5%的等位基因;比例≤25%和≤50%的共有等位基因数;无偏期望杂合度(呃e);Hardy-Weinberg平衡的位点特异性偏差(HWE).采用相同软件进行主坐标分析(PCoA)。利用Arlequin软件进行分子方差分析(AMOVA),以确定不同种质(地理来源、基因库来源)组内和组间的遗传变异[64].由于应用了成熟的标记,空等位基因的频率没有显示,而扩增是真实的和未被掩盖的。初步计算表明,不需要提供位点间的空等位基因结果。此外,对标记和群体多样性参数(如f统计量、遗传距离、HWE以及其他)没有显示出任何可能受到显著水平的无效等位基因影响的干扰。每个地理来源的基因流估计是通过使用Slatkin的私人等位基因方法计算迁移的有效数量来进行的[46]和Genepop 4.1.0软件[65,报告了巴顿和斯拉特金的更正估值[47].人口1.2.28软件[66]用于计算Nei’s标准遗传距离[45,并在bootstrapping(100次)下进行聚类分析,构建具有算术平均(UPGMA)树状图的非加权对组方法。使用TreeView软件将UPGMA的树状图可视化[67].树形图是生根的p . coccineus它最初并没有被定义为外群体,因为它被视为与p .寻常的外来群体给予无根树木极性,并允许重建祖先的特征状态和区域[68].Statgraphics Centurion XVI(2009)使用平方欧几里得方法和Ward 's算法在bootstrapping(100次)下定义地理起源之间的遗传亲缘关系的树形图。结构2.3.3软件[69]被用来使用贝叶斯方法推断种群结构,解释为每一个加入属于每个遗传簇的后验概率(问价值)。每个独立运行K(从1到15)的混合模型,燃烧周期为10,000,然后是100,000马尔可夫链蒙特卡罗重复。真正的K值的选择是基于运行之间的可能性比的增加,使用Evanno deltaK统计(70,并在Structure Harvester软件中实现[71].当一个成员的百分比在80%到100%之间时,一个加入被分配给一个特定的集群。在使用Structure软件时,在输入矩阵中使用了不同的信息来指定人口(起源)的数量(即,没有指定人口或地理起源[只有一个/欧洲起源];利用基因库来源的信息[八个基因库来源];并指定地理来源[12地理来源]),在相同的标准下为不同类型的分析选择最佳聚类数量。利用Genetix 4.05软件对来自不同地理来源的材料进行了全球因子对应分析(FCA)和连锁不平衡分析[72]和等位基因丰富(基于“增大化现实”技术),这些资料均使用FSTAT 2.9.3.2软件获取[73].特定的数据集在计算之前被标准化,以避免数据集大小的影响。
可用性数据和材料
在此包括支持本研究结论的数据集和补充材料。
缩写
- AMOVA:
-
分子方差分析
- 基于“增大化现实”技术:
-
等位基因丰富
- F:
-
固定索引
- FCA:
-
的阶乘对应分析
- He:
-
预期的杂合性
- HWE:
-
哈迪温伯格平衡
- 我:
-
香农信息指数
- 不:
-
有效等位基因数
- Np:
-
私人等位基因数量
- 个人电脑:
-
菜豆coccineus
- PCoA:
-
主坐标分析
- 图片:
-
多态信息含量
- 光伏:
-
菜豆
- SSRs:
-
简单序列重复
- 呃e:
-
无偏预期杂合性
- UPGMA:
-
算术平均数的非加权对组方法
参考
- 1.
belucci E, Bitocchi E, Rau D, Rodriguez M, Biagetti E, Giardini A,等。基因组学的起源,驯化和进化菜豆.出自:Tuberosa R, Graner A, Frison E, editors。植物遗传资源基因组学。柏林:施普林格;2014.p . 483 - 507。https://doi.org/10.1007/978-94-007-7572-5_20.
- 2.
Ganesan K, Xu B. Polyphenol-rich dry common beans (菜豆L.)及其健康益处。int j mol sci。2017; 18:2331。https://doi.org/10.3390/ijms18112331.
- 3.
ecpgr。欧洲搜索植物遗传资源目录。http://eurisco.ecpgr.org.访问日期:2018年7月15日
- 4.
D.普通豆子的起源、驯化和进化(菜豆l .)在:Van Schoonhoven A, Voysest O.普通豆类:作物改良研究。CAB Int: Wallingford, UK and CIAT, Cali 1991。7-53。
- 5.
Bitocchi E, Rau D, Bellucci E, Rodriguez M, Murgia ML, Gioia T, Santo D, Nanni L, Attene G, Papa R. Beans (phopeolus.SSP。)作为理解作物演变的模型。正面。植物SCI。2017; 8:722。https://doi.org/10.3389/fpls.2017.00722.
- 6.
Bitocchi E,Bellucci E,Giardini A,Rau D,Rodriguez M,Biagetti E等。分子分析普通豆的平行驯化(菜豆)在中美洲和安第斯山脉。新植醇。2013;197:300-13。https://doi.org/10.1111/j.1469-8137.2012.04377.x.
- 7.
Carović-Stanko K, Liber Z, Vidak M, Barešić A, Grdiša M, Lazarević B, Šatović Z.克罗地亚菜豆地方品种遗传多样性。植物学报,2017;https://doi.org/10.3389/fpls.2017.00604.
- 8.
意大利普通豆地方品种:多样性与种群结构。作物学报。2013;60:1515-30。https://doi.org/10.1007/s10722-012-9939-y.
- 9.
与SHATTERPROOF相似的基因组序列的核苷酸多样性(光伏SHP1)在家养和野生普通菜豆(菜豆l .)。Al Appl Genet。2011; 123(8):1341-57。
- 10.
Rendón-Anaya M,等。普通豆的起源和驯化的基因组历史推出了其最接近的姐妹物种。生物生物。2017; 18(1):60。
- 11.
Beebe S, Toro O, González AV, Chacón MI, Debouck DG。芸豆的野草作物复合体(菜豆L.,Fabaceae)在秘鲁和哥伦比亚的andes,以及它们对保护和繁殖的影响。遗传资源作物evol。1997年; 44:73-91。
- 12.
Payródelacruze,gepts p,colungagarcíamarínp,Zizumbo-Villareal D.野生种群遗传多样性的空间分布菜豆L.来自Guanajuato和Michoacán,墨西哥。Genet Res作物Evol。2005; 52:589-99。
- 13.
Martínez-Castillo J, Zizumbo-Villarreal J, Gepts P, Delgado-Valerio P, Colunga-GarcíaMarín P.利马豆野生居群结构和遗传多样性(种豆科来自墨西哥尤卡坦半岛。作物科学。2006;46:1071 - 80。
- 14.
陈志强,王志强,王志强,等。农户经营品种的遗传组成及其空间分布特征phopeolus.豆类种植:瓦哈卡的一个村庄的例子。作物学报。2012;52:1721-35。
- 15.
王志强,王志强,王志强,等。野生动物范围扩展的空间和时间尺度菜豆.生物医学进展。2017;35(1):119-31。
- 16.
CortésAJ,Chavarro MC,MadriñánS,这是D,Blair MW。在干旱相关的分子生态学和选择Asr野生和栽培菜豆的基因多态性(菜豆l .)。BMC麝猫。2012;13.1:58。
- 17.
Cortés AJ, D,查瓦罗C, Madriñán S,布莱尔MW。野生和栽培菜豆干旱相关DREB2编码基因的核苷酸多样性(菜豆l .)。Theor app Genet. 2012; 125.5:1069-85。
- 18.
Cortés AJ, Monserrate FA, Ramı ' rez-Villegas J, Madriñán S, Blair MW。野生植物种群的耐旱性:以普通豆类为例(菜豆l .)。PLoS ONE。2013; 8 (4): e62898。https://doi.org/10.1371/journal.pone.0062898.
- 19.
Blair MW,CortésAJ,这D.鉴定野生和栽培常见豆类的erecta基因及其干旱适应关联。植物SCI。2016; 242:250-9。
- 20.
Cortés AJ,布莱尔大师。通过测序和野生普通菜豆基因组-环境关联的基因分型预测广泛的不同的干旱适应。植物学报。2018;9:128。https://doi.org/10.3389/fpls.2018.00128.
- 21.
Cortés AJ, Skeen P, Blair MW, Chacón-Sánchez MIphopeolus.豆子胁迫适应和驯化的并行签名?植物科学学报2018;9:1816。https://doi.org/10.3389/fpls.2018.01816.
- 22.
等。遗传多样性和结构的世界范围的收集菜豆coccineus中国科学(d辑:地球科学)2011;122(7):1281-91。
- 23.
王志强,王志强,王志强,等。来自西巴尔干半岛的普通豆类遗传多样性的检验。acta Hortic sinica . 2015;140(4): 308-16。
- 24.
刘志刚,刘志刚。中国土地利用/覆被变化的时空特征。生态学报芸苔属植物显著L.维持遗传多样性和基因流动潜力:实证评价。伦敦:Intechopen;2018. p。27-44。https://www.intechopen.com/books/brassica-germplasm-characterization-breeding-and-utilization/spatial-and-temporal-assessment-of-brassica-napus-l-maintaining-genet ic-diversity-and-gene-flow-pote
- 25.
Sinkovič L, Pipan B, Meglič V, Kunstelj N, Nečemer M, Zlatić E, Žnidarčič D.斯洛文尼亚甘薯品种的遗传分化(Ipomoea Batatas.),以及不同培养基对其农艺和营养性状的影响。中国农业科学。2017;12(4):350-6。https://doi.org/10.4081/ija.2017.949.
- 26。
Pipan B,ŠuštarVozličJ,MegličV.栽培,品种结构和交叉授粉的可能性芸苔属植物显著L.在斯洛文尼亚。Acta Agric Slov。2011; 97(3):247-58。
- 27。
王志强,王志强,王志强,等芸苔属植物显著l . Genetika。2013;45(2):309 - 27所示。https://doi.org/10.2298/GENSR1302309P.
- 28。
Šuštar Vozlič J, Maras M, Zupin M, Sedlar A, Zadražnik T, Razinger J, Kidrič M, Vodnik D, Pipan B, Meglič V.普通大豆抗非生物胁迫育种的遗传、蛋白质组学和生理背景。正确答案:植物育种:将科学带入生命的艺术。摘要,2016年第20届EUCARPIA大会,瑞士苏黎世。苏黎世:Agroscope;Kölliker, R(ur.), Boller, B. 2016。p。299。
- 29。
黄志强,王志强,王志强,等。红薯种质资源的遗传评价。农业科学与技术。2017;19(5):1139-50。
- 30.
Pipan B,žnidarčičd,Megličv.甘薯遗传多样性评估[Ipomoea Batatas.(l)。]在不同倍性水平上应用两种毛细管平台。生物技术学报。2017;92(2):192-8。https://doi.org/10.1080/14620316.2016.1249963.
- 31.
以色列种质资源:本地葡萄株系的表型和基因分型(葡萄l .)。葡萄。2015;54:87-9。
- 32.
Rusjan D,Pipan B,PelengićR,MegličV.基因型和葡萄的表型歧视(葡萄' Vitovska '和' Garganja '教派的变种。acta Hortic sinica, 2012;77(2): 84-94。
- 33.
Campa A,Murube E,Ferreira J.遗传多样性,人口结构和联系在西班牙普通豆类多样性面板中,通过逐序列显示。基因。2018; 9(11):518。
- 34.
利用表型和全基因组dartseq生成的硅达特标记信息分析土耳其普通豆基因库的遗传多样性。《公共科学图书馆•综合》。2018;13 (10):e0205363。https://doi.org/10.1371/journal.pone.0205363.
- 35.
Smýkalp,纳尔逊Mn,Berger JD,冯Wettberg EJB。遗传变化在作物驯化过程中的影响。农艺学。2018; 8:119。https://doi.org/10.3390/agronomy8070119.
- 36.
关键词:大豆,有机栽培,多环境评价,遗传特性可持续性。2018;10:777。https://doi.org/10.3390/su10030777.
- 37.
黄豆中富含at的微卫星的特性研究(菜豆l .)。Al Appl Genet。2008; 118(1):91-103。https://doi.org/10.1007/s00122-008-0879-z.
- 38.
大豆全基因组锚定微卫星图的开发(菜豆l .)。Al Appl Genet。2003; 107(8):1362-74。https://doi.org/10.1007/s00122-003-1398-6.
- 39.
Gaitán-Solís E, duke MC, Edwards KJ, Tohme J.普通豆类的微卫星重复(菜豆):分离、鉴定和跨种扩增phopeolus.ssp。作物科学。2002;42(6):2128 - 36。https://doi.org/10.2135/cropsci2002.2128.
- 40.
Benchimol LL, Campos T, Carbonell SAM, Colombo CA, Chioratto AF, Formighieri EF等。普通菜豆遗传多样性结构(菜豆利用新开发的微卫星标记研究429个中美洲和安第斯起源的品种。植物资源学报。2007;54:1747-62。https://doi.org/10.1007/s10722-006-9184-3.
- 41.
Hanai LR, Campos T, Camargo LEA, Benchimol LL, Souza AP, Melotto M, Carbonell SAM, Chioratto AF, Consoli L, Formighieri EF, Siqueira MVBM, Tsai SM, Vieira MLC。从基因和基因组来源分离的普通菜豆SSR位点的开发、表征和多态性比较分析。基因组。2007;(3):266 - 77。https://doi.org/10.1139/g07-007.
- 42.
Hanai LR,Santini L,Camargo Lea,Fungaro MHP,Gepts P,Tsai Sm,Vieira MLC。使用EST-SSR,RGA,AFLP和推定功能标记扩展公共豆的核心图。mol品种。2010; 25(1):25-45。https://doi.org/10.1007/S11032-009-9306-7.
- 43.
Campos T, Oblessuc PR, Sforça DA, Cardoso JMK, Baroni RM, Sousa ACB, Carbonell SAM, Chioratto AF, Rubiano LLB, Souza AP.利用微卫星进行遗传连锁分析,检测普通豆类生长习性的遗传(菜豆l .)。摩尔品种。2011;27:549-60。
- 44.
Yu K, Park SJ, Poysa V, Gepts P. genetic analysis of simple sequence repeat (SSR) markers in common bean [j] .应用生态学报,2017,29(6):758 - 763。菜豆l .)。J在这里。2000;91(6):429 - 34。https://doi.org/10.1093/jhered/91.6.429.
- 45.
居群间遗传距离。我自然。1972;106(949):283 - 92。
- 46.
自然群体的基因流动。Ann Rev Ecol Syst. 1985; 16:393-430。
- 47.
Barton NH, Slatkin MA。稀有等位基因在细分群体中分布的准平衡理论。遗传。1986;56:409-16。
- 48.
MW布莱尔,Soler A, Cortes AJ。普通菜豆的多样性及种群结构(菜豆l .)。7。2012;(11):e49488。
- 49.
Gioia T,Logozzo G,Attene G,Bellucci E,Benedettelli S,Negri V,Papa R,Spagnoletti ZP。引言瓶颈的证据和广泛的基因间池(Mesoamerica×Andes)杂交在欧洲常见的豆类(菜豆l .)种质。《公共科学图书馆•综合》。2013;8:e75974。https://doi.org/10.1371/journal.pone.0075974.
- 50.
chávez-servia jl等。谷物豆类。在:Tech,Rijeka,克罗地亚,卷。10;2016. p。63439。
- 51.
Papa R, Nanni L, Sicard D, Rau D, Attene G.遗传多样性的进化菜豆L. In: Motley TJ, Zerega N, Cross H, editors。达尔文的收获:作物起源、进化和保护的新方法。纽约:哥伦比亚大学出版社;2006.121 - 42页。https://doi.org/10.7312/motl13316-007.
- 52.
布莱尔MW,CortésAJ,Penmetsa RV,Farmer A,Carrasquilla-Garcia N,Cook Dr。一种高吞吐量SNP标记系统,用于父母多态性筛选,以及普通豆中的多样性分析(菜豆l .)。acta physica sinica . 2013;126(2): 535-48。
- 53.
关键词:野生群体,SNP发现,基因多样性,连锁不平衡美洲山杨.植物基因组学。2012;8(4):821-9。
- 54.
Cortés AJ, Chavarro MC, Blair MW。普通菜豆的SNP标记多样性(菜豆l .)。acta physica sinica . 2011; 23(5):827。
- 55.
Blair Mw,CortésAJ,农民广告,黄W,Ambachew D,Penmetsa RV等。在共用豆的参考SNP地图中,重组率和连锁不平衡率(菜豆l .)。PLoS ONE。2018; 13 (3): e0189597。https://doi.org/10.1371/journal.pone.0189597..
- 56.
Galeano CH Cortés AJ, Fernández AC, Soler Á, Franco-Herrera N, Makunde G, Blair MW。普通菜豆单核苷酸多态性基因标记的遗传和关联作图。BMC麝猫。2012;13(1):48。
- 57.
Perseguini JMKC,Silva Gmb,Rosa JRBF,Gazaffi R,MarçalJF,Carbonell Sam,Benchimol-Reis LL。开发适合关联映射研究的共同豆核心集合。genet mol res。2015; 38(1):67-78。
- 58.
欧洲的豆类:欧洲地方品种的起源和结构菜豆L. Al Appl Genet。2010; 121(5):829-43。
- 59.
Sinkovič L, Pipan B, Sinkovič E, Meglič v。菜豆L.)和跑步者(菜豆coccineus豆类种质:斯洛文尼亚基因库的例子。BioMed Res. 2019;2019:1 - 13, ID 6376948。https://doi.org/10.1155/2019/6376948.
- 60.
Schuelke M. PCR片段荧光标记的经济方法。NAT BIOTECHNOL。2000; 18(2):233。
- 61.
Wagner HW, Sefc KM。1.0身份。维也纳:农业科学大学应用遗传学中心;1999.
- 62.
Park S.微卫星工具包。都柏林圣三一学院遗传学系;2001.
- 63.
Peakall R, mouse PE。遗传分析在excel。用于教学和研究的群体遗传软件。Mol Ecol Notes. 2006; 6:288-95。
- 64.
Excoffier L, Lischer H. Arlequin套件版本3.5:在Linux和windows下执行人口遗传学分析的新系列程序。分子生态学Res. 2010; 10:564-7。
- 65.
Rousset F. Genepop 4.1.0:完全重新实现Windows和Linux的Genepop软件。Mol Ecol Res。2008; 8:103-6。
- 66.
Langella O.人口1.2.28。Logiciel de Genetique des oplations。Laboratoire人口,Genetique et Evolution,Gif-sur-yvette,法国;2002年。
- 67.
Page rdm。TreeView:在个人计算机上显示系统发育树的应用。comp appl biosc。1996年; 12:357-8。
- 68.
科尔特斯AJ。关于蚕豆的起源(菜豆l .)。am j植物sci。2013; 4(10):1998。
- 69.
Pritchard JK, Wen X, Falush D. STRUCTURE软件文档:版本2.3。美国:芝加哥大学人类遗传学系、牛津大学统计学系;2009.
- 70。
利用结构化软件检测个体聚类数量:一项仿真研究。摩尔生态。2005;14:2611-20。
- 71。
伯爵大,冯·鲍姆。结构收割机:一个网站和程序可视化结构输出并实现Evanno方法。保守遗传资源。2011; 3:429-31。
- 72。
Belkhir K,Borsa P,Goudet J,Bonhomme F. Genetix:Logicel Sous Windows Pin LaGénétiqueDesopmulations 4.02。UniversitédeMontpellierII,Laboratorie Genome,群体,互动,1999. P.1。http://kimura.univ-montp2.fr/genetix/.2019年2月11日访问。
- 73。
Goudet J. Fstat视觉(1.2):计算f -统计的计算机程序。J在这里。2002;86:485-6。
致谢
作者感谢以下基因库为本研究提供的植物材料:AIS(斯洛文尼亚,阿尔巴尼亚),IBGR-UNI-BL(波斯尼亚和黑塞哥维那),CPD(匈牙利),CSIC(西班牙),DAB(意大利),IHAR(波兰),IPK(德国)和IRP-NS(塞尔维亚)。我们也要感谢马拉斯和杰尔卡Šuštar-Vozlič在选择队员方面的帮助。我们非常感谢Aleš Sedlar和Aleksandra Savić为DNA提取提供的初步技术支持。最后,我们也要感谢两位匿名审稿人,感谢他们对我们稿件提出的深思熟虑的意见和宝贵的建议。
资金
这项研究得到了斯洛文尼亚农林食品部和斯洛文尼亚研究机构的部分资助,资助号分别为P4-0072和L4-7520。在应用研究项目L4-7520下,我们设计了研究和收集,进行了包括数据解释在内的分析。农业生物多样性研究pre - game P4-0072项目的资助覆盖了手稿的整个写作过程。
作者信息
从属关系
贡献
概念化,VM,BP;植物识别,VM,BP;方法,BP;软件,BP;验证,VM,BP;正式分析,BP;调查,BP;资源,VM;数据策委,BP;写作 - 原始草案准备,BP; writing - review and editing, VM; visualisation, BP; supervision, VM; project administration, BP; funding acquisition, VM both authors have read and approved the final manuscript.
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1。
782个物种的结构图。
附加文件2:图S2。
基于Nei’s标准遗传距离和UPGMA聚类方法建立了完整的系统发育关系。
附加文件3:图S3。
种质的遗传结构,说明其起源种群。
附加文件4:表S1。
本研究使用的材料。
附加文件5:表S2。
SSR标记在本研究中的应用。
附加文件6:表S3。
考虑材料基因库来源的分子变异分析。
附加文件7:表S4。
基因座无统计学显著偏差HWE(p> 0.05)。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Pipan,B.,Meglič,V.欧洲西部东部进展的多样化和遗传结构菜豆l .种质。BMC植物杂志19,442(2019)。https://doi.org/10.1186/s12870-019-2051-0
收到了:
接受:
发表:
关键字
- 菜豆
- SSR标记
- 地理产地
- 基因库
- 基因库
- 基因簇
- 遗传多样性
- 欧洲人到达