- 研究文章
- 开放获取
- 发布:
甜叶草本植物的遗传多样性及群体结构甜菊糖甙rebaudianaB.,培育和地方品种种质的EST-SSR标记基因分型和甜菊糖苷表型评估
BMC植物生物学体积19文章编号:436.(2019年)
抽象的
背景
甜菊糖甙rebaudiana(菊科),来自巴拉圭的原生,将甜菊醇糖苷(SGS)累积进入其叶子。这些化合物表现出对消费者对减少日常糖摄入量的需求的厌芳甜味味道。尽管发展了美国rebaudiana在世界各地的栽培中,新品种的开发是最近才开始的,特别是由于(1)种质收集和育种,(2)遗传多样性及其结构的研究,(3)基因组工具的巨大缺乏。
结果
在这项研究中,我们developped 18 EST-SSR从150258 EST从加州大学戴维斯分校的菊科植物基因组计划(http://compgenomics.ucdavis.edu/data/).我们145美国rebaudiana来自世界各地的31个品种和31个地方品种。多态信息含量(PIC)在0.60 ~ 0.84之间。平均每个位点有12个等位基因,观察杂合度高达0.69。地方品种显示的私人等位基因是栽培品种的两倍。基因型可分为3个遗传群体。这些地方品种在同一类群中还发现了最古老的品种“Eirete”和“MoritaIII”型。其他两个簇只包含栽培基因型。其中一个发现了原始的遗传变异。SG表型不能区分3个遗传簇,但表型显示苦至甜SGs的组成范围很广。
结论
这是对遗传多样性的第一次研究甜菊糖甙rebaudiana涉及145个基因型,包括已知的品种和不同来源的地方品种群体。本研究指出美国rebaudiana对地方小种群体进行遗传改良,甚至对其组成性状进行遗传改良。
背景
甜叶菊雷劳迪安娜,从巴拉圭本土,是一种多年生的物种,积累甜菊糖苷(SGS)为它的叶子。这些天然化合物具有acaloric强烈的甜味。叶美国rebaudiana首先是由巴拉圭和巴西的原生人用作一般的甜味剂[1].消费者对减少每日糖摄入量和健康产品的需求的地方美国rebaudiana生产在这些需求的十字路口。与不断增长的需求相关,美国rebaudiana的全世界的种植都在增加。这种植物可以在气候条件广泛的地区生长。然而,培养美国rebaudiana作为一种作物,是最近的,并且在原产国的小规模上完成。1964年,在巴拉圭报道了其第一次商业种植(Katayama等人1976; Lewis 1992)。之后,苏米达于1971年制造了巨大的努力,建立美国rebaudiana在日本的栽培(Crammer & Ikan, 1986)。后来,它被作为一种作物引入许多国家。2016年,全球甜叶菊叶产量的80%来自中国,每年5 - 6万吨干叶[2].其他重要的生产国位于亚洲(印度尼西亚,印度,日本,韩国)和美国(墨西哥,美国和加拿大)[3.].最近的监管批准解释了欧洲新开始的生产领域[4].
这最近发育的作物缺乏高价值和可追踪品种。Angelini等人最近列出了九十品种。2016年[5].但是,农民的实践表明,生产的大多数品种都与被称为“Eirete”、“Criolla”和“Morita”类型的品种有关。它们作为种子出售,通常通过开放授粉生产,因为该物种是自交不亲和的[6].这些基因型主要是通过群体选择培育出来的。除了这些目前种植的种群品种外,还有一些品种获得了美国S&W公司的专利[7]或马来西亚谱赛科。这些基因型主要改善了SG的产量和SG的组合物制造的甜蜜味道。
然而,许多其他特征,如种子萌发率,开花日,空中生物质产量,对生物和非生物胁迫的反应较差。然后,显然需要鉴定生产遗传资源和育种。
种质资源的收集和研究是开展育种工作的重要支柱。植物遗传资源的多样性为植物育种工作者开发具有理想性状的新品种和改良品种提供了机会,也是限制遗传侵蚀的基础。没有可用的公共收藏,只有私人公司拥有不可用的收藏。对少数基因型的表型研究已在不同国家进行[8,9,10].多年以来,利用分子标记的有显著改善作物遗传多样性的管理和利用[11].在甜菊糖甙直到最近,缺乏的基因组信息作出了基因分型的瓶颈。一些分子标记如RAPD [12]及ISSR [13[以前的研究已被用于分析群体集合中的多样性。RAPD也被用来建造第一个美国rebaudiana1999年的遗传图谱[14].由于缺乏可重复性,这些标记被抛弃了。尽管近年来农作物测序技术取得了巨大的发展,美国rebaudiana已于2017年由私人财团pureccircle /Coca-Cola/Keygene测序[15].该联盟宣布已对一种基因型进行了测序,但序列尚未公布。SNP标记信息的缺乏证实了开发SSR标记的必要性。对于一些孤儿物种来说美国rebaudiana对于遗传群体,分析SSR仍然是第一选择标记[16,17].由于它们的高多态性、相对容易评分、可测试中性和孟德尔遗传,它们仍然具有很大的适用性。Kaur等人和Bhandawat等人[18,19]通过对从NCBI检索的叶片组织中提取的5548条甜菊est序列进行筛选,分别开发了52和17个SSR标记。这些标记用于从CSIR研究所(印度)的选择中随机选择的40个基因型或来自印度北部的12个本地基因型。这些研究为SSR标记的开发和应用奠定了基础美国rebaudiana.2013年,组合物基因组项目从15种参考转录组合组合组合物中释放了EST数据,用于组合作物或其野生亲属包括美国rebaudiana[20.].这一大量序列可用于SSR模式的筛选和EST-SSR标记的开发。据我们所知,在美国rebaudiana没有研究涉及开发分子标记来分类种质和质疑种群结构的问题。本研究的目的是:1)开发和评估EST-SSRs的适用性美国rebaudiana作为31个地方品种基因型和114个栽培品种基因型的标记;2)鉴定种群的遗传多样性和群体结构美国rebaudiana的地方品种和栽培品种和3),以检查在耕地和地方品种种群的遗传变异性和其结构和表型变异性SG之间的链路。
结果
EST-SSR基因分型
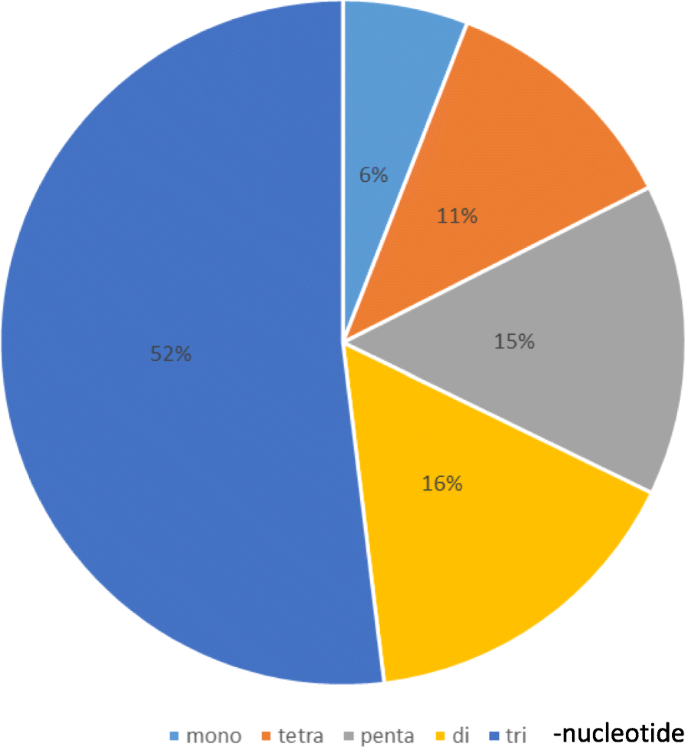
在150,258个unigenes中,可以检测到3401个SSR模式,1745个独特。将这些1745 SSR分为6%的单核苷酸,16%的二核苷酸,52%的三核苷酸,11%的四核苷酸和15%的五核苷酸(图。1和附加文件2:表S1)。在这些1745中,考虑1060,适用于底漆设计(表1).超过60%是三核苷酸重复类型。选择九十四有10到26个重复的底漆设计(附加文件1:图S1)。经5个基因型筛选,19个未扩增,5个PCR产物超过300 bp, 17个单型或多位点图谱。53对引物扩增出多态性片段。基于多路复用的可能性,其中18人被选为人口研究(附加文件)2:表S2)。
总结了从150 258个单基因中筛选出的1745个单基因的重复数分布情况甜菊糖甙rebaudiana在http://compgenomics.ucdavis.edu/data/cwassy_2012/iAssSta.fa(UC Davis的Compositae Genome项目)
遗传变异性
由145个单独植物组成的面板(表2和附加文件2:表S3)与18个选定标记物进行基因分型。在分析的145个个体植物中检测到总共213个等位基因(表3.).每个位点检测到的等位基因数为5 ~ 19个(stvia021;Stvia048),平均每个位点有12个等位基因。标记的pic在0.60 (stvia021)和0.84 (stvia025和stvia051)之间。平均观察到的杂合度和期望杂合度Ho = 0.69 (min-max: 0.46 (stv018)-0.87 (Stv036)), He = 0.78 (min-max: 0.66-0.86)。以私人等位基因的平均数量计算,等位基因丰富度的范围为栽培基因型的1.5到地方基因型的3(附加文件)1:图S2)。
人口结构
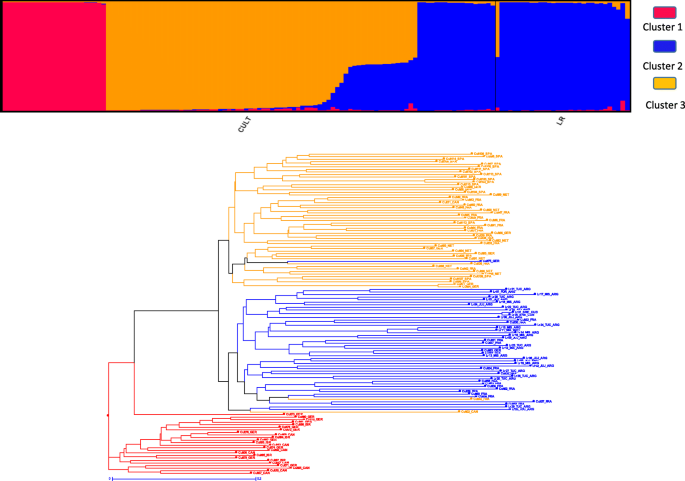
结构推断的连续K值(DK)之间的对数似然变化率揭示了三个相对较高的Δ簇K价值K= 3(无花果。2a).我们使用在K = 3时推断的STRUCTURE成员系数来定义在后续分析中使用的种群。在以后的分析中,如果某一群体的隶属系数≥0.80,则对该群体进行基因型分配。因此,归属于一个特定亚群体的相同值低于80%概率的基因型被认为是混合的。在此基础上,聚类1有24份,聚类2有48份,聚类3有54份。19个加入被混合(附加文件2:表S4)。当K = 3时(图。2a),培养的甜叶菊被证明属于三个集群(名为1,2和3),而阿根廷和古巴的体力成员属于群集2.在这个集群中,培养的甜叶菊属于最古老的选择“Eiretei“,”Eireteii“和”Moritaiii“类型,但也与”C“和”D“品种更近最近在德国选择并属于eustas系列。
利用基于模型的程序结构分析了甜菊栽培品种和地方品种在K = 3时的群体结构。一个栽培和体力植物的祖先的比例甜菊糖甙rebaudiana登记入册(n = 145) inferred with STRUCTURE for K = 3. Each individual is represented by a vertical bar, partitioned into colored segments in proportion of the estimated membership in the different genetic clusters inferred with STRUCTURE. Under the figure are depicted the two groups of genotypes cultivated (CULT) or landraces (LR) and color and names of the three clusters.b基于骰子异化指标的邻近的树木图显示了非混合126之间的关系甜菊糖甙rebaudianaindividuals (i.e. individuals assigned to one cluster at K = 3 with a membership coefficient > 0.80). Genotypes were colored according to their assignment to the three different genetic clusters, as inferred by STRUCTURE. Branch length is proportional to the distance between nodes
三组甜菊糖簇间的遗传变异与分化
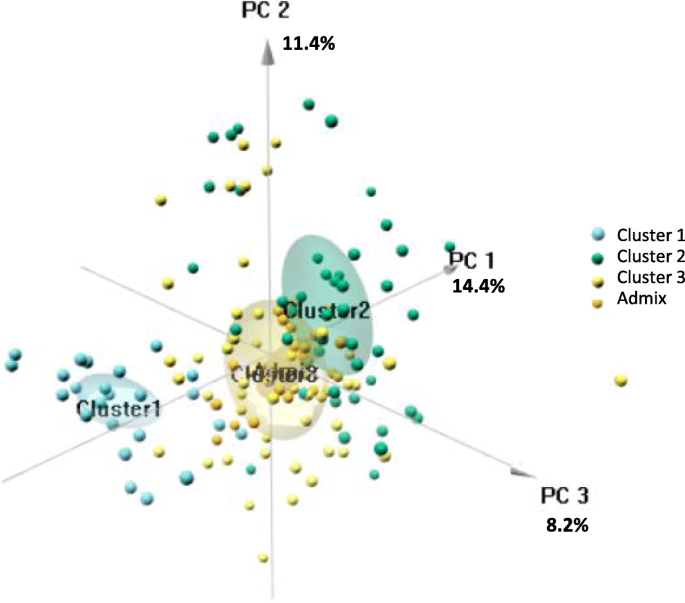
我们建立了126非混合个人的邻接树(附加文件2:表S4)。树状图显示了三个主要的进化枝(图2)。2b).树状图的结构与Structure推断的聚类一致,除CULT75_GER、CULT02_CAN和CULT40_FRA基因型外,其余3个分支分别对应于聚类1(红色)、2(蓝色)和3(橙色)。簇的树状图提供了一个有趣的模式,具有明显的分化栽培甜菊簇1。PCA(无花果。3.)揭示了与结构推断的类似模式,具有清晰分化的培养的甜叶菊簇1,清楚地与其他甜叶菊簇分离。
我们计算了三个遗传群体的遗传统计量(表)4).近交系系数(Fis)均较低,但均为正。这一结果与大多数标记物上Hardy-Weinberg平衡的显著卡方检验有关(表3.),与种子批定的推定血清关系有关。在培养的人群簇1和3中发现高遗传多样性分别= 0.725和0.753分别,在混合的体型和培养的簇2中(他= 0.801)。
三个甜叶菊种群的两两FST均显著5).它们表示三个人群之间的差异化。Amova被用来估计人群中的方差(表6).结果表明,大部分遗传变异是由显著的群体内变异引起的(98%;表格6).只有2%的遗传变异归因于人群的差异。
甜菊种群间甜菊醇苷组成的变异
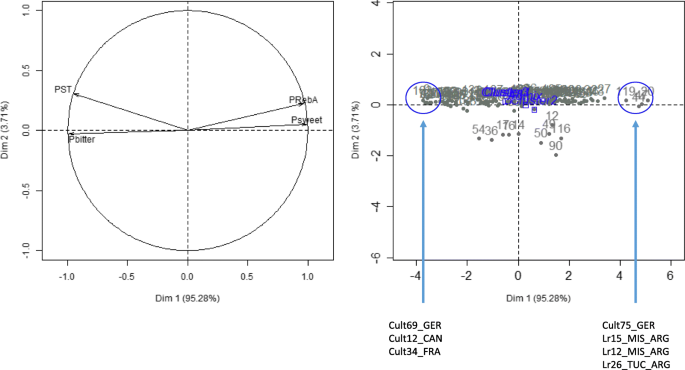
以上分析显示史蒂维亚地铁培养的高遗传多样性。为了评估根据其SG组成的不同群体的分类,我们估计了Steviol(ST),Reba,Sweet SG的比例作为rebm,rebd和rebbf和苦涩的sg作为rebc,dula的总和,摩擦和某人。相比之下,变量的图表对比甜味的SGS,例如Reba和Sweet SG,甜菊糖苷和苦味SG(图。4).个体的图表显示了沿着第一个维度的分布,在第一个维度中,基因型表现出最甜的味道,SG,直到那些表现出主要的苦味。有趣的是,3个遗传群体的内容性状在不同SGs中差异不大,3个群体聚在个体图的中心。第一组和第三组重叠。然而,值得注意的是,群集2,在SG的甜味最高的成分中脱颖而出。因此,解释SG甜味成分最多的基因型是阿根廷种群的3个基因型(Lr15_MIS_ARG、Lr12_MIS_ARG和Lr26_TUC_ARG)以及培养基因型Cult75_GER,即EUSTAS收集的改良基因型“C”(Hastoy et al., 2019)。解释SG中成分最多的基因型主要是栽培基因型,如Cult69_GER、Cult12_CAN和Cult34_FRA。
讨论
遗传资源的收集和深入研究是建立育种计划的重要步骤。美国rebaudiana具有认可的卫生兴趣,现在的兴趣和相关行业现在高度发达和全球。尽管如此,甜叶菊的优化改善是其早期阶段。育种计划相对较新。特别是,它们遭受危急缺乏关于与缺乏基因组工具相关的遗传资源的深入信息。根据尽可能多样化的145种基因型的收集,我们的研究具有目标(1),以制定强大的SSR标记(2)来研究来自31种栽培的遗传多样性和遗传结构的遗传结构和遗传结构,但也是来自阿根廷和古巴不同地区的31个地球场。
SSR多态性
SSR标记对于研究遗传多样性是有用的,因为它们是高度多态性的,多个等位基因及其发展需要有限的基因组信息。因此,它们是最简单,更丰富的分子标记。分子标记的发展美国rebaudiana是长期存在的。1999年,183个RAPD标记被开发出来,并用于从一个伪杂交F1中生成第一个部分遗传图谱[14].然后RAPD和ISSR分子标记在三个研究中分别以研究2008至2011年6个埃及和印尼种质之间的关系[13,212016年印度加入12个国家[22].如在[19],这些标记的优势性和低重复率导致了EST-SSR标记的发展。EST-SSR是从5548条公开的est序列中预测的2977条ungenes中开发出来的美国rebaudiana经过 [18,19].分别开发了五十二岁和十七个EST-SSR。它们用于研究来自印度CSIR的四十种随机基因型的遗传变异[19]和来自北印度的12个国家[18,22].这些作品报告了每位轨迹的许多等位基因在2到15之间,平均每SSR基因座平均低于我们所发现的。这可以由SSRS针对的基因组的区域和/或通过Bhandawat等人的较窄的遗传多样性来解释。学习 [19在kaur等人。学习 [18,主要研究印度人的基因型。硬粒小麦中也有这种现象[23茄子中的[24]或在罂粟[25基因SSRs比EST-SSRs具有更多的多态性。
然而,在研究和我们的研究中,平均HO很高,0.80和0.69,按照报告的交配系统美国rebaudiana[6].
人口结构和遗传多样性
群体结构和Dendography分析将遗传分为三个集群/亚步骤,尽管分析基于不同的方法。较少数量的混合基因型(19/145)可以通过收集的组成,78%(114/145),其中培养品种和22%(31/145)的体重标准。没有分析野生基因型。此外,种植的品种通常通过在两个鉴定的父母之间穿越而产生。使用有限数量的父母也可以解释较少的混合。
除两个中国品种属混合聚类外,未见区域聚类现象。特别是第1群,收集了来自加拿大、德国和Israël的基因型,表明甜菊糖存在大量的种子和等位基因交换。聚类1和3主要由栽培品种组成。集群1似乎揭示了原始的多样性。它包含在德国选择的基因型“Gawi”[26,27但起源不明[28].然而,在我们之前对法国西南部野外条件下不同来源的15个基因型的表型研究中,基于叶面生物量产量,“Gawi”属于不同于“Eirete”的形态[28].集群3还包括栽培品种,其中一些被出售为“Criolla”类型。以一种非常有趣的方式,阿根廷地体和古巴的旧样品都在集群中分组。兰片揭示了培养基因型的平均值的两倍的平均私有等位基因数。该集群与耕种品种共享,键入“Eireete”和“Moritaiii”,已知是以前改善的品种,非常普遍存在世界上。
AMOVA结果表明,遗传分化主要是群体内变异所致。因为我们的研究是第一个关于美国rebaudiana遗传资源方面,目前尚无参考研究。但是,在其他异交多年生植物中也观察到了类似的结果,如苜蓿(紫花苜蓿) [29达尔马提亚除虫菊(tanacetum cinerariifolium.) [30.]或农艺交换机(Panicum Virgatum.) [31].在这些物种的种群中观察到的高水平遗传变异最有可能是由于它们的部分或完全异体生殖系统。
SG表型
SGs的表型不允许构造不同的群体。基于这一特征,三个遗传群体的差异很小。令人惊讶的是,在SG中,地方品种群体似乎更倾向于甜味的品质,而仅由品种组成的1和3群则无法区分。它们由品种组成,产生各种各样的SGs,从苦到甜,这似乎令人惊讶,考虑到SG软/苦的比例是甜菊糖的首选标准[5].
结论
我们的研究是对遗传多样性的第一次分析美国rebaudiana涉及145个基因型,包括已知的品种和不同来源的地方品种群体。本研究产生了18个新的高度多态性和鲁棒的微卫星标记。这些标记对于遗传多样性评估和种质管理具有很大的潜力,这是繁殖的关键步骤。这些标记也可用于基因型可追溯性。145种基因型美国rebaudiana成功分型。他们发现三个遗传群体人口中一个显着的变化。地方品种透露他们的等位基因丰度和他们的甜蜜SG表型的长远利益。他们通过养殖怀有进一步改进的宝贵遗传变异在阿根廷中所示通过他们的表型32].
方法
植物材料
由145个独立的植物组成的面板属于美国rebaudiana从不同来源收集(表2和附加文件2:表S3)。从三十一品种中发出的一百十四种基因型是从三十一品种发出的“种植”,作为种子批次,如下方式分发:从加拿大,来自中国15人,来自法国的34个,来自德国的20,来自Isražl,5来自荷兰和来自西班牙的18岁。三十一位是地架。二十九岁是北阿根廷,福尔摩沙地区,石竹,枣和毫无米什的地区,属于INTA系列[32].二、目录编号1687090和1687 091,馆藏编号5353,由纽约植物园植物标本馆提供。它们分别于1927年和1931年在古巴收集。
SSR基因分型
识别EST-SSR标记
微卫星或SSRS于454次测序中产生的150,258个EST开发,并从UC Davis的Compositae Genome项目下载(http://compgenomics.ucdavis.edu/data/cwassy_2012/iAssSta.fa)根据附加文件中描述的管道1:图S1。Progration Sputnik(http://abajian.net/sputnik/)对含有SSR的EST进行鉴定。SSR识别的最小motif重复数定义为单个核苷酸单元20个重复,二核苷酸单元10个重复,三核苷酸单元7个重复,四核苷酸单元5个重复,五核苷酸单元4个重复。用Primer3Plus设计SSRs侧翼的引物对[33] with melting temperatures 52–57 °C, primer lengths 18–24 bp, expected fragment size 100–300 bp. All the designed primers were screened on five samples, Cult32_FRA, Cult33_FRA, Cult34_FRA, Cult36_FRA, Cult37_FRA. The primers producing clear and polymorphic bands were subsequently used for genetic diversity assessments.
DNA提取和分子基因分型
使用改进的先前公布的方案提取基因组DNA [34].幼叶在烘箱中在55℃下在烘箱中脱水48小时,然后在GRINOBENDER中研磨成细粉末。将一毫升缓冲液(0.1M Tris-HCl pH 8,0.7M NaCl,0.04M EDTA,1%HATMAB,加入在使用前添加的1%β-巯基乙醇和50μg/ mL蛋白酶K加入到30mg粉末中并在65℃下孵育60分钟,每15分钟通过反转轻轻混合。氯仿:异丙醇萃取,异丙醇沉淀和70%乙醇洗涤,DNA在100μl纯水中重悬。在EPOCH微孔板分光光度计(Biotek)上定量基因组DNA。
PCR amplifications were performed in 15 μl reaction volume containing 10 ng of template DNA, 1 X PCR buffer (10 mM Tris-HCl pH 8.3; 50 mM KCl), 2.5 mM MgCL2,每个引物0.2 μM,每个DNTP’s 200 μM, 0.5 U SurePRIME™DNA聚合酶(MP Biomedicals)。进行了放大的Mastercycler Pro(埃普多夫)后PCR协议:15分钟初始变性在95°C和35周期30年代在94°C, 45 s 55°C, 60年代在72°C,紧随其后的是最后一个扩展10分钟在72°C。
PCR扩增子在变性聚丙烯酰胺凝胶上分离,由4.5%聚丙烯酰胺(丙烯酰胺:双丙烯酰胺19:1)和7M尿素在1×TBE缓冲液中。运行后,使用银染色方案可视化扩增片段[35].
每个位点的片段进行基因分型,并用10-330 bp的梯形图(Invitrogen公司)对等位基因大小进行评分。
遗传变异
利用PowerMarker 3.25计算等位基因频率和遗传多样性[36]及GenAlEx 6.5 [37].这些措施包括:(Na)的等位基因数,主要的等位基因频率,多态信息含量(PIC),期望杂或基因多样性(HE),观测杂合度。近亲繁殖系数(FIS)用SPAGEDI 1.3 [计算38],并与GENETIX v4.05验证[39].私人等位基因丰度与锛软件计算调整为样本大小的差异[40].我们进一步探索了使用使用简单匹配的jaccard系数方法和2000多个复制的自动匹配相差指数构建的未加权邻接树,以达尔文软件包v6.0.010中实现的重复的匹配相差指数构建的遗传分化和关系41].使用Arlequin V3.5估计人口FST和NEI的指数[42].FST的意义是通过对1000个基因型数据进行随机重采样来评估的。
人口结构
用GENETIX v4.05执行的主成分分析(PCA)探索个体间遗传关系的表达[39].我们还使用了结构中实现的基于个人的贝叶斯聚类方法2.3.3 [43]调查人口细分。我们使用accixture和相关等位基因频率从k = 2到k = 10的结构使用,假设没有现有的人口信息。Markov Chain Monte Carlo迭代的烧坏和数量分别设定为10,000和100,000。对每k进行十个独立运行,并用ClumpP V1.2处理输出[44].结构条形图使用disstruct 1.1显示[45].我们检查了ΔK的分布,绘制了结构收割机(http://taylor0.biology.ucla.edu/structureHarvester/),根据Evanno等人(2005)[46].
甜菊醇苷的提取与定量
如Hastoy等人所述进行甜醇糖苷萃取和定量。(2019)[28].在202 nm检测到9种SGs (RebD, RebM, ST, RebA, RebC, DulA, Rub, RebB, SB),之前用纯化SG标准(Chromadex,美国)进行鉴定。每个SG用5 ~ 1000ng /μL纯化标准液定量。结果以单位叶干重含量(% w/w)和总叶干重含量(% w/w)表示,并以百分比(%)表示。
可用性数据和材料
在这项研究中产生的或分析所有的数据都包括在此发表的文章作为补充信息的文件英寸
参考文献
- 1.
甜菊和甜菊的植物学美国rebaudiana.甜菊:甜菊属。2002;泰勒安:18-39。
- 2.
甜叶菊的太阳J.发展甜菊糖苷产业在中国。PROC 9日甜菊SYMP 2016年,EUSTAS,瑞典,15-16 2016年9月,扬MC Geuns埃德。2016年145-50。
- 3.
甜菊属植物的地理分布、植物描述及自交不亲和机制。糖科技。2018;20:1-10。
- 4.
委员会条例(欧盟)。2011年11月11日的第1131/2011号于2011年11月11日修改附件二(EC)欧洲议会的第133/2008号和安理会关于甜菊糖糖苷。OFF J EUR UNION。2011;修改A:L295 / 205 L295 / 211。
- 5.
Angelini LG, Martini A, Passera B, Tavarini S.栽培美国rebaudiana贝尔托尼和相关挑战。:甜味剂。Cham:施普林格国际出版;2016.1-52。
- 6.
Yadav AK, Dhyani D, Ahuja PSSS。的改进综述甜菊糖甙rebaudiana(据)。acta botanica yunnanica(云南植物研究)2011;91:1-27。
- 7。
钱敏干叶与甜菊苷产量甜菊糖甙rebaudiana在美国西部HortScience。2016; 51:1220-7。
- 8。
以半多年生植物为研究对象甜菊糖甙rebaudiana温带气候下的培尔托尼:基因型、环境和植物年龄对甜菊醇苷含量和组成的影响。植物资源学报。2016:685-94。
- 9。
Grevsen K,Sorensen JN。第二年培育成绩丹麦“绿色甜叶菊”项目 - 有机食品的天然甜味剂。从野外到Fork,Proc 9th Stevia Symp 2016,Eustas,瑞典,2016年9月15日至16日,Jan Mc Geuns Ed。2016;:115-26。
- 10.
Lankes C,的哥洛莎P.评价甜菊糖甙rebaudiana在葡萄牙阿连特霍地区的一个地点发现了基因型。Proc 8th Stevia Symp 2015, EUSTAS, Bonn, Ger Geuns JMC, Ceunen S, Eds. 2015; 167-76。
- 11.
陈志强,王志强,王志强,等。作物遗传多样性评价的研究进展:基于遗传多样性分析的视角。Genet Res Int. 2015; 15:14。
- 12.
王志强,王志强,王志强,等甜菊糖甙rebaudiana使用RAPD和HPTLC分析。pharm biol。2013; 51:771-7。
- 13.
Hadia HA, Badawy O, Hafez AM。甜菊糖(甜菊糖甙rebaudiana基于ISSR分析的Bertoni)资料。细胞生物学杂志。2008;2:1-5。
- 14.
姚永强,潘志强,王志强甜菊糖甙rebaudiana.基因组。1999; 42:657-61。
- 15.
Schauer S,Huang T,Samuel P,Khazi F,Markosyan A.来自测序和注释的见解甜菊糖甙rebaudiana基因组及其在农艺和健康中的应用。ANN NUTR METAB。2017; 71:1316-7。
- 16.
孙敏,刘旭东,孙敏,等。关于我死亡的报道有些夸张:这是对21世纪使用微型卫星的研究人员的评论。4.中国农业科学(英文版);
- 17.
微卫星标记:它们的含义和为什么它们如此有用。基因生物学杂志。2016;39:312-28。
- 18。
基于SSR标记的表达序列标签的鉴定与功能注释甜菊糖甙rebaudiana.中国农业科学。2015;39:439-50。
- 19。
bhandawat a,sharma h,nag a,singh s,singh ahuja p,kumar sr。功能相关的新型微卫星标记,用于有效基因分型甜菊糖甙rebaudiana据。J麝猫。2014;93:75 - 81。
- 20。
哈金斯·卡,赖志,Oliveira LO, Still DW, Scascitelli M, Barker MS,等。菊科作物基因组学:参考转录组组合和与野生近缘种杂交的证据。生态学报。2014;14:166-77。
- 21。
Dyah S, Rina Sri K, Budi SD。甜菊糖(甜菊糖甙rebaudiana(贝尔托尼)基于分子特征的。Int Conf Biosci Biotechnol Proc. 2011; 1:37-43。
- 22.
陈志强,陈志强,陈志强,等。基于RAPD和ISSR标记的水稻遗传多样性评价甜菊糖甙rebaudianaBertoni Accessions。印度j biotechnol。2016; 15:95-100。
- 23.
基于EST-SSRS和基因组SSRS的栽培硬粒小麦基因型变异分析。Euphytica。2001;119:39-43。
- 24.
利用基因组SSRs和EST-SSRs分析茄子长白品系的多样性、亲缘关系和遗传指纹。Sci Hortic。2011;129:238-46。
- 25.
引用本文:刘志强,刘志强,刘志强,等。基于EST-SSR标记的罂粟EST-SSR分子标记研究。植物品种。2013;132:344-51。
- 26.
Zabala嗯。Optimierung Von Wachstum und Ertrag(Süßstoffbildund)Bei甜菊糖甙rebaudianaBertoni unter mitteleuropäischen Standortbedingungen。Rheinischen Friedrich-Wilhelms-Universitat波恩;2011.
- 27.
张志强,张志强甜菊糖甙rebaudiana基因型。Proc 5th Stevia Symp 2011,Eustas,比利时,Jan MC Geuns Ed。2011; 75-87。
- 28.
引用本文:王志强,王志强,王志强,等。15个基因型的解译性能甜菊糖甙rebaudiana在法国西南部通干生物质和甜菊糖苷的评价。IND作物PROD。2019; 128:607-19。
- 29.
YIN S,王Y,南Z.苜蓿种质遗传多样性研究(紫花苜蓿利用微卫星分析产于美国的L. subsp sativa)。Legum杂志2018;41:202-7。
- 30.
Grdisa M,Liber Z,RadoSavljevic I,Carovic-Stanko K,Kolak I,萨科维奇Z.达尔马提岛遗传多样性和结构(tanacetum cinerariifolium.在巴尔干庇护中的特雷维尔./sch./bip。,奥斯特卡伊)。Plos一个。2014; 9。
- 31。
柳枝稷的遗传多样性及其遗传结构研究(Panicum Virgatum.l .)的人口。基因资源作物学报。2013;60:1057-68。
- 32。
莫雷诺FE,所罗门V,ErazzúLE,Budeguer CJ,马尔多纳多LM。甜菊糖甙rebaudiana(Bertoni)Bertoni:Compentinacióndeesteviósidoyrebaudiósidoa en ena genotipos proMisorios de UnaColecciónnntucumán。Novena Reun Prod VegYSéptimaProdmanCelNoa S MTucumán。2016:393-8。
- 33。
Untergasser A,Nijveen H,Rao X,Bisseling T,Geurts R,Leunissen Jam。primer3plus,一个增强的web界面到primer3。核酸RES。2007; 35:1-4。
- 34.
从新鲜组织中分离植物DNA。焦点(麦迪逊)。1990; 12:13-5。
- 35.
Bassam BJ, Caetano-Anollés G, Gresshoff总理。快速和灵敏的银染色的DNA在聚丙烯酰胺凝胶。学生物化学肛门。1991;196:80-3。
- 36.
刘K,缪斯SV。PowerMarker:遗传标记分析的综合分析环境。生物信息学。2005; 21:2128-9。
- 37.
Speall R,Smouse Pe。Genalex 6.5:Excel中的遗传分析。教学和研究人口遗传软件 - 更新。生物信息学。2012; 28:2537-9。
- 38.
Hardy OJ,Vekemans X. Spagedi:一个多功能计算机程序,用于分析个人或人口水平的空间遗传结构。Mol Ecol Notes。2002; 2:618-20。
- 39.
Belkhir K,证交所P,Chikhi L,Raufaste N,伯恩霍姆F. GENETIX 4.05,logiciel苏视窗TM倒拉génétiqueDES种群。LABORATOIRE基因组人群,交互,CNRS UMR 5171,Üniversite电蒙彼利埃II,蒙彼利埃(法国)。2004年。
- 40.
Szpiech Za,Jakobsson M,Rosenberg Na。Adze:将私人私人私人的稀疏方法进行计数到人口组合。生物信息学。2008; 24:2498-504。
- 41.
Perrier X,JacqueMoud-Collet JP。达尔文软件。2006年。http://darwin.cirad.fr/.
- 42。
Excoffier L, Lischer HEL。Arlequin套件3.5:在Linux和windows下执行群体遗传学分析的新系列程序。生态学报2010;10:564-7。
- 43。
基于多位点基因型数据的群体结构推断。遗传学。2000;155:945-59。
- 44。
Jakobsson M,Rosenberg Na。CLUMPP:用于处理群体结构分析中的标签交换和多模的集群匹配和排列程序。生物信息学。2007; 23:1801-6。
- 45。
罗森博格NA。人口结构图形显示程序。Mol Ecol Notes, 2004; 4:137-8。
- 46.
Evanno G,Regnaut S,Goudet J.检测使用软件结构个体的簇的数目:一个模拟研究。Mol Ecol。2005; 14:2611-20。
- 47.
Weir BS,Cockerham CC。估计人口结构分析的F统计。进化(n y)。1984; 38:1358-70。
确认
作者感谢植物标本室主任和纽约植物园(Bronx, NY 10458-5126)的Tom Zanoni博士提供了来自古巴、Coralie Chesseron (INRA,波尔多,法国)的植物生产样本和Pierre Jannot (Rouages, Agen,法国)的科学支持样本。
感谢感谢S.博士玛丽特(INRA,UMR BIOGECO,法国)的手稿的批判性阅读。
资金
C. Hastoy得到了ANRT 2014/0915基金和法国Oviatis SA的支持。新阿基坦地区通过Cifre的专项资金支持这项工作。
作者信息
隶属关系
贡献
VSL在PC和DR的贡献下构思和设计了实验;PC开发了EST-SSR。PC和CH进行了实验。PB、LEE、CJB提供材料;VSL和PC对数据进行了分析。VSL在DR, LEE和CJB的校对下撰写论文,所有作者阅读并批准了手稿的最终版本。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1。
本研究中使用的18种SSRs的筛选流程总结。图S2。每个基因座的平均私人等位基因数量和培养和实地基因座的均外等位基因的平均数量的基因分型使用Adze软件计算
附加文件2:表S1。
利用150、258个ungenes对1745个SSR标记的重复模式进行了分析甜菊糖甙rebaudiana在http://compgenomics.ucdavis.edu/data/cwassy_2012/iAssSta.fa(UC Davis的Compositae Genome项目)。表S2。本研究使用的18个SSR标记及其引物及特性。表S3。的列表甜菊糖甙rebaudiana栽培和地方品种群体研究。表S4。通过结构分析,研究了不同基因型在不同群体中的分布情况
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Cosson,P.,Hastoy,C.,Errazzu,L.E.等等。甜叶草本植物的遗传多样性及群体结构甜菊糖甙rebaudianaB.,培养和LATERASE通过EST-SSRS基因分型和Steviol糖苷表型评估的种质。BMC植物杂志19,436(2019)。https://doi.org/10.1186/s12870-019-2061-Y.
已收到:
接受:
发表:
关键词
- 甜菊糖甙rebaudiana
- 遗传多样性
- 品种
- 长白猪
- Steviol糖苷