抽象的
背景
豆科是一个重要的经济和生态植物科,其细胞器基因组研究一直倾向于质体基因组(质体)。迄今为止,发表的豆科线粒体基因组(有丝分裂基因组)序列不到15条,除4条外,其余均属于凤蝶亚科,这限制了人们对豆科大小变异和内容的理解。为了解决这一问题,我们对来自3个亚科(Cercidoideae, Detarioideae和Caesalpinioideae)的4个有丝染色体进行了测序和组装。
结果
基于共享线粒体蛋白编码区域的系统发育分析产生了一个完全确定和支持的系统发育,与质体树完全一致。比较分析表明豆科植物中存在两种有丝分裂基因组的扩增。四属体型扩张(Tamarindus,Libidibia,血红素瘤,银合欢)在两个亚属(Detarioideae和Caesalpinioideae)中发生在相对深的节点中,主要由细胞间基因转移和/或三种横向基因转移(HGT)引起。由于天然线粒体序列重复,在Papilionoideae中发生了第二种,最近的扩张。家族范围的基因含量分析显示出11个基因损失,四(RPS2.,7,11和13)其中发生在Fabaceae的祖先。剩余七种基因的损失(cox2,rpl2,rpl10,RPS1.,rps19,sdh3,sdh4)局限于特定的谱系或独立发生在不同的支系中。三个基因的内含子(cox2,ccmFc和rps10)由于大的序列插入和缺失,显示了广泛的谱系特异性长度变化。Fabaceae毒蛛之间的共同DNA分析表明了代骨间隔物的大量衰减,并进一步了解了Caesalpinioideae和HoloParasitic的模突厥片之间的HGTLophophytum(蛇菰科)。
结论
本研究是迄今为止对豆科有丝分裂基因组进行的最全面的分析,扩展了对大小和基因/内含子含量动态变化的理解。本文报道的四个新测序的有丝分裂基因组将系统发育范围扩展到四个亚家族。该家族在古代和近代都经历了多次有丝分裂基因组大小的波动。这些大小变化的原因在不同的世系中是不同的。豆科有丝分裂基因组通过吸收外源DNA和复制本地线粒体DNA经历了广泛的大小波动。
背景
植物的线粒体基因组(麻醉剂)表现出大小,架构,基因含量和核苷酸替代率的激烈变化[1,2].促型介质估计估计,主要基于复重动力学和限制性片段分析,范围为200 kB至2.4 MB(12倍)[3.].最近使用完整的有丝分裂基因组序列进行的估计扩大了这一范围,从异常小的基因组毒蕈氏菌霉素6kb [4到11.3 Mb的大的多部基因组硅宾conica[5].这种广泛的大小变化归因于内部和外部因素。一个主要的内部原因是重复序列的增殖,包括成百上千的重复基序[6,7].这种重复的DNA也是产生有丝分裂基因组多部分结构的重排的主要原因[8,9].促使促进膨胀中的主要外部因素涉及来自来自其他基因组隔室的天然DNA和来自不同生物的外国DNA的增益[10].
细胞内不同基因组间的遗传物质交换称为细胞内基因转移(IGT) [11,12].涉及IGT的事件从细胞核和质体招募DNA或将线粒体DNA输出到细胞核或质体。起源于核基因组和质体基因组(plas质体)的有丝分裂基因组中的序列分别被称为核源线粒体DNA (MINC)和质体源线粒体DNA (MIPT) [13].通常,MINCs是通过与细胞核中编码的基因或转座元件(TEs)的序列相似性来检测的[14,15].在被子植物中,大多数MIPTs是通过细胞内的IGT事件产生的。然而,一些mipt有更复杂的历史,包括结合IGT和种间线粒体-线粒体水平基因转移(HGT)。例如,已确定的mipt由IGT转移到一个物种,然后由HGT转移到另一个不相关的物种[16].促毒素的活性和复杂的DNA交换能力[17,18通过结合直接和媒介介导的机制,不仅限于植物,而且也发生在节肢动物、线虫、原生动物、细菌、真菌和病毒中[19].因此,许多已经被探索的植物有丝染色体代表了来自不同来源的DNA镶嵌,既来自单个细胞内部,也来自与其他物种的交换[20.].
在促使催乳素的末端原点[21],核的IgT事件伴随着大多数真核生物的“射出的原始粒子”基因组大小减少[22].然而,在植物有丝分裂基因组中存在广泛的IGT和种间HGT [23尺寸和结构导致卓越的变化。线粒体基因的核拷贝可能随时间降级,但有时它们会获得用于表达和细胞内靶向信号的调节元素,将产品直接运输回线粒体[24].转移基因的激活和靶向使线粒体拷贝变得可有可无,往往导致线粒体基因丢失,许多核糖体蛋白和琥珀酸脱氢酶基因都发生过这种情况[25].豆科中一个显著的基因丢失是cox2[26].对相态谱系的广泛采样显示了许多不同条件的存在,一些类群具有副本cox2在细胞核和线粒体以及其他线粒体复制丢失的地方[27].
豆科(豆科)是比较基因组学的一个极好的科[28].在具有模型物种的Anuiaperm家族中,Fabaceae是最大的,包括六个亚属(Cercidoideae,Detarioideae,Duparquetioideae,Dialioideae,Caesalpinioideae,Papilionoideae),770属和20,000种[29].这种多样性也使家族成为进化研究的理想系统。利用Southern杂交对被子植物有丝分裂基因组进化的早期研究[27,30.,31,32]揭示了豆科植物核基因组线粒体基因的若干基因缺失和IGT。完整的有丝分裂基因组测序[33,34,35,36,37,38,39,40,41.,42.]表现出许多现象,包括基因丢失、IGT、HGT和有丝分裂基因组大小的相当大的变化。此外,最近的研究表明,线粒体DNA从caesalpininioideae的mimosoid分支向全寄生虫的大规模水平转移是前所未有的Lophophytum(蛇菰科)39,42.,43.].然而,这些比较仅包括11个完整的Fabaceae毒蛛,其中九个来自Papilionoideae。
最近测序技术的进步不断推进了对质体的理解[44.,45.]和有丝分裂基因组[2,46.进化)。然而,由于有丝分裂基因组的复杂性,这一进展偏向于质体,在大多数世系中,质体是高度保守的[47.].迄今为止探索的许多毒蛛含有重复性DNA的优势,未知的起源的混杂DNA,基因损失和剧烈基因组重新排列。促催化序列的缺乏阻碍了对变异的模式和原因的理解。这里,测序来自三种不同FABACEAE亚壳的四种线粒体基因组,并组装来自早期发散的亚壳(Cercidoideae和Detarioideae)的两种物种,来自非模骨CaeSalpinioideae的两种物种。这允许对Fabaceae Subcamilies之间的促滤蛋白酶进化进行调查,包括促发滤液的水平转移到HoloparasiticLophophytum.
结果
基因组装配和整理
线粒体阅读的初始组装产生一到四个大的contigs紫荆黄花,Tamarindus indica.,Libidibia coriaria和Haematoxylum brasiletto.最后用聚合酶链反应(PCR)和桑格测序产生了每个物种的单一主染色体。基因组大小从348,530到631,094 bp不等,每个有丝分裂基因组的平均覆盖率为150X到490X(见表)1和附加文件1:表S1)。在每个基因组中,使用配对末端、质体过滤的reads覆盖相当均匀(附加文件)2:图S1a)。总单端reads也被映射到每个完整的有丝分裂基因组,以显示MIPTs的分布(附加文件2:图S1B)。恒定基因组覆盖率与PCR和Sanger测序一起证实,塑性过滤的组装方法成功地区分了毒蛛中的塑料和MIPTS的读数。
有丝分裂基因组和质体系统发育的比较
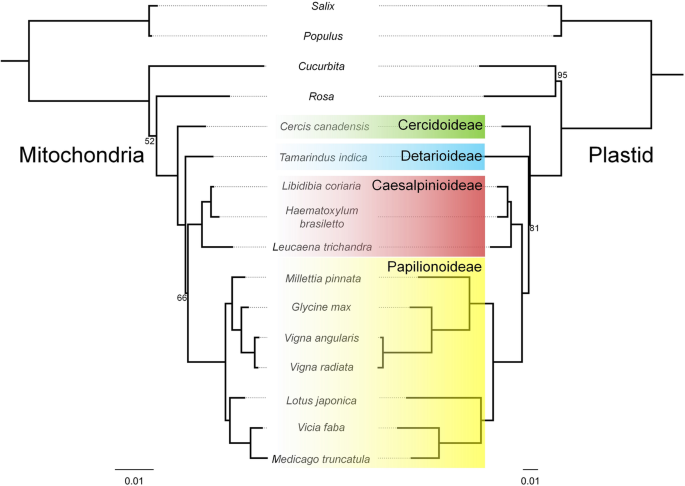
使用来自发丝器组(26cd; 25,265bp对齐长度)和塑性(68个Cds; 52,497bp对准长度)的所有共用蛋白质编码基因构建的最大可能性(ml)树木构成,以评估两个细胞器基因组之间的一致性(图。1).豆科植物的树状结构一致,支持度高(除有丝分裂基因组和质体树各2个节点外,其余节点支持度均为100%)。蠓亚科首先分化,其次是蠓亚科。灰蝶亚科和凤蝶亚科各为单系,形成一个支撑良好的枝,然后再分化。两株豆科植物虽然没有拓扑不一致现象,但在枝长上存在显著差异。非蝶类生物的质粒与有丝染色体的替代比为1:7. 7,蝶类生物的替代比为1:7.2。以有丝基因组树为框架,评估豆科植物有丝基因组特征的变化。
有丝分裂基因组组成和大小变异
对12个具有代表性的豆科有丝分裂基因组的基因、核转座元件(TEs)、MIPTs和重复序列进行了枚举(表)1).所有基因组含有三个RRNA,TRNA的数量范围为15至20.蛋白质编码基因的数量从31-37变化。推定的核衍生的TES贡献了3.5-5.1%的毒蛛,这与基因组大小密切相关(附加文件2:图S2a)。MIPTs对有丝分裂基因组大小的贡献极小(0.1-1.6%),与基因组大小的相关性低于TEs(附加文件)2:图开通)。
在12个豆科有丝分裂基因组中研究了两种串联和分散的重复家族(附加文件)1:表S2)。大部分重复序列是分散的,物种间串联重复序列的累计长度在0.1 ~ 1.8 kb之间。有丝分裂基因组具有大量且高度可变的短(< 100 bp)分散重复序列,这些重复序列部分或完全重叠于其他短、中(101-1000 bp)或大(> 1001 bp)重复序列。每个有丝分裂基因组的重复模式都极其复杂。因此,部分或全部重叠重复被计算为单个重复单元,以计算总重复次数。
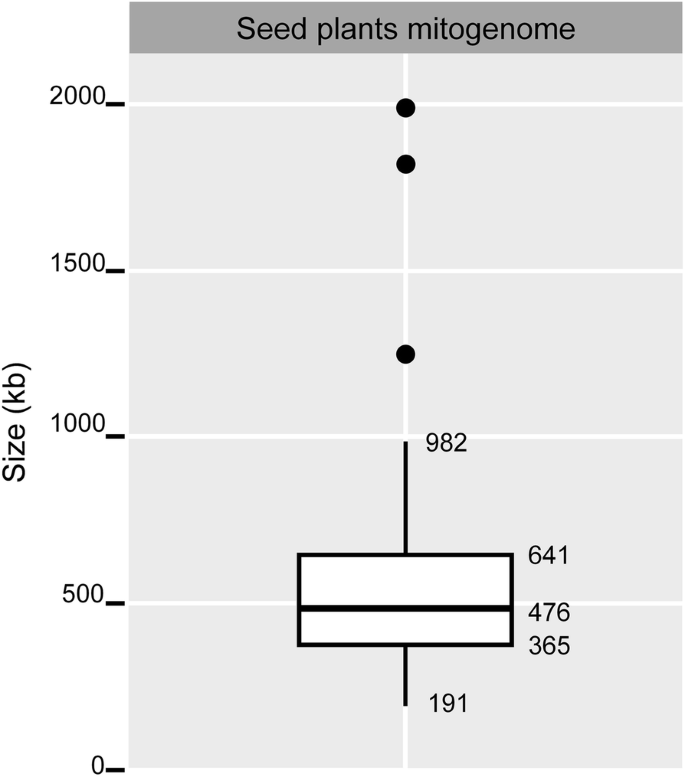
豆科植物的有丝分裂基因组大小差异很大,从271,618 bp不等Medicago truncatula)至729,504 bp银合欢trichandra)(CA. 2.7折;表1).种子植物有丝分裂基因组的中位大小(图。2)为476 kB,整体变化范围为191至982 kB(大约5倍,不包括异常值),并侧重位范围(IQR,中间50%)范围为365 kB至641 kB(大约2倍)。基于种子植物的毒蛛的中值(476 kB),Fabaceae毒蛛形成了两组非重叠尺寸组,大(588-730 kB)和紧凑(272-425 kB)。含有大型含有含有大的毒细胞的群体包括四个属(Tamarindus,Libidibia,血红素瘤,银合欢)在两个亚属(Detarioideae和Caesalpinioideae)中)维亚比亚法瓦(凤蝶亚科,蚕豆1和图。3.),而包含紧凑尺寸组紫荆(凤蝶亚科)和所有其他凤蝶亚科种。
种子植物中线粒体基因组的大小变异。种子植物数据基于来自NCBI的106种可用的完整毒蛛(https://www.ncbi.nlm.nih.gov/genome/organelle/) 2018年10月16日生效。没有单一主染色体的有丝染色体基因组不包括在内。方框显示四分位数范围(IQR,中间50%),方框内的水平线为平均值。黑色圆圈表示超过1.5 IQR值的异常值。须线覆盖了数据的范围,没有异常值
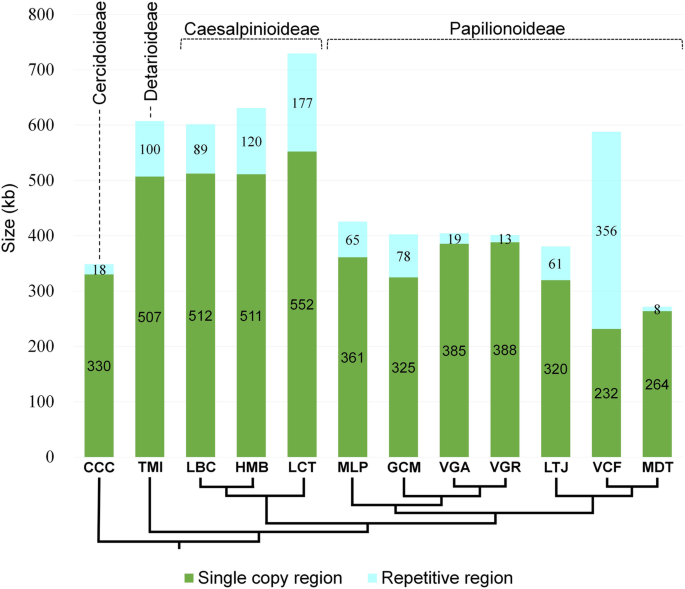
Fabaceae毒蛛中的单拷贝和重复DNA。解析单拷贝和重复区域并绘制在图1的Fabaceae的系统发育上。1.直方图中的值表示每个区域中包含的序列的数量(kb)。CCC =紫荆黄花,tmi = Tamarindus indica.,LBC = Libidibia coriariaHMB =Haematoxylum brasilettoLCT =银合欢trichandra中长期规划,=Millettia pinnata,gcm = 大豆VGA =豇豆属活动,VGR = 诉放射虫纲,ltj = 莲花japonicus.VCF =维亚比亚法瓦联合化疗=Medicago truncatula.重复内容的详细信息在附加文件中1:表S2
Mitogenome扩大在V. Faba.主要是由于最近重复序列的快速扩展。重复DNA占基因组(588.0 kb)的60.6% (356.3 kb)1)但除了一个大(> 1 kB)重复序列的所有副本中被排除在外,基因组大小是406.8 kB(附加文件1:表S2),类似于其他Papilionoideae毒蛛(图。3.).近期重复扩张在总重复积累中表现明显V. Faba.(附加文件1:表S2)。四属的有丝分裂基因组增大(Tamarindus,Libidibia,血红素瘤,银合欢)在两个亚科(Detarioideae和Caesalpinioideae)中不能单独归因于最近的重复生长,因为重复的比例适中(14.5-24.3%),单拷贝序列(500-552 kb)的数量与其他亚科相比非常高(表1和图。3.).
有丝分裂基因组基因的进化与内含子含量
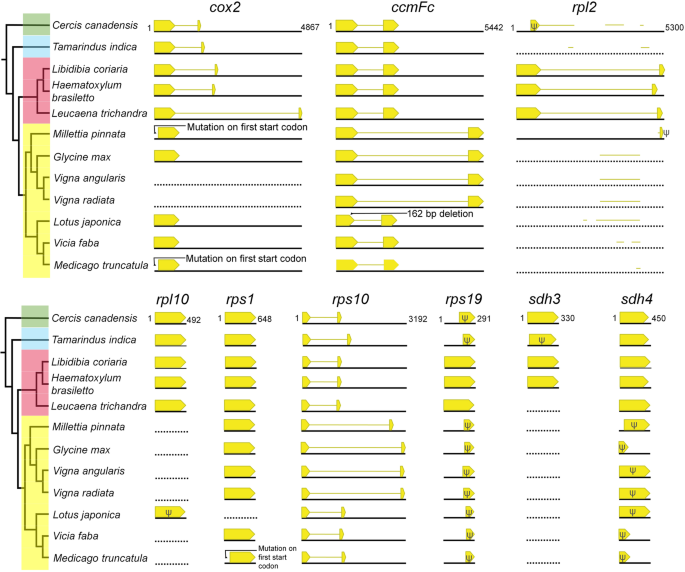
图中有丝分裂基因组系统发育树。1用于评估豆科植物中共享基因和独立基因及内含子损失。在所有豆科有丝分裂基因组中,41个蛋白编码基因中有30个是完整的2:图S3a)。其余11个基因中,4个核糖体蛋白基因(RPS2.,7,11,13)从所有豆科中丢失(假基因化、截短或删除)。七个基因的状况(cox2,rpl2,rpl10,RPS1.,rps19,sdh3,sdh4)在家族中是可变的(图。4).还有一个独特的162 bp内缺失(54个氨基酸)来自ccmFc的莲花相对于紫荆.凤蝶亚科的所有成员都有相同的损失rpl10和sdh4虽然剩余序列量不同于分类群。损失cox2和RPS1.是凤蝶属独有的吗豇豆属和莲花,分别。三个基因,rpl2,rps19和sdh3,在Fabaceae的亚属中失去了多次。的sdh3编码序列丢失了三次,来自Tamarindus(Detarioideae),银合欢(CaeSalpinioideae),以及所有取样的Papilionoideae。一个完整的rpl2和rps19仅存在于天麻科。对这两个基因的序列进行ML分析,得到的树支持豆科起源(截断在紫荆因为在这两种情况下,豆科在rosid分支中形成了一个强有力支持的单系群(92和89%的bootstrap值,附加文件2:图S4)。这些结果表明,该科保留了天然副本rpl2和rps19而其他的亚家族则经历了多次损失。
豆科的高度可变线粒体基因。分类群与相应亚科的关系(绿色=蠓科;蓝色= Detarioideae;红色= Caesalpinioideae;黄色=凤蝶亚科)在左边的分支图中被描绘。从图中所示的系统发生树绘制出进化图。1.黄条和线分别表示CDS区域和内含子区域。每个基因下面黑线的长度是基于物种中最长的基因拷贝,其值是给定的紫荆.没有可识别的编码序列的基因表示为虚线黑线。推定的假催化或截短的基因用psi(ψ)表示。不提出序列片段(<100bp)。序列片段的位置基于最长基因的相应区域测定。内含子和CDS长度对于同一基因成比例,但不属于基因之间
在三个基因中发现了内含子的存在/缺失和长度的广泛差异ccmFc(953 - 4100个基点),cox2(0;732 - 4080个基点)rps10(842 - 2829个基点)(图4).内含子ccmFc和rps10在papilionoideae的单胞内亚群中大大扩展(Millettia,甘氨酸和豇豆属).损失cox2凤蝶亚科均存在内含子。在非蝶类中,蝶的长度cox2内含子因种而异,并明显伸长银合欢,含麻糖蛋白的模突厥片的成员。多次对准揭示了另外的2.9kB序列银合欢cox2基因内区(附加文件2:图S5)。使用这个序列作为BLASTN查询在NCBI数据库中没有返回重要的匹配。与CENSOR数据库中已知的转座因子没有很强的匹配。在这项研究中完成的对四个有丝分裂基因组的BLAST搜索显示了与基因间间隔(IGS)的强烈匹配血红素瘤(位置15,062-17,424)。额外的对齐cox2内含子序列银合欢对应的IGS血红素瘤在2.4 kb上显示了94.9%的序列标识(附加文件2:图S5)。在有丝分裂基因组中银合欢这额外的cox2IGS区域中不存在内含子序列。串联重复序列(周期大小:51,拷贝数:4)位于独特序列的近3'末端,但未识别侧翼重复序列。Caesalpinioideae具有明显长长的内含子rpl2(4017 - 4307个基点)(图4).然而,尚不确定该内含子在caesalpininioideae中是否由于缺乏来自家族内近亲属的参考有丝分裂基因组而扩增。所有其他豆科有丝分裂基因组,包括缺乏可识别外显子序列的物种rpl2的片段序列类似于Caesalpinioideae中的内含子。
共享dna的成对变异和遗传距离
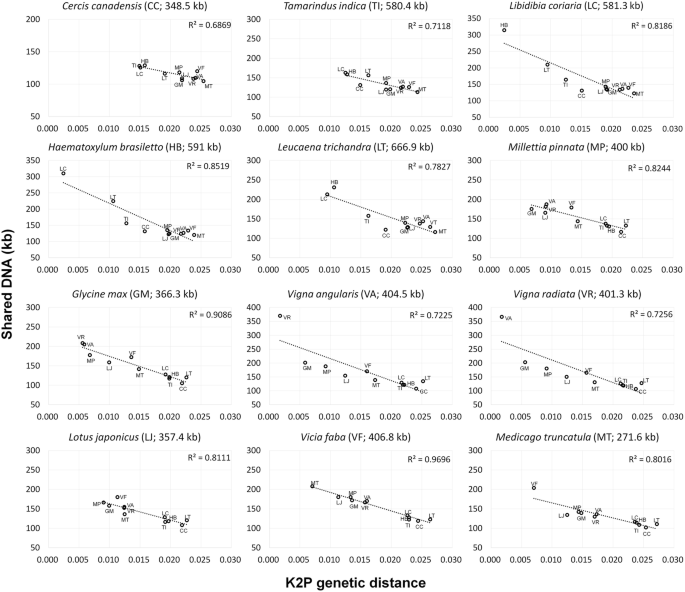
在Fabaceae Mitogomes之间评估共享DNA含量和Kimura 2参数(K2P)遗传距离(附加文件1:表S3和S4)和这两个参数的相关性测试。分析支持每个物种的强负相关(图。5).即使是总基因组大小相似的近亲物种,也很少共享DNA。例如,有两个秋葵科属(Libidibia和血红素瘤),差异为0.24%,仅共享约50%的有丝分裂基因组(附加文件1:表S3和S4)。凤蝶亚科的一个例子清楚地表明,在分化的早期阶段,共享DNA的迅速减少。Intrageneric变化豇豆属(0.1%的差异)显示92%的有丝分裂基因组DNA共享,而属间比较诉活动和甘氨酸(0.59%的差异)显示约50%的DNA是共享的。大多数基因间比较的共享率不到50%。在所有的比较中,至少有100 kb的DNA是共享的,这主要包括基因和它们的侧翼区域。
12个代表性豆科有丝分裂基因组共享DNA与K2P遗传距离的关系单个图上的分类单元名称代表查询的物种,每个图上的点代表11个学科豆科物种。每个被试物种用两个字母首字母缩写和有丝分裂基因组大小来表示,不包括大量的大重复副本(> 1 kb),在每个图表上方的括号中。关于共享DNA和K2P遗传距离的完整信息在附加文件中1:表S3和S4
豆科和Lophophytum
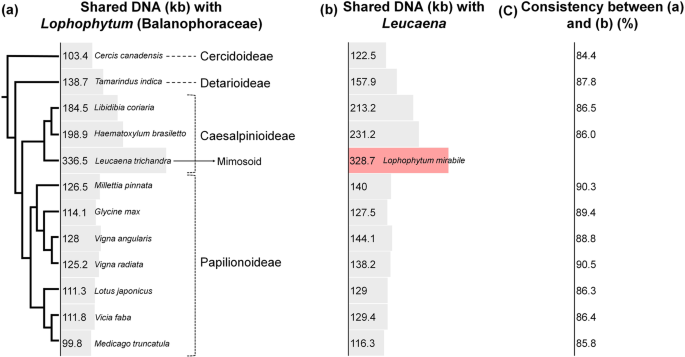
全寄生物有丝分裂基因组的BLAST比较Lophophytum(Balanophoraceae)与12个Fabaceae毒蛛中的每一个进行(图。6一个)。银合欢分享了最多的LophophytumDNA (336.5 kb)。在凤蝶亚科内,共有DNA的数量从99.8-126.5 kb不等。在非蝶类中,与亲缘关系相关的共享DNA逐渐增加银合欢.这种模式与使用促使促型的分析非常相似银合欢作为其他11种豆科植物的查询序列Lophophytum(图。6B.).在这个比较中,共享的dna银合欢与Lophophytum328.7 kb。这两种分析的一致性在于共享的DNA的数量,其中大多数代表了~ 100 kb的基因和保守的侧翼区域(图。6c)。
豆科与全寄生性植物DNA共享Lophophytummitogenomes(蛇菰科)。(一个的有丝分裂基因组Lophophytum mirabile查询包含12种豆科植物的主题数据库。(b的有丝分裂基因组银合欢trichandra是查询一个主题数据库,包括其他11个豆科和Lophophytum mirabile.(c) (一个)和(b)计算出来。从图中所示的系统发生树绘制出进化图。1.分类名称在每个共享DNA值旁边指示。来自灰吧的分类名称(一个)和(b)是相同的,而红色条则表示Lophophytum取代l . trichandra在分析
讨论
有丝分裂基因组大小变化的原因
在大多数真核生物谱系中观察到细胞器基因组大小的缩小,基因组压缩的选择性压力可能提供复制优势[22].然而,在陆地植物中,与大多数动物相比,有丝分裂基因组的大小通常是扩大的[23].豆科植物的有丝染色体组显示出两种不同的大小变化模式1和图。3.).对植物促型大小的约束仍然模糊,但是对基因组大小的放松选择是对高度可变和大型毒蛛的一种合理解释(图。2).考虑每株植物线粒体的低拷贝毒素数量(常常只有一个或部分拷贝)[48.,选择的一个来源,线粒体内有丝基因组之间复制的竞争[49.,相对于动物可能会减少。豆科的副源亚群中比较稳定的基因组大小。Tamarindus到血红素瘤,Millettia到莲花(图。3.和附加文件1:表S2)]表明,通过遗传和化学计量的转变为大量不同的基因组尺寸,诱导毒蛛的完整性在家庭中并不常见。
很明显,在Fabaceae中存在几种独立的促丝孔尺寸波动(图。3.).然而,考虑到豆科(770属,20000种)的总体大小和多样性,目前的分类单元取样仍然稀疏,因此很难确定波动的数量和时间[29].值得注意的是,有丝分裂基因组大小波动与核染色体进化之间存在一种一致的模式[50.,51.,52.].紫荆,最早发散的患有第二个小促滤器组的思考的成员(表1),具有非多倍体遗留核基因组[52.].整个基因组复制发生在其他5个亚科的基部附近[50.,51.].四个属Tamarindus,Libidibia,血红素瘤,银合欢在Detarioideae和Caesalpinioideae亚科中有较大的有丝分裂基因组。最近发表的有丝分裂基因组Styphnolobium是凤蝶亚科早期分化的属,其大小为484,916 bp,也较其他凤蝶属大[40].这些观察结果表明,大量有丝分裂型尺寸波动,可能伴随着Fabaceae的深层节点的核染色体演变。需要扩大含有毒素的分类物和整个家庭的核基因组,以确认这种模式。
通过参考基因组的缺乏和核和促型非基因序列的快速发展性质来解析促毒素内的混杂DNA的起源是挑战的10,53.].四个属的促扩音Tamarindus,Libidibia,血红素瘤和银合欢可能是来源不明的混杂DNA整合的结果(图。3.).缺乏参考核基因组和额外的混杂序列在物种中不保守的事实(图。6)妨碍追踪序列的起源。利用线粒体编码序列进行的系统发育分析没有显示出种间HGT的强烈信号(图。1和附加文件2MIPTs的贡献非常低,且与基因组大小相关性不强(见表4)1和附加文件2:图开通)。这些证据表明,DNA不太可能因特异性HGT事件与促滤板中的促滤器编码序列或IGT事件产生。扩大的诱变蛋白酶与序列的存在强烈的相关性,其具有核对核特性的身份(附加档案2:图S2a)。可能大的含有诱导的诱导毒素与核基因组更容易掺入核基因组的DNA(例如,有限的转移窗假设)[54.,55.].TEs可能代表来自核基因组(即MINCs)的大量DNA插入的残余轨迹[20.].为了阐明早期发散的Fabaceae谱系的进化事件,需要更多的核和线粒体参考基因组。
蝶类物种的有丝分裂基因组扩张维亚比亚法瓦是由于获取了大量重复序列。有几个类似扩大的案例V. Faba.[1.4折;407 kb(基因组大小不包括除一次大重复外的所有基因组)- 588 kb(基因组总大小)]已在各种作物植物的物种中被报道[例如,甜菜属(368 KB - 501 KB,1.4倍)[56.];玉米(535kb - 739kb, 1.4倍)[57.];Brassica oleracea(219 - 360kb, 1.6倍)[58.,59.];和栽培稻(402 kB - 637 kB,1.6倍)[60.].然而,这些物种的变异限于特定的载体。催乳素瘤的调查Brassica oleracea品种(61.揭示了基因组扩增是通过在所有品种中低频率存在的重复序列之间进行重组而实现的。
在Papilionoideae中,促进促进的促进型大小Medicago(图。3.).如上所述,豆科植物的副aphyletic亚群中相对稳定的有丝染色体大小不支持小缺失随时间积累的情况。然而,有证据表明植物有丝分裂基因组可以在短时间内丢失大量的DNA [4,62.,63.].一些的有丝染色体组硅宾物种包括数十种染色体,总总尺寸约为11 MB [5,64.].最近的种间和种内群体水平分析表明,有丝分裂基因组美国noctiflora经历了一个由整个染色体丢失引起的基因组减少的阶段[63.].放大的有丝染色体具有更分散的重复有丝染色体具有更高的概率形成多部分的有丝染色体,其中一些有丝染色体是紧凑的,不携带基因。例如,银合欢在豆科标本中,有丝分裂基因组最多,重复总数最多(附加文件)1表S2),包括一个假定的不含基因的亚有丝分裂基因组分子[39].形成无毒杀菌剂染色体和随后通过偏析排除(例如线粒体分裂,细胞分裂或遗传)可以解释毒蛛的压实[63.].豆科植物有丝分裂基因组染色体丢失、IGT和HGT获得外源DNA以及获得重复DNA可能是造成有丝分裂基因组大小波动的原因。这些过程可以解释种子植物有丝分裂基因组大小的增加和减少(图。2).
线粒体系统发育及其在豆科系统发育中的潜力
来自线粒体和塑性基因序列的树拓术在Fabaceae中完全一致(图。1).两种树之间的主要区别在于分枝长度,特别是凤蝶亚科。非蝶类的线粒体树与质体树的分枝长度比为1:7. 7,而蝶类的线粒体树与质体树的分枝长度比为1:7.2。类凤蝶胞浆内核苷酸取代率的加速[65.]的部分原因是世代时间较短,因为大多数凤蝶属植物是草本植物,而其他亚科则普遍为木质。与其他亚科相比,蝶类的有丝染色体组也表现出轻微的加速,这也可能是由于世代时间短造成的。有丝分裂基因组编码区的低核苷酸替代率被认为是解决更深层次的系统发育关系的优势[23,66.].然而,大多数被子植物系统发育是基于质体和核数据,而有丝分裂基因组数据未得到充分利用[67.].这可能是由于HGT事件的风险,导致外源线粒体基因纳入分析。这导致数据冲突,导致无法解决的树和降低支持值的系统发育估计基于多基因。然而,豆科植物有丝染色体和质体数据的整体拓扑一致性和高支持值表明,有丝染色体的编码区域没有经历太多的HGT,利用这两个细胞器的数据在该家族未来的系统发育研究中可能是有用的。
线粒体基因的多重丢失和谱系特异性内含子大小变异
在豆科中,我们推测有7个基因丢失(cox2,rpl2,rpl10,RPS1.,rps19,sdh3和sdh4) (图。4和附加文件2:图S3)。Adams等人。[32通过Southern杂交证明了被子植物中多个线粒体基因的多次丢失。目前的发现与Adams等人的观点大体一致[32,差异是由于假基因的存在(附加文件2图S3),其中一些可能在Southern的分析中产生了信号。三个基因(rpl2,rps19,sdh3)已经从豆科有丝分裂基因组中多次丢失(图。4).在系统发育分布上有3种损失sdh3.而其他豆类缺乏rpl2和rps19从丝分裂剂中,所有Caesalpinioideae都有完整的拷贝。与其他Angiasperms的系统发育分析(附加文件2图4)表明,这些基因在大多数豆科植物中的存在不是通过种间HGT的再捕获事件的结果,而是本地祖先基因的残余。
从不同谱系中的诱导型毒素的共享基因损失不一定反映多个IGT事件,因为对核基因组的单一肠梗阻转移可以以各种方式影响后代谱系[24,25].如果在线粒体或细胞核中没有选择优势,线粒体基因丢失或保留的斑块性系统发育分布是可以预期的[27].的例子rps19保留在禾本科(禾本科)[68.,69.]与保留相当rpl2和rps19在Fabaceae。核拷贝需要几个中间阶段在IGT后静音线粒体基因[24,70].中级步骤持续了6000万年rps19在雀麦草(叶片),而核拷贝的缺失可使水稻保留祖先的线粒体序列(栽培稻)[69.].同时,其他线粒体基因在不同丢失阶段(rpl10和sdh4)表明大多数基因损失在Fabaceae植物中平行进行(图。4和附加文件2:图S3)。
虽然基因含量的变化支持一个复杂的进化史,但内含子含量和长度的变化要简单得多,往往局限于豆科的单个分支(图。4).在…的情况下cox2,内含子的丢失仅限于凤蝶亚科。三种豆科线粒体基因的内含子大小差异(cox2,rps10,ccmFc)是相当大的,但与其他植物没有本质上的区别。大多数陆地植物有丝分裂基因组内含子小于6 kb [71.]但一个值得注意的例外是11.4 kBcox2内含子的Nymphaea.[72.].在光合植物有丝分裂基因组中已经报道了几种内含子伸长[73.].外来DNA的大量插入是内含子序列增加的主要来源rps10和ccmFc在豆科(37,40而不是像报道的其他陆生植物的重复序列的扩散。Psilotum(蕨类植物的盟友)[71.];铁树(裸子植物)74.];和Nymphaea.(基底被子植物)72.]]。Chang等[37]建议弥鼻病毒是额外的内含子序列的推定来源rps10,以及附加内含子序列的起源ccmFc是未知的(40].
核苷酸排列cox2基因银合欢和其他豆类建议序列插入(约2.9 kb)到内含子(附加文件2:图S5)。虽然BLAST分析使用独特的内含子序列查询NCBI数据库没有产生强匹配,但大部分(~ 82.8%)与caesalpininioid属IGS区域的核苷酸同源性高(94.9%)血红素瘤(附加文件2:图S5)。这说明越短越好cox2内含子的血红素瘤和Libidibia表示演化枝中祖先的长度,包括银合欢.的插入nad4L基因内nad1内含子被识别卷柏(蕨类植物的盟友)[75.].的cox2然而,从CaeSalpinioideae显示的内含子伸长率可能是由于IGS序列转移到内含子中,该现象尚未在种子植物的毒蛛中得到完全理解。
遗传距离的毒素衰变
12种Fabaceae物种中的所有成对关系都与Guo等人的研究结果一致。[53.],显示出共享DNA的数量与编码区序列差异之间有很强的负相关(附加文件1:表S3,S4;无花果。5).一个值得注意的发现是,当两个有丝分裂基因组序列的50%不再共享时,半衰期非常短。这支持了大多数有丝分裂基因组DNA可能没有功能的建议[76.].然而,在大多数情况下,每个豆科物种,包括最分散的类群,共享至少100 kb的序列,包括基因区域和接近基因的IGS。这些区域代表有丝分裂基因组的核心,这个值与菊科有丝分裂基因组中共享的DNA数量相当(约88 kb) [77.].克里斯腾森(2,6]假设,准确且容易出错的双链断裂修复机制的结合,以及在编码区域没有有害突变的情况下对有丝染色体分子的后续选择,可以产生这种现象。然而,详细的机制、外来序列迁移(即种间HGT)和已有序列变异对IGS快速变化的贡献[78.仍然模糊。无论是什么原因导致了共享的有丝基因组DNA的快速下降,在豆科中有明显的编码序列保存的证据(图。5).
从含羞草类物种向Lophophytum
对牛鞭草亚科、芫荽亚科和两个非粟粒类牛鞭草亚科4个新有丝染色体的分析(图。6)增强来自模突豆类的广泛水平促滤泡转移的支持Lophophytum[39,42.].然而,两个重要的问题仍然存在:1)含羞体寄主物种是唯一的贡献者Lophophytum豆科植物中的有丝分裂基因组[392)有多少DNA从豆科植物转移到Lophophytum?[42.[商务英语比较金额Lophophytum(图。6一)和银合欢(图。6b) 11个豆科植物有丝分裂基因组共享的DNA显示出相同的模式,排除了大量外来DNA进入的可能性Lophophytum起源于非含羞草豆科。DNA共享的程度银合欢和Lophophytum(~ 330kb)表明,含羞体寄主物种(例如:Anadenanthera,Enterolobium,印加,Piptidenia.,Pithecolobium.)[79.> 330kb(附加文件2:图S6)。此外,两种分析之间共享DNA的一致百分比(平均为87.5%)(图。6c)可能表明Lophophytum含有宿主含羞体有丝分裂基因组至少87.5%的DNA。事实上,即使在非常密切相关的属中,有丝分裂基因组衰变的前半衰期也很短。5)表明这个估计是保守的。
结论
由于完整序列的缺乏,植物毒蛛的复杂结构使得对比较分析进行了挑战性。在过去的十年中,随着更促进的促进型序列可获得的理解,了解奇异变化的模式和原因大大改善,然而,有很多促型促催化序列可用于比较分析。一个患有促型比较的焦点的一个家庭是Fabaceae,但大多数这些调查是基于来自六个公认的亚属的物种,Papilionoideae。这里报告的四种新测序的筛选毒素将系统发育覆盖范围延伸至四个亚壳。该家庭在古代和近期经历了几种大量的丝分感器大小波动。这些大小变化的原因在不同的世系中是不同的。在整个Falbaceae的进化历史中发生了多种,独立的七种基因损失。相反,内含子含量和长度的变化仅限于单个分支。最后,在FABACEAE上扩展的谱系采样提供了新的洞察力转移来自FABACEAE进入寄生植物的促滤波器序列Lophophytum.
方法
有丝分裂基因组组装和验证
豆科四种,紫荆黄花,柽柳属,马鞭草属(=石莲子coriaria),Haematoxylum brasiletto,进行有丝分裂基因组测序。四个品种的种子最初是从eBay (l . coriaria)和美国农业部国家植物种质资源系统(c .黄花,t .籼和h . brasiletto)在Schwarz等人[80].凭证标本由Erika N. Schwarz鉴定,并保存到德克萨斯大学奥斯汀植物标本馆(TEX-LL)。由Schwarz等人生成的原始读取[80]用于有丝分裂基因组组装。这些100 bp的配对末端序列是由Illumina (San Diego, CA)对包含约700 bp插入的文库进行测序生成的。在genei7.1.9 (https://www.geneious.com).在有丝分裂基因组组装之前,通过将所有reads映射到相应的完整的plasome,识别和排除了plasome reads [c .黄花(KF856619),t .籼(KJ468103),l . coriaria(KJ468095)和h . brasiletto(KJ468097)]使用自定义选项(允许每个间隙和不匹配的最大值为1%,只映射附近匹配的配对读取,保存未使用读取列表)。在geneius中,使用大约3000万个质体过滤读取数据,以低灵敏度进行从头组装。然后将所有经过质体过滤的读取序列映射到组装的contigs上。排除了低覆盖率(< 100X)的推定核基因组contigs。在剩余的contigs中,排除了高覆盖率的核contigs(如核糖体DNA重复单元),并通过BLAST搜索参考Fabaceae有丝分裂基因组序列(https://www.ncbi.nlm.nih.gov/genome/organelle/)使用BLASTN 2.8.0+ [81.]的默认选项。
为了完成和验证促滤蛋白组件,通过在底漆中设计的引物进行聚合酶链反应(PCR)[82.].引物序列和目标区域列在附加文件中1:表S5。通过PCR和Sanger测序证实了contigs之间的空白,分散重复,MIPTs大于约700 bp文库插入,以及大重复区域和单拷贝区域之间的连接。对于大于1.5 kb的MIPTs,采用TaKaRa PrimeSTAR GXL (TaKaRa Bio USA, Inc., Mountain View, CA, USA)进行长程PCR,扩增子的Sanger测序由位于奥斯汀的德克萨斯大学基因组测序与分析中心(University of Texas sequencing and Analysis Facility)采用巢式PCR完成。在Geneious中对每个有丝分裂基因组进行手工提纯。最后,通过两个不同的数据集和选项来检查组装和精炼的有丝分裂基因组的覆盖率。第一个映射使用了带有自定义选项的塑料过滤读取(每个缺口和不匹配的最大值为5%,只允许匹配附近的配对读取)。第二个映射使用总原始单端读取与低灵敏度选项。
基因组注释和分析
除了四个完整的有丝分裂基因组,八个以前发表的有丝分裂基因组被重新注释(附加文件1:表S6)进行比较分析。利用Geseq的豆科有丝分裂基因组注释[83.].选择了三种推动量的毒蛛(Psilotum无偿: KX171638和KX171639;银杏叶: KM672373;鹅掌楸tulipifera: NC_021152)作为BLAT参考序列,每个序列包含一组41个保守的祖先种子植物蛋白编码基因[53.,71.,84.].在Geneious中对注释进行评估和手工修正。开始和停止密码子被手动调整以适应开放的阅读框架。外显子和内含子的边界是通过与从NCBI (https://www.ncbi.nlm.nih.gov/genome/organelle/).Trnascan-SE v2.0检查TRNA [85.].所有4个完整和注释的有丝分裂基因组均保存在GenBank (MN017226 - MN017229)中。
假设转座因子(TEs)使用CENSOR网络服务器进行了调查[86.],并选择Viridiplantae作为序列源,如Park等人所述[87.].MIPTs的数量和长度由缺省选项BLASTN 2.8.0+评估。每个有丝分裂基因组被用作对包含相应质体的主题数据库的查询(附加文件)1:表S6)。推测的MIPTs小于100 bp被排除在估计之外。
研究了两种重复序列(串联重复序列和分散重复序列)在每个有丝分裂基因组中的分布。串联重复序列使用串联重复序列查找器4.09版本进行分析[88.]的默认选项。通过使用每个有丝分裂基因组作为实验对象和BLASTN分析查询来识别分散重复,单词大小为7,e值为1e-6,遵循Guo等人的方法[53.,可检测短至30 bp的重复序列。所有爆炸击中的部分都保留了下来。串联重复序列和分散重复序列的坐标信息被转移到每个有丝分裂基因组作为geneous的注释。重复序列之间的重叠区域被排除在估计之外,并计算有丝分裂基因组中重复和单拷贝DNA的百分比。在4个大小区间(30-100、101-300、301-1000和> 1001 bp)中估计重复的BLAST命中数。在估计重复次数时,将部分或全部重复次数视为单个重复单元。
种子植物有丝分裂基因组大小变异的估计
为了估算种子植物有丝分裂基因组的大小变化,我们评估了NCBI提供的106个完整的有丝分裂基因组(https://www.ncbi.nlm.nih.gov/genome/organelle/;于2018年10月16日)。
系统发育分析
为了推断豆科植物有丝分裂基因组之间的系统发育关系,并检验与质体系统发育的一致性,对两个基因组的数据进行了最大似然(ML)分析。除12种具有代表性的豆科植物外,分类单元取样还包括固氮分支的另外两个属(Cucurbita和罗莎)和两属的Malphigiales (杨树和柳树)作为外组(附加文件1:表S6)。从每个细胞链基因组中提取来自16个纳税群的所有共用线粒体(26)和体层(68)蛋白质编码基因(附加文件1:表S7)。来自每个细胞器基因组的基因序列在两个单独的数据集中倾斜,并与Mafft V.7.017对齐[89.]使用默认选项。对对齐的区域被删除或手动调整每个对准。通过JModeltest v.2.1.6中的Akaike信息标准(AIC)选择核苷酸替代模型[90.].使用RAxML v.8进行ML分析(GTR + I + G, 1000次bootstrap重复)[91.]在CIPRES科学门户[92.].
检验…的系统发育地位rpl2和rps19从已公布的线粒体序列中提取了四个Caesalpinioideae有丝基因组的基因,代表性被子植物的序列(附加文件)1:表S6)。紫荆黄花来自Cercidoideae也包括在该分析中。如上所述进行对准和ML分析(GTR + G)。
共同的DNA分析
利用与重复分析相同参数的BLASTN对12个豆科有丝分裂基因组的共享DNA进行了两两比较。为了避免因包含大重复而过高估计共享DNA,从所有有丝分裂基因组中手动删除了> 1 kb以外的所有重复。成对木村2参数(K2P) [93.]使用MEGA version 7.0计算12种豆科植物的遗传距离[94.基于26条线粒体CDS序列的比对,用于系统发育分析。
利用全寄生虫的多部有丝分裂基因组(KU992322-KU992380和KX792461)的连接序列估计共享DNA检测HGTLophophytum以12个豆科有丝分裂基因组为研究对象。进行了类似的比较银合欢作为一个疑问和12种(其他11种Fabaceae物种和Lophophytum)作为主题。通过除以价值来计算比较之间的共享DNA的百分比Lophophytum由银合欢.
数据和材料的可用性
所有4个完整和注释的有丝分裂基因组均保存在GenBank (MN017226 - MN017229)中。
缩写
- 英国石油公司:
-
完全
- cd:
-
编码序列
- HGT:
-
水平基因转移
- IGS:
-
基因间的间隔
- IGT:
-
细胞内的基因转移
- 差:
-
四分位范围
- K2P:
-
木村出现
- kb:
-
千杆杆
- MINC:
-
起源细胞核的线粒体DNA
- 初期:
-
质体起源的线粒体DNA
- ML:
-
最大似然
- 聚合酶链反应:
-
聚合酶链反应
- TE:
-
转座因子
参考
- 1.
割草机JP,Sloan DB,Alverson AJ。植物线粒体基因组多样性:基因组学革命。在Wendel JF,Greilhuber J,Dolezel J,Leitch IJ,编辑。植物基因组多样性Vol。1.纽约:斯普林克;2012.第123-144页。
- 2.
线粒体DNA修复与基因组进化。In: Logan DC,编辑。年度植物评论第50卷。纽约:著名;2018.11-32页。
- 3.
Scheffler即线粒体。纽约:Wiley-Liss。公司;1999.
- 4.
Skippington E Barkman TJ Rice DW Palmer JD寄生植物的微缩有丝分裂基因组毒蕈氏菌霉素极其不同,充满活力,丢失了所有河畔基因。Proc Natl Acad Sci U S A. 2015; 112:E3515-24。
- 5。
Sloan DB, Alverson AJ, Chuckalovcak JP, Wu M, McCauley DE, Palmer JD, Taylor DR.在开花植物线粒体中具有异常高突变率的巨大多染色体基因组的快速进化。公共科学图书馆杂志。2012;10:e1001241。
- 6。
植物线粒体修复机制和选择中的基因和垃圾。基因组生物学进展。2014;6:1448-53。
- 7。
植物线粒体基因组中不同寻常大小的重复:鉴定、发生率和进化。G3-Genes Genomes Genet. 2019; 9:549-59。
- 8。
环的奥秘:高等植物线粒体基因组的结构和复制。植物科学进展。1997;2:477-83。
- 9。
Ogihara Y,Yamazaki Y,Murai K,Kanno A,Terachi T,Shiina T,Miyashita N,Nasuda S,Nakamura C,Mori N.谷物线粒体基因组完全核苷酸测序揭示的谷物细胞基因组的结构动态。核酸RES。2005; 33:6235-50。
- 10.
Goremykin VV,Lockhart PJ,Vella R,Velasco R.的线粒体基因组马吕斯有明显种子植物线粒体基因组扩张的输入驱动假说。植物j . 2012; 71:615-26。
- 11.
berthorsson U, Adams KL, Thomason B, Palmer JD。开花植物中广泛的线粒体基因水平转移。大自然。2003;424:197 - 201。
- 12.
理查森AO,帕尔默JD。植物的水平基因转移。J Exp Bot. 2006; 58:1-9。
- 13.
Mower JP, Jain K, Hepburn NJ。水平转移在塑造植物线粒体基因组中的作用。Adv Bot Res. 2012; 63:41-69。
- 14.
Alverson AJ, Wei X, Rice DW, Stern DB, Barry K, Palmer JD。从完整的线粒体基因组序列深入了解线粒体基因组大小的进化Citrullus lanatus和Cucurbita浆果(葫芦科)。生物学报2010;27:1436-48。
- 15.
Alverson AJ, Rice DW, Dickinson S, Barry K, Palmer JD。黄瓜细菌大小的多染色体线粒体基因组的起源和重组。植物细胞。2011;23:2499 - 513。
- 16。
甘丝C,桑切斯 - 普埃塔米。植物线粒体的外塑序列经常通过线粒体到线粒体水平转移获得。SCI REP。2017; 7:43402。
- 17。
植物线粒体通过通透性过渡孔复合体积极导入DNA。EMBO j . 2003; 22:1245-54。
- 18。
Archibald JM,Richards Ta。基因转移:任何东西都进入植物线粒体。BMC BIOL。2010; 8:147。
- 19。
高C,仁X,梅森作为,刘H,萧米,李杰,富d。植物水平基因转移。Func综合基因组学。2014; 14:23-9。
- 20.
赵宁,王颖,华军。线粒体在植物基因组间基因转移中的作用:来源和库。国际分子生物学杂志。2018;19:547。
- 21.
关于有丝分裂细胞的起源。《经济学家》1967;14:225-74。
- 22.
Selosse M-A, Albert B, Godelle B.减少细胞器基因组大小有利于基因转移到细胞核。趋势生态进化。2001;16:135-41。
- 23.
陆生植物的线粒体DNA:系统发育角度的特性。咕咕叫麝猫。2004;46:123-39。
- 24.
Bonen L.线粒体基因离开家。新植物。2006; 172:379-81。
- 25.
亚当斯KL,帕尔默JD。线粒体基因内容的进化:基因丢失并转移到细胞核。中国生物医学工程学报。2003;29(4):489 - 494。
- 26.
苦涩的JM,Palmer JD。RNA介导的基因转移coxII在开花植物进化过程中从线粒体到细胞核。细胞。1991;66:473 - 81。
- 27.
亚当斯kl,歌曲,roesller pg,nugent jm,doyle jl,doyle jj,palmer jd。在作用中的细胞内基因转移:双重转录和核和线粒体的多个沉默cox2豆类的基因。美国国立科学学院1999;96:13863-8。
- 28.
Gepts P,Beavis WD,Brummer EC,Shoemaker RC,Stalker HT,Weeden NF,Young Nd。豆类作为模范植物家庭。通过基因组学会,交叉豆类的食物和饲料报告的基因组学。植物理性。2005; 137:1228-35。
- 29.
豆类系统发育工作组(LPWG)。基于分类系统发育的豆科新亚科分类。分类单元。2017;66:44 - 77。
- 30.
王志强,王志强,帕尔默。线粒体基因片段转移:核糖体蛋白基因的裂变rpl2部分或全部基因转移到细胞核。生物学报2001;18:2289-97。
- 31.
Adams KL, Rosenblueth M, Qiu Y-L, Palmer JD。被子植物进化过程中两个线粒体琥珀酸脱氢酶基因的多重丢失和转移。遗传学。2001;158:1289 - 300。
- 32.
Adams KL, Qiu Y-L, Stoutemyer M, Palmer JD。间断进化的线粒体基因含量:在被子植物进化过程中,线粒体基因丢失和转移到细胞核的高和可变率。美国国家科学学院学报2002;99:9905-12。
- 33.
Alverson AJ, Zhuo S, Rice DW, Sloan DB, Palmer JD。豆科植物的线粒体基因组豇豆属辐射动物以及短线粒体重复序列的重组分析。《公共科学图书馆•综合》。2011;6:e16404。
- 34.
Kazakoff Sh,Imelfort M,Edwards D,Koehorst J,Biswas B,Batley J,Scott Pt,Gresshoff PM。捕捉生物燃料井口和强国:豆科植物叶片和线粒体基因组Pongamia pinnata.《公共科学图书馆•综合》。2012;7:e51687。
- 35.
红豆细胞器全基因组序列的从头组装(豇豆属活动)使用下一代定序器。品种sci。2013; 63:176。
- 36.
豆科植物的线粒体基因组序列维亚比亚法瓦.前植物SCI。2013; 4:128。
- 37.
张某,王Y,卢茹,盖j,李继,楚p,guan r,zhao t.大豆线粒体基因组揭示了细胞内和系统发育水平的复杂基因组结构和基因演化。Plos一个。2013; 8:E56502。
- 38.
毕超,王旭,徐勇,魏胜,石勇,戴旭,尹涛,叶宁Medicago truncatula.线粒体DNA B. 2016; 1:22 2 - 3。
- 39。
Kovar L,Nageswara-Rao M,Ortega-Rodriguez S,Dugas DV,Straub S,Cronn R,Strickler SR,Hughes Ce,Hanley Ka,Rodriguez DN。基于PACBIO的线粒体基因组组装银合欢trichandra(豆科)和线粒体RNA编辑的基因内评估。基因组生物学进展。2018;10:2501-17。
- 40.
石y,刘y,张s,zou r,唐家,穆波,彭y,董生,全部线粒体基因组序列的组装与对比分析槐花“JinhuaiJ2”。《公共科学图书馆•综合》。2018;13:e0202485。
- 41.
于涛,孙丽,崔海涛,刘松,门俊,陈松,陈勇,陆超。一种第三纪常绿木本植物的线粒体基因组Ammopipthus蒙古斯.线粒体DNA B. 2018; 3:9-11。
- 42.
豆科植物线粒体DNA向全寄生物的基因组转移Lophophytum mirabile(蛇菰科)。mol phylocyet evol。2019; 132:243-50。
- 43.
Sanchez-Puerta M,GarcíaLe,Wohlfeiler J,Ceriotti LF。全身同源物在全洛杉矶植物中无与伦比的替代替代替代本地线粒体基因。新植物。2017; 214:376-87。
- 44.
Ruhlman Ta,Jansen RK。开花植物的塑性基因组。在:Maliga P,编辑。叶绿体生物技术:方法和协议Vol。1132.纽约:斯普林克;2014. p。3-38。
- 45.
割草机jp,vickrey tl。土地植物塑性基因组中的结构多样性。ADV BOT RES。2018; 85:263-92。
- 46.
Gualberto JM, Newton KJ。植物线粒体基因组:动态和突变机制。植物学报。2017;68:225-52。
- 47.
Jansen RK, Ruhlman TA种子植物质体基因组。在:博克R,努普V,编辑。叶绿体和线粒体的基因组学。纽约:施普林格;2012.103 - 26页。
- 48.
体细胞线粒体中基因拷贝数极低且可变。植物j . 2010; 64:948-59。
- 49.
王志强,王志强,王志强,等。植物线粒体基因组的动态变化:一个三层次选择过程的模型。遗传学。1996;144:369 - 82。
- 50。
Cannon SB, McKain MR, Harkess A, Nelson MN, Dash S, Deyholos MK等。结瘤和非结瘤豆科植物早期辐射中的多倍多倍体事件。生物医学进展。2015;32:193-210。
- 51。
Ren L, Huang W, Cannon SB. .祖先基因组的重建揭示了选择的豆科植物的染色体进化史。新植醇。2019;223:2090 - 103。
- 52。
Stai JS, Yadav A, Sinou C, Bruneau A, Doyle JJ, Fernànde-Baca D, Cannon SB紫荆:一般多倍体豆科的非多倍体基因组遗物。植物学报。2019;10:345。
- 53。
郭伟,范伟,杨gj,克努普V,帕尔默JD, Mower JP。银杏和千岁兰有丝染色体组揭示裸子植物线粒体进化的极端差异。摩尔生物进化。2016;33:1448-60。
- 54。
史密斯博士,克罗斯比K,李RW。核质体DNA丰度与质体数量之间的相关性支持有限转移窗假说。基因组生物学进展。2011;3:365-71。
- 55.
将有限转移窗口假说扩展到细胞器间DNA迁移。基因组生物学进展。2011;3:743-8。
- 56.
Satoh M,Kubo T,Nishizawa S,estiati a,itchoda n,mikami t。糖甜菜的细胞质雄性无菌型和正常型线粒体基因组共享相同的已知功能基因,但表达ORF的含量不同。Mol Gen Genomics。2004; 272:247-56。
- 57.
艾伦·乔Fauron厘米,风骚女子P,罗克L, Oddiraju年代,林GN, Meyer L,太阳H,金正日K,王c中比较两个肥沃和三个败线粒体基因组的玉米。遗传学。2007;177:1173 - 92。
- 58.
关键词:线粒体基因组测序,线粒体基因组,进化机制,线粒体基因组芸苔属植物.BMC基因组学。2011; 12:497。
- 59.
Grewe F, Edger PP, Keren I, Sultan L, Pires JC, Ostersetzer-Biran O, Mower JP。11个十字花科植物线粒体基因组与线粒体转录组的比较分析Brassica oleracea.线粒体。2014;19:135-43。
- 60.
Bentolila S,Stefanov S.基于雄性育和雄性无菌线粒体基因组完整序列的水稻线粒体进化重新评估。植物理性。2012; 158:996-1017。
- 61.
Tanaka Y,Tsuda M,Yasumoto K,Terachi T,Yamagishi H.完全线粒体基因组序列Brassica oleracea同时存在的有丝分裂型分析。咕咕叫麝猫。2014;60:277 - 84。
- 62.
Skippington E Barkman TJ Rice DW Palmer JD比较丝裂基因组学表明寄生虫的呼吸能力槲寄生尽管丧失了复杂的I和极端序列分歧,并且揭示了水平基因转移和基因组大小的显着变化。BMC植物BIOL。2017; 17:49。
- 63.
Wu Z, Sloan DB。线粒体基因组中完整染色体的存在和缺失的重组和种内多态性。遗传。2019;122:647-59。
- 64.
吴Z,切杂交效力,泰勒博士,斯隆DB。Agiosperm硅胶Noctiflora的大规模线粒体基因组通过整个染色体的增益或丧失而发展。Proc Natl Acad Sci U S A. 2015; 112:10185-91。
- 65.
Schwarz EN, Ruhlman TA, Weng M-L, Khiyami MA, Sabir JS, Hajarah NH, Alharbi NS, Rabah SO, Jansen RK。整个质体的核苷酸替换率揭示了凤蝶亚科的加速速率和与豆类亚科基因组特征的相关性。中国科学(d辑:地球科学)2017;
- 66.
KH, Li W-H,夏普总理。在植物线粒体、叶绿体和核dna中,核苷酸替代率差异很大。美国国家科学研究院1987;84:9054-8。
- 67.
邱玉玲,李磊,王斌,薛建勇,Hendry TA,李荣强,Brown JW,刘勇,Hudson GT,陈志东。从四个线粒体基因序列推断被子植物系统发育。中国科学:地球科学(英文版),2010;
- 68.
截短和分散rpl2和rps19在小麦线粒体中,伪基因与邻近的下游基因共转录。咕咕叫麝猫。2001;39:264 - 72。
- 69.
Atluri S,rampersadsn,粘性L.在线粒体和核中的S19核糖体蛋白的函数基因的保留超过6000万年。Mol Gen Genomics。2015; 290:2325-33。
- 70.
研究了线粒体与细胞核之间的基因转移。Adv Bot Res. 2012; 63:21-40。
- 71.
郭伟,朱安,范伟,Mower JP。蕨类植物的完整线粒体基因组Ophioglossum californicum和Psilotum无偿具有最大的细胞器内含子。新植醇。2017;213:391 - 403。
- 72.
董珊珊,赵超,陈飞,刘永强,张胜,吴华,张丽,刘永强。早花植物线粒体基因组的全基因组分析nymphaea colorata.是高度重复,低重组。BMC基因组学。2018;19:614。
- 73.
植物和光合微生物线粒体内含子的粘性L.。ADV BOT RES。2012; 63:155-86。
- 74。
周淑敏,春杰施安,王丹,吴永伟,刘淑敏,周天逸。裸子植物的线粒体基因组Cycas taitungensis.包含一个新颖的短散布元件家族,Bpu序列和丰富的RNA编辑位点。生物化学学报2008;25:603-15。
- 75。
关键词:RNA,编码岛,微卫星,重复序列卷柏moellendorffii线粒体:早期管胞植物线粒体dna重组频繁的根源。基因组生物学进展。2011;3:344-58。
- 76。
植物线粒体基因组进化可以用DNA修复机制来解释。基因组生物学进展。2013;5:1079-86。
- 77。
王胜,宋强,李胜,胡志东,宋超,黄华,刘玉玲菊花nankingense使用牛津纳米孔长的读物和菊科毒蛛的多样性和演化。基因。2018; 9:547。
- 78。
被子植物线粒体基因组的组织与变异。杂志。2007;129:6-13。
- 79.
陈志强。白藜芦醇科植物。1980;23:1-80。
- 80.
Schwarz EN, Ruhlman TA, Sabir JS, Hajrah NH, Alharbi NS, AL - malki AL, Bailey CD, Jansen RK。豆科植物质体基因组序列显示平行倒置和多重损失rps16在papilionoids。中国科学(d辑:地球科学)2015;
- 81.
Altschul SF, Madden TL, Schäffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ。gap BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸Res. 1997; 25:3389-402。
- 82.
Untergasser A, cutcuttache I, Koressaar T, Ye J, Faircloth BC, Remm M, Rozen SG。primer3 -新的功能和界面。核酸学报2012;40:e115。
- 83.
蒂利希M, Lehwark P, Pellizzer T, Ulbricht-Jones ES, Fischer A, Bock R, Greiner S. geseq -细胞器基因组的通用和准确注释。核酸学报2017;45:W6-W11。
- 84.
Richardson AO, Rice DW, Young GJ, Alverson AJ, Palmer JD。“石化”的线粒体基因组鹅掌楸tulipifera:祖先基因的内容和顺序,祖先的编辑位点,以及极低的突变率。BMC医学杂志。2013;>。
- 85.
Lowe TM,CHAN PP。Trnascan-SE在线:集成搜索和上下文以分析转移RNA基因。核酸RES。2016; 44:W54-7。
- 86.
Repbase中重复元素的标注、提交和筛选:RepbaseSubmitter和Censor。BMC生物信息学。2006;7:474。
- 87.
Park S, Ruhlman TA, Sabir JS, Mutwakil MH, Baeshen MN, Sabir MJ, Baeshen NA, Jansen RK。药用植物细胞器基因组的完整序列Rhazya一成不变(夹竹桃科)和跨越小行星的线粒体基因组进化的对比模式。BMC基因组学。2014;15:405。
- 88.
串联重复序列发现者:一个分析DNA序列的程序。核酸Res. 1999; 27:573-80。
- 89.
Katoh K,Misawa K. Kuma Ki。Miyata T Mafft:一种基于快速傅里叶变换核酸RES的快速多序列对齐的新方法。2002; 30:3059-66。
- 90.
jModelTest 2:更多的模型,新的启发式和并行计算。Nat方法。2012;9:772。
- 91.
Stamatakis a . RAxML版本8:系统发育分析和大系统发育后期分析的工具。生物信息学。2014;30:1312-3。
- 92.
基于CIPRES的大型系统发育树推断的科学门户。在:网关计算环境研讨会(GCE), 2010;2010.1 - 8页。Ieee。。
- 93.
Kimura M.通过核苷酸序列的比较研究估算基取代的进化率的简单方法。J Mol Evol。1980; 16:111-20。
- 94.
Kumar S,SteCher G,Tamura K. Mega7:用于更大数据集的分子进化遗传学分析版本7.0。mol Biol Evol。2016; 33:1870-4。
确认
我们感谢新墨西哥州立大学的C. Donovan Bailey教授提供的有丝分裂基因组序列银合欢trichandra在公开发行之前。我们还感谢德克萨斯大学奥斯汀分校的基因组测序和分析设施执行Illumina测序,德克萨斯大学奥斯汀分校的德克萨斯高级计算中心访问超级计算机,以及德克萨斯- ll作为代金证标本的存储库。这项研究已经作为海报在2019年7月27日至31日,美国亚利桑那州图森的《植物学》上展示。
资金
作者感谢沙特阿拉伯吉达阿卜杜勒阿齐兹国王大学(KAU)校长Abdulrahman O. Alyoubi教授的财政支持。资助方不参与研究设计、数据收集、数据分析、数据解释、手稿撰写或决定发表。
作者信息
从属关系
贡献
JSMS和RKJ设计和构思了研究;ISC进行了研究;ENS、TAR和RKJ收集样本和整理数据;ISC、MAK、NHH、MJS和SOR对数据进行分析和解释;ISC, RKJ和TAR撰写了手稿。所有作者阅读,修改,并批准了手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
四个完整的豆科有丝分裂基因组的装配信息。表S2。Fabaceae毒蛛重复序列综述。表S3。豆科物种线粒体基因组共享DNA (kb)的两两比较。表S4。豆科物种线粒体基因组的成对K2P距离。表S5。引物序列和有丝分裂基因组整理产生的扩增产物。表S6。比较基因组学和系统发育分析中使用的含有毒蛛的数量。表S7。包含在系统发生重建中的线粒体和体层基因。
附加文件2:图S1。
阅读覆盖四个完整的豆科有丝分裂基因组。图S2。有助于12个代表性的骨髓植物的促进型大小的因素。图S3。豆科12个有丝分裂基因组的基因含量。图S4。最大似然性文学rpl2和rps19.图S5。别变的cox2豆科有丝分裂基因组中的基因。图S6。豆科与全寄生物共享线粒体DNA的关系示意图Lophophytum(蛇菰科)。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
崔。,Schwarz, E.N., Ruhlman, T.A.et al。豆科线粒体基因组大小和含量的波动既有古代的,也有近代的。BMC植物BIOL.19,448(2019)。https://doi.org/10.1186/s12870-019-2064-8
收到了:
接受:
发表:
关键字
- 比较基因组学
- 基因损失
- 基因组大小
- 基因内区大小
- 重复