抽象的
背景
蓝莓具有很高的经济价值。大多数为新鲜市场选择的蓝莓品种都有一个吸引人的浅蓝色涂层或“开花”的果实,因为存在一个可见的重的表皮蜡层。这种蜡质层也可以作为防止水果干燥和变质的天然屏障。
结果
在这项研究中,我们试图鉴定其表达与蓝莓果实保护蜡涂层有关的基因,利用用于蜡质层的两个独特的种质群。我们从两种适应的兔子混合繁殖群体中膨胀了来自蜡质和非蜡状蓝莓后代的RNA('Nocturne'X T 300和'夜景'x US 1212),并产生了316.85亿RNA-SEQ读数。我们DE Novo组装了与其他公开的RNA-SEQ数据集成的数据集,并将组装修剪成91,861个蓝莓Unigene集合。所有ungenes都在功能上注释,导致79个基因可能与蜡积累有关。我们将蜡状和非蜡状后代的表达模式与使用的edger进行了比较并在T 300群体中鉴定了在T 300种群中的总体1125基因,并且在US 1212群中的2864个基因具有至少两倍的表达差异。通过RT-QPCR实验验证几种基因的差异表达,候选基因,FatB编码酰基 - 酰基载体 - 蛋白质]水解酶的水解酶,其表达与我们群体中蜡质涂层的偏析密切相关。该基因在蜡质中比两种群体的非蜡质植物在蜡质中的5倍较高水平。我们从每种群体的三种蜡状植物中扩增并测序该基因的cDNA,但不能从每种群体测试的三种非蜡状植物中扩增cDNA。我们对齐了Vaccinium将FATB蛋白质序列推导到其他植物物种的FATB蛋白序列。在PF01643结构域内,其给予FATB其催化功能,80.08%的氨基酸在蓝莓和氨基酸之间是相同的或具有保守的替代品Cucumis梅洛序列(XP_008467164)。然后我们放大和测序了很大一部分FatB基因本身来自两个种群的蜡质和非蜡质个体。cDNA和gDNA序列比对结果表明,蓝莓具有较好的抗氧化活性FatB基因由6个外显子和5个内含子组成。虽然我们没有通过两个非常大的内含子进行测序,但比较外显子序列发现蜡质植物和非蜡质植物之间没有显著的序列差异。这表明另一个基因,调节或影响FatB表达,必须在人群中隔离。
结论
本研究有助于进一步了解蓝莓表皮蜡的生物合成。此外,蓝莓单基因的收集有助于对即将到来的染色体水平的蓝莓基因组进行功能注释。
背景
蓝莓栽培正在全世界迅速扩展。美国最大的蓝莓生产国生产,从2010年到2015年增加了33%,从2005年到2015年增加了119%(美国农业部 - 国家农业统计服务(USDA-NASS))。近年来,全球生产也在增长,从2009年到2014年增长了58%,现在超过525,000公吨(联合国,粮食和农业组织)。Blueberries的消费量也有所增加,可能导致了对花青素的许多健康益处的更大意识。蓝莓已被证明是所有新鲜水果和蔬菜的花青素和抗氧化剂的最富有的来源之一1].在美国,人均蓝莓的年消费量从0.6磅上升。2000年达到1.5磅。2010年(2].
有三种主要种类的蓝莓种植,Tellaploid高杂蓝莓(Vaccinium corymbosum和杂交种),六倍百倍的兔子蓝莓(诉virgatum)和野生四倍体Lowbusburber(V. angustifolium.).蓝莓育种工作的重点是广泛适应气候、延长季节、抗病虫害、耐机械处理和高品质的性状[3.].水果的品质包括许多属性,如好的味道,大的尺寸,坚固,浅蓝色,等等。蓝莓的颜色是由于果实内的花青素和果实外部的蜡质角质层的存在。这种蜡涂层或“bloom”提供了理想的浅蓝色。除了在视觉上比黑色水果更吸引人之外,这种蜡状的表皮还能延缓水果在贮藏过程中的干燥和变质[4].
陆地植物的角质层是一层疏水层,覆盖着包括果实在内的所有器官的气相表面,由角质层和角质层蜡组成。研究认为,角质层是抵御非气孔水分流失的第一道保护屏障,可以降低生物/非生物胁迫的影响,并改变光的反射率[5].该角质层由三层组成:由Cutin,肠蜡和多糖组成的最内层或内在层(CL);皮层适当(CP)由迹象蜡,肠蜡和cutin组成;最后,最外层由奇迹蜡(EW)组成[6].角质层蜡是由长链脂肪族化合物、三萜和固醇、类黄酮等其他代谢物组成。脂肪酰基蜡是由vlc -脂肪酸(VLCFAs)通过两种不同的生物合成途径获得的,一种是生成醇的途径,生成伯醇和烷基酯,另一种是生成烷烃的途径,生成醛、烷烃、仲醇和酮[6].
利用气相色谱-质谱联用技术和扫描电子显微镜对蓝莓果实表皮蜡的化学成分进行了表征。三萜和β-二酮是主要化合物,分别占总蜡的64.2和16.4% [7].对小麦和大麦的研究认为,三萜主要在表皮层和表皮层形成无定形蜡质,而β-二酮主要在表皮层形成结晶蜡质,呈现明显的白霜状[8,9].
在本研究中,我们试图利用两个独特的蓝莓种质群体来鉴定与蓝莓果实蜡质保护涂层相关的基因表达。这些群体是我们蓝莓育种计划(北方适应兔眼杂交育种群体)中由于有无蜡涂层而明显分离的杂交组合的结果。RNA测序是在有蜡涂层的子代和没有蜡涂层的子代的大体积RNA上进行的。在两个群体之间差异表达的基因中,选择几个候选基因,并在组成原始群体的单个植株上通过实时qPCR检测它们的表达。从这些分析中,我们确定了最好的候选基因,它们在两个群体中都有差异表达,并且与蜡的生物合成或运输有关。在这项研究中,我们还生成了迄今为止最全面的蓝莓转录组组装,这将使整个蓝莓研究人员受益,并便于对蓝莓基因组的注释。
结果
蓝莓转录组测序和组装
试图鉴定与蓝莓果实上的保护蜡涂层相关的基因,使用了两个独特的种质种群。这些人群受到我们北方兔饲料计划中的十字架产生的,该程序被视觉隔离,用于蜡质涂层的存在/不存在(图。1).表中描述了罂粟涂层的抗涂层的分离比1.RNA-seq是从有蜡涂层的子代和没有蜡涂层的子代的膨大果实组织中提取的RNA。四个RNA-seq文库(来自两个种群的蜡质和非蜡质块)共产生了3.1685亿对端100 bp的Illumina reads(附加文件)1:表S1)。
除了从这项研究中产生的阅读,从NCBI的SRA中还下载了先前从蓝莓中产生的3.767亿对端和4.854亿单端Illumina阅读。经过质量修剪后,这些90.77英镑的干净阅读被映射到最新版本的蓝莓基因组组装可用(Robert Reid, UNC,和Allan Brown, IITA,个人通信)。在所有分析的图书馆中,我们研究的图书馆拥有最高的映射率(平均91%)。大多数其他库也有很好的映射率(平均74%),除了库SRR1187674,它的映射率太低,不能被认为是蓝莓转录组数据(附加文件1:表S2)。排除该文库后,其余87.34 Gbp的clean RNA-seq reads用于转录组组装。
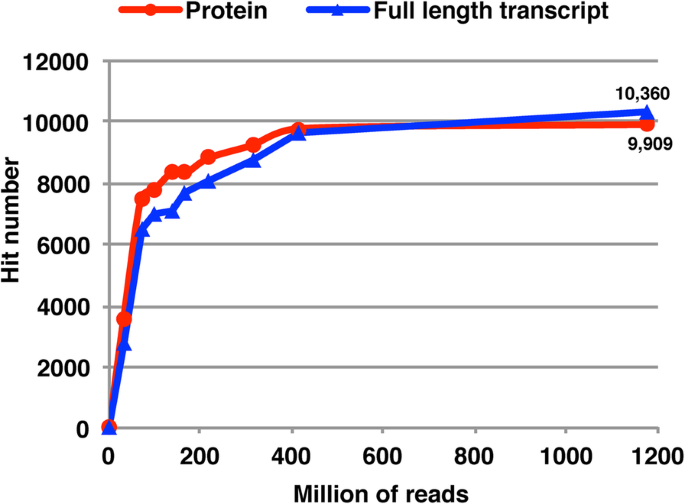
采用阶梯从头组装策略来评估该数据集是否足以提供一个全面的蓝莓转录组组装。随机选择一系列不同的数百万个读取数据,并根据每个数据集进行从头组装。在全长转录本表征和对非模式生物的蛋白质命中方面,随着读数的增加,更多的转录本被捕获,直到达到约4亿个读数的平台(图)。2).我们还使用454序列比较了先前由我们的实验室组装的Contig [10,11:结果,在37,524(86.67%)454个contigs中,有32,794个对新的de novo程序集(附加文件1:表S3)。因此,我们得出结论,本研究中用于蓝莓转录组装配的11亿reads的总干净数据集足以给出饱和装配。
我们还基于此读取数据集的基于参考组件,装配工作流程如附加文件所示1:图S1。然而,当映射回到组装的转录物时,与DE Novo版本相比,基于参考的组件具有更低的映射率(附加文件1:表S2)。这可能是由于该版本的参考基因组不完整和/或参考基因组注释中的错误。因此,我们选择在我们的研究中使用新版本的转录本组装,而只使用基于参考的组装作为转录本可信度的支持证据。
蓝莓Unigene收集和功能注释
总共生成251,974个三位一体的“基因”和352,293个三位一体的成绩单(初始Rawo Novo集会(附加文件)1:表S4)。该程序集的平均读回映射率为77.4%(附加文件1:表S2,第4列的平均值)。组装的转录物在Swissprot数据库中达到9909个蛋白质记录[12]并捕获了10,360个全长蓝莓基因(图。2).这些结果表明大会质量良好。然而,在通过TransDecoder识别编码区域之后,8037转录物具有完全相同的编码序列。我们通过选择每个三位一体的基因的最长预测的CDS序列(附加文件)来减少这种冗余1:图S2)。我们进一步应用了CD-BIT [13]抛光所选择的组件,导致91,861候选三位一体的基因'。然后我们使用tgicl [14再次检查这个候选人集合。仅发现460个集群,冗余度较低。因此,91,861个候选Trinity‘基因’集被认为是一个良好的、非冗余的蓝莓unigene数据集(表2).
所有unigenes在公共蛋白数据库SwissProt、TrEMBL、NCBI-Nr数据库和refPlant中进行功能注释。来自途径数据库PlantCyc和KEGG的酶记录也用于基因功能注释。总共有56696个unigenes(61.72%)被至少一次公共数据库的命中注释。NCBI-Nr数据库注释的unigenes最多。在所有refPlant注释的物种中,葡萄(葡萄)导致蓝莓Unigenes最多的命中(附加文件1:图S3)。来自847个PlantCyc代谢途径的14231条酶记录被分配到蓝莓unigene数据集。
蓝莓unigenes作为潜在的蜡相关蛋白质被注释
为了更好地了解植物表皮蜡沉积的遗传基础,我们查阅了相关文献47篇1:表S5)。From these sources, we collected 112 protein records encoded by 88 genes reported to be related to plant cuticular wax accumulation and incorporated 447 enzymes from the PlantCyc cuticular wax biosynthesis pathway, PWY-282, to establish our own wax database, which we call ‘waxybase’. We then applied stringent searching criteria between waxybase and the blueberry unigene data set for annotation. As a result, 79 blueberry genes were annotated by 46 waxybase proteins (Table3.).拟南芥和番茄(茄属植物lycopersicum)提供了该注释中最多的引用记录。这些与蜡相关的蓝莓基因被注释为各种关键酶,包括生物合成基因CER1/3/6/9/10、调控因子MYB41/106和蜡分泌相关基因ABCG11/12/32(附加文件1:表S6)。
与有粘面蜡积聚有关的差异表达基因(DEGS)
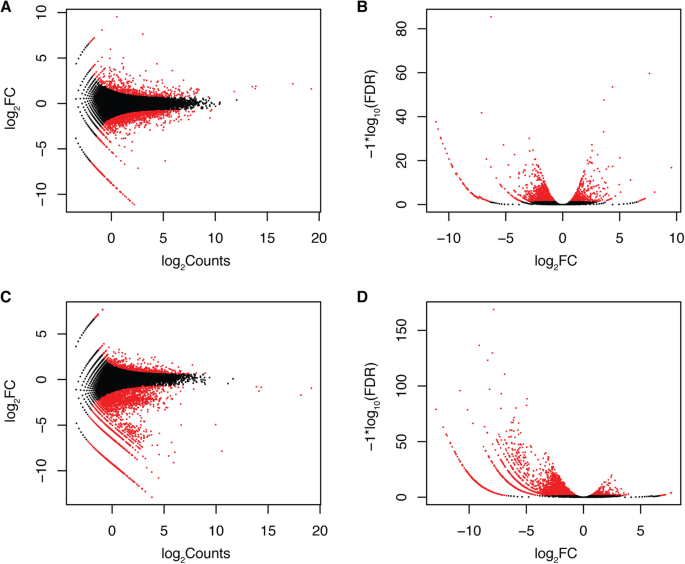
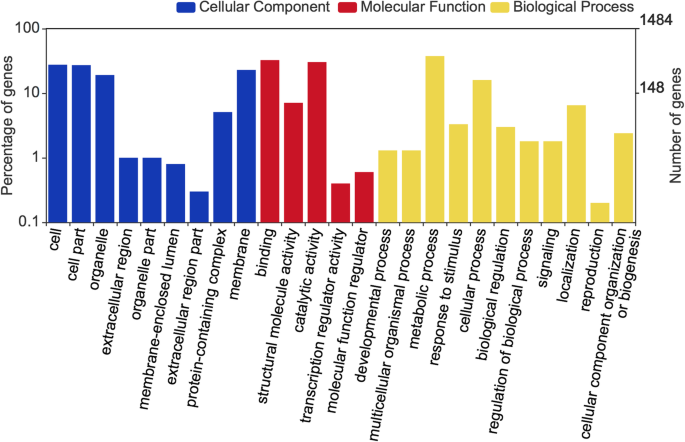
为了发现在我们独特的蓝莓群体中观察到的与果实上保护性蜡涂层分离相关的表达水平的基因,使用edgeR [15].我们在“Nocturne”x T 300群体中鉴定了1125个基因,在“Nocturne”x US 1212群体中鉴定了2864个基因,它们在蜡质和非蜡质文库中的表达水平至少有2倍的差异(补充文件)2:表S7和附加文件3.:表S8)。有趣的是,与两个群体的蜡质库相比,更多的基因在非蜡质蓝莓库中有更高的表达水平(图2)。3.).共有3333只(96.47%)发现患有NCBI NR数据库的命中,然后通过BLAST2GO映射到基因本体(GO)条目(图。4).25种氧化石墨烯功能类别受到热捧,包括“膜”功能和“含蛋白复合物”功能。其中4个被鉴定的deg也在蜡基上进行了注释,并在其他基因中选择了这些基因(如下所述)进行RT-qPCR验证。
对已鉴定的DEGs和其他已知参与蜡生物合成的基因进行表达分析
我们首先将edgeR预测的表达结果与功能注释结果结合,筛选出26个基因进行RT-qPCR验证(Additional file)4:表S9)。在这26个基因中,有4个基因预测了两个群体中蜡质块比非蜡质块更高的表达水平(Log)2Fc = 2, fdr = 0.001)。另外四个基因(总共85个)预测了非蜡质块的更高表达水平,而不是两种群体的蜡质块(日志2Fc = 2, fdr = 0.001)。此外,我们选择了七个顶级12基因的“夜曲”x T 300人口和四个基因的“夜曲”x我们1212人口预测上级表达的蜡质体积比non-waxy大部分只有一个人口(日志2Fc = 1, fdr = 0.005)。最后,在选择的RT-qPCR基因中,我们纳入了“Nocturne”x US 1212种群蜡质体中预测表达水平高于非蜡质体的仅有的两个基因,并在蜡质数据库(Log)中进行了比对2Fc = 1, fdr = 0.005)。我们还选择了在非蜡质体中预测表达水平高于“Nocturne”x US 1212种群蜡质体的前5个基因(总共8个),并在蜡质数据库(Log)中取得了成功2Fc = 1, fdr = 0.005)。其中一种基因在非蜡质体中的表达水平也被预测高于“Nocturne”x T 300群体的蜡质体。在' Nocturne ' x T 300群体中,没有预测到的其他差异表达基因与蜡基(Log2Fc = 1, fdr = 0.005)。
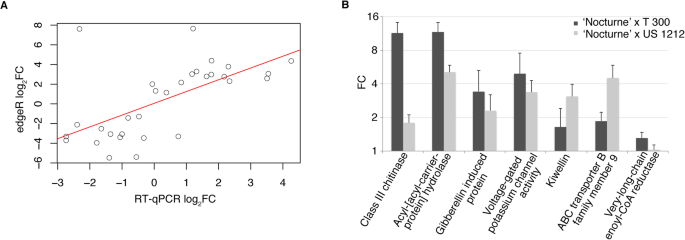
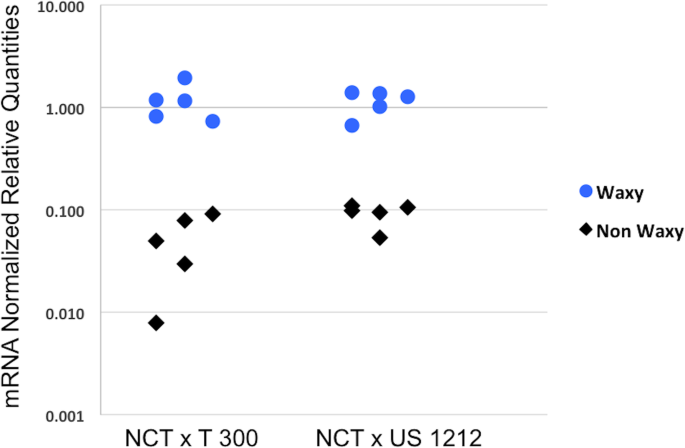
总体而言,差异表达预测结果与RT-qPCR结果吻合较好(图2)。5a)拟合系数为0.62的线性回归模型(Pearson相关,t = 4.27, df = 29,p值= 1.91 e-04)。在已验证的差异表达基因中,有两个基因可能与蜡的生物合成有关;一个与酰基[酰基载体蛋白]水解酶同源,另一个与hxxxd型酰基转移酶同源。从RT-qPCR结果,与同源基因,酰基-酰基载体蛋白质水解酶表达在一个11.68折和5.07折更高水平更高,平均而言,在个人的植物组成比个体植物蜡状大块组成“夜曲”non-waxy散货的x T 300和“夜曲”我们1212人口,分别(无花果。5b)。与同源性基因HXXXD-type acyl-transferase在0.36折低水平表达的蜡质大部分植物“夜曲”x T 300人口和2.26折更高层次的蜡质大部分植物“夜曲”x 1212人口,使它成为不可能的候选人蜡质基因在我们的人口(附加文件4:表S9)。在无花果。6,我们展示了来自两个群体的5个蜡质块和5个非蜡质块果实组织中的酰基-[酰基载体蛋白]水解酶mRNA(来自RT-qPCR)水平。在这20种植物中,蜡质植物的表达量均较高FatB而非蜡质植物则没有。所有单株的表达水平都有一定的范围(共38个;10个蜡质和7个非蜡质来自“Nocturne”x T 300种群,13个蜡质和8个非蜡质来自“Nocturne”x US 1212种群),它们显示在附加文件中的箱形图中1:图S4。
接下来,我们从文献中选择了17个已知与蜡积累有关的主要基因[6],所有这些都包含在我们的蜡质酶中,用于RT-QPCR分析。无论它们是否预计基于RNA-SEQ数据,都被选择而选择它们。RT-QPCR结果表明,这些基因中的任何一种群体的蜡质和非蜡质块之间都不会显着表达(附加文件5:表S10)。
蓝莓序列分析FatB互补脱氧核糖核酸和gDNA
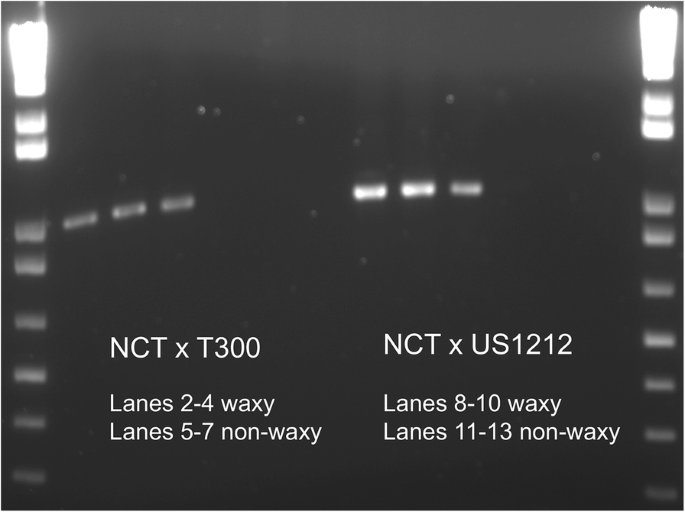
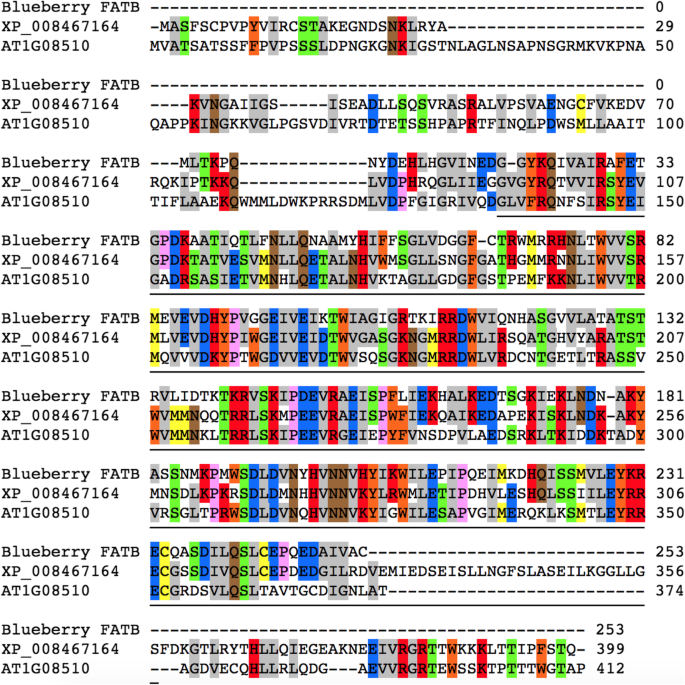
我们试图扩增差异表达的酰基-[酰基载体蛋白]水解酶同源物(也称为FATB)的cDNA,通过在转录序列末端附近设计引物,从两个群体中分别选取3个蜡质植物(该基因表达水平最高)和3个非蜡质植物(该基因表达水平最低)。扩增仅在蜡质植物中成功,推测是因为该基因在非蜡质植物中的表达过低(图。7).将6株蜡质植物的cDNA扩增产物不经克隆直接测序,并进行比较。在它们的序列中没有发现差异。然后进行了多次序列比对Vaccinium推导出脂肪b蛋白序列(253个氨基酸)Cucumis梅洛FATB (XP_008467164;注释为棕榈酰酰基载体蛋白硫酯酶,来自NCBI BLASTP搜索的最佳命中)蛋白序列(图。8来自其他几种物种的Fatb蛋白序列(附加文件1:图S5)。通过加入下划线显示的PF01643结构域(酰亚胺 - 乙酰丙基蛋白质]硫代酯酶,ACY-ACP-TE结构域)。8,通过水解脂肪酸中的酰基,使该蛋白具有终止脂肪酸酰基延伸的催化功能。在该区域内,80.08%的氨基酸在蓝莓和蓝莓之间完全相同或有保守替换Cucumis梅洛序列。
因为我们不能扩增从我们测试的非蜡质植物的cDNA序列,我们也试图测序FatB基因本身。根据蓝莓基因组设计引物,对两个群体的蜡质植物和非蜡质植物基因组DNA片段进行测序。我们捕获了该基因的所有外显子区域,但没有对两个非常大的内含子进行全部测序。将cDNA序列与gDNA序列比对后,我们确定蓝莓FatB基因由6个外显子和5个内含子组成1:图S6)。我们比较了FatB从两个种群的蜡质植物和非蜡质植物的序列中,只发现一些退化的核苷酸变异。
讨论
对于像蓝莓这样的多年生灌木,培育一个新的品种可能需要9到20年的时间[16].基因组辅助育种在一些主要作物上已被证明是有效的,特别是在世代时间较长的多年生果树和灌木上尤其有用。基因组资源在蓝莓中变得可用。二倍体的遗传连锁图[17]和商业四倍体[18蓝莓已经被构建,但需要进一步的饱和。流式细胞术估计蓝莓基因组为~ 600 Mbp [19].一种二倍体蓝莓(诉corymbosum) accession ' W85-20 '已经测序并使用罗氏454 Newbler汇编器组装到长度为358 Mbp(15,129个支架)[20.]并使用Illumina RNA-SEQ结合454序列数据进行注释,导致〜60,000个蓝莓基因模型[21].已经公布了几种蓝莓转录组研究,重点是揭示冷驯化的潜在机制[10,22,23,果实成熟[21,24,以及水果抗氧化剂含量[25].然而,迄今为止,仍然没有公开可用的专用基因注释的染色体水平蓝莓基因组。在本研究中,除了来自其他研究的RNA-SEQ数据外,我们还报告了使用我们自己的RNA-SEQ数据组装的91,861个蓝莓Unigenes的集合[21,25].该单基因集将为蓝莓基因组序列注释提供高质量的证据。
根据最近的一项调查报告,蓝莓育种特质优先事项,坚定,保质期和外表是行业最重要的水果质量特征之一[16].这种蜡质涂层使蓝莓果实呈现出诱人的浅蓝色“开花”。最近也有报道称,角质层蜡的各种成分与果实在贮藏过程中的失重和软化高度相关(有些是正相关,有些是负相关)[26].在我们的研究中,我们使用了两种适应的兔子杂交繁殖群体,这些植物共享普通父母的“夜曲”,这是一种六倍黑猩猩的品种,浆果上没有可见的蜡质涂层。群体对水果上的蜡质涂层隔离,这使水果为浅蓝色尘土飞扬的颜色,而不是黑色。通过组合膨胀分析分析和RNA-SEQ的概念,我们将基因表达谱与两种群体的蜡质和非蜡质块的基因表达谱进行比较,并鉴定了表达水平至少两倍差异的差异表达基因(DEGS)。然后通过RT-QPCR验证来自RNA-SEQ分析的蜡质涂层的最佳候选基因的表达。
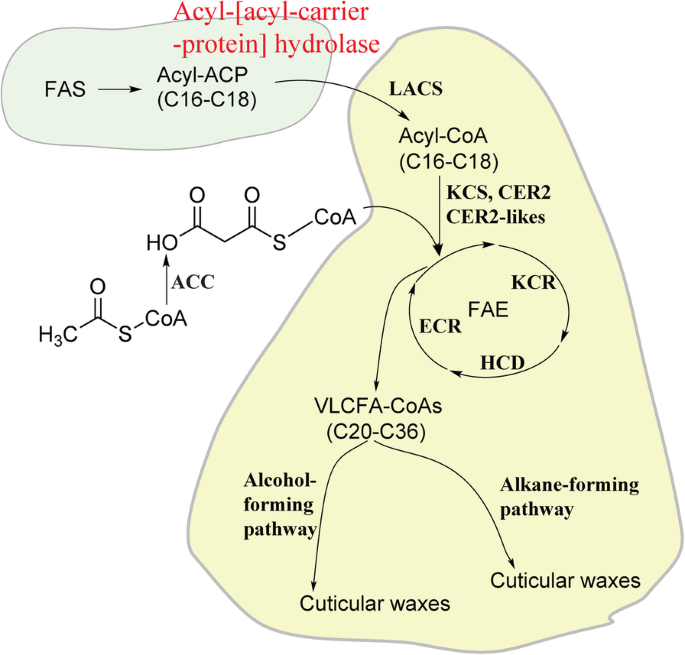
从这个工作,一个优秀的候选基因从列表中出现RNA-seq度的分析,及其微分表达式被RT-qPCR验证个体组成的植物钡的表达水平平均高出11.68和5.07倍,比non-waxy蜡状大块大块的人群。这是唯一出现的与蜡生物合成相关的DEG,表达量超过一个对数2两种种群中蜡质块的含量都比非蜡质块高两倍。该基因是蓝莓中酰基-[酰基载体蛋白]水解酶的同源物,也称为FatB基因拟南芥.在玉米中,插入FatB基因(GRMZM5G829544)已经被证明会导致种子中棕榈酸(16:0)含量的减少[27,28].在拟南芥,T-DNA插入FatB基因(AT1G08510)已经被证明会导致不同组织类型中棕榈酸酯(16:0)和硬脂酸酯(18:0)水平的降低,导致生长速度的降低。此外,FatB基因已经涉及为蜡生物合成提供脂肪酸,因为T-DNA插入/敲除突变拟南芥结果突变体叶片和茎中蜡质总量分别减少了20%和50% [29].这种突变导致非常长链脂肪酸(VLCFA)生物合成的脂肪酸供应严重减少。数字9展示如何将产物从酰基 - [酰基载体 - 蛋白质]水解酶反应漏斗中进入VLCFA生物合成途径,这又需要蜡生物合成。VLCFA通过醇形成途径或烷烃形成途径来修饰,以产生有切割蜡的主要成分[30.].
我们对蓝莓的cDNA进行了扩增和测序FatB基因,但不能扩增从三个蜡质植物测试,从三个非蜡质植物的cDNA,可能是因为它在这些植物的低表达。然后我们放大和测序了很大一部分FatB基因本身来自两个种群的蜡质和非蜡质个体。cDNA和gDNA序列比对结果表明,蓝莓具有较好的抗氧化活性FatB基因由6个外显子和5个内含子组成。虽然我们没有对两个非常大的内含子进行测序,但对外显子序列的比较发现,蜡质植物和非蜡质植物之间没有显著的序列差异,这表明有另一个基因在调控或以某种方式影响着蜡质植物FatB表达,必须在人群中隔离。
在我们的群体中,果实上蜡质涂层的分离表明,这种特性是定量的,因为蜡质植物似乎有不同的蜡水平。因此,蜡涂层的存在应该由至少几个基因决定,尽管一个基因可能负责大部分的基因型差异。我们的研究结果表明,蓝莓的表达FatB基因与果实蜡开花密切相关。然而,我们在蜡质和非蜡质植物中对该基因的测序并没有发现与非蜡质表型相关的基因突变。如果能找到控制FatB如果证明它对该蓝莓和其他蓝莓育种群体的果蜡分离起作用,那么它可以作为标记辅助选择的一种标记。
这项研究表明FatB该基因与蓝莓的蜡质表型有关,该基因与蜡质涂层的关系可在蓝莓的其他作图和亲缘群体中检验Vaccinium比如蔓越莓和越橘。目前,我们正在对蓝莓二倍体群体的果实颜色性状(从黑色到浅蓝色)进行定位,并希望确定该基因或另一个与蜡质相关的基因是否与二倍体群体中该性状的主要QTL相重合。此外,本研究还构建了蓝莓的转录组,为新出现的蓝莓基因组的功能注释提供了高质量的基因表达证据。
结论
在本研究中,我们利用两个北方适应的兔眼杂交育种群体分离的果实上的蜡涂层。我们进行了大量分离分析结合RNA-seq分析。我们获得了一组91,861个蓝莓ungenes,为蓝莓基因组序列注释提供了表达证据。利用此组装,我们探索了与果实蜡质涂层相关的DEGs,并鉴定了一个基因,FatB,其表达与弹性蜡质层的存在密切相关。
方法
植物材料
用于这项研究的两个北方兔眼繁殖种群是由' Nocturne ' [31[T 300(33株)和' Nocturne ' x US 1212(36株)。' Nocturne '和US 1212是复杂的混种六倍体,主要由诉virgatum和诉constablaei贡献较少的诉corymbosum,诉darrowii,诉tenellum.'Nocturne'和美国1212起源于新泽西州Chatsworth的USDA-ARS繁殖计划。T 300是100%诉virgatum.t300源于美国农业部和佐治亚大学之间的一个合作育种项目。夜曲(Nocturne)是黑色果实,很少或不含蜡。这两个种群由于果实上有无蜡涂层而明显分离。在2014年夏天,根据果实上肉眼可见的蜡量,植物被给予了0到5的等级。0分或1分表示没有或很少蜡,2分或3分表示存在中等数量的蜡,4分或5分表示存在重蜡涂层。对于散装的准备(下文讨论),只有评分为0的植物被用于非蜡散装;植物有二十种> 2 were used in the waxy bulks. Ripe fruit from each plant was flash frozen in liquid nitrogen and stored at − 80 °C for future RNA extractions (described below). The segregation ratios for this trait in both populations are described in Table1.
RNA提取,cDNA合成和质量检测
对于RNA-seq库,RNA是从体积较大的组织样本中提取的。“Nocturne”x T 300群体中有10个蜡质个体和9个非蜡质个体,而“Nocturne”x US 1212群体中有13个蜡质个体和10个非蜡质个体。为了制作每个大块,从每种类型的每个个体中提取等量的果实组织(0.5 g)。然后如前所述提取RNA [10].提取后,用NanoDrop ND-1000 (NanoDrop Technologies, Wilmington, DE, USA)检测RNA浓度和质量。此外,用1%琼脂糖凝胶用溴化乙锭染色检查质量。
对于实时PCR,从包含两种群体的蜡质和非蜡质小块的个体中提取RNA。使用四克果组织,并使用用于块的相同程序提取RNA [10].在纳米二-1000上测量RNA浓度和纯度。在进一步分析中,仅使用含有A260 / A280比率的RNA样品1.9和2.1和A260 / A230的比例。为了验证完整性,我们扩增了泛素羧基 - 末端水解酶基因的5'和3'区域的两个101bp长cDNA片段(UBP14)通过QPCR穿过CDNA样本。片段分别是来自cDNA的3'末端的1769和348bp。3':5'放大比例UBP14使用比较CQ方法从所有样品计算cDNA片段[32].所有比率在1.28-3.06的范围内(2.44±0.89;平均值±SD)。只有在比率> 4.4时,才会认为RNA质量不足[33].为了去除污染基因组DNA (gDNA),在合成cDNA之前,用TURBO™DNase I (Life Technologies, USA)处理RNA提取物,如前所述[22].在cDNA合成之后,如前所述,测试CDNA样品的GDNA污染[22].在GDNA污染的测试中,1140bp条带没有从任何样品中扩增。因为cDNA样本符合RNA质量的标准,并且没有GDNA污染,因此判断它们适用于QPCR分析。
排序和数据整理
根据Illumina HiSeq2500制造商的说明,从上述两个蓝莓群体的蜡质和非蜡质块中构建了两个RNA-seq文库。位于北卡罗来纳州坎纳波利斯的大卫·h·默多克研究所(David H. Murdock Research Institute)对图书馆进行了准备和测序。产生100 bp的配对端序列。原始读取是基于两个标准进行裁剪的。首先,丢弃5 '端10个核苷酸和3 '端5个核苷酸,去除末端的残基接头序列和低质量序列。在此之后,超过94%的剩余碱基对的每个碱基质量评分为> 30。其次,任何包含10个以上的模糊核苷酸(在85个bp中)的阅读被丢弃。
所有2016年前发布的蓝莓RNA-seq数据均来自美国国家生物技术信息中心(NCBI)短读档案(SRA),包括登录号为SRP039977、SRP039971和SRA046311的图书馆。下载的数据使用上述相同的标准进行质量调整。阅读质量分数统计由FastQC提供[34].
从头组装和基于参考的组装
使用本研究中的所有RNA-SEQ数据和从SRA下载的RNA-SEQ数据进行DE Novo蓝莓转录组组件。基于来自所有数据的不同数百万的随机选择的读取进行梯形组件。所有组件都使用Trinity进行[35,参数设置如下:——KMER_SIZE = 25,−-normalize_reads,−-normalize_max_read_cov = 60。
此外,参照导向组件使用TopHat-Cufflinks协议执行[36].使用TopHat将所有RNA-seq数据与蓝莓参考基因组进行比对,生成BAM文件。使用SAMtools对所有BAM文件进行排序[37].然后,Cufflinks使用经过排序的BAM文件进行转录。对于每个库,Cufflink生成一个GTF文件。然后使用CUFFMERGE将所有的GTF文件合并成一个单独的GTF文件。
采用两种不同的方法来评估每个装配件的质量。首先,使用TopHat将干净的reads映射回参考基因组,从而评估RNA-seq读取表示[38]并将清洁映射回到组件,包括基于参考和de novo版本,使用bowtie [39].其次,通过BLASTN到蓝莓基因组注释草案(Robert Reid, UNC, and Allan Brown, IITA, personal communication)和BLASTP到SwissProt/Trembl [12)记录的蛋白质。从BLASTN和BLASTP的搜索结果中选择最佳的比对结果。
基于参考的组装转录本也与使用BLASTN的从头组装转录本对齐。与身份分数> 80、查询长度> 80%和被试长度> 80比对的结果被标记为高置信度成绩单
Unigene分配和功能注释
使用TransDecoder进一步识别组装的转录本中的编码区域[40].orf使用BLASTP预测并映射回SwissProt蛋白数据库,使用HMMscan映射回Pfam域数据库[41].
为了消除装配体内部的冗余,对每个基因选择预测CDS最长的转录本,并丢弃预测CDS < 200个碱基的转录本。此外,CD-HIT [13使用默认参数(2017年5月1日构建的4.7版本)来消除冗余的文本。最后,tiger基因指数聚类工具(TGICL) [14]用于双重检查冗余(最小重叠长度= 120,重叠的最小百分数= 90)。
NCBI非冗余蛋白序列数据库(Nr)的蛋白记录,SwissProt/Trembl, PlantCyc [42]和NCBI RefSeq [43[release plant (refPlant)下载到我们的本地服务器并格式化到蛋白质数据库中。在功能注释方面,针对每个数据库对unigene推导的蛋白序列进行BLASTP注释。根据以下条件过滤得到的命中结果:身份> = 40,命中分数> =60,命中长度> =查询序列长度的一半。BlastKOALA [44] web服务器用于KEGG子数据库“genus_eukaryotes”。pep”注释。
差异表达转录本的鉴定
对于基于对准的丰富评估,使用Bowtie将来自四种不同RNA-SEQ库中的每一个(从两个人群的蜡状和非蜡质块的蜡块)的清洁读回我们的蓝莓Unigene数据集。然后通过预期最大化(RSEM)方法使用RNA-SEQ评估表达丰度[45]对于每个图书馆。使用Edger包识别差异表达水平[15].对于无对齐表达式量化,Salmon [46]是基于两相干涉程序进行的。
Waxybase建设
基于文献产生了与植物中蜡积累有关的基因列表。从植物沸石中检索这些基因的蛋白质序列[47或从NCBI。植物表皮蜡生物合成途径PWY-282的序列直接从PlantCyc下载[42].利用BLASTP来确定我们的蜡数据库(wax base)和蓝莓unigene数据集之间的最佳匹配。筛选标准:鉴定> 70,比对>蛋白序列长度70%,比对评分> 100。对于那些与蓝莓序列没有很好的同源性的基因,我们转而寻找葡萄同源性。
实时qPCR引物设计
NCBI BLAST软件用于测试所有PCR引物的特异性。用于实时QPCR的引物利用60±2°C,PCR扩增子长度为65-100bp的PCR扩增子长度为20-23个核苷酸的标准,GC含量为40-60%。从Mfold版本3.4软件预测了扩增子的二级结构(最小自由能量的默认设置,50 mm NA+, 3 mM Mg2+,退火温度60°C) [48].所选择的引物使扩增子具有最小的二级结构以及不会干扰退火的熔化温度。综合DNA技术公司(Coralville, IA, USA)合成了引物。
实时QPCR实验
PCR反应在IQ5 (Bio-Rad, Hercules, CA, USA)热循环器中使用iQ™SYBR®Green Supermix进行。以稀释后的cDNA为模板,每条引物为0.150 μM,总反应体积为20 μl。所有pcr均采用以下热剖面:聚合酶激活(95℃3 min),扩增和定量循环重复40次(95℃30 s, 60℃1 min)。通过熔融曲线分析检查引物对的特异性,扩增图如前所述分析[22].归一化相对量(NRQ)的确定如Hellemans等[49].每个引物对(E)的实时PCR扩增总平均效率由单个扩增图的指数阶段确定。为了计算效率,eq. (1 + E) =10坡使用LinReg软件。3 - 5个荧光数据点,R2 ≥ 0.998 defined the linear regression lines [50,51].使用先前鉴定的两个稳定表达的蓝莓参考基因进行归一化:UBC28和Vc4g26410[51]。为了确认目前材料中引用的参考的稳定表达,我们计算了两种群体的每个蜡质和非蜡质组的NRQ(平均值±SD)的平均值:(1)'夜曲的X T 300人口,UBC28蜡质植物= 1.16±0.29,非蜡状= 1.14±0.15,Vc4g26410蜡状植物= 0.91±0.22,non-waxy = 0.89±0.10;(2)“Nocturne”x US 1212 population,UBC28蜡厂= 1.19±0.18,非蜡状= 1.30±0.23,Vc4g26410蜡质植物= 0.86±0.14,非蜡状= 0.79±0.14。
最佳候选基因的cDNA和基因组DNA测序
人们试图扩增一个与蜡涂层相关的基因的近全长cDNAFatB基因编码酰基-[酰基载体蛋白水解酶]),从三个蜡质和三个非蜡质植物的每个种群,包括在原始块。根据供方的指示,使用SuperScriptIII逆转录酶,用oligo-dT12-18 (Life Technologies, USA)引物合成互补dna。将cdna稀释至50 μl。根据组装好的转录本序列和基因组注释信息设计PCR引物[20.](正向引物CatGctttttcacGTTGCAGAT;反向引物CCGTCTCTCCTTGGGATGATGA))。PCR反应体积为20μL含有1x Promega(Madison,Wi)GotaQ Flexi缓冲液,3 mm MgCl2Promega GoTaq Flexi DNA聚合酶0.5 units, cDNA 1 μL。在Bio-Rad (Hercules, CA) T100热循环器中进行扩增,初始变性值为95°5分钟,然后40个变性循环(92°, 40秒),退火(60秒°,30s),延伸(72°60岁),72岁时最后一次延长°10分钟。
放大FatB基因测序,如前所述,首次提取总基因组DNA [52研究结果表明,在原始种群中,每个种群中分别有3种蜡质植物和3种非蜡质植物。将FATB cDNA序列与蓝莓和蔓越莓基因组进行比对,建立基因模型,设计PCR引物。PCR反应体积为20 μL,含1x Promega (Madison, WI) GoTaq Flexi Buffer、3 mM MgCl2、0.2 mM dNTP、0.1 μM正反引物、0.5 units Promega GoTaq Flexi DNA聚合酶和25 ng基因组DNA。在Bio-Rad (Hercules, CA) T100热循环器中进行扩增,初始变性95°5 min,然后变性(92°,40s)、退火(60°,60s)、延伸(72°,120 s) 40个循环,最后72°延伸10 min。
扩增产物使用Zymoclean凝胶DNA回收试剂盒(Zymo Research, Irvine, CA)进行纯化,根据制造商的协议使用ABI Big Dye Terminator v3.1循环测序试剂盒(Applied Biosystems, Foster City, CA)进行直接测序,并在Applied Biosystems 3500基因分析仪上运行。
序列组装的FatB基因
原始测序读数是质量修整,然后通过引物的引物模板位置的顺序锚定,从而引用蓝莓基因组序列。锚定读数之间的重叠由BLASTN确定。通过Clustalw执行多个序列对齐[53来识别可能的变化。利用BLASTN将cDNA序列与组装好的gDNA序列进行比对,确定可能的外显子/内含子边界。
数据和材料的可用性
本研究生成的所有RNA-seq序列在NCBI Short Read Archive (SRA)中公开,登录号为SRR6281886、SRR6281887、SRR6281888和SRR6281889。组装好的转录组保存在DDBJ/EMBL/GenBank转录组Shotgun Assembly (TSA)数据库,登录号为GGAB00000000。蓝莓FATB的互补DNA序列以登录号SRR7879249保存。
缩写
- ABCG:
-
半转运体必须二聚形成功能性ABC转运体
- ACC:
-
乙酰辅酶a羧化酶
- ACLA2:
-
atp -柠檬酸裂解酶A2亚基
- BDG3:
-
水解酶保镖3.
- CD1:
-
番茄细胞外酰基转移酶
- CER:
-
eceriferum
- ECR:
-
Enoyl-CoA还原酶
- 技术工程师:
-
脂肪酸elongase
- 远:
-
脂肪酰coa还原酶
- FATB:
-
acyl-ACP thioesterases
- 外籍家庭:
-
fid
- GL8:
-
Glossy8基因,β-酮酰基还原酶
- GPAT6:
-
Glycerol-3-phosphate酰基转移酶6
- HCD:
-
β-羟基乙酰-CoA脱水酶
- HTH:
-
性急的人
- 九:
-
beta-Ketoacyl-CoA还原酶
- ;:
-
β-酮酰基-CoA合酶
- lac:
-
长链酰基-CoA合成酶
- 电感电容电阻测量:
-
编码细胞色素P450 CYP86A8,脂肪酸ω-羟化酶
- PAS2:
-
超长链羟基脂肪酸辅酶a脱水酶
- RDR1:
-
依赖RNA的RNA聚合酶
- SlTTS1:
-
单功能的β香树脂醇合成酶
- WIN1 / SHN1:
-
WAX诱导剂,ap2结构域蛋白转录因子
- WXP1:
-
包含AP2结构域的推定转录因子
参考文献
- 1.
曹国强,马志强,马志强,等。抗氧化能力受总酚和花青素含量、成熟度和品种的影响。农业食品化学学报1998;46(7):2686-93。
- 2.
Yousef Gg,Brown Af,Funakoshi Y,Mbeunkui F,Grace MH,Ballington JR等。高效定量商业品种中健康相关的花青素和酚醛酸谱和蓝莓育种选择(Vaccinium spp。).中国食品科学。2013;61(20):4806-15。
- 3.
Lobos GA,汉考克JF。为变化的全球环境培育蓝莓:综述。植物科学学报2015;6:782。
- 4.
Albrigo LG,Lyrene PM,Freeman B.蜡和土着果实和叶子的其他特征vaccinium elliotti chapm..acta Hortic sinica, 1980;105(2): 230-5。
- 5.
Shepherd T, Wynne GD。胁迫对植物表皮蜡质的影响。新植醇。2006;171(3):469 - 99。
- 6.
拟南芥角质层蜡的合成、出口和调控。《中华内分泌杂志》2013;52(1):110-29。
- 7.
楚W,Gao H,Cao S,Fang X,Chen H,肖S。蓝莓中有切割蜡的组成和形态(Vaccinium spp。)水果。食品化学。2017;219:436-42。
- 8.
Hen-Avivi S,Savin O,Racovita R,Lee W-S,Adamki N,Malitsky S等人。小麦中的代谢基因簇W1和大麦Cer-cqu基因座确定β-diketone生物合成和肌肉。植物细胞。2016; DOI:https://doi.org/10.1105/TPC.16.00197.
- 9。
Schneider LM, Adamski NM, Christensen CE, Stuart DB, Vautrin S, Hansson M,等。的Cer-cqu基因簇在β-二酮酮合成酶聚酮途径中确定三个关键球员,在迹象蜡中合成蛇磷酸盐。J Exp Bot。2016; 67(9):2715-30。
- 10.
Rowland LJ, Alkharouf N, Darwish O, Ogden EL, Polashock JJ, Bassil NV,等。从冷驯化到脱驯化的蓝莓叶片、发育果实和花蕾的转录组序列的生成和分析。植物学报。2012;12(1):46。
- 11.
Darwish O,Rowland LJ,Alkharouf NW。BBGD454:使用454序列的蓝莓转录组分析数据库。生物信息。2013; 9(17):883-6。
- 12.
TU. UniProt:通用蛋白质知识库。核酸Res. 2017;45(D1): D158-69。
- 13.
付玲,牛波,朱志华,吴胜,李伟。CD-HIT:用于下一代测序数据聚类的加速算法。生物信息学,2012,28(23):3150 - 2。
- 14.
黄雪梅,梁飞,黄雪梅,等。tiger基因指数聚类工具(TGICL):一个用于大型EST数据集快速聚类的软件系统。生物信息学。2003;19(5):651 - 2。
- 15.
罗宾逊MD,麦卡锡DJ,SMYTH GK。编辑:用于数字基因表达数据的差异表达分析的生物导体包。生物信息学。2010; 26(1):139-40。
- 16.
等。美国和加拿大蓝莓产业的育种性状优先。HortScience。2018; 53(7): 1021 - 8。
- 17.
Rowland LJ, Ogden EL, Bassil N, Buck EJ, McCallum S, Graham J,等。蓝莓种间二倍体群体遗传图谱的构建及耐冷性QTL的鉴定。摩尔品种。2014;34(4):2033 - 48。
- 18.
McCallum S, Graham J, Jorgensen L, Rowland LJ, Bassil NV, Hancock JF, et al.;同源四倍体蓝莓SNP和SSR连锁图谱的构建。摩尔品种。2016;36(4):41。
- 19。
流式细胞术测定蓝莓倍性水平和细胞核DNA含量。应用计算机学报。1993;86(8):1001-6。
- 20.
Bian Y,Ballington J,Raja A,Brouwer C,Reid R,Burke M等。在耕种的蓝莓中重复简单序列的模式(Vaccinium section青球藻属。)及其在揭示遗传多样性和群体结构方面的应用。摩尔品种。2014;34(2):675 - 89。
- 21.
等。通过RNA-Seq分析和注释一个拟蓝莓基因组组装,识别了参与果实成熟、生物活性化合物生物合成和阶段特异性选择性剪接的候选基因。Gigascience。2015;4(1):5。
- 22.
死JV,罗兰LJ。通过转录组分析阐明蓝莓的冷驯化途径。环境专家机器人。2014;106(增刊C): 87-98。
- 23.
蔡斌,宋gq。开花调控基因的转录谱VcFT-overexpressing蓝莓植物。PLoS ONE。2016; 11 (6): e0156993。
- 24.
Li L,张H,刘Z,Cui X,张T,Li Y等。比较转录组测序和DE Novo分析Vaccinium corymbosum在水果和颜色发展期间。BMC植物BIOL。2016; 16(1):223。
- 25.
李X,Sun H,Pei J,Dong Y,Wang F,Chen H等人。德诺维蓝莓转录组的测序和比较分析发现抗氧化剂相关的推定基因。基因。2012; 511(1):54-61。
- 26.
Moggia C, Graell J, Lara I, Schmeda-Hirschmann G, Thomas-Valdés S, Lobos GA。高丛蓝莓果实特性和表皮三萜与采后品质的关系。Sci Hortic。2016;211:449-57。
- 27.
郑平,Babar MDA, Parthasarathy S, Gibson R, Parliament K, Flook J,等。在玉米籽油中,一个核苷酸的插入会导致脂肪b被截断,从而减少饱和脂肪酸。中国科学(d辑:地球科学)2014;
- 28.
李丽丽,李强,杨晓东,郑东,等。一个11 bp的插入Zea Mays Fatb.降低了玉米籽粒脂肪酸中棕榈酸的含量。《公共科学图书馆•综合》。2011;6 (9):e24699。
- 29.
Bonaventure G, Salas JJ, Pollard MR, Ohlrogge JB。脂肪b基因的破坏拟南芥显示了饱和脂肪酸在植物生长中的重要作用。植物细胞。2003;15(4):1020 - 33所示。
- 30.
对表皮蜡的研究进展拟南芥和作物物种。植物学报2015;34(4):557-72。
- 31.
ehlenfeldt mk,马丁rb,罗兰lj。'Nocturne'混合蓝莓:冬季硬性,混合物种六倍六倍,具有观赏景观兴趣和新颖的果实品质。HortScience。2015; 50(12):1825-7。
- 32.
Schmittgen TD, Livak KJ。采用对比CT方法分析实时荧光定量PCR数据。Nat Protoc。2008;3:1101。
- 33.
Die JV, Obrero Á, González-Verdejo CI, Román学生物化学肛门。2011;419(2):336 - 8。
- 34.
FASTQC。http://www.bioinformatics.babraham.ac.uk/projects/fastqc/.2018年10月2日。
- 35.
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I,等。没有参考基因组的RNA-Seq数据的全长度转录组组装。Nat生物技术。2011;29(7):644 - 52。
- 36.
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR,等。TopHat和袖扣RNA-seq实验的差异基因和转录本表达分析。Nat Protoc。2012;7(3):562 - 78。
- 37.
李洪波,李洪波,李洪波,等。序列对齐/映射格式和SAMtools。生物信息学。2009;25(16):2078 - 9。
- 38.
Kim D, Pertea G, Trapnell C, Pimentel H, Kelley R, Salzberg SL. TopHat2:在插入、缺失和基因融合中转录组的精确比对。基因组医学杂志。2013;14 (4):R36。
- 39.
领结。http://bowtie-bio.sourceforge.net/index.shtml.2018年10月2日。
- 40。
TransDecoder。https://github.com/TransDecoder/TransDecoder.访问日期:2018年10月2日
- 41。
Mistry J, Finn RD, Eddy SR, Bateman A, Punta M.同源性搜索的挑战:HMMER3和线圈-线圈区域的收敛进化。核酸学报2013;41(12):e121-e121。
- 42。
PlantCyc。http://www.plantcyc.org/.访问日期:2018年10月2日
- 43.
NCBI参考序列(RefSeq):现状、新特征和基因组注释策略。核酸Res. 2012;40(D1): D130-5。
- 44.
Minoru K, Yoko S, Kanae M. BlastKOALA和GhostKOALA:用于基因组和宏基因组序列功能表征的KEGG工具。中华医学杂志。2016;428(4):726-31。
- 45.
Li B,杜威CN。RSEM:具有或不具有参考基因组的RNA-SEQ数据的准确转录物定量。BMC生物信息学。2011; 12(1):323。
- 46.
Salmon提供了快速和偏知的转录本表达量化。Nat冰毒。2017;14(4):417 - 9。
- 47.
Goodstein DM,Shu S,Howson R,Neupane R,Hayes Rd,Fazo J等人。植物血统:绿色植物基因组学的比较平台。核酸RES。2012; 40(D1):D1178-86。
- 48.
Zuker M. Mfold Web服务器用于核酸折叠和杂交预测。核酸RES。2003; 31(13):3406-15。
- 49.
用于实时定量PCR数据管理和自动化分析的qBase相对定量框架和软件。基因组医学杂志。2007;8 (2):R19。
- 50。
Ramakers C, rujter JM, Deprez RHL, Moorman AFM。定量实时聚合酶链反应(PCR)数据的无假设分析。>。2003;339(1):62 - 6。
- 51。
死JV,罗兰LJ。优越的跨物种参考基因:蓝莓案例研究。Plos一个。2013; 8(9):E73354。
- 52。
Panta gr,罗兰lj,arora r,ogden el,lim c-c。蓝莓二倍体映射群中冷耐寒性和脱氢基因的遗传。J作物改进。2004; 10:37-52。
- 53。
Thompson JD, Gibson TJ, Higgins DG。使用ClustalW和ClustalX进行多序列比对。Curr Protoc生物信息学2003;doi:https://doi.org/10.1002/0471250953.bi0203s00.
致谢
我们要感谢美国农业部动物基因组学和改进实验室的Steven G. Schroeder博士为我们的分析提供了一个计算平台。
资金
该项目由USDA-ARS项目提供资金8042-21000-279-00。在房屋资金中提供的USDA-ARS提供由XQ,ELO,MKE,JJP和LJR进行的所有部分进行资助,包括设计研究,收集,分析和解释数据以及编写稿件。Mention of a trademark, proprietary product, or vendor does not constitute a guarantee or warranty of the product by the U.S. Department of Agriculture or any of the other agencies involved in this research, and does not imply its approval to the exclusion of other products or vendors that also may be suitable.
作者信息
从属关系
贡献
XQ进行了生物信息学分析,包括De Novo集装,注释和差异表达分析。ELO提取RNA以制备RNA-SEQ文库并进行RT-QPCR。她还扩增了CDNA,提取的基因组DNA,并扩增了所选基因的序列分析的扩增GDNA。JVD为RT-QPCR设计引物并分析了RT-QPCR结果。MKE构建育种群体和收集的植物组织。MKE,ELO和LJR一起进行了种群的表型评级。JJP测序所选择的cDNA和GDNA序列。OD和NA进行了基于参考的转录组件。LJR设计并指导了研究并参与了数据的分析,包括选择基因的RT-QPCR。所有作者都致力于写作稿件。 All authors have read and approved the manuscript.
作者的信息
在1-2页,3-29行。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
额外的文件1。
补充信息。
附加文件2:表S7。
预测在“夜曲”X T 300人口中的次数。
附加文件3:表S8。
“Nocturne”x US 1212种群的预测DEGs。
附加文件4:表S9。
所选deg的实时qPCR数据。蜡质/非蜡质的折痕变化显示为标准化相对量±SEM的平均值。
附加文件5:表S10。
从文献中选择涉及蜡质积累的17个主要基因的实时qPCR数据。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
齐,X,奥格登,e.l.,狄尔维,J.V.et al。转录组分析在两个适应北方的兔眼育种群体中鉴定了与蓝莓果实蜡质涂层相关的基因。BMC植物杂志19,460(2019)。https://doi.org/10.1186/s12870-019-2073-7
收到了:
接受:
发表:
关键字
- 的隔离分析
- 微分表达式
- 水果蜡
- RNA-seq
- VacciniumSPP。