- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
面包小麦籽粒缺陷遗传位点的快速鉴定与鉴定gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba19gydF4y2Ba,文章号:gydF4y2Ba483gydF4y2Ba(gydF4y2Ba2019gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
小麦是一种重要的作物,养活了世界上数十亿人。提高小麦产量对保障世界粮食安全至关重要。籽粒的正常发育是小麦产量形成的重要保证。籽粒表型的遗传学研究和籽粒灌浆关键基因的鉴定对剖析小麦籽粒形态发生和产量潜力的分子机制具有重要意义。gydF4y2Ba
结果gydF4y2Ba
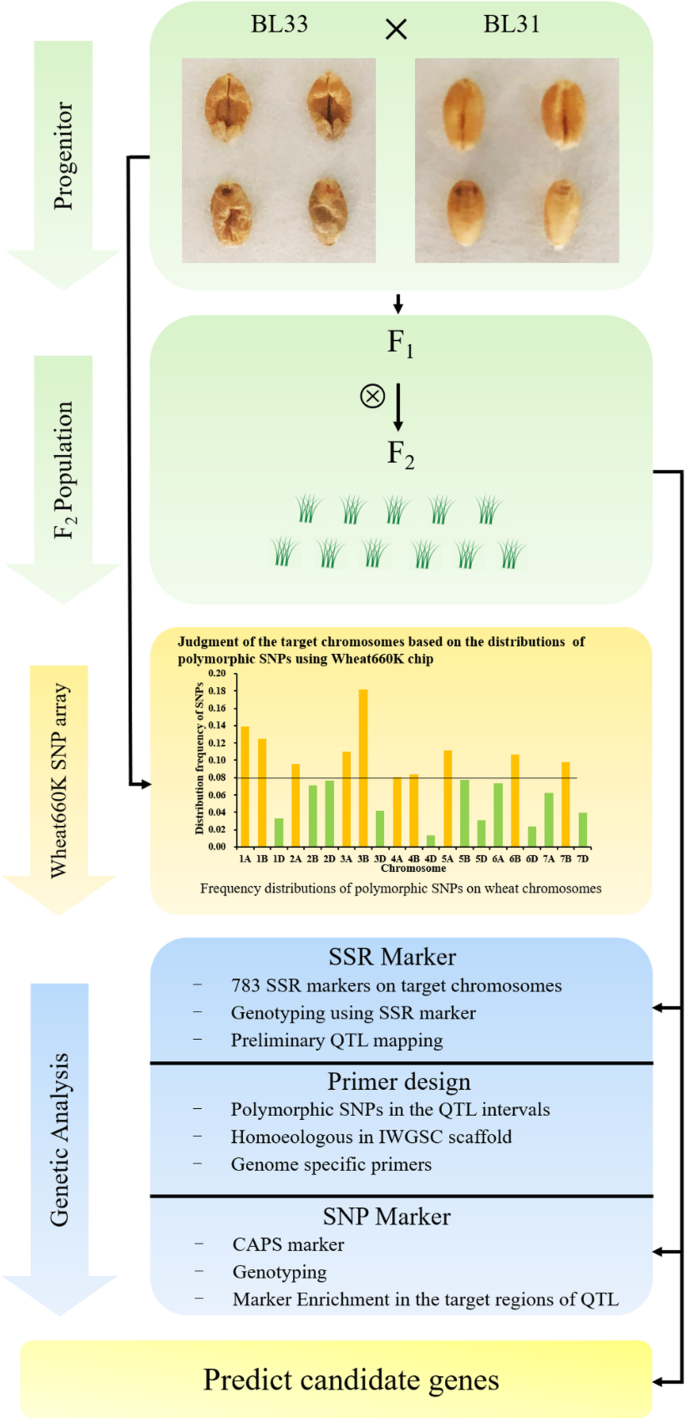
本研究鉴定了一对成熟粒饱满、成熟粒萎缩的缺陷粒(Dek)等基因系BL31和BL33,并构建了BL31/BL33的遗传群体。Wheat660K芯片检测BL31和BL33之间有10条染色体的多态性单核苷酸多态性(SNP)标记频率较高。从上述染色体中筛选出783个SSR标记,利用遗传群体将其中15个标记整合到两个连锁群中。遗传图谱鉴定出3个QTL,gydF4y2BaQDek.caas-3BS.1gydF4y2Ba,gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba,对表型方差的解释分别为14.78 ~ 18.17%、16.61 ~ 21.83%和19.08 ~ 28.19%。此外,从Wheat660K中提取的5个多态性snp成功转化为cleaved amplification polymorphism sequence (CAPS)标记,富集了上述QTL的靶区。生化分析表明,BL33灌浆期籽粒蔗糖含量显著高于BL31,成熟籽粒淀粉含量显著低于BL31,表明Dek QTL可能参与碳水化合物代谢。根据国际小麦基因组序列联盟(International Wheat Genome Sequence Consortium, IWGSC) RefSeq v1.0对每个QTL的候选基因进行预测。gydF4y2Ba
结论gydF4y2Ba
确定了小麦Dek性状的3个主要QTL,并预测了其致病基因,为小麦Dek性状的精细定位和调控机制的深入研究奠定了基础。gydF4y2Ba
背景gydF4y2Ba
小麦(gydF4y2Ba小麦gydF4y2BaL.)为全球超过35%的人口提供主食(gydF4y2Bahttp://www.wheatinitiative.org/sites/default/files/attached_file/wheat_facts.pdfgydF4y2Ba).人口的增加、耕地的减少和极端气候的频繁发生给小麦的消费带来了巨大的挑战。鉴定和挖掘小麦产量遗传位点对提高小麦产量潜力具有重要意义。产量由多个因素决定,如粒重和大小。籽粒饱满度是影响籽粒重量和形态品质的重要参数[gydF4y2Ba1gydF4y2Ba].在以往的研究中,已经发现了许多控制粒重的遗传位点[gydF4y2Ba2gydF4y2Ba],而小麦籽粒饱满度与籽粒灌浆相关的遗传分析鲜有报道。籽粒缺陷突变体(Dek)直接反映在籽粒饱满度上,通常表现为籽粒干瘪、粒重低。因此,鉴定Dek基因位点对提高产量潜力和外观质量具有重要意义。在玉米中发现了不少Dek突变体[gydF4y2Ba3.gydF4y2Ba].他们的基因gydF4y2Badek1gydF4y2Ba,gydF4y2Badek35gydF4y2Ba,gydF4y2Badek38gydF4y2Ba和gydF4y2Badek39gydF4y2Ba均与胚乳中淀粉和/或蛋白质的合成和积累有关[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba].许多Dek突变体已经在小麦中产生,但它们的遗传信息在很大程度上仍然未知。SSR标记具有高多态性、重复性好、共显性、全基因组分布等特点,有利于小麦QTL定位和分子育种[gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba].随着测序技术的快速发展,高通量测序或基于芯片的SNP基因分型平台已被用于QTL定位[gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba].此外,小麦参考基因组已于近期组装并发布[gydF4y2Ba15gydF4y2Ba].获得高质量的小麦基因组数据和高通量SNP基因分型系统对改善小麦遗传育种具有重要意义。我们在一个自发变异群体中鉴定了一个Dek系,发现其籽粒严重萎缩和凹陷。本研究的目的是通过多个基因分型系统、小麦参考基因组和生化分析,快速实现Dek的QTL定位和致病基因预测。gydF4y2Ba
结果gydF4y2Ba
表型分析gydF4y2Ba
BL31的籽粒正常饱满,而BL33的Dek率在所有环境中均为100%(附加文件gydF4y2Ba1gydF4y2Ba).Dek率在FgydF4y2Ba2gydF4y2Ba和FgydF4y2Ba2:3gydF4y2Ba种群在三种环境中呈高度相关,相关系数为0.70 ~ 0.92(表2)gydF4y2Ba1gydF4y2Ba).方差分析显示,两种基因型(gydF4y2BaPgydF4y2Ba< 0.001)和环境(gydF4y2BaPgydF4y2Ba< 0.05)对目标性状有显著影响(表gydF4y2Ba2gydF4y2Ba).基因型与环境的相互作用未达到统计学意义(gydF4y2BaP >gydF4y2Ba0.05)(表gydF4y2Ba2gydF4y2Ba)和F . Dek率的广义遗传力gydF4y2Ba2:3gydF4y2Ba两种环境的差异为0.95,说明Dek率主要由基因型决定。种群中Dek率呈连续分布(图2)。gydF4y2Ba1gydF4y2Ba, a-d),表明Dek由多个基因位点控制。此外,支撑表型的频率图呈“谷-峰”型异常分布,提示种群中存在控制Dek的主要QTL [gydF4y2Ba16gydF4y2Ba].gydF4y2Ba
Dek位点的染色体定位及连锁图的构建gydF4y2Ba
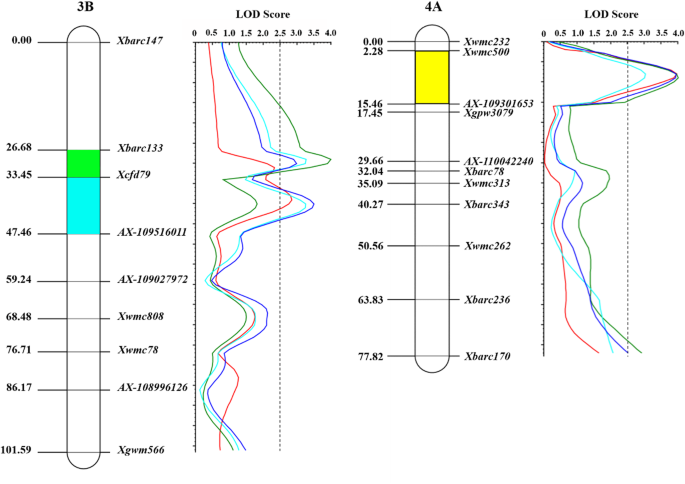
理论上,一条染色体的等基因系之间的多态性标记越多,该染色体具有目标性状QTL的概率越高。为了快速确定Dek QTL的染色体位置,我们使用Wheat660K SNP阵列对等基因系BL31和BL33进行了基因分型,并将其与籽粒表型进行了对比。基于多态性snp的频率分布,Dek QTL认为1A、1B、2A、3A、3B、4A、4B、5A、6B和7B 10条染色体是潜在的靶染色体(附加文件gydF4y2Ba2gydF4y2Ba).为了进一步验证Dek QTL的染色体位置,从上述目标染色体中选取SSR标记构建遗传图谱。从这些染色体上选取783个SSR标记构建连锁图谱进行QTL分析(附加文件)gydF4y2Ba3.gydF4y2Ba) [gydF4y2Ba8gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba].其中118个SSR标记在两个亲本间存在多态性,而在3B(13)、4A(19)和5A(3)染色体上,只有35个SSR标记在两个表型比较的品系(分别为40个缺陷核品系和40个正常核品系)之间存在一致多态性。随后,利用多态性SSR标记对整个F .进行基因分型gydF4y2Ba2gydF4y2Ba在3B和4A染色体上构建了2个连锁群,分别包含6个和9个SSR标记(图2)。gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
Dek QTL的LOD轮廓。遗传区域gydF4y2BaQDek.caas-3BS.1gydF4y2Ba,gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba在gydF4y2Ba绿色gydF4y2Ba,gydF4y2Ba蓝色的gydF4y2Ba和gydF4y2Ba黄色的gydF4y2Ba在染色体上。高义2017年、新乡2018年、高义2018年LOD等高线,平均值用gydF4y2Ba红色的gydF4y2Ba,gydF4y2Ba绿色gydF4y2Ba,gydF4y2BawathetgydF4y2Ba,gydF4y2BadeongareegydF4y2Ba分别行。LOD阈值2.5由虚线垂直表示gydF4y2Ba
QTL定位gydF4y2Ba
基于上述连锁群和遗传群体中的Dek率,对Dek性状进行QTL定位;在3B和4A染色体上检测到3个QTL,分别为gydF4y2BaQDek.caas-3BS.1gydF4y2Ba,gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba,分别为(表gydF4y2Ba3.gydF4y2Ba;无花果。gydF4y2Ba2gydF4y2Ba).gydF4y2BaQDek.caas-3BS.1gydF4y2Ba侧翼被gydF4y2BaXbarc133gydF4y2Ba和gydF4y2BaXcfd79gydF4y2Ba遗传间隔为6.77 cM;gydF4y2BaQDek.caas-3BS.2gydF4y2Ba位于gydF4y2BaXcfd79和Xwmc808gydF4y2Ba间隔35.03 cM;gydF4y2BaQDek.caas-4ALgydF4y2Ba被映射到gydF4y2BaXwmc500gydF4y2Ba和gydF4y2BaXgpw3079gydF4y2Ba在15.17 cM的遗传区间内(图;gydF4y2Ba2gydF4y2Ba).根据上面的初步映射,得到了gydF4y2BaQDek.caas-3BS.1gydF4y2Ba,gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba根据IWGSC参考基因组RefSeq v1.0 (gydF4y2Bahttp://plants.ensembl.org/Triticum_aestivum/Info/IndexgydF4y2Ba) [gydF4y2Ba15gydF4y2Ba].的物理间隔gydF4y2BaQDek.caas-3BS.1gydF4y2Ba,gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba3BS染色体为7.60 ~ 18.84 Mb, 18.84 ~ 32.75 Mb, 4AL染色体为704.34 ~ 710.05 Mb。基因分析表明gydF4y2BaQDek.caas-3BS.1gydF4y2Ba,gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba分别能解释14.78 ~ 18.17%、16.61 ~ 21.83%和19.08 ~ 28.19%的表型变异(表gydF4y2Ba3.gydF4y2Ba).这些QTL在各环境中均能确定10%以上的Dek表型变异,表明它们是主要QTL,符合遗传群体Dek率的“峰谷”分布规律。此外,所有控制Dek形成的等位基因都来自BL33。gydF4y2Ba
QTL靶区标记物富集gydF4y2Ba
为了进一步缩小目标QTL的遗传区间,从Wheat660K芯片中鉴定出的多态SNPs根据QTL初步作图确定的上述物理区域进行筛选。总共有60个多态snp被用于开发位点特异性CAPS标记,其中5个,gydF4y2Baax - 109027972gydF4y2Ba,gydF4y2Baax - 109516011gydF4y2Ba,gydF4y2Baax - 110042240gydF4y2Ba,gydF4y2Baax - 109301653gydF4y2Ba和gydF4y2Baax - 108996126gydF4y2Ba,成功映射到链接组中(附加文件gydF4y2Ba4gydF4y2Ba;额外的文件gydF4y2Ba5gydF4y2Ba;额外的文件gydF4y2Ba6gydF4y2Ba).随后,gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba遗传区间分别缩小至14.01 cM和13.18 cM(表gydF4y2Ba3.gydF4y2Ba;无花果。gydF4y2Ba2gydF4y2Ba).相应地,的物理间隔gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba,分别降至1.16 Mb和1.13 Mb(表gydF4y2Ba3.gydF4y2Ba).虽然多态位点gydF4y2Baax - 108996126gydF4y2Ba和gydF4y2Baax - 110042240gydF4y2Ba的遗传间隔没有进一步缩小吗gydF4y2BaQDek.caas-3BS.1gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba时,它们使目标区域饱和(图;gydF4y2Ba2gydF4y2Ba).Dek性状表现为籽粒发育异常,通常被认为影响籽粒重量。为了验证这一情况,我们分析了Dek QTL对千粒重的影响。与BL31相比,BL33的TGW显著降低24.12-28.81%gydF4y2Ba7gydF4y2Ba).基于上述连锁图谱的QTL分析表明,TGW只有一个QTL,命名为gydF4y2BaQTGW.caas-3BSgydF4y2Ba,在gydF4y2BaXcfd79gydF4y2Ba和gydF4y2Baax - 109027972gydF4y2Ba,解释了19.18 ~ 23.94%的表型变异(附加文件gydF4y2Ba8gydF4y2Ba).gydF4y2BaQTGW.caas-3BSgydF4y2Ba重叠gydF4y2BaQDek.caas-3BS.2gydF4y2Ba提示Dek QTLgydF4y2BaQDek.caas-3BS.2gydF4y2Ba可能影响TGW。其他两个QTL的遗传区间对TGW无显著影响,gydF4y2BaQDek.caas-3BS.1gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba。gydF4y2Ba
Dek QTL候选基因的预测gydF4y2Ba
克隆的gydF4y2BadekgydF4y2Ba玉米基因与籽粒生长发育有关,特别是淀粉和/或蛋白质在胚乳中的合成和储存[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba].我们的生化分析还表明,BL33成熟籽粒淀粉含量显著低于BL31(附加文件)gydF4y2Ba9gydF4y2Ba).相比之下,在灌浆阶段,BL33的蔗糖含量高于BL31,蔗糖还原速度较慢(补充文件)gydF4y2Ba10gydF4y2Ba).据此,我们假设这三个QTL的致病基因都参与了碳水化合物代谢。本研究将3个主要QTL定位到小于15 cM的遗传区间。的物理区域gydF4y2BaQDek.caas-3BS.1gydF4y2Ba为11.24 Mb,而gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDeg.caas-4ALgydF4y2Ba根据IWGSC RefSeq v1.0,分别只有1.16和1.13 Mb(表gydF4y2Ba3.gydF4y2Ba).3个QTL的物理间隔中共存在233个基因(附加文件)gydF4y2Ba11gydF4y2Ba).我们发现了9个基因(基因ID:gydF4y2BaTraesCS3B01G025200、TraesCS3B01G028100、TraesCS3B01G028200、TraesCS3B01G028300、TraesCS3B01G028500、TraesCS3B01G028700、TraesCS3B01G039100、TraesCS3B01G039800gydF4y2Ba,gydF4y2BaTraesCS4A01G446700gydF4y2Ba涉及碳水化合物代谢或在Dek qtl的目标区域或附近的谷物发育(附加文件)gydF4y2Ba11gydF4y2Ba).为了进一步确定Dek QTL的候选基因,利用WheatExp (gydF4y2Bahttps://wheat.pw.usda.gov/WheatExp/gydF4y2Ba).在这四个基因中,gydF4y2BaTraesCS3B01G025200gydF4y2Ba,gydF4y2BaTraesCS3B01G028700gydF4y2Ba,gydF4y2BaTraesCS3B01G039800gydF4y2Ba,gydF4y2BaTraesCS4A01G446700gydF4y2Ba,在颗粒显影期间有一个可观察的表达式(附加文件gydF4y2Ba12gydF4y2Ba) [gydF4y2Ba24gydF4y2Ba].根据IWGSC RefSeq v1.0的功能注释,分别编码果糖-二磷酸醛缩酶(Fba)、β-果糖呋喃糖苷酶(β-果糖呋喃糖苷酶)、脱落酸缺乏4 (ABA4)和蔗糖合成酶(Sus)的4个基因,暂定为gydF4y2BaTaFba-3BgydF4y2Ba,gydF4y2BaTaBff-3BgydF4y2Ba,gydF4y2BaTaABA4-3BgydF4y2Ba,gydF4y2BaTaSus-4AgydF4y2Ba。利用qPCR技术在三个不同阶段(5、15和25 DAF)收获的未成熟籽粒中证实了它们的表达模式(附加文件)gydF4y2Ba13gydF4y2Ba).结果表明,上述基因均在籽粒中表达,且在5 DAF时表达量最低。gydF4y2BaTaBff-3BgydF4y2Ba和gydF4y2BaTaSus-4AgydF4y2Ba主要检测到15 DAF,而gydF4y2BaTaFba-3BgydF4y2Ba和gydF4y2BaTaABA4-3BgydF4y2Ba在25-DAF粒中表达活性最高。gydF4y2Ba
讨论gydF4y2Ba
通过多个基因分型系统结合小麦参考基因组的快速和经济有效的QTL定位策略gydF4y2Ba
随着测序技术的快速发展,越来越多的小麦品种被测序,并鉴定出大量的SNPs [gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba].因此,一些基于测序和基于芯片的SNP基因分型平台已经开发出来并用于小麦的遗传分析[gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba].一些研究利用SNP芯片实现了QTL的精细定位[gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba].基于芯片的SNP阵列对于基因分析是高通量和有效的,但芯片非常昂贵。在本研究中,为了快速且经济有效地绘制Dek的QTL,我们仅使用Wheat660K芯片对具有对比表型的等基因亲本系进行基因分型。首先,根据多态性snp的频率分布,确定可能含有Dek QTL的目标染色体。其次,有必要通过基因分型遗传群体构建QTL连锁图谱。然而,众所周知,将SNP转化为灵活的标记(如KASP和CAPS)是费时费力的,特别是在具有三个同源亚基因组的六倍体小麦中。考虑到SSR标记的高多态性、高重复性、共显性和全基因组分布,我们在上述候选染色体中收获了方便的SSR标记,并快速构建了连锁图[gydF4y2Ba8gydF4y2Ba,gydF4y2Ba35gydF4y2Ba].第三,根据IWGSC RefSeq v1.0对Dek QTL进行遗传作图,并确定其物理区间。最后,我们成功地将物理靶区部分多态snp转化为CAPS标记,缩小了QTL的遗传区域。综上所述,我们通过将高通量SNP阵列与具有成本效益和灵活性的SSR标记以及小麦参考基因组相结合,建立了一种快速高效的QTL定位策略。gydF4y2Ba3.gydF4y2Ba).当然,要在所有染色体中找到QTL,全基因组连锁图是必要的。未来,我们将构建重组自交系(RIL)群体,实现Dek QTL的全基因组鉴定。gydF4y2Ba
快速高效的Dek性状QTL定位工作流程。我们无花果的制作。gydF4y2Ba3.gydF4y2Ba参考图的框架。gydF4y2Ba1gydF4y2BaRamirez-Gonzalez等人。[gydF4y2Ba36gydF4y2Ba]gydF4y2Ba
Dek QTL与先前报道的Dek相关基因位点的比较gydF4y2Ba
通过对dek相关性状的遗传位点比较,不仅可以加深对QTL遗传效应的认识,还可以为QTL精细定位提供遗传信息。gydF4y2BaQDek.caas-3BS.1gydF4y2Ba与gydF4y2BaXbarc133gydF4y2Ba(表gydF4y2Ba3.gydF4y2Ba;无花果。gydF4y2Ba2gydF4y2Ba).两个QTL,gydF4y2BaQGfrmax.nfcri-3BgydF4y2Ba和gydF4y2BaQGfd.nfcri-3BgydF4y2Ba,分别控制籽粒灌浆速率(GFR)和籽粒灌浆期(GFD)gydF4y2BaXgwm533gydF4y2Ba的物理距离为1mbgydF4y2BaXbarc133gydF4y2Ba[gydF4y2Ba37gydF4y2Ba].gydF4y2BaQTgw.nfcri-3BgydF4y2BaTGW也与gydF4y2BaXgwm533gydF4y2Ba[gydF4y2Ba37gydF4y2Ba].gydF4y2BaQDek.caas-3BS.1gydF4y2Ba和gydF4y2BaQDek.caas-3BS.2gydF4y2Ba在相邻区域被发现,两者都与gydF4y2BaXcfd79gydF4y2Ba(表gydF4y2Ba3.gydF4y2Ba;无花果。gydF4y2Ba2gydF4y2Ba).TGW的QTL与gydF4y2BaXcfd79gydF4y2Ba由Groos等人发现。[gydF4y2Ba38gydF4y2Ba].此外,gydF4y2BaQMswscf.acs-3B.1gydF4y2Ba花前期主茎的水溶性碳水化合物(WSC)与gydF4y2BaXcfd79gydF4y2Ba[gydF4y2Ba39gydF4y2Ba].gydF4y2BaQDek.caas-4ALgydF4y2Ba被gydF4y2BaXwmc500gydF4y2Ba和gydF4y2Baax - 109301653gydF4y2Ba(表gydF4y2Ba3.gydF4y2Ba).之间的物理距离gydF4y2BaQDek.caas-4ALgydF4y2Ba和gydF4y2BaWx-B1gydF4y2Ba大约是16mb。迄今为止,许多QTL为淀粉、蛋白质及直链淀粉和支链淀粉的含量,已报道在4AL染色体上与籽粒产量接近gydF4y2BaWx-B1gydF4y2Ba[gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba].gydF4y2BaQGsc4AgydF4y2Ba,是测定谷物淀粉含量的QTL,与gydF4y2BaWx-B1gydF4y2Ba[gydF4y2Ba42gydF4y2Ba].gydF4y2BaQftsc4AgydF4y2Ba谷物总淀粉含量和gydF4y2BaQfams4AgydF4y2Ba籽粒直链淀粉含量也与之相关gydF4y2BaWx-B1gydF4y2Ba[gydF4y2Ba46gydF4y2Ba].以确定之间的遗传关系gydF4y2BaQDek.caas-4ALgydF4y2Ba和gydF4y2BaWx-B1gydF4y2Ba, Nakamura等人开发的基因特异性功能标记。[gydF4y2Ba48gydF4y2Ba]对亲本BL31和BL33进行基因分型。结果显示它们之间没有检测到多态性(附加文件gydF4y2Ba14gydF4y2Ba),暗示gydF4y2BaWx-B1gydF4y2Ba对戴克性状影响不大。gydF4y2Ba
候选基因分析gydF4y2Ba
两个候选基因,gydF4y2BaTaFba-3BgydF4y2Ba和gydF4y2BaTaBff-3BgydF4y2Ba,在gydF4y2BaQDek.caas-3BS.1gydF4y2Ba分别编码Fba和β-果糖呋喃苷酶(表gydF4y2Ba4gydF4y2Ba).Fba是参与Calvin循环、控制光合器官固碳和淀粉合成的非调控酶之一,可催化磷酸二羟基丙酮与3-磷酸甘油醛可逆醛缩反应生成果糖-1,6-二磷酸[gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba].β-果糖呋喃糖苷酶是蔗糖和淀粉合成、代谢和转运的关键酶,可不可逆地催化蔗糖水解为葡萄糖和果糖[gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba].gydF4y2BaTaFba-3BgydF4y2Ba和gydF4y2BaTaBff-3BgydF4y2Ba可能调节碳水化合物代谢,从而影响Dek表型。gydF4y2BaTaABA4-3BgydF4y2Ba被预测为候选基因gydF4y2BaQDek.caas-3BS.2。gydF4y2BaABA4在新黄质合成中的作用[gydF4y2Ba53gydF4y2Ba].新黄质是维持叶片捕光叶绿素和光合功能完整性所必需的,涉及光合产物的代谢[gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba].gydF4y2BaTaSus-4AgydF4y2Ba可能是致病基因的gydF4y2BaQDek.caas-4AL。SusgydF4y2Ba缺乏症导致gydF4y2Bashrunken-1gydF4y2Ba玉米的突变[gydF4y2Ba56gydF4y2Ba].关联分析表明,两种蔗糖合成酶基因,gydF4y2BaTaSus1gydF4y2Ba和gydF4y2BaTaSus2gydF4y2Ba对小麦同源染色体7组和2组分别有显著影响[gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba].在不久的将来,我们将通过小麦转化实验鉴定这些候选基因的多态性位点,对目标QTL进行精细定位,并进一步确定Dek的致病基因。gydF4y2Ba
结论gydF4y2Ba
在这项研究中,我们检测到三个DEK qtl,gydF4y2BaQDek.caas-3BS.1gydF4y2Ba,gydF4y2BaQDek.caas-3BS.2gydF4y2Ba和gydF4y2BaQDek.caas-4ALgydF4y2Ba,每一种都可以解释10%以上的表型变异。此外,他们的候选基因,gydF4y2BaTaFba-3BgydF4y2Ba,gydF4y2BaTaBff-3BgydF4y2Ba,gydF4y2BaTaABA4-3BgydF4y2Ba,gydF4y2BaTaSus-4AgydF4y2Ba,根据生化分析和IWGSC RefSeq v1.0注释进行预测。这些结果为小麦Dek的QTL精细定位和分子机制剖析提供了重要信息。gydF4y2Ba
方法gydF4y2Ba
植物材料gydF4y2Ba
在甘肃省农业科学院小麦研究所(GAAS)培育了一批Dek系。通过多代自交鉴定出BL31和BL33等基因系,成熟粒饱满,成熟粒萎缩。遗传群体包括146个FgydF4y2Ba2gydF4y2Ba植物及其相应的FgydF4y2Ba2:3gydF4y2Ba将BL31和BL33的杂交系进行QTL定位。gydF4y2Ba
田间试验和表型评价gydF4y2Ba
FgydF4y2Ba2gydF4y2Ba2016-2017年在河北高邑(37°33′n, 114°26′e)种植。FgydF4y2Ba2:3gydF4y2Ba2017-2018年在河南新乡(34°53′n, 113°23′e)和高邑县播种。田间试验被随机地安排在完整的区块中,每个位置两个重复。每个地块包括一个2米长的行,种植20株植物,间隔30厘米。谷物以单株或行收割。水稻籽粒表型gydF4y2Ba2gydF4y2Ba用单株缺陷粒的百分比(定义为Dek率)对植株进行评分。FgydF4y2Ba2:3gydF4y2Ba在各株系中,每株系中选取10个具有代表性的株系,计算Dek率,然后以各株系的平均Dek率进行后续统计和遗传分析。gydF4y2Ba
利用SNP芯片初步定位目标染色体QTLgydF4y2Ba
Guillemaut和Laurence从新鲜树叶中提取了高质量的基因组DNA [gydF4y2Ba59gydF4y2Ba].由中国农业科学院作物科学研究所开发的高通量基因分型工具Wheat660K,包含630,517个SNP。gydF4y2Bahttps://wheat.pw.usda.gov/ggpages/topics/Wheat660_SNP_array_developed_by_CAAS.pdfgydF4y2Ba),用于检测两个等基因系BL31和BL33。gydF4y2Bahttp://www.capitalbio.com/gydF4y2Ba).利用Microsoft Excel 2016分析每条染色体中多态性snp的频率分布(gydF4y2Bahttps://www.microsoft.com/zh-cn/gydF4y2Ba).根据多态性snp在每条染色体上的频率分布,确定具有潜在QTL的目标染色体。gydF4y2Ba
SSR标记分型gydF4y2Ba
在15 μl含有7.5 μl 2 ×的混合液中进行SSR标记的聚合酶链反应(PCR)gydF4y2BaTaqgydF4y2BaPCR Mix(北京浩特生物科技有限公司,北京;gydF4y2Bahttp://www.ht-biotech.net/gydF4y2Ba),每个引物5 pmol和50-100 ng基因组DNA,使用ABI Applied Biosystems Veriti 96 Well Thermal Cycler (Gene Co., Ltd, Shanghai;gydF4y2Bahttp://www.genecompany.com/gydF4y2Ba).95°C PCR扩增5 min, 35个循环(94°C 30s, 55-60°C 30s, 72°C 30s), 72°C扩增7 min。PCR产物采用JY 600HE电泳仪(北京君益电泳有限公司,北京;gydF4y2Bahttp://www.bjjunyi.com/gydF4y2Ba)或2%琼脂糖凝胶,在220v下使用DYY-7C电泳仪(北京六一生物科技有限公司;gydF4y2Bahttp://www.ly.com.cn/gydF4y2Ba).gydF4y2Ba
将基于芯片的多态snp转化为CAPS标记gydF4y2Ba
为了丰富目标遗传区域,需要将BL31和BL33之间的多态性snp转化为灵活的标记,如竞争性等位基因特异性PCR (KASP)和CAPS标记。CAPS技术是snp基因分型的有效工具[gydF4y2Ba60gydF4y2Ba],结合PCR和限制性内切酶酶切,对具有多态性SNPs的目标等位基因进行鉴别。利用DNAMAN软件对目标区域亲本间的多态性snp进行比对(gydF4y2Bahttps://www.lynnon.com/index.htmlgydF4y2Ba)设计染色体特异性CAPS标记[gydF4y2Ba61gydF4y2Ba].用于CAPS标记的引物和限制性内切酶在附加文件中列出gydF4y2Ba4gydF4y2Ba。gydF4y2Ba
统计分析gydF4y2Ba
采用SAS 9.2软件(gydF4y2Bahttp://www.sas.comgydF4y2Ba)与PROC混合程序。广义遗传力(gydF4y2BahgydF4y2BaBgydF4y2Ba2gydF4y2Ba)按以下公式计算[gydF4y2Ba62gydF4y2Ba]:gydF4y2BahgydF4y2BaBgydF4y2Ba2gydF4y2Ba=σgydF4y2Ba2gydF4y2BaggydF4y2Ba/(σgydF4y2Ba2gydF4y2BaggydF4y2Ba+σgydF4y2Ba2gydF4y2Ba通用电气gydF4y2Ba/ r + σgydF4y2Ba2gydF4y2BaεgydF4y2Ba/ re)。gydF4y2Ba
在这,gydF4y2BaσgydF4y2Ba2gydF4y2BaggydF4y2Ba,gydF4y2BaσgydF4y2Ba2gydF4y2Ba通用电气gydF4y2Ba和gydF4y2BaσgydF4y2Ba2gydF4y2BaεgydF4y2Ba分别对基因型、基因型×环境相互作用和残差方差进行估计;gydF4y2BargydF4y2Ba和gydF4y2BaegydF4y2Ba分别为每个环境的重复数和环境数。采用PROC CORR程序和SAS 9.2软件计算3种不同环境间性状的相关系数。gydF4y2Ba
连锁图谱构建及QTL分析gydF4y2Ba
joinmap4.0软件(gydF4y2Bahttp://www.kyazma.nlgydF4y2Ba)用于构造连接图[gydF4y2Ba63gydF4y2Ba],利用Kosambi作图函数估计标记间的遗传距离[gydF4y2Ba64gydF4y2Ba].QTL分析采用基于Dek率的包容性复合区间映射(ICIM),采用IciMapping 4.0软件(gydF4y2Bahttp://www.isbreeding.netgydF4y2Ba) [gydF4y2Ba65gydF4y2Ba,gydF4y2Ba66gydF4y2Ba].基于1000个I型误差为的排列,确定显著QTL的对数概率(LOD)阈值评分为2.5gydF4y2BaPgydF4y2Ba< 0.05。通过逐步回归计算表型方差解释(PVE),以估计单个QTL效应。gydF4y2Ba
谷物淀粉和蔗糖含量测定gydF4y2Ba
六粒成熟的谷物在臼中被磨成细粉。所得粉末0.1 g,用淀粉含量测定试剂盒(北京太阳神科技有限公司,北京,gydF4y2Bahttp://www.solarbio.net/gydF4y2Ba).然后用以下公式将mg/g换算为mg/粒,对籽粒淀粉含量进行评分:籽粒淀粉含量(mg/粒)=淀粉含量(mg/g) * TGW / 1000。gydF4y2Ba
TGW数据显示在附加文件中gydF4y2Ba7gydF4y2Ba。在开花后5、10、15、20、25和30 d (DAF)采集2个亲本的6粒,验证灌浆期籽粒蔗糖含量的动态变化趋势。将干燥的谷物在砂浆中研磨成细粉,取0.1 g粉末,用植物蔗糖检测试剂盒测定谷物蔗糖含量(北京太阳生物科技有限公司,北京,gydF4y2Bahttp://www.solarbio.net/gydF4y2Ba).gydF4y2Ba
实时定量PCRgydF4y2Ba
用RNAprep pure Plant Kit(天根生物科技有限公司)从开花后5、15、25天的新鲜谷物中提取总RNA。gydF4y2Bahttp://www.tiangen.com/gydF4y2Ba).使用逆转录酶M-MLV (RNase HgydF4y2Ba−gydF4y2BaTakara生物医药科技有限公司gydF4y2Bahttp://www.takara.comgydF4y2Ba).使用iTaq™Universal SYBR Green Supermix on CFX Real-Time System (Bio-Rad)进行实时定量PCR (qPCR)。qPCR谱图如下:95°C for 2 min, 95°C for 5 s, 58°C for 20 s,循环40次。最后进行分离阶段,生成用于判断扩增特异性的熔解曲线。qPCR定量采用2gydF4y2Ba——ΔΔCtgydF4y2Ba方法(gydF4y2Ba67gydF4y2Ba].gydF4y2Ba伸长因子1 αgydF4y2Ba(gydF4y2BaEF1αgydF4y2Ba)基因作为内控,校准感兴趣基因的表达水平。附加文件显示qPCR中使用的引物(见附加文件)gydF4y2Ba15gydF4y2Ba).gydF4y2Ba
数据和材料的可用性gydF4y2Ba
支持本文结果的数据集包含在本文及其附加文件中。gydF4y2Ba
缩写gydF4y2Ba
- AGPP:gydF4y2Ba
-
二磷酸腺苷葡萄糖焦磷酸化酶gydF4y2Ba
- 方差分析:gydF4y2Ba
-
方差分析gydF4y2Ba
- 帽子:gydF4y2Ba
-
裂解扩增多态序列gydF4y2Ba
- 证照:gydF4y2Ba
-
细胞壁转化酶gydF4y2Ba
- DAF):gydF4y2Ba
-
开花后几天gydF4y2Ba
- 卡片:gydF4y2Ba
-
有缺陷的内核gydF4y2Ba
- 功能性:gydF4y2Ba
-
fructose-bisphosphate醛缩酶gydF4y2Ba
- GFD:gydF4y2Ba
-
灌浆时间gydF4y2Ba
- 肾小球滤过率(GFR):gydF4y2Ba
-
灌浆速率gydF4y2Ba
- GWAS:gydF4y2Ba
-
全基因组关联研究gydF4y2Ba
- ICIM:gydF4y2Ba
-
包含复合区间映射gydF4y2Ba
- IWGSC:gydF4y2Ba
-
国际小麦基因组序列联盟gydF4y2Ba
- KASP:gydF4y2Ba
-
竞争性等位基因特异性聚合酶链反应gydF4y2Ba
- LOD:gydF4y2Ba
-
概率对数gydF4y2Ba
- 通过:gydF4y2Ba
-
解释表型变异gydF4y2Ba
- qPCR:gydF4y2Ba
-
实时定量PCRgydF4y2Ba
- SNP:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
- 苏维埃社会主义共和国:gydF4y2Ba
-
简单序列重复gydF4y2Ba
- Sus:gydF4y2Ba
-
蔗糖合酶gydF4y2Ba
- TGW:gydF4y2Ba
-
千粒重gydF4y2Ba
- WSC:gydF4y2Ba
-
水溶性碳水化合物gydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
程硕,杨明生,张伯强,季克泽,赵伯博,高DR.小麦籽粒饱满度与灌浆指数(gydF4y2Ba小麦gydF4y2Bal .)。江苏农业科学1993;9:7-10。gydF4y2Ba
- 2.gydF4y2Ba
李凡杰,文伟,何志华,刘剑德,金辉,曹帅,等。利用高密度SNP标记对三个中国面包小麦群体产量相关性状的全基因组连锁定位。理论应用,2018;131:1903-24。gydF4y2Ba
- 3.gydF4y2Ba
Neuffer MG, Sheridan WF。有缺陷的玉米籽粒突变体。一、遗传和致死率研究。遗传学。1980;95:929-44。gydF4y2Ba
- 4.gydF4y2Ba
李立德,郭志刚,张志刚,张志刚,等。的gydF4y2Ba缺陷核1gydF4y2Ba(gydF4y2Badek1gydF4y2Ba)玉米籽粒胚乳中糊粉层细胞发育所需的基因编码钙蛋白酶基因超家族的膜蛋白。自然科学进展,2002;gydF4y2Ba
- 5.gydF4y2Ba
陈晓珍,冯峰,齐文文,徐丽敏,姚德升,王强,等。gydF4y2BaDek35gydF4y2Ba编码一种PPR蛋白,影响线粒体nad4内含子1的顺式剪接和玉米种子发育。植物学报。2017;10:427-41。gydF4y2Ba
- 6.gydF4y2Ba
李亚波,李志强,李志强,王志强,等。玉米TTI2基因ds元件插入对玉米籽粒缺陷突变体的影响。自然科学进展,2017;gydF4y2Ba
- 7.gydF4y2Ba
李新军,魏刚,孙世林,陈志林,景超,宋伟,等。缺陷核39编码玉米种子发育所需的PPR蛋白。中国生物医学工程学报,2018;gydF4y2Ba
- 8.gydF4y2Ba
Röder MS, Korzun V, Wendehake K, Plaschke J, Tixier MH, Leroy P,等。小麦的微卫星地图。遗传学。1998;149:2007-23。gydF4y2Ba
- 9.gydF4y2Ba
王勇,杨超,金庆军,周德杰,王士生,于玉军,等。禾本科植物SSRs的全基因组分布、比较及组成分析。BMC Genet 2015;16:18。gydF4y2Ba
- 10.gydF4y2Ba
UPOV-BMT, BMT/36/10技术委员会第36届会议进展报告,特别是生化和分子技术及dna分析技术工作组,日内瓦,2002年。gydF4y2Ba
- 11.gydF4y2Ba
Sukumaran S, Dreisigacker S, Lopes M, Chavez P, Reynolds MP。生长在温带灌溉环境的优良春小麦群体籽粒产量和相关性状的全基因组关联研究。《应用理论》2015;128:353-63。gydF4y2Ba
- 12.gydF4y2Ba
高丽丽,李志强,李志强,李志强。春小麦优良选育系幼苗和成株抗叶锈病全基因组关联研究。PLoS One, 2016;11:e0148671。gydF4y2Ba
- 13.gydF4y2Ba
Kassa MT, You FM,希伯特CW, Pozniak CJ, Fobert PR, Sharpe AG。高效筛选小麦抗叶锈病基因的高预测性SNP标记gydF4y2BaLr16gydF4y2Ba。BMC植物生物学,2017;17:45。gydF4y2Ba
- 14.gydF4y2Ba
Mourad AMI, salam A, Belamkar V, Wegulo S, Bowden R, Jin Y.基于全基因组关联研究的新型SNP标记的鉴定和验证gydF4y2BaSr6gydF4y2Ba面包小麦茎锈病抗性基因研究。中国植物科学,2018;9:380。gydF4y2Ba
- 15.gydF4y2Ba
张志刚,张志刚,张志刚,等。利用完全注释的参考基因组改变小麦研究和育种的限制。科学。2018;361年。gydF4y2Ba
- 16.gydF4y2Ba
曹帅,郭晓林,刘大东,张小强,张安明。小麦光周期温敏性雄性不育基因定位的初步研究(gydF4y2Ba小麦gydF4y2Bal .)。植物学报2004;31:293-8。gydF4y2Ba
- 17.gydF4y2Ba
宋qj, Fickus EW, Cregan PB。小麦三核苷酸SSR基序的鉴定。应用理论。2002;104:286-93。gydF4y2Ba
- 18.gydF4y2Ba
郭宏宏,刘志强,刘志强,刘志强。微卫星的可转移性研究gydF4y2Ba小麦属植物tauschiigydF4y2Ba对六倍体小麦和二倍体相关种进行扩增、杂交和序列比较。应用理论,2002;105:736-44。gydF4y2Ba
- 19.gydF4y2Ba
王晓明,王晓明,王晓明。小麦微卫星向二倍体转化的研究进展gydF4y2BaTriticeaegydF4y2Ba携带a、B和D基因组的物种。《应用理论》2001;103:346-52。gydF4y2Ba
- 20.gydF4y2Ba
王晓明,王晓明,王晓明,王晓明。基于多态微卫星标记的植物遗传多样性研究gydF4y2Ba山羊草属tauschiigydF4y2Ba以及对面包小麦d基因组的转移性。应用理论,2002;104:1164-72。gydF4y2Ba
- 21.gydF4y2Ba
Pestsova E, Ganal EW, Röder MS.面包小麦D基因组特异性微卫星标记的分离和定位。基因组。2000;43:689 - 97。gydF4y2Ba
- 22.gydF4y2Ba
苏迪勒P, Singh S, Cadalen T, Brown-Guedira GL, Gay G, Qi LL,等。基于微卫星缺失仓系统建立小麦遗传-物理图谱关系(gydF4y2Ba小麦gydF4y2Bal .)。功能性整合基因组学。2004;4:12-25。gydF4y2Ba
- 23.gydF4y2Ba
张晓明,王晓明,王晓明,等。面包小麦高密度微卫星一致性图谱研究(gydF4y2Ba小麦gydF4y2Bal .)。应用理论。2004;109:1105-14。gydF4y2Ba
- 24.gydF4y2Ba
张晓明,张晓明,张晓明。谷物生长发育阶段的十进制编码。杂草决议1974;14:415-21。gydF4y2Ba
- 25.gydF4y2Ba
IWGSC。基于染色体的六倍体面包小麦抽穗序列(gydF4y2Ba小麦gydF4y2Ba)基因组。科学。2014;345:1251788。gydF4y2Ba
- 26.gydF4y2Ba
李志强,王志强,王志强,等。野生二聚体基因组结构和多样性阐明了小麦的进化和驯化。科学。2017;357:93-7。gydF4y2Ba
- 27.gydF4y2Ba
罗春春,顾永强,邓培宇,王华,苏华哲,迪尔KR,等。小麦D基因组祖先Aegilops tauschii的基因组序列。大自然。2017;551:498 - 502。gydF4y2Ba
- 28.gydF4y2Ba
王思聪,黄东,傅立刚,黄斌,黄伟。利用高密度90,000个单核苷酸多态性阵列鉴定多倍体小麦基因组多样性。植物科学学报。2014;12(2):787 - 96。gydF4y2Ba
- 29.gydF4y2Ba
Winfield MO, Allen AM, Burridge AJ, Barker GLA, Benbow HR, Wilkinson PA,等。六倍体小麦及其二、三级基因库的高密度SNP基因分型阵列植物科学进展。2016;14(2):366 - 366。gydF4y2Ba
- 30.gydF4y2Ba
Allen AM, Winfield MO, Burridge AJ, Downie RC, Benbow HR, Barker GL,等。适用于六倍体面包小麦全球种质高通量SNP基因分型的小麦育种家阵列(gydF4y2Ba小麦gydF4y2Ba).中国生物工程学报,2017;15(3):369 - 369。gydF4y2Ba
- 31.gydF4y2Ba
金辉,文伟,刘建东,翟森,张勇,闫杰,等。高成8901/周麦16重组自交系小麦加工品质参数的全基因组QTL定位前沿植物科学,2016;7:1032S。gydF4y2Ba
- 32.gydF4y2Ba
崔峰,张宁,范晓玲,张伟,赵春杰,杨丽娟,潘锐,梅超,杰红,赵霞。利用小麦660k SNP阵列高密度遗传图谱进行核数主要QTL的高分辨率定位。科学代表2017;7:3788。gydF4y2Ba
- 33.gydF4y2Ba
田小林,文伟,谢林,付丽萍,徐达,付超,等。面包小麦低株高基因Rht24的分子定位。中国植物科学,2017;8:1379。gydF4y2Ba
- 34.gydF4y2Ba
刘建东,何志辉,吴林,白波,文伟,谢佳杰,等。面包小麦黑点反应QTL的全基因组连锁定位(gydF4y2Ba小麦gydF4y2Bal .)。理论应用,2016;129:1-12。gydF4y2Ba
- 35.gydF4y2Ba
王丽霞,邱娟,常丽龙,刘汉华,李海波,庞保生,等。利用SSR标记评价小麦品种差异。中国农业科学。2015;14:1923-35。gydF4y2Ba
- 36.gydF4y2Ba
Ramirez-Gonzalez RH, Segovia V, Bird N, Fenwick P, Holdgate S, Berry S,等。RNA-Seq散装分离分析能够识别高分辨率遗传标记,用于六倍体小麦育种。中国生物技术学报(英文版);2015;gydF4y2Ba
- 37.gydF4y2Ba
王仁祥,张晓霞,吴林,王锐,海林,闫春春,等。不同生态环境下小麦灌浆率和千粒重的QTL定位作物学报。2008;34:1750-6。gydF4y2Ba
- 38.gydF4y2Ba
陈志伟,陈志伟,陈志伟,等。面包小麦籽粒蛋白质含量、产量和千粒重的遗传分析。应用理论,2003;106:1032-40。gydF4y2Ba
- 39.gydF4y2Ba
胡玉军,李文明,杨德龙,刘勇,程海波,常林,等。小麦籽粒水溶性碳水化合物含量数量性状位点的整合与元分析。麦类作物,2016;36:989-95。gydF4y2Ba
- 40.gydF4y2Ba
王志刚,王志刚,王志刚。影响小麦4A染色体直链淀粉含量和农艺性状的遗传位点鉴定。应用理论。1999;98:977-84。gydF4y2Ba
- 41.gydF4y2Ba
Mccartney CA, Somers DJ, Lukow O, Ames N, Noll J, Cloutier S,等。春小麦组合RL4452 בAC域’品质性状QTL分析植物育种。2006;125:565-75。gydF4y2Ba
- 42.gydF4y2Ba
田波,刘波,朱志林,谢庆国,田景昌。小麦籽粒淀粉积累的条件和无条件QTL定位。中国农业科学。2011;44:4551-9。gydF4y2Ba
- 43.gydF4y2Ba
王建军,刘文华,王辉,李汉林,吴娟,杨晓明,等。小麦种质资源3228产量相关性状QTL定位Euphytica。2011;177:277 - 92。gydF4y2Ba
- 44.gydF4y2Ba
崔峰,赵超,丁爱明,李娟,王玲,李小峰,等。利用3个相关小麦RIL群体构建籽粒产量相关性状的综合连锁图谱及QTL定位。理论应用,2014;127:659-75。gydF4y2Ba
- 45.gydF4y2Ba
田斌,邓志智,谢庆国,江景涛。小麦籽粒总淀粉含量及其组分发育行为的遗传解剖。农学通报2015;66:445-55。gydF4y2Ba
- 46.gydF4y2Ba
邓志智,胡森,陈峰,李文军,陈建军,孙春林,等。小麦蛋白质与淀粉相互作用的遗传解剖。Mol Breed. 2015;35:12。gydF4y2Ba
- 47.gydF4y2Ba
邓志智,方WQ,郭旭,赵XX,郭宏,胡森,等。利用两种QTL作图方法对两个群体小麦粉淀粉及其组分间相互作用的遗传解剖。Mol Breed. 2018;38:41。gydF4y2Ba
- 48.gydF4y2Ba
中村勇,李志强,李志强,等。基于pcr技术的部分蜡质小麦的快速分类。基因组。2002;45:1150-6。gydF4y2Ba
- 49.gydF4y2Ba
Uematsu K, Suzuki N, Iwamae T, Inui M, Yukawa H.增加质体中果糖1,6 -二磷酸醛缩酶促进烟草植物生长和光合作用。中国科学(d辑),2012;gydF4y2Ba
- 50.gydF4y2Ba
Iwaki T, Wadano A, Yokota A, Himeno M. aldolase -在光合作用中控制核酮糖1,5 -二磷酸再生速率的重要酶。植物细胞生理学杂志。1991;32:10 - 91。gydF4y2Ba
- 51.gydF4y2Ba
王LJ。高等植物蔗糖转化酶的研究进展。安徽农业科学,2014;42:8108-11。gydF4y2Ba
- 52.gydF4y2Ba
廖建华,李建民,吴勇。小麦淀粉合酶的研究进展。贵州农业科学,2014;42:18-22。gydF4y2Ba
- 53.gydF4y2Ba
North HM, De Almeida A, Boutin JP, Frey A, To A, Botran L,等。拟南芥aba缺失突变体gydF4y2Baaba4gydF4y2Ba表明胁迫诱导ABA积累的主要途径是通过新黄质异构体。植物J. 2010;gydF4y2Ba
- 54.gydF4y2Ba
王强,文小刚,陆晨明,张启东。超高产水稻华安3号冠层不同衰老叶的光合功能植物生态学报2004;28:39-46。gydF4y2Ba
- 55.gydF4y2Ba
叶绿素a/b结合蛋白。光化学与生物化学。1995;62:367-82。gydF4y2Ba
- 56.gydF4y2Ba
Chourey PS, Nelson OE。酶的缺乏是由gydF4y2Bashrunken-1gydF4y2Ba玉米的突变。生物化学,1976;14:1041-55。gydF4y2Ba
- 57.gydF4y2Ba
侯杰,姜启勇,郝彩春,王永强,张海恩,张晓霞。一个世纪小麦育种中蔗糖合酶单倍型的全球选择。植物物理学报。2014;164:1918-29。gydF4y2Ba
- 58.gydF4y2Ba
蒋启勇,侯杰,郝春春,王丽玲,葛海峰,董玉生,张晓霞。小麦(gydF4y2Bat . aestivumgydF4y2Ba)gydF4y2Ba蔗糖合成酶2gydF4y2Ba基因(gydF4y2BaTaSus2gydF4y2Ba)在胚乳发育中活跃,与产量性状相关。Funct integrg Genomics, 2011; 11:49-61。gydF4y2Ba
- 59.gydF4y2Ba
植物DNA的分离:一种快速、廉价、可靠的方法。植物摩尔生物学报告。1992;10:60-5。gydF4y2Ba
- 60.gydF4y2Ba
Shahinnia F, said - tabatabaei BE。利用dCAPS方法将大麦单核苷酸多态性转化为基于pcr的标记。生物化学学报,2009;32:564-7。gydF4y2Ba
- 61.gydF4y2Ba
Thiel T, Kota R, Grosse I, Stein N, Graner a . SNP2CAPS:用于CAPS标记开发的SNP和INDEL分析工具。核酸研究,2004;32:e5。gydF4y2Ba
- 62.gydF4y2Ba
奈奎斯特,贝克,RJ。植物群体遗传力的估计和选择反应的预测。植物科学学报,2001;10(2):366 - 366。gydF4y2Ba
- 63.gydF4y2Ba
用一种新的计算机程序:连接图构建综合遗传连锁图。植物学报1993;3:739-44。gydF4y2Ba
- 64.gydF4y2Ba
Kosambi DD.从重组值估计映射距离。安优生学,1943;12:17 5 - 5。gydF4y2Ba
- 65.gydF4y2Ba
李海辉,叶勇,王建科。一种改进复合区间映射的改进算法。遗传学。2007;175:361 - 74。gydF4y2Ba
- 66.gydF4y2Ba
王杰。数量性状基因的包涵复合区间作图。作物学报。2009;35:239-45。gydF4y2Ba
- 67.gydF4y2Ba
Schmittgen TD, Livak KJ。用CgydF4y2BaTgydF4y2Ba方法。Nat protocol . 2008; 3:1101-8。gydF4y2Ba
致谢gydF4y2Ba
作者非常感谢悉尼大学植物育种研究所的R. A. McIntosh教授对这篇手稿的严格审查。gydF4y2Ba
资金gydF4y2Ba
国家重点研发计划项目(2016YFD0100502)、国家自然科学基金项目(31571663)、中国农业科学院科技创新计划项目资助。材料的种植和表型鉴定由国家自然科学基金(31571663)和中国农科院科技创新计划资助,基因分型、数据分析和稿件撰写由国家重点研发计划(2016YFD0100502)资助。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
FC、TXL、FLP、XDA、WY和XXT进行了实验和数据分析。DJY为研究创建并提供了DEK线。CSH和ZY设计了实验。FC和CSH撰写了草稿。XXC和HZH对稿件进行了修改。所有作者都阅读并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
不适用。gydF4y2Ba
发表同意书gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们之间没有利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:图S1。gydF4y2Ba
BL31 (a)和BL33 (b)籽粒表型。gydF4y2Ba
附加文件2:图S2。gydF4y2Ba
21条小麦染色体上多态性snp的频率分布。中突出显示有潜在Dek QTL的染色体gydF4y2Ba橙色gydF4y2Ba。频率阈值0.08用一条水平线表示。分布频率是每条染色体上多态性snp占总数的百分比。gydF4y2Ba
附加文件3:表S1。gydF4y2Ba
本研究使用的SSR标记。gydF4y2Ba
附加文件4:表S2。gydF4y2Ba
用于开发CAPS标记的snp。gydF4y2Ba
附加文件5:图S3。gydF4y2Ba
cap标记的设计gydF4y2Baax - 109027972gydF4y2Ba(一),gydF4y2Baax - 108996126gydF4y2Ba(b),gydF4y2Baax - 109301653gydF4y2Ba(c),gydF4y2Baax - 109516011gydF4y2Ba(d)和gydF4y2Baax - 110042240gydF4y2Ba(e).箭头和方框分别显示CAPS标记的目标地点和限制地点的位置gydF4y2Ba。gydF4y2Ba标记的CASP标记的正向和反向引物被标记在特定的目标位点上。gydF4y2Ba
附加文件6:图S4。gydF4y2Ba
PCR分析分子标记,丰富QTL的靶区。(一)gydF4y2Baax - 109027972gydF4y2Ba。(b)gydF4y2Baax - 108996126gydF4y2Ba。(c)gydF4y2Baax - 109301653gydF4y2Ba。(d)gydF4y2Baax - 109516011gydF4y2Ba。(e)gydF4y2Baax - 110042240gydF4y2Ba。M, Marker (DL2000DNA阶梯a, b, c和e;20bp DNA ladder in d;Takara生物公司)。目标片段用红色箭头显示gydF4y2Ba。gydF4y2Ba1、2车道分别为BL33、BL31;第三道显示FgydF4y2Ba1gydF4y2Ba植物;4-10车道为F车道gydF4y2Ba2gydF4y2Ba个人的植物。gydF4y2Ba
附加文件7:表S3。gydF4y2Ba
BL31和BL33的TGW比较gydF4y2Ba。gydF4y2Ba
附加文件8:图S5。gydF4y2Ba
采用包涵复合区间作图(ICIM)分析F染色体3B上TGW QTL的LOD等高线gydF4y2Ba2gydF4y2Ba和FgydF4y2Ba2:3gydF4y2BaBL33/BL31衍生的种群。基因区gydF4y2BaQTGW.caas-3BSgydF4y2Ba中突出显示。gydF4y2Ba红色的gydF4y2Ba。高义2016-2017年种植季和2017-2018年种植季LOD等值线为gydF4y2Ba红色的gydF4y2Ba和gydF4y2Ba绿色gydF4y2Ba分别行。LOD阈值2.5由虚线垂直表示。gydF4y2Ba
附加文件9:图S6。gydF4y2Ba
BL31和BL33成熟籽粒淀粉含量的比较。gydF4y2Ba
附加文件10:图S7。gydF4y2Ba
开花后籽粒蔗糖含量的变化。gydF4y2Ba
附加文件11:表S4。gydF4y2Ba
QTL物理区间标注的基因。gydF4y2Ba
附加文件12:表S5。gydF4y2Ba
发育籽粒中候选基因的转录模式。gydF4y2Ba
附加文件13:图S8。gydF4y2Ba
发育籽粒中四个候选基因的转录模式。gydF4y2Ba
附加文件14:图S9。gydF4y2Ba
PCR分析gydF4y2BaWx-B1gydF4y2Ba特定的标志。M, Marker (DL2000DNA阶梯;Takara生物公司)。目标片段用红色箭头显示gydF4y2Ba。gydF4y2Ba1巷为中国春天;2巷表示关东107;车道3和车道4分别为BL33和BL31。gydF4y2Ba
附加文件15:表S6。gydF4y2Ba
用于实时定量PCR的引物。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文根据创作共用属性4.0国际许可协议(gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)除另有说明外,适用于本条所提供的资料。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
傅,春,杜,杰,田,欣。gydF4y2Baet al。gydF4y2Ba面包小麦籽粒缺陷遗传位点的快速鉴定与鉴定。gydF4y2BaBMC植物生物学gydF4y2Ba19日,gydF4y2Ba483(2019)。https://doi.org/10.1186/s12870-019-2102-6gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-019-2102-6gydF4y2Ba
关键字gydF4y2Ba
- 有缺陷的内核gydF4y2Ba
- 灌浆期gydF4y2Ba
- 快速QTL定位gydF4y2Ba
- SNP数组gydF4y2Ba
- 小麦属植物aestivemgydF4y2BalgydF4y2Ba