- 研究文章
- 开放访问
- 发表:
野生花生中磷脂酰乙醇胺结合蛋白的分子和转录特性arachis duranensis.和arachis ipaensis.
BMC植物生物学体积19., 文章编号:484.(2019年)
摘要
背景
磷脂基乙醇胺结合蛋白(PEBPS)参与了植物建筑和开花时间的调节。职能PEBP在许多植物物种中研究了基因。但是,关于特征和表达型材的知名知之甚少PEBP野生花生种类的基因,arachis duranensis.和arachis ipaensis.那栽培花生的二倍体祖先。
结果
在该研究中,使用基因组鉴定方法来鉴定和表征总共32个花生PEBP两种野生花生各16个基因,A. Duranensis.和A. iPaensis..这些PEBP基因分为3组(终端FLOWER1-像,开花轨迹T.- 它和ft和tfl1的母亲基于它们的系统发育关系。这些基因结构,图案和它们中的每一个的染色体位置PEBPs分析了。此外,还确定了4个同胞变性重复和1个串联复制A. Duranensis.和2个中间瘤细胞糖尿病和1个串联帕拉拉在A. iPaensis..九十五不同独联体-作用元素在PEBP基因启动子区域和大多数基因具有不同的数量和类型独联体元素。因此,它们的转录模式PEBP基因在不同的组织中和在不同生长阶段的漫长一天和短日情况下变化,表明功能多样性PEBPs在不同组织中及其在植物光周期依赖性发育途径中的潜在功能。此外,我们的分析显示AraduF950M/AraduWY2NX在A. Duranensis., 和ARAIP344D4/Araip4V81G在A. iPaensis.是用于调节植物建筑的良好候选人,以及Aradu80YRY AraduYY72S,和AraduEHZ9Y在A. Duranensis.和AraipVEP8T在A. iPaensis.可能是调控花期的关键因子。
结论
十六PEBP从两个二倍体野生花生基因组中分别鉴定和表征了基因,A. Duranensis.和A. iPaensis..遗传特征和时空表达分析表明它们在植物生长发育中的重要作用。这些发现进一步加深了我们对PEBP植物物种的基因功能。
背景
开花时间是影响植物后代产量的关键因素。因此,植物已经进化了复杂的系统,该系统调节从植物生殖阶段的过渡。开花时间的精确调整由内部和环境提示控制[1那2那3.那4.].已经鉴定了几种调节开花时间的途径,例如光周期,春化,葛根酸和自主途径[1那2那5.那6.那7.].此外,许多分子调控成分已被证明参与这些开花调控途径。例如,许多MADS-box家族成员和磷脂酰基乙醇胺结合蛋白(pebp)已经被证明参与了从茎尖分生组织到花序分生组织的转换[1那8.那9.].
这PEBP基因家族是一组古老、保守的基因,编码蛋白质,在所有真核生物界(包括细菌、动物和植物)中高度相似[10.那11.那12.].pebp是通过其与磷脂酰乙醇胺脂相结合的偏好而识别的[13.].以前对功能的研究PEBP基因的发现PEBPs编码在许多物种中调节生长和分化的多种信号通路中涉及的蛋白质[12.那14.那15.].在植物中,PEBP基因主要参与开花时间和工厂建筑规定[11.那16.那17.].许多最近的研究已经调查了个体PEBP然而,基因是完整的PEBP科仅在少数植物物种中进行了研究,如拟南芥、大豆、水稻、玉米和棉花[8.那18.那19.那20.].
在植物中,PEBP家庭基因一般分为三组:终端FLOWER1(TFL1)式,开花轨迹T.(英国《金融时报》)式,ft和tfl1的母亲(MFT.) - 般的子家庭[1那8.那18.那21.那22.].在拟南芥中,英国《金融时报》- 类似的子家庭包含两个成员,英国《金融时报》和ft的孪生姐妹(TSF.),两者都促进从植物转变为生殖增长[1那23.那24.].英国《金融时报》是一种生物钟基因,其蛋白质已被证明从叶向茎尖分生组织移动,并与转录因子FD相互作用,加速开花[25.那26.那27.那28.那29.].TSF.的最接近的同系物英国《金融时报》,具有类似的功能。过度的TSF.促进开花tsf突变体具有延迟开花时间表型[1那30.].这TFL1麦克/地区有三名成员,TFL1那拟南芥CENTRORADIALIS(ATC.),英国《金融时报》的兄弟(BFT),所有这些都据报道延迟开花时间。TFL1参与花期和植物结构的调节。突变的TFL1导致早花期和确定的生长习性[1那31.].ATC.是短日诱导的花卉抑制剂,其蛋白质通过脉管系统到射击顶点来影响开花时间[1那32.].BFT显示傍晚峰值的日振荡表达式模式,类似于英国《金融时报》.过度的BFT导致延迟开花时间和严重的花卉缺陷,而倒闭BFT对开花时间没有影响。因此,BFT在确定开花时间的决定中冗余工作[1那33.].这MFT.-like子组只包含一个成员,MFT.,这有弱点英国《金融时报》- 类似的活性,主要参与种子萌发信号途径[34.那35.].
花生是世界各地的重要油豆科植物。栽培的花生是同种异体一体化(AABB,2N= 4X= 40),并被认为是源于两个二倍体物种的杂交和多倍体化,A. Duranensis.(AA Genome)和A. iPaensis.(BB Genome)[36.那37.那38.那39.].改善工厂建筑和开花时间可以帮助增加花生生产。功能基因的分析有助于鉴定可以对花生品种的修饰来增加产量。对野生花生基因的调查将提供培养花生基因的进一步函数表征的基本信息[40那41.].在这项研究中,PEBP对两种野生花生的基因进行了鉴定和特征分析。A. Duranensis.和A. iPaensis..这些的许多特征PEBP基因分析包括基因进化关系、基因结构、保守基序和基因表达模式。我们的发现将使进一步的特性PEBP花生的基因和蛋白质功能。
方法
植物材料和生长条件
荒野A. Duranensis.PI219823和A. iPaensis.PI468322物种用于基因表达分析。花生种子在自来水中发芽,然后在具有不同光周期的生长室的盆中种植。将生长条件设定为16小时24°C光/ 8小时24°C暗和10小时24°C光/ 14小时24°C漫长的一天和短日光周期。湿度约为30%。在不同的生长阶段发光后,植物叶子被取样2小时。阶段1至第6阶段(S1-S6)被认为是前两个,第三,第四,第五,第六和第七叶的阶段是完全扩展的。确认表达水平PEBP不同组织中的基因,栽培花生Tiffrunner和野生花生在青岛,中国的田地中生长,并收集了不同组织进行分析。在RNA提取之前将样品储存在-80℃。
识别的花生PEBP成员
PEBP保守结构域(PF01161)的氨基酸序列和来自拟南芥的PEBP氨基酸序列[1]和大豆[8.]作为对花生基因组数据库(https://www.peanutbase.org/).使用PFAM数据库进行分析所有输出基因(http://pfam.xfam.org/search)和国家生物技术信息(NCBI)中心,以确认保守的PEBP领域。没有保守域序列的基因被丢弃。使用protparam测定蛋白质分子量和理论异等物电点(https://web.expasy.org/protparam/).使用ProtComp工具预测pebp的亚细胞定位(http://linux1.softberry.com/berry.phtml?topic=protcomppl&group=programs&subgroup=proloc).
系统发育关系分析
来自两种野生花生、栽培花生、拟南芥、大豆、普通大豆和苜蓿的PEBP全长氨基酸序列[8.使用Clustal-X2对齐。对准结果用于使用Mega 7中的邻接方法构建系统发育树[42.那43.].
分析PEBP基因结构和保守的主题
野生花生的外显子内组织PEBPs使用基因结构显示服务器程序(GSD)使用从花生基因组数据库获得的编码域序列(CD)和基因组序列来确定[44.].使用MEME工具分析花生PEBP保守的主题(http://meme-suite.org/)以下参数:最大数量为15个图案和6-50个氨基酸残基的最佳主题宽度。
染色体定位、基因复制、共同和独联体表演元素
为了分析染色体分布,PEBP从花生基因组数据库中获得基因位置,并将其映射到物理染色体位置。根据Zhang等人的描述,大豆、普通大豆和花生之间进行了共序分析。[45.].利用OrthoMCL软件对花生PEBP序列进行聚类,利用Circos软件进行同源性分析[46.那47.].这独联体-花生的作用成分PEBPPlantCare预测基因(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)[48.,利用启动子区上游2 kb的翻译起始密码子PEBP基因。
亚细胞定位分析
代表性野生花生的亚细胞本地化PEBP如Li等人所述分析基因。[49.].每一个完整的PEBP基因和GFP扩增,克隆到修饰的pCAMBIA1300载体上。然后这些构念被瞬时表达烟草benthamiana用农杆菌介导的渗透法处理。
RNA提取和表达分析
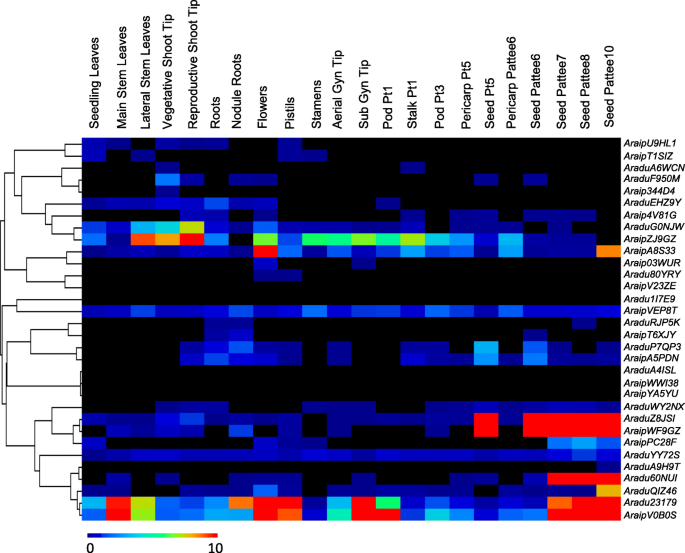
如Li等人所述进行RNA提取和定量实时PCR(QRT-PCR)。[49.].简单地说,用RNeasy迷你试剂盒(Qiagen)分离植物总RNA。使用SuperScript II逆转录酶(Promega)和1.5 μg总RNA合成第一链cdna。使用LightCycler480 SYBR Green I Master Kit (Roche Diagnostics)在LightCycler480机器(Roche Diagnostics)上进行qRT-PCR。qRT-PCR扩增程序如下:94℃10 s, 58℃10 s, 72℃10 s, 40个循环。基因表达量归一化至野生花生肌动蛋白表达基因(Araduw2Y55.要么ARAIPFY50U.).对于栽培花生中的基因表达分析,基因表达水平被标准化为肌动蛋白-表达基因,如Sui等人所述[50.].使用三种生物重复分析每个样品。本研究中使用的所有引物列于其他文件中10..确定转录模式PEBPRNA-seq数据集取自花生数据库(https://peanutbase.org/gene_expression.).这22个组织被Clevenger等人鉴定为[51.],详情如下:‘幼苗叶’(出苗后10天的幼苗叶),‘主茎叶’,‘侧茎叶’,‘营养茎尖’(来自主茎),‘生殖茎尖’(来自第一个侧叶),‘根’(10日生根),‘根瘤根’(25日生根瘤),‘花’(花被),‘雌蕊’(雌蕊),‘雄蕊’(雄蕊),“空中妇科提示”(空中雌蕊柄尖),“子妇科提示”(地下雌蕊柄尖),“PodPt1”(帕蒂阶段1 pod),“StalkPt1”(帕蒂阶段1杆),“PodPt3”(帕蒂第三阶段pod)的果皮Pattee5”(帕蒂阶段5果皮),“种子Pattee5”(帕蒂阶段5种子),“果皮Pattee6”(帕蒂阶段6果皮),“种子Pattee6”(帕蒂阶段6种子),'种子Pattee7 '(帕蒂第7阶段种子),'种子Pattee8 '(帕蒂第8阶段种子),和'种子Pattee10 '(帕蒂第10阶段种子)。这A. hypogaea.基因表达谱被定位到A. Duranensis.和A. iPaensis.热图分析[51.那52.那53.].
结果
的识别PEBP两种野生花生种类的基因
识别PEBP在野生花生物种中表达的基因A. Duranensis.和A. iPaensis.,PEBP保守结构域(PF01161)的氨基酸序列(PF01161)和来自拟南芥和大豆的全长PEBP蛋白序列用作针对花生基因组数据库的爆炸询问。然后使用PFAM和NCBI工具确认这些候选人中的保守PEBP域PEBP基因。总共,32PEBP从两个野生花生基因组中鉴定基因(表1).多个PEBP使用基因组,Cd和氨基酸序列分析基因特性(表1).野生花生的基因组长PEBP基因范围从989 BP(AraduP7QP3)至8770bp (AraduWY2NX), CDS长度为327 bp (AraduWY2NX)至630 bp(Aradug0njw),推测氨基酸数目为108 ~ 209。预测每个PEBP的分子量和等电点(由于缺乏相关信息,将AraduG0NJW丢弃)。分子量为12,058.74 ~ 20,482.26 kDa,等电点为4.93 ~ 9.68。此外,对PEBP的亚细胞定位进行了预测。未发现Aradu23179、Aradu60NUI或AraipV0B0S的定位信息.所有其他野生花生pebp均定位于细胞质和细胞核(见表)1),类似于大豆pebp [8.].几个PEBP的亚细胞本地化A. Duranensis.,包括AraduWY2NX、AraduYY72S和AraduQIZ46A. iPaensis,鉴定包括ARAIP4V81G,ARAIPVEV8T和ARAIPA8S33。结果证实了它们在细胞质和核中的亚细胞局部(附加文件1).
染色体定位分析
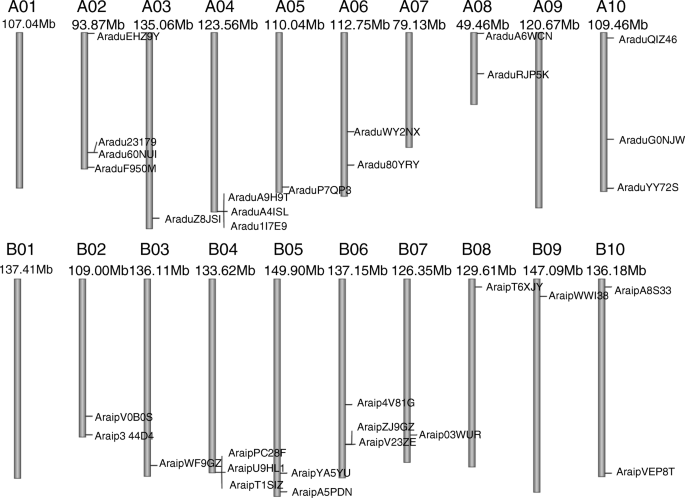
确定野生花生的染色体位置PEBPs, 这PEBP利用花生基因组数据库将基因定位到相关染色体位置。1).染色体分布图显示了16个染色体的位置PEBP两种野生花生的基因(图。1和表格1).对于AA基因组野生物种,PEBP基因分布在10条染色体中的7条上PEBP在染色体1,7或9上发现基因。染色体2含有最多PEBP基因,总共4(图。1).相比之下,BB野生花生基因组的10条染色体中有9条含有PEBP基因,除染色体外。染色体4和6含有最多PEBPBB基因组中的基因,3PEBP基因(图。1).在这些当中PEBP基因,大多数位于染色体臂中(图。1).只有四个基因,包括Araduwy2nx,Aradurjp5k,Aradug0njw,和Araip4V81G的染色体靠近中间(图。1).
分类和系统发育分析PEBP基因
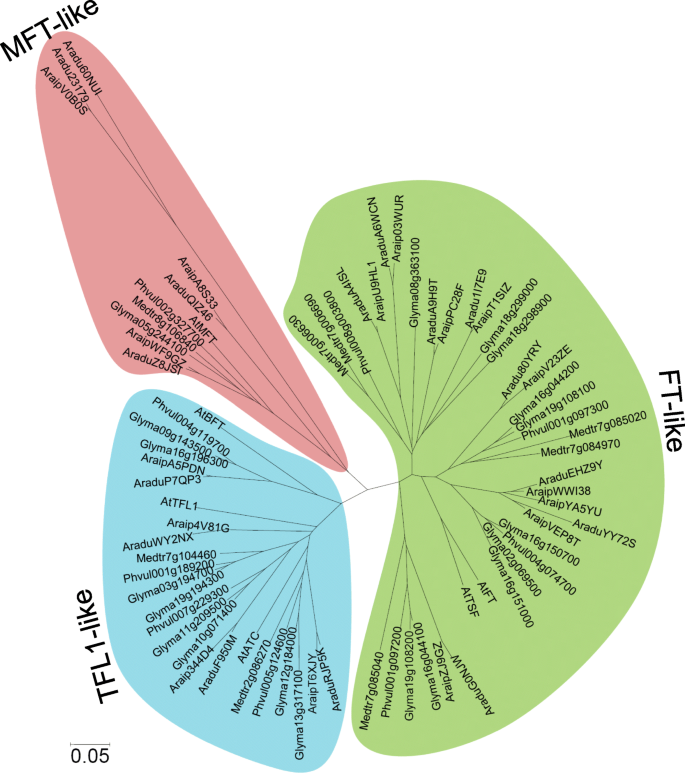
在植物中,PEBP基因可以分为3个亚家族,TFL1-像,英国《金融时报》- 它和MFT.- 根据他们的基因结构[1那8.].分类野生花生PEBP将基因分为不同的亚家族,并根据研究充分的同源性推断信息PEBP来自其他植物物种的基因,构建了系统发育树PEBP来自野生花生,拟南芥,大豆,常见豆和Medicago的基因[8.].系统发育关系分析分类为野生花生PEBP基因进入3个子家庭(图。2和表格1).在这些成员中,8PEBP基因属于TFL1- 般的子家庭,17名成员被分组成了英国《金融时报》- 般的子家庭和7PEBPs被归为MFT.- 般的子家庭。AA基因组包含8个英国《金融时报》- 它,4.TFL1- 它和4MFT.- 样基因。相比之下,BB基因组有9英国《金融时报》- 它,4.TFL1- 它和3MFT.这可能反映了AA和BB基因组中基因的功能分化。由于栽培的花生是一种异源四倍体,由杂交和多倍体衍生而来A. Duranensis.和A. iPaensis.,栽培花生(A. hypogaea.)PEBP基因也进行了调查PEBP成员被确定(附加文件2).系统发育分析表明A. hypogaea.基因组含有8TFL1- 它,16.英国《金融时报》- 它和7MFT.类成员(附加文件2).
在野生花生AA和BB基因组中发现了许多同源基因对[40那41.那54.那55.].因此,直观PEBP利用野生花生PEBP氨基酸序列生成的系统发育树(图。3.一种)。十五否tthologousPEBP基因对(全部除外araipya5yu和Aradu60nui.)被归为同一类(图。3.一种)。大多数基因对具有高度相似的CD和蛋白质序列(图。3.一个和表2),表明同源基因具有相似的功能。染色体定位分析表明,15对同源基因中有11对位于同一位点A. Duranensis.和A. iPaensis.染色体(图。1和表格2).然而,AraduEHZ9Y那AraduA6WCN,和Aradug0njw与相关的同源基因位于不同的染色体上A. iPaensis..Aradurjp5k.位于染色体8的中间部分,而其正交基因,AraipT6XJY,位于染色体臂(图。1和表格2).这表明二倍体花生基因组中可能发生过染色体重排[54.那56.].
的外显子-内含子结构和保守结构域PEBP基因
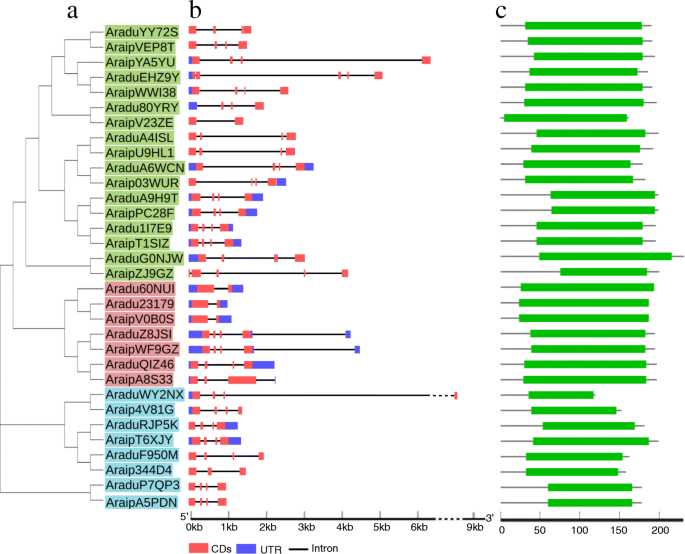
经典PEBP成员具有保守的4-外显子结构[19.].以确定野生花生的基因结构PEBP基因,基因结构显示服务器程序用于调查PEBPexon-intron组织(44.].我们的调查结果显示32中的23个PEBP成员的基因结构为4外显子保守,9个基因的组织结构不一致(图2)。3.b).对于这9个基因英国《金融时报》- 麦克风AraduEHZ9Y那Aradu80yry.那araipv23ze., 和AraipZJ9GZ分别包含5、3、2和5个外显子。这MFT.- 麦基基因Aradu60nui.那Aradu23197那Araduv0b0s,和AraipA8S33分别有2、2、2和3个外显子,TFL1例如基因ARAIP344D4有3个外显子(图。3.b).此外,对每个野生花生PEBP蛋白的PEBP结构域进行了分析,发现PEBP结构域的长度超过了每个PEBP蛋白的一半,除了AraipZJ9GZ(图)。3.C和附加文件3.).为了进一步分析PEBP的结构,我们对PEBP蛋白的保守基序进行了研究。在32种野生花生pebp中发现了15种不同的基序(图1)。4.和额外的文件4.).但在所有PEBP蛋白上均未发现单一motif,说明其功能存在多样性。
分析PEBP基因复制和同步
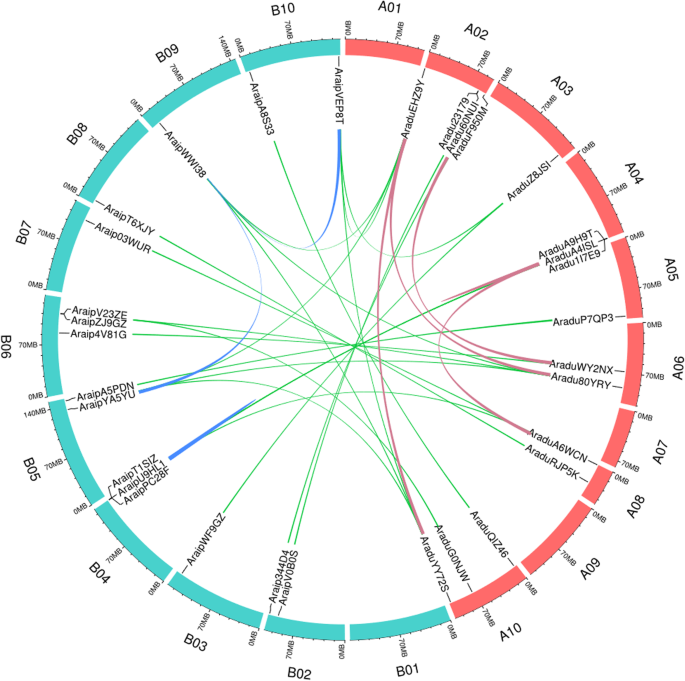
多倍性在开花植物中很常见,基因复制常发生在基因进化过程中[57.,对野生花生基因组进行同源性分析。在这两种基因中都发现了许多副同源基因A. Duranensis.和A. iPaensis.(无花果。5.).4个染色体间重复和1个串联重复A. Duranensis..三染色体间重复,Araduyy72s / Araduehz9y.那Aradu80YRY / AraduEHZ9Y, 和Aradua6wcn / Aradua4isl.,属于英国《金融时报》例如sub-family和AraduWY2NX / AraduF950M属于TFL1-subfamily。串联复制由三个基因组成:AraduA9H9T那AraduA4ISL,和Aradu1i7e9..两个中间瘤细胞糖尿病,ARAIPWWI38 / ARAIPVEP8T.和ARAIPWWI38 / ARAIPYA5YU,发现一个串联重复A. iPaensis..这两对重复的基因属于英国《金融时报》-like子类,串联帕拉戈尔载有三名成员:Araippc28f.那AraipT1SIZ,和araipu9hl1..此外,同源性分析也在介于之间A. Duranensis.和A. iPaensis.鉴定了25个同源基因对。在这些基因对中,串联重复AraduA9H9T那AraduA4ISL,和Aradu1i7e9.在A. Duranensis.被发现与串联副鸟同源Araippc28f.那AraipT1SIZ,和araipu9hl1.在A. iPaensis.(无花果。5.).
拟南芥TFL1调节植物建筑及其在豆类中的同源基因是具有保守功能的混合性[1那58那59那60那61].探讨野生花生的潜在功能TFL1般的基因,围绕大豆生长习性基因的基因组区域GMDT1(Glyma19G194300),普通豆类生长习性基因PvTFL1y (Phvul001g189200)和野生花生TFL1与同源物相比,以确定它们是否是Syntelogs。野生花生基因之间的同步AraduF950M那AraduWY2NX那ARAIP344D4那araip4v81g,大豆GmDt1和常见的豆子PvTFL1y在所有地区发现(附加文件)5.),表明这些基因可能是同源基因,这些野生花生基因可能参与植物结构调控。
分析独联体-野生花生的作用成分PEBP基因启动子区
调查野生花生的调节PEBP基因表达,独联体分析了翻译起始密码子上游2 kb启动子区域的作用元件。总共有95种独联体- 在这些上游发现元素PEBP其中56个被预测具有推定功能,包括发育相关元件7个,环境胁迫相关元件4个,激素响应元件10个,光响应元件26个,启动子相关元件5个,位点结合相关元件4个(表)3.和额外的文件6.).在这些功能中独联体- 为元素,光响应元素是每个最丰富的类型PEBP启动子,以及所有的PEBP基因含有激素响应元素,光响应元件和启动子相关元素,表明所有的PEBPs参与相关的信号传导途径(表3.).此外,在所有32个野生花生中发现了启动子相关元素塔塔盒PEBP推动者区域,表明TATA盒是表达的必要条件PEBP基因。三十一PEBP促销者包含光响应元件盒4,除了英国《金融时报》例如基因araipya5yu(附加文件6.).此外,还有类型和数量独联体-作用要素在各区域间呈现出不同的分布PEBP启动子(表格3.和额外的文件6.),建议PEBP基因在功能上是不同的,可能在不同的信号通路中起作用。
野生花生PEBP多种组织中的基因表达模式
为进一步研究野生花生的潜在功能PEBP基因,PEBP利用该数据集分析了22个不同组织的基因转录模式A. hypogaea.基因表达映射到A. Duranensis.和A. iPaensis.[51.那52.].表达水平随机选取几个PEBP在几个野生花生和培养的花生组织中检查基因,发现它们的表达模式类似于公布的数据集(附加文件7.).许多野生花生PEBP基因显示组织特异性表达谱。例如,所有的MFT.类成员,包括4AdPEBP成员(Aradu60nui.那AraduQIZ46那Aradu23179, 和Araduz8jsi.)和3AiPEBP基因(AraipV0B0S那AraipWF9GZ和AraipA8S33),在种子中含量很高,表明它们可能在种子的生长发育中起作用(图。6.).这英国《金融时报》例如基因AraipZJ9GZ显示侧茎叶,营养射击提示和生殖射击提示的高表达水平。相比之下,5英国《金融时报》类成员,包括2AdPEBP基因(Aradu1i7e9.和Aradua4isl.)和3.AiPEBP基因(araipwwi38,araipya5yu,和araipv23ze.),在所有测试组织中低水平表达(图。6.),建议他们在开花时间监管中可能具有弱或无功能。还研究了重复的基因对表达,并且一些重复的基因对在一些组织中显示出类似的表达模式(图。6.).例如,Aradu80yry.和AraduEHZ9Y在花中显示出类似的表达水平,和AraduWY2NX和AraduF950M在生殖射击尖端中彼此非常相似,表明它们可能在这些组织中具有类似的功能。相反,一些重复的基因对显示出明显的表达模式。例如,Aradua6wcn.在营养芽尖端和茎Pt1的高水平表达,而其重复的基因,AraduA4ISL,所有测试组织中的表达水平低(图。6.),表明在进化期间的功能分歧。因为许多野生花生PEBP基因与栽培花生同源PEBP基因(附加文件8.),比较直际基因表达水平.许多同源基因表达水平相近。例如,AraipYA5YU / ArahyFW8Z6T那AraipWWI38 / Arahy5H2LSK,和Aradu1i7e9 / Arahyxgva1e.在大多数被测试的组织中有相似的表达模式(附加文件9.).
表达概况为32PEBP来自两种野生花生22个不同组织的基因。22个花生组织(由Clevenger [51.]使用。用于热图分析,A. hypogaea.基因表达被映射到A. Duranensis.和A. iPaensis., 分别
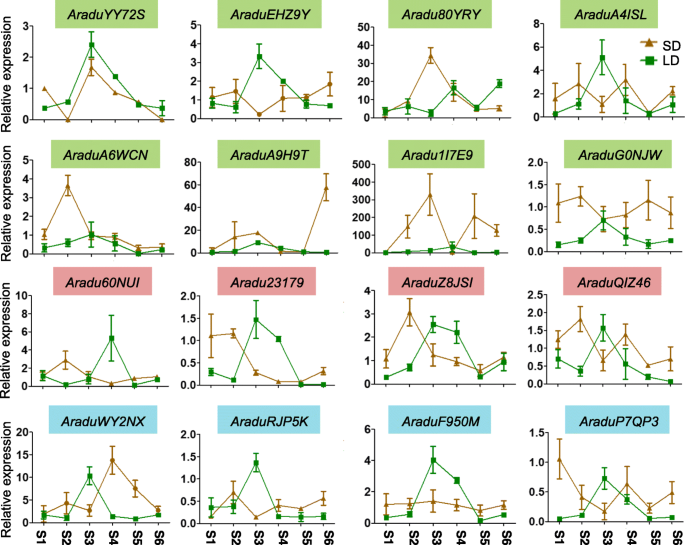
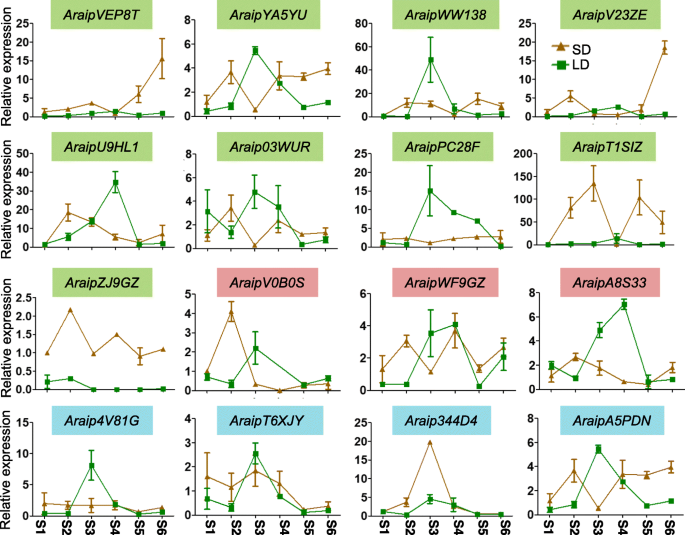
野生花生的表达分析PEBPs不同生长期长日照和短日照条件下
许多light-responsive独联体在野生花生中发现了-作用元素PEBP推动者区域。就这样PEBP在不同的生长阶段在不同光周期下的野生花生中研究了基因表达。在一些生长阶段,不同的光周期改变了大多数的表达式模式PEBP基因(图。7.和8.),表明它们在发育相关阶段的光响应中发挥重要作用。一些PEBP基因在漫长的一天条件下,在所有生长阶段都显示出一致的表达水平,包括Aradua6wcn.那Aradua9h9t,和Aradu1i7e9.在A. Duranensis., 和AraipVEP8T那araipv23ze.那AraipT1SIZ,和AraipZJ9GZ在A. iPaensis.,所有这些属于英国《金融时报》例如sub-family(无花果。2那7.和8.).在短昼光周期下,AraduF950M那AraipPC28F,和Araip4V81G在不同的生长相中展示了一致的表达水平(图。7.和8.).此外,表示AraipZJ9GZ在短的一天条件下比所有测试生长阶段的长期条件更高(图。8.).在长日照条件下,一些重复的基因对在整个生长阶段表现出相似的表达模式,如Araduyy72s / Araduehz9y.和ARAIPWWI38 / ARAIPYA5YU(无花果。7.和8.),建议这些重复的基因可以在功能上是多余的。
讨论
描述的PEBP基因极大地增加了我们对许多植物物种中调控开花时间和植物结构的分子机制的认识[8.那18.那19.那20.].花生是全世界重要的油籽作物,以及鉴定花生PEBP基因有助于我们进一步了解花生开花时间和植株结构调控的功能。此外,对野生花生的调查PEBP基因为花生栽培相关基因的进一步功能表征提供了必要的信息[40那41.].在我们的研究中,有32个PEBP对两个野生花生基因组进行了基因鉴定。
不同的植物物种具有不同的数量PEBP基因(8.那18.那19.].两种二倍体野生花生,A. Duranensis.和A. iPaensis.,有16个PEBP基因(表1).旨在源自两倍类二倍体野生物种的杂交和多倍化的同种异体一种覆盖的花生,以及基因组大小A. hypogaea.是接近的和A. Duranensis.和A. iPaensis.基因组[36.那37.那38.那39.].这A. hypogaea.基因组含有31.PEBP会员(附加文件2),这也接近了这一总和PEBP基因在A. Duranensis.和A. iPaensis.基因组。这两种野生花生基因组含有8TFL1- 它,17英国《金融时报》- 它和7MFT.- 状成员(图。2和表格1),虽然A. hypogaea.基因组包含8TFL1- 它,16.英国《金融时报》- 它和7MFT.类成员(附加文件2).可能是其中之一英国《金融时报》- 般的基因A. hypogaea.在进化过程中消失了。此外,拟南芥、水稻、大豆和玉米分别有6个、19个、23个和24个PEBP成员,分别8.那18.那19.].基因组大小A. Duranensis.那A. iPaensis.那a . hypogaea拟南芥,大米,大豆和玉米是1.25 GB,1.56 GB,2.7 GB,125 MB,466 MB,1.1 GB和2.3 GB,分别[40那62那63那64那65].因此,这些植物中的基因组尺寸与数量不相关PEBP基因。
基因复制在植物中尤为普遍,为新的基因功能提供了资源。一些复制的基因变成假基因,没有功能,而其他的基因复制对进化出新的功能[8.那66].例如,重复PEBP由于关键氨基酸的改变,大豆中的基因在功能上发生了分化[8.].4个染色体间重复和1个串联重复A. Duranensis.,发现2个染色体间重复和1个串联重复A. iPaensis.(无花果。5.).部分野生花生复制基因对表达模式相似,而其他基因对在不同组织或生长阶段的表达水平不同。6.那7.和8.).例如,Aradu80yry./AraduEHZ9Y和AraduWY2NX/AraduF950M这些基因在生殖茎尖的表达量相近,说明这些基因在功能上是保存的。相反,表达Aradua6wcn.在营养射击尖端和茎PT1中很高,而其复制品,AraduA4ISL,在这两个组织中有低表达水平(图。6.),表明这些复制的功能有分歧PEBP基因。
在这些当中PEBP成员,英国《金融时报》和TFL1在许多物种中都很顺利[1].花期的调节由英国《金融时报》已经受到了高度调查。这英国《金融时报》- 麦基基因Aradu80YRY AraduYY72S,和AraduEHZ9Y在A. Duranensis., 和AraipV23ZE、AraipVEP8T AraipYA5YU,和AraipWWI38在A. iPaensis.,在拟南芥中高度保守英国《金融时报》和TSF.基因(图。2).然而,araipv23ze.那araipya5yu,和AraipWWI38在所有测试组织中具有非常低的表达水平(图。6.).可能就是这样Aradu80YRY AraduYY72S,和AraduEHZ9Y在A. Duranensis.和AraipVEP8T在A. iPaensis.对花期的调控有重要作用。TFL1是参与植物构型调控的重要基因之一。同源的基因TFL1已被证明可以参与植物建筑规则在许多豆类中,如大豆(GmDt1),常见的豆(PvTFL1y)和绿豆(VRDET1.),这三个基因是Syntelogs [1那58那59那60那61].同步之间AraduF950M/AraduWY2NX那ARAIP344D4/Araip4V81G, GmDt1, 和PvTFL1y观察到A. Duranensis.和A. iPaensis.,分别(附加文件5.),表明它们在植物结构调控中起作用。只有一个TFL1在大豆,常见的豆类和床上涉及植物建筑监管的基因。虽然两个基因具有与大豆同步GmDt1和菜豆PvTFL1y在A. Duranensis.和A. iPaensis.分别在两种野生花生种中,只有一个关键构件也只有一个涉及的植物架构调节。MFT.通过脱落酸和赤霉酸途径在种子萌发中起重要作用[34.那35.].在野生花生,建议-就像PEBP成员在种子中的表达水平高于其他组织(图。6.),表明这些基因可能在种子发育和种子萌发途径中发挥关键作用。未来的工作需要充分阐明的参与PEBP在开花时间和植物建筑学调节途径的基因在野生花生。
独联体- 为元素是结合转录因子和活性基因表达的重要因素。花生PEBP发现启动子区域含有各种各样的顺式作用元件包括发育元件、环境胁迫相关元件、激素响应元件、光响应元件、启动子相关元件、位点结合相关元件(表)3.和额外的文件6.).这表明了PEBP基因可能在这些信号途径中具有重要作用。此外,表达模式PEBP基因在不同的组织和不同的生长阶段是不同的。6.那7.和8.),进一步支持功能差异。全部PEBP基因包含光响应元件,大部分花生PEBP基因在漫长的日期与短日生长条件下表现出改变的表达模式(图。7.那8.和表格3.),表明它们可能通过光周期依赖性途径参与植物开发。虽然大多数英国《金融时报》- 样基因含有这6种功能独联体- 元素元素(表3.和额外的文件6.),10英国《金融时报》在长期条件下,在不同组织或在生长阶段的不同组织中具有低表达水平或不变的表达水平(图。6.那7.和8.).这可能是由于这些基因在长日照条件下,在这些组织或生长阶段的功能较弱或没有作用。一些PEBP成员,比如英国《金融时报》例如基因(1那23.那49.,是生物钟基因,可能在本研究未检测的一天中的其他时间表达。此外,英国《金融时报》例如基因AraipZJ9GZ在短日情况下表现出更高的表达水平(图。8.),建议AraipZJ9GZ参与在短日短日落的开花时间监管,而不是漫长的一天条件。总而言之,变异独联体-element数量和基因表达模式英国《金融时报》- 般的基因可能反映出他们在开花时间监管中的功能多样性。TFL1- 克里MFT.-像PEBP基因可能在其他功能途径中类似地不同。另外,许多野生花生PEBP基因与栽培花生基因同源(附加文件)8.),因此,野生花生的表达模式PEBP基因可以用来推断PEBP基因表达A. hypogaea.在漫长的一天和短日条件下的生长阶段。
结论
用全基因组分析方法鉴定和表征了16PEBP来自两个二倍体野生花生种类的基因,A. Duranensis.和A. iPaensis..这些的许多特征PEBP研究了基因,包括染色体分布,基因结构和基序。32.PEBP基因被分为TFL1-像,英国《金融时报》- 它和MFT.- 类似的子家庭。在野生花生物种中鉴定了间同胞形式重复的基因对和串联复制事件。此外,还发现了四种可能在植物架构中发挥重要作用的四种基因,并且可以识别出可能调节开花时间的另外四个。九十五独联体-作用元件被鉴定,其中56个具有假定的功能,可能负责组织和光周期表达模式的差异。详细了解野生花生PEBP基因对于未来的努力将有助于修改花生植物开花时间和植物建筑。
可用性数据和材料
在当前研究期间生成和分析的数据集可从相应的作者获得合理的请求。
缩写
- 答:(duranensis
-
arachis duranensis.
- A. iPaensis:
-
arachis ipaensis.
- AA:
-
氨基酸
- 空中交通管制:
-
拟南芥蒂利亚纳Centroradialis.
- BFT:
-
英国《金融时报》的兄弟
- CD:
-
编码域序列
- 装备:
-
染色体数目
- 英国《金融时报》:
-
开花轨迹T.
- 通用汽车:
-
甘氨酸最大
- GSDS:
-
基因结构显示服务器程序
- LD:
-
漫长的一天;SD,短短
- 建议:
-
ft和tfl1的母亲
- Mol.Wt:
-
分子量
- n / a:
-
不适用
- NCBI:
-
国家生物技术信息中心
- PEBP:
-
ethanolamine-binding磷脂蛋白
- PI:
-
等电点
- Pv:
-
phoudolusulus vulgaris.
- QRT-PCR:
-
定量实时聚合酶链反应
- S1:
-
阶段1
- S2:
-
第2阶段
- S3:
-
第3阶段
- S4:
-
第4阶段
- S5:
-
第5阶段
- S6:
-
第6阶段
- TFL1:
-
终端FLOWER1
- TSF:
-
ft的孪生姐妹
参考文献
- 1。
Wickland DP,Hanzawa Y.的开花基因座T /终端花1基因家族:功能进化和分子机制。莫尔植物。2015; 8(7):983-97。
- 2。
徐S,Chong K.通过vernalisation记住冬天。Nat植物。2018; 4(12):997-1009。
- 3.
Beinecke FA, Grundmann L, Wiedmann DR, Schmidt FJ, Caesar AS, Zimmermann M, Lahme M, Twyman RM, Prufer D, Noll GA。白天中性种烟草(nictiana tabacum)在长日照条件下依赖FT/ fd的开花期起源于兼性短日照祖先绒毛状烟草(Nicotiana tomentosiformis)。植物j . 2018; 96(2): 329 - 42。
- 4.
Eom H,Park SJ,Kim Mk,Kim H,Kang H,Lee I.TAF15b,参与开花的自主途径,抑制的转录开花基因座C..工厂J. 2018; 93(1):79-91。
- 5。
Boss PK, Bastow RM, Mylne JS, Dean C.决定开花的多种途径:启用,促进和重置。植物细胞。2004;16 (5):S18-31。
- 6.
陈志强。植物开花控制的分子和遗传机制。植物细胞。2004;16 (5):S1-17。
- 7.
Baurle I,Dean C.植物发育过渡的时间。细胞。2006; 125(4):655-64。
- 8.
王Z,周Z,刘y,刘t,李q,ji y,li c,fang c,王m,wu m等。大豆和拟南芥中磷脂酰乙醇胺结合蛋白的功能演变。植物细胞。2015; 27(2):323-36。
- 9.
郑XM,吴FQ,张X,林QB,王j,guo xp,雷克兰,程祖,zou c,zou c,wan J.演化的演变PEBPft样分枝的基因家族和选择性标记。地球科学进展。2016;54(5):502-10。
- 10。
Schoentgen F,Jolles P.从结构起作用:新的广泛蛋白质家庭结合疏水性配体并显示核苷酸结合位点的可能生物学作用。费用。1995年; 369(1):22-6。
- 11.
王志强,王志强,王志强,等。抗鼻草花序结构的控制。大自然。1996;379(6568):791 - 7。
- 12.
Banfield MJ,Barker JJ,Perry AC,Brady RL。来自结构的功能?人磷脂酰乙醇胺结合蛋白的晶体结构表明在膜信号转导中的作用。结构。1998年; 6(10):1245-54。
- 13。
Bernier I, Tresca JP, Jolles P.配体结合研究从牛脑细胞质纯化的23 kDa蛋白。Acta botanica yunnanica(云南植物研究)1986;871(1):19-23
- 14。
杨坤,李胜,杨国华,等。RKIP对Raf-1激酶活性和MAP激酶信号通路的抑制。大自然。1999;401(6749):173 - 7。
- 15.
Chautard H,Jacquet M,Schoentgen F,Bureaud N,Benedetti H.TFS1P,PEBP系列的成员抑制了IRA2P,但不是Saccharomyces Cerevisiae中的IRA1P Ras GTPase-Activating蛋白。Eukartot细胞。2004; 3(2):459-70。
- 16.
Kobayashi Y,Kaya H,Goto K,Iwabuchi M,Araaki T.一对带有拮抗作用的一对相关基因,介导开花信号。科学。1999年; 286(5446):1960-2。
- 17.
Kardailsky I,Shukla VK,Ahn Jh,Dagenais N,Christensen SK,Nguyen JT,Chory J,Harrison MJ,Weigel D. Flock诱导者FT的激活标记。科学。1999年; 286(5446):1962-5。
- 18.
chardon f,tameryal c.系统核发科学分析PEBP谷物的基因家族。中国科学(d辑:地球科学)2005;61(5):579-90。
- 19.
关键词:基因工程,基因工程,基因工程,表达谱PEBP来自玉米的基因家族。植物理性。2008; 146(1):250-64。
- 20.
关键词:陆地棉,PEBP家族基因,分子标记,功能分析《公共科学图书馆•综合》。2016;11 (8):e0161080。
- 21.
Karlgren A, Gyllenstrand N, Kallman T, Sundstrom JF, Moore D, Lascoux M, Lagercrantz uPEBP植物基因家族:种子植物演化中的功能多样化。植物理性。2011; 156(4):1967-77。
- 22。
Leeggangers H,Rosilio-Brami T,Bigas-Nadal J,Rubin N,Van Dijk Adj。Nunez de Caceres Gonzalez FF,Saadon-Shitrit S,Nijveen H,Hilhorst HWM,immink RGH等:Tulipa Gesneriana和Lilium Longiflorum Pebpp基因及其在开花时间控制中的推定作用。植物细胞生理。2018; 59(1):90-106。
- 23。
引用本文:王志强,王志强,王志强开花位点t (ft)作为储存器官分化的信号的同源物。CurrOp植物BIOL。2015; 23:45-53。
- 24。
PUTERILL J,VARKONYI-GALIC E. FT和Florigen在植物中的长途开花控制。CurrOp植物BIOL。2016; 33:77-82。
- 25。
Abe M,Kobayashi Y,Yamamoto S,Daimon Y,Yamaguchi A,Ikeda Y,Ichinoki H,Notaguchi M,Goto K,Araaki T.Fd,一种来自拍摄顶点的花卉通路积分器FT的Bzip蛋白。科学。2005; 309(5737):1052-6。
- 26。
Corbesier L, Vincent C, Jang S, Fornara F, Fan Q, Searle I, Giakountis A, Farrona S, Gissot L, Turnbull C,等。拟南芥花诱导中FT蛋白的运动与花的长距离信号传导有关。科学。2007;316(5827):1030 - 3。
- 27。
Notaguchi M,Abe M,Kimura T,Daimon Y,Kobayashi T,Yamaguchi A,Tomita Y,Dohi K,Mori M,Araaki T.长距离,拟南芥开花基因座T蛋白的远程传播动作促进开花。植物细胞生理。2008; 49(11):1645-58。
- 28.
罗晓霞,高志强,王颖,陈智,张伟,黄静,余华,何亚梅。核因子-康斯复合物拮抗Polycomb抑制抑制开花轨迹T.拟南芥对诱导长日照的表达。植物j . 2018; 95(1): 17 - 29日。
- 29.
Luccioni L,Krzymuski M,Sanchez-Lamas M,Karayekov E,Cerdan Pd,Casal Jj。康斯坦斯在短时间内延迟拟南芥开花。工厂J. 2018。https://doi.org/10.1111/tpj.14171.
- 30.
山口A,小林Y,后藤K,安倍M,荒木T。FT(TSF)的双妹妹acta botanica botanica(云南植物研究). 2005;46(8):1175-89。
- 31。
Bradley D,Ratcliffe O,Vincent C,Carpenter R,Coen E.花序的开放承诺和拟南芥建筑。科学。1997年; 275(5296):80-3。
- 32。
黄尼克,简·吴,陈俊,余Ts。拟南芥蒂利亚纳Centroradialis.同源物(ATC.)系统性上起作用以抑制拟南芥中的花香。工厂J. 2012; 72(2):175-84。
- 33。
yoo sj,chung ks,jung sh,yoo sy,lee js,ahn jh。ft和tfl1的兄弟(BFT) 拥有TFL1- 密封的活动和功能TFL1对拟南芥花序分生组织发育的影响。植物j . 2010; 63(2): 241 - 53年。
- 34。
刘诗,Kardailsky I, Lee JS, Weigel D, Ahn JH。通过过度表达加速开花MFT.(ft和tfl1的母亲).摩尔细胞。2004;17(1):95 - 101。
- 35。
xi w,liu c,hou x,yu h.ft和tfl1的母亲在拟南芥中通过负反馈环路调节ABA信号调节种子萌发。植物细胞。2010;22(6):1733 - 48。
- 36。
Kochert G,Stalker Ht,Gimenes M,Galgaro L,Romero-Lopes C,Moore K.RFLP和细胞遗传学证据,了解所有OTetroTaploid驯化花生的起源和演化,落花生hypogaea(豆科植物).我是J机器人。1996年; 83:1282-91。
- 37。
Freitas fo,Moretzsohn Mc,Valls JF。巴西印度体重的遗传变异落花生hypogaea L.genet mol res。2007; 6(3):675-84。
- 38。
Moretzsohn MC, Gouvea EG, Inglis PW, Leal-Bertioli SC, Valls JF, Bertioli DJ:栽培花生关系的研究(落花生hypogaea)及其最接近的野生物种,利用内含子序列和微卫星标记。安机器人2013;111(1): 113 - 126。
- 39。
王p,高c,卞x,赵某,赵c,xia h,宋h,侯l,湾s,王x。野生植物中细胞苷-5 dna甲基转移酶和脱盐酶家族的基因组鉴定和对比分析花生。前植物SCI。2016; 7:7。
- 40.
Bertioli DJ,Cannon SB,Froenicke L,Huang G,Farmer Ad,Cannon Ek,Liu X,Gao D,Clevenger J,Dash S等人。基因组序列arachis duranensis.和arachis ipaensis.是栽培花生的二倍体祖先。Nat麝猫。2016;48(4):438 - 46。
- 41.
陈X,李h,pandey mk,杨q,王x,garg v,李h,chi x,doddamani d,hong y等。花生A-基因组祖细胞的基因组草案(arachis duranensis.)提供进入Geocarpy,油脂合成和过敏原的见解。Proc Natl Acad Sci U S A. 2016; 113(24):6785-90。
- 42.
MEGA7:用于更大数据集的分子进化遗传学分析7.0版本。生物化学学报。2016;33(7):1870-4。
- 43.
李S,王RH,金HQ,丁yh,CM C. Mungbean中热冲击转录因子的分子特征及表达谱分析。前群体。2019; 9:736。
- 44.
胡b,金吉,郭艾,张h,罗j,高g. gsds 2.0:升级基因特征可视化服务器。生物信息学。2015; 31(8):1296-7。
- 45.
张D,赵M,李S,Sun L,Wang W,Cai C,Dierking Ec,MA J.古代多倍素大豆重复基因介导的种子油含量下源性调节机制的可塑性与创新。工厂J. 2017; 90(6):1120-33。
- 46。
Krzywinski M,Schein J,Birol I,Connors J,Gascoyne R,Horsman D,Jones Sj,Marra Ma。电讯:对比较基因组学的信息审美。Genome Res。2009; 19(9):1639-45。
- 47。
侯L,张Z,窦S,张Y,庞X,李玉。中国枣突蛋白基因家族的基因组鉴定,表征及表达分析(Ziziphus Jujuba Mill。).足底。2019;249(3):815 - 29。
- 48。
Lescot M,Dehais P,Thijs G,Marchal K,Moreau Y,Van de Peer Y,Rouze P,Rombauts S. Plantcare,植物数据库独联体-作用调控元件和启动子序列硅分析工具的门户。核酸学报2002;30(1):325-7。
- 49。
李士,ying y,secco d,王c,narsai r,whelan j,shou h. pho2和gigantea之间的分子相互作用显示出开花时间和磷酸盐稳态之间的新串扰奥雅萨苜蓿.植物细胞环境。2017; 40(8):1487-99。
- 50。
Sui J,Li R,风扇Q,歌曲L,Zheng C,Wang J,Qiao L,Yu S.响应响应小GTP结合蛋白AhRabg3b在花生中的分离和特征(arachis hypogaea l。).Euphytica。2013;189:161 - 72。
- 51。
Clevenger J,Chu Y,Scheffler B,Ozias-Akins P. Allotetroploid的发育转录组Map落花生hypogaea.植物学报2016;7:1446。
- 52。
Dash S, Cannon EKS, Kalberer SR, Farmer AD, SB. C: peanut base和其他花生生物信息资源(第8章).《花生遗传学、加工和利用》,由Stalker HT和WilsonvRF编辑。家出版社。2016;241 - 252。ISBN 9781630670382。
- 53。
宋H,郭Z,陈T,Sun J,Yang G.含LRR序列的基因组鉴定和这些序列对甘氏榴坦斯的线虫感染的响应。BMC植物BIOL。2018; 18(1):279。
- 54。
宋H,王P,林JY,赵C,毕y,王X.基因组鉴定与表征WRKY基因家庭在花生。前植物SCI。2016; 7:534。
- 55。
王鹏,宋辉,李超,李鹏,李爱华,关辉,侯丽,王雪梅。热激转录因子家族基因的全基因组分析落花生.前植物SCI。2017; 8:106。
- 56。
丁m,陈俊,姜y,林l,cao,王m,张y,荣j,ye w.基因组 - 范围的调查和转录组分析WRKY基因家庭Gossypium.Mol Gen Genomics。2015; 290(1):151-71。
- 57。
Adams KL,Wendel JF。植物中的多倍体和基因组进化。CurrOp植物BIOL。2005; 8(2):135-41。
- 58。
田志,王旭,李锐,李勇,Specht JE, Nelson RL, McClean PE,邱林,马军。大豆生长习性的人工选择。中国科学院院刊2010;107(19):8563-8。
- 59。
刘B,Watanabe S,Uchiyama T,Kong F,Kanazawa A,夏Z,Nagamatsu A,Arai M,Yamada T,Kitamura K,等。大豆干生长习性基因DT1.是拟南芥的同源物吗终端FLOWER1.植物理性。2010; 153(1):198-210。
- 60.
普通豆类生长习性基因PvTFL1y是拟南芥的功能性同源物TFL1.acta physica sinica . 2012;124(8): 1539-47。
- 61.
李S,丁Y,张D,王X,唐X,戴D,金H,李Sh,Cai C,MA J.并行驯化在豆科作物中测定茎生长习性的广泛突变谱。工厂J. 2018; 96(4):761-71。
- 62.
拟南芥基因组I.分析开花植物拟南芥的基因组序列。自然。2000; 408(6814):796-815。
- 63.
于军,胡胜,王军,王国康,李胜,刘波,邓艳,戴丽,周勇,张旭,等。水稻基因组草图(水稻籼稻).科学。2002;296(5565):79 - 92。
- 64.
张建平,张建平,张建平,等。B73玉米基因组:复杂性、多样性和动态。科学。2009;326(5956):1112 - 5。
- 65.
Schmutz J,Cannon SB,Schlueter J,MA J,Mitros T,Nelson W,Hyten DL,Song Q,Thelen JJ,Cheng J,等。古代聚丙烯大豆的基因组序列。自然。2010; 463(7278):178-83。
- 66.
Kondrashov Fa,Rogozin Ib,狼义,千宾ev。选择基因重复的演变。基因组Biol。2002; 3(2):Research0008。
致谢
感谢青岛农业大学张晓军提供的野生花生种子PI219823和PI468322。
资金
国家自然科学基金面上项目(no . 31971898, no . 31601372);青岛农业大学科研基金面上项目(no . 6631115039, no . 6631119010)。资助方在研究的设计、数据的收集、分析和解释以及手稿的撰写中没有任何作用。
作者信息
从属关系
贡献
SL构思并设计了本研究。HJ, XT, MX, HZ, JS, CC进行了实验并分析了数据。SL写了手稿。所有作者阅读并批准了手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1。
PEBP蛋白在烟草叶片细胞中的亚细胞定位。
附加文件2。
野生花生与栽培花生PEBP蛋白的进化关系分析
附加文件3。
野生花生PEBP结构域的比对。沃特,金针黄,和水的颜色表明TFL1-像,MFT.- 克里英国《金融时报》例如sub-families,分别。
附加文件4。
野生花生PEBP蛋白中15个motif的序列标志。“位点”和“宽度”分别表示含有每个基序的野生花生PEBP蛋白的数量和每个基序的氨基酸数量。
附加文件5。
大豆间同源性分析GmDt1,常见的豆子PvTFL1y,和野生的花生TFL1- 样基因。研究了Soybean,常见豆类和野生花生之间分析的同源基因周围的同性区域。(a)之间的同时分析Aradurjp5k.那AraipT6XJY,和GMDT1(Glyma19G194300), 和PvTFL1y (Phvul001g189200)。(b)之间的同音分析AraduF950M那ARAIP344D4,GMDT1, 和PvTFL1y。(c)联合症分析AraduWY2NX那Araip4V81G, GmDt1, 和PvTFL1y。红色的方框表示我们的目标基因,绿色的方框表示同源基因周围的基因。通用,甘氨酸最大;PV,phoudolusulus vulgaris。
附加文件6。
函数的独联体- 在野生花生的启动子区发现的元素PEBP基因。
附加文件7。
几种野生花生和栽培花生的表达模式PEBP基因。
附加文件8。
野生和栽培的花生局部基因。
附加文件9。
表达概况PEBP栽培和野生花生22种不同组织的基因。
附加文件10。
本研究中使用的引物。
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
金,H.,唐,X.,邢梅。et al。野生花生中磷脂酰乙醇胺结合蛋白的分子和转录特性arachis duranensis.和arachis ipaensis..BMC植物BIOL.19,484(2019)。https://doi.org/10.1186/s12870-019-2113-3
已收到:
公认:
发表:
关键字
- 野生花生
- 磷脂酰乙醇胺结合蛋白
- 基因家族
- 开花时间
- 工厂建筑