摘要
背景
炭疽菌辣椒炭疽病是一种影响辣椒产量和品质的主要病害。辣椒spp)。斯氏炭疽菌在我国广泛存在,具有较强的致病性和耐药性,在辣椒果实中引起炭疽病,严重减产。之前是一个抗炭疽病位点AnR去5根据对青果成熟期果实的分析,将其定位在P5染色体上。本研究的目的是缩小该位点的区间,并确定产生抗性的基因。
结果
根据基因重组的结果辣椒“PBC932”和c .建立“77013”,我们开发了Kompetitive等位基因特异性PCR(KASPar)标记和插入-删除(InDel)标记,这些标记与AnR去5在绿色成熟果实阶段,利用它们构建遗传连锁图(42个标记,24.4 使用BC表型和基因型分析中获得的数据4年代1,公元前4年代2,及公元前4年代3.人口,AnR去5标记位于P5in-2266-404和P5in-2268-978之间,距离为164kb。这个区域包含五个基因,包括CA05g17730.CA05g17730编码' R1C-3-like '推测的晚疫病抗性蛋白同源物。的转录水平CA05g17730' PBC932 '和' 77013 '之间的差异。它的结构CA05g17730“PBC932”和“77013”之间也存在差异。
结论
我们将QTL区间缩小到一个包含5个基因的区域,这些结果将有助于进一步研究辣椒抗炭疽病的机理,为辣椒抗炭疽病品系的分子标记辅助选择奠定基础。
背景
炭疽病是辣椒生产的主要经济制约因素之一(辣椒全世界,特别是热带和亚热带地区[1].在雨季,炭疽病在收获前和收获后造成的水果产量损失可超过80%[2].炭疽病的症状包括坏死组织凹陷和出现脓疱[3.].到目前为止,辣椒炭疽病已被归因于24炭疽菌种类[4].中国有三种胡椒,最初鉴定为c . capsici,c、和c . acutatum[1,3.,5,最近被重新归类为c . truncatum[6,7],c . siamense和c . scovillei[8,9,10].病原体C scovillei使用被称为c . acutatum入选本研究。
炭疽病通常通过施用杀菌剂来控制,但这些化合物会对环境和人类健康产生负面影响。病果的清除和排水沟的清理是一项劳动密集型的农业防治工作。生物防治剂如芽孢杆菌服务提供商.其推定的过氧化氢酶可能有助于保护辣椒免受炭疽病[11].然而,培育抗病品种是防治该病的最佳长期策略,是育种工作者的重要目标。关于辣椒炭疽病的宿主和致病病原体之间的相互作用的信息仍然很少[12].亚洲已经尝试了20多年的抗炭疽病育种,但收效甚微[13].
anthracnose-resistant的品种辣椒“PBC932”和辣椒baccatum“PBC80”是1998年由avrdc -世界蔬菜中心确定的,从那时起,亚洲胡椒育种家就共享了这种辣椒[14].它们是泰国辣椒作物抗炭疽病的两个主要来源[15].不同辣椒基因型中发生的不同寄主反应是果实成熟不同阶段特定寄主和病原相互作用的结果炭疽菌物种被确定,菲奥里尼亚,果珊瑚,c、,c . scovillei,c . truncatum流行的。c . scovillei是中国pepepr的优势种之一,主要分布于东亚[8,9].
在之前的一项研究中c . capsici(现在c . truncatum),分析种内疾病评分的频率分布c . baccatum“PBC1422”×c . baccatum“PBC80”杂交表明,一个隐性基因负责成熟绿果期的抗性,一个显性基因负责成熟果期的抗性[16].抗性数量性状位点(QTL)定位c . scovillei(前acutatum),C.truncatum(前辣椒)已使用三种抗性评估方法进行了评估:疾病发生率、真实病斑直径和总病斑直径c . baccatum“PBC81”至c . scovillei(前acutatum),c . truncatum(前辣椒)处于不同的位置[17].抵抗c . capsici(现在元宝枫)在‘PBC932’中发现由一个隐性基因控制[15].“AR”系是一个抗炭疽病的育种系,由风轮菜“中国人民银行932年”。抵抗C .truncatum(前辣椒)也被发现是由一个隐性基因控制的[18].
抵抗c . acutatum(现在scovillei)源自风轮菜据报道,' PBC932 '在绿色果实中由两个互补显性基因控制,但在红色果实中由两个隐性基因控制[19].影响基因表达的两个显性基因c . acutatum(现在scovillei)在种内试验中鉴定了抗性c .建立简历“邦昌” × 风轮菜“PBC932”人群[20].一个包含12个连锁群(LGs)和214个单核苷酸多态性(SNPs)以及824个 cM覆盖率是由来自c .建立“Bangchang”×风轮菜在LG2上,在14个SNP的区间内,在两个SNP之间鉴定出“PBC932”和两个与成熟绿色水果抗炭疽病相关的QTL 厘米[14].另有14个LGs、385个SSR标记(简单序列重复,SSR;insertion-deletion InDel;1个绿色成熟期抗炭疽病的主效QTL和4个次效QTL定位在P5染色体上[21].
在许多类型的分子标记中,单核苷酸多态性(SNPs)对育种具有吸引力[22].竞争性等位基因特异性PCR (KASPar)标记,由SNP位点设计,为标记辅助选择和QTL精细定位等应用提供了成本效益和可扩展的灵活性[23].InDel多态性(碱基插入或缺失)在基因组中相对丰富,InDel标记已经开发并在前期研究中使用[24,25].在本研究中,我们利用KASPar和InDel标记构建了炭疽病抗性qtl图谱c .建立“77013”×风轮菜“PBC932”。精细定位分析使我们能够缩小具有抗性的QTL的间隔。
结果
表型鉴定
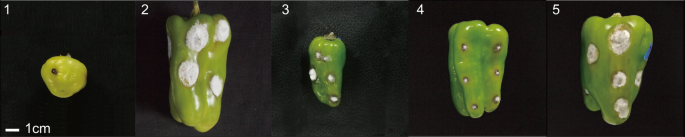
根据以下标准确定抗病表型:抗病(R):0 嗯 ≤ O ≤ 12 mm;易感疾病:O > 12 “PBC932”和F的所有工厂1“77013”的所有植株均为S。R的遗传为显性遗传4年代2–1和BC4年代2–2个群体来自BC的不同抗病单株4年代1.不列颠哥伦比亚省4年代1,公元前4年代2公元前1,4年代2-2居群相对于R和S植物的分布呈正态分布(图。1).分离比分别为250:182、140:102、117:45,这与两个互补显性基因预期的分离比9:7或3:1相符[Χ2 = 0.461(< 3.84), Χ2 = 0.252(< 3.84), Χ2 = 0.667(< 3.84)和Χ2= 0.378(< 3.84)](表1).
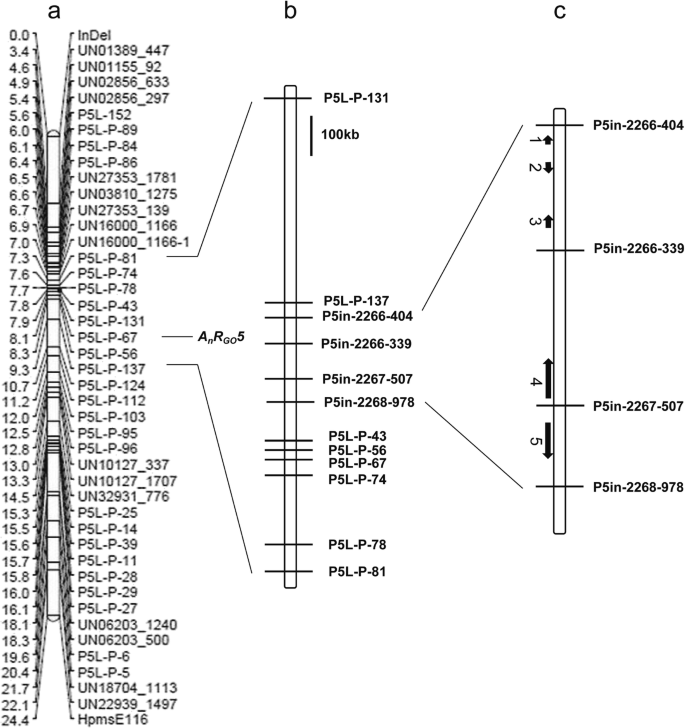
根据BC建立的遗传连锁图谱4年代1群体共包含44个标记,包括42个KASPar标记、1个InDel标记和1个SSR标记,全长图谱为24.4 标记间的平均遗传距离为0.55 cM.通过将图谱信息与真实病斑直径数据相结合,预测了一个与炭疽病抗性相关的QTL。最紧密连锁的QTL标记为P5L-P-67,贡献率为69.3%,LOD为24.37。一个95%置信区间的QTL,AnR去5.标记在P5L-P-137与UN16000_1166-1之间,二者之间的遗传距离为2.33 cM。为了缩短定位间隔,我们从BC中选择了R株4年代1建造卑诗省4年代2–1和BC4年代2–2个群体。使用P5L-P-137和UN16000_1166–1之间的9个标记筛选这些群体中的单株,以获得BC的交换单株4年代2-44年。BC的后代4年代2-44和非交换的单个工厂BC4年代2–47株分别种植,7日测定绿色成熟果实的抗性表型 两株植物后代的真实病斑直径值显著不同。因此,一个nR去5位于标记P5L-P-137和P5L-P-81之间(图。2a) .该区间包含13个预测基因。
为了进一步缩小本地化间隔,BC4年代3.构建了883株群体,并对其进行了分析。物理位置AnR去5根据“CM334”和“Zunla”的基因组序列发现了基因簇标记。根据亲本重新测序数据,为这一精细定位区间开发了四个InDel标记,即P5in-2268-507、P5in-2268-339、P5in-2266-404和P5in-2267-978。交换的单株BC4年代3.–6个在P5L-P-137和标记的P5L-P-81之间用11个标记进行筛选。公元前4年代3.–6和未交换的单个工厂BC4年代3.-417分别种植,接种7 d后测定果实表型。两种植物后代的真病灶直径值有显著差异。通过比较交换植物BC的基因型和表型4年代2公元前-44年,4年代3.-6,主要抗炭疽病QTLAnR去5在辣椒青绿成熟期,定位在P5in-2266-404和P5in-2267-978之间。2b, c).这个区间包含5个预测基因。
Blastn在该定位区间内对5个基因进行搜索,发现CA05g17700编码一个果胶甲基酯酶相关蛋白;CA05g17710和CA05g17720编码未知蛋白;CA05g17730和CA05g17740编码抗马铃薯晚疫病蛋白“R1C-3-like”同源物(见表1)2)将CA05g17730和CA05g17740基因序列与番茄基因组数据库(SGN)中的序列进行比较http://solgenomics.net).在核苷酸水平上,番茄第5染色体上CA05g17730与Solyc05g043420.1.1的相似性为85%,CA05g17740与Solyc05g043420.1.1的相似性为88.1%。Solyc05g043420.1.1编码一个“核苷酸结合位点+亮氨酸丰富重复”(NBS-LRR)蛋白。这类蛋白质是由一大家族抗病基因编码的。
基因表达分析
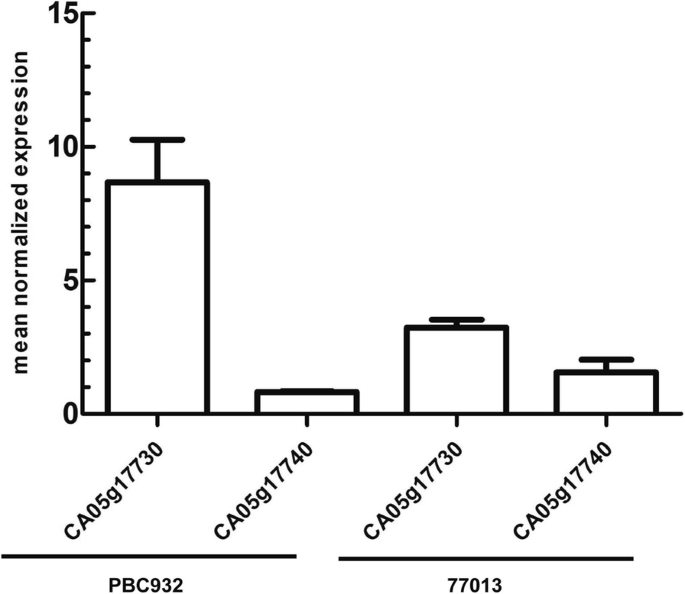
使用特异性荧光定量引物通过RT-qPCR分析CA05g17730和CA05g17740的转录水平。在“PBC932”和“77013”之间,CA05g17740的转录水平没有显著差异,但在“PBC932”和“77013”之间,CA05g17730的转录水平存在显著差异(图。3.).
基因结构分析
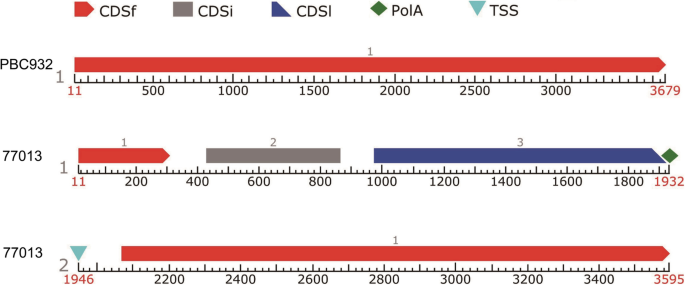
从CA05g17730基因序列设计引物,扩增' PBC932 '和' 77013 '中CA05g17730基因的全长序列。CA05g17730在' PBC932 '中长度为3673-bp,在' 77013 '中长度为3589-bp。与‘pbc932’中的CA05g17730序列相比,‘77013’在177 bp处有一个19 bp的缺失,在204 bp处有一个8 bp的缺失,在472 bp处有一个15 bp的缺失,在704 bp处有一个42 bp的缺失,在3644处有一个3 bp的缺失(附加文件)2:图S1)。利用Sol Genomics Network和Softberry网站的工具对CA05g17730的外显子区域进行了预测,发现‘77013’序列的所有缺失都在外显子区域。“PBC932”与“77013”之间CA05g17730基因缺失部分可能导致这两个品系之间CA05g17730蛋白的功能差异。利用Softberry网站上的工具预测基因结构,CA05g17730中的1个CDS在' PBC932 '中预测,77013中预测了2个基因和4个CDS。CA05g17730在' 77013 '中的转录被预测由于碱基缺失而在1932 bp处过早终止,导致CA05g17730在' 77013 '中的功能丧失(图)。4).
讨论
在这份报告中,我们精细地绘制了抗性位点C.scovillei从P5染色体上164Kb物理间隔的PBC932中提取。这比我们实验室曾经定位的间隔约为9.6的位点要贵 同一染色体上的cM和P7、P10和P12染色体[21].基于最近获得的一个“PBC932”图谱和一个不同的抗性QTL炭疽菌P2上发现的物种是另一个结果[14]与该位点相关的几个标记(P5in-2266-404、P5in-2266-339、P5in-2267-507、P5in-2268-978)已被开发出来,可用于分子标记辅助选择。
基因CA05g17730是目前我们部分证实的最可疑的候选抗病基因之一。在164Kb的物理间隔内,仅发现了包括CA05g17730在内的5个基因。我们的分析表明,CA05g17730基因编码Solyc05g043420.1.1的同源物,即“R1C-3样”假定的晚疫病抗性蛋白大多数R基因编码“核苷酸结合位点+富含亮氨酸重复序列”(NBS-LRR)类蛋白质,其中包含对ATP或GTP结合至关重要的保守核苷酸结合(NB)位点[26,27]以前的研究探讨了NBS-LRR蛋白在不同植物中的作用,包括拟南芥[28],土豆[29,30)、大米(31),玉米(32],还有西红柿[33].CA05g17730的功能以及是否具有更详细的抗性还需要进一步研究。
在PBC932基因中,还有其他被怀疑与抗药性有关的基因C.scovillei?在以前的研究中,Lin等人认为两个互补的显性基因控制抗药性c . scovillei在“PBC932”(19].随后Sun等人证实,正如Mahasuk等人报道的[2,16]PBC932的遗传曾被证明在绿色和红色果实期的抗性是由不同的基因控制的,这些基因与重组个体在相同的P5基因组间隔内[21].Sun等在P5, P7, P10, P12染色体上定位了几个qtl [21]因此,在不同的基因座上有更多的基因参与抗性。即使在P5染色体上,在本研究中,抗性和易感性的分离比c . acutatum在BC4S中1,BC4S2–1和BC4S22个群体均符合1:3或9:7的孟德尔模型,表明大多数遗传变异是由两个互补的显性基因解释的。事实上,CA05g17710和CA05g17720基因编码未知的功能蛋白。
结论
我们缩小了抗性的主要QTLc . scovillei包含五个基因的164KB区域的间隔。同源物和转录本分析表明,CA05g17730是可能的抗肿瘤候选基因c . scovillei在辣椒成熟的青果期。这一发现为研究两者之间的关系提供了新的见解c . scovillei这些结果将有助于进一步研究辣椒抗炭疽病机理,为辣椒抗炭疽病品系的分子标记辅助选择奠定基础。
方法
绘制种群和病原体图谱
母本为自交系“77013”(c .建立L.),由中国农业科学院蔬菜花卉研究所(IVF)培育[21]。父系父母为“PBC932”(风轮菜),抗c . acutatumAVRDC王天成博士提供[21].F1这一代是由小麦的种间杂交产生的c .建立“77013”×风轮菜“PBC932”。F1与‘77013’回交数代,得到BC4人口。植物抗c . acutatum是从BC4人口和公元前4年代1群体通过自交获得。然后,种间公元前1年代1,公元前1年代2,及公元前1年代3.后代[('77013' × ‘PBC932') × ‘获得77013']。
使用单孢子分离方法[34],分离物“Ca”,于2009年从中国湖南省辣椒植物的病果中收集和分离。该分离物经鉴定为阳性c . acutatum基于其菌落形态和rDNA its(ITS4/ITS5)序列(Genbank登录号KC936995)[21].通过在NCBI中爆破rDNA-ITS (ITS4/ITS5)序列,将分离物‘Ca’重新分类为c . scovillei.
炭疽病抗性评价
采用1999年AVRDC开发的方法,对离体成熟青果进行微注射接种,稍加修改。成熟的青果用蒸馏水和75%乙醇洗涤。的c . scovillei分离物“Ca”培养4-6天 在新鲜制备的液体马铃薯葡萄糖琼脂(PDA)培养基中,在100℃下摇动10天 28转/分 在黑暗中°C以促进分生孢子的形成。然后,液体PDA培养基通过四层无菌纱布过滤以获得分生孢子悬浮液。通过刺穿成熟绿色水果的表面形成1 mm深的伤口,1 μl分生孢子悬浮液(浓度5 × 105 毫升·毫升− 1)注射到每个伤口。根据水果的大小,每个水果都有2到6个伤口。接种后,将果实置于四层潮湿无菌滤纸的塑料盒中,保持湿度在95%左右。在26°C孵卵7天后,测量真实病变直径(定义为所有病变的平均病变直径(mm)),作为抗性表型的指标[35].
簇靶分子标记及连锁图谱的研究进展
的AnR去5在亲本和BC中筛选了一个InDel标记和一个SSR标记的基因簇标记1人口[21,36].辣椒品系“CM334”和“Zunla”的物理地图数据[37,38]用于这些分析。这个AnR去5利用在线BlastN选项将基因簇标记与“CM334”基因组序列进行比对,以确定物理位置(http://passport.pepper.snu.ac.kr/?t=PGENOME)然后,我们开发了物理间隔内的标记。我们基于转录组序列与辣椒基因组信息的比较开发了KASPar标记(http://peppergenome.snu.ac.kr/download.php(Wang等人,未发表数据)。从它们中,我们选择了16个KASPar标记(附加文件1:表S1)位于染色体5上AnR去5先前报告[21].基于“PBC932”和“77013”的重新测序数据,我们搜索了该物理间隔内的SNP位点,并使用Primer 5.0软件设计了26对多态性KASPar引物。
从亲本系和BC中提取总基因组DNA4年代1,公元前4年代2,及公元前4年代3.使用CTAB方法的种群[39].标记物由生工生物技术公司(中国北京)合成。利用Join Map 4.0软件对42个标记进行分析和连接[40].使用Kosambi函数计算地图距离。使用LOD为3.0、步长为0.5的MAPQTL6.0对炭疽病评分进行QTL分析[41].根据表型变异解释比例(% E)确定主要QTLAnR去5,使用Student t检验比较每个子代家族中纯合重组子和纯合非重组子的真实病变直径值。
基因功能预测
物理位置AnR去5-连锁标记是根据“CM334”和“Zunla”基因组的blastn搜索确定的。关于包含以下内容的区域中预测基因的信息:AnR去5通过Sol基因组学网络(SGN)的blastn搜索收集http://solgenomics.net)和国家生物技术信息中心(NCBI)http://www.ncbi.nlm.nih.gov)数据库。
RT-qPCR与候选基因测定
根据“CM334”和“Zunla”基因组数据库搜索基因序列(http://passport.pepper.snu.ac.kr/?t = 基因组;http://peppersequence.genomics.cn/page/species/index.jsp).特异荧光定量引物(100-200 根据基因编码序列(CDS)设计bp(附加文件)1:表S2)。
以PBC932和77013的健康成熟青果为材料,用孢子悬浮液接种。果实附着在植物上。7天后,采集水果皮损附近的果肉,立即用液氮冷冻。用SV总分离系统(Promega, Madison, WI, USA)分离总RNA。使用5X All-In-One RT MasterMix (Applied Biological Materials Inc., Richmond, Canada)从RNA中合成第一链cDNA。采用基因特异性引物和GoTaq®qPCR Mix进行PCR分析。PCR分析使用Light Cycler 480 II仪器(Roche Diagnostics Ltd., Rotkreuz, Switzerland)进行。每种PCR混合物(体积为20 μl)的热循环条件如下:95℃,10 min;95°C 15 s, 55°C 30 s, 72°C 30 s共40个循环;和95°C为15 s; 60 °C for 15 s; and 95 °C for 15 s. Three biological replications were included for each experiment. Relative expression levels were calculated using the 2−∆∆计算机断层扫描方法(42].经RT-qPCR分析,候选基因为‘PBC932’与‘77013’之间转录水平差异显著的基因。
基因序列分析与基因结构预测
为了获得候选基因的全序列,利用基因特异性引物对从' PBC932 '和' 77013 '的基因组中扩增每个基因(附加文件)1:表S3)。产品由Sangon Biotechnology Co. (Beijing, China)测序,序列使用SeqMan软件组装。利用DNAMAN V6软件对PBC932和77013的基因序列进行比较。这两种亲本的基因结构是利用Softberry网站(http://linux1.softberry.com/berry.phtml?topic=fgenesh&group=programs&subgroup=gfind).
数据和材料的可用性
本研究中使用和/或分析的数据集可根据合理要求从相应作者处获得。
缩写
- O:
-
所有病灶的平均直径(mm)
- R:
-
抗病
- S:
-
疾病的易感
工具书类
- 1.
Than P,Jeewon R,Hyde KD,Pongsupasamit S,Mongkolporn O,Taylor PWJ.的特性和致病性炭疽菌属与辣椒上的炭疽病(辣椒属《植物病理学》2008a;57(3):562-72。
- 2
Mahasuk P,Khumpeng N,Wasee S,Taylor PWJ,Mongkolporn O.辣椒(辣椒属)苗期和结果期对炭疽病(辣椒炭疽菌)抗性的遗传.植物品种.2009年a;128(6):701-6。
- 三。
Montri P,Taylor PWJ,Mongkolpon O.辣椒炭疽病病原菌在泰国的病理类型.植物学报,2009;93(1):17-20。
- 4
Mongkolporn O, Taylor PWJ。辣椒炭疽病:炭疽菌分类和致病性。植物病理学研究。2018;67(6):1255 - 63。
- 5.
Mongkolporn O,Montri P,Supakaw T,Taylor PWJ.三种病毒感染成熟绿色和成熟辣椒果实的差异反应炭疽菌物种。植物分布。2010;94:306-10。
- 6.
Damm U, Woudenberg JHC, Cannon PF, Crous PW。草本寄主具有弯曲分生孢子的炭疽菌种。真菌潜水员。2009;39:45 - 87。
- 7.
Ranathunge NP,Mongkolporn O,Ford R,Taylor PWJ.辣椒上的截形炭疽病系统:感染、定殖和防御机制.澳大利亚植物病理学杂志.2012;41:463-73。
- 8.
Damm U, Cannon PF, Woudenberg JHC, Crous PW。Colletotrichum acutatum种复合体。螺栓Mycol。2012;73:37 - 113。
- 9
刁永忠,张超,刘芳,王文忠,刘玲,蔡玲,刘小玲。炭疽菌中国引起炭疽病的物种。Persoonia。2017;38(2017):20-37。
- 10.
De Silva DD, Ades PK, Crous PW, Taylor PWJ。在澳大利亚与辣椒炭疽病有关的炭疽菌种。植物病理学研究。2017;66:254 - 67。
- 11.
关键词:辣椒,炭疽病,芽孢杆菌,生防剂辣椒刺盘孢.生物防除。2018年,63(6):833 - 42。
- 12
比P, Haryudian P, Sitthisack P, Paul WJT, Kevin DH。辣椒炭疽病引起的炭疽菌浙江大学学报,2008b;9(10):764。
- 13
Mongkolporn O, Taylor PWJ。辣椒。出自:Kole C,编辑。野生作物亲缘:基因组和育种资源,第5卷。纽约:施普林格;2011.43-57页。
- 14
Mahasuk P,Struss D,Mongkolporn O.在两个国家鉴定的抗炭疽病QTL辣椒资料来源:Mol.2016;36(1):10。
- 15
Pakdeevaraporn P,Wasee S,Taylor PWJ,Mongkolporn O.辣椒炭疽病抗性遗传.植物品种.2005;124(2):206-8。
- 16
Mahasuk P,Taylor PWJ,Mongkolporn O.辣椒炭疽菌抗性两个新基因的鉴定.植物病理学.2009b;99(9):1100-4。
- 17
李杰,洪杰,杜杰,尹杰,李杰,洪杰,杜杰,尹建斌.辣椒炭疽病两种炭疽菌抗性QTL的鉴定.作物科学与生物技术杂志.2010;13(4):227-33。
- 18
关键词:辣椒,炭疽病,抗性,隐性基因Sci繁殖。2008;58(2):137 - 41。
- 19
林世华,彭格尼夫克,王聪聪.炭疽菌对辣椒炭疽病抗性的遗传.学报.2007;760:329-34。
- 20.
Mahasuk P,Chinthaisong J,Mongkolporn O.两种炭疽病菌致病型和接种方法对辣椒炭疽病的不同抗性。品种科学。2013;63(3):333-8。
- 21.
孙晨,毛少林,张振华,Palloix A,王丽华,张宝祥。对炭疽病的抵抗力(炭疽菌acutatum)辣椒成熟绿色果实和成熟果实的遗传多样性由染色体P5上的主要显性QTL簇控制。Sci Hortic.2015;181:81–8。
- 22.
Ashrafi H, Hill T, Stoffel K, Kozik A, Yao J, Chin-Wo SR, Van Deynze A.辣椒转录组的新组装(甜椒):单核苷酸多态性(SNPs)、单核苷酸多态性(SSRs)和候选基因的硅发现基准。BMC基因组学。2012;13(1):571。
- 23.
基于竞争等位基因特异性PCR (KASP)的单核苷酸多态性基因分型技术及其在作物改良中的应用。摩尔品种。2014;33:1-14。
- 24
刘波,王颖,翟伟,邓军,王辉,崔勇,程飞,王鑫,吴军。基于全基因组重测序的油菜InDel标记的建立。[j] .计算机应用。2013;126(1):231-9 .]
- 25
李伟,程杰,吴Z,秦C,谭S,唐X,崔J,张L,胡KL。基于InDel的辣椒连锁图谱(甜椒).摩尔品种。2015;35(1):32。
- 26
丹格尔,琼斯.植物病原菌与对感染的综合防御反应.自然.2001;411(6839):826。
- 27
卢卡锡E,塔肯FLW。持久的强抗性蛋白质是植物防御的策划者。植物生物学。2009;12(4):427–36.
- 28
Meyers BC, Kozik A, Griego A, Kuang H, Michelmore RW。拟南芥nbs - lrr编码基因的全基因组分析植物细胞。2003;15(4):809 - 34。
- 29
Pritchard L, Etherington GJ, Mackenzie K, Cock PJ, Wright F, Sharma SK, Bolser D, Bryan GJ, Jones JD, Hein I. NBS-LRR基因家族的鉴定和定位。生物医学中心基因组学。2012; 13:75 - 88。
- 30.
罗志强,王志强,王志强,等。马铃薯耐药基因的克隆及基因定位。《公共科学图书馆•综合》。2012;7:e34775。
- 31.
周涛,王勇,陈建强,Araki H, Jing Z, Jiang K, Shen J, Tian D.粳稻NBS基因的全基因组鉴定显示non-TIR NBS- lrr基因的显著扩展。中国生物医学工程学报。2004;32(4):441 - 446。
- 32.
帕特森·阿、鲍尔斯·杰、布鲁格曼·R、杜布恰克·I、格里姆伍德·J、甘达拉赫·H、哈伯勒·G、赫尔斯滕·U、米特罗斯·T、波利亚科夫·A.《高粱双色基因组与草的多样性》《自然》2009;457(7229):551-6。
- 33.
Nieri D, Di Donato A, Ercolano mr对番茄减数分裂重组图谱的分析揭示了NB-LRR基因的染色体优先位置。Euphytica。2017;213(9):206。
- 34.
Ho WC,Ko WH.一种获得真菌单孢子分离物的简单方法.博特公牛科学院,1997;38:41-4。
- 35.
炭疽病的QTL定位(炭疽菌spp)两者交叉的阻力甜椒和风轮菜《Theor Appl Genet》2004;109(6):1275-82。
- 36
黄浩,张志中,张志中,毛思亮,王力宏,张本x。EST数据库中辣椒SSR信息的分析。中国农业科学院。2011;10:1532-6。
- 37
Kim S,Park M,Yeom S,Kim YM,Lee JM,Lee HA,Seo E,ChoiJ CK,Kim KT,Jung K,Lee GW,Oh SK,Bae C,Kim S,Lee HY,Kim SY,Kim MS,Kang BC,Jo YD,Yang HB,Jeong HJ,Kang WH,Kwon JK,Shin C,Lim JY,Park JH,Huh JH JH,Kim JS,Kim BD,Cohen O,Paran O,Paran Kys,Park Kys,Kim HA,Kim HJ,Bosland PW、Reves G、Jo SH、Lee BW、Cho HT、Choi HS、Lee MS、Yu Y、Choi YD、Park BS、Deynze A、Ashrafi H、Hill T、Kim WT、Pai HS、Ahn HK、Yeam I、Giovannoni JJ、Rose JKC、Sørensen I、Lee SJ、Kim RW、Choi IY BS、Lim JS、Lee YH、Choi D.辣椒的基因组序列为辣椒品种辛辣性的进化提供了见解enet.2014;46:270–8。
- 38
方秦C, C,沈Y, X,陈L, Min J,程J,赵年代,徐M,罗Y,杨Y, Z,毛L,吴H, Ling-Hu C,周H, H, Gonzalez-Morales年代,Trejo-Saavedra D,田H,唐X,赵M,黄Z,周,姚明X,崔J,李W,陈Z,冯Y, Y妞妞,Bi年代,杨X,李W, Cai H,罗X, Montes-Hernandez年代,Leyva-Gonzalez M,熊Z,他X,白,棕褐色,张唐X,刘D,刘J,年代,陈M, L, L,张Y,廖W,张Y,王M, Lv X,温家宝B,刘H, H烹调的菜肴,张Y,杨年代,王X,徐J,李X,李,王J, Palloix, Bosland P,李Y,克罗,Rivera-Bustamante R, Herrera-Estrella L,阴Y, Yu J, K胡,栽培辣椒和野生辣椒的全基因组测序为辣椒的驯化和专门化提供了深刻的见解。中国科学院院刊。2014;111(14):5135-40。
- 39
富尔顿TM,春旺瑟J,唐克斯利SD。从番茄和其他草本植物中提取DNA的微准备程序。植物学报。1995;13(3):207-9。
- 40.
Van Ooijen JW,Voorrips RE.Joinmap(R)3.0版:遗传连锁图计算软件。Wageningen:Plant Research International;2001。可在www.kyazma.nl/index.php/mc上查阅。
- 41.
Van Ooijen JW.JoinMap®4,《实验种群遗传连锁图谱计算软件》,第33卷。瓦格宁根:Kyazma BV;2006年。第10.1371页。
- 42.
Livak KJ,Schmittgen TD.使用实时定量PCR和2−∆∆Ct方法。方法。2001;25:402–8。
致谢
我们感谢来自Edanz中国集团Liwen Bianji的Jennifer Smith博士(www.liwenbianji.cn/ac),用于修改本手稿草稿的英文文本。
资金
本研究得到了国家自然科学基金(31572121)、国家重点研究开发项目(2017YFD0101901)、中国农业科学院合作科技创新项目(CAAS-ASTIP-IVFCAAS)、广西科技项目(桂科AB16380065)的资助中国农业部项目(CARS-25)。
作者信息
从属关系
贡献
WL和GJ构思设计了实验。ZY, LY和WL构建了这份手稿。ZY、LY、ZZ、CY对植物材料进行抗病性鉴定。WL、ZY、ZZ、CY、ZB、WR、YH进行分子标记的开发和检测。ZY、LY和MW对数据进行了分析。ZY、ZZ、CY、ZB、YH对最终稿件进行了改进。所有作者阅读并批准了最终的手稿。
通讯作者
道德宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明他们没有相互竞争的利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充资料
附加文件1:表S1。
利用SSR、InDel和KASPar标记进行序列定位AnR去5在5号染色体上。表S2。用于RT-qPCR实验的引物序列。表S3。用于克隆候选基因的引物序列。
附加文件2:图S1。
乳腺癌CA05g17730基因的序列分析辣椒“PBC932”和甜椒通过PCR扩增和测序获得“77013”。
权利和权限
开放存取本文根据知识共享署名4.0国际许可的条款分发(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
赵彦,刘彦,张志强。等。炭疽病抗性主要QTL的精细定位AnR去5在辣椒“PBC932”。植物生物学20.189 (2020). https://doi.org/10.1186/s12870-019-2115-1
收到了:
接受:
发表:
关键词
- 胡椒
- 炭疽病
- 良好的映射
- 斯氏炭疽菌
- QTL