摘要
背景
广泛种植的辣椒(辣椒Spp .)是最多样化的蔬菜之一;然而,很少有研究关注中国商业品种的遗传多样性和亲缘性特征。在这项研究中,使用来自35个不同基因的重测序数据,确定了92个完美单核苷酸多态性(SNPs)c .建立行。在此基础上,设计了一种将完美SNPs的多重扩增与Illumina测序相结合的Target SNP-seq基因分型方法,对271个商品辣椒品种进行多态性检测。
结果
由于多态性信息含量、观察杂合度、期望杂合度和小等位基因频率的平均值分别为0.31、0.28、0.4和0.31,完美SNPs面板具有较高的识别能力。值得注意的是,所研究的辣椒品种根据果实形状分为块状、长角型、短角型和线形果型。长角果居群的遗传多样性最高,其次为短角果居群、线形果居群和块状果居群。然后将一组35个核心SNPs用作竞争性等位基因特异性PCR (KASPar)标记,这是另一种用于品种鉴定的可靠基因分型技术。主成分分析和系统进化树构建的遗传相关性分析表明,4个果形居群分别聚在一起,重叠部分有限。基于结构聚类分析,可将品种划分为与果实形态相关的5个亚群体。此外,根据随机化测试,亚群在统计学上是不同的F圣统计数据。分别位于染色体1、2、3、4、6和12上的9个位点与果实形状指数显著相关(p< 0.0001)。
结论
本研究开发的目标SNP-seq可作为辣椒遗传多样性、群体亲缘性和分子育种检测的有效工具。此外,本研究还表明,以果实形状为重点的育种方案对辣椒品种的遗传结构有显著影响。
背景
胡椒是该属的成员辣椒,原产于南美洲,是全球最具经济价值的蔬菜作物之一[1,2,3.].迄今为止,有38种辣椒已被报道(USDA-ARS, 2011)。其中,c .建立,c . frutescens,c .摘要,c . baccatum,c下毛竹被认为已经被驯化了[4].在全球范围内,最主要的物种是c .建立美国有许多商业品种,在大小、形状、刺激性和颜色上都有很大差异。
随着种子贸易的发展和全球化,以真实性和纯度为基础的种子商业质量变得越来越重要[5].传统上,品种鉴定是通过田间形态性状调查完成的;然而,这一过程费时费力,因此不适合现代检验要求[6].用分子标记来区分品种是一种更高通量的方法[5].事实上,遗传标记已被用于多种商业作物的DNA指纹、多样性分析、品种鉴定和标记辅助育种[7,8].此外,一些基于pcr的工具已被用于检测辣椒的遗传多样性,包括随机扩增多态性(RAPD)、限制性片段长度多态性(RFLP)和扩增片段长度多态性(AFLP) [9,10,11,12].
最近,两个c .建立Zunla-1和CM334 [3.,13],为全基因组简单序列重复序列(SSR)和插入或删除(InDel)标记的检测和开发提供了重要平台[14,15,16,17,18,19,20.].虽然已经有大量的SSR和InDel标记,但这些技术还不适合大规模的种质鉴定。因此,对能够表征数千种种质资源的高效、快速和高通量系统的需求尚未得到满足。
满足这种高标准的一种方法是使用单核苷酸多态性(SNPs),它是很好的基因分型标记,因为它们覆盖了整个基因组,主要是双等位基因。因此,已经开发了多个高通量SNP基因分型平台,包括GoldenGate [21]和Infinium [22], TaqMan [23],以及KASPar平台(KBiosciences,www.kbioscience.co.uk).近年来,高通量转录组测序和测序基因分型(GBS)已成功应用于辣椒,产生了信息丰富的全基因组SNP数据[24,25,26,27,28,29,30.].然而,SNP标记物基因分型需要一个全面的技术平台和特殊的设备和试剂,因此被认为是昂贵的。
目标测序基因分型(GBTS)是一种有针对性的序列捕获策略,可以使用高通量测序技术对数千个ssr或snp进行基因分型。GBTS的两种主要类型是多重PCR和基于探针-溶液的目标测序;该技术已被商业化为AmpliSeq [31], NimbleGen [32], SureSelect [33]、GenoBaits和GenoPlexs [34].迄今为止,该技术已广泛应用于医疗应用,但很少用于农业物种。然而,基于多重pcr的Target SSR-seq技术已成功应用于382个黄瓜品种的遗传多样性和结构研究[35].本研究结果表明,GBTS是一种可定制、灵活、高通量、低成本、准确的测序工具。
中国出产的辣椒占世界辣椒产量的三分之一[36].迄今为止,中国辣椒种质资源的遗传多样性研究主要是利用SSR标记进行的,但这些研究仅对几个中国种质资源(多达32个)进行了研究[37]或少量SSR标记(最多28个)[36].然而,用于辣椒品种基因分型和鉴定的高通量SNP平台明显落后于SSRs,对中国辣椒品种间遗传多样性的研究尚未得到广泛分析。因此,本工作的主要目标是:1)开发适合辣椒品种基因分型的Target SNP-seq技术;2)利用KASPar平台对复合核心- snp标记进行鉴定,以最大限度地进行品种鉴定;3)研究271个辣椒品种的遗传多样性、结构和分化水平。这项研究表明,一种新的Target SNP-seq可以作为一种快速有效的辣椒基因分型工具,这些栽培品种的遗传结构受到选择果实形状的育种程序的强烈影响。
结果
用于目标SNP-seq的全基因组完美snp
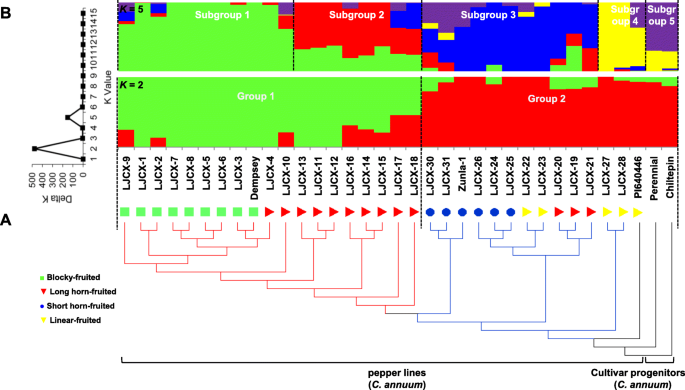
重新排序31个辣椒品种(c .建立)生成了872 Gb的成对端序列数据,平均深度为~ 8.4。在绘制到Zunla-1基因组后[3.],在31个重新测序的品系和4个先前发表的品种(Dempsey、Zunla-1、Perennial和Chiltepin)的基因组序列中检测到40,700,040个SNPs [3.,13].大约11.3%c .建立基因组包含不同的SNP位点。共有21,237,194个小等位基因频率(MAF)为> 5%,缺失数据< 10%的snp被认为是用于下游分析的高质量snp。使用c .建立作为一个外类群,系统发育树显示辣椒系一般可以根据果实形状进行分类,除了三个长角果系与线形果系分组。根据遗传距离,果实形状的转变从奇尔特宾型辣椒开始,然后是线状果型、短角果型、长角果型,最后是块状果型辣椒,块状果型辣椒与奇尔特宾型辣椒距离最远(图2)。1a).此外,35行可以根据最优的数量分为两大类K结构= 2(图;1b);第1组由9个钟果系和10个长角果系组成,其余包括3个长角果系、全部线形果系和全部短角果系,以及2个品种祖先多年生和Chiltepin被分配到第2组。这些辣椒系的聚集似乎与果实类型更相关,当K= 5。类群1分为亚类群1(多为块状果)和亚类群2(长角果),类群2由亚类群3(多为短角果)、亚类群4(线形果)和亚类群5(品种祖代小果)组成。
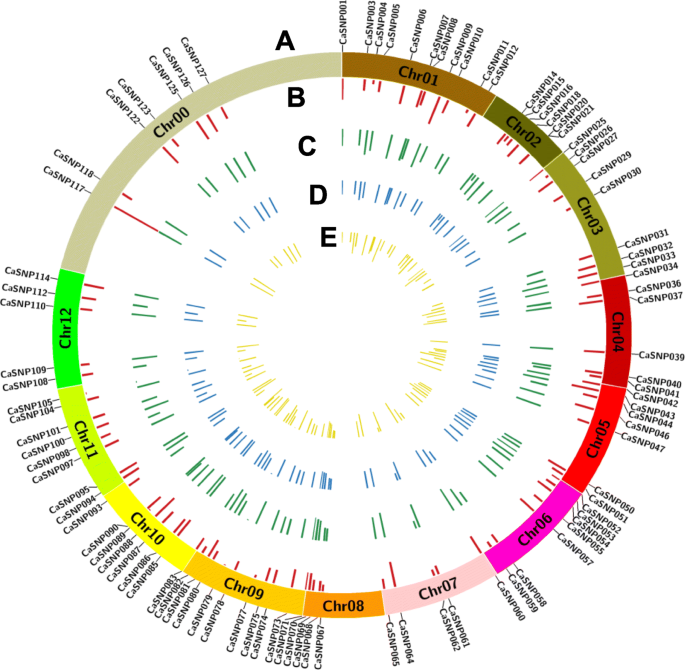
鉴于辣椒基因组具有高度的重复性,研究人员采用了严格的标准来鉴定完美的snp(参见方法).总共鉴定出521个完美snp,其中92个分布在整个基因组(图2)。2;额外的文件9:表S2),作为多重PCR靶点。根据前面的注释[3.], 83个和9个完美snp分别位于基因间区和基因区。每个完美SNP的最近侧翼注释基因显示在附加文件中9:表S2。
利用Target SNP-seq对辣椒品种进行基因分型的最佳snp特征。一个92个完美snp在水稻基因组表意图中的分布c .建立Zunla-1 [3.].b观察杂合度(何),用红色表示。c预期杂合度(他)以绿色表示。d每个SNP位点的多态性信息含量(PIC)以蓝色表示。e每个SNP位点的小等位基因频率(MAF)用黄色表示。这个数字是由Circos (http://circos.ca/), SNP区域放大到2 Mb
基于目标SNP-seq的辣椒品种基因分型分析
使用Target SNP-seq对271个辣椒品种进行了基因分型,其中包括90个块状辣椒品种、113个长角辣椒品种、25个短角辣椒品种和43个线果辣椒品种8:表S1)。271个品种共产生5590万次reads,平均目标reads深度为2064,约82%的样品测序深度大于1000 ×(额外文件2:图S2A)。271个品种中,有238个(87.8%)符合Zunla-1基因组[3.]的比率超过90%(附加文件2:图S2B)。在这些对齐的reads中,221个品种(81.5%)与目标SNP区域的对齐率超过80%(附加文件)2:图S2C)。此外,还分析了Target SNP-seq均匀性指数,该指数用于计算每个品种覆盖超过平均深度值10%的比例。本研究的平均均匀度指数为93.68%(附加文件2:图S2D),表明92个SNPs之间的序列深度具有较高的一致性。
271个辣椒品种的完美snp
遗传参数MAF何,他,以及每个完美SNP所揭示的PIC在附加文件中给出10:表S3。MAF是衡量标记物辨别能力的指标;因此,双等位基因标记的MAF越接近0.5,识别性能越好。在这项研究中,28.26%的完美snp的MAF在0.4到0.5之间,而只有4个snp的MAF低于0.1(附加文件)3.:图S3A)。的何每个SNP值在0.01 (CaSNP079) ~ 0.59 (CaSNP009)之间,平均为0.28,其中11个SNP值较高何(> 0.4)(附加文件3.:图S3B;额外的文件10:表S3)。此外,他值范围从0.01 (CaSNP079)到0.5 (CaSNP043和CaSNP094)(附加文件3.:图S3C;额外的文件10:表S3),而PIC值在完美snp之间的变化范围为0.01 (CaSNP079)至0.38 (CaSNP043, CaSNP094和CaSNP117),平均值为0.31(附加文件3.:图S3D;额外的文件10:表S3)。71.74%的完美snp的PIC值大于0.30,只有4个snp的PIC值小于0.2。上述结果表明,完美snp图谱具有较高的品种识别能力,其中CaSNP043、CaSNP94、CaSNP117和CaSNP009的品种识别能力最好。总的来说,结果表明,目标SNP-seq可以作为辣椒基因分型的快速工具。
水果形状上的完美snp
对4个果形居群遗传多样性的遗传参数平均值进行了比较,结果表明,块状果形居群遗传多样性的平均值最低他(0.18),何(0.16), PIC (0.15)1),表明该种群的遗传多样性最低。相比之下,长角果居群表现出最高的遗传多样性他(0.39),何(0.36), PIC(0.31)。
在某些果居群中,共有21个SNP位点没有显示出任何多样性(PIC = 0),其中块状果、长角果、短角果和线形果居群分别有16个、1个、3个和5个位点(附加文件)10:表S3)。这些果实形状特异性位点可能是在育种过程中被选择的,也可能是由于与决定果实性状的基因有联系而被选择的。
一个核心snp集的识别
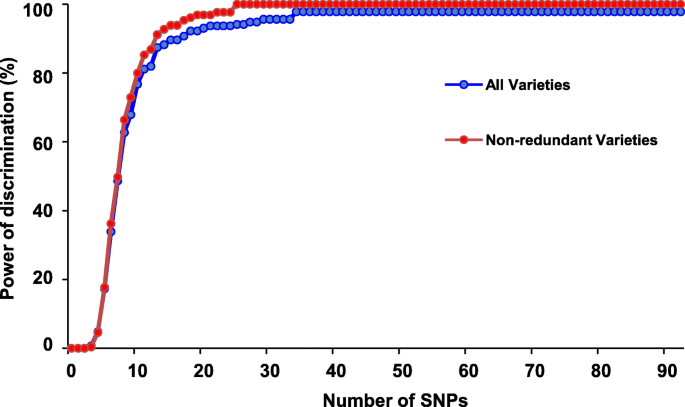
完美的SNP面板区分了271个辣椒品种中的97.7%(图2)。3.),其余的表现出相同的多位点基因型,也难以与田间表型区分。考虑到一些品种可能存在多个名称,具有相同基因型的品种可能是冗余的,因此被丢弃以建立非冗余基因型品种。因此,至少有27个完美SNPs可以区分所有非冗余的品种(图2)。3.).
为了为KASPar平台开发核心SNP集,每个完美SNP标记都在23到95个辣椒品种上进行了测试,使用两个等位基因特异性正向引物和一个通用反向引物。结果显示,35个SNP引物(附加文件11:表S4;额外的文件4:图S4)使用Target SNP-seq产生了一致且可重复的结果。最后,提出了35个在所有品种中识别率高达97%,在非冗余品种中识别率为100%的SNPs,作为KASPar平台使用的核心SNPs集(图2)。3.和附加文件4:图S4;额外的文件11:表S4)。
辣椒品种的遗传结构
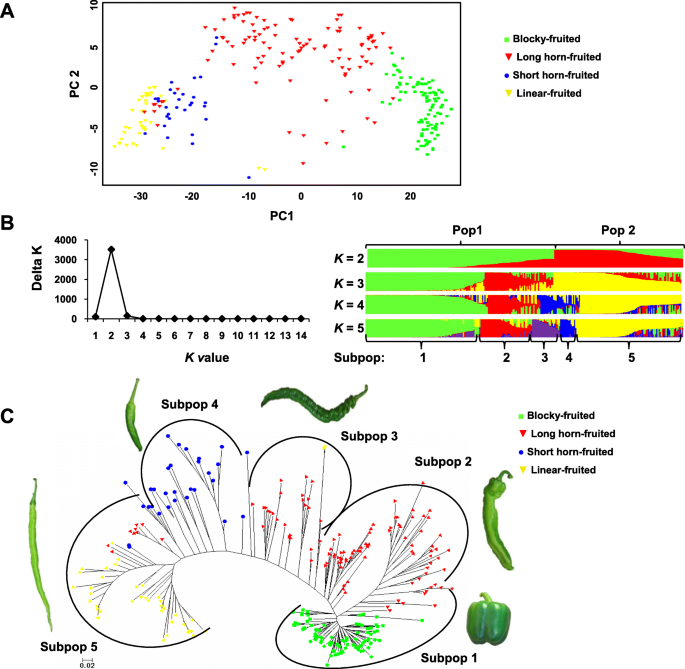
使用92个完美snp进行主成分分析(PCA),以调查271个品种的群体聚类(图2)。4a).根据PCA图,四个果形居群总体上是单独聚类的。块状果品种分布非常集中,而长角果品种分布相对分散。线形果品种与短角果品种的亲缘关系比与长角果或块状果品种的亲缘关系更密切。线性和块状果的种群是最多样化的,这些集群没有重叠,这表明在整个育种历史中存在相当大的遗传差异。值得注意的是,长角果和短果品种的选择显示出与线形果种群的密切关系。
利用聚类程序structure进一步推断271个品种的群体结构,对2 ~ 5个聚类数进行测试(K).伊万诺修正[38]显示了三角洲的峰值K在K= 2,表示存在两个主要种群,记为Pop1和Pop2。Pop1有160个品种(59.0%),包括所有块状果品种,60.2%的长角果品种,只有两个线形果品种(图2)。4b;额外的文件8:表S1)。其余111个品种(41.0%)归属于Pop2,包括所有短角果和线形果品种,以及39.8%的长角果品种(图2)。4b;额外的文件8:表S1)。当K= 3时,Pop1又细分为块状和长角果型两个簇。在K= 4时,将56%短角-、15长角-和2个线果品种的混合分配到Pop2的一个新聚类中,将这些短角-和一个新长角果组分别分配到独立聚类中K= 5。值得注意的是,线性结果类型从未被分配到一个独立的集群K是增加了。考虑到种群分类与果实类型高度相关K= 5时,将两个主居群进一步细分为5个亚居群(Subpop1~Subpop5;无花果。4b;额外的文件8:表S1)。Subpop1、2、3和4的结构清晰,没有或只有很少的外加剂。Subpop1共有98个品种,其中90个属于块果品种,8个属于长角果品种。长角果品种同时是Subpop2和Subpop3的成员,这并不奇怪,因为长角果品种同时分布在Pop1和Pop2中。Subpop2由44个长角果品种组成。Subpop3共24个品种,其中长角果品种22个,线果品种2个。Subpop4由14个短角果品种组成。与PCA分析结果一致,外加剂主要分布在Subpop5中,包含41个长线形果品种,以及少数短角果和长角果品种。
无根系统发育树(图;4c)与前面提到的主成分分析和基于模型的种群结构一致,并表明尽管有添加剂,但四种水果形状有明显的区别。根据对应亚群体内与其他品种的最低平均遗传距离选择的代表性品种的图像如图所示。4c.来自Subpop2和Subpop3的两个长角果品种的代表图像清晰地显示出不同的形态。
总之,三种独立的分析方法有力地支持了辣椒品种划分为五个分化良好的遗传群体,这些遗传群体与不同的果实形状相关,表明这些栽培品种的遗传结构可能受到育种实践中果实形状选择的强烈影响。
辣椒居群遗传变异评价
利用分子方差分析(AMOVA)对Pop1和Pop2的结果进行比较,发现总遗传变异的33.04%分布在Pops之间,8.47%分布在Pops内部,其余58.49%分布在品种内部(表2)2).AMOVA分析进一步表明,变异最大(63.83%)发生在品种内,变异最小(3.54%)发生在子品系内,变异在子品系间占32.63%2),表明subpop之间的差异相对适中。
为了测试Pops之间和Subpops之间的显著差异,执行了随机化测试(附加文件5:图S5)。输出显示了代表随机化地层分布的六个直方图。输出的观测结果表明,考虑到pop和subpop地层的所有层次,pop和subpop的结构有明显的差异(附加文件)5:图S5)。这些结果也支持了两个Pops和五个Subpops的分离。的两两估计F圣表明Pop1和Pop2的种群分化较高(F圣= 0.35)。成对的F圣Subpop2和Subpop3(主要由长角果品种组成)之间的差异为0.13,Subpop1(主要是块状果品种)和Subpop4(短角果品种)之间的差异为0.483.).值得注意的是,高遗传分化(F圣Subpop1和Subpop5之间的遗传分化率为0.43,而Subpop4和Subpop5之间的遗传分化率较低(F圣= 0.14)。
果实形状相关基因座的鉴定
271个辣椒品种的果形指数(FSI)变化范围很大(附加文件)12:表S5)。块状果、长角果、短角果和线形果群体的平均FSI分别为1.34、4.98、4.70和16.56。在块状、角状和线形种群中观察到显著差异(p< 0.01),长角果群与短角果群间无显著差异。FSI值大于9.5是线性水果的典型特征。
观察到种群结构和果实形状之间的一致性(图;2),接下来我们对271个品种和165个遗传位点的FSI进行了关联分析,其中包括92个SNPs和另外73个SSRs,这些都是通过Target测序检测到的(附加文件)6:图S6)。使用k + q在混合线性模型(MLM)下,共鉴定出9个位点(CaSSR013、CaSSR090、CaSSR105、CaSSR091、CaSSR039、CaSSR044、CaSSR107、CaSSR077和CaSNP112)在一定阈值下与FSI显著相关p-value of 0.0001(附加文件7:图S7;额外的文件13:表S6)。为了与先前确定的数量性状位点(QTL)配对,Zunla-1参考基因组中9个位点的物理位置[3.]及CM334 [13]在表中提供4.casr091位点和casr039位点位于同一染色体的820 kb内,被认为是一个单位。因此,这9个位点分别位于1、2、3、4、6和12号染色体上的8个染色体区域,每个位点解释的表型变异在7.9 ~ 12.7%之间。CaSSR044和CaSSR107这两个在6号染色体上约39 Mb的位点解释了最高的表型变异,分别为12.4%和12.7%(表2)4和附加文件13:表S6;额外的文件7:图S7)。
讨论
目标SNP-seq高通量基因分型
高通量基因分型技术已成为有效作物育种计划的必要条件。目标SSR-seq结合了完美ssr的多重扩增和高通量测序,最近被开发出来并应用于黄瓜品种的鉴定,导致了一组核心ssr的表征[35].该测序技术可在72小时内获得数千个数据点,成本低于7美元/份,由于覆盖率高,基因分型准确率可达100%。本研究开发的Target SNP-seq的成本与Target SSR-seq的成本相似,因为这两种技术在构建目标库时使用了相同的程序。
在这项研究中,使用重测序工具从35人的基因组中鉴定出92个完美snpc .建立基于严格的筛选标准.只有9.8%的完美SNPs落在基因区域内,这与之前的结果一致,即基因区域的变异密度明显低于基因间区域[30.].然后,研究人员将这些完美SNP用于目标SNP测序,以评估271个中国流行辣椒品种的遗传多样性。结果表明,完美SNP面板具有较高的品种鉴别能力,71.74%的完美SNP的PIC值为> 0.3010:表S3)。此外,至少有27个完美snp可以区分所有非冗余的品种(图2)。3.).值得注意的是,PIC平均值为0.31,低于SSR标记研究得出的值[17,39].这些差异可以用不同类型标记的性质来解释;SSRs是多等位基因,比单核苷酸多态性(SNP是双等位基因)更具多态性[40].差异的另一个原因可能是由于商业品种与地方品种相比变化较少或种质资源收集广泛。
一组35个核心SNPs与92个完美SNPs具有相同的辨别能力,成功地转化为KASPar标记,代表了辣椒品种的另一种可靠的基因分型选择(图2)。3.;额外的文件11:表S4)。与SSR标记不同,SNP标记不需要在每个实验中包含参考品种,也将克服实验室之间关于SSR等位基因的混淆。
近交系种群结构c .建立行
自从辣椒最初在墨西哥被驯化以来,它们的果实形状和大小就受到了强烈的选择。56].全球各地的消费习惯和辣椒类型偏好各不相同。仅在美国,就有超过20种市场类型被认可和消费。57].在中国,大多数商业栽培的辣椒品种都属于该物种c .建立,市场类型按水果形状分类,如流行的块状水果、长角水果、短角水果和线形水果[58,59].迄今为止,大多数实验都评估了几种基因之间的关系辣椒物种(29,30.,36,37,60,61,62的遗传多样性c .摘要而且c . baccatum来自相对限制地区的种质[26,63].基于分子标记、泛基因组测序和GBS的系统发育分析证实了这一点c .摘要而且c . frutescens彼此之间的关系比c .建立[29,61,64].几项研究试图描述栽培植物的种群相关性c .建立在受限制的地理范围内[29,36,40,41,42,65].他们揭示了该物种的种群结构c .建立在植物和果实描述符方面,品种主要与不同的品种类型有关,因此主要是人类根据消费模式选择品种类型和适应高度多样化的农业气候条件的结果。值得注意的是,35个基因之间的关系被重新排序c .建立本研究中描述的行与以前的报告分组一致c .建立根据果实性状[29,41,65].此外,在之前的研究中,也可以观察到块状果辣椒相对于小的辣味Chiltepin-like类型的最远位置的聚集[29,41,65].
遗传结构c .建立品种
尽管之前的研究已经表明c .建立中国地方品种按品种类型聚类[36],这些关系在商业上很重要c .建立来自不同公司的品种还没有用一套精细的遗传标记进行研究。在本研究中,在中国栽培的广泛辣椒品种中评估了四种果形种群之间的关系。遗传参数比较显示最低何在块果种群中观察到,而在角果种群中检测到最高(表1).这些发现与早期的研究一致,即相对于刺激性品种,非刺激性块状果系的多样性减少有关[41,42,43,44].与块状果实品种相关的狭窄遗传多样性可能是与有限基因库的近亲繁殖的结果。
此外,主成分分析和系统发育树表明,四个果形群体分别聚在一起,很少或没有重叠。这与果实形状分类系统一致,表明中国辣椒品种的遗传结构受到选择果实形状的育种计划的显著影响。相似地,STRUCTURE分析将这些品种划分为Pop1和Pop2两个主要居群,并进一步划分为Subpop1 ~ Subpop5 5个亚居群(图2)。4b).此外,亚群体与果实形状相关。Subpop1、Subpop4和Subpop5分别对应块状、短角和线形果实品种。然而,大多数长角果品种被分为两个亚群体,Subpop2和Subpop3,这两个亚群体在统计学上是唯一的(附加文件)5:图S5)。本研究将辣椒品系和品种遗传结构的最优拟合分为两组,与368份中国辣椒品种遗传结构的最优拟合结果不同c .建立Zhang等人分析的资料[36,18],采用28个SSR标记,将材料分为3个结构组。这些差异可能归因于两项研究中使用的辣椒材料类型和标记数量的不同。然而,两项研究中对水果类型的聚类似乎有些相似,尽管使用了不同的水果形状分类。例如,Group1主要包括矩形、方形和三角形水果类型[36],也主要聚集在本研究的Pop1中。第3组主要由果实小而长的品种组成,其特点是果实长宽比非常高[36],这是本研究Pop2中线果和部分短角果品种的特征。综上所述,我们的研究为辣椒品种果实形状的群体结构提供了有价值的见解,并证实了育种家果实形状选择对辣椒品种遗传结构的强烈影响。
果实形状相关基因座的鉴定
果实形状是辣椒育种中一个重要的性状。在甜椒和小果辣椒杂交的种内和种间群体中已鉴定出许多控制FSI的qtl [45,46,47,48,49,50,51,52,53].辣椒中第一个FSI QTL,命名为fs3.1,在连锁群3上检测到[48].该QTL随后在其他连锁分析中被检测到[46,47,51].Colonna等人(2019)最近通过对373个辣椒品种的全基因组关联研究,发现位于基因CA03g16080外显子的3号染色体上的SNP 3:183386147与FSI显著相关[30.].在目前的研究中,FSI相关位点casr105及其最近的上游标记CaSNP029位于3号染色体上约26 Mb的间隔内(附加文件)13:表S6)。该FSI相关区域覆盖了基因中已报道的FSI相关位点SNP 3:183386147叶1(CA03g16080) [30.].Zygier等人(2005)绘制了一个水果形状的QTL (fs2.1)在第2号染色体上[50].该QTL在其他研究中也被检测到,并被发现接近卵圆形的CM334基因组中约158 Mb的CA02g22830基因[52,53].在目前的研究中,FSI相关位点CaSSR090及其最近的下游位点CaSSR024,在2号染色体上的间隔跨度约为12.6 Mb,覆盖了所报道的卵圆形的基因(CA02g22830)。的卵圆形的基因最初是在番茄中发现的,它控制着果实形状从圆形到梨形的转变[54,55].单个FSI QTL (fs4.2),这解释了26.1%的表型变异[50].我们发现两个FSI相关位点casr091和CaSSR039位于CaSNP041和4号染色体末端之间,覆盖约12.9 Mb(附加文件)13:表S6)。本研究还检测到染色体1、6和12上存在FSI关联位点(附加文件)6:图S6和附加文件7:图S7;表格4和附加文件13:表S6)。在对这些关联位点的蛋白质功能进行筛选后,我们发现两个卵圆形的CA06g21580和CA12g07370基因可以作为候选基因,因为它们对果实形状有显著影响。
辣椒中SNP-seq靶标的研究方向
值得注意的是,适用于特定引物设计的前景选择标记也可以添加到我们的Target SNP-seq中使用的完美SNP面板中。例如,基于功能的网站Tobamovirus耐药基因l3.而且l4[66),疫霉capsici抗性基因CaDMR1和Phyto5NBS1 [67,68],细菌斑点抗性基因Bs3[69),而马铃薯Y病毒耐药基因pvr1[70],已成功开发出功能位点侧翼区域的特定引物(附加文件14:表S7),并添加到完美的SNP面板。这些抗性基因的功能位点,结合完美的SNP和SSR标记,可以通过Target SNP-seq在数百个辣椒品种中同时检测到。这项技术的商业应用有可能提高标记相关选择程序的效率,以及帮助品种鉴定。
结论
本研究开发的目标SNP-seq是辣椒遗传多样性研究、品种鉴定和群体结构表征的高通量可靠工具。利用主成分分析(PCA)、系统发生树生成和结构分析(STRUCTURE)表明,中国市上辣椒品种的遗传结构受到育种果实形状选择的显著影响。最后,对有限数量的标记进行关联分析,可以识别先前报道的和控制水果形状的新基因组区域。
方法
植物材料、果实形态分类、DNA提取
本研究对国内60家不同育种公司提供的271个辣椒品种进行了分析。关于这些杂交种子的信息,包括品种名称和来源,可在附加文件中找到8:表S1。利用北京蔬菜研究中心辣椒遗传育种组和高通量分子育种平台进行了果实性状调查和遗传鉴定。
品种在北京市通州区蔬菜品种展示中心的温室条件下种植。温室温度为25 - 30℃(08:00-20:00)和20-25℃(20:00-08:00),有自然光。每个品种至少由四株植物组成。果实被分为四种水果形状之一:块状、长角状、短角状和线形果型(附加文件)8:表S1)。水果形状分类的例子在附加文件中给出1:图S1。使用游标卡尺测量每株植物中4至10个成熟果实的最大高度和宽度(杭州工具测量工具公司,中国杭州)。
采用基于ctab的方法,从每个品种的个体中随机选择4株幼苗提取DNA [71].DNA完整性使用1.5% (w/v)琼脂糖凝胶电泳进行评估,浓度使用Nanodrop 2000分光光度计(Thermo Fisher Scientific, DE, USA)测定。
重新测序,完善SNP鉴定
总共有31种不同的辣椒品种(c .建立),包括来自我们正在进行的BVRC育种项目和美国国家植物种质资源系统提供的PI640446的30个自交系,这些自交系被选择在上海马约彼生物harm科技有限公司的Illumina X Ten平台上重新测序。这31个辣椒系具有不同的遗传背景和园艺性状,包括8个块状果系、13个长角果系、5个短角果系和5个线状果系(图2)。1).
31个重新测序系和4个先前测序品种的原始读数;Zunla-1 (c .建立)和它的野生祖先Chiltepin (c .建立var。glabriusculum) [3.],c .建立简历.常年和c .建立简历.邓普西(13],使用Trimmomatic [72].然后将干净的reads映射到Zunla-1染色体2.0版本的参考基因组[3.]使用默认参数的burws - wheeler比对工具(BWA),使用基因组分析工具包(GATK, v2.4-7g5e89f01)调用snp [73].将MAF > 5%、缺失数据< 10%的SNPs导入MEGA,以栽培品种前体Chiltepin为外群,采用邻域连接法构建根系系统发育树[74].群体结构分析使用structure v2.3完成。人口数目(K)按标准程序确定[75磨合周期为10万次,马尔可夫链蒙特卡洛为10万次。进行了20次独立下入K从1到15不等。最优K是根据埃瓦诺函数定义的吗K方法(38].
为了获得用于后续目标SNP-seq分析的全基因组snp数据集,使用以下标准确定了完美的snp:(i) MAF > 0.4以过滤掉无信息的snp;(ii)漏报率< 0.2;(iii)杂合度< 0.2;(iv) SNP位点100 bp侧翼序列无序列变化;(v)每个位点有2个等位基因。
目标SNP-seq
目标SNP-seq过程如前所述使用上述确定的snp完成[35].简而言之,目标SNP-seq的文库构建包括以下两轮PCR:第一轮使用完美SNP引物的多路面板扩增并捕获DNA样本中的目标SNP(附加文件)9:表S2);第二轮为每个DNA样本的捕获产品添加了唯一的条形码。因此,样品是根据不同的条形码区分的。在含有50 ng基因组DNA模板、8 μl多重snp捕获面板引物(10 μM)、10 μl 3m酶(Molbreeding Biotechnology Company, China石家庄)的30 μl反应混合液中进行多重PCR。PCR混合物在95°C下加热5分钟,然后在95°C下加热30秒,60°C下加热4分钟,72°C下加热4分钟,最后在72°C下延长4分钟。PCR产物用磁珠悬浮液和80%乙醇纯化。同样,第二次PCR扩增在30 μl的反应体积中进行,反应体积中含有11 μl的前一轮PCR纯化产物,10 μl的3 MTaq酶(Molbreeding Biotechnology Company, China石家庄),无核酸酶水8 μl,引物1 μl,引物序列为正向5′- AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGA - cgctcttccg -3′和反向5′- caagcagaagacggcatacgagat -XXXXXXXXGTGACTGGAGTTCCTTGGCACCCGAGA-3 '(条形码由下划线序列表示)。PCR过程为95℃,时间为3 min;在95°C持续15秒,58°C持续15秒,72°C持续30秒,最后在72°C持续4分钟。用100 μl 80%乙醇和23 μl Tris-HCl缓冲液(10 mM, pH 8.0 ~ 8.5)纯化PCR产物。在此之后,在Molbreeding Biotechnology Company(中国石家庄)使用Illumina X Ten平台对Target SNP-seq文库进行测序。
目标SNP-seq的SNP基因型分析
使用Illumina bcl2fastq管道(Illumina, San Diego, CA, USA)对来自Target SNP-seq的原始数据进行解复用,以基于样本特异性条形码确定每个品种的确切基因型。使用Trimmomatic软件过滤干净的数据,将每个品种的reads映射到Zunla-1辣椒参考基因组[3.]使用缺省参数的BWA。计算各品种的序列深度、比对率以及目标比对率和均匀度,评价目标比对结果。
均匀性推断为深度为>的SNPs占平均深度的10%。
其中S为Target SNP-seq生成的总碱基;M为品种总数;N为snp的总数;L:平均读长度。
用GATK进行SNP基因型的命名。根据高通量测序结果,读取次数最多和读取次数第二多的SNP等位基因分别作为每个目标SNP位点的主、次等位基因。当主等位基因的读频大于0.7时,该位点被描述为纯合位点。如果主、次等位基因的读频均大于0.35,则该位点为杂合位点。
每个品种的序列深度=品种生成的总碱基/ 92个目标基因组区域的总碱基长度。
确定每个完美SNP的遗传参数
完美snp的遗传参数统计,包括观察到的杂合度(Ho),期望杂合度(He),以及多态资讯含量[76使用Perl脚本计算,公式如下:
在哪里l等位基因位点,和P我而且Pj表示的总体频率我th而且jth等位基因。使用Circos软件绘制完美SNPs的染色体分布(http://circos.ca/), SNP区域放大到2 Mb。
遗传结构分析
用三种不同的方法研究了品种间的遗传关系:PCA、STRUCTURE和系统进化树。使用R中的FactoMineR包进行主成分分析[77].STRUCTURE v2.3实现的基于贝叶斯的模型过程[75,78]也被用来确定人口结构。人口数目(K)按上述方法测定,并使用猿而且Poppr基于使用MEGA v5.1查看的树的邻居连接方法的R中的包[79,80].
种群多样性分析
不同水果形状的种群,以及由结构推断出的亚种群,何,他采用上述方法计算PIC、MAF分析。为了测量群体和亚群体的遗传差异,采用AMOVA和两两比较F圣使用popprR包和函数成对。Neifst在HierfstatR包,分别为[81,82].进一步进行随机化检验,以检验差异的意义,采用函数随机检验ade4包(81].AMOVA和随机化试验的详细说明可在https://grunwaldlab.github.io/Population_Genetics_in_R/AMOVA.html而且https://rdrr.io/cran/poppr/man/poppr.amova.html.
完美snp的鉴别能力
为了确定区分最大辣椒品种数量的最小SNPs数量,开发了一个Perl脚本,根据以下算法确定1到92个完美SNPs的最佳识别能力。
1)第一个SNP的选择:a)对每个SNP进行品种间两两比较,每个SNP 36585个比较;b) Xij如果j存在基因型差异= 1thI的两两比较thSNP (i = 1,2,3,…,92;J = 1,2,3,…,36,585)。Xij如果j的基因型差异不存在,则= 0thI的两两比较thSNP (i = 1,2,3,…,92;J = 1,2,3,…,36,585);c)最大值为的SNP\({\sum}_{j=1}^{36585}\math {Xij} \)在92个SNP中被选为第一SNP。2)选择最好的两个SNP: a)第一个选择的SNP和其余91个SNP组成91个SNP组合;b)对91个SNP组合中的每个组合进行了36583个配对比较;c) X乔丹如果j存在基因型差异= 1thm的两两比较thSNP组合(m = 1,2,3,…,91;J = 1,2,3,…,36,583)。X乔丹如果j的基因型差异不存在,则= 0thm的两两比较thSNP组合(m = 1,2,3,…,91;J = 1,2,3,…,36,583);d)最大值为的SNP组合\({\sum}_{j=1}^{36585}\math {Xmj} \)被选为最佳的两个snp。如果SNP组合值相同,则优先选择与第一个SNP位于不同染色体上的第二个SNP的SNP组合作为最好的两个SNP。3)最佳3个SNP的选择:a)最佳2个SNP和其余90个SNP组成90个SNP组合;b), c), d)步骤与步骤2)相同,选择最佳的3个snp。如果SNP组合值相同,则优先选择位于不同染色体上的第3个SNP作为第1个和第2个SNP作为最佳3个SNP。如步骤3所示,逐步筛选出4 ~ 92个最佳SNPs,其中1 ~ 92个最佳SNPs的辨别能力按以下公式计算:辨别能力=具有独特基因型的品种数/ 271。饱和度曲线由1到92个最佳snp的判别能力绘制。3.).高分辨能力指的是高饱和度和高SNP辨别能力。
设置核心SNPs用于品种鉴别
为了利用KASPar平台开发一套能够区分不同品种的核心SNP,为每个完美SNP标记设计了两个等位基因特异性的正向引物和一个通用的反向引物。然后,通过KASPar平台对23 - 95个商业品种进行SNP标记的潜在效用评估;如前所述,检测荧光[83].详细说明可在www.kbioscience.co.uk.如上所述,使用Perl脚本从成功验证的SNP标记中选择核心SNP集。最后,将具有最大品种识别力(饱和度值最高)的SNP标记鉴定为核心SNP集。核心snp标记的引物序列见附加文件11:表S4。
关联分析
每个品种的FSI以最大高度与最大宽度之比计算。92个SNP位点(附加文件9:表S2)结合73个SSR位点(附加文件6:图S6),均为本研究开发的271个品种的目标测序检测样本,用于关联分析。用于SSRs目标库构建和检测的方法与target SNP-seq中使用的方法相同。采用TASSEL 5.2.25软件进行关联分析。同时考虑果形群体(Q矩阵)和亲缘矩阵(K矩阵)和以果形群体(Q矩阵)为固定因子的一般线性模型(GLM)进行果形位点的关联识别。标记-性状关联的显著性在p-value小于10−4.因为它已被普遍证明,传销+ Q +K模型在检测位点方面比其他模型更有效[84,85],本研究仅给出了MLM + Q + K模型的数据。每个完美SNP所解释的表型变异为R2-从传销中获取的值。从使用CM334基因组发表的蛋白质注释中鉴定出与显著相关位点最近的上游和下游SNP位点之间的候选基因[3.].
数据和材料的可用性
本文报道的原始序列数据已存入中国科学院北京基因组研究所(BIG)大数据中心(大数据中心成员,2019)基因组序列资料馆,登录号为CRA001576。这些数据可以在http://bigd.big.ac.cn/gsa.
缩写
- 妊娠:
-
扩增片段长度多态性
- AMOVA:
-
分子方差分析
- 投资策略基金会:
-
果实形状指数
- GATK:
-
基因组分析工具箱
- GBS:
-
Genotyping-by-sequencing
- GBTS:
-
通过目标测序进行基因分型
- 全球语言监测机构:
-
一般线性模型
- 他:
-
预期的杂合性
- 何:
-
观察到的杂合性
- InDel:
-
插入或删除
- 卡斯帕·:
-
竞争性等位基因特异性PCR
- 加:
-
小等位基因频率
- 传销:
-
混合线性模型
- 主成分分析:
-
主成分分析
- 图片:
-
多态性信息内容
- RAPD:
-
随机放大多态
- RFLP:
-
限制片段长度多态性
- SNP:
-
单核苷酸多态性
- 苏维埃社会主义共和国:
-
简单序列重复
参考文献
- 1.
Moscone EA, Scaldaferro MA, Grabiele M, Cecchini NM, Sánchez García Y, Jarret R, Daviña JR, Ducasse DA, Barboza GE, Ehrendorfer F.辣椒的进化(辣椒-茄科):细胞遗传学的观点。植物学报。2007;745:137-70。https://doi.org/10.17660/ActaHortic.2007.745.5.
- 2.
Olmstead RG, Bohs L, Migid HA, Santiago-Valentin E, Garcia VF, Collier SM。茄科的分子系统发育。分类单元。2008;57:1159 - 81。https://doi.org/10.1002/tax.574010.
- 3.
秦超,于春春,沈洋,方晓东,陈琳,闵建民,程建文,赵顺生,徐敏,罗勇,等。栽培和野生辣椒的全基因组测序提供了深入了解辣椒驯化和专业化。美国国家科学研究院。2014; 111:5135-40。https://doi.org/10.1073/pnas.1400975111.
- 4.
Andrews J. pepper:被驯化的人辣椒.奥斯汀:德克萨斯大学出版社;1984.
- 5.
高鹏,马华,栾峰,宋华。甜瓜DNA指纹图谱为籽粒品质评价提供了证据支持。公共科学图书馆,2012;7:e52431。https://doi.org/10.1371/journal.pone.0052431.
- 6.
田海林,王芳芳,赵建荣,易红梅,王磊,王锐,杨勇,宋伟。中国玉米品种DNA指纹鉴定高通量兼容SNP阵列玉米esnp3072的构建。Mol Breed. 2015;35:136。https://doi.org/10.1007/s11032-015-0335-0.
- 7.
McCouch SR, Chen XL, Panaud O, Temnykh S,徐亚波,Cho YG, Huang N, Ishii T, Blair M.微卫星标记的开发、定位及其在水稻遗传育种中的应用。植物分子生物学杂志,1997;35:89-99。https://doi.org/10.1023/a:1005711431474.
- 8.
Nagaraju J, Kathirvel M, Kumar RR, Siddiq EA, Hasnain SE。利用基于荧光的ISSR-PCR和SSR标记对传统和进化的巴斯马蒂和非巴斯马蒂水稻品种进行遗传分析(第99卷,第5836页,2002年)。美国国家科学研究院。2002; 99:13357。https://doi.org/10.1073/pnas.212463799.
- 9.
杨晓明,李志强,李志强,等。黄曲霉致病性及RAPD分析疫霉nicotianae突尼斯辣椒的致病性。中国生物医学工程杂志,2007;70:142-8。https://doi.org/10.1016/j.pmpp.2007.08.002.
- 10.
Lanteri S, Acquadro A, Quagliotti L, Portis E.一个地方辣椒品种遗传变异的RAPD和AFLP评价(甜椒L.),产于意大利西北部。作物进化,2003;50:723-35。https://doi.org/10.1023/a:1025075118200.
- 11.
Lefebvre V, Palloix A, Rives M.辣椒品种间的核RFLP (甜椒L). Euphytica. 1993; 71:189-99。https://doi.org/10.1007/BF00040408.
- 12.
Tanksley SD, Bernatzky R, Lapitan NL, Prince JP辣椒和番茄基因序列的保存与排序不一致。美国国家科学研究院。1988; 85:6419-23。https://doi.org/10.1073/pnas.85.17.6419.
- 13.
金森,朴敏,严思,金玉敏,李建民,李夏,徐娥,崔杰,郑坤,金KT,等。辣椒的基因组序列为人类辛辣感的进化提供了新的视角辣椒物种。Nat Genet, 2014; 46:270-8。https://doi.org/10.1038/ng.2877.
- 14.
郭国军,张光亮,潘广斌,刁伟平,刘建军,葛伟,高长忠,张勇,蒋超,王顺生。InDel标记的开发与应用辣椒sp .基于全基因组重测序。科学通报2019;9:3691。https://doi.org/10.1038/s41598-019-40244-y.
- 15.
李伟文,程建文,吴志明,秦超,谭松,唐旭,崔俊杰,张磊,胡建林。基于indel的辣椒连锁图谱(甜椒).Mol Breed. 2015;35:32。https://doi.org/10.1007/s11032-015-0219-3.
- 16.
谭松,程建文,张磊,秦春,农大栋,李伟平,唐霞,吴志明,胡建林。基于InDel和SSR的辣椒花原基启动qtl的构建(辣椒spp)。科学通报。2015;10:e0119389。https://doi.org/10.1371/journal.pone.0119389.
- 17.
Yumnam JS, Tyagi W, Pandey A, Meetei NT, Rai M.基于形态学、SSR标记和遗传多样性的印度东北地方辣椒品种遗传多样性评价Pun1轨迹。植物摩尔生物学报告。2012;30:1470-9。https://doi.org/10.1007/s11105-012-0466-y.
- 18.
张晓峰,孙海辉,徐勇,陈斌,余顺生,耿世生,王强。基于辣椒RIL群体的大量SSR和InDel标记的开发及高密度遗传图谱的构建(甜椒l .)。Mol Breed. 2016;36:92。https://doi.org/10.1007/s11105-012-0466-y.
- 19.
Aguilar-Meléndez A, Morrell PL, Roose ML, Kim SC.半野生和驯化辣椒的遗传多样性和结构(甜椒;茄科)来自墨西哥。中国科学(d辑),2009;https://doi.org/10.3732/ajb.0800155.
- 20.
杨晓明,王晓明,王晓明,王晓明,等。家蚕分类与遗传多样性研究辣椒安第斯地区的物种。作物学报,2012;49:1077 - 88。https://doi.org/10.1007/s10722-011-9744-z.
- 21.
Fan JB, Oliphant A, Shen R, Kermani BG, Garcia F, Gunderson KL, Hansen M, Steemers F, Butler SL, Deloukas P,等。高度平行SNP基因分型。清华大学学报(自然科学版),2003;68:69-78。https://doi.org/10.1101/sqb.2003.68.69.
- 22.
Steemers FJ, Gunderson KL.基于BeadArray™平台的全基因组基因分型技术。生物技术学报。2007;2:41-9。https://doi.org/10.1002/biot.200600213.
- 23.
Livak KJ, Flood SJA, Marmaro J, Giusti W, Deetz K.末端带有荧光染料的寡核苷酸提供了一种用于检测PCR产物和核酸杂交的淬灭探针系统。基因组决议1995;4:357-62。https://doi.org/10.1101/gr.4.6.357.
- 24.
姜建辉,杨海波,郑海辉,崔平,权志杰,姜bc。在标记辅助回交的转录组测序中发现单核苷酸多态性标记辣椒.科学通报。2014;32:535-43。https://doi.org/10.7235/hort.2014.14109.
- 25.
Taranto F, D 'Agostino N, Greco B, Cardi T, Tripodi P.辣椒全基因组SNP的发现及群体结构分析(甜椒)使用基因分型测序。BMC Genom. 2016;17:943。https://doi.org/10.1186/s12864-016-3297-7.
- 26.
Nimmakayala P, Abburi VL, Saminathan T, Almeida A, Davenport B, Davidson J, Reddy CV, Hankins G, Ebert A, Choi D, Stommel J.全基因组差异和连锁不平衡分析辣椒baccatum由基因组锚定的单核苷酸多态性揭示。PlantSci前面。2016; 7:1646。https://doi.org/10.3389/fpls.2016.01646.
- 27.
Nimmakayala P, Abburi VL, Saminathan T, Alaparthi SB, Almeida A, Davenport B, Nadimi M, Davidson J, Tonapi K, Yadav L, Malkaram S, Vajja G, Hankins G, Harris R, Park M, Choi D, Stommel J, Reddy UK。辣椒素类物质与果实重量的全基因组多样性及其关联图谱甜椒L.科学代表2016;6:38081。https://doi.org/10.1038/srep38081.
- 28.
Taitano N, Bernau V, Jardón-Barbolla L, Leckie B, Mazourek M, Mercer K, McHale L, Michel A, Baumler D, Kantar M, van der Knaap E.一个新的墨西哥辣椒收集的全基因组基因分型揭示了之后的地方种族分化历史甜椒l .驯化。科学通报。2018;12:78-92。https://doi.org/10.1111/eva.12651.
- 29.
Pereira-Dias L, Vilanova S, Fita A, Prohens J, Rodríguez-Burruezo A.辣椒遗传多样性、群体结构及亲缘关系(辣椒通过基因分型测序(GBS)发现来自西班牙多样性中心的本地人种。园艺决议2019;6:54。https://doi.org/10.1038/s41438-019-0132-8.
- 30.
Colonna V, D 'Agostino N, Garrison E, Albrechtsen A, Meisner J, Facchiano A, Cardi T, Tripodi P.基因组多样性与果实形态的新型全基因组关联辣椒,来自746k个多态性位点。科学通报2019;9:10067。https://doi.org/10.1038/s41598-019-46136-5.
- 31.
李林,方志文,周建峰,陈辉,胡泽峰,高丽芬,陈红林,任松,马友友,卢林,张文喜,彭慧。一种精确高效的SSR大规模基因分型方法及其应用。核酸,2017;45。https://doi.org/10.1093/nar/gkx093.
- 32.
Krasileva KV, Vasquez-Gross HA, Howell T, Bailey P, Paraiso F, Clissold L, Simmonds J, Ramirez-Gonzalez RH, Wang XD, Borrill P, Fosker C, Ayling S, Phillips AL, Uauy C, Dubcovsky J.多倍体小麦隐藏变异的发现。美国国家科学研究院。2017; 114:913-21。https://doi.org/10.1073/pnas.1619268114.
- 33.
姜丽,刘霞,杨娟,王海峰,蒋建良,刘丽丽,何松,丁小东,刘建峰,张强。GWAS位点定向重测序发现乳产量性状的新遗传变异。BMC Genomics. 2014;15:1105。https://doi.org/10.1186/1471-2164-15-1105.
- 34.
郭泽峰,王华伟,陶俊杰,任永华,徐超,吴克胜,邹超,张金娟,徐一兵。通过玉米目标测序(GBTS)基因分型,开发育种家负担得起的多个SNP标记面板。Mol Breed. 2019;39:37。https://doi.org/10.1007/s11032-019-0940-4.
- 35.
杨俊杰,张军,韩仁旭,张峰,毛俊杰,罗军,董宝宝,刘辉,唐辉,张金娟,温春林。目标SSR-seq:一种新型SSR基因分型技术,可用于黄瓜品种遗传分析。中国植物科学,2019;10:531。https://doi.org/10.3389/fpls.2019.00531.
- 36.
张晓明,张振华,顾晓珍,毛世林,李XX, Chadoeuf J, Palloix A,王lh,张bx。辣椒的遗传多样性(辣椒中国种质资源反映了品种类型的选择和空间分布。《中国农业科学》2016;15:1991-2001。https://doi.org/10.1016/s2095 - 3119 (16) 61364 - 3.
- 37.
孟CY,魏晓春,赵媛媛,袁玉玉,杨世杰,王志智,张晓文,孙金文,郑晓玲,姚启杰,张强辣椒通过SSR标记。植物学报。2017;8:70-8。https://doi.org/10.5376/mpb.2017.08.0008.
- 38.
李志强,李志强,李志强,等。利用软件STRUCTURE检测个体聚类的数量:模拟研究。分子生物学,2005;14:2611-20。https://doi.org/10.1111/j.1365-294X.2005.02553.x.
- 39.
李建民,李建民,金玉敏,金东彬。辣椒微卫星基因座的分子遗传定位。应用理论,2004;108:619-27https://doi.org/10.1007/s00122-003-1467-x.
- 40.
Taranto F, D'Agostino N, Greco B, Cardi T, Tripodi P.辣椒全基因组SNP的发现及群体结构分析(甜椒)使用基因分型测序。BMC Genomics, 2016;17。https://doi.org/10.1186/s12864-016-3297-7.
- 41.
Hill TA, Ashrafi H, reye - chin - wo S, Yao JQ, Stoffel K, Truco MJ, Kozik A, Michelmore RW, Van Deynze A甜椒基于30K单基因辣椒基因芯片并行多态性发现的遗传多样性和群体结构。科学通报。2013;8:e56200。https://doi.org/10.1371/journal.pone.0056200.
- 42.
所罗门AM,韩凯,李建辉,李海燕,张松,姜伯承。埃塞俄比亚的遗传多样性与群体结构辣椒种质。PLoS One, 2019;14:e0216886。https://doi.org/10.1371/journal.pone.0216886.
- 43.
Nicolai M, Cantet M, Lefebvre V, st -Palloix AM, Palloix a .辣椒基因分型研究(辣椒SSR位点的分析,为栽培的野生起源提供了新的证据c .建立通过人对品种类型的选择构建遗传多样性。作物学报,2013;https://doi.org/10.1007/s10722-013-0006-0.
- 44.
Tam SM, Lefebvre V, Palloix A, Sage-Palloix AM, Mhiri C, Grandbastien MA。ltr -反转录转座子Tnt1和T135标记揭示了驯化辣椒的遗传多样性和进化关系。应用理论,2009;119:973-89。https://doi.org/10.1007/s00122-009-1102-6.
- 45.
Chaim AB, Paran I, Grube RC, Jahn M, van Wijk R, Peleman J.辣椒果实相关性状QTL定位(甜椒).应用理论。2001;102:1016-28。https://doi.org/10.1007/s001220000461.
- 46.
韩坤,郑海杰,杨海波,姜思敏,权志强,金森,崔丹,姜bc。超高密度仓图有助于辣椒园艺性状的高通量QTL定位(甜椒).DNA决议2016;23:81-91。https://doi.org/10.1093/dnares/dsv038.
- 47.
亚奈斯·SC, Ashrafi H, reys - chino - wo S, Hill TA, Stoffel KM, VanDeynze A.辣椒素、果实品质和植株结构性状qtl的鉴定辣椒瑞来斯的人口。基因组。2013;56:61 - 74。https://doi.org/10.1139/gen-2012-0083.
- 48.
蔡志刚,杨晓明,李志刚,李志刚。花青素基因A位点与花青素存在的连锁关系fs10.1是辣椒中主要的果形QTL。应用理论,2003;106:889-94。https://doi.org/10.1007/s00122-002-1132-9.
- 49.
顾饶,王晓明,杨晓明,王晓明。辣椒品种间杂交产量QTL的定位甜椒而且c . frutescens.理论应用,2003;106:1457-66。https://doi.org/10.1007/s00122-003-1204-5.
- 50.
Zygier S, Chaim AB, Efrati A, Kaluzky G, Borovsky Y, Paran I.辣椒第2和4染色体果实大小和形状的QTL定位及其与番茄QTL图谱的比较应用理论,2005;111:437-45。https://doi.org/10.1007/s00122-005-2015-7.
- 51.
巴奇,李建平,李建平,李建平,李建平。辣椒植株发育和果实性状的QTL分析及其选择性表型表现。理论应用,2009;118:1157-71。https://doi.org/10.1007/s00122-009-0970-0.
- 52.
王晓明,王晓明,王晓明,王晓明,等。植物器官大小的基因组学研究进展甜椒.植物基因组,2017。https://doi.org/10.3835/plantgenome2017.03.0026.
- 53.
朱春华,李志刚,李志刚,李志刚。控制甜椒果实大小和其他园艺性状的数量性状位点分析(甜椒).植物基因组。2018;11:160125。https://doi.org/10.3835/plantgenome2016.12.0125.
- 54.
van der Knaap E, Chakrabarti M, Chu YH, Clevenger JP, Illa-Berenguer E,黄志军,Keyhaninejad N, Mu Q,孙玲,王亚萍,吴松。番茄果实重量和形状调控的分子机制。植物科学进展(英文版);https://doi.org/10.3389/fpls.2014.00227.
- 55.
Wu S, Zhang BY, Keyhaninejad N, Rodriguez GR, Kim HJ, Chakrabarti M, Illa-Berenguer E, Taitano NK, Gonzalo MJ, Diaz A, Pan YP, Leisner CP, Halterman D, Buell CR,翁yq, Jansky SH, van Eck H, Willemsen J, Monforte AJ, Meulia T, van der Knaap E.果实和其他植物器官形态多样性的共同遗传机制。Nat Commun. 2018;9:4734。https://doi.org/10.1038/s41467-018-07216-8.
- 56.
Kraft KH, Brown CH, Nabhan GP, Luedeling E, Ruiz JDL, d'Eeckenbrugge GC, Hijmans RJ, Gepts P.驯化辣椒起源的多条证据,甜椒在墨西哥。美国国家科学研究院。2014; 111:6165 - 70。https://doi.org/10.1073/pnas.1308933111.
- 57.
Bosland PW, Votava E.辣椒:蔬菜和香料辣椒.卡比:牛津,沃灵福德;2000.
- 58.
耿士生,陈波,张晓峰,孙景涛。中国辣椒育种发展及品种分布。中国辣椒。2011;1:1-5。可以从:https://www.ifabiao.com/lj/201103/15393478.html.
- 59.
耿士生,陈波,张晓峰,杜海生。中国辣椒品种的市场需求趋势及育种策略。中国蔬菜。2015;3:1-5http://www.cnveg.org/UserFiles/File/3-1.pdf.
- 60.
莫雷拉AFP,鲁亚斯PM,鲁亚斯CD,巴巴VY, Giordani W, Arruda IM, Rodrigues R, Goncalves LSA。果实性状的遗传多样性、群体结构和遗传参数辣椒.科学通报。2018;236:1-9。https://doi.org/10.1016/j.scienta.2018.03.012.
- 61.
欧丽娟,李东,吕建华,陈卫军,张志强,李晓峰,杨伯忠,周世德,杨松,李卫军,等。栽培辣椒全基因组(辣椒)及其在基因存在-缺失变异分析中的应用。New Phytol. 2018; 220:360-3。https://doi.org/10.1111/nph.15413.
- 62.
李hy, Ro NY,郑海杰,权JK, Jo J, Ha Y, Jung A, Han JW, Venkatesh J, Kang BC。遗传多样性和群体结构分析构建了一个庞大的核心集合辣椒种质。BMC Genet 2016;17:142。https://doi.org/10.1186/s12863-016-0452-8.
- 63.
杨晓明,王晓明,王晓明,等。基于微卫星技术的中国水稻遗传结构与多样性分析辣椒在新热带。作物学报。2014;61:741-55。https://doi.org/10.1007/s10722-013-0069-y.
- 64.
Baral JB, Bosland PW。解开物种困境辣椒frutescens而且c .摘要(茄科):使用形态学、分子分析和性相容性的多证据方法。环境科学学报,2004;29(2):344 - 344。https://doi.org/10.21273/JASHS.129.6.0826.
- 65.
冈萨雷斯-佩雷斯S,加尔塞斯-克拉弗A,马洛C,德米埃拉LES,法约斯O,波马尔F,梅里诺F,西尔瓦C辣椒SPP的亲缘关系与多元化过程甜椒在西班牙。科学通报。2014;9:e116276。https://doi.org/10.1371/journal.pone.0116276.
- 66.
杨海波,刘文伟,姜华辉,金志华,赵海杰,刘志华,姜bc。L等位基因特异性标记的开发与验证辣椒.Mol Breed. 2012; 30:819-29。https://doi.org/10.1007/s11032-011-9666-7.
- 67.
Rehrig WZ, Ashrafi H, Hill T, Prince J, Van Deynze A。CaDMR1与QTL进行协同隔离Pc5.1为了抵抗疫霉capsici胡椒(甜椒).植物基因组。2014;7:1-12。https://doi.org/10.3835/plantgenome2014.03.0011.
- 68.
刘文文,姜志华,郑海辉,崔海杰,杨海波,金KT,崔丹,崔桂杰,姜敏,姜bc。结合使用散装分离分析和微阵列揭示SNP标记,精确定位一个主要的QTL的抗性疫霉capsici在胡椒。理论应用,2014;127:2503-13。https://doi.org/10.1007/s00122-014-2394-8.
- 69.
罗默P, Hahn S, Jordan T, Strauss T, Bonas U, Lahaye T.辣椒启动子激活介导的植物病原体识别Bs3耐药基因。科学。2007;318:645-8。https://doi.org/10.1126/science.1144958.
- 70.
杨敏,姜建平,林德曼W, Frantz JD, Faber N, Jahn MM.基于点突变的水稻抗性等位基因CAPS标记pvr1编码eIF4E的基因座辣椒.理论应用,2005;112:178-86。https://doi.org/10.1007/s00122-005-0120-2.
- 71.
Fulton TM, Chunwongse J, Tanksley SD。从番茄和其他草本植物中提取DNA的微制备方案。植物摩尔生物学报告,1995;13:207-9。https://doi.org/10.1007/bf02670897.
- 72.
Bolger AM, Lohse M, Usadel B. Trimmomatic:一种用于Illumina序列数据的灵活修剪器。生物信息学。2014;30:2114-20。https://doi.org/10.1093/bioinformatics/btu170.
- 73.
McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo MA。基因组分析工具包:用于分析下一代DNA测序数据的mapreduce框架。基因组Res. 2010; 20:1297-303。https://doi.org/10.1101/gr.107524.110.
- 74.
Price MN, Dehal PS, Arkin AP. FastTree 2-用于大对齐的近似最大似然树。科学通报。2010;5:e9490。https://doi.org/10.1371/journal.pone.0009490.
- 75.
王晓燕,王晓燕,王晓燕,等。基于多位点基因型数据的种群结构推断。遗传学,2000;155:945-59https://www.ncbi.nlm.nih.gov/pubmed/10835412.
- 76.
Botstein D, White RL, Skolnick M, Davis RW。利用限制性片段长度多态性构建人类遗传连锁图谱。阿姆·J·胡姆·热内。1980;32:314-31可从:https://www.ncbi.nlm.nih.gov/pubmed/6247908.
- 77.
胡森,乔西,页J.主成分方法-层次聚类-分区聚类:为什么我们需要选择可视化数据?技术报告- agrocampus,应用数学系。2010http://www.agrocampus-ouest.fr/math/
- 78.
法鲁什D,斯蒂芬斯M,普里查德JK。利用多位点基因型数据推断群体结构:相关位点和相关等位基因频率。遗传学,2003;164:1567-87https://www.ncbi.nlm.nih.gov/pubmed/12930761.
- 79.
来自少数个体的平均杂合度和遗传距离的估计。遗传学,1978;89:583-90https://www.ncbi.nlm.nih.gov/pubmed/17248844.
- 80.
王晓明,杨晓明,Grünwald;Poppr:一个R包,用于克隆、部分克隆和/或有性生殖群体的遗传分析。PeerJ。2014; 2: e281。https://doi.org/10.7717/peerj.281.
- 81.
Excoffier L, Smouse PE, Quattro JM。从DNA单倍型之间的度量距离推断的分子方差分析:应用于人类线粒体DNA限制数据。遗传学。1992;131:479 - 91。
- 82.
de Meeus T, Goudet J.一个循序渐进的教程,使用HierFstat来分析在多个层次上的层次结构的人口。《感染Genet Evol》2007;7:731-5。https://doi.org/10.1016/j.meegid.2007.07.005.
- 83.
苏田培,李培荣,杨俊杰,隋桂兰,余玉军,张丹娟,赵晓阳,王文华,温春林,余顺生,张福龙。用于遗传多样性分析的高性价比单核苷酸多态性标记方法的建立芸苔属植物拉伯.Mol Breed. 2018;38:42。https://doi.org/10.1007/s11032-018-0795-0.
- 84.
李文杰,李文杰,李文杰,李文杰,等。玉米幼苗根系发育的全基因组关联分析(玉米l .)。BMC Genomics. 2015;16:47。https://doi.org/10.1186/s12864-015-1226-9.
- 85.
王海红,杨卫东,王晓明,王晓明。番茄菌斑抗性在定向选择复合育种群体中的关联分析。植物病理学。2015;105:1437-45。https://doi.org/10.3835/plantgenome2017.03.0026.
确认
作者感谢张建南博士在生物信息学分析过程中的协助。
资金
国家重点研究与发展基金(No. 2017YFD0102004 - CW, No. 2017YFD0101901 - SG, No. 2016YFD0101704 - BC),国家自然科学基金(No. 31701913) - HD,北京新星计划(No. 31701913)资助。中国农业农村部农业农村部(批准号11162130109236051),北京市农林科学院(批准号:Z181100006218060),北京市组织部农业农村部(批准号:2016000021223ZK22);KJCX20170402, KJCX20161503, QNJJ201810和KJCX2017102)。资助机构在研究设计、数据收集、分析和解释或撰写手稿方面没有任何作用。
作者信息
从属关系
贡献
CW和SG设计了这项研究。HD和JY进行了生物信息学分析。KY, BC, XZ提供资料,协助数据分析。HD、BC、XZ和JZ进行实验。房屋署起草了手稿。所有作者都阅读并批准了手稿的最终版本。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:图S1。
水果形状分类的例子。果实形态分为四种类型:(A)块状:块状,肩宽5.0 ~ 12.5 cm,长7.0 ~ 18 cm,裂片3 ~ 4瓣,有芳角、长芳角、麻腊角,中国名;(B)长角果:角形长,肩宽3.0-8.0厘米,长10.0-35.0厘米,无瓣,有牛角角、阳角角、洛斯角,中国名;(C)短角果型:圆锥形,中热,基部直径1.0-3.0厘米,长3.5-10.0厘米,果皮极薄,中国名干椒、潮天椒;(D)线果型:辣椒型,宽1.0-3.0 cm,长10.0-35.0 cm,无肩、无瓣,包括鲜椒、跳椒、美仁椒,国名。
附加文件2:图S2。
目标SNP-seq基因分型分析结果。271个辣椒品种的平均reads深度分布(A)、reads对辣椒参考基因组的比对率(B)、靶区比对率(C)和均匀性分布(D)。
附加文件3:图S3。
271个辣椒品种92个完美snp的遗传多样性分析小等位基因频率(MAF;A),观察杂合度(Ho;B),期望杂合度(He;C)、多态性信息含量(PIC;D)。
附加文件4:图S4。
对23 ~ 95个辣椒品种的35个核心SNP标记进行竞争性等位基因特异性PCR (KASPar)分析。
附加文件5:图S5。
Pops和Subpops之间差异的显著性检验。该图显示了在所有水平上显著的种群分化,因为观察到的线(黑色)不属于排列的预期分布。
附加文件6:图S6。
用于与水果形状指数(FSI)关联分析的标记的一个子集的染色体图。每个标记在12条染色体上的物理位置c .建立Zunla-1 [3.]显示在括号之间。显著相关的标记用红色表示。
附加文件7:图S7。
271个辣椒品种果实形状指数(FSI)的曼哈顿图(A)和分位数-分位数图(B)。红色虚线表示高概率与FSI相关位点。
附加文件8:表S1。
本研究所用辣椒品种的分类及资料。
附加文件9:表S2。
用于目标SNP-seq的92个完美snp的多路引物面板。
附加文件10:表S3。
在271个辣椒品种和4个果形群体中检测到92个完美SNPs的特征和多样性。
附加文件11:表S4。
本研究建立了35个核心snp标记的引物序列。
附加文件12:表S5。
收集辣椒品种果实形状指数(FSI)的范围、平均值和标准差(SD)。
附加文件13:表S6。
CM334和Zunla-1参考基因组中与果实形状指数(FSI)相关的区域。
附加文件14:表S7。
利用Target SNP-seq检测辣椒四种病害的功能抗性位点。
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
杜,洪,杨,杰,陈,B。et al。目标测序揭示了辣椒品种的遗传多样性、群体结构、核心snp标记和果实形状相关位点。BMC植物生物学19日,578(2019)。https://doi.org/10.1186/s12870-019-2122-2
收到了:
接受:
发表:
关键字
- 胡椒
- 单核苷酸多态性
- 基因结构
- 目标SNP-seq
- 关联分析