摘要
背景
花药发育在转录水平上已被广泛研究,但在全基因组尺度上对全长转录本的系统分析尚未发表。在这里,太平洋生物科学公司(PacBio)的Sequel平台和下一代测序(NGS)技术相结合,在大白菜花药中生成全长序列和完整的转录本结构。
结果
采用单分子实时测序(SMRT)技术,共生成1098,119条循环一致性序列,平均长度为2664 bp。超过75%的ccs被认为是全长非嵌合(FLNC)读取。经过误差校正后,725731个高质量FLNC reads估计携带19503个位点的51501个异构体,其中38992个来自已知基因的新异构体和3691个来自新基因的新异构体。在这些新型异构体中,我们鉴定出407个长非编码rna (lncRNAs)和37549个开放阅读框(orf)。此外,共鉴定出453,270个替代剪接(AS)事件,且花药中的大多数AS模型被确定为近似外显子跳过(XSKIP)事件。在花药发育过程中调控的关键基因中,AS事件主要发生在基因中SERK1,CALS5,NEF1,CESA1/3.此外,我们鉴定了104个融合转录本和5806个具有选择性多聚腺苷酸化(APA)的基因。
结论
我们的工作证明了大白菜花药发育的转录组多样性和复杂性。这一发现为进一步研究大白菜的基因组注释和转录组提供了基础。
背景
基因测序是生物学研究领域的一项革命性技术。这些技术中的第一项是桑格测序;但由于通量低、自动化程度低,Sanger测序在基因组和转录组分析中的应用受到严重限制[1].NGS技术的出现,如ABI SOLiD、Illumina Solexa和Roche 454系统,促进了不同植物物种的结构和功能基因组学研究。在这些技术中,Illumina测序具有高精度、高通量、高灵敏度、低成本等优点,是目前应用最广泛的基因组测序平台[2].c .巨大成功是第一种用NGS完成全基因组从头测序的蔬菜作物。随后,主要的作物基因组美国tuberosum,t . aestivum,显著,g . raimondii就,并对其他作物进行了测序。NGS的短读RNA-Seq经常用于转录组分析。利用短读RNA-Seq,研究人员可以获得全基因组表达基因的谱,包括低丰度基因,以及新基因和snp [3.].该属花粉及花药发育的基因表达谱研究芸苔属植物已累积于近年[4,5,6,7,8,9,10,11].然而,尽管NGS技术是有效的,但它们仍然存在一些缺点,包括产生相对较短的读取,这可能导致误组装和间隙[12].此外,短读不太适合准确检测AS事件产生的结构变异(SVs)和转录本异构体[13,14].受限于NGS方法,短RNA-Seq reads必须组装成较长的DNA contigs [15],这一过程容易使从高重复区域转录的短序列读文或多个基因家族的相似成员误组装[16].对于多倍体植物来说,这个问题可能会变得更加严重,因为它们在共存的亚基因组之间往往具有更高的序列相似性,这经常间接导致注释错误。此外,短读RNA-Seq无法区分单个转录本的交替拼接形式,而这些转录本可能构成很大一部分转录本。例如,大约83.4%的多外显子基因易患AS答:芥,这有助于生物蛋白质的多样性,而不会大量增加基因的数量[17].
第三代测序(Third generation sequencing, TGS)技术近年来发展起来,以单分子测序(SGS)和实时测序著称[18].第一个TGS技术平台是由Helicos Biosciences提供的,但它在市场上被证明是不可用的,因为它相对较慢,昂贵,并且产生的读取值较短(~ 32 bp) [19].不久之后,PacBio公司的单分子实时测序(SMRT)成为构建全长转录本的独特机会[20.].SMRT技术的显著特点是产生长读取。最初,SMRT技术产生的读取的平均长度只有~ 1.5 kb,但现在是10-15 kb [21].因此,SMRT可以提高基因模型的准确性,因为它可以生成覆盖全长转录本的reads [14].但SMRT测序仍存在较大的技术缺陷和局限性,即成本较高、吞吐量较低、错误率较高。因此,目前NGS技术和SMRT测序的结合是更可取的:共识序列读取从原始PacBio子读取构建,并与从适当的NGS平台生成的读取对齐。使用这种方法,多个复杂的基因组已经成功地从头组装或改进[22,23,24,25,26,27,28,29,30.].
SMRT测序此前已有效应用于转录组分析。特征良好的全长转录本不仅有利于基因结构分析和选择性剪接,而且极大地提高了重要位点的功能研究[15].早期SMRT测序在转录组上的应用相对狭窄,大多数集中在模式生物,如人类[13]和酵母[31].自2015年以来,SMRT技术已被广泛应用于不同物种基因组和转录本的全长序列表征。SMRT促进了普通六倍体小麦的结构基因组学和籽粒转录组研究[15].在丹参中,将SMRT测序应用于不同的根组织,发现约40%的检测到的基因位点发生了选择性剪接(alternative splicing, AS)事件[32].在玉米B73中,从六个组织中鉴定出超过111000个转录本,通过SMRT测序揭示了转录组的复杂性[14].PacBio SMRT用于高粱转录组,超过11000个新的剪接异构体,约11000个表达基因的选择性多聚腺苷酸化(APA), 2100多个新基因以前所未有的规模被发现[33].的答:芥通过SMRT分析转录组,增强了对正常条件下差异表达AS异构体和ABA处理应答的理解[17].在毛竹中,发现了超过42,280种不同的剪接异构体和25,069个聚腺苷酸位点[34].在石斛兰officinale,茎叶全长cDNA转录本揭示了多个参与多糖合成的基因[35].通过SMRT测序分析了红三叶草的转录组,结果发现了大约29,730个来自已知基因的新亚型和2194个来自新基因的新亚型,此外还有超过5000个AS事件,超过4300个长非编码rna (lncRNAs)和3700个融合转录本[36].利用SMRT技术,从苜蓿叶片三个不同发育阶段共获得113321个转录本;测序数据揭示了约7568个AS事件和17740个lncRNAs [37].以上工作对于深入了解其基因组和转录本具有重要意义。
属芸苔属植物包括多种重要的经济蔬菜和油料作物.“U三角形”已经确定,指的是三个二倍体物种,b·拉伯(基因组,2n = 20),b .黑质(B基因组,2n = 16),和b . oleracea(C基因组,2n = 18),以及三个双二倍体物种,显著(AC基因组,2n = 38),b . juncea(AB基因组,2n = 36),和b . carinata(BC基因组,2n = 34)。2011年,第一个属芸苔属植物基因组草案,b·拉伯Genome v1.5出版。利用新一代测序(NGS)技术获得了283.8 Mb的基因组,contig N50大小为46 kb,极大地促进了基因组学和分子生物学研究,以及大豆的通用育种b·拉伯和其他芸苔属植物物种(38].第二版(v2.0)于2017年组装。进一步完善了脚手架的秩序,进行了升级b·拉伯基因组v2.5为389.2 Mb, contig N50大小为53 kb [39].但受NGS技术读取长度的限制,上述基因组版本存在连续性差、装配误差大、重复序列装配率低等缺点。最近发布的一个版本,b·拉伯基因组v3.0采用单分子测序(PacBio)、光学定位(BioNano)和染色体构象捕获(Hi-C)技术重新组装和注释。的总长度b·拉伯基因组v3.0为353.14 Mb, contig N50大小为1.45 Mb,支架N50大小为4.45 Mb,其中支架1301个,间隙389个[40].高质量的参考基因组信息为遗传和功能基因组学的发展奠定了坚实的基础b·拉伯重点研究了重要农艺性状调控基因的克隆及遗传背景分析。只有在遗传水平上分析性状形成的分子机制,才能进行定向遗传育种、分子标记辅助育种乃至分子设计育种,大大提高育种效率,加快培育优良新品种。
由于适应性广,用途多,大白菜是中国种植和消费最广泛的蔬菜b·拉伯.虽然使用PacBio Sequel平台已经改进了参考基因组,但在大白菜中组织特异性mRNA的序列和结构数据仍然很少。本研究的主要目的是利用新兴的SMRT测序技术描述大白菜花药的全长转录本,以揭示花药发育的转录组复杂性。利用SMRT测序数据,经短读NGS技术校正,分析花药全长转录本,进一步揭示大白菜AS事件、lncRNAs和融合异构体。这项研究为进一步的基因组重新注释提供了宝贵的资源,并增加了我们对花药转录组的理解。
结果
转录组测序和纠错
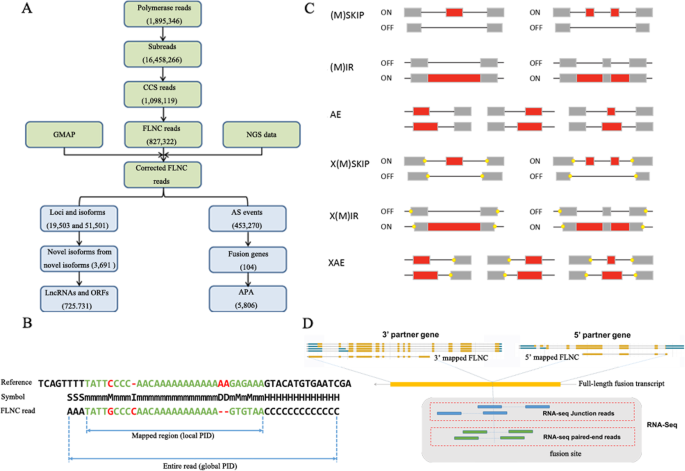
受限于Illumina平台上短读RNA-Seq的容量,大白菜双单倍体(DH)系' FT '的花药特异性转录组分析(图2)。1a-c)使用PacBio Sequel平台进行。为了尽可能完整地识别转录本,从花药发育过程中获得的每个池样本中提取和混合高质量的总mrna,以获得全长序列和剪接变体。整个流程如图所示。2.
生物信息学分析。一个PacBio Sequel平台流程图。b说明FLNC映射和PID计算。m:匹配。M:不匹配;soft-clipping。H: hard-clipping。D:删除;我:插入。PID的计算公式是:当地PID = m / (m + m + D + I), gobal PID = m / (m + m + S + H + D + I)。cAS事件分类图。(M)SKIP:(卡带外显子)外显子跳过;(M)IR:保留(多个)单内含子;AE:替代外显子末端(5 ',3 '或两者都有);X(M)SKIP:近似(盒式外显子)外显子跳过;X(M)IR:(多个)单内含子的近似保留;XAE:近似替代外显子末端;d融合转录本检测示意图
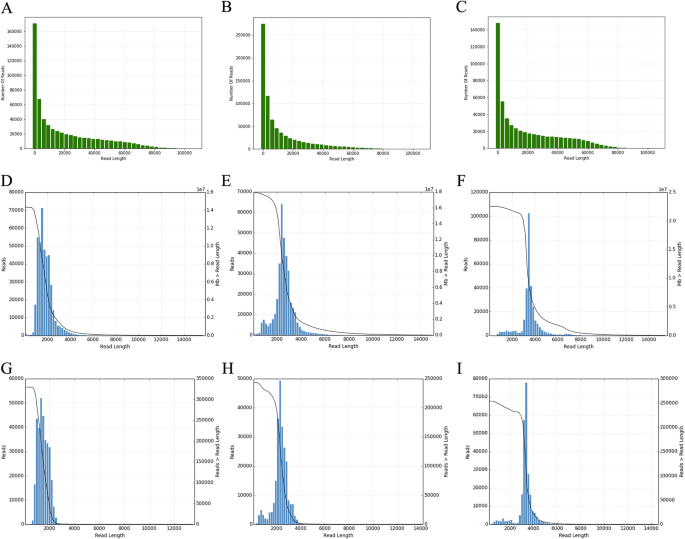
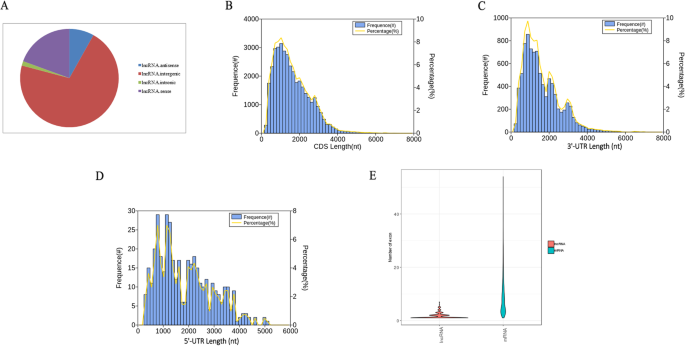
使用PacBio Sequel平台构建了三个不同的SMRT钟形文库,cDNA插入大小分别为1-2 kb、2-3 kb和> 3 kb。经过过滤,共捕获到1,895,346个聚合酶reads,代表超过33.14个G碱基,平均长度为17,965 bp, N50为39,750 bp(附加文件2:表S1;无花果。3.a - c)。从聚合酶reads中去除适配器后,大约获得16,458,266个过滤子reads,平均长度为2121 bp(附加文件2:表S2)。通过多次测序合并和纠错后的子读,在三个库中共生成1098119个循环共识序列(circular consensus sequences, ccp),平均深度为11.33次(Additional file .)2:表S3)。ccs的长度分布与三个库的预期大小一致(图2)。3.d-f)。CCSs分别计数如下:5 '引物,3 '引物,poly-A尾,全长,全长非嵌合(FLNC)。我们总共检测到863,281个全长reads,包含5 '引物,3 '引物和poly-A尾。然后,827,322个reads被认为是低人工连接体的FLNC,占ccs的75.34%1).在1-2 K、2-3 K和> 3 K库中FLNC reads的平均长度分别为1499 bp、2324 bp和3288 bp。3.胃肠道;表格1).各库FLNC读取量分布基本一致,但1 ~ 2 kb库略大于其他两个库。总的来说,我们已经全面获得了全长转录本,使得准确构建剪接变体成为可能。
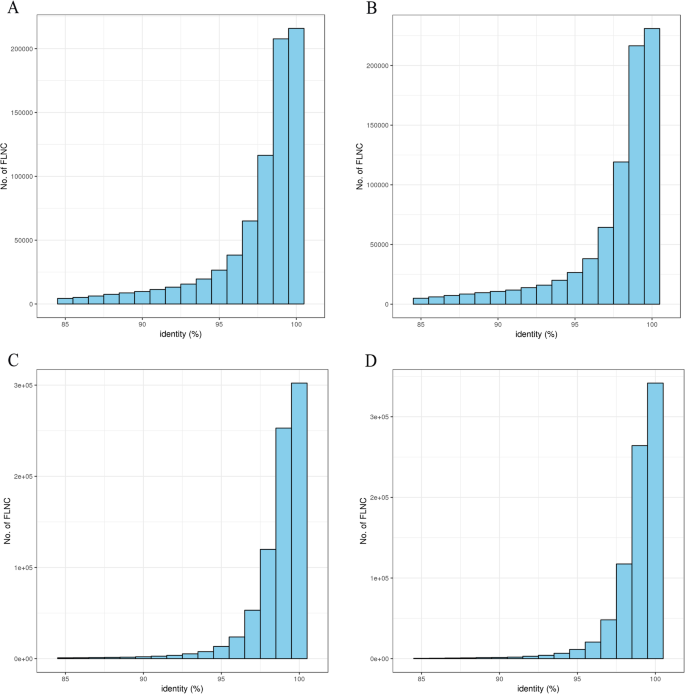
SMRT序列具有很高的碱基错误率(高达12 - 15%),主要是由于额外的碱基插入。为了进一步校正PacBio Sequel平台测序的FLNC reads,验证软件使用了Illumina HiSeq 2000花药转录本。使用GMAP2在软件中,将纠错前后的FLNC reads与参考基因组进行比较,计算全局和局部的身份百分比(PID)(图2)。4).纠错前,全局PID均值为94.97%。经过错误修正后,该值高达97.04%(附加文件2:表S4)。更新后,我们获得了725,731个高质量FLNC reads供后续研究使用(表2).
基因座和异构体检测和表征
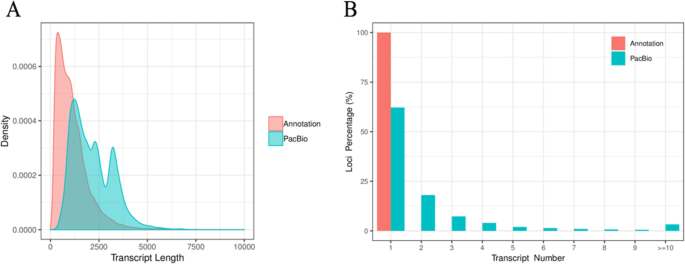
误差校正分析允许FLNC读数精确映射到参考基因组,包括起始位点、终止位点和剪接位点。基于这些信息,可以识别基因位点和亚型。为了评估异构体长度密度,我们将PacBio数据集的基因座覆盖率与b·拉伯v3.0注释。在我们的数据中,共有725,731个错误校正的FLNC reads覆盖了51,501个异构体,并分配到19,503个位点。长度为1 - 2 kb的位点约9102个,其次为2-3 kb(3867个),> 3 kb(3355个)和< 1 kb(3179个)。在参考基因组中,约46250个异构体覆盖了46250个位点,其中主要分布在< 1 kb(24937个),其次是1 - 2 kb(15700个),2-3 kb(3959个),> 3 kb(1654个)(表3.;无花果。5).同样地,我们对基因座密度的亚型数进行了评估,表明每个基因座在参考基因组中可以产生一个独特的亚型。然而,在我们的数据中,大约有12124个(62.16%)位点可以产生一种独特的异构体,5种以上的异构体覆盖了大约6.83%的PacBio注释位点(图2)。5 b).A06.1469基因的亚型数最多,约为524个。因此,与参考基因组相比,PacBio数据集可以提供更丰富的异构体长度多样性和位点数密度,有助于更全面地揭示花药转录组的复杂性。此外,我们还评估了通过PacBio Sequel平台获得的每个基因座和亚型的外显子-内含子结构。19503个位点中,单外显子位点2911个(14.93%),多外显子位点16592个(85.07%)。在51501种异构体中,4.188种(8.13%)为单外显子,47,313种(91.87%)为多外显子3.).
基于图书馆建设的特点,我们无法保证抄本5′端结构的完整性。因此,PacBio Sequel平台生产的FLNC和异构体的全长评价仅在5 '端进行估计。以基因组注释的多外显子转录本为参考,从PacBio数据集中筛选出方向相同且重叠大于20%的异构体。如果从PacBio数据集中获得的异构体的第一个剪接供体位点确实包含基因组注释5 '端的第一个剪接供体位点,则认为该异构体为全长异构体,相应的FLNC也被认为是全长FLNC。我们的数据表明,约76.66%的多外显子isoforms和88.22%的多外显子FLNC在5’端包含与参考注释相同的剪接供体位点,被认为是全长的,这意味着结构的完整性相对较高(附加文件2:表S5)。
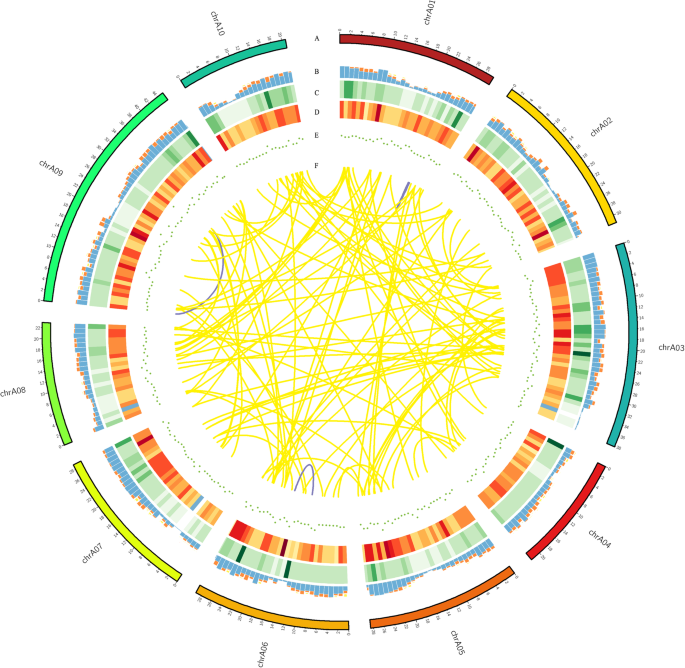
接下来,将测序的基因位点和异构体与参考注释进行比较,确定新的位点或新的异构体。已发布的b·拉伯基因组注释包含46,250个位点和46250个异构体。在我们的PacBio数据集中,从19503个基因的51501个异构体中,我们从已知基因中鉴定出16821个已知异构体。此外,有2682个转录本与无注释基因重叠被认为可能是新基因(附加文件2:表S6;无花果。6d).这些新基因被发现产生3691种新异构体(图。6c).我们还从11398个已知基因中发现了38992个新的异构体。3691种新型异构体中,1455种(39.42%)为单外显子异构体,2236种(60.58%)为多外显子异构体。上述新基因和异构体的存在有利于提高植物的完整性b·拉伯基因组注释。
新型异构体的功能注释
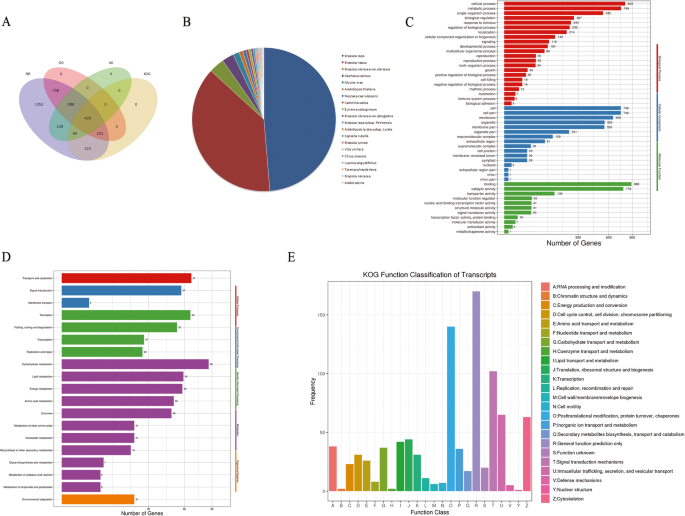
在本研究中,通过NCBI非冗余蛋白序列(NR)(89.76%)、基因本体(GO)(44.57%)、真核同源群(KO)(24.36%)、真核同源群(KOG)(22.70%)和Swiss-Prot蛋白序列(Swiss-Prot)数据库对3691个新异构体进行了功能注释,共有377个(10.21%)未注释(附加文件)2:表S7)。总共有420种新异构体在所有五个数据库中都有显著的命中(图2)。7一个).在NR数据库中,新异构体最多的3组分布在b·拉伯(1578),显著(1236)和b . oleracea(112)(图。7 b).GO分析将1645种异构体的富集划分为三个本体,即生物过程、细胞成分和分子功能。我们在“生物过程”中发现了1602个GO术语,其中“细胞过程”(48.75%)、“代谢过程”(45.53%)和“单生物过程”(32.52%)占较大比例。“生物过程”中的许多术语都与花药发育有关,如花粉萌发、脂肪酸代谢过程、花粉外壁形成、花粉管发育、花药开裂、授粉、花粉-花柱相互作用等。在“细胞成分”中共检测到358个GO词,其中“细胞”(45.40%)、“细胞部分”(45.40%)和“膜”(39.51%)是最多的三个富集词。在“细胞成分”中,有11种和2种新的异构体分别归属于GO术语“花粉管尖端”和“花粉管”。我们的数据显示,769个GO术语被分配到“分子功能”,其中“结合”(53.98%)和“催化活性”(47.29%)是最丰富的术语(图2)。7 c).为了鉴定富集途径,共有899种新亚型被用于101种KEGG途径。KEGG通路中的新型异构体包括“细胞过程”、“环境信息处理”、“遗传信息处理”、“代谢”和“有机体系统”五个层次。在这些术语中,层次最多的是“新陈代谢”(71.29%),其次是“遗传信息处理”(11.88%)(图1)。7 d).此外,我们还发现了与“脂肪酸”相关的三种代谢途径,这对花药发育至关重要[41].KOG分析显示,838种新异构体被划分为24类,其中数量最多的3类分别是“一般功能预测”(20.29%)、“翻译后修饰、蛋白质转换、伴侣”(16.71%)和“信号转导机制”(12.18%)(图2)。7 e).
新型异构体的LncRNA和ORF预测
lncrna具有调节功能,对转录后、转录和表观遗传学至关重要[42].通过CPAT软件预测来自新基因的新异构体和来自上述功能注释数据库中未命中的已知基因的新异构体,以识别PacBio数据集中的lncrna。为了获得高置信度的lncrna,我们保留了长度超过200 bp的具有最佳截断值的亚型。共预测407种新型异构体为lncrna,占所有新型异构体的1%,平均长度为1127 bp(附加文件2:表S8)。长度超过1000 bp的lncrna约有168个(41.28%),长度超过4000 bp的lncrna有4个。预测的lncrna分为四种类型,其中反义lncrna 33个(8.11%),基因间型lncrna 289个(71.01%),内含子型lncrna 6个(1.47%),有意义lncrna 79个(19.41%)(图。8).将预测的lncrna映射到b·拉伯的使用Circos可视化软件显示10条染色体,显示407个lncrna随机分布,其中3个lncrna没有锚定在染色体上(图2)。6 e).通过transDecoder软件预测开放阅读框架(ORF),得到37549种具有预测ORF的新异构体。接下来,研究编码序列(CDS)的密度和长度分布,平均长度为915 bp(图15)。8 b).编码的肽序列在附加文件中列出2:表S9。对5’和3’边界未翻译区(UTRs)分布的密度和长度进行了识别,结果显示3’UTRs 415个,平均长度为641 bp, 5’UTRs 8791个,平均长度为788 bp(图2)。8 c, d).进一步分析具有预测ORF和lncRNA的新型异构体的外显子结构,发现每个mRNA和每个lncRNA的平均外显子数分别为8.78和1.65。8 e).
AS的各种模型
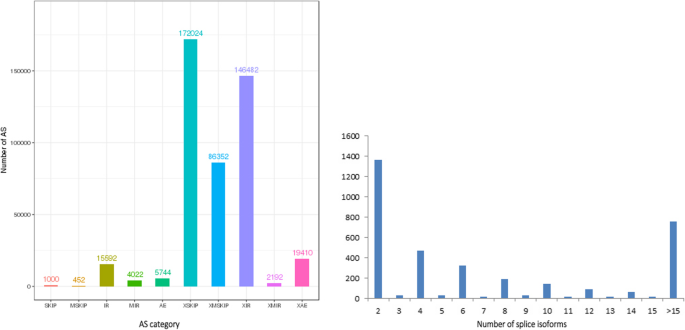
AS根据不同的剪接模式增加转录组和蛋白质组的多样性,而不是通过大量放大细胞或组织中的基因数量[43,44].传统上,AS事件包括几种不同类型:外显子跳过(SKIP)和盒式外显子(MSIP),单个(IR)和多个(MIR)内含子的保留,替代外显子末端(5 ',3 ',或两者都有)(AE),近似外显子跳过(XSKIP)和盒式外显子(AMSKIP),单个(XIR)和多个(XMIR)内含子的近似保留,以及近似替代外显子末端(XAE)(图。2摄氏度).最新的b·拉伯v3.0参考基因组未纳入AS模型和剪接异构体。然而,早期共检测到156,516个独特的剪接连接b·拉伯基因组v1.5, IR事件在参考基因组中占主导地位,类似于物种,如m . truncatula,p . trichocarpa,答:芥,o .漂白亚麻纤维卷,c . reinhardtii,b . distachyon[45].在我们的研究中,我们将pacbio测序的亚型与b·拉伯基因组v3.0,并发现共19503个位点对应51501个异构体发生了453270次AS事件,表明AS事件在花药中的分布非常高(附加文件)2:表S10)。在我们的研究中产生的AS事件总数为:1000 SKIP、452 MSKIP、15,592 IR、4022 MIR、5744 AE、172,024 XKIP、86,352 XMSKIP、146,482 XIR、2192 XMIR和19,410 XAE(图。9).此外,我们观察到XSKIP(37.95%)是主要的,而MSKIP(0.1%)是最不常见的事件。这一发现极大地丰富了花药转录信息。在我们的PacBio Sequel平台分析中,在3576个基因中发现了两种或更多的亚型。在1115个基因中检测到10种或更多的剪接异构体(图。9 b).剪接异构体最多,为64266个BraA06g022340.3C;这个基因是同源的拟南芥H(+)- atp酶8 (AHA8)。为了验证SMRT检测AS事件的准确性,随机选择3个基因,设计横跨预测剪接事件的基因特异性引物用于RT-PCR。RT-PCR和Sanger测序在花药中的表达结果与PacBio数据集中检测到的剪接异构体完全一致,证明这些数据是可靠的(附加文件1:图S1)。
融合转录本和APA鉴定
融合转录本是指将两个或多个独立基因拼接在一起形成的新基因,称为嵌合转录本。导致融合转录本产生的机制包括基因组结构变异、转位或转录后的反式剪接。在这项研究中,我们鉴定了104个融合转录本,涉及187个注释基因(附加文件)2:表S11)。融合转录本在A03染色体上分布最多,其次为A09和A01染色体。根据染色体分布,我们检测到101个染色体间融合转录物和3个染色体内融合转录物(图;6 f).这一结果与玉米等其他物种的结果一致[14]和红三叶草[39].先前的研究表明,大多数融合转录本由两个基因组成[46].与这些研究一致的是,我们数据中的104个融合转录本全部由两个基因组成。此外,随机选择了3个SMRT检测到的融合转录本,并在花药和其他四个花器官中进行了实验验证。实验结果证实了这些嵌合rna的真实性(附加文件1:图S1)。
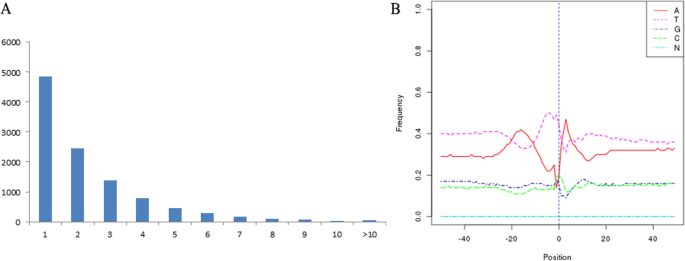
pre-mRNA到成熟mRNA的转录后修饰过程主要包括在5 '端添加7-甲基鸟苷帽,内含子剪接,3 '端通过裂解和多聚腺苷酸化形成[47].poly-A尾巴在3 '端的具体位置是可变的,这种变化可能会影响microRNA或RNA结合蛋白与mRNA的结合,以及RNA剪接和翻译的过程。采用Tapis软件准确识别花药中多聚腺苷酸位点。通过研究我们的PacBio数据集中的3 '端转录本,从10,661个基因中检测到24,816个poly-A位点,其中5806个基因具有选择性多聚腺苷酸化(APA)(图。6b、附加文件2:表S12)。共有4855个基因具有至少一个poly-A位点,而733个基因具有5个以上poly-A位点(图2)。10).平均每个基因的poly-A位点数为2.33个。聚a位点最多的有19个,在BraA02g029650.3C而且BraA04g001890.3C.接下来,我们分析了所有poly-A位点的上游和下游50个nts的核苷酸分布。与其他物种的结果一致,我们的PacBio数据集中的poly-A位点显示出核苷酸偏倚,上游的尿嘧啶(U)和下游的腺嘌呤(a)富集(图2)。10 b).
讨论
目前,大白菜的参考基因组已通过单分子测序更新到3.0版本。然而,大白菜的全长转录本、替代剪接转录本、融合基因和APA位点在转录水平上尚未得到很好的探索。花药是植物的雄性生殖器官,可以产生花粉粒。花药发育的调控网络是一个涉及一系列生物学事件的极其复杂的过程[48].在拟南芥,花药发育分为14个阶段,其中包括两个阶段:小孢子发生和小配子体发生[49].简而言之,花药的发育源于雄蕊原基的形成,小孢子母细胞经过减数分裂形成单倍体小孢子四分体。小孢子被胼胝质包裹,从四分体中释放单个小孢子需要绒毡层分泌的胼胝质酶的作用。然后合成小孢子壁,绒毡层降解,花粉有丝分裂分裂,间隔细胞变性,气孔分化,最后花药开裂,释放成熟花粉粒。这些事件是相对独立的,在时间和空间上具有协调性。其中一个事件的基因结构或表达异常可能导致花粉功能丧失,从而产生雄性不育系。植物雄性不育的一个重要应用是杂交制种,杂交制种的优势在于可以提高种子产量和抗逆性[50,51].因此,有必要研究全长mRNA信息,以提供花药发育中剪接异构体的全面视图。
PacBio测序是一种有效的全长转录本测序平台,因为它可以生成平均长度为12 kb的长reads [52].PacBio测序平台可以全面分析每个基因的剪接异构体,而无需组装。在我们的工作中,我们使用PacBio Sequel平台分析了大白菜花药的全长转录组,共获得了1,098,119个ccp。其中827322个转录本被鉴定为FLNCs,每个测序文库的长度均符合文库标准(图2)。3.;表格1).单分子测序的碱基错误率较高,约13%,主要是由于额外碱基的加入,特别是在均聚物中[53].然而,由于这种误差是随机发生的,因此不存在误差偏差,这与用NGS技术观察到的误差不同。目前,进一步校正PacBio测序最常见和有效的方法是使用来自Illumina平台的高精度数据。使用短读RNA-Seq进行纠错后,鉴定出725731个高质量flnc,获得51501个异构体,其中38992个新异构体来自11398个已知基因,3691个新异构体来自2682个新基因(附加文件)2:表S6)。这些结果表明,PacBio转录组测序可以提高获得全长转录本的能力,并丰富新的或未特征的异构体或基因。在获得的新异构体中,鉴定出407个高置信lncrna和37549个具有预测orf的新异构体(附加文件)2:表S8和表S9)。在花粉发育和受精过程中b·拉伯,共检测到12501个假定的lncrna,平均长度为373 bp [42].在我们的数据中,来自新异构体的预测lncrna的平均长度为1127 bp(附加文件)2:表S8)。在此之前,b·拉伯基因组仅使用orf进行注释,因此没有定义5 '和3 ' utr。2013年,Tong等人提供了全球转录景观b·拉伯加入Chiifu-401-42并定义了5 '和3 ' utr。5 '和3 ' UTRs的平均长度分别为139 bp和184 bp [45].在拟南芥, 5 '和3 ' UTRs的平均长度分别为88 bp和184 bp [54].在我们的PacBio测序数据中,来自具有预测ORF的新型异构体的5 '和3 ' utr的平均长度分别为788 bp和641 bp。8c, d)。
除了捕获全长转录本外,PacBio测序的另一个优势是能够检测AS事件,这些事件在调节细胞分子、细胞生理学和发育途径中起着至关重要的作用[45,55,56].AS基因在水稻、玉米、b·拉伯,答:芥分别为33、37、42和61% [57,58,59].受限于短读,以前使用NGS技术对转录组的研究只能提供单个剪接连接,而PacBio测序技术可以应用于每个mRNA的交替剪接形式[39].IR是各种基因组中最常见的事件,它支持前mrna剪接的内含子定义机制[60].在我们的研究中,我们收集了所有发育阶段的花药,以获得相对全面的拼接异构体。然而,我们总共检测到453,270个AS事件,其中大多数AS事件是XSKIP(图3)。9).以往研究表明,选择性剪接转录本在不同植物中具有组织特异性表达[61,62,63,64].对于新颖的剪接连接b·拉伯,仅在一个组织中检测到34.4%的替代性香料转录本[45].因此,AS事件患病率的差异可能与组织特异性有关。这些发现说明了花药特异性转录组的复杂性。遗憾的是,PacBio测序检测到的转录本的表达水平还没有分析,也没有办法分析AS事件引起的同一基因不同亚型的表达模式。
以模型植物为例拟南芥例如,花药发育过程中的关键调控基因已被广泛报道,主要涉及小孢子发生、绒毡层形成、胼胝质层形成、花粉壁形成和花药开裂[65].大白菜和拟南芥均属芸苔科,亲缘关系密切,序列相似性高。因此,我们收集了34个已被证实参与花药发育的基因拟南芥(附加文件2:表S13)。除了在芸苔科中出现的三种全基因组复制(WGDs)外,芸苔属基因组还经历了另外一种古老的三倍复制,并伴有基因分馏[38].因此,基于Brassica数据库中最好的BLASTX搜索,我们从PacBio注释数据中获得了53个注释基因(附加文件2:表S13)。在这些基因中,无性生殖的(AG)),SPOROCYTELESS /喷嘴(SPL/出处),几乎没有分生组织(BAM1/2),额外的孢子细胞/1 .小孢子细胞过多(EMS1 /练习),体细胞胚胎发生受体样激酶1(SERK1),绒毡行列式1(TPD1)对花药发育早期阶段的小孢子发生进行了注释。对于绒毡层发育和程序性细胞死亡(PCD),检测到的关键基因是拟南芥MYB DOMAIN PROTEIN 80/103(AtMYB80/AtMYB103),功能失调的绒毡层1(DYT1),绒毡层发育与功能(TDF1),夭折的小孢子(自动对盘及成交系统),男性不育(MS1).对于花粉外壁形成,胼胝合酶5(CALS5),周期蛋白依赖性激酶G1(CDKG1),生长素反应因子17(ARF17),无外膜形成1(NEF1),花粉粒破裂(RPG1),外缘形成有缺陷的(DEX1),无原墨辛和质膜波动(转专业),CYP703A2,乙酰辅酶A合成酶(ACOS5),男性不育2(一份),粘附花粉少(LAP5),atp结合盒式G26(ABCG26 / WBC27)。对于花粉梗的形成,纤维素合成酶1/3(CESA1/3.),阿拉伯半乳聚糖蛋白6/11(APG6/11),束素样阿拉伯半乳聚糖蛋白3(FLA3)。对于另一个裂口,Myb结构域蛋白26(MYB26),Nac次生壁增厚促进因子(NST1)。此外,在我们的PacBio数据集中,一些基因座被发现含有不同的选择性剪接异构体。例如,两个位点(BraA07g036270.3C而且BraA07g029410.3C)注释为SERK1,这是重要的花药细胞规格,但只是BraA07g036270.3C表达了两种交替拼接的异构体早在减数分裂阶段,小孢子母细胞质膜外就开始沉积胼胝质层,这是花粉壁发育的开始。在拟南芥, 12CALS基因被鉴定CALS5在四分体时期胼胝质的合成中起重要作用。在突变体cals5时,小孢子周围胼胝质产生不足,导致原乳素形成缺陷,进而影响孢粉在花粉外壁的沉积[66].在我们的数据中,CALS5(BraA09g010050.3C)有大约1065个拼接变体,并且XMSKIP在AS模型中占主导地位。对于primexine的形成,有两个基因座(BraA10g025410.3C而且BraA02g004840.3C)注释为NEF1.检测到2个AS事件,IR和XAEBraA10g025410.3C中找到XSKIPBraA02g004840.3C.在拟南芥、多中国极限运动协会克隆了与花粉肠内壁形成相关的纤维素合成酶基因;敲除突变体cesa1而且cesa3表现为配子体不育表型,花粉壁异常[67].两者都有注解CESA1而且CESA3在大白菜中各含有两个基因座。BraA01g005650.3C是CESA1其中检测到40个交替剪接的异构体,包括12个IR, 6个XIR, 12个XMSKIP和10个XAE。CESA1(BraA03g057280.3C)有12个交替拼接的异构体,包括8个IR, 2个XIR和2个XAE。类似地,两个位点被注释为CESA3(BraA03g002020.3C而且BraA02g001600.3C).8例AS事件:检测到IR 6例,XAE 2例BraA03g002020.3C中检测到2个IRBraA02g001600.3C.我们的研究确定了在大白菜花药发育过程中活跃的关键基因中的AS事件。
结论
利用全长转录组技术研究了大白菜花药发育过程中存在的转录本和剪接异构体。使用PacBio Sequel平台共鉴定出51501种异构体。同时,检测到453,270个AS事件,XSKIP事件在另一个中广泛发生。在我们的PacBio测序中,共检测到53个在花药发育过程中活跃的关键基因,其中8个注释位点具有交替剪接的异构体。此外,本研究还预测了104个融合转录本和24816个poly-A位点。这些新发现为完整的花药特异性转录组数据表征和改进大白菜基因组注释提供了有价值的资源。
方法
植物材料
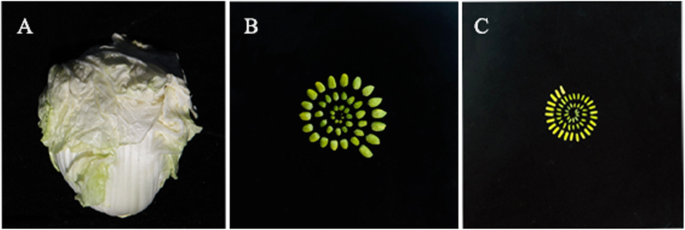
优质大白菜DH系‘FT’是我实验室(辽宁省十字花科蔬菜遗传育种重点实验室)采用分离小孢子培养技术自主选育的。DH系FT的特点是早熟、耐热、卵形叶头和白色花瓣(图2)。1).2018年8月,DH系“FT”种子在4℃冰箱中春化,然后在沈阳农业大学温室中播种。在盛花期,随机选取生长一致的3株植株,将每株植株一个完整花序的整个芽分别收集在铝箔片中(图2)。1 b).然后,从每个芽中分离花药,在液氮中冷冻,并在SMRT测序之前在−80°C保存(图。1 c).
PacBio文库构建和测序
使用Trizol试剂(Invitrogen, CA, USA)从三个样本中提取总RNA。RNA纯度和完整性由nanoophotometer®分光光度计(IMPLEN, CA, USA)和Bioanalyzer 2100系统(Agilent Technologies, CA, USA)监测。用1%琼脂糖凝胶检测RNA污染。RNA浓度检测使用Qubit®2.0荧光仪(Life Technologies, CA, USA)。将每个样品中总RNA的等量混合在一起。使用SMARTer™PCR cDNA合成试剂盒(Takara Biotechnology, Dalian, China)制备全长cDNA。使用BluePippin™尺寸选择系统(Sage Science, Beverly, MA)实现全长cDNA的大小分割(1 - 2,2 - 3和> 3)。将过滤后的全长cdna进行再扩增、末端修复、SMRT适配器连接和外切酶消化。经BluePippin™二次筛选后,使用Pacific biosciences DNA Template Prep Kit 2.0构建三个SMRTbell文库。使用Qubit®2.0荧光计(Life Technologies, CA, USA)和Bioanalyzer 2100系统(Agilent Technologies, CA, USA)测量文库的定量和大小。 Subsequently, SMRT sequencing was performed on a PacBio Sequel platform by Frasergen Bioinformatics Co., Ltd. (Wuhan, China).
Illumina RNA-Seq文库构建与测序
同时,使用Bioanalyzer 2100和RNA 6000 Nano LabChio Kit (Agilent, CA, USA)分析同等混合RNA的数量和纯度。聚t寡聚磁珠(Invitrogen)分离Poly (A) mRNA。裂解后,用mRNASeqample Preparation Kit (Illumina, San Diego, USA)处理后,将裂解的RNA片段反转录到cDNA文库中。在评估文库质量后,我们在LC科学(中国杭州)按照供应商推荐的方案对Illumina Hiseq 2500进行了PE300测序。
质量过滤和纠错
使用SMRT Link v5.0对PacBio原始读取进行预处理和过滤。简单地说,使用以下参数从subread SAM文件生成ccs:最小subread长度= 50;最小通过次数= 1,最小预测准确率= 0.8,最小阅读分数= 0.65。然后,通过评估5 '和3 '适配器和聚(A)尾的存在,将ccs分为全长或非全长读取。FLNC reads是包含所有三种元素的完整的ccs,在DNA片段中没有额外的适配器序列副本。
使用高质量的Illumina短读码对FLNC读码进行纠错。proovread软件v2.12广泛有效地应用于迭代短读共识校正FLNC序列[68].使用GMAP2软件中,误差校正前后的FLNC序列进行了比较b·拉伯V3.0参考基因组[69,70],使用“-no-chimeras and -n 100”来计算PID值,包括全局PID和局部PID(图1)。2 b).PID值越高,表明测序数据与参考基因组的一致性越高。分别统计纠错前后基因组比较的PID值,并更新PID值较高的基因组比较。然后,利用唯一映射的高PID(全局PID > 95%,局部PID > 97%)的FLNC序列标记位点和异构体。
基因位点和亚型的发现
根据校正后FLNC reads的比对位置鉴定基因位点和异构体。对于基因座,两个转录本在同一链上至少有20%的起始位点重叠,并且至少有一个外显子重叠超过20%,被认为是相同的基因座转录本。这些相同的基因座转录本被进一步分析以进行亚型鉴定。该过程主要包括去除冗余转录本和过滤低可靠性转录本。去除冗余转录本的方法如下:首先,如果相同基因座转录本的所有剪接位点都相同,则可以认为是一个亚型;其次,如果一种亚型在5 '末端区域降解,但其余区域与其他亚型一致,则应将其过滤掉。对于假阳性,当全局PID < 99%时,每个异构体结构模型必须至少支持两个FLNC读取;否则,如果只有一个序列,则该序列的所有连接位点都被基因组注释或Illumina RNA-Seq数据完全支持。
新基因及异构体鉴定
将上述基因位点和异构体与参考注释进行比较,鉴定已知基因和异构体,以及新基因和异构体。符合下列条件之一的基因被判定为新基因:(1)注释基因无重叠或重叠率小于20%;或(ii)与标注的基因重叠大于20%,但基因方向不一致。此外,如果测序的异构体包含一个或多个新的剪接位点,或者如果测序的异构体和注释的异构体都不是单外显子,则认为它是一种新的异构体。
功能注释
利用NR、KOG、KO和Swiss-Prot数据库对新异构体进行了注释。71,72].KOBAS v2.0搜索KEGG通路[73].GO注释采用BLASTX v2.2.26和BLAST2GO v2.3.5软件[74].
LncRNA和ORF鉴定
为了鉴定LncRNA,首先在NR、KOG、KO和Swiss-Prot数据库中以默认参数搜索已知基因的新异构体或PacBio数据获得的新基因的新异构体。筛选出BLAST命中1E-5的亚型,其余亚型通过CPAT v1.2.2进一步评估蛋白编码能力(http://lilab.research.bcm.edu/cpat/).
为了预测orf,使用transDecoder软件识别潜在的编码序列(http://transdecoder.sf.net).默认情况下,TransDecoder预测的orf长度。LongOrfs至少含有100个氨基酸。为了提高orf的敏感性,使用BlastP将可能的orf翻译蛋白与Swiss-Prot数据库进行同源蛋白鉴定。同时,通过Hmmscan从Pfam数据库中确定蛋白质结构域[75,76].随后,TransDecoder。根据上述结果使用Predict对所有预测的orf进行筛选,保留与Swiss-Prot数据库同源或具有相同域的orf。
AS、融合转录本和APA鉴定
使用ASprofile软件确定替代剪接(AS)事件[77].利用ASprofile对同一基因位点上的不同异构体进行比较,对剪接类型(M) SKIP、(M) IR、AE、X (M) KIP、X (M) IR和XAE进行分类和特征分析。2摄氏度).融合转录本是5 '和3 '序列映射到参考基因组中两个或多个基因位点的转录本,对应于5 '伴侣和3 '伴侣基因。融合基因检测采用中国武汉Frasergen公司自主研发的iso-seq融合转录本检测软件。软件原理图如图所示。二维.Poly-A位点是RNA转录后的重要修饰位点。Tapis软件获得可靠的APA位点[33].
rt - pcr验证
从DH系FT的花器官(包括花药、萼片、花丝、花瓣和雌蕊)中提取总RNA,按上述方法混合。使用FastQuant RT Super Mix (TIANGEN, China)进行反转录。RT-PCR在10 μl体积,含50 ng DNA, 1.0 μl 10 Taq反应缓冲液(含Mg2+), 2.5 mM dNTP 0.8 μl, 0.5 μm正、反引物各1 μl, Taq DNA聚合酶1u (TIANGEN, China)。扩增在iCycler热循环仪(Bio-Rad, USA)上进行,循环参数如下:95°C初始变性5分钟,95°C 30秒,56°C 30秒,72°C 30秒,最终在72°C延伸10分钟,循环35次。基因特异性引物采用GENEWIZ(苏州)Primer Premier 5.0设计。PCR产物在2%琼脂糖凝胶上分析,然后进行Sanger测序。所有引物都列在附加文件中2:表S14。
数据和材料的可用性
支持本文结论的数据集包含在本文及其附加文件中。我们将原始SMRT数据保存在国家生物技术信息中心(NCBI)的序列阅读档案(SRA)中,登录号为SRR10259626, SRR10259627和SRR10259628的生物项目PRJNA576779。Illumina RNA-Seq数据上传到SRA,登录号为SRR10247439,生物项目ID为PRJNA576332。基因组序列和基因注释信息B.rapa已于http://brassicadb.org/brad/datasets/pub/Genomes/Brassica_rapa/V3.0/.
缩写
- APA:
-
可变聚腺苷酸化
- 为:
-
可变剪接
- CCS技术:
-
循环一致性序列
- FLNC:
-
全身non-chimeric
- LncRNA:
-
长链非编码RNA
- 门店:
-
新一代测序
- 子:
-
开式阅读架
- PID:
-
percentage-of-identity
- SMRT:
-
单分子实时测序
参考文献
- 1.
桑·F,尼克伦·s·科尔森。DNA测序链终止抑制剂。《美国国家科学院学报》上。1977年,74(12):5463 - 7。
- 2.
van Dijk EL, Auger H, Jaszczyszyn Y, Thermes C.十年的下一代测序技术。植物学报,2014;30(9):418-26。
- 3.
王志强,王志强,王志强。RNA-Seq:转录组学的革命性工具。植物学报。2009;15(1):57-63。
- 4.
安华,杨忠,易斌,文娟,沈娟,涂军,马超,付涛。中国pol CMS花蕾可育与不育的比较转录谱分析B显著.BMC Genomics. 2014;15:258。
- 5.
刘超,刘震,李超,张勇,冯慧。大白菜遗传雄性不育系可育芽和不育芽的转录组比较分析。中国生物医学工程学报。2016;52(2):130-9。
- 6.
刘小强,于春春,董建国,徐阿希,胡文雄。新创油菜温敏雄性不育突变体的转录组重建(芸苔属植物显著;十字花科)。应用植物科学,2017;5(12).pii:apps.1700077。
- 7.
裴霞,景芝,唐志,朱艳。比较转录组分析研究西兰花细胞质雄性不育相关差异表达基因(芸苔属植物oleracea意大利语)。科学通报。2017;217:234-42。
- 8.
王松,王超,张XX,陈旭,刘俊杰,贾晓峰,贾淑琪。转录组新创白菜细胞质雄性不育相关差异表达基因的组装与分析。植物物理学报,2016;
- 9.
徐红梅,孔晓东,陈峰,黄建新,楼晓霞,赵建勇芸苔属植物显著使用RNA-Seq和脂类相关候选基因的鉴定。BMC Genomics. 2015;16(1):1 - 10。
- 10.
闫霞,董超,于娟,刘伟,姜超,刘娟,胡强,方霞,魏伟。新型恢复系NR1与Nsa CMS杂交自花后代可育和不育植株幼芽转录组谱分析芸苔属植物显著.生物化学学报。2013;14(3):1-16。
- 11.
周欣,刘震,季荣,冯慧。大白菜多等位基因遗传雄性不育可育花蕾与不育花蕾的转录谱比较(芸苔属植物定l . ssp。学报).中国生物医学工程学报。2017;29(5):967-90。
- 12.
古德温,麦克弗森,JD,麦康比WR。成年:下一代测序技术的十年。植物学报,2016;17(6):333-51。
- 13.
Sharon D, Tilgner H, Grubert F, Snyder M.人类转录组的单分子长读研究。中国生物工程学报。2013;31(11):1009。
- 14.
王波,曾娥,Regulski M, Clark TA,韩涛,焦杨,陆震,Olson A, Stein JC, Ware D.利用单分子长读测序揭示玉米转录组的复杂性。Nat Commun, 2016;7:11708。
- 15.
董玲,刘宏,张娟,杨松,孔刚,褚建军,陈楠,王东。单分子实时转录本测序为普通小麦基因组注释和籽粒转录组研究提供了便利。BMC Genomics. 2015;16:1039。
- 16.
李B, Fillmore N, Bai Y, Collins M, Thomson JA, Stewart R, Dewey CN。评价德诺来自RNA-Seq数据的转录组组装。中国生物医学工程学报。2014;15(12):553。
- 17.
朱飞飞,陈明明,叶宁华,石磊,马kl,杨建峰,曹玉玉,张玉杰,Yoshida T, Fernie AR,范光杰,温波,周瑞,刘天宇,范涛,高波,张丹,郝广峰,肖山,刘永刚,张杰。蛋白质基因组分析揭示了拟南芥幼苗脱落酸反应的选择性剪接和转译。中国植物学报,2017;29(3):344 - 344。
- 18.
Schadt EE, Turner S, Kasarskis A.第三代测序的窗口。中国生物医学工程学报。2010;19(R2): R227-40。
- 19.
普什卡列夫D,内夫NF,奎克博士。单个人类基因组的单分子测序。生物技术学报。2009;27(9):847-50。
- 20.
Roberts RJ, Carneiro MO, Schatz MC. SMRT测序的优势。中国生物医学工程学报。2013;14(7):405。
- 21.
van Dijk EL, Jaszczyszyn Y, Naquin D, Thermes C.测序技术的第三次革命。植物学报,2018;34(9):666-81。
- 22.
Allen SL, Delaney EK, Kopp A, Chenoweth SF。锯齿果蝇基因组的单分子测序。G3(贝塞斯达)。2017; 7(3): 781 - 788。
- 23.
Clavijo BJ, Venturini L, Schudoma C, Accinelli GG, Kaithakottil G, Wright J, Borrill P, Kettleborough G, Heavens D, Chapman H, Lipscombe J, Barker T, Lu FH, McKenzie N2, Raats D, Ramirez-Gonzalez RH, Coince A, Peel N, Percival-Alwyn L, Duncan O, Trösch J3, Yu G, Bolser DM, Namaati G, Kerhornou A, Spannagl M, Gundlach H, Haberer G, Davey RP, Fosker C, Palma FD, Phillips AL, Millar AH, Kersey PJ, Uauy C, Krasileva KV, Swarbreck D, Bevan MW,改良的异源六倍体小麦基因组的组装和注释确定了完整的农艺基因家族,并为染色体易位提供了基因组证据。中国生物工程学报,2017;27(5):885-96。
- 24.
Csabai Z, Tombácz D, Deim Z, Snyder M, boldogkzyi Z.小说全基因组序列分析。东南欧洲分离的伪狂犬病毒株可感染传播微生物。2019;2019:1806842。
- 25.
Edger PP, VanBuren R, Colle M, Poorten TJ, Wai CM, Niederhuth CE, Alger EI, Ou S, Acharya CB, Wang J, Callow P, McKain MR, Shi J, Collier C, Xiong Z, Mower JP, Slovin JP, Hytönen T, Jiang N, Childs KL, Knapp SJ。单分子测序和光学作图改良了林地草莓(草莓属vesca)染色体尺度上的相似性。Gigascience。2018;7(2):1 - 7。
- 26.
李艳,魏伟,冯军,罗红,皮敏,刘志,康晨。基于illumina和smrt的野生草莓Fragaria vesca基因组序列的重新注释。DNA决议2017。https://doi.org/10.1093/dnares/dsx038.
- 27.
彭智,胡勇,谢娟,Potnis N, Akhunova A, Jones J,刘智,White FF,刘森。Long read和单分子DNA测序简化了基因组组装和TAL效应基因分析黄translucens.BMC Genomics. 2016;17:21。
- 28.
Prakash G, Kumar A, Sheoran N, Aggarwal R, Satyavathi CT, Chikara SK, Ghosh A, Jain RK。利用PacBio单分子实时测序和Illumina NextSeq 500测序获得珍珠谷瘟病病原菌Magnaporthe grisea菌株PMg_Dl的基因组序列。微生物资源公告。2019;8(20)。pii: e01499-18。
- 29.
张玲,胡娟,韩霞,李娟,高艳,Richards CM,张超,田勇,刘刚,居红,王东,田勇,杨春,孟M,袁刚,康刚,吴勇,王凯,张宏,王东,丛萍。一个高质量的苹果基因组组装揭示了一个逆转录转座子与红色果实颜色的关系。自然科学进展。2019;10(1):1494。
- 30.
邓赵L, L,李G,金H, Cai J,商H,李Y,吴H,徐W,曾庆红L,张R,赵H,吴P,周Z,郑J, Ezanno P,杨AX,燕问,认为MW,他J .单分子测序的M13没有扩增病毒基因组。PLoS One. 2017;12(12):e0188181。
- 31.
Lipson D, Raz T, Kieu A, Jones DR, Giladi E, Thayer E, Thompson JF, Letovsky S, Milos P, Causey M.酵母转录组的单分子测序。生物技术学报。2009;27(7):652-8。
- 32.
徐震,Peters RJ, Weirather J,罗红,廖波,张旭,朱勇,吉阿,张波,胡松,Au KF,宋娟,陈松。丹参不同根组织与丹参酮生物合成结合测序平台获得的转录组全长序列及剪接变异。中国植物学报,2015;26(6):344 - 344。
- 33.
Abdel-Ghany SE, Hamilton M, Jacobi JL, Ngam P, Devitt N, Schilkey F, BenHur A, Reddy AS。高粱转录组的单分子长读研究。Nat Commun, 2016;7:11706。
- 34.
王涛,王红,蔡东,高艳,张红,王艳,林超,马玲,顾玲。毛竹根状茎相关选择性剪接和选择性聚腺苷酸化的综合分析。中国植物学报,2017;29(4):344 - 344。
- 35.
许他L,傅年代,Z,燕J,徐J,周H,周J,陈X, Y,李盟KF,姚明H .杂交测序茎和叶的全长cDNA成绩单石斛兰officinale。基因(巴塞尔)。2017; 8(10)。pii: E257。
- 36.
赵颖,袁娟,李松,贾松,韩玲,徐玲。红三叶草转录本和剪接异构体分析(三叶草借口L.)单分子长读测序。中国生物医学工程学报。2018;18(1):300。
- 37.
晁颖,袁杰,郭涛,徐玲,穆志,韩玲紫花苜蓿l.单分子长读测序。中国生物医学工程学报,2019;39(3):319 - 319。
- 38.
太阳王X,王H,王J, R,吴J,刘年代,白Y, Mun JH,班克罗夫特,程F,黄年代,李X,华W,王J,王X, Freeling M,皮雷JC,帕特森啊,Chalhoub B, B,海沃德,夏普AG)、公园BS, Weisshaar B,刘B, B,刘B,通C, C, C杜兰,彭C, C耿,Koh C, C林,爱德华兹D,μD,沈D, Soumpourou E, F, F弗雷泽,科南特G, G Lassalle,国王GJ, Bonnema G,唐H, H, Belcram H,周H, Hirakawa H,安倍H,郭H, H,金H,帕金IA, Batley J, Kim JSJ,李江,徐J,邓J,金正日是的,李J, Yu J,孟J,王J, Min J, J,也不会王J, K畠山直哉,吴K, L,方L,技巧,链接毫克,赵M,金米,Ramchiary N, Drou N,伯克曼PJ, Cai Q,黄Q, R, Tabata年代,程年代,张,张年代,黄,佐藤年代,太阳年代,Kwon SJ,老崔,李TH,风扇W,赵X, X,徐X, Y,邱Y,阴Y, Y, Y Du,廖Y, Y Lim, Narusaka Y, Y小王,小王Z,李Z,王Z,熊Z,张Z;芸苔属植物拉伯基因组测序项目联盟。中多倍体作物物种的基因组芸苔属植物拉伯.中国科学。2011;43(10):1035-9。
- 39.
蔡超,王旭,刘波,吴娟,梁娟,崔勇,程峰,王旭。芸苔属植物拉伯2.0:通过序列重新组装和基因重新注释进行参考升级。植物学报,2017;10(4):649-51。
- 40.
张磊,蔡旭,吴娟,刘敏,Grob S,程飞,梁娟,蔡超,刘铮,刘波,王峰,李松,刘峰,李旭,程磊,杨伟,李明华,Grossniklaus U,郑辉,王旭芸苔属植物拉伯参考基因组单分子测序和染色体构象捕获技术。Hortic Res. 2018;5:50。
- 41.
宁刚,程旭,罗鹏,梁峰,王志,于刚,李霞,王东,鲍明。混合测序和图谱发现(HySeMaFi):广泛破译无参考基因组生物基因剪接和表达的可选策略。科学代表2017;7:43793。
- 42.
黄玲,董辉,周东,李敏,刘艳,张芳,冯勇,于东,林松,曹娟。黄龙花粉发育和受精过程中长链非编码rna的系统鉴定芸苔属植物拉伯.植物学报,2018;36(1):344 - 344。
- 43.
Kelemen O, Convertini P, Zhang ZY, Wen Y, Shen ML, Falaleeva M, Stefan S.选择性剪接函数。基因。2013;514(1):行。
- 44.
王et, Sandberg R, Luo S, Khrebtukova I, Zhang L, Mayr C, Kingsmore SF, Schroth GP, Burge CB。人类组织转录中的替代异构体调控。大自然。2008;456(7221):470 - 6。
- 45.
童超,王鑫,于娟,吴娟,李伟,黄娟,董超,华伟,刘松芸苔属植物拉伯.BMC Genomics. 2013;14:689。
- 46.
Weirather JL, Afshar PT, Clark TA, Tseng E, Powers LS, Underwood JG, Zabner J, Korlach J, Wong WH, Au KF。融合基因及其在乳腺癌中显著表达的融合异构体的杂交测序鉴定。核酸工程学报,2015;43(18):e116。
- 47.
de Almeida SF, García-Sacristán A, Custódio N, Carmo-Fonseca M.核RNA监测、人外泌体和RNA聚合酶II转录终止之间的联系。中国生物医学工程学报,2010;38(22):8015-26。
- 48.
王志刚,王志刚,王志刚。雄蕊结构与功能。植物科学。2004;16(增刊):S46-60。
- 49.
砂光机。
- 50.
Schnable PS, Spriger NM。作物杂种优势研究进展。植物学报,2013;
- 51.
植物花药发育的转录调控Arabidpsis.基因。2019;689:202-9。
- 52.
范文彬,李志刚,李志刚,李志刚。端粒到端粒的研究进展新创组装恶性疟原虫基因组通过长读(>11kb)、单分子、实时测序。DNA res 2016;23(4): 339-51。
- 53.
Quail MA, Smith M, Coupland P, Otto TD, Harris SR, Connor TR, Bertoni A, Swerdlow HP, Gu Y.三个下一代测序平台的故事:离子流的比较。BMC Genomics. 2012;13:341。
- 54.
Alexandrov NN, Troukhan ME, Brover VV, Tatarinova T, Flavell RB, Feldmann KA。的特性拟南芥使用全长cdna发现的基因和基因组。植物分子生物学。2006;60(1):69-85。
- 55.
Kalsotra A, Cooper TA。发育调节选择性剪接的功能后果。植物学报。2011;12(10):715-29。
- 56.
徐普杰,朴MJ,朴CM植物低温胁迫响应中转录因子的选择性剪接:机制与功能。足底。2013;237(6):1415 - 24。
- 57.
菲利奇金SA,牧师HD,吉万SA,沈R,科比DW,福克斯SE,王wk,莫克勒TC。可选剪接的全基因组图谱拟南芥.基因组学报,2010;20(1):45-58。
- 58.
Li P, Ponnala L, Gandotra N, Wang L, Si Y, Tausta SL, Kebrom TH, Provart N, Patel R, Myers CR, Reidel EJ, Turgeon R, Liu P, Sun Q, Nelson T, Brutnell TP。玉米叶片转录组的发育动态。中国科学,2010;42(12):1060-7。
- 59.
张刚,郭刚,胡霞,张艳,李强,李锐,庄茹,陆震,何震,方欣,陈琳,田伟,陶勇,Kristiansen K,张欣,李松,杨红,王娟,王洁。水稻转录组的单碱基对深度测序显示出较高的复杂性。基因组学报,2010;20(5):646-54。
- 60.
关婷,Benovoy D, Dias C, Gurd S, Provencher C, Beaulieu P, Hudson TJ, Sladek R, Majewski J.人类转录异构体变异的全基因组分析。植物学报。2008;40(2):225-31。
- 61.
晁强,高泽峰,张东,赵广兵,董丰强,付彩霞,刘丽娟,王bc。杨树茎转录组的发育动态。植物科学进展,2019;17(1):206-19。
- 62.
乔东,杨超,陈杰,郭艳,李艳,牛松,曹凯,陈哲。茶树次生代谢通路相关全长转录本的综合鉴定及选择性剪接。科学通报2019;9(1):2709。
- 63.
孙勇,侯辉,宋辉,林凯,张震,胡娟,庞娥。黄瓜多组织选择性剪接的比较。中国生物医学工程学报。2018;18(1):5。
- 64.
王敏,王鹏,梁峰,叶震,李娟,沈超,裴林,王峰,胡娟,涂林,Lindsey K,何东,张霞。异源多倍体棉花选择性剪接的格局、复杂性和调控研究。中国生物工程学报,2018;29(1):344 - 344。
- 65.
张伟,宋杰,杨晨。花药开裂的调控。中国科学(d辑),2011;
- 66.
董x,洪泽,Sivaramakrishnan M, Mahfouz M, Verma DP。胼胝合酶(CalS5)是小配子体发生时外壁形成和花粉存活所必需的拟南芥.植物学报,2005;42(3):315-28。
- 67.
Persson S, Paredez A, Carroll A, Palsdottir H, Doblin M, Piondexter P, Khitrov N, Auer M, Somerville CR拟南芥.《美国国家科学院学报》上。2007; 104(39): 15566 - 71。
- 68.
李文杰,李志强,李志强,陈志强。基于PacBio算法的PacBio修正算法。生物信息学。2014;30(21):3004 - 11。
- 69.
吴道明,汪达边CK。GMAP:用于mRNA和EST序列的基因组定位和比对程序。生物信息学。2005;21(9):1859 - 75。
- 70.
唐元D, Z,王M,高W,涂L,金X,陈L, Y,张L,朱L,李Y, Z,梁问杨X,刘N,金,Lei Y, Y叮,李G,阮X, Y阮,张X岛棉花基因组序列(海岛棉)为更好的可纺纤维的异源多倍体化和发展提供了见解。科学通报2015;5:17662。
- 71.
布芬克,谢C,胡森DH。快速和敏感的蛋白质定位使用金刚石。Nat method . 2015;12(1): 59-60。
- 72.
贾steiger E, Jung E, Bairoch a . SWISS-PROT:通过蛋白质数据库连接生物分子知识。分子生物学,2001;3(3):47-55。
- 73.
谢超,毛晓霞,黄娟,丁勇,吴娟,董松,孔林,高刚,李春春,魏林。KOSAS 2.0:一种用于丰富通路和疾病标注和识别的web服务器。Nucleic Acids Res. 2011;39(Web Server issue): W316-22。
- 74.
Conesa A, Götz S, García-Gómez JM, Terol J, Talón M, Robles M. Blast2GO:功能基因组学研究中注释、可视化和分析的通用工具。生物信息学。2005;21(18):3674 - 6。
- 75.
基于概率推理的新一代同源搜索工具。基因组学报,2009;23(1):205-11。
- 76.
Finn RD, Mistry J, Tate J, Coggill P, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer EL, Tate J, Punta M. Pfam:蛋白质家族数据库。核酸研究数据库,2010;38:D211-22。
- 77.
Florea L. Song L和Salzberg SL.成千上万的外显子跳过事件在16种人类组织中的剪接模式中进行区分。F1000Res。2013;2:188。
确认
感谢中国武汉弗雷泽根生物信息学有限公司的冯彦博提供了PacBio后续平台的相关文献,并积极协调与技术人员的沟通,促成了本稿件的完成。我们也感谢Editage (www.editage.cn)进行英文编辑。
资金
国家重点研发计划项目(No. 2016YFD0101701)和国家自然科学基金项目(No. 31772298、31201625)资助。每个资助机构根据一份研究提案授予资金。他们对研究的设计、数据的收集、分析和解释以及手稿的撰写没有任何影响。
作者信息
从属关系
贡献
ZL和FH构思并设计了这项研究。CT分析数据并撰写稿件。CT和HL进行了验证实验。CT、JR、XY进行数据分析。所有的作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
所有的作者都宣称他们没有竞争利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1图S1。
RT-PCR验证AS事件(1-3)和融合转录本(4-6)。M, DNA标记DL2000;一个花药;年代,萼片;F,灯丝;体育,花瓣;π,雌蕊;1、m54191_180531_084316/71238183/3459_97_CCS;2、m54191_180531_084316/15467311/43_3034_CCS;3, m54045_180508_172253/21365668/2097_84_CCS; 4,BraA03g009520.3C;5,BraA01g012300.3C;6,BraA02g020980.3C.
附加文件2表S1。
来自PacBio Sequel平台的聚合酶阅读摘要。表S2。来自PacBio Sequel平台的子阅读摘要。表S3。来自PacBio Sequel平台的ccp摘要。表S4。序列纠错前后全局PID统计。表S5。PacBio数据集中全长转录本的评估。表S6。定位到参考基因组的位点和异构体分类。表S7。PacBio Sequel平台对所有新颖异构体进行功能注释。表S8。PacBio Sequel平台lncrna信息。表S9。PacBio Sequel平台检测ORF信息。表S10。PacBio Sequel平台检测剪接异构体。表S11。PacBio Sequel平台检测融合基因。表S12。PacBio Sequel平台检测Poly-A位点。表向.花药和花粉发育相关基因b·拉伯基因组v3.0。表S14系列.用于RT-PCR验证的引物。
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
谭超,刘宏,任杰,刘志强。et al。利用单分子实时测序技术,对大白菜花药的转录本和剪接异构体进行分析(芸苔属植物拉伯l . ssp。学报).BMC植物生物学19日,517(2019)。https://doi.org/10.1186/s12870-019-2133-z
收到了:
接受:
发表:
关键字
- 白菜
- 花药
- 完整的成绩单
- 可变剪接
- 融合成绩单