摘要
背景
种质库保存着代表作物改良可用的最全面的本地遗传多样性目录的收藏。种质库的使用者感兴趣的是尽可能广泛地代表更广泛的收藏中所存在的多样性的固定数量的样本。一个相关的问题是,是否有必要开发完全独立的种质样品,或者有可能从预先定义的核心集面板中选择嵌套集,而不是从整个收集。本研究利用国际玉米种质资源库(CIMMYT)的15384个玉米地方品种的数据,研究了以下8种多样性标准对样本代表性的影响:(1)两种核心选择策略,统计抽样(DM)或数值最大化方法(CH);(2)选择不同大小的样本;(3)选择不同大小的样本,相互独立或嵌套。
结果
样本量大于10%的样本量保留了75%以上的多态性标记,适用于所有选择策略和样本类型;较低的样本量在重复中表现出更大的变异性(不稳定性);样本量对CH-independent组合的影响最大。独立样本和嵌套样本在DM法的所有标准中表现相似,但在CH法中却存在差异。DM方法比CH方法能更好地逼近种群中的已知值;CH方法的采集和样本的二维多维标度图突出了样本选择趋向于多样性的极值,而DM方法的抽样更能代表多样性的整体基因型分布。
结论
使用大于或等于整个集合10%的核心子集,很好地满足了代表性和多样性的要求。巢式样本与独立样本具有相似的多样性和代表性,为种质资源库的样本定义提供了一种经济有效的方法。对于大多数评估标准来说,DM方法比CH方法更接近总体中的已知值,也就是说,它从收集的样本中产生了更具有统计代表性的样本。
背景
全球的种质库保存着世界上最重要的食品和饲料品种,造福人类。这些收藏构成了最全面的本地遗传多样性目录,为面临人口增长、气候变化、饮食变化等挑战的作物改良提供了宝贵的未开发资源。[1].尽管这些系列的固有价值,但在尝试为其特定用例选择适当的材料时,许多种质库客户都面临着令人生畏的任务。纯粹的集合数量和稀疏护照和特征数据通常可用,使选择具有挑战性。
为了解决从种质库中进行可理解选择的一些挑战,一些倡议已经采用了下一代测序和基因分型来更全面地描述收集多样性的某些方面。以玉米和小麦为重点的“发现种子”计划(https://seedsofdiscovery.org/)和以水稻为重点的“3000个基因组”计划(http://iric.irri.org/resources/3000-genomes-project)是两个例子,旨在研究储存在玉米、小麦和水稻种质资源库中的巨大多样性。这种基因组特征,无论是单独还是与其他数据资源结合,都为种质资源库的收集提供了一个新的视角,有可能促进更多用户相关的种质选择。尽管这一数据资源具有巨大的价值,但客户通常无法评估或利用所有感兴趣的材料,需要对整个收集或感兴趣的部分进行某种形式的子集设置。在这种情况下,代表性的概念很重要,在不同的实际方法下对其进行量化,对于维持从整个种质库采集的样本的遗传基础是相关的。在选择材料进行评估和潜在育种时,代表性尤其重要,以避免迅速限制遗传变异的瓶颈。遗传标记多年来一直被用作信息来源,可用于评估种质样品在遗传保护活动中的代表性,如加入、再生和收集。
传统上对异地种质库的遗传资源进行取样,目的是形成核心亚群,用于种质库的保存和遗传多样性研究。在形态、表型或分子标记数据的基础上,可以形成核心亚群(或核心样品),并将其组装起来,以便于异地种质资源的研究、评价和利用[2,3.,4].核心子集通常包括总接入数量的5 - 20% [3.,4,5,6,7,8],因此,与原始集合相比,核心子集代表遗传和表型多样性的减少。在遗传资源保护中,核心馆藏和/或核心子集的形成对于在核心内尽可能地保存原始馆藏中所存在的多样性(代表性)至关重要。通常,一个对保护领域以外的领域有兴趣的种质库客户(例如,植物育种家或分子遗传学家)对固定比例的收集不感兴趣,例如,一个经典的核心子集;相反,她/他更感兴趣的是固定数量的“代表性样本”,这些样本可能具有价值特征,例如代表一个特定的地理区域,适应或起源于一个特定的重音流行的地方。这些“繁殖子集”,就像核心子集一样,需要尽可能广泛地代表更广泛的集合中存在的多样性。
考虑到这些需求,抽样策略应定义抽样方法和分配方法[9].分层采样策略表明,首先将遗传分类为非重叠组(或集群),然后是用于将群集中的载体分配到样本中的方法。90年代后期和2000年代初,发表了对采样和分配策略的激烈研究以及形成核心子集的方法[10,11,12,13,14].提出了一种采用连续和离散形态数据同时形成核心样本的顺序聚类策略。主要的想法是使用几何方法(如Ward方法)来形成组(簇)(使组内的方差最小化)。然后采用“混合正态分布”统计方法,通过改变分组的形状、方向和体积,最大化似然函数,确定每个加入属于每个分组的概率,对以前的分组起作用。这种两阶段分类方法被[15]形成多个热带玉米地方品种的不同核心亚群(玉米L)。
就使用连续和离散(分类)表型特征的混合物时对群集进行采样的分配策略而言,采样可能是跨群集的常量,与群集中的个体数量成比例或与群集中的附加之间的距离成比例,such Gower’s distance, the D allocation method [16].抽样的D分配法也可以应用于分子标记,使从每组抽取的样本应与每个聚类内的遗传距离或等位基因多样性成正比[17].因此,无论每个聚类内的个体总数如何,最终的子集将按比例代表更多的个体,而不是从一个遗传多样性较低的聚类中代表更多的个体。该策略保证了核心样本具有良好的代表性和较高的等位基因丰富度,而遗传距离的应用则提供了遗传距离的欧几里得表示。
在现代基因组技术对种质库加入的预测精度方面,[18研究了从8416个墨西哥小麦样品中获得的核心样品的预测准确性,以及储存在种质群中的2403个伊朗小麦样品。作者根据两个标准定义了10%和20%的核心样本。一个标准是与预测误差方差相关的可靠性措施,其通过应用使用标记数据的前100个主组件的方法来最小化的目标函数[19];他们被称为,在研究[18]预测核心样本。用于选择10和20%核心样本的其他标准基于[16利用改良的罗杰斯遗传距离。genomic-enabled的最后分析预测精度在这项研究表明,使用10%或20%的核心没有不利影响预测精度的特征与整个样本相比,进一步支持,形成多元化的核心样本保持足够的多样性和代表性的人口研究。
为了保持核心样品的等位基因丰富度,还研究和提出了其他一些策略。一种有效的策略使每个标记位点上的等位基因数量最大化;这就是M策略[5].另一种策略是根据群体内遗传多样性的比例从群体中取样,从而使核心样本中的等位基因数量最大化。此外,形成核心样本的其他策略都试图最大化核心样本中的等位基因多样性,而其他方法则最大化核心样本中遗传多样性的代表性[20.].另一方面,其他方法避免在集合的极端处选择相似的加入,从而使每个加入与核心中最近的其他加入之间的平均距离最大化[21].
作者在22[研究了几种用于计算不同标记等位基因的特异性的公式,参考它们的分布跨越换档;根据其等位基因的特异性评估加入稀有性;用于计算kullback-Leibler公式定义的分歧;估计整个集合中的等位基因丰富;并且用于计算丢失的等位基因(丢失不在核心样本的等位基因)以及香农分复指数。这些公式以及改性的罗杰遗传距离(MR)与HCORE和其他形成核心子集的策略(REMC,MIXREP,MSTRAT和随机样品)一起使用[23].上述作者[22把这些公式和方法应用到一大批小麦采集中。对于10%的核心样本,Kullback-Leibler标准略优于MR遗传距离(0.442),但MR克服了其他方法的差异。对于20%的核心样本,Kullback-Leibler标准与MR遗传距离(0.434)相同,但优于其他方法。使用Kullback-Leibler的有效方法有:(i)根据等位基因的平均特异性来确定加入稀有度,(ii)根据其特异性对等位基因进行排序,并检测仅在某些品种中常见的等位基因,以及(3)根据稀有度和差异对这些品种进行排序。因此,检测一组罕见的和特定的亲缘,可能有某些潜在的重要表型性状。
另一种形成不同大小不同核心样本的方法提出了一种拟指数,用于整合遗传距离和多样性指数[24,该指标可作为一种方法,根据分配给标准措施的权重,同时优化多个遗传措施。上述作者[24]提出了Core Hunter (CH)算法,该算法使用一种先进的随机局部搜索算法来最大化伪指数,对多个多样性指数的结果略优于d方法的性能(见[24]),但只有在单一措施正在优化时才。最近改善了初始核心猎人(核心猎人1和2),核心猎人3(CH3)来自[25]包括两种距离汇总方法,即从入口到最近入口和从入口到最近入口[21].此外,CH3包含了两种新的、改进的距离总结方法,用于量化核心集合的多样性或代表性,在最大化改进的多样性度量方面比core Hunter 1和2更有效。
鉴于选择核心子集和最大数量的不同核心样本的问题的高度维度,问题已经多种方式达到了多种方式,今天的解决方案和提案可分为两种主要方法方法:(1)统计数据使用D分配方法“分层随机采样选择”的基本概念的方法,以及使用“数值最大化方法”的基本概念的数值算法方法,例如由核心亨特(CH3)使用的基本概念。它们都集中在相同的目标:获得含有集合中存在的大多数遗传多样性的样本,但前者基于随机样本的代表性的统计概念(特别是核心遗传多样性的代表性,当然,它们的不同措施),而后者基于选择子集的数学概念,最大限度地提高一些标准(用于描述遗传多样性的一个或多个措施)。
对于这两种方法(D-allocation方法或Core Hunter方法),在为种质库经理或种质库客户选择样本时,都会出现两个主要问题。第一个问题是:要最理想地代表整个集合或特定价值(如特定种族、物种等)集合的那一部分的多样性,需要的最小样本量是多少?这个问题改变了传统的10-20%的收集方法来定义种质集。随着基因组重新测序成本的持续降低,以及种质库客户开始询问应该对多少份和哪些种质进行测序,以获得最大的变异,这一研究领域就显得尤为重要。第二个问题反映了种质库客户对获得一定数量的条目的日益增长的需求,例如,150份具有多样性和代表性的种质。在这种情况下,从集合的角度经理,相关要求如果有必要开发完全独立的种质集或是否有可能形成嵌套集抽样的方式面板完成而不是整个收集大量预定义的面板。在这种情况下,嵌套系统很简单,可以维护足够的种子/克隆储备以供分发,好处是客户可以潜在地将评估数据众包,围绕一组公共资源构建丰富的知识。
基于上述考虑,本文的目的是评估,使用数据来自15000多个玉米一起存储在国际玉米和小麦改良中心的玉米种质资源库,对多样性和代表性的影响(1)选择的样本大小5、10、20、30、40 - 50%整个集合(样本尺寸效应),(2)一个集合的独立抽样对嵌套抽样的影响(样本方法效应);(3)采用以具有MR遗传距离的d -方法为代表的统计抽样策略或以CH3和MR距离为代表的数值最大化方法(策略方法)的相对优点。
结果
d方法(DM)中抽样的定义
如上所述,DM-Method是3级方法:首先完成分类(聚类),然后从每个簇中选择的载体比例按比例地定义为群集分集(由组测量)mrd平均值),最后从分层随机抽样过程产生的1000个候选样本中选择最佳(最具多样性)的样本(表)1).
多样性分析
我们在本研究中使用的八个标准来评估与面板定义的不同方法相对于样本大小(收集大小的5%至50%),采样类型(独立或嵌套)和建筑方法核心(统计分层采样与数值最大化的采样)。因为所有标准(除了保留变体和诊断标记的数量除外)具有0到1之间的可能值范围,我们还使用样本值与种群值的比率来比较方法(图3和4中的红线。1和2)评估考虑整体人口的方法的代表性(表格2和3.).
下面我们总结了研究的采样过程如何影响各种标准,并通过与人口级别指标相比。应该指出的是,没有出现比较的统计测试,因为与平均值相比,标准误差太小;因此,几乎所有的比较都产生非常低p值(表2和3.),甚至使用假设指数为Beta、泊松或二项分布的广义线性模型。
修正罗杰斯距离(mrd)
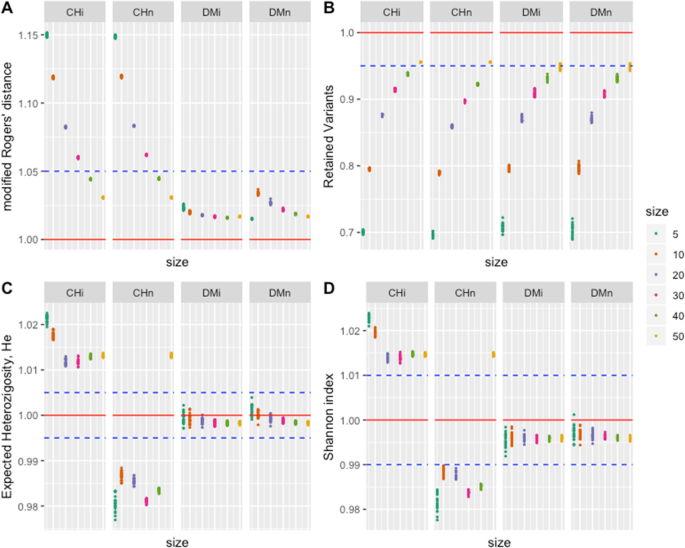
作为所有方法中确定样本的基础,mrd是一个关键的评估指标。的意思是mrd对于所有形成的样本,都高于总体(表)3.).这是可以预期的,因为形成样品的过程是最大的mrd通过遗漏由总体人口中相似个体产生的冗余信息。的意思是mrd值随着样本量的增加而减小,在DM和CH采样方法中,独立样本和嵌套样本之间的值相似(图3)。1a).抽样方法对均值有很强的影响mrd这是一个预期的结果,说明了CH在寻找目标函数的最大值时的有效性(我们正在寻找最大化的样本)mrd通过减少人口中存在的冗余的平均值)。另一方面,DM方法显示比CH方法更接近群体值的值。对于CH独立(CHI)和CH嵌套(CHN)样品,估计mrd只有当样本大小大于或等于S40时,值才接近群体值(从收集值,蓝线的距离的5%的间隔内),而所有样本尺寸和类型的样本都在该间隔内DM-方法的距离(图。1一个)。
多态标记保留数量(poly)
多态性标记数量是衡量多样性的一个重要指标,但与我们评估的其他多样性标准表现不同。最大样本量为50,poly接近总体值的95%,而对于较小的样本量,如s5,poly大约在人口中发现的70%(图。1b).样本量与多态位点保留量之间并非线性关系。在s10或s10以上,样本保留了接近或超过人群中发现的80%的位点,随着样本量的增加,保留的增加减少。所有四种组合方法(CH和DM)和样本类型(独立或嵌套)(Chi、CHn、DMi和DMn)的多态标记的保留模式在所有样本量中非常相似(图3)。1b)。
期望杂合度(he)与Shannon指数(Shan)
两者值的分布他和山对于任何选择的方法都是相似的(图。1C和D)。方法Chi达到了最高值他和山对于所有样本大小,CHN具有高于和低于人口的最低值。方法以类似的方式执行DMI和DMN,具有更接近已知群体的值的值。这些结果很重要,如他和山被认为是非常有用的遗传多样性措施;的确,他通常被称为“遗传多样性”[26,27].从统计学的角度来看,我们可以说dm方法比ch方法更精确地估计总体值,因为dm方法得到的值对于两种类型的样本和所有的样本容量都更接近已知的总体值。也就是说,DM得到了所测量的总体多样性的更好的抽样表示他要么山,而CH方法产生的样本过多代表(CHi)或过少代表(CHn)已知总体值。
观察杂合度(ho)和ho -ho测量
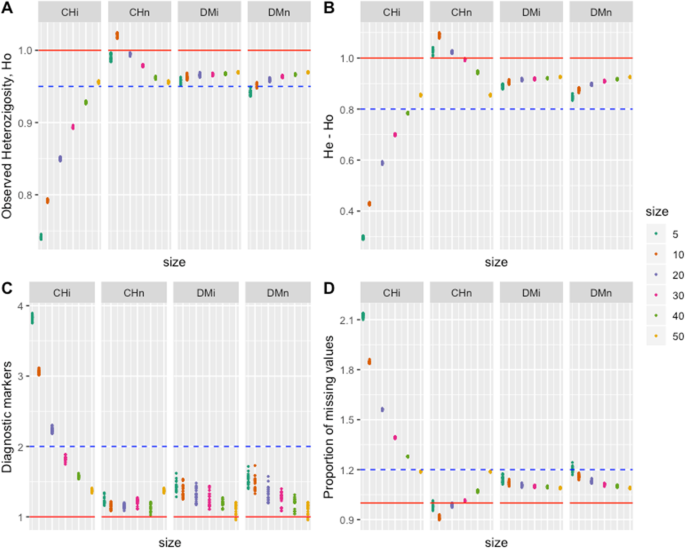
观察到的杂合度显示出不同的模式他和山:除了所选组的值的所有(S10)都属于人口值。CHN,DMI和DMN更接近所有样本尺寸的人口值,而CHI值低于上述值,特别是对于低于或等于S30的样本尺寸。低估何除CHn外,所有方法的样本量均随样本量的减小而增大,表现出非预期的性能(图3)。2a).再一次,DM似乎总是更接近(并且在5%的区间内)观察到的整个集合的杂合度。
的他,何差异是近交的一种度量(\(号= 1 - \压裂{ho}{他}\)),当纯合子亲本比预期的多时,则为正值他值,即近交系数大于预期;当纯合子数小于预期时,为负值,即近交系数小于预期。整个种群近交系值为1-0.0655/0.0400 =−0.642).的he-ho测量表现类似于观察到的杂合度,表明有更多的变异性何比他可变性。再一次,he-ho对CHn、DMi和DMn的估计较好,而对CHi的估计较低(表明近亲繁殖更多),特别是当样本量小于或等于s30时(图3)。2b)总之,用于标准何和he-ho,与CHi和CHn样本法相比,DMi和DMn在整个集合值附近更稳定,始终在总体均值附近的5%区间内。
诊断标记(ndiag)
诊断标记是指那些在人群或样本中只出现在少数供体中的变异,它们的存在对这些供体具有指示性或诊断性。当研究不同品种间的遗传差异时,由于样本中基因型总数的减少,样本量的减少应导致该标准的增加。CHn的性能与DMn和DMi相似,因为它们的值接近或高于(小于2倍)总体值;然而,CHn在样本大小上没有明显的变化,而DMn和DMi则随着样本大小的增加而减小(图2)。2c).当样本量小于s30时,CHi值大于群体值的2倍,表明对对比等位基因种质的选择较强。
缺失值比例(pmiss)
缺失值的比例是一个重要的度量,不是对于多样性本身,而是对于样本所代表的信息的质量(完整性)。几乎所有样本的缺失值比例都高于总体(图。2D,见红线)。当样本量小于s30时,CHi方法得到的比例大于总体缺失值的1.25倍。对于其他方法和样本量,缺失值所占比例不大于总体值的1.25倍。从s10开始,CHn样本中缺失值所占的比例随着样本量的增加而增加。相比之下,两种DM方法都呈现出相反的关系,即采购经理人指数值随着样本量的增加而减小(图。2D),然而样本量的影响较小,数值更密切地跟踪采购经理人指数所有的收藏品。
如何选择样本:整个集合的多维缩放2D图形表示
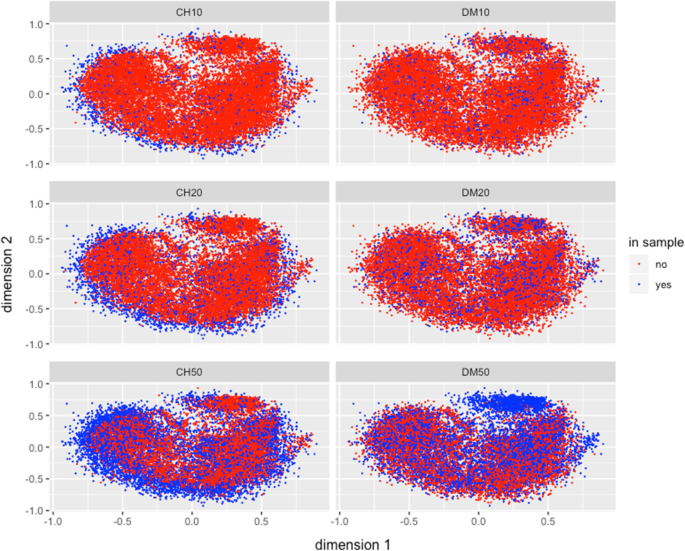
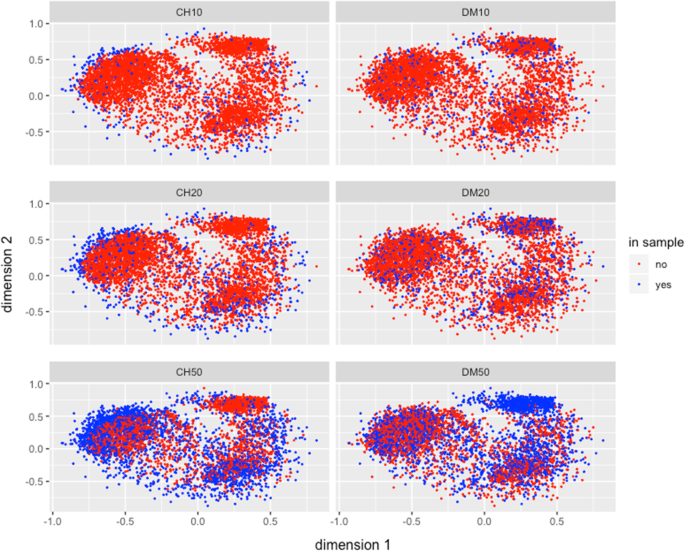
CH和DM方法观察到的差异对多样性的措施和取样的代表性,可以更好的理解通过观察到达被CH和DM方法选择不同大小的样本在mrd的多维标度表示两个维度。数字3.显示了两种方法收集的最佳独立s10、s20和s50样品;这说明,来自CH的s10样本从整个种群的边界捕获了更多的多样性,而来自DM的样本捕获了分布在整个种群的基因型(蓝点)。对于其他样本量s20和s50,观察到样本的同样行为。方法CH通过对整个集合的极端样本进行抽样,使多样性最大化,而DM方法则对整个集合的所有部分进行均匀抽样。s20和s50也有类似的结果(图。3.), DM方法的样本更均匀地反映了15384份玉米种质资源的分布情况。
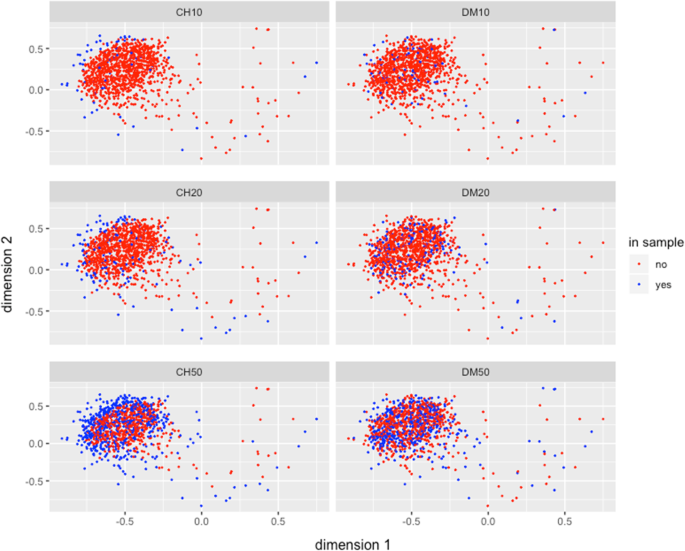
由CH和DM分别为玉米品种Conico选择的独立样本s10, s20, s50的mrd的多维尺度的二维表示如图所示。4.与已经描述的考虑整个集合的情况的结果相似(图。3.), DM方法比CH方法更完整地反映了Conico玉米小种存在的总变异性,CH方法集中了三个独立样本大小(s10, s20和s50) Conico材料分布的极端取样。.
此外,在研究高原适应区选择的玉米资源的mrd多维尺度的二维表示时(图。5),使用大小为S1、S20和S50的独立样本,我们再次观察到DM方法的样本比CH方法选择的样本更具有代表性,CH方法选择的样本更多地集中在分布的边缘。综上所述,对于整个玉米采集和基于种族和适应的样本,Conico玉米和高原玉米样本,CH基本上选择多样性分布的极端边界,而DM选择多样性分布的整个谱。
稳定性:同一过程重复的可变性
表格4显示在每种方法中重复(20重复)之间的变化的变化,样本类型和样本大小;清楚我们介绍了标准偏差的比率:stdev_sample / stdev_s50。最重要的结果是低样本尺寸意味着重复之间的变化暗示,即获得“坏”(或“非常好”)样本的概率对样本大小增加。第二次观察是CHI比其他方法和类型(独立或嵌套)产生更类似的重复。采购经理人指数,山,多态标记和具体标记,而DMi则产生更多类似的重复他,何,mrd标准;第三个观察是最不稳定的标准,也就是显示更多不同重复的标准poly和n诊断接头.
外部信息的恢复(种族和适应区域)
表5和6显示收集到的23个种族和收集到的8个适应地区的品种的分类。对于每个种族和地区,一个好的样本应该选择与收集的比例相似的比例。表中的最后两列5和6显示每个种族和地区在采集中的比例与CH法和DM法获得的样本的差异。在品种恢复方面,CH法和DM法的差异最大值分别为2.63和1.76%,而在适应区,差异最大值分别为4.62和3.70%。DM方法显示出轻微的优势,但总的来说,两种方法都选择了适当比例的两个外部(对分析)变量。
最后,表7显示了最常用的遗传多样性测量和Wright(1951)的种族和适应样本统计。该表还显示了CH和DM方法(最佳s20样本)的收集(总体)值及其估计。样本估计值与采集值相似;这是一个有趣的点,特别是对于F圣(AMOVA Fst):适应区域(群体2.67%,DM样本2.89%,CH样本1.92%)和小种(群体7.89,DM样本8.48,CH样本8.38%)之间的差异。一般来说,DM比CH更接近收集值(种群)。然而,与所研究的收集值相比,这两种策略都能很好地估计种族和地区之间的差异。
讨论
样本大小的影响
样本大小对杂合度影响最大的是CHi方法(他和he-ho指标),诊断标志物数量(ndiag)和样本中缺失值的比例(采购经理人指数);对于这些标准,当样本量减少时,差异更大。而对于其他方法,缩小样本量的作用则没有同样强的作用。对于所有方法和类型的样本,大于s20的样本保留了85%以上的多态标记。在所有情况下,样本量的减少与重复之间的标准偏差的增加有关,也就是说,过程更不稳定。
样本效应类型
对于dm方法的所有标准,独立的和嵌套的样本表现出相似的性能。而CH-method得到的嵌套样本与独立样本之间存在差异,尤其是期望杂合度与观察杂合度、Shannon指数、诊断标志物数量、样本缺失值比例等方面存在差异。在这些情况下,嵌套样本比独立样本表现更好,对不同的标准更稳定。这一发现是有价值的,因为使用嵌套样本,避免了对不同样本大小选择非常不同的资源,这对集合管理人员是有好处的,因为可以集中精力维护集合的定义子集的足够的种子/克隆,以便更频繁地分发给客户。
样本选择策略(CH、DM方法)
结果表明,除保留的多态标记数外,所有标准中均包括mrd对于所有样本容量,对于两种类型的样本(独立或嵌套),统计DM方法比CH方法更接近已知的总体值(即样本/总体比率更接近于1)。这一结果是预期的,因为统计分层随机抽样策略的主要优势在于给予来自同一阶层的个体(基于群体或聚类)mrd距离)在样本中选择相同的概率,在采样过程的每个步骤中选择它们中的任何一个,并分配给每个组的样本大小与其多样性成比例。当用于构建嵌套样本时,CH方法会产生类似的结果作为DM,但为最重要的标准,遗传多样性显示出不同的结果(他)和香农指数(山),两者都被低估了。
在收集的多维尺度二维表示中观察了CH和DM方法获取样本的差异,并比较了(由CH和DM)选择的最佳独立s10、s20和s50样本。虽然这些表征说明了DM-method在选择过程中出现的代表性比CH-method的优势,但也显示了DM-method可能存在的弱点:当一组(DM第一阶段获得的聚类)表现出高度多样性时,该方法为样本选择了大量的基因型;如果群体规模不够大,该方法可以选择全部或几乎全部群体基因型(见图中右上角蓝点云)。3.,DM50,并将其与表中的DM-方法从组4和6中选择的分配数量的基因型进行比较1).综上所述,两种策略(CH和DM)可以同时使用,以获得各自的优势,避免各自的缺点。
结论
本研究从CIMMYT种质库的大量玉米材料中发现,CH法比DM法对样本量的影响更大,其代表性和遗传多样性对样本量的影响更大。在CH和DM方法中,占居群总数20%以上的样本量保留了85%以上的多态性标记。独立样本和嵌套样本在DM方法的所有标准上表现相似,而CH方法得到的嵌套样本和独立样本之间存在差异。
一般来说,对于大多数标准,统计DM方法比CH方法更接近已知的总体值。前两个维度的多维标度的情节的收集和最好的(重复)样本选择CH和DM独立样本的大小从10到50%明显显示了选择的矿样中的偏见CH方法,相比更加完整,在统计DM下选择偏倚更小、更均匀的核心样本。两种采样方法对种族及其适应区域信息的恢复结果比较,DM方法优于CH方法,可以更好地恢复整个收集的信息。
方法
基因型种质库
我们使用了来自CIMMYT种质库的22903份原始基因型种质库的数据,这些种质库是根据《粮食和农业植物遗传资源国际条约》的标准材料转让协议(SMTA)提供的可供分发的材料(http://www.fao.org/3/a-bc083e.pdf).这些地方品种采用达特seq™技术进行基因分型。基因分型是对每个加入DNA样本中代表的复合样本(30个个体)进行的。共鉴定出616967个双等位基因单核苷酸多态性(SNP)标记。每个样本中SNP等位基因的频率由每个等位基因的序列计数来确定。通过过滤缺失值(最多允许20%)和标记覆盖率(大于2.0)的结果数据,形成一个包含161,104个SNP标记的最终数据集。种质筛选到最后一组15384个玉米地方品种,收集地点来源的地理数据可用性作为选择标准。表格8描述了15384个地方种族的种族适应组成,表9结果表明,在15384份玉米材料中最终使用的标记具有一定的特性。最终分析中使用的所有数据以及相关的标识符、使用的加入描述和标记过滤参数可通过CIMMYT研究数据和软件存储库网络的CIMMYT种子发现存储库(https://data.cimmyt.org/dataverse/seedsofdisoverydvn.),在题为“玉米地方种质库15384份SNP等位基因频率和描述数据”的研究中,http://hdl.handle.net/11529/10548315.此数据可在许可证和使用条款中描述http://hdl.handle.net/11529/10548315与SMTA下的种质可用性对齐。
抽样方法
三阶段分层随机抽样:d方法(DM)
简要介绍d方法[13,16,17[基于修改的罗杰斯的遗传距离,将遗传分类为组(群集)分类mrd)[28采用“组内最小方差”聚类方法,如[29].适当数量的组是以图形方式定义的,使用正方形与其相关的“伪F”统计量之间的关系。然后将要从每个群集中选择的待选择的数量与平均值成比例地定义mrd每个集群。在确定每个聚类样本的加入数量后,得到了1000个独立的分层随机样本,并得到了均值mrd计算每一个的值;显示最大平均值的样本mrd以Value overall作为最优种质面板。
核心猎人3:CH-method
如[25“Core Hunter是一个多用途的核心子集选择工具,它使用本地搜索算法,根据一个或多个指标生成子集,包括几个距离指标和等位基因丰富度。”它是在R [30.]包Core Hunter (http://www.Core Hunter.org, review October 2018),并允许用户定义和使用不同的选项。在本工作中,我们将ch方法应用于mrd距离矩阵和默认选项。如[25“核心猎人3构建了高多样性的核心集合(高进入距离到最近进入距离;E-NE),最大限度地代表了整个收藏中的个人收藏。”
抽样过程
嵌套和独立的样本代表了两种方法,为用户提供了一种种质银行的换乘器。独立的表示每次采用新样本都与前一个项目无关;嵌套表示当拍摄集合的大样本时,从原始样本中的归档被扣除,以满足用户对该加入的要求。从整个集合的嵌套和独立样本大小的嵌套和独立样本大小的重复(S50 = 7692,S40 = 6154,S30 = 4165,S20 = 3077,S10 = 1538和S5 = 769分别使用两种先前提到的采样方法选择了accessions。
多样性的措施
遗传多样性通常从两个角度进行研究:等位基因遗传多样性,遗传学家和分类学家的观点;个体间遗传多样性,育种家的观点[20.].我们利用等位基因频率来产生6个多样性指标:期望和观察的杂合度及其差异(近交系系数)、Shannon熵指数、多态等位基因或标记的数量,以及诊断标记(仅针对收集的少数品种的标记)。从“繁殖者的角度”,我们使用mrd遗传距离在成对的个人之间,最后,样品中缺失值的比例作为信息恢复的量度。使用以下多样性标准。
预期的杂合性(26,或基因多样性[27],他,是最常用的索引。定义为:\(0 \ Le {He} _i = 1- \ Sum \ limits_ {j = 1} ^ 2 {\ hat {p}} _ {ij} ^ 2 \ le 0.5 \),一个我th二倍体标记(位点),和\(he= frac{1}{L}\sum \limits_{i=1}^L{he}_i \)也就是总体上所有基因位点的平均值。该指标对遗传变异进行了总结,当等位基因频率均为0.5(最大位点多样性)时,二倍体位点的遗传变异最大值为0.5。
观察到的杂合性,何我,为位点杂合子的比例我,并取平均值以进行种群特性描述,何.它受到近亲繁殖和其他进化过程的影响,然后与他,产生近交系系数f轨迹:f我= 1−何我/他我,以及它们在总体中的平均值。的f系数为Hardy-Weinberg均衡下近亲繁殖的最大似然估计[27].我们使用了他,何差异作为近交的衡量标准:负值表示近交高,正值表示近交低,零表示无近交。
香农多样性指数为了我th轨迹:le {sh} \ (0 \ _i = - \总和\ limits_ {j = 1} ^ 2{帽子\ p {}} _ {ij} \子弹{\ mathit {\ log}} _2 \离开({帽子\ p {}} _ {ij} \右)le 1 \ \),以及总体的平均值。我们使用以2为底的对数,因为当等位基因频率等于0.5时,指标值是1.0,多样性的最大值。
改良的罗杰斯距离-个体间的遗传距离
基于其良好的数学和遗传特性[28,我们选择了mrd两个人之间x, y,通过一组L SNP标记测量:
使用专门为自由软件R的脚本进行数据处理[30.].使用了包含4个节点的高性能计算机,每个节点由94个核和512 Gb RAM内存组成。
保留的多态标记数
抽样过程产生的基因型数量的减少,由于在具有相同遗传结构的基因型样本中进行选择,可以减少某些标记的多态性;相反的结果是不可能的,因为在集合中是单态的标记将在任何提取的样本中继续是单态的。由于多态标记的数量是一个集合多样性的衡量标准,我们将其在样本中的减少作为抽样对测量多样性的影响的衡量标准。
诊断标记
当一个等位基因在集合中只有少数基因型是固定的,我们将该等位基因(或标记)定义为诊断型,因为它识别和区分了集合中其他的几组基因型。我们观察了这些等位基因(标记)在不同的样本量和方法的表现。
缺失值的比例
收集或样本中缺失值的比例不是其遗传多样性的衡量标准,但它是任何统计数据集的质量的衡量标准:产生低比例的缺失数据的进程是更好的过程。
代表性
从统计学的角度来看,对一个总体进行抽样最重要的目标是“样本代表性”:一个好的样本应该用测量到的特征值和个体在总体中的频率分布来代表总体。这些原则应用于遗传多样性时,测量“特征”是一个示例的位点的基因结构,意味着一个好的样本必须是个体的一个子集代表大多数人口的遗传结构,即测量和non-measured位点。我们衡量遗传样本代表性的一种方法是,在群体和样本之间比较一套与多样性相关的标准,使样本的值更接近于群体中的已知值。
稳定性或过程的可重复性
取样方法的另一个重要特征是其稳定性或重复性。在本文中,我们对每个“策略-样本类型-样本大小”组合重复20次,以衡量抽样过程的可重复性。计算各标准重复之间的标准偏差,获得重复性的测量值;当标准偏差较低时,该过程被认为更具有可重复性和稳定性。
外部信息的恢复(种族和适应区域)
种质库基因型的特点是不同的外部(非遗传)变量。在这种情况下,我们发现了两个这样的变量:基因型种族和基因型收集的气候适应区。当采用抽样过程时,我们期望外部变量保持属于每个外部组的基因型的比例,就像在收集中一样。每个变量(种族或地区)的基因型比例从整个收集和最好的s20独立样本(CH, DM)中计算。然后对它们进行比较,并用差异的绝对值(总体-样本)作为恢复外部特征的能力的衡量标准。最后,我们进行了分子方差分析(AMOVA),计算了Wright [31]的统计数据,以比较每一种方法在开发样本时的相对表现,这些样本与总体有密切的代表性。
数据处理
我们使用了专门为自由软件R [30.,图是使用ggplot2 [32].进程作为包含5个节点的高性能计算机运行,每个节点由94个核和512 Gb RAM内存组成。
数据和材料的可用性
支持本文结论的数据集以及相关标识符可通过CIMMYT研究数据和软件存储库网络的CIMMYT种子发现存储库获得(https://data.cimmyt.org/dataverse/seedsofdisoverydvn.),在题为“玉米地方种质库15384份SNP等位基因频率和描述数据”的研究中,http://hdl.handle.net/11529/10548315.此数据可在许可证和使用条款中描述http://hdl.handle.net/11529/10548315根据《粮食和农业植物遗传资源国际条约》SMTA规定的种质可得性。
缩写
- CH:
-
猎人核心战略
- 气,中文:
-
独立或嵌套样本从CH方法
- 糖尿病:
-
大小分配与多样性成比例的分层随机抽样策略
- DMi,静:
-
DM方法的独立或嵌套样本
- 他,何:
-
预期和观察到的杂合性
- mrd:
-
改良罗杰遗传距离
- ndiag:
-
“诊断标记物”数量
- 采购经理人指数:
-
缺失值的比例
- poly:
-
多态性标记数
- 山:
-
香农熵指数
- SNP:
-
单核苷酸多态性
参考
- 1.
Hellin J, Bellon MR, Hearne SJ。墨西哥的玉米地方品种和对气候变化的适应。作物改良学报,2014;28(4):484-501。https://doi.org/10.1080/15427528.2014.921800.
- 2.
弗兰克尔哦,棕色AHD。目前植物遗传资源 - 批判性评估。在:遗传学:新的边疆。卷。4.新德里:牛津&IBH出版有限公司;1984. p。1-11。
- 3.
布朗和。核心集合的案例。在:Brown Ahd,Frankel Oh,Marshall Dr,Williams JT,编辑。植物遗传资源的使用。剑桥:剑桥大学出版社;1989. p。136-56。
- 4.
布朗和。核心收藏:遗传资源管理的实用方法。基因组。1989;31:818-24。
- 5.
Schoen DJ, Brown AHD。野生作物亲缘关系的等位基因丰富度的保护借助于遗传标记的评估。美国国立科学研究院1993;90:10623-7。
- 6.
棕色AHD,Spillane C.实施核心收藏 - 原则,程序,进步,问题和承诺。in:rc rcj,hodgkin t,编辑。今天和明天的核心收藏。罗马:国际植物遗传资源研究所;1999. p。1-9。
- 7.
创建核心收藏的一般方法。在:约翰逊RC,霍奇金T,编辑。今天和明天的核心收藏。罗马:国际植物遗传资源研究所;1999.
- 8.
范欣图。植物遗传资源的研究进展。罗马:国际植物遗传资源研究所,公报第3号;ISBN 92-9043-454-6;2000.
- 9.
《抽样与实验》。Environmetrics。2002;13:429-36。
- 10。
Franco J, Crossa J, Villasenor J, Taba S, Eberhart SA。利用层次和密度搜索方法对墨西哥玉米品种进行分类。作物科学。1997;37(3):972 - 80。
- 11.
Franco J, Crossa J, Villaseñor J, Taba S, Eberhart SA。通过分类变量和连续变量对遗传资源进行分类。作物科学。1998;38:1688 - 96。
- 12.
Franco J,Crossa J,VillaseñorJ,Castillo A,Taba S,Eberhart SA。用于在多种环境中对遗传资源进行分类的两阶段三元化方法。农作物科学。1999年; 39:259-67。
- 13.
陈志强,陈志强,陈志强,等。一种多变量环境性状交互作用的研究方法。作物科学。2003;43:1249-58。
- 14.
基因型分类的统计方法。Euphytica。2004;137:19-37。
- 15.
王志强,王志强,王志强,等。基于遗传算法的玉米种质资源评价[J]。作物科学。1998;38:1378 - 86。
- 16.
陈志强,王志强,王志强,等。核心亚群形成过程中保存遗传多样性的采样策略。作物科学。2005;45:1035-44。
- 17.
利用遗传标记构建玉米核心亚群的方法。作物科学。2006;46:854 - 64。
- 18.
Crossa J, Jarquín D, Franco J, Pérez-Rodríguez P, Burgueño J, Saint-Pierre C, Vikram P, Sansaloni C, Petroli C, Akdemir D, Sneller C, Reynolds M, tataris M, Payne T, Guzman C, Peña R, Wenzl P, Sukhwinder S.小麦地方品种基因库基因组预测。G3 Genes Genomics Genetics. 2016; 3:1819-34。
- 19.
Akdemir D, Sanchez JI, Jannink J-L。用遗传算法优化基因组选择训练群体。Genet Sel Evol. 2015。https://doi.org/10.1186/s12711-015-0116-6.
- 20。
Marita JM, Rodríguez JM, Nienhuis J.开发识别最大多样性核心集合的算法。植物遗传资源学报,2000;47:515-26。
- 21。
董立国,杨志强,王志强,等。遗传资源有效利用的核心种质资源质量评价。Theo Appl Genet. 2013; 126:289-305。
- 22。
Reyes-valdésm,burgueñoj,sukhwinder s,Martínezo,Sansaloni Cp。种质银行管理与保护的加入罕见与等位基因特异性的信息观点。柱骨。2017; 13(2):E0193346。https://doi.org/10.1371/journal.pone.0193346.
- 23.
Gouesnard B, Bataillon TM,去夫克G, Rozale C, Schoen DJ, David JL。Mstrat:一种通过最大化等位基因或表型丰富度来构建种质核心集的算法。J在这里。2001;92(1):93 - 4。
- 24.
Core hunter:一种基于多种遗传测度的遗传资源采样算法。BMC生物信息学。2009;10:243-55。https://doi.org/10.1186/1471-2105-10-243
- 25.
分子标记辅助植物育种的离散优化算法。根特大学理学院应用数学、计算机科学与统计学系博士论文。2018。
- 26.
伯格e,哈里克jl。丙酶基因座遗传多样性的定量。可以j for res。1997年; 27:415-24。
- 27.
堰BS。遗传数据分析2。桑德兰:西诺尔联合公司出版商;1996.
- 28.
相似和不同系数的遗传和数学性质在植物育种和种子库管理中的应用作物科学。2005;45:1-7。
- 29.
分层分组优化目标函数。J Am Stat Assoc. 1963; 58:236-44。
- 30.
R:统计计算的语言和环境。维也纳:统计计算R基金会;2018.URLhttps://www.r-project.org/
- 31.
种群的遗传结构。安尤金。1951;15:323-54。
- 32。
韦翰h .用户!Ggplot2,用于数据分析的优雅图形。纽约:施普林格-弗拉格;2016.
确认
N/A
资金
该工作得到了墨西哥政府的秘书园De Tryantura Y Desarrollo Rural(农业和农村发展部)在墨西哥政府(传统农业的可持续现代化)倡议中得到了支持。该资助者在研究和收集,分析和解释的设计中没有直接作用或写作稿件。
作者信息
隶属关系
贡献
SJH和JF-D提出了问题和方法;SJH和JC2生成并策划了原始基因型数据;JF-D进行了分析并写了第一选秀;JC1提供了关于方法和分析的咨询。所有作者审查并纠正了稿件。所有作者阅读并认可的终稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
陈志强,陈志强。et al。样本选择策略对种质库遗传多样性和代表性的影响。BMC植物杂志19,520(2019)。https://doi.org/10.1186/s12870-019-2142-y
收到了:
接受:
发表:
关键字
- 采样基因库的加入
- 遗传多样性代表性
- 样本大小
- 独立和嵌套的样本
- 单核苷酸多态性