摘要
背景
巴黎(Melanthiaceae)是一个经济上重要但分类上困难的属,在被子植物中是独一无二的,因为一些物种具有非常大的核基因组。系统发育关系巴黎一直以来都备受争议。基于完整的质体和核糖体DNA (nrDNA)序列,本研究旨在重建一个健全的系统发育树,并探索该属的历史生物地理和分支多样化。
结果
目前确认的29个物种巴黎被取样。全质体和nrDNA序列由基因组撇脂方法生成。利用最大似然和贝叶斯推理方法重建了系统发育关系。基于系统发育框架和分子定年,生物地理情景和历史多样性巴黎进行了探讨。质体和核数据集之间存在显著冲突,质体树与过去的形态学解释高度一致。祖先区域重建表明巴黎可能起源于东北亚和中国北方,在其多样化过程中经历了多次分散和分隔事件。中新世/上新世边界以来,演化支的多样化速度急剧加快。
结论
我们的结果为澄清一些长期存在的分类学争论提供了重要的见解巴黎.细胞核不一致可能是由该属中古代和最近的杂交引起的。晚中新世以来的气候和地质变化,如亚洲季风的增强和青藏高原的快速隆升,以及更新世时期的气候波动,在推动青藏高原活动范围扩大和辐射多样化方面发挥了重要作用巴黎.我们的发现挑战了大基因组可能限制物种形成的理论预测。
背景
巴黎是一个小属,曾经被归入延叶科[1],但现属墨兰科[2,3.,4,5,6].本属包括连续分布于欧亚大陆的林下多年生草本植物约29种[7,8,9].大多数种(24/29)分布在中国和喜马拉雅山,巴黎可能在亚热带东亚经历了显著的物种多样化(21°-34年°N) (7,9,10].本属的大多数品种由于其各种治疗特性而在中国和邻近国家是非常有价值的传统草药[11,12,13].的根状茎巴黎重楼var。对而且p .重楼var。云南裂(粉末Paridis)在中国已作为传统药物使用超过2000年[14].迄今为止,已经开发了40多种商业药物和保健品粉末Paridis作为原材料[15],年销售总额约15亿美元[16].此外,在越南、缅甸、尼泊尔、不丹和印度,几乎所有具有粗根状茎的品种都被收集用于药用目的[13,14].
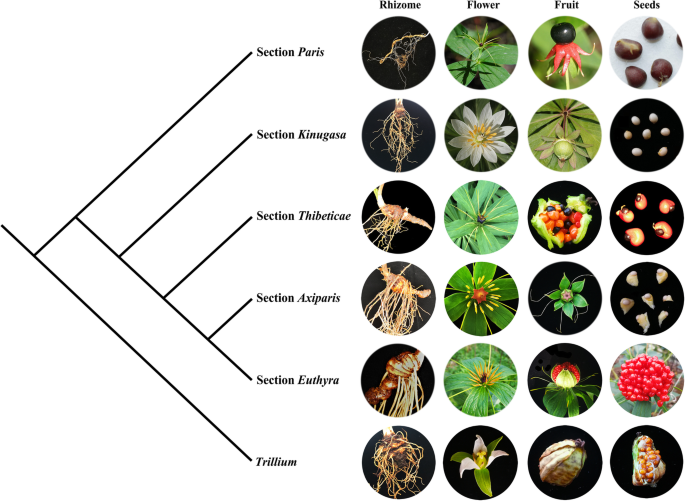
巴黎在形态上有独特的单轮叶(> 3)和单生的顶端花,4 - 15瓣。然而,根茎、叶、花、雄蕊、子房、果实和种子等已被广泛用于构建分类的植物,其种间差异较大[7,17].自Linneaus建立该属以来[18,这本书经过了多次关键性的修订。基于根茎和果实形态,19],他建立了第一个非fragenic分类系统巴黎将当时已知的物种分为两部分:Euthyra而且巴黎.Hara [17]描述了第三部分,Kinugasa.相反,Takhatajan [20.]认为这三个部分是属:巴黎s。.(=教派。巴黎),Daiswa(=教派。Euthyra),Kinugasa(=教派。Kinugasa).在最全面的修订中,李[7将该属分为两个亚属,Daiswa而且巴黎,和八个部分,Axiparis,Dunnianae,Euthyra,Fargesianae,Kinugasa,Marmoratae,巴黎,Thibeticae。基于分子和形态学证据,Ji等[21]提出了一种更新的李[7结合部分Dunnianae,Fargesianae而且Marmoratae与Euthyra.
最近的一些研究试图重建内部的系统发育关系巴黎基于单个或多个DNA位点[21,22,23,24].由于序列变化不足或分类单元采样有限,这些研究未能提供满意的分辨率或支持非异构关系。因此,缺乏一个可靠的系统发育计划阻碍了令人满意的解决长期以来对分类的分歧巴黎限制了我们对这个重要的经济属的进化和生物地理历史的理解。
基于有限序列区域的植物系统发育通常分辨率低,支持度低,特别是发生了快速多样化或杂交事件的进化支[25,26,27,28,29].最近,下一代测序技术,一种能够产生比Sanger测序更多数量级数据的技术,已越来越多地用于系统发育重建[30.,31,32,33,34,35].这为解决系统发育困难类群中的顽固关系提供了新的方法[36,37,38,39,40,41,42,43].黄等人。[44]和杨等人。[45]试图应用质体基因组(质体)来解决其中的系统发育关系帕里s.尽管质体数据极大地提高了系统发育的分辨率和支持度,但有限的分类单元采样阻止了他们对该属建立一个可靠的整体视图。因此,有必要扩大样本量,覆盖所有描述的部分,甚至所有物种,并使用具有不同遗传模式的标记来全面了解的进化史巴黎.
通过核糖体DNA序列(nrDNA)基因转换的双亲本遗传但通常为单亲本进化和非重组的、主要由母系遗传的质体包含大量进化信息变异,适合进行系统发育分析[46,47,48,49,50].通过相对低覆盖率的鸟枪测序总基因组DNA的基因组略读是一种恢复整个质体和nrDNA的有效方法[47].近年来,基因组略读已被广泛用于重建较低分类水平和近缘物种之间的进化关系[51,52,53,54,55],以及研究不同植物分支的网状进化[52,56,57,58].在这项研究中,我们从目前已知的所有质体和nrDNA序列中生成巴黎用基因组略读法研究物种。基于系统基因组学分析,我们的目标是:(1)阐明内部的进化关系巴黎;(2)探索该属的生物地理情景和历史多样性。
结果
Illumina测序和组装
低覆盖率基因组测序每个样本产生857 - 3573万对末端清洁reads (150 bp)(附加文件)1:表S1)。其中,7.25 × 104到2.10 × 106和4.81 × 103.到4.59 × 104分别映射到参考质体和核nrDNA。基于这些数据,我们组装了所有样本的完整质体和nrDNA,平均测序深度分别为68.72-1998.57次和75.15-1136.47次。
从头组装生产了33个巴黎质体,表现为典型的四方结构,大小在156,139-158,643 bp之间2:图S1)。巴黎质体在基因含量和排列上是保守的。所有质体包含114个基因,包括80个蛋白质编码基因,30个tRNA基因和4个质体rRNA基因(附加文件)3.:表S2)。对质体的比对产生了166,726个位点的矩阵,其中我们确定了5899个可变位点(3.53%)和3225个(1.93%)为吝啬信息位点(表1)1).此外,我们的新生nrDNA组装完全覆盖18S, ITS1, 5.8S, ITS2和26S区域。的序列长度巴黎nrDNA在5840 ~ 5859 bp之间。对nrDNA序列的比对产生了443个可变位点(7.56%),其中264个(4.50%)为简约信息位点(表2)1).
系统发育关系
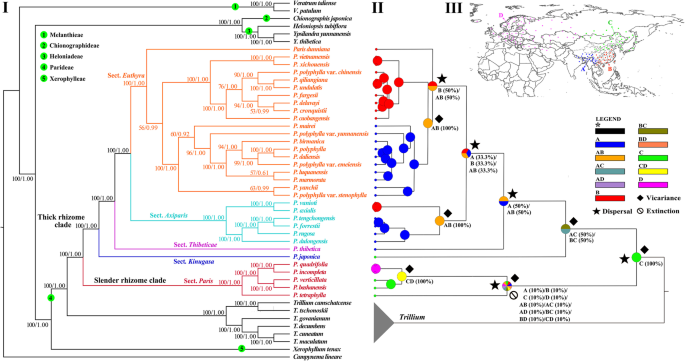
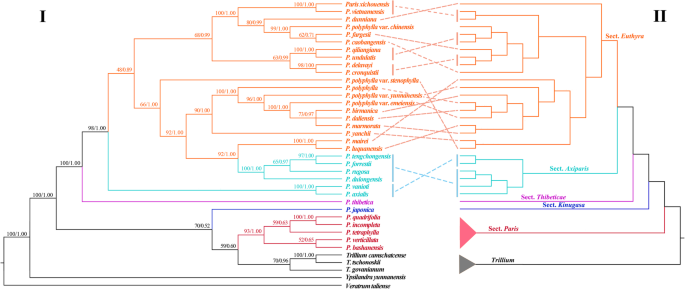
完整质体的标准最大似然(ML)和贝叶斯推理(BI)分析生成了相同的树拓扑(图2)。1).5个高度支持的支系(自举百分比,BP = 100;分析了Melanthiaceae的后验概率,PP = 1.00),与Zomlefer所识别的5个部落相对应[3.].他们之间的关系与先前的研究一致[3.,5,6,44,59].的单一性巴黎强烈支持(BP = 100, PP = 1.00),与延龄草(bp = 100, pp = 1.00)。在巴黎, 5个支撑良好的演化支,对应Ji等划定的5个剖面。[21被发现。我们的结果支持的连续分歧P。部分巴黎,Kinugasa,Thibeticae,Axiparis而且Euthyra.除少数末端种关系外,大多数亲缘关系获得了较高的支持度。例如,之间的关系p . luquanensis而且p . marmorataML和BI树分支支持度均较弱(BP = 57, PP = 0.67)。
不一致长度差(ILD)检验显示显著不一致(pnrDNA和质体数据集之间< 0.001)。nrDNA序列的系统发育分析(图;2)分裂的部落(巴黎而且延龄草)种分为2个演化支(BP = 100, PP = 1.00)。在第一枝内(BP = 70, PP = 0.50),剖面巴黎是姐妹延龄草(BP = 59, PP = 0.60),这一对是姐妹Kinugasa.在第二支(BP = 100, PP = 1.00)内,剖面Thibeticae是所有种类的节的姐妹吗Axiparis而且Euthyra,非单系。在比较细胞核和质体拓扑结构时,我们在不同的分类水平上观察到三个细胞核不一致的实例(图2)。2).第一个是非单性的巴黎在核数据集中。二是章节的单一性Axiparis而且Euthyra:它们在nrDNA树中是傍型的,而在质体树中是单系的。细胞核不一致的第三个例子涉及节内的种间关系Euthyra(无花果。2).
祖先区域重建,分子定年,历史多样化
统计-扩散距离分析(S-DIVA)1)重建了东北亚和中国北部(C)作为最近期共同祖先(MRCA)的祖先区巴黎.它可能经历了向西或向南扩散到中国西南部和喜马拉雅山脉或中国东部、中部、南部和印度支那北部[AC (0.50/BC(0.50)],从而进化出粗根状分枝的MRCA。然后,推导出分隔来分割截面Kinugasa(日本列岛)从剩余分类单元(节Thibeticae,Axiparis,Euthyra、东亚亚热带);在后者中,推断出三次分散和两次分隔事件。虽然S-DIVA分析未能重建剖面的祖先区域巴黎在演化枝中推断出一次分散、一次灭绝和两次地理分隔事件。
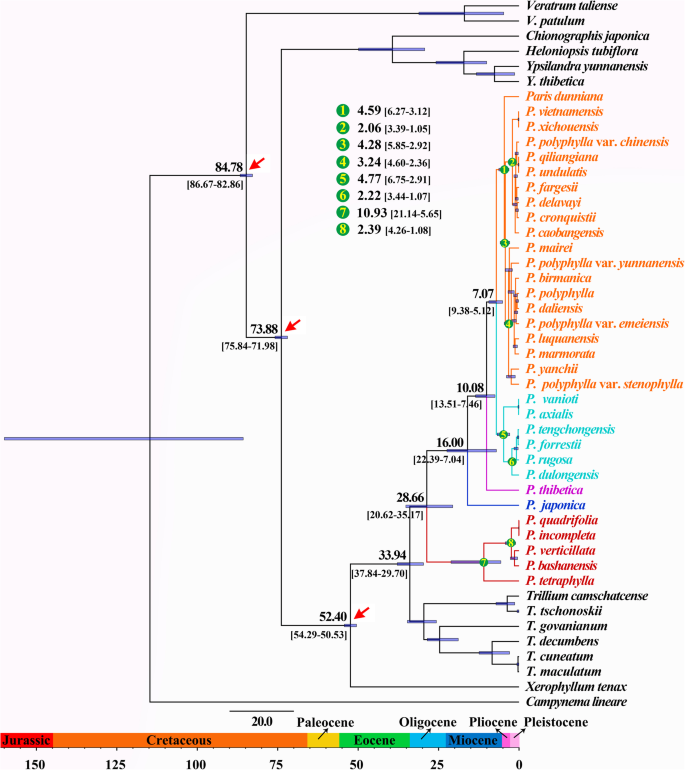
野兽分析(图。3.)表明姐妹属之间的分化,巴黎而且延龄草,发生在33.94 Mya (95% HPD: 37.84-29.70 Mya)。在巴黎在早渐新世前后的28.66 Mya (95% HPD: 35.17-20.62 Mya),粗而细的根状分枝与MRCA分道扬镳。大约中新世早期,粗根状枝的多样化开始于16.00 Mya (95% HPD: 22.39-7.04 Mya),导致单型剖面的分化Kinugasa从剩余的粗根茎类群中。随后,单株节Thibeticae偏离截面MRCAAxparis而且Euthyra10.08 Mya (95% HPD: 13.51-7.46 Mya),晚中新世。章节的分割Axparis而且Euthyra日期为7.07 Mya(95%火奴鲁鲁pd: 9.38-5.12 Mya),在中新世/上新世边界附近。此外,部分的多样化巴黎,Axiparis,Euthyra分别发生在10.93 Mya (95% HPD: 21.14-5.65 Mya)、4.77 Mya (95% HPD: 6.75-2.91 Mya)和4.59 Mya (95% HPD: 6.27-3.12 Mya)。
半对数沿袭时间(LTT)图分析(图。4的起源巴黎在中新世/上新世边界附近,生物多样性急剧增加。上新世和更新世保持了这种上升趋势。大进化混合物的贝叶斯分析(BAMM)发现,物种净多样化的速率变化巴黎,这是随着教派之间的分歧而发生的。Euthyra而且Axiparis(无花果。4).
讨论
系统发育推论和分类学含义
以往基于少量DNA位点或有限分类单元采样的系统发育分析未能可靠地重建其主干巴黎树(21,22,23,24,44,45].包括所有目前已确认的物种,质体分析完全解决种间的关系巴黎在大多数节点具有强大的支持。我们的研究进一步证实,基于更多的DNA位点的系统发育分析可以显著提高低分类学水平的分辨率[36,43,52,53,60].
以质体为基础的系统发育强烈支持的单系性巴黎并恢复了与Ji等人先前提出的剖面相对应的5个强烈支持的主要演化支。[21].在巴黎,沿截面树的主干连续发散巴黎,Kinugasa,Axiparis,Thibeticae,Axiparis,Euthyra是被推断出来的。一些形态学特征可以支持这种分化模式(图2)。5).简而言之,细长的根茎和圆形更像浆果的果实区分段巴黎从其他部分。然而,没有包膜的种子(或假种皮,大概是一个胚形的特征)分开的节巴黎而且Kinugasa从其他地方。虽然种粗根状分枝(包括节Kinugasa,Thibeticae,Axiparis,Euthyra)果实一般有棱角,节Kinugasa具有艳丽的白色萼片,是其中的独特之处。第三个分支部分Thibetica类似于Euthyra在有开裂的蒴果,但它的种子形态(与不完整的假种皮)是类似的Axiparis.因此,质体树与Ji等人基于形态学的分类高度一致。[21].
质体树为解决长期以来在分类上的分歧提供了有价值的见解巴黎.所有巴黎种间花和叶的形态突触形态均为4 - 15个延龄草(无花果。5),且两属的单系性均得到充分支持,使其有合理的认识巴黎作为单一属[7,17,21]而不是将其分为三个属(Daiswa,Kinugasa,巴黎。) [20.].新树进一步支持了Ji等人的分类学处理。[21结合部分Dunnianae,Fargesianae而且Marmoratae与Euthyra.鉴于其重要的经济价值,解决长期以来的分类学争议将有利于对其的探索和保护巴黎物种。
的当前分类学p .重楼质体和nrDNA结果均不支持。这些品种可能被认为是不同的物种,但考虑到这些品系观察到的细胞核不一致性,这也是有可能的。2杂交可能与它们的起源有关。用更合适的群体遗传和细胞学技术对这一群体进行进一步研究是有必要的。
Cytonuclear不整合
与Ji等人之前的研究相似[21],我们检测到nrDNA与质体树之间的不一致巴黎在深节和浅节。细胞核不一致是植物系统发育中相当普遍的现象[25,56,61,62,63,64,65,66,67,68].在大多数情况下,核树更符合形态分类学[43,56,61,62,64,67,68,69,70],而这种不一致主要是由于细胞质多态性的不完全分选或细胞质基因组通过杂交渗入到另一个物种的核背景[25,63,68,71,72].然而,在我们的研究中,质体树巴黎与形态学证据基本一致,提示无细胞质基因流的nrDNA渐渗可能是检测到的不一致性的原因[68,71,72,73,74].
考虑到在巴黎可能是由于影响nrDNA树的现象,其中包括一个亲本拷贝的快速基因转换[75,76,77,78,79,80],通过该数据集恢复的系统发育关系可能不能代表与不受基因转换影响的核基因组的其他部分获得的系统发育关系。尽管如此,这些结果可能为过去的杂交事件提供有用的信息,否则可能不是核基因组的大多数模式。恢复失败巴黎在nrDNA树中是单系的。2)表明在各部分之间可能存在杂交巴黎而且延龄草后部分巴黎从属的其余部分分裂出来的。值得注意的是,最大的真核生物基因组,那p .粳稻[81,82],被认为是一个异体八倍体之间巴黎而且延龄草根据先前的细胞学调查[83].这种混合假设是由它作为section姐妹的位置支持的巴黎而且延龄草在nrDNA树上。同样,分段的非单性Axiparis而且Euthyra在nrDNA树中观察到(图;2)也可以归因于古代的杂交。姐妹关系p . luquanensis/p . mairei(部分Euthyra)和四种剖面的演化枝Axiparis(p . dulongensis,p . forrestii,p .玫瑰而且p . tengchongensis)表明杂交可能发生在这些类群的祖先之间。此外,剖面的种间广泛不一致Euthyra(无花果。2)支持了前人的结论,即种间自然杂交Euthyra是可能的,如果传粉者是相同的,但很少有人知道这方面的生物学巴黎.大多数物种之间的人工异交实验是有效的[84].种间杂交是可能的原因,细胞核不一致之间观察到的物种在部分。此外,如前所述,各品种的位置也不一致p .重楼这表明杂交可能在它们的起源中也发挥了作用。
值得注意的是,在本研究中检测到的细胞核不一致性仅仅反映了质体和nrDNA数据集之间的冲突,nrDNA数据集实质上是质体和核糖体基因的两个连接群,充其量只能代表两个大的单位点DNA区域。在如此有限的数据集上,是否存在核全基因组和质体不一致是一个巨大的飞跃。为了进一步验证这一点,通过限制性位点相关DNA测序(RAD-seq)甚至全核基因组测序产生的大量未链接的核位点很可能是需要的。
生物地理学和谱系多样化
因为检测到强烈的细胞核不一致巴黎质体树与基于形态的分类很符合,我们解决了生物地理学和历史多样性的问题巴黎基于质体数据集。S-DIVA分析恢复了亚洲东北部和中国北部的祖先地区巴黎.与扩散和分隔事件相关(图。1的冠节点巴黎年代为28.66亿年(图;3.),在渐新世早期,当时全球气候恶化[85]导致欧亚大陆大部分地区适应干燥和寒冷气候的植被扩张[86].因此,早期的分歧巴黎可能是这些事件造成的。此外,S-DIVA分析显示日本特有种的分化p .粳稻而且p . tetraphylla由两个独立的地理分隔事件触发(图;1).它们的散度时间为16.00 Mya (p .粳稻)及10.93亿亚(p . tetraphylla)形成的中新世,对应着日本海的开放,将日本列岛与东亚大陆分开[87].
在粗根状枝中,S-DIVA分析推断了三次扩散事件(图2)。1),分别发生在10.08、7.07和4.59 Mya。3.),分别。新世气候变化可能在引发这些事件中发挥了重要作用。渐新世/中新世过渡时期亚洲季风的开始,在东亚低纬度地区和高纬度地区的森林之间建立了联系[88].中新世晚期以来,亚洲夏季风的增强,使东亚亚热带地区形成了湿润气候[89,90,91],并导致东亚森林的大面积扩张[88,92].这些气候和环境的变化将创造有利的栖息地,促进各剖面的扩散和分化Thibeticae,Axiparis而且Euthyra产于东亚亚热带地区。此外,随着东亚高纬度地区森林的扩张,MRCA也逐渐增加p . quadrifolia而且p . incompleta可能已经迁徙到欧洲了
LTT和BAMM分析均显示其内部存在枝型多样化巴黎在中新世/上新世边界附近突然加速,这可能是由于夏季季风气候的进一步加强,以及中新世晚期以来在亚热带东亚逐渐建立的两种不同的季风制度的开始[88,92,93].此后,中国东部、中部、南部和印度支那北部以太平洋季风为主,中国西南部和喜马拉雅地区则以印度季风为主[92,93,94,95].S-DIVA分析(图;1)和分子测年(图;3.)表明分隔事件在分段中独立发生Axiparis(4.77 Mya)和Euthyra(4.28 Mya)。这意味着由气候分化引起的深刻的生态异质性可能推动了两个地区显著的异域物种形成[96,97,98].此外,LTT和BAMM分析(图。4)揭示了最广泛的分歧巴黎在上新世和更新世时期,有超过一半的现存类群出现在这一时期。青藏高原在晚中新世(约10~8 Ma)至上新世早期(约3.6 Ma)急剧上升[99,One hundred.],从而极大地改变了全球气候[94,101]从而深刻地影响了东亚的生物过程,如物种范围的扩张/收缩和地理分隔[102].更新世时期,东亚地区至少有四次主要冰川期[103],这些可能在东亚创造了显著的隔离和多样化的栖息地[104,105].如此复杂的地质、生态和环境异质性,预计推动了广泛范围的植物分支的多样化[104,106,107,108,109也会引发地理隔离,并促进物种的辐射巴黎.
在植物中,属级多样性与属平均基因组大小呈负相关[110,111].奈特等人。[111]提出了大基因组约束假说,该假说认为具有大基因组的植物类群的多样性更慢。随后,Suda等人。[112]的研究发现,许多经历适应性辐射的马卡罗尼西亚被子植物的岛屿支的基因组大小较小,并假设快速多样化更可能发生在基因组大小较小的被子植物中。值得注意的是巴黎在被子植物中具有较大的基因组。该属中记录的最小基因组大小(p . verticillata, 1C = 30.52 Gb)远大于被子植物的平均基因组大小(1C = 5.7 Gb) [113,114].此外,已知最大的真核生物基因组p .粳稻, 1 C = 148.88 Gb,属于巴黎[81,82].在这项研究中,我们发现巴黎可能经历了中新世/上新世边界以来的物种辐射(图。4),这与大基因组大小会限制物种形成的预测并不一致[111,112].还必须承认,虽然谱系多样化急剧增加,但与其他辐射相比,所涉及的物种总数并不多,例如,石竹类植物在地中海[115),而Aizoaceae在南非[116].大基因组约束假说的普遍性需要进一步评估,尽管在本研究中发现了谱系多样化的增加巴黎并不会与之产生重大矛盾。
结论
本研究是一项全面的系统发育研究巴黎这是一个在经济上很重要但在分类学上很困难的属。完整质体和nrDNA序列的分析重建了一个可靠的系统发育,并为澄清一些长期存在的分类学争论提供了启示巴黎.我们还发现了质体数据集和核数据集之间的显著冲突。中观察到的细胞核不一致巴黎可能是古代和近代杂交造成的。祖先区域重建表明巴黎可能起源于东北亚和中国北方,在其多样化过程中经历了多次分散和分隔事件。基于系统发育框架和分子定年,我们认为中新世以来的气候和地质变化在触发陆域形成和演化枝多样化方面发挥了重要作用巴黎.我们的发现为阐明动物的进化史提供了重要的见解巴黎,并将有利于勘探和保护巴黎物种。
方法
植物取样,DNA提取和Illumina测序
我们对33个品种进行了采样,以代表李氏所识别的全部29个物种和5个变种。7]及其后所描述的[9,84,117,118,119].本研究中使用的植物材料的原始来源和凭证信息在附加文件中提供4:表S3。凭证标本由纪云衡博士鉴定。采用CTAB(十六烷基三甲基溴化铵)方法从约20 mg硅胶干叶中提取基因组DNA [120].超声切取约5 μg纯化基因组DNA。使用TruSeq DNA Sample Prep Kit (Illumina, Inc., USA)根据制造商的协议制备平均插入尺寸为350 bp的配对端文库。这些文库在Illumina HiSeq 2000平台上进行配对端测序。使用NGS QC Toolkit过滤原始读取以删除适配器和低质量读取[121],将读取长度百分比的分界值设置为80,PHRED质量分数设置为30。
组装和基因注释
的完整质体序列巴黎quadrifolia(GenBank Accession: KM067394)作为组装新测序的参考巴黎质体系。在Bowtie v2.2.6中,质体contigs根据文献进行组织,通过重叠的末端序列连接得到完整的质体[122]使用默认参数。质体使用Dual Organellar Genome Annotator数据库进行注释[123].手动检查蛋白质编码基因的起始密码子和终止密码子以及内含子/外显子边界。tRNAscan-SE 1.21进一步验证了注释的tRNA基因[124],使用默认参数。视觉检查每个质体的大单拷贝(LSC)、小单拷贝(SSC)和反重复(IR)区域的边界,并根据geneous V10.2中的参考质体的边界手动调整[125].
对于nrDNA序列组装,我们首先排除了所有质体样reads。基于剩余的reads,使用完整的nrDNA序列(包括26S, 18S和5.8S核糖体RNA基因和内部转录间隔子)进行从头组装Lillium tsingtauense(GenBank登录号:KM117263)作为参考。外部转录间隔巴黎物种拥有太多的重复序列和反转,这可能会使组装不准确,因此我们没有组装该区域。使用上述过程组装映射到参考nrDNA的Contigs。核核糖体RNA基因及其与ITS区域的边界通过与geneous V10.2中的参考文献的比较进行注释和定义[125].
系统发育分析
研究的系统发育位置巴黎在紫兰科中,有15个其他的质体,代表了在紫兰科中公认的五个部落(附加文件)5:表S4)与新测序的33例进行整合巴黎归根结底是质体。此外,33巴黎nrDNA和五个rDNA序列Veratrum加入(1),Ypsilandra(1加入)和延龄草将Melanthiaceae(3个种质)纳入一个核数据集(附加文件)4:表S3)。Campynema线性而且Veratrum taliense根据前人研究分别对质体树和核树进行生根[3.,5,6,59].利用MAFFT对质体和nrDNA序列进行比对[126]集成在genous v.10.2中[125,并在必要时手动编辑。使用Modeltest v3.7选择最合适的质体序列替换模型(GTR + G)和nrDNA序列替换模型(GTR + I + G) [127]用赤池信息准则[128].我们认为整个质体是一个单一的遗传单位。接下来,我们使用PartitionFinder v. 2.1.1对小拷贝区域和大拷贝区域以及反向重复确认了相同的模型[129].质体和核数据集之间的冲突使用不一致长度差(ILD)检验[130在PAUP* 4.0b10中实现[131]进行1000个重复。
采用ML和BI方法进行系统发育分析。使用RAxML-HPC BlackBox v8.1.24进行ML分析[132]进行1000次快速自举。BI分析采用MRBAYES v.3.1.2进行[133].每个数据集的运行都从一个随机的起始树开始,每100代进行一次采样。最初25%的样本树木被丢弃。后验概率值由剩余的树确定。当分裂频率的平均标准差< 0.01时,认为达到平稳。
分子定年和多样化率估计
值得注意的是,没有化石被确定为黑桫椤科及其近亲。此前一项对被子植物单子叶和主要分支的17个化石的研究表明,Melanthiaceae的花冠年龄约为84.8 Mya,而分支Parideae-Xerophyllideae和Chionographideae-Heloniadeae的花冠年龄约为74 Mya,部落Parideae和Xerophyllideae的花冠年龄约为52.3 Mya [134].我们利用这些时间来校准系统发育树(图。3.).在BEAST v.2.4.7中进行分子测年[135].BEAST分析在不相关对数正态放松时钟方法下运行,具有圣诞树先验。马尔可夫链蒙特卡洛链运行10,000,000次,每1000代采样一次。用Tracer v. 1.5监测链的平稳性和BEAST分析的收敛性。
多样化率随时间的变化采用半对数沿袭时间(LTT)图方法。通过APE v.5.3计算从分子定年结果推断出的一致年表[136]在R环境内[137].我们进一步研究了年净多元化率的潜在变化巴黎基于时间校准的最大分支可信度树(具有最高后验概率),使用宏观进化混合物贝叶斯分析(BAMM) [138].
祖传地区重建
对于生物地理重建,巴黎根据目前的分布情况,将其划分为4个地区:A)中国西南部和喜马拉雅山脉;B)中国东部、中部、南部和印度支那北部;C)东北亚和中国北部;D)欧洲和高加索地区。祖先分布巴黎用统计色散-距离分析(S-DIVA)重建[139]在rasp4.0中实现[140].从BI分析中得到的浓缩树和4000棵老化后的贝叶斯树被用作输入树。随机树被定义为1000,其他参数被设置为默认值。
数据和材料的可用性
本研究生成的序列可在GenBank上获得(登录号见表S3)。手动调整的序列对齐被保存在Treebase (http://purl.org/phylo/treebase/phylows/study/TB2:S25166).
缩写
- BI:
-
贝叶斯推理
- 英国石油公司:
-
碱基对
- 英国石油公司:
-
引导百分比
- BS:
-
引导
- CTAB:
-
十六烷基三甲基溴化铵
- 背景:
-
脱氧核糖核酸
- HPD:
-
最高后验密度
- ILD:
-
不一致长度差
- 红外光谱:
-
反向重复
- 其:
-
核糖体DNA内部转录间隔
- 姜昆:
-
中国科学院昆明植物研究所植物标本室
- LSC:
-
大单副本
- 图片:
-
时间的半对数谱系
- 密度:
-
马尔科夫链蒙特卡洛
- ML:
-
最大似然
- MRCA:
-
最近的共同祖先
- 米娅:
-
几百万年前
- nrDNA:
-
核糖体DNA序列
- 页:
-
后验概率
- QTP:
-
青藏高原
- 核糖体rna:
-
核糖体核糖核酸
- S-DIVA:
-
统计-扩散地理差异分析
- SSC:
-
小单份
- tRNA:
-
转移核糖核酸
- 美元:
-
美元
参考文献
林德利,蔬菜王国。伦敦:布拉德利和埃文斯;1846.
被子植物系统发育类群。开花植物科的有序分类。安。密苏里。波特。加德。1998;85:531-53。https://doi.org/10.2307/2992015.
Zomlefer WB, Williams NH, Whitten WM, Judd WS。百合科(百合科,百合科)的属界和关系Zigadenus:来自ITS和trnL-F序列数据。中国科学(d辑),2001;https://doi.org/10.2307/3558411.
佐勒夫WB,贾德WS,惠顿WM,威廉姆斯NH。文章标题百合科植物简介,重点讨论了百合科部落的性状进化。在:哥伦布JT,修士EA,波特JM,王子LM,辛普森MG,编辑。单子叶:比较生物学和进化(不包括极子)。加利福尼亚州:圣安娜牧场植物园;2006.p . 564 - 76。
金俊杰,洪俊杰,蔡伟,范文明,金俊华。基于4个质体位点的百合单子叶目家族关系分析:matK, rbcL, atpB而且atpF-H.机械工程学报,2013;https://doi.org/10.1111/boj.12039.
金志刚,李志刚,李志刚,李志刚。百合科植物叶绿体DNA序列分析。机械工程学报,2016;https://doi.org/10.1111/boj.12405.
李H.属巴黎(Trilliaceae)。北京:科学出版社;1998.
季勇,周泽,李华。属中四个新同义词巴黎(Trilliaceae)。植物分类学报,2007;45:388-90。https://doi.org/10.1360/aps06163.
杨健,王永华,李辉。巴黎qiliangiana(桔梗科),湖北一新种。中国植物学报。2017;329:193-6。
梁思智,Soukup VG。巴黎L. In: Wu ZY, Raven PH,编辑。中国植物志。北京:科学出版社、密苏里植物园出版社;2000.p . 88 - 95。
龙春林,李华,欧阳志,杨霞,李强,Trangmar B.农业生物多样性保护与促进对策——以云南为例。中国生物多样性研究。2003;12:1145-56。https://doi.org/10.1023/a:1023085922265.
何俊,张松,王辉,陈彩霞,陈顺丰。的研究和应用进展巴黎重楼var。云南裂(Trilliaceae)。云南学报,2006;28:271-6。
Cunningham AB, Brinckmann JA,毕玉峰,裴世杰,Schippmann U,罗鹏。巴黎春季:回顾从野生收获到种植的贸易、保护和机会巴黎重楼(Trilliaceae)。中华民族药典杂志,2018;222:208-16。https://doi.org/10.1016/j.jep.2018.04.048.
李华,苏波,杨勇,张震。我国罕见医疗现状评价巴黎以探索其种植的未来发展。科学通报。2015;44:1-6。
王艳红,牛红梅,张志智,胡晓霞,李慧巴黎.中华医学杂志2015;40:833-9。
黄丽强,肖鹏鹏,王友友。中国珍稀濒危药用植物资源调查。第1版。上海:科学技术出版社;2012.
Hara H.变异巴黎重楼史密斯,关于其他亚洲物种。南京工业大学学报(自然科学版)。1969;10:141-80。
林奈植物属。斯德哥尔摩:Salvius;1753.
弗朗谢·A.专著巴黎.《历史与社会》,1888;24:267-91。
Takhtajan A.对Daiswa(Trilliaceae)。Brittonia。1983;35:255 - 70。
季勇,Fritsch PW,李华,肖廷杰,周志科。的系统发育和分类巴黎从DNA序列数据推断。Ann Bot. 2006; 98:245-56。https://doi.org/10.1046/j.1365-2885.2002.00426.x.
加藤,田内荣,江野。延叶草科植物的分子系统学研究理智lato根据推断:序列数据。分子系统进化学报,1995;4:184-93。https://doi.org/10.1006/mpev.1995.1018.
张志刚,张志刚。延叶蕨科植物分子系统学研究。的系统发育分析延龄草和它的盟友使用序列:而且matKcpDNA基因和18S-26S nrDNA内部转录间隔子。植物学报,1999;14(2):366 - 366。https://doi.org/10.1046/j.1442-1984.1999.00009.x.
Farmer SB, Schilling EE。基于形态和分子资料的延铃科植物系统发育分析。系统工程学报。2002;27:674-92。https://doi.org/10.2307/3093915.
植物细胞质基因流动的系统发育结果。进化趋势植物。1991;5:64-84。https://doi.org/10.1007/BF00021248.
Rokas A, Carroll SB,更多基因还是更多类群?基因数和分类单元数对系统发育准确性的相对贡献。中国生物医学杂志2005;22:1337-44。https://doi.org/10.1093/molbev/msi121.
Whitfield JB, Lockhart PJ。破译古代快速辐射。生态环境学报。2007;22:258-65。https://doi.org/10.1016/j.tree.2007.01.012.
Philippe H, Brinkmann H, Lavrov DV, Littlewood DTJ, Manuel M, Wörheide G,等。解决复杂的系统发育问题:为什么更多的序列还不够。公共科学图书馆。2011;9:e1000602。https://doi.org/10.1371/journal.pbio.1000602.
孙敏,Soltis DE, Soltis PS,朱欣,Burleigh JG,陈哲。被子植物蔷薇科分支深度系统发育不一致。生物信息学杂志,2015;https://doi.org/10.1016/j.ympev.2014.11.003.
Jansen RK, Cai Z, Raubeson LA, Daniell H, de Pamphilis CW, Leebens-Mack J,等。分析了64个质体基因组中的81个基因,揭示了被子植物基因组尺度的进化模式。《美国国家科学院学报》上。2007; 104:19369 - 74。https://doi.org/10.1073/pnas.0709121104.
Moore MJ, Bell CD, Soltis PS, Soltis DE.利用质体基因组尺度数据来解决基底被子植物之间的神秘关系。自然科学进展,2007;26(3):366 - 366。https://doi.org/10.1073/pnas.0708072104.
Moore MJ, Soltis PS, Bell CD, Burleigh JG, Soltis DE. 83个质体基因的系统发育分析进一步揭示了球果的早期多样化。自然科学进展(英文版),2010;https://doi.org/10.1073/pnas.0907801107.
Philippe H, Derelle R, Lopez P, Pick K, Borchiellini C, Boury-Esnault N,等。系统基因组学复兴了关于动物深层关系的传统观点。生物学报,2009;19:706-12。https://doi.org/10.1016/j.cub.2009.02.052.
周旭,徐松,徐娟,陈波,周凯,杨刚。系统基因组分析揭示了laurasiasian哺乳动物的序间关系和快速多样化。生物工程学报。2012;61:150-64https://doi.org/10.2307/41515183.
麦凯,杨阳。植物系统基因组学的研究进展。植物科学通报2018;6:e1028。https://doi.org/10.1002/aps3.1038.
李志强,李志强,李志强,等。利用叶绿体基因组大规模并行测序提高系统发育分辨率的研究。BMC生物学,2009;7:84。https://doi.org/10.1186/1741-7007-7-84.
巴雷特CF,斯佩克特CD,利本-麦克J,史蒂文森DW,佐姆勒弗WB,戴维斯JI。解析古代辐射:完整的质体基因集能否阐明热带姜(Zingiberales)之间的深层关系?Ann Bot. 2014; 113:119-33。https://doi.org/10.1093/aob/mct264.
马普芬,张艳霞,曾彩霞,郭志华,李德泽。叶绿体系统基因组分析解决了一个棘手竹族的深层关系Arundinarieae(禾本科)。《生物工程学报》2014;63:933-50。https://doi.org/10.1093/sysbio/syu054.
王志刚,王志刚,王志刚,王志刚,王志刚,王志刚,王志刚,等。应用质体尺度数据集研究荔枝科植物的基层状系统发育。中国科学(d辑),2015;https://doi.org/10.3732/ajb.1500298.
Attigala L, Wysocki WP,杜瓦尔MR, Clark LG。植物的系统发育估计和形态进化Arundinarieae(竹科:竹科)基于质体系统基因组分析。分子系统进化学报,2016;https://doi.org/10.1016/j.ympev.2016.05.008.
张士德,金俊杰,陈淑云,蔡文伟,Soltis DE,李海涛,等。基于质体系统基因组学的晚白垩世以来蔷薇科植物的多样化。新植物,2017;214:1355-67。https://doi.org/10.1111/nph.14461.
Carlsen MM, Fér T, Schmickl R, Leong-Škorničková J, Newman M, Kress WJ。解决热带Zingiberales早期分化谱系的快速植物辐射:推动基因组数据的极限。分子进化学报,2018;https://doi.org/10.1016/j.ympev.2018.07.020.
林海燕,郝玉军,李建华,付彩霞,Soltis PS, Soltis DE,等。由物种多样化后的远古渐渗引起的系统基因组冲突Stewartia s.l。(山茶科)。分子系统进化学报,2019;https://doi.org/10.1016/j.ympev.2019.02.018.
黄燕,李霞,杨忠,杨超,杨军,季勇。全叶绿体基因组序列分析提高了植物的系统发育分辨率巴黎(Melanthiaceae)。前沿植物学报2016;7:1797。https://doi.org/10.3389/fpls.2016.01797.
杨林,杨忠,刘超,何忠,张忠,杨洁,等。叶绿体系统基因组学分析为最大的真核生物基因组持有者的进化提供了见解,巴黎粳稻(Melanthiaceae)。BMC植物生物学2019;19:293。https://doi.org/10.1186/s12870-019-1879-7.
Linder CR, Goertzen LR, Heuvel BV, Francisco-Ortega J, Jansen RK。18S-26S rDNA完整的外部转录间隔:在菊科和紧密亲缘科中低分类水平的扩增和系统发育用途。分子系统进化学报,2000;https://doi.org/10.1006/mpev.1999.0706.
Straub SC, Parks M, Weitemier K, Fishbein M, Cronn RC, Liston A.导航基因组冰山一角:植物分类学的下一代测序。中国科学(d辑),2012;https://doi.org/10.3732/ajb.1100335.
Bock DG, Kane NC, Ebert DP, Rieseberg LH。基因组略读揭示了耶路撒冷洋蓟块茎作物物种的起源:既不是来自耶路撒冷,也不是洋蓟。新植物,2014;201:1021-30。https://doi.org/10.1111/nph.12560.
Hollingsworth PM, Li DZ, Michelle VDB, Twyford AD。用DNA区分植物物种:从条形码到基因组。Philos Trans R Soc B. 2016;371:20150338。https://doi.org/10.1098/rstb.2015.0338.
曾彩霞,Hollingsworth PM,杨健,何志生,张志荣,李德泽,等。用于DNA条形码和系统基因组学的基因组略读标本室标本。植物学报。2018;14:43。https://doi.org/10.1186/s13007-018-0300-0.
Simpson MG, Guilliams CM, hasenstabb - lehman KE, Mabry ME, Ripma L.爆米花花的系统发育:利用基因组略读评价亚部落的单系和相互关系Amsinckiinae(紫草科)。分类单元。2017;66:1406-20。https://doi.org/10.12705/666.8.
瓦格斯,欧提兹,辛普森,BB。相互冲突的系统基因组信号揭示了最近高安第斯多样化的网状进化模式(Asteraceae: Astereae):Diplostephium).新植物,2017;214:1736-50。https://doi.org/10.1111/nph.14530.
乌里韦-康弗斯S, Carlsen MM, Lagomarsino LP, Muchhala N.的系统发育关系Burmeistera(桔梗科:半边莲科):在最近的一次辐射中结合整个质体和靶向位点数据。分子进化学报,2017;https://doi.org/10.1016/j.ympev.2016.12.011.
Fonseca LHM, Lúcia GL.结合高通量测序和靶向位点数据推断“Adenocalymma-Neojobertia支系(龙舌兰科,龙舌兰科)。分子系统进化学报,2018;https://doi.org/10.1016/j.ympev.2018.01.023.
hekenhauer J, Paun O, Chase MW, Ashton PS, Kamariah AS, Samuel R.部落分子系统基因组学研究Shoreeae(龙心科)使用整个质体基因组。Ann Bot. 2019; 20:1-9。https://doi.org/10.1093/aob/mcy220.
Bruun-Lund S, Clement WL, Kjellberg F, Rønsted N.首次质体系统基因组学研究揭示了进化历史中潜在的细胞-核不一致性热带榕属植物l .(桑科)。生物信息学杂志,2017;https://doi.org/10.1016/j.ympev.2016.12.031.
马吉赞纳,杨杨,魏基特,麦凯恩,波利尤JM。探索植物生命树的方法。植物科学通报2018;6:e1309。https://doi.org/10.1002/aps3.1039.
刘硕,Edwards CE, Hoch PC, Raven PH, Barber JC。基因组略读提供了新的见解的关系Ludwigia部分Macrocarpon,多倍体复合体。Am J Bot. 2018; 105:875-87。https://doi.org/10.1002/ajb2.1086.
金C,金SC,金JH。黑桫椤科的历史生物地理:通过白令海陆桥进入北美的一个案例。中国植物科学,2019;10:396。https://doi.org/10.3389/fpls.2019.00396.
马志智,文杰,Ickert-Bond SM,聂志林,陈丽琪,刘小强。葡萄的系统基因组学、生物地理学与适应性辐射。分子进化学报,2018;https://doi.org/10.1016/j.ympev.2018.08.021.
索提斯,德,库佐夫,k。植物核和叶绿体系统发育的不一致Heuchera组(石菖蒲科):叶绿体捕获和过敏性的证据。进化。1995;49:727-42。https://doi.org/10.2307/2410326.
杨晓明,李志强,李志强,等。植物ITS与叶绿体拓扑结构的不一致性Boykinia组(Saxifragaceae)。系统工程,1996;21:169-76。https://doi.org/10.2307/2419746.
王志强,王志强,王志强。植物系统基因组学研究。科学通报。2005;36:541-62。https://doi.org/10.1146/annurev.ecolsys.35.112202.130205.
张文杰,张文杰,张文杰,等。基因组系统发育不一致的研究进展。入职:Soltis D, Soltis PJD,编辑。植物分子系统学II DNA测序。纽约:克鲁威学术出版社;1998.p . 265 - 96。
恩斯JJ。结合具有不同系统发育历史的数据集。中国生物医学工程学报。1998;47:568-81。https://doi.org/10.1080/106351598260581.
快速DL, Jones OR, Epstein DR.纠正不一致长度差检验中由于噪声不平衡而导致的错误不一致问题。生物工程学报。2007;56:496-503https://doi.org/10.1080/10635150701429974.
项小刚,Schuiteman A,李德泽,黄卫文,钟文文,李建文,等。分子系统学石斛兰(兰科,石斛科)产于亚洲大陆,基于质体和核序列。生物信息学杂志,2013;https://doi.org/10.1016/j.ympev.2013.06.009.
Folk RA, Mandel JR, Freudenstein JV。在被子植物谱系中,祖先基因流动和平行细胞器基因组捕获导致了极端的系统基因组不和谐。系统生物学,2017;66:320-37。https://doi.org/10.1093/sysbio/syw083.
文德尔JF,道尔JJ。系统发育不一致:基因组历史和物种形成的窗口。入职:Soltis PS, Soltis DE, Doyle JJ,编辑。植物分子系统学。纽约:查普曼和霍尔;1998.p . 265 - 96。
李志刚,李志刚,李志刚,李志刚。植物发育过程中细胞与核的不一致热带榕属植物部分Galoglychia植物和传粉者之间的联系也发生了变化。中国生物医学工程学报,2009;https://doi.org/10.1186/1471-2148-9-248.
Rieseberg LH, Wendel JF。植物的渗透及其后果。入:哈里森RG,编辑。混合区和进化过程。纽约:牛津大学出版社;1993.p . 70 - 114。
杂交和适应性辐射。生态环境学报。2004;19:198-207。https://doi.org/10.1016/j.tree.2004.01.003.
Arnold ML, Buckner CM, Robinson JJ。花粉介导的路易斯安那鸢尾花渗透和杂交物种形成。中国自然科学学报(英文版),2001;https://doi.org/10.2307/2356002.
Setoguchi H, Watanabe I.岛种间的交叉基因流冬青属植物在小笠原群岛和琉球群岛。中国科学(d辑),2000;https://doi.org/10.2307/2656887.
希利斯DM,莫里茨C,波特CA,贝克RJ。核糖体DNA协同进化中偏倚基因转换的证据。科学。1991;251:208 - 310。https://doi.org/10.1126/science.1987647.
李文杰,李文杰,李文杰。一种不同寻常的核糖体dna序列Gossypium gossypioides揭示了古老的,神秘的基因组间渗入。分子系统进化学报,1995a;4: 398 - 313。
李文杰,李文杰,李文杰,等。棉花异源多倍体物种形成的双向基因间协同进化(Gossypium).中国科学院学报(自然科学版),1995b;92: 281 - 42。
李志强,李志强,李志强,等。烟草核糖体DNA的基因转换与低甲基化、去致密和可能活跃的基因单位有关。染色体。2000;109:161 - 72。https://doi.org/10.1007/s004120050424.
柯伟华,张志强,张志强,张志强,等。核糖体DNA的快速协同进化Tragopogon同种异体多倍体最近和复发起源。遗传学。2005;169:931-44。https://doi.org/10.1534/genetics.104.032839.
李丽娟,李丽娟,李丽娟。协同进化能快速消除rDNA编码区序列变异,但不能消除基因间间隔区序列变异烟草异源四倍体。植物科学进展。2017;https://doi.org/10.1007/s00606-017-1442-7.
佩利塞J,费伊MF,雷奇IJ。最大的真核生物基因组?中国机械工程学报,2010;43(4):344 - 344。https://doi.org/10.1111/j.1095-8339.2010.01072.x.
Dodsworth S Leitch AR Leitch IJ。被子植物基因组大小多样性及其对基因空间的影响。Curr Opin Genet Dev. 2015; 35:73-8。https://doi.org/10.1016/j.gde.2015.10.006.
染色体补体Kinugasa粳稻特别提到了它的起源和习性。Cytologia。1938;8:137-41。https://doi.org/10.1508/cytologia.8.137.
季勇,李华,周志科。巴黎caobangensis纪永华,李海华,周泽科(延铃科),越南北部一新种。植物分类学报。2006;44:700-3。https://doi.org/10.1360/aps050112.
Zachos J, Pagani M, Sloan L, Thomas E, Billups K. 65 Ma至今的全球气候变化趋势、节奏和异常。科学。2001;292:686 - 93。https://doi.org/10.1126/science.1059412.
歌我。西欧和中欧始新世/渐新世界线周围的植被和植物区系变化。在:Prothero DR, Berggren WA,编辑。始新世-渐新世气候与生物演化。普林斯顿:普林斯顿大学出版社;1992.437 - 50页。
超大陆的历史及其与日本列岛起源的关系。中国地质大学学报(自然科学版)。https://doi.org/10.5026/jgeography.120.100.
孙晓军,王鹏飞。亚洲季风系统有多久的历史了?中国古植物学记录。中国地质大学学报(自然科学版)2005;https://doi.org/10.1016/j.palaeo.2005.03.005.
万思敏,李ac,克里夫特PD,斯图尔特JBW。东亚季风的发展:南海北部20 Ma以来的矿物学和沉积学记录。古地理古气候古生态。2007;254:561-82。https://doi.org/10.1016/j.palaeo.2007.07.009.
Jacques FMB,郭世祥,苏涛,邢玉文,黄玉军,刘玉生,等。中国西南晚中新世季风气候的定量重建——以云南临沧区系为例。古地理古气候古生态。2011;304:318-27。https://doi.org/10.1016/j.palaeo.2010.04.014.
张qq, Ferguson DK, Mosbrugger V,王玉峰,李春春。青藏高原隆升对中国西南地区植被和气候变化的响应。中国地质大学学报(自然科学版)。https://doi.org/10.1016/j.palaeo.2012.08.009.
姚玉峰,Bruch AA, Mosbrugger V,李春春。基于植物化石的中国南方中新世气候格局及其演化定量重建中国地质大学学报(自然科学版)。https://doi.org/10.1016/j.palaeo.2010.04.012.
李凡杰,卢梭达明,吴nq,郝庆忠,裴玉平。中国西部黄土高原陆生软体动物记录揭示的晚新近纪东亚季风演化:由冬季到夏季为主的亚体系。地球行星科学,2008;274:439-47。https://doi.org/10.1016/j.epsl.2008.07.038.
An ZS, Kutzbach JE, Prell WL, Porter SC.晚中新世以来亚洲季风的演变与喜马拉雅-青藏高原的阶段性隆升。大自然。2001;411:62-6。https://doi.org/10.1038/35075035.
姚涛,Thompson L,杨伟,于伟,高原,郭晓军,等。青藏高原及周边地区不同冰川状况与大气环流的关系。张国昌。2012;2:663-7。https://doi.org/10.1038/nclimate1580.
李小文,李娟。田中-开永系——东亚植物区系研究的重要区系。安莫·波特·加德。1997;84:888-92。https://doi.org/10.2307/2992033.
王玲,施耐德H,张晓春,项泉平。喜马拉雅山脉的崛起通过改变季风体制,迫使东南亚蕨类植物多样化。BMC植物生物学2012;12:210。https://doi.org/10.1186/1471-2229-12-210.
陆敏,毛丽芬,杨涛,叶剑峰,刘波,李海林,等。标题中国被子植物区系的进化史。大自然。2018;554:234-8。https://doi.org/10.1038/nature25485.
刘志刚,刘志刚,刘志刚。东亚温带植物的多样性研究。大自然。2001;413:129-30。https://doi.org/10.1038/35093166.
Royden LH, Burchfiel BC, Van DHRD。青藏高原的地质演化。科学。2008;321:1054-8。https://doi.org/10.1126/science.1155371.
史玉峰,崔志军,苏震。中国第四纪冰川与环境变化。石家庄市:河北科学技术出版社;2005.
李俊杰,方晓明。青藏高原隆升与环境变化。科学通报。1999;44:2117-24。https://doi.org/10.1007/BF03182692.
施YF。第四纪中期青藏高原冰冻圈演化及其与全球变化的关系冰川冻土,1998;20:197-208。
艾克斯罗德·迪,阿尔·谢巴兹一世,雷文·PH.中国现代植物区系史。入:张爱玲,吴光生,编辑。东亚植物区系特征及多样性。北京:中国高等教育出版社;1998.43-55页。
邱YX,傅CX,惠普来了。中国及邻近地区植物分子系统地理学:追踪世界上最多样化的温带植物群第四纪气候和环境变化的遗传印记。分子系统进化学报,2011;https://doi.org/10.1016/j.ympev.2011.01.012.
钱海燕,李志强,李志强,等。温带植物物种多样性的大规模过程与亚洲偏向。大自然。2000;407:180-2。https://doi.org/10.1038/35025052.
吴志智,孙华,周志凯,彭华,李德智。中国特有植物区系的起源与分化。云南学报,2005;27:57 - 604。https://doi.org/10.1007/s11515-007-0020-8.
文健,张建强,聂志林,钟勇,孙慧。青藏高原植物的进化多样性。Front Genet. 2014;5:4。https://doi.org/10.3389/fgene.2014.00004.
法夫尔A, Päckert M, Pauls SU, Jähnig SC, Uhl D, Michalak I,等。青藏高原隆起对西藏生物进化的作用。中国生物医学杂志,2015;https://doi.org/10.1111/brv.12107.
Vinogradov AE。自私的DNA是不适应的:来自植物红色名录的证据。趋势,2003;19:609-14。https://doi.org/10.1016/j.tig.2003.09.010.
奈特,莫利纳里,彼得罗夫,检察官。大基因组约束假说:进化、生态学和表型。张志强,2005;https://doi.org/10.1093/aob/mci011.
苏达,杨晓东,杨晓东,Jarolímová V.中国大陆被子植物基因组大小的变化:加那利特有植物区系的40%已完成。植物科学学报,2005;26(2):326 - 326。https://doi.org/10.1007/s00606-004-0280-6.
Leitch IJ, Beaulieu JM, Chase MW, Leitch AR, Fay MF。单子叶植物基因组大小、动态和进化。安·波特,2010。https://doi.org/10.1155/2010/862516.
佩利塞J,凯利LJ,雷奇IJ,佐勒弗WB,费伊MF。宇宙的侏儒和巨人:基因组大小和染色体进化在单子叶科黑桫椤科。新植物,2014;201:1484-97。https://doi.org/10.1111/nph.12617.
范伟林,李志强,李志强,等。欧洲物种多样化的空前速度。Proc R Soc B. 2010; 277:1489-97。https://doi.org/10.1098/rspb.2009.2163.
Klak C, Khunou A, Reeves G, Hedderson T.基于四个质体DNA区域的Aizoaceae系统发育假说。中国科学(d辑),2003;https://doi.org/10.2307/4123697.
季勇,杨超,黄勇巴黎教派。Axiparis(桔梗科)产于中国云南。Phytotaxa。2017;306:234-6。https://doi.org/10.11646/phytotaxa.306.3.6.
尹浩,张浩,薛东。巴黎重楼var。emeiensis尹鸿祥,张宏,薛东,四川延铃科一新变种。植物分类学报,2007;45:822-7。https://doi.org/10.1360/aps06160.
李华,雷乐林,杨永明。巴黎yanchii是一种新物种巴黎云南莲属(紫花科)。科学通报。2017;46:1-5。https://doi.org/10.16473/j.cnki.xblykx1972.2017.01.001.
道尔JJ,道尔JL。一种用于少量新鲜叶片组织的快速DNA分离程序。植物化学通报1987;19:11-5。
Patel RK, Jain M. NGS QC工具包:用于下一代测序数据质量控制的工具包。公共科学图书馆,2012;7:e30619。https://doi.org/10.1371/journal.pone.0030619.
Langmead B, Salzberg SL. Bowtie2快速间隙读取对齐。Nat Methods. 2012; 9:357-9。https://doi.org/10.1038/nmeth.1923.
Wyman SK, Jansen RK, Boore JL。利用DOGMA自动注释细胞器基因组。生物信息学。2004;20:3252-5。https://doi.org/10.1093/bioinformatics/bth352.
Schattner P, Brooks AN, Lowe TM。用于检测的tRNAscan-SE、snoscan和snoGPS web服务器trna和snorna。核酸研究,2005;33:W686-9。https://doi.org/10.1093/nar/gki366.
宋志强,李志强,李志强,等。genous basic:一个用于组织和分析序列数据的集成和可扩展的桌面软件平台。生物信息学。2012;28:1647-9。https://doi.org/10.1093/bioinformatics/bts199.
Katoh K, Standley DM. MAFFT多序列比对软件版本7:性能和可用性的改进。中国生物医学杂志,2013;30:772-80。https://doi.org/10.1093/molbev/mst010.
波萨达D,克兰德尔KA。MODELTEST:测试DNA替代模型。生物信息学。1998;14:817-8。https://doi.org/10.1093/bioinformatics/14.9.817.
张志刚,张志刚。系统发育中的模型选择和模型平均:赤池信息准则和贝叶斯方法优于似然比检验。生物工程学报。2004;53:793-808。https://doi.org/10.1080/10635150490522304.
兰弗瑞,彭志强,李志强,李志强。基于分子和形态系统发育分析的进化分区模型选择新方法。中国生物医学杂志,2017;34:772-3。https://doi.org/10.1093/molbev/msw260.
Farris JS, Källersjö M, Kluge AC, Bult C.不一致性检验的显著性。支序分类学。1994;10:315-9。
斯沃福德DL。PAUP:使用简约(和其他方法)进行系统发育分析,4.0 beta。马萨诸塞州:Sinauer Associates;2002.
Stamatakis A. RAxML-VI-HPC:基于最大似然的系统发育分析,涉及数千个类群和混合模型。生物信息学。2006;22:2688 - 90。https://doi.org/10.1093/bioinformatics/btl446.
Ronquist F, Huelsenbeck JP。MrBayes 3:混合模型下的贝叶斯系统发育推断。生物信息学。2003;19:1572-4。https://doi.org/10.1093/bioinformatics/btg180.
Givnish TJ, Zuluaga A, Marques I, Lam VKY, Gomez MS, Iles WJD,等。百合属单子叶目的系统基因组学和历史生物地理学:出澳大利亚和穿越南极洲。支序分类学。2016;32:581 - 605。https://doi.org/10.1111/cla.12153.
张志强,张志强,张志强,谢东,等。野兽2:贝叶斯进化分析软件平台。科学通报。2014;10:e1003537。https://doi.org/10.1371/journal.pcbi.1003537.
Paradis E, Schliep K.猿5.0:现代系统发育和进化分析的环境,R.生物信息学,2019;35:526-8。https://doi.org/10.1093/bioinformatics/bty633.
R开发核心团队。R:统计计算的语言和环境。维也纳:R统计计算基金会;2017.
刘志刚,刘志刚,刘志刚,等。BAMMtools:一个R包,用于系统发生树的进化动力学分析。方法中国生物医学工程学报,2014;https://doi.org/10.1111/2041-210X.12199.
俞勇,Harris AJ,何晓杰。S-DIVA(统计分散-地理分布分析):推断生物地理历史的工具。分子系统进化学报,2010;https://doi.org/10.1016/j.ympev.2010.04.011.
于勇,何旭东,何旭东。RASP(系统发育中祖先状态的重建):历史生物地理学的工具。分子进化学报,2015;https://doi.org/10.1016/j.ympev.2015.03.008.
确认
我们感谢皇家植物园(爱丁堡)、Li Heng、Liu Bing和Pascal Bruggeman为本研究提供了一些材料;易思荣,安海成,甘其亮,赵仁,赵廷州,杨成金,周国华和王章明对现场工作的帮助。
资金
感谢国家自然科学基金重大项目(31590823)、国家自然科学基金(31872673)、云南省国家自然科学基金联合基金(U1802287)、中国科学院大型科学基金(No. 2017-LSF-GBOWS-02)、云南省科技计划重大项目(2019ZF011-2)对本研究的设计支持。并收集、分析和解释资料,以及撰写稿件。
作者信息
从属关系
贡献
YJ, JBY和TSY构思了这项研究;YJ, LY, JBY收集数据;LY, CL, ZY, JY对数据进行分析;YJ撰写稿件;MWC和TSY讨论了结果并修改了手稿。所有作者均已阅读并批准稿件。
相应的作者
道德声明
伦理批准并同意参与
所有样品的采集完全符合国家和地方法规的许可。研究中使用的植物样本并非来自国家公园或自然保护区。根据国家和地方立法,收集这些植物不需要特别许可。
发表同意书
不适用。
相互竞争的利益
作者声明,这项研究是在没有任何商业或财务关系的情况下进行的,这些关系可能被解释为潜在的利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:表S1。
Illumina测序综述。
附加文件2:图S1。
质体图巴黎物种。
附加文件3:表S2。
基因含量巴黎质体系。
附加文件4:表S3。
在研究中使用的样本,凭证和来源信息,以及Genbank的收录。
附加文件5:表S4。
从Genbank下载序列。
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
季勇,杨良,蔡文伟et al。质体系统基因组学,生物地理学和分支多样化巴黎(Melanthiaceae)。BMC植物生物学19,543(2019)。https://doi.org/10.1186/s12870-019-2147-6
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-019-2147-6
关键字
- 质体phylogenomics
- 生物地理学
- 辐射多样化
- Cytonuclear不整合
- 大基因组大小
- Parideae
- 巴黎
- Melanthiaceae
- Trilliaceae