摘要
背景
农艺性状位点的鉴定及其遗传结构的表征是标记辅助选择(MAS)的关键。全基因组关联研究(GWAS)已越来越多地被用作识别标记-性状关联(MTAs)的有力工具。在不同遗传背景下引入新的自适应等位基因可能有助于提高老小麦或新开发小麦品种的产量,以平衡全球的供需。从不同气候区收集的地方品种可以成为这种适应性等位基因的宝贵资源。
结果
利用收集的298个伊朗面包小麦品种和地方品种进行了GWAS,以探索在正常(充足灌溉)和应激(雨水灌溉)条件下,2016-2018种植季节农艺性状的遗传基础。基于W7984参考基因组,利用所有原始单核苷酸多态性(SNP, 10938个SNPs)或附加的46,862个SNPs获得高质量的基因分型测序(GBS)数据集。结果表明,B基因组在两个品种(49,880对,27.37%)和地方品种(55,086对,28.99%)中均具有最高的显著标记对。标记对之间的连锁不平衡(LD)在2D染色体上最强(0.296)。与A和B基因组相比,D基因组的LD衰减较低。在两个测试环境下的关联映射总共产生了313和394个显著的(−log . log)10P>3)原始SNP数据集和估算SNP数据集的mta。基因本体分析结果表明,在水培条件下,原始集合中有27%和27.5%的SNPs MTAs位于蛋白编码区。而在估算数据集中,水分充足和雨水灌溉条件下,蛋白编码基因中MTAs的比例分别为22.6%和16.6%。
结论
我们的发现表明,伊朗面包小麦的地方品种拥有有价值的等位基因,可以适应干旱胁迫条件。位于编码基因内的MTAs可用于小麦新品种的基因组育种。虽然缺失数据的补充增加了mta的数量,但这些位于编码基因中的mta的比例在不同的亚基因组中都减少了。
背景
面包小麦(小麦L.)是发展中国家和发达国家的主要作物,因此经常需要平衡供应和需求[1].预计人口的增长将增加小麦的需求,从而突出了植物育种家需要利用一切可获得的工具,寻找新的方法,在未来几十年可持续地增加面包小麦的产量[1,2].20世纪60年代和70年代绿色革命后,通过改善农场管理措施和引进适应性良好的小麦品种,小麦产量显著提高。然而,由于人口激增,全球小麦消费量也在稳步增长[1].1962年和2012年小麦产量和消费量的比较表明,对小麦的需求大幅增加,中国、欧盟、印度和美国是主要消费国。然而,从过去50年的需求增长来看,很明显,包括印度尼西亚、孟加拉国和泰国在内的亚洲国家处于榜单的前列。1].有几个因素极大地限制了世界各地的小麦生产,如缺水、盐和冷胁迫,导致粮食和生物量的重大损失[3.,4].
了解耐旱性机制和确定介导耐旱性的基因座是任何旨在提高面包小麦水分缺乏引起的耐旱性的育种方法的关键步骤。测序技术的最新进展已经成为可供模型植物使用的基因组数据库,例如拟南芥[5),栽培稻[6],以及一些重要的作物品种,包括大麦芽[7),t . aestivum[8].大规模基因组资源的可用性为发现植物对不同环境胁迫反应背后的遗传和分子机制提供了机会。由于大多数重要农艺性状可能由大量基因控制,数量性状位点(QTL)定位已广泛用于分析这些性状的遗传结构[9,10,11].然而,qtl作图有一些缺点,例如分辨率低和每次研究可以筛选的等位基因数量有限。高通量基因分型技术提供了大量的单核苷酸多态性(SNP)数据,通过提供高分辨率的连锁图,极大地提高了QTL定位的分辨率[12].
此外,由于大规模基因组资源的可用性增加,全基因组关联研究(GWAS)现在是解剖数量性状遗传结构的QTL作图的可行替代方法[13].与qtl映射相比,GWAS有助于以时间和成本效益的方式加速评估更具代表性的个体集合[14].GWAS基于建立基因型和表型之间的相关性,其思想是,在几代人群中已经形成了连锁不平衡(LD),因此即使因果突变不一定包括在可用的遗传标记集中,也可以检测到含有qtl的区域。覆盖整个基因组的高密度基因分型使GWAS成为识别所观察到表型的基因组区域的宝贵工具。最近的几项研究已经成功地应用GWAS来确定包括水稻在内的许多作物物种的重要性状的遗传基础[15],大麦[16],玉米[17]和小麦[18].这些研究还提供了关于mta的信息,这可以帮助育种人员进行标记辅助选择。特别是,最近的一些研究通过确定与粮食产量和相关性状相关的qtl来关注小麦[19,20.,21].
通过测序进行基因分型(GBS)可以以经济高效的方式获得大量snp,但它经常受到高比例缺失数据的困扰,这可能限制任何全基因组关联研究的准确性。处理缺失数据的一种方法是通过imputation,这已在许多关于人类和植物基因组的研究中成功实施[22,23,24,25,26].在考虑了没有直接基因分型的snp后,通过依赖来自普通单倍型的连锁信息,Imputation可以增加GWAS中包含的变异数量[27].低深度测序和文库复杂性可能会导致SNP数据中的信息缺失,因此基因型imputation可以通过可用的参考基因组部分弥补这类问题,而不需要额外昂贵的重测序[28,29].本研究的主要目的是利用GBS-SNP数据进行GWAS实验[30.]和基于面包小麦W7984参考基因组估算的SNPs,此前已被证明可产生最高的估算精度[31].在伊朗小麦地方品种和品种中测量了三类农艺性状,并将其用于一项关联研究,以探索假定的qtl,以鉴定可能参与提供耐旱性的重要发育途径的基因。本研究的第二个目标是通过评估归因对mta的影响,确定使用原始SNP数据与归因SNP数据产生的结果是否存在差异。
结果
表型评价
分别分析了丰水和雨养条件下的数据集。方差分析发现显著差异(P < 0.01)在两种环境下,除了雨养条件下的千粒重外,所有性状均在两种环境下进行了两年的研究(附加文件)2:表S2和表3)。在雨水灌溉条件下,早期羽化被推迟,但基因型的生理成熟度比水分充足条件下降低了14.9天,从而提前完成了它们的寿命(表S2和表3)1).丰水条件下灌浆期为27.2 d,雨水条件下灌浆期为24.0 d。在水分充足条件下,每穗粒数和千粒重变异最大(标准差分别为7.35和7.10),而在雨养条件下,株高和花梗长度变异最大(标准差分别为15.55和7.26)。丰水条件下,籽粒产量、穗重、粒数、千粒重、叶片绿度与灌浆期呈极显著正相关(p < 0.01),而物候性状和冠层温度与籽粒产量呈负相关(p < 0.01而且0.05,附加文件2:表S4)。雨育条件下,籽粒产量与物候性状、株高、花序梗长、穗长、冠层温度呈显著负相关,与穗重、穗粒数、千粒重呈显著正相关(p < 0.05)。p < 0。01、附加文件2:表S5)。
SNP标记的评价
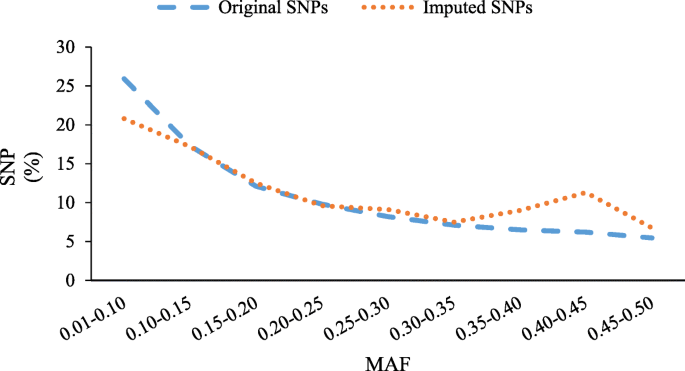
测序后,在566,439,207个reads中共鉴定出458,363,607个unique reads(~ 81%非冗余reads)。经过重复去化和比对,共征集到133039个snp,其中10938个缺失数据< 10%,杂合度< 10%,小等位基因频率(MAF) >1%。选择这些snp进行进一步分析。在鉴定的10938个snp中,MAFs的标记数量分别在0.01-0.1和0.45-0.50范围内最高(2835,25.92%)和最低(597,5.46%)。1).此外,我们利用W7984参考基因组获得了一组46,862个估算snp,这些snp也被用于估计遗传多样性。
连杆不平衡(LD)
连锁不平衡分析表明,LD在亚基因组、染色体和每条染色体之间存在差异,并且LD通常随着snp之间距离的增加而下降。共有368,310个标记对(MP,根据不同染色体上snp的组合)具有平均平方等位基因频率相关性或r2各品种间的连锁系数为0.132,其中96541个(26.2%)在P < 0.01(表2).大约93%的所有MPs和94%的重要MPs位于距离<10 cM。基因组B和D中MPs数量最多(182271,49.49%),最少(50395,13.68%)。此外,在2D染色体上的MPs之间的LD最强(0.296),其次是1D染色体(0.214)。
对地方品种进行类似分析,共鉴定出405,738个MPs,平均LD为0.097,大大低于品种(表2)2).然而,在本地人种数据中观察到较大比例的显著MPs(29.31%)。89%的总MPs和88%的显著MPs距离< 10 cM。此外,在B基因组中观察到的MPs数量最多(190,001)。本地种不同亚基因组的SNPs总数略高于品种。同样,LD在2D染色体上最高(0.198)。LD衰减在附加文件中显示3.:无花果。S2-4。D基因组染色体上的LD表现出明显的趋势,与a或B基因组相比,LD的衰变发生得更慢。对于A和B基因组的大多数染色体,当距离< 5 cM时,LD下降到0.1,而d基因组的相应距离为5 ~ 10 cM。
种群结构与亲缘矩阵
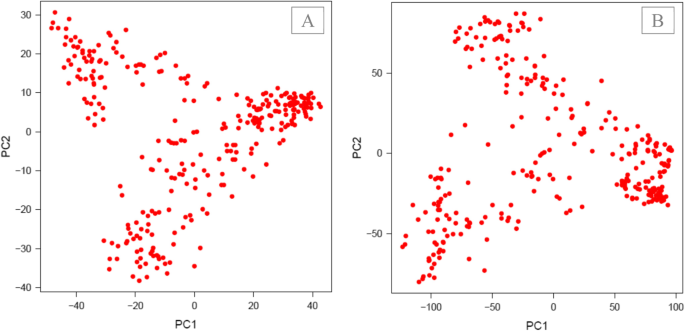
我们使用从原始和估算的snp中获得的个体方差-协方差矩阵(亲属矩阵)来评估群体结构。对于这两个数据集,分析确定了三个主要的不同程度的外加剂组。对于原始数据集,前两个主成分解释了17.2%的遗传方差(图2)。2a),而估算snp的方差为23.2%(图2)。2b).此外,在种群结构分析中,当K = 3时,∆K值最大。
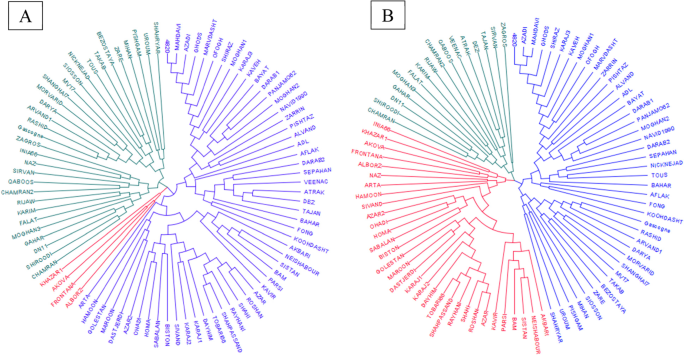
I组共69份,66个品种,3个地方民族;类群II包含120个品种,102个地方品种和18个品种;类群III包含103个地方品种和6个品种(图2)。3.a).当我们使用估算的snp时,材料也分为三个主要类群,其中类群I包含113份材料,108个地方品种和5个品种;类群II包含74份研究,70个品种和4个地方品种;第三组共110份,包括97个地方品种和13个品种。3.b).原始SNP数据显示有24个品种与两个地方民族群体混种,而估算SNP数据显示只有19个品种混种。杂交种来自伊朗地方品种,杂交种包括Shahi、4820、Mahdavi、Azadi、Ghods、Neishabour和Sivand来自其他材料。
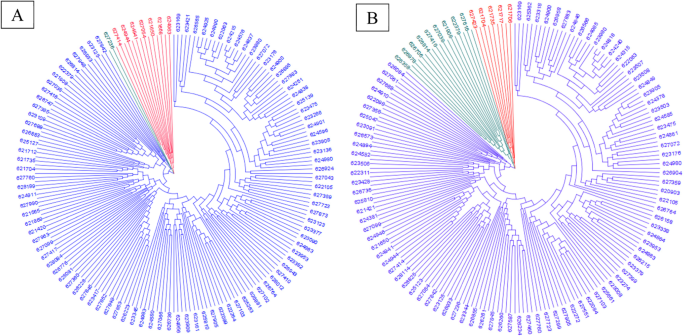
所有品种的邻居连接树也清楚地显示了两个数据集的聚类为三个子组(图。4除了Khazar1、Akova、Frontana和Alborz这几个品种转移为两个相邻组的12个和25个品种的两个系列(图。4b).即使根据两个SNPs数据集将本地人种聚为三个组,但使用估算SNPs可以更清楚地区分它们的差异(图。5).利用原始snp将PI627236和PI625433归为同一类群,而将PI625433归为最大类群,利用估算snp进行聚类时,这两个小类群的分布发生了变化。
农艺和生理性状mta
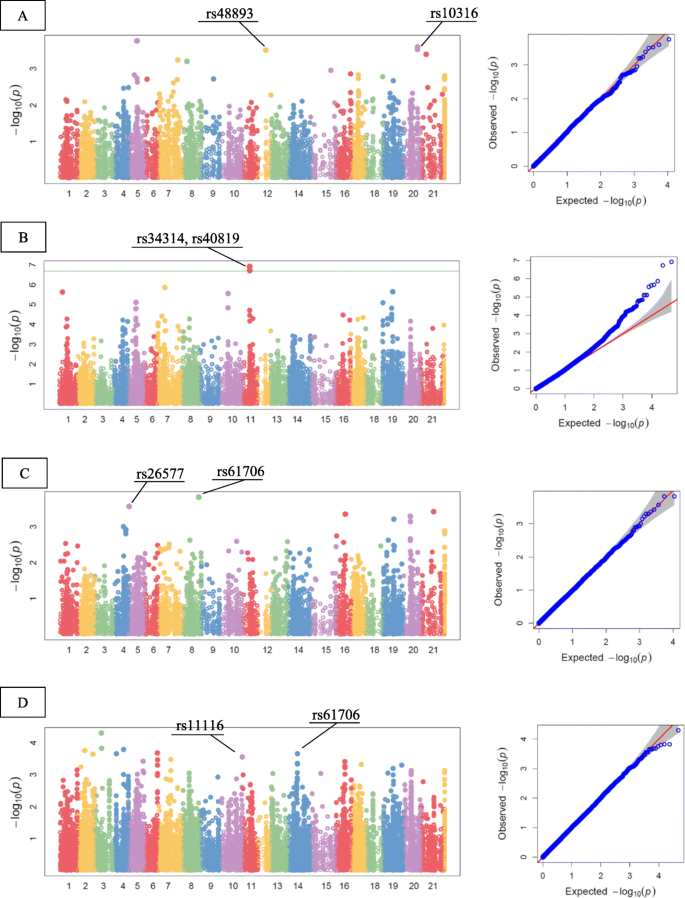
使用原始SNP数据集共识别出313个mta,显著性值为-log10P>3适用于充足灌溉和雨水灌溉条件。对估算数据共检测到394个mta3.).无论是原始snp还是估算snp, B基因组的MTAs数量最多,而D基因组的MTAs数量最少。在水分充足条件下研究的性状中,籽粒灌浆期和穗长分别表现出最高的原始SNPs和估算SNPs相关MTAs数量。此外,利用原始数据集和估算数据集,在水分充足条件下,单株籽粒产量分别观察到13个和4个mta。在干旱胁迫条件下,基于非估算SNPs,有23个mta与籽粒灌浆期和花梗长度有显著相关,而基于估算SNPs,有10个和18个mta与这些性状有显著相关。然而,每穗种子数的估算MTAs数量最高(25 MTAs)。
对于原始SNP数据,在水分充足的条件下,2B和7B染色体上的显著标记数量最多,分别为12个和11个,其次是2A、3A和6B染色体,每条染色体上有8个标记。在雨养条件下,在染色体5B和6B上共鉴定出29个和15个相关snp。对于估算数据集,在水分充足的条件下,4B、1A和5B染色体上识别出47、32和26个显著标记,而在雨水灌溉条件下,使用估算snp时,5B、4A和1A染色体上识别出85、40和28个显著标记。A和B染色体上的标记在不同性状间表现出多效性。
基因注释
我们使用原始SNP数据集识别的313个mta的基因本体表明,在充足的水和雨水喂养的条件下,分别有27%和27.5%的mta位于蛋白质编码基因内(附加文件)4:表S6和表7)。相比之下,在使用估算SNP数据集识别的394个mta中,分别有22.6和16.6%位于水分充足和雨水灌溉条件下的编码基因内(补充文件)4:表S8和9)。具有mta的基因主要编码的蛋白质涉及泛素化、氧化还原、蛋白质磷酸化、组蛋白去泛素化、转录负调控、对脱落酸的反应、分解代谢过程、多细胞生物发育、叶黄素生物合成、对紫外线的反应、离子运输、细胞分裂素生物合成、DNA甲基化、DNA复制、细胞对DNA损伤刺激的反应、氧化应激反应、细胞蛋白修饰过程,碳水化合物代谢过程。
我们总结了表中显示最强关联的snp的结果4来7.在水分充足的条件下,编码基因中的5个和7个标记分别位于染色体1A、4B、4D、5A、5B、6B和7B上,分别为原始和估算的snp(表4而且5).这些标记与冠层温度、穗粒数、千粒重、灌浆期、籽粒产量、穗重和穗长、叶片绿度和出苗天数有关。这些snp大多位于B基因组上,其次是A基因组,最后是D基因组(表4而且5).在雨养条件下,14和11个SNPs与调节穗重和穗长、千粒重、株高、花序柄长、叶片绿度、灌浆期、种子数、籽粒产量、冠层温度、出苗天数、抽穗期和生理成熟度的基因相关(表2)6而且7).在雨养条件下,这些标记均位于B和A基因组上,D基因组上无显著相关标记。总的来说,B基因组包含了相当一部分农艺性状的高显着snp。染色体1A上的标记rs36032和染色体7A上的标记rs56337分别与在充足水和雨养环境下提供粮食产量的基因相关。
挖掘高度相关的有利等位基因
本研究将籽粒产量、籽粒数、千粒重、籽粒灌浆期、穗重、叶绿度增加、出苗天数、抽穗天数、开花天数、生理成熟天数、冠层温度、株高、花序柄长、穗长等具有积极作用的SNPs定义为有利等位基因。使用原始数据集和估算数据集,在充足的水条件下,强相关snp的表型效应被量化人工智能,冠层温度rs62576降低0.72,穗长rs36808降低1.09,rs40819降低0.70,穗长rs36808降低0.50,穗长rss57386降低0.118).rs48893和rs10316对穗粒数的正增加效应分别为0.96和1.20。千粒重、灌浆期、产量和叶片绿度分别提高0.77 g、0.90d、0.01 g和1.82。穗重与两个标记rs36032和rs736相关,分别使性状增加0.02 g和0.33 g。相关snp解释的表型方差(PVE)范围为7 ~ 40%(表2)8).snp rs48893、rs10316和rs36032均表现出较高的相关性状方差。
如表所示9雨育条件下,3、2、2、2、1、1个原snp与穗重(分别增加0.02、0.03、0.03 g)、千粒重(分别增加1.59、1.90 g)、籽粒产量(分别增加0.03、0.03 g)、灌浆期(分别增加1.31、1.57 d)、穗粒数(增加0.58)、叶片绿度(增加1.69)呈正相关。rs2234、rs25700、rs46075和rs30520对穗长(增加0.53 cm)、株高(增加11.06 cm)、花梗长(增加1.76 cm)和出苗期(增加0.28 d)均有正向影响,但对穗长、穗重、花梗长、抽穗天数、开花天数、生理成熟天数、冠层温度和叶片绿度的有利等位基因数分别为2、1、2、2、1、1、2和1。Rs11116和rs25700使花梗长度分别减少了2.66和4.56 cm, rs59282和rs17806使抽穗、开花和成熟天数分别减少了4.24、7.59、7.04和6.78 d, rs64750使冠层温度下降了2.61℃。在雨养条件下,PVE在7 ~ 38%之间。与籽粒产量、穗重和粒数相关的snp均能解释相当比例的表型方差。此外,SNP rs17806将开花和生理成熟的天数分别调整了约36%和38%。农艺性状高相关单倍型的Manhattan图和qq图如图所示。6.
讨论
提高小麦产量是小麦育种者的首要任务,以满足世界范围内日益增长的需求。在目前的研究中,我们探索了伊朗六倍体小麦群体的多样性,并对一些重要的农艺性状进行了关联作图研究。这些性状直接或间接地影响丰水或雨养条件下的粮食产量。这些性状之间存在显著的正相关或负相关关系,可用于衡量它们对目标性状如粮食产量和粮食产量相关属性的影响。鉴于大多数农艺性状是多基因的,而耐旱性是一个涉及多种途径的复杂机制,我们利用大型多样性面板重点研究了三类农艺性状。此外,使用多样化的基因库有助于提高关联映射的分辨率。因此,我们对历史和现代品种以及来自不同气候区的代表性地方品种进行了测试,以包括足够的遗传变异,以便能够绘制性状相关的变异。
大多数伊朗品种来自国际玉米和小麦改良中心(CIMMYT)的材料,有24个品种的先进和分离系,源自该中心。至少有40个品种通过杂交育种项目获得,其中一个CIMMYT的高级品系是亲本之一1:表S1)。这些品种在绿色革命后大量释放。先前对来自巴基斯坦的历史小麦品种的研究表明了CIMMYT种质资源的巨大贡献,该种质资源也被用于伊朗品种的开发[21].我们确定了三个具有混合遗传背景的品种集群,与发布年份没有明确的关系。来自伊朗的品种数量相对较少,这表明在当前和旧品种中,伊朗地方品种的利用相对狭窄,这可能是一个实质性的遗传瓶颈。
与早期的研究一致,我们在B和A基因组中发现了大多数snp,而较年轻的D基因组显示出较低数量的snp [32,33].我们在LD的标记对数量上也观察到了同样的趋势,其中定位到B基因组的snp比定位到D基因组的snp大约多三倍。在两个品种和地方品种中,最显著的标记对都在染色体2B和3B上观察到(表2)2).在A和B基因组中看到的更高的多样性可能是它们更古老的进化背景的结果,并且由于基因流从t . turgidum而不是缺乏基因流动Ae。tauschii面包小麦[34,35].此外,在现代品种育种计划中,由于对祖先六倍体地方品种的强烈选择,可能发生了瓶颈效应,这可能对D基因组产生进一步的影响。这种瓶颈会导致有效种群规模的减少,从而增加了a和B基因组中低频等位基因的损失率,另一方面,D基因组中罕见等位基因比例较高,表明年轻基因组的等位变异减少[36].我们的结果表明,大多数显著性LD标记位于距离< 10 cM处。然而,D基因组的LD和标记距离远高于其他两个亚基因组。品种中所有基因组之间的更大程度的联系清楚地表明了在这些遗传历史中选择的影响(表2)2).选择、重组、突变、遗传漂变、交配制度和群体亲缘关系都是影响连锁失衡的主要因素[37,38,39].事实上,与本地品种相比,品种表现出明显更高的LD,特别是在D基因组中,这可能是育种过程中对重要农艺性状选择的结果[40,41].
虽然定位遗传力低的性状可能不会产生理想的增益,但利用高度相关的性状可以增加功率,因此有助于快速推进育种计划。尽管在不同的目标环境中,特别是在缺水条件下,粮食产量是最可靠的选择标准,但迄今为止,这一性状的复杂遗传结构限制了直接基于基因组的选择。然而,控制该性状的基因的多效性效应以及粮食产量与耐旱机制之间的密切联系突出了其他形态、物候和生理性状的作用,在任何选择策略中都应考虑这些因素。
在313个和394个鉴定出的mta中,分别有86个和76个属于编码基因P值<0.001.为了消除可能的假阳性关联,我们选择了相关性最强的标记,在所有染色体上产生了19个和17个标记,并使用两个SNP数据集在两种环境中进行了识别。利用水分充足条件下的原始snp,鉴定了冠层温度(1A)、种子数(4D和7B)、千粒重(5B)和灌浆期(6B)的qtl。在1A和4B染色体上观察到籽粒产量和穗重的QTL,在1A染色体上观察到叶片绿度的QTL,在4B和5B染色体上观察到3个穗长QTL,在5A染色体上观察到1个苗期QTL。这些结果与之前检测到的穗长qtl一致[42],粮食产量[42],每穗粒数[43]和千粒重[44].
在雨育条件下,在1B、2B染色体上鉴定了穗重qtl, 6B和7A染色体上鉴定了籽粒产量qtl, 1B染色体上鉴定了穗长qtl, 2B和3B染色体上鉴定了千粒重qtl, 3B染色体上鉴定了花序柄长qtl, 5B染色体上鉴定了株高qtl, 5B染色体上鉴定了叶片绿度qtl, 5B染色体上鉴定了每穗粒数qtl, 6B染色体上鉴定了灌浆期qtl, 6B染色体上鉴定了籽粒灌浆期qtl, 6B染色体上鉴定了出苗天数qtl。这些结果与Ain等人的发现一致。[21]对株高、千粒重和籽粒产量有显著影响。波索里尼等人。[45]和Acuna-Galindo等人[46]分别在6B和5B染色体上鉴定出稳定的籽粒产量和株高qtl。5B染色体上每穗种子数的mta以前也有报道[47,48].诺伊曼等人[14]在2B染色体上发现了穗重的QTL。在雨育条件下,所有鉴定的mta均位于染色体4A、4B和5B上,qtl分别为冠层温度(4A和4B)、花梗长度(4A和5B)、穗重(4B)、穗长(4B和5B)、叶片绿度(5B)和抽穗天数、开花天数、成熟期(5B)。这些结果与之前报道的这些性状的qtl一致[14,33,47,48].此外,我们还发现了一些新的染色体区域,这些区域含有用于生理参数和物候生长阶段的MTAs。例如,在水分充足的条件下,rs62576 (1A)、rs34075 (1A)和rss57386 (5A)与冠层温度、叶片绿度和羽化天数相关。在雨养条件下,标记rs64750 (4A)和rs55557 (4B)分别与冠层温度相关,rs4607 (3B)和rs41689 (5B)分别与花梗长度和叶绿度相关。此外,标记rs17806 (5B)对抽穗天数、开花和生理成熟具有多效性作用。
尽管缺失数据的归因显著增加了mta的潜在数量,主要是在A和B基因组上,但编码基因中出现的SNP的比例下降了,从原始SNP数据集中的约27%下降到归因数据集中的19.6%。这表明,大多数缺失数据的SNP位于非编码DNA区域,在大多数植物基因组中,读取映射和SNP调用已知更有问题。例如,通过位置克隆来解剖强相关的染色体区域,以确定假定的因果基因是关联映射研究之后的下一个逻辑步骤。除了使用比较基因组学方法来确定相关基因的功能外,还需要独立的功能验证来保证位置克隆或转基因实验的成功[21,49,50,51].我们获得了假定snp的侧翼序列,并将其与IWGSC RefSeq v1.0进行比对。这一信息表明,通过关联研究发现的大多数基因都参与了重要的生物合成途径,如氧化还原、碳水化合物代谢、离子运输和细胞壁生物发生。这些基因编码的蛋白主要参与dna结合、atp结合、过氧化物酶活性、蛋白激酶活性、金属离子结合、酶抑制剂活性、丝氨酸型内肽酶活性、水解酶活性、反转运蛋白活性和跨膜活性。在早期的研究中也报道了这种联系[52,53,54,55,56,57,58].这些基因均位于染色体区域,与重要的农艺性状有很强的相关性,可作为今后育种的合适靶基因。正如Dong等人所描述的,我们计算了有利等位基因的表型效应。[59结果表明,它们对籽粒产量的影响很小,但对千粒重、穗长和叶片绿度的影响要大得多。另一方面,有助于提高千粒重和叶绿度的等位基因也具有多向效应,从而降低胁迫条件下的株高、花序梗长和冠层温度。大多数已确定的mta存在于涉及多层过程和复杂网络的基因之间,因此它们对农艺性状的轻微影响并不太牵强。
结论
本研究对伊朗收集的298个面包小麦品种和地方品种的重要农艺性状进行了GWAS分析。在B基因组上,品种和地方品种的标记对数量最多。利用原始snp和估算snp分别鉴定出14个物候、农艺和生理性状的313个和394个MTAs。已确定的标记和性状之间的关联通常在10个范围内−3和10−4.这种数量性状的复杂遗传和大量的控制基因似乎排除了更大的关联。然而,发现的mta的主要部分解释了相关性状总表型变异的20%以上。不过,还需要进一步的研究来验证本研究中检测到的标记物是否适用于其他人群和环境。基因本体分析表明,约27%的标记分布在编码基因内,具有应用于新品种基因组选育的潜力。虽然缺失数据的补充可以增加相关标记的数量,但不同亚基因组中位于编码区域的mta的百分比降低了。本研究鉴定的标记可为启动标记辅助选择、精细定位和克隆潜在基因和qtl提供有用的遗传资源。
方法
植物材料和实验条件
一套320种伊朗小麦,包括1942年至2014年间发布的102个品种,以及1931年至1968年间收集的218个地方品种(附加文件)1:表S1)在德黑兰大学农学和植物育种系的农业研究用地上,采用alpha-lattice设计,在灌溉系统和雨养条件下进行了两次重复试验。植物材料由德黑兰大学和种子和植物改良研究所(SPII)提供,卡拉杰,伊朗。共获得298份(90个品种和208个地方品种)的表型和基因型数据。该油田位于n35 '。80°和东经50′。95° in Karaj, Iran, and experiments were conducted during the cropping seasons of 2016–17 and 2017–18 (weather conditions are given in Additional file3.:图S1)。
现场试验
根据Zadoks量表对植株发育进行评分,包括i)到出苗期(Zadoks 12天)、ii)到抽穗期(Zadoks 50天)、iii)到开花期(Zadoks 65天)、iv)到生理成熟期(Zadoks 91天),以及v)每块有一半达到相应阶段的灌粒期。利用土壤植物分析开发(SPAD, Minolta Camera Co.,大阪,日本,SPAD502 Plus叶绿素仪)和LIHERO红外测温仪分别测量了Zadoks 60的叶片绿度和冠层温度。两年收获后测定籽粒产量及其相关性状,包括穗重、穗长、穗粒数和千粒重。
基因分型采用测序和植入法
Alipour等人曾描述过伊朗小麦GBS库的开发和排序。[30.].简单地说,在将测序reads修剪到64 bp并将它们分组到序列标签中后,使用允许不匹配的内部比对来识别snp,误差可达3 bp。UNEAK (Universal network enabled Analysis Kit) GBS管道用于snp调用,其中低质量评分(<15)的reads和低次要等位基因频率<1%的snp被删除,以避免测序错误引起的假阳性标记。数据还使用BEAGLE v3.3.2进行了imputation [60]基于指定所有个体的单倍型阶段后获得的可用等位基因频率。在植入过程中评估了四个不同的参考基因组,其中W7984参考基因组被证明具有最高的植入精度[31].使用RStudio中的ggplot2包获得不同染色体的LD衰变[61,基于黄土回归分析。
种群结构与亲缘矩阵
样本中的总体结构使用structure v.2.3.4估计[62],并为K = 1到10的值设置了10,000步的老化和模拟阶段。∆K为连续的K值,用于确定最可能的亚种群数量。使用观察到的和期望的等位基因频率值来计算TASSEL v.5中标记之间的LD [63].然后获得用于关联研究的所有材料的结构矩阵(Q-matrix)。为了确定品种与地方品种之间的关系,基于TASSEL v.5中计算的成对距离矩阵构建了邻居连接树[63],并使用始祖鸟(https://sites.google.com/site/cmzmasek/home/software/archaeopteryx).
全基因组关联研究
我们使用一般线性模型(GLM)和混合线性模型(MLM)来获得标记效应的无偏估计。传销方法产生了最准确的标记性状关联,不同版本的传销模型,包括Q、K或Q + K,被用于控制群体结构(Q)的影响和使用TASSEL v.5的亲本之间更扩散的关系(K) [63].关税化措施[64]对RStudio中的MLM模型进行关联映射[61].TASSEL [63]及GAPIT [64基于相关基因座使用的重要性进行评估t测试。一般而言,政府税务关税法[64]提供了对混杂效应更强的控制。因此,我们只报告了GAPIT的结果[64].在传销模型中,个体被视为随机效应,个体之间的亲缘关系通过亲缘矩阵来表达。在聚类分析中,以亲缘矩阵元素作为相似度度量,通过热图图采用算术平均非加权对组法(UPGMA)对聚类进行可视化。曼哈顿图是表型和基因型之间联系的可视化形式,其中snp根据其染色体和碱基对位置进行排序。在曼哈顿图中,x轴因此表示每个SNP的基因组位置,y轴表示的负对数P-测试H时f检验生成的值0.在这里,热图和曼哈顿地块都是从使用GAPIT包的增强比较场景中获得的[64].
基因注释
从小麦90k SNP数据库中获得了所有显著相关SNP的序列[65]用于评估基因注释的Gramene (http://www.gramene.org/),将它们对准IWGSC RefSeq v1.0注释(https://wheat-urgi.versailles.inra.fr/Seq-Repository/Annotations).通过对编码酶参与的途径的研究,探索了假定基因的功能。将SNPs序列与参考基因组比对后,选取同源率和blast评分最高的重叠基因进行进一步处理。从集成格兰烯数据库中提取每个基因的基因本体,包括分子功能和生物学过程(http://ensembl.gramene.org).
表型数据分析和计算有利的等位基因效应
使用SAS v.9.4分析表型数据[66]分别用于两个环境。然后从用于高级线性分析的alpha-lattice设计中获得调整后的均值。使用GLM和Mixed程序估计调整后的平均值。有利等位基因的表型效应(人工智能)用以下公式估算:
在那里,xij是否有表型价值j第Th个人我等位基因,n我携带人数是多少j等位基因,NK是所有条目的第n个个体表型值,和nK是个体的数量。所有等位基因的正、负效应均由人工智能> 0,人工智能分别< 0。
数据和材料的可用性
在当前研究期间生成和分析的数据集可在Figshare中获得。5、6、7和8次运行的SNP数据文件可以分别在以下位置访问:
https://figshare.com/s/92cd9f23d9dddb224304;
https://figshare.com/s/b402031d86d969a7cb0f;
缩写
- CT:
-
冠层温度
- 大卫·爱登堡:
-
开花天数
- 德:
-
出现的日子
- DH:
-
出发天数
- 糖尿病:
-
生理成熟的天数
- GBS:
-
基因测序分型
- 女友:
-
灌浆期
- GWAS:
-
全基因组关联研究
- 孔侑:
-
粮食产量
- H:
-
株高
- LD:
-
连锁不平衡
- 格林:
-
叶绿色
- 加:
-
小等位基因频率
- MAS:
-
分子标记辅助选择
- 议员:
-
标记对
- MTA:
-
Marker-trait协会
- PL:
-
花梗长度
- QTL:
-
数量性状位点
- SL:
-
穗长
- SN:
-
每穗粒数
- SNP:
-
单核苷酸多态性
- 西南:
-
体重飙升
- TKW:
-
千粒重
参考文献
- 1.
Curtis T, Halford NG。粮食安全:提高小麦产量的挑战和不损害粮食安全的重要性。中国生物医学工程学报,2014;26(3):354 - 372。
- 2.
Tilman D, Cassman KG, Matson PA, Naylor R, Polasky S.农业可持续发展与集约化生产实践。大自然。2002;418(6898):671 - 7。
- 3.
O’leary GJ, Christy B, Nuttall J, Huth N, Cammarano D, Stöckle C, Basso B, Shcherbak I, Fitzgerald G,罗强2在自由空气证书下2半干旱环境下的富集(FACE)实验和模拟。张格学报。2015;21(7):2670-86。
- 4.
理查兹RA,亨特JR,柯克加德JA,西帕斯oura JB。澳大利亚小麦产量改良及对缺水环境的适应——个案研究。作物草地学报,2014;65(7):676-89。
- 5.
倡议AG)。开花植物基因组序列分析拟南芥.自然。2000;408(6814):796 - 815。
- 6.
高夫,王瑞,王瑞,杜恩,Glazebrook J, Sessions A, Oeller P, Varma H,等。水稻基因组草图(栽培稻l . ssp。粳稻).科学。2002;296(5565):92 - 100。
- 7.
Beier S, Himmelbach A, Colmsee C, Zhang X-Q, Barrero RA, Zhang Q, Li L, Bayer M, Bolser D, Taudien S,等。大麦参考基因组图谱序列的构建。科学数据。2017;4:170044。
- 8.
Mayer KFX, Rogers J, Dolezel J, Pozniak C, Eversole K, Feuillet C, Gill B, Friebe B, Lukaszewski AJ, Sourdille P,等。基于染色体的六倍体面包小麦抽穗序列(小麦)基因组。科学。2014;345(6194)。
- 9.
Brondani C, Rangel PHN, Brondani RPV, Ferreira ME。水稻产量相关性状的QTL定位与渐渗选用glumaepatula栽培稻(栽培稻)使用微卫星标记。《理论与实践》,2002;29(6):344 - 344。
- 10.
布斯特迈尔H,潘T,安德森JA。QTL定位和标记辅助选择镰刀菌素小麦抗头疫病研究进展。植物学报。2009;28(1):1 - 26。
- 11.
羽衣甘蓝BCY, Jahufer MZZ, Brouwer JB, Pang ECK。介绍作物改良中的标记、数量性状位点(QTL)定位和标记辅助选择:基本概念。Euphytica。2005;142(2):169 - 96。
- 12.
黄超,聂旭,沈超,游超,李伟,赵伟,张旭,林哲。利用高密度snp的全基因组关联研究揭示中国陆地棉农艺性状的群体结构和遗传基础。中国生物工程学报,2017;15(11):1374-86。
- 13.
Saidou A-A, Thuillet A-C, Couderc M, Mariac C, Vigouroux Y.环境相互作用的基因型关联研究:前景和限制。BMC Genet, 2014;15(1):3。
- 14.
Neumann K, Kobiljski B, Dencic S, Varshney RK, Boerner a .面包小麦全基因组关联图谱研究(小麦l .)。Mol Breed. 2011;27(1): 37-58。
- 15.
赵魏黄X, X,唱T, Q,冯Q,赵Y,李C,朱C,陆T,张Z, et al。水稻地方品种14个农艺性状的全基因组关联研究。中国科学,2010;42(11):961-76。
- 16.
Pasam RK, Sharma R, Malosetti M, van Eeuwijk FA, Haseneyer G, Kilian B, Graner a .全球春大麦农艺性状的全基因组关联研究。BMC植物生物学。2012;12:16。
- 17.
李辉,彭震,杨旭,王伟,付杰,王杰,韩勇,柴勇,郭涛,杨楠,等。玉米籽粒油脂生物合成基因结构的全基因组关联研究。中国科学,2013;45(1):43-50。
- 18.
Sukumaran S, Dreisigacker S, Lopes M, Chavez P, Reynolds MP。生长在温带灌溉环境的优良春小麦群体籽粒产量和相关性状的全基因组关联研究。应用理论与实践。2015;28(2):353-63。
- 19.
Bordes J, Ravel C, Jaubertie JP, Duperrier B, Gardet O, Heumez E, Pissavy AL, Charmet G, Le Gouis J, Balfourier F.全球小麦核心收集中与氮限制响应相关的基因组区域应用理论与实践。2013;26(3):344 - 344。
- 20.
Zanke C, Ling J, Plieske J, Kollers S, Ebmeyer E, Korzun V, Argillier O, Stiewe G, Hinze M, Beier S,等。欧洲冬小麦抽穗期主要效应QTL的遗传结构。植物科学进展(英文版);
- 21.
Q-u A, Rasheed A, Anwar A, Mahmood T, Imtiaz M, Mahmood T, Xia X, He Z, Quraishi UM。巴基斯坦历史小麦品种在雨养条件下籽粒产量的全基因组关联。植物科学进展(英文版);
- 22.
Elliott LT, Sharp K, Alfaro-Almagro F, Shi S, Miller KL, Douaud G, Marchini J, Smith SM。英国生物库中脑成像表型的全基因组关联研究。大自然。2018;562(7726):210 - 6。
- 23.
Khera AV, Chaffin M, Aragam KG, Haas ME, Roselli C, Choi SH, Natarajan P, Lander ES, Lubitz SA, Ellinor PT,等。常见疾病的全基因组多基因评分可识别与单基因突变风险相当的个体。植物学报,2018;50(9):1219-24。
- 24.
马志刚,李志刚。全基因组关联研究中的基因型imputation。植物学报。2010;11(7):499-511。
- 25.
何松,赵颖,Mette MF, Bothe R, Ebmeyer E, Sharbel TF, Reif JC,蒋勇。欧洲优质小麦定量遗传标记植入的前景与局限性(小麦l .)。BMC基因组学.2015; 16(1): 168。
- 26.
傅得到。带有imputation的高度不完全SNP基因型数据的遗传多样性分析:一项经验评估。G3:基因基因组遗传,2014;4(5):891-900。
- 27.
李艳,王志强,王志强,王志强,等。中国生物医学工程学报。2009;10:387-406。
- 28.
Nyine M, Wang S, Kiani K, Jordan K, Liu S, Byrne P, Haley S, Baenziger S, Chao S, Bowden R,等。利用第一代单倍型图谱snp对冬小麦进行基因型植入,改善了全基因组关联作图和性状基因组预测。中国生物医学工程学报,2019;9(1):125-33。
- 29.
Subirana I, Gonzalez JR.纵向研究中估算snp的遗传关联分析和元分析。热内流行病学,2013;37(5):465-77。
- 30.
Alipour H, Bihamta MR, Mohammadi V, Peyghambari SA, Bai G, Zhang G.伊朗小麦地方品种和栽培品种分子遗传多样性的基因分型分析。中国植物科学,2017;8:1293。
- 31.
李丽波,白戈,张庚,毕汉塔,马默迪V, Peyghambari SA。利用大麦和小麦基因组参考资料的小麦基因测序分型(GBS)数据的Imputation精度。PLoS One. 2019;14(1):e0208614。
- 32.
Berkman PJ, Visendi P, Lee HC, Stiller J, Manoli S, Lorenc MT, Lai K, Batley J, Fleury D, Simkova H,等。分散和驯化塑造了面包小麦的基因组。中国生物工程学报,2013;11(5):564-71。
- 33.
王志强,王志强,王志强,等。群体测序技术在六倍体小麦基因分型中的应用。中国生物医学工程学报。2015;5(12):2547-53。
- 34.
德沃夏克J,阿胡诺夫ED,阿胡诺夫AR,迪尔KR,罗敏聪。驯化四倍体小麦诊断DNA标记的分子特征为基因从野生四倍体小麦流向六倍体小麦提供了证据。分子生物学杂志,2006;23(7):1386-96。
- 35.
Jordan KW, Wang S, Lun Y, Gardiner L-J, MacLachlan R, Hucl P, Wiebe K, Wong D, Forrest KL, Sharpe AG, Consortium IWGS。异源六倍体小麦的单倍型图谱揭示了同源基因组的独特选择模式。中国生物工程学报。2015;16:48。
- 36.
赵山,张伟,Akhunov E, Sherman J,马勇,罗明春,Dubcovsky J.美国小麦基因来源SNP标记多态性分析(小麦l .)品种。Mol Breed. 2009;23(1):23 - 33。
- 37.
gat BS, Long AD。连杆不平衡的内幕。植物学报。2003;15(7):1502-6。
- 38.
刘辉,周辉,吴勇,李霞,赵娟,左涛,张旭,张勇,刘松,沈勇,等。遗传关系与连锁失衡对基因组选择的影响。公共科学学报。2015;10(7):e0132379。
- 39.
闫娟,王晓燕,王晓燕,王晓燕,王晓燕。基于SNP标记的玉米遗传特性及连锁不平衡分析。科学通报。2009;4(12):e8451。
- 40.
Abdullaev AA, Salakhutdinov IB, Egamberdiev SS, Khurshut EE, Rizaeva SM, Ulloa M, Abdurakhmonov IY。遗传多样性、连锁不平衡和关联作图分析海岛棉l .种质。《公共科学图书馆•综合》.2017; 12 (11): e0188125。
- 41.
Oraguzie NC, Wilcox PL, Rikkerink EHA, de Silva HN。连锁不平衡。在:Oraguzie NC, EHA R, Gardiner SE, De Silva HN,编辑。植物中的关联映射。纽约,纽约:施普林格纽约;2007.p零部件。
- 42.
Marza F, Bai GH, Carver BF, Zhou WC。小麦群体Ning7840 × Clark产量及相关性状的数量性状位点理论与应用,2006;(4):688-98。
- 43.
王仁祥,海林,张晓霞,尤国祥,闫春春,肖世生。中国冬小麦群体禾尚麦X Yu8679籽粒灌浆率和产量相关性状的QTL定位。应用理论。2009;118(2):313-25。
- 44.
陈志伟,陈志伟,陈志伟,等。面包小麦籽粒蛋白质含量、产量和千粒重的遗传分析。应用物理学报,2003;106(6):1032-40。
- 45.
bosolini E, Wicker T, Knobel PA, Keller B.小草基因组同源位点的比较Brachypodium水稻:对小麦基因组学和草基因组注释的影响。植物J. 2007; 49:704-17。
- 46.
Acuna-Galindo MA, Mason RE, Subramanian NK, Hays DB。小麦适应干旱和高温胁迫相关QTL区域的元分析作物科学,2015;55(2):477-92。
- 47.
Lopes MS, Dreisigacker S, Pena RJ, Sukumaran S, Reynolds MP。小麦关联作图计划(WAMI)面板的遗传特征,用于解剖春小麦复杂性状。应用物理学报,2015;28(3):453-64。
- 48.
Pinto RS, Reynolds MP, Mathews KL, McIntyre CL, Olivares-Villegas J-J, Chapman SC.小麦群体抗热抗旱适应性QTL的研究。应用理论与实践。2010;21(6):1001 - 1001。
- 49.
张志强,张志强,张志强,等。基于基因组学的作物抗旱性研究。植物科学进展,2006;11(8):405-12。
- 50.
Cattivelli L, Rizza F, Badeck F- w, Mazzucotelli E, Mastrangelo AM, Francia E, Mare C, Tondelli A, Stanca AM。作物抗旱性的改善:从育种到基因组学的综合观点。农田作物,2008;105(1-2):1-14。
- 51.
王晓明,王晓明,王晓明,等。高粱抗旱性候选基因的筛选、鉴定和功能注释(高粱二色的(l)Moench)。BMC Genet 2017;18:119。
- 52.
Sairam RK, Saxena DC。小麦抗氧化应激与抗氧化剂基因型:耐水胁迫的可能机制。中国农业科学,2000;26(1):59 - 61。
- 53.
Antao CM, Malcata FX。植物丝氨酸蛋白酶:生化、生理和分子特征。植物化学学报。2005;43(7):637-50。
- 54.
张慧,毛晓霞,王春,景瑞。小麦常见基因TaSnRK2.8的过表达增强了小麦抗旱、耐盐和低温的能力拟南芥.《公共科学图书馆•综合》.2010; 5 (12): e16041。
- 55.
Nguema-Ona E, Vicre-Gibouin M, Gotte M, Plancot B, Lerouge P, Bardor M, Driouich A.细胞壁o -糖蛋白和n -糖蛋白的生物合成和功能。植物科学进展(英文版);
- 56.
小麦植物在胁迫下的抗氧化反应。中国生物医学杂志,2016;39(1):1 - 6。
- 57.
Karacic Z, Vukelic B, Ho GH, Jozic I, Sucec I, Salopek-Sondi B, Kozlovic M, Brenner SE, ludwigg - muller J, Abramic M.一种具有双重活性的新型植物酶:非典型Nudix水解酶和二肽基肽酶III。中国生物医学工程学报,2017;36(1):344 - 344。
- 58.
Dragwidge JM, Scholl S, Schumacher K, Gendall AR. nhx型Na+(K+)/H+反转运蛋白是TGN/EE转运和内体离子稳态所必需的拟南芥.细胞科学.2019年,132 (7):jcs226472。
- 59.
董超,王娟,陈强,于勇,李波。陆地棉产量及产量构成有利等位基因的关联作图检测。基因与基因组学。2018;40(7):725-34。
- 60.
Browning BL, Browning SR.对大数据集的三组和不相关个体进行基因型imputation和单倍型-相位推断的统一方法。王志刚,王志刚。2009;(2):10 - 10。
- 61.
团队R: RStudio: R的集成开发。RStudio, Inc,波士顿,马萨诸塞州URL http://www RStudio com2015年,42:14。
- 62.
王晓燕,王晓燕,王晓燕,等。基于多位点基因型数据的种群结构推断。遗传学。2000;155(2):945 - 59。
- 63.
白伯利,张哲,张文杰,张文杰,张文杰,张文杰。TASSEL:用于不同样本中复杂性状关联映射的软件。生物信息学。2007;23(19):2633 - 5。
- 64.
利AE,田F,王Q, Peiffer J,李米,布拉德伯里PJ,马戈尔,盾牌,张z GAPIT:基因组协会和预测集成的工具。生物信息学。2012;28(18):2397 - 9。
- 65.
王s, Wong D, Forrest K, Allen A, Chao S, Huang BE, Maccaferri M, Salvi S, Milner SG, Cattivelli L,等。利用高密度9万单核苷酸多态性阵列鉴定多倍体小麦基因组多样性。中国生物工程学报,2014;12(6):787 - 791。
- 66.
研究所S: Base SAS 9.4程序指南:统计程序:SAS研究所;2017.
致谢
我们非常感谢乌普萨拉大学生态与遗传系的Thomas Richards博士的建议和修改手稿。
资金
这项工作得到了德黑兰大学和伊朗国家科学基金会的支持。96008453)。资助机构参与了材料创作、研究设计、数据分析和撰写手稿。
作者信息
从属关系
贡献
YR:进行现场试验,分析数据集,并撰写手稿。MRB:协助数据分析和撰写稿件。AT:协助数据分析和修改稿件。HA和PKI:协助数据分析和撰写稿件。所有作者都对手稿进行了修改和编辑。所有作者均已阅读并认可最终稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。资助机构参与了材料创作、研究设计、数据分析和撰写手稿。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1表S1。
伊朗小麦品种的信息包括1942年至2014年发布的品种,以及1931年至1968年收集的地方品种
附加文件2表S2。
灌溉条件下伊朗小麦品种农艺性状的方差分析。表S3。雨养条件下伊朗小麦品种农艺性状的方差分析。表S4。在水分充足的条件下,两年以上伊朗小麦品种农艺性状之间的人的相关系数。表S5。在雨养条件下,两年以上伊朗面包小麦品种农艺性状相关性的Pearson置信度。
附加文件3图S1。
气候条件包括2016-17种植季和2017-18种植季田间试验点的降水和温度。图S2。A基因组不同染色体的LD衰变模式t . aestivum基于原始snp。图S3。B基因组不同染色体的LD衰变模式t . aestivum基于原始snp。图S4。D基因组不同染色体的LD衰变模式t . aestivum基于原始snp。
附加文件4表S6。
利用原始SNPs对暴露于充足水分条件下的伊朗小麦农艺性状的预期mta的描述。表S7。利用原始snp对雨养条件下伊朗小麦农艺性状的预期mta进行描述。表S8。利用估算的单核苷酸多态性(SNPs)对暴露于充足水分条件下的伊朗小麦种质农艺性状的预期mta进行描述。表S9。利用估算SNPs对雨养条件下伊朗小麦农艺性状的预期mta进行描述。
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
拉希米,Y.,比哈姆塔,m.r.,塔利伊,A.。et al。面包小麦农艺性状的全基因组关联研究为未来育种计划揭示了新的假定等位基因。BMC植物生物学19日,541(2019)。https://doi.org/10.1186/s12870-019-2165-4
收到了:
接受:
发表:
关键字
- 小麦
- 农艺性状
- GWAS
- 放在
- 基因注释