抽象的
背景
蔗糖合酶(SUS)被广泛认为是参与高等植物蔗糖代谢的关键酶,是作物库强度的生化标记。然而,尽管在表征SUS基因家族的生理功能方面取得了重大进展,但对该家族在高等植物中的进化过程和意义的了解仍然不完全。
结果
在该研究中,我们在19种植物物种中鉴定了100多种基因,并重建了它们的系统发育,呈现在高等植物中的SUS基因家族演化的潜在框架。三种古代分叉的SU基因亚脲(SU,II和III)基于它们的系统发育关系和血管植物中的独特内含子/外显子结构,并且发现它们在单子叶和双叶片中独立地发展。SUS基因的每个亚家族在各种植物中表现出不同的表达模式,这意味着在单焦点和奇特的分歧之前发生它们的功能分化。此外,SUS III基因在Dicots中的松弛净化选择下进化,并显示出狭窄的表达型材。另外,对于SUS基因的所有三个亚颗粒,GT-B结构域比“调节”结构域更加保守。
结论
本研究揭示了SUS基因家族在高等植物中的进化过程,为被子植物SUS基因的进化保护和功能分化提供了新的认识。
背景
蔗糖是高等植物光合作用的主要最终产物,从源叶向库器官输出。植物中的蔗糖分解代谢是世界上最大的代谢通量之一,在碳资源分配和糖信号启动中起着关键作用[1那2].蔗糖通过转α(INV)将葡萄糖和果糖或SUS水解,其催化蔗糖和尿苷二磷酸(UDP)至果糖和UDP-葡萄糖的可逆转化率[1那2那3.].有令人信服的证据表明,SUS在促进碳的进入非分水合成植物细胞的代谢以及测定作物物种中的水槽强度的作用。例如,rugosus4(Rug4.)豌豆的突变减少种子质量和淀粉含量[4.),shrunken1(sh1)玉米中的突变导致沉积的种子表型由于胚乳细胞壁整合的破坏[5.].对SU基因特异性同种型的反义抑制降低了番茄中幼果的果实环境和蔗糖卸载能力[6.]减少马铃薯块茎中的淀粉积累[7.]影响胡萝卜中纤维素和淀粉的生物合成[8.,并抑制棉花纤维细胞的起始、伸长和种子发育[9.].土豆的过度表达Sus4.基因增加淀粉,腺苷二磷酸二磷酸(ADP) - 葡糖和UDP-葡萄糖的水平和马铃薯的总产量[10.,提高玉米种子胚乳中淀粉和adp -葡萄糖含量[11.,减少种子败育,提高棉花纤维产量[12.].
SUS是由一个多基因小家族编码的高等植物。对单个物种SUS基因的研究表明,结构保守、表达和功能分化与基因家族进化密切相关。的拟南芥SUS基因家族仅包含6个表达谱不同但部分重叠的SUS基因[13.],他们的角色通过相应的敲除突变体进行了研究[14.].ATSUS1和ATSUS4.在缺氧条件下表现出显著的诱导作用,并且这两个基因的双突变体在水培培养中生长速度降低[14.].ATSUS5和ATSUS6.在韧皮部中特异表达,在胼胝质合成中具有特异功能[15.].Pea拥有一个SUS基因家族,至少包含三个不同的基因,即:Sus1.那Sus2.和Sus3.,这三种基因显示出不同器官和器官发展中的不同表达模式。这些基因,Sus1.显示组成型表达模式,在显影种子中高度表达,缺乏Sus1.活动Rug4.豌豆的突变体不能用Sus2.和Sus3.[16.].在棉花、杨树、柑橘和葡萄等其他植物中,不同SUS基因的组织特异性和发育依赖性表达模式表明,每个SUS基因可能具有特定的生理功能[17.那18.那19.那20.].然而,植物SUS基因在表达和功能上的差异是否发生在特定物种出现后,或者至少在一定程度上发生在被子植物的共同祖先中,目前尚不清楚。
尽管在确定SUS基因的生理功能和监管机制方面取得了令人信服的进步,但我们对高等植物中SUS基因家族进化过程的了解仍然不完整。分子遗传研究提供了对个体蛋白质的生理功能的实质性见解,而进化分析将阐明基因家族的起源和扩张史,并进一步向进化角度提供新的洞察力。几年来,我们对植物中SUS基因家族的演变的理解一直基于个体血症种类的调查[17.那20.那21.].因此,为了更好地了解高等植物SUS基因的进化,有必要在更大范围内进行全面的进化分析。在本研究中,我们选择了16种被子植物和3种裸子植物、蕨类植物和苔藓植物来研究当前的SUS基因是如何从原始被子植物SUS谱系中进化和分化出来的。我们对植物SUS基因的分类、基因复制、结构特征、选择压力和表达谱进行了研究。这些结果将为了解SUS基因的进化史以及进化差异对SUS基因功能多样性的影响提供基础参考。
结果
Angiosperms中SU基因的分类
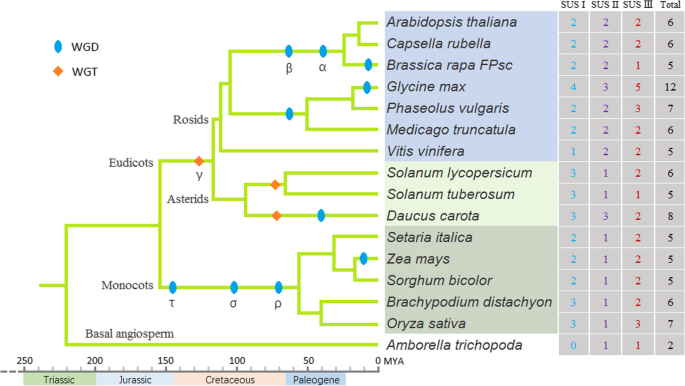
共有96个SUS基因在16个贪心植物种类中鉴定出10种Dicot植物,5种单焦植物和基础植物,Amborella trichopoda,使用隐马尔可夫模型(HMM)和BLASTP搜索(图。1).在调查的所有物种中,大豆拥有最多的SUS基因(12个),比观察到的多6倍A. Trichopoda.(2)其余14个物种均含有5 ~ 8个SUS基因。
本研究调查的16种被子植物占据了重要的系统发育位置,因为它们包括三个主要的被子植物谱系(单子叶植物、小行星和蔷薇植物)和一个基生被子植物谱系(图。1).为了了解SUS基因在被子植物中的系统发育关系,我们构建了16种被子植物中96个SUS基因的无根系统发育树。系统发育树将SUS基因家族清晰地划分为三个亚家族,分别为SUS I、II和III。每个亚科进一步聚为3个类群:单子叶类群、双子叶类群和基生被子植物类群。2).ATSUS5和ATSUS6.包含一个3 '扩展名[13.].在本研究中,几乎所有SUS III亚家族基因的蛋白质表现出C末端延伸(附加文件1图S1),表明被子植物的SUS III基因可能是从共同祖先进化而来的。
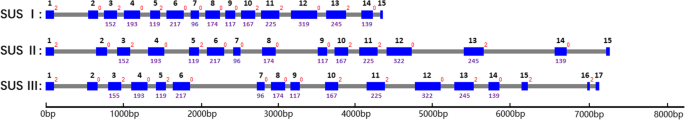
内含子/外显子装置通常被认为是基因文理中的重要参数[29.那30.].为了确认SUS基因的分类,我们在开始和止锁密码子之间分析了96个SUS基因的预测内含子/外显子结构(附加文件2:图S2)。基于内含子/外显子布置和外显子长度(附加文件6.:表S1)已经研究过的96个SUS基因,我们重建了Angiosperms的SU,II和III基因的拟议的古代内含子结构(图。3.).古SUS I和SUS II基因有15个外显子,而古SUS III基因有17个外显子,包含3 '延伸。3个古SUS基因的第3 ~ 14外显子长度高度保守。内含子缺失是被子植物中SUS古老基因后代普遍存在的现象,尤其是SUS I和SUS III基因的双子叶组。对于SUS I基因的双子叶组,内含子在第5和第6外显子之间和第12和第13外显子之间丢失。在SUS III基因的双子叶组中,内含子也在第12和第13个外显子之间丢失。内含子丢失仅发生在少数SUS II基因中(附加文件)2:图S2)。总的来说,SUS I、II和III基因具有不同的内含子/外显子排列。综上所述,我们对被子植物中96个SUS基因的系统发育和内含子/外显子结构的分析表明,在单子叶和双子叶分化之前,SUS基因有3个祖先亚家族。
被子植物中三个SUS基因的内含子/外显子结构。基于96个被子植物SUS基因的内含子/外显子结构和外显子长度(附加文件2:图S2;额外的文件6.:表S1),我们重建了服从中的三个祖先SU基因的内含子/外显子原理图。蓝色框表示编码区域内的外显子,连接它们的灰线表示内含子。我们使用了Sobic.010G072300.1,AMTR_V1.0_SCAFFOLD00106.5和AMTR_V1.0_SCOFFOLD000444444.21的内含子/外显子结构,以分别代表ACOISPERM祖先SU,II和III基因的内含子/外显子原理图。蓝框下的紫色数字代表了相应的保守外显子的尺寸(BP)。灰色线左上角的红色表示相应内含子的内部阶段。灰色线的长度不代表内含子的实际尺寸
高等植物中SUS基因的进化轨迹
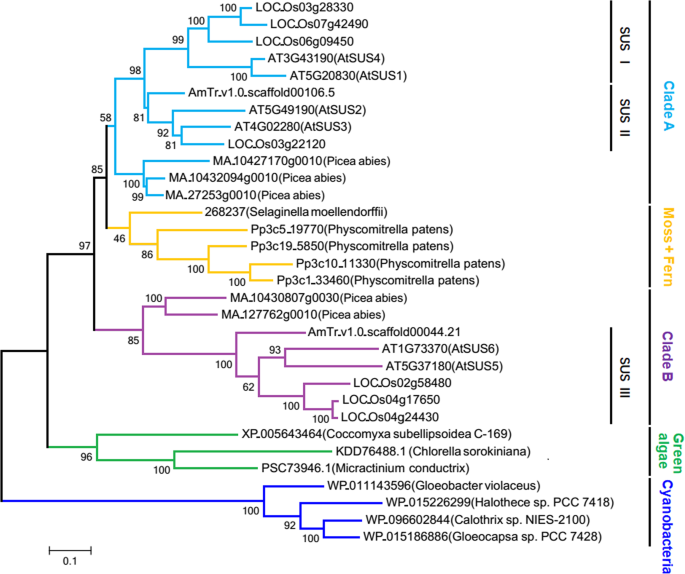
为了追踪SUS基因在高等植物中的进化,我们获得了25条同源序列Physcomitrella金属盘(苔藓),Selaginella Moellendorffii(蕨类植物),Picea amies(裸子植物)和3种Agiosperm(A. Trichopoda.那拟南芥蒂利亚纳和栽培稻).以7条来自蓝藻和绿藻的SUS序列为外群,构建系统进化树,推测SUS基因的进化轨迹。SUS基因在种子植物中有两个进化枝(进化枝A和进化枝B),都包含裸子植物分支和被子植物分支。SUS I和SUS II基因构成A枝的被子植物分支,SUS III基因构成B枝的被子植物分支(图2)。4.).在系统发育树中,分支A的SUS基因与苔藓和蕨类植物分支的SUS基因相近。4.),它们也有类似的内含子/外显子排列(图。3.;额外的文件3.图S3),说明进化枝A的基因比进化枝B的基因更保守。先前的研究揭示了祖先种子植物全基因组复制事件(ε)和祖先被子植物全基因组复制事件(ζ)分别发生在现存种子植物和现存被子植物多样化之前不久[31.].根据图1所示的系统发育关系。4.其中,SUS I和SUS II基因可能来源于原始被子植物WGD事件,而A和B支SUS基因可能来源于原始种子植物WGD事件。进化枝B的SUS基因的蛋白包含一个类似于苔藓植物和蕨类植物SUS蛋白的c端延伸4.:图S4)。而枝B的SUS基因则多了2个3 '端外显子,这在枝A、苔藓植物和蕨类植物中均未发现(图2)。3.;额外的文件3.:图S3),表明CLADE B SUS基因是种子植物中的一种新型SUS基因。
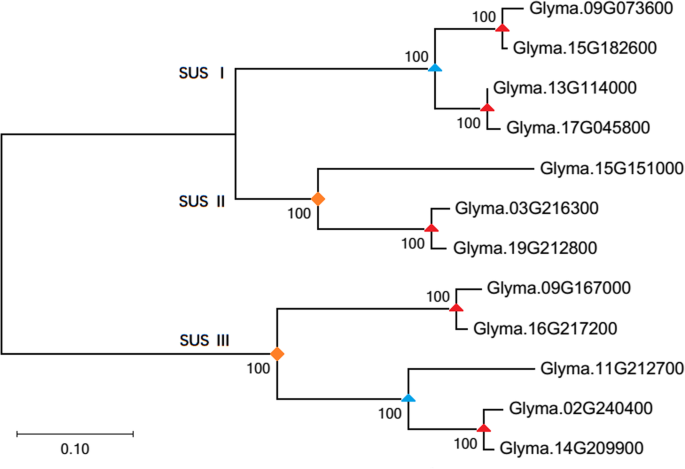
进一步推测了SUS基因在被子植物中的扩展模式。SUS基因的串联复制很少发生,而被子植物中SUS基因的扩增主要通过WGD进行。在单子叶植物中,一个WGD事件(τ)已经被推断出发生在它们从双子叶进化枝分化之后[32.]而且全基因组三次事件(γ)可能由所有核心销售分享[33.那34.那35.].一系列成功的WGDS与白垩纪 - 古烯(K-PG)边界相关联36.那37.].还有一些最近的WGDs似乎是在许多不同的植物谱系中独立发生的(图。1).在大豆中以SUS基因作为示例,“伽玛”事件,纸质谱系WGD事件和大豆谱系特异性WGD事件分别增加了SUS基因的数量2,2和5(图。5.;额外的文件7.:表S2)。由于大豆谱系特异性WGD事件中重复的SU基因的高保留比,大豆中SUS基因的数量几乎是其他物种的两倍(图。1).有趣的是,我们调查的五种单子叶植物中的SUS II基因中没有发现扩张;它们每个只包含一个SUS II基因,与在中的数字相同A. Trichopoda..
综上所述,在种子植物中,SUS基因有两个单系进化枝,而进化枝B的SUS基因可能是一个新的进化枝,因为它包含了另外两个3 '端外显子。在被子植物中,SUS基因有3个亚科,其中被子植物中SUS基因的扩展主要通过WGD进行,而草中SUS II基因尚未扩展。
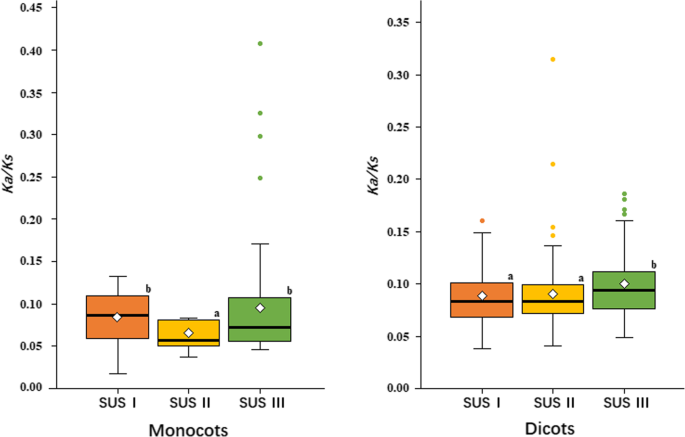
SUS III基因是在双子叶植物放松纯化选择下进化的
越来越多的证据表明,来自同一基因家族的不同亚家族可能会经历不同的纯化选择压力,形成相对固定的表达模式,具有不同的功能[38.那39.].为了确定SUS基因是否在Anuiaperms中的不同进化约束下,计算了计算SUS基因三个亚壳的每个同义位点(ω)值的同一位点的非纯类替换的总比率(图。6.;额外的文件8.:表S3)。SUS II基因的整体ω值(0.0630)显著低于SUS我基因(0.0842)和SUS三世在单子叶植物基因(0.0962),这表明,SUS II基因在单子叶植物净化选择强,可能是因为我们调查五单子叶植物只有一个SUS II基因(图。1).在双子叶植物中,SUS III基因的总ω值(0.0990)显著高于SUS I基因(0.0865)和SUS II基因(0.0889),表明SUS III基因受到了宽松的纯化选择,可能获得了新的功能。
SUS基因的表达谱
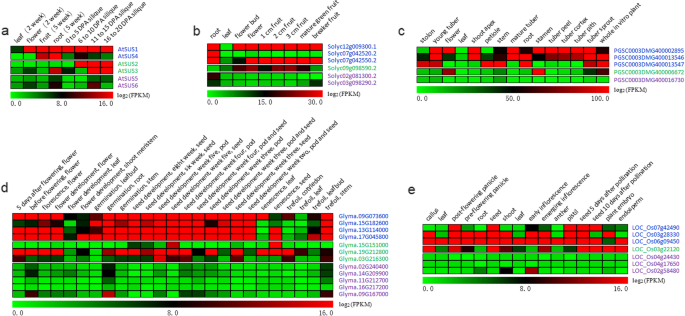
此外,我们研究了SUS基因的三个亚属植物的表达谱A. Thaliana.那g·马克斯那Solanum lycopersicum那茄属植物tuberosum和o.苜蓿从公开可用的RNA-seq数据(图。7.).在拟南芥那ATSUS5和ATSUS6.来自SUS III亚家族的基因仅在特定组织中表达,而SUS I (ATSUS1和ATSUS4.)及二级技能提升计划(ATSUS2.和ATSUS3)基因的表达量比SUS III基因的表达量大(图3)。7.a).在其他四种植物(西红柿、土豆、大豆和水稻;无花果。7.c)。这些表达数据表明,SUS III基因比SUS I和SUS II基因具有更多的组织特异性表达模式。此外,来自5个物种的SUS I基因在不同的营养和生殖器官中均有组成性表达(图1)。7.).
SUS基因表达谱。一种A. Thaliana.;B.S. lycopersicum.;C美国tuberosum;D.g·马克斯;E.o.苜蓿.来自五种物种的各种器官和发展阶段的转录数据从表达式ATLAS下载[40].不同颜色标记的基因名称表明这些基因来自不同的SUS亚家族。蓝色:SUS I亚科;绿色:SUS II亚家族;紫色:SUS III亚科。FPKM:每千碱基外显子模型的片段每百万读图
SUS基因在双子叶植物中的独特表达模式(图。7.)同意他们经历的不同选择压力(图。6.).SUS III基因在双子叶松弛纯化选择条件下进化,表现出组织特异性表达模式,而SUS I基因受到更大的进化限制,表现出更广泛的表达模式。有趣的是,虽然SUS III基因并没有在宽松的进化约束下进化,但与单子叶植物中的SUS I基因相比,水稻中的SUS III基因仍然表现出组织特异性的表达模式(图2)。7.e)。这些发现揭示了SUS I基因在维持细胞蔗糖代谢方面的保守功能,并且SUS III基因可能在高血管植物的演变中获得新功能。观察到蔗糖代谢的另一个基因的类似模式。中性/碱性反转酶基因显示宽或组成型表达模式,并且经历了比酸转化酶基因更大的进化约束,其表现出更多的组织特异性表达模式[38.].
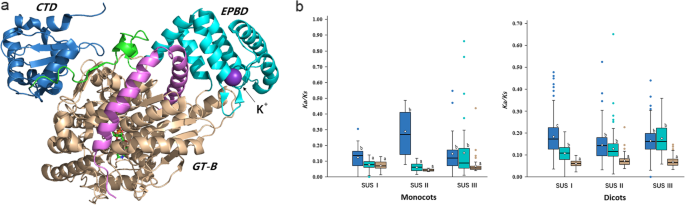
CTD、EPBD和GT-B域受到不同的选择压力
植物SUS多肽链包括一个细胞靶向结构域(CTD)、一个早期结节蛋白40 (ENOD40)肽结合结构域(EPBD)、一个典型的GT-B结构域和一个c端(图)。8.一种) [41.那42.].n端“调控”结构域,包括CTD和EPBD,参与细胞靶向,GT-B结构域参与糖基转移反应。6个SUS基因的一般动力学特性拟南芥,它们依赖于GT-B域,彼此之间关系密切[14.].根据代谢环境的不同,SUS通过与各种细胞器膜的相互作用,改变其细胞位置,从胞质到纤维素、胼胝质和淀粉生物合成的位置[43.那44.那45.[cytoskeletal actin [46.]通过其“监管”领域[41.].SUS的CTD,EPBD和GT-B域具有不同的功能,并且这些域也可能经历不同的选择压力。我们计算了单焦和平塔中SUS基因三种亚颗粒的ω值。为了我们的意外,SU基因所有三个亚壳的GT-B结构域的Ω值低于“调节性”域的GT-B结构域(图。8.c;额外的文件9.:表S4)。GT-B域的活动位点几乎是相同的,在三个亚家族内部和之间(附加文件5.:图S5)。因此,GT-B结构域比“调控”结构域更保守,受到更多的进化约束。
植物苏的结构与域演化。一种结构从AtSUS1A.thaliana(PDB代码3S27)[41.].结构域用不同的颜色突出显示:CTD是蓝色的;EPBD为青色;CTD和EPBD之间的连接子为绿色,GT-B结构域为小麦结构域,c端延伸子为粉色;B.单子叶和双斑点三种SU基因亚壳的CTD(蓝色),EPBD(青色)和GT-B(小麦)结构域的总体ω值。方框的上边沿和下边沿表示上、下四分位数,中央黑线表示中位数,菱形表示平均值。晶须延伸到盒子边缘外的四分位间距的1.5倍。方框上方的不同字母表示显着的差异(单程-Anova和Dunnett T3测试; P <0.05)
讨论
蔗糖广泛存在于生物中,包括蓝藻、单细胞藻类,尤其是高等植物。它通常是在盐或渗透胁迫下在蓝藻中合成的,被认为有助于维持渗透平衡[47.那48.].然而,在大多数植物中,蔗糖是光合作用的主要最终产物,蔗糖代谢在碳资源分配和糖信号的启动中发挥枢轴作用[1那2].蔗糖被SUS分解成udp -葡萄糖和果糖或者被INV分解成葡萄糖和果糖[49.].Wan等人已经阐明了三种INV的进化步骤和功能含义之间的关系。[38.].由于SUS基因在高等植物中的进化历史尚不完整,全面了解其进化轨迹、结构特征、表达谱和功能意义,将有助于优化碳资源配置,提高作物产量。
植物SUS基因的起源、进化与分类
SUS基因可能起源于变形菌纲或变形菌纲与蓝藻纲的共同祖先,植物可能是从蓝藻纲遗传而来[48.].通过对不同植物的全基因组测序,比较基因组方法已鉴定出大量的SUS基因,可用于研究植物SUS基因的起源、进化和分类。在本研究中,系统发育分析表明,植物的SUS基因形成了一个单系群,表明所有植物的SUS基因可能起源于一个共同的祖先[48.(图。4.).WGD或多倍体通常随后进行大量基因丧失和代级化,是植物中的常见现象[31.].保留的重复基因不仅为生物创新提供了必要的遗传物质,而且还引起了植物同源基因的多样性。来自种子植物的SUS基因形成了两个单系进化枝(进化枝A和进化枝B)。4.),可能是因为种子植物的祖先经历了WGD事件[31.],复制的SUS基因拷贝被保留。此外,在分支A中还发现了被子植物SUS基因的两个亚科(SUS I和SUS II)。4.)可能源于原始被子植物WGD事件[31.].根据系统发育关系,植物SUS基因历史上被分为三个主要亚科(Sus1, SUS A和New Group/NG) [13.那50.].我们对被子植物SUS基因的系统发育分析与这一分类相一致,我们将其分别命名为SUS I、II和III(图2)。2).此外,进化枝A的基因比进化枝B的基因更接近于原始类型植物的SUS基因,因为前者的基因在系统发育树中与苔藓植物和蕨类植物的SUS基因聚类(图2)。4.),具有与苔藓植物和蕨类植物SUS基因相似的古老内含子/外显子结构(图2)。3.;额外的文件3.:图S3)。相反,思工B基因可能是出现在种子植物中的新型。
被子植物SUS基因的每个亚科至少由两个独立的组组成,即单子叶组和双子叶组。2),这与被子植物中55个SUS基因的分类一致[51.].在其他涉及Anuiosperm Sus基因的分类的研究中,SUS II基因也由单子叶和双斑点的基因组成;然而,来自单焦点和双点的基因没有独立组织[17.那19.,这可能是因为所使用的单子叶SUS基因数量相对较少。我们的研究结果支持了单子叶和双子叶SUS基因独立进化的观点。在单子叶和双子叶分裂后,它们的祖先经历了特定的WGD事件[32.那33.那35.那52.].在随后的演变期间出现了许多物种,经验丰富的血统特定的WGD事件[36.].WGD事件和随后特定SUS基因的保留和丢失导致不同物种SUS基因的进化轨迹不同[17.那18.那20.].
在一定程度上,内含子/外显子结构允许我们预测SUS基因的可能起源和关系[13.那50.].在我们的研究中,高度保守的Anuiaperms的三个古老SUS基因的前14个外显子(图。3.),进一步表明SUS基因的所有三个亚属亚属可能来自共同的祖先[48.].三个SUS亚家族中内含子/外显子结构最明显的差异是,大多数SUS III基因在3 '端还有两个外显子(Additional file)2:图S2),中未在白细胞和Pteridophyte SU基因中发现(附加文件3.:图S3)。然而,SU III基因中的3'延伸的功能仍然未知。SUS基因的每个亚家族具有不同程度的内含子损失,并且在SU和III基因的双斑点中,第12和第13次外显子之间的内含子损耗(附加档案2:图S2) [13.那50.].SUS基因内含子损失的进化和功能性意义需要进一步研究。
SUS I基因可能在o中蔗糖新陈代谢的关键作用2- 缺货环境
蔗糖代谢对多细胞植物至关重要,可被SUS或INV降解;然而,这些酶在特定植物中的确切作用在很大程度上仍是未知的[2那15.].两个SUS和INV都显示为多个,不同的同种型。细胞质INV(CIN)基因是古老的,可能在维持细胞溶质糖稳态和细胞功能方面发挥关键作用。细胞壁INV(CWIN)和真空INV(VIN)基因受到松弛净化选择压力,并且CWIN基因已经与血管植物共束,这可能是韧皮卸载的功能成分[38.].在被子植物中,SUS基因可以被清晰地分为三个亚科。2)[13.那20.];然而,每个亚科的确切功能尚未阐明。我们推测SUS基因的每个亚家族可能具有不同的功能。
蔗糖通过SUS转化为己糖磷酸只需要INV转化所需ATP的一半,而在O型糖中,SUS途径被认为比INV途径更有效2- 可以限制ATP合成的缺陷环境[1那14.那15.].在单子叶植物和双子叶植物中,通过缺氧或缺氧诱导某些SUS基因是普遍存在的现象。的表达Sus1.和Sh1在玉米、SS1在小麦,和苏珊∗Dc1在低氧或缺氧条件下诱导或增强[53.那54.那55.[所有这些基因来自SUS I基因亚家族。同样,在A. Thaliana.,转录水平增加ATSUS1和ATSUS4.但缺氧下的其他SU基因(附加档案10.:表S5) [13.那56.[这两个基因的双突变体显示缺氧下显着的生长迟缓[14.].反义抑制黄瓜SU基因的抑制(CsSUS3)减低对缺氧压力的耐受性[57.].始终如一地,一些SUS基因长期被认为是用于沉降强度的生物化学标志物,特别是在代谢高度活性或笨重的器官中,其中内源性氧气水平可能是低的。例如,玉米的突变缺乏Sus1.要么Sh1导致降低淀粉含量[5.],而反义抑制特定的SUS基因大大减少了马铃薯块茎中的淀粉积累[7.],抑制棉花纤维伸长和种子发育[9.].此外,我们研究了与水槽强度相关的SU基因,发现几乎所有这些来自SUS I的基因(表1).因此,我们推测SUS I基因而不是SUS II和III基因负责O细胞中蔗糖的转化2- 进行环境。符合此视图,单倍型协会显示两个SUS I基因(TaSus1和TaSus2)来自小麦与千粒重量相关,主要取决于淀粉合成的速率和量[58.那59.].SUS I基因显示不同植物和生殖器官中的组成型表达(图。7.),在正常氧水平下,它们在蔗糖代谢中的作用可被INV基因替代[14.].然而,在氧气含量不足的情况下,SUS I基因是不可替代的。
SUS II和SUS I基因具有相似的古老内含子/外显子结构,可能来源于原始被子植物WGD事件。这两个SUS亚家族的基因表达谱不同。7.)[13.,表明它们的功能可能经历了一定程度的分化。例如,两个SUS II基因来自拟南芥,即ATSUS2.和ATSUS3,没有响应o引起的2缺乏,这两个基因的双突变体与野生型(WT)控制没有明显不同,尽管这两个基因在种子中强烈表达[13.那14.].江等人。[60.]报道了一个棉花SUS II基因(表1),GhSusA1,这与生产力密切相关,作为水槽强度的关键调节器,表明某些SUS II基因可能与特定植物中的SUS I基因具有功能重叠。
SUS III基因表现出狭窄的表达谱,尽管它们的功能尚不清楚
SUS III基因具有不同于SUS I和SUS II基因的内含子/外显子结构2:图S2),显示一个狭窄的表达式轮廓(图S2)。7.),并在双子叶中受到宽松的净化选择压力(图。6.),提示SUS III基因可能具有不同于SUS I和SUS II基因的功能。Wan等人[38.]报道了CWIN基因在高等植物中出现并表现出组织特异性表达模式,可能是与高等植物维管发育共同进化的。两个拟南芥SUS III基因,即ATSUS5和ATSUS6.,仅在特定的组织和器官中表达(图。6.),由这两个基因编码的蛋白质特别存在于韧皮部中[15.].这两个基因的双突变体在筛板孔内壁显示出比野生型植物更薄的胼胝质层。这两个SUS III基因被认为参与筛板中胼胝质的形成[15.].因此,我们认为SUS III基因可能也参与了高等植物维管发育过程。
植物SUS基因的进化保守与分化
如上所述,所有植物SUS基因可能从共同的祖先演变。在迄今为止迄今为止的植物物种中,SUS由小型多岛家族编码。对SU基因的三个亚壳的内含子/外显子结构的比较筛查确实揭示了内含子的数量和位置在高管培养中高度保守,尽管在特定的SU基因中丢失了一些内含子(图。3.;额外的文件2:图S2)。此外,AtSUS1的GT-B域的16个活性位点在被子植物SUS基因中几乎完全相同[41.](附加文件5.:图S5),GT-B结构域经历了强大的净化选择(图。8.).此外,来自不同亚属的SUS的同种型拟南芥具有相似的动力学性质[14.].以上结果表明,SUS基因在植物中具有结构和功能上的保守性。因此,我们推测不同的SUS基因可能在不同的细胞类型或细胞器中,在不同的发育阶段或不同的胁迫条件下,发挥相似的功能。我们的分析表明,被子植物SUS基因的三个亚科表现出不同的表达谱,这些表达谱可能是在单子叶和双子叶分化之前形成的。一般来说,每个亚家族中源自WGD事件的同源或复制的SUS基因继承了其祖先的表达模式(图。7.).此外,SUS根据代谢环境改变其细胞位置,通过与各种细胞器膜和细胞骨架肌动蛋白的相互作用,参与纤维素、胼胝质和淀粉的生物合成[41.].SUS基因的时空表达的特异性和蛋白质亚细胞定位的变异性决定了其功能的多样性。识别单个SUS基因的功能是一项挑战,这不仅是因为重复的SUS基因之间存在功能冗余[14.]但也因为vin基因可以部分功能替代SUS基因[15.].
结论
Angiosperm SUS基因家族可以基于它们的系统发育关系和独特的内含子/外显子结构分为三种亚属(SUS I,II和III),并且发现它们在单子叶和双叶中独立地发展。SUS基因的每个亚家族在各种植物中表现出明显的表达模式,并且SUS III基因在Dicots中的松弛净化选择下进化并显示出狭窄的表达型材。这项工作应该为了解SUS基因的进化史以及进化分歧如何导致SUS基因的功能多样性的基础。
材料和方法
数据采集
的基因组序列、注释和基因模型A. Thaliana.那危害风疹那Brassica Rapa.那g·马克斯那Pinguicula寻常魅力那Medicago Truncatula.那vitis Vinifera那S. lycopersicum.那美国tuberosum那胡萝卜胡萝卜那Setaria italica那Zea Mays.那高粱双色那Brachypodium distachyon那o.苜蓿那A. Trichopoda.那美国moellendorffii那p .金属盘, 和Coccomyxa subellipsoidea均采自Phytozome (https:///phytozome.jgi.doe.gov/pz/portal.html.).数据p .冷杉属从Congenie下载(http://congenie.org/).AtSUS1的四聚体结构来源于A. Thaliana.是从结构生物信息学(RCSB)蛋白质数据库(PDB代码3S27)的研究协作中获得的。
鉴定SUS基因
我们结合HMM和BLASTP搜索来确定19个物种的SUS基因。首先,从Pfam网站(http://pfam.xfam.org/),然后利用这两个HMM谱图作为查询,使用HMMER (V3.0)软件识别所有可能的SUS基因。二是六种氨基酸的序列阿斯图斯基因[13.), 7OsSUS基因[61.使用阈值期望值设定为1E-10的阈值期望值来运行BLASTP搜索以针对每个物种中的所有蛋白质序列。从HMM和BLASTP搜索获得的所有命中都被合并在一起,删除了冗余命中。最后,所有候选序列都进行了在线PFAM分析(http://pfam.xfam.org/),进一步证实它们同时具有SUS结构域和糖基转移酶1组结构域。去除缺少SUS结构域或糖基转移酶1组结构域的蛋白序列。
系统发育和基因结构分析
使用ClustalW对氨基酸序列进行比对,手工去除缝隙和未对齐的切片。使用MEGA v7.0中对齐的氨基酸序列,采用最大似然(ML)方法进行系统发育树重建。参数如下:模型,WAG;救赎,1000复制;缺失数据,部分删除。从GFF文件中分析了SUS基因的结构,并利用植物内含子外显子比较与进化(PIECE) (https://wheat.pw.usda.gov/piece/gsdraw.php.).
估计K.一种/K.S.比率
使用基于氨基酸序列的CLUSTALW对准同源基因对的密码子序列。每种非同义网站的非同义替换的比率(K.一种)为每个同义地点的同义替换(K.S.)(ω值),使用DnaSP version 5计算。饱和效应是通过丢弃基因对来避免的K.S. > 2. Theω值通常被认为是蛋白质水平上选择的量度,值为ω > 1, =1 and < 1 indicating positive selection, neutral evolution and negative or purifying selection, respectively.
数据和材料的可用性
支持本手稿的结论的数据集包含在文章及其附加文件中。
缩写
- ADP:
-
二磷酸腺苷
- ATP:
-
三磷酸腺苷
- CIN:
-
细胞质INV.
- 仪:
-
细胞目标域
- CWIN:
-
细胞壁发票
- 与:
-
果糖早期40
- EPBD:
-
与peptide-binding域
- FPKM:
-
每千碱基外显子模型的片段每百万读取映射
- 唔:
-
隐藏的马尔可夫模型
- inv:
-
逆变酶
- ML:
-
最大似然
- mya:
-
百万年前
- PDB:
-
蛋白质数据银行
- SUS:
-
蔗糖合成酶
- UDP:
-
尿苷二磷酸酯
- 文:
-
空泡的发票
- 摇:
-
Whelan和高盛
- WGD:
-
全基因组重复
- 重量:
-
全基因组三倍
- WT:
-
野生型
参考文献
- 1。
KOCH K.蔗糖新陈代谢:糖感测和植物发育中的监管机制和关键作用。CurrOp植物BIOL。2004; 7:235-46。
- 2。
阮yl。蔗糖新陈代谢:碳酸多样化和糖信号的网关。Annu Rev植物Biol。2014; 65:33-67。
- 3.
陈志强,陈志强,陈志强,等。植物中氨基葡萄糖的合成机制。植物生态学报。2010;29:191-203。
- 4.
Craig J,Barratt P,Tatge H,DéjardinA,Handley L,Gardner CD,Barber L,Wang T,Hedley C,Martin C等人。突变在这方面Rug4.该基因座通过对蔗糖合酶的影响改变豌豆植株的碳氮代谢。植物j . 1999; 17:353 - 62。
- 5。
Chourey PS,Taliercio EW,Carlson SJ,Ruan Yl。遗传证据表明玉米胚乳中存在的蔗糖合成酶的两种同工酶至关重要,一种用于细胞壁完整性,另一个用于淀粉生物合成。Mol Gen Genet。1998年; 259:88-96。
- 6。
D'aoust Ma,yelle s,nguyen-quoc b.番茄水果蔗糖合成酶的反义抑制降低了果实的果实设置和蔗糖卸载能力。植物细胞。1999; 11:2407-18。
- 7。
关键词:马铃薯,蔗糖合酶,库强度茄属植物tuberosuml .)。工厂J. 1995; 7:97-107。
- 8。
胡萝卜中蔗糖合酶的反义抑制(胡萝卜胡萝卜L.)影响生长而不是蔗糖分区。植物mol biol。1999; 41:465-79。
- 9。
抑制蔗糖合酶基因表达抑制棉纤维细胞起始、伸长和种子发育。植物细胞。2003;15:952 - 64。
- 10。
Baroja-Fernández E, Muñoz FJ, Montero M, Etxeberria E, Sesma MT, Ovecka M, Bahaji A, Ezquer I, Li J, Prat S, et al. .提高转基因马铃薯蔗糖合酶活性(茄属植物tuberosum结果表明,马铃薯块茎中淀粉、adpg和udpg含量增加,总产量增加。acta botanica yunnanica(云南植物研究). 2009;
- 11.
李茹,巴洛哈 - 费尔尼德斯e,bahaji a,muñozfj,ovecka m,montero m,sesma mt,alonso-casajúsn,almagro g,sánchez-lópezam,等。增强蔗糖合成酶活性导致玉米中淀粉和ADP-葡萄糖的水平增加(Zea Mays.l .)种子胚乳。acta botanica yunnanica(云南植物研究). 2013;54:282-94。
- 12.
徐姆,布里尔e,llewellyn dj,furbank rt,阮yl。棉花加速叶片膨胀,降低种子流产,增强纤维生产的棉花蔗糖合成酶基因的过度表达。莫尔植物。2012; 5:430-41。
- 13。
王志强,王志强,王志强,等。蔗糖合酶多基因家族的结构和表达谱拟南芥.J Exp Bot. 2004; 55:397-409。
- 14。
Bieniawska Z, Paul Barratt DH, Garlick AP, Thole V, Kruger NJ, Martin C, Zrenner R, Smith AM。拟南芥蔗糖合酶基因家族的分析。植物j . 2007; 49:810-28。
- 15.
Barratt DH, Derbyshire P, Findlay K, Pike M, Wellner N, Lunn J, Feil R, Simpson C, Maule AJ, Smith AM。正常的生长拟南芥需要细胞溶溶剂转化酶但不是蔗糖合成酶。Proc Natl Acad Sci U S A. 2009; 106:13124-9。
- 16。
王天良,王志强,王志强,等。大豆蔗糖合酶基因的克隆与表达分析。植物杂志。2001;127:655 - 64。
- 17。
关键词:棉花,蔗糖合酶,基因家族,结构,系统发育,表达模式BMC Plant Biol. 2012;12:85。
- 18。
伊斯兰教MZ,Hu XM,Jin LF,刘yz,彭萨。柑橘蔗糖合成酶基因的基因组鉴定和表达谱分析:糖积累调控中可能作用的调查。Plos一个。2014; 9:E113623。
- 19。
x,陈z,王俊,叶m,ji l,王j,廖w,ma h.识别与表征杨树蔗糖合成酶基因家族。基因。2014; 539:58-67。
- 20.
朱X,王M,李X,九氏,王建,方j。葡萄蔗糖合成酶基因家族的基因组分析(vitis Vinifera):结构,进化和表达配置文件。基因(巴塞尔)。2017; 8:111。
- 21.
关键词:蔗糖合酶,基因家族,基因结构,系统发育,表达谱Theobroma可可l .)。J Genet。2015; 94:461-72。
- 22.
被子植物系统学。开花植物目和科被子植物系统发育分类的最新进展:APG IV. Bot J Linn Soc. 2016; 181:1-20。
- 23.
Iorizzo M,Ellison S,Senalik D,Zeng P,Satapoomin P,Huang J,Bowman M,Iogene M,Sanseverino W,Cavagnaro P等人。高质量的胡萝卜基因组组件为类胡萝卜素积累和分子基因组进化提供了新的见解。NAT Genet。2016; 48:657-66。
- 24.
马铃薯基因组测序联盟。块茎作物马铃薯的基因组序列和分析。NAT。2011; 475:189-95。
- 25.
Schmutz J,Cannon SB,Schlueter J,MA J,Mitros T,Nelson W,Hyten DL,Song Q,Thelen JJ,Cheng J,等。古代聚丙烯大豆的基因组序列。NAT。2010; 463:178-83。
- 26.
番茄基因组联盟。番茄基因组序列提供了肉质果实演化的见解。NAT。2012; 485:635-41。
- 27。
Kumar S,SteCher G,Tamura K. Mega7:用于更大数据集的分子进化遗传学分析版本7.0。mol Biol Evol。2016; 33:1870-4。
- 28。
基于最大似然方法的蛋白质进化的一般经验模型。Mol Biol Evol. 2001; 18:691-9。
- 29。
Sánchez D, Ganfornina MD, Gutiérrez G, Marin A. Lipocalin基因家族的外显子-内含子结构与进化。生物化学学报2003;20:775-83。
- 30.
邵寨,薛耶,吴P,张扬,吴y,恒y,王b,陈杰克。高级核苷酸结合位点 - 富含富含含量的富含进化模式的古代分散的课程的大规模分析。植物理性。2016; 170:2095-109。
- 31。
Jiao Y, Wickett NJ, Ayyampalayam S, Chanderbali AS, Landherr L, Ralph PE, Tomsho LP, Hu Y, Liang H, Soltis PS, et al.;种子植物和被子植物的祖先多倍体。Nat。2011;473:97 - 100。
- 32。
焦勇,李建军,唐华,李建军。综合共线和系统基因组分析揭示了一个古老的基因组复制在单子叶植物。植物细胞。2014;26:2792 - 802。
- 33。
Jaillon O, Aury JM, Noel B, Policriti A, Clepet C, Casagrande A, Choisne N, Aubourg S, Vitulo N, Jubin C, et al.;葡萄基因组序列表明主要被子植物门的祖先六倍体化。Nat。2007;449:463-7。
- 34。
唐华,王旭,Bowers JE, Ming R, Alam M, Paterson AH。通过多重排列的被子植物基因图谱揭示古代的六倍性。基因组研究》2008;18:1944-54。
- 35。
Jiao Y, Leebens-Mack J, Ayyampalayam S, Bowers JE, McKain MR, McNeal J, Rolf M, Ruzicka DR, Wafula E, Wickett NJ,等。与核心双二子体早期多样化相关的基因组三倍化。基因组医学杂志。2012;13:R3。
- 36。
Vanneste K,Baele G,Maere S,Van de Peer Y. 41植物基因组的分析支持与白垩纪 - 古近边界相关的成功基因组重复浪潮。Genome Res。2014; 24:1334-47。
- 37。
范德培。恐龙与恐龙的演化:K/Pg边界的演化。植物生态学报2016;30:62-9。
- 38。
万H,吴L,杨y,周g,阮yl。蔗糖新陈代谢的演化:倒置酶和超越的二分法。趋势植物SCI。2018; 23:163-77。
- 39。
徐X,冯y,方s,xu j,王x,guo w。β1,3-葡聚糖酶基因家族的基因组鉴定Gossypium通过比较分析。Sci众议员2016;6:29044。
- 40.
et al., et al., et al., et al.等。表达图谱更新——人类、动物和植物中基因和蛋白质表达的综合数据库。核酸Res. 2016;44: D746-52。
- 41.
郑义,安德森,张烨,加拉夫托罗姆。蔗糖合成酶-1的结构拟南芥蒂利亚纳及其功能含义。J Biol Chem。2011; 286:36108-18。
- 42.
蔗糖合酶:一种用于生物催化糖基化过程的糖基转移酶。Biotechnol放置2016;34:88 - 111。
- 43.
高等植物蔗糖代谢的调控:关键酶的定位和活性调控。生物化学学报2000;35:53 - 89。
- 44.
Etxeberria E,Gonzalez P.蔗糖合成酶的脊髓瘤蔗糖合成酶的探测及其潜在的载体从液泡中培养。J Exp Bot。2003; 54:1407-14。
- 45.
玉米蔗糖合酶的线粒体定位和信号转导功能。J Biol Chem. 2006; 281:15625-35。
- 46.
邓肯卡,Huber Sc。通过蔗糖浓度和磷酸化调节蔗糖合酶寡聚化和F-actin关联。植物细胞生理。2007; 48:1612-23。
- 47.
盐诱导的蔗糖积累是由蓝藻中蔗糖-磷酸合酶介导的。acta botanica sinica(植物学报)1999;
- 48.
卢恩我。蔗糖合成的进化。植物杂志。2002;128:1490 - 500。
- 49.
王莉,阮玉玲,王莉,王莉,等。蔗糖韧皮部的调控及其对作物产量和粮食安全的影响。J Exp Bot. 2014; 65:1713-35。
- 50.
小松A,Moriguchi T,Koyama K,Omura M,Akihama T.柑橘中蔗糖合成酶基因的分析表明不同的作用和系统发育关系。J Exp Bot。2002; 53:61-71。
- 51.
张丹,徐斌,杨晓霞,张震,李斌杨树:结构,表达和进化。树遗传学基因组织。2010; 7:443-56。
- 52.
汤,鲍德·杰,王X,帕特森啊。Angiosperm基因组比较揭示了单子地域谱系中的早期多倍体。Proc Natl Acad Sci U S A. 2010; 107:472-7。
- 53.
关键词:干旱胁迫,干旱胁迫,小麦,蔗糖合酶基因,抗旱性基因。1990;88:167 - 72。
- 54。
Zeng Y,Wu Y,Avigne Wt,Koch Ke。缺氧和缺氧的糖敏感蔗糖合成酶的差异调节表示互补的转录和后颅面反应。植物理性。1998年; 116:1573-83。
- 55。
胡萝卜中两种蔗糖合酶基因的组织特异性表达(胡萝卜胡萝卜l .)。植物学报1999;39:349-60。
- 56。
Klok EJ, Wilson IW, Wilson D, Chapman SC, Ewing RM, Somerville SC, Peacock WJ, Dolferus R, Dennis ES。拟南芥根培养中低氧反应的表达谱分析。植物细胞。2002;14:2481 - 94。
- 57。
Wang H, Sui X, Guo J, Wang Z, Cheng J, Ma S, Li X, Zhang Z.Cucumis sativus蔗糖合酶3 (CsSUS3)减少缺氧应力耐受性。植物细胞环境。2014; 37:795-810。
- 58。
江Q,侯j,郝c,王l,ge h,dong y,zhang x.小麦(T. Aestivum.)蔗糖合酶2基因(TaSus2)活跃于胚乳的发展与产量特征有关。Funct综合基因组学。2011; 11:49-61。
- 59。
侯军,姜强,郝超,王勇,张慧,张欣。小麦蔗糖合酶单倍型的全球选择。植物杂志。2014;164:1918-29。
- 60.
江y,郭文,朱h,阮yl,张t。过度表达GhSusA1增加植物生物量,提高棉纤维产量和质量。生物技术学报。2012;10:301-12。
- 61.
Cho J-I, Kim H-B, Kim C-Y, Hahn T-R, Jeon J-S。水稻蔗糖合酶复制基因的鉴定与特性分析OsSUS5和OsSUS7它们与质膜有关。摩尔细胞。2011;31:553 - 61。
致谢
感谢张永霞、刘清泉、王银杰和王伟林对我们研究工作的宝贵帮助和意见。
资金
该工作得到了国家自然科学基金(31701497)的支持,江苏省植物资源研究和利用重点实验室(JSPKLB201801和JSPKLB201832)(中国)。资助者在研究设计,数据收集,分析和解释或稿件的准备方面没有作用。
作者信息
从属关系
贡献
XX和Hy设计了研究。XX,YY,CL,YS,TZ和MH进行了实验并分析了数据。xx,sh和hy写了稿件。所有作者都阅读并批准了最后一篇论文。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:
图S1。SUS基因c端氨基酸比对。
附加文件2:
图S2。内含子/外显子结构组织的Anviosperms Sus基因。蓝色框表示编码区域内的外显子,连接它们的灰线表示内含子。其他颜色的盒子代表融合的外显子,上面的数字表明了哪些外显子被融合。红色盒子表示外显子的分裂。由于SUS III亚家族基因的3'末端的复杂性,我们没有显示出外显子的融合。
附加文件3:
图S3。SUS基因的内含子/外显子结构组织p .金属盘和美国moellendorffii.蓝色框表示编码区域内的外显子,连接它们的灰线表示内含子。灰色线左上角的红色表示相应内含子的内部阶段。其他颜色的盒子代表融合的外显子,上面的数字表明了哪些外显子被融合。
额外的文件4:
图S4。来自SUS基因的C-末端氨基酸对准A. Thaliana.那o.苜蓿那p .金属盘和美国moellendorffii。
额外的文件5:
图S5。SUS基因GT-B结构域的氨基酸比对。在AtSUS1的GT-B结构域中鉴定到的16个活性位点(His-287, Gly-302, Gly-303, Gln-304, Arg-382, His-438, Met-578, Arg-580, Gln-648, Asn-654, Glu-675, Phe-677, Gly-678, Leu-679, Thr-680, Glu-683)呈橙色。
额外的文件6:
表S1。外显子长度(BP)Anuiosperms Sus基因的统计数据。
额外的文件7:
表S2。Ks亚家族内SUS基因对的值g·马克斯.
额外的文件8:
表S3。的估计Ka / Ks(ω)值SUS基因。
额外的文件9:
表S4。的估计Ka / Ks(ω)SU基因三个域的值。
附加文件10:
表S5。的表达模式拟南芥缺氧下的SUS基因。6个SUS基因的表达数据拟南芥来源于GEO数据集(GSE119327)。我们分析了6个SUS基因的表达拟南芥在缺氧条件下发现ATSUS1和ATSUS4.缺氧诱导。adj。P.Val:P.-多次测试调整后的值。logFC:两个实验条件之间log2倍的变化。
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
徐,X.,Yang,Y.,Liu,C。et al。高等植物蔗糖合酶基因家族的进化史。BMC植物BIOL.19,566(2019)。https://doi.org/10.1186/s12870-019-2181-4
已收到:
公认:
发表:
关键字
- 蔗糖合成酶
- 基因家族
- 进化
- 种系发生
- 表达式模式