摘要
背景
盐度是限制棉花产量的最重要的环境因素之一。然而,盐碱地棉花减产的主要遗传因素尚不清楚。
结果
在这里,我们评估了皮棉产量的三个主要组成部分,单铃重(SBW),皮棉率(LP)和单株铃数(BNPP)g .分子在四种盐条件下,在两年内加入。表型分析表明,LP在不同盐分条件下没有变化,但BNPP显著降低,SBW在高盐条件下略有增加。基于57,413个高质量单核苷酸多态性(snp)和全基因组关联研究(GWAS)分析,分别鉴定出42个、91个和25个稳定数量性状位点(qtl)。表型和QTL分析表明,3个性状间相关性不大。在4种不同的盐条件下,LP同时检测到8个稳定的qtl,而SBW和BNPP的重复qtl较少。基因本体(Gene Ontology, GO)分析表明,它们的调控机制也存在较大差异。通过转录组谱数据,我们检测到8个稳定LP qtl中的10个基因在纤维发育期间主要表达。此外,单倍型分析发现MYB基因(GhMYB103),在顺式调控区和编码区存在两个SNP变异,与皮棉率显著相关,表明在皮棉产量中起着至关重要的作用。我们还从BNPP qtl中鉴定出40个候选基因是盐诱导的。在高盐条件下生长的植物中,与碳水化合物代谢和细胞结构维持相关的基因丰富,而与离子运输相关的基因在低盐条件下生长的植物中活跃,这表明高盐和低盐条件下BNPP的调控机制不同。
结论
该研究为阐明棉花耐盐机制奠定了基础,为培育高产耐盐旱棉品种提供了基因资源。
背景
盐度是限制作物生产力的最重要的环境因素之一[1]。盐胁迫影响全球约10亿公顷的干旱和半干旱地区[2]并且由于气候变化、不科学的灌溉和过度施肥而日益严重[3.]。科学家和农学家花了很大的努力来改进和提高盐碱地的利用率。除了利用物理和化学方法降低含盐量外,利用现代分子手段筛选或选育高耐盐作物是解决现状的经济有效途径。
在过去的几十年里,一些方法,如分子标记[4]、转基因技术[5]、转录组测序[6],以及基于单核苷酸多态性(SNP)的全基因组关联研究(GWAS) [7],已被用于研究植物耐盐机制和挖掘植物的精英等位基因。GWAS能够有效地将基因型与表型关联起来,并利用自然群体同时检测许多天然等位基因变异,因此在植物中得到了广泛的应用。在水稻中,Shi等人(2017)通过筛选478份材料的6361,920个snp,报道了11个与活力指数(VI)和平均发芽时间(MGT)的胁迫敏感性指数(ssi)相关的位点,这在未来的分子辅助育种中具有潜在的价值。8]。棉(Gossypium spp。)由于耐盐性高,可作为盐碱地的先锋作物进行土壤复垦[9]。然而,它的生长和发育仍然会受到不利的盐条件的影响。土壤含盐量8 ~ 18ds /m导致棉花产量损失15% ~ 55% [10]。因此,发现棉花耐盐反应通路的限制因子,鉴定相关基因是提高棉花产量的有效途径。
许多决定棉花纤维产量和质量等性状的候选基因[11,12,13,14]、籽油成分及蛋白质含量[15,已被揭露。此外,还对棉花的抗逆性进行了一些研究。利用145个SSR标记,检测到10个耐盐相关性状的60个qtl [4]。通过GWAS和RNA-seq分析,鉴定出33个与棉花耐盐性综合评价值相关的显著snp [9]。通过基因分型测序(GBS)的全基因组SNP分析,共鉴定出10个与盐相关性状的66个qtl,其中12个候选基因可能在棉花耐盐性中起关键作用[j]。16]。利用CottonSNP63K基因阵列,发现陆地棉苗期耐盐相关性状(相对成活率和耐盐水平)与7个基因组区域共有23个snp显著相关[j]。7]。利用基因组重测序数据,报道了9个与盐条件下亚洲棉相对鲜重、相对茎长、相对含水量和耐盐综合指数相关的SNP富区[j]。17]。一些研究集中在耐盐相关性状上,然而,很少有报道研究盐胁迫等胁迫条件下的纤维产量,尽管这对盐碱地的利用具有更大的实用价值。
在本研究中,我们研究了316株棉花产量的三个主要组成部分,单铃重(SBW)、衣分(LP)和单株铃数(BNPP)g .分子在四种不同的盐条件下,以不同的来源加入两年。不同盐环境下皮棉产量3个组成部分的表型变异表明,BNPP是导致单株皮棉产量降低的主要限制因素。此外,利用57,413个snp进行GWAS分析,鉴定与LP、SBW和BNPP相关的qtl和候选基因。在本研究中,系统地阐明了LP是最稳定的性状,BNPP最容易受盐田条件的影响。该研究结果为盐胁迫机制的研究提供了新的思路,并为提高棉花耐盐性提高铃数提供了新的思路。
结果
盐条件下衣棉产量各组成部分的表型变异
对316份陆地棉材料进行了SBW、LP和BNPP测定(附加文件)1表1)在四种不同的盐条件下生长(附加文件)2:图S1A和附加文件3.表2)。SBW、LP和BNPP性状的平均值在5.28 ~ 6.84 g之间4:表S3), 37.89至40.39%(附加文件5:表S4)及5.01至7.74(附加文件)6:表S5)。BNPP变异系数最大(22.53 ~ 48.08%),LP最小(8.15%~ 12.68%)。为了减小环境误差,我们进一步评估了在相同盐分条件下3个性状的最佳线性无偏预测(BLUP)值。BLUP值显示SBW分布在5~7 g(附加文件)2:图S1B)和LP在35~45%(附加文件2:图S1C)。但在a、B盐条件下,BNPP差异较大,分别为3~7,C、D盐条件下为5~92:图S1D)。
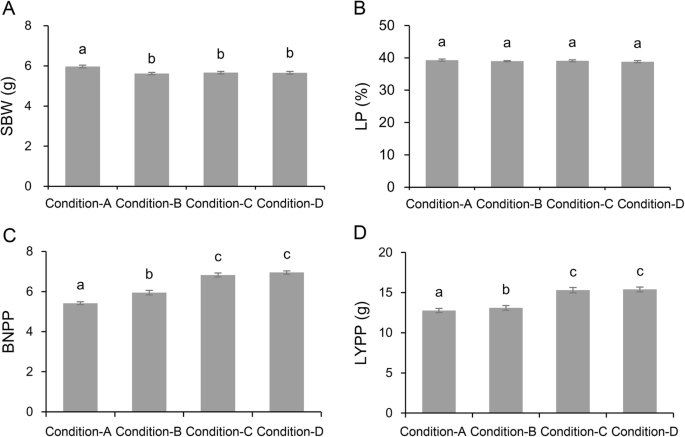
成对样品t -进行了进一步研究表型变异的试验。条件A的SBW略高于条件B、C和D(图2)。1一个);然而,在四种盐条件下,LP没有显著差异(图2)。1b).最显著的是,BNPP随着盐浓度的增加而降低(图2)。1c)。此外,LYPP也随着盐浓度的增加而降低,主要是由于BNPP的变化(图2)。1d).我们比较了最高和最低盐条件下SBW、LP、BNPP和LYPP的差异,发现SBW增加了5.29%,LP保持不变,BNPP减少了17.75%,LYPP减少了16.79%。综上所述,棉花的皮棉产量主要受铃数减少的影响,但铃重略有增加。这可能是由于高盐条件下铃数严重偏低所致。
对四种盐条件下的SBW、LP和BNPP进行了相关分析(附加文件)7:图S2)。结果表明,3个性状间相关性不大。不同盐环境下的LP值高度相关,R值在0.75 ~ 0.83之间,其次是SBW, R值在0.43 ~ 0.63之间。4种盐条件下BNPP均无相关性或相关性较弱,进一步说明BNPP受盐浓度影响较大。
皮棉产量相关性状的GWAS
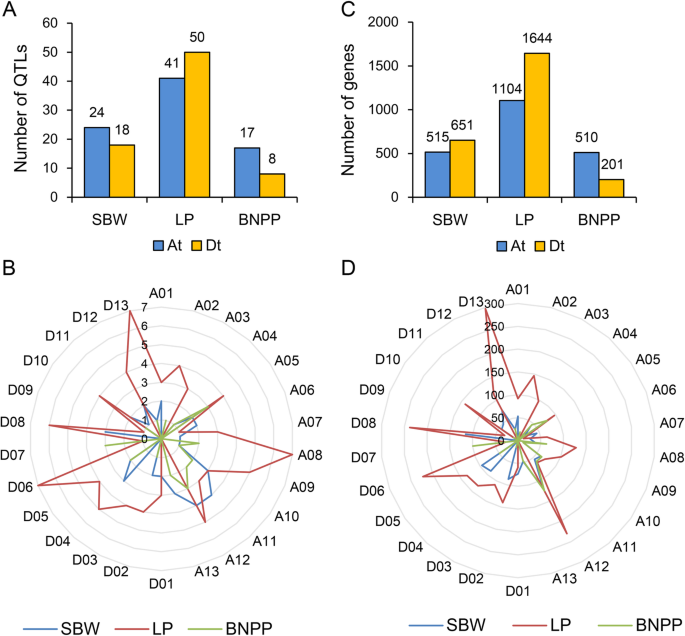
利用57,413个高质量snp的基因型数据[18],利用R软件包“mrMLM”中的多位点混合线性模型(MLM)模型的“mrMLM”、“FASTmrMLM”、“FASTmrEMMA”、“pKWmEB”、“ISIS EM-BLASSO”和“pLARmEB”6种方法,对3个性状在不同环境下及BLUP值进行GWAS分析[19]。总共有26条染色体上的854个数量性状核苷酸(QTNs)被鉴定出与这三个性状显著相关8表6)。我们在上一份报告[18]并计算每条染色体的平均LD,进一步选择最低的LD,约200kb作为LD阈值,用于将qtn合并到同一个QTL中。6个多位点MLM模型在不同环境和不同blps下共检测了600个qtl,其中SBW的151个,LP的417个,BNPP的112个,共检测1446次(表1)1)。对于每个性状,大多数qtl (SBW的85个,LP的235个,BNPP的65个)只检测到一次,这表明这些qtl容易受到环境条件的影响(附加文件)9:图S3)。为了提高相关qtl的可靠性和稳定性,我们选择了那些在不同方法或环境中被检测三次或更多次的qtl作为稳定的qtl进行进一步分析。结果,在SBW、LP和BNPP中分别鉴定出42、91和25个qtl(表2)1)。
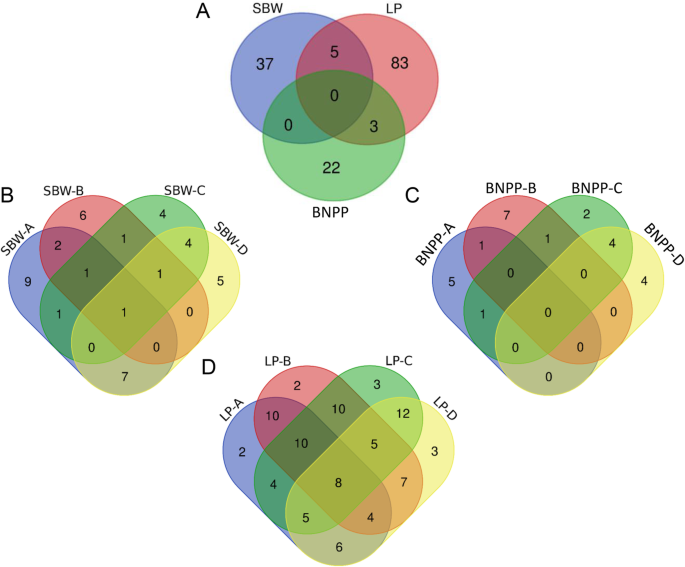
染色体分布表明,这些稳定的qtl广泛分布在26条染色体上,其中SBW和BNPP位于At亚基因组的qtl多于位于Dt亚基因组的qtl,而LP的qtl则相反(图2)。2a).大多数SBW qtl位于A11和A12染色体上,LP位于A08、D06和D13染色体上,BNPP位于A05、A12和D07染色体上(图2)。2b).这些稳定QTL的维恩图显示,在3个性状中没有检测到共定位的QTL,大多数QTL是单个性状特异性的(图2)。3.A),这意味着这三种性状的遗传控制存在很大差异。我们进一步分析了各性状在不同盐条件下的qtl。在所有四种盐条件下,只检测到SBW的一个QTL,而在BNPP中没有检测到重叠的QTL(图2)。3.b, c)。然而,在不同的盐条件下,大多数LP的qtl是重复检测的:在所有四种盐条件下,LP的qtl同时检测到8个(图2)。3.d).这些结果表明,不同盐胁迫条件下SBW和BNPP的调控机制复杂多变,但LP对盐胁迫的调控稳定且具有高度遗传性。
qtl候选基因的鉴定
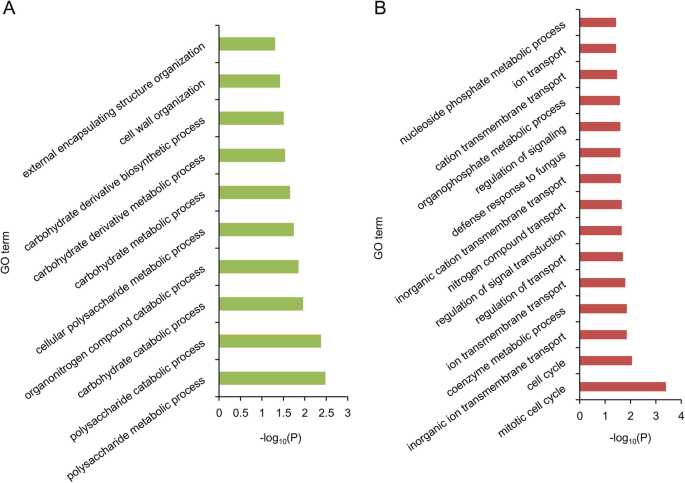
在这些稳定QTL区域中提取潜在的候选基因g .分子TM-1基因组[20.]。在SBW、LP和BNPP的稳定QTL区域共鉴定出1166、2748和711个候选基因(图2)。2c), SBW、LP和BNPP的大部分基因分别分布在A12、D13和A12染色体上(图2)。2d).通过氧化石墨烯分析,发现SBW QTL区域的基因在“胚胎发育”和“细胞形状调节”方面富集(另附文件)10:图S4和附加文件11表S7)。LP qtl基因在“赤霉素介导的信号通路调控”、“活性氧代谢过程正向调控”和“油菜素内酯生物合成过程”等激素和ROS调控途径中富集。它们还富含碳水化合物代谢过程,如“葡萄糖代谢过程”和“己糖生物合成过程”,这与之前的报道一致,这些氧化石墨烯项目在纤维发育中起着至关重要的作用[11,21]。此外,我们分别鉴定出14个、21个和10个与“高尔基囊泡运输”、“植物型次生细胞壁生物发生”和“葡萄糖代谢过程”相关的基因,但这些过程相关的基因均未在BNPP和SBW的QTL区域中发现(附加文件)12:图S5和附加文件11表S7)。与BNPP相关的基因功能主要富集于“有丝分裂细胞周期”、“离子跨膜运输”和“多糖分解代谢过程”(另附文件)13:图S6和附加文件11:表S7),表明BNPP与应激反应密切相关。
LP相关基因
通过组织和器官转录组分析[22],我们从LP qtl中鉴定出182个在纤维发育过程中显性表达的基因。其中在10 DPA、15 DPA、20 DPA和25 DPA时高表达的基因分别为29、35、73和45个(附加文件)14:图S7和附加文件15表8)。此外,我们对所有4种盐条件下8个稳定qtl区域的基因进行了关注,鉴定出10个在纤维发育过程中主要表达的基因(附加文件)16表S9)。他们,Gh_A05G2488该基因编码生长素转运促进因子家族成员PIN-FORMED like蛋白(PILS),位于高频相关QTL (A05: 32377816-33,100,112)中,通过多种方法和环境检测了21次。生长素对植物的生长发育至关重要,包括棉纤维的发育。在植物发育过程中起重要作用的生长素载体是PIN- formed (PIN)蛋白[23,24]。两个基因,Gh_D13G0342编码RAB GTPase同源物G3F (RABG3F)Gh_A10G2138这两个基因分别位于QTL (D13: 3297533-3,912,736)和QTL (A10: 99896949-100,396,471)。之前的报告显示RAB基因在细胞极性生长中起着至关重要的作用,包括花粉管的伸长[25],根毛[26]和棉纤维[27]。
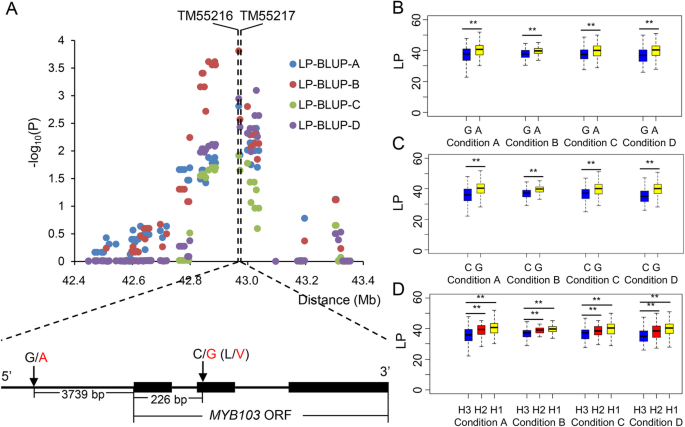
棉花种子转移细胞富含胼胝质,胼胝质调节纤维伸长和次生壁增厚[j]。28]。Zhang等(2007)发现转录因子MYB103影响花药发育过程中的胼胝质溶解拟南芥[29]。我们发现Gh_D03G1419该基因编码MYB103转录因子(名为GhMYB103),位于高频相关QTL (D03: 42299450-43,529,568)。序列分析显示,与LP相关的两个qtn分别位于该基因上游3739 bp (TM55216, D03: 42969311)和外显子(TM55217, D03: 42973276)区域(图2)。4a). TM55217位点的单核苷酸突变(从C到G)导致氨基酸从亮氨酸(L)变为缬氨酸(V)(图5)。4a).通过学生的t经检验,我们发现TM55216中A基因型的LP值显著高于G基因型(图2)。4b),并且在TM55217中G基因型显著高于C基因型(图2)。4c).两个qtn可产生3个单倍型;H1: AG, H2: AC, H3: GC。AG和AC单倍型的LP值显著高于GC单倍型。而AG和AC单倍型间差异不显著,说明QTN TM55216可能在LP中发挥更重要的作用(图5)。4d)。
此外,我们将LP qtl与以前报告中发表的GWAS信号整合在一起[11,12],发现3个qtl (A12: 602614 - 743324;D13: 58792627 - 59289811;A09: 4676815-5,076,815),与GWAS信号重叠。三个qtl在不同的盐条件下被检测到。此外,从QTL区域中鉴定出10个基因在纤维发育期间主要表达(附加文件)16表S9)。他们,转化[30.),CPK17[31]与下胚轴生长或纤维发育有关。
综上所述,这些稳定的LP qtl的基因特征及其在纤维发育过程中的优势表达表明,它们可能在育种实践中对LP的改善发挥重要作用。
BNPP相关基因
BNPP的qtl易受环境变化的影响。在所有4种盐环境下均未检测到重复的qtl,大多数qtl仅在1或2种盐环境中被鉴定出来。例如,主要TM57617_TM57620 (D05: 24.3 - -24.8 Mb,检测到16倍)和TM74225 (D10: 24.1 - -24.5 Mb,发现7次)被确定在盐条件下C和d .然而,QTL TM52041_TM52044 (D02: 49.2 - -49.7 Mb,发现6次)被认定只有在盐条件,和QTL TM29006 (A08: 81.7 - -82.1 Mb,检测5次)仅被盐条件。这些结果表明,基因调节吐絮期不同的数量不同的盐条件。
通过氧化石墨烯分析发现大量与离子传递相关的物质被富集(附加文件)13:图S6和附加文件11表S7)。为了进一步探索胁迫耐受性与铃数之间的关系,我们利用盐胁迫转录组进行了RNA-seq分析Gossypiumacc。TM-1。经FPKM筛选,共获得QTL区基因3500个(根446个,叶395个)。通过GO注释和差异表达分析,我们重点研究了40个盐诱导胁迫反应基因。其中6个在根和叶中常见17:图S8,附加文件18表S10和附加文件19(表S11)。Gh_A04G1216编码一个高亲和力的K+运输船1号(HKT1)。AtHKT1限制根到茎的钠运输,被认为是盐耐受性的必要条件拟南芥[32]。Gh_A05G3239编码一种过氧化物酶超家族蛋白(POD),该蛋白已被证明在棉花盐胁迫下的抗氧化中起重要作用[33]。Gh_A08G1183编码一种丝裂原活化蛋白激酶(MAPK),该蛋白激酶已被广泛报道与棉花耐盐性有关[34]。除了离子转运GO项外,盐胁迫下碳水化合物代谢也很活跃,如富集GO项“多糖分解代谢过程”中鉴定出10个基因,其中3个基因在盐胁迫下差异表达。细胞周期调控对植物生长发育至关重要[35]。在富集的氧化石墨烯术语“细胞周期”中共鉴定出33个基因,其中8个基因在盐胁迫下差异表达。这些候选基因可能对不同盐环境下棉铃数的增加有贡献。
为了探究高盐和低盐条件下铃数调控的关键基因和机制,我们比较了条件A和D之间BNPP QTL区域的基因,在盐条件A和D下分别鉴定出204个和265个基因。氧化石墨烯富集分析显示,高盐和低盐条件下基因的功能分类存在显著差异。与“多糖代谢过程”、“碳水化合物分解代谢过程”和“细胞壁组织”相关的GO术语在高盐条件下富集A(图2)。5a、附加文件20.:表S12),而低盐条件下“细胞周期”、“离子跨膜转运”和“信号转导调节”则富集(图12)。5b).这表明在高盐条件下碳水化合物代谢和细胞结构维持起着至关重要的作用,而离子转运是一个基础过程,在低盐条件下更为重要。在高盐条件下,检测到几个与BNPP相关的候选基因。Gh_A11G1551编码脯氨酸脱氢酶1 (ProDH1),也称为早期脱水反应5 (ERD5),已被广泛研究,特别是在非生物胁迫下[36]。对于能量代谢,Gh_A05G1912编码一种异淀粉酶3 (ISA3),它有助于淀粉的分解。在低盐条件下,三个候选基因在钠钾平衡中发挥重要作用。在细节,Gh_A10G0441编码钾转运蛋白1 (KUP1) [37],Gh_A12G0074编码一个高亲和力的K+运输工具5 (HAK5) [38),Gh_A12G0061编码氢交换钠2 (NHX2) [39]。
讨论
随着世界范围内耕地面积的减少和土壤环境的恶化,迫切需要提高作物的抗逆性。新疆是中国棉花主产区,但土壤盐碱化程度较高。挖掘盐碱条件下能提高棉花产量的优良等位基因,对选育高产抗逆性棉花新品种具有重要意义。随着高通量测序技术和新的统计方法的发展,GWAS为植物功能基因的挖掘提供了一种快速有效的方法。在以往的研究中,已经在萌发和播种阶段检测到与耐盐性相关的几种营养生长指数的GWAS信号[4,17]。然而,胁迫条件下棉花产量性状的GWAS研究很少。本研究重点研究了4种盐条件下的3个皮棉产量性状(SBW、LP和BNPP),并鉴定了有利的相关qtl和精英等位基因。研究结果为棉花耐盐性的遗传基础、耐盐性状变异的新等位基因和候选基因的鉴定提供了新思路,为加快棉花耐盐育种进程提供了新思路。
盐胁迫下棉花产量下降。已有研究报道,盐胁迫条件下SBW、LP和BNPP显著降低[40,41,42]。本研究选择4种不同的田间土壤盐环境,2年重复,研究棉花材料皮棉产量构成因素。通过表型分析,LP在不同盐分条件下变化不显著。最高盐浓度下的SBW略高于其他3种低盐浓度,这可能与BNPP降低有关。值得注意的是,随着总盐含量的增加,BNPP显著降低,导致棉绒产量降低。总体而言,与其他两个性状相比,BNPP是最易受盐胁迫的因子。与最低盐条件相比,最高盐条件下LYPP损失16.79%,主要是由于BNPP减少所致。因此,在盐胁迫下保持棉花铃数对保持棉棉高产具有重要意义。此外,SBW、LP和BNPP性状之间的相关性较小,说明在盐胁迫下,不同的生物过程调节着这3个性状。
提高产量是棉花育种计划的主要目标。LP是棉花产量的重要组成部分,是棉花品种的重要经济指标。虽然本研究中表型分析显示LP在不同盐条件下没有显著变化,但GWAS结果表明,与之前报道的非盐条件下相比,重叠qtl较少[11,12],这意味着一些特定的基因参与了盐度条件下LP的改善。我们还在A08和D08染色体上发现了大量与LP相关的qtl,据报道这些qtl中含有许多与纤维发育相关的qtl或关键基因[11,12]。然而,与LP相关的qtl分布与Su et al.(2016)的报道有很大不同[43]。综上所述,这些结果表明LP是一个复杂的数量性状,在我们的研究中检测到的大多数位点都是新的,可能与盐胁迫有关。其中8个qtl在4种盐胁迫条件下同时被鉴定出来,这可能与盐胁迫下LP的增加有关。此外,我们在这些稳定的LP qtl中鉴定了10个与纤维发育密切相关的基因,如得利),RAB和MYB。在拟南芥, overexpressing得利)基因降低了下胚轴和根的生长[44]。然而,生长素的积累可以促进纤维细胞的细胞起始(−2 ~ 2 DPA)。转基因实验表明,该基因对胚珠有多种特异性抑制作用GhPIN基因抑制棉花的纤维形成和伸长[45],表明Gh_A05G2488在纤维发育中起重要作用。RAB和RAB受体蛋白在棉纤维发育中也具有潜在的重要功能。在拟南芥,RabA4d是正确调控花粉管生长所必需的。的损失RabA4d导致花粉管生长的破坏和细胞壁结构的改变[46]。此外,PRA1结构域蛋白是作为Rab gtpase受体调节囊泡运输的小跨膜蛋白。AtPRA1蛋白定位于内质网、高尔基体和核内体/泡前室,表明在分泌和内吞细胞内运输途径中都有功能[47]。众所周知,MYB转录因子在纤维发育中也起着至关重要的作用。GhMYB212RNAi植物(GhMYB212i)在发育纤维中积累的蔗糖和葡萄糖较少,纤维较短,绒毛指数较低[48]。的表达分析MYB家庭证明GhMYB103在25种DPA种子纤维中高度表达,与纤维素合成相关[49]。在我们的研究中,两个重要的qtn位于基因的上游和外显子区域GhMYB103分别引起基因表达水平变化和氨基酸变异,进而影响棉花的LP。此外,该SNP位于的上游调控区GhMYB103单倍型分析可能在LP中发挥更重要的作用。虽然GWAS分析已经鉴定了许多与LP相关的基因,但本研究的候选基因可能在盐胁迫下LP的改善中发挥更重要的作用。我们还鉴定出3个与GWAS分析中报道的LP位点共定位的QTL,并在QTL区域进一步鉴定出10个候选基因。这些研究可为盐环境和正常环境下LP的改良提供遗传资源。
对BNPP的GWAS分析相对较少,特别是在盐胁迫下。我们的研究表明,盐胁迫可导致单株铃数显著下降,这与之前的报道一致[40,50],说明铃数是胁迫环境下棉绒产量提高的第一限制因素。我们还发现,在不同的盐条件下,与BNPP相关的重复qtl数量较少,表明BNPP的调控机制复杂。GO分析表明,与BNPP相关的基因主要参与“有丝分裂细胞周期”、“离子跨膜运输”和“多糖分解代谢过程”。其中,丰富了大量与离子转运相关的过程,说明优良的离子转运能力在棉花耐盐性中起着关键作用。Na+积累可导致离子中毒,引起作物生物量下降和产量损失[1]。在盐胁迫下,通过离子吸收和区隔来维持离子稳态对植物生长至关重要。通过rna序列分析,我们发现HKT1已知它在Na的去除中起作用+在盐胁迫条件下,从木质部向根传递的基因被下调。过度的HKT1可以减少钠的含量+在茎部积累,显著提高耐盐性拟南芥[51]。有趣的是,HKT1也被发现在盐胁迫条件下下调g . davidsonii一个具有重要抗盐胁迫特性的棉花d基因组二倍体品种[52]。这表明的功能HKT1可以在棉花上进行改良,提高抗逆性。通过比较高盐和低盐条件下的qtl,我们发现与BNPP相关的qtl没有重叠,表明不同盐条件下的调控机制复杂。高盐条件下富集的基因主要与能量代谢和细胞形态维持有关。高盐胁迫可导致植物光合效率降低[53]。在非胁迫条件下,植物利用大部分能量维持营养和生殖生长。然而,随着盐浓度的增加,植物需要分配更多的能量来抵抗压力[1]。此外,高盐浓度也会增加渗透胁迫,植物需要合成更多的渗透酶来维持细胞形态。在这项研究中,我们发现ISA3在能量代谢和ProDH1有助于维持细胞形态。ISA3在淀粉颗粒的表面起作用,并从颗粒表面去除可以提高淀粉分解率的短分枝。Atisa3突变体比野生型植物有更多的叶片淀粉和更慢的淀粉分解速度[54]。为了对抗盐胁迫引起的渗透胁迫,一些植物积累了脯氨酸、甘氨酸甜菜碱、糖醇等多种相容的渗透产物,以保护大分子,维持细胞膜内外的渗透平衡[j]。55]。的表达式ProDH2,高度同源的基因ProDH1,能促进脯氨酸在逆境条件下的积累[56]。此外,低盐条件下富集的基因主要与离子转运有关。作为一种耐盐作物,棉花在低盐条件下受到的盐害比其他植物要小,这可能是由于棉花具有高效的离子运输能力。在这项研究中,KUP1,HAK5的和NHX2被认为有助于离子稳态。这表明,低盐浓度下活跃的钠钾离子交换能力是棉花耐盐性的基础。此外,这些结果也反映了棉花在不同盐胁迫条件下对抗逆性的不同需求。
一些报道表明,可以通过改变棉花耐盐基因的表达来提高棉花产量。例如,过度表达AvDH1在棉花中可减少膜离子泄漏,提高超氧化物歧化酶活性,从而达到耐盐增产的目的[j]。50]。过度的SNAC1,属于胁迫相关转录因子NAC超家族,可以通过促进根系发育和降低蒸腾速率来提高抗旱性和耐盐性[57]。在本研究中,我们首次报道了盐胁迫下棉花三个皮棉产量组成部分的表型和GWAS分析。结果表明,在盐碱地环境下,BNPP是影响棉花产量的最重要因素。此外,我们还鉴定了大量在盐条件下对皮棉产量提高有贡献的精英等位基因。这些发现将有助于我们了解棉花耐盐机理,并为盐碱地环境下棉花品种的选育提供参考。
结论
阐明盐碱地棉花产量构成因素的遗传变异,对盐碱地的开发利用具有重要的实用价值。本研究通过对316份陆地棉材料在不同盐胁迫条件下的表型分析表明,棉花皮棉产量主要受铃数减少的影响。通过GWAS分析,分别鉴定出42个、91个和25个稳定qtl。此外,10个候选基因包括得利),RAB和MYB,与纤维发育密切相关,用于LP改善HKT1,圆荚体和MAPK,对盐环境下铃数的增加有重要意义。此外,高盐和低盐条件下BNPP的调控机制不同,表明不同盐条件下BNPP的调控较为复杂。本研究为理解棉花盐胁迫机理提供了新的思路,并为盐胁迫下棉铃数的提高提供了基因资源。
方法
植物材料和田间试验
本研究共使用了316份陆地棉材料,包括来自中国不同地区的303个品种/系和从美国引进的13个地方品种1:表1)。所有文献由南京农业大学收藏保存。从中国南京农业大学获得了种植和调查自然种群所需的所有许可。
2016年和2017年,在新疆农业大学试验站(43°20′~ 45°20′′,84°45′~ 86°40′′)4种不同盐田浓度下,种植316份旱棉材料。从中国新疆农业大学获得了对这些材料进行实地评价所需的所有许可。采用5点采样法测定土壤总盐含量。4种环境的总含盐量分别为19 g/Kg(条件A)、10 g/Kg(条件B)、7 g/Kg(条件C)和5 g/Kg(条件D),每种盐条件设2个重复样地(另附文件)2:图S1A和附加文件3.表2)。采用宽/窄行交替种植模式(窄行10 cm,宽行66 cm),每个接穗分两行种植,行长2 m,每亩间株距0.10 m。采用地膜下滴灌施肥促进植物生长。其他农艺操作对所有处理都是相同的。
表型调查和数据分析
在每行中间随机选取每株10株,进行标记鉴定,记录SBW、LP和BNPP的数据。在植株成熟时(开铃率约为70%),用10个生物重复计数BNPP。从标记植株的中枝上采集20个发育良好的开铃样品(每株2个铃),称重用于SBW和LP计算。为了减少环境影响,利用lme4包中的“lmer”函数,基于混合线性模型估计了每种盐条件下这三个性状的最佳线性无偏预测因子(blbp) [58]。为探讨不同盐浓度对棉棉产量的影响,采用SBW、LP和BNPP相乘计算LYPP。
成对样品t-采用SPSS软件进行不同盐条件和性状间的检验和相关性分析。使用R软件包“Performance Analytics”进行相关分析的可视化。
GWAS分析
根据Paterson et al.(1993)的方法提取316份棉花的基因组DNA [59]。采用CottonSNP80K阵列对316份棉花材料进行基因分型。SNP基因分型和群体结构在我们之前的研究中有报道[18]。共使用57,413个snp(召唤率≥0.9,次要等位基因频率(MAF)≥0.05)进行GWAS分析。为了探究snp -性状之间的关联,我们建立了一个多位点随机- snp -效应混合线性模型(mrMLM) [19]使用R软件包“mrMLM”,参数如下P-在rMLM中的值:0.001;候选基因搜索半径(Kb): 100;mrMLM的临界LOD得分:3。采用Q + K模型。种群结构(Q)矩阵采用k = 3的混合物1.3计算,亲缘关系(k)矩阵采用R软件包“mrMLM”计算。分别利用3个性状在不同盐分条件下的BLUP值和单一环境进行GWAS。
qtl与候选基因鉴定
我们参考了先前一份报告[18],计算每条染色体的平均LD,并选择最低的LD(约200 kb)作为LD阈值,用于将qtn合并到同一个QTL中。如果两个qtn之间的距离小于200kb,则合并为一个QTL。每个性状在不同方法或不同环境中发现3次或3次以上的qtl被选为稳定qtl。从参考基因组TM-1中通过自编写壳脚本提取位于稳定QTL区域的候选基因[20.]。基因本体(GO)分析使用AgriGO V2.0,采用SEA方法[60]。
RNA-seq分析
为了确定哪些基因与皮棉产量相关,从NCBI Sequence Read Archive collection PRJNA490626中下载了TM-1组织的转录组图谱[22]。通过计算z分数,确定了花后- 3、0、1、3、5天,10、15、20和25天胚珠,10、15、20和25天纤维组织的根、茎、叶、花瓣、环面、萼片、苞片、花药、花丝、雌蕊、胚珠和纤维组织在23个组织或发育阶段的表达模式。为了研究与胁迫耐受相关的基因,从NCBI Sequence Read Archive collection PRJNA532694和PRJNA490626中分别下载了盐处理过的TM-1根和叶片的转录组图谱。两个RNA-seq读数都被映射到g .分子acc。TM-1基因组使用默认参数的Tophat拼接比对器[61]。每个文库中基因组匹配的读数用袖扣组装起来[62]。然后使用Cuffmerge将单个转录本程序集合并为单个转录本集。最后,采用Cuffdiff法检测差异表达基因(DEGs),截断q值为0.05。每个样本的3个生物重复用于RNA-seq实验。主要表达基因和盐胁迫应答基因的热图由Mev软件(http://mev.tm4.org)。
数据和材料的可用性
本研究的RNA-Seq数据已存放在国家生物技术信息中心(NCBI),http://www.ncbi.nlm.nih.gov/),编号为PRJNA490626、PRJNA532694和PRJNA490626。
缩写
- BLUP:
-
最好的线性无偏预测
- BLUPs:
-
最好的线性无偏预测器
- BNPP:
-
每株铃数
- 简历:
-
变异系数
- GBS:
-
测序基因分型
- 走:
-
基因本体论
- GWAS:
-
全基因组关联研究
- LD:
-
连锁不平衡
- LP:
-
线头百分比
- LYPP:
-
单株皮棉产量
- 加:
-
次要等位基因频率
- 经济价值:
-
数量性状位点
- 本考察团:
-
数量性状核苷酸
- 南偏西:
-
单铃重
- SNP:
-
单核苷酸多态性
- 苏维埃社会主义共和国:
-
简单序列重复
参考文献
- 1.
赵建军,张建军,张建军。盐度与作物产量。植物学报,2019;21(S1): 31-8。
- 2.
Dagar JC, Minhas PS.盐渍化土壤农林业管理的全球视角。编辑:Dagar J, Minhas P。用于管理涝渍盐渍土壤和劣质水的农林业。新德里:Springer India;2016.5-32页。
- 3.
韩静,石静,曾丽,徐静,吴丽。施氮对温室土壤酸碱度的影响。环境科学与污染研究,2015,22(4):2976-86。
- 4.
杜丽,蔡超,吴生,张飞,侯生,郭伟。棉花盐胁迫相关性状有利QTL等位基因的评价与探索(g .分子l .)。科学通报,2016;11(3):e0151076。
- 5.
李忠,李林,周坤,张勇,韩霞,丁艳,葛霞,秦伟,王鹏,李峰,等。GhWRKY6在这两种转基因中起负调控作用拟南芥棉花在干旱和盐胁迫下。前沿热纳。2019;10:39 . 92。
- 6.
张斌,陈霞,陆霞,舒宁,王霞,杨霞,王森,王杰,郭玲,王东,等。转录组分析陆地棉L.揭示了NaCl、NaOH和Na之间不同的作用机制2有限公司3.强调宽容。科学通报,2018;8:13527。
- 7.
孙中伟,李海林,张勇,李正康,柯海峰,吴立强,张光远,王晓峰,马志远。棉花苗期耐盐相关snp及候选基因的鉴定(陆地棉l .)。植物科学,2018;9:1011。
- 8.
石云英,高丽丽,吴志成,张晓军,王敏,张春春,张峰,周玉玲,李志康。水稻种子萌发期耐盐性的全基因组关联研究。植物学报,2017;17(1):92。
- 9.
袁勇,邢辉,曾伟,徐军,毛丽,王丽,冯伟,陶杰,王辉,张辉,等。植物耐盐性的全基因组关联及差异表达分析陆地棉L在萌发阶段。植物学报,2019;19(1):394。
- 10.
李春华,李春华,李春华,等。盐碱化条件下基于卫星植被指数的作物产量预测。作物学报,2016;19(2):134 - 43。
- 11.
方丽,王强,胡勇,贾勇,陈健,刘波,张志,管旭,陈生,周斌,等。棉花的基因组分析确定了与纤维品质和产量性状相关的选择特征和位点。物理学报,2017;49(7):1089-98。
- 12.
马忠,何生,王旭,孙健,张勇,张刚,吴磊,李忠,刘忠,孙刚,等。对陆地棉核心材料进行重测序,确定了影响纤维质量和产量的基因组变异和位点。植物物理学报,2018,50(6):803-13。
- 13.
王敏,涂林,林敏,林忠,王鹏,杨强,叶忠,沈超,李军,张磊,等。棉花驯化过程中的不对称亚基因组选择和顺式调控分化。物理学报,2017;49(4):579-87。
- 14.
宋超,李伟,裴翔,刘勇,任忠,何坤,张飞,孙坤,周翔,马翔,等。陆地棉衣分遗传变异及候选基因的全基因组关联分析。理论应用学报,2019;32(7):1991-2002。
- 15.
袁勇,王鑫,王磊,邢宏,王强,赛义德梅,陶杰,冯伟,张刚,宋晓玲,等。全基因组关联研究确定了与大豆种子油成分和蛋白质含量相关的候选基因陆地棉[j] .植物科学,2018;9:1359。
- 16.
Diouf L,潘泽,何绍平,龚文峰,贾玉华,Magwanga RO, Romy KRE, Rashid HO, Kirungu JN,杜晓明。利用基因分型测序(GBS)构建陆地棉苗期耐盐qtl高密度连锁图谱及定位。中华医学杂志,2017;18(12):2622。
- 17.
Dilnur T,彭志,潘志,Palanga KK,贾勇,龚伟,杜霞。亚洲棉耐盐性的关联分析(木本棉),有SNP标记。中华生物医学杂志,2019;20(9):2168。
- 18.
侯松,朱刚,李勇,李伟,付健,牛娥,李丽,张东,郭伟。棉花干旱胁迫相关性状的全基因组关联研究揭示遗传变异及候选基因(陆地棉l .)。植物科学,2018;9:1276。
- 19.
王绍斌,冯建勇,任伟,黄斌,周磊,温玉军,张军,Dunwell JM,徐生,张彦明。通过多位点混合线性模型方法提高全基因组关联研究的能力和准确性。科学通报,2016;6:1944。
- 20.
异源四倍体棉花(陆地棉l . acc。TM-1)为光纤的改进提供了资源。生物工程学报,2015,33(5):531-7。
- 21.
王峰,张军,陈勇,张超,龚军,宋忠,周军,王军,赵超,焦敏,等。关键纤维相关qtl候选基因的鉴定及有利等位基因的衍生陆地棉重组自交系g .取得渐渗现象。植物工程学报,2019。https://doi.org/10.1111/pbi.13237。
- 22.
海岛棉和陆地棉基因组提供了异源四倍体棉花的起源和进化的见解。中国生物医学工程学报,2019;51(4):739-48。
- 23.
张晓明,张晓明,张晓明,等。植物生长素在植物器官形成过程中的作用机制。细胞。2003;115(5):591 - 602。
- 24.
Dubrovsky JG, Sauer M, napsuciy - mendivil S, Ivanchenko M, Friml J, Shishkova S, Celenza J, Benkova E.生长素对侧根建立细胞的局部形态发生的影响。科学通报,2009;35(5):391 - 391。
- 25.
彭杰,Ilarslan H, Wurtele ES, Bassham DC。AtRabD2b和AtRabD2c在花粉发育和花粉管生长中具有重叠的功能。植物学报,2011;11:25。
- 26.
布兰科FA,梅斯奇尼EP,萨内蒂ME,阿吉拉尔OM。一个小的rabb家族的GTPase在根毛形成和普通豆根瘤菌共生关联的感染前阶段是必需的。植物学报,2009;21(19):2797-810。
- 27.
李鹏,郭伟。猪Rab基因家族的全基因组分析Gossypium通过比较分析。植物学报,2017;58(1):26。
- 28.
gerriero G, Fugelstad J, Bulone V.我们对高等植物纤维素生物合成的真正了解?植物学报,2010;32(2):591 - 591。
- 29.
张志斌,朱军,高建峰,王超,李红,李红,张海清,张生,王德明,王启祥,等。转录因子AtMYB103是否通过调节绒毡层发育、胼胝质溶解和外壁形成来调控花药发育拟南芥。植物学报,2007;32(3):528 - 538。
- 30.
Dahlke RI, Fraas S, Ullrich KK, Heinemann K, Romeiks M, Rickmeyer T, Klebe G, Palme K, Luthen H, Steffens B.生长素信号通路对原生质体膨胀和下胚轴生长的影响。植物生理学报,2017;17(2):982-94。
- 31.
肖刚,赵鹏,张艳。激素在棉花纤维发育调控中的关键作用。植物科学,2019;10:87。
- 32.
安丹,陈建刚,高玉青,李霞,晁志峰,陈志荣,李qq,韩明丽,王玉玲,王云峰,等。AtHKT1驱动适应拟南芥通过降低花的钠含量来降低盐分。科学通报,2017;13(10):e1007086。
- 33.
易卜拉欣文,邱文武,张超,曹峰,朱生,吴峰。两种耐盐基因型棉花单株及组合的耐盐干旱比较生理分析。植物物理学报,2019;16(2):559 - 561。
- 34.
张军,邹东,李勇,孙旭,王宁宁,龚世义,郑勇,李小兵。GhMPK17是一种棉花丝裂原活化蛋白激酶,参与植物对高盐度和渗透胁迫的反应以及ABA信号的传递。科学通报,2014;9(4):95642。
- 35.
李建军,李建军,李建军,等。植物细胞周期调控的研究进展。热论。2006;40:77-105。
- 36.
Rizzi YS, Cecchini NM, Fabro G, Alvarez ME。差动控制及功能拟南芥ProDH1和ProDH2生物营养性和坏死性病原体感染的基因。植物学报,2017;18(8):1164-74。
- 37.
王艳,陆军,陈东,张军,祁坤,程锐,张华,张生。梨KT/HAK/KUP家族的全基因组鉴定、进化及表达分析。基因组。2018;61(10):755 - 65。
- 38.
张建军,李建军,李建军,李建军,陈建军,陈建军,陈建军,陈建军,陈建军ILK1和高亲和K+转运体HAK5的是先天免疫和非生物应激反应所必需的。植物生理学报,2016;17(2):1470-84。
- 39.
Barragan V, Leidi EO, Andres Z, Rubio L, De Luca A, Fernandez JA, Cubero B, Pardo JM。离子交换剂NHX1和NHX2介导液泡对钾的活性吸收,调节细胞膨胀和气孔功能拟南芥。植物学报,2012,24(3):1127-42。
- 40.
易卜拉欣文,朱彦明,陈艳,邱文武,朱生,吴峰。干旱和盐胁迫下棉花叶片次生代谢、植物激素和产量的基因型差异。植物物理学报,2019;36(2):343 - 355。
- 41.
Hussein MM, about - baker NH。纳米锌对缓解棉花盐胁迫的作用。应用科学学报,2018;5(8):171809。
- 42.
彭健,刘健,张磊,罗健,董辉,马勇,赵旭,陈斌,隋宁,周忠,等。土壤盐分对棉花叶片蔗糖代谢的影响。科学通报,2016;11(5):e0156241。
- 43.
苏军,范生,李林,魏华,王超,王宏,宋敏,张超,顾林,赵生,等。GWAS检测中国陆地棉衣分有利等位基因和候选基因。植物科学,2016;7:1576。
- 44.
Barbez E, Kubes M, Rolcik J, Beziat C, Pencik A, Wang B, Rosquete MR, Zhu J, Dobrev PI, Lee Y,等。一个新的推测生长素载体家族调节植物细胞内生长素稳态。大自然。2012;485(7396):119 - 22所示。
- 45.
张敏,曾建勇,龙辉,肖艳华,闫晓燕,裴宇。生长素通过ghpin介导的生长素运输调控棉花纤维形成。植物生理学报,2017;58(2):385-97。
- 46.
Rab GTPase RabA4d调控花粉管顶端生长拟南芥。植物学报,2009;21(2):526-44。
- 47.
Alvim Kamei CL, Boruc J, Vandepoele K, Van den Daele H, Maes S, Russinova E, Inze D, De Veylder L.PRA1基因家族拟南芥。植物生理学报,2008;47(4):1735 - 1749。
- 48.
孙文杰,高志勇,王健,黄玉青,陈毅,李建峰,吕明林,王健,罗明,左克军。棉纤维伸长需要转录因子GhMYB212调节蔗糖进入扩张纤维的运输。植物学报,2019;22(2):864-81。
- 49.
MacMillan CP, Birke H, Chuah A, Brill E, Tsuji Y, Ralph J, Dennis ES, Llewellyn D, Pettolino FA。棉花组织和细胞特异性转录组揭示了植物次生细胞壁多样性背后的基因调控的微妙之处。生物医学工程学报,2017;18(1):539。
- 50.
陈静,万生,刘辉,范生,张勇,王伟,夏敏,袁锐,邓峰,沈峰。罗布麻死盒解旋酶基因的过表达(AvDH1)能使棉花耐盐,提高盐碱地的产量。植物科学,2015;6:1227。
- 51.
Moller IS, Gilliham M, Jha D, Mayo GM, Roy SJ, Coates JC, Haseloff J, Tester M. Shoot Na+通过细胞类型特异性的Na改变来排除和增加耐盐性+运输在拟南芥。植物学报,2009;21(7):2163-78。
- 52.
朱刚,李伟,张峰,郭伟。盐胁迫下棉花选择性剪接的RNA-seq分析Gossypium davidsonii。生物医学工程学报,2018;19(1):73。
- 53.
作物耐盐性——成本是多少?植物学报,2015,32(3):668-73。
- 54.
张建军,张建军,张建军,张建军,等。植物淀粉颗粒分解机制的研究进展。生物化学学报,2006;28(1):1250 - 1290。
- 55.
Delauney AJ, Verma DPS。植物中脯氨酸的生物合成和渗透调节。植物学报,1993;4(2):215-23。
- 56.
李建军,李建军,李建军,等。大豆脯氨酸脱氢酶的非冗余功能拟南芥。植物生理学报,2010;10(10):790。
- 57.
刘刚,李霞,金生,刘霞,朱丽,聂燕,张霞。水稻NAC基因的过表达SNAC1通过促进根系发育和降低蒸腾速率提高转基因棉花的耐旱性和耐盐性。科学通报,2014;9(1):86895。
- 58.
张建军,张建军,张建军,等。线性混合效应模型的拟合。统计软件学报,2015;67(1):1 - 48。
- 59.
Paterson AH, Brubaker CL, Wendel JF。快速提取棉花的方法(Gossypium适合RFLP或PCR分析的基因组DNA。植物学报,1993;11(12):122 - 127。
- 60.
田涛,刘宇,严宏,游强,易鑫,杜志,徐伟,苏忠。AgriGO v2.0:面向农业社区的GO分析工具包,2017更新。中国生物医学工程学报,2017;45(5):522 - 522。
- 61.
张建军,张建军,张建军,等。基于RNA-Seq技术的剪接连接研究。生物信息学,2009,25(9):1105 - 11所示。
- 62.
trappnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR, Pimentel H, Salzberg SL, Rinn JL, Pachter L. TopHat和袖链的差异基因和转录本表达分析。地理学报,2012;7(3):562-78。
致谢
感谢南京农业大学生物信息学中心高性能计算平台提供的数据分析设施。非常感谢南京农业大学农学院Waqas Amjid博士的帮助编辑和评论。
资金
国家转基因计划项目(2018ZX0800918B)、国家作物育种重点研发计划项目(2018YFD0100400)、国家自然科学基金项目(U1903203;31771885),中央高校基本科研业务费专项资金(kyyj20101;KYYJ201801)、江苏省现代作物生产协同创新中心项目(No.10)、新疆维吾尔自治区天山青年计划(2018Q016)。资助者不参与研究的实验设计、数据收集、分析和解释,也不参与撰写论文。
作者信息
从属关系
贡献
实验采用WZG和QJC设计。实验采用GZZ、WWG、XHS、FLS、SH、NL、YJH、DYZ和ZYN进行。GZZ和WZG起草稿件,WZG修改稿件。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们没有相互竞争的利益
额外的信息
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
附加文件1表S1。
本研究使用的316种棉花资料。(xlsx20kb)
附加文件2图S1。
土壤盐浓度示意图及3个皮棉产量组成部分的表型数据分布。A:不同总含盐量的土壤盐分分布示意图。试验田分为8个部分,包括4种不同的总含盐量,每种盐态2个重复。B-D: 4种盐条件下单铃重(B)、衣分(C)和单株铃数(D)的表型数据分布。(TIFF 469 kb)
附加文件3表S2。
四种不同环境下土壤总含盐量的测定。(xlsx11kb)
附加文件4表S3。
4种盐条件下单铃重表型数据统计。(XLSX 10kb)
附加文件5表S4。
4种盐条件下衣分的表型数据统计。(XLSX 10kb)
附加文件6表S5。
四种盐条件下单株铃数表型数据统计。(XLSX 10kb)
附加文件7图S2。
皮棉产量3个组成部分与各性状不同盐条件下的相关分析。红框表示三个皮棉产量成分之间的相关关系。绿框表示各性状在不同盐条件下的相关性。方框中的数字表示相关系数(R阀)。表示*、**和***P值分别在0.05、0.01和0.001水平。(TIFF 2605kb)
附加文件8表S6。
利用多位点MLM模型检测单铃重、衣分和单株铃数的qtn和qtl。(XLSX 151kb)
附加文件9图S3。
3个皮棉产量组成部分600个相关qtl的检测次数分布。x轴表示检测到的次数;y轴为qtl数量。(TIFF 216kb)
附加文件10图S4。
单铃重相关候选基因的富集生物学过程。(TIFF 230kb)
附加文件11表S7。
氧化石墨烯富集与这三个性状相关的基因。(XLSX 21kb)
附加文件12图S5。
与衣分相关的候选基因的富集生物学过程。(TIFF 552kb)
附加文件13图S6。
与单株铃数相关的候选基因的富集生物学过程。(TIFF 430kb)
附加文件14图S7。
棉花纤维发育中与皮棉率相关的优势表达基因热图。数量表示不同的组织或发育阶段,1代表根;2:阀杆;3:叶;4:花瓣;5:环;6:萼片;7:苞片;8:花药;9:长丝; 10: pistil; 11: -3DPA ovule and fiber; 12: 0DPA ovule and fiber; 13: 1DPA ovule and fiber; 14: 3DPA ovule and fiber; 15: 5DPA ovule and fiber; 16: 10DPA ovule; 17: 15DPA ovule; 18: 20DPA ovule; 19: 25DPA ovule; 20: 10DPA fiber; 21: 15DPA fiber; 22: 20DPA fiber; 23: 25DPA fiber. (TIFF 586 kb)
附加文件15表S8。
182个优势表达基因在不同组织器官纤维发育过程中的表达模式。(XLSX 64kb)
附加文件16表S9。
在稳定qtl中鉴定出衣分相关的候选基因。(xlsx11kb)
附加文件17图S8。
盐胁迫下单株铃数相关候选基因热图。位于qtl上的胁迫响应基因在盐胁迫下分别在根(A)和叶(B)中被盐诱导。红色标记的基因名称代表根和叶的差异表达。(TIFF 639kb)
附加文件18表S10。
盐胁迫下19个单株铃数相关候选基因在根系中的表达模式(XLSX 13kb)
附加文件19表S11。
盐胁迫下单株铃数相关27个候选基因在叶片中的表达模式(XLSX 13kb)
附加文件20表S12。
盐条件A和d下单株铃数相关基因的氧化石墨烯富集(XLSX 12kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
朱国强,高伟,宋翔。et al。全基因组关联揭示棉花盐田条件下皮棉产量组成部分的遗传变异(陆地棉l .)。BMC Plant Biol20.23(2020)。https://doi.org/10.1186/s12870-019-2187-y
收到了:
接受:
发表:
关键字
- 全基因组关联研究
- 线头百分比
- 每株铃数
- 单株铃重
- 盐胁迫
- 转录组分析
- 陆地棉