摘要
背景
狭窄的遗传基础、复杂的异体四倍体基因组和重复元件的存在导致了单核苷酸多态性(snp)的发现芸苔属植物juncea(AABB;2n = 4x = 36),速度较慢。双消化RAD (ddRAD)是一种基因组复杂性降低技术,随后采用NGS技术,从印度(天塔拉,RSPR-01和Urvashi)及异国情调(Donskaja四世,Zem 1和EC287711)下。
结果
序列数据分析在两个基因库的基因型中鉴定出6个基因型的10399个snp,读取深度为10倍覆盖。基因组共发现44个高变区(核苷酸变异热点),其中93%为编码基因/区域的一部分。鉴定出的snp的功能是通过在MassARRAY®平台上对一组不同的snp进行基因分型来估计的b . juncea基因型。基于SNP基因分型的遗传多样性和群体研究将基因型分为两个不同的集群,主要基于起源地点。6个形态性状的基因型分析表明,6个性状的平均值在印度和外来基因库之间存在显著差异。对6个性状进行关联分析,在a组和B组祖先基因组的11条染色体上共鉴定出45个显著的标记性状关联。
结论
尽管多样性较窄,但ddRAD测序能够识别两个基因库之间的大量核苷酸多态性。关联分析鉴定了开花和成熟性状之间的共同snp /基因组区域,从而强调了控制开花和成熟的共同染色体区域基因的可能作用芸苔属植物juncea。
背景
芸苔属植物juncea俗称印度芥菜,是印度次大陆、中国北方和东欧国家重要的油料作物。它被广泛种植,用于生产精油和调味品的种子;然而,它的嫩叶也被用作蔬菜或与其他沙拉蔬菜混合。芸苔属植物juncea有两个不同的基因库:印度基因库和东欧基因库(外来的)[1]。东欧基因库在分子水平上表现出更多的多样性,具有更大的产量潜力,而印度基因库的遗传多样性较窄,产量潜力较低[2,3.]。尽管有两个形态多样化的库,但由于复杂的异源四倍体基因组和驯化,该作物经历了狭窄的遗传基础。4]。这种狭窄的遗传基础阻碍了种质改良的进程,因为它减少了发现重要农艺性状的多样等位基因并使其渗入优质种质的机会[5]。
这种遗传增强可以通过利用传统的植物育种方法或标记辅助选择(MAS)在外来(欧洲)和印度基因库之间转移等位基因来实现。MAS要求鉴定和使用与目标性状紧密联系的分子标记,而关联作图不需要事先的分子作图信息,仅基于连锁不平衡(LD)作为鉴定标记-性状关联的重要工具。关联分析通过考虑基因组中不同位置的共分离(或共传播)等位基因,推断出显著的标记-性状关联[6],当与一组密集的分子标记一起使用时,可以对特征进行精细的映射。在油料芸苔属植物。(b . juncea和显著)采用不同类型的分子标记,结合各种模型(GLM、Q、PCA和K),找出各性状与标记之间的密切关系。在大多数关联作图研究中,SSR标记被用于群体研究,因为它们在群体遗传学推断中很有用,而且与双等位基因标记相比,它们具有很高的信息量[7]。然而,snp的高遗传力也使其成为具有古老基因组重复的作物物种遗传多样性和系统发育的良好指标,如玉米b . juncea。各种基于ssr的全基因组关联图谱研究在b . juncea[8,9),显著[10,11,12多种重要的农艺性状。此外,单核苷酸多态性(SNPs)也是精细定位研究的首选,如显著[13,14,15,16,17]。然而,没有这样的研究报告b . juncea,主要是由于无法获得SNP标记。
snp的发现b . juncea进展缓慢主要是由于其狭窄的遗传基础、复杂的异源四倍体基因组和高度重复的区域[18,19]。由于同源和同源DNA序列的存在,两个亚基因组(A和B)的存在使得SNP发现和基因分型更加困难和麻烦。由于多倍体事件导致A和B基因组的重复和三倍,SNP发现的过程进一步复杂化[20.]。为了降低基因组的复杂性,有多种基因组还原方法,利用一组限制性内切酶和特定的选择过程,只对多个基因型中选定的限制性内切片段进行测序,从而同时进行SNP发现和基因分型。生物信息学软件的进步也支持在个体中快速识别真正的snp。
在本研究中,采用改进的ddRADseq方法对6个基因型(分别来自印度和欧洲基因库)的部分基因组进行了测序b . juncea用于snp鉴定和基因分型。使用CLC Genomics工作台上可用的工具开发了一个生物信息学管道,用于检测snp(附加文件)1:图S1)。然后利用这些snp来评估不同群体之间的分子多样性水平和群体结构,并通过关联作图来确定6个形态性状的显著标记-性状关联。
结果
ddrad文库的制备和测序
基于微流体的电泳分析显示,在库制备过程中选择大小后,大多数片段在300 - 400个碱基范围内1:图S2)。在Illumina HiSeq 2000平台上对6个基因型进行序列条形码和汇总测序,共产生约2300万个对端reads,平均每个基因型有383万个reads。6个样本的平均读取质量(Phred评分)为35.02,约89%的样本的Q评分> 30,说明大部分原始数据质量较好。读1的平均质量评分(R1)略好于读2的平均质量评分(R2)1: Table-S3)。读取1的质量略好于读取2的质量,这是由于流池中每个簇内的克隆由于反复冲洗流池而受到的损害最小。两次读取之间的质量分数差异也归因于相位误差[21]。
序列分析和SNP鉴定
在对低质量序列进行修剪后,将处理后的reads组装成contigs,然后将其与参考基因组比对b . juncea(GenBank: LFQT00000000)使用默认参数,约92-94%的reads被映射到参考基因组。单个样本的定位百分比从92.56 (Zem 1)到94.35 (Donskaja IV)。在所有被定位的reads中,超过80%的reads唯一地映射到单个位点(表1)1).
采用概率变异检测方法,将序列与参考基因组比对进行snp鉴定。同源物筛选后,共发现10399个深度至少为10个reads的单核苷酸变异分布在6个基因型中。
高变区snp与蛋白质预测
在大小选择和SNP鉴定过程中所遵循的严格条件导致在基因型中检索到的SNP标记数量较少。尽管SNP分布在所有染色体上,但与其他区域相比,各种染色体上的SNP频率较高的区域被称为高变区或SNP热点。除A08和B03外,其余染色体上共有44个snp高变区或热点。这些染色体上的高变区总数从1到5个不等,这些区域的snp总数从4到11个不等。BlastX对这些高可变序列进行分析,发现近93.2%的高可变区域是编码序列的一部分(表2)2).
不同基因库的形态分析
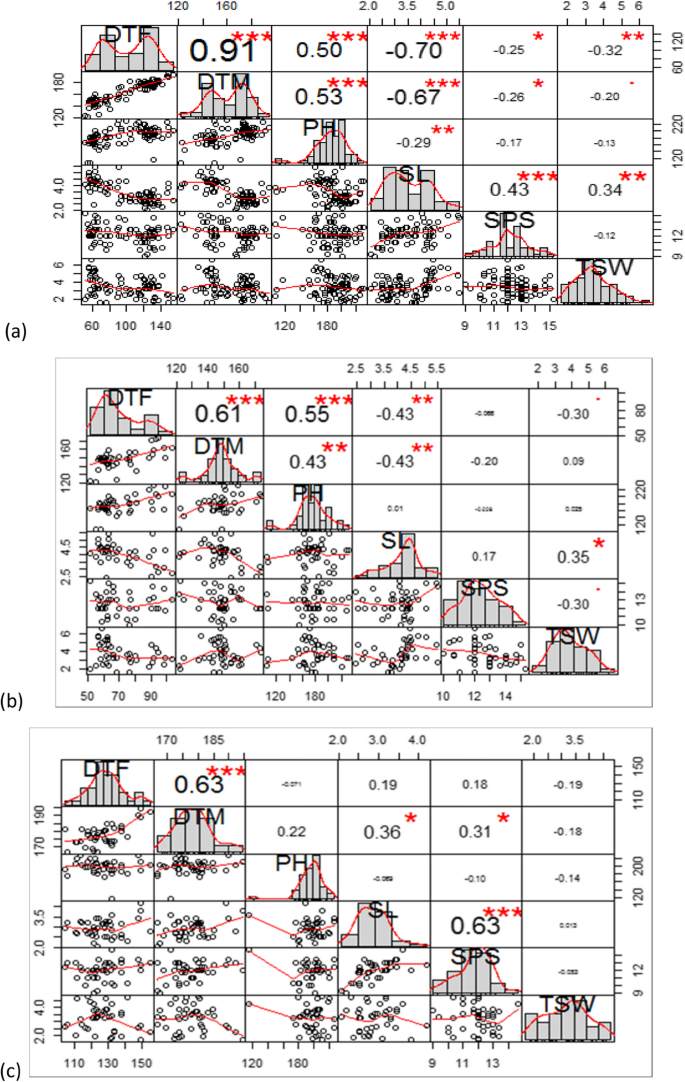
多样化的核心设置b . juncea在2015-16和2016-17两个地点对80个基因型进行了生长和产量性状的鉴定。利用在两个地点采集的数据计算各基因型的平均开花天数(DTF)、成熟天数(DTM)、株高(PH)、穗长(SL)、每穗种子数(SPS)和千粒重(TSW)。对各性状的图相关性分析表明,除DTF和DTM外,各性状在不同核心集中均呈正态分布(图2)。1).考虑到DTF和DTM的双峰分布,利用这两个性状的平均值将不同的核心集划分为两个基因库,即欧洲(外来)基因库和印度基因库。两个基因库的个体图相关分析表明,DTF和DTM等性状均呈正态分布。
在两个基因库中,性状与其他性状的相关性存在差异。在印度基因库中,DTF与除SPS外的其他性状均有较高的相关性;而欧洲基因中的DTF仅与DTM有显著相关。印度基因库的DTM与PH(0.43)和SL(- 0.43)显著相关;该性状与PH不显著相关,与SL(0.36)和SPS(0.31)正相关。PH值与DTF和DTM的相关性在印度显著,而在欧洲不显著。SL与DTF呈负相关,与DTM呈显著负相关;与印度基因库中的TSW呈正相关。而在欧洲基因库中,SL与DTM呈显著负相关。SPS与印度基因库的产量性状均不相关,但与SL和DTM显著相关。 In both the genepools, TSW was not significantly correlated with any of the traits but SL in European. The t-test for means for two genepools indicated that the average values for two genepools were significantly different. ThepStudent’s t检验的-值表明,两个基因库中所有性状的平均值差异极显著(表2)3.).
基于SNP标记的多样性分析与群体结构
共有61个SNP标记广泛分布于整个基因组b . juncea利用基因组对核心集进行鉴定,得到80个基因型的多样性谱。在61个标记中,发现48个SNP标记是多态性的。由于该标记为双等位基因,共扩增了98个等位基因(表2)4).次要等位基因频率范围为0.00 ~ 0.46,平均为0.16。基因多样性和杂合性也表明基因型之间存在显著的差异。基因多样性值为0.013 ~ 0.49,杂合度值为0.012 ~ 0.69,平均为0.16。多态性信息含量(PIC)的取值范围为0.012 ~ 0.371,平均值为0.19。
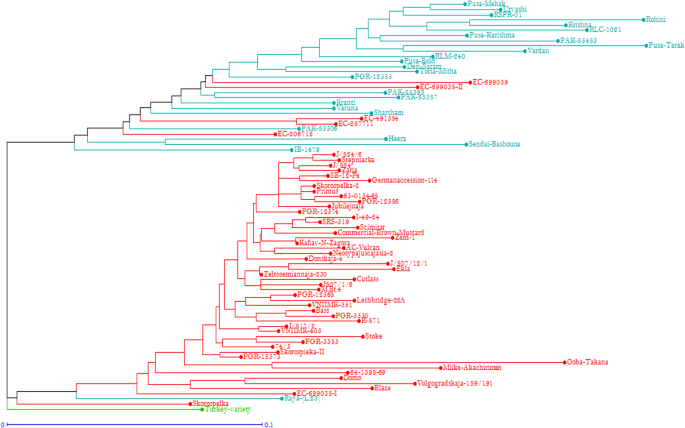
利用structure V2.3.4软件对80个基因型在Hardy-Weinberg平衡下的群体结构进行估算。根据最大似然和ΔK (ΔK)值,确定最优组的数量为2个(图2)。2).利用标记等位基因数据构建的树状图也将80个基因型分为两个不同的集群,来自土耳其的当地选择形成一个单独的组。80个基因型均分为3个主要聚类,其中聚类I、II和III分别含有29个、50个和1个基因型。图1和图2也显示了基因型的亚群(图2)。3.).聚类表明SNP标记能够以较高的准确性将来自地理区域的相关基因型组合在一起。集群I由主要来自印度次大陆的基因型组成,集群II由外来基因型组成。然而,一些外来基因型(EC287711、EC206712、EC491584、EC699038-II和EC699059)与印度基因型被归为一类,这可能是由于这些基因型在本研究中考虑的一些位点上的等位基因组成相同。与61个SNP标记的小子集相比,通过在更多的遗传位点上对它们进行表征,可能进一步完善它们的分组模式。
关联映射分析
利用均匀分布在水稻所有染色体上的61个单核苷酸多态性,对6个产量贡献性状进行了关联分析b . juncea。为了确定真正的标记-性状关联,我们使用了这两种方法p值和标记2的值,并且只有那些重要的关联才被考虑p值< 10−6。在使用的61个snp中,18个snp涉及6个不同性状的45个显著标记-性状关联(表2)5).这些关联定位于18条染色体中的11条b . junceaa染色体共有23个标记性状关联,b染色体共有22个标记性状关联。DTF的关联最多,为16个,其次是SL 13个,DTM 12个,TSW 2个,SPS和ph各1个。除A07和B02各1个外,几乎所有snp都涉及不同性状的多重关联。B04上的一个SNP标记与4个不同的性状相关;发现8个snp与3个不同的性状相关,随后发现6个snp分别与2个性状相关。与DTF、DTM和SL相关的SNP标记分布在A染色体和b染色体上,并且这些关联存在于多条染色体上。SPS、PH和TSW的snp仅分布在b基因组的单染色体上。的p所有关联的-值都小于由错误发现率的p值(0.015)确定的阈值。的p-值范围为1.26E-05 ~ 1.15E-18,表型方差贡献(r2)范围为0.20至0.89。
讨论
大量的基于分子标记的研究已经导致了对基因组成的更深入的了解芸苔属植物物种。SNP标记对于农艺重要基因的精细定位至关重要,目标是实现标记辅助育种的优良作物品种。snp在基因组中的分布频率要高得多,并已用于开发高密度分子遗传图谱和感兴趣区域的精细图谱。基因组中SNPs的丰度、低突变率和高遗传力抵消了双等位基因的缺点。snp随机分布在整个基因组的重复和非重复区域,但存在于基因/非重复区域的snp非常重要。异源多倍体基因组祖细胞中同源区域的存在,除了重复元素外,还增加了基因组的复杂性。然而,最近在降低基因组复杂性方面的进展加上NGS技术已经非常成功地开发了作物的全基因组snp。
在目前的研究中,一对限制性内切酶(均方误差我和囊I)用于对的独特区域进行ddRAD测序b . juncea。类似的降低基因组复杂性的技术也被用于几种作物[22,23,24动物[25]和昆虫[26,27)的物种。已经提出了对这种技术的一些修改。对于其他多倍体作物(棉花),采用GR-RSC (Genome Reduction-Restriction Site Conservation,基因组还原限制性位点保护)技术,并结合生态国际扶轮和论坛I限制性内切酶的大小选择在450到600 bp之间[28而另一项研究更倾向于使用的组合生态国际扶轮和Msp尺寸选择在200-400 bp之间[25]。
基因型测序后,共获得6个品种的2300 MB配对端序列数据b . juncea每个基因型平均有383.33 MB。同样,水稻dd-RAD测序平均获得147.3 MB数据[29]。考虑到基因组的大小b . juncea955 MB和1150 MB 6个基因型的单读测序数据,单个基因型平均占整个基因组的20%,从而将基因组复杂性降低了近5倍。另一项对芸苔属物种的研究报告了ddRAD后几乎相似的基因组部分的减少[30.]。两个reads的平均质量分数在34.63 ~ 35.40之间,90%的序列数据的Q分数至少为30,表明测序reads具有高质量的参考基因组比对和SNP鉴定。用不同的基因组复杂性降低方法(SLAF-seq)对茶叶进行高通量测序的质量分数相似[31]。由于Q值较高,大部分(近83%)的序列读段被定位到参考基因组中的独特位置,这表明ddRAD方法可用于定位基因组中的独特区域。读取到唯一区域的映射也确保了来自重复或平行区域的snp被排除在进一步的分析之外。
通常,snp分布在整个基因组中,并且发现snp的平均分布频率在100 nt到500 nt之间。在本研究中,93%的snp高变区(热点)发生在芸苔属植物junceasnp分布在编码区的上游、下游和基因间区。这些高变区多数SNP频率小于10nt。此外,在携带SNP热点的染色体区域检测到40个基因/编码序列,可能指出这些SNP在这些基因的表达中可能具有调节作用。虽然,以往的研究很少报道重复区域的SNP热点,这主要是由于DNA聚合酶的错误导致链滑移和不平等交换[32,33]或由于突变热点或重组热点的存在[34]。本研究发现,每条染色体上的SNP热点是随机分布的,在50 nt染色体区域内,这些热点涉及的SNP数量从4到11不等。环境胁迫造成的高选择压力可能导致基因区域中突变等位位点的积累,从而提高作物在不利环境条件下的存活率[35,36]。
在一组高度多样化的基因型中,功能性snp的高比例(97%)表明ddRAD技术在文库制备过程中始终针对不同个体的相同位点的准确性,部分原因是用于序列定位和复杂和多倍体作物snp鉴定的生物信息学工具的改进。通过RAD-seq鉴定的snp及其在先前研究中的修饰也在其他作物中显示出类似的功能水平[37,38]。在本研究中,从61个功能性snp的子集中获得的双等位基因数据能够分组多样化b . juncea基因型分为两个主要集群-印度和外来(欧洲)基因库。多样性和聚类结果与前人基于SSR和其他标记系统的研究结果一致。基于snp的多样性分析还得出结论,均匀分布的一小部分snp将对各种遗传分析非常有用。
6个性状的形态特征在相关矩阵上显示出非常有趣的模式。综合各基因型分析,DTF和DTM的双峰分布表明,这两个性状在印度和欧洲的基因库中受不同组基因控制。欧洲基因库传统上是在低温短日条件下驯化的,而印度基因库更有利于在印度次大陆西北部平原的中低温条件下播种。印度和欧洲基因库DTF和DTM分别在相关矩阵中呈单峰分布,进一步强化了印度和欧洲基因库DTF和DTM由不同基因控制的假设。然而,基因型和表型之间的详细相互作用可以通过QTL分析和其他遗传分析来研究。
在本研究中,使用61个snp的共同子集进行多样性,种群结构和关联分析。对于多样性和群体结构分析,snp子集能够将80个基因型分为两个不同的集群,每个集群都由来自印度和欧洲(外来)基因库的基因型过度代表;这表明了在各种遗传分析中使用稀疏但统一定位的snp策略的有效性。
SNPs的一个子集,代表染色体的所有区域b . juncea用于识别显著的标记-性状关联。利用SNP亚群进行关联分析,可以对不同染色体上的各种农业形态性状进行基因定位,确定基因组区域,利用大量分子标记进行性状/基因组区域的精细定位。大多数snp被鉴定为与多个性状相关,因此本质上表明要么是多个性状的基因聚类,要么是同一基因组区域内调节多个性状的同一组基因参与。在这些性状中,DTF和DTM都有共同的SNP/基因组区域与它们相关,这意味着这两个性状的基因聚集在一起,或者可能有相关/协调的基因表达。最近的一项研究,使用F2人口地图,在芸苔属植物显著还发现了与开花时间和各种生长相关形态性状有关的qtl(和eqtl)在染色体A10的一个共同基因组区域的共定位[39]。在另一项研究中,各种品质和营养性状的qtl再次被定位到双单倍体群体遗传图谱的共同区域芸苔属植物显著[40]。利用染色体片段代换系(cssls)也报道了这种多性状的qtl集群芸苔属植物拉伯[41]。在目前的研究中,在印度和欧洲的基因库中,DTF和DTM性状之间的高相关性也表明,在早期的一项研究中,这两种性状的共同基因组区域(和snp)很可能存在关联[42]。
三个多倍体物种之间存在共同的祖先基因组,导致了关联分析结果的鉴定和比较。在目前的研究中,DTF的关联被定位到A-和B-基因组染色体上。类似的开花时间基因已经在植物的A-和B-亚基因组上被鉴定出来b . juncea[43]。在本研究中分别鉴定出6.8 MB (A06_6796237)和23.4 MB (A06_23478761)与DTF高度显著相关,这与开花时间相关(FTR)基因的结果一致。利用转录组分析,在A06染色体7.2 MB ~ 21.6 MB区鉴定出33个开花时间相关基因[j]。44]。当前研究的关联分析结果表明,稀疏但均匀定位的snp子集将非常有用,可以为感兴趣的性状划分基因组区域。
结论
这是第一个使用ddRAD-seq来开发snp的报道芸苔属植物juncea。单核苷酸多态性最初仅从6个基因型的序列比较中得到;然而,当对不同的基因型进行测试时,发现这些snp具有功能。用于关联分析的snp也被发现与6个形态性状显著相关。考虑到芸苔属植物juncea由于遗传基础较窄,本研究鉴定的snp将为连锁作图、精细作图和关联分析等各种遗传研究提供良好的来源。
方法
植物材料和DNA提取
一套六个b . juncea选择基因型(来自印度和外来种质各3个)用于ddRAD文库的制备。天塔拉(BJI-1),Urvashi(BJI-2)和RSPR-01(BJI-3)是从印度基因库中选育的Zem 1(BJE-1),Donskaja四世(BJE-2)和EC287711(BJE-3)均选自欧洲(外来)基因库。种子采购自印度德里大学(南校区)(Deepak Pental博士)和印度新德里国家植物遗传资源局。对80个品种进行SNP基因分型b . juncea从植物基因资源,农业和农业食品,加拿大和遗传与植物育种部,skuast -查谟,印度采购的基因型(附加文件)1: Table-S4)。采用改进的SGS缓冲法分离总基因组DNA [45],用纯化的DNA制备dd-RAD文库。
形态学资料评价与统计分析
不同核心组的表型数据b . juncea在2015-16年和2016-17年的两个不同地点也有记录。收集了6个性状的数据:天开花(DTF -从播种日期天数的50%,植物的花在每个情节),天到期(DTM -天数从播种日期豆荚在75%的植物在每个情节都晒黑),株高(PH值——米)长角果长度(厘米)SL -,每长角果种子(SPS -平均数量的种子在单一舱/长角果)和千粒重(天水围-重量在1000克的随机种子收集)。每个基因型的性状值定义为同一位置两个重复的平均值。性状间的相关系数用学生的学习任务并对方差分量进行了计算。
ddRAD文库开发和NGS测序
ddRAD-seq协议[25,46]用于从6个序列条形码简化表示库(RRLs)中构建芸苔属植物juncea基因型。制备ddRAD文库时,用10微克纯化DNA酶切完成Mse我和囊我。酶切DNA用0.8%琼脂糖凝胶分离;300 - 400bp的片段凝胶切除并洗脱。然后将洗脱和纯化的DNA进行末端修复,按照制造商的协议连接短dA-tail并与适配器连接。用PCR扩增连接的DNA,并将Illumina特异性指数和流式细胞退火序列添加到片段DNA中。对每6个基因型使用6个不同的索引序列,以便于池化过程。将6份DNA样本归一化至终浓度为50 ng/μl,汇总至终体积为300 μl,生成简化的表示库。然后使用Illumina HiSeq 2000对汇集的dd-RAD文库进行测序,产生100 bp的成对末端reads。
序列预处理和SNP检测
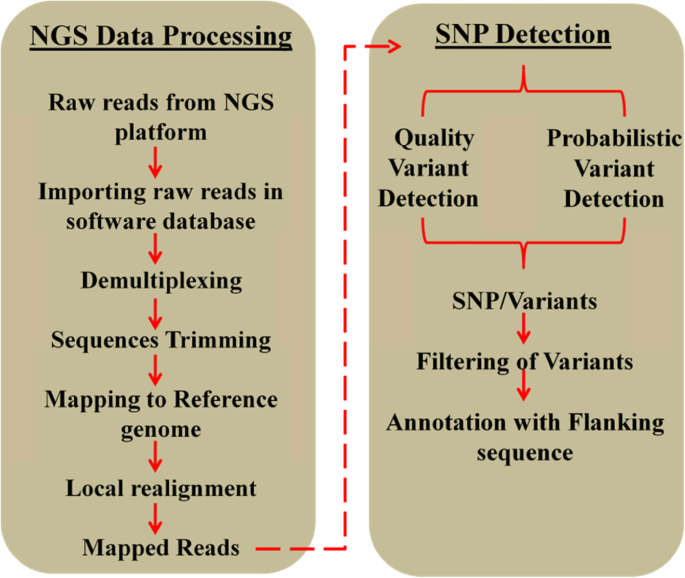
测序后获得的ddRAD-seq reads使用CLC Genomics Software进行生物信息学分析,以获得高质量的SNP集。配对的末端测序reads通过管道进行一系列步骤(解复用、修剪、与参考基因组作图、局部重组、snp检测和与侧翼序列注释)。下面的滤波方案(图1)4)来最大限度地保留真基因多态性snp:(1)从正端裁剪13个碱基,从反端裁剪3个碱基;(2)将作图参数设置为-错配成本:2,插入成本:3,缺失成本:3,长度分数:0.5,相似分数:0.95,我们选择进行局部比对而不是全局比对,因为如果末端与参考文献存在许多差异,则允许末端未对齐。(3)使用概率SNP检测方法对映射reads进行SNP检测,参数为最小覆盖率:4,变异概率:98.00,倍性:2,(4)侧翼序列为400bp。对于读的映射,芸苔属植物juncea基因组作为参考基因组[47]。
snp验证和基因分型
从每条染色体中选择3-4个SNP位点,共61个SNP位点的子集,并在不同的基因组中进行验证b . juncea。每个SNP侧翼的序列被用来合成正向、反向和反向iPLEX通用扩展引物使用Agena xassay设计套件V2.0软件。将正、反向PCR引物稀释至100 μM浓度iPLEX通用延伸引物稀释至500 μM浓度。实验步骤包括:(1)利用正、反向引物进行多重PCR,(2)对虾碱性磷酸酶(SAP)清理反应,(3)iPLEX(4)树脂清洗反应去除盐,(5)引物延伸产物在光谱芯片上斑点化,(6)使用MALDI-TOF质谱进行光谱芯片检测。通过MassARRAY TYPER 4.0软件进行基因型鉴定。
种群与多样性分析
SNP基因分型数据用于群体结构和遗传多样性分析[48在Singh等人之后[49]。后验概率(qK)估计有10,000次重复,然后是100,000次迭代。在结构分析中,假定多样性群体遵循混合模型和相关等位基因频率,而没有先验群体信息。进行结构分析,每组5个重复K1 ~ 5。的ΔK采用Structure Harvester软件计算[50的最优值K。每个结构分析重复的隶属系数阈值为70%,使用软件CLUMPP生成Q矩阵[51];然后用DISTRUCT软件绘制Q矩阵[52]。使用Powermarker v3.51软件计算多态信息含量(PIC)值和等位基因频率[53]。在Darwin5软件中实现了无加权邻居连接树方法[54构建系统发育树;这棵树的自举值是通过在1000次的位置重新采样来确定的。
利用数据库进行基因鉴定和注释
将snp /高变区侧翼序列与对照进行比较b . juncea使用BLASTX(截止e值为1E-10)在蛋白数据库中识别相应的序列[55]。
关联分析
利用该品种的基因型(snp)和表型数据进行关联分析芸苔属植物基因型和群体结构数据(Q矩阵)56]。标记-性状关联分析采用TASSEL 3.0软件进行,GLM程序保持显著关联阈值为P< 0.01。
数据和材料的可用性
本研究中从6个基因型中鉴定出的snp已提交至EMBL-EBI数据库的欧洲变异档案馆,登录号为PRJEB26751。
缩写
- BJE:
-
芸苔属植物juncea异国情调的
- BJI:
-
芸苔属植物juncea印度
- ddRAD:
-
双消化,Restriction一个ssociatedDNA
- DH:
-
迄今己双
- DTF:
-
开花的日子
- DTM:
-
到期
- 全球语言监测机构:
-
广义线性模型
- LD:
-
连锁不平衡
- MALDI-TOF:
-
基质辅助激光解吸/电离飞行时间
- MAS:
-
分子辅助选择
- 主成分分析:
-
主成分分析
- PH值:
-
株高
- 图片:
-
信息内容
- QTL:
-
数量性状位点
- RRLs:
-
简化表示库
- SL:
-
长角果长度
- SLAF-seq:
-
特异性位点扩增片段测序
- 单核苷酸多态性:
-
单核苷酸多态性
- SPS:
-
每西里克种子数
- SSRs:
-
简单序列重复
- 天水围:
-
千粒重
参考文献
- 1.
Dhaka N, Rout K, Yadava SK, Sodhi YS, Gupta V, Pental D, Pradhan AK。利用QTL分析分析印度和东欧基因库系种子重的遗传解剖及等位基因变异检测芸苔属植物juncea。理论与应用,2017;130(2):293-307。
- 2.
Yadava SK, Arumugam N, Mukhopadhyay A, Sodhi YS, Gupta V, Pental D, Pradhan AK。小麦产量相关性状的QTL定位芸苔属植物juncea荟萃分析和上位相互作用使用两个不同的杂交东欧和印度基因库系。理论与应用,2012,25(7):1553-64。
- 3.
Pradhan AK, Sodhi YS, Mukhopadhyay A, Pental D.印度芥菜杂种优势育种(芸苔属植物junceaL. Czern & Cross):对产量贡献杂种优势的组成性状分析。Euphytica。1993;69:219-29。
- 4.
Lukens LN, Quijada PA, Udall J, Pires JC, Schranz ME, Osborn TC。古代和近代芸苔属作物物种的基因组冗余和可塑性。中国生物医学工程学报,2004;32(4):564 - 564。
- 5.
Srivastava A, Gupta V, Pental D, Pradhan AK。基于aflp的农艺重要天然系和部分新合成系遗传多样性评价芸苔属植物juncea。理论与应用,2001;102:193-9。
- 6.
王晓明,王晓明。植物遗传研究进展。大自然。2003;456:720-3。
- 7.
罗旭,马超,岳勇,胡凯,李勇,段志,等。揭示油菜籽收获指数的复杂性状(芸苔属植物显著)与关联映射。BMC基因组学。2015; 16(1): 379。
- 8.
李建军,李建军,李建军,等。水稻根系性状与产量性状的全基因组关联图谱芸苔属植物juncea(l)Czern & Coss。分子育种,2015;35(1):48。
- 9.
Dhaka N, Mukhopadhyay A, Paritosh K, Gupta V, Pental D, Pradhan AK。基因ssr的鉴定及ssr连锁图谱的构建芸苔属植物juncea。Euphytica。2017;213(1):15。
- 10.
蔡东,肖宇,杨伟,叶伟,王斌,尤纳斯明,吴健,刘凯。油菜6个产量相关性状的关联图谱(芸苔属植物显著l .)。理论与应用,2014;127(1):85-96。
- 11.
Gyawali S, Harrington M, Durkin J, Horner K, Parkin IA, Hegedus DD, Bekkaoui D, Buchwaldt L.部分抗性的微卫星标记全基因组关联定位菌核病sclerotiorum在一个世界的收藏芸苔属植物显著。动物学报,2016;36(6):72。
- 12.
傅Y,张D,格里森M,张Y, B,华年代,丁H,允许妇女M,李J,钱W, Yu H .籽油含量的QTL分析芸苔属植物显著通过关联映射和QTL映射。Euphytica。2017;213(1):17。
- 13.
曲春明,李思敏,段小军,范建华,贾立德,赵海燕,陆凯,李金宁,徐晓峰,王荣。利用关联定位技术鉴定种子硫代葡萄糖苷含量候选基因芸苔属植物显著[j] .基因工程,2015,6(4):1215-29。
- 14.
李F,陈B,徐K,高G,燕G,乔J,李J,李H,李L,张小X, t的全基因组关联研究在油菜株高和主要分支数量(芸苔属植物显著).植物科学,2016;42(2):169 - 177。
- 15.
罗欣,薛铮,马超,胡坤,曾铮,窦生,涂静,沈健,易斌,傅涛。联合全基因组关联和转录组测序揭示了油菜籽下胚轴延长的复杂多基因网络。芸苔属植物显著l .)。科学通报,2017;7:41561。
- 16.
赵瞿C,贾庆林L,傅F, H,陆K, L,徐X, Y,李,王R,李j .全基因组关联映射和脂肪酸组成的候选基因的识别芸苔属植物显著L.使用SNP标记。BMC基因组学。2017; 18(1): 232。
- 17.
赵慧,王旭,赵伟,陈玲,郭玲,李波,侯冬,濮松,张玲,王辉。利用全基因组数量性状位点定位分析水稻收获指数及相关性状的遗传分析芸苔属植物显著。品种学报,2019;69(1):104-16。
- 18.
刘建军,刘建军,刘建军,等。植物遗传多样性的研究进展拟南芥基因组。植物学报,2000;12(7):1093-101。
- 19.
刘建军,李建军,李建军,等。十字花科植物基因组学研究进展。植物科学进展,2006;11(11):535-42。
- 20.
杨天杰,金钟生,权思杰,林国宝,崔宝宝,金佳,金美,朴建勇,林明辉,金海,等。植物三重花C区二倍体化过程的序列水平分析芸苔属植物拉伯。植物学报,2006;18(6):1339-47。
- 21.
Schirmer M, D 'Amore R, Ijaz UZ, Hall N, Quince C. Illumina误差谱:解决宏基因组测序数据的精细尺度差异。生物信息学,2016;17:12。
- 22.
李建军,李建军,李建军,等。利用下一代限制性位点相关DNA (RAD)测序技术检测花生全基因组多态性。动物学报,2015;35(7):145。
- 23.
张建军,张建军,张建军,张建军。利用限制性相关DNA (RAD)测序发现普通豆的SNP,用于遗传多样性和群体结构分析。中国生物医学工程学报,2016;11(11):1182 - 197。
- 24.
Marrano A, Birolo G, Prazzoli ML, Lorenzi S, Valle G, Grando MS.葡萄种质资源中单核苷酸多态性的rad测序研究[j]。诉酿酒用葡萄l .)。PloS one。2017; 12 (1): e0170655。
- 25.
张建军,张建军,张建军,张建军。双消化RADseq:一种在模型和非模型物种中重新发现SNP和基因分型的廉价方法。PloS one。7(5): e37135。
- 26.
Carmichael SN, Bekaert M, Taggart JB, Christie HR, Bassett DI, Bron JE, Skuce PJ, Gharbi K, Skern-Mauritzen R, Sturm a。Lepeophtheirus salmonis),采用RAD测序。科学通报,2013;8(10):e77832。
- 27.
Burford MO, Coyle K, Daniels HV, Labadie P, Reiskind MH, Roberts NB, Roberts RB, Schaff J, Vargo EL。一组具有重要生态和经济意义的非模式昆虫和鱼类分类群的通用双消化RAD测序方法的建立。中国生物医学工程学报,2016;16(6):1303-14。
- 28.
Byers RL, Harker DB, Yourstone SM, Maughan PJ, Udall JA。异源四倍体棉花SNP检测方法的建立与定位。理论应用学报,2012;24(7):1201 - 1414。
- 29.
杨国强,陈彦明,王建平,郭超,赵玲,王学勇,郭勇,李玲,李大志,郭志。一种用于被子植物SNP发现和基因分型的通用简化ddRAD文库制备方法的建立。植物学报,2016;12(1):39。
- 30.
陈翔,李翔,张斌,徐军,吴忠,等。利用伪参考序列检测多倍体作物限制性内切片段相关多态性并进行基因分型:以异源四倍体为例芸苔属植物显著。生物医学工程学报,2013;14:346。
- 31.
马建强,黄丽,马春林,金建强,李春春,王荣科,郑洪洪,姚明忠,陈磊。基于SLAF-seq的茶树高密度遗传图谱的大规模SNP发现与分型研究。科学通报,2015;10(6):e0128798。
- 32.
刘建军,刘建军,刘建军,等。基于遗传基因复制滑移的生物适应性研究。中华生物医学工程学报,2016;33:557 - 566。
- 33.
周强,周晨,郑伟,Mason AS,樊森,吴超,付东,黄勇。基于slf -seq的全基因组SNP标记揭示油菜育种痕迹(英文)芸苔属植物显著l .)。植物科学学报,2017;8:648。
- 34.
李建军,李建军,李建军,等。植物减数分裂的分子生物学研究。植物学报,2015;33(6):357 - 357。
- 35.
张建军,张建军,张建军,等。分子标记技术在植物育种中的应用。方法:中华医学杂志。2015;45(5):13 - 27。
- 36.
魏格尔D, Nordborg M.种群基因组学对野生植物物种适应的理解。科学通报,2015;49:315-38。
- 37.
吴旭,任超,乔希涛,王涛,徐东,阮海涛。大豆高通量测序发现SNP。中国生物医学工程学报,2010;11:469。
- 38.
张建军,张建军,张建军,等。利用限制性限制相关DNA (RAD)测序发现普通豆的SNP,用于遗传多样性和群体结构分析。生物化学学报,2016;29(3):1277 - 1291。
- 39.
李锐,郑凯,Davis JT,等。整合的QTL和eQTL定位为一个新的合成异源多倍体F2群体的脂肪酸组成、开花时间和生长性状提供了见解和候选基因芸苔属植物显著。植物科学,2018;9:1632。
- 40.
Fakheri BA, Solouki M, Möllers C, Rezaizad a .冬季油菜(Brassica napus L.)双单倍体群体QTL控制农艺性状的定位。中国生物医学工程学报,2018;37(5):1389-406。
- 41.
李欣,王伟,王忠,李坤,林玉平,朴铮。染色体片段代换系的构建及其对水稻开花和形态性状的QTL定位芸苔属植物拉伯。植物科学,2015;6:432。
- 42.
吕红,王强,刘霞,韩峰,方志,杨林,等。白菜全基因组图谱揭示与主要农艺性状相关的新QTL群(甘蓝变种l .)。植物科学前沿。2016; 7:989。
- 43.
赵宁,刘超,孟艳,胡忠,张敏,杨健。异源多倍体开花调控基因的鉴定芸苔属植物juncea。园艺学报,2019;5(3):109-19。
- 44.
简慧,张安,马军,王涛,杨波,双利生,刘磊。联合QTL定位和转录组测序分析揭示了中国小麦开花时间候选基因芸苔属植物显著。生物医学工程学报,2019;20(1):21。
- 45.
李建军,李建军,李建军,等。高质量种子DNA提取技术的研究进展。作物改良学报,2017;31(5):637-47。
- 46.
杨国强,陈彦明,王建平,郭超,赵玲,王学勇,郭勇,李玲,李大志,郭志。一种用于被子植物SNP发现和基因分型的通用简化ddRAD文库制备方法的建立。植物学报,2016;12(1):39。
- 47.
杨军,刘东,王旭,纪超,程飞,刘波,胡忠,陈生,等。异源多倍体的基因组序列芸苔属植物juncea差异同源基因表达对选择的影响分析。植物学报,2016,48(10):1225-32。
- 48.
Pritchard JK, Stephens M, Donnelly P.基于多位点基因型数据的种群结构推断。遗传学。2000;155(2):945 - 59。
- 49.
Singh R, Bollina V, Higgins EE, Clarke WE, Eynck C, Sidebottom C, Gugel R, Snowdon R, Parkin IA。单核苷酸多态性鉴定及基因分型亚麻荠漂白亚麻纤维卷。动物学报,2015;35(1):35。
- 50.
伯爵哒。结构收割机:一个可视化结构输出和实现埃文诺方法的网站和程序。资源保护,2012;4(2):359-61。
- 51.
雅各布森M,罗森博格NA。在种群结构分析中处理标签切换和多模态的聚类匹配和排列程序。生物信息学。2007;23(14):1801 - 6。
- 52.
罗森博格NA。用于图形显示人口结构的程序。生物化学学报,2004;4(1):137-8。
- 53.
刘凯,缪思伟。PowerMarker:用于遗传标记分析的集成分析环境。生物信息学。2005;21(9):2128 - 9。
- 54.
佩里尔X,杰奎姆-科莱JP。2006.达尔文的软件http://darwin.cirad.fr/darwin。
- 55.
刘松,周忠,陆军,孙峰,王松,刘海,姜勇,kuuktas H, Kaltenboeck L, Peatman E,刘铮。鲶鱼基因组相关SNP的生成及其高密度SNP阵列的构建。生物医学工程学报,2011;12(1):53。
- 56.
张志强,张志强,张志强,张志强。TASSEL:软件的关联映射复杂性状在不同的样本。生物信息学。2007;23(19):2633 - 5。
致谢
不适用。
资金
作者确认收到了印度新德里政府生物技术部2013年至2016年的研究资助(BT/PR3946/AGR/2/839/2011)。
作者信息
从属关系
贡献
RS设计全实验;RS和JS进行生物信息学分析;RS、JS、SS和SMZ进行遗传分析;JS、VS、GS、IS、SKS设计并执行MassARRAY分析;RS, JS, SKG和RKS撰写手稿。所有作者都阅读并认可了稿件。
相应的作者
道德声明
伦理批准并同意参与
-不适用。
发表同意书
-不适用。
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
附加文件1:图S1。
CLC Genomics Workbench中snp /变异体检测工作流程设计(红色实线表示输入文件;蓝色虚线表示输出文件)。图S2。对制备的文库进行生物分析仪分析(蓝色峰表示阶梯峰,红色峰在35和10,380 bp处表示内部标准)。表S3。6个基因型ddrad序列数据综述。表S4。的细节芸苔属植物juncea用于关联分析的基因型。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
苏丹,J.,辛格,R.,夏尔马,S.。et al。基于ddRAD序列的基因库间snp鉴定及关联分析芸苔属植物juncea。BMC Plant Biol19日,594(2019)。https://doi.org/10.1186/s12870-019-2188-x
收到了:
接受:
发表:
关键字
- 年代炉火nucleotidepolymorphisms(单核苷酸多态性)
- 双消化,Restriction一个ssociatedDNA (dd-RAD)
- MassARRAY
- 多样性分析
- 关联映射