抽象的
背景
雌雄异株存在于雄性不育的雌性和雌雄同体的物种中。高等植物的雄性不育通常是由线粒体基因组编码的细胞质雄性不育基因与核编码的恢复系基因之间的核相互作用引起的。线粒体和核编码转录组在雌性和雌雄同体中被深入研究,但对质体中性别特异性基因表达知之甚少。我们比较了一个雌雄异株物种的两个单倍型中雌性和雌雄同体的质体转录组硅宾寻常的已知的CMS候选基因。
结果
我们从五个单倍型生成完全的体液基因组序列美国寻常的包括单倍型KRA和KOV,其完整的线粒体基因组序列已经发表。我们根据其质体序列构建了系统发育树美国寻常的.而低地美国寻常的单倍型(KRA和KOV)聚在一起,欧洲高山植物的亲缘关系较早发生分化。美国寻常的属于中硅宾但我们仍然检测到该物种的两个遗传资源之间的212个替换和112个插入。我们估计在ndhF基因,可能反映适应的美国寻常的高海拔,或放松选择。我们比较了雌性和雌雄同体体积体转录瘤之间的覆盖和编辑率的深度,发现两种性别之间没有显着差异。我们确定了51个独特的C到U编辑网站的塑植基因组美国寻常的,其中38个在蛋白质编码区,28中,内含子和11个中的11个。编辑网站psbZ基因只在两个质体基因组中的一个被编辑。
结论
结果表明,两种单倍型的质体转录组无显著性差异美国寻常的.它表明塑性基因的基因表达不受花蕾的CMS的影响美国寻常的,尽管两性在特定组织中的质体基因表达仍有差异。我们揭示了两者质体转录组之间的差异美国寻常的单倍型在编辑率和几个反义转录物的覆盖范围内。我们的结果记录了塑性基因组和转录om的变异美国寻常的.
背景
雌雄异株是一种雌雄同体(H)和雌性(F)个体同时存在于同一种群中的植物繁殖系统。大约2%的被子植物属有这种现象[1].Mitococondrial编码的细胞质雄性不育(CMS)基因的相互作用和核恢复剂的相互作用,雄性不育(但不完全)牙科生殖植物物种编码。RF.)基因2].CMS用于农业中以生产高产率高的杂种种子,因此在作物中广泛研究,例如,水稻[3.,4],向日葵[5)、玉米(6或糖甜菜[7].
尽管雌雄异株现象在被子植物中广泛存在,但对野生植物CMS的研究却很少。硅宾寻常的(膀胱剪枝)作为自然种群中雌雄异株和CMS研究的模型出现于20多年前[8].属硅宾具有多样化的交配系统的物种 - 雌雄同体,雌雄同体和DIOECY [9,10].交配系统影响细胞器位点的DNA序列变异,这在雌雄异株中通常较高硅宾最可能是由于作用于CMS基因座和整个细胞器基因组的平衡选择[11,12].
除了选择模式外,替代率是影响植物细胞器中DNA序列变异的另一种基本因素。虽然植物细胞素基因组通常替代率通常低,但在某些系统发育谱系中高度升高,包括该属硅宾[13].特别是两个物种硅宾noctiflora和硅宾conica- 在线粒体和塑性基因组中实现了极端的序列和结构演变[14,15].
硅宾寻常的具有高于高止性的细胞内基因组替代率,但与其他相比相对缓慢地发展硅宾物种 [16,17].然而,线粒体基因组的种内结构重排美国寻常的是极端的,不仅涉及基因间DNA的频繁丢失和获取,而且还涉及编码序列的变化[16,18].
完全测序的线粒体基因组的可访问性美国寻常的RNA-seq方法的发展使得该物种线粒体综合转录组的构建成为可能。这种F和H植物之间转录组的比较美国寻常的已经在他们的线粒体基因组中发现了候选CMS基因,并发现了不同单倍型之间的RNA编辑率差异,这增加了种内蛋白质的变异[18,19].
的质体基因组美国寻常的对这一物种线粒体基因组的研究程度要低一些。仅一个单倍型质体基因组的完整序列已发表[15],目前还没有对该物种进行全面的质体转录组分析。
更详细地了解塑性基因组进化的洞察力美国寻常的,我们组装了来自该物种的五个单倍型的完全体层序列,其中来自高山的两种牧草,适应高海拔,遗传遥远来自源自低地群体的剩余三个单倍型[20.].CMS是一种复杂的表型,与一些线粒体和许多核基因表达的深刻变化有关[21,22,23].然而,质体转录组的研究很少与CMS有关,尚不清楚雌雄异株植物质体基因的转录是否受雄性不育表型的影响。因此,我们比较了F和H两种单倍型植物的质体转录组美国寻常的(KRA和KOV),线粒体转录组也可用[18,19].在这两种单倍型中都发现了CMS候选基因,这可能将CMS对质体转录组的潜在影响与特定的CMS候选基因联系起来。我们没有发现两性之间的任何显著差异,但发现了两性转录组之间有趣的差异美国寻常的单倍型。
结果
完全质体基因组美国寻常的
我们组装了五种完整的塑性基因组美国寻常的来自欧亚大陆的单倍型D11, VS1, ZE2, KRA和KOV(表1).它们的长度在151463 bp到151572 bp之间,包含一个长单拷贝区(LSC)、一个短单拷贝区(SSC)和两个反向重复序列(IRs)2).重复区和单拷贝区之间的界限和基因含量与之前发表的质体基因组相同美国寻常的SD2 [15].
我们鉴定了简单序列重复序列(SSR)的位置,串联重复单元的短序列,在六个质体基因组美国寻常的在研究。我们发现超过5个核苷酸的单核苷酸重复序列有871个,但其中超过9个核苷酸的只有46个(附加文件)1此外,有62个二核苷酸,8个三核苷酸,8个四核苷酸,只有一个五核苷酸。SSRs由于其变异性,在群体遗传研究中是有用的标记。在大多数单核苷酸区域,由于异质性而产生的单核苷酸单位数量的个体内差异被观察到。重复单位高于两个核苷酸的SSRs,不受异质性影响,因此可以推荐用于质体基因分型美国寻常的.我们发现了19个二、三、四核苷的位置,这些位置在分析的质体基因组中存在差异美国寻常的.
系统发育关系和序列多态性
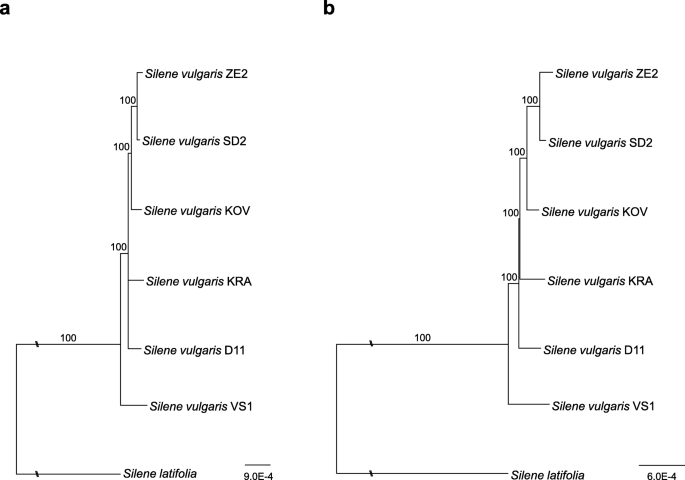
单倍型VS1和D11为高山质体美国寻常的人群,在高于1200米的海拔地区发生。s。L,首先在基于级联蛋白质编码序列构建的系统发育树上分叉美国latifolia作为一个小组(图。1b)。当使用除均聚物之外的整个体层序列除了大于五个之外的整个体层序列时,确认了相同的拓扑(图。1a).在后者的系统发育树中,西伯利亚的KRA单倍型和阿尔卑斯山的D11单倍型的位置未被确定。最大似然(maximum-likelihood, ML)方法得到的结果与MrBayes生成的输出结果一致,只是始终要解析KRA和D11的位置(额外的文件2:图S1,附加文件3.:图S2)。
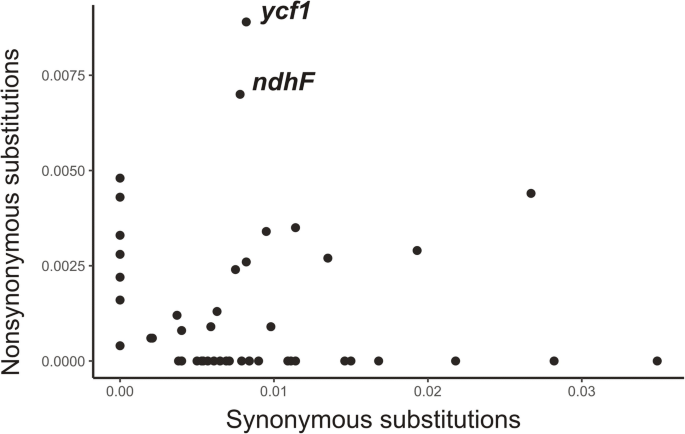
双塑性基因组成对比对的诱导数美国寻常的从33到112变化,核苷酸取代范围为35至212(表2).与Jeseníky Mts中VS1单倍型的等位基因多态性最多,与该单倍型在系统发育树中的基本位置一致美国寻常的单倍型。诱导在63个基因区域,10内内含者中发生,以及两个编码区(ndhD和YCF1.),这也显示了非同义替换的数量增加(附加文件1:数据集3)。
美国寻常的质体基因的多态性程度各不相同。77个独特的蛋白质编码基因中有30个是相同的,另外25个基因只携带相同的分离位点,因此编码的蛋白质在6个质体单倍型中是相同的。只有22个基因,包括accD,matK,RPOB.或YCF2.,携带至少一个非同义分离位点(图。2).的代换rpl20单倍型SD2和ZE2中的基因产生了一个提前终止密码子,该密码子将rpl20蛋白缩短了最后三个氨基酸。
两个高度多态基因YCF1.和ndhF-包含了绝大多数的多态性:69个非同义分离位点中的48个。(附加文件1.详细检测这两个基因的Ka/Ks值> 1.0,表明放松选择或积极选择。所有的两两比较都是这样美国寻常的单的YCF1..但是,只有ndhF由山地单倍型VS1和D11组成的比对显示出高Ka/Ks(附加文件)1:数据集4。
质体的转录组美国寻常的热泪盈眶,KOV
我们生成了两个单倍型的质体转录组美国寻常的KRA及KOV [18,19利用之前用于构建线粒体转录组的数据集。我们比较了F和H个体以及两个单倍型之间的花蕾的基因覆盖率和RNA编辑率。
我们比较了蛋白质编码基因的深度覆盖,因为在RNA提取过程中丢失了cDNA文库制备之前的rRNA,并且在包括TRNA的小RNA(<100nt)。在单倍型Kov和KRA中,F和H植物中的覆盖深度类似,在性别之间没有显着表达基因。在KOV和KRA塑性基因组之间不能直接比较覆盖的深度,因为在不同平台上进行illumina测序,并为每个体积转录组产生不同长度的读取。因此,我们比较了两种单倍型之间的高度低盖的基因。基因psbA,:,psbE,RPS14和rps16是最表达的,而是psbN是Kra和Kov植物中最不表达的基因(附加档案1:数据集5),这表明两个质体转录组之间大致相似。的ndhF基因显示比在附加文件中的热图中编码NADH复合蛋白的其他基因的转录物水平降低4:图S3。
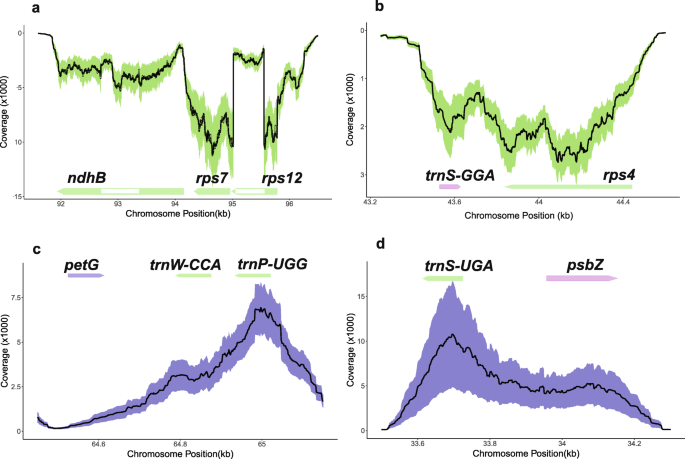
在大多数塑性基因中,内含子覆盖到比相邻外显子的较低程度,但它们的覆盖率达到与某些基因中的外显子相当的水平,例如ndhB(无花果。3.一个).
反义非编码转录物的体积转录组美国寻常的热泪盈眶,KOV
我们鉴定了5个反义非编码转录本> 100 nt,其转录本丰度可与蛋白编码基因相媲美,重叠的环境基因(额外的文件1:数据集6),其中三个是在KRA和Kov转录组中发现的,仅在KOV中揭示了两种。的的环境S-GGA和的环境W-CCA -的环境p-ugg反义转录物对应于3'UTRSrps4基因和佩特格基因,分别。的的环境S-uga反义转录转录与5'UTR的分层psbZ基因(图。3.b, c, d).
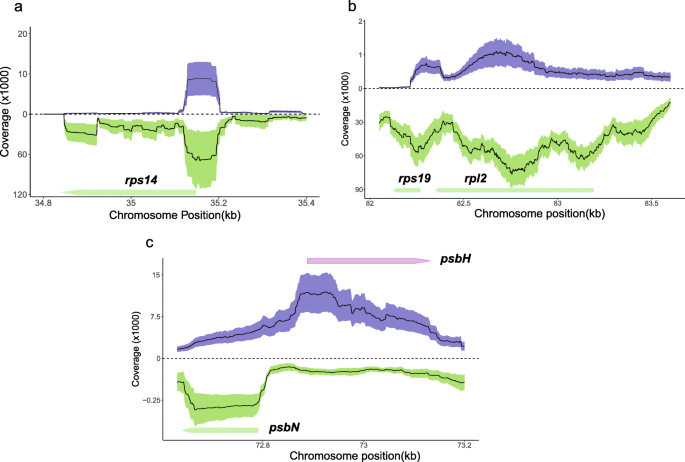
反义转录本横跨5'UTR和RPS14基因(图。4一个),是KOV中源自蛋白编码基因的最丰富的atisense转录本,但在KRA转录组中缺失。的另一个反义转录本RPS19和rpl2.两者都揭示了基因美国寻常的转录组研究(图。4b).的psbN用于编码照相系统的小跨膜蛋白的基因编码[24[较长度的反义方向,从有义义取向和相反的股线中转录psbT-psbH成绩单。的反义psbN文字记录的覆盖深度远高于感觉psbN对PSBN蛋白的转录物编码(图。4c).
与蛋白质编码基因类似,在F和H植物之间没有发现反义转录水平的统计学显着差异。相比之下,kov和kra转录组之间的反义转录物的丰度不同美国寻常的超过转录水平的蛋白质编码基因。最显著的区别在于反义RPS14转录物(图。4a),在KOV中高度覆盖,但在KRA中完全缺失。
氯seq管道估计的KRA和KOV质体转录组覆盖深度[25]的结果与上述结果一致。上述结果是根据[19)(附加文件1:数据集5)。
质体基因组中的RNA编辑位置美国寻常的热泪盈眶,KOV
我们确定了51个独特的C到U编辑网站的塑植基因组美国寻常的其中38个位于蛋白编码区,2个位于内含子区,11个位于基因间区。rrna和tRNAs的编辑位点由于样品制备方法的影响,其覆盖面存在偏差,未进行评估。编码序列的大部分编辑(95%)是非同义的,改变了氨基酸组成;只有两个编辑网站保持沉默。编辑频率最高的基因是ndhB编辑(9),ndhD(4网站)ndhA(4)网站。
我们比较美国寻常的对8种被子植物的编辑位点进行了全面的质体编辑组研究:Amborella trichopoda.,Cucumis巨大成功[26],Spirodela polyrhiza.[27],Aegilops Tauschii.[28],拟南芥[29],橡胶树取代巴西橡胶树[30.],烟草[31],豇豆属辐射动物[32)(表3.).蛋白质编码区域中的大部分38编辑美国寻常的被编辑,或者在比较的大多数血管培养物中替换为DNA水平的T.两个沉默的编辑在高管植物上没有保守。高度编辑的位置rps16内含子也被编辑进去答:tauschii并被DNA中的T取代答:trichopoda和答:芥,这可能表明它在功能上的重要性。在比较下,被子植物的基因间区域不可能可靠地对齐。
RNA的速率编辑在vulartias kra和Kov的塑体转录组中
其中至少有一项的编辑率高于80%美国寻常的转录om在38个编辑中测定的蛋白质编码基因中的26个,所有这些都是非同义词(附加文件1:数据集7)。两个静音位点仅被编辑约50%或更低。编辑事件在大约10%的时间内引入过早的停止密码子ndhJ转录物,但是这种位置未在比较下的其他高管中编辑。在创建起始密码子需要编辑是必要的ndhD基因,其中C存在于编码区的第二位置。然而,所有的KRA和KOV植物在这个位置被编辑< 15%,这意味着只有一小部分ndhD转录本编码了一种功能蛋白。
与蛋白质编码基因相比,在基因间区11个编辑位置中,只有2个编辑量超过80%。其中一个位于3'UTR沥青第2位位于KRA质体基因组的64,933位的环境W-CCA -的环境P-UGG反义转录。
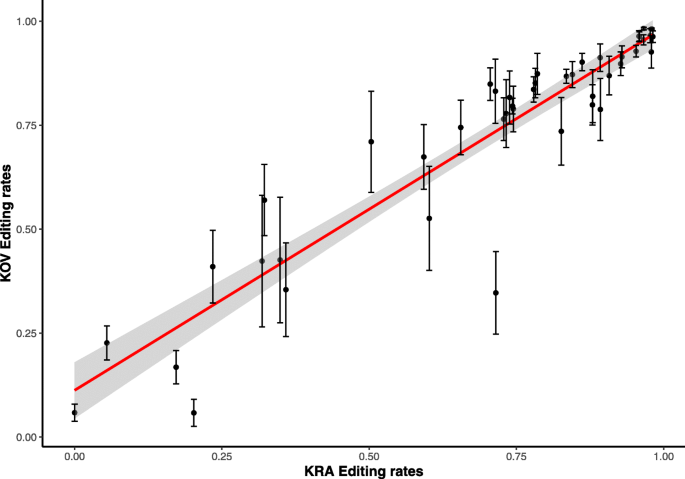
Kra和Kov Plastid转录率的编辑率相互一致,在中间速率40-70%的位置上表现出适度的衍射(图。5).最显著的差异发现在50的位置psbZ编码区,将亮氨酸改为丝氨酸。在KRA单倍型的这个位置没有观察到编辑,而大约6%psbZ六种KOV植物中的每一种都对转录本进行了编辑,这些转录本代表了大约400个reads3.).对这个位置的编辑在被子植物中有所不同。同样的位置被编辑进去了答:芥和S. Polyrhiza.,虽然没有报告编辑答:tauschii,H. Brasiliensis.,或c .巨大成功.T取代了C在质体基因组中的这个位置n .烟草和V. Radiata..要验证在Caryophyllales中的对该位置的编辑,我们下载了四个转录组数据硅宾物种,Agrostemma githago和Spinacia oleracea从SRA存档并映射它们psbZ序列。我们发现高编辑的位置50psbZ在美国conica菠菜没有编辑,美国noctiflora和Silene Paradoxa..该职位在更低的范围内被编辑硅宾latifolia和答:githago(附加文件1:数据集8)psbZ在大多数数据集中是低的,它显示了在美国寻常的,类似于高分类学水平上的分散编辑模式。
在KOV或Kra Haplotypes之间观察到F和H个体之间的编辑率没有统计学显着的差异。GSNAP提供的编辑率的估计[19]和氯seq管道[25一致(补充文件5:图S4)。
讨论
六个质体基因组的变异美国寻常的
6个质体基因组全序列的比较美国寻常的揭示了相同的结构和中度序列差异,这与先前报告的缓慢塑性进化率相同硅宾不同于快速进化的物种美国noctiflora和美国conica[15),或Silene Paradoxa.和Lychnis Chalcedonica[17].的美国寻常的质体基因组更类似于质体基因组美国latifolia在结构和进化速度方面。我们的研究包括两份来自山区种群和一份来自海边沙滩的种群。山地单倍型在系统发育树中首先分化,证实了山地单倍型与低地群体的遗传距离。相比之下,海滩加入ZE2与其余种群聚集在一起,表明它最近发生了分化,可能是在适应盐度增加的生态分化的海岸栖息地的过程中被隔离。
质体进化率的高变异不仅存在于硅宾物种,但也存在于单个质体基因中[17].在种内水平上,质体基因的替代率也存在显著差异美国寻常的.主要是两个基因,YCF1.和ndhF,累积了大量的非同义替换。而这件事YCF1.各基因均表现出较高的替代率硅宾物种和其他被子植物ndhF基因属于进化较慢的基因[17].我们的研究发现ndhF包括至少一个山的单倍型的成对排列美国寻常的.Ka/Ks值接近1表示松弛选择,> 1可能表示积极选择。我们的结果表明,不同的选择作用于ndhF与低地植物相比,山上的基因美国寻常的.以前的栖息地暴露于包括紫外线的强烈灯。山区人口中黄酮类化合物的增加美国寻常的被解释为对过度光照的防御[33].有可能,非同义替换在ndhF也可能与适应高海拔地区更高的光照强度有关。
质体转录组中的RNA编辑美国寻常的
我们发现F和H植物花芽的质体转录组在覆盖率和RNA编辑率方面没有显著差异,在KRA和KOV单倍型中也没有显著差异美国寻常的.这表明质体转录组不受两个具有不同CMS类型的单倍型花粉败育过程的影响[18,19].然而,随着我们分析来自整个花芽的总RNA,不能排除一些特定组织的塑体中的转录,例如Tapetum仍然受CMS的影响。
共发现38个RNA编辑位点美国寻常的转录组。该值可与其他eudicots中报告的值相比较答:芥[29],40个编辑V. Radiata.[32或51编辑c .巨大成功[26].该数量远低于基础高血管植物的体积蛋白编码基因中发现的138个编辑部位答:trichopoda,这与开花植物在进化过程中质体编辑逐渐丢失的一般趋势一致[26].基因组DNA中C替换T导致的编辑缺失同样在类似程度上发生,但它影响了不同谱系中的不同位点(见表)3.).
蛋白质编码基因(36)中绝大多数编辑位点是非同义的,改变编码氨基酸。单一例外(低速率编辑引入过早的停止密码子ndhJ),所有的非同义位点都被保存下来——它们要么被编辑,要么在至少一个物种中被T取代。
大多数,但不是所有的非同义网站被编辑超过80%。一个有趣的例子,一个基本的位置,很大程度上未经编辑的观察ndhD基因编辑仅在15%的转录本中建立起起始密码子,这大大降低了正确mRNA的丰度,并可能降低了功能性NdhD蛋白的产量。同样的位置在非光合器官(根、黄化苗)中低编辑答:芥[34].由于我们的转录组来自幼芽,其中包含光合和非光合组织,低编辑ndhD起始密码子可能反映了一些花组织缺乏编辑。由于其可能对NDH蛋白的丰度和NDH复合物的功能有很强的影响,因此,开始密码子的编辑可能被用来调节NdhD的表达ndhD基因。虽然细胞细胞RNA在植物中编辑的主要功能存在于保守氨基酸的恢复过程中[35],其在前术后基因表达控制中的作用应考虑[36].之前在番茄质体中描述了发育阶段和组织特异性RNA编辑的其他例子[37或马铃薯[38].
两个单倍型美国寻常的不同的编辑程度psbZ基因。将一个单倍型被编辑为低程度,但始终如一的两种性别,另一个根本没有编辑。编辑率50的位置psbZ在比较的被子植物中,可以观察到所有的可能性,即编辑、C替换T和零编辑(表4).没有综合的塑体转录组硅宾已发布,但是从Genbank开采可用的转录组数据显示出类似的变异模式psbZ近亲之间的编辑率美国寻常的在被子植物。psbZ蛋白是光系统II超分子结构的重要组成部分[39,40,其亚基属于最不分化的基因硅宾[17,很可能是由于净化选择的作用。的变化psbZRNA编辑血管植物,血涡轮虫,甚至在单一物种中美国寻常的因此,令人惊讶。位置50的编辑将psbZ第一个跨膜结构域中间的S替换为L [40].该交换可能对蛋白质功能没有明显的影响。RNA编辑中的变异拟南芥影响功能不太重要的地点的物种,最近被描述[41的位置psbZ是统一编辑的拟南芥根据这项研究的物种。作为第50的位置psbZ完全编辑在模型植物中答:芥,识别负责的核因子psbZ编辑将成为可能,这将有助于明确该编辑事件的功能。
反义RNA在塑体转录组中美国寻常的
我们找到了长长的反义文字记录psbN基因,它比该基因的意义转录本更丰富。的psbN基因位于与之互补的股线上psbT-psbH代际区域,这是保守的一部分psbB操纵子。对psbN基因答:芥由质体编码的RNA聚合酶识别的特定启动子和核编码的sigma因子SIG3共同控制[42].的反义psbN发现转录物影响裂解psbT-psbHintercistronic RNA (43并影响翻译psbT信使rna在答:芥[44].因此反义是可能的psbN转录本具有类似的调节功能美国寻常的.相反,我们发现互补链的覆盖率非常低或为零ndhB基因。的反义ndhB在答:芥,烟草和杨树,可能在mRNA稳定性控制中发挥作用[45].其表达受温度和发育阶段的影响。它可能不会在花蕾中表达,或在美国寻常的在所有。
由于大小限制,我们没有估计包括tRNAs在内的小rna的表达,但我们检测到从互补链转录的较长的反义rna的环境基因。的反义trnS-GGA和反义trnW-CCA位于3'Utrrps4和佩特格, 分别。它们形成二级结构,其可以通过调节塑性mRNA的转录的RNA结合蛋白来识别[46,47].同样,反义TRNS.-佐治亚大学可以稳定5 '端psbZ并影响其翻译。
在Plastid转录om中描述了许多反义RNA,例如107个推定的反义转录物答:芥[48,或137个反义候选人萨尔维亚米尔蒂希萨[49].我们只检测到8种推测的长反义rna美国寻常的,这可能是由两个因素引起的。首先,我们通过将覆盖率阈值提高到蛋白质编码基因水平来缩小搜索。其次,我们小心地消除了线粒体基因组中衍生自体层基因组的读数,这可以错误地被错误地识别为塑性编码的转录物。
反义RNA的积累可能受到环境和发育阶段的影响[44,45,这也许可以解释为什么一些反义rna只在一个单倍型中表达美国寻常的.另一方面,所有推定的反义RNA都识别在美国寻常的转录组也在答:芥[48],这表明他们的进化保护和可能的功能性重要性。
结论
我们发现从花蕾制备的塑体转录蛋白中的f和h个体之间没有显着差异(可能预期两性之间的差异)的牙科生物植物的两个单倍型美国寻常的说明CMS与该物种质体基因表达的变化无关。然而,我们不能排除质体转录组的差异存在于花芽的特定组织中。我们观察到两个单倍型之间的差异美国寻常的50位RNA的编辑率psbZ基因,它在一些被子植物中被编辑,包括答:芥,但不在其他人。在单倍型中也检测到反义转录物表达水平的差异。我们的结果记录了内部内部水平的塑体转录om的变异美国寻常的.
构建转录组的质体单倍型KRA和KOV在质体全基因组序列构建的系统发育树中属于一个主簇。美国寻常的在高欧洲山脉收集的人群,向其中测序完全塑性基因组,以增加文学分析,以增加内部采样。他们占据了Phylograph的基础位置,可能更接近祖先美国寻常的.
方法
植物材料
我们收集了种子美国寻常的来自欧洲山区木材线以上的两个种群(Dachstein和Vřesová studánka,分别为D11和VS1单倍型),以及来自荷兰海平面以上沙丘上生长的一个种群(Zandvoort, ZE2)(表2)1).山上的人口美国寻常的表现出与低地植物不同的形态特征(花卉颜色,叶形,脱垂生长)。这些人群有时被视为单独的亚种美国寻常的无性系种群。prostrata或美国寻常的无性系种群。glareosa[50[沿着高度梯度的无限基因流动和薄膜变化沿着高度梯度的无限制流动和抗疱疹性变化[20.,33].因此,我们将山区人口简单地称为美国寻常的在我们的研究中。
如前所述,种子在布拉格实验植物学研究所(IEB)的温室中发芽和栽培[19].美国寻常的采集于达赫斯坦,平卧,开淡紫色花朵。它有时被归类为美国寻常的无性系种群。prostrata[50].然而,有报道称,在高海拔和低海拔植物之间存在中间表型的个体[20.],这表明群体中无限基因流动。粪脂植物对水分非常敏感,在温室里越来越差。因此,它们在21岁的ICB栽培室的受控条件下培养oC,16/8 H光/暗,在含有珍珠岩,蛭石和椰子益官(1:1:1)的盆中,受精(Kristalon-Start和Kristalon-Fruit和Flower,1:1)每周或每一个第二周。植物材料由HelenaŠtorchová确定。各种种子美国寻常的单倍型在布拉格IEB沉积,可根据要求提供。本研究中收集和使用的所有样品都不需要任何特殊许可。目前研究中使用的植物材料符合政府法规。
完全塑性基因组序列来自美国寻常的D11,VS1和ZE2
我们执行了三种塑体单倍型的Novo组装美国寻常的.大约100毫克来自单个H个体的幼芽美国寻常的D11,VS1或ZE2(表2)在液氮中冷冻并用裂解缓冲液(QIAGEN基因组DNA缓冲液组)用中国杵和砂浆进行研磨。使用qiagen基因组尖端(20g)制备高分子基因组DNA的方案按照制造商的说明进行。将约8μg的DNA溶于100μl10μm的Tris-HCl缓冲液(pH = 8.3)中送到Pacific Bioscience rsii P上的SMRT测序的GATC Biotech(Konstanz,Germany)。来自每个的类似等分类的基因组DNA三个进入的美国寻常的被送往Olomouc植物结构与功能基因组学中心IEB进行Illumina MiSeq测序(2 × 300个周期,片段大小约1000 nt),利用Nextera化学制备DNA库。
SMRT测序产生大约39,000个reads, N50 Read长度= 16,500 nt美国寻常的这里;98,000读N50读长度= 16,800 nt for美国寻常的VS1;94,000读取N50读取长度= 18,100nt for美国寻常的ZE2。一种混合校正管线proovread[51]用于用短而准确的MiSeq读取来纠正长但容易出错的SMRT读取。的proovread输出'修剪'组成的错误纠正读取被用作一个本地爆破数据库和读取与之同源的美国寻常的质粒基因组(JF715057)blastn用截止e-20. canu v 1.3搜索[52]申请了大会proovread纠正读取。由此产生的两个COLIGS对应于长单拷贝区域(LSC)加反向重复(IR)和短单拷贝区域(SSC)。在Genbank登录号MK473866-8(ZE2,VS1,D11)下沉积完整的体层基因组序列。
质体基因组完整序列JF715057 [15]衍生自美国寻常的携带线粒体单倍型SD2 [16为新组装的质体基因组的注释提供了参考美国寻常的.
完全塑性基因组序列来自美国寻常的KOV和KRA
从Roche 454 GS-FLX平台获得的数据集与Titanium试剂(来自构建的3 kb对端文库),以前用于组装线粒体基因组美国寻常的haplotypes kov [16]及KRA [18,用于生成两种单倍型的质体序列。虽然DNA标本中线粒体DNA丰富,但含有大量质体reads,质体基因组覆盖面积为10-20 ×。罗氏的GS de novo Assembler v.2.6(“Newbler”)用于初始组装。所得到的contigs被定位到叶绿体基因组中美国寻常的(JF715057)和间隙由个体修剪454填充,读取映射以反对相同的参考。通过重新映射到它们的重新映射来确认KOV和KRA塑性序列。手动检查对齐,分别检查潜在的SNP,诱导和插入并进行编辑。通常观察到均聚物> 5的个体变化,这可能反映出可能的异质性,或者在同一个体中的两个或更多个变体序列的共存。得到的KOV和KRA完整的体积序列(GenBank登录号MH890612和MH890613)用作以下转录组分析的参考基因组。
叶绿体基因组的功能
利用snp-dists (v. 0.6,github.com/tseemann/snp-dists),而对于indels的两两比较,R内ape包体的dist.dna功能采用“indel”和“indelblock”模型。利用微卫星识别软件MISA-web估算SSR位点[53单核苷酸为5个核苷酸,二聚体和三核苷酸为4个重复单元,四、五、六核苷酸为3个重复单元。基因和编码区根据质体基因组进行了注释美国寻常的(JF715057),并经包装参考[54,证实了起始密码子和终止密码子中存在编辑位点。
系统发育分析
首先,六个质体基因组美国寻常的在一起硅宾latifolia作为外组(JF715055)与MAFFT v.7.388对齐[55使用L-INS-i模式,手动编辑错位位点。反向重复区域A (IRA)区域被切出核苷酸序列。在系统发育树计算方面,比较了两种不同的比对方法;首先,当非编码序列(CDS)中超过5个核苷酸时,同源聚合物区域等非信息位点被掩盖;第二组只有CDS(不含tRNAs和rnas)。此外,indel字符使用“简单indel编码”算法进行编码,如[56]用2matrix进行两种对齐[57].系统发育树使用RAxML进行计算[58]及贝叶斯先生[59]在CIPRES入门网站[60].
采用最大似然(ML)方法,使用CIPRES门户网站RAxML v. 8.2.10和1000个bootstrap和GTRGAMMA模型进行bootstrap和树推理。索引在分区文件中以二进制字符的形式给出,描述了索引的大小和分布情况。此外,通过MrBayes v. 3.2.6使用贝叶斯方法构建系统发育树[59]使用Markov Chain Monte Carlo算法和默认型号4by4在两个运行中为5000代,树木采样每1000代。不同的分区Nexus文件由序列对齐和indel编码组成。固定的字符频率为Indel数据集和Dirichlet(1.0,1.0,1.0,1.0)和前25%的拓扑丢弃(烧焦)。当运行之间的分流频率的标准偏差低于0.01时,分析被停止。
内部的代名词和非唯一替换的数量美国寻常的用DNASP V5测定体积蛋白编码基因[61].如果在6个对齐的质体序列中至少有一个在各自的位置上发现了可选择的核苷酸,则确定了分离位点美国寻常的.
Illumina公司读映射
我们使用存储在Short Read Archive登录号PRJNA321915下的读取。通过Illumina对3个F和3个H个体花蕾总RNA的cDNA序列进行测序得到美国寻常的热泪盈眶(18](GenBank登录号码SRX3102769 - SRX3102774)和三个F和三个人的花蕾美国寻常的kov [19(GenBank登录号为SRX272140 - SRX272145)。
使用GSNAP v. 2017-05-03 [62在已知剪接位点的对端模式下[14].单倍体型KOV和KRA的质体基因组序列美国寻常的用作参考。将参考体层基因组内的IRA切除以确保使用GSNAP进行适当的读取映射。通过SAMTOOLS V.1.17中的视图函数通过链分离所得对准。1.9 [63,根据读取对方向利用SAM标志进行过滤,如[18对这些比对进行筛选,以获取插入到相应单倍型线粒体基因组的质体DNA (GenBank登录号为JQ771300和MH455602)的潜在线粒体阅读4).在这些已知的区域,所有不匹配的参考序列和没有呈现潜在的RNA编辑被过滤,部署SAMtools视图功能,一个定制的AWK脚本和seqtk v. 1.2 (https://github.com/lh3/seqtk)作后续分析。通过整合基因组查看器(IGV)将映射的reads可视化[64].
为了比较,我们还使用了氯化物管道[25的质体转录组分析美国寻常的KOV,热泪盈眶。这个管道依赖于几个不同的、开源的生物信息学程序,如SAMtools和BEDtools v. 2.25.0来正常运行。bowtie v. 2.2.6使用了与GSNAP最终对齐相同的参考、已知的拼接站点和过滤过的读取[65]和tophat v. 2.1.1 . [66]用于读取映射,如[25].
RNA编辑利率
在GenomeAnalysisTK v. 3.7中使用HaplotypeCaller进行初始变异发现[67关于减去和搁浅的对齐美国寻常的设置最小调用和发出阈值(stand_call_conf)为20。所有带有C-to-T(正链)和G-to-A(负链)变化的变异位点都经过SAMtools v. 1.2 mpileup的手动检查和验证,以便在后续的最终调用中使用每个观察等位基因的高质量碱基数量的DPR输出标签(从v. 1.3起停止)。在低读映射但RNA编辑明确的情况下,DP4值被用来评估所有个体的编辑率。根据这些值计算RNA编辑率,即t的计数除以特定编辑位点的Cs和Ts之和。最终的编辑站点作为列表,用氯喹计算编辑率。编辑率低于5%的阈值(或10个被编辑的核苷酸,无论哪个更小),或低覆盖率(不足200个reads mapping),最终结果中不会显示出来。
转录丰度估计
根据[中的描述是对转录物丰度的估计19使用bedtools中的覆盖率函数和自定义AWK脚本。覆盖率按基数计算,在各自感兴趣的特征长度上平均,首先不考虑链,然后对每个样本进行链特定计算,然后归一化为TPM,如[68].计算单倍型和性别TPM的平均值和标准差。反义转录本覆盖深度超过300-500时被识别,对应于表达最少的蛋白编码基因的TPM值。网络工具Morpheus (https://software.broadinstitute.org/morpheus.)用于热图构建。
可用性数据和材料
本研究数据已保存在NCBI中,生物工程登录号为PRJNA321915。rna序列从雌雄同体中读取美国寻常的KRA存储在SRS2438489号码下,在SRS2438490下的女性中的读数。RNA-SEQ从六个人中读取美国寻常的KRA在GenBank登录号SRX272140 - SRX272145下存放。完整的质体基因组可以在GenBank登录号MH890612和MH890613 (KOV和KRA)和MK473866-8 (ZE2, VS1,D11)下找到。
缩写
- accD:
-
乙酰辅酶a羧化酶D亚基
- 英国石油公司:
-
碱基对
- 互补脱氧核糖核酸:
-
互补脱氧核糖核酸
- CMS:
-
细胞质雄性不育
- F:
-
女性
- H:
-
雌雄同体
- kb:
-
千碱基
- LSC:
-
长期单一副本
- matK:
-
成熟酶K
- ndhA:
-
NADH脱氢酶一
- ndhB:
-
NADH脱氢酶B
- ndhD:
-
NADH脱氢酶D
- ndhF:
-
NADH脱氢酶F
- psbA:
-
光系统II蛋白A
- psbE:
-
光系统II蛋白E
- psbH:
-
光系统II蛋白
- psbN:
-
光系统II蛋白
- psbT:
-
光系统II蛋白T
- psbZ:
-
光系统II蛋白Z
- ::
-
Rubisco亚基L.
- RPOB.:
-
RNA聚合酶B.
- RPS14:
-
核糖体蛋白S14系列
- rps16:
-
核糖体蛋白S16
- rrna:
-
核糖体核糖核酸
- SSC:
-
短的单一副本
- SSR:
-
简单序列重复
- TRNA:
-
转移核糖核酸
- UTR:
-
翻译区
参考文献
- 1.
植物性系统的相对频率和绝对频率:雌雄异株、雌雄同株和雌雄异株。中国科学(d辑:地球科学)2014;https://doi.org/10.3732/ajb.1400196.
- 2.
线粒体和核基因的相互作用对雄性配子体发育的影响。植物细胞。2004;16:s154 - 69。https://doi.org/10.1105/tpc.015966.
- 3.
Kazama T. Nakamura,Watanabe,M. Sugita Kt。BT型细胞质雄性无菌水稻RF1蛋白的线粒体蛋白累积抑制机制。工厂J. 2008; 55:619-28。https://doi.org/10.1111/j.1365-313x.2008.03529.x.
- 4.
王克,高f,ji y,liu y,dan z,杨p等。ORFH79通过与电子传输链复合体III的亚基相互作用损害线粒体函数,在红莲细胞质雄性无菌大米中。新植物。2013; 198:408-18。https://doi.org/10.1111/nph.12180.
- 5.
Sabar M,Gagliardi D,Balk J,Leaver C. OrfB是F1FO-ATP合成酶的亚基:以向日葵的细胞质雄性不育的基础上的洞察力。Embo Rep。2003; 4:381-6。https://doi.org/10.1038/sj.embor.embor800.
- 6.
Allen Jo,Fauron Cm,Minx P,Roark L,Oddiraju S,Guan Nl等。玉米两种肥沃和三种雄性无菌线粒体基因组的比较。遗传学。2007; 177:1173-92。https://doi.org/10.1534/genetics.107.073312.
- 7.
Darracq A, Varré JS, Maréchal-Drouard L, Courseaux A, Castric V, Saumitou-Laprade P,等。甜菜线粒体基因组的结构和内容多样性:比较基因组分析。基因组生物学进展。2011;3:723-36。https://doi.org/10.1093/gbe/evr042.
- 8.
Charlesworth D,Laporte V.男性无菌多态性硅宾寻常的:遗传资料分析:来自两个种群并与之比较胸腺寻常的.遗传学。1998;150:1267 - 82。
- 9.
Desfeux C,Maurice S,Henry JP,Lejeune B,突云博士。属的生殖系统硅宾.属生殖系统的进化硅宾.中国生物医学工程学报。1996;263:409-14。
- 10.
Casimiro-Soriguer I,Buide Ml,Narbona E.在属的不同谱系中的性系统多样性硅宾.AOB植物。2015;7:plv037。doi:https://doi.org/10.1093/aobpla/plv037
- 11.
德莱勒T,德尔照片。古代线粒体单倍型和颅内重组在牙科植物中的证据。Proc Natl Acad Sci U S A. 2002; 99:11730-5。https://doi.org/10.1073/pnas.182267799.
- 12.
Touzet P, Delph LF。育种制度对silene线粒体基因多态性的影响。遗传学。2009;181:631-44。https://doi.org/10.1534/genetics.108.092411.
- 13.
Mower JP, Touzet P, Gummow JS, Delph LF, Palmer JD。种子植物线粒体基因中同义替换率的广泛变异。BMC Evol Biol. 2007;7:7。https://doi.org/10.1186/1471-2148-7-135.
- 14.
史隆·德,Alverson AJ, Chuckalovcak JP, Wu M, McCauley DE, Palmer JD, et al.;开花植物线粒体中具有异常高突变率的巨大多染色体基因组的快速进化。公共科学图书馆。2012;10:e1001241。doi:0.1371 / journal.pbio.1001241.
- 15.
Sloan DB,Alverson AJ,Wu M,Palmer JD,Taylor Dr。最近的塑性序列和结构演变的加速度与Ageniosperm属的极端线粒体分歧均匀硅宾.基因组生物学进展。2012;4:294-306。https://doi.org/10.1093/gbe/evs006.
- 16.
陈志强,陈志强,陈志强,等。线粒体基因组序列、结构和基因含量的种内变异硅宾寻常的,一种具有普适细胞质雄性不育的Angiosperm。新植物。2012; 196:1228-39。https://doi.org/10.1111/j.1469-8137.2012.04340.x.
- 17.
Sloan DB,Triant Da,Forrester NJ,Bergner LM,Wu M,Taylor Dr。高血管植物部落硅藻(蛛网苜蓿)加速塑性基因组进化的重复综合征。mol phylocyet evol。2014; 72:82-9。https://doi.org/10.1016/j.ympev.2013.12.004.
- 18.
Štorchováh,石头jd,sloan db,abeyawardana oaj,muller k,walterováj,pažoutovám。同源重组在线粒体基因组中改变细胞色素b转录的背景硅宾寻常的热泪盈眶。BMC基因组学。2018;19:874。https://doi.org/10.1186/s12864-018-5254-0.
- 19.
Stone JD, Koloušková P, Sloan DB, Štorchová h硅宾寻常的.J Exp Bot。2017; 68:1599-612。https://doi.org/10.1093/jxb/erx057.
- 20.
自然植物-病原体系统的海拔病害分布:来自宿主种群和气候变化的见解。Oikos。2014;123:1126-36。https://doi.org/10.1111/oik.01001.
- 21.
李志峰,张永昌,陈永强。mirna和lncrna在生殖发育中的作用。植物科学。2015;238:46-52。https://doi.org/10.1016/j.plantsci.2015.05.017.
- 22.
吴军,张敏,张波,张旭,郭磊,齐涛,等。陆地棉CMS-D2及其保持系和恢复系的全基因组比较转录组分析。BMC基因组学。2017;18:454。https://doi.org/10.1186/s12864-017-3841-0.
- 23。
Hamid R,Tomar Rs,Marashi H,Malekzadeh S,Golakiya Ba,Mohsenpour M.转录组剖析和编目舒适的棉花花芽中的花芽中的差异差异基因表达(陆地棉l .)。基因。2018;660:80 - 91。https://doi.org/10.1016/j.gene.2018.03.070.
- 24。
Plöchinger M, Schwenkert S, von Sydow L, Schroder WP, Meurer J. PSII维护和装配中辅助TerC和ALB3的功能更新。植物科学学报,2016;https://doi.org/10.3389/fpls.2016.00423.
- 25。
Castandet B,Hotto Am,Strickler SR,Stern DB。氯化氯,优化的叶绿体RNA-SEQ生物信息管道,揭示了在热应激下的细胞内转录组的重塑。G3-基因基因组群。2016; 6:2817-27。https://doi.org/10.1534/g3.116.030783.
- 26。
王志强,王志强,王志强,等。开花早枝植物叶绿体RNA的频繁编辑:对编辑位点及其核特异性因子共存的初步研究。BMC Evol Biol. 2016;16:23。https://doi.org/10.1186/s12862-016-0589-0.
- 27.
王W,张W,吴y,maliga p,乱七八型杂志编辑Spirodela polyrhiza.,水生单子叶鱼类。《公共科学图书馆•综合》。2015;10:e0140285。https://doi.org/10.1371/journal.pone.0140285.
- 28.
王敏,刘辉,葛立国,王敏,魏宁,等。叶绿体转录本RNA编辑位点的鉴定与分析Aegilops Tauschii.l基因。2017;8:13。https://doi.org/10.3390/genes8010013.
- 29.
李志伟,李志伟,李志伟。拟南芥叶绿体定量编辑型。2月。2013;587:1429-33。https://doi.org/10.1016/j.febslet.2013.03.022.
- 30.
唐法特sornruang S, uthaaipaisanwong P, Sangsrakru D, Chanprasert J, Yoocha T, Jomchai N, et al.;全叶绿体基因组的特征橡胶树取代巴西橡胶树揭示基因组重排、RNA编辑位点和系统发育关系。基因。2011;475:104-12。https://doi.org/10.1016/j.gene.2011.01.002.
- 31.
Hirose T, Kusumegi T, Tsudzuki T, Sugiura M.烟草叶绿体转录本的RNA编辑位点:作为叶绿体RNA聚合酶活性可能调节因子的编辑。1999; 262:462-7。
- 32.
Lin C, Ko C, Kuo C, Liu M, Schafleitner R. Transcriptional滑脱and RNA editing increase Transcriptional Transcriptional diversity in chloroplasts: deep sequencing . acta optica sinica(生物谷bioon.com豇豆属辐射动物基因组和转录组。Plos一个。2015; 10:E0129396。https://doi.org/10.1371/journal.pone.0129396.
- 33.
Berardi Ae,Fields Pd,Abbate JL,Taylor Dr。花卉颜色和叶子化学的高度分歧和抗液体变异硅宾寻常的.我是J机器人。2016; 103:1508-23。https://doi.org/10.3732/AJB.1600106.
- 34.
Tseng CC,Lee CJ,Chung Yt,Sung Ty,Hsieh MH。非光合组织中拟南芥塑性基因表达和RNA编辑的差异调节。植物mol biol。2013; 82:375-92。https://doi.org/10.1007/s11103-013-0069-5.
- 35.
MAIER RM,Reckermann K,Igloi GL,Kossel H.玉米叶绿体基因组的完整序列:基因含量,发散的热点和遗传信息的微调编辑。J Mol Biol。1995年; 251:614-28。
- 36.
斯隆DB。核和线粒体RNA编辑系统对蛋白质多样性有相反的影响。医学杂志。2017;13:20170314。https://doi.org/10.1098/rsbl.2017.0314.
- 37.
番茄果实发育和叶绿体到染色体分化的质体转录组学和翻译组学:染色体基因表达主要服务于单一蛋白质的生产。植物细胞。2008;20:856 - 74。https://doi.org/10.1105/tpc.107.055202.
- 38.
Valkov VT, Scotti N, Kahlau S, Maclean D, Grillo S, Gray JC,等。马铃薯叶片叶绿体和块茎淀粉体质体基因表达的全基因组分析:转录和转录后控制。植物杂志。2009;150:2030-44。https://doi.org/10.1104/pp.109.140483.
- 39.
Swiatek M,Kuras R,Sokolenko A,Higgs D,Olive J,Cinque G,等。叶绿体基因YCF9.编码光系统II (PSII)核心亚基,PsbZ,参与PSII超分子结构。植物细胞。2001;13:1347 - 67。
- 40。
Wei X,Su X,Cao P,Liu X,Chang W,Li M,等。3.2Å分辨率菠菜光照系统II - LHCII超复杂的结构。自然。2016; 534:69-74。https://doi.org/10.1038/nature18020.
- 41。
川边A,富烈hata HY, Tsujino Y,川边T, Fujii S,吉田T拟南芥物种。植物科学。2019;280:241-7。https://doi.org/10.1016/j.plantsci.2018.12.009.
- 42.
Zghidi W, Merendino L, Cottet A, Mache R, lebs -Mache S.核编码质体sigma因子SIG3特异性转录psbN塑性中的基因。核酸RES。2007; 35:455-64。https://doi.org/10.1093/nar/gkl1067.
- 43.
Chevalier F,Ghulam MM,Rondet D,Pfannschmidt T,Merendino L,Lerbs-Mache S.表征psbH前体rna显示一个精确的核糖核酸内切酶裂解位点PSBT / PSBH.基因间区域依赖于psbN基因的表达。植物生态学报。2015;88:357-67。https://doi.org/10.1007/S11103-015-0325-Y..
- 44.
黄志强,李志强,李志强,等psbT意义和反义rna。核酸学报2011;39:5379-87。https://doi.org/10.1093/nar/gkr143.
- 45.
王志强,王志强。植物叶绿体中长反义RNA的快速报道。新植醇。2010;4:615-22。https://doi.org/10.1111/j.1469-8137.2010.03203.x.
- 46.
Bollenbach TJ,Sharwood Re,Gutierrez R,Lerbs-Mache S,Stern DB。RNA结合蛋白CSP41A和CSP41B可以调节拟南芥中叶绿体编码RNA的转录和翻译。植物mol biol。2009; 69:541-52。https://doi.org/10.1007/S11103-008-9436-Z..
- 47。
王志强,王志强,王志强,等。维管植物叶绿体中rna稳定因子的研究进展。学生物化学论文。2018;62:51 - 64。https://doi.org/10.1042/ebc2170061.
- 48。
Hotto Am,Schmitz RJ,Fei Z,Ocker Jr,Stern DB。叶绿体不应过度的叶绿体的意外变化,如深度测序所揭示的拟南芥转录组。基因组学杂志。2011;1:559-70。https://doi.org/10.1534/g3.111.000752.
- 49。
Chen H,张J,元G,刘C. DNA修饰中的复杂相互作用,非分量RNA表达和蛋白质编码RNA表达萨尔维亚米尔蒂希萨叶绿体基因组。Plos一个。2014; 9:E99314。https://doi.org/10.1371/journal.pone.0099314.
- 50.
Marsden-Jones Em,Turrill WB。膀胱罐。伦敦:光线社会;1957年。
- 51.
Hackl T,Hedrich R,Schultz J,Forster F.序列分析Provread:通过迭代简短读取共识,大规模高精度PacBio校正。生物信息学。2014; 30:3004-11。https://doi.org/10.1093/bioinformatics/btu392.
- 52.
karenn S, Walenz BP, Berlin K, Miller JR, Bergman NH, phillip AM。Canu:通过自适应k -mer加权和重复分离,可扩展和精确的长读组装。基因组研究》2017;27:722-36。https://doi.org/10.1101/gr.215087.116.
- 53.
基于序列分析的微卫星预测网络服务器。Bioiformatics。2017;33:2583 - 2585。doi: 0.1093 /生物信息学/ btx198。
- 54.
Robison Ta,狼PG。参考:用于在塑性基因组中注释RNA编辑的R包。苹果厂SCI。2019; 7:E1216。https://doi.org/10.1002/aps3.1216.
- 55.
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性文章快速轨道的改进。mol Biol Evol。2013; 30:772-80。https://doi.org/10.1093/molbev/mst010.
- 56.
系统生物学家协会在基于序列的系统发育分析中以空白为特征。系统医学杂志。2000;49:369 - 81。
- 57.
DR Salinas, Little DP。2MATRIX:一个用于indel编码和系统发生矩阵连接的工具。应用植物学报。2014;2:1300083。https://doi.org/10.3732/apps.1300083.
- 58.
Stamatakis a . RAxML版本8:系统发育分析和大系统发育后期分析的工具。生物信息学。2014;30:1312-3。https://doi.org/10.1093/bioinformatics/btu033.
- 59.
Ronquist F, Teslenko M, van der Mark P, Ayres DL, Darling A, Hohna S, et al.;MrBayes 3.2:跨大模型空间的高效贝叶斯系统发育推理和模型选择。系统医学杂志。2012;61:539-42。https://doi.org/10.1093/sysbio/sys029.
- 60。
关键词:进化树,CIPRES,进化树abstract:网关计算环境研讨会(GCE)论文集,2010年11月14日,新奥尔良。洛杉矶:IEEE;p 1 - 8。https://doi.org/10.1109/gce.201010.5676129.
- 61。
Librado P, Rozas J. DnaSP v5: DNA多态性数据综合分析软件。生物信息学。2009;25:1451-2。https://doi.org/10.1093/bioinformatics/btp187.
- 62。
吴丹丹,吴志强,吴志强。短读序列中复杂变异和剪接的快速和耐单核苷酸多态性检测。生物信息学。2010;26:873 - 81。https://doi.org/10.1093/bioinformatics/btq057.
- 63。
李洪波,李洪波,李洪波,等。序列对齐/映射格式和SAMtools。生物信息学。2009;25:2078-9。https://doi.org/10.1093/bioinformatics/btp352.
- 64。
Thorvaldsdottir P.综合基因组观察者(IGV):高性能基因组学数据可视化和探索。简短生物形式。2012; 14:178-92。https://doi.org/10.1093/bib/bbs017.
- 65。
Langmead B,Salzberg SL。与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9:357-9。https://doi.org/10.1038/nmeth.1923.
- 66.
Trapnell C, Pachter L, Salzberg SL. TopHat:用RNA-Seq发现剪接连接。生物信息学。2009;25:1105-11。https://doi.org/10.1093/bioinformatics/btp120.
- 67.
等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组研究》2010;20:1297 - 303。https://doi.org/10.1101/gr.107524.110.
- 68.
Wagner GP, Kin K, Lynch VJ。使用RNA-seq数据测量mRNA丰度:RPKM测量在样本之间是不一致的。理论Biosci。2012;131:281-5。https://doi.org/10.1007/s12064-012-0162-3.
确认
作者感谢Dan B. Sloan和James D. Stone,用于有价值的评论和语言修正。我们感谢奥洛穆茨的MiroslavValárik进行Miseq测序。获得缔约方和项目所拥有的计算和储存设施,该项目涉及国家电网基础设施介入,该项目根据“研究,开发和创新的大型基础设施”(LM2010005)(LM2010005)提供了“大型基础设施项目”(LM2010005)。
资金
该项目由捷克共和国拨款机构16-09220S拨款HŠ资助。欧洲区域发展基金项目“实验植物生物学中心”提供了额外的支持。CZ.02.1.01/0.0/0.0/16_019/0000738)。资助者只提供资金支持,他们在研究的设计、数据的分析和解释、决定发表或手稿的准备中没有作用。
作者信息
从属关系
贡献
MK进行了大部分的数据分析和解释,MJ参与了数据分析,OAJA和CK进行了湿实验室工作,HŠ构思和设计了实验并提供解释,HŠ和MK撰写了手稿。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
相应作者[Helena Storchova]是BMC植物生物学编辑委员会的成员,提交人声明他们没有其他竞争利益。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
额外的文件1。
数值表。数据集S1.质体基因组中的简单序列重复美国寻常的.数据集S2。质体基因的置换率美国寻常的.S3数据集。KAKS矩阵。数据集S4。质体基因的基因覆盖率美国寻常的.数据集S5。塑性基因组中的反义转录覆盖美国寻常的.数据集S6。质体基因组的编辑率美国寻常的.数据集S7。psbZ编辑范围
附加文件2:图S1。
不同塑体单倍型的最大似然性系统发育树硅宾寻常的。一个。根据质体基因组的所有位点(大于五个核苷酸的均聚物位点除外);b。只有质体编码区。硅宾latifolia被用作外群体。长枝缩短50%,有两条斜线。Indels用Simmon & Ochoterena(2000)编码。比例尺表示每个站点的替换数量。自举支持度低于60%的分支被折叠成多分支。通过用RaxML v的CIPRES WebPortal计算系统发育树。8.2.10使用1000个引导和Gtrgamma模型
附加文件3:图S2。
不同质体单倍型的贝叶斯50%多数规则系统发育树硅宾寻常的基于一个。质体基因组的所有位点,但大于五个核苷酸的均聚物区域除外;b。只有质体编码区;C。质体基因组的所有位点,除了Simmon & Ochoterena(2000)后大于5个核苷酸、具有简单indel编码的均聚物区域;天。质体编码区只有简单的indel编码。硅宾latifolia被用作外群体。长枝缩短50%,有两条斜线。比例尺表示每个站点的替换数量。系统发生树通过CIPRES网站门户与MrBayes v. 3.2.6计算,使用5000代
附加文件4:图S3
热图显示了质体的转录水平ndh六个个体的基因美国寻常的热泪盈眶,KOV。的ndhF基因是两种单倍型中最不表达的基因,其他基因的表达在两种单倍型之间变化
附加文件5:图S4。
GSNAP和ChloroSeq管道估计的编辑速率的比较。从6个个体中计算的平均值(±SD)被绘制出来,观察数据的95%置信区间在图中以灰色表示
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Krüger, M., Abeyawardana, o.a.j., Juříček, M.。等等。雌雄异株物种质体基因组的变异硅宾寻常的.BMC植物杂志19,568(2019)。https://doi.org/10.1186/s12870-019-2193-0
收到了:
接受:
发表: