抽象的
背景
克罗顿德拉科是一个树栖物种及其乳胶以及植物的其他一些部分,传统上用于治疗各种疾病和疾病。生物碱,例如致马诺氟,防止早期动脉粥样硬化进展,同时表现为乳胶丰富的乳胶成分,已被描述为伤口治疗师和抗肿瘤剂。尽管对这些和其他次级代谢产物具有很大的兴趣,但对于物种而言,这些生物碱的生物合成途径仍然很大程度上是未知的。
结果
了解木兰花碱和塔斯品碱生物合成的途径c·德拉科并鉴定这些方法中的关键酶,我们在该物种的主要器官(根,茎,叶子,花序和花序)中对转录组和代谢物进行了综合分析。通过高通量RNA测序分析产生转录物谱,同时还进行了靶向和高分辨率未确定的代谢物分析。这些化合物的生物合成似乎发生在所检查的植物器官中,但中间体可以从它们产生它们积累的其他细胞的细胞中旋转中介机构。
结论
我们的结果为更好地理解木兰花碱和塔斯品碱的生物合成提供了一个框架c·德拉科.此外,我们展示了多个OMIC方法的潜力来识别涉及感兴趣的生物合成途径的候选基因。
背景
克罗顿德拉科var。德拉科是属于大戟科家族的树栖物种,广泛分布在美洲。它通常被称为'Sangregado','Palo Sangrikeo'(出血树),'Llora sangre'(血液哭泣)或'Sangre de Dragon'(Dragon的血液),因为这些树木产生的强烈乳胶乳胶[1].对于其他产乳植物,乳汁管细胞已被描述巴豆物种 [1].这些生活专门的细胞以管状系统的形式合成和储存乳胶,独立于木质和韧皮肌[2].乳汁管细胞最引人注目的方面,是那些与它们的生理学和它们在乳胶生产中的作用有关的方面,特别是在橡胶树(橡胶树取代巴西橡胶树)、罂粟(果实),并在某些物种巴豆属[1,3.,4,5,6].含羞草参与植物防御食草动物和一些次生代谢物的运输[7,8,9],虽然它们的确切的个体起源,它们的分化机制和乳管系统的组织往往存在于植物的几个器官,但仍不确定。
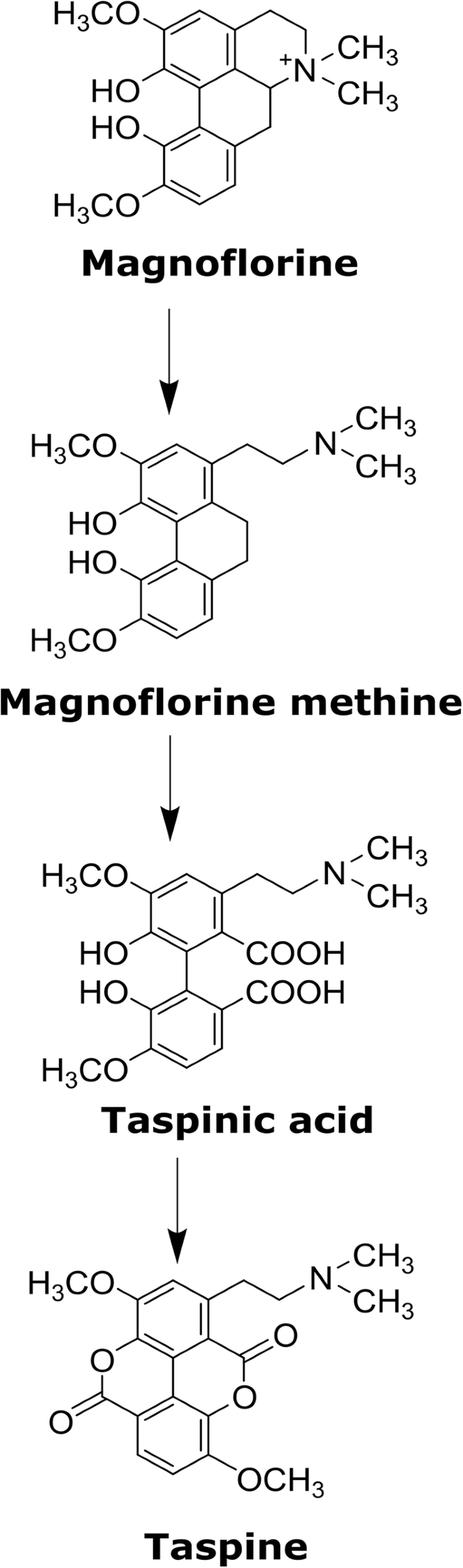
c·德拉科在传统上被用于治疗广泛的疾病和疾病。这种植物的药用特性可以归因于其所有器官中存在大量的次级代谢物,包括生物碱、单宁、二萜和精油[10.,11.,12.].乳胶在传统医药中应用广泛,其化学成分已被鉴定为几种胶乳巴豆.最丰富的次级代谢物(高达90%的干重)是原花质粘蛋白和几种多酚化合物,如(+) - 儿茶素,( - ) - EpicaTechin,GallocateChin,EpigallocateChin和Dimeric Procyanidins [13.].其他次要成分也被识别,包括Korberins [14.如Bincatriol,Crolechinol和Croolechinic acid [15.]其中还[16.,17.].在c . lechleri是该属的最多研究的种类之一,生物碱Taspine也被鉴定为乳胶的丰富成分,其干重约9%[18.].Taspine是苄基异喹啉型生物碱,被描述为伤口治疗师[19.]及抗肿瘤药物[20.].后者属性已经通过乳胶和咔哒声来实验证明了c . lechleri[21.],也有报道称,其他的塔斯品衍生物对几种癌细胞谱系也有类似的效果[22.,23.].虽然Taspine的生物合成途径仍然未知,但其一些植物物种的合成涉及催化Hofmann消除(+) - 氧化氯(季腹啡醛)的酶反应,导致摩尔多甲烷的形成和C9之间的双键的氧化氧化和C10及其沉思化最终产生Taspine [24.].在近缘种中已检测到木兰碱的存在C. Cumingii.[25.],C. Xalapensis.[26.] 和c . lechleri[18.].
尽管有药理潜力巴豆目前尚无这些物种的基因组数据,塔斯品碱和其他次级代谢物生物合成的完整途径尚不清楚。因此,我们对从已建立的不同器官收集的RNA库进行了测序和从头组装c·德拉科通过芽接繁殖的树木。此外,我们还为公共数据库提供注释,并根据其功能和代谢途径对转录本进行分类。最后,在此基础上,分别通过液相色谱-高分辨质谱(LC-HRMS)和液相色谱-串联质谱(LC-MS-MS)进行非靶向和靶向代谢组学分析;提出了塔斯品的生物合成途径,并首次确定了一些编码酶的unigenes。
结果
unigenes的构建克罗顿德拉科
总共产生87,884,170个原始Illumina配对读数来自所有器官样本(叶,茎,根,水果,花序和花)。每个样品7.78至1940万次序列,每个样品平均为1460万。由于质量不足(有关详细信息,请参阅方法,丢弃了大约16.79%的原始Illumina成对末端读数。为了提高计算时间和质量,在装配之前,通过将配对的读取通过重叠区域加入相继的读取(附加文件)来生成更长的读取1:表S1)。两个数据集(长读和未合并的对端读)被用来组装转录组c·德拉科这包括总共441,543个unigenes,范围为200至17,193bp,平均长度为691.32 bp(附加文件2和附加文件3.).
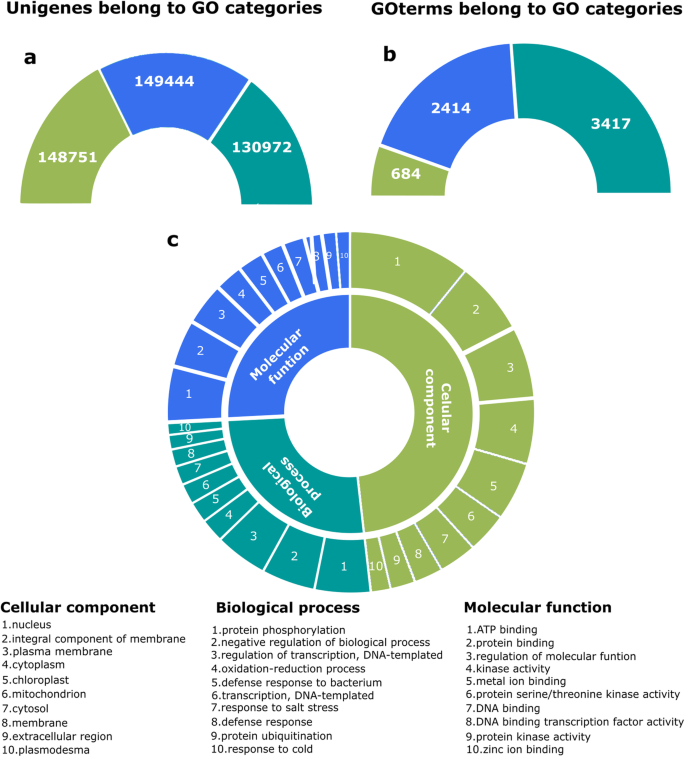
作为c·德拉科通过基于聚(a)选择的RNA-SEQ方法产生转录体,鉴定了通过错误插入/缺失产生的框架(266,967 unigenes,60.46%的60.46%总共使用基于同源性的方法使用AleringWise流水线[27.].剩余序列可能表示未转换的区域(UTRS)或非编码RNA(NCRNA)。然而,这些也可以表示具有几种常见装配人工的序列,包括嵌合器,抗褶皱中的杂散插入和结构异常,如不完全,碎裂或局部杂散的Contigs。由于发现与先前描述的蛋白质相似的可能性取决于查询序列长度[28.[只有至少75个氨基酸的非冗余编码序列是注释并使用的未来分析(共174,925人;附加文件2和3.;额外的文件4:图S1和附加文件5:表S2)。
的同源搜索和功能注释c·德拉科unigenes
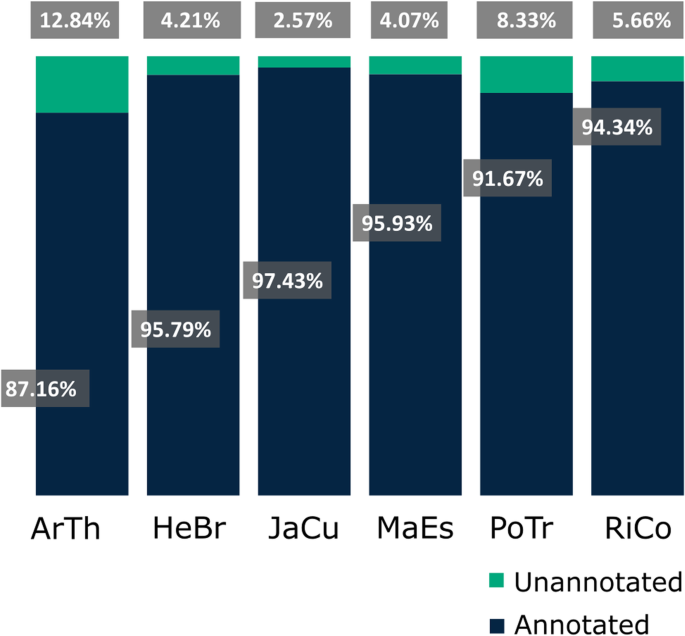
我们确定了c·德拉科可以使用所选蛋白质om(来自Malpighiales命令的五个高昂的Agenperps和来自Malpighiales命令的五个高昂的Angiospers来注释蛋白质拟南芥蒂利亚纳).大多数相应的蛋白质(98.20%)显示出高相似性(E值≤10−5)与至少来自一个参考物种的蛋白质(附加文件5:表S2)。从…c·德拉科非冗余unigenes(167071), 97.43%基于麻风树图同源蛋白。也相对于含有同源蛋白木薯耐(95.93%),橡胶树取代巴西橡胶树(95.79%),里纳斯市政府(94.34%),杨树trichocarpa(91.67%)和拟南芥蒂利亚纳(87.16%)(图。1).计数唯一的登录号(或位点标识符)对应于鉴定为同源的参考蛋白c·德拉科Unigenes,我们估计独特基因的数量c·德拉科在转录组中表示的基因组。根据物种的不同,这些数字在16942到24544个蛋白质之间变化,这代表了在基因中发现和表达的最小数量的最严格的估计c·德拉科organs采样(附加文件5:表S2)。我们进一步比较c·德拉科对PFAM域数据库的unigenes [29.].总共116,981c·德拉科unigenes包含至少一个Pfam结构域,6391个不同的Pfam结构域由unigenes表示(附加文件5:表S2)。
基因本体(GO)功能表征[30.]基于相应同源物中的信息拟南芥蛋白质组。大约6515个独特的GO术语被分配给了总共149444个c·德拉科unigenes,包括3417从“生物过程”类别,2414来自“分子函数”,684来自“细胞成分”(图。2A和B)。平均而言,超过八个GO-术语分配给每个c·德拉科unigenes(附加文件6:表S3)。在生物过程、分子功能和细胞组分这三个类别中,GO最突出的术语分别是蛋白磷酸化、ATP结合和细胞核(图1)。2c)。
最后,分配了c·德拉科通过BLAST比对KEGG数据库,采用双向最佳hit方法,获得了1958个独特的KEGG orthology (KO)标识符和1023个独特的酶委员会(EC)编号,共计38514个c·德拉科unigenes(附加文件6:表S3)。通过指定的KO标识的位置来评估组装的转录组的代表性和完整性c·德拉科将基因整合成代谢途径的全球地图。将unigenes集合中所代表的代谢途径与基因组中所存在的代谢途径进行比较拟南芥蒂利亚纳和离散差异被确定(附加文件5:图S2)。我们量化了组装转录组的完整性,其基于鉴定预定义的1375个胚胎单拷贝直脑道鉴定估计为81.3%。这是使用Busco v3.0.1(基准通用单拷贝正轨)管道完成的[31].这些数据表明转录组包含蛋白质编码基因的广泛覆盖c·德拉科基因组。
多个基因表达图集c·德拉科器官
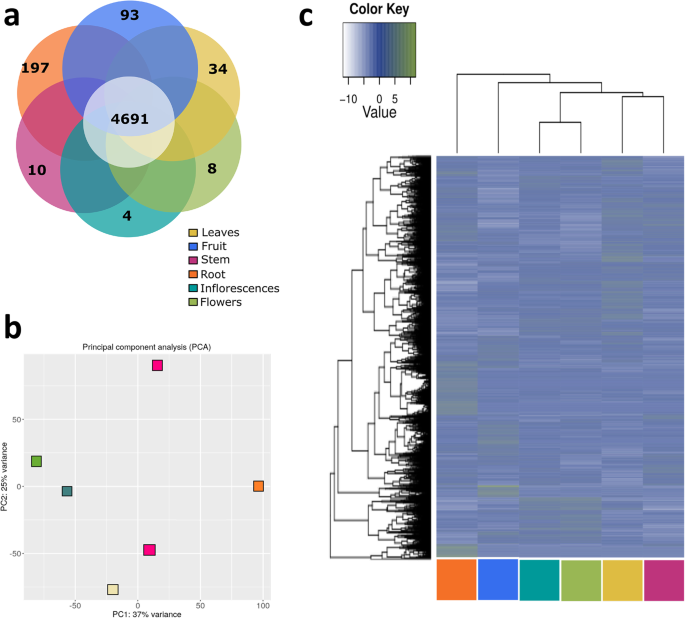
该图谱包含了各主要植物器官中ungenes的表达谱c·德拉科.此数据集很重要,因为它可用于建立每个器官的标识(附加文件7:表S4)。优先表达基因被定义为器官特异性转录水平至少是其他器官转录水平的两倍(logFC≥1)的ungene,并且在FDR调整后这种差异具有统计学意义P≤0.001。
总共10556人c·德拉科unigenes在至少一个器官中优先表达(附加文件)7:表S5)。其中一些可能被视为器官特定的基因,因为映射到这些unigenes的所有读取源自单个库(附加文件7:表S5)。其余基因,尽管它们的优先表达,也可能被认为是普遍存在的,因为它们的转录水平高于背景值的三倍以上(FPKM值≥3),在所有被分析的器官(图。3.a).尽管多数优先表达c·德拉科unigenes(约占总数的93%)被归为无所不在,它们的表达谱可以通过主成分分析(PCA)和层次聚类(HC)进行分类(图2)。3.B和C分别)。
对于采样的器官类型,HC和PCA都反映了器官之间的已知异质功能的相似性。例如,来自茎和叶片的转录物作为相邻组聚集,反映其生理相似性(营养器官)。同样地,生殖器官转录om(花序和花序)被分组在一起,而根和水果的基因产物保持为独立人物,并与其余部分分开。
对于每个被分析的器官,我们调查了那些富集了大量优先表达基因的类别。有趣的是,这些GO类别与每个器官所扮演的特定功能角色相关(附加文件)4:图S3)。等主要器官的茎、根、叶、浓缩主要类别有关植物营养(如离子的运输、肽和激素运输),而在花序和花,过多类别主要是与生殖器官的发展(例如,花、雄蕊、心皮螺纹和花粉)。
对某些特定基因的调查产生了有趣的结果。例如,一些c·德拉科unigenes (UniGene120823和UniGene211389),与拟南芥与光收获络合物II相关的蛋白质(分别为1G29930和AT3G61470),优先于叶片表达。这些蛋白质涉及在照相I亚单元f(AT1G31330)和亚基L(AT4G12800)之间的激发能量分布的调节中,(分别为Unigene104983和Unigene91612)[32]和光系统II (PS II的一个外部亚基,AT5G66570| UniGene92418) [33].在成员中发现了类似的表达谱拟南芥RBCS Multigene系列(AT1G67090 | Unigene120289),其为光合作用产生Rubisco(核糖糖-1,5-双磷酸羧酸盐酶/氧气)[34].上述所有蛋白质通过特异性表达的基因进行编码拟南芥叶子(35].的c·德拉科unigene UniGene70470编码的同源物拟南芥与干燥相关的蛋白质(AT1G47980)以花朵表示。在拟南芥,的基因AT1G47980在开花,花瓣分化和膨胀阶段,在花,花瓣,种子,萼片和雄蕊中表达。此外,据报道米娅(男性配子生成的花药受损)插入突变体,其产生未从四边形解离四肢的脆弱花粉颗粒,显着增加了表达AT1G47980基因(36].同样,在水果中,c·德拉科编码十字花植物的同源物(AT5G44120)的基因(UNIGENE37221)以器官特异性方式表达。这是可预测的,因为十字花素基因家族以协调的方式表达,但在种子填充和发展期间具有组织特异性差异[37,38,39].此外,在名单c·德拉科单独表达在根中的unigenes,对以前报道的几种转录因子有一个群体同源拟南芥,并且是特定的根源(AT1G21860 | Unigene25896,Unigene17133,AT1G74150 | Unigene54724,AT2G27880 | Unigene6800,AT3G06460 | Unigene78483,AT3G45300 | Unigene68073和AT3G47570 | Unigene14449)[40].
为了验证归一化读计数(FPKM)获得的表达谱,对上述10个unigenes进行了RT-qPCR分析。所有评估的unigenes的RT-qPCR表达谱与通过read计数分析计算的表达谱完全一致(附加文件)7:图S4)。
鉴定植物物种相比的矫形组
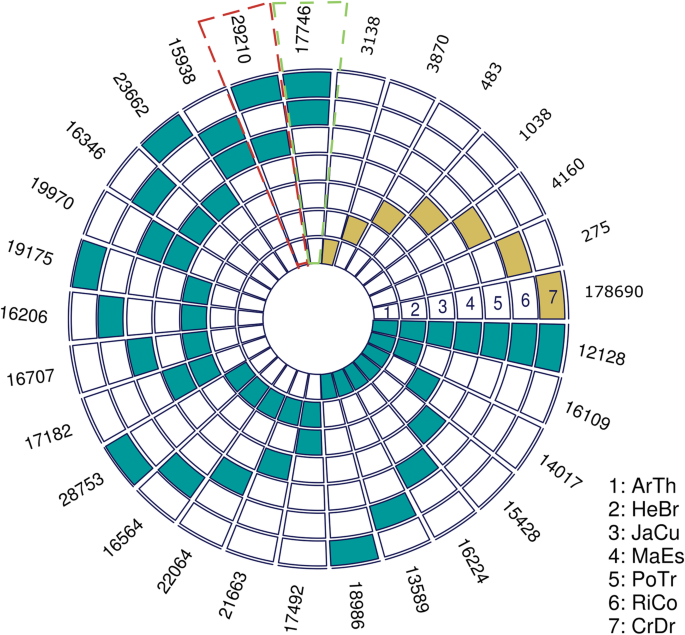
使用ORHMCL算法[41我们在本研究中使用的所有参考物种中鉴定了同源基因。总共281,493个蛋白质(127,919个来自c·德拉科;22463年从答:芥;33,563来自h .取代巴西橡胶树;19865年从j . curcas;25,361来自m .耐;34145年从P. Trichocarpa.;18177年从r .普通的)被分配到41,392个Orthogroups(附加文件8:表S7)。其中,7种参考种共有9717个正群;1326个正群含有来自Malpighiales目植物物种的蛋白质,但不在拟南芥和525个Orthogroups包含一对一单拷贝基因(图。4和附加文件7:表S6)。
在比较植物物种之间鉴定的数量共同的正交和独特基因。每个同心圆对应于一个物种[(1)arth,拟南芥蒂利亚纳;(2)希伯来圣经,橡胶树取代巴西橡胶树;(3)jacr,麻风树图;(4)MAES,木薯耐;(5)Potr,杨树trichocarpa;(6) RiCo,里纳斯市政府;(7) CrDr,克罗顿德拉科].蓝色的块表示成对的交集(两个物种之间的同源基因),黄色的块表示每个物种的独特基因(物种特有的基因)。共享的同心圆的数目显示在最后一个同心圆的旁边。c·德拉科分享最大数量的orthologsP. Trichocarpa.(用深蓝色虚线突出显示的盒子)和最少的r .普通的(盒子突出显示粉红色虚线)。要生成此数字并可视化多种字段,我们使用了R包超克Xacttest [42]
镁生物合成中候选基因的鉴定及表达分析
阿波啡生物碱木兰花碱可能是塔斯品的生物合成前体[24.].为了鉴定木兰花碱生物合成的主要酶,我们进行了广泛的文献综述。目前对木兰花碱生物合成的认识主要来自于一项研究Coptis japonica.[43],其中C-C酚偶联(年代)的细胞色素P450 (CYP80G2)的网状素似乎负责产生(年代),随后可n -甲基化产生木兰花碱。罂粟(果实)(()的N-甲基化(年代- - 甘氨酸产生季铵碱细胞碱,其也可以用作通过替代路线形成摩托氯的CYP80G2样酶的基材[44](附加文件7:图S5)。木兰花碱生物合成中涉及的C-C苯酚偶联反应类似于吗啡生物合成中所需的(R)-网氨酸转化为salutaridine [45,46].吗啡和相关生物碱的生物合成p .果实相关物种通过以L-酪氨酸开始的多步途径发生[45],其在几个步骤中转化为 - 乙烯。(S) - 然后将其转化为其立体异构体(R) - reteRINELEN,其中许多已知的许多苄基异喹啉途径中的独特案例[47].在随后的步骤(R)中 - 将其转换为吗啡(附加文件7:图S5)。涉及从酪氨酸到吗啡的主要生物合成步骤的大多数酶[48]和/或木兰[44的功能特征p .果实有些已经在其他植物中被发现,包括Coptis japonica.[43,49],Eschscholzia加利福尼亚州[50],Podophyllumspp。51] 和丘脑蓝[52].
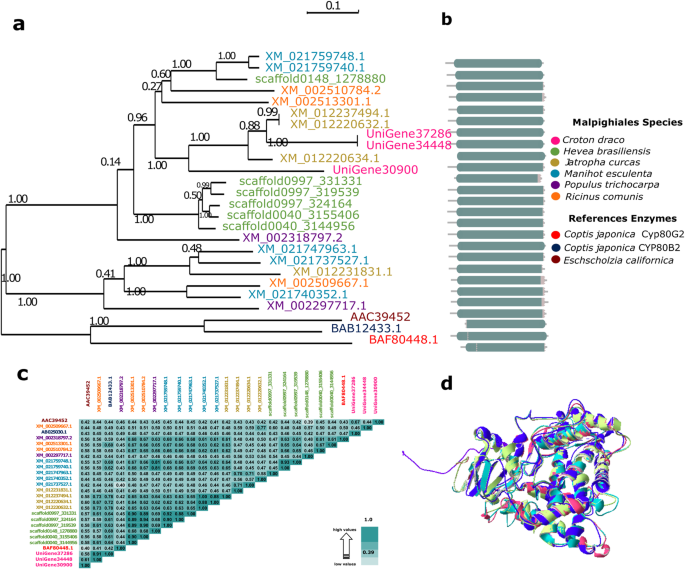
基于已发布的信息,使用OrthoMCL软件结合系统发育分析,我们鉴定了c·德拉科可能参与木兰生物合成的候选基因(附加文件)7:图S5及表格1).含一些参照酶[酪氨酸脱羧酶(TYDC), (S)-去甲氧氯酸合酶(NCS), (S)-去甲氧氯酸6- o -甲基转移酶(6OMT), (S)-氯氯酸n -甲基转移酶(CNMT), (S)-n -甲基氯氯酸3 ' -羟化酶(NMCH;从OrthoMCL输出中提取CYP82B亚家族)、(S)3 ' -羟基n -甲基钴酸盐4 ' - o -甲基转移酶(4 ' omt)、(S)-corytuberine合成酶(CTS, CYP80G2)和网状n -甲基转移酶(RNMT)](补充文件)7:表S6)为了鉴定本研究中使用的一些参考物质中存在的末端的序列,包括c·德拉科unigenes。然后,我们进行所有鉴定的蛋白的对准以产生相应的系统发育树并确认存在从每种类型的酶具有独特的稳定域的存在。包含在含有一些参考蛋白质的替代蛋白中包含的蛋白质的对准是手动烹调的(我们仅考虑那些代表完全蛋白的长度的至少70%的蛋白质)。数字5显示了最大似然(ML)系统发生树,一个图形表示,其中对齐的直系同源蛋白的保守域表示,恒等矩阵和最后(仅用于本例)的结构模型叠加c·德拉科蛋白质及其矫形器用作(S)-Corytuberine合酶酶(CTS)的参考。
(S)-corytuberine synthase (CTS;CYP80G2,参比蛋白,登录号:AB288053.1)。一个描绘蛋白质与比较的不同植物物种分组的蛋白质之间关系的最大似然性系统发育树。b细胞色素p450 (CYP450)保守结构域(PF00067)中的一级蛋白结构用青灰色框表示。c对准CTS Orthologs之间的百分比标识百分比。dCTS蛋白的结构模型叠加,包括来自的CTS蛋白c·德拉科物种[UNIGENE30900(绿色),UNIGENE34448(蓝色)和UNIGENE37286(粉红色)]和CTS参考蛋白来自Coptis japonica.(紫色的)。有关更多详细信息,请参阅方法
根据身份矩阵(附加文件8表S7-S13),各组同源酶的相似性在23 - 70%之间。在所有参考种中均鉴定出同源物种的情况下,系统发生树上的分支与种树上的分支是一致的。这是意料之中的,因为根据系统发育定义,如果两个同源基因从一个共同的物种形成事件中分离出来,它们就是同源基因。因此,一组同源基因(任意一对同源基因的一组基因)的系统发育树必然与相应的物种树具有相同的拓扑结构。所有经上述分析鉴定的unigenes都被认为是主要候选基因c·德拉科与木兰花碱生物合成途径相关的基因,(图。6),Taspine的原发性前体。
木兰花碱生物合成途径示意图及相关基因表达谱c·德拉科编码所涉及的酶的物种。在该图中,表示每个提出的中间体的分子结构,然后是酶(红色显示的缩略乳)及其相应的正交结果c·德拉科转录组。表示分析的每个器官中的转录物(FPKM)的丰度的表达曲线也被显示为每个c·德拉科未鉴定为参考酶的直脑的unigenes。首字母缩略词:TYDC(酪氨酸脱羧酶);NCS((s) - 诺尔科霉素合成酶);600((s)-norcoclaurine 6-O-甲基转移酶);CNMT((s)-coclaurine n-甲基转移酶);NMCH((S)-N-甲基氯鱼3'-羟化酶,CYP82B亚家族);4'Omt((s)3'-羟基N-甲基钴磺胺4'-O-甲基转移酶);CTS((s)-corytuberine合成酶,CYP80G2)
木兰花碱的生物合成始于多巴胺和对羟基苯乙醛的缩合反应,这两种物质都来自于l -酪氨酸。norcoclauth合成酶催化这个反应,但有趣的是,属于两个不同蛋白家族的两种酶能够催化这个反应,这两种蛋白都在我们的参考蛋白列表中。这些酶中的一种丘脑蓝[55]是由防御反应引起的,属于PR10/Betv1家族蛋白。另一种酶,在Coptis japonica.,是依赖2-氧戊二酸的双加氧酶家族的成员[54].只有一个异常(含N-甲基转移酶,RNMT)从含氧化物合成中涉及的所有参考酶的原序表中鉴定在c . drado转录组。此外,酶-Norcoclaurine 6-O-甲基转移酶和(S)3'-羟基N-甲基CORAURINE 4'-O-甲基转移酶(分别为60甲基转移酶(60MT和4'OMT),以独特的c·德拉科Unigene(附加文件7:表S6)。
缺乏与RNMT酶的同源物强烈提示(s)-网氨酸合成木兰花碱的途径是通过(s) -corytuberine作为中间体c·德拉科(图。6,请参阅附加文件7:图S5)。在这一途径中,(s)-reticuline是C8-C2 '底物偶联,生成(s) -corytuberine,这一步是由(s) -corytuberine合成酶(CTS, CYP80G2)的活性催化的,随后是木兰花碱合成所需的n -甲基化。这一步骤可由(S)-环氯嘌呤n -甲基转移酶(CNMT)催化c .粳稻种能接受(S)-corytuberine作为次要底物[49].此外,中心异喹啉生物碱中间体(S)网状物的生物合成需要6OMT和4'OMT酶[56,57,58.].即使这些酶在高度的序列同一性中,几种植物种类中也存在两种酶的存在表明它们可能具有严格的底物特异性,并且可以仅能够耐受体内假定底物的轻微变化。然而,有证据表明,一些O-甲基转移酶在它们可以甲酸盐的基材的范围内令人惊讶地允许[59.,60.].因此,有可能c·德拉科一种独特的o -甲基转移酶可以将不同的底物甲基化,例如在其他物种中被6OMT和4'OMT酶识别的底物(图)。6,请参阅附加文件7:图S5)。
除了基于RNA-seq数据进行鉴定外,我们还分析了所有可能参与木兰生物合成途径的候选基因的表达模式(图)。6).这一分析表明,这些基因是普遍存在的,正如它们的转录本在所有植物器官评估的丰度所示。而且,即使它们在不同器官中的表达水平不同,也没有一个基因的表达有差异或以器官特异性的方式表达。这一结果是可以预测的,因为除了从树皮中提取的乳胶样本外,木兰花碱存在于每个取样器官中c·德拉科.
木兰花碱可能是塔斯品碱的生物合成前体
根据Shamma和Moniot提出的生物合成途径[24.[需要几种酶促反应达到摩尔诺罗兰转化为Taspine(图。7).然而,涉及这些酶的生物分析还没有进行。同样,目前还没有能够催化这些反应的酶的功能特征,因此通过寻找同源基因来鉴定参与这一途径的候选基因并不是一种可行的策略。根据LC-HRMS结果,并将其光谱指纹图谱与METLIN-Scripps数据库中报道的相比较,初步鉴定了塔斯品生物合成途径中的前体中间体以及木兰花碱生物合成的中间体。考虑误差小于3.0 ppm(附加文件4:图S6和附加文件9:表S14系列)。HRMS代谢组学分析经常用于识别生物合成途径中涉及的前体[61.,62.以及整合代谢组学和下一代测序数据来阐明植物的途径,为新的重要发现做出了贡献[63.].
从镁氟到Taspine的生物合成途径c·德拉科.该途径中的主要中间体是根据先前提出的途径显示的[24.]
(s) - 是摩洛汀的 - 核苷酸核苷酸的核核,以茎,叶子和花朵的浓度增加。有趣的是,在根中未检测到 - reticuline。相比之下,致氧化氯在根和鲜花中最丰富而不是茎和叶子(附加文件4:图S6和附加文件9:表S14系列)。有趣的是,在分析的任何器官中都没有检测到tembetarine,它是一种中间体,可能是某些植物的前体,而木兰碱就是在其中合成的。在取样的器官中,通过LC-HRMS鉴定出的(S)-网状网氨酸和(S)-corytuberine的缺失与RNMT酶的同源物以及(S)-网状网氨酸和(S)-corytuberine的缺失相一致,为木兰花碱生物合成途径的提出提供了额外的证据c·德拉科.关于Taspine及其中间体,氧化氯甲基和Taskinic acid,对每个器官中的每种代谢物观察到类似的型材(附加文件4:图S6和附加文件9:表S14系列)。在分析的器官中,木兰花碱和塔斯品碱始终是检测到的最丰富的代谢物。基于这些结果,我们的数据为木兰花碱和塔斯品碱的生物合成途径提供了有力的证据c·德拉科物种。
Taspine和Magnoforline的定量和身份证实
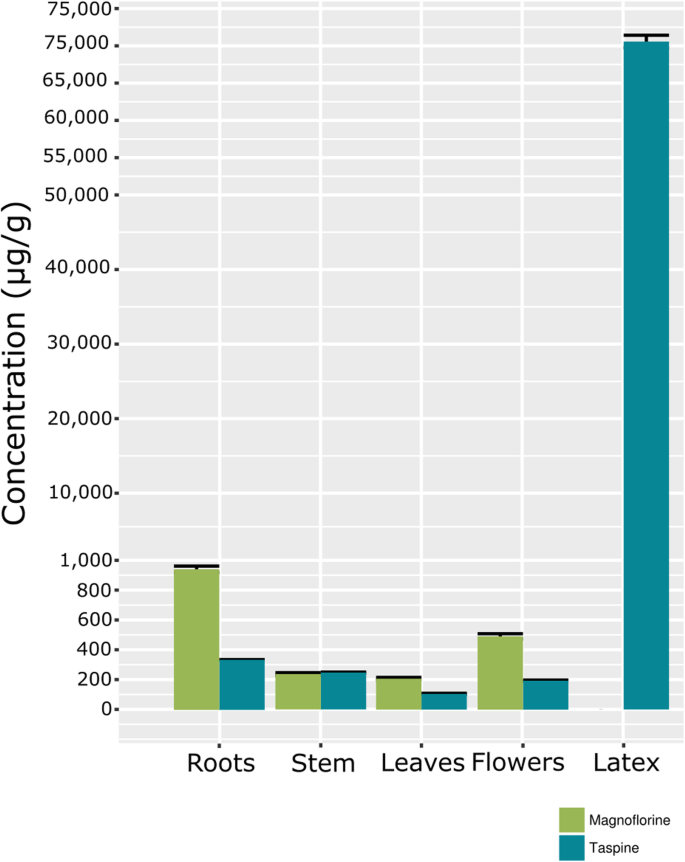
采用液相色谱-质谱联用技术定量测定了不同器官和乳胶中木兰花碱和塔斯品的代谢物含量。木兰花碱和塔斯品碱的分布模式与LC-HRMS分析结果一致(花和根中含量略高于茎和叶;无花果。8).Taspine,它存在于其他一些物种中巴豆属[18.],也被发现在乳胶中的丰富c·德拉科(约为干重的7%)。与取样的其他器官相比,树干树皮的乳胶中有大量的塔斯匹碱。塔斯品只在乳管细胞中积累,这种代谢物占乳胶总干重的相当一部分。有趣的是,没有检测到木兰花碱(图。8).这与先前的报告一致,可在某些组织中检测到镁氯,但不在乳胶中c . lechleri[18.].
在器官中丰富的含含乳丝细胞克罗顿德拉科
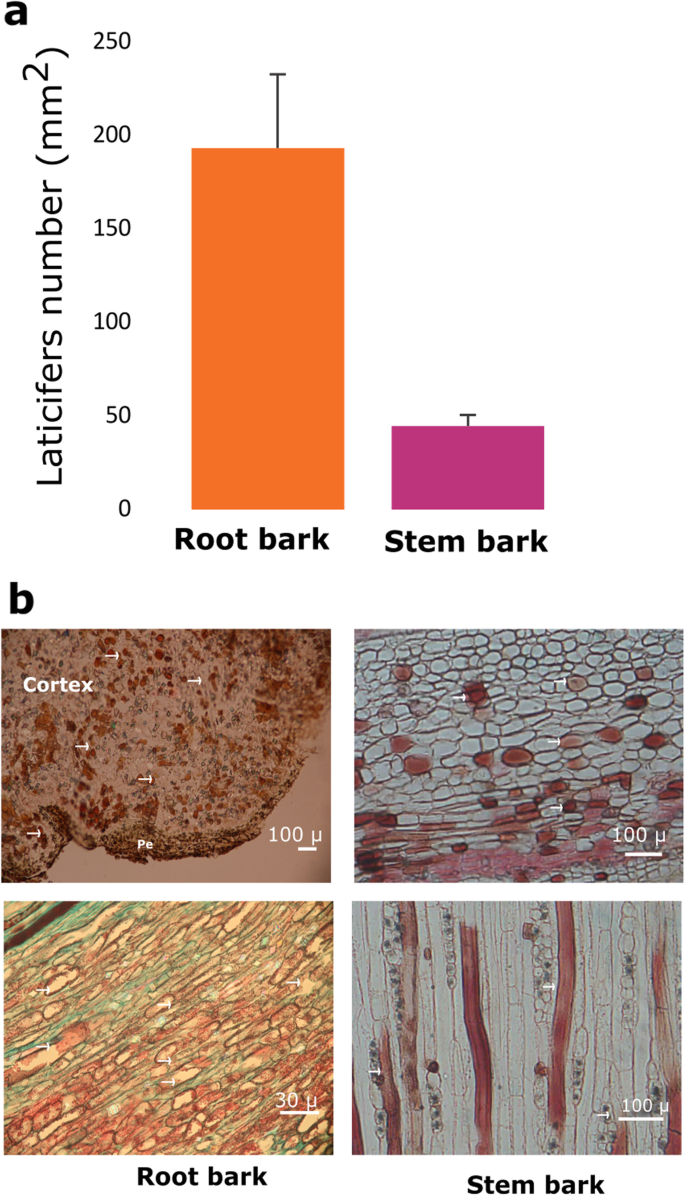
确定是否有丰富c·德拉科含型细胞与诸如Taspine等次级代谢物的浓度有关,我们量化了茎和根的皮质区域中的皮质细胞密度。我们观察到,Taspine在根中比在茎中更丰富,并且是具有最高密度的含型细胞的根的皮质区域(图。9).
讨论
在乳胶植物中,含型细胞主要存在于叶片和/或茎中,尽管在其他植物物种中,含型细胞可以在其他器官和组织中发现它[64.].在巴豆乳汁管细胞主要位于树皮韧皮部,其丰度取决于植物年龄、树的位置和环境[4,65.].含羞草比年轻茎更旧的含量较少[66.];树枝的乳汁管密度比树干高,热带雨林树木的乳汁管比半落叶热带森林树木的乳汁管多[1].如在其他物种中,特定代谢物的积累与乳管细胞的丰度有关[67.], 在c·德拉科塔斯品的积累似乎与乳管细胞的丰度有关。在这项研究中,我们证明了木兰花florine,塔斯品生物合成的前体,存在于从几个器官获得的甲醇馏分中。然而,这种生物碱并不存在于从树皮中提取的乳胶中。这些结果表明Podophyllum物种,一些参与木兰生物合成的基因可以在特定的细胞类型中转录[51].很可能巴豆在一种特定的细胞中合成木兰花碱后,这种化合物必须转移到乳汁管细胞中,以转化和储存为塔斯品。我们假设木兰花碱不能在乳胶中检测到,因为它是一种短暂的代谢物和一种高度反应的中间体,能迅速产生塔斯品及其前体。
除了在植物的四个不同器官中提供镁氯和干峰存在的证据,该研究使用转录组数据和植物化学分析来提出这两种代谢物的生物合成途径。主要发现,例如没有rnmt酶,表明在c·德拉科CNMT可能是一种双功能酶,能够识别两种不同的底物:(S)-氯代酸和(S)-corytuberine。同时,考虑到一个单一的c·德拉科Unigene是60Mt和4'Omt酶的正交结果,似乎氧化镁合成中的额外步骤需要识别两种不同底物的另一种O-甲基转移酶。
不出所料,转录图谱c·德拉科unigenes揭示了植物器官的形态和功能角色不同。我们观察到每个人的表达方式c·德拉科被视为参与将酪氨酸转化为氧化镁的不同酶促步骤的素候选的未成年人未以器官特异性方式表达,其中一些在某些器官比其他器官更丰富。
这种明显的表达动力可能与某些代谢物或中间体从产生它们的器官转位到它们优先积累的另一个器官有关。一些负责生物碱或其他次级代谢物转运的转运体已被描述[68.,69.,70]并显示在实验研究中的功能[70,71.].例如,表现出强生物活性的生物碱作为植物防御机制对抗病原体和草食虫,有时需要通过蛋白酶从它们合成到积累位点(例如,在不同器官中细胞的泡沫的泡沫中的组织中展开。这种活性运输可以让植物应对这些化合物的毒性。这种现象的规范实例是尼古丁,主要的生物碱烟草通过从根组织中通过木瓜旋转的物种,在叶片的液泡中的积累位点上旋转72.].
考虑到magnnoflorine和taspine都属于aporphine生物碱,它们在不同的癌细胞系中的毒性已经被广泛研究[21.,73.],它们的生物合成可能涉及一种复杂的方法,该过程需要不同的器官,不同的细胞类型和必须易于转移的一些中间体的主动传输。
由于单个细胞类型可能含有数千种代谢物,其中许多代谢物的结构、功能和药理潜力都不为人所知,因此药用植物不仅是植物化学家面临的巨大挑战,而且是生物活性化合物、酶、转运体、或者可以用于合成生物学项目、医学和兽医应用的监管机构。幸运的是,下一代序列技术和精确的质谱技术,以及核磁共振波谱技术,可以用来对“非模型”植物转录组进行测序,并鉴定参与这些化合物生物合成的酶。将这些研究与基于质谱的代谢组学相结合,将使我们能够识别新的化合物并阐明它们的生物合成途径。在目前的研究中,我们采用了一种综合的方法,结合多组学工具,产生了关于木兰花碱和塔斯品碱合成的酶的第一个证据c·德拉科.
结论
鉴定与吗啡生物碱合成相关的基因,如木兰花碱和塔斯品碱,将有助于今后植物的功能研究,并为通过代谢工程提高植物甚至微生物宿主的生产水平提供依据。在我们的数据集中,我们不仅识别了这些具有抗癌和植物防御特性的代谢物的代谢途径中涉及的酶的编码转录本,而且我们还通过识别一些中间产物展示了证据,这一途径在植物中占有一席之地c·德拉科生产这些重要的化合物。此外,我们注释了各种代谢途径涉及的许多基因。组装的数据集c·德拉科本研究中提出的未成年人是可用于物种的第一组OMIC数据,并为其他功能和比较基因组研究提供基础。
方法
生物材料
本研究中使用的所有样本都是从生长良好的三岁树木中采集的。这些树被认为是无性系(至少在地上部分),因为它们是由单个个体的分枝嫁接到由种子生长的三个月大的砧木上繁殖的。的c·德拉科种植园位于墨西哥韦拉克鲁斯州特佐纳帕市国家林业、农业和牲畜研究所(INIFAP)的“El Palmar”实验中心。有代表性的标本保存在XAL植物标本室(INECOL a.c., Xalapa,墨西哥),登录号为FRF-316。本研究中所采集的样本是来自同一棵树的叶、茎和根。果实、花序和花,后两个器官分别有花茎和没有花茎。所有样品立即用液氮冷冻,带回实验室,-80℃保存至使用。
虽然在RNA-seq实验中推荐生物复制是正确的,但当样本来自于尽管属于同一群体,但基因可能不同的个体时,这一点尤其重要。然而,本研究中使用的样本来自相同条件下生长的克隆植物;因此,在使用克隆的情况下,预计不会有相当大的遗传变异。我们取样的主要目的是获得来自不同器官的转录图谱,其中包含尽可能多的基因。此外,一些最近发表的评估不同治疗或情况的研究,没有显示独立测序的生物复制;尽管如此,他们已经显示出了自信和结论性的结果[74.].由于上述原因,我们从五棵不同的树木中采集不同器官的样本,然后将它们汇集在一起,并在RNA分离和文库制备时将其视为独特的样本。
RNA分离,文库制备,测序
将收获的样本粉碎,并从200 mg匀浆组织中分离总RNA。根据先前描述的方法从含有大量多糖和多酚化合物的植物中分离出RNA [75.].使用生物分析仪系统(安捷伦技术公司)的芯片毛细管凝胶电泳评估RNA的完整性。用紫外分光光度计(BioSpectrometer Eppendorf)在260 nm处测定RNA浓度。以500ng的RNA为输入材料制备cDNA文库。使用标准TruSeq RNA样本制备试剂盒(Illumina)制备每个植物器官的一个文库,并使用Illumina NextSeq500平台测序,获得150 bp的配对端reads。包含序列读取和质量分数的文件存放在国家生物技术信息中心(NCBI)的短读档案(SRA)中。加入SRP156345数量。
测序序列和从头转录组组装的质量控制
在装配之前,使用来自下一代测序(NGS)工具包的QualityControl.py脚本进行低质量配对终端读取(https://github.com/czh3/ngstools/blob/master/quitalcontrol.py.).用于选择高质量(HQ)对端读取的严格参数为:-q 30(要保持的最低质量分数),-p 90(必须具有[-q]质量的最低碱基百分比)和-a 30(所选对端读取的最低平均质量)。使用SeqPrep程序合并重叠的测序序列(https://github.com/jstjohn/seqprep.),除去条形码和污染适配器序列,避免孤儿读取的产生。通过将读取(R1和R2)合并通过其重叠区域,进行转录组组合物,包括可自由的HQ成对终端读取和更扩展的序列,从而通过其重叠区域合并读取(R1和R2)。Trinity汇编程序(v2.4.0)[76.在所有生成的数据集(来自叶、茎、根、果实、花序和花的库)被合并时,使用了默认参数。在生成的contigs (unigenes)中,使用SeqClean软件(http://compbio.dfci.harvard.edu/tgi/),而来自环境的污染物序列则使用DeconSeq软件去除[77.].考虑到DeconSeq需要一个预先建立的潜在污染物序列数据库,我们从重复DNA元素(RepBase;http://www.girinst.org/repbase/)和所有来自已测序基因组中预测的基因模型的转录本,这些转录本可在GenBank中找到,用于昆虫、细菌和真菌(https://www.ncbi.nlm.nih.gov/genome/).最后的unigenes来自c·德拉科从无污染的Contig生成,最小长度为200bp。
蛋白质编码区的鉴定和注释
从中生成准确和非冗余数据集c·德拉科转录组,与编码区对应的序列从全长unegen中提取。考虑到插入和删除导致开放阅读帧的损失,两者都是从下一代技术组装的序列中的常见错误[78.,编码序列中可能出现的移码错误在翻译相应蛋白质之前得到了纠正。AlignWise管道(27.用于这个目的。一旦编码序列(CDS)被识别和校正,BLASTClust程序(ftp://ftp.ncbi.nih.gov/blast/documents/blastclust.html/)用于创建非冗余序列集。为了避免冗余,将那些CDS与另一个CDS长度至少90%相同的Unigenes去除。此外,导致序列不超过75个氨基酸的CDS被移除,因为找到与前面描述的蛋白质相似的可能性高度依赖于查询序列的长度[28.].作为注释过程的一部分,使用单向最佳点击方法(E-Value 10−05进行)以鉴定在每组参考蛋白中,那些与蛋白质同源的那些c·德拉科unigenes。作为参考蛋白,我们使用衍生自几种完整植物基因组的预测基因模型的蛋白质组。只有一个例外(拟南芥蒂利亚纳;http://www.arabidopsis.org/),所有参考蛋白质素是乱胡子的有神分植物的那些[橡胶树取代巴西橡胶树[79.],麻风树图[80],里纳斯市政府[81.],杨树trichocarpa[82.] 和木薯耐[83.]]。基于相似性分配基因本体(GO)注释答:芥蛋白质组序列。对KEGG通路和酶编码进行了分类c·德拉科使用在线Kegg自动注释服务器(KAAS)的Unigeneshttp://www.genome.jp/tools/kaas/KEGG(京都基因和基因组百科全书)数据库。最后,我们使用HMMER软件包(v3.2.1)筛选和鉴定保守蛋白域([84.];http://hmmer.org/)和pfam数据库(http://pfam.sanger.ac.uk.)包含0.0001的包含阈值。
直接同源组标识
通过Markov聚类算法(MCL)鉴定出直偶和副酰基基因,其充气值为1.5 [85.].OrthoMCL程序(41用于这个目的。OrthoMCL [41图]是一种图形聚类算法,设计用于识别基于序列相似性的同源蛋白,并区分从副骨关系的外观,而无需计算地强烈的系统发育分析。对于聚类过程,我们认为所有比较蛋白质中的30个氨基酸的最小输入长度,以及10的阈值−10在BLAST步骤中。选择这个严格的临界值是为了避免假阳性结果。从每个比较的蛋白质组中去除剪接变异,并选择最长的预测亚型作为每个基因的代表转录本。同源分析中包括的蛋白质组(均来自前一节中提到的物种Malpighiales和答:芥)是通过添加来自其他植物的某些蛋白质序列来补充的。这些酶被包括进来,因为它们都已经被功能上的描述和已知参与木兰花的生物合成(参考酶在文本中被描述)。
鉴别差异表达基因(转录组Atlas)
为了产生不同植物器官的全局转录概况的全面地图集,每个样品的高质量R1-R2读对对齐与注释和非冗余对齐c·德拉科被视为参考转录组的unigenes集合。为了最大限度地减少偏差并避免差异表达分析中的假阳性结果,在映射过程中使用大约700万个随机选择的读取。为此,我们使用了RSEM(RNA-SEQ通过期望最大化)管道[86.],处理和直接执行Bowtie2 [87.,并根据预期的读计数计算基因的最大似然丰度估计、后验均值估计和95%可信区间。RSEM还计算了标准化表达值,如每百万千基片段(FPKM)和每百万转录本(TPM)。基于这些信息,我们从每个被采样的器官中,用表示unigenes及其对应的转录水平(用预期读计数、TPM和FPKM值表示)的行和列构建一个表达矩阵。FPKM≥3的阈值用于区分至少一个器官中的表达基因,这些器官的计算值可视为背景噪声[88.].的表达式级别比较c·德拉科分析的器官之间的未成年人,Trinity软件包中包含的Perl脚本[76.使用使用TMM(M-VALUE的加权修剪均值)方法进行归一化过程[89.,90.[通过使用Edger Biocucons封装,随后的随后鉴定差异表达的基因[89.].差异基因表达分析集中于鉴定在每个器官中特异性或优先表达的那些转录物(未成果),并区分可视为普遍存在的那些。对于器官编辑配对比较,估计分散体然后用于LOGFC(日志2折叠变化)计算。此外,调整了假发现率(FDR)P-值采用Benjamini-Hochberg方法计算[91.].FDR≤0.001和logfc≥1用作阈值以判断同一个转录性丰度的差异的重要性c·德拉科两个不同的器官之间的未亚姻。为每个UNIGENE估计的FPKM值用于比较样品中的表达水平。最后,为了鉴定分析的器官中的过度持有的生物学功能,使用宾果旁的差异表达基因进行基因本体论(GO)功能性富集分析(https://www.psb.ugent.be/cbd/papers/bingo/home.html.).分析是用a进行的P-value < 0.05,基于超几何检验和benjamin - hochberg FDR校正。
定量PCR分析
使用逆转录酶定量PCR(RT-QPCR)分析进行RNA-SEQ数据的验证。用于QPCR的RNA与用于RNA-SEQ的RNA相同。使用上标III逆转录酶(Invitrogen)从分离的RNA中制备QPCR的cDNA模板。使用Primer3 V.0.4.0设计每个基因的引物(http://bioinfo.ut.ee/primer3-0.4.0/),生成附加文件中显示的引物序列9:表S15。RT-qPCR在STRATAGEN MX3005P(安捷伦技术)实时热循环仪中使用SYBR Green®PCR Master Mix (Life Technologies)进行。放大程序包括在95°C下初始变性步骤30 s,在95°C下5 s, 60°C下30 s, 72°C下30 s, 40个循环。PCR的特异性通过熔化曲线分析进行评估。的表达actin-2(AT5G09810 | Unigene122200)用作内部控制。值得一提的是,它以前报告过一些植物物种,actin-2在比较不同的组织和/或发育阶段时,它是最好的管理基因之一[92.].它的稳定c·德拉科通过比较所有采集器官的FPKM值(8.685±1.337 SE)得到确认。每一个反应对所选的对象进行三次重复c·德拉科unigenes和每个基因的荧光强度,以周期阈值(Ct)值测量,使用2(-ΔΔct)方法(93.].
系统发育分析
在我们的分析中,来自所有植物物种的蛋白质被归类为参照酶的同源物,并由SeaView处理[94.].首先,使用MUSCLE v3.8.31程序[95.],以氨基酸序列引导其相应编码序列的比对。使用一般时间可逆(GTR)替代模型在最大似然框架(ML)下生成系统发育树。PhyML v3.0软件[96.用于这个目的。此外,优化了平衡频率,拓扑和分支长度,选择了最近的邻居交换(NNI)和子树修剪和重新生成(SPR)的最佳算法。通过近似似然比测试(ALT)分析分支鲁棒性[97.].最后,使用BioEdit软件估计比对序列之间的身份矩阵(http://www.mbio.ncsu.edu/bioedit/bioedit.html.).
蛋白质建模
的c·德拉科的3D蛋白结构及其同源性作为(S)-corytuberine synthase (CTS)酶的例子的参考(如图所示)。5),使用瑞士模型工作区([98.];http://swissmodel.expasy.org/).CY17A1蛋白(PDB条目6B82.1)的结构,可用于蛋白质数据库[99.,作为模板。对生成的每个模型进行各种参数检查,包括Z、GMQE(全球模型质量估计)和QMEAN(定性模型能量分析)分数,以评估模型的准确性。一旦蛋白质被独立建模,使用SWISS-PDB viewer v4.1.0程序将它们全部叠加[100.].
通过液相色谱 - 高分辨率质谱(LC-HRMS)的Taspine和致氧化物前体的推定鉴定
叶,茎,根和花样(n=每个组织3)在冷冻干燥机(Labconco FreeZone®)中冻干。与花序(带有花茎的花序)相对应的样本不包括在植物化学分析中,因为从花茎中去除花会导致无叶的花茎,我们认为这可能与茎或花相似,这两者都是单独分析的。水果不包括在内,因为和花朵一样,水果也是季节性器官,其数量取决于授粉频率。采用加速溶剂萃取(ASE)系统(ASE 350, Dionex, Thermo Scientific)制备植物提取物。简单地说,将3 g干燥材料与1 g硅藻土混合,放置在直径2.9 cm的34 mL迪奥尼克斯不锈钢电池中。在10.342 MPa的压力下充满甲醇,加热到60°C 5 min。然后用甲醇(高效液相色谱级,Sigma-Aldrich)洗涤细胞至细胞体积的30%,为了避免偏差,将三个重复样品的甲醇提取物池化,从每个器官制备单个样品(1 mL)。采用Waters I类UPLC耦合Synapt G2-Si HDMI质谱仪作为分析平台。采用Waters Acquity BEH (1.7 μm, 2.1 × 50 mm)柱,将ASE系统获得的每个样品提取池注入2 μL。样品温度为15℃,柱温为40℃。 Water (A) and acetonitrile (B), both with 0.1% formic acid (MS grade, Sigma-Aldrich), were used as mobile phase. The solvent gradient was 0–13 min, 1–80% B; 13–14 min, 80% of B; 14–15 min 80–1% of B with a constant flow rate of 0.3 mL/min. It was used an electrospray ionization source in positive mode with a capillary, sampling cone and source offset voltages of 3000, 40 and 80 V, respectively. The source and desolvation temperatures were 100 and 20 °C, respectively. The desolvation gas flow was 600 L/h, and the nebulizer pressure was 0.65 MPa. As lock mass a mixture of leucine-enkephalin (556.2771, [M + H]+),设置为:质量范围50-1200 Da,功能1 CE, 6 V,功能2 CER 10-30 V,扫描时间0.5 s。使用MassLynx软件(v4.1, Waters™)分析生成的光谱信息。根据准确的质谱和碎片模式(允许的最大误差为3ppm)与METLIN-Scripps数据库中报道的数据(https://metlin.scripps.edu/).
通过液相色谱 - 质谱(LC-MS)定量Taspine和Magnoforline
在串联LC-MS平台中进行镁氯和干峰的定量和身份证实。分析了用于LC-HRMS测定的相同样品,但是从额外产生另外的甲醇提取物c·德拉科乳胶以5-10厘米的分支和主干的树皮在5-10厘米的间隔收获。乳胶冻干,将1mg溶于1ml甲醇中,0.1%的甲酸(MS级,Sigma-Aldrich)以分析LC-MS-MS。使用耦合到6460三重四极其(QQQ)质谱仪的安捷伦技术1290 UPLC进行色谱分离和检测Taspine和Magnoforline,用Jetstream电喷雾(ESI)源。所用的柱是安捷伦ZORBAX SB-C18,2.1×50mm,1.8μm,塔温为40℃。流动相是水(A)和乙腈(B),其含有0.1%甲酸(MS级,Sigma Aldrich),其流速为0.3ml / min。溶剂梯度为0-10分钟,B的5-30%;10-11分钟,B的30-95%;11-12分钟,95%的B且最终12-14分钟,B的95-5%,1分钟保持不变。注射体积为1μL,并将各自的提取物池一式三份注入。质谱仪设置为气温300°C,流量为5升/分钟。 The nebulizer pressure was set at 0.31 MPa, and the sheath gas temperature and flow were 250 °C and 11 L/min, respectively. The capillary and nozzle voltages used were 3500 and 500 V, respectively. The polarity used for the analysis was positive with a fragmentor voltage of 135 V, and a collision energy of 10 V. The acquisition method used was dynamic multiple reaction monitoring (dMRM) with the transitions 342.1 > 296.7 and 370.1 > 325.1 for magnoflorine and taspine, respectively. The magnoflorine commercial standard was purchased from Sigma-Aldrich and taspine was purified in-house by using the protocol described previously [17.].为具有10个校准点的每个化合物(0.25,0.5,1,3,5,7,9,11,13,15和17μm)制备校准曲线。将每个校准点注射两次,并且绘制区域的浓度。执行二次回归以获得r2每种化合物和数量的值> 0.99的值是通过使用MassHunter工作站软件VB.06.00(安捷伦技术)建立的。结果表示为样品(干重)的μg/ g。
含有根和茎的树皮中的含税细胞丰富
为了分析不同植物器官中的Taspine含量与含型含型细胞的丰度直接相关,我们量化了根系中的含型乳脂细胞的密度和茎上的树皮。我们分析了这些器官,因为它们在独特的器官中的丰富根据植物物种而异;2,101.,102.].在属的种类中巴豆,大量乳管细胞分布于根的皮层区域和茎的树皮中[1],收获乳胶。从上述同一树收集的样品(参见植物材料部分),用植物固定剂FAA(福尔马林:冰酸乙酸:70%乙醇,5:5:90,体积),通过一系列乙醇脱水溶液和嵌入石蜡(熔点54-56°C)[103.].用旋转切片体获得12-15μm厚度为12-15μm的横向和切向纵向截面,并用Safranin和Fast-Green染色[103.].对于永久性制剂,使用合成树脂固定液。尼康Eclipse E600显微镜配备了数码相机(佳能Eos Rebel t3i 600D),用于获取堆叠图像。用大约15张不同的图像来确定乳汁管的密度。
数据和材料的可用性
本研究期间生成或分析的所有数据都包含在本发布的文章及其补充信息文件中。
缩写
- 4米:
-
(S) 3 '羟基N-methylcoclaurine 4 ' -O-methyltransferase
- 6欧特:
-
(s)-norcoclaurine 6'-O-甲基转移酶
- aLTR:
-
近似可能性 - 比率测试
- CNMT:
-
(S) -coclaurine N-methyltransferase
- CTS:
-
-corytuberine合成酶
- 罗斯福:
-
错误发现率
- FPKM:
-
每百万每千季碎片
- 去:
-
基因本体论
- GTR:
-
一般时间可逆
- HC:
-
分层群集
- LC-HRMS:
-
液相色谱 - 高分辨率质谱
- LC-MS-MS:
-
液相色谱-串联质谱法
- ml:
-
最大似然
- ncrnas:
-
非编码RNA
- NCS:
-
-norcoclaurine合成酶
- NMCH:
-
(S)-N-甲基氯磺胺3'-羟化酶
- NNI:
-
最近的邻居交换
- PCA:
-
主成分分析
- RNMT:
-
reticaline n-甲基转移酶
- RT-qPCR:
-
逆转录酶定量PCR
- SPR:
-
子树修剪和重新重建
- TPM:
-
记录每百万
- TYDC:
-
酪氨酸脱羧酶
- UTRS:
-
未经翻译的地区
参考
- 1。
Farias FR,威廉姆森JS,Rodriguez SV,Angeles G,葡萄牙vo。树皮解剖学克罗顿德拉科var。德拉科(大戟科)。植物学报。2009;96(12):2155-67。
- 2。
Castelblanque L,Balaguer B,Marti C.进入乳房细胞组织的新颖见解:包括统一侦察系统的细胞。2016; 172(2):1032-44。
- 3.
D'Auzac J,Jacob JL,Chrestin H.橡胶树乳胶生理学:含含型细胞和乳胶 - 一种细胞质模型,1 EDN:CRC压力机;1989年。
- 4.
维管形成层和木材中的乳状管巴豆SPP。(大戟科)。IAWA J. 1989; 10(4):379-83。
- 5.
关键词:“射线侵入”乳汁管巴豆切片环柱头(大戟科),第30卷;2009.
- 6。
Nessler Cl,Mahlberg PG。亚太福尔斯在雄蕊果实L. planta。1976; 129(1):83-5。
- 7。
哈格尔,杨永杰,法奇尼。有牛奶吗?乳汁管的秘密生活。植物学报。2008;13(12):631-9。
- 8。
皮卡德WF。乳汁管和分泌管:植物中的另外两种管系统。新植醇。2008;177(4):877 - 88。
- 9。
作为植物防御系统的植物乳胶和其他分泌物:其中包含的各种防御化学物质和蛋白质的作用。植物化学。2011;72(13):1510 - 30。
- 10。
Murillo RM, Jakupovic J, Rivera J, Castro VH。二萜和其他成分克罗顿德拉科(大戟科)。翻转Biol roph。2001; 49(1):259-64。
- 11.
Salatino A,Salatino MLF,Negri G.传统用途,化学和药理学巴豆物种(大戟科)。J Braz Chem Soc. 2007; 18:11-33。
- 12.
MOTTA LB,Furlan CM,Santos Dyac,Salatino MLF,Duarte-Almeida JM,Negri G,JED C,Altg R,Cordeiro I,Salatino A.来自叶子的提取物的成分和抗增殖活性巴豆macrobothrys.Rev Bras。2011; 21:972-7。
- 13.
Cai Y,Evans FJ,Roberts MF,Phillipson JD,Zenk MH,Gleba Yy。来自的多酚化合物克罗顿莱切王.植物化学。1991;30(6):2033 - 40。
- 14.
蔡勇,陈志平,飞利浦JD。Clerodane二萜来自克罗顿莱切王.植物化学,1993;34(1):265 - 8。
- 15.
蔡勇,陈志平,飞利浦JD。迪尔替代克罗顿莱切王.植物化学,1993;32(3):755 - 60。
- 16。
从南美龙血中分离二氢苯并呋喃木脂素(巴豆作为细胞增殖的抑制剂。中国科学(d辑:地球科学)1993;56(6):899-906。
- 17。
De Marino S, Gala F, Zollo F, Vitalini S, Fico G, visoli F, Iorizzi m克罗顿莱切王(Muell-Arg)和对抗氧化活性的评价。分子。2008; 13(6):1219-29。
- 18。
Milanowski DJ, Winter RE, Elvin-Lewis MP, Lewis WH三种生物碱化学型的地理分布克罗顿莱切王.中国科学(d辑:地球科学)2002;65(6):814-9。
- 19。
Vaisberg AJ, Milla M, Planas MC, Cordova JL, de Agusti ER, Ferreyra R, Mustiga MC, Carlin L, Hammond GB。塔斯匹碱是桑热德的愈合剂克罗顿莱切王.Planta Med。1989; 55(2):140-3。
- 20.
陈志平,蔡勇,飞利浦JD。龙血抗肿瘤、抗菌、创面愈合特性的研究。足底医学。1994;60(6):541 - 5。
- 21.
蒙托波里M,Bertin R,Chen Z,Bolcato J,Caparrotta L,Froldi G.克罗顿莱切王SAP和孤立的生物碱表现出对人黑素瘤SK23和结肠癌HT29细胞系的抑制作用。J属植物。2012; 144(3):747-53。
- 22。
张继,张Y,潘X,王某,他是新型对称毒素衍生物作为抗癌剂的合成和细胞毒性评价。Med Chem。2011; 7(4):286-94。
- 23。
张军,张艳,单颖,李娜,马伟,何磊。新型塔斯品衍生物的合成及其抗癌活性的初步评价。中华医学杂志。2010;45(7):2798-805。
- 24。
Shamma M,Moniot JL。将氧化氟化物转化为干峰的生物地图案化转化。J Chem SoC D Chem Communce。1971; 18:1065-6。
- 25。
Silva Ga:estudofarmacognósticode克罗顿乌鲁库塔纳Baillon(Sangra d'Agua).Tesis Moftal en Insumosfarmacêuticos.圣保罗:DeSãoPaulo大学;1999.
- 26。
Arvalo A,Lotti C,Piccinelli A,Russo M,Ruiz I,Rastrelli L.叶片和酚醛衍生物的叶子巴豆xalapensisL.(大戟科),卷4;2009.
- 27。
Evans T,松散的M. SimplyWise:一种用于识别蛋白质编码序列和校正框架偏移的工具。BMC生物信息学。2015; 16:376。
- 28。
Novaes E,Drost Dr,Farmerie Wg,Pappas GJ JR,Grattapaglia D,Sederoff Rr,Kirst M.高通量基因和SNP发现桉树祖母,一个不表征的基因组。BMC基因组学。2008; 9:312。
- 29。
Finn Rd,Coggill P,Eberhardt Ry,Eddy SR,Mistry J,Mitchell Al,Potter SC,Punta M,Qureshi M,Sangrador-Vegas A等。PFAM蛋白质家族数据库:迈向更可持续的未来。核酸RES。2016; 44(D1):D279-85。
- 30.
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT等:基因本体:生物学统一的工具。基因本体学报2000,25(1):25 - 29。
- 31。
Simao Fa,Waterhouse Rm,Ioannidis P,Kriventseva EV,Zdobnov Em。BUSCO:评估基因组组装和注释完整性,单拷贝正端。生物信息学(英国牛津大学)。2015; 31(19):3210-2。
- 32。
张某,Scheller HV。光收获综合体II与照相系统I的几个小亚基结合.J Biol Chem。2004; 279(5):3180-7。
- 33。
织机TM,罗斯JL,Fagerlund Rd,Frankel Lk,伊顿黑麦JJ。照相系统II的外在蛋白。Biochimica et Biophysica Acta(BBA) - 生物能器学。2012; 1817(1):121-42。
- 34。
Izumi M,Tsunoda H,Suzuki Y,Makino A,Ishida H. RBCS1A和RBCS3B,两个主要成员拟南芥RBCS多基因家族,具有产生足够的Rubisco含量以维持叶片光合能力的功能。中国生物医学工程学报。2012;63(5):2159-70。
- 35。
Yamakawa S,Ando K,Chisaka A,Yoshida K,Shinmyo A,Kohchi T.通过用CDNA微阵列鉴定的基因表达分析鉴定的叶特异性基因的启动子活动的系统瞬态测定拟南芥蒂利亚纳.J Biosci Bioeng。2004; 98(2):140-3。
- 36。
Jakobsen MK,Poulsen LR,Schulz A,Fleurat-Cardard P,Moller A,Schiott M,Amtmann A,Palmgren Mg。花粉开发与施肥拟南芥依赖于编码V p型ATP酶的雄性配子生成受损的花药基因。基因开发。2005; 19(22):2757-69。
- 37。
Sjodahl S,Gustavsson Ho,Rodin J,Lenman M,Hoglund AS,Rask L.Cruciferin基因家族的协调,但在组织特异性差异芸苔属植物显著种子发展。植物学报。1993;23(6):1165-76。
- 38.
Hajduch M,Hearne Lb,Miernyk Ja,Casteel Je,Joshi T,Agrawal GK,Song Z,周M,Xu D,Thelen JJ。种子填充的系统分析拟南芥:使用一般线性建模来评估转录物和蛋白质表达的一致性。植物理性。2010; 152(4):2078-87。
- 39.
Hegedus DD,Coutu C,Harrington M,Hope B,Gerbrandt K,Nikolov I.多个内部分拣决定簇可以促进贩运十字花蛋白到蛋白质储存液泡。植物mol biol。2015; 88(1-2):3-20。
- 40。
Czechowski T,Bari RP,Stitt M,Scheible WR,Udvardi Mk。实时RT-PCR分析超过1400拟南芥转录因子:前所未有的敏感性揭示了新的根和幼虫基因。工厂J. 2004; 38(2):366-79。
- 41。
李L, Stoeckert CJ Jr, Roos DS。OrthoMCL:真核生物基因组同源组的鉴定。基因组研究》2003;13(9):2178 - 89。
- 42。
王敏,赵颖,张博。多集交叉口的高效测试与可视化。Sci众议员2015;5:16923。
- 43.
细胞色素P450在木兰花碱生物合成中催化C-C酚偶联反应的分子克隆和特性研究Coptis japonica.细胞。J Biol Chem。2008; 283(14):8810-21。
- 44.
莫里斯js,facchini pj。鸦片罂粟中白血管生物碱氧化钒生物合成中涉及的网状氨基N-甲基转移酶的分离与表征。J Biol Chem。2016; 291(45):23416-27。
- 45.
Ziegler J, Facchini PJ, Geißler R, Schmidt J, Ammer C, Kramell R, Voigtländer S, Gesell A, Pienkny S, Brandt W.罂粟中吗啡生物合成的进化。植物化学,2009;70(16):1696 - 707。
- 46。
Gesell A,Rolf M,Ziegler J,Diaz Chavez ML,黄FC,Kutchan TM。CYP719B1是Salutaridine合成酶,鸦片罂粟中吗啡生物合成的C-C酚偶联酶。J Biol Chem。2009; 284(36):24432-42。
- 47。
Farrow SC, Hagel JM, Beaudoin GA, Burns DC, Facchini PJ:通过细胞色素P450融合在罂粟中(S)网状素的立体化学转化。2015年,11(9):728 - 732。
- 48。
Wijekoon CP,Facchini PJ。使用病毒诱导的基因沉默在鸦片罂粟中吗啡途径酶的系统敲低。工厂J. 2012; 69(6):1052-63。
- 49。
陈志强,陈志强,陈志强,等。N-甲基转移酶基因的克隆与表达分析Coptis japonica..J Biol Chem。2002; 277(1):830-5。
- 50.
Pauli Hh,Kutchan TM。两种等位基因编码的分子克隆和功能异源表达式 - 甲基克霉素3'-羟化酶(CYP80B1),一种新的甲基丙氨酸诱导的细胞色素p-450依赖性单氧酶的苄基异喹啉生物合成。工厂J.998; 13(6):793-801。
- 51.
Marques JV,Dalisay DS,Yang H,Lee C,Davin LB,Lewis Ng。多OMICS策略解决了生物碱的难以捉摸的性质Podophyllum物种。摩尔BioSyst。2014; 10(11): 2838 - 49。
- 52.
Luk Ly,Bunn S,Liscombe DK,Facchini PJ,Tanner Me。苄基异喹啉生物碱生物合成诺尔科霉素合成酶的机理研究:酶促体育术 - 辛酸反应。生物化学。2007; 46(35):10153-61。
- 53.
罂粟中酪氨酸/多巴脱羧酶基因家族的差异表达和组织特异性表达。生物化学学报1994;269(43):26684-90。
- 54.
黄志强,王志强,王志强,等。谷丙草酸合成酶的功能分析Coptis japonica..中国生物化学杂志。2007;32(9):6274-82。
- 55。
Samanani N,Liscombe DK,Facchini PJ。NorcoClaurine合酶的分子克隆与表征,催化苄基异喹啉生物合成中的第一种承诺步骤的酶。工厂J. 2004; 40(2):302-13。
- 56。
oounaroon a,decker g,schmidt j,lottspeich f,kutchan tm。(R,S) - 甲磺酸氢碱7-O-甲基转移酶和(R,S)-NORCOCLAURINE 6-O-甲基转移酶果实- 鸦片罂粟中生物碱生物合成甲基转移酶的cDNA克隆与表征。工厂J. 2003; 36(6):808-19。
- 57。
陈志强,陈志强,陈志强,等。氨基甲基转移酶的分子克隆Coptis japonica.细胞。EUR J Biochem。2002; 269(22):5659-67。
- 58。
3 ' -羟基- n -甲基氯代嘌呤4 ' - o -甲基转移酶在异喹啉生物碱生物合成中的应用Coptis japonica..J Biol Chem。2000; 275(30):23398-405。
- 59。
Frick S,Kutchan TM。异喹啉生物碱和苯丙烷生物合成共同的O-甲基转移酶的分子克隆和功能表达。工厂J.17(4):329-39。
- 60.
Frick S,Ohnaroon A,Kutchan TM。植物组合生物化学:O-甲基转移酶的情况。植物化学。2001; 56(1):1-4。
- 61.
Hadjipieri M,Georgiadou Ec,Marin A,Diaz-Mula HM,Goulas V,Fotopoulos V,Tomas-Barberan Fa,Manganaris Ga。枇杷果实果皮组织在树木发育过程中的语说和转录释放肉毒状生物合成途径。BMC植物BIOL。2017; 17(1):102。
- 62.
潘辉,杨伟,姚超,沈勇,张勇,石昕,姚胜,吴伟,郭德成。基于质量缺陷过滤分类和前驱离子列激发的高分辨率质谱分析在吲哚生物碱中发现的应用钩藤制成.J Chromatogr A. 2017; 1516:102-13。
- 63.
scossa f,Benina M,Alseekh S,张Y,Fernie AR。代谢组织和下一代测序数据的整合阐明了药用植物中天然产物代谢的途径。Planta Med。2018; 84(12-13):855-73。
- 64。
SACCHETTI G,Ballero M,Serafini M,Romagnoli C,Bruni A,Poli F. Laticifer组织分布和生物碱位置Vinca Sardoa.(搁凳)演讲。(apocynaceae),撒丁岛的地方植物(意大利),Vol。39;1999.
- 65。
Rudall PJ。大戟科乳汁管。acta botanica yunnanica(云南植物研究). 1987;94(1-2):143-63。
- 66。
rudall p.乳酪在克罗托康康科(Euphorbiaceae):同源性和演化。安莫僵尸加德。1994年; 81(2):270-82。
- 67.
Rush Md,Kutchan TM,COSCIA CJ。萌发中吗啡南生物碱和含型细胞的外观的相关性Papaver Bracteatum.幼苗。植物细胞代表1985; 4(5):237-40。
- 68.
次级代谢物的转运体。植物学报。2005;8(3):301-7。
- 69.
Shitan N,Yazaki K.植物生物碱的积累和膜运输。Curr Pharm Biotechnol。2007; 8(4):244-52。
- 70。
黄志强,林志田,叶志强,等。烟草中烟碱转运体的时空变化及其对烟碱转运和积累的影响尼科尼亚塔哈瓦姆.植物信号表现。2015; 10(7):E1035852。
- 71.
El-Sayed M,Verpoorte R.凯蒂斯希斯萜类吲哚生物碱的生物合成与调控。Phytochem启2007;6(2):277 - 305。
- 72.
shji T, Yamada Y, Hashimoto T.茉莉酸诱导的腐胺n -甲基转移酶基因在根烟草的抗旱性.植物生理学报。2000;41(7):831-9。
- 73.
Mohamed Sm,Hassan Em,Ibrahim Na。细胞毒性和抗病毒活性的间歇性生物碱洋玉兰[j] .环境科学与技术,2010;24(15):1395-402。
- 74.
刘超,范超。德诺维菠萝花粉的转录组合和鉴别血管诱导鉴定差异表达基因的鉴定。前植物SCI。2016; 7:203。
- 75.
黄志强,王志强,王志强,等。一种多酚和多糖丰富的多肉植物RNA分离方法的改进。植物学报。2000;18(4):369-76。
- 76.
Grabherr Mg,Haas Bj,Yassour M,Levin JZ,Thompson Da,Amit I,Adiconis X,Fan L,Raychowdhury R,Zeng Q等。来自RNA-SEQ数据的全长转录组件没有参考基因组。NAT BIOTECHNOL。2011; 29(7):644-52。
- 77.
从基因组和宏基因组数据集快速识别和去除序列污染。《公共科学图书馆•综合》。2011;6 (3):e17288。
- 78.
杨X,Chockalingam SP,Aluru S.下一代测序的误差校正方法调查。简短生物形式。2013; 14(1):56-66。
- 79。
唐C,杨米,方y,罗y,gao s,xiao x,z,周b,张b,tan x等。橡胶树基因组揭示了橡胶生产和物种适应的新见解。Nat植物。2016; 2(6):16073。
- 80.
Sato S,Hirakawa H,Isobe S,Fukai E,Watanabe A,Kato M,Kawashima K,Minami C,Muraki A,Nakazaki N等。含油树基因组的序列分析,麻风树图L. DNA Res。2011; 18(1):65-76。
- 81.
Chan AP,Crabtree J,Zhao Q,Lorenzi H,Orvis J,Puiu D,Melake-Berhan A,Jones Km,Redman J,Chen G等人。油籽种类草案的基因组序列里纳斯市政府.生物科技Nat》。2010;28(9):951 - 6。
- 82.
托斯康········乔西奥,詹森斯,波利曼·j,格里戈里艾尔,我,地狱,普通n,拉尔夫斯,rombauts s,萨拉玛夫A等。黑色棉布的基因组,杨树trichocarpa(托尔。&灰色)。科学(纽约,纽约)。2006; 313(5793):1596-604。
- 83.
王伟,冯波,肖建军,周昕,李鹏,张伟,王颖,张鹏,等。从野生祖先到栽培品种的木薯基因组。Nat Commun。2014;5:5110。
- 84.
Finn Rd,Clement J,Eddy SR。HMMER Web服务器:交互式序列相似性搜索。核酸RES。2011; 39(Web服务器问题):W29-37。
- 85.
Enright j, Van Dongen S, Ouzounis CA.一种大规模检测蛋白质家族的高效算法。核酸学报2002;30(7):1575-84。
- 86.
李斌,杜威出版社。RSEM:从有或没有参考基因组的RNA-Seq数据中准确的转录本定量。BMC生物信息学。2011;12:323。
- 87.
Langmead B,Salzberg SL。与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9(4):357-9。
- 88。
Mortazavi A,Williams Ba,McCue K,Schaeffer L,Wold B.用RNA-SEQ进行测绘和量化哺乳动物转录om。NAT方法。2008; 5(7):621-8。
- 89。
罗宾逊医学博士,麦卡锡DJ,史密斯GK。edgeR: Bioconductor包,用于数字基因表达数据的差异表达分析。生物信息学(英国牛津大学)。26(1): 139 - 2010; 40岁。
- 90。
狄米尔MA,RAU A,Aubert J,Hennequet-Antier C,Jeanmougin M,Servant N,Keime C,Marot G,Castel D,Estelle J等。Illumina高通量RNA测序数据分析综合评价。简短生物形式。2013; 14(6):671-83。
- 91。
Benjamini Y,Hochberg Y.控制虚假的发现率:多种测试的实用和强大的方法。j r stat soc ser b方法。1995年; 57(1):289-300。
- 92。
Jian B, Liu B, Bi Y, Hou W, Wu C, Han T. quantitative real-time PCR用于大豆基因表达的内控验证。BMC Mol Biol. 2008;9:59。
- 93。
Livak KJ,Schmittgen TD。使用实时定量PCR和2的相对基因表达数据分析(-Delta Delta C(T))方法。方法。2001; 25(4):402-8。
- 94。
海景版本4:序列比对和系统发育树构建的多平台图形用户界面。acta botanica sinica, 2010;27(2):221 - 224。
- 95.
埃德加钢筋混凝土。MUSCLE:多序列比对,高精度,高通量。核酸学报2004;32(5):1792-7。
- 96.
Guindon S,Gascuel O.一种简单,快速,准确的算法,通过最大可能性来估计大的系统发育。系统中的生物学。2003; 52(5):696-704。
- 97.
Anisimova M,Gascuel O.分支机构的近似似然比测试:快速,准确,强大的替代品。系统中的生物学。2006; 55(4):539-52。
- 98.
Arnold K,Bordoli L,Kopp J,Schwede T.瑞士模型工作区:一种基于Web的蛋白质结构同源性建模的环境。生物信息学(英国牛津大学)。2006; 22(2):195-201。
- 99.
Kiefer F, Arnold K, Kunzli M, Bordoli L, Schwede T. SWISS-MODEL库和相关资源。核酸Res. 2009;37(数据库版):D387-92。
- One hundred.
guex n,peitsch mc。瑞士型号和瑞士PDBViewer:对比较蛋白质建模的环境。电泳。1997年; 18(15):2714-23。
- 101.
Condon JM, Fineran BA。铰接乳汁管的分布和组织Calystegia Silvatica.(络vulaceae)。BOT GAZ。1989; 150(3):289-302。
- 102.
ABD Razak SB。植物含锰的开发与分布。达勒姆:达勒姆大学;2000年。
- 103.
Ruzin SE。植物显微技术和显微镜。牛津大学出版社:美国;1999.
致谢
我们要感谢Diana Sánchez-Rangel和Jazmín Blaz进行的富有成果的讨论和他们提出的宝贵建议。我们也感谢Alan Josué Pérez-Lira的技术支持。我们感谢特雷弗·威廉姆斯在审阅手稿时给予的宝贵帮助。最后,我们也感谢匿名审稿人的积极和相关的评论,这提高了本文的质量。
资金
这项工作得到了Consejo Nacional de Ciencia YTecnologí(Conacyt)的支持,授予223323(EI)。该资助者在研究和收集,分析和解释的设计中没有作用,以及编写手稿。
作者信息
从属关系
贡献
EI构思并领导了这项研究,并撰写了这篇论文。AC进行转录组组装、注释、统计分析和额外的生物信息学分析。FR和GA进行了解剖研究。EV建议使用高性能计算系统和生物信息学分析。AA监督RNA库的制备和测序。AG、LM和FR进行植物化学筛选。AC、FR、AG、LM、EV、AA、AP、GA、EI对数据进行分析和收集。所有作者阅读并批准了最终的手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者宣称,这项研究是在没有任何商业或财务关系的情况下进行的,这些关系可以被解释为潜在的利益冲突。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
摘要的测序数据产生克罗顿德拉科.
附加文件2:
单基因序列大于1000 bp。(FASTA 51035 kb)
3:额外的文件。
单基因序列短于1000 bp。(FASTA 67268 kb)
附加文件4:图S1。
的频率c·德拉科unigenes长度分布。图S2。c·德拉科转录组映射到KEGG全球代谢网络,图S3。基因本体(GO)富集的术语富含大量优先表达的基因,并在分析的器官(叶,茎,根,水果和花序中鉴定,有和没有花卉景观),图S4。RNA-Seq数据的RT-qPCR验证,图S5。木兰花碱和吗啡的生物合成途径图S6。木兰花碱、塔斯品碱及其部分中间体的离子搜索和HRMS鉴定(PPTX 4505kb)
附加文件5:表S2。
注释的c·德拉科冗余(nr) unigenes。
附加文件6:表S3。
功能分类c·德拉科unigenes。
附加文件7:表S4。
表达谱矩阵c·德拉科unigenes。表S5.优先表达基因的表达谱。表S6.正交蛋白质(Orthogroups)。
附加文件8:
与参考蛋白同源的每对蛋白的百分率鉴别矩阵:酪氨酸脱羧酶(表S7(S) -norcoclaurine合成酶表S8和表S9),(s)-norcoclaurine 6'-O-甲基转移酶表S10),(s)-coclaurine n-甲基转移酶(表S11)、(S)- n -甲基氯代酸3 ' -羟化酶(表S12), (S)-corytuberine合成酶(表向).
附加文件9:表S14
:木兰花碱和塔斯品碱的推定鉴定,以及通过离子搜索和HRMS分析鉴定的一些直接前体。表S15.用于RT-QPCR测定的引物
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Canedo-Téxon,A.,Ramón-Farias,F.,Monribot-Villanueva,J.L。et al。通过转录组学和代谢组学分析,发现木兰碱和塔斯品碱生物合成途径的新发现克罗顿德拉科(大戟科)。BMC植物杂志19,560(2019)。https://doi.org/10.1186/s12870-019-2195-y
已收到:
接受:
发表:
关键字
- 克罗顿德拉科
- Aporphine生物碱
- 镁氟
- 卡斯巴尼
- RNA-seq