摘要
背景
玉米经历了大约5至12万年前的全基因组重复事件。因为这种事件发生在来自高粱的物质后,所以可以通过将同义区域映射到高粱染色体来部分地重新构建预复制子组。在进化期间,玉米在每个古代亚群之间存在不均匀的基因丧失。这些基因组之间的分馏和分歧持续到今天,不断变化遗传构成和表型和影响农艺性状。
结果
在这里,我们对最新的玉米参考基因组组件进行了亚基因重建。基于多种组织的职业基因对的表达和丰度数据,我们观察到亚因子中基因的功能分歧。虽然较大玉米亚组中的基因通常在较小的亚群中的物质中表达更高,但我们观察到在不同组织中的家庭表达优势开关的情况下观察到病例。我们首次证明蛋白质丰富在较大的亚因子中的第一次,但它们还显示出组织特异性的优势,一种类似于RNA表达优势的模式。我们还发现花粉表达与蛋白质丰富唯一地解耦。
结论
我们的研究表明,较大的亚因子具有更大范围的功能分配,并且在基因函数方面存在亚因子之间的相对缺乏重叠,而不是通过基因表达和蛋白质丰度的类似模式提出。我们的研究还透露,一些反应由较大和较小的亚因子唯一催化。这里观察到的组织特异性的非等级表达级别优势模式意味着调节控制的变化,其利用差异化的保留重复液的选择性压力导致基因功能的最终变化。
背景
通过多倍体的全基因组重复发生在许多真核生物中,包括小麦,大豆和棉[1].通过一些估计,在50%至95%之间所有的Angiospers都经历了至少一个多倍体事件[2那3.].据信玉米在古老的品质活动中偏离高粱[4.],其次是在5至12万年之间发生的全基因组重复事件,导致玉米祖先成为四倍体的[5.].基因组重复和随后的融合后,玉米减少到二倍体状态[6.].基因流失在基因组的一种拷贝中,比其他副本更大的速率 - 一种称为通过短缺失机制作用的分馏偏差的现象[7.],导致较大的(麦芽1)和更小(MAIZE2)亚因子。以前的工作由Brohammer等。[8.]发现分馏模式在多种玉米线之间看起来类似。
重复基因的功能分歧[9.]先前已在拟南芥中进行过研究[10.),芸苔属植物11.], 棉 [2]和玉米[12.].推测,单一核苷酸多态性(SNP)和插入/缺失(吲哚)的累积以及对上游调节元件的变化和转座子干扰导致功能性分歧。POL IV介导的玉米介导的基因沉默研究[13.]发现转发元件(TE)确实影响表达,并且在麦芽2上更常见的(对于RPD1 / rMR6)而不是MAIZE1,而非同步基因更可能具有上游的TES(即,通过TE Shuffling的可能产生)。
以前的工作由Moore等人。[14.[图]示出了一些可能的姓氏复制基因可能会给足够的进化时间体验。虽然我们无法将现代基因直接与古代同行进行比较,但我们可以通过比较彼此的现代重复项来探视在进化时间的可能结果。如果我们假设全基因组复制事件导致具有类似功能和表达模式的重复基因,则应推出保守重复基因之间的表达模式的变化将导致一个在其中一个中的监管函数的增益或丧失副本。
保留重复剂表达之间的关系通常被描述为优势(MAIZE1)亚群[9.那15.那16.在总);然而,有证据表明,保留副本之间的关系并不总是简单的优势关系[16.].在棉花中,有互惠沉默的证据表明[2]使得保留重复对中的一种拷贝在一些组织中表达但不是其他组织,而另一副本在不同的组织中表达。以前的表达数据分析[16.[平均母猪2基因组均表达玉米1基因组的普遍趋势,但也鉴定了被描述为保留重复率的内容,其中在不同组织中的重复基因切换了优势。此外,他们发现抑制基因倾向于体验更大的转座载荷,但与非抑制基因相比,在从开放阅读框的5'末端到最近的上游转座子的距离显着差异。
在这项工作中,我们使用先前描述的协议重新生成子组组分配[15.]在使用最新的玉米参考基因组组件时,B73 Refgen_v4 [17.].通过从现有表达式Atlas看基因表达和蛋白质丰富模式[18.我们研究了职业副本之间的功能差异。我们还看看基因本体[19.那20.]来自Maizecyc的注释[21.]和uniprot [22.],来自Corncyc的途径[23.]和基因组组装组提供的结构注释中每个基因模型的同种型数量[24.].我们发现,Maize1亚基因组具有更大的GO赋值范围,在GO注释方面,亚基因组之间相对缺乏重叠,这比表达和丰度数据所提示的要少。我们首次证明,玉米1号的蛋白质丰度高于玉米2号,但它们也显示出组织特异性优势,类似于玉米1号的RNA表达优势。我们还发现花粉表达与蛋白质丰度是独特的解耦。最后,提出了一种基于表达模式的保留对分类方法。
结果
RefGen_v4中的亚基因组分配
通常认为同义突变在弱的选择性约束下,随着时间的推移逐渐积累。通过将同步区域的ks(同义突变率)与除群中的ks(同义突变率)比较,可以利用该前提来估算类似年龄的区域。我们遵循Schnable等人之前描述的方法。[15.]使用以下修改生成B73 Refgen_v4中的基因模型的子组组分配。通过将未掩蔽的玉米B73 Refgen_V4与未掩蔽的遮挡玉米B73 Refgen_V4进行比较来确定玉米和高粱之间的同时性区域高粱双色位于CoGe上的SynMap工具中的v3.1 [25.].我们过滤出含有少于12个基因和同步块的小同步块,其块平均值高于1.0。我们利用贪婪的方法来组合同步块,其尺寸与相对于高粱的非重叠基因模型。
最近的一项研究表明,玉米和当前日与乌苏里亚/伏索之间的共同进化关系,这可能会为祖传玉米和祖先高粱(祖先玉米)的父母的父母的性质提供线索[26.].有人提出,这些父母可以与玉米中的现代亚因素联系起来,可以揭示亚因素的起源,但是对于本研究的目的,我们继续使用Schnable等人提到的Maize1和Maize2的定义。[15.].我们分配给玉米1亚基和较小,更分馏的设定的较大,较少分馏的设置到玉米2亚群。另外,密码子偏置和侵犯对同学突变的弱选择性约束的假设,这些突变已经在其他生物中观察到[27.那28.],可能会影响群组的分配方式,但是我们目前不了解累积沉默突变的基准,这将允许校正这种可能性。额外的文件中提供了亚群分配和保留重复基因的列表1。
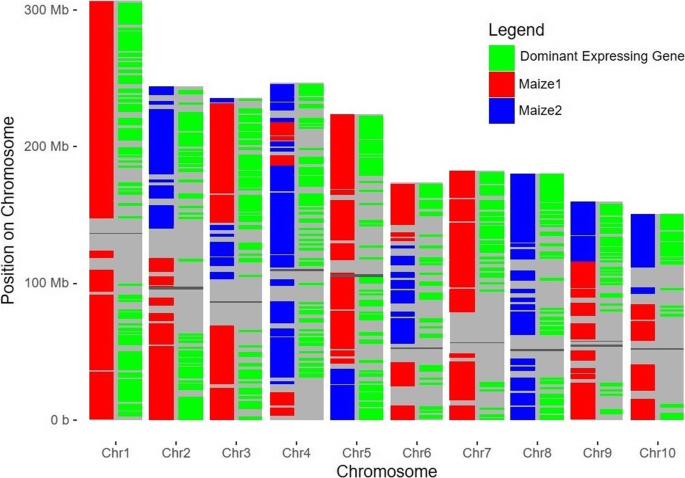
每个基因组都分散了占优势的家庭
玉米1和麦芽2的位置绘制在图2中的染色体上。1。在每个染色体的右侧标记在所有染色体的右侧至少2倍的保留重复副本至少2倍(参见附加文件2).其中一个或另一副本的统治性案例广泛分布在所有染色体上。分配给Maize1的染色体4的远端显着不存在显性保留的重复态。
玉米1的基因表达量和蛋白质丰度通常更高,但并不总是在相同的组织和相同的生长水平
使用STAR测量每百万FPKM转录本的每千碱基片段的基因表达[29.]及袖扣[30.关于B73可用的测序数据[18.].Cufflinks在这些位置之间平均分配映射到多个位点的读取,这使得它能够处理映射到多个同源基因的读取产生的歧义。在所有组织中,maze1同源基因的表达量是其重复的maze2同源基因的2倍或更高。在各组织中,有51.1 ~ 57.9%的玉米ze1基因的表达量高于玉米ze2,但在FPKMs中,玉米ze2基因的表达量远高于玉米ze1,在成熟花粉和果皮中表达量略高于玉米ze1。
我们发现,与平均玉米2职业相比,也观察到玉米1职业有2倍或更大的蛋白质丰富。成熟花粉中的平均蛋白质在相对于其他组织中的两种子组中特别高,并且在玉米1中的玉米1中大得多。但是DNSAF(分布式标准化光谱丰度因子)的平均玉米蛋白丰度[31.]在成熟叶、根分生组织、根伸长、胚乳12 DAP、次根、初生根和营养分生组织等7个组织中均较大。
发现B73成熟花粉比其他组织的表达基因较少,以及较少的可检测的蛋白质丰富。花粉具有表达与丰度之间的最低相关性(图。2)并含有几种具有非常高的表达。以前的其他玉米数据集的研究发现,通常具有更高的转录因子表达,并具有较高的玉米基因表达[32.].虽然我们发现玉米1基因高于其玉米2对应于反向,但MAIZE2中的基因的平均FPKM均高于B73成熟花粉和胚乳冠27DAP的玉米1。此外,如果将来自每个亚基因组的所有基因的FPKM(不仅仅是具有保留的复制品)的FPKM,则平均,在23种组织中只有5个在玉米组织中具有更高的平均表达。
表达和丰富显示了保留重复的组织特异性主导模式
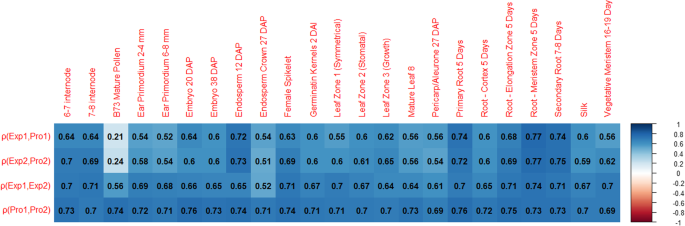
基因表达的分析通常使用平均值或其他聚集体来确定折叠变化和表达优势。然而,在保留的重复项之间表达优势的较大趋势可以掩盖在检查不同组织,条件或时间点的表达模式时发现的单个细微差。无花果。3.呈现四种代表性案例,其中可以将其中一个保留的重复项称为在表达式中仅在可用组织的平均FPKM时表示“显性”职业。这些情景包括但不限于:a)具有一个职业的平均优势的高度相关性表达,b)一个职业层面具有很少或没有检测到的表达,c)一个职业表达比所有可用组织下的另一个物质表达得多)两家家庭都是不同条件下的主导表达者,但一个职业仍然具有更高的平均表达。
在我们的研究中保留了3660对重复对,我们发现37对,其中两个基因在所有23个组织中没有发现表达。有274对,其中一个或另一个基因没有可检测的表达,但不是两者。一组930对(成对的25%)表现出交替的主导地位,其中两个基因至少有1种组织,它们表达比其职业高2倍。
剩余的保留重复对,265具有相关> 0.95。剩下的2154对不符合这些目标模式。这些对全部在至少一种组织中表达两种基因,并且不会表现出明显的优势,也没有具有高正相关性。有几种可能的场景可以描述这些基因对的功能。一种可能性是它们通过剂量补偿选择性地约束,该剂量补偿防止了一个或两个基因的分馏。另一种可能性是这些基因在功能上通过其表达模式清楚地揭示的方式在功能上发挥作用。进一步调查可用于区分这些病例。
使用蛋白质丰富数据进行类似的分析。我们发现8对,其中两个蛋白质在所有23个组织中没有检测到丰度。有133对的那种蛋白质有可检测的丰富,另一个蛋白质没有。成对的一组353(10%)表现出交替的主导地位,其中两个蛋白质至少有1种组织,其中它们比其职业高2倍。剩余的对中,98对具有相关> 0.95。其余3068对不符合这些模式。
对于1372个保留的重复对,23个组织中的至少1个中的表达和23个组织中的至少1个组织中的至少1个(在额外文件中称为“DATAcompete”中的至少1个2).这些成对总共122(9%)表现出交替的优势,其中两个基因至少有1种组织,它们表达比其职业高2倍,并且蛋白质丰度高于其职业。其余对的,50对的表达和丰度都有0.95。
功能注释意味着MAIZE1的更大多样性
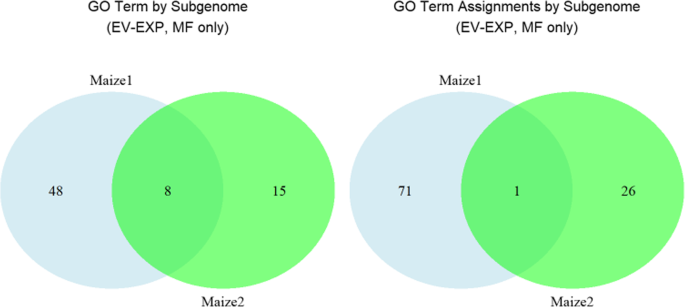
分配给MAIZE1和MAIZE2基因的GO注释的比较揭示了MAIZE1中的更多GO术语。在考虑到计算分配和手动策划的GO注释时,我们发现麦莉斯1和MAIZE2中的基因的1254个GO条款,MAIZE1具有599级,玉米1和MAIZE2具有158个独特的术语(参见附加文件3.).使用实验确定的证据代码手动分配基因的GO术语被认为是注释分配的黄金标准。仅比较这些亚基因组之间的金标准GO注释,我们发现Maize1和Maize2的基因都有8个GO项,而Maize1有48个独特项,Maize2有15个独特项,如图所示。4.(剩下)。考虑到每个转让给个体基因,与图26中所示的26相比,Maize1具有71个独特的任务。4.(对)。在MAIZE1和MAIZE2之间只重叠了1个转让。
玉米1基因比玉米2基因有更多的同型异构体
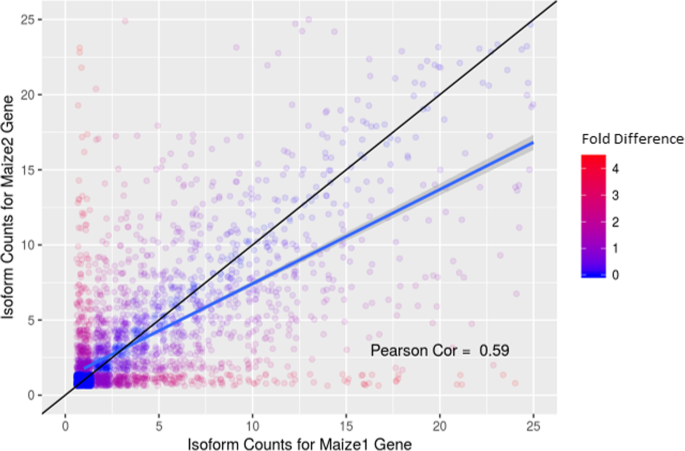
替代剪接已经与多功能基因有关,并改变基因产物的定位,因此独特同种型的增加应指示与基因相关的功能中的可能膨胀。我们比较了图2中的每个保留重复对中的两个家庭所预测的同种型数。5.。我们发现玉米1中的基因更容易具有比B73 Refgen V4组件提供的注释中的玉米2对应物的替代剪接形式更高。
有些反应是由Maize1和Maize2亚基因组唯一催化的
通过比较Maize1和Maize2的途径和反应关联,我们发现许多反应在CornCyc中只注释到一个亚基因组。有687个唯一的反应分配给Maize1, 255个唯一的反应分配给Maize2。如果我们只考虑保留的重复基因和与至少一个途径相关的反应,我们发现Maize2有更独特的分配反应(见附加文件)4.).玉米1亚血管催化在玉米2中不可用的副反应2,而玉米2催化在玉米中不可用的26个反应。尽管玉米2酶催化了较大的独特反应,但这些反应代表了较小的途径:玉米1基因参与其中14个途径,其中玉米2没有直接贡献,而MAIZE2仅涉及7个途径。
讨论
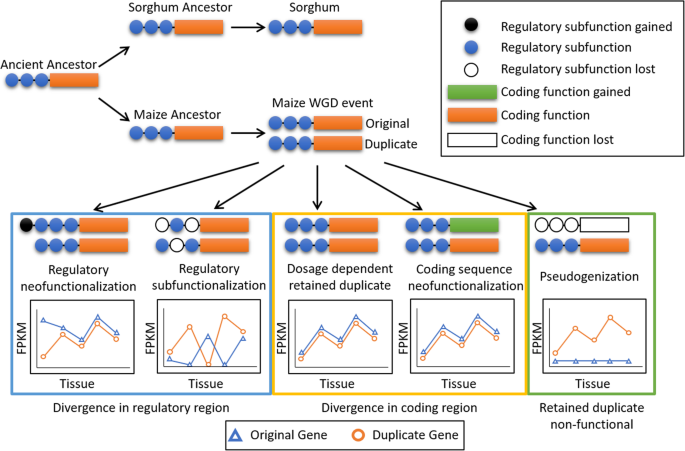
在一系列组织中观察玉米保留重复态的表达和丰度模式提供了对复制基因的进化分歧的见解。图2中描绘的表达式图案包括:3.建议进化压力如何改变转录调节。假设保留的同源基因最初是相同的,这允许我们将现代同源基因表达模式与[33.].基因上游区域的序列变化具有相对于其工业的基因的表达,包括调节子功能的增益或丧失,或者如图2所示。6.。不会预期编码区域的变化影响表达式模式。假置化表示如前所述的分馏的情况15.那36.].
保留基因的默认假设是其表达模式应该非常相似。事实上,这是许多假定的保留副本的情况下,共同表达到一个高度(基于相关性)和相似的水平。这也可能是这些类型的保留副本与高度相关的共同表达,但有一个基因在一个低得多的水平表达。在全基因组复制(WGD)事件后,发现剂量敏感基因以更高的速率被保留[37.].
不经常讨论的有趣情景是案例是不同组织中的主要保留重复开关。这些病例暗示了它们意味着1)两种基因表达并且可能具有功能性产品,2)其优势模式变化,因此它们可能在不同的监管控制下。虽然这并不排除两个基因具有相同的分子功能的可能性,但它强烈意味着它们在单独的进化压力下进化,因此更可能在功能上发散。
研究经常发现,当全基因组聚集时,基因表达测量和蛋白质丰度测量之间只有适度的相关性(通常在40%到50%之间)[18.那38.那39.那40那41.]。一些研究发现差异表达基因更大的相关性[42.].我们报告了基因和蛋白质丰富值之间的相关程度(Spearman相关= 0.549)。我们发现,当在所有组织中平均时,玉米含量比玉米含量较大。尽管在整个基因组中总体基因组在整体上具有检测到的非零表达,但是检测到的非零蛋白质丰富的预留复制品比基因表达更多。特别是,我们已经缩小了具有类似模式表达和丰富的小型基因列表,尽管我们发现发现没有可检测的表达的基因通常不能易于言,但没有可检测的蛋白质(参见附加文件2).
由于获得蛋白质丰富数据的难度,表达数据通常用于科学研究,这提出了表达数据是否与蛋白质丰富相关的问题。因为mRNA水平通常是瞬态的,因此情境敏感,并且可能不会影响由于翻译后调节导致的表型,蛋白质丰度水平通常是影响表型的基因的更可靠指标。实际上,我们的结果表明,使用蛋白质丰度数据实际上可以提供额外的见解,因为几乎没有检测到的表达的几个基因实际上是可检测的蛋白质丰富。
保留的重复项不仅在组织跨组织的表达和丰度模式中不等,它们也有不同的功能注释。我们报告说,计算分配的术语显示亚因素之间的大差异。由于两个家庭都保留重复,并且被发现具有相似的同义区域,我们预计它们的注释之间更大的重叠。部分原因可能是其他生物和序列相似性的同意/矫正性通常只是计算分配术语的标准之一。This result suggests either 1) the homeologs are not similar enough for the computational algorithms to apply the same homology transfer to both homeologs (i.e. they have diverged) or 2) that the computational methods used to perform homology transfer are not set up to handle cases involving retained duplicates (i.e. they enforce a 1-to-1 match in orthology) in which case these GO terms should be viewed skeptically, or 3) gene loss in one or both subgenomes has left the other subgenome with unique functions.
实验衍生的注释也有利于玉米1。虽然该结果可能正在捕获文献中的一些偏差,表明MAIZE1中的基因更可能被注释,而不是在面值上拍摄的玉米2,因为某些功能注释比其他功能更容易获得,这结果似乎似乎强化了玉米1和玉米2子组织在功能上发散的假设。
偏见分馏进化原因的一个工作假设是玉米整个基因组重复事件被认为是国内多元化的结果。它已经理解父母进入了一个Allopolyploid事件Brassica Rapa.[11.和其他物种的转座子负载水平不同,这可能也适用于玉米异源多倍体事件。因为TE沉默可以传播到基因区域[43.],它们可能导致基因表达中的耗尽,使得进入多倍体事件的单独谱系具有不同的全基因组RNA表达水平[44.].表达较低的亚基组在较少的选择压力下[45.,从而积累更多的删除。因此,分割程度较低的亚基因组(我们武断地将其分配给Maize1)有望成为表达水平较高的亚基因组。已知或预测的同源多倍体似乎没有显示出分块偏差的证据,这一事实支持了这一假设[46.].
结论
玉米保留重复率之间的表达和丰度模式的比较揭示了与新官能化框架一致的作业之间的功能分歧的证据,其中原始基因的功能保持不变,并且重复的基因通过中性突变获取新功能[33.].有趣的是,伪析化似乎并未成为偏差的主要形式,因为该对中的哪一种基因没有显示出表达,并且甚至更少的对该基因也没有任何蛋白质丰富证据。这表明玉米中的职业对继续提供一种选择性优势。组织特异性交替优势模式的患病率表明,这种优点与功能性分歧有关。手动和计算分配的GO术语注释中的差异可能反映了来自其祖先功能的一个或两个副本的分歧。
以前的研究发现,玉米1的大量基因平均高于其玉米2家族。利用当前版本的玉米参考基因组上的子宫内的更新定义,我们发现在广泛的组织上发现基因表达也显示出这种偏向致玉米1基因的更大表达。我们发现玉米1有更多的GO注释,但感到惊讶地认为玉米2预计催化比玉米1更具独特的反应。已经观察到的分馏偏差可能导致MAIZE2中更大的基因损失转化为与MAIZE1中的基因相比,MAIZE2基因将进化新功能的更大可能性。我们观察到保留重复态的蛋白质丰度值遵循类似于玉米子组织的组织特异性表达的模式。在考虑基因表达时,物质之间丰富模式的差异可以提供功能性分歧的支持证据。
方法
Zea Mays.基因组序列
这Zea Mays.基因组序列从(ftp://ftp.ensemblinomes.org/pub/release-37/plants/fasta/zea_mays/dna/zea_mays.agpv4.dna.toplevel.fa.gz.)和来自(ftp://ftp.ensemblinomes.org/pub/release-37/plants/gtf/zea_mays/zea_mays.agpv4.37.gtf.gz.).
RNASEQ表达和蛋白质丰富数据集
Walley等人描述的公开表达和蛋白质丰富数据集。[18.用于这个项目。该数据集包括Illumina Hiseq2500单链读数和23种组织收集的蛋白质丰度。短读文件从Genbank的序列读取存档(SRA)下载[47.],格式为Fastq [48.].使用MaizeGDB可用的映射文件将蛋白质数据从Refgen_v2投影到Refgen_v4。在病例是一种基因模型被认为分为两种或更多种新的基因模型,将refgen_v2基因模型的DNSAF值分配给每个refgen_v4基因模型。在认为两种或更多种基因模型合并的情况下,将每个Refgen_v2基因模型的DNSAF值的总和分配给Refgen_v4基因模型。(请参阅附加文件5.对于表达数据)。
将RNASEQ数据与REFGEN_V4对齐
表达数据使用Star对齐至B73 Refgen_V4组件[29.] 2.5.2b版本。FASTQC [49.]揭示了用Trimmomatic去除的适配器污染[50.]版本0.36使用以下参数:SE -phred33 Illuminaclip:Truseq3-Se.fa:2:30:10尾随:3 Minlen:36。使用以下参数对AGPV4.37基因模型引导的AGPv4序列进行索引步骤:--RunMode GenomeGenere -Sjdboverhang 100.默认默认所有其他参数。使用以下参数完成FASTQ格式读取的FASTQ格式的读取读取的读取:--RUNMODE OppletReads -QuantMode GeneCounts。默认默认所有其他参数。袖扣[30.] 2.2.1用于使用参数 - Library-Firststrand来定量FPKM中的基因表达。对所有组织的生物重复平均FPKM。Star报告了4%的读取映射到多个基因座。默认情况下,携带覆盖的袖扣均匀分布到均匀的所有位置都可以映射到的所有位置。请参阅附加文件6.和额外的文件7.获取详细信息。
数据和材料的可用性
本研究中使用的该软件和数据可供选择https://github.com/jrwalsh/subgenome.。
缩写
- DAP:
-
种植后的日子
- DNSAF:
-
分布归一化谱丰度因子
- FPKM:
-
每百万千兆比赛每千碱基
- 去:
-
基因本体论
- INDELs:
-
插入/删除
- Ks:
-
同义突变率
- RNA:
-
核糖核酸
- SNPS:
-
单核苷酸多态性
- SRA:
-
序列读取存档
- te:
-
可转换元素
- WGD:
-
全基因组重复
参考
- 1。
亚当斯KL,文德尔JF。植物的多倍体和基因组进化。植物学报。2005;8:135-41。
- 2。
亚当斯KL,克伦R,珀西菲尔德R,温德尔JF。通过多倍体复制的基因对转录组和器官特异性的相互沉默的贡献是不平等的。《美国国家科学院学报》上。2003; 100:4649-54。
- 3.
Dodsworth S,Chase MW,Leitch Ar。后期多倍化子系分交金对子植物的进化成功的关键吗?Bot J Linn Soc。2016; 180:1-5。
- 4.
Swigoňováz,莱j,ma j,ramakrishna w,llaca v,bennetzen jl等。近距离分裂高粱和玉米基因组祖细胞。Genome Res。2004; 14:1916-23。
- 5。
王志强,王志强,王志强,等。玉米基因组的四倍体起源。Comp funcgenomics . 2004; 5:281-4。
- 6。
Gaut Bs,Le Thierry D'Ennequin M,Peek As,Sawkins MC。玉米作为植物核基因组进化的模型。Proc Natl Acad Sci U S A. 2000; 97:7008-15。
- 7。
Woodhouse Mr,Schnable JC,Pedersen BS,Lyons E,Lisch D,Subramaniam S等人。在玉米中四倍体之后,短缺失机理从两个家业中的一个中除去了基因。Plos Biol。2010; 8:E1000409。
- 8。
Brohammer Ab,kono tjy,springer nm,mcgaugh se,hirsch cn。差异分馏在玉米基因组含量变化和功能中的有限作用(Zea Mays.L.)自交系。工厂J. 2018; 93:131-41。
- 9。
梁Z,Schnable JC。全基因组重复后亚因子和基因对之间的功能分歧。莫尔植物。2018; 11:388-97。
- 10。
Blanc G,Wolfe Kh。拟南芥进化期间多倍体形成的重复基因的功能分歧。植物细胞。2004; 16:1679-91。
- 11.
王雪峰,陈峰,王学军。多倍体基因组显性基因的起源、遗传和调控。《美国国家科学院学报》上。2014; 111:5283-8。
- 12.
Schnable JC,由可见突变体表型鉴定的Freeling M.基因显示出玉米两种亚因子中的一个增加的偏差。Plos一个。2011; 6:E17855。
- 13。
Forestan C,Farinati S,Aiese Cigliano R,Leanardon A,Sanseverino W,Varotto S.玉米RNA PLIV影响附近TE插入的基因的表达,并对转录产生基因组抑制作用。BMC植物BIOL。2017; 17:161。
- 14。
Moore Rc,Purugganan MD。植物重复基因的进化动态。CurrOp植物BIOL。2005; 8:122-8。
- 15.
Schnable JC,Springer NM,Freeling M.通过基因组优势和古老和持续的基因丢失来分化玉米子组织。PROC NATL ACAD SCI U S A. 2011; 108:4069-74。
- 16。
Pophly SD,Tellier A.人口水平纯化选择和基因表达形状玉米群进化。mol Biol Evol。2015; 32:3226-35。
- 17。
焦友,佩鲁佐P,Shi J,Liang T,Stitzer Mc,Wang B等。用单分子技术改善玉米参考基因组。自然。2017年;前进在线出版物。https://doi.org/10.1038/nature22971。
- 18。
Walley JW,Sartor Rc,Shen Z,Schmitz RJ,Wu KJ,Urich Ma,等。OMIC网络在玉米发展地图中的集成。科学。2016; 353:814-8。
- 19。
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM,等。基因本体论:生物学统一的工具。基因本体论联盟。Nat麝猫。2000;25:25-9。
- 20。
基因本体论联盟。扩大基因本体知识库和资源。核酸RES。2017; 45:D331-8。
- 21。
Monaco Mk,Sen Tz,Dharmawardhana Pd,Ren L,Schaeffer M,Naithani S等人。玉米代谢网络施工和转录组分析。植物基因组。2013; 6:0。
- 22。
TU. UniProt:蛋白质信息中心。核酸学报2015;43:D204-12。
- 23。
Corncyc 8.0 |植物代谢网络。https://www.plantcyc.org/databases/corncyc/8.0.。访问2017年12月18日。
- 24。
Andorf CM, Cannon EK, Portwood JL, Gardiner JM, Harper LC, Schaeffer ML,等。MaizeGDB更新:玉米模型生物数据库的新工具、数据和接口。核酸Res. 2016;44: D1195-201。
- 25。
非模型基因组的价值和在CoGe中使用SynMap来解剖蔷花籽之前的六倍体的一个例子。热带植物学报。2008;1:181-90。
- 26。
Mckain Mr,Estep Mc,Pasquet R,Layton dj,DíazDMV,Zhong J等人。玉米两种亚因素的祖先。Biorxiv,Vol。352351;2018年。
- 27。
Lawrie DS, Messer PW, Hershberg R, Petrov DA。D melanogaster同义位点的强纯化选择。公共科学图书馆麝猫。2013;9:e1003527。
- 28。
植物密码子偏倚决定因素相互作用的新认识。DNA研究》2015;22:461 - 70。
- 29。
Dobin A,Davis Ca,Schlesinger F,Drenkow J,Zaleski C,JHA S等人。星:超快通用RNA-SEQ对齐器。生物信息学。2013; 29:15-21。
- 30。
Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR,等。TopHat和袖扣RNA-seq实验的差异基因和转录本表达分析。Nat Protoc。2012;7:562。
- 31。
张Y,Wen Z,Washburn MP,Florens L. Refinements标记免费蛋白质组定量:如何处理多种蛋白质共享的肽。肛门化学。2010; 82:2272-81。
- 32。
王B,Condski M,Tseng E,Olson A,Goodwin S,McCombie WR,等。单分子测序获得的玉米和高粱的比较转录景观。Genome Res。2018年。https://doi.org/10.1101/gr.227462.117。
- 33。
Innan H,Kondrashov F.基因重复的演变:模型之间的分类和区分。NAT Rev Genet。2010; 11:97-108。
- 34。
强制A,Lynch M,Pickett FB,Amores A,Yan Y1,Mostlethwait J.通过互补,退行性突变保存重复基因。遗传学。1999; 151:1531-45。
- 35。
Ohno S.基因复制的进化。柏林,海德堡:施普林格柏林海德堡;1970.https://doi.org/10.1007/978-3-642-86659-3。
- 36。
Freeling M,Scanlon MJ,Fowler Je。基因组重复后分馏和子官能化:驱动基因含量的机制及其后果。CurrOpin Genet Dev。2015; 35:110-8。
- 37。
Abrouk M,张R,Murat F,Li A,Pont C,Mao L等。Grass MicroRNA Gene Paleohistory在全基因组重复后亚基组分区中的基因剂量平衡推出新的洞察。植物细胞。2012; 24:1776-92。
- 38。
Guimaraes JC,Rocha M,Arkin AP。大肠杆菌蛋白质丰富和噪声的转录水平和序列决定因素。核酸RES。2014; 42:4791-9。
- 39。
Vogel C,Marcotte Em。从蛋白质组学和转录组分析中欣赏蛋白质丰富的洞察。NAT Rev Genet。2012; 13:227-32。
- 40。
Ponnala L,Wang Y,Sun Q,Van Wijk KJ。MRNA和蛋白质丰度在玉米叶中的相关性。工厂J. 2014; 78:424-40。
- 41。
吴克杰,沈志强,王志强,等。从玉米种子蛋白质型图谱重建蛋白质网络。《美国国家科学院学报》上。2013; 110: E4808-17。
- 42。
Koussounadis A,Langdon Sp,Um Ih,Harrison DJ,Smith VA。异种移植模型体系中差异表达mRNA和mRNA蛋白质相关性的关系。SCI批准。2015; 5。https://doi.org/10.1038/srep10775。
- 43。
霍利斯特博士,Smith LM,郭永林,Ott F, Weigel D, Gaut BS。转座元件和小rna导致拟南芥和拟南芥基因表达差异。《美国国家科学院学报》上。2011; 108:2322-7。
- 44。
鞭毛L,UDALL J,Nettleton D,Wendel J.在AllopolyPloid Gossypiumreveals中的重复基因表达式表达演化的两个时间逐个不同阶段。BMC BIOL。2008; 6:16。
- 45。
Renny-Byfield S,Rodgers-Melnick E,Ross-Ibarra J.基因分级和玉米古代亚因子的功能。mol Biol Evol。2017; 34:1825-32。
- 46。
Zhao M,张B,Lisch D,Ma J.亚血管差异的模式和后果提供了植物古聚倍性本质的见解。植物细胞。2017; 29:2974-94。
- 47。
Leinonen R, Sugawara H, Shumway M.序列读取档案。核酸Res. 2011;39(数据库版):D19-21。
- 48。
Cock PJA, Fields CJ, Goto N, Heuer ML, Rice PM。sanger FASTQ文件格式的序列与质量分数,和Solexa/Illumina FASTQ变体。核酸学报2010;38:1767-71。
- 49。
Andrews S. Babraham BioInformatics - FASTQC用于高吞吐量序列数据的质量控制工具。https://www.bioinformatics.babraham.ac.uk/projects/fastqc/. .
- 50.
Bolger Am,Lohse M,Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014; 30:2114-20。
致谢
在CERES计算群集中执行RNASEQ映射,作为由USDA-AR提供的Scinet资源的一部分。特别感谢在MaizeGdb维护的愈合Go注释(Ethalinda Cannon,Mary Schaeffer,Brittney Dunfee,Mandy Dietze,Miranda Huerta)。John Portwood创建了B73 Refgen_V4对齐表达式数据的Gbrowse曲目。USDA是一个平等的机会提供者和雇主。
资金
工资和开放使用费用由美国农业部-农业研究服务CRIS项目5030-21000-068-00-D和2030-21000-024-00-D支付。资助机构在研究的设计、数据的收集、分析和解释或手稿的撰写中没有发挥作用。
作者信息
隶属关系
贡献
JRW,TZS和CMA,构思,设计和协调项目。JRW开发并记录了代码,执行了分析,并起草了稿件。MRW有助于分析。所有作者均致力于稿件写作流程并批准了最终手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
提交人声明他们没有竞争利益。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
沃尔什,J.R.,Woodhouse,M.R.,Andorf,C.M.et al。在玉米中,组织特异性基因表达和蛋白质丰度模式与分化偏倚相关。BMC植物BIOL.20,4(2020)。https://doi.org/10.1186/s12870-019-2218-8
已收到:
公认:
发表:
关键字
- 亚群
- 基因表达
- 蛋白质丰富
- 玉米
- 功能分歧