摘要
背景
锌指A20/AN1基因家族编码应激相关蛋白的转录因子(SAP的描述很好拟南芥还有米饭,包括14AtSAP和18OsSAP与多种非生物胁迫的可变耐受性相关的基因。的SAP基因家族在结构上表现出很大的多样性,在不同的植物种类中也表现出很大的多样性。这项研究的目的是确定所有HvSAP大麦基因(大麦芽L.),分析选择的基因在大麦叶片中对盐度响应的表达,并开发SNP标记HvSAP12通过田间试验评价大麦植物基因型与籽粒产量的关系。
结果
在我们的研究中,17HvSAP在大麦中发现了与水稻基因高度同源的基因。五个基因,HvSAP5,HvSAP6,HvSAP11,HvSAP12而且HvSAP15半定量RT-PCR和qPCR分析结果显示,与对照相比,在水培条件下大麦植株叶片中对盐胁迫响应高表达。开发了类amplifluor SNP标记KATU-B30,并用于HvSAP12基因分型。强烈的联想(R2KATU-B30与50 F单株产量之间存在= 0.85的差异3.在哈萨克斯坦北部和中部干旱和低至中等盐度的田间试验中,育种系源于granar × Baisheshek杂交。
结论
一群HvSAP基因,HvSAP12特别是在大麦植物耐盐耐旱方面起着重要作用,在田间试验中与较高的籽粒产量相关。利用SNP标记KATU-B30进行标记辅助选择,可以在非生物胁迫条件下提高大麦产量。
背景
大量的应力相关蛋白(SAP)是由含有两个保守结构域A20/AN1的锌指蛋白编码的。在人类中,这些基因发挥肿瘤坏死因子(TNF)的作用[1但在植物中,SAP基因在不同类型的非生物和生物胁迫中扮演着重要的角色,它们是众所周知的植物免疫反应的一部分(综述在[2])。广泛存在于植物物种中,SAP基因在结构上非常保守,但功能却非常多样化[3.].首次在水稻中发现[4), 18OsSAP基因显示与14有很强的相似性AtSAP基因的拟南芥[5].SAP其他植物的基因也被研究过,比如玉米[3.,西红柿[6),草季而且羊草[7,8,小麦[9,以及棉花[10李子和苹果[11,12),芸苔属植物显著而且Medicago truncatula[13,14].然而,大麦(大麦芽)是一种重要的谷类作物,代表了我们在这方面知识的空白SAP基因尚未被描述和研究。
和人类一样,SAP植物中的基因通过膜结合受体激酶参与病原体识别过程[15],以及对抗细菌和真菌感染的防御机制,正如最近番茄的例子所示SlSAP3而且SlSAP4沉默和过度表达[16,17].然而,更强烈的反应SAP在植物中发现了应对各种非生物胁迫的基因,包括干旱、盐碱、寒冷和其他(综述在[2])。
不论植物种类,不同SAP基因对非生物胁迫有特定的表达谱。所有18个OsSAP水稻和13个基因被鉴定出来SlSAP在广泛的非生物胁迫中,番茄的基因对一种或多种胁迫都有反应。5,6].AtSAP10被报道强烈参与了拟南芥植物对重金属和高温的反应[18].对于盐度,大多数SAP水稻的基因,拟南芥和番茄在苗期对NaCl有响应[5,6,而37人中的33人GhSAP基因在一个月大的棉花植株暴露于强盐胁迫(300 mM NaCl) 2 h后表达[10].盐盐:同样高浓度的盐作用于Medicago truncatula2-12小时导致4MtSAP基因和其他五个基因的下调[14].多年生草的一个月大的植物,羊草,暴露于更高浓度的NaCl (400 mM) 6-24 h,和转录水平LcSAP持续增加[8].
据报道,干旱会引发强烈的表达MdSAP15,MdSAP25而且MdSAP29苹果基因[12但是,相反地,OsSAP7基因在水稻幼苗中下调[19].的表达FaSAP的最接近的同系词OsSAP8)在草类中,季,PpSAP1在李子(碧桃)也在盐碱化或干旱条件下植株中高度上调[7,11].显示出类似趋势的是,2SAP基因(BnaA03g47350D而且BnaC07g39590D),对应于AtSAP10而且OsSAP3/OsSAP5用盐诱导和PEG脱水处理芸苔属植物显著由RNA-Seq技术测定[13].
表达的AtSAP10,OsSAP7而且MdSAP15的最接近的同源词OsSAP4/OsSAP8)转基因拟南芥分别对提高植物对重金属和干旱的耐受性非常有益[12,18,19].转基因烟草植物过表达LmSAP从装饰性分析来看,Lobularia maritima,该基因高表达,其中A20和AN1结构域分别专门参与盐/渗透和氧化应激反应[20.].在转基因小麦和过表达水稻中SAP研究发现,在不同非生物胁迫下,水稻籽粒产量较高或无净效应(见[2])。
变化SAP对非生物胁迫的基因表达在各种植物物种中有很好的记录(在[综述]2]),但关于这种表达与单倍型、突变或遗传多态性相关的信息记录甚少。在这种情况下,可以设计分子标记来指示单倍型和特定表达之间的强或弱关联SAP基因。例如,据报道,三个分子标记围绕TaSAP1-A1在干旱和水充足条件下生长的300个面包小麦品种中,基因和农艺上重要的性状如千粒重、每穗粒数、穗长、花序梗长和每穗总小穗数[9].在众多的分子标记中,SNP(单核苷酸多态性)是非常受欢迎的,最近开发的类amplifluor SNP标记[21]被选择并应用于当前的研究,从非常不同的SNP检测方法中选择(在[22])。
除了自然变异外,杂交群体及其后代也可用于优选单倍型的遗传分析SAP基因的分离模式和基因遗传类型。关联分析SAP遗传背景和植物在逆境环境中的表现可以提供直接的信息支持基因的作用和功能SAP基因。
大麦(大麦芽L.)是一种二倍体植物,属于草科和小荞科。大麦单倍体基因组大小约5.3 Gb,有7条染色体,是迄今为止最近测序的最大的二倍体物种基因组之一[23].大麦植株对几种非生物胁迫表现出一定的耐受性,可以在相对恶劣和不利的环境中生长。然而,如果能更好地理解大麦对干旱和盐等非生物胁迫的耐受机制,就可以提高大麦的产量。24].在大麦植物非常复杂的基因调节网络系统中,HvSAP基因在对抗非生物胁迫的协调反应和信号系统中发挥着潜在的重要作用。
在目前的研究中,在一个国际合作项目的框架内,如资助部分所示,使用了来自哈萨克斯坦的当地大麦品种和一个产生的杂交品种。哈萨克斯坦有这么大的土地可供农田作物种植,严重干旱和高盐度是一些地区非常典型的现象。动物饲料对大麦种子的需求很大,因此,更好地了解大麦植物对非生物胁迫耐受性的遗传机制不仅可以帮助当地农业企业,而且对许多国家研究大麦和其他作物的研究人员和育种人员也有好处。
本研究的目的是:(1)确定一个全面的HvSAP大麦基因组中存在的基因并与水稻的参考基因组进行比较(2)研究和选择HvSAP半定量和qRT-PCR分析对盐度最敏感的基因;(3)寻找天然多态性HvSAP12在大麦杂交种的亲本中;(4)开发用于杂交植物基因分型和标记辅助选择的SNP标记;(5)评价在哈萨克斯坦北部和中部强干旱和中低盐环境下,所选杂交基因型及其籽粒产量性状之间的相关性。
结果
的识别HvSAP使用生物信息学方法的候选基因
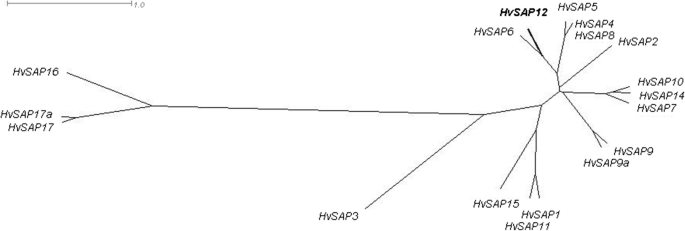
在大麦数据库中筛选snp时(http://bioinf.scri.ac.uk/barley_snpdb), SNP ID: ABC08579被发现是一个潜在的候选基因。水稻亲缘蛋白AAP37480的注释,推测A20/ an1型锌指蛋白栽培稻、组粳稻是表示。利用IPK技术寻找大麦同源基因https://webblast.ipk-gatersleben.de/barley_ibsc/viroblast.php)和NCBI数据库(https://www.ncbi.nlm.nih.gov)的研究表明,该SNP存在于HvSAP12编码应激相关蛋白的基因。和大米类似,HvSAP12属于转录因子A20/ an1型锌指蛋白群。从IPK和NCBI数据库中分别鉴定了大麦基因HORVU2Hr1G053670和AK363382HvSAP12和17岁。HvSAP在大麦基因组中共发现了与水稻基因高度同源的基因1).大麦基因组中的小重排很明显,其中两个HvSAP基因被复制(HvSAP9a而且HvSAP17a),但其他三个基因(HvSAP8,HvSAP13而且HvSAP18)比18人少OsSAP水稻的基因[5].
17人的分子系统发育树HvSAP基因(图。1)的基因核苷酸序列与推导出的蛋白质氨基酸序列的顺序和模式一致。分析显示两者之间的分子相似性最接近HvSAP12和HvSAP6,与的相似度相对较近HvSAP5而且HvSAP4 / HvSAP8.HvSAP3被孤立,但仍然比最孤立的群体更接近HvSAP16和配对HvSAP17而且HvSAP17a基因(图。1).
17株已鉴定的分子系统发育树HvSAP基于大麦基因组序列的基因。用SplitsTree4 (http://www.splitstree.org).的位置HvSAP12带有描述SNP的基因用粗体表示
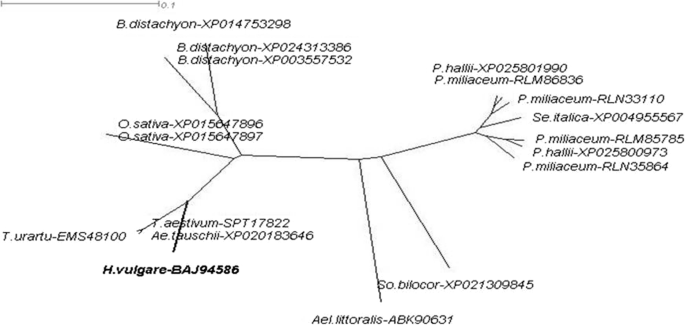
为了更好地确定编码蛋白HvSAP12(登录号BAJ94586)与其他单子叶植物SAP蛋白序列之间的相似性,基于NCBI数据库(https://www.ncbi.nlm.nih.gov)(图。2).
HvSAP12蛋白的分子系统发育树(以粗体显示)。其他单子叶植物的同源蛋白序列可在NCBI数据库中找到。用SplitsTree4 (http://www.splitstree.org).这些物种的植物学名称(按顺时针顺序排列)如下:大麦芽,小麦属植物urartu,山羊草属tauschii,小麦,栽培稻,Brachypodium distachyon,黍halli,黍miliaceum,Setaria italica,高粱二色的而且Aeluropus littoralis.在物种名称后添加相应的蛋白质序列登录id
分析HvSAP半定量RT-PCR表达
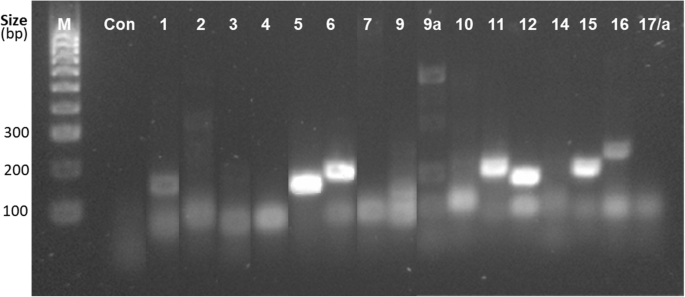
半定量RT-PCR分析17例HvSAP从胁迫植物中提取的大量cDNA样本显示了5个高表达基因:HvSAP5,HvSAP6,HvSAP11,HvSAP12而且HvSAP15(无花果。3.).两个基因,HvSAP1而且HvSAP16的表达水平较低,但仍清晰,而其余的HvSAP基因没有表达或表达很差,包括多条带HvSAP9a基因。选择5个高表达基因进行进一步研究。
表达分析的五种选择HvSAP基因
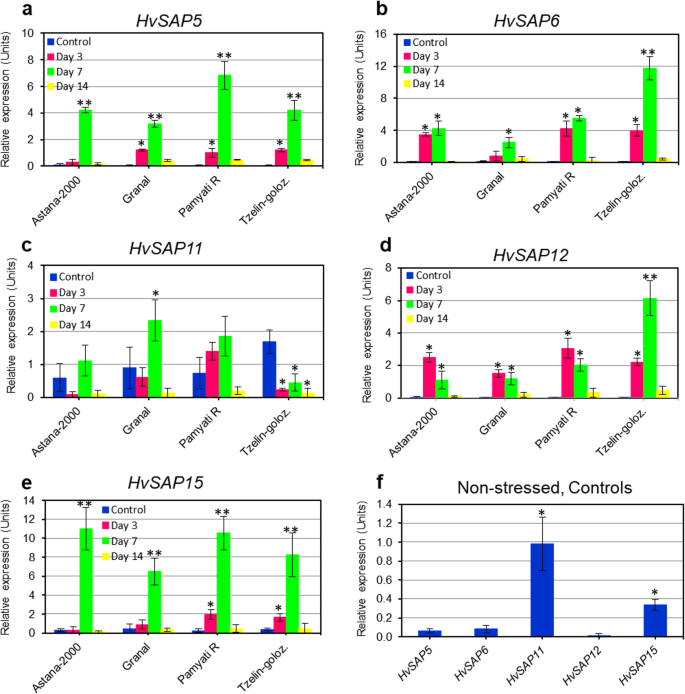
五分之四HvSAP在暴露于盐胁迫3天后,特别是7天后,被检测的基因表达相对较高或非常高(图1)。4).转录水平增长最快的是HvSAP5(7倍),HvSAP6(12),HvSAP12(3)HvSAP15),“碳足迹”(11倍.两个基因,HvSAP6而且HvSAP12(无花果。4b和d),在第3天和第7天,4个品种中有3个的表达量相似,而HvSAP5而且HvSAP15(无花果。4a和e)在第7天均显著升高。在对照(非胁迫)植株中,这4个基因的表达量从非常低(0.35相对表达单位)不等HvSAP15)到极低的HvSAP5,HvSAP6而且HvSAP12(无花果。4f).只有一个基因,HvSAP11(无花果。4C),与其他四种被研究的基因表达谱非常不同HvSAP基因。首先,初始水平HvSAP114个研究品种的表达量在0.5 ~ 1.7个相对表达单位之间变化。4C),显著高于其他四组的记录HvSAP基因(图。4f).盐胁迫下,表达HvSAP11在试验第7天,Granal显著(2.5倍)升高,而Tzelinniy golozerniy在整个试验期间(3 ~ 14 d)均下调(图4)。4c).现在就研究的4个大麦品种在盐和干旱影响环境下的产量与5个研究品种的表达谱之间的相关性得出任何结论还为时过早HvSAP基因。
小麦分离群体Granal × Baisheshek的基因分型HvSAP12使用Amplifluor SNP标记的基因
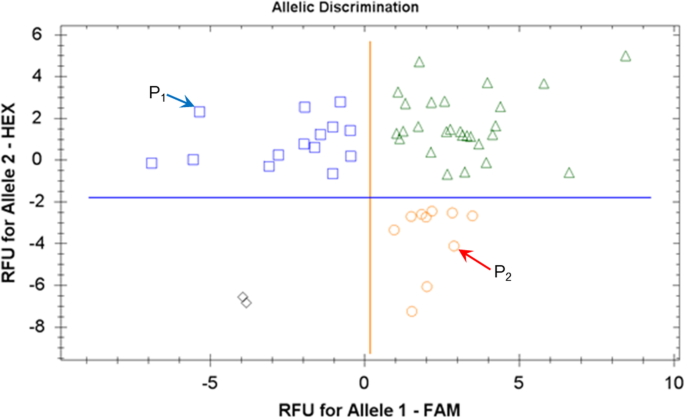
在该基因的3 ' -UTR区发现了一个明确的SNPHvSAP12F .基因的亲本Granal和Baisheshek3.隔离人群(附加文件1图S1)。在此基础上,开发了类似于amplifluor的SNP标记KATU-B30,并用于对50个F3.这种杂交的后代使用KATU-B30 SNP标记的基因分型结果显示,与亲本相同的等位基因具有良好的识别力(图1)。5).基因分型结果HvSAP12用于与植物表型的进一步比较。
F3.选育系来自分离居群Granal × Baisheshek
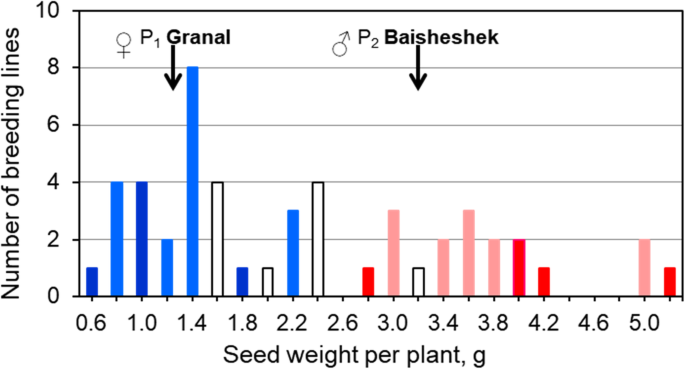
在干旱和中低盐胁迫的田间试验中,对50个小麦品种的产量进行了表型分析3.后代呈连续分布(图。6).然而,KATU-B30 SNP标记得分不同的基因型组在最低和最高的籽粒产量(每株分别< 2.6和> 2.6 g)之间有明显的区别,并且没有重叠(图5)。6).产量最低和最高的两组选育系包括纯合子(图中分别用深蓝色和红色表示)。6)和杂合子的混合物(显示为浅蓝色和粉红色)。
等位基因的HvSAP12,源自母系亲本Granal,图中为蓝色方格。5图中深蓝色和浅蓝色柱状图显示,与产量较低的育种系相关。6.分别HvSAP12来自父本Baisheshek的等位基因用红圈表示(图1)。5)以及单株籽粒产量较高的基因型的红条和粉条(图。6).一个非常强烈的联想(R2= 0.85)HvSAP12基因分型(无花果。5)和粮食产量表型(图。6).
讨论
一个庞大的家族HvSAP在对照(非胁迫)和NaCl处理的水培试验以及中低盐干燥环境下的田间试验中,首次鉴定了这些基因的表达特征。
有关的公开资料很少SAP在良好条件下生长的不同植物的基因表达。在番茄,SlSAP基因表现出更强的相似性拟南芥比单子叶植物种;尽管如此,番茄和大麦的比较HvSAP成绩单值得注意。非常高的表达SlSAP1而且SlSAP10在没有任何胁迫的9日龄番茄幼苗中发现[6].这些基因与OsSAP1,OsSAP11而且OsSAP15在大米中,这与我们的结果完全吻合HvSAP11而且HvSAP15,表明在非胁迫大麦植株中转录水平最高(图。4f).我们研究中发现的另外两个大麦基因,HvSAP5而且HvSAP6的研究表明,在有利条件下生长的植物中,基因表达水平较小,但仍可识别。因此,我们的结果与番茄相似,其中SlSAP4,SlSAP5而且SlSAP9聚集在系统发育树的同一分支中[6].
在我们的研究中,17个中有5个被识别了HvSAP在盐胁迫下,基因表现出高表达谱HvSAP基因没有或转录水平很低。作为对比,在水稻中,所有5个同源基因在盐度胁迫下都上调了表达[5].最高水平的表达增加了3.5倍OsSAP5而且OsSAP12,其后是OsSAP6而且OsSAP11(三倍)OsSAP15(1.5倍)。总的来说,我们的结果是HvSAP基因显示出与水稻的相似之处,除了这一点HvSAP15相比OsSAP15转录本在大麦和水稻中的表达量分别很高(6 - 11倍)和相对较低(1.5倍)。
然而,在其他的比较中,更多的是相互矛盾的观察OsSAP而且HvSAP基因。三个基因,OsSAP7,OsSAP10而且OsSAP14,盐胁迫下水稻幼苗上调5.5-、9-和10倍[5],而同源大麦基因(HvSAP7,HvSAP10而且HvSAP14)在半定量RT-PCR中未发现增体cDNA扩增,因此被排除在进一步研究之外。同源基因表达差异如此显著,可能有几个原因。首先,我们的研究中使用了4个大麦品种,与Vij和Tyagi发表的单一水稻品种相比,这明显更具代表性[5].在该试验中,水稻幼苗在涂有湿棉的托盘中生长到7天,而我们的试验中大麦幼苗在3周大,用生长液水培。水稻幼苗暴露在200 mM NaCl中很短的时间(6 h),而我们的实验中大麦植株的叶片是在3、7和14天后取样的,作为150 mM NaCl的长期盐度处理。
然而,从我们的角度来看,大米实验的主要区别是[5我们用大麦做的实验是方法论上的,这可以极大地改变对结果的解释。尽管没有明文规定,但我们可以假设水稻幼苗简单地从湿托盘转移到含有200 mM NaCl的容器中,并暴露6小时[5].植物从非胁迫条件下突然(一步)转移到200 mM NaCl溶液中可能会引起“渗透冲击”和细胞浆解,原生质体从细胞壁分离,特别是在根系中。因此,报告的基因表达(包括SAP基因)可能是“盐冲击”的结果,而不是“盐胁迫”的结果,如前所述,随着NaCl的逐步(分几个步骤)施用而发生[25].在我们对大麦植物的实验中,五个基因的高转录水平(HvSAP5,HvSAP6,HvSAP11,HvSAP12而且HvSAP15)在NaCl浓度逐渐升高的生长溶液中发现,这些基因确实对盐胁迫有响应。相比之下,在水稻中报道的三种基因,OsSAP7,OsSAP10而且OsSAP14[5],可能是对强渗透/盐冲击而不是盐胁迫的反应,因此这些结果应该与NaCl的施用方法有关。
的比较也出现了类似的情况HvSAP大麦基因表达SlSAP9日龄番茄幼苗基因转入200 mM NaCl培养基1 h和8 h [6].所有五个HvSAP对逐渐盐胁迫响应的基因与番茄中相应的基因具有相似性,而包括SlSAP6,SlSAP12而且SlSAP13基因表达可能与盐休克反应有关。
一般来说,绝对表达水平并不总是基因功能重要性的可靠指标。然而,研究过的HvSAP基因对盐胁迫的响应表现为高表达或低表达(转录量或多或少)。进一步的研究可以证明这些候选基因中哪一个在植物对胁迫反应的功能上更重要。然而,研究中记录了对盐度反应的低表达和少量转录的基因似乎不太可能在植物的耐受性中有更重要的功能作用。
耐盐和耐脱水有一个共同的反应成分——细胞的渗透调节。因此,也就不足为奇了SAP在离体(脱水)水稻和番茄叶片中,基因在响应盐度或快速脱水时表现出类似的共表达趋势[5,6].在自然环境中,干旱和盐碱也经常相伴而生。在这方面,大麦中表达较高的基因之一,HvSAP12,似乎与植物对干旱和盐的同步反应有关,特别是在田间试验中。在文献中,只有一项关于小麦的研究,报告了一种单倍型与有益的等位基因的强烈关联TaSAP1具有重要的农艺性状,如在干旱和水充足条件下的粒重、穗生产力成分和单株粒产量[9].因此,我们利用来自F3.一个G × B杂交的子代,在KATU-B30 SNP标记之间HvSAP12在干旱和盐影响的田间试验中,它们的单株籽粒产量表型相似,现在补充了Chang等人对面包小麦的研究结果。[9].
然而,在我们的研究中,用于KATU-B30 SNP标记的等位基因区分的簇是相当松散的,这是自开发的Amplifluor-like SNP标记的典型。然而,qPCR仪软件自动测定的等位基因需要人工验证,并与亲本基因型进行比较,以确保结果的置信度。
基于成熟的和经过实验验证的功能分子标记的标记辅助选择可以有效地选择生长在干旱环境中的面包小麦植株的最佳基因型[9]和在干旱和盐胁迫下的大麦植株(目前的研究)。因此,应用这里开发的像KATU-B30 SNP这样的分子标记,可以帮助加快大麦育种过程,在哈萨克斯坦严酷的胁迫影响环境下提高粮食产量,并有可能在许多其他国家。
结论
大麦是17HvSAP在我们的研究中发现了编码应激相关蛋白的基因。五个基因,HvSAP5,HvSAP6,HvSAP11,HvSAP12而且HvSAP15半定量RT-PCR和qPCR分析结果显示,与对照相比,在水培条件下大麦植株叶片中对盐胁迫响应高表达。SNP标记KATU-B30用于HvSAP12.这个和那个HvSAP基因在大麦植物的耐盐性和耐旱性中起着明确的作用,并在田间试验中与更高的谷物产量有关。利用SNP标记KATU-B30进行标记辅助选择,可以在非生物胁迫条件下提高大麦产量。
材料与方法
植物材料
本研究采用哈萨克斯坦为动物饲料生产开发的4个大麦品种:哈萨克斯坦高产、耐干旱、耐盐的标准大麦品种Astana-2000;Granal,具有优良籽粒品质的分离群体的亲本[26]对非生物胁迫相对敏感;哈萨克斯坦高产耐寒大麦饲料优良品种Pamyati Raisi以及Tzelinniy golozerniy,一种无壳或“裸籽”的当地品种,在干旱和盐碱条件下产量更少。根据之前的结果,F2杂交种由大麦品种Granal和Baisheshes(干旱高产)杂交而成,采用人工去雄、分离和受控授粉的方法,由Grigory Sereda(哈萨克斯坦卡拉干达地区Khristenko Karaganda农业实验站)提供。大麦品种的种子和繁殖的F2分离的种群是由G. Sereda在国际合作研究项目框架下提供的。应研究人员的要求,可以提供少量种子。
水培中的盐胁迫和叶片样品的采集
种子在培养皿中的湿滤纸上在室温下发芽5天,然后使用所述方法将幼苗转移到水培装置中[27],但须作以下修改。四个容量为12升的桶盖上了直径1厘米的孔。在转移的幼苗中间轻轻包裹一个泡沫片作为支撑,使植物的根悬浮在生长溶液中固定在孔中。介质的恒定曝气由水族泵提供。进一步的细节,包括生长溶液的组成和有生长植物的水培装置的图像,见[中的图5a和图b。28])。在整个实验过程中,水培溶液的pH值保持在接近中性的范围内(pH = 6.5-7.0),这是通过pH计(Activon, Model 20, Adelab Scientific, Australia)定期监测确认的,无需进一步调整。生长液每天加满水,每10天完全更换一次。当植株长到三周大时,将NaCl添加到两个盒子(指定为“盐胁迫”)中,每天两次,连续三天增加25 mM NaCl,使最终浓度达到150 mM NaCl,以避免突然的盐冲击[25].补充CaCl2与初始生长溶液(0.98 mM Ca2 +) [29].在不添加NaCl和氯化钙的情况下,与对照植株生长一致2.
将三株植物的所有叶片分别放入10毫升塑料管中,分别在施盐后第0、3、7和14天对每种基因型、处理和收集时间点进行3次独立的生物重复。装有叶片样本的试管立即在液氮中冷冻,并保持在−80°C,直到RNA提取。
现场试验
2017年和2018年在干旱条件下,在哈萨克斯坦北部努尔苏丹S. Seifullin哈萨克斯坦农业技术大学和哈萨克斯坦中部卡拉干达地区A.F.Khristenko Karaganda农业实验站的研究领域进行了田间试验。在湿润土壤中播种后,土壤含盐量极低(EC = 1 ~ 3 dS/m),但在干旱生长季逐渐增加,直至达到温和水平(EC = 4 ~ 6 dS/m)。营养生长期的总降雨量为107-130毫米,低于这些地区过去多年观测到的150-166毫米的平均降雨量。播种两行,1米长,每行植株间5厘米,行与行之间20厘米。在两年的试验中,每个田间试验使用4个随机重复。测量每个地块收获的所有作物的籽粒总产量,并重新计算为每株克。
利用生物信息学和分子系统发育比较分析鉴定“感兴趣基因”
大麦SNP数据库(http://bioinf.scri.ac.uk/barley_snpdb)用于搜索和选择单个目标基因或“感兴趣的基因”进行进一步研究。利用NCBI数据库中大麦的核苷酸集合,对含有SNP的基因片段进行BLAST分析,确定其全长靶基因(https://blast.ncbi.nlm.nih.gov).
采用生物信息学的方法对其全核苷酸序列进行了鉴定HvSAP,其对应的多肽序列用于NCBI和IPK大麦BLAST Server的BLASTN和BLASP (https://webblast.ipk-gatersleben.de/barley_ibsc/viroblast.php).利用水稻基因注释网站(http://rice.plantbiology.msu.edu/annotation.shtml).所有的染色体位置HvSAP大麦基因组中的基因是通过植物基因组和系统生物学,大麦项目的网站(http://pgsb.helmholtz-muenchen.de/plant/barley/fpc/searchjsp/index.jsp),并与IPK大麦爆炸服务器进行了检查。
中两个核苷酸序列的分子树状图SAP利用SplitsTree4程序构建了大麦和其他单子叶植物SAP多肽的基因和氨基酸序列(http://www.splitstree.org) [30.].采用无根共识树算法和等角树状图算法进行两种方法的优选SAP基因和SAP多肽系统发育树。
RNA提取,cDNA合成,半定量PCR和qPCR分析
每个收集时间点从每个水培盆(盐胁迫和对照)中选择每个品种的3株单株。冷冻的叶子样本被研磨在10毫升的管子和两个9毫米的不锈钢球轴承使用涡流搅拌机。使用trizol样试剂进行RNA提取,遵循前面描述的协议[27],然后在1.5%琼脂糖凝胶上对每个RNA样品进行1 μl电泳,并在NanoDrop (ThermoFisher, USA)上对RNA进行定量,从而检查RNA质量。所有的cdna均由2 μg每个通过质量控制的RNA样本合成,经过1 μl DNase处理(NEB Biolab, England),在室温(22℃)下孵育15分钟,并按照制造商说明使用Protoscript-II逆转录酶试剂盒(NEB Biolab, England)。所有cDNA样本的质量都通过PCR得到了预期大小的产物。
用水(1:5)稀释的cDNA样本进行半定量和qPCR分析。半定量RT-PCR从每个合成样品和4个品种(包括盐处理和对照)中收集1 μl cDNA,并将其装入一个试管中。常规PCR的反应量为15 μl,含1 μl扩增的cDNA, 1 ×供应的反应缓冲液,1.8 mM MgCl2dNTP各0.2 mM,引物各0.25 μM, Go-Taq DNA聚合酶1.0单位(Promega, USA)。扩增在热MyCycler (BioRad, USA)中进行,程序如下:94°C 2分钟;30个周期,94°C 10 s, 55°C 10 s, 72°C 15 s;最后在72°C下延长1分钟。PCR产物的扩增子大小在136 - 165 bp之间变化1表S1)。PCR产物在1.5%琼脂糖凝胶中电泳分离,GelRed (Biotium, USA)用100 bp DNA Ladder (Bioline, USA)染色,使用GelDoc系统(BioRad, USA)在紫外线下可视化。
对于qPCR表达分析,采用实时qPCR系统,模型CFX96 (BioRad, USA)遵循前面描述的qPCR协议[31].每孔qPCR总体积(10 μl)包括5 μl 2 × KAPA SYBR FAST (KAPA Biosystems, USA)、4 μl稀释cDNA和1 μl 2个基因特异性引物(每个引物3 μl;额外的文件1表S1)按照制造商的建议。用基因表达相对于两个参比基因的平均表达的归一化方法计算靶基因的表达数据:adp -核基化因子1样蛋白(HvADP)、AJ508228和糖酵解甘油醛-3-磷酸脱氢酶(HvGAPDH), x60343 [32].每次qPCR实验至少使用3个生物重复和2个技术重复。
DNA提取和SNP荧光扩增基因分型
从田间试验生长的分离群体中,从每个品种和育种系中选择5株均匀植株,将最年轻的发育完全的叶片分别收集到10毫升的塑料管中(不膨大),代表每个品种和育种系的5个生物重复。如我们早期论文所述,用苯酚-氯仿从树叶样本中提取DNA [33].在0.8%琼脂糖凝胶上检测1 μl总基因组DNA以评估质量,并使用Nano-Drop (ThermoFisher, USA)测量浓度。
使用CFX96 Real-Time PCR检测系统(BioRad, USA)进行类荧光扩增SNP分析,使用的DNA样本如前所述[21,34],并对大麦基因分型进行以下调整。每个总体积为10 μl的反应包含:3 μl调整为20 ng/μl的模板DNA, 1 μl两个荧光标记通用探针混合物(每个0.125 μM), 1 μl等位基因特异性引物混合物(两个正向引物各0.075 μM,常见反向引物各0.39 μM), 2 μl 5 × Go-Taq Master-mix (Promega, USA),最终组分浓度如下:1.75 mM MgCl20.2 mM的dNTP和0.05单位的Go-Taq聚合酶(Promega, USA)。标记的SNP位点被用来设计等位基因特异性引物。通用探针和引物的序列以及生成的扩增子的大小在附加文件中给出1(图S2)。
PCR使用的是根据早先发表的计划进行调整的程序[21,35]:初始变性,95°C, 2分钟;20个“双重”循环,95°C 10秒,60°C 10秒,72°C 20秒,95°C 10秒,55°C 20秒,72°C 50秒;每个周期后记录等位基因特异性荧光。通过SNP调用的基因分型由仪器软件自动确定,但每个SNP结果也通过扩增曲线和最终的等位基因鉴别进行手动检查。实验重复两次,进行两次技术重复,确认了SNP呼叫的置信度。
统计分析
采用IBM SPSS统计软件,用方差分析计算和分析均数和标准误差,用Student 's估计显著性概率t以及。相关分析2采用受试者间效应测试(IBM SPSS, Statistics Desktop 25.0.0.0)进行。
数据和材料的可用性
在这项研究中产生和分析的所有数据都包含在这篇发表的文章及其附加文件中。
缩写
- KATU:
-
哈萨克农业技术大学
- SAP:
-
应激相关蛋白
- SNP:
-
单核苷酸多态性
- 肿瘤坏死因子:
-
肿瘤坏死因子
参考文献
- 1.
Dixit VM, Green S, Sarma V, Holzman LB, Wolf FW, O'Rourke K, Ward PA, Prochownik EV, Marks RM。肿瘤坏死因子-α诱导人内皮细胞中包括巨噬细胞特异性趋化素的新基因产物。中国生物化学杂志1990;265(5):2973-8。
- 2.
Giri J, Dansana PK, Kothari KS, Sharma G, Vij S, Tyagi AK。sap作为植物非生物胁迫反应的新型调节剂。Bioessays。2013;35(7):639 - 48。https://doi.org/10.1002/bies.201200181.
- 3.
金燕,王敏,付娟,宣宁,朱莹,连莹,贾铮,郑洁,王刚。植物ZnF-AN1基因的系统发育及表达分析。基因组学,2007;90(2):265 - 75。https://doi.org/10.1016/j.ygeno.2007.03.019.
- 4.
Mukhopadhyay A, Vij S, Tyagi AK。水稻锌指蛋白基因的过表达使转基因烟草能够耐受寒冷、脱水和盐胁迫。中国环境科学与技术2004;29(6):369 - 369。https://doi.org/10.1073/pnas.0401572101.
- 5.
Vij S, Tyagi AK。水稻A20/AN1锌指(s)胁迫相关蛋白(SAP)基因家族的全基因组分析及其系统发育关系拟南芥.生物技术学报,2006;27(6):565-75。https://doi.org/10.1007/s00438-006-0165-1.
- 6.
Solanke AU, Sharma MK, Tyagi AK, Sharma AK。环境胁迫响应的特征及其系统发育分析SAP编码番茄A20/AN1锌指蛋白的基因家族。分子基因组学,2009;282(2):153-64。https://doi.org/10.1007/s00438-009-0455-5.
- 7.
Martin RC, Glover-Cutter K, Baldwin JC, Dombrowski JE。盐胁迫诱导锌指蛋白的鉴定与鉴定季.BMC Res Notes。2012;5:66。https://doi.org/10.1186/1756-0500-5-66.
- 8.
刘杰,杨旭,杨旭,徐敏,刘杰,薛敏,马鹏LcSAP,一个羊草增强盐耐受性的基因酿酒酵母.Mol Biol 2017;44(1): 5-9。https://doi.org/10.1007/s11033-016-4091-y.
- 9.
常静,张静,毛旭,李安,贾静,景瑞TaSAP1-A1以及它与小麦农艺性状的关系。足底。2013;237(6):1495 - 508。https://doi.org/10.1007/s00425-013-1860-x.
- 10.
高伟,龙龙,田旭,金晶,刘宏,张宏,徐峰,宋聪。棉花含A20/AN1锌指胁迫相关蛋白(SAPs)的全基因组鉴定与表达分析。中国生物医学工程学报,2016;29(6):369 - 369。https://doi.org/10.1007/s00438-016-1252-6.
- 11.
Lloret A, Conejero A, Leida C, Petri C, Gil-Muñoz F, Burgos L, Badenes ML, Ríos g跟压力的蛋白质(SAP)基因在李属.科学通报2017;71:332。https://doi.org/10.1038/s41598-017-00471-7.
- 12.
董强,段东,赵松,徐斌,罗军,王强,黄东,刘超,李超,龚旭,毛凯,马峰。苹果胁迫相关蛋白基因家族的全基因组分析及克隆揭示MdSAP15在转基因作物中,它赋予了对干旱和渗透胁迫的耐受性拟南芥.国际分子科学杂志2018;19(9):2478。https://doi.org/10.3390/ijms19092478.
- 13.
何霞,谢松,谢鹏,姚明,刘伟,秦琳,刘铮,郑明,刘红,关明,华伟。与非生物胁迫响应相关的A20/AN1锌指结构域应力相关蛋白(SAP)的全基因组鉴定芸苔属植物显著.环境专家机器人2019;165:108-19。https://doi.org/10.1016/j.envexpbot.2019.05.007.
- 14.
周艳,曾亮,陈锐,王艳,宋杰。植物中锌指蛋白A20/AN1胁迫相关蛋白(SAP)基因家族的全基因组鉴定与鉴定Medicago truncatula.中国生物医学工程学报(英文版);2018;https://doi.org/10.2298/ABS170529028Z.
- 15.
Stegmann M, Monaghan J, Smakowska-Luzan E, Rovenich H, Lehner A, Holton N, Belkhadir Y, Zipfel c受体激酶FER是ralf调控的植物免疫信号转导支架。科学。2017;355(6322):287 - 9。https://doi.org/10.1126/science.aal2541.
- 16.
刘松,王杰,姜松,王宏,高原,张宏,李东,宋峰。番茄SlSAP3是胁迫相关蛋白家族的成员,是一种免疫调节因子两pv。番茄DC3000。植物病理学杂志2019;20(6):815-30。https://doi.org/10.1111/mpp.12793.
- 17.
刘松,袁霞,王勇,王宏,王俊,沈铮,高原,蔡杰,李东,宋峰。番茄胁迫相关蛋白4对番茄抗腐营养真菌免疫有积极作用葡萄孢菌.植物学报2019;32(5):566-82。https://doi.org/10.1094/MPMI-04-18-0097-R.
- 18.
Dixit AR, Dhankher OP.一种新的压力相关蛋白AtSAP10拟南芥耐镍、锰、锌和高温应力。科学通报,2011;6(6):e20921。https://doi.org/10.1371/journal.pone.0020921.
- 19.
沙玛G,吉里J,提亚吉AK。大米OsiSAP7负调控ABA胁迫信号,使拟南芥对水分亏缺胁迫敏感。植物科学学报,2015;https://doi.org/10.1016/j.plantsci.2015.05.011.
- 20.
Ben Saad R, Safi H, Ben Hsouna A, Brini F, Ben RW。LmSAP蛋白功能域分析揭示了锌指A20结构域在非生物胁迫耐受中的重要作用。原生质》2019。https://doi.org/10.1007/s00709-019-01390-2.
- 21.
Jatayev S, Kurishbaev A, Zotova L, Khasanova G, Serikbay D, Zhubatkanov A, Botayeva M, Zhumalin A, Turbekova A, Soole K, Langridge P, Shavrukov Y. ampliflor -like SNP标记在植物基因分型中的优势BMC Plant Biol 2017;17:254。https://doi.org/10.1186/s12870-017-1197-x.
- 22.
施ramm C, Kurishbaev A, Jatayev S, Anderson P, Shavrukov Y.单核苷酸多态性(SNP)标记在谷物育种和作物研究中的应用:现状与展望。作者:奥登F,弗里特W,编辑。作物育种技术进展。剑桥:Burleigh Dodds科学出版社;2019.https://doi.org/10.19103/AS.2019.0051.16.
- 23.
国际大麦测序联盟。2019年10月5日评估。https://imagefpc.public.iastate.edu/IBSC%20Webpage/IBSC%20Template-home.html.
- 24.
Hasunuma K,编辑。大麦:物理特性、遗传因素和环境对生长的影响。纽约:新星科学出版社;2014.
- 25.
盐应激或盐休克:我们在研究哪些基因?J Exp Bot 2013; 64:119-27。https://doi.org/10.1093/jxb/ers316.
- 26.
贝尔W,杰西诺e。春皮大麦品种的化学成分及营养价值。植物学报,2013;19(4):721-7。
- 27.
Shavrukov Y, Bovill J, Afzal I, Hayes JE, Roy SJ, Tester M, Collins NC。HVP10编码V-PPase是大麦的主要候选基因HvNax3钠排斥基因:来自精细定位和表达分析的证据。足底。2013;237:1111-22。https://doi.org/10.1007/s00425-012-1827-3.
- 28.
杨文华,杨文华,杨文华,等。水培技术在植物抗非生物胁迫研究中的应用。正确位置:Asao T,编辑。水培法。植物生物学研究的标准方法。Rijeka: InTech开放获取出版商;2012.39 - 66页。
- 29.
Shavrukov Y, Gupta NK,宫崎J, Baho MN, Chalmers KJ, Tester M, Langridge P, Collins NC。HvNax3-一种来自野生大麦的芽钠排除基因座(大麦芽ssp。spontaneum).功能整合基因组学2010;10:277-91。https://doi.org/10.1007/s10142-009-0153-8.
- 30.
系统发育网络在进化研究中的应用。Mol Biol Evol. 2006; 23:254-67。https://doi.org/10.1093/molbev/msj030.
- 31.
Shavrukov Y、Zhumalin A、Serikbay D、Botayeva M、Otemisova A、Absattarova A、Sereda G、Sereda S、Shvidchenko V、Turbekova A、Jatayev S、Lopato S、Soole K、Langridge P.利用Amplifluor SNP标记鉴定的dreb2型基因表达量与哈萨克斯坦北部面包小麦品种的生产性能和脱水耐受性相关。植物科学进展2016;7:1736。https://doi.org/10.3389/fpls.2016.01736.
- 32.
弗道斯·J,李勇,里德·N, Langridge P,史丕杰,Tricker PJ。不同胁迫条件下大麦microRNAs和mrna定量表达分析的内参基因鉴定。科学通报,2015;10(3):e0118503。https://doi.org/10.1371/journal.pone.0118503.
- 33.
Zotova L、Kurishbayev A、Jatayev S、Khassanova G、Zhubatkanov A、Serikbay D、Sereda S、Sereda T、Shvidchenko V、Lopato S、Jenkins C、Soole K、Langridge P、Shavrukov y转录因子TaDREB5和TaNFYC-A7基因在面包小麦叶片中对干旱、脱水和ABA的响应差异表达。植物科学进展2018;https://doi.org/10.3389/fpls.2018.01441.
- 34.
Zotova L, Kurishbayev A, Jatayev S, Goncharov NP, Shamambayeva N, Kashapov A, Nuralov A, Otemissova A, Sereda S, Shvidchenko V, Lopato S, Schramm C, Jenkins C, Soole K, Langridge P, Shavrukov Y.一般转录抑制子TaDr1共同表达为TaVrn1而且TaFT1在干旱下种植面包和小麦。2019;10:63。https://doi.org/10.3389/fgene.2019.00063.
- 35.
Khassanova G, Kurishbayev A, Jatayev S, Zhubatkanov A, Zhumalin A, Turbekova A, Amantaev B, Lopato S, Schramm C, Jenkins C, Soole K, Langridge P, Shavrukov Y.细胞内囊泡运输基因,RabC -GTP蛋白在盐胁迫和快速脱水条件下表达高,而在干旱条件下表达下调。中投arietinuml .)。2019;10: -40。https://doi.org/10.3389/fgene.2019.00040.
确认
我们要感谢南澳大利亚弗林德斯大学(澳大利亚)和哈萨克斯坦农业技术大学(努尔苏丹)的工作人员和学生,感谢他们对这项研究的支持和对手稿的批评意见。
关于这个补品
本文已发布的一部分BMC植物生物学第20卷增刊1,2020:第五届国际科学会议“植物遗传学、基因组学、生物信息学和生物技术”(PlantGen2019)的文章精选。该补刊的全部内容可在网上查阅//www.cinefiend.com/articles/supplements/volume-20-supplement-1.
资金
该研究得到了教育和科学部的支持。哈萨克斯坦,研究项目BR05236500 (SJ)。出版费用由该计划提供。资助者不参与研究设计、数据收集、分析和解释、决定发表或准备手稿。
作者信息
从属关系
贡献
AB和MA进行了植物生长和基因表达的实验部分;AK和SJ在哈萨克斯坦建议、安排和监督实验;MM和AZ进行了PCR、测序和基因分型;GS、RP、SS进行杂交并进行田间试验分析;PL建议和监督混合分析,并编辑最终版本的手稿;CS协助分析水培实验数据,编辑初稿;CLDJ为生物信息学数据分析提供建议和监督,并编辑了手稿的改进版本;KLS在澳大利亚建议和监督基因表达分析;YS协助并监督了所有的实验,并撰写了第一版的手稿。所有作者都阅读并认可了最终稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者声明他们没有竞争利益。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
附加文件1:图S1。
的3 ' -UTR区序列片段HvSAP12两个大麦亲本Granal和Baisheshek的基因。图S2。SNP的位置,引物设计,通用探针和SNP特异性引物的序列,扩增子的大小。表S1。引物用于半定量RT-PCR和qPCR分析17HvSAP基因和两个内参基因,包括扩增子大小和寡核苷酸序列。
权利与权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
阿尔达马斯(M. Aldammas),库里什巴耶夫(A. Kurishbayev)。et al。锌指A20/AN1胁迫相关基因的鉴定、基因表达及遗传多态性HvSAP用的是哈萨克斯坦的盐胁迫大麦。植物生物学20.156(2020)。https://doi.org/10.1186/s12870-020-02332-4
收到了:
接受:
发表:
关键字
- 大麦
- 基因表达
- 遗传多态性
- HvSAP
- 分子标记辅助选择
- SNP标记
- 跟压力的蛋白质
- 盐度
- 锌指A20/AN1转录因子