摘要
背景
Globodera rostochiensis属于马铃薯的主要病原菌,与寄主植物的根有复杂的相互作用机制。商业品种的抗性通常是基于特定的R基因从近缘野生物种的自然种群和安第斯高原本地马铃薯品种中渗入。研究抗病分子机制,筛选抗病基因的自然群体,对于了解植物病原菌相互作用的基础知识和培育持久抗性具有重要意义。这里我们利用了茄属植物phureja在南美洲采集到的植物,与之形成对比的抗性G. Rostochiensis..
结果
的侵扰美国phureja与G. Rostochiensis.幼体导致创伤应激,随后激活细胞分裂和组织再生过程。与敏感美国phureja基因型,通过快速诱导各种应力响应相关基因的耐药性反应。该分子事件链伴有幼稚侵袭性位点的过敏反应并提供高液位阻力。转录组分析还揭示了分析之间的相当大的差异美国phureja基因型和参考基因组。
结论
植物根系中与基因表达模式变化相关的分子过程G. Rostochiensis.讨论了虫害和抗性或敏感表型的建立。转录组从头组装被认为是发现新的抗性性状的重要工具美国phureja加入。
背景
马铃薯包囊线虫起源于南美洲安第斯地区[1].金黄色马铃薯包囊线虫(GPCN,Globodera rostochiensis)是一种在世界范围内发现的检疫性害虫,对马铃薯生产造成相当大的损失[2,3.].目前,G. Rostochiensis.(Ro1型)在俄罗斯联邦61个领土实体登记,包括861个行政区,占地面积1,794,442公顷[4,5].根据马铃薯品种的不同,产量损失可能在19%到90%之间[6],而GPCN卵子可以在囊肿内休眠和存活30年[7].预测的影响G. Rostochiensis.也可能因气候变化而增长[8].
马铃薯包囊线虫对植物的保护是复杂的,通常涉及到杀线虫剂的使用[8,9和诱捕作物(例如,茄属植物sisymbriifolium)[3.,10.].然而,许多化学杀线虫剂要么效率低[11.,12.欧洲的毒性和禁止,GPCN的控制主要基于主要抗性基因的部署(R-Genes)(例如,[13.])。对GPCN致病型Ro1具有较强抗性的R基因已渗入南美起源种的商品品种中H1来自栽培物种的基因茄属植物tuberosum无性系种群。andigenum[14.),Gro1-4来自玻利维亚野生物种的基因S. Spegazzinii.[15.,16.].由于存在线虫进化的威胁,人们普遍认为新的R基因的渗入具有重要意义[13.,17.].
GPCN的幼体渗透植物的根,在其中诱导喂养合胞体的形成。来自食道腺体的效应蛋白触发代谢和形态变化链,导致几个根细胞融合成一个喂养结构,以及抑制植物免疫反应[18.].在一些植物品种对静止线虫的抗性研究中,所揭示的抗性机制主要基于不亲和互作和超敏反应:死细胞区在虫灾的早期或后期形成的死细胞区,使幼体无法获得营养,无法完成其生命周期[19.].尚未评估对土豆盐久坐线虫线虫的抗性机制的自然各种各样。最近的研究强调了植物线涂层相互作用的生物学复杂性,例如不寻常的R基因的作用(例如大豆RHG1α-克拉蛋白有效,伴有各种囊肿线虫)[20.,21.]、新的线虫效应体及免疫抑制的相关机制(如:rha1b.)[22.].
植物-线虫亲和和不亲和的相互作用为详细研究植物细胞重编程的基本遗传机制、防御系统的预防和宿主-病原共同进化提供了独特的模型。相互作用的过程是相当复杂的:线虫接种的一个重要特征是显著的组织损伤导致非特异性的伤害胁迫。这种非特异性伤害反应可能与对GPCN的特异性反应重叠,或成为其组成部分[23.].
在这里,我们提供了两个二倍体马铃薯的根转录组的详细比较分析结果美国phureja与GPCN抗性形成对比。在线虫幼体接种后,转录组的动态变化揭示了与细胞分裂、组织再生、对病原体的非特异性防御以及一些未知作用的过程(如质体相关代谢链的变化)相结合的生物学过程。讨论了线虫繁殖和植物抗性的分子机制。另一项观察是关于研究安第斯本地马铃薯品种的一般方法。研究发现,参考基因组注释不足以分析这些来自VIR收集的二倍体马铃薯的转录组,而从头转录组组装可以填补这一空白(至少部分)。本研究的目的之一是通过常规育种或其他生物技术方法(如[24.,25.,26.,27.,28.])。
结果
gpcn耐药与易感的转录组比较美国phureja基因型
的根的样本美国phureja分别在接种前(0)、接种后(24)和接种后(72)h取i-0144786和i-0144787 (hpi),接种GPCN幼体或水(每个时间点/类型3株)。获得30个配对reads文库,共1265,033,228个配对reads作为原始测序数据。过滤后的967,259,304配对reads仍然是干净的测序数据。平均库大小大约是4200万读对之前和3200万读对后过滤(附加文件1).
平均而言,65%的读基因对与参考基因组是一致的[29.],使用STAR工具。计算了所有注释的39021个基因的读计数。在差异表达评估前对低表达基因进行过滤,得到21,113个基因样本,用于进一步分析。
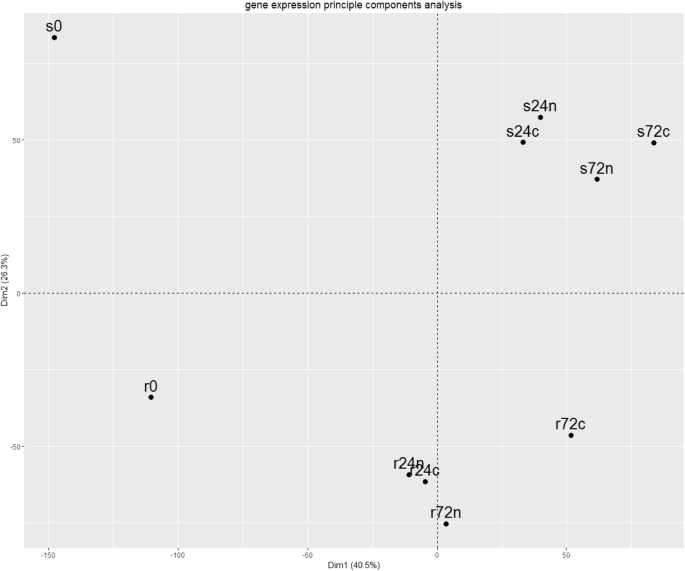
两个比较的转录om之间的差异美国phureja主成分分析评价基因型(PCA,图。1).第一主成分(轴X)对应于不同时间点(0,24,72hPI)的基因表达模式的变化,用于抗性和易感基因型。第二组分(Y轴)表征两种分析的基因型之间基因表达模式的差异。
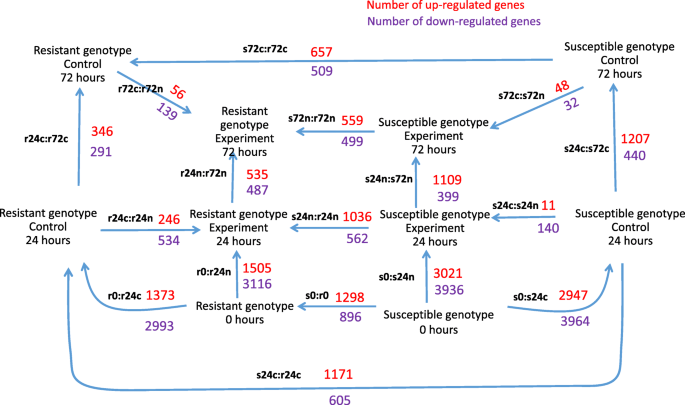
人们可以看到美国phurejagpcn耐药和易感基因型具有不同的转录组,这可能涉及这些材料的不同来源;(ii)基因表达模式在24 hpi处变化最大,72 hpi处变化较小;(iii)易感植物的转录组变化比抗性基因型更明显。(1)在相同的实验条件下,检测易感基因型中基因表达上调或下调的基因(s0:r0, s24n:r24n, s24c:r24c, s72n:r72n, s722c:r72c);(ii)揭示同一基因型植物在不同hpi点(r0:r24c, r24c:r72c, r0:r24n, r24n:r72n, s0:s24c, s24c:s72c, s0:s24n, s24n:s72n)的转录组差异;(iii)揭示同一基因型植物的转录组差异,但接种水(c,对照)或线虫(n) (r24c:r24n, r72c:r72n, s24c:s24n, s72c:s72n)。总的来说,结果是17个两两比较(图。2).
图(图。2)反映已识别的DEG的数量。箭头显示比较方向。例如,对比较S0:易感基因型之间的易感基因型(S0)和耐药性耐腐蚀(R0)产生1298个基因,其表达水平较高,表达水平低于抗性基因型的896个基因。差异表达基因的完整列表可用作附加文件2.
这些数据与主成分分析(图。1).首先,在0和24hpi的根转录om之间的Degs的数量大于两种基因型的24和72 HPI之间。在0和24hpi接种水后抗性基因型的根数为1373(上)和2993(下),而24 HPI和72 HPI的次数为346(上升)和291(下降)).观察到类似的情况用于抗性美国phurejaNematode接种后的加入:R0:R24N(1505(上),3116(下))与R24N:R72N(535(上),487(下))。其次,在易感品种的根部通常越大越大。最后,用水或线虫幼虫接种后,在接种后,对易感植物的根部的特征在于较小的差异。例如,S24C:S24N比较揭示了11个和140个下调基因,而根部转录组的比较美国phureja接种GPCN或水(r24c:r24n)后,抗性植株中有246个基因表达上调,534个基因表达下调。
基因本体论
在AgriGO的帮助下,获得了不同植物样本对的基因集富集分析结果(附加文件)3.包含所有比较的GO项的完整列表)。观察到的趋势摘要呈现在表格中1.
从头转录组组装
美国phureja正在调查的基因型是在南美洲收集的,它们的基因组可能与参考基因组不同[29.].因此,仅基于与参考基因组的对准的转录组的注释未完成,并且可以从考虑中错过一些重要的基因。至少部分地填补这个差距,我们还研究了美国phureja不使用参考基因组而重新构建转录组。为此,组装了两个主转录组。原始转录组由538,200个(抗性accession i-0144787)和643,926个contigs(敏感accession i-0144786)组成。在利用evidentigene软件减少冗余装配体后,168,254和172,210个contigs仍然存在于抗性和敏感的非冗余装配体中美国phureja分别转录om。BUSCO分析显示,占抗性和74.4%的抗分线抗性的89.9%,占易感基因型的74.4%和13.3%的碎片化BUSCO。
平均而言,78%的干净读对与抗病品种的非冗余重叠集对齐,72%与感病品种的非冗余重叠集对齐。此外,参照基因组、CDS和蛋白质序列对非冗余contigs序列进行了比对。将contigs与参考基因组和CDS序列进行TPM筛选和比对的结果汇总于表中2.
人们可能会发现,有一组与参考基因组没有显著同源性的新转录本(由独立blastn软件定义的e值e < 10)−30).实际上,不能通过与参考基因组的对准预测这些新转录物的功能,并且它们的功能分析需要其他方法。因此,我们在诸如HMM封装的借助于Novo组装的Contig潜在地编码的蛋白质中的域中的域(Contigs与CD和基因组的比对的结果以及PFAM域预测的额外的蛋白质4).研究发现,相当一部分新的转录组编码与pfam注释的功能域同源的多肽,即这些mrna可能编码功能蛋白(见表)2).
事实上,转录组分析需要额外的支持,以验证潜在的功能性转录本在翻译组和蛋白质组水平上的表达(例如,[30.,31.,32.])。然而,所提供的数据为进一步对特定基因的实验研究提供了背景,例如,编码NBS-LRR受体的基因。众所周知,抗GPCN的r基因通常属于NBS-LRR受体家族。早前有报道称i-0144787的抗性美国phureja加入不是由于已知的R基因的存在[4].由于一些重新组装的转录本在参考基因组注释中没有对应的基因,因此应该在这些contigs上扩展对新r基因的搜索。为了揭示潜在的负责GPCN耐药的NBS-LRR基因,我们利用NLR-Parser软件对从头组装的转录本进行了表征[33.].研究发现,许多cdna编码具有NBS-LRR结构域的蛋白,其中一些contigs与参考基因组没有注释甚至不具有同源性。对易感美国phureja其中30个contigs被预测为NBS-LRR候选基因,所有候选基因均可与参考基因组进行比对,但仅有26个与注释蛋白编码基因的基因组位点进行比对(即有4个cDNA可能由注释中缺失的基因编码)。对于GPCN-resistant美国phureja基因型,预测了编码NBS-LRR蛋白的33个CDNA。可以将三十二个聚合物对准在参考基因组序列上,其中28个被映射到已知的蛋白质编码基因座。因此,两者的转录组美国phureja加入包含新的NBS-LRR编码基因的额外转录本(附加文件4).有趣的是,通过NLR-PARSER预测为NBS-LRR的一种CONTIG与NBS-PARSER编码cDNA,对核苷酸序列水平的参考基因组或CDS序列没有同源性。它的尺寸为3112个核苷酸,其预测的翻译产品长度为865氨基酸。CONTIG具有具有显着性值E = 1.1°10的NB弧域−39和LRR-8域预测E = 4.5°10- 6.将其与NCBI非冗余基因序列进行比对,结果与表中列出的5个条目同源3..
此外,该cDNA contig与第3染色体的核苷酸序列具有同源性美国lycopersicum和美国penellii.同时,Contig预测的翻译产品的氨基酸序列显示出在NCBI蛋白质数据库中呈现的许多蛋白质序列的同源性。有趣的是,100个序列中最好的击中,没有序列美国tuberosum或美国phureja.十首最佳歌曲包括美国pennellii,美国lycopersicum,n .烟草,n tomentosiformis,n的抗旱性.
候选主基因提供名单美国phureja抗GPCN品种i-0144787
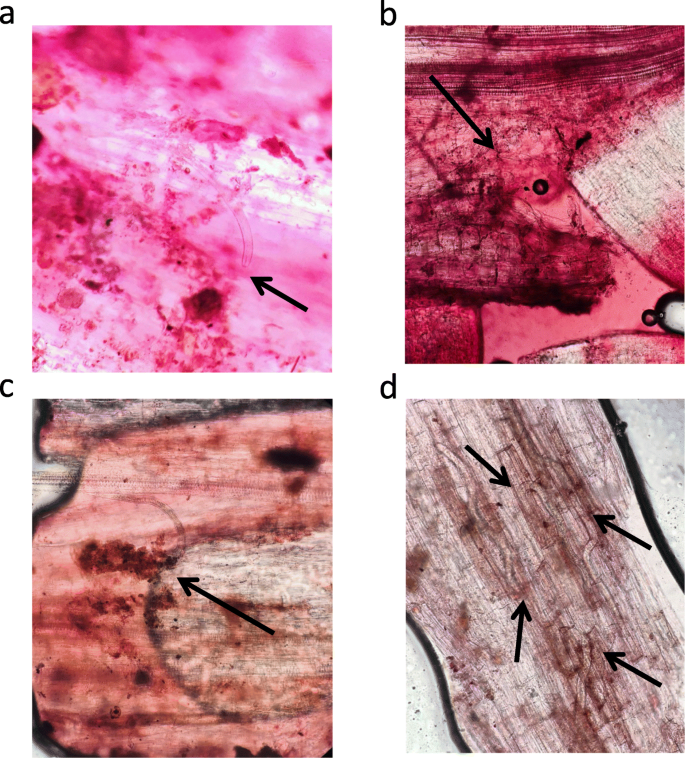
本研究的任务之一涉及到基因组中r基因的鉴定美国phurejaI-0144787加入为GPCN提供高抗性。根据所得的结果,通过HR响应和编程细胞死亡介导的抗性(图。3.),通常与NBS-LRR型受体相连。
因此,我们根据以下假设编制了候选r基因列表:(1)候选基因应属于NBS-LRR组;(ii)候选基因在抗性根中的表达水平要高得多美国phureja基因型(最好在易感基因型中无表达);(iii)应检测候选基因在抗性根中的表达水平美国phureja基因型在水或GPCN幼体接种前。
(Jupe et al., 2013)中描述的419个潜在R基因列表[34.进行了分析。寻找与的同源性美国phureja转录本显示抗性基因根中有17个基因普遍表达上调,13个基因表达下调(附加文件)5).其中一些转录物用于QRT-PCR以验证RNA-SEQ结果(表4).
大多数情况下,qRT-PCR结果支持RNA-seq数据,所有这些基因都可以作为进一步r -基因搜索的主要候选基因(关于qRT-PCR的详细信息可参见附加文件)6和7).
讨论
本研究比较了来自南美不同地区的两个二倍体马铃薯标本。早前已有研究表明,即位i-0144786对GPCN敏感,而即位i-0144787高度耐药[4].有趣的是,所观察到的抗性与广泛分布的已知R基因的存在无关美国tuberosum商业品种(GRO1-4,H1).对i-0144786和i-0144787的比较分析显示,它们转录组的一般组成以及nbs - lrr编码基因的表达集存在相当大的差异[35.].因为这些美国phureja南美不同地区的基因型进化不同,我们对它们的根转录组进行了详细的比较研究,从根转录组水平评估GPCN抗性的潜在机制和遗传差异。
得到的结果支持了早期的观察结果,即两者之间存在相当大的差异美国phureja基因型(无花果。2,3.).与抗性相关的分子机制G. Rostochiensis.在AgriGO的帮助下进一步分析(表)1).实验包括用水或GPCN幼体接种马铃薯植株的根,并在0 hpi(即接种前)、24 hpi和72 hpi下分析样品。事实上,根的接种过程本身就会造成组织机械性损伤。在接种水前(0 hpi)和接种水后(24 hpi),对抗性和敏感基因型的根转录组进行比较,发现细胞周期相关项上调,而多种调节和代谢过程受到抑制。这一趋势在72 hpi时间点更为明显。转录组的动态可能反映了试验设计导致的中等损伤胁迫下马铃薯根系的反应,主要与损伤后的组织再生有关。有趣的是,抗性基因型在24 hpi点对水接种诱导的伤害有相当强的响应,而在72 hpi点与敏感基因型没有这种差异(表)1).
用GPCN接种土豆根源导致组织损伤的相当强烈的组织损伤,因为在根汽缸和血管系统中的血管渗透和运动。GPCN接种后易感基因型的根细胞的转录组的变化与水控制相似:上调次数的农场术语主要与细胞周期相关,有丝分裂(24 HPI,72 HPI)和各种代谢过程(72 HPI)。由于成功的线虫侵扰导致Syncytia(饲养部位)的发育,细胞周期和组织再生相关的农场术语也可能反映了这一过程。抗性美国phureja对接种GPCN的反应非常不同:在24 hpi点,各种应激反应相关基因上调。这一列表包括了与氧化应激、过氧化物酶活性、肽酶抑制剂、糖基化酶相关的术语。在72 hpi时,抗性基因型的根转录组对生物胁迫及其相关过程(氧化还原、过氧化物酶活性、糖基酶、内肽酶、蛋白酶、核糖核酸酶等)反应强烈。还检测到与组织再生相关的基因的激活(细胞壁生物发生、细胞分裂等,表1).这些术语可能反映了被迁移的GPCN幼体损伤的根组织的再生,以及一些其他过程,例如合胞体的启动,以及由于过敏反应导致的局部程序性细胞死亡后的再生。因此,抗性根和敏感根的转录组动态有很大的差异美国phureja基因型在0,24和72 HPI。有趣的是,在抗性和易感基因型之间的转录om之间的比较似乎不那么富有信息,可能是因为这些摘要之间的遗传分歧。另一个观察结果指出与塑体定位的代谢链有关的次数。塑性体中的方法可能具有重要性建立抵抗力,并且应该进行进一步分析。
一般来说,抗美国phureja基因型在24 hpi点具有较强的生物胁迫反应和较强的生物胁迫反应基因诱导的特点。抗性机制整合了局部超敏反应(图。3.),以及防御相关基因的系统性诱导(表1).GPCN侵染伴随着抗性和易感基因型细胞分裂相关基因的高表达,这可能反映了线虫分泌的调控因子在幼虫运动和细胞重编程后的组织再生。
发现根部转录om美国phureja研究结果包含了大量未在参考基因组中注释的转录本,并可能编码具有pfam注释功能域的蛋白(见表)2).马铃薯和其他栽培种的天然品种作为新育种性状的来源被广泛使用。在这里,我们证明了从头转录组组装提供了额外的数据,以揭示R基因注释不当或甚至在参考基因组中缺失(表)3.).几种NBS-LRR编码转录本的特征美国phureja选择耐药基因型以进一步绘制主要的GPCN抗性特征(表4).
在本研究中两种自然美国phureja标本的选择是由于它们对GPCN的相对抗性。这些采集于南美洲偏远地区的标本很可能是在不同病原体种类的压力下进化而来的,它们具有特定的R基因子集。转录组分析为抗病建立的遗传机制提供了有价值的数据,并为其渗入育种程序提供了新的、可能有用的R基因。
结论
二倍体马铃薯的接种美国phureja与GPCN的幼体伴随根损伤应激和再生过程的激活。在根抗gpcn美国phureja基因型,分子事件链在幼体的入侵位点启动超敏反应,并提供高水平的抗性。比较转录组分析是揭示抗病机制和候选r基因的有效工具。特别是,从头转录组组装突出了新的功能基因变异,并大大扩展了参考基因组注释。
方法
植物材料
两种二倍体栽培种的加入美国phureja从Vir Potato收集获得K-11,291(秘鲁收集)和K-9836(来自玻利维亚)(每种加入用Vir Ins I-0144787和I-0144786分别由一个克隆(基因型)代表)。根据Plastid SSRS数据,这些access具有不同的单倍型,表明其不同的母体起源[36.,37.].基因型i-0144786对GPCN (Ro1)敏感,而i-0144787对GPCN (Ro1)高抗,但不含GPCN (Ro1)的DNA标记Gro1-4和H1(TG689, 239E4 left/Alu I, and Gro1-4) [4,35.].
线虫接种
人口的G. Rostochiensis.(pathotype Ro1)起源于俄罗斯列宁格勒地区(Belogorka),以前曾被描述[4].将线虫种群在感病品种“Nevsky”上繁殖。用浮选技术从土壤中提取包囊,并在4°C保存。美国phureja如前所述进行了植物准备和接种GPCN [35.].用酸性品红染色的感染根,在AxioScope A1光学显微镜(卡尔蔡司,德国)下扫描线虫的存在。
RNA提取
RNA-seq,美国phureja分别在接种前(0 h)、接种后24 h和72 h采集根G. Rostochiensis.(致病型Ro1)。对于每个基因型,取3株感染植株和3株对照(水接种)植株。需要注意的是,线虫接种技术会造成一些根伤,因此水接种根为转录组比较提供了重要的控制。这些植物的根用无菌蒸馏水彻底冲洗,固定在液氮中,用于RNA提取。用RNeasy Plant Mini Kit (Qiagen)提取总RNA。
RNA-SEQ.
使用生物分析仪2100(Agilent)评估RNA样品的质量,所有样品均具有载体8.2或更高。使用Mirvana™miRNA分离套件(AMALION)提取小RNA和总RNA级分。RNA-SEQ.library preparations were carried out with 1.5 mkg of total RNA fraction using TruSeq® Stranded mRNA LT Sample Prep Kit (Illumina) according to the manufacturer’s instructions for barcoded libraries with small modifications (4 min RNA fragmentation time and 12 PCR cycles were used). Final libraries quantification was performed with a Bioanalyzer 2100 and a DNA High Sensitivity Kit (Agilent). After normalization, barcoded libraries were pooled and sequenced on a NextSeq550 sequencer 2 × 150 bp using a High Density Cassette and NextSeq® 500 High Output v2 Kit 300 cycles (Illumina).
中存在
RNA样本用DNAse (QIAGEN RNase-Free DNAse Set)处理。通过反转录制备单链cDNA,使用RevertAidTM试剂盒(Thermo Fisher Scientific Inc., Waltham, MA, USA)提取总RNA样本1 μg。利用OLIGO软件设计qRT-PCR引物。β-微管蛋白基因作为参考(GenBank: Z33382;正向引物5 ' -AGCTTCTGGTGGACGTTATG-3 ',反向引物5 ' -ACCAAGTTATCAGGACGGAAGA-3 ')。使用SYNTOL SYBR Green I试剂盒(SYNTOL, Moscow, Russia)进行qRT-PCR。对于每个反应,进行三次技术重复。
RNA-seq数据的生物信息学分析
库预处理
对Illumina NextSeq550测序平台获得的30个对端短reads文库进行分析。测序质量和长度分布采用FastQC软件(https://www.bioinformatics.babraham.ac.uk/projects/fastqc/0.11.5)版本。Cutadapt软件版本1.9.1 [38.]用于移除测序适配器,Prinseq-lite软件版本0.20.4 [39.]实施以筛选以下标准:长度不小于100个基础,平均验证质量得分不小于30.如果在一对中读取的情况下不符合上述标准,则从分析中丢弃全对。此外,采用prinseq选项'-derep 1',以便去除可能的光学复制。
短读文库与取自Ensembl植物数据库的参考基因组版本SolTub_3.0进行了比对[29.]使用STAR Aligner 2.5.3a [40].
基因表达分析
R的EdgeR包[41.,以鉴定差异表达基因(DEGs)。首先,对阅读计数表进行过滤,以去除表达水平低于阈值的基因(30个文库中不少于2个文库的表达水平不低于3个/百万)。利用R的PlotMDS功能,根据基因表达水平对文库进行多维聚类可视化。其次,使用EdgeR函数calcNormFactors对读取计数进行规范化。用EdgeR的估计disp函数计算色散。得到的伪计数矩阵和色散值对EdgeR的glmQLFit函数进行处理。最后,利用glmQLFTest功能对不同实验条件下收集的根转录组中的DEGs进行鉴定。此外,使用TPM程序对读计数进行归一化,并计算TPM值的平均值。基因分为不同表达如果一笔的意思TPM的实验值不小于10相比,实验之间的表达水平的变化计算磨边机是不少于两倍(| log2 (FC) |≥1),和错误发现率值小于或等于0.05(罗斯福≤0.05)。
此外,使用AgriGO v2的在线工具“奇异富集分析”进行了基因本体术语富集分析[42.]默认参数(使用yekutieli校正的Fisher测试,术语p< 0.05则认为显著富集)。分别对上调组和下调组进行分析。
从头转录组组装
从头组装美国phureja转录组采用Trinity软件2.6.5版本。从15个抗性或敏感基因型文库中分别组装了2个基因型的转录组。因此,两个“主转录组”被组装,每个对应于一个美国phureja基因型。进一步分析组装的转录om包括以下步骤:冗余降低,转录量分析,基因组表示搜索和PFAM引向域预测,包括NB和LRR域预测。
为了减少组装转录om的冗余,实施了证据基因管道版本'18may07'的Tr2aacds.pl效用。此实用程序识别组装Contigs中的开放阅读帧(ORF),并丢弃缺少至少一个ORF的Contig。然后,它将CONDIG合并在一起基于其预测的ORFS - 如果一个ORF的一个ORF的序列是另一个CONTIG的较长ORF的子序列,则移除具有较短ORF的CONTIG。将结果的Contig集被归类为非冗余。另外,该实用程序识别对应于预测ORF的蛋白质的氨基酸序列。
Busco(基准通用单拷贝Orthologs)软件版本3.0.2 [43.,44.利用]用于评估组件的质量。对胚胎搜索的非冗余组合体对胚胎搜索进行胚胎搜索。Busco专门设计用于评估基因组或转录om质量。
为了通过RNA-seq库的短读来评估contig覆盖率,使用bowtie -build实用程序(version 2.2.9)对contig的非冗余集进行了索引[45.].然后,应用Trinity软件包的几个实用软件分析contigs的转录水平。首先,使用了' abundance_estimate_to_matrix '效用。选择基于比对的丰度估计方法,利用eXpress 1.5.1版本软件估算所有库中每个contig的丰度值。' filter_low_expr_transcripts '实用程序用于删除表达式值低于基于' Transcript Per Million ' (TPM)值所选阈值的contigs。三个Trinity实用程序用于研究组装的contigs的差异表达:' run_DE_analysis ', ' DE_results_to_pairwise_summary ', ' pairwise_DE_summary_to_DE_classification '。使用先前获得的表达式值矩阵运行' run_DE_analysis '实用程序。
比较的转录组美国phureja与参考基因组,Blat软件版本34 [46.使用了)。此外,借助ncbi-blast独立包版本2.7.1+的blastn实用程序[47.,非冗余的contigs对齐美国tuberosum从Ensembl植物数据库获得的CDS序列版本3.0。将预测的非冗余contigs氨基酸序列进行比对美国tuberosum在blastp工具的帮助下,从Ensembl植物数据库获得的肽序列版本3.0。对于功能分析和域结构预测,hmmscan利用HMMER包(hmmer.org实现了3.1b2版本。Pfam-A。Pfam release 31.0的hmm数据库[48.]被用作参考数据库。
De Novo组装转录物的预测潜在地编码NBS-LRR蛋白
nlr解析软件,MEME套件版本4.9.1 [49.桅杆公用事业用于此目的。Jupe和Co-authors提供的MEME.xml文件[34.]从GitHub库下载,用作主题定义文件。在预测的非冗余氨基酸序列中寻找NBS-LRR基序美国phureja利用MAST进行转录组分析。xml格式的输出文件作为nlr解析器软件的输入[33.].
另外分析了与参考基因组无同源性的nbs - lrr样蛋白的特异性cdna。利用Nucleotide blast工具将contigs序列与NCBI nr数据库进行比对,并将相应氨基酸序列与NCBI非冗余蛋白序列数据库进行比对。10个最佳的蛋白质序列使用ClustAl Omega web创建一个多重比对文件。
可用性数据和材料
原始测序数据保存在NCBI Sequence Read Archive中,编号为PRJNA515801 (https://www.ncbi.nlm.nih.gov/bioproject/PRJNA515801).
改变历史记录
2021年1月15日
这篇文章的修订本已经发表,可以通过原始文章访问。
缩写
- GPCN:
-
金黄色马铃薯包囊线虫
- R基因:
-
耐药基因
- 梵尔:
-
瓦维洛夫植物遗传资源研究所
- 度:
-
差异表达基因
- 走:
-
基因本体论
- 子:
-
开放阅读框
- 车身:
-
基准通用单拷贝Orthologs
- TPM:
-
每百万成绩单
- CD:
-
编码DNA序列
参考文献
- 1。
Hockland S, Niere B, Grenier E, Blok V, Phillips M, den Nijs L, Anthoine G, Pickup J, Viaene N.对非欧洲群体毒力影响的评估Globodera pallida和G. Rostochiensis.在欧洲种植马铃薯。线虫学。2012;14:1-13https://doi.org/10.1163/138855411X587112.
- 2。
Evans K, Trudgill DL。马铃薯生产中的有害生物。第1部分。马铃薯的线虫害虫。In: Harris P, editor。马铃薯作物。伦敦:Chapman & Hall;1992.
- 3.
Dandurand LM, Zasada IA, Wang X, Mimee B, De Jong W, Novy R, Whitworth J, Kuhl JC。马铃薯包囊线虫在北美的现状。植物病理学。2019;57:117-33。https://doi.org/10.1146/annurev-phyto-082718-100254.
- 4.
Limantseva L, Mironenko N, Shuvalov O, Antonova O, Khiutti A, Novikova L, Afanasenko O, Spooner D, Gavrilenko tGlobodera rostochiensis来自瓦维洛夫植物工业研究所的栽培和野生马铃薯品种的致病型Ro1。植物品种。2014;133:660-5https://doi.org/10.1111/pbr.12195.
- 5。
Khiutti AV, Antonova OY, Mironenko NV, Gavrilenko TA, Afanasenko OS。马铃薯对检疫病害的抗性。中国生物医学工程学报2017;7(8):833-44https://doi.org/10.1134/S2079059717050094.
- 6。
不知道在美国发生或有限分布的害虫,第68号:黄金线虫:美国农业部,动植物卫生检查服务,植物保护和检疫;1985.p。10。https://archive.org/details/CAT31375597/mode/2up.
- 7.
Winslow Rd,Willis RJ。马铃薯的线虫疾病。II。土豆囊肿线虫,heterodera rostochiensis..见于:韦伯斯特J编辑。经济线虫学。纽约:学术;1972.- 34页。
- 8.
Jones LM, Koehler AK, Trnka M, Balek J, Challinor AJ, Atkinson HJ, Urwin PE。据预测,气候变化将改变目前的害虫状况Globodera pallida和G. Rostochiensis.在英国。中国科学:地球科学。2017;23(11):4497-507。https://doi.org/10.1111/gcb.13676.
- 9.
基于氟砜对马铃薯包囊线虫的新型杀线虫作用,研究了氟砜对马铃薯包囊线虫的进行性代谢损伤Globodera pallida.Perfist Biochem physiol。2017; 142:83-90。https://doi.org/10.1016/j.pestbp.2017.01.009.
- 10.
Dandurand LM, Zasada IA, LaMondia JA。诱捕作物的影响,茄属植物sisymbriifolium, 在Globodera pallida,globodera tabacum.,Globodera ellingtonae.J Nematol。2019;51:1-11。https://doi.org/10.21307/jofnem-2019-030.
- 11.
Trudgill DL, Elliot MJ, Evans K, Phillips MS.白薯包囊线虫(Globodera pallida)——对英国面临的威胁的批判性分析。Ann Appl Biol. 2003; 143:73-80https://doi.org/10.1111/j.1744-7348.2003.tb00271.x.
- 12.
俄罗斯联邦境内使用的农药和农用化学品目录,2017年。https://www.agroxxi.ru/upload/pia/handbook64.zip.
- 13。
Whitworth JL, Novy RG, Zasada IA, Wang X, Dandurand LM, Kuhl JC。马铃薯选育无性系和品种对三种马铃薯包囊线虫的抗性。工厂说。2018;102 (11):2120 - 8https://doi.org/10.1094/PDIS-12-17-1978-RE.
- 14。
J,他赞美上帝E,奥肯博U, van Vliet J, Peleman J,塞格尔B, van der Heijden年代,Linde P, Graveland R, Hutten R, van Eck H, Coppoolse E, der Vossen E, J,他Goverse A高分辨率的H1轨迹地图窝藏抗线虫马铃薯囊肿Globodera rostochiensis.Al Appl Genet。2004; 109:146-52。https://doi.org/10.1007/S00122-004-1606-Z..
- 15.
利用限制性片段长度多态性定位马铃薯抗包囊线虫的主要显性基因Globodera rostochiensis.Mol Gen Genet。1990年; 224:177-82https://doi.org/10.1007/BF00271550.
- 16。
马铃薯抗线虫基因Gro1的标记富集及高分辨率图谱分析孟根。1995;249:82-90https://doi.org/10.1007/BF00290239.
- 17.
Strachan SM, Armstrong MR, Kaur A, Wright KM, Lim TY, Baker K, Jones J, Bryan G, Blok V, Hein iGlobodera pallida致四倍体马铃薯的致病型PA1。Al Appl Genet。2019; 132(4):1283-94https://doi.org/10.1007/s00122-019-03278-4.
- 18.
王志强,王志强,王志强,等。寄生性线虫效应分子对植物生长发育的影响。植物学报。2019;10:970。https://doi.org/10.3389/fpls.2019.00970.
- 19.
Kaloshian I,Desmond Oj,Atamian HS。抗病基因和防御反应在不相容的相互作用中。在:Jones J,Gheysen G,Fenoll C,编辑。植物线虫相互作用的基因组学与分子遗传学。纽约:斯普林克;2011. p。309-24。
- 20.
一种非典型的n -乙基马来酰亚胺敏感因子使抗线虫的Rhg1大豆存活。中国科学(d辑:地球科学)2018;https://doi.org/10.1073/pnas.1717070115.
- 21.
关键词:大豆,包囊线虫,抗性基因座,抗性基因座植物病理学。2019;109(12):2107 - 15所示。https://doi.org/10.1094/PHYTO-07-19-0225-R.
- 22.
Kud J, Wang W, Gross R, Fan Y, Huang L, Yuan Y, Gray A, Duarte A, Kuhl JC, Caplan A, Goverse A, Liu Y, Dandurand LM, Xiao F.马铃薯包囊线虫效应蛋白RHA1B是一种泛素连接酶,并利用两种不同的机制抑制植物免疫信号。公共科学图书馆Pathog。2019;15 (4):e1007720。https://doi.org/10.1371/journal.ppat.1007720.
- 23.
王志强,王志强,王志强,等。植物对线虫的基础抗性研究进展。J Exp Bot. 2016; 67:2049-61。https://doi.org/10.1093/jxb/erw005.
- 24。
关键词:花生nss - lrr基因AhRRS5,烟草,抗烟草青枯病生物技术学报2017;15(1):39-55。https://doi.org/10.1111/pbi.12589.
- 25。
Kochetov AV, Titov SE, Kolodyazhnaya YS, Komarova ML, Koval VS, Makarova NN, Ilyinskyi YY, Trifonova EA, Shumny VK。携带脯氨酸脱氢酶反义抑制基因的烟草转化子具有较高的脯氨酸含量和细胞质渗透压的特点。中国科学(d辑:地球科学)2004https://doi.org/10.1023/B:RUGE.0000016999.53466.e1.
- 26。
Trifonova EA, Sapotsky MV, Komarova ML, Scherban AB, Shumny VK, Polyakova AM, Lapshina LA, Kochetov AV, Malinovsky VI.表达牛胰岛核糖核酸酶的转基因烟草植株对烟草花叶病毒的保护。植物细胞代表2007;26:1121-6。https://doi.org/10.1007/s00299-006-0298-z.
- 27。
细胞外核糖核酸酶基因的表达提高了烟草对黄瓜花叶病毒的抗性。BMC Plant Biol. 2016;16(supl 3):246。https://doi.org/10.1186/s12870-016-0928-8.
- 28。
李晓霞,张颖,尹玲,陆军。病原菌诱导的葡萄TIR-NB-LRR基因VaRGA1的过表达增强了葡萄植株的抗病性和耐旱耐盐性烟草benthamiana.原生质。2017;254:957 - 69。https://doi.org/10.1007/s00709-016-1005-8.
- 29.
凯西PJ,艾伦我,分配,胡须,Boddu年代,螺栓BJ, Carvalho-Silva D,克里斯滕森M,戴维斯P, Grabmueller C, Kumar N,刘Z, Maurel T,摩尔B MD医学博士Maheswari U, Naamati G,纽曼V, Ong CK, Paulini M,佩德罗H,佩里E,罗素M,麻雀H, Tapanari E,泰勒K, Vullo,威廉姆斯G, Zadissia,奥尔森,斯坦J,魏年代,Tello-Ruiz M,Ware D, Luciani A, Potter S, Finn RD, Urban M, Hammond-Kosack KE, Bolser DM, De Silva N, Howe KL, Langridge N, Maslen G, Staines DM, Yates A. Ensembl基因组2018:非脊椎动物物种的集成组学基础设施。核酸Res. 2018;46(D1): D802-8。https://doi.org/10.1093/nar/gkx1011.
- 30.
Kochetov AV, Sirnik OA, Rogozin IB, Glazko GV, Komarova ML, Shumny VK。高等植物mRNA 5 ' -非翻译区域的上下文特征。36杂志。2002;(4):649 - 56https://doi.org/10.1023/A:1019852427093.
- 31.
Kochetov Av,Sarai A.翻译多态性作为植物蛋白质常见的潜在来源拟南芥.生物信息学。2004;20:445-7。https://doi.org/10.1093/bioinformatics/btg443.
- 32.
Ventoso I,Kochetov A,Montaner D,Dopazo J,Santoyo J.在哺乳动物细胞中的ER应激反应期间广泛的翻译重塑。Plos一个。2012; 7(5):E35915。https://doi.org/10.1371/journal.pone.0035915.
- 33.
Steuernagel B, Jupe F, Witek K, Jones JD, Wulff BB。NLR解析器:快速标注植物NLR补语。生物信息学,2015,31(10):1665 - 7。https://doi.org/10.1093/bioinformatics/btv005.
- 34.
Jupe F, Witek K, Verweij W, Śliwka J, Pritchard L, Etherington GJ, Maclean D, Cock PJ, Leggett TM, Bryan GJ, Cardle L, Hein I, Jones JD。抗性基因富集测序(RenSeq)能够从已测序的植物基因组中重新注释NB-LRR基因家族,并在分离群体中快速定位抗性位点。植物j . 2013; 76:530-44。https://doi.org/10.1111/tpj.12307.
- 35.
Kochetov AV, Glagoleva AY, Strygina KV, Khlestkina EK, Gerasimova SV, Ibragimova SM, Shatskaya NV, Vasilyev GV, Afonnikov DA, Shmakov NA, Antonova OY, Gavrilenko TA, Alpatyeva NV, Khiutti A, Afanasenko OS。nbs - lrr编码基因在两种植物根转录组中的差异表达茄属植物phureja基因型具有对比的抗性Globodera rostochiensis.BMC植物BIOL。2017; 17(SOMPL 2):251。https://doi.org/10.1186/s12870-017-1193-1.
- 36。
Gavrilenko T, Antonova O, Ovchinnikova A, Novikova L, Krylova E, Mironenko N, Pendinen G, Islamshina A, Shvachko N, Kiru S, Kostina L, Afanasenko O, Spooner DA。俄罗斯国家马铃薯收藏的微卫星和形态评估。植物生态学报。2010;57:1151-64。https://doi.org/10.1007/s10722-010-9554-8.
- 37。
Gavrilenko T,Antonova O,Shuvalova A,Krylova E,Alpatyeva N,Spooner D,Novikova L.基于塑性微卫星多态性的培养土豆遗传多样性和起源。Genet Res作物Evol。2013; 60:1997-2015。https://doi.org/10.1007/s10722-013-9968-1.
- 38。
Martin M. Cutadapt从高通量测序读取中移除适配器序列。EMBnet.Journal。2011;https://doi.org/10.14806/ej.17.1.200.
- 39。
Schmieder R,Edwards R.质量控制和偏见的数据集预处理。生物信息学。2011; 27(6):863-4。https://doi.org/10.1093/bioinformatics/btr026.
- 40.
Dobin A,Davis Ca,Schlesinger F,Drenkow J,Zaleski C,JHA S,Batut P,Chaisson M,Gingeras TR。星:超快通用RNA-SEQ对齐器。生物信息学。2013; 29(1):15-21。https://doi.org/10.1093/bioinformatics/bts635.
- 41.
罗宾逊医学博士,麦卡锡DJ,史密斯GK。edgeR:用于数字基因表达数据差异表达分析的生物导体包。生物信息学,2010,26(1):139 - 40。https://doi.org/10.1093/bioinformatics/btp616.
- 42.
杜志华,周旭,凌云,张志华,苏志华。一种面向农业社区的GO分析工具。核酸学报2010;38:W64-70。https://doi.org/10.1093/nar/gkq310.
- 43.
Simão FA, Waterhouse RM, Ioannidis P, Kriventseva EV, Zdobnov EM. BUSCO:用单拷贝同源性评估基因组组装和注释完整性。生物信息学,2015,31(19):3210 - 2。https://doi.org/10.1093/bioinformatics/btv351.
- 44.
Waterhouse RM, Seppey M, Simão FA, Manni M, Ioannidis P, Klioutchnikov G, Kriventseva EV, Zdobnov EM. BUSCO从质量评估到基因预测和系统基因组学的应用。生物医学进展。2018;35(3):543-8。https://doi.org/10.1093/molbev/msx319.
- 45.
Langmead B,Salzberg S.与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9:357-9。https://doi.org/10.1038/nmeth.1923.
- 46.
肯特WJ。BLAT -类似爆破的对准工具。基因组研究》2002;12(4):656 - 64。https://doi.org/10.1101/gr.229202.
- 47.
Camacho C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K, Madden TL. BLAST+:建筑与应用。BMC Bioinf。2009;10:1-9。https://doi.org/10.1186/1471-2105-10-421.
- 48。
Finn Rd,Coggill P,Eberhardt Ry,Eddy SR,Mistry J,Mitchell Al,Potter SC,Punta M,Qureshi M,Sangrador-Vegas A,Salazar Ga,Tate J,Bateman A. PFAM蛋白质家族数据库:走向更多可持续的未来。核酸RES。2016; 44(D1):D279-85。https://doi.org/10.1093/nar/gkv1344.
- 49。
Bailey TL, Boden M, Buske FA, Frith M, Grant CE, Clementi L, Ren J, Li WW, Noble WS。MEME套件:motif发现和搜索工具。核酸Res. 2009;37: W202-8。https://doi.org/10.1093/nar/gkp335.
致谢
作者非常感谢Daria Kochetova的英文编辑工作。
关于这个补充
本文已作为一部分发布BMC植物生物学卷20增刊1,2020:第五届国际科学会议“植物遗传学、基因组学、生物信息学和生物技术”(PlantGen2019)的精选文章。该补充的全部内容可在网上找到//www.cinefiend.com/articles/supplements/volume-20-supplement-1.
资金
这项研究得到了俄罗斯科学基金会(RSF) No. 16-16-04073的资助(包括发表费用、研究设计、植物实验、转录组测序、手稿撰写)。此外,用于植物基因组和转录组分析的生物信息学软件项目获得了IC&G Kurchatov Genomics Center的资助(075-15-2019-1662;S.V.G, D.A, N.A.S.和g.v.v)。在IC&G温室设施中栽培和准备实验用植物(由ICG预算项目0324-2019-0039提供支持)。NGS测序在ICG基因组研究中心进行。
作者信息
从属关系
贡献
AVK设计了这个研究并撰写了手稿。OSA和AK为GPCN接种和抗性评价的实验部分。GVV编写库,并在NextSeq550平台上对库进行测序,参与数据解读。NVS参与了图书馆的编制和排序工作。KVS、AYG、SVG参与了RNA的提取、cDNA的制备、qRT-PCR内参基因引物的设计、qRT-PCR的性能以及手稿的起草。NAS, AAE, DAA对测序数据进行硅分析,并参与了手稿的起草。EKK和SMI对研究的设计和协调以及对手稿的严格修改做出了贡献。OYA和NVA准备了美国phureja用于GPCN接种的植物材料,进行了RNA的提取美国phureja参与数据解释工作。建议使用TAG选定的美国phureja本研究的基因型,提供了本材料并参与了本研究的协调。所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
本文已更新。最初的出版物包含了一个错误的历史日期。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
科切托夫,a.v.,埃戈罗娃,a.a.,格拉戈列娃,A.Y.等等。马铃薯抗病性的机理Globodera rostochiensis:抗性和敏感根转录组的比较茄属植物phureja基因型。BMC植物BIOL.20.350(2020)。https://doi.org/10.1186/s12870-020-02334-2
收到了:
接受:
发表:
关键词
- 茄属植物phureja
- Nematode.
- 电阻
- Globodera rostochiensis
- 转录组
- 土豆