摘要
背景
降低作物生产对化肥的依赖及其相关成本、碳足迹和其他环境问题是农业面临的一项挑战。解决这一问题需要新的解决方案,而高氮利用效率或耐低氮有效性的作物育种已被广泛认为是一种有前途的途径。然而,作物植物高氮利用效率或低氮耐受性的分子机制仍有待研究,包括长链非编码rna (long non-coding RNAs, lncRNAs)的作用。
结果
在这项研究中,我们在大麦中鉴定出498个lncrna (大麦芽)长亲本B968(柳竹头紫大麦),其中487个为新亲本,56个为低氮胁迫响应系。为了对差异表达的lncrna进行功能分析,我们分析了共表达和共定位蛋白编码基因的基因本体(GO)和京都基因与基因组百科全书(KEGG)富集情况,并进一步预测了与注释的共表达蛋白编码基因或微rna (miRNAs)的相互作用。差异表达的lncrna和mirna之间的目标拟态预测确定了40个假定的lncrna目标拟态和58个目标mirna。通过qPCR进一步验证了6个差异表达的lncrna,其中一个在两种技术中均表现出一致的差异表达。大多数lncrna的表达水平都很低,这可能是RNA-seq和qPCR数据明显不一致的原因。
结论
分析低氮胁迫下差异表达的lncrna,以及它们的共表达或共定位的蛋白质编码基因和目标模拟物,可以阐明大麦和其他作物植物适应低氮胁迫的复杂和迄今尚未确定的机制。
背景
过去半个世纪农作物生产的显著变化和进步主要是由于将矮化基因纳入育种计划和无机化肥的使用:这些发展通常被定义为“绿色革命”[1,2].然而,基于作物育种和施用化肥(特别是氮肥)的产量增长已达到一个平台期[3.]而过量使用化肥会造成严重的环境问题[4,5].另一方面,在许多发展中国家和贫困地区,缺乏化肥仍然是一个大问题。因此,必须开发和采用新的解决方案来提高产量,同时保持或减少化学氮肥的施用。因此,已经存在了几十年的高氮利用效率作物育种正变得越来越重要。
虽然氮利用效率的概念早在20世纪80年代就被提出[6],化肥的可获得性和低成本,加上作物育种中对矮化/半矮化基因的关注,意味着在改善这一重要性状方面几乎没有进展[2].此外,由于氮素利用效率高的分子机制复杂,目前尚不清楚[2,7].目前,随着化肥价格的上涨,其碳足迹和环境成本越来越难以接受,越来越多的研究人员试图解决这一问题,通过开展低氮耐受性研究,为提高作物氮素利用效率提供新的解决方案[8,9,10].
长链非编码rna (Long non-coding RNAs, lncrna)是指长度超过200个核苷酸且没有蛋白质编码潜能的rna。根据编码它们的DNA区域相对于蛋白质编码基因的位置关系,它们可以分为三种类型:长基因间非编码rna (lincRNAs);反义lncRNAs;内含子lncRNAs (incRNAs) [11,12].lncrna多年来很少受到关注,但在动物系统中发现x不活跃的特异性转录本(XIST)和下一代测序(NGS)的发展后,这种情况发生了改变[11,13].lncrna目前被认为在转录和转录后调控、组蛋白修饰、RNA加工和小RNA通路中发挥重要作用[13].LncRNAs也在不同胁迫下的许多作物中进行了研究,包括氮缺乏[10,12,14,15],但关于大麦中lncrna的相关信息很少,特别是与低氮耐受性相关。
大麦是世界上第四大谷物作物,也是谷物研究的重要模式植物。它是最早被驯化的谷物之一,由于适合麦芽,在人类文化中发挥了重要作用[16].尽管基于农民和育种者偏好的作物选择对现代作物品种产生了重大影响[17],过去大麦长地生产缺乏化肥供应可能意味着一些耐氮性低的种质被保留下来,如果将这些种质纳入育种计划,可能有利于提高氮的利用效率。因此,我们对一个长白大麦品种进行了低氮耐受性筛选,发现生长限制主要集中在胁迫初期的嫩枝上,而长白大麦品种B968(柳竹头紫大麦)表现出较强的低氮耐受性[18,19,20.].在此之前,我们已经发现即使在施加低氮胁迫后1小时,大麦芽的基因表达也会发生变化[21],而在低氮胁迫水稻中,也观察到基因调控的重要性[22].
本研究的目的是进行比较转录组分析,以鉴定长大麦B968芽对1小时低氮胁迫的lncrna。这些lncrna可能被利用来提高大麦的低氮耐受性和/或氮利用效率。长大麦B968被认为是相对耐受低氮胁迫的,因为它的茎部生物量不受低氮胁迫时期的影响,低氮胁迫导致大多数长大麦生物量减少[19,20.].即便如此,在低氮胁迫下,地上部氮(N)浓度(每g地上部干重mg N)和氮积累量(每株mg N)均显著降低,而根系生物量显著增加。这表明根和梢对低氮胁迫的响应不同,梢部生物量更直接地反映了对低氮胁迫的抗性,尽管根系生物量的增加可能有利于低氮适应[19,20.,21].
该研究首次在整个转录组水平上研究了大麦中lncrna对低氮胁迫的响应,并预测了lncrna与蛋白质编码基因或mirna之间的相互作用。
结果
lncrna的鉴定和鉴定
以正常氮供氮和低氮胁迫条件下生长的大麦长青品种B968为材料,进行cDNA文库构建和RNA-seq分析,每个样品设置2个生物重复。RNA-seq数据已存入国家生物技术信息中心:提交ID SUB6290350;生物项目ID PRJNA566107。
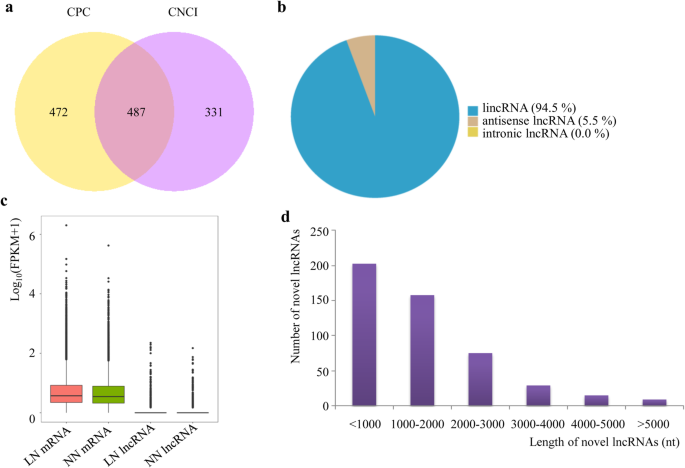
RNA-seq数据中共鉴定出498个独特的lncrna,其中487个是新的lncrna,包括460个基因间lncrna (lincrna)和27个反义lncrna(图2)。1a和b,附加文件2).它们分布在所有染色体上,2号染色体上lncrna含量最高,6号染色体上lncrna含量最低,而它们对正义或反义链没有偏好(附加文件)2).我们还比较了正常N和低N条件下lncRNAs和mrna的表达水平,发现两种处理之间lncRNAs和mrna的表达水平相似,但lncRNAs的整体表达水平低于mrna(图2)。1c).我们进一步分析了这些新型lncrna的长度,发现大多数长度都小于2000nt(图。1d)。
lncrna对低氮胁迫的响应
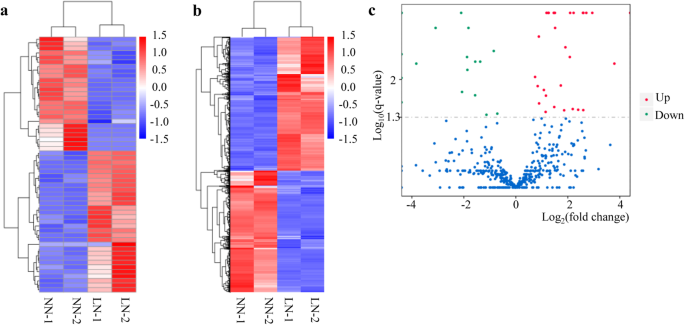
为了在大麦嫩枝中鉴定低氮胁迫响应的lncrna,比较了正常氮处理和低氮处理之间lncrna的归一化表达。聚类显示lncrna在正常N和低N样本中分为两类(图2)。2a).然后比较正常N处理和低N处理之间的归一化mrna表达,聚类也显示正常N和低N样品之间存在明显差异(图。2b). 56个lncrna(均为新lncrna)在正常氮处理和低氮处理之间表达差异,其中31个在低氮处理下表达上调,25个在低氮处理下表达下调(图2)。2c、附加文件2).低氮胁迫响应型lncrna分布在所有染色体上,2号和6号染色体上的lncrna最多。6号染色体低氮响应型lncrna比例最高(附加文件)2).
低氮胁迫响应型lncrna的功能分析
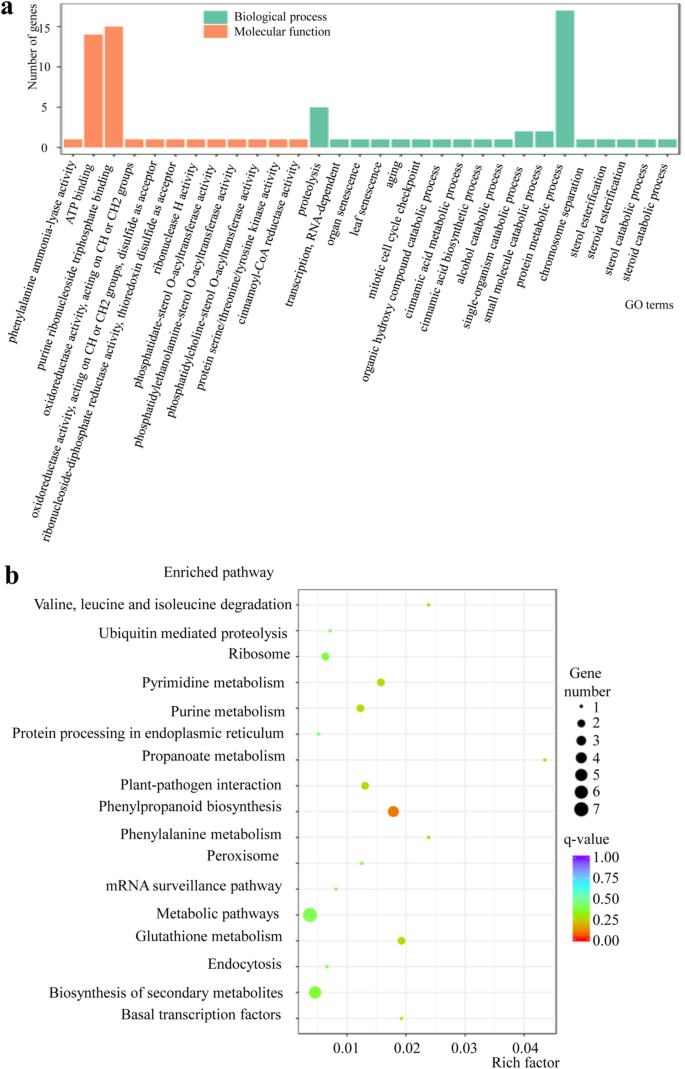
为了分析低氮胁迫响应型lncRNA的潜在功能,共定位靶基因,即位于基因组中距离差异表达lncRNA区域不到100 kb的基因,被用于GO和KEGG分析(附加文件)2).对这些共定位基因的GO分析显示,排名前30位的词条主要富集在生物过程和分子功能的类别中,尤其是ATP结合、嘌呤核糖核苷三磷酸结合和蛋白质代谢过程这3个词条,尽管这3个词条的富集程度不显著(图2)。3.a和附加文件2).KEGG富集结果表明,这些共定位基因被分配到17条KEGG通路上,但只有苯丙类生物合成通路是显著的(p-adj < 0.05)(图;3.b和附加文件2).
共表达蛋白编码基因(即与lncRNA共表达的Pearson相关系数大于0.95的基因)p值小于0.001)用于GO和KEGG分析(附加文件2).对这些共表达蛋白编码基因的GO分析表明,前30个词条主要富集在细胞成分类,且均显著(p-adj < 0.05),细胞成分种类最多(图;4a和附加文件2).KEGG分析表明,前20个富集通路均显著(p-adj < 0.05)。代谢途径、次生代谢产物生物合成和核糖体三大类富集因子最多,丙酮酸代谢、氧化磷酸化和抗坏血酸和钙酸盐代谢富集因子最高(图。4b和附加文件2).
大麦嫩枝低氮胁迫下与差异表达的lncrna共表达的蛋白编码基因功能分析一个去分析;bKEGG分析;c差异表达的lncrna和共表达的蛋白质编码基因的细胞壁网络:红色三角形代表lncrna,绿色圆圈代表蛋白质编码基因,网络的大小和复杂性反映了所涉及的相互作用的数量(附加文件)2)
为了阐明低氮胁迫下差异表达的lncrna的功能以及lncrna与蛋白质编码基因之间的关系,我们进一步利用Cytoscape软件将注释的共表达的蛋白质编码基因和差异表达的lncrna建立假定的相互作用网络(图2)。4c和附加文件2).分析表明,蛋白质编码基因与lncRNA之间的相互作用非常复杂,一个lncRNA可以与多个蛋白质编码基因相互作用,一个蛋白质编码基因也可以与多个lncRNA相互作用。显然,那些可以与许多蛋白质编码基因相互作用的lncrna,如lnc000327,可能对低氮胁迫的响应有很大的影响。
lncrna对m个iRNAs的目标拟态
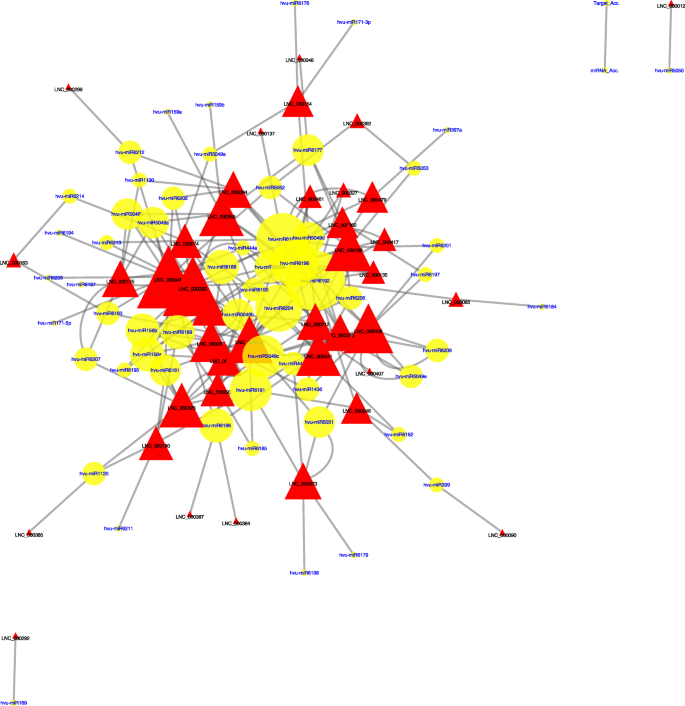
共鉴定出40个假定的lncRNA靶模拟物和58个靶miRNA,其中一个lncRNA可与多个miRNA相互作用,一个miRNA可与两个或两个以上不同的lncRNA相互作用(图2)。5和附加文件2).虽然lncrna对mirna的作用还有待进一步验证,但该预测仍然提供了对目标拟态的潜在机制的概述。
差异表达的lncRNAs和miRNAs的胞壁网络。红色三角形代表lncrna,黄色圆圈代表mirna,网络的大小和复杂性反映了所涉及的相互作用的数量(附加文件)2)
qPCR验证差异表达的lncrna
选择正常N和低N条件下读数大于30的差异表达lncrna进行qPCR验证。它们是:lnc000161, lnc000189, lnc000274, lnc000356, lnc000382, lnc000470和lnc000182,尽管lnc000182被放弃了,因为它的引物设计被证明非常困难。在其他基因中,只有lnc000470与RNA-seq结果一致,在低氮条件下表达上调(图4)。6a和b)。lncrna的表达水平远低于内参基因,与lncrna的预期一致(图。1c),除lnc000161外,lnc000161在两种处理中均较高表达(图。6a). lncrna的低表达可能是导致qPCR和RNA-seq结果明显差异的原因,可能需要开发替代qPCR的方法来验证lncrna的表达。尽管如此,lnc000470的qPCR结果验证了RNA-seq数据,以及其相互作用的蛋白质编码基因和mirna,是进一步研究的良好候选者。
讨论
在非常高的含氮条件下提高氮的利用效率可能非常困难,甚至不可能[2,因此,专注于提高对低氮或氮饥饿胁迫的抵抗力的机制被认为是提高氮利用效率和适应贫瘠土地的更好选择。一个成功的例子是在水稻中发现了早期结瘤蛋白基因,该基因是在低氮胁迫条件下通过转录分析确定的,与氮利用效率的提高有关[8].该基因的发现及其与提高低氮胁迫抗性和更好的氮利用效率的关系清楚地证明,相关研究不应局限于直接参与氮代谢的基因。与此同时,大麦因其对贫瘠土地的良好适应性,正成为类似研究的首选模式作物,特别是随着最近新的测序技术的发展[9,23,24].
在本研究中,我们首次在转录组水平上鉴定出487种新型大麦lncrna,其中大多数组成核苷酸少于2000个,与棉花的情况很相似[14].此外,在低氮胁迫下,56个lncrna在长白种人B968中差异表达,因此可能在低氮胁迫响应中发挥重要作用,其中55个被归类为lincrna。对于响应低氮胁迫的lncrna的功能分析,我们主要关注其与蛋白编码基因和mirna的关系。对于共定位蛋白编码基因,氧化石墨烯分析中没有显著富集项,只有苯丙类生物合成一条通路显著富集。值得注意的是,在低氮环境下,西藏野生大麦中苯丙类生物合成途径也得到了丰富[9].这表明,与苯丙烷生物合成相关的基因可能在大麦对低氮胁迫的响应中起重要作用。在这项分析中,我们设置了100 kb的分离距离限制,这可能解释了为什么相对较少的基因被鉴定出来,只有一条通路被显示出富集。
相比之下,共表达蛋白编码基因的分析发现了大量的基因,GO分析显示富集前30位的词汇都是显著的,大部分属于细胞成分的范畴。KEGG分析还显示,所有前20个富集途径都是显著的。这表明功能术语或途径与低氮胁迫的响应广泛相关,与之前的研究一致[9,22,23,25].为了更直观地描述差异表达的lncrna和共表达的蛋白编码基因之间的关系,细胞景观网络清晰地识别了枢纽差异表达的lncrna,该网络也可以帮助我们研究不同枢纽lncrna及其靶向蛋白编码基因对低氮胁迫的抗性。
拟南芥中首次提出了目标拟态,并将一个lncRNA称为IPS1被鉴定出可以与miRNA (miR399)结合以防止其目标mRNA的降解,PHO2,以控制Pi稳态[26].lncrna的这一重要功能不太可能局限于磷酸饥饿[10,13,27].本研究中确定的40个假定的lncrna靶模拟物可能用于研究它们对mirna的调控及其靶mrna,以揭示更复杂的适应低氮胁迫的机制。此外,我们还发现lnc00090和lnc000248是hvu-miR399的模拟靶标,这表明miR399在大麦低氮胁迫下也可能发挥重要作用。
结论
该研究表明,分析低氮胁迫下差异表达的lncrna,以及它们的共表达或共定位的蛋白编码基因和目标模拟物,可以阐明大麦和其他作物植物适应低氮胁迫的复杂且迄今尚未确定的机制。
方法
植物材料和低氮处理
大麦长种B968是一组大麦基因型之一,最初作为种子从上海农业生物基因中心获得,并由陈志伟和蒋琦保存在上海农业科学院生物技术研究所。长白地植物B968在人工气候室中生长,生长条件如Chen等所述。[21].NH4没有3.为氮源,设两种氮条件:正常氮(N)(对照),NH为1.43 mM4没有3.,低氮胁迫,NH 0.24 mM4没有3..低氮处理从幼苗发育3叶阶段开始(在此阶段之前,我们认为种子胚乳仍可向生长中的幼苗提供氮)。用于转录组分析和lncRNA表达验证的嫩枝在两种氮条件下处理1小时后取样,并保存在−80°C的冰箱中。每个样本2个重复进行转录组分析,每个样本3个重复进行qPCR分析。为了进行性状研究,根据Chen等人的研究,在两种氮处理条件下,幼苗在处理2周后收获。[20.,21].
lncRNA文库制备及rna测序
使用TRIzol试剂(Invitrogen, USA)从每个大麦芽样品中分离总RNA。用1%琼脂糖凝胶电泳监测RNA降解和污染情况;使用nanoophotometer分光光度计(IMPLEN, CA, USA)检查纯度;使用Qubit RNA检测试剂盒和Qubit 2.0荧光计(Life Technologies, CA, USA)测量浓度;完整性使用Bioanalyzer 2100系统(Agilent Technologies, CA, USA)的RNA Nano 6000检测试剂盒进行评估。每个样品的总RNA总量为3 μg,作为RNA样品制备的输入材料,核糖体RNA使用Epicentre riboo -zero rRNA去除试剂盒(Epicentre, Madison, WI, USA)去除。然后使用NEBNext®Ultra™Directional RNA Library Prep Kit for Illumina (NEB, USA)根据制造商的说明,使用rrna耗尽的RNA生成测序文库。文库在Illumina Hiseq Xten平台(Illumina Inc., San Diego, CA)上测序,生成150 bp的配对末端reads。干净的核苷酸序列数据范围为16.14 ~ 19.64 Gb(均为> 12 Gb), Q30百分比均为> 95%(表2)1,附加文件1,图A1)。这些结果表明,所得数据充分可靠,可供进一步分析。Spearman相关分析也表明,每个样品的两个生物重复均满足要求(均大于0.8)(附加文件)1,图A2)。
新型lncRNA鉴定
通过从原始数据中删除包含适配器序列或poly-N的读取和其他低质量的读取来获得干净的数据。计算Q20、Q30和GC信息,对洁净数据进行评价。参考基因组和基因模型注释文件已从EnsemblPlants (http://plants.ensembl.org/Hordeum_vulg/Info/Index, IBSC v2)。使用Bowtie v2建立参考基因组索引,使用TopHat v2.0.9将干净的reads与参考基因组对齐。每个样本的映射reads通过Cufflinks (v2.1.1)以参考注释为基础的转录本(BRAT)方法组装[28,29].通过6个步骤鉴定新型lncrna: 1)选择外显子数≥2的转录本;2)选择长度为> 200 bp的转录本;3)选择袖扣计算覆盖> 3的成绩单;4)通过Cuffcompare去除已知mrna(蛋白质编码)或ncrna的转录本;5)选择FPKM (fragments per kilobase of transcript per million mapped reads, FPKM)表达量≥0.5的转录本;6)使用CPC(编码潜能计算器)(0.9 - r2)检测非编码潜能的转录本[30.]及CNCI(编码非编码索引)(v2) [31].
lncrna的差异表达
使用Cuffdiff v2.1.1提供统计例程,用于使用基于负二项分布的模型确定数字转录本或基因表达数据中的差异表达[28].成绩单带p-形容词值(调整p值)< 0.05为显著差异表达。
GO和KEGG分析
为了预测低氮胁迫响应型lncrna的功能,分别通过GOseq R包对差异表达的lncrna共定位或共表达的蛋白编码基因进行基因本体(GO)富集分析,GO项为ap-adj值小于0.05为显著富集。京都基因及基因组百科全书(http://www.genome.jp/kegg/)通路富集分析采用KOBAS软件进行。
共表达蛋白编码基因与lncRNA相互作用
注释共表达蛋白编码基因,Pearson相关系数大于0.99p值小于0.001,差异表达的lncrna使用Cytoscape进行假定的交互网络预测。
M iRNA和lncRNA相互作用
所有大麦mirna均从Mirbase 22下载(http://www.mirbase.org/)和所有显著差异表达的lncrna被psRNATarget用于目标拟态预测。预测目标模拟的准则和原则主要基于Deng等人[14]和吴等人。[27].利用Cytoscape将差异表达的lncrna及其靶mirna用于假定的交互网络预测。
实时定量PCR和统计学验证
用TRIzol试剂(Invitrogen,美国)分离总RNA,用无rnase - DNase I (Promega,美国)处理。使用PrimeScript®RT reagent Kit (TaKaRa, Japan)将约1 μg RNA反转录为第一链cDNA。定量实时PCR (qRT-PCR)使用SYBR®Select Master Mix和ABI 7500 Fast仪(Applied Biosystems, USA)进行。qRT-PCR反应对每个cDNA样本进行3次重复,退火温度为60℃,共扩增40次,每次反应含5 μL 2 × mix,每个引物(10 mM) 0.6 μL, 10 ×稀释cDNA模板1 μL,终体积为10 μL。用于lncrna和内参基因的引物在附加文件中列出2.lncrna的引物由NCBI网站上的Primer-BLAST设计,内参基因的引物直接取自Chen等人。[24];采用LinRegPCR软件检测PCR效率。各lncRNA归一化相对数量根据Chen等计算[32]以及Rieu和Powers [33],并通过LinRegPCR软件获得Cq值。计算使用三个内参基因:HvGAPDH(glyceraldehyde-3-phosphate脱氢酶),HvARF1(adp -核糖基化因子1样)和HvTUBB6(β微管蛋白6)。这是在低氮胁迫下大麦嫩枝中发现的三个最稳定的内参基因[24].统计分析主要依据Chen等[32使用转换后的Cq值[Cq = log .2(1 / NRQ)]。低氮胁迫与对照基因表达差异采用0.05水平的t检验(p< 0.05)(附加文件1).
数据和材料的可用性
RNA-seq数据已存入国家生物技术信息中心:提交ID SUB6290350;生物项目ID PRJNA566107。
缩写
- CNCI:
-
编码非编码索引
- 中国共产党:
-
编码势计算器
- Cq:
-
定量循环
- FPKM:
-
每千碱基的片段数
- 走:
-
基因本体论
- KEGG:
-
京都基因与基因组百科全书
- lncRNA:
-
长链非编码RNA
- lincRNA:
-
长基因间非编码RNA
- incRNA:
-
内含子非编码RNA
- microrna的:
-
微RNA
- 门店:
-
下一代测序;
- qPCR:
-
定量聚合酶链反应
- p-adj:
-
调整p价值
- RNA-seq:
-
RNA序列
- XIST:
-
非x活性特异转录本
参考文献
- 1.
那个宿舍叫赖茨LP。新小麦与社会进步:改良小麦品种有助于实现前所未有的高水平粮食生产。科学。1970;169:952-5。
- 2.
Han M,冈本M, Beatty PH, Rothstein SJ, Good AG。作物植物氮素利用效率的遗传。《热内年鉴》2015;49:1-9。
- 3.
Fischer RA, Byerlee D, Edmeades GO。技术能解决2050年的产量挑战吗?入:Conforti P,编辑。世界粮食和农业展望:2050年展望。罗马:联合国粮食及农业组织(粮农组织);2011.p . 389 - 462。
- 4.
好的AG, Beatty PH.肥沃的自然:公地过度的悲剧。公共科学学报,2011;9:e1001124。
- 5.
张强,何坤,霍宏。净化中国空气。大自然。2012;484:161-2。
- 6.
Moll RH, Kamprath EJ, Jackson WA。对影响氮素利用效率的因素进行分析和解释。Agron J. 1982; 74:562-4。
- 7.
徐刚,范X,米勒AJ。植物氮同化与利用效率。安。植物科学进展(英文版)。2012;
- 8.
毕ym, Kant S, Clark J, Gidda S, Ming F, Xu J, Rochon A, shelbj, Hao L, Zhao R, Mullen RT, Zhu T, Rothstein SJ。从水稻表达谱中鉴定的氮响应性早期结节蛋白基因,在过度表达的转基因水稻植株中提高了氮利用效率。植物细胞环境学报。2009;32:1749-60。
- 9.
全鑫,曾娟,叶玲,陈刚,韩忠,沙吉明,张刚。两个西藏野生大麦基因型对低氮响应的转录组分析。BMC植物生物学2016;16:30。
- 10.
申雪雪,郑俊杰,林志勇,金涛,朴志华,金建奎,申昌。水稻转录组分析(栽培稻)基因和非编码rna在氮饥饿下使用多重组学技术。BMC Genomics. 2018;19:532。
- 11.
马玲,Bajic VB,张震。长链非编码rna的分类。生物工程学报。2013;10:924-33。
- 12.
王慧,牛庆文,吴宏文,刘健,叶健,于宁,蔡新华。对水稻和玉米非编码转录组的分析揭示了与农业性状相关的保守lncRNAs的作用。植物学报(英文版);
- 13.
刘杰,王辉,蔡新华。植物长非编码RNA转录组。植物科学进展。2015;13:319-28。
- 14.
邓峰,张旭,王伟,袁锐,沈峰陆地棉长链非编码rna (lncRNAs)在盐胁迫下的表达。BMC植物生物学,2018;18:23。
- 15.
陈玲,石松,姜宁,Khanzada H,瓦桑GM,朱超,彭鑫,徐娟,陈勇,余强,何霞,付军,陈霞,胡玲,欧阳林,孙霞,何红,边娟。水稻镉胁迫响应早期根系发育的长链非编码rna全基因组分析。BMC Genomics. 2018;19:460。
- 16.
马舍尔M, Gundlach H, Himmelbach A,等。一个染色体构象捕获大麦基因组的有序序列。大自然。2017;544:427-33。
- 17.
Duvick DN。育种对玉米产量提高的贡献(玉米l .)。Adv Agron. 2005; 86:83-145。
- 18.
陈忠文,卢荣军,邹林,杜志忠,高红红,何涛,黄建辉。上海地区大麦地方品种和栽培品种遗传多样性分析。Genet Mol res 2012; 11:644-50。
- 19.
蒋强,陈忠,刘超,何涛,郭刚,高锐,徐辉,李勇,卢荣,黄娟。大麦地方品种苗期耐低氮性筛选与鉴定指标。华北农业学报。2019; 34:148-55。
- 20.
陈震,蒋强,徐娟,张伟,何婷,郭刚,王婷,马勇,黄娟,刘超,卢荣。不同低氮胁迫对大麦地方品种苗期低氮耐性的影响。植物物理学报,2019;49:442 - 448。
- 21.
陈志,刘超,王勇,何涛,高锐,徐辉,郭刚,李勇,周玲,卢荣,黄娟。不同大麦品种苗期氮代谢相关基因表达分析揭示了低氮胁迫下两种大麦品种的适应差异。中国生物医学工程学报。2018;2018:1 - 10。
- 22.
Coneva V, Simopoulos C, Casaretto JA, El-Kereamy A, Guevara DR, Cohn J, Zhu T, Guo L, Alexander DC, Bi YM, McNicholas PD, Rothstein SJ。与水稻硝酸盐反应相关的代谢和共表达网络分析。BMC Genomics. 2014;15:1056。
- 23.
魏志,曾晓,秦超,王勇,白龙,徐强,袁红,唐勇,尼玛婷。通过比较转录组分析发现了西藏无皮大麦叶片中常见的硝酸盐胁迫响应基因。前沿植物科学,2016;7:1067。
- 24.
陈震,李颖,刘超,王艳,何婷,郭刚,方超,高锐,徐辉,周玲,黄娟,Halford NG, Lua R.基于RNAseq数据的大麦低氮胁迫定量RT-PCR归一化内参基因筛选。中国农业科学,2018;
- 25.
毕玉梅,王瑞林,朱涛,Rothstein SJ。全局转录谱揭示了拟南芥对慢性氮胁迫的不同响应和假定的氮调控成分。BMC基因组学,2007;8:281。
- 26.
Franco-Zorrilla JM, Valli A, Todesco M, Mateos I, Puga MI, Rubio-Somoza I, Leyva A, Weigel D, García JA, Paz-Ares J.靶标拟态为microRNA活性调控提供了一种新的机制。Nat Genet, 2007; 39:1033-7。
- 27.
吴海军,王志明,王敏,王新军。广泛的长非编码rna作为植物microrna的内源性目标模拟物。植物科学进展。2013;
- 28.
Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ, Salzberg SL, Wold BJ, Pachter L. RNA-Seq转录本组装和定量揭示了细胞分化过程中未注释的转录本和异型转换。生物技术学报。2010;28:11 - 5。
- 29.
Cabili MN, Trapnell C, Goff L, Koziol M, Tazon-Vega B, Regev A, Rinn JL。人类大型基因间非编码rna的综合注释揭示了整体性质和特定的子类。基因开发2011;25:15 15 - 27。
- 30.
孔玲,张勇,叶志强,刘晓乔,赵树清,魏玲,高光。CPC:利用序列特征和支持向量机评价转录本的蛋白质编码潜力。核酸决议2007;35(Web服务器问题):W345-9。
- 31.
孙玲,罗红,卜东,赵刚,于凯,张超,刘勇,陈锐,赵艳。利用序列本征组成对蛋白质编码和长非编码转录本进行分类。核酸研究,2013;41:e166。
- 32.
陈忠,黄娟,Muttucumaru N, Powers SJ, Halford NG。脱落酸(ABA)和代谢信号因子在大麦胚乳和胚发育中的表达分析。中国粮食科学,2013;58(2):255-62。
- 33.
Rieu I, Powers SJ。实时定量RT-PCR:设计、计算和统计。植物细胞。2009;21:1031-3。
确认
不适用。
资金
本文由中国农业科学研究系统(No.;car -05- 01a -02),上海市自然科学基金项目(No. 17ZR1425300),国家自然科学基金项目(No. 31601295)。英国生物技术和生物科学研究委员会(BBSRC)通过“设计未来小麦计划”(BB/P016855/1)在洛桑研究所支持了该项目。这些资助者在研究的设计、数据的收集、分析或解释,或手稿的写作中没有任何作用。
作者信息
从属关系
贡献
QJ进行植物生长、处理和样品制备;ZC和QJ制备RNA样品;ZC和WZ进行qPCR实验;ZC和PJ对数据进行了生物信息学和统计学分析;ZC和NGH撰写稿件;CL, RL和JH是项目负责人。作者们阅读并批准了手稿。
作者的信息
不适用
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
陈志伟和蒋琪是共同第一作者。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
陈震,蒋启刚,蒋平。et al。大麦品种B968幼苗期低氮胁迫响应长链非编码rna (lncRNA)BMC植物生物学20.142(2020)。https://doi.org/10.1186/s12870-020-02350-2
收到了:
接受:
发表:
关键字
- 长非编码rna
- 大麦
- 大麦芽
- 低氮胁迫
- 氮利用效率
- RNA-seq