摘要
背景
根际微生物群落是动态的,受病原入侵等周围环境因素的影响。我们研究了Ralstonia solanacearum用高通量测序和GC-MS技术对根际微生物组和代谢物的病原体丰富。
结果
两种流根间细菌群落的较高或降低的病原体丰度之间存在显着差异,并且微生物体的这种差异甚至忽略了病原体的存在。较高的病原体丰度降低了alpha.根际细菌群落的多样性以及共同发生网络的联系。几个细菌群体如芽孢杆菌和几丁根与病原体丰富呈负相关。GC-MS分析显示两组根际土壤中的显着不同的代谢物,即下毛的根际土壤,如果糖,蔗糖和青霉素,如高病原体丰富。
结论
两根际土壤中不同的代谢组可能解释了Mantel试验中细菌群落的差异。芽孢杆菌和几丁根糖类化合物与病原菌丰度呈负相关,表明其潜在的生防能力。
背景
与宿主植物相关的微生物群,尤其是根,确定土壤传播病原体的感染[29.].组装一个自我服务的根际微生物群对植物和病原体都是至关重要的。植物吸收有益的微生物来刺激植物生长,引发植物系统防御,并拮抗病原体[3.,32.].这种招聘是通过释放特异性化合物的释放,并通过植物中的激素表达水平的改变来实现[4,35.].对于植物病原体,通过根渗出物的宿主植物的识别是成功入侵的第一步[7,12.];此外,抑制宿主防御和获取必需营养素也控制了入侵事件[7,15.,44.].病原体之间或病原体与其他微生物之间的相互作用可能通过物质交换或效应器基因表达增强来促进致病性[16.,31.].这些研究表明,在发病过程中有一个病原体助手。通过对病原菌入侵后根际微生物群的研究,可以了解寄主植物与病原菌的相互作用以及病原菌与其他微生物的关系。
土壤微生物群落受土壤理化性质、pH值、温度和土壤水分等非生物因素的因果影响[13.,25.].而土壤代谢组对土壤微生物的影响,如糖、有机酸和其他低分子量化合物,作为土壤微生物的有效养分,负责植物与根际微生物的相互作用,却很少受到关注[26.].根出渗出物是根际土壤低分子量分子的主要来源,并且根部渗出物组合物的变化会影响根际土壤微生物群落的组成[17.,26.,48.].宿主植物的土壤养分状况和健康状况也可以通过影响根部渗出物来改变根际微生物群的组成[30.].
物种入侵通过与本地微生物的资源和生态位竞争,对原始生态系统产生了强烈的影响[43.].在根际土壤中,病原菌入侵也可以通过间接影响宿主而影响微生物群落[38.,41.,例如,Berendsen等[4)表明,拟南芥蒂利亚纳在霜霉病病原体的叶面防御激活时明确促进根际的三种细菌种类Hyaloperonospora Arabidopsidis..作为革兰氏阴性菌植物病原性,Ralstonia solanacearum导致全球性的青枯病[51,52,53].它显示出在入侵木马血管后蔓延到地上的快速容量[54,55,56]然后过量产生细胞外多糖以阻断水流[57,58].
在这项研究中,根际土壤样品从7岁的单一种植温室特点是高丰度的收集r . solanacearum在前面的果实阶段之前有100%的疾病发病率。将番茄植物与土壤一起在延迟发育阶段进行采样。通过Q-PCR检测总病原体密度,通过GC-MS技术测定土壤的低分子量分子,并且使用高通量测序来确定根际细菌群落的组成的变化。我们设置了这项研究以回答两个问题:(1)r . solanacearum入侵决定根际微生物群落组成?(2)根际土壤低分子量分子是否对根际微生物群落组成的变化起作用?
结果
丰富的Ralstonia solanacearum在番茄根际
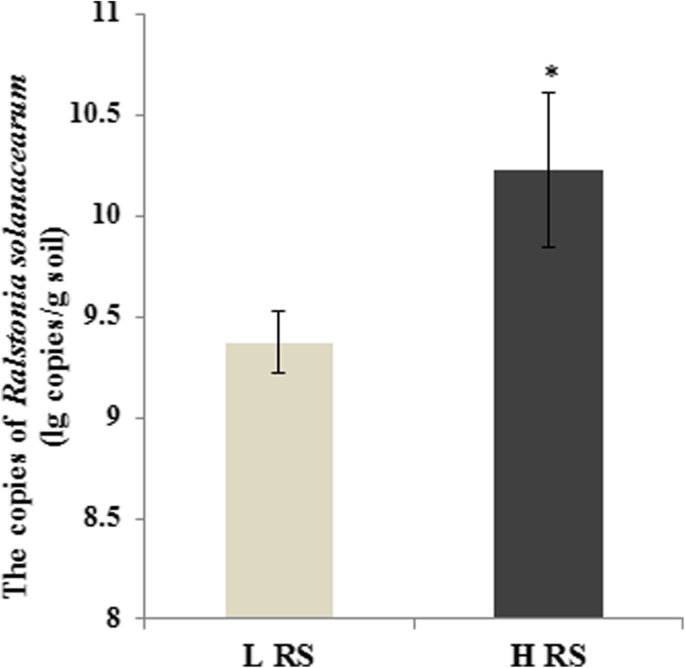
我们选择了12种土壤样品并将它们分成了更高的RS丰度组(HRS)和低级丰度组(LRS),因此每组6重复。丰富的卢比显着不同(p< 0.05, t检验)。1).利用Illumina Hiseq技术对12个样品进行16S rRNA测序,揭示根际细菌群落结构。
细菌群落组成
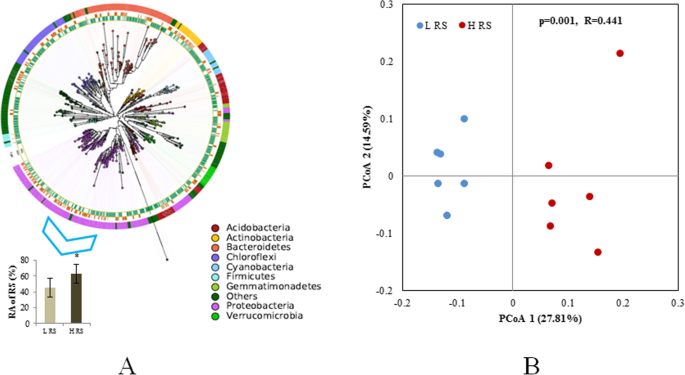
所有样品的平均读数为15,143(标准偏差(SD):3416)。OTU的数量可以反映细菌群落的多样性,每种样品的213和410之间的oTus范围的数量平均为340(SD:59)。相对丰度最高的前四个字段是proteobacteria.(76.72%),伯啉(13.63%),蓝藻(4.36%),和actinobacteria.(1.98%)(图。2a).主坐标分析(PCoA)评价的两个群落组成差异明显,相似度分析(PERMANOVA)评价的群落组成差异有统计学意义(p = 0.001, R2 = 0.44) (Fig.2b).对这些细菌群落进行仔细检查,发现一个OTU属于拉尔塞尼亚是占主导地位的proteobacteria.(无花果。2a)。测序数据拉尔塞尼亚与定量数据的结果相似,HRS组的相对丰度显著高于LRS组(图2)。2a).排除后拉尔塞尼亚,两组间差异仍有统计学意义(p = 0.001在PERMANOVA,补充图。1).
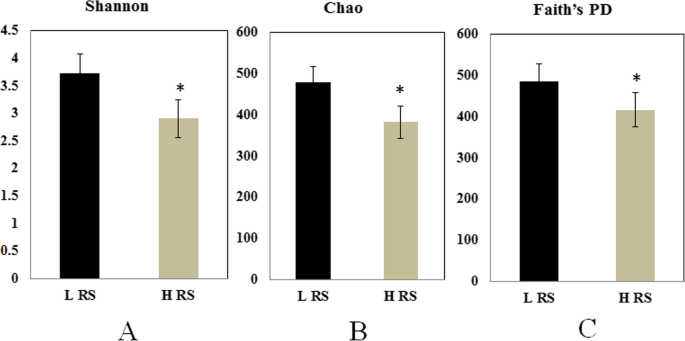
的alpha.多样性指标计算考虑所有观测到的OTUs,丰度高拉尔塞尼亚减少了alpha.与LRS组相比,HRS的根际细菌群落的多样性(图。3.).
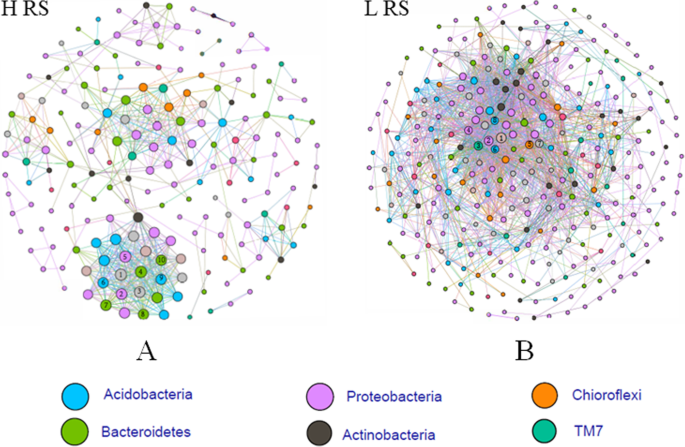
比较根际细菌共生网络的变化
为了进一步了解病原菌入侵对细菌群落的影响,我们通过对高、低两种细菌分类图谱的相关性分析,构建了细菌共生网络拉尔塞尼亚番茄的丰度根际(图。4).网络之间的网络不同。一般来说,丰富的丰富拉尔塞尼亚导致连接数量的清晰减少。分别有197和310个节点,分别为HRS和LRS组666和2153个链接。该网络分析的其他全局统计数据列于补充表中1.两种网络的主导枢纽存在一定的差异,具体来说,细菌的主导枢纽是什么Paenibacillus,Flavisolibacter,Chitinophagaceae,Sphingobacteriales,MND1,独立,红螺菌科和GOUTA19在HRS根际Xiphinematobacter,Xanthomonadaceae,III1-15,H39,EW055,MND1和DS-18在LRS根际。的拉尔塞尼亚只与一个典型的典型归类为联系Kaistobacter,这可能是与之相关的重要细菌组拉尔塞尼亚入侵。
rs(HRS)样本高丰度的根际细菌群体的共进发生网络分析(一个)及低丰度的遥感样本(b).基于分类图谱的相关分析,构建了番茄根际细菌共生网络。在显著(P< 0.01;Spearman秩相关检验)与高度(Spearman’s r > 0.96)相关。根据网络中的连接数,对每个网络的Hub细菌进行排名。对于(A) 1): O_Solibacterales;2): G_Flavisolibacter;3): G_Paenibacillus;4): F_Chitinophagaceae;5): O_Sphingobacteriales;6): O_MND1;7): O_Solibacterales; 8): O_Sphingobacteriales; 9): G_GOUTA19; 10): F_Rhodospirillaceae. (B) 1): O_DS-18; 2): O_iii1–15; 3): Unassigned; 4): G_Candidatus Xiphinematobacter; 5): O_H39; 6): O_EW055; 7): O_MND1; 8): F_Xanthomonadaceae
与病原菌抗性相关的潜在细菌分类
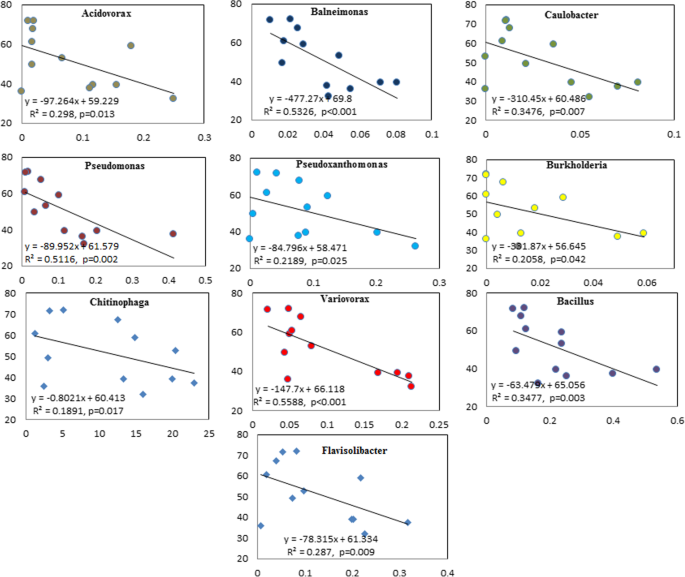
调查涉及的病原体增殖的潜在细菌群,我们进一步筛选在属水平的相对丰度。似乎与他们的相对丰度病原体大量负相关,和他们的相对丰度10属均在LRS组显著高于HRS组(图5).例如,几丁根明显(p < 0.05, t-test) higher in LRS group than in HRS group with the relative abundance of 17.95 and 4.6%, respectively.
根际土壤代谢产物特征及其与细菌群落组成的关系
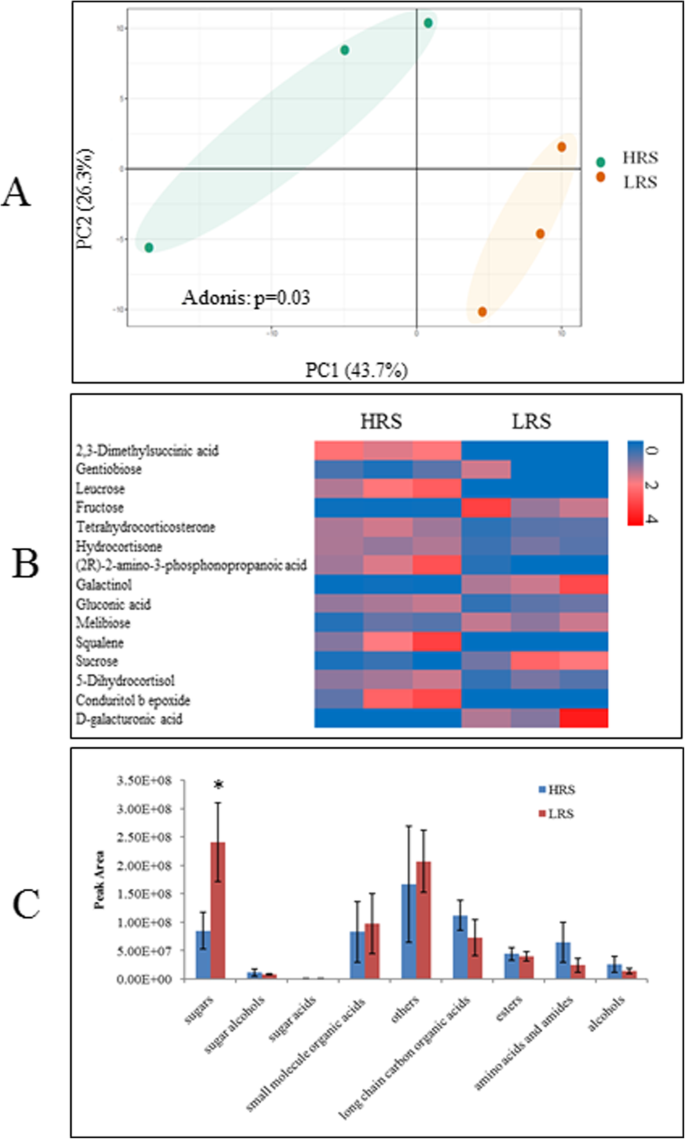
为了建立病原菌丰度对根际土壤微生物群影响的机理解释,采用气相色谱-飞行时间质谱(GC-TOF-MS)对高、低RS丰度根际土壤代谢产物进行了采集和分析。基于694个检测峰的主成分分析(PCA)揭示了LRS和HRS处理根际土壤代谢产物(AMOVA、p< 0.05,无花果。6a).根据分子结构,694个检测峰中有249个被分为8类,包括糖醇(8个)、糖(38个化合物)、糖酸(4个)、低分子量有机酸(31个)、长链有机酸(37个)、酯(13个)、酰胺(40个)、氨基酸、醇(22个)或其他(56个)。差异分析显示,其中16种化合物在两组间差异显著(t检验,p < 0.05)(图2)。6b).在上述分类水平上评价,LRS处理中糖和小分子有机酸含量较高,但只有糖存在显著性差异(t-检验,p < 0.05)(图4)。6c)。总体而言,来自LRS集团的根际土壤覆盖着更丰富的糖,尤其是果糖,蔗糖和Melibiose。
Mantel试验表明根际土壤代谢物和细菌系统之间的正相关性和显着相关性(人:r = 0.44,p= 0.006)。基于偏cca的变异分配分析(VPA)表明,根际土壤代谢物对17.2%的变异进行了解释。
讨论
本文报道了番茄根际微生物群落的变化与病原菌丰度的高低有关r . solanacearum.结果表明,根际病原菌含量显著影响细菌群落组成和多样性。相对丰度伯啉在卢比内部大大统治proteobacteria..当番茄根际感染时发生了类似的趋势P. Parasitica.[21.].通常,根际土壤中的低微生物多样性有利于病原体侵袭[24.].的确,在我们的研究中,病原菌入侵的成功降低了微生物的多样性,Shannon, Chao和Faith的PD指数在高RS丰度样品中显著下降,这表明病原菌入侵破坏了宿主相关的微生物群落[43.].主要坐标分析表明,病原体丰度的改变改变了细菌群落的组成,并在去除Rs的OTU后进一步支持了这一结论。几种有益的属性的丰富(假单胞菌,洋葱,和芽孢杆菌)与病原体呈负相关。据报道,这些有益属的种类通过产生抗微生物二次代谢物和挥发性有机化合物来发挥重要作用,用于控制抑制土壤中的土壤中疾病。6,26.,34.,45.].
有益微生物和病原体丰度的相关分析已被用于在一些出版物中识别潜在的抗病原体微生物[9,28.].然而,我们不能认为它们只有通过简单的相关性分析,对于几乎所有的分类,由于病原体较高,几乎所有的分类群几乎所有的分类都相对较低。需要进一步调查来设计有效的筛选以分离抗议性拉尔塞尼亚微生物。
根际微生物间的相互作用不仅对群落的稳定具有重要意义,而且对寄主的发育也具有重要意义。一般来说,社区内的联系越多,表明其具有抑制病原体入侵的能力[39.,43.].在这项研究中,发现高级丰度摧毁了番茄根际的细菌联系。r . solanacearum相对丰富的相对丰富显示出与其他微生物的最小连接。只有一个otu归类为Kaistobacter在HRS和LRS样本中均与RS相关。Kaistobacter据报道,抑制土壤中更普遍[18.,20.,23.],它是潜在的PGPR组,因为它可以产生激素[19.].共同发生网络分析表明,RS入侵影响了根际微生物网络,此外Flavisolibacter与Rs是负相关的HRS网络中的一个重要枢纽。两者Kaistobacter和Flavisolibacter能从生物学上控制由青枯病引起的青枯病吗r . solanacearum但是,这一假设需要进一步验证。
直接影响植物健康和抗病原体的根际微生物群[27.,33.]被根际土壤代谢物掌握[26.,47.].根除渗透依赖于许多非生物和生物因子,前者包括病原体侵袭,尤其是殖民化。例如,尖孢镰刀菌殖民化可以刺激酚醛酸的分泌[46.].叶面病原体侵染增加了根分泌物中某些氨基酸和脂肪酸的浓度[47.].虽然直到现在,虽然略微了解受病原体影响的根际土壤代谢产物。我们的研究表明,与较低的病原体丰度样品相比,番茄根际的较高病原体丰度显着改变了土壤代谢物型材。低RS样品中丰富的糖表明高糖没有有益r . solanacearum增长。这可能是因为糖,如果糖和蔗糖,不是碳源r . solanacearum[44.].事实上,低RS根际样品中富含果糖和蔗糖。氨基酸与r . solanacearum入侵[15.,因为在高海拔地区根际土壤样品中相对浓度高于低海拔地区根际土壤样品。其他化合物与病原菌丰度呈显著正相关或负相关,但其对病原菌定殖的作用有待进一步研究。
方法
实验说明和土壤取样
番茄的温室实验(“石吉 - 宏 - 冠”(生产ID:1880518)是从九泉泉种子有限公司购买的中国商业品种。,Ltd,Ltd,尚未存放在任何公共植物标目中)进行在南京蔬菜科学研究所(31°43'N,118°46'e),中国南京,中国南京,是一个7岁的单一养,用于研究番茄细菌枯萎病。对于这项研究,番茄植物于2017年3月22日至20日,2017年6月20日,并在2017年6月7日收获之前收集了根际样品。详细介绍了番茄细菌枯萎症状的视觉观察,从实验现场之前,从实验部位出发,从实验现场进行健康和弱西红柿的根(9次重复)。然后,将所有根样品无菌转移到储存袋中并在立即运输到实验室之前保持在冰上。根际土壤样品根据FU等[14.].将番茄根系上黏附松散的土壤摇掉丢弃,在无菌条件下,用无菌手术刀将番茄根系组织及其根际土壤切成1cm的段。用无菌水冲洗与番茄根段紧密结合的土壤。为了收集低分子量的分子,取出一半悬浮液进行冻干。其余标本15,000 g离心20 min,用沉淀提取DNA。
土壤基因组DNA提取Ralstonia solanacearum量化
随后,根据制造商的方案,使用超净土壤DNA分离试剂盒(MoBio Laboratories Inc., Carlsbad, USA)提取根际土壤样品的总DNA。每个样品采用2个技术重复,以尽量减少DNA提取偏倚,同时由于不可控DNA污染,也进行了阴性对照[36.,42.].Samples were stored at − 20 °C, and performing polymerase chain reaction was performed with the pooled DNA samples of technical replicates. The NanoDrop ND-2000 spectrophotometer (NanoDrop, ND2000, Thermo Scientific, 111 Wilmington, DE) was used to assess all DNA sample quality based on the 260/280 nm and 260/230 nm absorbance ratios. The concentration of extracted DNA ranged between 40 ng/μl and 60 ng/μl.Ralstonia solanacearum用靶向的引物通过定量PCR测定丰度警察编码鞭毛亚基的基因(正向引物:5 ' -GAA CGC CAA CGG TGC GAA CT-3 '和反向引物:5 ' -GGC GGC CTT CAG GGA GGT C-3 ')在Applied Biosystems StepOne Plus (Applied Biosystems, CA, USA) [37.].标准曲线的生成和qPCR检测是按照我们之前发表的文章进行的[49.].基于Rs的丰度分成两组样品(Ralstonia solanacearum):高丰度和低丰度。
PCR扩增和测序
引物338F/806R扩增提取DNA的16S rRNA基因的V3-V4部分[11.].PCR扩增循环如下:95°C 5 min - 30个循环,94°C 30 s + 52°C 30 s + 72°C 30 s - 72°C 10 min。放大,25μL反应混合物1μL每个引物(10μM), 5μL 5 x Q5 GC高增强剂,2μL deoxynucleoside三磷酸腺苷(核苷酸),9.75μL消毒超纯水,5μL 5 x PCR缓冲,0.25μL 5 U /μL Q5聚合酶和8]和1 μL DNA (20 ng/μL)。amplicon过程是根据报道的最佳实践进行的[36.,42.].的条带切出,并从1.2%琼脂糖使用的MinElute PCR纯化试剂盒(Qiagen,德国),并且QiagenQIAquick凝胶提取试剂盒(Qiagen,德国)纯化凝胶。PCR产物混合和双末端测序广东Magigene生物科技有限公司中国Illumina的MiSeq测序平台上。
扩增序列处理与分析
16S rRNA基因序列用QIIME1.9.1处理[5, USEARCH 10.0和内部脚本。对端Illumina读取由FastQC过滤,并由join_paired_ends.py脚本连接。基于高可信度16S代表序列,由USEARCH(−usearch_global and uc2otutab.py scripts, cutoff = 0.97)生成OTU表。另一个过滤步骤是使用PyNAST (align_seqs.py脚本)将所有OTUs的代表性序列与Greengenes_13.5数据库进行比对,以去除非细菌16S rRNA基因序列。使用Greengene_13.5分类器对OTUs进行分类。
为了描述根际群落的组成,每种样品随机提取9519序列以计算alpha.多样性指数包括Shannon、Chao和Faith的PD指数,用于估算alpha._diversity.py脚本由qiime。使用脚本make_phylogyy.py构建系统发育树(默认设置 - FastTree)。在计算之前bet我们首先使用CSS方法通过normalize_table.py脚本来标准化OTU配置文件[59].使用beta_diversity.py脚本编制Bray-Curtis相似性矩阵。主坐标分析(PCoA)图由R版本3.4.3中使用ggplot2包创建的Bray-Curtis相似性矩阵生成。为了更好地评价RS对根际细菌群落的影响,我们重新绘制了PCoA图,不包括RS的16S rRNA基因序列。此外,系统发育分子生态网络分析(MENA) [10.,50[用于探索两种治疗中的相互作用网络,对于MENA分析,通过去载到管道之前的oTus相对丰富的oTus滤波,静电滤波。其中至少在所有样品中的三个中的OTU是保留的,一些具有缺失值的OTU保持坯料,并且选择了Pearson相关系数以计算OTU的相似性。用相关值≥0.96滤出相关矩阵的相关性,p-value < 0.05. The global properties of the networks were calculated in MENA pipeline. Gephi (v. 0.92) was used to visualize the networks finally [1].
GC-MS检测和分析低分子量分子
随机选取高RS丰度组和低RS丰度组3个根际土壤样品(共6个)进行代谢物分析。对于低分子量分子的提取,将土壤样品分为两部分(每部分0.2 g),分别用2 mL EP管和24 μL Adonitol (dH中1mg /mL原液)进行提取2O)作为内标添加。一部分土壤样品在0.5 mL甲醇溶液中以45 Hz的频率在球磨机中均化4分钟(V甲醇:V水= 3:1),超声处理5min(冰水孵育)5次。上清0.4 mL, 4℃,10000×g离心15 min后转移到新鲜离心管中。用0.5 mL乙酸乙酯萃取残渣,再用0.4 mL乙酸乙酯萃取至甲醇萃取。土壤样品的另一部分先用乙酸乙酯萃取,然后用甲醇萃取。总的来说,从一个土壤样品中提取1.6 mL (0.4 × 4)溶液,取40 μL作为质控样品,将1.2 mL转移到一个新鲜的2ml GC/MS玻璃瓶中,用氮气干燥。甲氧基胺化盐酸20 μL (20 mg mL- 1将在吡啶中加入干燥的样品中,并在80℃下用30μl的BSTFA(双(三甲基甲硅烷基)三氟乙酰胺)孵育(1%TMC(三甲基氯硅烷),V / V),在80℃下孵育30分钟。最后将混合物在70℃下孵育1.5小时。与Li等人报告的相同,进行GC-TOF-MS分析和原始峰值分析。[22.]
根际土壤代谢组分析,主成分分析(PCA) [60]用来可视化使用R包装纯素的两个样品的组成。代谢分析显示途径分析(https://www.metaboanalyst.ca//faces/ModuleView.xhtml).
统计分析
组间alpha-diversity指标的差异采用R修订3.4.3中的非参数t检验。采用PERMANOVA (permutationmultivariate analyses of variance, Adonis, Bray-Curtis transformed data, permutation= 999)和AMOVA (molecular variance analysis, AMOVA)分析两种处理间细菌群落组成和根系分泌物的差异。为了确定根际分泌物的变化百分比,我们对根际土壤代谢组中所有化合物的相对丰度进行t检验,以测定这些丰度在两个样品之间的显著差异。的P-数值经Benjamini-Hochberg FDR程序进行多次比较校正[2].除了代谢途径情节的富集分析之外,使用R创建所有图。
数据和材料的可用性
原始序列数据保存在NCBI Sequence Read Archive (SRA)数据库中,登录号为SRP246782 (https://www.ncbi.nlm.nih.gov/sra/?term=SRP246782).
缩写
- OTU:
-
运营分类单位
- GC-MS:
-
气相色谱 - 质谱法
- 卢比:
-
Ralstonia solanacearum
- LRS:
-
较低的Rs丰度组
- HRS:
-
高RS丰度群
- PCOA:
-
主坐标分析
参考资料
- 1。
巴斯蒂安男,海曼S,Jacomy M(2009)Gephi:一个开源软件,用于研究和操作网络。在博客和社交媒体的第三次国际会议AAAI。
- 2。
控制错误发现率:一种实用而强大的多重测试方法。皇家统计学会杂志。系列1995;57:289 - 300。
- 3.
Berendsen RL, Pieterse CM, Bakker PA。根际微生物群与植物健康。植物生态学报。2012;17:478-86。
- 4。
Berendsen RL,Vismans G,Yu K,Song Y,De Jonge R,Burgman WP,Burmolle M,Herschend J,Bakker P,Pieterse CMJ。疾病诱导的植物有益细菌联盟的组合。ISME J. 2018; 12:1496-507。https://doi.org/10.1038/s41396-018-0093-1.
- 5.
Caporaso JG,Kuczynski J,Stombaugh J,Bittinger K,Bushman FD,Costello Ek,Fierer N,Pena AG,Goodrich JK,Gordon Ji,Huttley Ga,Kelley St,Knights D,Koenig Je,Ley Re,Lozupone CA,麦当劳,Muegge BD,Pirrung M,Reeder J,Sevinsky JR,Turnbaugh PJ,Walters WA,Widmann J,Yatsunenko T,Zanefeld J,Knight R. Qiime允许分析高吞吐量社区测序数据。NAT方法。MBIO。2010; 7:335-6。https://doi.org/10.1038/nmeth.f.303.
- 6.
carion VJ, Cordovez V, Tyc O, Etalo DW, de Bruijn I, de Jager VCL, Medema MH, Eberl, Raaijmakers JM。伯克氏菌科和含硫挥发物在抑病土壤中的作用。ISME j . 2018; 12:2307-21。https://doi.org/10.1038/s41396-018-0186-x..
- 7.
Chuberre C,Plancot B,Driouich A,Moore JP,Bardor M,Gugi B,vicre M.植物免疫是划分的,专门从事根部。前植物SCI。2018; 9:1692。https://doi.org/10.3389/fpls.2018.01692.
- 8.
Claesson MJ, O'Sullivan O, Wang Q, Nikkilä J, Marchesi JR, Smidt H, de Vos WM, Ross RP, O'Toole PW。焦磷酸测序和系统发育微阵列用于探索人类远端肠道微生物群落结构的比较分析。《公共科学图书馆•综合》。2009;4:e6669。https://doi.org/10.1371/journal.pone.0006669.
- 9.
交叉ML.微生物对抗微生物:由益生菌乳酸菌产生的免疫信号及其在保护对抗微生物病原体中的作用。微生物学与免疫学。2002;34:245-53。
- 10。
邓勇,蒋永华,杨勇,何智,罗峰,周继杰。Mol Ecol网络分析。2012;13:113。https://doi.org/10.1186/1471-2105-13-113.
- 11.
Dennis KL, Wang Y, Blatner NR, Wang S, Saadalla, Trudeau E, Roers A, Weaver CT, Lee JJ, Gilbert JA, Chang EB, Khazaie K.腺瘤性息肉由微生物刺激的局灶炎症驱动,并由产生il -10的T细胞控制。癌症研究》2013。https://doi.org/10.1158/0008-5472.can-13-1511.
- 12.
Finkel OM, Castrillo G, Herrera Paredes S, Salas Gonzalez I, Dangl JL。了解和利用植物有益微生物。植物生态学报。2017;38:465 - 465。https://doi.org/10.1016/j.pbi.2017.04.018.
- 13。
Flores-Rentería D, Rincón A, Valladares F, Curiel Yuste J.农业基质不同程度地影响地中海栎破碎化森林土壤微生物群落的α和β结构和功能多样性。土壤生物化学。2016;92:79-90。https://doi.org/10.1016/j.soilbio.2015.09.015.
- 14。
傅L,Ruan Y,Tao C,Li R,Shen Q.致癌生物无机肥料诱导与香蕉枯萎病抑制相关的弹性培养细菌群落。SCI批准。2016; 6:27731。https://doi.org/10.1038/srep27731.
- 15.
黄建军,王旭,王雪梅,徐颖,沈强,黄建军。微生物入侵对土壤微生物群落组成的影响。生物肥料土壤。2016;52:997-1005。https://doi.org/10.1007/s00374-016-1136-2.
- 16.
Hanemian M, Barlet X, Sorin C, Yadeta KA, Keller H, Favery B, Simon R, Thomma BP, Hartmann C, Crespi M, Marco Y, Tremousaygue D, Deslandes L.拟南芥CLAVATA1和CLAVATA2受体通过mir169依赖途径参与茄科Ralstonia的致病性。新植物学家。2016;211:502-15。https://doi.org/10.1111/nph.13913.
- 17.
黄X-F,Chaparro JM,Redenton Kf,张R,沉Q,Vivanco JM。根际相互作用:根出渗出物,微生物和微生物社区。植物学。2014; 92:267-75。
- 18.
江j,yu m,hou r,li l,ren x,jiao c,yang l,xu h.土壤微生物群落的变化与Panax喹甲粒素L.根腐病的发生有关。2019; 438(1):143-56。
- 19.
生物炭刺激下的植物生产性能与根际微生物多样性和代谢潜力密切相关。新植醇。2017;213:1393 - 404。
- 20。
Kyselkovám,KopeckýJ,Frapolli M,DéfagoG,萨尔戈瓦瓦GL,Moğnne-Loccoz Y.土壤抑制或有利于烟草黑根腐病的根瘤菌群落组成的比较。ISME J. 2009; 3:1127。
- 21。
Larousse M, Rancurel C, Syska C, Palero F, Etienne C, Industri B, Nesme X, Bardin M, Galiana E.番茄根系微生物群与疫霉菌相关疾病。微生物。2017;5:56。https://doi.org/10.1186/s40168-017-0273-7..
- 22。
李米,宋吉,马Q,孔子D,周Y,江X,Parales R,Ruan Z,张Q.探讨了少核糖菌生物降解的特点和新机制。LAM1902。JAgric Food Chem。2020; 68:826-37。
- 23。
刘旭,张胜,姜强,白艳,沈刚,李胜,丁伟。利用群落分析探讨烟草青枯病抑制的细菌指标。Sci众议员2016;6:36773。
- 24。
Locey KJ, Lennon JT。比例法则预测全球微生物的多样性。中国科学院院刊2016;113:5970-5。
- 25。
马建军,杨春华,王志强。不同地理位置对土壤微生物多样性和组成的影响。环境科学学报。2016;563-564:199-209。https://doi.org/10.1016/j.scitotenv.2016.04.122.
- 26。
王志强,王志强,王志强,等。植物根际代谢产物在土壤微生物中的作用[j]。植物j . 2017; 90:788 - 807。https://doi.org/10.1111/tpj.13543.
- 27。
门德斯LW,门德斯R, Raaijmakers JM, Tsai SM。抗土传病原菌育种对菜豆根际活性微生物群的影响。ISME j . 2018; 12:3038-42。https://doi.org/10.1038/s41396-018-0234-6.
- 28.
Mendes R,Garbeva P,Raijmakers JM。根际微生物组:植物有益,植物病原和人致病微生物的意义。有限元微生物版本2013; 37:634-63。
- 29.
Mendes R, Raaijmakers JM。微生物组功能的跨界相似性。ISME j . 2015; 9:1905。
- 30.
Mommer L,Kirkegaard J,Van Ruijven J.根互动:朝向根际框架。趋势植物SCI。2016; 21:209-17。https://doi.org/10.1016/j.tplants.2016.01.009..
- 31。
Peyraud R,Cottreet L,Marmiesse L,Genin S.通过毒力调节网络控制初级代谢促进植物病原体中的鲁棒性。NAT Communce。2018; 9:418。https://doi.org/10.1038/s41467-017-02660-4.
- 32。
philippe L, Raaijmakers JM, Lemanceau P, Van Der Putten WH。回到根:根圈的微生物生态学。2013;11:789。https://doi.org/10.1038/nrmicro3109.
- 33。
Pieterse CM, Zamioudis C, Berendsen RL, Weller DM, Van wee SC, Bakker PA。诱导有益微生物的全身抗性。植物病理学杂志。2014;52:347-75。https://doi.org/10.1146/annurev-phyto-082712-102340.
- 34。
拉扎W,泠嗯,羊L,黄Q,番茄青枯病菌青枯菌于由防菌芽孢杆菌产生的挥发性有机化合物沉Q.响应杆菌SQR-9。SCI代表2016; 6:24856。https://doi.org/10.1038/srep24856.
- 35。
Rudrappa T,Czymmek KJ,Pare PW,BAIS HP。根系分泌的苹果酸新增有益的土壤细菌。植物理性。2008; 148:1547-56。https://doi.org/10.1104/pp.108.127613.
- 36。
学者A,Jacquiod S,Vestergaard G,Schulz S,Schloter M.基于标记基因的扩增子测序的土壤微生物群落分析。土壤生物学和生育能力。2017; 53(5):485-89。
- 37。
SchönfeldJ,Heuer H,Van Elsas J,Smalla K.基于PCR扩增Flic碎片的PCR扩增的土壤中Ralstonia Solanacearum的特异性和敏感性检测。申请环境微生物。2003; 69:7248-56。
- 38。
植物-细菌共生的趋化信号系统。植物学报。2016;90:549-59。https://doi.org/10.1007/s11103-016-0432-4.
- 39。
Shi S,Nuccio Ee,Shi ZJ,He Z,周J,Firestone MK。互连的根际:高网络复杂性主导根际组装。ECOL LETT。2016; 19:926-36。https://doi.org/10.1111/ele.12630.
- 40。
Thomashow Ls,Kwak Ys,Weller DM。可持续农业中的根系微生物:模型,代谢物和机制。害虫管理科学。2019年。https://doi.org/10.1002/ps.5406..
- 41。
Venturi V,龙骨C.信号传导在根际。趋势植物SCI。2016; 21:187-98。https://doi.org/10.1016/j.tplants.2016.01.005.
- 42。
大数据的智能化——如何利用宏基因组学来了解土壤质量。土壤生物学与肥力。2017;53(5):479-84。
- 43。
魏Z,胡锦涛,顾y,尹s,xu y,jousset a,shen q,friman vp。Ralstonia solanacearum病原体在侵袭过程中破坏了细菌根际微生物。土壤Biol Biochem。2018A; 118:8-17。https://doi.org/10.1016/j.seilbio.2017.11.012..
- 44。
魏志强,杨涛,徐勇,沈强,朱素华。植物根系相关细菌群落的营养网络结构对病原菌入侵和植物健康的影响。Nat Commun。2015;6:8413。https://doi.org/10.1038/ncomms9413.
- 45。
袁杰,阮y,王b,张继,逃离黄芩植物生长促进植物生长芽孢杆菌菌株甲嘧硫唑胺抑制镰刀菌枯萎病株,促进香蕉植物的生长。JAgric Food Chem。2013; 61:3774-80。https://doi.org/10.1021/jf400038z.
- 46。
袁家,吴义,赵米,文T,黄Q,沉Q.酚醛酸渗透蛋白对芽孢杆菌的根定子和病原体抑制性能的影响。BIOL控制。2018A; 125:131-7。https://doi.org/10.1016/j.biocontrol.2018.05.016.
- 47。
元J,赵J时,温家宝T,赵M,李R,古森斯P,黄问,白Y,比万科JM, Kowalchuk GA, Berendsen RL,沈问:根分泌物把地上的土传遗产病原体感染。微生物。2018 b; 6:156。https://doi.org/10.1186/s40168-018-0537- 0.018-0537-X..
- 48。
Zhalnina K,Louie Kb,Hao Z,Mansoori N,Da Rocha联合国,Shi S,Cho H,Zhao H,Karaoz U,Northeng TR,Firestone MK。动态根出渗出物化学和微生物底物偏好在根际微生物群落组件中的驱动模式。Nat microbiol。2018; 3:470。https://doi.org/10.1038/s41564-018-0129-3.
- 49。
赵M元J,沈Z,董M,刘H,温家宝T,李R,沈问:优势在根际微生物群的土壤和根效果重新组装。微生物生态学杂志。2019;95。https://doi.org/10.1093/femsec/fiz139..
- 50。
周J,Deng Y,Luo F,HE Z,Tu Q,XJM Z.功能分子生态网络。2010; 1:E00169-10。
- 51。
海沃德AC。Pseudomonassolanacearum引起的细菌枯萎生物学和流行病学。ANN Rev Phytopathol。1991; 29(1):65-87。https://doi.org/10.1146/annurev.py.29.090191.000433.
- 52。
江G,魏Z,徐军,陈H,张Y,她X,马乔AP,丁W,廖B.青枯病在中国:历史,现状和未来前景。前植物SCI。2017; 8。https://doi.org/10.3389/fpls.2017.01549.
- 53。
Yabuuchi E,Kosako Y,矢野我,堀^ h,二伯克霍尔德氏菌和产碱杆菌属,以根雷尔氏菌的西内Y.转移。十一月:青枯pickettii(罗尔斯顿,palleroni和doudoroff 1973)梳的建议。11月青枯雷尔氏菌(史密斯1896年)梳。十一月和富养产碱(戴维斯1969)梳。十一月微生物免疫。1995; 39:897-904。
- 54。
茄科Ralstonia solanacearum利用无机氮代谢在含氧有限的宿主木质部环境中产生毒力、ATP和解毒。Mbio 2015; 6。https://doi.org/10.1128/mBio.02471-14.
- 55。
多聚半乳糖醛酸酶是番茄青枯菌快速定殖和全毒力的必需酶。植物病理学杂志。2000;57:77-83。https://doi.org/10.1006/pmpp.2000.0283.
- 56。
赛乐E,麦克加维JA,马绍尔,丹尼TP。细胞外多糖和内切葡聚糖酶在番茄青枯菌入侵和定殖中的作用。植物病理学87:1264 1997;1271年。https://doi.org/10.1094/phyto.1997.87.12.1264.
- 57。
Denny TP, Baek s .遗传学证据:细胞外多糖是茄荚假单胞菌致病因子。植物微生物相互作用1991;https://doi.org/10.1094/Mpmi-4-198.
- 58。
Genin S, Denny TP。茄科Ralstonia solanacearum种复合体的病理基因组学。Ann Rev Phytopathol. 2012; 50:67-89。https://doi.org/10.1146/annurev-phyto-081211-173000.
- 59。
Paulson J,Stine O,Bravo H,Pop M等。微生物标志物基因调查的差异丰度分析。NAT方法2013; 10:1200-1202。https://doi.org/10.1038/nmeth.2658.
- 60.
王志强,王志强。代谢组学的多变量分析。咕咕叫代谢组学。2013;1(1):92 - 107。
致谢
没有一个
资金
本研究受到江苏省自然科学基金(BK20170724),中央大学的基础研究资金(KJQN202017),中国自然科学基金(31902107),创新人才(BX201600075)和特别基金用于公共利益的农业科学研究:作物枯萎病的综合管理技术(201503110)。融资机构没有任何作用,但为本研究提供了财务支持,包括实验实施,采样和数据分析。
作者信息
隶属关系
贡献
JY,TW和TL:进行的所有试验;JY和QS设计的研究,并编写了大部分的稿件;MZ和QH:这项研究提供了重要的意见,并帮助写论文。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1。
主要坐标分析(PCOA)利用整个OTU表与属于oTus的整个OTU表进行根际细菌社区的Bray-Curtis差异。Ralstonia solanacearum.
附加文件2:表S1。
研究了RS丰度不同的两群共现网络的特征。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于在这篇文章中提供的数据,除非在信用额度的数据另有说明。
关于这篇文章
引用这篇文章
Wen,T.,Zhao,M.,Liu,T.et al。大量的高Ralstonia solanacearum改变了番茄根际微生物组和代谢组。BMC植物BIOL.20,166(2020)。https://doi.org/10.1186/s12870-020-02365-9
收到:
接受:
发表:
关键字
- 根际土壤中微生物
- 根际代谢物
- Ralstonia solanacearum
- 病原体丰富
- 共同存在网络