摘要
背景
Polycomb抑制复合物1和2通过翻译后修饰特定的组蛋白残基在表观遗传基因调控中发挥重要作用。Polycomb抑制复合物2负责组蛋白H3上赖氨酸27的三甲基化;Polycomb抑制复合物1催化组蛋白H2A在赖氨酸119位点的单倍化。这两种配合物在拟南芥,但是多元素,特别是那些具有复杂的全多大曲调起源的多胶质组织基因家族的演变是未知的。
结果
在这里,我们在allohexaploid面包小麦中介绍了硅胶抑制复合物1和2(PRC2,PRC1)亚基的硅鉴定,重建其进化历史和一系列33种发育阶段的转录分析。我们确定了PRC2 [E(Z),SU(Z),FIE和MSI]的四个主要亚基,以及PRC1(PC,PSC和SCE)的三个主亚基,并确定了它们的染色体位置。我们发现,编码亚基蛋白的大多数基因在面包小麦中作为副磷酸存在。使用来自不同组织的面包小麦RNA-SEQ数据以及整个植物的发育阶段ontoberis揭示了个体寄生虫的可变转录活性。系统发育分析显示了温带谷物中的高水平蛋白质守恒。
结论
面包小麦中Polycomb抑制复合物1和2核心成分的鉴定和染色体定位,可能使人们更深入地了解普通冬小麦的发育过程,包括春化过程。
背景
在较高生物中的基因表达的调节包括各种作用于转录,后幕和后期性水平的机制。坐标适当的基因活性所需的更复杂调节还包括通过组蛋白修饰(甲基化,乙酰化,磷酸化和泛素)的染色质重塑的调节,这导致特异性染色质变化。突出的后翻译改变是特定氨基酸残基发生的组蛋白修饰。组蛋白H3(H3K4ME)上的赖氨酸4的甲基化主要与转录激活有关,而赖氨酸9和27的二甲基化和三甲基化分别导致转录抑制[r3K27ME31].H3K9ME2与小双链RNA和DNA高甲基化一起有助于重复DNA序列的沉默[2那3.].基因的抑制表观遗传调控过程通常由Polycomb组蛋白(PCG)控制,这些蛋白质(PCG)在基本水平处,在植物和动物中进化地保守[4.].最初确定的果蝇,Polycomb抑制复合物1 (PRC1)和2 (PRC2)是参与发育基因调控的两个主要复合物(综述于[4.那5.那6.])。传统上,已建议在分层PRC2→PRC1的方式中工作[PRC2 [7.]但最近,已经提出了PRC2的独立功能[8.那9.].根据层次模型,PRC2与赖氨酸27(H3K27ME3)的特定DNA序列基序结合在附近的核桃中的赖氨酸27(H3K27ME3),募集PRC1,该PRC1催化组蛋白H2A(H2AK119U1)并稳定H3K27ME3改性染色质重塑[10].PRC2:PRC1-independent模型提出,PRC1和PRC2有它们自己特定的结合PRE的适配器蛋白,因此,PRC1/2是通过与它们特定的适配器蛋白相互作用而独立招募的[8.].
果蝇PRC1包含四个核心组件,Polycomb(PC),Polyhomeotic(pH),后性爱梳理(PSC)和性梳子额外(SCE);据报道,第五组分,在Midleg(SCM)上的性梳子(审查[6.])。PRC1的存在在植物中尚不清楚,直到在中文中描述了戒指蛋白拟南芥[11那12].在答:芥,LIKE异染色蛋白1(AtLHP1)可替代PC功能[13].利用其染色体,LHP1识别和结合组蛋白H3甲基化赖氨酸27(H3K27ME3)[14].A. Thilana B淋巴瘤MO-MLV插入区域1同源物(ATBMI1A到C.)是Psc的三个同系物和非常有趣的新基因(atring1a.和atring1b.)是Sce的两种同源物(在[15])。植物中没有鉴定pH homolog [16].然而,与PRC1相关的植物特异性蛋白质,例如答:芥胚胎花1(ATEMF1)[17]或者答:芥Vernalization1(ATVRN1)[18已经提出了。EMF1参与控制射击建筑和开花拟南芥[19]与PRC1的AtBMI1和AtRING1同源物[相互作用20.那21].相比之下,没有关于ATVRN1之间的相互作用的报告,它涉及vernalization拟南芥[22],以及迄今为止的其他PRC1组件[23].因此,关于VRN1是否是PRC1的核心组分没有共识。最近,报道了具有类似PRC1的功能的替代复合物[24].在拟南芥,两个同源BAH(溴 - 邻同源)结构域的蛋白质形成植物特异性复合物与胚胎FLOWER1(AtEMF1),并且该BAH-EMF1复杂读取和效果的H3K27me3标记和介导基因组范围的转录抑制。一个BAH结构域蛋白的同源物也已经在单子叶植物(稻),其可指示在开花植物[其保护发现24].编码PRC1亚基的基因也曾在单像,例如,Zea Mays.和栽培稻[23]但不是在农艺上的重要温带谷物,如小麦或大麦。
PRC2复合物由四个亚基形成:Zeste的增强子[E(Z)],额外的性梳理(ESC),Zeste 12的抑制剂[Su(Z)12]和WD蛋白P55 [25];然而,类似于PRC1,已经提出了另外的第五核心组分(Jing)果蝇[6.].在植物中,PRC2已经彻底研究过拟南芥(审核[4.])。PRC2的催化活性是与E(z)中的SET结构域相关的组蛋白甲基化。到目前为止已经描述了三个E(z)同源物:卷曲叶(CLF) [26, swinger (swwn) [27]和medea(mea)[28].类似地,已经鉴定了三种SU(Z)的同源物:减少vernalizate2(VRN2)[29],胚胎花2(EMF2)[30.]和施肥独立种子2(FIS2)[31].的esc.同源物受精独立胚乳(f)作为单一基因存在;相比之下,五个基因(MSI1至MSI5)已发现WD40 P55同源物(ira1的多拷贝抑制因子那MSI)在拟南芥[32].每个人拟南芥e(z)和su(z)同源物在不同的发育阶段起作用(在[33])。E(Z)同源物MEA在早期的胚乳开发期间活跃[34];SWN和CLF在营养发育和春化过程中起重要作用。春化后的开花起始是由开花抑制因子控制的开花轨迹C(FLC.) [35那36].还表明H3K27ME3水平增加和逐渐沉默FLC.在春化(37];此外,FLC.在寒冷期结束时完全关闭[38].该状态在下一代重置,因此,工厂必须经过春化到花卉。
在拟南芥,clf swn双突变体完全失去H3K27me3,提示PRC2可能失活[39].但是,clf swn植物只形成诸如偶尔的体细胞胚胎的愈伤组织结构[40].SU(Z)同源物FIS参与雌性配子体和种子发展的调节[41]但是SU(Z)同源物EMF2控制过渡到开花[42].草PRC2的同系物已经在玉米,水稻和大麦[确定硅43那44那45那46那47那48那49],其功能主要与种子和胚乳发育有关[49那50.];有关详细摘要,请参阅[51.].虽然Kapazoglou等人。[49]鉴定出大麦PRC2同源物HvFIE那hve(z)那HVSU(Z)12A和HVSU(Z)12B那P55.尚未找到。
最近,Lomax等人。[52.]确定A.Brachypodium distachyon突变体春化没有要求。在突变Zeste的增强剂(EZL1),的同源物答:芥CLF,导致H3K27me3和H3K27me2在b . distachyon VERNALIZATION1(BDVRN1.)并且因此,早些时候开花而不进行春化。几个地区的H3K27ME3水平显着降低Tavrn1.春化过程中也有报道在面包小麦Triticum aestivum.,与冷时期的长度正相关[53.].这些发现表明在草丛中春化过程中的PRC2介导的H3K27ME3沉积中的重要作用。
尽管面包小麦的社会经济重要性,但由于缺乏注释的参考基因组,我们对生物过程的理解受到限制,直到最近,当国际小麦基因组测序联盟(IWGSC)发表了中国春季的参考基因组[54.].总体而言,由于其多倍体性质和高重复含量,复杂的小麦基因组难以解码。面包小麦(2n = 6x = 42)是最近形成的骨核基因组大小(16,974mb / 1c,[55.])从三种同源性亚因子(A,B和D)组装,并具有超过85%的重复元素。因此,深入分析基因及其生化途径以及中央农艺性状的分子基础落后于其他作物和模型植物物种,如答:芥.
在这里,我们报告了编码PRC2和PRC1的个体亚基的面包小麦基因的鉴定和染色体位置。我们分析了不同发育阶段的个体基因的mRNA水平,并与其他人发现序列保护Triticeae物种,如Triticum Urartu.那山羊草属tauschii和Triticum dicoccoides.,采用系统发育学方法。我们还讨论了PRC2和PRC1在面包小麦春化过程中的作用。
结果
小麦中PRC2和PRC1核心成分的硅质鉴定
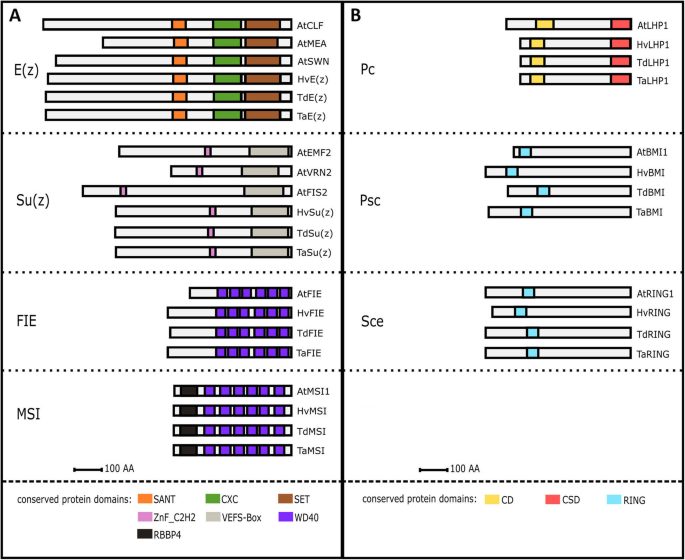
使用蛋白质序列拟南芥PcG同源物,我们鉴定了小麦组分及其各自的染色体位置。如预期的那样,在所有三个小麦亚基因组A, B和D中单个组分的同源性也被定位。面包小麦成分用前缀“Ta”表示Triticum aestivum.然后是A, B或D来表示亚基因组的位置。如果在不同的染色体上或同一染色体但在不同的位置上发现了额外的条目,则分别添加相应的数字以区分单独的并行对数,例如,TaSu(z)-2A1(表1).使用可用的参考基因验证染色体位置T. Urartu.(2n = 2x = 14)T. dicoccoides.(野生二粒小麦,2n = 4x = 28,登录Zavitan)和h . vulgare(2n = 2x = 14,品种Morex)(附加文件1:表S1)。
Zeste的增强者[E(z)]位于染色体4和7上(表1).4号染色体上E(z)在短手臂上发现了[Tae(z)-4a1]并且在长臂上[TaE (z) 4 b1那TaE (z) 4 d1];7号染色体,E(z)在短臂上找到了(附加文件1:表S1)。职位Tae(z)-4a1在4A染色体短臂上与六倍体小麦中报道的中心倒位相对应[54.那56.].7号染色体上各自短臂上的两个副对数被鉴定出来,相隔仅几十个碱基,表明它们起源于一个本地基因复制事件(附加文件)1:表S1)。此外,由于多重插入和缺失(吲哚),位于染色体7a上的副葡萄醇不同86个氨基酸,并且染色体7b和7d的那些含有85个氨基酸的氨基酸,长度最长为137个氨基酸(附加文件2:图S1D)。
Kapazoglou等。[49)报道Zeste的抑制器[Su(z)]同源基因,位于2H和5H染色体上。同样,我们在染色体2和5上发现了小麦同源物。有趣的是,在2AS和2BS染色体上发现了两个同源物,但在2DS染色体上只发现了一个同源物(见表)1).第5组的所有三个同源性都位于长臂上。面包小麦二倍体祖细胞T. Urartu.在2号染色体短臂上发现了2个同源基因,位置≈1.5 Mb和≈2.4 Mb;在5号染色体长臂上发现了1个同源基因。野生二粒小麦登录Zavitan还携带两个同源物在2AS和2BS上,以及5AL和5BL上的同源物(附加文件1:表S1)。
由基因编码的两种蛋白质tasu(z)-2a2和tasu(z)-2b2携带32个氨基酸的插入。该插入也发现在编码的蛋白质中tridc2ag000370.14基因T. dicoccoides.而且到了h . vulgare基因horvu.morex.r2.2hg0078790.1位于2染色体上(附加文件2:图S1G)。
Esc亚单位报告果蝇已被指定依赖于受精ENDOSPERM1(HvFIE1)在大麦中[49],我们遵循了这一风格,并命名为小麦同源Tafie..我们在7次上发现了两个同源物(TaFIE-7A2.1和TaFIE-7A2.2)和一个上7AL(tafie-7a1.)(表1和额外的文件1:表S1)。染色体7D港口一个基因位于短臂上(TAFIE-7D1)和长臂上的一个基因(Tafie-7d2.).最初,没有使用IWGSC的中国春天的参考序列定位7B同源物。令人惊讶的是,在染色体的长臂的远端部分中发现了副病虫病。这与染色体4的该区域含有一部分染色体7b [56.].具有4AL同源物(TAFIE-4A1)的互惠爆炸与先前位于Zavitan的7AL / 7BL上的基因具有高相似性,并在7H染色体上与大麦基因。预测的大麦蛋白作为FIE [57.那58.].随后,我们利用TGAC的小麦基因组组装数据,在Ensembl植物数据库中使用BLAST搜索,鉴定了7BL同源物TRIAE_CS42_7BL_TGACv1_580129_AA1912160.1 [59.](附加文件1:表S1)。
p55亚基包含WD40结构域(与FIE相同)以及组蛋白结合蛋白RBBP4的n端结构域,被命名为MSI1 (IRA1的多拷贝抑制子)拟南芥.在面包小麦中,有两个同源词(Tamsi1.)存在于5组的每种染色体上,其中一个例外:最好的爆炸结果之一没有锚定任何染色体(traescsu02g072700.).与序列比较T. Urartu.和t . turgidum透露了5Al染色体的高度良好;因此,我们指定了这种未分配的加入TaMSI1-A1,说明其在染色体5A上的位置1和额外的文件1:表S1)。
然而,小麦PRC1组分的定位更为复杂,因为到目前为止还没有在谷类中进行过描述,使得验证结果较为困难。因此,我们使用的参考序列h . vulgare含有预测蛋白的注释。
像异铬蛋白蛋白1(LHP1)小麦同种型在染色体7的长臂上发现BMI1染色体5的短和长臂上的同源物。拟南芥有三个BMI1同源物(AtBMI1A来AtBMI1C)但爆炸了AtBMI1A和AtBMI1B在染色体的长臂上鉴定了小麦的相同基因。令人惊讶的是,爆炸搜索AtBMI1C在短臂上不仅发现了相同的同源基因,还发现了其他的同源基因。短臂上的基因与大麦基因的位置相对应,也在5H染色体短臂上。该基因被注释为泛素连接酶dreb2a相互作用蛋白2(DRIP2,一个同义词BMI1) [58.]和对应于拟南芥指定。长臂上的基因对应于大麦基因的位置也被注释为泛素连接酶DRIP2[58.]和位于染色体5H的长臂。
ring1.在3组的三条染色体的长臂上都发现了同源物。ring2.存在于第1组所有三条染色体的长臂上。
小麦同系物TaEMF1未被鉴定拟南芥蛋白序列用于BLAST搜索。然而,在EMF1蛋白序列中发现了与位于3A、3B和3D染色体上的同源蛋白Z. Mays.使用(23].这些基因的位置与位置相关联HvEMF1在大麦中,表明它们可能是同源的ATEMF1.
我们还鉴定了单个PcG小麦组分的主要蛋白结构域(图。1).面包小麦的比较拟南芥那h . vulgare和T. dicoccoides.显示出高域保护,进一步支持小麦同源物鉴定的准确性。
系统发育分析
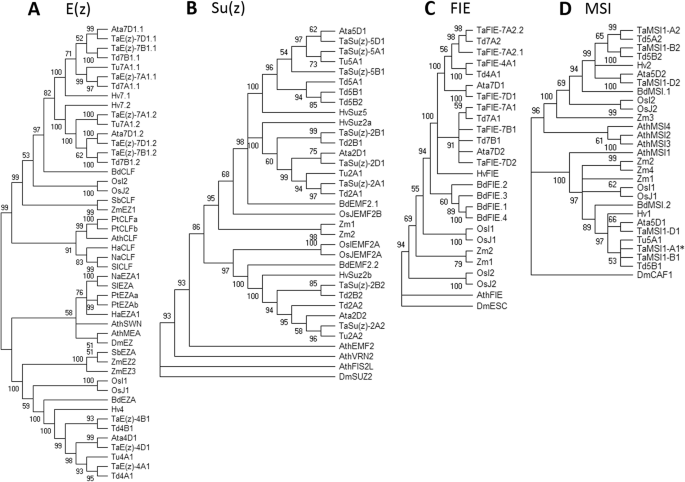
构建了小麦PRC2和PRC1组分的系统发育树,揭示了小麦PRC2和PRC1组分之间的进化关系拟南芥,大麦,大米,玉米,所有面包小麦同源物和面包小麦祖细胞(图。2和3.).
植物PRC2组分E(Z)的系统发育分析(A.)、苏(z) (B.),FIE(C.)和msi(D.).采用最大似然法和MEGA x中基于JTT矩阵的模型进行分析,从1000个重复中推断出bootstrap共识树。E(z)树是中点根的。Su(z)、FIE和MSI树扎根于外群黑腹果蝇(DM)。山羊草属tauschii(ATA),拟南芥蒂利亚纳(Ath),Brachypodium distachyon(Bd),Helianthus Annuus.(哈),尼古利亚娜attenuata.(na),杨树trichocarpa(PT),Solanum lycopersicum(Sl),高粱双色(SB),大麦芽(HV),oryza sativa indica.(OsI),Oryza sativa japonica(OsJ),Triticum aestivum.(Ta)的,Triticum dicoccoides.(TD),Triticum Urartu.(你),Zea Mays.(ZM)。星号(*)表示未基于爆炸搜索分配给任何染色体的基因 - 染色体地点是由过上层的T. Urartu.和t . turgidum
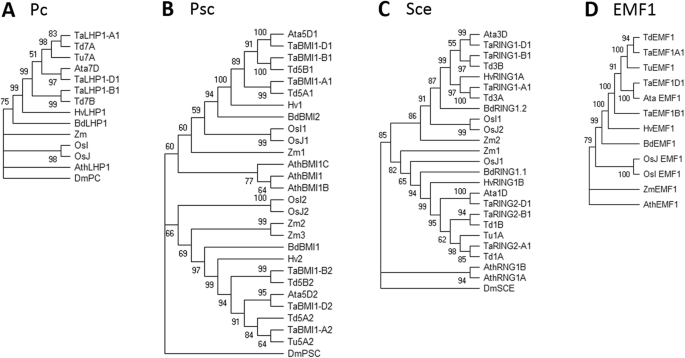
植物PRC1组分LHP1 (A.), RING1 (B.),BMI1(C.)和EMF1(D.).采用最大似然法和MEGA x中基于JTT矩阵的模型进行分析,从1000个重复中推断出bootstrap共识树。树木根植于外群黑腹果蝇(Dm),但EMF1树除外,它是根拟南芥蒂利亚纳(Ath)。山羊草属tauschii(ATA),Brachypodium distachyon(Bd),大麦芽(HV),oryza sativa indica.(OsI),Oryza sativa japonica(OsJ),Triticum aestivum.(Ta)的,Triticum dicoccoides.(TD),Triticum Urartu.(你),Zea Mays.(Zm评选)
系统发育分析表明,位于染色体4和7上的小麦E(Z)同源物分别落入单独的曲面,包括ATSWN和另一个包括ATCLF。这表明了E(z)小麦4号染色体上的基因被认为是AtSWN但是7号染色体上的基因被认为是AtCLF(图。2).
Su (z)在2号和5号染色体上发现了基因。染色体2上的基因聚为一个分支,染色体5上的基因聚为第二个分支。系统发育分析表明Su (z)是同源ATEMF2(图。2 b).
同源物的f位于7号染色体上,但最好的爆击是为4A染色体。有趣的是,4AL染色体上的基因同源物(TAFIE-4A1)用7as染色体同源物(TaFIE-7A2.1和TaFIE-7A2.2与7AL同系物不属于分支(图。2摄氏度).
MSI发现同源物在染色体5的长臂上有两个位置,除了traescsu02g072700.,没有分配给任何染色体(附加文件1:表S1)。然而,在同一分支这个非锚定的基因进化聚集在一起TaMSI1-B1和TaMSI1-D1表明它可能代表塔姆西复制5A染色体上(表1).
PRC1组分的系统发育分析不起眼:小麦LHP1同源物根据亚因子A,B和D聚集。虽然拟南芥有三个BMI1同源,小麦BMI1的同源物分成只有两个分支。这与我们的基于定位结果一致(附加文件1:表S1)。环状同源物根据它们在染色体1和3上的位置聚集成两个曲片(图。3 b).
RNA-SEQ分析表明A,B和D同源物的保守转录模式
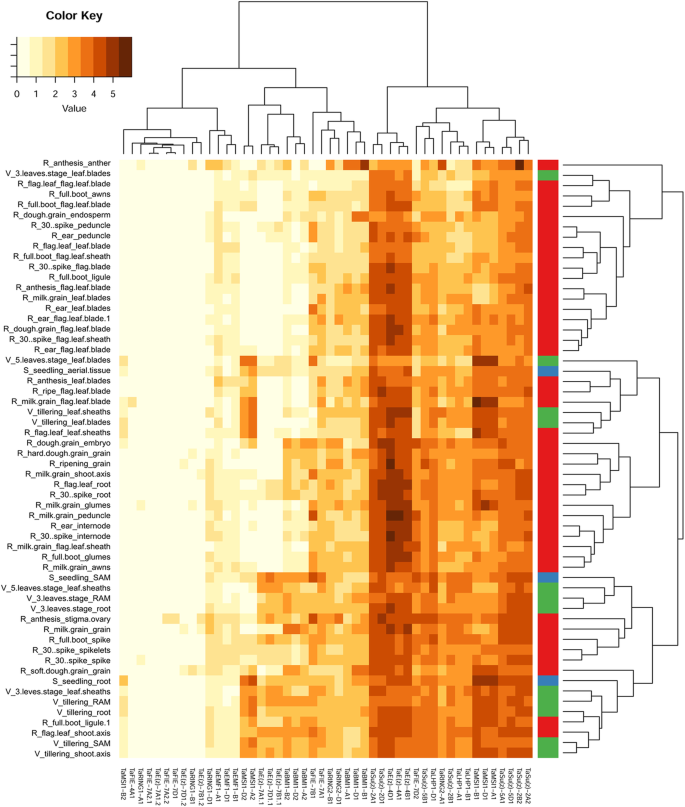
为了评估单个PRC1和PRC2亚基的转录活性和潜在的组织特异性,我们利用Azhurnaya登录(expVIP数据库)中公开的58个面包小麦发育阶段和组织的rna测序数据进行了转录组学分析。提取上述所有基因的百万分之转录本(TPM)值,根据其在组织中的转录谱相似性聚类,并在热图中显示(图2)。4.和额外的文件3.:表S2)。log2转化后使用TPM值,便于对许多低转录水平的基因进行分析。
PRC1和PRC2 mRNA水平的热图在不同的小麦发育阶段。根据基因(行)和组织(柱)之间的转录简谱相似,聚集来自品种Azhurnaya的候选基因的公开的RNA-SEQ数据。每个组织的特征在于“高级龄_GAGE_TISSUE”。通过水平颜色条纹也突出了高级阶段S - 幼苗(蓝色),V - 营养型(绿色)和R - 繁殖(红色)。有关发育样本和输入值的详细描述,请参阅附加文件3.:表S2。彩色键在LOG2转换后显示每百万(TPM)的成绩单
我们发现A,B和D亚骨内的同源物学经常显示出高度相似的转录型材(例如,Tae(z)-4a1,b1,d1;Tae(Z)-7A1.2,B1.2,D1.2;TaBMI1-A1, B1, D1;和TaBMI1-A2 B2, D2;TaMSI1-A1,B1,D1).这表明祖细胞中建立的发育规则仍然存在于现代小麦的亚因素中,并表明同源基因拷贝之间的功能分化低。可能的例外是苏(z) 2 b2,在分析中获得了61.82TPM,在分析中的所有基因中的最高值。实际上,这种mRNA水平高于其同源层的5倍苏(z) 2 a2(TPM 12.39)。然而,这两个基因在所有其他组织中显示出相似的mRNA水平(注意SU(Z)-2D2没有找到T. Aestivum.基因组)。尽管RNA-SEQ数据提供了用于许多PRC1和PRC2基因的转录的固体支持,但还有几乎没有在分析的组织中转录的拷贝,即使整个同源群也是如此。例如,Tae(z)-7a1.2,b1.2,和D1.2副本,代表直肠的拟南芥CLF.在很大程度上没有在整个发展中表达;相比之下,Tae(z)在染色体4上的同种型,代表直肠拟南芥SWN,是TPM值最高的基因之一。观察到略微不同的模式Tamsi1-A2,B2和D2和TaMSI1-A1, B1和D1,分别代表组织特异性和一般MSI组。然而,这种相关性并未普遍适用于一个PRC1或PRC2亚基的所有同源物。组织聚类(log2 plot)揭示了三个主要群体,尽管差异相对较少。前两个块(从图1中的左右到左右。4.)主要由处于生殖阶段的植物组织组成,其特点是只表达特定的副本。相反,第三个聚类中含有更多的幼苗和营养期植株组织,表达的PRC1和PRC2组分数量最多。
讨论
植物PcG蛋白参与植物的发育过程,例如从营养阶段到生殖阶段的过渡,开花和种子的发育[31那60.那61.].PCG蛋白形成梳压制性配合物如PRC1和PRC2的组.PRC2通过组蛋白H3K27的甲基化控制染色质重塑[5.].压抑基因的表观遗传学这个标记是相当普遍的。据报道,近4500(16%)基因拟南芥携带镇压标记H3K27ME3 [62.那63.].在单子叶植物中,许多基因也有H3K27me3标记。有趣的是,在水稻中,抑制性标记H3K27me3与转录水平之间存在显著的一致性,其中大多数H3K27me3标记(几乎85%)与基因区域有关。事实上,H3K27me3标记的基因有近53%的表达,并且发现基因表达水平与H3K4me3/H3K27me3和H3K27me3/H3K4me3的比值相关[64.].在玉米中,H3K27ME3也主要存在于基因 - 致密染色体臂中,并且它靶向具有重要监管作用的基因[65.].在大麦中,H3K27me3高密度分布于端粒近端区,覆盖基因和基因间DNA,该标记指定兼性异染色质。与水稻和玉米类似,H3K27me3优先覆盖未表达基因,但不专属于它们,也可以在一些转录活性基因上发现[66.].尽管存在这种复杂模式的可能性,但H3K27me3的组织特异性差异和/或ChIP和转录组学方法的不同敏感性可能会造成潜在的伪影。
提出了植物物种中H3K27ME3靶标的保护。H3K27ME3在玉米中的目标[65.将与标记为H3K27ME3的基因进行比较拟南芥[39[米[64.].研究发现,34%的玉米基因与拟南芥H3K27me3标记。以H3K27me3标记的同源基因在两种单子叶植物(水稻和玉米)中的数量几乎都是单子叶植物(水稻和玉米)的2倍拟南芥[65.].PRC2在春化反应中也起着关键作用拟南芥.在春化之前,主要开花启动子的表达开花轨迹T(英国《金融时报》)被高水平的压抑FLC.,但冷治疗触发PRC2依赖的沉默FLC.,这与H3K27ME3的水平增加相关[37那67.].什么时候FLC.变得不活跃,表达英国《金融时报》启动并触发过渡到开花(审查[68.])。相比之下,H3K27ME3标记在温带谷物中的春化之前存在高水平[52.那53.那69.],可能是由于PRC2活动,如[70].这可能导致染色质压实和VRN1.镇压。在寒冷时期,H3K27ME3标记消失,导致染色质重塑,这可能能够表达VRN1..因此,可以发生从营养阶段到生殖阶段的过渡。由于缺乏面包小麦中PcG成分的详细信息,阻碍了诸如春化等分子机制的研究。基于同源性搜索,我们在面包小麦中识别并定位了推定的PRC2和PRC1基因。在所有三个小麦亚基因组(A, B和D)中,大多数亚基都是同源的。
小麦PRC2组件的染色体位置对应,在大麦此前公布的PRC2基因[49].有趣的是,一些旁系同源是相同的染色体上发现的,位于不同染色体上的旁系同源也发现了。这些多个站点可以由小麦基因组,其中经历了频繁的染色体重排的性质异源六倍体来解释。个别旁系同源物之间的比较还显示缩短的蛋白质(附加文件4.:表S3,附加文件2:图。S1)和不同的低表达水平。这些发现表明基因的可能改变和/或副官能化。我们还确定了关于各个副本之间的距离而不同的帕拉戈尔科。tasu(z)-2a1和tasu(z)-2a2,而两个副本Tafie.基因(TaFIE-7A2.1和TaFIE-7A2.2)仅被37 kB的区域分开(附加文件1表S1),这表明不同的机制促成了小麦基因重复。不幸的是,它们基于expVIP数据库的表达级别非常低。
有趣的是,在染色体组4和7上发现了E(z)副对数。4号染色体和7号染色体之间的易位已被报道[54.那56.].简而言之,当前日常小麦染色体4的结构是Allohexaploid小麦基因组内的动态染色体重排的说明性例。染色体的最终组成是由古代长臂的渗透反演产生的,这成为现代短臂,随后从5Al和7bs的易位完成了染色体的重新排列。同意这一点,副本TAFIE-4A1基因与7as染色体上的同源物保持更紧密的系统发育关系(TaFIE-7A2.1和TaFIE-7A2.2)(图。2摄氏度).
此外,系统发育分析表明,4号染色体上的基因是推测的同源基因AtSWN但染色体7上的基因更接近AtCLF.保守域的蛋白质对准拟南芥SWN和CLF具有来自TAE(Z)的结构域显示催化设定结构域中的氨基酸的9个独立诊断变化。染色体4对染色体4对ATCLF和TAE(Z)拷贝分享这些九个位置,7(附加文件)5.:图。S2)。这种间接表明,在单焦点和Dicots的进化分裂之前已经存在了CLF和SWN样蛋白[71.].CLF和SWN在很大程度上是功能冗余的拟南芥,它们在植物中同时敲除会产生含有体细胞胚的愈伤组织样结构[72.].目前,功能冗余的程度在两者之间Taswn样式和TaCLF-like群体是未知的,但是Taswn样式同源物学比是更强烈的表达TaCLF-like同源物,它与图案形成鲜明比拟南芥[73.].在mRNA水平上也有很大的差异(高达11倍)CLF-like7号染色体上旁系同源物,其可以指示独联体一些拷贝的调控元件要么发生了突变,要么丢失了。未来的实验将揭示这些拷贝是否可能在组织特异性水平上被亚功能化,或者朝着从面包小麦基因组中去除的方向发展。表达谱分析表明,虽然总有至少一个基因表达水平高,但并非所有代表单个核心成分的副对数都表达相似。这可能是因为模拟序列不相同(附加文件2:图S1);因此,它们的功能和表达可能会发生改变。
与组装植物PRC1核心成分的LHP1、RING1和BMI1不同,识别其他可能是该复合物一部分的植物特异性蛋白非常困难。EMF1的化学性质和功能与Psc相似果蝇和它的同源物,BMI1,在拟南芥[74.].EMF1的保守性较差,与任何其他已知功能的蛋白质没有明显的同源性[19].EMF1中没有注释的域,但预测了整个EMF1同源组共享的5个保守基序[17那23].尽管EMF1存在于单子叶和双子叶[17那19那23]中未发现直接同源物T. Aestivum.使用EMF1蛋白质序列拟南芥对于同源性搜索。因此,我们使用了一系列单子床植物(玉米),表明EMF1在Dicots和单焦点之间不保守。AtVRN1,它在以前的研究中分配给PRC1 [18那75.]显示在单像中不存在[23].在拟南芥那AtVRN1在春化过程中起着重要作用。应该强调的是VERNALIZATION1(VRN1.)基因与小麦无关VRN1.in.拟南芥而是同源的APETALA1、花椰菜和富有成果的(AP1,CAL,和富尔语),在没有任何作用拟南芥vernalization [76.].但是,当ATVRN1蛋白质序列拟南芥用于在小麦中进行同源性搜索,得到类似于位于染色体5a,5b和5d上的基因的类似蛋白质。这些蛋白质含有四个B3结构域,而ATVRN1蛋白质拟南芥只包含两个域。综上所述,我们鉴定了面包小麦中PRC1的所有核心亚基(单子叶中包括LHP1、RING1和BMI1)。植物特异性蛋白EMF1和VRN1的鉴定尚不明显。PRC1的单个亚基在同源基因之间也有相同的保守蛋白结构域,但并不是所有的同源基因都有相同的表达水平,表明在顺式调控水平上存在分化。
结论
面包小麦中单个PCG组分的鉴定将有助于揭示重要的生物过程的分子机制。更详细的研究(表达研究,小麦品种之间的序列变异等)是必要的,以揭示单一基因,包括旁系蛋白酶,以及它们在影响植物发育的多元菌抑制络合物的形成中可能的作用。
方法
在Silico PCG组件识别中
T. Aestivum.通过BLAST搜索获得PCG组分蛋白序列T. Aestivum.基因组在ensemble Plants (http://plants.ensembl.org/index.html.)使用答:芥具有默认参数的蛋白质序列。对数据库中不可用的所有研究的蛋白质序列在根据基因组序列重建的硅中T. Aestivum.利用基因组数据从Ensembl植物中获得的参考品种(中国春)T. Urartu.和AE。Tauschii..数据T. dicoccoides.均从ensemble Plants中获得。获得的核苷酸序列与T. Aestivum.序列通过Mafft多对准器(1.3.3版)在遗传8.1.9软件中https://www.geneious.com.使用默认设置。在基因组序列的对准之后,提取编码序列(CDS)区域并将其翻译成蛋白质。一些基因组序列不充分组装,因此,对应于参考的序列有时散射到几个支架/折叠。通过从几个支架中提取部分序列来重建这些基因,将CDS区连接并将其转化为蛋白质(附加文件4.:表S3)。
蛋白质序列大麦芽从GenBank获得的https://www.ncbi.nlm.nih.gov/及大麦DB [58.];蛋白质B. Distachyon.那Helianthus Annuus.那尼古利亚娜attenuata.那Oryza sativa japonica那水稻,白杨那Solanum lycopersicum和Z. Mays.从UniProt (https://www.uniprot.org/)和ENSEMBL植物。在系统发育研究中使用的所有序列中的其他文件提供4.:表S3。
对鉴定的小麦PCG蛋白的互惠爆炸搜查是针对的答:芥Ensemblplants中的数据库TAIR10(https://plants.ensembl.org/Arabidopsis_thaliana/Info/Index)以验证结果。
系统发育分析
在Mega X中进行系统发育分析的蛋白质对准[77.通过Clustalw。对于PRC1和PRC2复合物中的所有基因,使用最大似然方法和基于JTT矩阵的模型推断出进化历史[78.]在mega x [77.].从1000个副本推断出的引导共识树[79.,以代表所分析的类群的进化史[79.].序列果蝇PcG蛋白被用来作为外的除了EMF1所有树木拟南芥序列作为外群。除E(z)根在中点外,所有系统发生树均根在外群。
转录组分析
RNA-SEQ数据库“EXPVIP”http://www.wheat-expression.com作为单个PcG核心亚基表达分析的数据源[80那81.].我们使用从春小麦品种Azhurnaya的根,叶子/芽,尖峰和谷物中收集的数据在58个不同的时间点,对应于22个组织或器官(附加文件)3.:表S2)。Azhurnaya品种的数据代表了发育的时间过程,仅使用了从3个或3个以上的生物重复中收集的数据。用R软件制作热图(https://www.r-project.org/)使用gplot, heatmap3和RColorBrewer包。根据不同实验点的mRNA数量的相似性对基因和发育阶段进行聚类。
蛋白质域识别
聪明http://smart.embl.de/(在正常模式SMART)[82.]和pfam.http://pfam.xfam.org/[83.]蛋白数据库用于预测PRC2和PRC1组分的保守蛋白结构域答:芥那h . vulgare那T. dicoccoides.和T. Aestivum..对所有的PRC2和PRC1亚基同源蛋白进行多序列比对答:芥那h . vulgare那T. dicoccoides.和T. Aestivum.是用MAFFT进行的v7.388[84.那85.].
可用性数据和材料
本研究期间生成或分析的所有数据都包含在本发表的文章[及其补充信息文件]中。所有数据都是从公开的数据库获得的(NCBIhttps://www.ncbi.nlm.nih.gov/,集成植物http://plants.ensembl.org/index.html.和expVIPhttp://www.wheat-expression.com/).
改变历史记录
2021年1月15日
这篇文章的修订本已经发表,可以通过原始文章访问。
缩写
- AP1:
-
APETALA1
- 呸:
-
溴相邻的同源性
- BMI1:
-
B淋巴瘤MO-MLV插入区域1同源物
- cal:
-
花椰菜
- CLF:
-
卷发叶
- DRIP2:
-
DREB2A-INTERACTING实验研究
- e(z):
-
Zeste的增强者
- EMF:
-
胚胎的花
- ESC:
-
额外的性爱梳子
- EZL1:
-
Zeste的增强剂
- FIE:
-
受精独立胚乳
- FIS2:
-
施肥独立种子2.
- 方法:
-
开花轨迹C
- 英国《金融时报》:
-
开花轨迹T
- 富裕:
-
富有成果的
- LHP1:
-
像异铬蛋白蛋白1
- 意味着:
-
梅德亚
- MSI:
-
IRA的多拷贝抑制因子
- PC:
-
Polycomb.
- PCG:
-
Polycomb组蛋白
- Ph值:
-
Polyhomeotic
- 中国:
-
Polycomb压抑复合体
- 前面:
-
Polycomb响应元素
- PSC:
-
后性爱梳子
- RBBP4:
-
视网膜母细胞瘤结合蛋白4
- RING1:
-
非常有趣的新基因
- SCE:
-
额外性梳
- 供应链管理:
-
在midleg上的性梳子
- 苏(z):
-
Zeste的抑制器
- SWN:
-
赶时髦的人
- TPM:
-
记录每百万
- VRN1:
-
VERNALIZATION1
- VRN2:
-
减少vernalization响应2
参考资料
- 1.
吴吉,李志强。染色质调控的研究进展。细胞。2009;136:200-6。
- 2.
Matzke MA, Mosher RA。rna导向的DNA甲基化:一个日益复杂的表观遗传途径。Nat Rev Genet. 2014; 15:394-408。
- 3.
Fultz D,Choudury SG,Slotkin RK。植物中活性转置元素的沉默。CurrOp植物BIOL。2015; 27:67-76。
- 4.
Polycomb组蛋白调控网络。植物学报2015;66:269-96。
- 5。
Margueron R,Reinberg D. Polycomb Complex PRC2及其在生活中的标志。自然。2011; 469:343-9。
- 6。
施瓦茨YB,Polycombs的Pirrotta五,一个新的世界:意想不到的伙伴关系和新兴的功能。NAT Rev Genet。2013; 14:853-64。
- 7。
王l,棕色jl,cao r,张y,kassis ja,琼斯卢比。PolycomB组沉默复合物的层次招聘。Mol细胞。2004; 14:637-46。
- 8。
王志强,王志强,王志强。Polycomb复合体的分层招聘重新访问。核。2017;8:496 - 505。
- 9。
Kahn Tg,Dorafshan E,Schultheis D,Zare A,Stenberg P,Reim I等。PRC1和PRC2招募到Polycomb响应元件的相互依存。核酸RES。2016; 44:10132-49。
- 10。
的Endo-H男,远藤TA,的Endo-H T,矶野K,谢里夫Ĵ,大原O,等人。组蛋白H2A的单泛素化是介导发育基因的PRC1依赖性抑制以维持ES细胞身份的关键步骤。公共科学图书馆遗传学2012; 8。
- 11.
徐L,沉Wh。Polycomb Suncenting Knox基因局限于拟南芥中的芽茎细胞。Curr Biol。2008; 18:1966-71。
- 12.
陈D,李兰特A,刘C,沉WH。拟南芥PRC1样的戒指蛋白是在营养生长过程中抑制胚胎性状的必要条件。细胞res。2010; 20:1332-44。
- 13.
图尔克楼Roudier楼Farrona S,马丁-Magniette ML,纪尧姆E,Buisine N,等人。拟南芥TFL2 / LHP1特异性地与由组蛋白H3赖氨酸27.公共科学图书馆遗传学的三甲基化标记基因的关联。2007; 3:0855-66。
- 14.
张晓东,张志强,张志强,等。拟南芥组蛋白H3赖氨酸27三甲基化的全基因组分析。Plos Biol。2007; 5:1026-35。
- 15.
陈德,黄烨,阮y,沉wh。绿色谱系中PRC1核心成分的进化景观。Planta。2016; 243:825-46。
- 16。
Bemer M,Grossniklaus U.植物开发过程中PolycomB组活动的动态调节。CurrOp植物BIOL。2012年; 15:523-9。
- 17。
Calonje M,Sanchez R,Chen L,Sung Zr。胚胎花液参与拟南芥的Polycomb组介导的AG基因沉默。在线植物细胞。2008; 20:277-91。
- 18。
Mylne JS, Barrett L, Tessadori F, messnage S, Johnson L, Bernatavichute YV等。LHP1是异染色质蛋白1的拟南芥同源基因,是FLC表观遗传沉默的必需基因。中国科学院院刊2006;103:5012-7。
- 19。
Aubert D,Chen L,Moon Yh,Martin D,Castle La,Yang Ch,等。EMF1是一种新的蛋白质,参与了征集建筑和拟南芥开花的控制。植物细胞。2001; 13:1865-75。
- 20。
Bratzel女,洛佩兹托雷洪G,科赫男,德尔波索JC,在拟南芥Calonje M.饲养小区标识需要PRC1 RING指同系物,其催化H2A单泛素化。Curr Biol。2010; 20:1853-9。
- 21。
Bratzel F,Yang C,Angelova A,López-Torrejóng,koch m,del pozo jc等。新拟南芥印迹基因ATBMI1的调节需要相互作用的不同表观遗传机制。莫尔植物。2012; 5:260-9。
- 22。
拟南芥VRN1在春化和花期调控中的多重作用科学。2002;297:243-6。
- 23。
Berke L,Snel B.植物Polycomb压抑综合体1(PRC1)存在于种子植物的祖先并具有复杂的重复历史。BMC EVOL BIOL。2015; 15:1-10。
- 24。
李Z,傅X,王Y,刘r,他在植物中由BAH-EMF1复合物沉默的Polycomb介导的基因。NAT Genet。2018; 50:1254-61。
- 25。
Bantignies F,Cavalli G.Polycomb群蛋白:3D中的镇压。趋势类型。2011; 27:454-64。
- 26。
Chanvivattana Y.拟南芥开花的多元菌蛋白的相互作用。发展。2004; 131:5263-76。
- 27。
多梳组基因调控拟南芥同源性基因表达。大自然。1997;386年。
- 28。
Grossniklaus U,Vielle-Calzada J-P,Hoeppner M,Gagliana WB。母体控制胚胎发生梅德亚,APolycomb.组基因in.拟南芥.科学。1998年; 280:446-50。
- 29。
Gendall AR,利维YY,威尔逊A,在拟南芥中春化的迪安C.春化2基因介导的表观遗传调控。细胞。2001; 107:525-35。
- 30。
拟南芥(Arabidopsis thaliana)芽发育与开花的关系。植物学报2001;13:2471-81。
- 31。
Luo M,Bilodeau P,Koltunow A,Dennis Es,孔雀WJ,Chaudhury Am。控制拟南芥施肥的种子发育的基因。PROC NATL ACAD SCI。1999; 96:296-301。
- 32。
Hennig L. Arabidopsis MSI1是对生殖发展的表观遗传维持所必需的。发展。2003; 130:2555-65。
- 33。
Derkacheva M,Hennig L.主题的变化:植物中的Polycomb蛋白蛋白。J Exp Bot。2014; 65:2769-84。
- 34。
Köhler C, Hennig L, Bouveret R, Gheyselinck J, Grossniklaus U, Gruissem W. Arabidopsis MSI1是MEA/FIE Polycomb group complex的一个组成部分,是种子发育所必需的。EMBO j . 2003; 22:4804-14。
- 35.
Sheldon CC, Burn JE, Perez PP, Metzger J, Edwards JA, Peacock WJ,等。FLF MADS box基因:一个受春化和甲基化调控的拟南芥开花抑制基因。植物细胞。1999;11:445。
- 36.
Michaels S, Amasino R.开花位点C编码一个新的MADS结构域蛋白,作为开花的抑制因子。植物细胞。1999;11:949-56。
- 37.
天使A,Song J,Dean C,Howard M.基于Polycomb的交换机底层定量表观遗传记忆。自然。2011; 476:105-9。
- 38.
Sheldon CC, Rouse DT, Finnegan EJ, Peacock WJ, Dennis ES。春化作用的分子基础:春化的中心作用开花轨迹C(FLC.).PROC NATL ACAD SCI。2000; 97:3753-8。
- 39.
Lafos男,克罗尔P,Hohenstatt ML,索普FL,Clarenz O,拟南芥分化期间H3K27三甲基化的舒伯特D.动态调节。Plos Genet。2011; 7:e1002040。
- 40.
H3K27ME3,陈X,Huang H,Huang H,H3K27ME3的重新编程对于获取培养拟南芥组织的多能性至关重要。Plos Genet。2012; 8。
- 41.
杨志强,杨志强,杨志强。拟南芥受精独立的种子发育。中国科学院院刊1997;94:4223-8。
- 42。
杨C-H,Chen L-J,Sung Z. raciCopsis-armidopsis-of eMF基因作用的遗传调节。Dev Biol。1995年; 169:421-35。
- 43。
Springer NM,Danilevskaya On,Hermon P,Helentjaris TG,Phillips RL,Kaeppler HF等。多元组,ESC和E(PC)玉米同源物的序列关系,保守结构域和表达模式。植物理性。2002; 128:1332-45。
- 44。
Thakur JK,Malik Mr,Bhatt V,Reddy Mk,Sopory Sk,Tyagi Ak等。水稻的Polycomb组基因(栽培稻(L. subspecies indica), OsiEZ1,核定位蛋白的编码,在幼苗和生殖发育中优先表达。基因。2003;314:1-13。
- 45。
Hennig L,Bouveret R,Gruissem W. MSI1样蛋白:染色质组件和重塑复合物的护送服务。趋势细胞BIOL。2005; 15:295-302。
- 46。
xu WJ, Laoueillé-Duprat S, O’connell MJ, Spillane C, Grossniklaus U, Phillips AR, et al.;玉米胚乳中Mez1基因的印迹、甲基化和分子进化植物j . 2007; 49:325-37。
- 47。
陈丽娟,刁志勇,Specht C,宋志荣。含vef结构域的PcG基因在植物中的分子进化。摩尔。2009;2:738-54。
- 48。
罗m,platten d,chaudhury a,孔雀wj,dennis es。多元组基因水稻同源物的表达,印迹和演化。莫尔植物。2009; 2:711-23。
- 49。
Kapazoglou A,Tondelli A,Papaefthimiou D,Ampatzidou H,Francia E,Stanca Ma,等。大麦的表观培素染色质调味剂:IV。种子发育过程中大麦多元群组(PCG)基因及外部ABA响应的研究。BMC植物BIOL。2010; 10:73。
- 50。
陈志刚,陈志刚。多冠抑制复合物2在小麦胚乳发育中的作用。植物学报。2015;6:1-5。
- 51。
Polycomb-group介导的植物表观遗传机制。生物化学学报。2011;18:395 - 406。
- 52。
Lomax A,Woods DP,Dong Y,Bouchéf,荣Y,Mayer Ks等。在BroChypodium distachyon中适当开花需要Zeste的卷叶/增强剂的正牙齿。植物J. 2018; 93:871-82。
- 53。
肖Ĵ,徐S,利C,徐Y,兴L,牛Y等。O型的GlcNAc介导的冬小麦春化过程中VER2和TaGRP2引起TaVRN1 mRNA的积累之间的互动。NAT Communce。2014; 5:1-13。
- 54。
国际小麦基因组测序联盟(IWGSC), IWGSC RefSeq首席研究员,Appels R, Eversole K, Feuillet C, Keller B,等。利用完全注释的参考基因组改变小麦研究和育种的限制。科学。2018;361:eaar7191。
- 55。
Bennett Md,史密斯杰布。核DNA在血管植物中的量。Philos Trans R SoC Lond B Biol Sci。1991; 334:309-45。
- 56。
王志强,王志强,王志强,等。对小麦4A染色体流分选臂进行新一代测序和共线整合,揭示了染色体结构和基因含量。植物j . 2012; 69:377 - 86。
- 57。
王志强,王志强,王志强,等。大麦基因组的染色体构象捕获排序序列。大自然。2017;544:427-33。
- 58。
Monat C,Padmarasu S,Lux T,柳条T,Gundlach H,Himmelbach A等人。Tritex:带有开源工具的Triticea Genomes的染色体级序列组装。基因组Biol。2019; 20:284。
- 59.
Clavijo的BJ,范德瑞尼L,Schudoma C,Accinelli GG,Kaithakottil G,赖特J,等。一种改进的装配和异源六倍体的小麦基因组标识农艺基因的完整家庭的注释,并提供了染色体易位的基因组证据。Genome Res。2017; 27:885-96。
- 60。
姜东,王亚平,王亚平,何亚平。拟南芥多冠抑制复合物2对开花位点C和T的抑制作用。PLoS ONE。2008; 3。
- 61.
徐勇,张丽,吴刚。植物幼-成过渡期的表观遗传调控。植物学报。2018;9:1-8。
- 62.
张晓东,张志强,张志强,等。拟南芥组蛋白H3赖氨酸27三甲基化的全基因组分析。公共科学图书馆杂志。2007;5:e129。
- 63.
Farrona S,Thorpe Fl,Engelhorn J,Adrian J,Dong X,Sarid-Krebs L等人。组织特异性表达开花轨迹Tin.拟南芥独立于Polycomb组蛋白抑制。植物细胞。2011;23:3204-14。
- 64.
他g,zhu x,eaking aa,chen l,wang x,guo l等。两种稻米亚种和互惠杂交种中的全球表观遗传和转录趋势。植物细胞。2010; 22:17-33。
- 65.
Makarevitch I, Eichten SR, Briskine R, Waters AJ, Danilevskaya ON, Meeley RB, et al.;H3K27三甲基化标记的玉米兼性异染色质的基因组分布。植物细胞。2013;25:780 - 93。
- 66。
Baker K, Dhillon T, Colas I, Cook N, Milne I, Milne L,等。对大麦表观基因组的染色质状态分析显示,大麦表观基因组具有高阶结构H3K27me1和H3K27me3丰度。植物j . 2015; 84:111-24。
- 67。
春化——冷诱导的表观遗传开关。中华细胞杂志2012;125:3723-31。
- 68。
Whittaker的C,迪安℃。FLC.基因座:表观生物学和适应中发现的平台。Annu Rev Cell Dev Biol。2017; 33:555-75。
- 69。
春化诱导的谷物开花与VERNALIZATION1基因组蛋白甲基化的变化有关。中国科学院院刊2009;106:8386-91。
- 70.
阿隆索-Peral的MM,奥利弗SN,Casao MC,格里纳普AA,Trevaskis B.谷物VERNALIZATION1基因的启动子是足以转录诱导通过延长冷。PLoS ONE。2011; 6:e29456。
- 71.
Spillane C,Schmid Kj,Laoueillé-duprat S,Pien S,Escobar-Restrepo J-M,Baroux C,等。正面达尔文选择在植物中的印迹肠道基因座。自然。2007; 448:349-52。
- 72。
Mozgovái,muñiz-Viana R,Hennig L. PRC2在拟南芥植物组织中抑制激素诱导的体细胞胚胎发生。Plos Genet。2017; 13:E1006562。
- 73。
Schmid M,Davison Ts,Henz Sr,Pape Uj,Demar M,Vingron M等。拟南芥发展的基因表达图。NAT Genet。2005; 37:501。
- 74。
Beh Ly,Colwell LJ,Francis NJ。多元菌抑制复合物1的核心亚基在功能中逐渐保守但不是主要序列。PROC NATL ACAD SCI。2012; 109:E1063-71。
- 75。
Holec S,Berger F.Polycomb组复合物在植物中调解发育过渡。植物理性。2011; 158:35-43。
- 76。
Yan L,Loukoianov A,Tranquilli G,Helguera M,Fahima T,Dubcovsky J.小麦冬季化基因VRN1的位置克隆。PROC NATL ACAD SCI。2003; 100:6263-8。
- 77。
王志强,王志强,王志强,等。基于遗传算法的分子进化遗传分析。Mol Biol Evol. 2018; 35:1547-9。
- 78。
Jones Dt,Taylor WR,Thornton JM。蛋白质序列的快速产生突变数据矩阵。计算Appl Biosci。1992年; 8:275-82。
- 79。
系统发育的置信限:一种使用bootstrap的方法。进化。1985;39:783 - 91。
- 80。
expVIP:一个可定制的RNA-seq数据分析和可视化平台。植物杂志。2016;170:2172 - 86。
- 81。
Ramírez-González RH, Borrill P, Lang D, Harrington SA, Brinton J, Venturini L,等。多倍体小麦的转录景观。科学。2018;361:eaar6089。
- 82。
Letunic I, Bork P. 20年SMART蛋白域注释资源。核酸学报2018;46:493-6。
- 83。
El-Gebali S, Mistry J, Bateman A, Eddy SR, Luciani A, Potter SC,等。2019年Pfam蛋白家族数据库。核酸Res. 2019; 47:427-32。
- 84。
Katoh K,Misawa K,Kuma K,Miyata T. Mafft:一种基于快速傅里叶变换的快速多序列对齐的新方法。核酸RES。2002; 30:3059-66。
- 85.
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30:772-80。
致谢
我们感谢匿名审稿人提出的建设性建议和意见。
关于这个补充剂
本文已作为BMC Plant Biology Volume 20 Supplement 1, 2020: Selected articles from the 5th International Scientific Conference“Plant genetics, genomics, bioinformatics, and biotechnology”(PlantGen2019)的一部分发表。该补充的全部内容可在网上找到//www.cinefiend.com/articles/supplements/volume-20- supplement-1..
资金
胡说,r .ČZ.M。和J.S.获得捷克科学基金会(批准号:)资助。19-05445S)。B.S.和A.P.获得ERDF“植物作为全球可持续发展的工具”资助(cz.02.1.1/0.0 /0.0/16_019/0000827)。发表费用由捷克科学基金会资助。19 - 05445)。资助机构捷克科学基金会和ERDF在研究的设计、数据的收集、分析和解释以及手稿的撰写中没有发挥作用。
作者信息
隶属关系
贡献
Z.M.构思了这项研究。B.S.进行了手稿的生物信息学分析和技术准备。r .Č。从支架中重建核苷酸序列并进行系统发育分析。A.P.分析RNA-seq数据。J.S.对结果的解释做出了贡献。所有作者都讨论了结果,阅读并批准了最终手稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
本文已更新。原始出版物包含错误的历史日期。
电子补充材料
附加文件1
S1:表。中所标识的PcG单独组件的清单Triticum aestivum.和它们的同源物Triticum Urartu.那Triticum dicoccoides.和大麦芽.该表显示PCG组件在谷物中保存。(XLSX 21 KB)
附加文件2
:图S1。该图显示了植物PRC1和PRC2核心成分的蛋白序列。这种排列包含的蛋白质序列Triticum aestivum.那Triticum dicoccoides.那大麦,和拟南芥蒂利亚纳。保守的蛋白质结构域以不同的颜色突出显示。(PDF 13 Mb)
附加文件3.
:表S2.用于转录组分析的面包小麦PCG基因的RNA-SEQ数据。所有数据都是从小麦品种Azhurnaya收集的,并根据主要阶段(幼苗营养阶段,生殖阶段)进行分类,进一步精炼到各个发育阶段以及主要组织(根,叶子/芽,尖峰和谷物)分类.(XLSX 51 KB)
附加文件4.
:表S3.用于系统发育分析的PcG组分的蛋白序列。该表包含单子叶植物和双子叶植物,并已通过BLAST鉴定。(SLSX 160 kb)
附加文件5.
:图。S2.该图显示了在设定结构域中显示九种氨基酸交换的e(z)同源物的蛋白质对准,允许将Tae(z)寄生虫酸分裂成SWN样和ClF样基团。(PDF 661 KB)
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
Strejčková, B., Čegan, R., Pecinka, A.。et al。肝细胞抑制络合物1和2六倍体面包小麦中的核心成分的鉴定。BMC植物杂志20,175(2020)。https://doi.org/10.1186/s12870-020-02384-6
收到:
接受:
发表:
关键字
- Polycomb压抑复合体
- 表观遗传学
- PRC2.
- 小麦
- 组蛋白甲基化