摘要
背景
CRISPR/Cas近年来已成为包括植物在内的各种生物体中广泛使用的基因组编辑工具。CRISPR/Cas的应用通常需要将多个表达单元导入植物中,因此需要快速简便的克隆过程。模块化的克隆(MoClo),基于金门(GG)方法,使发展与标准化的基因克隆系统的部件,如启动子、编码序列或终端,可以很容易地交换和组装成表达单位,这在自己的又可进一步组装成高阶基因结构。
结果
在此,我们展示了一个扩展的克隆工具包,其中包含103个编码各种基于CRISPR/Cas的核酸酶及其相应的引导RNA主干的模块。在其他组件中,该工具包包括许多允许表达CRISPR/Cas核酸酶(或任何其他编码序列)的启动子以及它们在单子叶植物和双子叶植物中的指导RNA。作为工具包的一部分,我们提供了一组模块,可以在不涉及PCR步骤的情况下快速、简便地组装tRNA-sgRNA多顺反子单元。我们还证明了我们的tRNA-sgRNA系统在小麦原生质体中的功能。
结论
我们相信,目前所提供的CRISPR/Cas工具箱是一个巨大的资源,将有助于植物科学界的研究人员更广泛地采用CRISPR/Cas基因组编辑技术和模块化克隆。
背景
CRISPR/Cas技术最近已成为包括植物在内的许多生物体的易于访问的基因组编辑工具[1]在许多植物系统中,产生基因敲除已经成为一种相当简单的CRISPR/Cas应用[2那3.那4.]然而,由于植物中同源定向修复(HDR)的效率较低,更复杂的应用,如等位基因替换或靶向基因插入,仍然是一个挑战[5.].
在其传统形式中,CRISPR/Cas系统包括一个DNA核酸酶,如Cas9,它被引导RNA引导到特定的基因组位置。因此,为了进行靶向诱变足底通常,编码CRISPR/Cas核酸酶的基因使用RNA聚合酶II(Pol II)启动子(如35Sp)表达,而指导RNA在RNA聚合酶III(Pol III)启动子(如U6p或U3p)下表达,具有定义的转录起始核苷酸(“G”表示U6p或“a”表示U3p)CRISPR/Cas的优点之一是复用,即通过共表达多个特定于这些位点的导向RNA,可以在多个基因组位置靶向DNA。导向RNA可以作为单个转录单元表达,每个转录单元位于其自身的Pol III启动子下[4.,或作为tRNA-sgRNA多顺反子转录本[6.].在后一种情况下,引导rna与tRNA在单个转录本中间隔,该转录本通过植物细胞内高度保守的tRNA加工机制加工成单个引导rna [6.].
如基因组编辑在植物中的应用通常依赖于多个表达单元递送到植物细胞中,包括选择标记,一个CRISPR / CAS核酸酶编码基因和一个或多个导向的RNA,以便能够装配编码此类表达的DNA构建体是很重要的单位容易和快速。基于金门(GG)克隆法[模块化克隆(MoClo)系统7.]是高度灵活和通用性,并提供了快速的,并使用标准遗传部分,如启动子,终止子,编码序列等容易组装的多表达单元构建体的一种方式的系统已经被成功地用于基因组中的植物编辑应用程序[3.那4.那8.那9.那10.]但是缺少编码许多最新基因组编辑试剂的模块。在这里,我们报告了一个扩展的GG克隆工具包,用于单子叶植物和双子叶植物的基因组编辑应用。我们相信,该工具包将成为现有基于GG的植物基因组编辑工具的一个有价值的补充,并被世界各地的植物研究人员广泛使用社区。
结果
在这项研究中,我们生成了一组103个GG模块,使人们能够在单子叶植物和双子叶植物物种中执行基因组编辑(附加文件2:表S5)。的克隆工具包是一个除了先前公布的GG模块[7.那9.那10.那11.]和includes, among many new elements, modules encoding FnCas12a, LbCas12a, Cms1 nucleases, C-G to T-A and A-T to G-C base editors, Cas9 variants with alternative protospacer adjacent motif (PAM) specificities (SaCas9, StCas9, ScCas9 etc), Pol II and Pol III promoters, as well as guide RNA backbone modules. The latter enable insertion of the guide sequence by cloning in a pair of annealed complimentary oligos without a PCR amplification step involved. The toolkit enables assembly of CRISPR/Cas constructs that target a single as well as multiple targets with guide RNAs expressed either under individual Pol III promoters (Fig.1)或使用多顺反子tRNA-sgRNA结构(图。4.).用经过验证的tRNA的-因组系统是在单子叶植物(水稻,小麦)高效[6.那12.]和双子叶植物(拟南芥)[13.我们的模块能够直接无pcr组装多顺反子tRNA-sgRNA阵列,同时编辑多个基因组靶标,这是该工具包的亮点之一。
CRISPR / CAS核酸模块
CRISPR/Cas核酸酶0级模块包括化脓性链球菌Cas9(SpCas9;pFH13、pFH24和pFH25)以及来自其他细菌物种的Cas9变体:金黄色葡萄球菌(SaCas9;pFH14),乳酸链球菌(StCas9;pFH15)和链球菌犬属(ScCas9;pFH76)(附加文件2:表S5)。SpCas9 [14.是在包括植物在内的各种生物体中进行基因组编辑时最常用的Cas9变体。SaCas9 (' NNGRRT ' PAM)和StCas9 (' NNRGAA ' PAM)不太常见,但也成功地用于水稻、烟草[15.]和拟南芥[16.那17.那18.].ScCas9 (pFH76)、SpCas9-NG (pFH32)和spcas9衍生的xCas9 (pFH22)的特征是PAM motif需求扩大:ScCas9的' NNG ' [19],和“NG”的SpCas9-NG [20]和xCas9 [21]我们还包括了来自美国的含有Cas12a(Cpf1)CRISPR核酸酶的模块弗朗西斯新凶手弗朗西斯菌(FnCas12a; pFH16和pFH46)和紫胶螺科细菌(LbCas12a;pFH17和pFH47)以及四种相关的Cms1核酸酶(pFH18-21)(附加文件2:表S5).LbCas12a[22],FnCas12a[23]和Cms1[24]所有这些都被证明在工厂里工作。
碱基编辑器是最近添加到一系列可用的基因组编辑工具,并允许DNA碱基对的定向转换如下:25]及A-T至G-C [26],但不引入双链断裂(DSB)。前者基本编辑器是基于胞苷脱氨酶,而后者 - 在腺苷脱氨酶。现在这两个基编辑已经被证明是在各种植物,包括小麦,大米和番茄[官能27那28那29那30那31]因此,我们生成了编码胞苷脱氨酶(pFH55和pFH79)和腺苷脱氨酶(pFH45和pFH92)的基于碱基编辑器的0级模块(附加文件2:表S5)。
EvolvR CRISPR引导的易出错DNA聚合酶最近被证明能够在目标基因组位点引入随机点突变[32].基于Halperin等人(2018)的手稿[32],我们用enCas9–PolI3M–TBD(pFH77;附加文件)的小麦密码子优化版本生成了一个0级模块2这可能被证明是植物,尤其是单子叶植物逆向遗传的有用工具。
我们使用上述CRISPR/Cas 0级核酸酶模块组装25个核酸酶表达单元,插入1级GG载体,用于单子叶植物和双子叶植物(附加文件)2:表S5)。
引导RNA模块
由于CRISPR/Cas是一种RNA引导核酸酶,因此引导RNA是其必不可少的组成部分,必须与核酸酶共同表达才能实现对靶DNA的切割。引导rna通常在Pol III启动子(如U3p或U6p)下表达,这些启动子具有明确的转录起始核苷酸(分别为' a '和' G ')。CRISPR/Cas通过共同表达多个引导rna,可以同时对少数基因组位点进行靶向,模块化克隆系统非常适合于组装含有多个表达单元的结构体,如CRISPR/Cas核酸酶和引导rna。
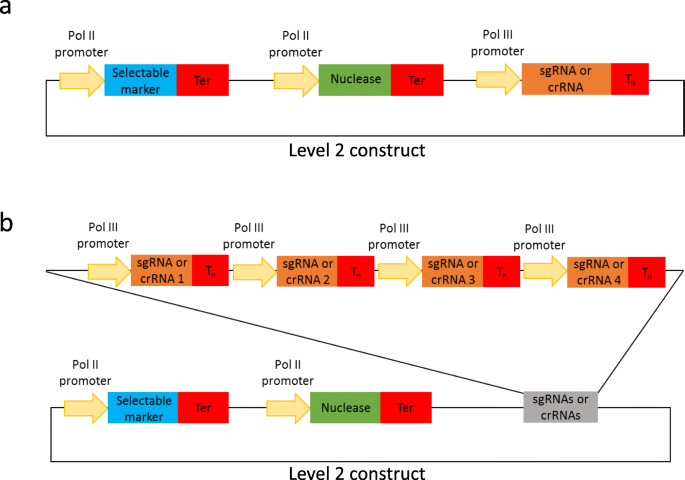
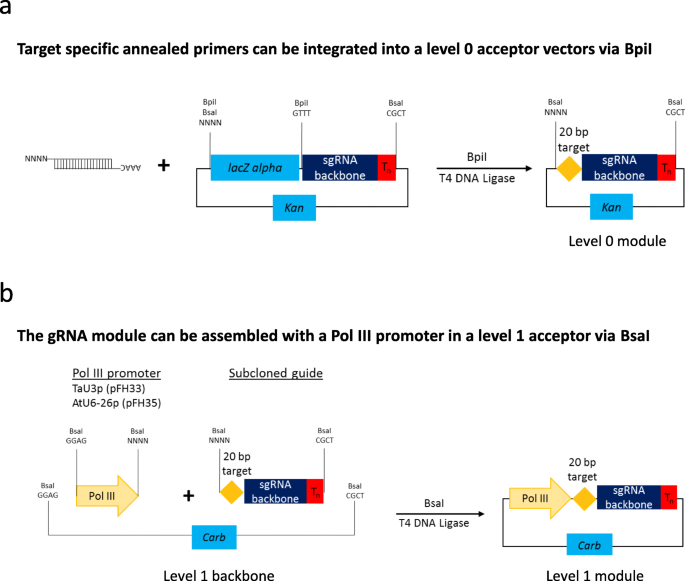
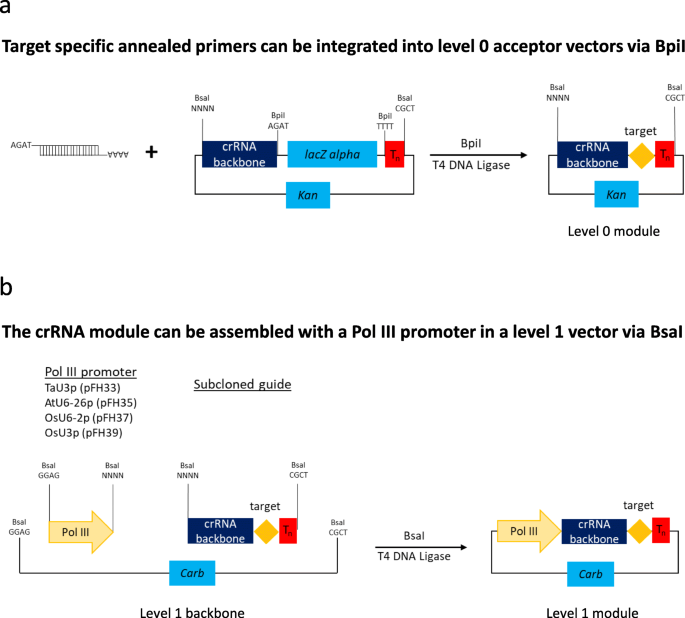
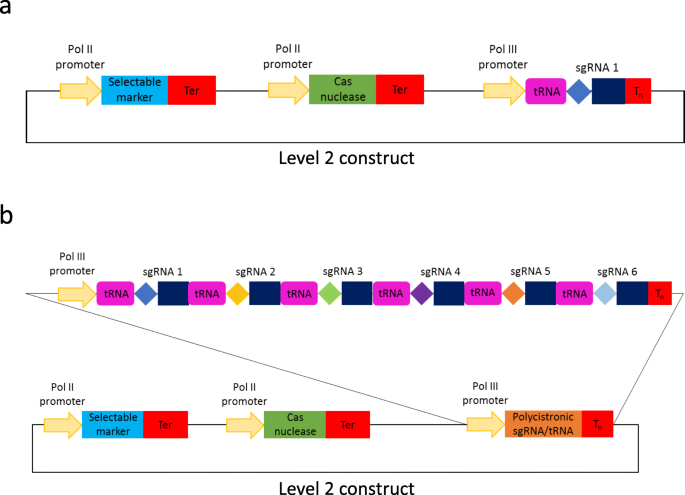
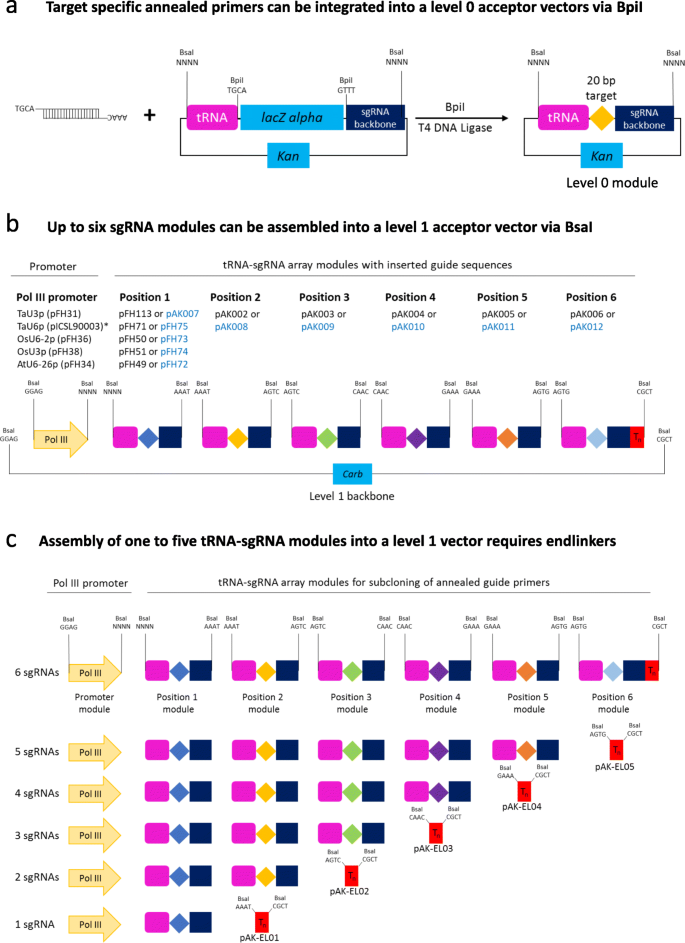
作为这项研究的一部分,我们生成了一些0级Pol III启动子模块(TaU3p、OsU3p、OsU6-2p和AtU6-26p;附加文件2:表S5)。此外,我们已经产生的多个引导RNA骨架水平0构建体,其可以被用于组装单个或多个导RNA表达单元,而不涉及PCR扩增步骤(附加文件2:表S5)。克隆系统,我们本允许下个别聚合酶III启动子(图任何明示导的RNA。1那2那3.;附加文件1:第2节;附加文件4.图S2)或从一个多顺反子tRNA- sgrna构建,其中包括sgrna与tRNA支架间距[6.(无花果。4.和5.;附加文件1:第3;附加文件4.:图S3)。使用在单个启动子下表达的引导RNA的方法能够表达CRISPR/Cas核酸酶Cas9家族的sgRNA,其在sgRNA的5′端携带引导序列(图。2),以及Cas12a (Cpf1)核酸酶和相关Cms1核酸酶的crRNA,它们在crRNA的3 '端有引导序列(图。3.).使用先前的克隆程序,可在单个Pol III启动子下组装多达四个导向RNA(图。1b) 每个多顺反子结构最多6个sgRNAs–使用后一个(图。4.b).需要注意的是,如果不需要可选择的标记,单个启动子下的引导rna数量可以增加到5个(图2)。1
b) ,或如果使用M级/P级向量,则更多[11.那36]。对于tRNA-sgRNA多顺反子结构,组装成2级目的载体的sgRNA总数可能多达24个(每1级6个),带有可选择标记,如果不使用可选择标记,则最多可达30个。同样,使用M级/P级载体可以添加30多个sgRNA[11.那36].我们的GG工具包使用户能够建立一个星期内的这种复杂的结构(附加文件1:图S1)。图中显示了携带在单个Pol III启动子下表达或作为多顺反子阵列一部分的grna的2级结构的组装管道的流程图。S2和S3(附加文件4.), 分别。
小麦原生质体tRNA-sgRNA-CRISPR/Cas构建体的检测
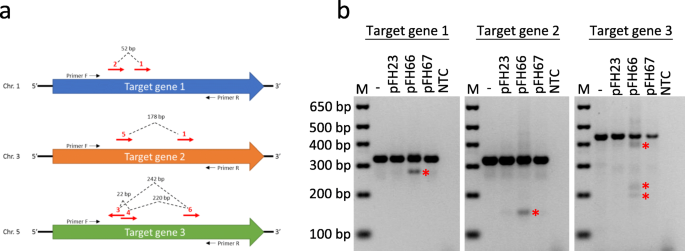
因此,GG工具包通过简化克隆过程,允许快速并行组装结构。由于使用GG构建多个CRISPR结构是一个简单的过程,因此在继续进行克隆之前,比较瞬态表达系统(如原生质体)中几个实验CRISPR设置的活动是合理的h稳定的植物转化,这可能是一个非常费时费力的过程。我们的CRISPR工具包包括三个小麦密码子优化的SpCas9版本(0级构建pFH13、pFH24和pFH25;附加文件2:表S5)及其各自的1级转录单元(pFH23、pFH66和pFH67;附加文件2:表S5)。这些变体SpCas9通过例如不同核定位信号(NLS)版本,或亲和标签。因此我们比较三个Cas9的活性通过共转化每个电平1构建体(pFH23,pFH66和pFH67)与含六个sgRNAs级别1质粒的小麦变体原生质体(图6.a)组装成tRNA-sgRNA阵列。这使得我们可以同时瞄准三个不同的小麦基因(图。6.一种)。我们已经通过至少两个与sgRNAs由于DNA条带移位预期为通过PCR检测Cas9切割位点之间大的缺失靶向每个基因如先前所述[4.]目的基因的PCR扩增显示,在用pFH66转化的原生质体中,与携带大量CRISPR/Cas诱导的缺失的等位基因相对应的明显附加条带。来自一些移位DNA条带的PCR扩增片段的Sanger测序结果可在附加文件中找到4.:图S4(.ab1序列跟踪文件在Figshare上可用,https://doi.org/10.6084/m9.figshare.11961975).相比之下,应用另外两个Cas9版本(pFH23和pFH67)会产生与携带缺失的扩增子相对应的非常微弱的条带(图)。6.b)。因此,我们的结果提示pFH66编码SpCas9的显著更高的活性,相比于其他两个Cas9变体,在小麦原生质体。
小麦原生质体中tRNA-sgRNA-CRISPR/Cas结构的检测三个小麦基因(一种)目标:目标基因1(同源基因)TraesCS1A02G338200那TraesCS1B02G350600和TraesCS1D02G340400),目标基因2(同源基因TraesCS3A02G289300那TraesCS3B02G323900和TraesCS3D02G289100)和靶基因3(同源基因TraesCS5A02G116500那TraesCS5B02G117800和TraesCS5D02G129600).红色箭头显示了20的位置 bp-sgRNA靶位。虚线表示预期的CRISPR/Cas诱导的缺失(B.)小麦原生质体与携带3个SpCas9变异(pFH23、pFH66或pFH67)之一的1级构建物(pFH94)和携带6个sgrna (一种)在tRNA-sgRNA数组中。通过PCR进行基因分型发现,与携带CRISPR/ cas诱导缺失的扩增子相对应的DNA带发生了位移(用红色星号标记)。显示的三个面板来自同一DNA凝胶的不同部分(附加文件4.:图S5)。' M '是DNA标记;' NTC '是无模板控件
讨论
在研究中提出的模块化克隆试剂盒使得能够快速和容易的装配DNA构建体对于基因组编辑在植物中的应用,并且是除了兼容GG模块先前公布的集合[7.那9.那10.那11.].所述试剂盒包括编码数,可以用来作为一种替代,以最常用的SpCas9 CRISPR / CAS核酸酶(SaCas9,StCas9,LbCas12a等)的模块。SaCas9,例如,已被证明是用于几种植物物种生成基因敲除[一个有效的工具15.那16.那17.那18.此外,已经证明可以提高植物的HDR效率[17.]由于SaCas9和StCas9具有比SpCas9更长的PAM基序,因此它们在DNA目标识别方面也可能更具特异性。
Cas12a(Cpf1)在DNA中产生交错切割[37],而Cas9 - 钝切[14.].由于这个原因,Cas12a (Cpf1)可能是CRISPR/Cas核酸酶的首选,当涉及到基于hdr的基因组编辑应用,如靶向基因插入[38那39].值得注意的是,模块化的克隆系统非常适合于基于HDR-应用作为DNA修复模板可以很容易地克隆为1级模块为等级2目的载体。Also, since Cas12a (Cpf1) has a T-rich PAM motif (‘TTTN’ for LbCas12a (LbCpf1) and ‘TTN’ for FnCas12a (FnCpf1)), it could be a better choice for targeted mutagenesis in plant species with AT-rich genomes as compared to SpCas9 (‘NGG’ PAM).
作为我们研究的一部分,我们已经生成了许多与相应的Pol III启动子和CRISPR/Cas核酸酶兼容的引导RNA主干0级模块(附加文件1:表S1)。该引导RNA骨架模块可以通过在退火对编码指导序列(图免费寡克隆被用于引导RNA表达单元无PCR装配。2和3.).当人们希望在单个启动子下表达引导RNA时,可以使用这些引导RNA骨干,最多可以将4个引导RNA组装成一个2级载体,与编码CRISPR/Cas核酸酶的1级模块和一个可选择的标记(如BAR、NPTII等;无花果。1额外的文件2:表S5,附加文件3.:表S6和附加文件4.:图S2)。我们还想指出,除了本研究中提出的插入CRISPR/Cas9导向序列的无PCR策略外(图。2a),也可以通过pcr扩增sgRNA主干(在其5 '端携带20 bp引导序列的正向引物),并将其置于Pol III启动子下,如前面所述[8.].后一种策略节省了一个克隆步骤,但可能不适用于Cas12a的情况,因为Cas12a的crRNA主干(大约40个核苷酸)可能太短,无法进行PCR扩增。
除了在自己的启动子下表达每个引导RNA,我们还生成了模块,允许多顺反子tRNA-sgRNA结构的组装,使用单个Pol III启动子表达多达6个sgrna(图)。4.).tRNA的-因组系统,最初由解等人描述。(2015)在水稻,后来被成功地在小麦施加[12.]和拟南芥[13.].然而,之前报道的tRNA-sgRNA系统依赖于一个相当繁琐的DNA构建组装过程,因为它涉及到携带重复序列的DNA片段的PCR扩增。我们已经生成的0级GG模块(图2)。5.和附加文件1表S3)可以直接、高效地组装tRNA-sgRNA阵列,而不需要PCR步骤。该系统提供了五个单子叶和双子叶Pol III启动子的选择,其中TaU6p是一个已发布的模块[35],以及两种不同的SpCas9 sgRNA骨干(经典的和改进的;附加文件1:表S3)。
因为稳定转化仍然是许多植物基因组编辑应用的主要瓶颈,包括像小麦这样的主要作物[40,在启动一个通常是漫长和劳动密集型的稳定转化过程之前,能够在瞬时表达系统(如原生质体)中检测CRISPR/Cas结构的活性是非常有利的。利用小麦原生质体系统,我们比较了三个不同的小麦密码子优化SpCas9变异的活性(图。6.),主要在c端nss上有所不同。事实上,其中一个结构(pFH66)比其他两个(图。6.b)中可能是由于在组蛋白H2B NLS,位于pFH66变体的C-末端,处于进口Cas9进入细胞核相比在其他两个构建体的SV40或nucleoplasmin NLS存在更有效。不同的NLS版本和Cas9活动之间可能存在的联系在拟南芥此前报道9.].
用于Cas9多路复用的tRNA-gRNA系统已被证明在单子体中有效[6.那12.]和双子叶植物一样[13.].在双子体中,研究表明,将trna与优化后的sgRNA主链融合[33]增加编辑效率[13.].然而,在单子叶植物中,只有典型的sgRNA主干[34]小麦原生质体分析使我们能够在单子叶植物物种中验证携带优化的sgRNA主干的tRNA-sgRNA阵列的功能。我们的结果表明,使用优化的sgRNA主干组装的tRNA-sgRNA阵列也可以在单子叶植物物种中产生更高的CRISPR/Cas效率同时,我们意识到,由于Cas9/sgRNA表达模式的差异以及DNA修复能力的差异,使用原生质体获得的结果可能不一定反映稳定转基因系的情况。
方法
DNA结构组装
根据制造商的说明,使用Q5®DNA聚合酶(新英格兰生物实验室)进行所有PCR扩增。根据所述方案(附加文件)进行所有GG切割连接反应1:第1部分)。
所有连接都转化为One Shot™TOP10化学活性大肠杆菌(赛默飞世尔科技),并构建物测序(欧陆基因组学)来验证。
提供了本研究中报告的所有GG模块组装相关的具体细节(附加文件1:第4节)。研究中使用的所有PCR引物的序列见表S4(附加文件)1).作为本研究的一部分生成的所有DNA构建物均存放在Addgene中(www.addgene.org)与Addgene公司的ID对于每个构建体所指示(附加文件2:表S5)。包含95个结构的工具包也将在96孔板上提供,参考编号为1000000159。其余的材料(103个结构中的8个)可以通过联系相应的作者来要求。所有构造的序列信息可以在GenBank (.gb)文件中找到www.addgene.org及在Figshare (https://doi.org/10.6084/m9.figshare.11961975).
原生质体分析
Protoplasts were isolated from etiolated 10 day old wheat seedlings (cv. Cadenza, sourced from CPB Twyford Ltd) as previously described [41]经过一些修改后,从Duchefa Biochemie(荷兰哈勒姆)获得纤维素酶R10和马其顿酶R10,并在26℃进行酶消化 摄氏四度 h、 随后,50000个原生质体,体积为100 μL转化为20 每个质粒μg(2 μg/μL),用质粒Maxi试剂盒(德国Qiagen)纯化。一批原生质体用等量的水处理,用作阴性对照(未转化)。然后,原生质体在6孔板中培养60天 h、 通过使用DreamTaq DNA聚合酶(ThermoFisher Scientific)和引物对FH432/FH433(目标基因1)、FH436/FH437(目标基因2)和FH440/FH441(目标基因3)对目标基因进行PCR扩增,分离DNA并检测CRISPR/Cas诱导的突变(附加文件1:表S4)。测序时,根据制造商的说明,使用Q5®DNA聚合酶(新英格兰生物实验室)和之前相同的引物对扩增目标基因。PCR产物用零钝体法进行亚克隆™ 地形™ PCR克隆试剂盒(Thermo Fisher Scientific)。使用引物FH48(附加文件)对单个克隆进行Sanger测序(Eurofins基因组学)1:表S4)。使用Geneious®版本10.2(生物标记)对齐并可视化测序文件。
数据和材料的可用性
大多数材料(DNA结构)可通过Addgene (www.addgene.org)下于表S5中指定个别的参考数字(附加文件2).包含95个结构的工具包也将在96孔板上提供,参考编号为1000000159。其余的材料(103个结构中的8个)可以通过联系相应的作者来要求。DNA构建序列文件可在GenBank格式www.addgene.org和Figshare(https://doi.org/10.6084/m9.figshare.11961975).图S4所示的Sanger序列读取可在Figshare (https://doi.org/10.6084/m9.figshare.11961975).
缩写
- CRISPR / CAS:
-
聚集的规则间隔的短回文重复序列/CRISPR相关
- crRNA:
-
CRISPR RNA
- 双边带:
-
双链断裂
- GG:
-
加洲的金门大桥
- HDR:
-
同源定向修复
- MoClo:
-
模块化克隆
- 帕姆:
-
Protospacer相邻主题
- 因组:
-
单导RNA
工具书类
- 1。
Belhaj K,查加西亚A,Kamoun S,赞助新泽西州,涅克拉索夫五,编辑植物基因组与CRISPR / Cas9。CurrOgin Biotechnol。2015; 32:76-84。
- 2.
哈恩楼曼特加扎O,格雷纳A,HEGEMANN P,Eisenhut男,韦伯APM。一种有效的视觉屏幕拟南芥CRISPR / Cas9活性。前植物科学。2017; 8。https://doi.org/10.3389/fpls.2017.00039。
- 3.
Nekrasov V,Staskawicz B,Weigel D,Jones JD,Kamoun S.使用Cas9 RNA引导的内切酶在模式植物烟草中进行靶向突变。Nat Biotechnol.2013;31:691–3。
- 4.
涅克拉索夫V,王C,运Ĵ,兰兹C,威格尔d,Kamoun S.快速产生通过基因组缺失一个自由转基因白粉病抗性番茄的。SCI代表2017; 7:482。
- 5.
Hahn F,Eisenhut M,Mantegazza O,Weber APM.基于Cas9基因打靶的拟南芥中缺陷无毛基因的同源性定向修复.前沿植物科学杂志,2018;9。https://doi.org/10.3389/fpls.2018.00424。
- 6.
谢凯,杨志强,杨志强。利用内源性trna加工系统增强CRISPR/Cas9多重编辑能力的研究。中国科学院院刊2015;112:3570-5。
- 7.
一种用于多基因结构标准化组装的模块化克隆系统。《公共科学图书馆•综合》。2011;6:e16765。
- 8.
布鲁克斯C,涅克拉索夫V,李普曼ZB,在番茄中的第一代使用群集规则间隔开的短回文重复序列/ CRISPR-Associated9系统凡埃克J.高效基因编辑。植物理性。2014; 166:1292-7。
- 9.
Castel B,Tomlinson L,Locci F,Yang Y,Jones JDG.拟南芥中Cast9介导的突变的T-DNA结构优化.公共科学图书馆,2019;14:e0204778。
- 10。
Raitskin O,Schudoma C,西A,赞助新泽西州。CRISPR相关(CAS)核酸酶效率和特异性的植物比较:用于精密基因组工程的扩展工具包。Plos一个。2019; 14:e0211598。
- 11.
Engler C,Youles M,Gruetzner R,Ehnert T-M,Werner S,Jones JDG等。植物的金门模块化克隆工具箱。ACS Synth Biol.2014;3:839–43。
- 12
王伟,潘强,何凡,赵胜,等。跨代CRISPR-Cas9活性促进异源多倍体小麦的多基因编辑。CRISPR Journal. 2018; 1:65-74。
- 13
张Q,邢H-L,王志平,张海义,杨菲,王X-C,等。CRISPR/Cas9在小鼠体内诱导的潜在高频离靶突变拟南芥《植物分子生物学》2018;96:445–56。
- 14
Jinek M,Chylinski K,Fonfara I,Hauer M,Doudna JA,Charpentier E.自适应细菌免疫中的可编程双RNA引导DNA内切酶。科学。2012;337:816–21。
- 15
Kaya H,Mikami M,Endo A,Endo M,Toki S.使用金黄色葡萄球菌Cas9。Sci众议员2016;6:26871。
- 16
斯坦纳特Ĵ,Schiml S,Fauser女,使用来自嗜热链球菌和金黄色葡萄球菌Cas9直向同源物普赫塔H.高效遗传植物基因组工程。植物杂志2015; 84:1295-305。
- 17
Wolter F,Klemm J,Puchta H.利用金黄色葡萄球菌Cas9核酸酶的卵细胞特异性表达在拟南芥中有效地进行植物基因靶向。植物杂志,2018;94:735–46。
- 18
Schmidt C,Pacher M,Puchta H.利用CRISPR/Cas系统有效诱导植物基因组中的可遗传倒位.植物杂志,2019;98:577–89。
- 19.
Chatterjee P,Jakimo N,Jacobson JM.高度相似的SpCas9同源基因的最低PAM特异性。Sci Adv.2018;4:eaau0766。
- 20。
Nishimasu H,Shi X,Ishiguro S,Gao L,Hirano S,Okazaki S等。扩大靶向空间的工程CRISPR-Cas9核酸酶。科学。2018;361:1259–62。
- 21。
胡JH,米勒SM,Geurts MH,唐W,陈蕾,孙N,等人。演化的CAS9变体,具有宽PAM兼容性和高DNA特异性。自然。2018; 556:57-63。
- 22。
唐X,Lowder LG,张T,Malzahn AA,郑X,Voytas DF,等.植物中高效基因组编辑和转录抑制的CRISPR–Cpf1系统.自然植物.2017;3:17018。
- 23。
Endo A,Masafumi M,Kaya H,Toki S.利用来自日本的Cpf1对水稻和烟草基因组进行有效的靶向诱变弗朗西斯新凶手弗朗西斯菌.Sci Rep.2016;6:38169。
- 24
Begemann MB,灰色BN,一月E,辛格A,凯斯勒直流,他Y,等人。表征和用于体内基因组编辑2个核酸酶的新颖组类型V,类的验证。生物XIV。2017年:192799。https://doi.org/10.1101/192799。
- 25
Komor AC,Kim YB,Packer MS,Zuris JA,Liu博士。基因组DNA中目标碱基的可编程编辑,无需双链DNA切割。自然杂志。2016;533:420–4。
- 26
Gaudelli NM, Komor AC, Rees HA, Packer MS, Badran AH, Bryson DI,等。基因组DNA中A•T到G•C的可编程碱基编辑,无需DNA切割。大自然。2017;551:464 - 71。
- 27
Shimatani Z, Kashojiya S, Takayama M, Terada R, Arazoe T, Ishii H,等。利用CRISPR-Cas9胞苷脱氨酶融合技术在水稻和番茄中的靶向碱基编辑。Nat生物技术。2017;35:441-3。
- 28
宗颖,王颖,李超,张锐,陈凯,冉艳,等。精准的碱基编辑水稻,小麦和玉米与cas9 -胞苷脱氨酶融合。Nat生物技术。2017;35:438-40。
- 29
宋强,李超,金胜,张东,王勇,等。利用nCas9和人类APOBEC3A的融合在植物中进行高效的c -t碱基编辑。生物科技Nat》。2018;36:950-3。
- 30。
李超,宗义,王勇,金胜,张东,宋强,等。利用cas9 -腺苷脱氨酶融合扩大水稻和小麦中的碱基编辑。基因组医学杂志。2018;19:59。
- 31。
华坤,陶鑫,朱建坤。利用Cas9变种扩大水稻碱基编辑范围。Plant Biotechnol J. 2019; 17:499-504。
- 32。
霍尔珀林SO,头CJ,黄EB,Modavi C,谢弗DV,Dueber的JE。CRISPR制导DNA聚合酶能够在可调谐窗口的所有核苷酸的多样化。自然。2018; 560:248-52。
- 33。
党勇,贾光国,蔡建军,马海峰,叶春华,等。优化sgRNA结构,提高CRISPR-Cas9敲除效率。基因组医学杂志。2015;16:280。
- 34。
Mali P,Yang L,Esvelt KM,Aach J,Guell M,DiCarlo JE,等。通过Cas9进行RNA引导的人类基因组工程。科学。2013;339:823–6。
- 35。
李春华,李春华,李春华,等。利用rna引导的Cas9核酸酶在大麦和甘蓝中诱导靶向的遗传突变。基因组医学杂志。2015;16:258。
- 36
沃纳S,恩氏C,韦伯E,Gruetzner R,Marillonnet S.快车道使用金门克隆和MoClo系统的多基因构建体的组装。生物工程。2012; 3:38-43。
- 37
Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, et al。Cpf1是一个2类CRISPR-Cas系统的单个rna引导内切酶。细胞。2015;163:759 - 71。
- 38
Begemann MB,灰色BN,一月E,戈登GC,他Y,刘H,等人。精确插入和使用Cpf1 CRISPR核酸酶高等植物基因组的指导编辑。SCI代表2017; 7:11606。
- 39
沃尔特楼普赫塔H.在植物中基因靶向可以通过使用CRISPR / Cas12a来增强。植物杂志2019; 100:1083至94年。
- 40.
Sparks CA, Jones HD。小麦粒子轰击的遗传转化。印于:Henry RJ, Furtado A, editors。谷物基因组学:方法和协议。Totowa, NJ: Humana出版社;2014.201 - 18页。https://doi.org/10.1007/978-1-62703-715-0_17。
- 41.
单Q,王燕,李杰,高聪。利用CRISPR/Cas系统在水稻和小麦中编辑基因组。Nat Protoc.2014;9:2395–410。
致谢
我们感谢Lucy Hyde(英国布里斯托尔大学)在管理载体数据库方面的贡献,以及她在建立原生质体协议方面的帮助。我们感谢Mark Youles和Laurence Tomlinson (Sainsbury Laboratory, Norwich, UK),以及Sylvestre Marillonnet (Leibniz Institute of Plant Biochemistry, Halle, Germany)的金门DNA结构。我们感谢Robert Hoffie, Ingrid Otto和Nagaveni Budhagatapalli (IPK Gatersleben, Germany)在原生质体检测方面提供的有用建议。
资金
Vladimir Nekrasov(Rothastd Research)获得了生物技术和生物科学研究委员会(BBSRC)设计未来小麦(DFW)的资助作者声明,资助机构在研究设计、数据收集、分析和解释以及撰写手稿方面没有任何作用。
作者信息
隶属关系
贡献
VN构思了手稿的构思;FH设计了pfh结构并进行了实验;AK设计pak结构并辅助克隆;LSL与FH一起进行原生质体实验,并辅助克隆;VN和FH撰写了手稿。所有作者阅读并批准了手稿。
通讯作者
道德宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者声明他们没有相互竞争的利益。
额外的信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充资料
附加文件1:
补充方法和协议。图S1。2级CRISPR/Cas结构组装的时间表。表S1。一系列编码模块引导与CRISPR/Cas核酸酶和Pol III启动子匹配的RNA主干。表S2。引物设计用于将引导序列插入到各自的引导RNA骨干模块中。表S3。tRNA-sgRNA序列中的Pol III启动子模块和匹配位置1模块。表S4。在这项研究中使用的引物。
附加文件2:表S5。
本研究期间创建的GG结构列表。
附加文件3:表S6。
其他组创建的附加GG构造。
附加文件4:图S2。
的流程图,示出2构建体与下个别聚合酶III启动子表达水平gRNAs的组装。图S3。一个流程图说明了用tRNA-sRNA多顺反子单位表达的GRNA组装2级结构。图S4。Sanger测序显示了小麦原生质体中由CRISPR/Cas引起的基因缺失。图S5。的DNA凝胶的未处理的图像在图1中。6.湾
权利和权限
开放存取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条中提供的数据,除非数据信用额度中另有规定。
关于这篇文章
引用这篇文章
哈恩,F.,科罗列夫,A.,桑朱尔乔·卢雷斯,L。et al。用于植物基因组编辑的模块化克隆工具包。植物生物学20,179 (2020). https://doi.org/10.1186/s12870-020-02388-2
已收到:
公认:
发表:
关键字
- CRISPR
- Cas9
- 植物
- 基因组编辑
- 金门
- MoClo