摘要
背景
红树林已经适应了潮间带——陆地和海洋生态系统之间的界面。各种研究表明,红树林在生理、生态和基因组水平上都发生了适应性进化。然而,这些研究很少关注通过转录组谱对盐适应的基因调控。
结果
我们对转录组进行测序Sonneratia阿尔巴在低盐(淡水)、中盐(海水盐度的一半)和高盐(海水盐度)条件下,研究了盐适应的潜在转录调控。在叶片组织中,64%的潜在盐相关基因在盐度从淡水增加到中等水平时表达不差异,而在盐度进一步增加到海水水平时表达上调或下调,表明这些基因很好地适应了中等盐度条件。我们推断细胞环境稳态的维持和调节都是重要的适应过程美国阿尔巴.i)叶片中硫代谢、黄酮和黄酮醇生物合成KEGG通路在基因上调时显著富集。它们都参与清除ROS或植物体内渗透相关代谢产物的合成和积累。ii)转录因子编码基因在上调转录本中的比例显著增加。在高盐条件下,耐盐相关TF家族高表达。(3)部分基因在盐胁迫下表达上调,在氨基酸水平上表现出适应进化的迹象,可能有助于适应波动的潮间带环境。
结论
本研究首次通过盐梯度实验在全转录组水平上阐明了红树林的高盐适应机制。结果表明,在高盐环境的适应过程中,有若干候选基因(包括盐相关基因、tf编码基因和PSGs)和主要途径参与。我们的研究也为未来极端环境下的适应性进化研究提供了有价值的资源。
背景
物种如何适应极端环境是进化生物学的一个基本课题。潮间带位于陆地生态系统和海洋生态系统的交界面,给当地生物带来许多恶劣或极端的环境,包括高盐度、缺氧、盐度波动、强紫外线和高温[1].然而,红树林已经移居到这些栖息地,并很好地适应了这些栖息地,进化出一系列高度特化的特征,包括耐盐性、胎生胚胎、气根和高单宁含量[2,3.,4].红树林是海洋盐生植物的一个主要群体,它们在高盐度环境中茁壮成长并完成它们的生命周期,这是由于海水水位波动造成的。因此,它们是研究适应性进化的良好模型系统。事实上,这一群体的适应性已经在表型、生态、生理和基因组水平上进行了研究[5,6,7,8,9,10].然而,尽管转录调节已被强烈选择用于快速适应环境条件的变化,并可能在生态和进化时间尺度上发挥关键作用,但很少有人关注它[11].
盐适应是一个长期的动态过程,由多个基因参与许多形态学、生理、分子和细胞过程[12,13,14].通过对模式植物的基因分析,发现了多种盐反应基因和重要的信号通路拟南芥[12,15],以及农作物水稻[16]和大豆[17].盐过度敏感(SOS)信号通路Ca2 +依赖性信号通路和ABA信号通路有助于介导盐胁迫下的细胞信号通路,维持细胞环境稳态[18].许多分子机制已经在糖植物中被揭示,但主要的限制因素是它们不能在高和波动的盐度中生存。这些研究还不足以理解植物盐适应的分子方面。
Sonneratia阿尔巴栖息在下游河口系统的低潮间带,是最普遍和耐盐的红树林物种之一。它在5 - 50%的海水中生长最佳,表明它有耐受高盐度和低氧的能力[19].它能合成有机溶质以调节细胞渗透势,并产生抗氧化酶以清除活性氧(ROS)以对抗高盐度[20.].因此,研究盐适应的遗传机制是一种很有吸引力的生态模式。然而,只有有限的RNA-seq数据来自Sonneratia已报道至今[21,22].转录组信息的缺乏美国阿尔巴跨越盐度条件是识别适应盐环境的分子基础的主要障碍。在本研究中,我们设计了一个实验来阐明高盐适应的转录调控美国阿尔巴.我们对叶和根的转录组进行了测序美国阿尔巴用人工海水灌溉幼苗。在基因组引导的转录组组装后,我们在低、中、高盐度条件下对转录组进行了表征,以进行进一步的功能分析。我们希望从遗传学、生物化学和生理学等方面对植物盐适应的潜在机制有新的认识美国阿尔巴.我们的研究也为进一步研究极端环境下红树林的适应性进化提供了宝贵的资源。
结果
鉴别差异表达基因和表达模式
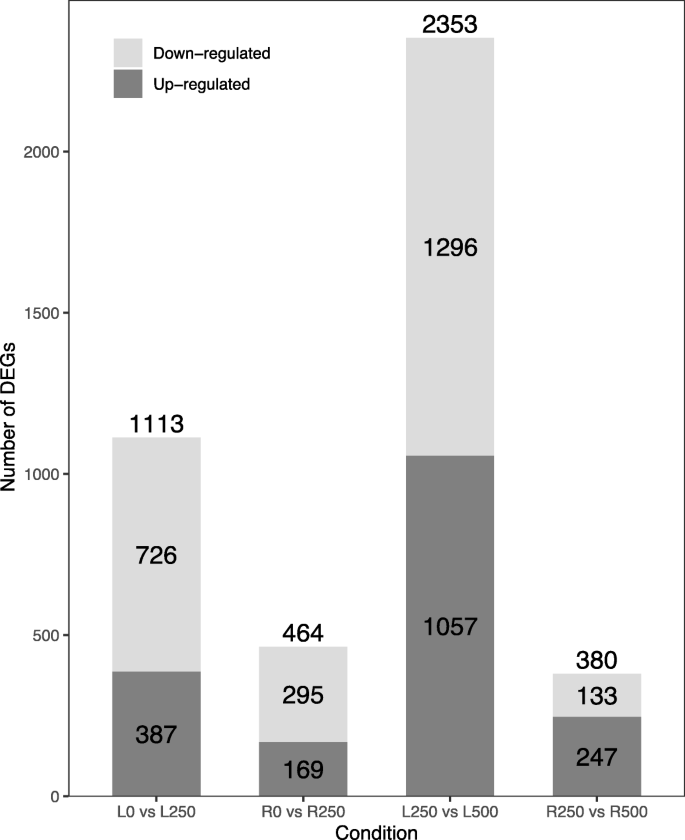
生成基因组资源,并开始阐明植物耐盐机制美国阿尔巴,我们对在不同盐水平培养基中饲养的植物的叶子和根进行了RNA-seq实验。表中列出了读取汇总统计信息和组装结果1.我们从12个库中生成了4420到6197万个100 bp的配对端读取。质量过滤后(见方法我们保留了41.94 ~ 5921万个99 bp的高质量reads,并成功地将其中93.85 ~ 94.83%的reads映射到美国阿尔巴参考基因组。这些测序结果分别在每个样本中进行组装,得到33,930 ~ 40,937个转录本,平均长度为3298 ~ 3530 bp, N50值为4214 ~ 4437 bp1).各条件下生物重复间的相关系数为0.87 ~ 0.971:图S1)。
为美国阿尔巴,其对潮间带环境的适应是一个长期的动态过程,其生长环境为海水浓度的5 ~ 50%,即25 mM ~ 250 mM [19].因此,500 mM和0 mM分别提供了高盐(压力)和低盐条件。大量介于0和250之间的基因有望具有相似的表达水平。应力在250毫米到500毫米之间。接下来,我们对转录本丰度进行了两组比较:低盐浓度(0 mM) vs中盐浓度(250 mM),中盐浓度vs高盐浓度(500 mM)方法详情)。在低-中NaCl处理中,叶片中显著上调基因387个,下调基因726个,根系中显著上调转录本169个,下调转录本295个。在对比中盐和高盐条件时,我们在叶片中发现了1057个上调基因和1296个下调基因,在根系中发现了247个上调转录本和133个下调转录本。1).叶片中差异表达基因(DEGs)明显多于根系。此外,我们发现中盐到高盐对比中比低盐到中盐对比中有更多的上调基因。
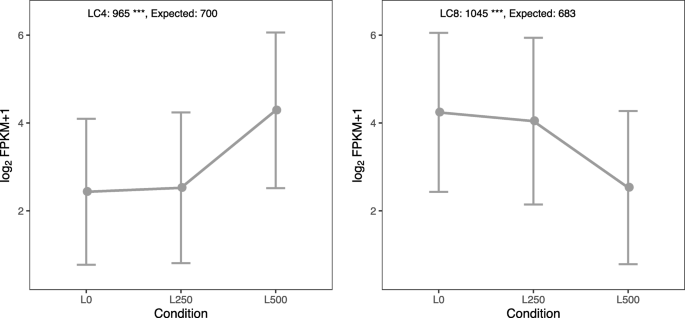
为了确定DEGs之间的共表达模式,我们根据它们在盐度条件和组织中的表达变化将它们分为8组(附加文件)2:图S2)。有趣的是,两个最大的叶片簇,LC8(1045个基因)和LC4(965个基因),仅由在高盐处理下显著下调或上调的基因组成。2).然而,同样的模式并不适用于根。为了评估LC8和LC4簇是否偶然比预期的更大,我们进行了置换试验(见方法详情)。我们确实看到这些簇比预期的要大。2).大多数基因在不同盐度条件下表达没有差异。LC4和LC8在所有8个星团中占大多数(64%)。只有当盐水平变高(500 mM)时,它们才有差异表达,但在中低NaCl比较中没有变化。因此,只有极高水平的盐处理才能显著改变美国阿尔巴转录组,正如预期的,考虑到这个物种的高耐盐性。
功能富集和转录调节
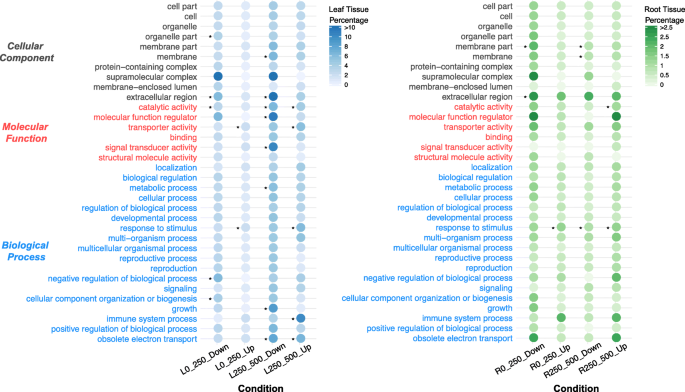
我们将功能基因本体(GO)分类分配给我们识别的deg(附加文件)3.表S1)和四个主要的共表达集群,包括LC4, LC8, LC4-RC4 (LC4和RC4之间重叠),LC8- rc8(附加文件4:表S2)。使用全基因组注释作为背景,我们发现在我们的差异表达基因中有几个类别是过度代表的(图。3.).值得注意的是,叶片中上调的许多基因涉及分子功能类别“转运蛋白活性”。生物过程类别“对刺激的反应”在所有四种比较(包括叶和根)中都显著过量。此外,在4个比较中,许多上调基因被分配到“对刺激的反应”类别(L0 vs L250 57个,R0 vs R250 35个,L250 vs L500 158个,R250 vs R500 36个)。对上调基因也进行了详细的注释。我们注意到许多基因都被注释了与耐盐有关的功能,特别是钠/氢交换器(NHX)编码基因(SA_23236L0 vs L250和L250 vs L500SA_23120(R250 vs R500),膜层固有蛋白(TIP)编码基因(SA_14179(L250 vs L500),质膜固有蛋白(PIP)编码基因(SA_2543L0 vs L250, L250 vs L500,SA_09545L0 vs L250,SA_08276,SA_20957,SA_24093,SA_26218,SA_26346L250 vs L500),Mangrin(SA_08086,SA_14631L250 vs L500),几种热休克蛋白编码基因,以及主要转录因子编码基因(下一节描述)。Mangrin与AOC编码基因部分同源。一些基因在高盐环境下上调,直接调节细胞渗透电位或整合代谢、激素和环境信号,以适应逆境。它们包括谷胱甘肽过氧化物酶编码基因(SA_08946在根和SA_18901在叶片中,单脱氢抗坏血酸还原酶编码基因(SA_20647在叶片中),醛酮还原酶编码基因(SA_02762在两种组织中,SA_02758,SA_02759在树叶中,和SA_15935在根中),在天冬酰胺、半胱氨酸、半乳糖醇、海藻糖-6-磷酸和水苏糖生物合成中的一些关键酶。我们还发现了未知功能的转录本上调。这些可以进一步研究,以获得盐适应的新见解。
有趣的是,在叶片中上调的转录本中,有965个属于LC4簇,在上调的158个基因中,有89.24%的基因被分配到“对刺激的反应”GO类别(附加文件)4:表S2)。这表明该簇在应对异常高盐胁迫中起着重要作用。
在下调基因中显著富集的GO术语是叶片中的“细胞外区域”和“催化活性”类别,以及根系中的“膜部分”(图2)。3.).此外,在叶片中下调的转录本中有1045个属于LC8簇,在“碳水化合物代谢过程”、“细胞外围”、“分子功能负调控”、“催化活性”等类别中富集(附加文件)4:表S2)。它们与基础代谢、植物生长调节、分子功能调节、信号换能器活性和电子传递密切相关。
接下来,我们通过交叉引用我们的上调基因列表和的KEGG注释,研究了在DEGs中显著富集的代谢途径美国阿尔巴参考基因组(附加文件5:表S3)。在高盐下差异表达的转录本中,代谢途径的过度表达(4个高度富集,3个轻度富集,表2)、硫代谢、内质网蛋白质加工、黄酮和黄酮醇生物合成是最有趣的,因为它们已知参与其中美国阿尔巴盐适应。黄酮生物合成、烟酸盐和烟酰胺代谢途径在根高盐处理下上调的基因中也有过高的表达(表2)2).与低盐条件相比,在培养基中表达差异的基因中,没有代谢途径突出。
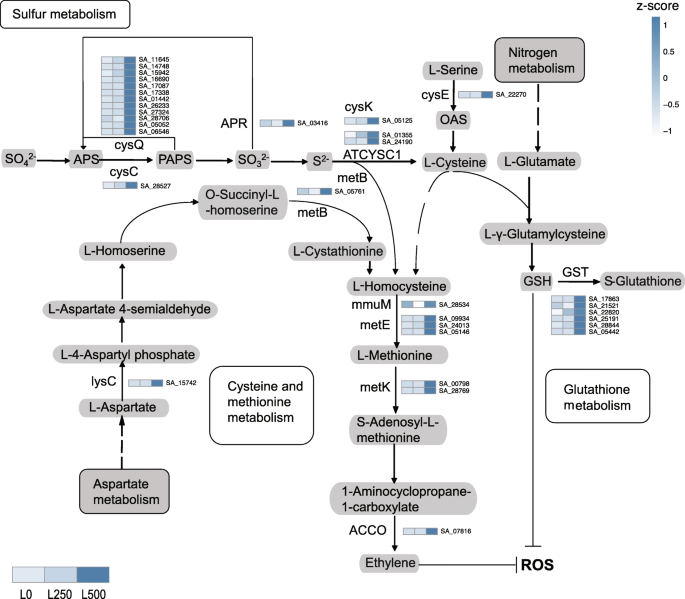
为了进一步探索盐处理下生物通路的调控,我们可视化了主要通路转录本中上调基因的表达谱(图2)。4).突出的途径涉及活性氧(ROS)清除,解毒和调节细胞渗透压。我们认为这些途径在盐适应中起着重要作用。值得注意的是,这些功能富集分析指出细胞环境是分子盐适应的可能位点美国阿尔巴.这似乎是合理的,因为候选蛋白质发生在细胞内。
参与盐适应的转录因子
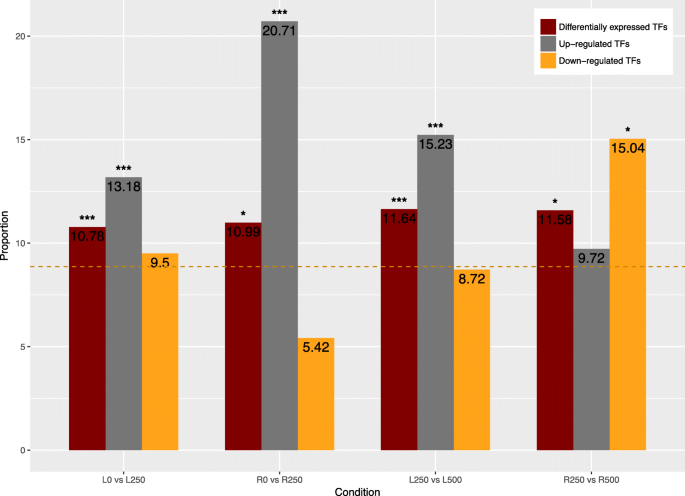
转录因子(Transcription factors, TFs)是一种调节蛋白,因此有可能通过影响下游的几个因子来调节盐适应的多个方面。以往的研究表明,tf在非生物应激反应和适应中发挥着重要作用[23,24,25,26,27].有趣的是,我们发现更多的转录因子,作为所有注释基因的比例,在美国阿尔巴(2595, 8.86%)和美国caseolaris(2729, 9.72%)p . granatum(1847年,6.34%)和大肠茅(2185年6.01%)。此外,在我们进行的所有对比中,TF编码基因在差异表达基因中被过度表达,但在叶片中尤其如此。在3个对比中,叶片对比和根系低盐和中盐条件下,tf的上调模式均显著,在根系中盐和高盐条件下,几个下调的tf略有富集(图2)。5).将转录因子分为公认的家族(附加文件)6:图S3),我们发现AP2/EREBP,南汽,WRKY,bZIP家族在上调基因中富集。
不同盐度对比(0 mM vs 250 mM NaCl和250 mM vs 500 mM NaCl)下叶片和根系中编码基因的差异表达转录因子的比例。橙色虚线表示编码基因的转录因子占整体的比例美国阿尔巴基因组。红柱表示差异表达的tf在所有DEGs中的比例。灰色条形图表示tf在上调基因中的比例。橙色条形图表示tf在下调基因中的比例。采用Fisher精确检验(Fisher’s exact test)评价差异表达/上调/下调转录因子与转录因子编码基因在全基因组中比例差异的显著性。P-value范围用single (corrected .)表示P-value < 0.1)或triple(已校正P-value < 0.05)星号
上调基因的正向选择
不寻常的耐盐性是红树林的一个派生特性。Ka/Ks值沿美国阿尔巴及其相关的计算(附加文件7:图S4)。结果表明,土壤中Ka/Ks比值升高美国阿尔巴相对于亲戚。因此,我们可以预计,一些因NaCl处理而上调的基因是在最近的进化过程中获得它们的作用的。我们想知道它们的氨基酸组成是否随着表达水平的变化而变化。为了验证这一想法,我们使用PAML包来识别阳性选择基因(PSGs)美国阿尔巴(见方法详情)。我们确定了16个上调的psg(附加文件8:表S4)。的SA_12151基因座Ka/Ks比值特别高(Ka/Ks = 4.42),可能发生正向选择。功能注释使我们进一步相信这些蛋白与耐盐性有关。
实时定量PCR (qPCR) DEG验证
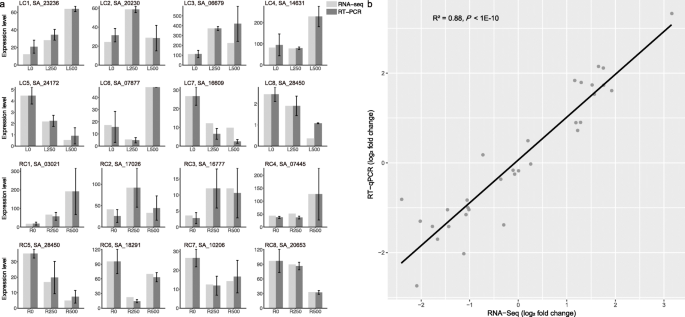
为了评估RNA测序对DEG鉴定的可靠性,每个聚类中16个基因的转录本用qPCR进行了评估。所选基因的表达模式与来自叶和根的8个簇的RNA-seq数据一致。qPCR结果如图所示。6a.日志与日志之间的相关性2RNA-seq实验和qPCR的表达量的倍增变化如图所示。6b.这些方法之间的相关性强且正(R2= 0.88,P-value <1E-10),表明RNA-seq结果具有很高的可重复性。
讨论
比较基因组学已被广泛用于研究适应极端环境的遗传基础[6,7,9,10,14,28,29,30.,31].在本研究中,从叶和根的综合转录组分析美国阿尔巴在不同盐度条件下,研究了盐适应的遗传机制。本研究结果为进一步研究盐生植物的盐适应提供了可能的基因和机制假设。
盐适应是一个受多种基因影响的长期动态过程,涉及多种形态、生理、分子和细胞过程[12,14,32,33,34].大量蛋白质合成的要求作为全球防御战略的一部分拟南芥已在先前的研究中得到广泛证明[35].在目前在这里,你可以体验到完整的海水+和K+反端口显示为一个典型的组件。耐盐H+- atp酶在维持低Na中起重要作用+由于它在营养组织中强烈表达,因此在海草细胞中的浓度[36,37].在适应过程中还需要硝酸盐的同化和离子分区化[38].而红树林诱导的基因在特定的盐反应通路和影响细胞环境的转录因子中起作用。在我们的研究中,大多数跨盐水平差异表达的基因都被叶片中最高盐浓度诱导或抑制。根系的变化模式不同,说明植物对盐的适应性美国阿尔巴是组织。
在我们的研究中发现的一组主要上调基因涉及离子运输和渗透调节,包括NHX,提示,皮普,Mangrin.据报道,这些基因在多种植物的耐盐性中起着重要作用[39,40,41,42].特别是,Mangrin与AOC编码基因部分同源。它被高盐度上调,其过表达增强了转基因酵母和烟草细胞的耐盐性[39].大多数转录本在高盐条件下下调涉及基础代谢、植物生长调节、分子功能调节、信号换能器活性和电子传递。这表明长期的高盐处理可能会抑制正常的代谢过程,破坏植物的稳态平衡。事实上,高盐度是一种重要的农业污染,会对植物造成损害[43].在拟南芥,盐胁迫并不是简单地降低稳态生长速率。虽然盐胁迫强烈抑制了几个离散相的生长,但其他相的生长却增加了[44].
高盐胁迫往往导致二次氧化应激,导致活性氧积累。我们确实观察到叶片中涉及硫、半胱氨酸、蛋氨酸以及谷胱甘肽代谢的多个关键酶(如cysQ、ATCYSC1、cysK、GST)均上调(图。4).这些途径增强了富硫肽(如s -谷胱甘肽)的合成,以清除ROS [45].此外,我们发现黄酮和黄酮醇生物合成途径基因在我们的差异表达转录本中被过度表达。类黄酮是天然抗氧化剂,可降低ROS水平,有助于植物的生存和生长[46].
盐可以促进烟酰胺和烟酸盐循环用于吡啶核苷酸的合成,以保护红树林中用于核苷酸合成的嘧啶和嘌呤衍生物工业sexangula[47].我们还发现烟酸盐和烟酰胺代谢途径的基因在高盐处理下的叶片和根中均有差异表达。
乙烯信号也影响植物对盐胁迫的反应拟南芥.高盐度诱导EIN3/EIL1蛋白的积累和激活。活化的EIN3减轻了过量ROS的积累,从而提高了植物的耐盐性[48].我们发现盐处理下叶片中半胱氨酸、蛋氨酸和谷胱甘肽代谢通路中的关键酶的表达上调,而根中没有上调。这表明乙烯和谷胱甘肽优先在叶片组织中合成,并在长期的盐适应中发挥作用。
多项研究表明,转录因子在植物中起着调节适应的作用。某些tf的功能(如AP2 / EREBP,南汽),尤其是对耐盐性的反应[35,49,50,51].我们还观察到,随着盐浓度的增加,许多TF编码基因表达上调。转录因子AP2/EREBP,WRKY,bZIP,南汽,MYB而且bHLH调节下游因子参与黄酮类生物合成、硫和硫代葡萄糖苷代谢等主要途径[52,53,54,55,56,57].这一结果证实了氧化石墨烯富集和途径富集的分析,并表明在盐适应过程中存在广泛的转录调控。然而,需要进一步的研究才能找到红树林盐反应的确切调控网络。
自然选择可能是主要驱动力美国阿尔巴盐对波动潮间带环境的适应。我们缺乏比较转录组数据来测试多少基因表达对盐处理的反应是适应性进化的结果。然而,我们确实发现了一些基因,它们在氨基酸水平上的进化要么加速,要么显示出正选择的迹象美国阿尔巴沿袭(附加文件8:表S4)。的确,SA_12151是生长素反应家族的一员,对水稻的各种非生物胁迫(生长素、高盐度胁迫)有反应[58].SA_26719(peroxisomal 3-ketoacyl-CoA thiolase)是一个过氧化物酶体相关基因。先前的研究报道了盐胁迫会影响过氧化物酶体酶活性和蛋白质表达[59].盐诱导过氧化物酶体相关基因的表达需要脱落酸和其他信号通路[60].ABA是一种应激激素,在多种非生物应激反应中起着核心作用。最后,SA_14625编码一种水解酶,参与植物的重要生理过程。在耐盐水稻中,它在高盐度胁迫下被上调[61].虽然我们的数据表明这些基因与耐盐性有关,但这些上调的PSGs的进化机制必须通过生理实验进一步证实。这将有助于我们更好地理解盐适应的进化动力学。
对于生活在潮间带的物种来说,最极端的挑战是由于不断的潮汐波动而造成的不稳定和高盐度。我们对DEGs的分析指出,细胞环境是分子盐适应的可能位点美国阿尔巴.因此,我们认为细胞环境稳态的维持和调节是重要的适应过程,有助于进化创新。
结论
我们进行了全面的转录组分析美国阿尔巴叶和根在盐度条件下。该研究证实了广泛的盐适应转录调控。叶片和根采用不同的动态基因调控策略进行盐适应美国阿尔巴.叶片中大部分差异表达基因都很好地适应了中等盐度条件。我们的分析报告了几个候选基因(包括盐相关基因、tf编码基因和PSGs)和主要途径,包括硫代谢、黄酮和黄酮醇生物合成、烟酸盐和烟酰胺代谢、半胱氨酸和蛋氨酸代谢,这些都与适应高盐环境有关。我们推断细胞环境稳态的维持和调节是重要的适应过程。我们的研究也可以作为未来极端环境下适应性进化研究的资源。随着今后对甲基组和蛋白质组的进一步研究,植物盐适应的确切机制有望被揭示。
方法
植物材料和生长条件
十八岁美国阿尔巴幼苗(附加文件)9:图S5)经许可采集自海南东寨港国家级自然保护区苗圃。所有样品均经东寨港国家级自然保护区管理局钟彩荣先生鉴定。然后在中山大学温室(23°C ~ 35°C,日照13.5 h,光照强度足以正常生长)栽培。幼苗被转移到沙质土壤中,使用一半霍格兰的溶液生长3周,以帮助植物适应新环境。将幼苗分为3组,每组2个生物重复。这三个实验组分别使用含0、250和500 mM NaCl的溶液灌溉7天。在自然条件下,美国阿尔巴通常在河口附近发现。雨水和上游河流使它很少受到正常海水浓度的影响。美国阿尔巴在5 ~ 50%的海水中达到最佳生长。中等盐度(250mm NaCl)约为正常海水盐度的一半,接近自然盐度的上限美国阿尔巴增长(19].虽然500毫米和0毫米分别提供了高盐(压力)和低盐条件,但0 - 250毫米是红树林在自然界中很好的耐受范围。相比之下,250-500 mM的压力确实很大。治疗时间以我们之前的研究为基础[62].
样本采集、测序和比对
每棵幼苗在7天后采集健康的幼叶和根,并立即在液氮中冷冻,并在−80°C保存。来自同一组的三株植物汇集在一起,以尽量减少单个植物之间的差异。根据制造商的说明,使用植物RNA试剂盒(Omega Bio-Tek, Doraville, USA)提取总RNA。对于每个样本,至少20 μg总RNA被发送到Novogene生物信息技术有限公司,使用Illumina HiSeq 2000平台进行测序。原始序列读取首先通过修剪适配器序列进行纯化。为了减少测序误差,满足以下条件的配对端低质量reads被定制脚本删除:i)未知碱基大于5% (N);Ii)质量<= 10的碱基含量超过20%;(3)平均质量在20以下。根据FastQC的结果,对过滤后的读取的最后一个碱基也进行了修整。
从每个样品的清洁读数对准美国阿尔巴TopHat v2.1.1参考基因组[10][63]和Bowtie2 v2.2.9 [64].然后,我们使用Cufflinks v2.2.1进行基因组引导的转录组组装和差异转录本表达分析[65].我们报告的表达水平为每千碱基片段的外显子模型每百万映射读取(FPKM)值。然后,我们使用基因表达数据计算所有重复之间的Pearson相关系数。
差异表达基因鉴定及表达模式分析
我们从两个比较中识别出了deg: 250 mM NaCl vs 0 mM NaCl和500 mM NaCl vs 250 mM NaCl。我们考虑了成绩单问值(FDR-adjustedP-values)低于5%,且褶皱变化大于2。我们把表达随盐浓度增加的基因称为“上调基因”,把表达随盐浓度相反方向变化的转录本称为“下调基因”。其余基因记为“无显著差异”。通过对3种表达模式(上调、下调或无显著差异)在叶和根中的表达进行配对比较,将这些DEGs分为8个不同且具有代表性的簇(LC1 ~ LC8, RC1 ~ RC8)。为了确定哪些集群在分配的基因数量上具有统计上显著的富集性,我们使用了10,000次排列测试。在每个排列中,我们首先随机打乱在不同条件下获得的所有表达量,以破坏条件与表达量之间的任何关联,并将它们分配到原始基因组中。然后我们对真实数据进行聚类分析,记录排列中每个聚类的基因数量是否小于实际观察。在10,000次排列运行后,我们估计将基因分配到一个集群的频率不少于实际表达数据中的频率,从而允许进行显著性测试。
基因注释与本体丰富分析
我们获得的美国阿尔巴基因组注释和指定的基因本体(GO)和KEGG orthology (KO)术语对所有基因。GO术语使用Web基因本体注释图(WEGO 2.0)分配[66]并通过Cytoscape_v3.7.2进行可视化[67].转录因子注释美国阿尔巴以及与之相关的基因组序列:Sonneratia caseolaris(真红树)[10],石榴(non-mangrove) [68),而桉树茅(non-mangrove) [69]使用iTAK1.7进行[70].我们检测了差异表达中tf的富集情况美国阿尔巴使用费雪精确测试基因。进行KEGG注释,将DEGs分配到代谢途径。我们将叶片和根中所有上调的基因都归为KEGG通路,并将这些通路的基因数量与全基因组背景进行比较。KEGG路径数据来自R包clusterProfiler [71],支持下载最新的KEGG注释。有三个以上上调基因的通路使用Fisher精确测试进行处理。如果Benjamini-Hochberg纠正,富集被宣布为显著P -价值下降到5%以下。我们鉴定了关键的盐反应基因美国阿尔巴基因的基因组注释和BLASTP搜索美国阿尔巴反对拟南芥基因组数据(TAIR10,www.arabidopsis.org)带截断e-value < 10−6.
选择分析
为了检测可能发生正选择的上调基因美国阿尔巴,我们首先确定了高质量的骨科医生美国阿尔巴以及它的近亲,包括石榴,Trapa bispinosa,Lagerstroemia叶,Duabanga开大花的,桉树茅.这些种属于桃金娘属同一目(APG IV,www.mobot.org/MOBOT/research/APweb/welcome.html).的全基因组序列石榴,桉树茅已从phytozome及NCBI下载[68,69].更接近的物种Trapa bispinosa,Lagerstroemia叶,Duabanga开大花的,我们在之前的研究中对转录组进行了测序[72].使用OrthoMCL识别的正交正交[73]作为初始数据集,我们使用倒数BLASTP最佳命中方法美国阿尔巴以及五个相关物种。然后使用MUSCLE [74]并使用对齐的蛋白质序列使用PAL2NAL生成密码子对齐[75].去除带有模糊数据的站点后,长度小于150bp的对齐被丢弃。这一过程产生了3936个高质量的骨科美国阿尔巴以及它的亲戚。这些密码子与系统发育树一起排列[76]用于后续分析。
我们使用PAML包中的“自由比”模型计算了系统发生树每个分支的Ka/Ks比[77].所有直系同源基因连接成一个单一的超级基因。我们使用PAML包中的分支模型来识别积极选择的基因,设置美国阿尔巴分支为前景[78].我们引入了备选模型和零模型之间的似然比较,前者允许前景分支以不同的速率进化,后者假设所有分支对每个正交向量集都以相同的速率进化。我们也在PAML中使用了分支站点模型,设置model = 2, NSsites = 2 [79].假设前景中至少有一个地点经历了正选择的替代模型和假设没有发生的零模型之间的可能性比较被用作正选择的检验。似然比检验方法与分支模型相同。采用R函数中实现的Benjamini-Hochberg方法对多重测试进行校正p.adjust.我们认为FDR低于5%的上调基因为PSGs。最后,我们手动过滤了所有在对准中存在潜在错误的psg,以最大限度地降低假阳性率。
RNA-seq数据实时定量PCR验证
进行qRT-PCR验证来自16个不同簇的16个转录本的RNA-seq结果(SA_28450均来自LC8和RC5)。qRT-PCR的基因特异性引物使用Primer Premier软件设计,并在附加文件中列出10:表S5。
使用PrimeScript™RT reagent Kit with gDNA Eraser (TaKaRa)从1 μg总RNA合成第一链cdna。β-肌动蛋白基因的表达作为内控。qRT-PCR采用Real-Time PCR系统在ABI 7900HT上进行,程序如下:95°C 30 s(95°C 5 s, 55°C 10 s, 72°C 15 s), 40个循环。每个样品采用3个技术重复进行分析。所选基因的相对表达谱用2-△△CT方法(80].从RNA-seq和qRT-PCR中选择的基因的表达谱之间的相关性使用简单的线性回归模型估计lmR中的函数。
数据和材料的可用性
RNA-seq数据已存入Genome Sequence Archive数据库(Accession No.;PRJCA001412)和NCBI Sequence Read Archive数据库(登录号:;PRJNA615770)。
缩写
- AOC:
-
烯氧化物环化酶
- 度:
-
差异表达基因
- FPKM:
-
每百万外显子模型映射读取的每千碱基片段数
- 走:
-
基因本体论
- 柯:
-
KEGG orthology
- NHX:
-
/钠氢交换器
- 皮普:
-
质膜内禀蛋白
- 观测:
-
积极选择的基因
- qPCR:
-
实时定量PCR
- ROS:
-
活性氧
- 紧急求救信号:
-
盐过度敏感
- TFs:
-
转录因子
- 提示:
-
膜内源性蛋白
- 紫外线:
-
紫外线
- WEGO:
-
Web基因本体注释图
参考文献
吉丽C,龙J,阿巴斯S,穆拉利RM,卡默尔FM,彭格拉B,等。南亚红树林的分布与动态。环境科学学报,2015;
红树林的生态生理学。树木。1988;2:129-42。
李志强,李志强。红树林耐盐机制研究进展。树木。2010;24:199 - 217。
汤姆林森PB。红树林植物学第二版。剑桥:剑桥大学出版社;2016.
黄国义,王永生。两种红树植物(秋茄和木榄根)叶片暴露于多种重金属的生理生化反应。J危险材料,2010;182:848-54。
徐松,何震,郭震,张震,Wyckoff GJ, Greenberg A,等。红树林从木本植物进化过程中的全基因组趋同。中国生物医学杂志,2017;34:1008-15。
徐松,贺震,张震,郭震,郭伟,吕华,等。通过全基因组测序揭示了红树林主要分支(根菌科)的起源、多样化和适应性。自然科学版2017;4:721-34。
郭忠,李霞,何忠,杨勇,王伟,钟超,等。红树林类群极低的遗传多样性反映了过去海平面的变化,并暗示了未来的不良反应。《生物学报》2018;24:1741-8。
吕华,何忠,吴超,石山。边缘环境下红树林基因组的趋同适应进化:卸载转座因子是红树林基因组的一种常见策略。新植物,2018;217:428-38。
何震,徐松,张震,郭伟,吕华,钟超,等。陆海交界面木本植物基因组的趋同适应。自然科学,2020年版。https://doi.org/10.1093/nsr/nwaa027.
案例I, de Lorenzo V, Ouzounis CA.细菌转录调控与环境适应。微生物学,2003;11:248-53。
关木M, Narusaka M,石田J, Nanjo T,藤田M, Oono Y,等。利用全长cDNA芯片检测7000个拟南芥基因在干旱、寒冷和高盐度胁迫下的表达谱。植物学报,2002;31:29 - 32。
M.耐盐机理。植物学报。2008;39:651 - 81。
马涛,王杰,周刚,岳忠,胡强,陈勇,等。沙漠杨树盐适应的基因组研究。Nat Commun. 2013;4:2797。
丁峰,崔鹏,王震,张松,阿里生,熊璐。盐胁迫下拟南芥pre-mRNA选择性剪接的全基因组分析。BMC Genomics. 2014;15:431。
Shankar R, Bhattacharjee A, Jain M.不同水稻品种的转录组分析为干旱和盐度胁迫响应提供了新的见解。科学通报2016;6:23719。
Belamkar V, Weeks NT, Bharti AK, Farmer AD, Graham MA, Cannon SB.大豆脱水和盐胁迫下HD-zip转录因子家族的综合表征和RNA-Seq分析。BMC Genomics, 2014;15:950。
Ji H, Pardo JM, Batelli G, Van Oosten MJ, Bressan RA,李霞。盐过度敏感(SOS)通路:已建立和新兴的作用。植物学报。2013;6:75 - 86。
Ball MC, Pidsley SM。在澳大利亚北部,两种红树林的生长对盐度的响应与分布的关系,海桑和杉木。生态学报,1995;9:77-85。
杨娥,易松,白峰,牛东,钟娟,吴强,等。耐盐红树胞质铜锌超氧化物歧化酶(SaCSD1)的克隆、鉴定及表达模式分析。中国生物化学杂志(英文版);2016;
陈松,周荣,黄勇,张敏,杨刚,钟超,等。利用Illumina平台对高耐盐红树海桑的转录组测序。基因组学杂志,2011;4:129-36。
杨勇,郭伟,沈鑫,李娟,杨松,陈松,等。红树海桑属进化保守的选择性剪接事件的鉴定和特征。科学通报2018;8:4425。
朱镕基j - k。拟南芥耐盐性的遗传分析。植物物理学报。2000;124:941-8。
胡红,尤军,方勇,朱鑫,祁忠,熊玲。水稻耐寒耐盐转录因子基因SNAC2的鉴定。植物化学学报,2008;
陈玲,宋勇,李松,张玲,邹超,于东。WRKY转录因子在植物非生物胁迫中的作用。生物化学学报。1819;2012:120-8。
林涛,徐旭,阮娟,刘松,吴松,邵旭,等。Taraxacum kak -saghyz Rodin的基因组分析为橡胶生物合成提供了新的见解。自然科学版2017;5:78-87。
杨东,杨涛。植物抗旱和耐盐胁迫转录因子及其在细胞转录网络中的意义。植物学报,2011;30:1383-91。
邱强,张刚,马涛,钱伟,王娟,叶铮,等。牦牛基因组及其对高海拔生活的适应。Nat Genet, 2012; 44:946-9。
何忠,李旭,杨敏,王旭,钟超,杜克宁,等。通过隔离和迁移循环的基因流的物种形成:来自多个红树林类群的见解。自然科学版2019;6:275-88。
李娟,高志,周玲,李玲,张娟,刘勇,等。比较转录组分析表明K+转运蛋白基因与茄子耐盐性有关。BMC植物生物学2019;19:67。
他气X Y, Ouzhuluobu LS,李J,张H,等。高原缺氧条件下西藏人一氧化氮调节的钝化。自然科学,2018;5:16 - 29。
Sicilia A, Testa G, Santoro DF, Cosentino SL, Lo Piero AR. RNASeq分析揭示了长期盐胁迫下的叶片转录组动态。BMC植物生物学2019;19:355。
刘建国,韩旭,杨涛,崔文华,吴爱民,付春霞,等。杨树对盐胁迫的全基因组转录适应。BMC植物生物学2019;19:367。
费塔多,李志刚,李志刚,李志刚,等。转录组分析和环境与盐度的关系。BMC植物生物学2019;19:427。
龚强,李鹏,马山,因杜·鲁帕萨拉,洪杰。嗜盐极生植物拟南芥对盐胁迫的适应能力。植物学报,2005;
Olsen JL, Rouzé P, Verhelst B, Lin YC, Bayer T, Collen J,等。海草Zostera marina的基因组揭示了被子植物对海洋的适应性。大自然。2016;530:331-5。
杨春华,杨春华,杨春华,杨春华,杨春华,等。盐胁迫下植物抗盐atp酶活性的研究进展。植物营养学报,2002;
吕晓霞,于平,邓伟,李艳。基于转录组分析的黄藻对NaCl胁迫的适应性。植物生理学报,2018;
Yamada A, Saitoh T, Mimura T, Ozeki Y.表达红树烯氧化物环化酶增强大肠杆菌、酵母和烟草细胞的耐盐性。植物细胞物理学报,2002;43(3):366 - 366。
杨晓明,杨晓明,杨晓明,等。含氮螺旋藻的大麦幼苗对盐胁迫的响应及水通道蛋白表达。中国生物医学工程学报,2011;29(3):349 - 349。
谭文文,林青,林涛,郭晓平,陆华昌。红树盐腺分泌对盐环境变化的响应。植物细胞环境学报。2013;36:1410-22。
吕红,李霞,郭志,何忠,石山。白地黄转录组的从头组装与注释。Mar Genomics, 2018; 39:3-6。
张志刚,杨振华,杨振华。植物耐盐性的分子生物学研究。植物物理学报,1997;19:427-33。
耿勇,吴荣,韦长青,谢峰,魏晓霞,陈佩敏,等。盐胁迫响应过程中拟南芥生长调控的时空研究。植物学报,2013;
Anjum NA、Aref IM、Duarte AC、Pereira E、Ahmad I、Iqbal M.谷胱甘肽和脯氨酸能够协同使植物抵御金属(样体)和盐胁迫的联合攻击。植物科学进展(英文版);
Hernández I, Alegre L, Van Breusegem F, Munné-Bosch S.类黄酮作为抗氧化剂在植物中的相关性如何?植物科学进展;2009;14:125-32。
杨春梅,杨春梅,杨春梅,杨春梅。短期盐胁迫对红树细胞嘧啶、嘌呤和吡啶核苷酸代谢谱的影响,工业sexangula.物理学报。2006;128:405-14。
彭健,李志,文霞,李伟,史华,杨林,等。盐诱导的EIN3/EIL1稳定通过阻止ROS积累而赋予拟南芥耐盐性。PLoS Genet, 2014;10:e1004664。
蒋艳,Deyholos MK. nacl胁迫拟南芥根系的综合转录谱分析揭示了一类新的响应基因。BMC植物生物学。2006;6:25。
格鲁伯V, Blanchet S, Diet A, Zahaf O, Boualem A, Kakar K,等。截叶苜蓿根尖对盐胁迫响应的转录因子鉴定。中国生物医学工程学报,2009;
杨晓明,杨晓明,杨晓明,等。缅甸红树对盐胁迫和渗透胁迫响应的转录和生理研究。植物分子生物学杂志,2008;
宋昌平,Agarwal M, Ohta M,郭勇,Halfter U,王鹏,等。拟南芥AP2/ erebp型转录抑制因子在脱落酸和干旱胁迫响应中的作用《植物科学》2005;17(4):344 - 344。
蒋娟,马珊珊,叶娜,姜敏,曹娟,张娟。WRKY转录因子在植物逆境响应中的作用。中国生物医学工程学报,2017;29(5):529 - 529。
马马涛,李志强,李志强,等。葡萄ttg2样WRKY转录因子参与调节液泡运输和类黄酮生物合成。植物科学,2017;7:1979。
马丽娟,李志强,李志强,等。葡萄VvibZIPC22转录因子参与类黄酮生物合成的调控。中国科学(d辑),2016;
Morishita T, Kojima Y, Maruta T, Nishizawa-Yokoi A, Yabuta Y, Shigeoka S.拟南芥NAC转录因子ANAC078在强光下调控类黄酮生物合成植物生理学报。2009;30(4):344 - 344。
徐伟,李志强,李志强,等。MYB-bHLH-WDR复合物对黄酮类化合物合成的转录调控。植物科学进展2015;20:176-85。
Jain M, Khurana JP。转录谱分析揭示了生长素反应基因在水稻生殖发育和非生物胁迫中的不同作用。Febs j . 2009; 276:3148-62。
Corpas FJ, Gómez M, Hernández JA, Luis A.对氯化钠敏感性不同的两个Pisum sativum品种活性氧代谢的研究。中国植物科学。1993;21(3):344 - 344。
Charlton WL, Matsui K, Johnson B, Graham IA, OHME-TAKAGI M, Baker A.盐诱导过氧化物酶体相关基因的表达需要乙烯、茉莉酸和脱落酸信号通路的组成部分。植物生态学报。2005;28:513-24。
Opassiri R, Pomthong B, Onkoksoong T, Akiyama T, Esen A, Cairns JRK。水稻糖基水解酶家族1的分析及Os4bglu12 β-葡萄糖苷酶的表达。BMC植物生物学。2006;6:33。
梁松,方林,周瑞,唐涛,邓松,董松,等。一种红树林物种,Ceriops tagal,在盐环境中的转录稳态,通过微阵列分析揭示。科学通报。2012;7:e36499。
Kim D, Pertea G, Trapnell C, Pimentel H, Kelley R, Salzberg SL. TopHat2:存在插入、缺失和基因融合时转录组的精确对齐。中国生物工程学报。2013;14:R36。
朗米德B,萨尔茨堡SL.快速间隙阅读对齐领结2。Nat Methods. 2012; 9:357-9。
特拉普内尔,罗伯茨A,戈夫L, Pertea G,金D, Kelley DR,等。TopHat和袖扣RNA-seq实验的差异基因和转录物表达分析。Nat protocol . 2012; 7:562-78。
叶健,方林,郑辉,张勇,陈杰,张震,等。WEGO:用于绘制GO注释的网络工具。核酸研究,2006;34:W293-7。
王俊杰,王俊杰,王俊杰,等。细胞景观:生物分子相互作用网络集成模型的软件环境。基因组决议2003;13:2498-504。
秦刚,徐超,R明,唐华,Guyot R, Kramer EM,等。石榴(Punica granatum L.)基因组及石榴苷生物合成基因组学研究。植物学报,2017;21(2):344 - 344。
杨晓东,张志强,张志强,等。巨桉的基因组。大自然。2014;510:356 - 62。
郑勇,焦超,孙华,Rosli HG, Pombo MA,张萍,等。植物转录因子、转录调节因子和蛋白激酶全基因组预测和分类的iTAK程序。Mol Plant, 2016; 9:1667-70。
于刚,王立国,韩勇,何启勇。clusterProfiler:一个R包,用于比较基因集群之间的生物学主题。组学。2012; 16:284-7。
李娟,杨勇,杨松,张震,陈松,钟超,等。红树海桑(Sonneratia caseolaris)及其非红树亲缘(Trapa bispinosa和Duabanga grandflora)的比较转录组分析。基因工程学报。2017;31:13-5。
李,施托克特CJ,罗斯DS。真核生物基因组正正交基的鉴定。基因组res 2003; 13:2178-89。
埃德加钢筋混凝土。MUSCLE:多序列比对,精度高,通量大。核酸决议2004;32:1792-7。
Suyama M, Torrents D, Bork P. PAL2NAL:蛋白质序列对齐到相应密码子对齐的鲁棒转换。核酸研究,2006;34:W609-12。
黄艳,石山。荔枝科植物的系统发育:基于叶绿体rbcL基因、psaA-ycf3间隔子和核rDNA内部转录间隔子(ITS)序列的初步分析。植物科学进展(英文版);2002;
杨志。PAML 4:最大似然系统发育分析。生物化学学报,2007;24:1586-91。
杨志。似然比试验检测阳性选择及其在灵长类动物溶菌酶进化中的应用。生物化学学报,1998;15:568-73。
杨震,杨志军,杨志军,等。基于密码子替代的分子适应性检测方法研究。分子生物学杂志2002;19:908-17。
Livak KJ, Schmittgen TD。使用实时定量PCR和2(−Delta Delta C(T))方法分析相关基因表达数据。方法。2001;25:402-8。
确认
我们感谢吴忠义和郭子晓的深刻评论;以及安东尼·j·格林伯格的手稿修改。
资金
国家自然科学基金(31830005、31971540)资助;国家重点研发计划(2017FY100705);广东省基础与应用基础研究基金(2019A1515010752);中国博士后科学基金(BX201700300、2017 M622857);中山大学常鸿塔科学基金。资助机构没有参与研究的设计和数据的收集、分析、解释或手稿的撰写。
作者信息
从属关系
贡献
S.S.和Z.H.构想了这项研究;X.F, S.X, J.L.和Q.C.分析了数据;j.l., y.y., H.L.和C.Z.收集材料并进行实验;x.f.、s.x.、Z.H.和S.S.撰写了手稿;所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:图S1。
所有样本间的基因表达相关性。皮尔逊的相关图可视化相关系数。比例条表示显示值的范围。叶片0 mM、叶片250 mM、叶片500 mM、根系0 mM、根系250 mM和根系500 mM条件下生物重复间的相关系数分别为0.92、0.97、0.90、0.97、0.87、0.90。
附加文件2:图S2。
差异表达基因(DEGs)的聚类分析和排列检验。在不同盐度对比下,8个簇在叶和根中均有表达谱。叶片中的DEGs在LC8、LC4、LC5三个簇中显著富集,而根中的DEGs在RC1和RC5中显著富集。在每个聚类中,黑线表示表达式级别(日志的平均值)10(FPKM值+ 1)),误差条表示标准差。用置换检验来评价差异的显著性。deg数量差异有统计学意义的聚类用三重(P-value < 0.01)星号。属于每个聚类的观察到的和预期的(只有显著的聚类)基因数量被标记在每个聚类的顶部。
附加文件3:表S1。
deg的GO注释。URG为上调基因;DRG代表下调基因。括号内的数字为GO ID基因的百分比,括号外的数字为基因编号。
附加文件4:表S2。
共表达簇中基因中富集的GO术语总结。
附加文件5:表S3。
上调基因的KEGG注释。URG代表上调基因。括号内的数字为KO ID基因的百分比,括号外的数字为基因或途径号。
附加文件6:图S3。
不同盐度(0、250和500 mM NaCl)下叶片(A)和根系(B)转录因子基因的不同表达模式在热图的右侧给出了在不同条件下表现出差异表达模式的各种TF家族。中上尺度表示经过z-score归一化的FPKM值。
附加文件7:图S4。
沿着系统发生树的每个分支的Ka/Ks比值。红树林谱系(美国阿尔巴)被涂成红色,设置为前景。
附加文件8:表S4。
与盐适应相关的PSGsSonneratia阿尔巴.
附加文件9:图S5。
秧苗美国阿尔巴.
附加文件10:表S5。
实时定量PCR分析引物序列。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
冯晓霞,徐寿,李俊,李志强。et al。红树林对热带潮间带环境盐度波动的分子适应Sonneratia阿尔巴.BMC植物生物学20.178(2020)。https://doi.org/10.1186/s12870-020-02395-3
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-020-02395-3
关键字
- 体内平衡
- 红树林
- 盐适应
- Sonneratia阿尔巴
- 转录因子
- 转录组的概要文件