摘要
背景
Sclerotinia stew腐烂(SSR),由菌核病sclerotiorum是大豆产量损失的一个重要原因。虽然许多论文报道了不同的位点对部分抗性有贡献,但很少有证明在不同群体中产生相同的表型影响。
结果
本研究采用全基因组关联作图法(GWAM)鉴定了一个与主茎抗SSR进展相关的主要数量性状位点。利用基因分型测序(GBS)和全基因组测序(WGS)获得的1.5 M单核苷酸多态性对127份大豆材料进行了基因分型,确保了广泛的基因组覆盖和SSR抗性的表型。snp -性状关联在1号染色体(Chr01)上发现了一个新的QTL,抗性株系茎秆上的病变缩短了29 mm。一个单基因(Glyma.01 g048000)与峰值SNP位于同一LD区,但功能未知。该QTL在携带Chr01等位基因的两系杂交后代中影响更为显著。携带抗性等位基因的个体发生病变的时间比携带敏感性等位基因的个体短近50%。
结论
这些结果表明,1号染色体上的新区域是一个有希望的SSR抗性QTL,可用于大豆育种。
背景
在美国北部和加拿大,大豆菌核病(Sclerotinia stem rot, SSR)是造成大豆产量和品质损失的重要病害。这种病是由菌核病sclerotiorum,是一种坏死性子囊菌属,可感染超过408种不同的物种[6].真菌通过花感染植物,然后通过茎蔓延引起漂白,严重的枯萎和粉碎组织[7].1994年在加拿大、1994年、2004年和2009年在美国,SSR被报道为第二大致病产量损失[18,32].然而,这种疾病的影响是很难预测的,因为真菌的发展受到温度和湿度的高度影响[20.].使用化学或生物防治可以降低其影响,但结果可能是可变的,因为当疾病发病率高于50%时,这些方法可能会失败[33].即使SSR在不利的气候条件下不构成威胁,每年进行多次预防处理也能取得最好的效果。因此,提高大豆品种的遗传抗性是降低SSR危害的最有效途径。
SSR抗性的评估在可变环境条件下具有挑战性。然而,由Bastien等人开发了可靠的接种方法。[3.],其中菌丝悬浮液施用在控制的温室条件下的花蕾上。它已被证明产生一致的结果,并被用于调查大豆SSR抗性的遗传决定因素[4,13,15].
到目前为止,大豆还没有完全抗性的报道。部分抗性由多个基因或数量性状位点(QTL)控制。已经进行了大量的作图研究,并通过传统的双亲本作图鉴定了114个以上的QTL [1,11,13,16,17,29,34].虽然该方法已被广泛应用于QTL定位,但仍局限于两亲本存在的遗传多样性。近年来,随着基因分型技术的进步,利用GWAM的数千个标记对多个大豆品系进行定量部分抗性筛选成为可能。利用该方法,在不同群体中也发现了130多个qtl [4,15,21,30.,31,34].如此多的位点对它们的可信度提出了一些疑问,特别是当这些位点中很少有被证明在不同的遗传背景下具有相同的等位基因效应时。一种解释是,基于不同的评估方法鉴定的一些qtl可能与逃逸或回避机制相混淆,而不是真正与对SSR的生理抗性有关[4,17].作为证明,通过在受控环境下进行抗性评价,在双亲本杂交中唯一鉴定出具有相同表型效应的QTL位于15号染色体上[4].这些结果表明,可靠的表型方法是本研究的关键因素。
与双亲本作图相比,多样性图谱提供了较低水平的标记与qtl之间的连锁不平衡(LD)。因此,根据种群大小和多样性的不同,GWAM需要较高的标记密度。对于较高的QTL检测力,QTL与任一侧翼标记的差值应大于0.8。为了实现这样的覆盖,Bastien等人[4]估计,如果要覆盖整个基因组,总共需要超过68 K的分布良好的SNPs,则在中端粒区域至少需要12900个SNPs,在端粒区域需要55700个SNPs。为了定位大豆SSR抗性位点,人们尝试了许多不同的基因分型方法,如基因分型法(GBS) [4,15,30.或特异性位点扩增片段测序(SLAF-seq) [34].迄今为止,在两项研究中使用Soysnp50k阵列实现了最大数量的信息SNP。466名中获得35,683个SNP [21[其他在915条改进线和405种大豆地板的人口中分别达到了31,600和35,708个SNP [31].很可能在这些最近的论文中获得的标记覆盖率仍然低于确保彻底覆盖基因组所需的数量。
先前使用的基因分型方法的一种替代方案是全基因组测序(WGS)。然而,这种方法仍然昂贵,特别是在使用大群时。在以前的工作中,Torkamaneh等人。[28]提出了一个被称为“扫描和填充”的两步方法。在第一步中,一个庞大的群体可以通过数以万计的SNP位点(使用GBS或数组)进行基因分型。在第二步中,可以对这些株系的子集(例如20%)进行WGS,这些株系可以作为参考面板,将数百万个SNP标记注入到整个选育集中。
在这项工作中,我们使用了这种联合GBS和WGS基因分型方法,对数百万个snp的关联面板(包括优秀的加拿大大豆株系)进行基因分型。利用这一完整的标记集,在关联面板中进行GWAM分析,确定了加拿大大豆对SSR部分抗性的qtl。
结果
SSR抗性在关联面板的线
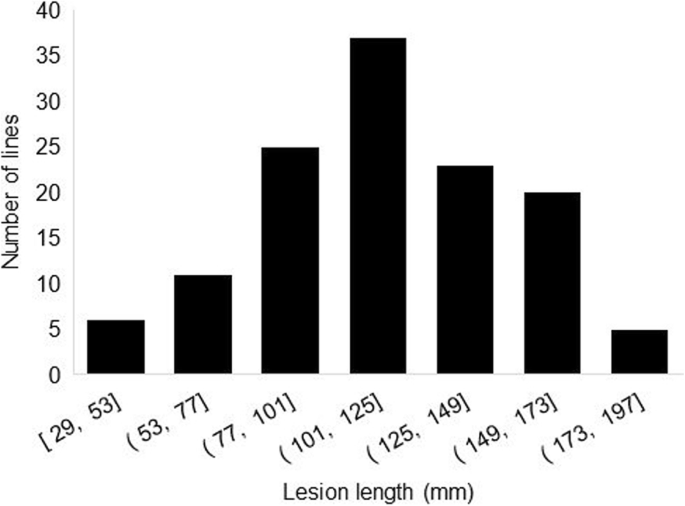
接种幼芽7天后测量病变长度,各基因型的平均值见补充表S1.如图1中所示。1,病变长度范围广泛,从低至29毫米到最大192毫米,人群中病变长度平均为114毫米。病变长度的分布呈钟形,表明有几个基因控制这一性状。耐药检查(lkarlo RR s19 - 90和枫Donovan)排名在最短的线路病变(127年1日,3日和21日)而高度敏感的检查Nattosan第二最长病变(177毫米)和两个中度敏感的检查(威廉姆斯82和贝菲尔德OAC)显示病变人口略高于平均水平。
标记分布
为了实现广泛的基因组覆盖,我们重新分析了之前获得的序列数据(940 M单端reads猿KI GBS文库由530个精英加拿大大豆线的DNA制备)使用改进的SNP呼叫管道(FAST-GBS)和更新的大豆参考基因组。这在包括所有关联面板的所有线路的530行的面板上产生了近150克的SNP。然后,我们使用了从102条线的WGS获得的4.1M SNP的目录,也包括在530条线中,作为参考面板,以赋予所有缺失基因座的基因型,从而导致4.1米标记的完整数据集。其中,3.5M的SNP在我们的结合面板中是多态(即,至少在127条线中至少携带交替等位基因)。在去除SNP映射到支架上(49.7K SNPS),3.4M SNP映射到20种大豆染色体中的一个。最后,我们删除了MAF低于0.05的标记,因此导致1,493,960个SNP的最终目录,我们进行了GWAM分析。
人口结构和血缘关系
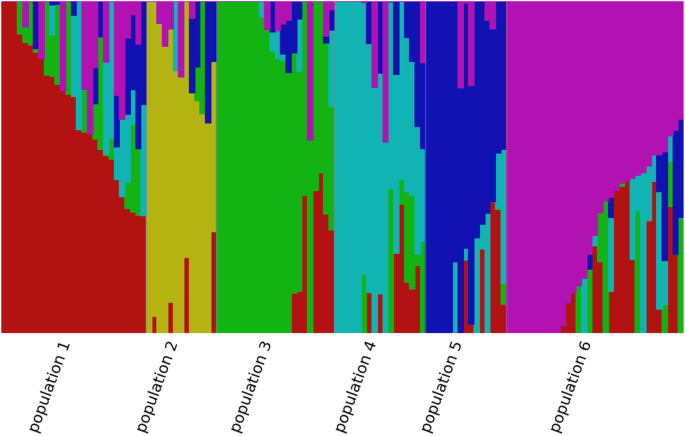
为了表征群体结构,我们对高LD (r2≥0.9;其余84,708个SNPs在fastSTRUCTURE中被使用。结果表明,该小组由3至6个亚群体组成。基于这两个结果,我们选择使用K = 6进行接下来的分析,图如图所示。2.为了进一步减少混淆,我们估计了关联面板之间的亲属矩阵。
SSR抗性的全基因组关联作图
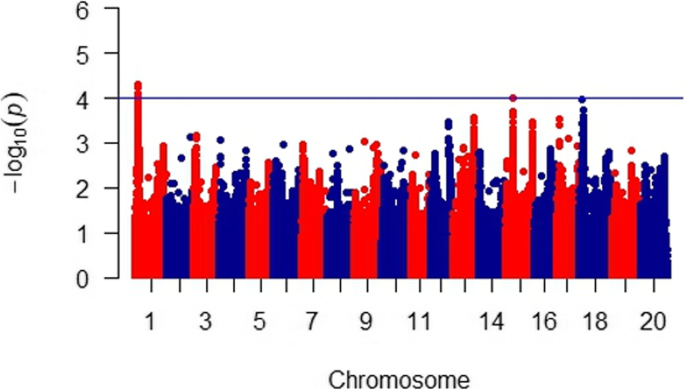
利用表型数据(平均病变长度)和完整套过滤SNP标记(接近1.5 M标记)估计标记-性状关联。这些分析使用传销(Q + K)和关联p-Values对应于FDR <0.1被认为是显着的。总共只发现两种染色体区域具有至少一个超过该阈值的峰值SNP(在CHR01 CH111上;图。3.).如表所示1,CHR01上的峰值SNP在5,594,597处,显示出一个p-价值5.08 × 10−5并解释了32%的表型变异。如图所示。4,在该位点携带有利等位基因T(频率= 0.38)的供试材料比携带可接受等位基因c的供试材料损伤更短。尽管在Chr01上携带抗性等位基因,但仍有部分供试材料出现平均超过100 mm的损伤。在15号染色体(chr15)上发现了第二个相关区域,该区域有一个显著相关的标记,位于13,665,369 (p-Value = 9.76×10−5;FDR = 0.04),并解释了15%的变异。次要等位基因A(频率= 0.32)固定的病灶比主要等位基因G固定的病灶短15 mm(补充图)1).
验证实验
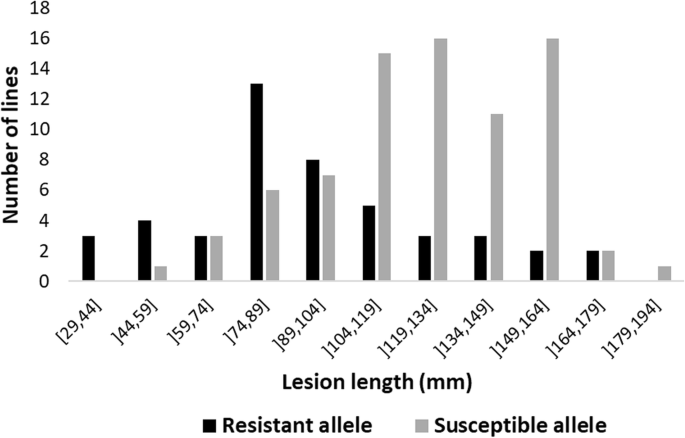
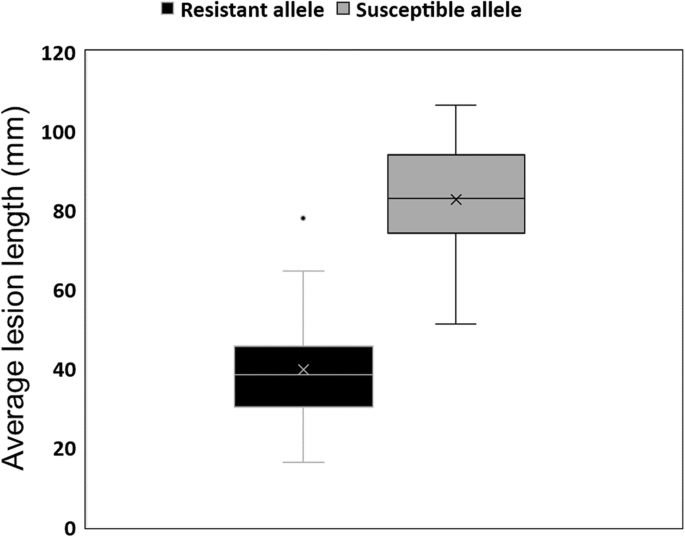
由于CHR15协会已经在以前的工作中验证了[4,重点对Chr01上的SSR抗性候选区域进行了验证。为了做到这一点,我们使用了F的总体8对OAC Bayfield (S)和Maple Donovan (R)的亲本进行了SSR抗性的高峰标记和SSR抗性的对比;Maple Donovan携带抗性等位基因,比OAC Bayfield的病变短78.3 mm。在验证试验中,除了选择47个重组自交系(ril)作为验证群体外,还使用亲本作为检验。利用CAPS标记对每个株系4个植株进行基因分型,分析其在Chr01上的QTL。在47个ril中,抗性等位基因21个纯合,感病等位基因26个固定。然后在两个温室试验中对这些ril和亲本进行SSR抗性评价。双亲间病变长度的对比仍很明显(60mm)。在RILs中,病变的平均长度为63 mm,范围为16 ~ 107 mm。有趣的是,几乎所有抗性等位基因固定的基因型都出现了平均水平以下的病变,范围在16到78毫米(平均40毫米)之间,而易感等位基因纯合株系的病变长度平均为83毫米,病变长度从51到107毫米。两个基因型类群之间的表型对比(43mm)(图。5)很重要(p= 0.007)。
在CHR01上QTL周围的基因组景观
在5.4和5.8 MB之间的所有标记对之间估计LD,以研究染色体上相关区域中的基因组景观1.在几乎完美的LD(R.)的基础上定义LD块(R.2 ≤ 0.98), all of the markers showing a significant association with lesion length resided in a small block (10 kb; block B1 in Supplementary Figure2)包含一个单基因(Glyma.01 g048000)。SNP的峰值位于Glyma.01 g048000的内含子内,因此不太可能引起功能的改变,而其他所有重要的SNP都位于基因的启动子内(ATG的1 kb以内)。由于这些标记之间的高LD,这些多态性本质上定义了两个单倍型(等位基因),频率较低的一个与提高抗性有关。基于峰值SNP和该基因之间的高度关联,它代表了一个强有力的候选基因。
讨论
数量的标记
在本研究中,我们使用了近1.5 M的高质量SNPs。Hyten等人基于对三个染色体片段的研究[14]估算了获得充分覆盖所需的snp数量(r2≥0.8),在9600 ~ 29000个标记之间。最近,一项对端粒和中端粒区域LD的全基因组估计带领Bastien等人[3.得出的结论是,大约需要6万个snp才能实现广泛覆盖。以前研究SSR抗性的GWAM研究都没有达到这样的标记覆盖率。在两个使用SNP标记的早期研究中,使用GoldenGate分析进行了基因分型,达到了858 [19]和1142个单核苷酸多态性[12].后来的GWAM研究使用了7864例gbs衍生的SNP目录[4), 839715]及11,811 [30.],而另一项研究达到了使用SLAF-SEQ获得的25,179个SNP的覆盖率更高的覆盖率[34].在这项工作之前,最近使用SoySNP50K BeadChip实现了最高覆盖率,为两个AM群体提供了31,600和35708个SNPs [31].在这里,组合GBS数据,WGS数据和归责缺失基因型,我们显着增加了SNP覆盖率,确保首次在我们的关联面板中赋予了可能赋予详细基因组覆盖的标记覆盖范围。前面表明,这种避税数据具有高精度,具有96.4%的缺失基因型与在与大豆阵列中的局部相同的锁相中获得的缺失基因型进行一致[28].鉴于最近大量大豆种质收集的WGS数据的可用性增加,我们认为在这项工作中使用的两步基因分型方法将极大地提高我们在全标记覆盖下进行全基因组扫描的能力。
CHR01上的新QTL
在我们对127个加拿大大豆品系的关联图谱中,我们鉴定了7个与SSR抗性显著相关的SNPs,仅位于2条染色体1和15的小片段。在Chr01上的第一个相关区域扩展超过10 kb,是全新的,因为它没有与之前报道的任何qtl重叠。事实上,这个区域不携带任何gbs衍生的SNPs或SoySNP50K BeadChip支持的SNPs。
在染色体15上发现了第二个QTL,并且是Bastien等人之前报道的完全相同的标志性关联。[4].考虑到我们使用了本质上相同的关联面板(除去的三个品系除外)和相同的表型数据,这些结果是预期的。在之前的工作中,已经报道了另外3个qtl (Chr01、Chr19和Chr20),但在本次工作中均未发现。这三个关联的FDR值(范围在0.04 ~ 0.09之间)都远高于第15个位点的FDR值(0.01)。为了研究这种明显缺乏重现性的可能原因,我们比较了所有相关SNP的SNP基因型和它们对应的SNP基因型,这些SNP基因型来自我们最近的大规模数据集。令人惊讶的是,对于第15染色体上的SNP, 98%的基因型调用是相同的,而对于其他相关的SNP(在Chr01、Chr19和Chr20上),在关联面板的各株系中,基因型调用的一致性水平要低得多(60 - 82%)。基于Torkamaneh和Belzile的研究[26,我们认为在Bastien等人的研究中发现的snp的数量[4事实证明,不足以充分捕获该协会面板中的单倍型,并导致缺失基因型数据的归因不准确。
QTL验证
在不同的遗传背景、环境和不同的采样群体中发现了大量的SSR抗性qtl。在考虑使用这些qtl进行标记辅助选择之前,强烈建议先进行验证。Chr15上的QTL之前已被Bastien等人验证[4抗性等位基因使两个RILs群体的病变长度分别缩短了12.3和17.6 mm。这种验证在其他研究中很少被报道。在这里,我们希望通过评估在该位点携带对比等位基因的ril中的SSR部分(或定量)抗性(病变长度)来验证在Chr01上发现的新标记-性状关联。结果表明,抗性等位基因纯合子株系(遗传自Maple Donovan)比敏感等位基因纯合子株系(遗传自OAC Bayfield)发生的病斑短43 mm。这种表型对比是非常显著的,并解释了很大一部分的表型变异的父母。此外,在分离的RILs中,估计的等位基因效应比关联面板中测量的更重要,表明双亲本群体中限制SSR抗性的qtl比关联面板中更少。这些数据表明,Chr01区与SSR抗性有关。
QTL检测效率及其在基因组选择中的应用
在本作工作中发现的两个区域,CHR01和CHR15,分别在我们的协会面板中解释了32和15%的该特征的表型变异。在以前的工作中,据报道,QTLS解释了3到23%的方差[4,15,21,30.,31,34].因此,在染色体1上报告的QTL构成了对这一特征报告的最有影响力的QTL。由于在小组中的少数群体(38%)的少数群体(38%)中存在有利等位基因,通过选择未来的育种努力中的这种部分电阻QTL,有相当大的范围来提高SSR阻力。
尽管如此,尽管有广泛的标记覆盖,该性状的大部分变异似乎仍未在我们的关联小组中捕捉到。因此,在该群体中可能存在其他未被发现的、对SSR有潜在影响的qtl。然而,在这样的图谱中,由于相关标记被过滤掉,很难捕捉到可能导致抗性的罕见等位基因(MAF < 5%)。将这项工作扩展到更大、更多样化的面板可以帮助发现更多的qtl。同时,剩下的未捕获的遗传也可能被解释成无数小的影响主要将很难发现(考虑到困难与表型相关的测量),将有限的育种项目中使用作为一个小的好处增加SSR阻力可能无法证明的分子标记辅助选择成本这样的小法。
候选基因在Chr01 SNP峰附近
当我们对Chr01上的相关区域进行研究时,我们发现在高LD中有一个具有峰值标记的单基因。除了位于内含子中的SNP峰外,Glyma.01 g048000的可能启动子中发现了所有其他显著相关的标记。由于峰值SNP与该基因之间的强相关性,它代表了一个强候选基因。然而,由于缺乏注释,人们可以假设该基因在菌核病耐药中的作用,因此需要进行功能研究,以提供该基因在抗药中的作用的确切证据。由于许多相关的SNPs都在5 '上游区域,因此研究该基因的调控将会特别有趣。
结论
我们得出结论,本研究中出现的知识将促进在可持续农业惯例下促进SSR挑战的解决。我们认为,这项工作是向植物育种更适用的GWAM数据进行前进的另一个阶梯。我们预计本研究中确定的遗传区域将成为SSR大豆育种计划中的关键工具,使遗传学家和分子生物学家能够在不久的将来识别SSR的因果抵抗基因。
方法
关联映射面板
本研究使用的关联图谱由来自私人育种计划(Semences programain Inc.)的127个株系组成,它们对SSR的响应存在很大差异[4].这些材料是在前人研究的基础上从530份具有代表性的加拿大大豆遗传多样性的材料(品种/高级育种系)中选出的[28].这127个品系都属于成熟组(MG),范围从000到II,只有一个品系Williams 82属于MG III。6个检测品种包括:3个已知具有良好SSR部分抗性的品种(Karlo RR、Maple Donovan和S19-90), 2个中抗性品种(OAC Bayfield和Williams 82)和1个高度敏感品种(Nattosan) [3.].Maple Donovan和Nattosan是来自东部谷物和油籽研究中心(加拿大农业和农业食品研究中心,渥太华,加拿大)的商业栽培品种,而S19-90是先正达种子公司的商业栽培品种。Williams 82是从美国种质资源信息网获得的。其余株系的种子均从Semences programain中获得。
验证小组
总共47华氏度8选择在染色体1 (Chr01)上分离的候选QTL的品系作为验证面板。这些品系是由部分抗性的枫树多诺万和易感的OAC Bayfield杂交产生的。
表型
使用Bastien等人描述的棉焊盘方法评估SSR偏电阻的线。[3.].对于关联面板,表型数据是之前由Bastien等人报道的[4].简单地说,在温室中播种植物采用随机完全区组设计,四个区组在时间上分开(2009年9月25日、11月6日、12月8日和2010年1月29日)。实验单元包括三盆6升的植物(每盆两株)。使用相同的实验设计来描述验证面板的特征,但只有两个区块在时间上分开(2017年5月4日和9月7日),每个实验单元有四个工厂。
使用黑地球(50%),珍珠岩(30%)和普发(20%)(20%)(Rivière-du-Loup,QC,Canada)的混合物制备灌封混合物。播种时,用Rhizostick®incululant(Becker Underwood,Ames,Ia)接种种子。在16-H光周期下生长植物,并将日/夜温度保持在26/22℃。
接种物由菌株NB-5制备(由加拿大QC QC魁北克市CEROM的S. Rioux博士提供),如Bastien等人所述[3.].简要地,S. sclerotiorum.在马铃薯葡萄糖肉汤(PDA)中培养3天,直至几乎达到饱和。在植物开始开花时就进行接种。首先,悬浮液在搅拌器中均质30秒。然后,将2.7 × 5.5 cm的棉垫片浸泡在悬浮物中。接种物施用于最低结花节的叶柄上。接种后移栽至昼夜温度22℃/18℃、湿度保持在2.5 g/m的不同温室3.有雾化系统。对于验证组,所有植株都在同一天接种,而对于关联组,由于花期的差异,需要几天的时间。接种后7 d测量病变长度。
SNP基因分型和估算
该协会小组是530条加拿大精英线的一部分,我们之前对这些线进行了GBS (猿KI,根据[10)随着时间的推移[4,28].为了最大化数据质量和一致性,所有读操作(940 M 108-bp单端Illumina读操作)都在改进的snp调用流水线(Fast-GBS [27),以及Williams 82参考基因组的最新版本(Wm82.a2.v1) [24].这导致了一个150 K SNPs的目录,所有缺失的数据使用BEAGLE v5进行归位[8正如Torkamaneh和Belzile所说[26].随后,对一组102线(其中15个是关联面板的一部分)进行全基因组测序(WG)[28].所得SNP目录(> 4 M SNPs)作为参考面板,对缺失位点进行大规模的归位。使用vcftools v0.1.16提取并过滤127个关联面板株系的SNP数据(4.1 M位点)[9].我们保留了少量等位基因数(MAC)≥1和少量等位基因频率(MAF)≥0.05的SNPs。估计了连锁不平衡(LD)(使用r2)使用PLINK 1.9的滑动窗口中的所有标记对[22].
人口结构分析
由于SNP目录的大小很大(近1.5 M SNP),使用PLINK 1.9进行修剪[22]删除高ld中的标记(r2 ≥ 0.9). The resulting set of 85 K SNPs was used to assess population structure using fastSTRUCTURE [23, K在1到12之间。使用fastSTRUCTURE中的chooseK工具估计了最可能的亚种群数量[23].
全基因组关联分析
考虑到GWAM,在关联面板中只使用了少量等位基因频率(MAF)≥5%的SNPs,这导致了接近1.5 M过滤的SNPs目录。利用上述表型(平均病变长度)和基因型数据,利用基因组关联预测集成工具(GAPIT version 2)对SSR部分抗性进行关联作图分析[25].为了纠正假阳性关联,使用混合线性模型(Q + K模型),考虑了人口结构(Q矩阵)和亲属关系(K矩阵)。Q矩阵(对于K = 6)是由fastSTRUCTURE导出的,而K矩阵是在TASSEL中生成的。当被测量时,标记-特质关联被认为是显著的p- 值低于一个关键p-对应于错误发现率(FDR)为0.1的值[5].
QTL验证
设计共显性cleaved amplified polymorphic sequence (CAPS)标记,对位于Chr01位点SNP峰的单倍区(染色体1:5594765)中的一个候选SNP进行基因分型。利用两条特异性引物(5 ' - gttgtatggaagtgcaactaaagttct -3 '和5 ' - ggtacttttttttttttaccttac GATGA-3 ')扩增包含靶标SNP的800 bp区域。这两个等位基因可以通过消化产生的扩增子来区分NmuCI。对SSR部分抗性相关等位基因(存在于Maple Donovan中)的PCR产物将被切割一次,从敏感亲本(OAC Bayfield)的等位基因扩增后得到的产物将不切割一次。所有47个F8验证面板的线(上文所述)以及使用该盖标记的两个父母线进行基因分型。
围绕SNP峰值的基因组景观
来自plink的ld值[22提取最显著相关SNP周围的2 Mb窗口,并使用Haploview (V4.2)可视化LD块[2]基于r2值。在含有峰关联的LD区段中发现的基因信息来自SoyBase (www.soybase.org.).利用SnpEFF对该区域核苷酸变异进行功能注释[35].
可用性数据和材料
VCF格式的SNPs完整目录可在FigShare (https://figshare.com/s/20a340d7b7cfe26396c9)和所有原始测序数据公开可在NCBI序列读取档案(SRA)与SRP#研究加入SRP059747 (GBS)和SRP094720 (WGS)。
参考文献
- 1.
Arahana VS,GRAEF GL,Specht Je,Steadman Jr,Eskridge Km。识别QTL抵抗力菌核病sclerotiorum在大豆。作物科学。2001;41:180-8。
- 2.
巴雷特JC,弗莱B,马勒J,戴利MJ。Haploview: LD和单体型图的分析和可视化。生物信息学》2005。https://doi.org/10.1093/bioinformatics/bth457.
- 3.
一种可重复性测定抗结核药物部分抗性的方法菌核病sclerotiorum在大豆。可以j植物sci。2012年。https://doi.org/10.4141/cjps2011-101.
- 4.
Bastien M,Sonah H,Belzile F. Genome宽协会映射菌核病sclerotiorum大豆抗性的基因分型测序方法。植物基因组。2014。https://doi.org/10.3835/plantgenome2013.10.0030.
- 5.
Benjamini Y,Yekutieli D.使用虚假发现率的定量特质基因座分析。遗传学。2005; 171:783-90。
- 6.
植物寄主指数菌核病sclerotiorum.可以j植物pathol。1994年。https://doi.org/10.1080 / 07060669409500766..
- 7.
博尔顿MD,Thomma BPHJ,纳尔逊BD。菌核病sclerotiorum(lib。)de bary:使用过多的病原体的生物学和分子特征。莫尔植物途径。2006;DOI:https://doi.org/10.1111/j.1364-3703.2005.00316.x。
- 8.
利用局部单倍型聚类进行全基因组关联研究的快速和准确的单倍型相位和缺失数据推断。2007; 81:1084-97。
- 9.
等。不同的调用格式和VCFtools。生物信息学》2011。https://doi.org/10.1093/bioinformatics/btr330.
- 10.
liu j, liu j, liu j, et al., et al. .一个强大的,简单的基因分型测序(GBS)方法的高多样性物种。《公共科学图书馆•综合》。2011。https://doi.org/10.1371/journal.pone.0019379.
- 11.
郭X,王D,Gordon S,Helliwell E,Smith T,Berry S等人。QTL潜在局部抵抗的遗传映射菌核病sclerotiorum大豆PI 391589A和PI 391589B。作物科学。2008;48:1129-39。
- 12.
郝东,程华,尹震,崔胜,张东,等。大豆产量及产量构成因素单核苷酸多态性和单倍型的鉴定(大豆)跨多个环境的地方种族。Theor Appl Genet, 2012。https://doi.org/10.1007/S00122-011-1719-0..
- 13.
关键词:大豆,白霉菌,抗性,qtl定位作物科学》2010。https://doi.org/10.2135/cropsci2009.06.0311.
- 14.
宋庆杰,张志强,张志强,等。大豆多居群连锁不平衡的高度可变模式。遗传学》2007。https://doi.org/10.1534/Genetics.106.069740.
- 15.
Iquira E,Humira S,Belzile F. QTL的关联映射菌核病利用基因分型测序(GBS)方法在大豆植物引种收集中的茎腐抗性。BMC Plant Biol. 2015。https://doi.org/10.1186/s12870-014-04087.1186/s12870-014-0408-Y..
- 16.
刘建军,刘建军,刘建军,等。大豆种质对白霉菌的抗性评价及QTL验证植物科学学报,2018;https://doi.org/10.3389/fpls.2018.00505.
- 17.
金HS,DIES BW。偏离部分抵抗的遗传菌核病大豆茎腐病。作物科学。2000;40:55 - 61。
- 18.
Koenning SR, Wrather JA。2006 - 2009年植物病害对美国大陆大豆产量潜力的抑制植物健康计划2010。https://doi.org/10.1094/php-2010-1122-01-rs..
- 19.
Mamidi S, Chikara S, Goos RJ, Hyten DL, Annam D, et al.;全基因组关联分析确定了与大豆缺铁褪绿病相关的候选基因。植物基因组。2011。https://doi.org/10.3835/plantgenome2011.04.0011.
- 20.
Mila AL, Yang XB。土壤温度和水势波动对苗木菌核萌发和药剂生产的影响菌核病sclerotiorum.工厂说。2008;92:78 - 82。
- 21.
Moellers TC,Singh A,Zhang J,Brungardt J,Kabage M,Mueller DS等。大豆的主要和背景基因座研究菌核病sclerotiorum电阻显示多环境中多种电阻模式。SCI REP。2017。https://doi.org/10.1038/s41598-017-03695-9.
- 22.
李志刚,李志刚,李志刚,等。PLINK:全基因组关联和基于群体连锁分析的工具集。Am J Hum Genet, 2007;81http://pngu.mgh.harvard.edu/purcell/plink/.
- 23.
Raj A, Stephens M, Pritchard JK。fastSTRUCTURE:大SNP数据集中群体结构的变异推断。遗传学。2014;197:573 - 89。https://doi.org/10.1534/genetics.114.164350.
- 24.
Schmutz J,Cannon SB,Schlueter J,MA J,Mitros T,Nelson W等人。古代聚丙烯大豆的基因组序列。自然。2010; 463:178-83。
- 25.
唐勇,刘旭,王军,李敏,王强,等。GAPIT版本2:用于基因组关联和预测的增强集成工具。植物基因组。2016。https://doi.org/10.3835/plantgenome2015.11.0120.
- 26.
扫描与填充:结合基因分型、SNP阵列和全基因组重测序数据的超密SNP基因分型。《公共科学图书馆•综合》。2015;10:e0131533。
- 27.
Fast-GBS:从基因分型测序数据中高效、高精度调用SNPs的新途径。BMC生物信息学,2017 b。https://doi.org/10.1186/s12859-016-1431-9.
- 28.
张志强,张志强,张志强,等。短季大豆全基因组核苷酸和结构变异的综合描述。生物技术学报,2017a。https://doi.org/10.1111/pbi.12825.
- 29.
黄志强,王志强,王志强,等。大豆菌核病抗性QTL的鉴定。作物科学。2008;48:2209-14。
- 30.
魏伟,吴旭东,马志强,等。对巴西分离物抗性的全基因组关联图谱菌核病sclerotiorum大豆基因型主要来自巴西。BMC基因组学。2017年。https://doi.org/10.1111/tpj.12810.
- 31。
张绍平,袁军,杜伟,等。结合GWAS和基因表达数据对大豆抗白霉菌功能特性进行研究。生物技术学报。2018。https://doi.org/10.1111/pbi.12918.
- 32.
伤痕贾瓦,霍宁斯SR。美国2003年至2005年美国大豆产量的疾病影响估计。j Nematol。2006; 38:173-80。
- 33.
曾伟,陈建平,郝杰。大豆菌核病的生物防治。控制杂志》2012。https://doi.org/10.1016/j.biocontrol.2011.09.012.
- 34.
赵x,韩y,李y,刘d,sun m等。基因座和候选基因鉴定抗性菌核病sclerotiorum在大豆(大豆L. MERR。)通过关联和联系地图。工厂J. 2015。https://doi.org/10.1111/pbi.12918.
- 35.
王丽萍,王丽萍,王丽萍,等。一个用于注释和预测单核苷酸多态性影响的程序,SnpEff:果蝇黑腹菌株w1118基因组中的SNPs;iso作业2;iso作业3。飞2012;80 - 92。https://doi.org/10.4161/fly.19695.
确认
作者希望感谢所有的资助者。这项工作得到了SoyaGen基金(www.soyagen.ca.),由加拿大基因组(Genome Canada)和其他合作伙伴资助。
资金
这项工作得到了SoyaGen基金(www.soyagen.ca.)由加拿大基因组和其他合作伙伴资助,详情如下[项目#5801至F.B.]。资助方没有参与研究设计、收集、分析、数据解释、撰写这篇文章或决定将其提交出版。
The complete list of funders includes Génome Québec, Genome Canada, the government of Canada, the Ministère de l’Économie, Science et Innovation du Québec, Semences Prograin Inc., Syngenta Canada Inc., Sevita Genetics, Coop Fédérée, Grain Farmers of Ontario, Saskatchewan Pulse Growers, Manitoba Pulse & Soybean Growers, the Canadian Field Crop Research Alliance and Producteurs de grains du Québec.
作者信息
从属关系
贡献
CB, DT和FB构思了这个项目。CB和MB进行温室表型。形式分析。CB和FB写作-原始草案。作者们阅读并批准了这个版本的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
127个大豆品系接种7 d后对SSR的响应。(*)表示用于WGS的行(补充文件“补充表S1”)
附加文件2:图S1。
根据Chr15标记的峰值等位基因,127个品系的病变长度分布
附加文件3:图S2
.Chr01上区域的LD地块图。箭头表示显著相关snp的位置。缩写词。GBS: Genotyping-by-sequencing。全基因组关联图谱。LD:连锁不平衡。马铃薯葡萄糖琼脂。PCR:聚合酶链反应。QTL:数量性状位点。 RILs: Recombinant inbred lines. SLAF-seq: Specific-locus amplified fragment sequencing. SNP: Single nucleotide polymorphism. SSR: Sclerotinia stem rot. USDA: United states department of agriculture. WGS: whole genome sequencing
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
Boudhrioua C., Bastien M., Torkamaneh, D.。等等。全基因组关联图谱菌核病sclerotiorum使用全基因组重构数据在大豆中的抵抗力。BMC植物杂志20.195(2020)。https://doi.org/10.1186/s12870-020-02401-8
已收到:
接受:
发表:
关键词
- WGS
- 菌核病sclerotiorum
- 大豆
- Gwam.