抽象的
背景
长度非编码RNA(LNCRNA)具有若干已知的功能,涉及植物中各种生物调控过程。然而,LNCRNA在花生种子发育期间的可能作用尚未得到充分探索。
结果
本研究采用花生重组自交系(RIL8种子大小的不同,用于调查开花(DAF)后15天和35天源自种子发育的综合LNCRNA谱。我们鉴定了总共9388年的已知和4037个新型LNCRNA,从中有1437次差异表达LNCRNA。有趣的是,许多LNCRNA的表达模式可以在两个密切相关的自交系之间存在非常不同,并且这些LNCRNA在35个DAF的一个ril中仅表达。发现一些差异表达的LNCRNA与推定的顺式作用靶基因有关,并预测参与转录,运输,细胞分裂和植物激素生物合成。通过QPCR验证了几种代表性LNCRNA和12个蛋白质编码基因的表达模式。在大多数LNCRNA和其靶基因之间观察到相同的表达模式。11 LNCRNA,XR_001593099.1,MSTRG.18462.1,MSTRG.34915.1,MSTRG.41848.1,MSTRG.12404.1,MSTRG.26719.1,MSTRG.35761.1,MSTRG.20033.1,MSTRG.13500.1和MSTRG.9304.1及其CIS及其CIS- 治疗靶基因可能在花生种子发育中发挥关键作用。
结论
这些结果为lncrna介导的花生种子发育调控提供了新的信息,有助于全面理解花生种子发育的分子机制。
背景
近年来,下一代测序方法的使用揭示了真核生物的转录是复杂的。许多真核生物基因组被普遍转录,产生编码和非编码rna (ncrna)。非编码rna (ncrna)是一类不能编码蛋白质的rna;然而,它们在许多生物过程中起着重要的调节作用[1].ncrna可根据其长度分为小rna (< 200 nt)和长非编码rna (lncrna,大于200 nt)。小RNA进一步分为microRNAs (miRNAs)、小核仁RNA (snoRNAs)、小干扰RNA (sirna)和小核RNA (snrna) [2].根据基因组位置和背景,lncrna可分为长基因间非编码rna (lincRNAs)、自然反义转录本(NATs)和内含子rna (incRNAs) [3.,4].
lncrna表现出组织和细胞特异性的表达模式,在不同物种之间往往表现出较差的保守性[5,6].前期研究发现,lncrna参与多种活动,包括聚集、转运蛋白、通过近端调控启动子活性等CIS.- 或者反式-作用序列,表观遗传修饰,沉默或抑制[7,8].LNCRNA还涉及生长和发育,哺乳动物的疾病[9,10].
在各种植物中表征了几种LNCRNA的生物学功能。例如,COOLAIR和冷风规范表达开花轨迹C(方法),确定拟南芥开花的时间11,12].在杂交水稻中,光周期敏感的雄性不育性与LNCRNA相关,称为漫长的雄性生育率相关的RNA(LDMAR)[13].在棉花幼苗中,两个LNCRNA,GHLNCNAT-ANX2和GHLNCNAT-RLP7提供了对真菌病原体的抗性增强verticillium dahliae.和葡萄孢属cinereal[14].在Medicago truncatula, lncRNAENOD40通过诱导核RBP的重新定位,与土壤根瘤菌相互作用形成根瘤[15].尽管有了这些进展,但关于lncRNA在植物,尤其是花生中的作用的研究仍然很少。迄今为止,已经在几种植物中进行了lncrna的全基因组分析,但花生中参与种子发育的lncrna的数量和特征尚未被探索。
花生(落花生hypogaeaL.),异源四倍体种(2n = 4x = 40;AABB (AABB)是一种重要的全球石油和蛋白质生产作物。花生种子大小是花生育种的主要农艺性状,因此了解花生种子发育的分子机制是花生育种工作者和育种工作者的当务之急。最近的研究集中在对花生种子发育有重要贡献的一些基因的鉴定上[16].此外,涉及花生种子发育中的几种miRNA及其靶基因的功能已经表现为[17].但是,没有报告提供有关LNCRNA参与的任何信息及其在花生种子发育机制中的靶向基因的任何信息。在这项研究中,我们使用了两个花生rils来更好地了解Lncrna在花生种子发育中的功能。开花(DAF)后15天和35天的LNCRNA表达谱的特征在于来自RIL的两个姐妹线8,其中线8106具有中等大小的吊舱和线8107具有具有不同种子尺寸的超大荚。我们在花生中识别和验证了LNCRNA;然后通过比较和分析两个花生rils之间的不同种子发育阶段的LNCRNA表达谱来鉴定差异表达的LNCRNA(DEL);因此,了解DELS在花生种子发展中可能的作用。
结果
排序花生lncrnas
为了研究花生种子发育过程中LNCRNA的动态变化,在两个种子发育阶段(15个DAF和35个DAF)下进行两种花生rils的全转录组合组特异性RNA测序,用三种生物重复进行。总共通过高通量测序产生了超过1亿原始读取。快速QC具有验收的算法,提供了大于95%的平均质量得分(Q30%),表明RNA-SEQ数据非常可靠。从原始读取,超过97%的读数是干净的读取(附加文件1:表S1)。
花生中lncrna的鉴定与鉴定
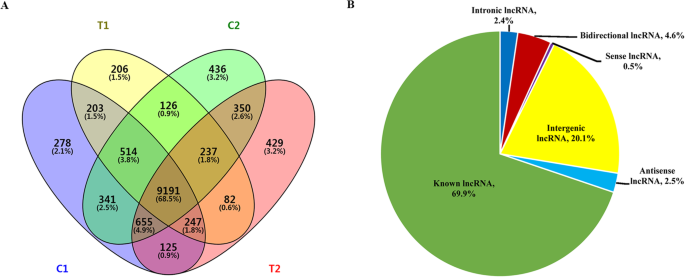
清洁读数被映射到两个二倍体胰岛基因参考基因组(A. Duranensis.和A. iPaensis.)使用tophat。然后,使用Stringtie组装并注释该转录物。鉴定,73,180,72,725,74,702,74,324,74,027,73,964,69,645,69,155,69,645,69,155,69,645,70,516,69,935和72,215个独特的MRNA,包括C1(RIL8106-15DAF),C2(RIL8107-15DAF),T1(RIL8106-35DAF)和T2(RIL8107-35DAF),每个分别有三个复制样本(附加文件2:表S2)。其余的转录本根据其长度和编码潜力进行过滤;舍弃小于200 bp的转录本,剔除CPC评分可能为>−1、CNCI评分为> 0的转录本[18,19].剩余的转录物被认为是LNCRNA,从该12528,9429,10,177,10,274,10,089,10,131,9005,8867,9044,9388,9398和9693个独特的LNCRNA分别从十二个样本中鉴定出来(附加文件2:表S2)。在本研究中确定了13,425个独特的LNCRNA(附加文件3.:表S3)。在15个DAF(C1和11,850中的11,554中,C2中11,554中的11,554中发现的LNCRNA的数量远远高于35 DAF(T1和11,316中的10,806),并且9191 LNCRNA对所有四个样品常见(无花果。1一种)。
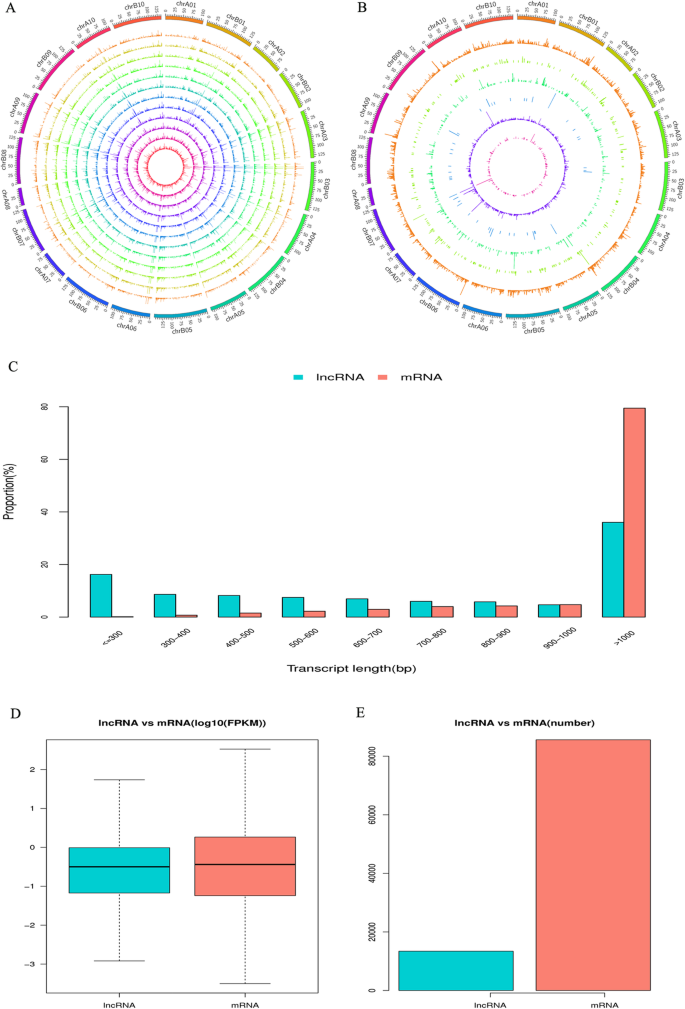
所有的lncrna都被定位到花生基因组的20条染色体上。我们的结果表明,lncrna均匀分布在这些染色体上,没有位置的偏好(图。2A和B)。根据这些LNCRNA的基因组位置,2693代苯基(20.1%),616双向(4.6%),340反义(2.5%),320次内肾上腺素(2.4%),鉴定为68次感测LNCRNA(0.5%)。此外,在本研究中还检测到9388名已知的LNCRNA(69.9%)(图。1b)。这些LNCRNA的长度的估计表明,大多数LNCRNA具有超过1000bp(图。2c).此外,利用外显子模型每百万映射reads (FPKM)的每千碱基片段(fragments per kilbase of exon model per million mapping reads)对每个转录本的表达水平进行评估,发现lncrna的总体表达水平和数量都低于mrna(图)。2d和e)。结果与上一次在高地棉花的研究一致[20.].
花生lncrna差异表达分析
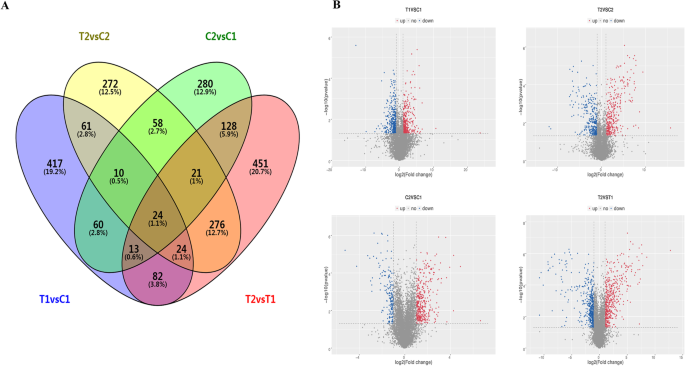
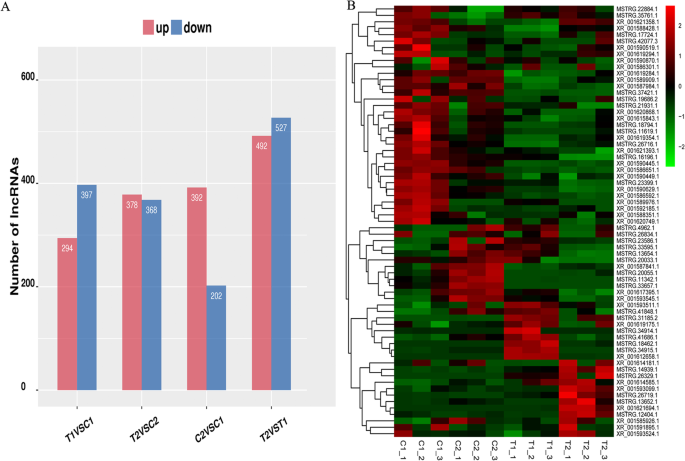
为了分析两个花生RILs在35 DAF和15 DAF位点lncrna的表达差异,我们比较了lncrna在所有文库中的归一化表达(FPKM)。采用以下标准鉴别不同组比较中差异表达的lncrna:(1) log2(倍变)> 1或log2(倍变)<−1,(2)统计学意义(p值< 0.05)。我们最终从四种不同的比较中鉴定出2178个差异表达的lncrna。其中,C2与C1、T2与T1、T1与C1、T2与C2比较,差异表达的lncrna分别为594、1019、691、746个,在四种比较中差异表达的lncrna共有24个(图2)。3.和附加文件4:表S4-1、S4-2、S4-3、S4-4)。此外,差异表达lncRNAs四两个瑞来斯之间的比较表明,数量的增长和衰减lncRNAs关系密切,如294年和397年衰减lncRNAs T1与C1, 378年和368年在T2与C2,和492年和527年在T2和T1(无花果。4a).然而,在15 DAF期,C2和C1中分别鉴定出392个上调和202个下调的lncrna,其中大种子系(RIL8107)和中种子系(RIL8106)差异显著(图8106)。4一种)。
此外,我们基于两种花生rils之间的两个发育阶段的表达模式聚集了67个差异表达的LNCRNA。我们的结果表明,在两个rils中,35 DAF阶段的LNCRNA之间的表达差异。例如,XR_001590629.1和MSTRG.37421.1被测调节,而XR_001593099.1在两个rils中上调(图。4b)。有趣的是,许多LNCRNA在35个DAF阶段的一个rils中主要表达。十五(15)LNCRNA表达,包括XR_001593511.1,MSTRG.18462.1,MSTRG.31185.1,MSTRG.34915.1,MSTRG.41848.1仅在RIL8106的中型种子线上上调,而仅包括九(9)个LNCRNA,包括XR_001614585.1,XR_001593524.1,MSTRG.22884.1,XR_001621358.1,XR_001621358.1,XR_001621694.1,MSTRG.12404.1,MSTRG.13652.1,MSTRG.26719.1,MSTRG.26719.1,MSTRG.35761.1仅在RIL 8107的大型种子线上进行上调(图。4b).这些结果表明,这些差异表达的lncrna可能在调节花生种子发育中发挥重要作用。
差异表达lncrna的验证
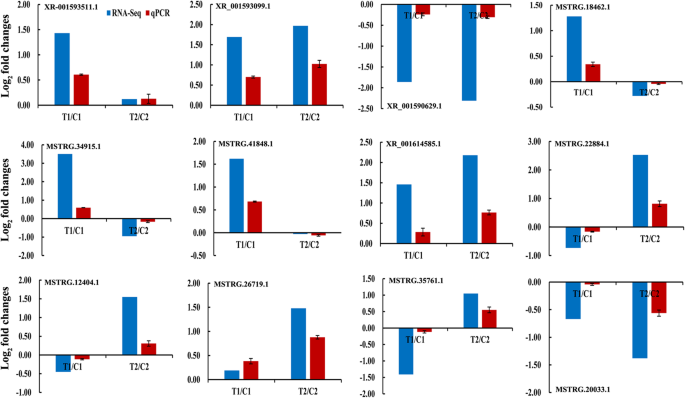
要从RNA-SEQ确认数据,我们随机选择了12个LNCRNA以验证其表达式QPCR(图。5).这些LNCRNA的QPCR结果与通过高通量测序的QPCR结果一致。例如,在PEANUT rils中确认LNCRNA XR_001593099.1的表达水平为上调,而LNCRNA XR_001590629.1在两个rils中下调。然而,LNCRNA XR-001593511.1,MSTRG.18462.1,MSTRG.34915.1和MSTRG.41848.1仅在RIL8106中显示出上调的表达,通过两种方法。类似地,LNCRNA XR_001614585.1,MSTRG.2404.1,MSTRG.12404.1,MSTRG.26719.1和MSTRG.35761.1仅通过两种方法在RIL8107中进行上调。此外,来自QPCR方法的结果还验证了LNCRNA MSTRG.20033.20033.1主要在RIL8107中均下调,如RNA-SEQ所示。这些结果表明RNA-SEQ分析是对LNCRNA表达模式的高度可靠预测。
花生种子发展中差异表达的LNCRNA的功能
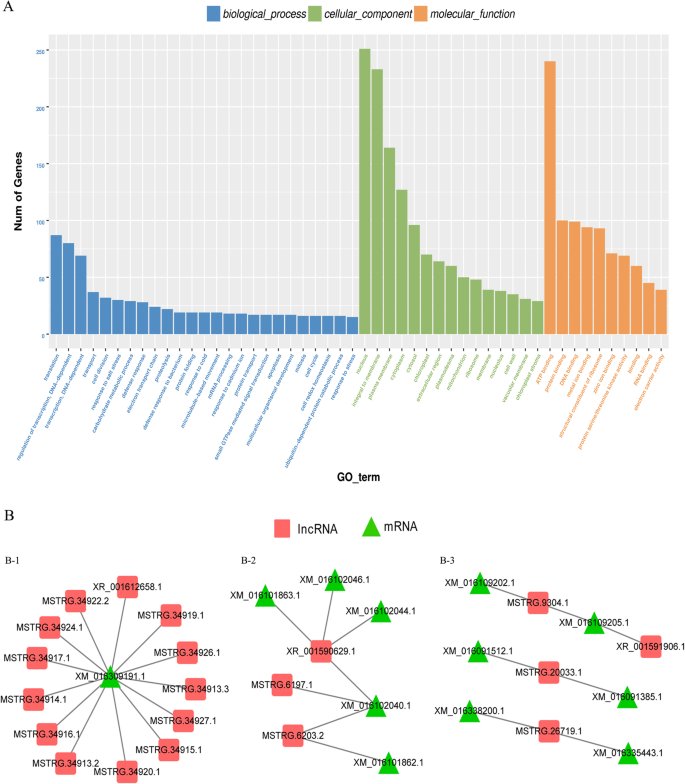
lncrna被证明优先接近它们的目标基因[21,22,23].为了探讨LNCRNA的潜在功能,我们定义了与每个相应的调节的LNCRNA为可预测的靶标的共表达蛋白质编码基因。在15个DAF和35个DAF阶段的两个花生rils之间鉴定差异表达的LNCRNA,以便将LNCRNA描述在种子开发阶段(附加文件5:表S5)。进行基因本体(GO)分析以对这些蛋白质编码基因进行分类。有25类生物过程,这些蛋白质编码基因主要富集“转录调节”,“翻译”和“运输”。此外,将一些重要的生长和发展相关基因鉴定为LNCRNA靶标,包括“细胞分裂”,“碳水化合物代谢过程”,“细胞周期”等(图。6一种)。这些发现表明,这些差异表达的LNCRNA可以通过调节相关蛋白质编码基因的表达来参与花生的种子发育。
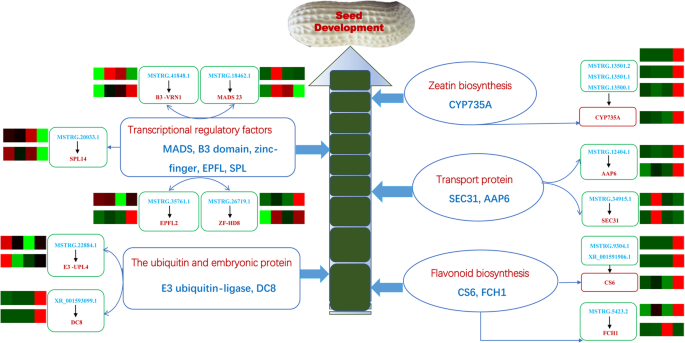
为了进一步了解差异lncrna在种子发育中的作用,我们构建了推测的交互网络以揭示lncrna与蛋白编码基因之间的关系(图)。6b).其中5个蛋白编码基因参与物质运输,包括蛋白质运输(XM_016309191.1)、铁运输(XM_016101863.1)、糖运输(XM_016101862.1)、生长素运输(XM_016102040.1)和翻译过程(XM_016102046.1)。在种子发育过程中被16种lncrna调控(图。6b - 1和6 b - 2)。3个转录因子,含B3结构域的转录因子(XM_016091512.1)、squamosa启动子结合样蛋白(XM_016091385.1)和锌指同源结构域蛋白(XM_016335443.1)被包含在Fig的网络中。6B-3,参与在种子发育期间在下游激活生长相关基因。此外,几种重要的生物过程,如Go:0009734,助长介导的信号通路;GO:0009740,赤霉酸介导的信号通路;GO:0051301,细胞划分,也被发现在本研究中(附加文件5:表S5)。这些结果提示,差异表达的lncrna可能调控调控花生种子发育的转录、运输、激素信号转导和翻译等多种生物学过程中的基因。
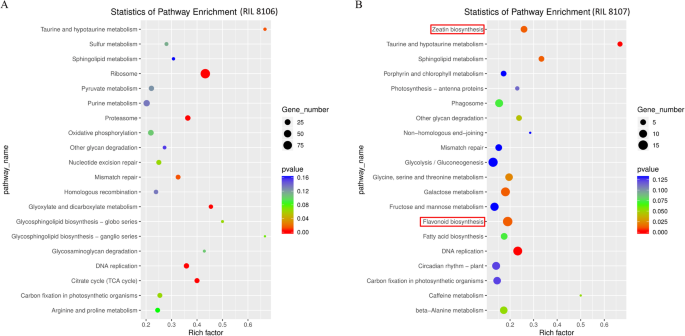
此外,基于KEGG分析,这些蛋白编码基因分别在两个花生ril的20条通路中显著富集(图2)。7a和b)。在这些途径中,有三个最常见的类别,包括核糖体、嘌呤代谢和氧化磷酸化,在中等大小的种子系RIL 8106中显著富集。然而,在大粒种子系RIL 8107中,三个最常见的途径是糖酵解/糖异生、类黄酮生物合成和半乳糖代谢。有趣的是,一些lncrna靶基因仅在RIL8107中参与了玉米素的合成(图)。7b).在该途径中,mstrg . 135001、MSTRG.13501.1和MSTRG.13501.2参与了玉米素的生物合成和CYP735A蛋白的积累水平(附加文件)5:表S5)。这些研究结果表明,通过影响内源激素积累水平,LNCRNA对花生种子发育的调节具有显着影响。
通过qPCR验证lncRNA靶基因
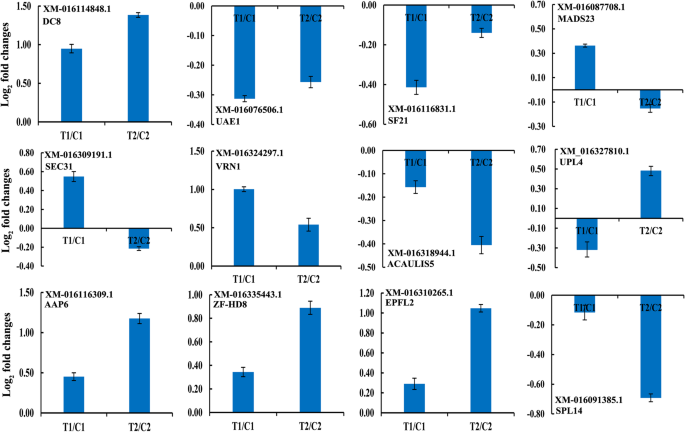
为了确定lncrna与相关蛋白编码基因的关系,我们通过qPCR验证了12个推测的顺式作用蛋白作为12个差异lncrna的靶蛋白的表达模式。在35 DAF阶段,大多数lncrna及其假定的顺式作用靶点共同表达并上调或下调(图)。5和无花果。8).在两种花生rils中调节LNCRNA XR_001593099.1的表达和胚胎蛋白DC-8样(XM_016114848.1)的靶标。然而,LNCRNA MSTRG.18462.1的表达和浊箱转录因子23样(XM_016087708.1),MSTRG.34915.1和蛋白质转运蛋白SEC31样靶(XM_016309191.1),MSTRG.41848.1和含B3结构域的转录因子VRN1样(XM_016324297.1)的靶标在35dAF中仅在RIL 8106中调节。同时,MSTRG.22884.1和E3泛素 - 蛋白质连接酶UPL4(XM_016327810.1)的表达式),MSTRG.12404.1和氨基酸透气6样(XM_016116309.1),MSTRG.26719.1和锌 - 手指同源域蛋白8样(XM_016335443.1),MSTRG.35761.1和表皮图案化因子样蛋白2(XM_016310265。1)were up-regulated at the 35 DAF only in the RIL 8107. Moreover, lncRNA MSTRG.20033.1 and squamosa promoter-binding-like protein 14 (XM_016091385.1) was down-regulated in the RIL 8107. These results indicate that lncRNAs are involved in regulating peanut seed development by modulating the expression of their cis-acting target genes.
讨论
花生种子发育过程中lncrna的鉴定及其功能验证
LNCRNA的调节作用越来越多地在植物中解开。已发现LNCRNA参与植物生长,发育,繁殖和非生物应激反应,如玉米,水稻和拟南芥[24,25,26,27].在花生中,以往对非编码RNA的研究主要集中在miRNA的鉴定和功能分析上[17,28,29,但lncrna的作用,特别是与花生种子发育的关系尚未见报道。花生种子发育是一个复杂的生物学过程,受基因协调表达调控。根据前面对花生种子发育的各个阶段的描述[17],荚果发育的第一个标志出现在15 DAF,荚果在35 DAF左右膨大到最大,称为定型果。因此,我们从两个花生RILs中选择这两个阶段的种子,使用高通量测序技术对lncrna进行全基因组分析。我们共鉴定了13,425个不同发育阶段的独特lncrna。这些lncRNAs 4037小说lncRNAs,包括MSTRG.37421.1 MSTRG.18462.1, MSTRG.31185.2, MSTRG.34915.1, MSTRG.41848.1, MSTRG.22884.1, MSTRG.12404.1, MSTRG.13652.1, MSTRG.13500.1, MSTRG.13501.1,等等,首次报道了在花生种子,种子发展的指示他们的角色。此外,我们在35 DAF阶段鉴定了1437个差异lncrna,而在两个ril之间鉴定了15个DAF。为了更好地了解差异lncrna在花生种子发育中的功能,我们分析了与这些lncrna共同表达的蛋白编码基因(附加文件)5:表S5)。与一些蛋白质编码基因共表达的一些LNCRNA在两个rils之间出现在35个DAF阶段,无论种子大小如何。例如,LNCRNA XR_001593099.1在花生rils中上调,LNCRNA XR_001593099.1的靶基因的表达也增加。靶基因,胚胎蛋白DC-8样(DC8)已被证明在胚胎组织的胚胎发生和胚乳组织的细胞壁中强烈表达[30.,31],表明这一点DC8在花生种子发展中发挥作用。我们的工作提供了对研究界有用的花生LNCRNA的重要资源。
特定的LNCRNA参与中等尺寸种子线Ril 8106的种子发育
在本研究中,许多lncrna及其假定的顺式作用靶基因主要在35 DAF阶段的RIL8106中共同表达。例如,MSTRG.18462.1、MSTRG.34915.1和MSTRG.41848.1这三种lncrna仅在35 DAF时在RIL 8106中上调(图1)。5).lncRNA的靶基因MSTRG.18462.1编码MADS-box转录因子23-like (疯了).此前的研究表明,MADS家族成员是控制开花和果实发育的遗传网络的关键元素[32].Tagl1是属于疯子家族的转录因素,并且已被证明涉及许多生物处理的番茄中成熟的果实[33].在这幅作品中,层次表达的淋漓尽致疯了随着lncRNA MSTRG.18462.1的表达增加,可能调控了MSTRG.18462.1的表达疯了为种子发育的过程。另一个上调的lncRNA MSTRG.34915.1在转运蛋白sec31-like的编码区上游表达(SEC31)在35个DAF阶段。在拟南芥中,证券交易委员会被证明基因对于配子体发育至关重要,并且是制育花粉的分泌贩运所必需的[34].这些结果提示,lncRNA MSTRG.34915.1可能调控MSTRG.34915.1的表达SEC31这可能有助于花生种子的发育。与MSTRG.34915.1类似,lncRNA MSTRG.41848.1位于包含B3结构域的转录因子VRN1-like编码区上游约10 kb处(VRN1.)(额外的文件5:表S5)。在RIL8106基因中,35 DAF表达均上调。此前的研究表明,B3结构域蛋白参与种子发育、激素反应和开花时间[35,36].因此,LNCRNA MSTRG.41848.1可以通过调节表达式来调节花生的种子发育VRN1..这些lncrna及其假定的顺式作用的靶基因的高表达是RIL 8106中特定种子发育的假设机制。
在大尺寸种子系RIL 8107中,特异性的lncrna参与了种子发育
许多LNCRNA及其推定的蛋白质编码基因仅在RIL8107中调节,包括LNCRNA MSTRG.22884.1,MSTRG.12404.1,MSTRG.26719.1和MSTRG.35761.1(图。5和无花果。8).lncRNA MSTRG.22884.1在RIL 8107及其靶酶E3泛素蛋白连接酶(UPL4.).E3家庭成员在拟南芥种子发育过程中调节配子发生和细胞周期过程中发挥着重要作用[37,38,39].增加的表达UPL4.基因可能与其在花生种子发育中的作用有关。另一个上调的LNCRNA MSTRG.12404.1靶标氨基酸允许6状(AAP6)基因,该基因编码植物特异性氨基酸跨膜转运体,并参与氨基酸摄取[40].在本研究中,表示AAP6基因在35 DAF表达上调,说明AAP6也可以参与花生种子发展。LNCRNA MSTRG.26719.1的靶基因是锌手指Homeodomain蛋白8样(ZF-HD8).先前的研究表明ZF-HD基因在花卉和叶片发展中编码一组转录调节剂[41,42].我们鉴定了位于编码序列的上游的LNCRNA MSTRG.26719.1ZF-HD8(附加文件5:表S5)。结果表明,MSTRG.26719.1可能通过调控的表达参与了种子发育的调控ZF-HD8.类似地,另一个上调的LNCRNA MSTRG.35761.1的靶基因是表皮图案化因子样蛋白2(EPFL2),它编码参与细胞分化的植物特异性转录因子。在大米、RAE2(一个EPFL基因)可以促进AWN伸长率的血管系统细胞的增殖[43].拟南芥中报告了类似的现象atepfl4.和atepfl6.坐标开发花序架构[44].在我们现在的工作中,我们发现的是水平EPFL2-特异性mRNA在35 DAF期增加(图。5和无花果。8).增加的表情EPFL2也可能通过调节细胞分化在花生种子发育中发挥作用。
相比之下,lncRNA MSTRG.20033.1及其可预测的靶基因squamosa启动子结合样蛋白14 (SPL.),显著下调,主要发生在RIL8107(图。5和无花果。8).的SPL.在植物发育阶段过渡,花卉和果实开发,吉布林斯信令等中起重要作用。[45,46,47].在我们目前的研究中SPL.基因被lncRNA MSTRG.20033.1调控,并参与花生种子发育,只是延长了发育阶段的变化。我们的研究结果表明,这些lncrna及其推测的蛋白编码基因仅在RIL 8107中有特定的调控,这可能解释了RIL 8107和RIL 8106种子大小的差异。
依赖LNCRNA的监管网络可能涉及花生的种子发展
植物内源激素在各种发育过程中发挥重要作用。植金素在植物中调节大量的生长和发育过程[48].细胞分裂素被认为是参与植物许多生物过程的主要激素,如顶端优势[49],根系增殖[50]和生殖发展[51].先前的研究表明CYP735A基因在Trans-Zeatin(内源性细胞蛋白)生物合成中起着重要作用麻风树[52].已经发现使用CRISPR-CAS9系统的JCCYP735A突变体具有显着降低的反式蛋白和反式蛋白质 - 核糖苷e显示出严重的延迟生长。在这项研究中,我们发现几种LNCRNA,包括MSTRG.13500.1,MSTRG.13501.1和MSTRG.13501.2,通过调节表达来参与Zeepin生物合成CYP735A仅在RIL8107(图。7b,无花果。9,以及附加文件5:表S5)。这些发现表明,这些LNCRNA可以通过影响内源性细胞蛋白生物合成来发挥花生种子发育的关键作用。此外,黄酮类化合物在植物开发中发挥着重要作用。在拟南芥中,特定组织中的黄酮类化合物,花青素和花青蛋白在特定组织中的积累导致生物方法的调节,包括光衰减,氧化应激保护和更重要的种子涂层发育[53].在我们目前的工作中,我们发现LNCRNA MSTRG.9304.1和XR_001591906.1通过调节Chalcone合酶6的表达,参与黄酮类生物合成,如(密室)仅在RIL8107中(图。7b,无花果。9,以及附加文件5:表S5),表明他们在花生种子发展中的潜在作用。
除了上述lncrna外,还评估了它们推测的蛋白质编码基因在花生种子发育中的功能(图。9).例如lncRNAs MSTRG.18462.1和targetMADS23;mstrg.41848.1和目标B3-VRN1.;MSTRG.22884.1和目标E3-UPL4;MSTRG.26719.1和目标ZF-HD8;MSTRG.35761.1和目标EPFL2;MSTRG.20033.1和目标SPL14.也被确认参与种子发育。识别这些与生长发育相关的lncrna,并对其调控网络进行表征,可以增强对花生和植物种子发育相关分子机制的理解。
结论
花生种子发育是一个复杂的过程,涉及一系列生理、形态和转录的变化。本研究首次报道了花生种子发育过程中lncrna的表达情况。共发现已知lncrna 9388个,新发现lncrna 4037个。其中,1437个lncrna在两个ril的种子发育过程中有差异表达。通过对这些lncrna及其相关蛋白编码基因的功能分析,发现这些lncrna - mrna中至少有11个调控模块在花生种子发育中发挥重要作用。我们的研究结果为lncrna及其在花生中的可能功能提供了新的见解,以及它们在种子发育过程中的表达模式;为花生产量相关性状的遗传改良提供了新的途径。
方法
植物材料
以花生品种花优7号和花优4号为材料,通过人工杂交获得了2个8代重组自交系RIL8106和RIL8107。这两个ril都是直立的弗吉尼亚型,有8-10个分枝,但它们的种子大小有主要区别。RIL8106的种子比RIL8107小,相应的100粒重为100 g,相应的100粒重为182 g。分别在15 DAF和35 DAF时从两个ril中收获新鲜种子,分别命名为C1、C2、T1和T2。随机抽取4个种子样品,在−80℃下进行液氮处理。每个处理采集3个重复的样品。
cDNA文库的构建及深度测序
在制造商的程序之后,使用Trizol Reagent(Invitrogen,CA)从每个花生种子样品中提取总RNA。根据震中核糖零金试剂盒(Illumina,San Diego,USA)中提供的协议,核糖体RNA从约10μg的总RNA耗尽,代表特定的脂肪型。净化后,将聚(a) - 或聚(a)+ RNA级分切割成小片段并逆转录,以构建基于RNA-SEQ样品制备试剂盒(Illumina,San Diego的所述方案的所述最终cDNA文库, 美国)。然后,在供应商推荐的协议之后,我们在LC Biotech(杭州)的Illumina Hiseq4000序列仪上进行了成对结束测序(150 bp)。
读取映射和转录组件
首先,使用CENCADAPT去除低质量读取(包括适配器污染,低质量碱基和未确定基础)[54].然后使用FASTQC验证序列质量(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/).然后我们使用TopHat [55]通过将它们映射到两个二倍体花生物种的参考基因组来获得清洁的配对末端读取,A. Duranensis.和A. iPaensis.,从peanutbase数据库(https://www.peanutbase.org/).要构建转录组,使用stringtie组装映射的读取[56].在生成最终转录组后,Stringtie和Ballgown [57]用于估计所有转录物的表达水平。
lncrna鉴定
根据LNCRNA的特征,我们采用以下步骤从转录组组件的转录物中鉴定LNCRNA [58].首先,将所有与参考基因组重叠的转录本定义为“已知lncRNA”。其次,在剩余的转录本中,选择长度超过200 bp的转录本进行蛋白编码评分测试,计算编码潜能计算器(CPC) [18]及编码-非编码索引(CNCI) [19].最后,具有CPC得分< - 1和CNCI得分<0的转录物被定义为新的LNCRNA。
差异表达模式的分析
StringTie [56]通过计算FPKM来计算所有转录本的表达水平,包括mrna和推测的lncrna [59].差异表达分析采用R package - Ballgown [57]与| log2(折叠变化)|> 1和p值< 0.05。
lncrna靶基因功能分析
为了探究lncrna在花生种子发育中的作用,我们预测了lncrna的顺式靶基因。lncrna可能对邻近的靶基因起顺式作用[60].在本研究中,我们通过python脚本选择lncRNA上、下游10万bp的编码基因[61].此外,我们利用BLAST2GO对lncrna的靶基因进行了功能分析[62, p值< 0.05表示显著性。
定量实时聚合酶链反应
我们通过RT-qPCR检测选定的lncrna和蛋白编码基因的转录水平(附加文件)6表S6),根据CFX96实时系统(Bio-Rad, Hercules, CA, USA)和TB Green Premix Ex Taq II (TaKaRa,大连,中国)的制造商说明。采用RNAsimple Total RNA Kit(天根生物技术,北京,中国)从不同发育阶段的种子中提取总RNA,并使用PrimeScript RT试剂Kit (TaKaRa,大连,中国)进行逆转录。逆转录反应按照供方的方案进行。以花生基因Actin 7作为内对照进行RT-qPCR。反应条件为:94℃30 s, 94℃5 s, 60℃30 s,循环40次。所有反应进行三次重复。通过2−ΔΔCT方法 [63].
可用性数据和材料
与本研究有关的所有数据已包含在稿件的数字/补充文件中。在本文中分析的两种花生重组近交系(Ril 8106和Ril 8107),在河南农业大学(郑州,中国),并从相应的作者提供合理的要求。RNA-SEQ数据集可在序列读取归档(SRA)数据库中(登录ID:PRJNA627334;https://www.ncbi.nlm.nih.gov/sra//term=prjna627334).
缩写
- lncRNAs:
-
长非编码rna
- 瑞来斯:
-
重组自交系
- DAF):
-
几天后开花
- DELS:
-
差异表达的lncrna.
- FPKM:
-
每百万百万片碎片每千比赛的碎片映射了读物
- 走:
-
基因本体论
- Kegg:
-
Kyoto基因和基因组的百科全书
- CPC:
-
编码潜在的计算器
- CNCI:
-
Coding-non-coding指数
参考
- 1.
白杨,戴晓霞,陈敏。植物和动物的RNA调控网络:一个长链非编码RNA的观点。功能基因组学。2015;14(2):91-101。
- 2.
周泽,李米,adeola ac,刘yh,伊尔文DM,谢赫,等。基因组鉴定长期非基因非沉积RNA基因及其与猪归算的潜在关联。基因组Biol Evol。2014; 6(6):1387-92。
- 3.
Dogini DB, Pascoal VD, Avansini SH, Vieira AS, Pereira TC。《rna的新世界》。2014; 37:285-93。
- 4.
王H,NIU QW,Wu HW,Liu J,Ye J,Yu N等。稻米和玉米非编码转录组分析揭示了与农业特征相关的保守LNCRNA的作用。工厂J. 2015; 84:404-16。
- 5.
Cabili Mn,Trapnell C,Goff L,Koziol M,Tazon-Vega B,Regev A等人。人类大的非基础非编码RNA的整合性注释揭示了全局性质和特定子类。基因开发。2011; 25(18):1915-27。
- 6.
马磊,张志强。长链非编码rna的分类研究。RNA杂志。2013;10(6):924 - 33所示。
- 7.
Kornienko AE, Guenzl PM, Barlow DP, Pauler FM。长链非编码RNA转录的基因调控。BMC医学杂志。2013;12。
- 8.
刘杰,王辉,蔡宁华。植物的长非编码RNA转录组。植物学报;2015;13(3):319-28。
- 9.
王建平,徐旭,王建平,等。非编码rna对人HOX位点活性和沉默染色质结构域的功能划分细胞。2007;129(7):1311 - 23所示。
- 10.
剪接促进哺乳动物细胞中mRNA的快速和有效输出。中国科学院院刊2008;105(9):3386-91。
- 11.
春化介导的非编码RNA的表观遗传沉默。科学。2011;331(6013):76 - 9。
- 12.
Swiezewski S,Liu FQ,Magusin A,Dean C.冷诱导的拟义转录物的拟南芥Polycomb靶标。自然。2009; 462(7274):799-802。
- 13.
丁J,Lu Q,欧阳Y,毛鹤,张P,姚杰,等。长度非编码RNA调节杂交水稻的必需组分,调节光周期敏感的雄性不育性。Proc Natl Acad Sci U S A. 2012; 109(7):2654-9。
- 14.
张丽,王敏,李宁,王华,邱鹏,裴磊,等。长链非编码rna参与棉花对黄萎病(一种真菌病害)的抗性。生物技术学报2018;16(6):1172-85。
- 15.
Campalans A, Kondorosi A, Crespi M. Enod40是一个包含开放阅读框的短mRNA,诱导了截断苜蓿核RNA结合蛋白的细胞质定位。植物细胞。2004;16:1047-59。
- 16.
张军,梁胜,段军,王军,陈胜,程志,等。花生种子发育过程中转录组的从头组装和表征,以及基因ssr标记的生成(落花生hypogaeal .)。BMC基因组学。2012;13:90。
- 17.
马旭,张旭,赵凯,李飞,李凯,宁林,等。小RNA和降解组深度测序揭示了microrna在花生种子膨胀中的作用(落花生hypogaeal .)。前植物SCI。2018; 9:349。
- 18.
孔林,张勇,叶志强,刘学强,赵树清,魏林,等。CPC:利用序列特征和支持向量机评估转录本的蛋白编码潜力。核酸学报2007;35(增刊2):W345-9。
- 19.
孙丽,罗华,卜东,赵刚,余凯,张超,等。利用序列的内在组成对蛋白质编码和长非编码转录本进行分类。核酸学报2013;41(17):e116。
- 20。
邓飞,张欣,王伟,袁锐,沈飞。盐胁迫下陆地棉长链非编码rna (lncRNAs)的鉴定。BMC Plant Biol. 2018;18:23。
- 21.
yu w,gius d,onyango p,muldoon-jacobs k,karp j,feinberg ap,等。其反义RNA肿瘤抑制基因P15的表观遗传沉默。自然。2008; 451(7175):202-6。
- 22.
Hanano S,Goto K. Arabidopsis Terminal Flower1参与了通过转录抑制的开花时间和花序发育的调节。植物细胞。2011; 23(9):3172-84。
- 23.
Pauli A, Valen E, Lin MF, Garber M, Vastenhouw NL, Levin JZ,等。斑马鱼胚胎发育过程中表达的长链非编码rna的系统鉴定。基因组研究》2012;22(3):577 - 91。
- 24.
张永昌,陈永强。长链非编码rna:植物发育中的新调控因子。生物化学学报。2013;436(2):111-4。
- 25.
李磊,叶志涛,吴伟,等。玉米长链非编码rna的全基因组发现和特征分析。基因组医学杂志。2014;15 (2):R40。
- 26.
张勇,廖军,李振宇,张军,李强,等。通过全基因组筛选和功能分析,鉴定出大量参与水稻有性生殖的长链非编码rna。基因组医学杂志。2014;15:512。
- 27.
Amor BB,Wirth S,Merchan F,Laporte P,D'Aubenton-Carafa Y,Hirsch J等人。新型长的非蛋白质编码RNA涉及拟南芥分化和应激反应。Genome Res。2009; 19(1):57-69。
- 28.
Chi X,Yang Q,Chen X,Wang J,Pan L,Chen M,等。从花生鉴定和表征microRNAS(落花生hypogaea通过高通量测序。《公共科学图书馆•综合》。2011;6 (11):e27530。
- 29.
高C,王p,赵某,赵c,x j,hou l等。小RNA分析和降低体分析显示了花生胚胎发生和早期荚的MicroRNA调节。BMC基因组学。2017; 18:220。
- 30。
哈佐波洛斯P,方芳,宋志仁。胡萝卜胚胎基因DC8的脱落酸调控。植物杂志。1990;94:690-5。
- 31。
成J,Seeley Ka,Goupil P,Sung Zr。DC8的表达与但不依赖于胚胎发生。植物mol biol。1996; 31(1):127-41。
- 32。
利用RNA-Seq转录组分析揭示草莓果实发育和成熟特性相关基因的差异表达(Fragaria×ananassa.).peerj。2018; 6:E4976。
- 33.
Garceau DC,Batson Mk,Pan IL。水果开发主题的变化:番茄(Tagl1)和其他物种的疯子箱基因的PLE谱系。Planta。2017; 246(2):313-21。
- 34.
El-Kasmi F, Pacher T, Strompen G, Stierhof Y-D, Müller LM, Koncz C, et al.;拟南芥SNARE蛋白SEC22在拟南芥配子体发育和高尔基叠体完整性维护中起重要作用。植物j . 2011; 66(2): 268 - 79。
- 35.
Swaminathan K, Peterson K, Jack t,植物B3超级家族。植物科学进展。2008;13:647-55。
- 36.
King GJ,Chanson Ah,McCallum Ej,Ohme-Takagi M,Byriel K,Hill JM等。拟南芥B3结构域蛋白质vernalization1(VRN1)涉及开发必不可少的方法,具有揭示其DNA结合表面的结构和突变研究。J Biol Chem。2013; 288(5):3198-207。
- 37.
夏涛,李能,李军,等。泛素受体DA1与E3泛素连接酶DA2相互作用,调节拟南芥种子和器官的大小。植物细胞。2013;25:3347-59。
- 38.
李娜,李勇。植物种子大小调控的信号通路。植物生态学报2016;33:23-32。
- 39.
拟南芥中HECT泛素蛋白连接酶(UPL)家族:UPL3在毛状体发育中具有特定的作用。植物j . 2003; 35(6): 729 - 42。
- 40。
宋鹏,陈旭,吴斌,高磊,智华,崔晓霞。大豆花叶病毒P3N-PIPO蛋白互作宿主因子的鉴定。植物生理学报。2016;38:131。
- 41。
谭QK-G,爱尔兰VF。拟南芥锌指 - 同型域基因编码蛋白质,具有在花卉发育期间协调的独特生化特性。植物理性。2006; 140:1095-108。
- 42。
Khatun K, Nath UK, Robin AFK, Park J-I, Lee D-J, Kim M-B,等。锌指同源结构域(ZHD)家族基因的全基因组分析和表达谱分析揭示了锌指家族基因在番茄器官发育和胁迫反应中的可能作用。BMC基因组学。2017;18:695。
- 43。
besshoo - uehara K, Wang DR, Furuta T, Minami A, Nagai K, Gamuyao R., RAE2功能缺失是亚洲栽培水稻无awnlessness所必需的。中国科学(d辑:地球科学)2016;
- 44。
内田,李JS,霍斯特RJ,赖h - h, Kajita R, Kakimoto T,等。内胚层和韧皮部间组织层配体受体通讯对花序结构的调控。中国科学院院刊2012;109(16):6337-42。
- 45.
USAMI T,Horiguchi G,Yano S,Tsukaya H.拟南芥的越来越较小的细胞突变体识别对偏异毛囊控制中的Squamosa启动子结合蛋白质基因的新颖作用。发展。2009; 136:955-64。
- 46.
于松,Galvão VC,张勇,霍雷尔,张涛,郝勇,等。赤霉素通过mir156靶向的SQUAMOSA启动子结合样转录因子调控拟南芥的成花转变。植物细胞。2012;24:3320-32。
- 47.
Wang Z - Z, Wang Y - Y, Wang Y - Y, han oufa A. SQUAMOSA PROMOTER BINDING PROTEIN-LIKE 2通过激活非对称叶片2调控拟南芥花器官发育和植物育性。植物学报。2016;92(6):661-74。
- 48.
基于SCFTIR1/ afb的生长素感知机制及其在植物生长发育中的作用。植物细胞。2015;27:9-19。
- 49.
Shimizu-Sato S,Tanaka M,Mori H.Auxin-cytokinin射击分支控制中的相互作用。植物mol biol。2009; 69(4):429-35。
- 50。
细胞分裂素的代谢与长距离转运。acta botanica yunnanica(云南植物研究),2010;52(1):53-60。
- 51。
徐志强,李志强,李志强,等。细胞分裂素氧化酶调节稻米产量。科学。2005;309(5735):741 - 5。
- 52。
蔡玲,张玲,傅强,徐志强。生物燃料植物细胞分裂素代谢基因IPTs、CYP735A和CKXs的鉴定与表达分析麻风树.Peer J. 2018; 6:E4812。
- 53。
黄志强,王志强,王志强,等。拟南芥中类黄酮生物合成基因的表达及其在种子发育中的应用[j]。BMC Res Notes. 2010;3:255。
- 54。
Martin M. Cutadapt从高通量测序读取中移除适配器序列。EMBnet杂志。2011;17(1):10。
- 55。
Trapnell C, Pachter L, Salzberg SL. TopHat:用RNA-Seq发现剪接连接。生物信息学,2009,25(9):1105 - 11所示。
- 56。
Pertea M, Pertea GM, Antonescu CM, Chang TC, Mendell JT, Salzberg SL. StringTie能够从RNA-seq读取改进的转录组重建。生物科技Nat》。2015;33(3):290 - 5。
- 57.
Frazee AC, Pertea G, Jaffe AE, Langmead B, Salzberg SL, Leek JT。Ballgown在转录组组装和表达分析之间架起桥梁。生物科技Nat》。2015;33(3):243 - 6。
- 58.
KAPUSTA A,Feschotte C.长的非数性RNA曲目的挥发性演变:机制和生物学意义。趋势类型。2014; 30:439-52。
- 59.
特拉普奈尔C, Williams BA, Pertea G, Mortazavi A, Kwan G, Van Baren MJ,等。RNA-Seq转录本组装和定量揭示了细胞分化过程中未注释的转录本和亚型转换。生物科技Nat》。2010;28(5):511 - 5。
- 60。
Mercer Tr,Dening Me,Mattick JS。长期非编码RNA:洞察功能。NAT Rev Genet。2009; 10(3):155-9。
- 61。
廖强,刘灿,袁学勇,康少林,苗瑞日,肖华,等。编码-非编码基因共表达网络中长链非编码RNA功能的大规模预测。核酸学报2011;39(9):3864-78。
- 62。
Blast2GO:功能基因组学研究中用于注释、可视化和分析的通用工具。生物信息学。2005;21(18):3674 - 6。
- 63。
Livak KJ,Schmittgen TD。使用实时定量PCR和2分析相对基因表达数据−ΔΔCT方法。方法。2001; 25:402-8。
确认
我们感谢匿名审查员,了解稿件的有用建议。
资金
基金资助:国家自然科学基金重点项目-河南联合基金资助项目(No. 20141201);U1704232),河南省重点科技项目(161100111000;S2012-05-G03),河南省创新科技队伍建设项目(2018JR0001)。这些资助机构在研究的设计、数据的收集、分析和解释以及手稿的编写方面没有作用。
作者信息
隶属关系
贡献
Dy设计并构思了这项研究。XM和GH写了稿件。XM,XZ和ST分析了数据。LN,KL,KZ和ZL进行了QPCR实验。ZX,GH,St和Dy编辑了手稿。作者阅读并批准了手稿。
通讯作者
道德声明
伦理批准和同意参与
本研究中的植物材料的集合符合机构,国家或国际指南。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1
.的意思是样品质量。
附加文件2:表S2
.RNA-Seq的统计数据读取四个样本。
附加文件3:表S3
.所有的lncrna都在花生中。
附加文件4:表S4
.已知lncrna与新lncrna的差异表达四项比较。(S4-1) 35 DAF位点RIL8106的lncrna差异表达(T1 vs C1)。(S4-2) 35 DAF位点RIL8107的lncrna差异表达(T2 vs C2)。(S4-3) C2与C1中lncrna的差异表达。(S4-4) lncrna在T2和T1中的差异表达。
附加文件5:表S5
.差异表达LNCRNA的靶基因预测。(S5-1)T1 VS C1中差异表达的LNCRNA的靶基因预测。(S5-2)T2 VS C2中差异表达的LNCRNA的靶基因预测。
附加文件6:表S6
.本研究的所有引物。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
马,X.,张,X.,Traore,S.M。等等。花生种子发育过程中长链非编码rna (lncrna)的全基因组鉴定与分析(落花生hypogaeal .)。BMC植物杂志20,192(2020)。https://doi.org/10.1186/s12870-020-02405-4
已收到:
接受:
发表:
关键词
- 花生
- lncRNA
- 种子发展
- RNA-SEQ.
- 转录调控