抽象的
背景
奥贝辛那雷斯三次来到全罗马纳米碱,提供了一个理想的系统,用于研究植物中全洛杉矶的生活方式的演变。由于Holoparasitic植物失去了光合作用的能力,可以通过塑性基因组降解和协调变化来揭示核解虫主义的演变。在奥洛库尼亚群的三种植物中,只有CLADE VI没有用于全洛杉矶植物的可用体积基因组序列。在这项研究中,我们测序了塑料和转录组Aeginetia籼,奥比丘西的汉族志综合体植物,研究其塑料进化和核基因组的相应变化作为光合作用丧失的响应。
结果
质体系的答:籼稻减少到86,212bp的大小,几乎所有与光合作用的基因都丢失了。将损伤基因的大规模碎片转移到线粒体和/或核基因组中。这些片段无法在其转录组中检测到,这表明它们是非功能性的。塑料中的大多数蛋白质编码基因显示出净化选择的松弛信号。塑性和转录组分析表明光合作用途径完全丧失,并且卟啉和叶绿素代谢途径部分保留,尽管不可能是叶绿素合成的。
结论
我们的研究表明,光合作用相关功能的丧失答:籼稻在细胞核和质体基因组中。丢失的质体基因被转移到它的核和/或线粒体基因组中,并以非常小的片段存在,没有表达,因此是无功能的。的Aeginetia籼塑料还提供了对奥古氏菌核查群重复演化的比较研究的资源。
背景
叶绿体(质体)是植物进行光合作用的细胞器,在大多数开花植物中,叶绿体基因组的结构和基因含量高度保守[1].典型的叶绿体DNA (cpDNA)是圆形的,长度主要在110 ~ 160 kb之间[2],它包含由大单拷贝区域(LSC)和小单拷贝区域(SSC)分隔的两个反相重复(IR)序列[3.].全寄生植物由于丧失了光合能力,为质体基因组进化研究提供了一个良好的系统。它们通常表现为质体基因组的减少,包括基因组大小和基因含量的减少。Orobanchaceae包含了从自养植物到半寄生植物和全寄生植物的全营养谱,特别适合研究叶绿体(质体)基因组进化。在Orobanchaceae中,全寄生虫存在于6个支持良好的分支中的3个,即分支III (Orobancheae, ~ 180种全寄生虫)、分支V (Rhinantheae, ~ 7种全寄生虫)和分支VI (Buchnereae, ~ 70种)[4.那5.那6.].到目前为止,Orobanchaceae的Holoparasites的塑性基因组序列主要来自Clade III [7.那8.那9.那10.那11.那12.].在进化枝V中,只有一种全寄生虫的质体基因组,Lathraea squamaria,最近已进行了测序[12.].虽然Clade VI包括来自四个Holoparasitic属的〜70种(Hyobanche那哈韦塔那Aeginetia和Christisonia),已经表征了来自该思路的全洛杉矶植物的体积基因组序列。
Orobanchaceae分支III和分支V中全寄生虫的质体基因组在基因组大小和基因含量上存在显著差异。进化枝III全寄生虫质体基因组大小为45,673 (Conopholis美国)至120,840 bp (Orobanche californica)[9.].然而,质体基因组的大小Lathraea squamaria来自CLADE V是150,504 BP [12.],比思克III中的那些大得多。塑性基因组中的完整基因的数量Conopholis美国和Orobanche物种范围从21到34 [9.],几乎所有与光合作用有关的基因(宠物那psa那公安局,rbcl.)丢失或成为伪原。而在塑性基因组中Lathraea squamaria,有46个完整基因,包括许多与光合作用有关的基因(例如psa那公安局和宠物).这可能是由于进化枝V的全寄生谱系比进化枝III的全寄生谱系更年轻[12.].
除了塑性降解之外,Holoparasitic植物的核基因组还预期作为光合作用丧失的响应而发展,因为塑体基因组中的光合作用与核基因组中的许多基因有关的基因[7.那13.].通过转录组测序可以揭示核基因的表达变化。例如,在一些寄生植物中研究了光合作用和叶绿素合成途径中基因的表达[7.那14.那15.].
Aeginetia是Orobanchaceae的一个小型全寄生属,约有四种分布于亚洲南部和东南部[16.].根据Orobanchaceae的系统发育分析,Aeginetia, 随着Hyobanche那哈韦塔和Christisonia,在CLADE VI中形成单胞嘧啶Holoparasitic谱系[4.那5.].Aeginetia籼是这个属中分布最广的物种[17.].它通常寄生在像泥矿植物的根源上miscanthus和糖果[18.].在最近的一项研究中答:籼稻已用于检测来自Fabaceae和Poaceae物种的水平转移的基因[19.].到目前为止,在该全洛杉矶植物中尚未研究体液基因组序列和光合作用相关途径的降解。
在这项研究中,我们组装了体积基因组答:籼稻使用基因组撇渣产生的illumina短读。我们还测序了来自多种组织的转录om,以检查光合作用中涉及的基因的表达变化。此外,我们研究了丢失的塑性基因的进化序列答:籼稻.本研究的结果将有助于我们理解质体和核基因组的协调进化,也将有助于对Orobanchaceae全寄生的趋同进化进行比较分析。
结果
严重收缩答:籼稻损伤基因的体积基因组和进化序列
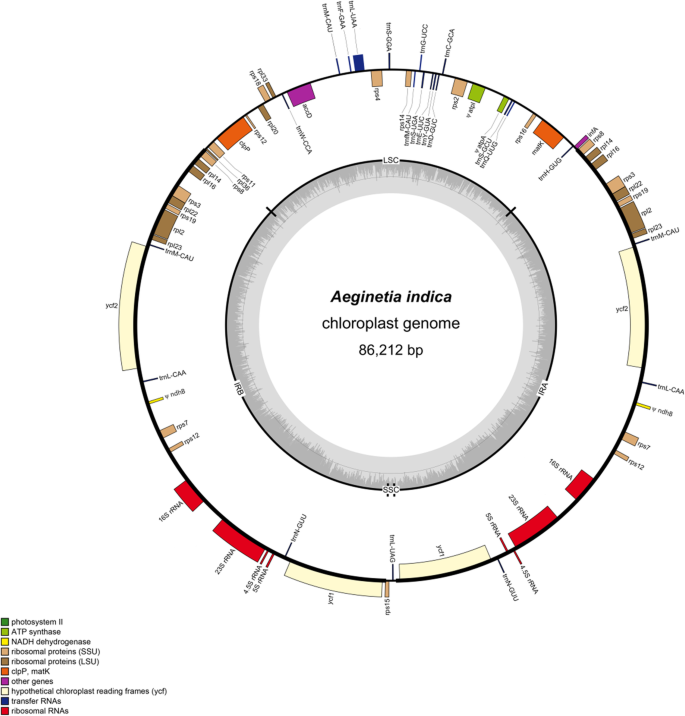
完全塑性基因组答:籼稻是86,212bp的长度,相对于大多数Angiospers的大小高度降低。它具有典型的四重结构,具有22,301bp的LSC区域,SSC区域的529bp,以及31,691bp的每一个IR区域(图。1).质体基因组AT含量为65.64%。基于DOGMA和GeSeq注释,获得答:籼稻包含48个推测的完整基因和3个假基因。这些完整的基因包含18个tRNA基因,4个rRNA基因,8个rRNA基因rpl.基因,12rps.基因和6个其他基因,即,YCF1.那YCF2.那accD那mat那infA和clpP(表1).这三个伪基因是ψatpa.,ψATPI.和ψndhB.ψatpa.和ψATPI.LSC区域的基因答:籼稻由于在88处被截断,质体变成了假基因TH.密码子和32的过早停止密码子nd密码子,分别。ψndhB由于53位的内部终止密码子,IR区域的基因变成了假基因rd.密码子。
胞浆内的SSC区答:籼稻显示大小和两个基因的严重减少,rpl15和trnl-uag.,在该区域发现(图。1).两个红外区域对LSC和SSC区域进行了扩展。在自同学的叶绿体基因组中Lindenbergia philippensis和其他自养植物一样,完好无损YCF1.基因通常跨越IR和SSC区域rps8那rpl14那rpl16那rps3那rpl22和RPS19基因位于LSC区域。然而,在答:籼稻塑料,完好无损YCF1.每个IR区域的基因rps8那rpl14那rpl16那rps3那rpl22和RPS19基因都转移到IR区域。
质体中的基因含量答:籼稻,四种全洛杉矶包括Cistanche Deserticola.那Orobanche austrohispanica和Epifagus virginiana从思工三世,和Lathraea squamaria从Clade V和自养相对l . philippensis比较(表S1).相比之下,l . philippensis,就会有大量的基因丢失答:籼稻质体基因组。十个NDH.(ndhA那ndhC那ndhD那ndhE那ndhF那ndhG那ndhH那ndhI那ndhJ和ndhK)基因丢失,ndhB基因变成假基因,它们编码nadh -脱氢酶复合物的亚基。所有五个psa(PSAA,PSAB,PSAC那psaI和psaJ)和15公安局(psbA那psbB那psbC那psbD那psbE那psbF那psbH那psbI那psbJ那psbK那psbL那psbM那psbN那psbT和psbZ)光系统I和光系统II的相关基因丢失。同时,所有六个宠物(善待动物组织那petB那皮特那petF那petH和petI)基因,其在光合电子传输中编码具有功能的细胞色素B6 / F复杂亚基。另外,四个三磷酸腺苷(ATPB.那atpe.那ATPF.和沥青)编码F型ATP酶亚基的基因,编码DNA依赖性RNA聚合酶的四种基因(rpoA那rpoB那rpoC1和rpoC2)和编码包膜膜蛋白的基因(CEMA.),Rubisco的大亚基(rbcl.)、血液附着因子(ccsA)和光系统组装因子(YCF3.和YCF4.)也迷失了。在两个全洛洛西菌物种的塑料中发现了类似的基因损失Orobanche austrohispanica和Epifagus virginiana从第三进化枝。尽管这些基因中的大多数并没有在质体中丢失Lathraea squamaria进化枝V和Cistanche Deserticola.从进化枝III开始,它们中的许多变成了伪基因S1).
来研究在答:籼稻,我们通过提取与64种植物参考质体匹配的基因组略读和其他分析确定了它们的基因组位置(见方法)。从提取的reads中共组装了339个长度≥150 bp的contigs,其中76个被注释为缺失质体基因片段答:籼稻质体基因组。这些76个片段的长度为150至3086bp,代表27个丢失的塑性基因,包括四个三磷酸腺苷基因(atpa.那ATPB.那atpe.和ATPF.), 7NDH.基因(ndhA那ndhB那ndhD那ndhE那ndhF那ndhH和ndhJ),四个psa基因(psaA那psaB那psaC和psaI), 6公安局基因(psbA那psbB那psbC那psbD那psbE和psbJ),三个RPO.基因(rpoB那rpoC1和rpoC2),每个基因petB那rbcl.和YCF3.(表S2).此外,我们发现15个代表九个基因的片段答:籼稻体液基因组也转移到线粒体和/或核基因组中,并存在于小片段中。在这些基因中,七个(atpa.那ndhA那ndhB那ndhF那petB那rpoB和rpl23)转移到线粒体和核基因组,11 (ndhD那ndhE那ndhH那ndhJ那rpl14那rpl16那rpl2那RPS12.那RPS14那rps3和rps4)仅转移到线粒体基因组,18(ATPB.那atpe.那ATPF.那psaA那psaB那psaC那psaI那psbA那psbB那psbC那psbD那psbE那psbJ那rbcl.那rpoC1那rpoC2那YCF3.和YCF2.)基于其测序深度仅转移到核基因组。在多种组织的转录组中未检测到所有这些片段,表明它们是非功能性的。
质体基因组的多重结构重排答:籼稻相对于它的自养亲戚
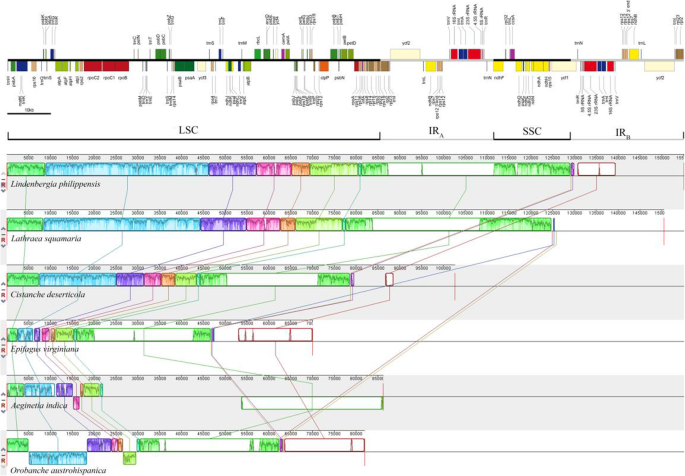
用紫红色2.4.0,血液塑料的序列对准答:籼稻(枝VI)和上述五种Orobanchaceae物种如图所示。2.我们为这6个物种鉴定了9个局部共线性块(LCBs)答:籼稻塑性基因组相对于叶绿体基因组经过两种倒置l . philippensis.其中一个反演位于LSC区域,包含一个完整的accD基因,另一个包含完整的SSC区域和IRB.地区。与之相比l . philippensis叶绿体基因组,塑料中没有倒置Lathraea squamaria那Cistanche Deserticola.和Epifagus virginiana,虽然有两个不同的反向Orobanche austrohispanica(图。2).
轻松净化选择答:籼稻质体基因
Orobanchaceae的7个物种共有20个蛋白编码基因,其中10个为同源基因rps.基因,7rpl.基因和accD那infA和mat基因用于系统发育分析。强烈支持最大似然树,所有分支的引导值为100(图S1).三str物种聚集成一个分支布什纳亚亚洲是他们的姐妹。Aeginetia籼是姐妹,思克是前四种物种。非同义(DN)/同义(DS)替换率比(Ω)可以被认为是选择压力的指示。将双比率模型(M2)与单比率模型(M0)进行比较。所有基因的ω值但是rpl20和RPS18在寄生植物分支大于非嗜磷植物分支(表S3),似然比检验显示M2在9个基因上显著优于M0。accD那infA那rpl22那RPS11那RPS14那RPS19那rps2那rps3和rps7,表明这些基因在寄生植物中稀释净化选择。使用三比例分支模型(M3),我们发现偏瘫物种比18个基因中的13个基因的13个ω细胞钠物种更高或更高Ω(剩余的两个基因的ω值),而全洛尼酸盐物种略高于Ω只有五个基因的偏瘫物种(表S3).这表明蛋白质编码基因保留在质体中答:籼稻仍然发挥着重要的功能作用,而不是经历比半寄生物种更宽松的选择压力。
光合作用途径的降解答:籼稻通过转录组分析揭示
我们分别获得21.05,19.04,18.34和18.02 GB清洁四种组织,即花,萼片,水果和茎。通过来自四种组织的读取数据的De Novo组装,我们获得了总共205,380的转录物,其中153,986℃被提取为unigenes。这些unigenes的平均长度和N50分别为623.18和880bp。通过Transdecoder的所有Unigenes预测的47,480个ORF(开放阅读框架),其中42,007个可以在瑞士-PLIC数据库中注释,在42,007瑞士 - Prot注释中,8466可以分配给131 kyoto基因组和基因组(Kegg途径。
为了验证转录组测序得到的基因表达的可靠性,我们还用qRT-PCR检测了10个基因的表达。两种方法获得的基因表达水平具有较高的相关性(Pearson相关系数R2 = 0.71; FigureS2),表明从转录组测序获得的基因表达是可靠的。
KEGG途径数据库中的光合途径(ko00195)包含63个基因(30个质体基因和33个核基因)。在答:籼稻塑料,编码参与照相I和II,细胞色素的蛋白质的基因B6F.复杂时,光合作用的电子传递就完全失去了。仅有的两个f型atp酶相关基因(atpa.和ATPI.)是假基因。通过转录组分析,只有14个在光合作用途径富集的核单基因有表达(表1)S4).14个基因包括一种编码psii 6.1kda蛋白的一个基因,编码蛋白质涉及光合电子传输的蛋白质和编码f型Atpase的组分(图S3).未检测到该途径中其他基因的表达,表明这些基因丢失或非表现。来自塑料和转录组分析的结果表明光合作用途径答:籼稻完全失去了。
卟啉和叶绿素代谢途径(KO00860)在植物中复杂化。卟啉是血红素和叶绿素的中间体,叶绿素生物合成需要血红素[20.].在从谷氨酸到原生卟啉IX的途径中,八个基因的表达(丙烯酸-那HemB那HemC那HemD那亚铁血红素那HemF那HemL和铁窗栏栅在转录组中观察到答:籼稻(数字S4).但是,由于没有表达氯化乙烯基氯化物A 8-乙烯基还原酶[EC:1.3.1.75],其催化二乙烯基硫代氯化物与蛋白氯化物[21.,叶绿素合成途径似乎结束于二乙烯基原叶绿素的产生答:籼稻(数字S4).显然,叶绿素合成途径在后期不完整,叶绿素不能合成答:籼稻.
讨论
的特点答:籼稻质体基因组
在Orobanchaceae现有质体基因组中,Orobanchaceae的质体基因组最多答:籼稻比那些小得多str物种(S. Aspera.那S. forbesii.和S. Hermonthica),但与Orobanchaceae分支VI相似Orobanche物种,如O. Pancicii(88525个基点)O. Crenata.(87,529 BP)来自Clade III [10.].然而,它们的质体基因组结构有很大的不同:LSC (22,301 bp)和SSC (529bp)区域的大小答:籼稻比那些小得多O. Pancicii(42,763和7608 bp)O. Crenata.(43,054和7925 bp),而IR的大小(31,691 bp)远大于O. Pancicii(19077个基点)O. Crenata.(18,275 bp)。塑料中的IR和SSC区域答:籼稻分别经历了膨胀和收缩。这些现象也在四种半寄生虫(str),源自Orobanchaceae同一分支VI [1].叶绿体基因组中的IR区域通常高度保守尺寸和基因含量,并且在稳定叶绿体基因组结构中起着重要作用[22.].在本研究中,IR地区的扩张发生在答:籼稻表明,塑料的戏剧性变化伴随着光合作用的丧失。此外,我们的研究表明,奥比丘西的全洛杉矶植物和血液化基因组的不同布置,例如,与l . philippensis叶绿体基因组,塑料中没有倒置Lathraea squamaria那Cistanche Deserticola.和Epifagus virginiana,而每一个都有两个截然不同的倒置答:籼稻和Orobanche austrohispanica塑料。
质体中的48个基因答:籼稻序列长度是否与自养的相似Lindenbergia philippensis.蛋白质编码基因答:籼稻在塑性函数中发挥基本作用,包括大和小核糖体蛋白质亚基(rpl.和rps.基因),乙酰-CoA羧化酶β亚基参与脂质生物合成(accD),内含子剪接(mat),翻译启动因子(infA),ATP依赖性蛋白酶亚基P(clpP)和蛋白质进口和营业额(YCF1.)[10.那23.].功能的功能YCF2.具有保守的开放阅读框架的基因仍然未知。四个基因,atpa.那clpP那rpl2和rpl23,含有内含子,这与内含子的保留一致mat作为内含子拼接的功能。
光合作用相关基因的丧失是全洛博马西植物的常见现象,例如Aphyllon和Orobanche[10.那11.].在其他全寄生植物中也观察到管家基因的丢失,例如,质体基因组Balanophora laxiflora.只有15505 bp大小,大多数基因丢失[24.], 和大花草属lagascae甚至失去了整个质体基因组[25.].一些管理基因已经转移到核基因组,它们的蛋白质可以移回质体执行它们的功能[26.].在这项研究中,27种失失的塑性基因答:籼稻然而,转移到核和/或线粒体基因组,然而,可以从我们的转录组数据中检测到这些片段的表达,表明它们是非功能的。
以前的研究提出了寄生虫中的塑性进化模型和基因损失的顺序[27.那28.那29.].这些模型中的五个阶段包括“光合”,“劣化I”,“静止”,“降解II”和“不存在”阶段。基因损失的顺序开始NDH.基因,然后是psa /公安局基因和RPO.那么三磷酸腺苷的基因,rbcl.基因,非必要的内政基因和其他代谢基因如accD那clpP那YCF1.和YCF2.,最后是剩下的管家基因,比如rpl.和rps.基因。根据他们的模型,塑料答:籼稻处于“静止”阶段。
光合作用途径的丧失答:籼稻
Aeginetia籼无光合作用,通过与寄主连接获得全部碳[19.].在本研究中,光合作用途径的丧失答:籼稻基于其塑料中的光合作因的丧失证实了,并且在其转录组中没有发现光合作用途径中许多基因的表达。叶绿素是在光合作用中吸收光能的主要颜料。叶绿素合成是不可能的答:籼稻因为在卟啉和叶绿素代谢途径的后期,一些关键基因未被检测到并表达。光下原叶绿素的积累可能会增加氧化风险[30.],所以降解叶绿素合成途径答:籼稻可能有利于它的生存和进化。相反,在全寄生植物中发现了完整的叶绿素合成途径Phelipanche aegyptiaca,表明叶绿素合成途径的表达除光合作用外还有其他功能(如代谢信号,前期研究已证实mg -原卟啉IX是一种候选信号分子)[31.].
结论
塑性基因组Aeginetia籼是来自奥比丘西的vi的全洛洛西能植物,在本研究中组装了De Novo。其塑性基因组显示尺寸的减小,伴随着几乎所有光合作用相关基因的损失或假置化和一些结构重排。将丢失的塑性基因转移到其核和/或线粒体基因组中,并且大多数存在于具有表达的非常小的片段中,因此是非功能性的。来自多种组织的转录组分析表明光合作用途径答:籼稻尽管叶绿素合成是不可能的,但是部分保留卟啉和叶绿素代谢途径的卟啉和叶绿素代谢途径。我们的结果表明,在全洛博马西植物的塑性和核基因组中的光合作用相关功能的协调丧失。塑料和转录组数据答:籼稻本研究为今后的研究提供了遗传资源,并将为Orobanchaceae全寄生趋同进化的比较分析提供参考.
方法
植物材料,DNA分离和illumina测序
所有的组织Aeginetia籼在本研究中使用的是从中国广东省雪山森林公园收集。本植物是由仁建州博士鉴定的基于其植物群本地稻草(Sinicae)中描述的形态特征[16.].代金券标本(周20190721)保存于中山大学植物标本馆。为研究目的而采集该物种的组织样本,并不需要特定的许可证。这些植物被取样并立即在液氮中冷冻,然后保存在−80°C以便进一步分析。使用HiPure Plant DNA Mini Kit (Magen公司,广州,中国)按照制造商的说明从花茎中分离出总DNA。分别用1%琼脂糖凝胶电泳和Qubit 3.0荧光仪(美国Invitrogen公司)检测DNA的质量和数量。在Illumina Hiseq X Ten平台(IGE Biotechnology Ltd., Guangzhou, China)上进行了150 bp配对端reads的Shotgun基因组测序。测序数据保存在NCBI Sequence Read Archive中,登录号为srr978563。
质体基因组的组装、注释和比对
塑性基因组答:籼稻采用novo成形术从Illumina测序数据重新组装[32.],参数为插入尺寸(300 bp)、K-mer(37)和覆盖率切断(1500)。的RPS16基因序列(EU572719.1)答:籼稻被用作种子结合DOGMA程序进行质体基因组注释[33.和GeSeq在MPI-MP CHLOROBOX (https://chlorobox.mpimp-golm.mpg.de/)默认参数。含有一个或多个过早止血密码子或框架突变的基因被认为是潜在的假生素。带注释的塑性基因组序列答:籼稻在登录号MN529629下存放在Genbank。用OGDRAW绘制塑性基因组的圆形图[34.].为了比较Orobanchaceae 3个分支全寄生植物的基因丢失和潜在的基因组重排,我们下载了4个全寄生植物的质体基因组序列,分别是Cistanche Deserticola.(NC021111.1),Orobanche austrohispanica(NC031441.1),Epifagus virginiana(NC001568.1)从Clade III和Lathraea squamaria(NC027838.1)从CLADE V和一个自养相对Lindenbergia philippensis(NC022859.1) from GenBank of Orobanchaceae分枝I。使用progressiveMauve对这些序列进行比对[35.],使用默认参数。
鉴定缺失质体基因的基因组位置答:籼稻
丢失的塑性基因答:籼稻可能转移到它的核和/或线粒体基因组。为了确定它们的基因组位置,共收集了来自4个目21个科的64个质体S5)从Genbank下载并作为参考诱饵序列。使用Magic-Blast V1.5.0将干净的配对端读数与所有参考诱饵序列对齐[36.],使用默认参数。所有的质体和质体样的读取答:籼稻使用Samtools v1.10提取[37.]使用命令:samtools视图-b -s -f 4.所有提取的读取都与samtools v1.10合并,然后用天鹅绒v1.2.10组装de novo [38.通过设置插入长度350,插入长度标准差100和最小连续长度150。这些contig和答:籼稻利用Magic-BLAST v1.5.0获得质体。利用Seqtk v1.2-r94 (https://github.com/lh3/seqtk),使用默认参数。所有的蛋白质序列Lindenbergia philippensis使用叶绿体基因组来构建蛋白质数据库,并且使用BLASTX v2.5.0 +将提取的折叠与该数据库进行注释,[39.] E值≤10- 5.从提取的褶皱中搜索丢失的塑性基因。作为塑体基因或具有侧翼线粒体或核序列的基因片段被注释的Contig用于确定它们的基因组位置(线粒体或核)。然而,对于大多数Contig,序列太短,无法确定其基因组位置,因此我们通过BWA-MEM计算了这些Contig的平均深度[40.绘制图谱以推断缺失质体基因的基因组位置。这是基于线粒体DNA的拷贝数通常比核DNA高一到两个数量级。注意,对于那些与质体基因有碰撞的contigs答:籼稻,我们计算了无击中部件的深度,以避免塑体基因的映射。在组装的转录om(如下所述)中搜索这些片段的序列以评估它们是否表达。
质体基因放松纯化筛选标记的检测答:籼稻
从GenBank中下载了6种Orobanchaceae植物的质体基因组,其中包括4种半寄生植物Striga Aspera.(MF780872.1),Striga forbesii(MF780873.1),Striga hermonthica(mf780874.1),和布什纳亚亚洲(MF780871.1)从湿度VI和两个自养植物Lindenbergia philippensis(nc_022859.1)和地黄(NC_034308.1)。六种系统发育和答:籼稻使用20个蛋白编码基因(7rpl.基因,10rps.的基因,accD那infA和mat)共享在质体基因组中。使用MAFFT进行序列比对后[41.]使用RaxML中的最大似然算法重建系统发生,[42.]有1000个引导复制,带地黄作为一个小组。最大可能性树显示为Figtree V1.4.3(http://tree.bio.ed.ac.uk/software/figtree/).
然后以系统发生树作为输入树,检测质体基因的松弛纯化选择答:籼稻.在七种物种中共享的每个基因的编码区域由Clustalw(密码子)对齐,默认设置在Mega-x版本10.5中[43.].使用密码子分析(codeml)估算了20个共享基因的非同义(dN) /同义(dS)替代率(ω) [44.].使用不同的分支模型来分析这些物种中的选择性压力。首先执行空单比率模型(M0,它假设所有分支的所有分支具有一个ω),然后是双比率模型(M2)的可能性,具有前景ω1,用于寄生物种以及用于自营养物种的背景ω2,与M0的比较。此外,将具有三个比率(M3)的分支模型分别为Holoparasitic,HealaSitic和forotootcicics的三种不同ω值分别与M2进行比较。M0 VS M2和M2 VS M3的似然比测试用Chi-Square分布进行,自由度等于模型参数数量的差异,以评估数据的适合替代分支模型。
转录组测序
从鲜花、萼片、果实、茎等组织中分离得到总RNA答:籼稻, 分别。使用1%琼脂糖凝胶电泳和Qubit分光光度计测定RNA的质量和浓度。用寡核苷酸DT纯化这四种组织的MRNA,然后用来构建cDNA文库。使用PE150(配对端150bp)战略,对Illumina Hiseq2000平台(IgE Biotechnology Ltd.)进行测序四个cDNA文库。花,萼片,水果和干组织的原始RNA测序数据答:籼稻在NCBI Sequence Read Archive中,登录号分别为SRR9959046、SRR9959049、SRR9959048和SRR9959047。
转录组分析
通过去除适配器和低质量读取来过滤原始测序数据。通过通过Trinity v2.8.4使用来自四个组织的清洁读数组装转录物[45.],然后使用Perl脚本get_longest_isoform_seq_per_trine_gene.pl从这些转录物中提取unigenes .pl中。使用RSEM确定所有ungenes的表达水平(每百万百万,TPM)[46.].所有unigenes在NCBI非冗余数据库(NR,http://www.ncbi.nlm.nih.gov/genbank/)使用钻石(http://ab.inf.uni-tuebingen.de/software/diamond/).预测开放阅读框架(ORF)并使用转码器工具获得推定的蛋白质序列(http://transdecoder.sourceforge.net/).推定的蛋白质序列以瑞士 - prot(http://www.uniprot.org/)和京都基因和基因组百科全书(KEGG) [47.通路(http://www.genome.jp/kegg/pathway.html)使用Blastx和Ghostkoala的e值<1e-5标准的数据库(https://www.kegg.jp/ghostkoala/), 分别。在Kegg途径内,我们仅关注光合途径(KO00195)和卟啉和叶绿素代谢途径(KO00860),并记录了这两种途径中基因的表达。
实时荧光定量PCR实验
为了检验转录组测序获得的基因表达的可靠性,使用CFX Manager (Bio- rad,美国)和TB Green®Premix Ex Taq™II (Tli RNaseH Plus) (Takara Bio Inc.,日本)对10个基因进行实时定量PCR (qPCR)。qPCR的起始步骤为95℃2 min, 95℃5 s, 60℃30 s,共40个循环,最后进行熔化曲线分析。每个反应分别有3个生物重复和3个技术重复。的肌动蛋白基因作为内部控制。用于qPCR的引物列于表S6.每个基因的表达量用2-ΔΔct方法(48.].
数据和材料的可用性
猎枪基因组测序数据Aeginetia籼在登录号为srr978563的NCBI序列读取档案中保存;注释的质体基因组序列答:籼稻以登录号MN529629存入GenBank;以及花、萼片、果实和茎组织的原始RNA测序数据答:籼稻在NCBI Sequence Read Archive中,登录号分别为SRR9959046、SRR9959049、SRR9959048和SRR9959047。支持本研究结果的数据集包括在本手稿及其附加文件中。
缩写
- cpDNA:
-
叶绿体DNA
- 红外光谱:
-
反向重复
- LSC:
-
大单副本区域
- SSC:
-
小单副本区域
- 教条:
-
双细胞器基因组注释器
- LCB:
-
本地共线的块
- ORF:
-
开放阅读框架
- KEGG:
-
Kyoto基因和基因组的百科全书
- PSII:
-
光系统II
参考文献
- 1。
Frailey DC,Chaluvadi SR,Vaughn Jn,Coodney CG,Bennetzen JL。羚羊族五次血管基岩体积的基因丧失和基因组重排。BMC植物BIOL。2018; 18(1):30。
- 2。
陈志强,王志强,王志强,等。陆地植物质体染色体的进化:基因含量、基因顺序、基因功能。植物学报。2011;76(3-5):273-97。
- 3.
肖J,李奇EB, Schilling EE,小RL。被子植物系统发育研究中叶绿体全基因组序列选择非编码区域的比较:乌龟和野兔3。植物学报。2007;94(3):275-88。
- 4.
Bennett Jr,Mathews S.寄生植物家庭的系统发育奥比丘西植物从Phytochrome a推断。我是J机器人。2006; 93(7):1039-51。
- 5。
McNEAL JR,Bennett Jr,Wolfe Ad,Mathews S.Malobanceae的Mathews S。我是J机器人。2013; 100(5):971-83。
- 6。
付伟,张宁,张伟,杨建军,王永强,宋志刚,王永强,杨建军,王永强。植物全寄生性多起源假说的检验:来自最后两个完全寄生性属的系统发育证据。Gleadovia和phacellanthus.前植物SCI。2017; 8:1380。
- 7。
Wolfe KH, modern CW, Palmer JD。非光合寄生植物最小质体基因组的功能和进化。中国科学(d辑:地球科学)1992;89(22):10648-52。
- 8。
李旭,张涛涛,乔强,任忠,赵军,Yonezawa T, Hasegawa M, Crabbe MJC,李俊,钟勇。全寄生植物叶绿体基因组的全序列分析Cistanche Deserticola.(Orobanchaceae)揭示了来自其宿主Haloxylon Ammodendron(Chenopodiaceae)的基因损失和水平基因转移。Plos一个。2013; 8(3):3。
- 9。
张亚平,张亚平,张亚平,张亚平,张志强,张志强,张志强。油菜科植物功能基因组和物理基因组减少机制的研究进展。植物细胞。2013;25(10):3711 - 25所示。
- 10。
陈志强,陈志强,陈志强。Orobanchaceae的叶绿体基因组还原过程中大量的细胞内基因转移。新植醇。2016;210(2):680 - 93。
- 11.
Schneider AC,Chun H,Tefanovic S,Baldwin BG。色调塑性塑性塑性塑性植物植物属植物属植物属植物属植物属植物属植物的水平基因转移。P ROY SOC LOND B:BIO。2018; 285(1887):20181535。
- 12.
Samigullin TH, Logacheva MD, Penin AA, Vallejo-Roman CM。最近的全寄生虫鳞状板条虫的完整质体基因组揭示了orobanchaceae中最早期的质体还原阶段。《公共科学图书馆•综合》。2016;11(3):3。
- 13。
陈志强,陈志强,陈志强。核基因参与叶绿体基因表达的研究进展。Biochimie。2000;82(6):559 - 72。
- 14。
吴淑梅,李新文,马- isa MN, Aizat-Juhari MA, Adam JH, Mohamed R, Wan KL, firdaus - rah M.核编码质体靶向蛋白的比较分析大花草属cantleyi对抗光合和非光合代表揭示了具有潜在发散功能的正交系统。SCI Rep-UK。2018; 8(1):17258。
- 15.
Ravin NV, Gruzdev EV, Beletsky AV, Mazur AM, Prokhortchouk EB, Filyushin MA, Kochieva EZ, Kadnikov VV, Mardanov AV, Skryabin KG。非光合真菌异养真二体的质体和核基因组中光合途径的丧失Monotropa hypopitys.BMC Plant Biol. 2016; 16:38。
- 16。
张z列当科。见:王伟,编辑。中华民国植物群。北京:科学出版社;1990.p . 69 - 124。
- 17。
Morawetz JJ,Randle CP,Wolfe广告。奥贝丘西热带思想内的系统发育关系。分类。2010; 59(2):416-26。
- 18。
Kato Y,Hisano K.根寄生虫的体外培养,Aeginetia籼L. - I.综合调查。Bot Mag Tokyo。1983; 96(3):203-9。
- 19。
王志强,王志强,王志强,等。Orobanchaceae 5种寄生植物的水平基因转移。基因组生物学进展。2018;10(12):3196-210。
- 20。
Oborníkm,绿色br。光合真核生物中血红素生物合成途径的马赛克起源。mol Biol Evol。2005; 22(12):2343-53。
- 21。
黄志强,王志强。叶绿素生物合成的研究进展。出自:Pessarakli M,编辑。手册的光合作用。博卡拉顿,伦敦,纽约:泰勒和弗朗西斯;2005.37-54页。
- 22。
佩里,沃尔夫·哈哈。豆类叶绿体DNA中的核苷酸取代速率取决于倒置重复的存在。J Mol Evol。2002; 55(5):501-8。
- 23。
赵文彬,崔BH,金智华,李dh,李智华。完整的质体测序显示在半寄生虫中一个极度减少的SSC区域Pedicularis Ishidoyana.(列当科)。关键词:神经网络,神经网络,神经网络
- 24.
Su Hj,Barkman TJ,Hao W,Jones SS,Naumann J,Skippington E,Wafula Ek,Hu Jm,Palmer JD,Depamphilis CW。新遗传典型和富集富含血液植物Balanophora的高增殖基因组中的富含素质。Proc Natl Acad Sci U S A. 2019; 116(3):934-43。
- 25.
Molina J, Hazzouri KM, Nickrent D, Geisler M, Meyer RS, Pentony MM, Flowers JM, Pelser P, Barcelona J, Inovejas SA, et al.;寄生开花植物叶绿体基因组的可能丢失大花草属lagascae(大花草科)。生物医学进展。2014;31(4):793-803。
- 26.
链接本文:Börner T, Zhelyazkova P, Legen J, Schmitz-Linneweber C.叶绿体基因表达与rna合成。在:Theg SM, Wollman F-A,编辑。质体生物学。纽约:施普林格纽约;2014.3-47页。
- 27.
Wicke S,MüllerKF,Depamphilis CW,Quandt D,Bellot S,Schneeweiss Gm。进化率变化的机理模型在植物中非空白生活方式的途径。Proc Natl Acad Sci U S A. 2016; 113(32):9045-50。
- 28.
陈志强,陈志强,陈志强,等。寄生性开花植物质体基因组的分子进化。在:Chaw S-M, Jansen RK,编辑。植物学研究进展。麻萨诸塞州:学术出版社;2018.315 - 47页。
- 29.
Naumann J, Der JP, Wafula EK, Jones SS, Wagner ST, Honaas LA, Ralph PE, Bolin JF, Maass E, Neinhuis C,等。非光合寄生植物高度分化质体基因组的检测和特征Hydnora visseri(菌花科)。基因组生物学进展。2016;8(2):435 - 63。
- 30.
Schoefs B,Franck F.蛋白氯化物减少:机制和进化。Photochem photobiol。2003; 78(6):543-57。
- 31.
质体向细胞核的信号传递:信使仍然迷失在迷雾中?趋势麝猫。2009;25(4):185 - 92。
- 32.
Dierckxsens N,Mardulyn P,Smits G. Novoplasty:来自整个基因组数据的细胞器基因组的De Novo组装。核酸RES。2017; 45(4):4。
- 33.
Wyman Sk,Jansen Rk,Boore JL。用教条自动注释细胞细胞基因组。生物信息学。2004; 20(17):3252-5。
- 34。
Lohse M,Drechsel O,Kahlau S,Bock R. Consterellargenomedraw - 一种用于生成塑体和线粒体基因组的物理图和可视化表达数据集的工具套件。核酸RES。2013; 41(Web服务器问题):W575-81。
- 35。
亲爱的AE,MAU B,Perna NT。进步:多种基因组对齐与基因增益,损失和重排。Plos一个。2010; 5(6):E11147。
- 36。
Boratyn Gm,Thierry-Mieg J,Thierry-Mieg D,Busby B,Madden TL。Magic-Blast,一种精确的RNA-SEQ对齐器,用于长短读取。BMC生物信息学。2019; 20(1):405。
- 37。
Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R.序列比对/映射格式和SAMtools。生物信息学。2009;25(16):2078 - 9。
- 38。
Zerbino Dr,Birney E. Velvet:De Bruijn图表的De Novo短读组装的算法。Genome Res。2008; 18(5):821-9。
- 39。
Camacho C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K, Madden TL. BLAST+:建筑与应用。BMC生物信息学。2009; 10(1): 421。
- 40。
Li H.用BWA-MEM对准序列读取,克隆序列和组装折叠。arxiv预印刷品。2013; 00(2013):1-3。
- 41。
kaoh K, Standley DM. MAFFT多序列比对软件版本7:性能和可用性的改进。生物医学进展。2013;30(4):772-80。
- 42。
Stamatakis A. Raxml版本8:一种系统发育分析和大脑发育后的分析。生物信息学。2014; 30(9):1312-3。
- 43。
王志强,王志强,李敏。基于遗传算法的分子进化遗传分析。生物医学进展。2018;35(6):1547-9。
- 44。
杨志。PAML 4:最大似然法的系统发育分析。中国生物医学工程学报。2007;24(8):1586-91。
- 45。
Grabherr Mg,Haas Bj,Yassour M,Levin JZ,Thompson Da,Amit I,Adiconis X,Fan L,Raychowdhury R,Zeng Q等。来自RNA-SEQ数据的全长转录组件没有参考基因组。NAT BIOTECHNOL。2011; 29(7):644-52。
- 46。
Li B,杜威CN。RSEM:具有或不具有参考基因组的RNA-SEQ数据的准确转录物定量。BMC生物信息学。2011; 12:323。
- 47。
Kanehisa M, Goto S. KEGG:京都基因和基因组百科全书。核酸学报2000;28(1):27-30。
- 48.
Livak KJ, Schmittgen TD。利用实时荧光定量PCR技术分析相关基因表达数据−ΔΔCT方法。方法。2001;25(4):402 - 8。
致谢
不适用。
资金
国家自然科学基金项目(no . 31811530297);中国科学院华南植物园广东省应用植物学重点实验室项目(no . AB2018014);广州市科技计划项目(no . 201707020035);资助机构在研究的设计、数据的收集、分析和解释以及手稿的撰写中没有发挥作用。
作者信息
隶属关系
贡献
RZ构思了该研究并设计了实验;JC,RY和JD执行了实验和数据分析;JC和RZ写了稿件;RZ和YL修改了手稿。所有作者阅读并认可的终稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:图S1。
基于20种血浆基因的序列,奥博法西亚河七种七种物种的最大可能性树。节点中的数字是引导值。每个网站的替换量。
附加文件2:图S2。
转录组分析与qRT-PCR检测的相关性。每个qRT-PCR反应分别进行3个生物学重复和3个技术重复。
附加文件3:图S3。
在观察到的光合作用途径中基因的表达Aeginetia籼转录组。检测到表达的基因在红框中。礼貌的©www.genome.jp/kegg/kegg1.html..
附加文件4:图S4。
检测到卟啉和叶绿素代谢途径中基因的表达Aeginetia籼转录组。检测到表达的基因在红框中。礼貌的©www.genome.jp/kegg/kegg1.html..
附加文件5:表S1。
五种全寄生植物和一种自养亲缘植物的质体基因含量Lindenbergia philippensisin.黑匣子表示没有基因,白色盒子表明存在基因,灰色盒子表明假生素。
附加文件6:表S2。
缺失和转移质体基因的基因组位置Aeginetia籼.其注释、序列比对、比对位置和序列识别均基于叶绿体基因Lindenbergia philippensis.*表示基因也存在于质体中Aeginetia籼.
附加文件7:表S3
.基于七种酿酒座共用的20蛋白编码基因的分支模型分析,放松杂种植物中的纯化选择。似然比测试用于比较三个模型(M0:单个比率模型; M2:两个比率模型; M3:三比率模型)。P.- 当小于0.05时,值得粗体。
附加文件8:表S4。
的ungenes表达水平Aeginetia籼在光合作用途径中基于转录组分析。
附加文件9:表S5。
64个参考质体基因组的数据来源。
附加文件10:表S6。
用于定量实时PCR的引物。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
陈,J.,Yu,R.,Dai,J.等等。光合作用途径的丢失和缺失质体基因在全寄生植物中的基因组位置Aeginetia籼.BMC植物BIOL.20,199(2020)。https://doi.org/10.1186/s12870-020-02415-2
收到了:
公认:
发表:
关键词
- Aeginetia籼
- 质体基因组
- 转录组