摘要
背景
氮素是作物生长的主要营养元素。在植物中,肽转运蛋白(PTR)基因家族的成员可能参与硝酸盐的吸收和转运。在这里,我们鉴定了水稻PTR基因家族,并分析了它们在近等基因系中的表达谱。
结果
在Nipponbare、R498和Nipponbare中分别鉴定出96、85和78个PTR基因光稃稻亚洲栽培稻和非洲栽培稻的系统发育树相似。花生(125)和大豆(127)的PTR基因数较高。水稻、玉米、高粱、花生、大豆和大豆中的521个PTR基因拟南芥可分为4组,单子叶植物和双子叶植物的分布不同。在日本粳稻基因组中,25个PTR基因分布在1、2、3、4、5、7、8、9和10号染色体的5个片段重复区,其中16个包含NPF(NRT1/PTR家族)结构域。RNA序列分析结果表明,NIL15和NIL19在三个时期的差异表达基因数分别为928、1467和1586个。在低氮条件下,差异表达的PTR基因数量显著增加。使用WGCNA分析RNA-seq数据以预测基因间的潜在相互作用。我们将表达模式相似的基因分为一个模块,得到25个目标模块。在这些模块中,有三个模块可能参与水稻氮素的吸收和利用,尤其是棕色模块,其中hub基因被注释为调节水稻氮素代谢的蛋白激酶。
结论
本研究对水稻PTR基因家族进行了综合分析。在日本七鳃鳗基因组中发现96个PTR基因,其中25个位于5个大片段重复区。Ka/Ks比值表明许多PTR基因发生了正选择。RNA-seq结果表明,许多PTR基因参与了水稻氮素利用效率(NUE),蛋白激酶可能在这一过程中起重要作用。这些结果为通过分子育种改良水稻核苷利用率提供了基础。
背景
水稻是世界上最重要的粮食作物之一。随着人口的不断增长,粮食安全问题变得越来越突出1]. 氮肥的施用一直是提高水稻产量的主要手段,但氮肥的过量施用也增加了生产成本,对环境造成了严重的破坏。随着“绿色超级稻”概念的提出[2]因此,发展高效利用氮肥的水稻品种已成为在保护环境的同时提高农业生产的关键。
nrt1在水稻硝酸盐吸收和转运中发挥重要作用[3.那4.].NRT1家族主要包括低亲和力硝酸盐运输司,属于PTR家族。因此,NRT1 / PTR系列也被命名为NPF。在稻米中鉴定了主要的NUE相关的PTR基因[5.].osnrt1.是水稻中鉴定的第一个PTR基因[6.]并位于与OsNPF8.9型[7.];osnrt1.有两份笔录,OsNRT1.1a和OsNRT1.1b.OsNRT1.1b编码一个具有6个跨膜结构域的PTR蛋白,该蛋白在高氮或低氮条件下均能提高水稻的氮素吸收OsNRT1.1a仅在高氮条件下工作[8.]. 基于与ATNRT1.1., Plett等人[9.]将三种同源基因命名为稻米OsNRT1.1A那OsNRT1.1B,和osnrt1.1c。过度表达OsNRT1.1A可以上调与硝酸盐和铵转运相关的基因[10.]. 胡等[11.)发现,loc_os10g40600.编码一个硝酸盐转运蛋白NRT1.1B,它可能影响indic和japonica.通过调节水稻根微生物种群并改变根际微环境[12.].osnpf2.2.可以从水稻木质部卸载硝酸盐,参与硝酸盐从根向茎的运输,从而影响维管系统甚至整个植株的生长发育[13.].过度表达OSPTR6.提高铵转运蛋白基因的表达,激活谷氨酰胺合成酶,对水稻生长有促进作用,但在高铵条件下降低了NUE[14.].Fang等人[15.]表明改变OSPTR9.影响氮素利用效率、植物生长和水稻产量。osnpf2.4.没有扮演一个角色3.-吸收、长途运输和再分配;此外,改变其表达间接影响钾在根和茎中的再利用。胡等[16.]结论了osnpf7.2.在高硝态氮条件下,对根系中硝态氮的细胞内分布起一定作用,并影响水稻的生长。最近,Gao等人[17.]利用氮素利用效率的差异籼稻和japonica.米饭克隆一种编码NADH / NADPH依赖性硝酸还原酶OSNR2的基因,其与硝酸盐转运蛋白OSNRT1.1b相互作用,以通过籼稻促进硝酸盐的吸收。Tang等人。[18.]双亲和硝酸盐转运蛋白基因的克隆osnpf6.1.发现了转录因子锇酸钠42可以激活osnpf6.1.,进而提高水稻氮素吸收和氮素利用效率。综上所述,这些水稻PTR基因可能在氮素利用中发挥重要作用,而且在水稻基因组中可能还有其他PTR基因参与氮代谢途径。
在先前的研究中,我们发现了一个NUE相关的QTLqNUE6米饭,还有loc_os06g15370.可能是理想的候选基因[19.].诠释loc_os06g15370.是肽转运蛋白,我们将此基因命名为OsPTR10型,因为OsNPF3.1型[7.].在这里,我们使用最新的Nipponbare Genome来鉴定PTR基因,并将它们与R498中的PTR基因进行比较,光稃稻玉米、高粱、花生、大豆和拟南芥. 本研究的主要目的是:(1)测定水稻PTR基因的数目(ii)了解水稻和其他5种植物PTR基因的进化关系((三)分析水稻核发育过程中PTR基因的调控网络。
结果
栽培稻PTR基因的鉴定
在27日选用品种,亚洲栽培水稻(栽培稻L.)和非洲种植的水稻(光稃稻Steud.)是人类驯化和利用的两种物种[20.].使用Ptr基因家族的保守区域序列,在Nipponbare中鉴定了96个PTR基因(栽培稻L. SSP..japonica.)(表2)在R498中鉴定出85个(栽培稻l . ssp。籼稻)(表3.)和78人被确定在光稃稻(表4.).
PTR基因的系统发育分析
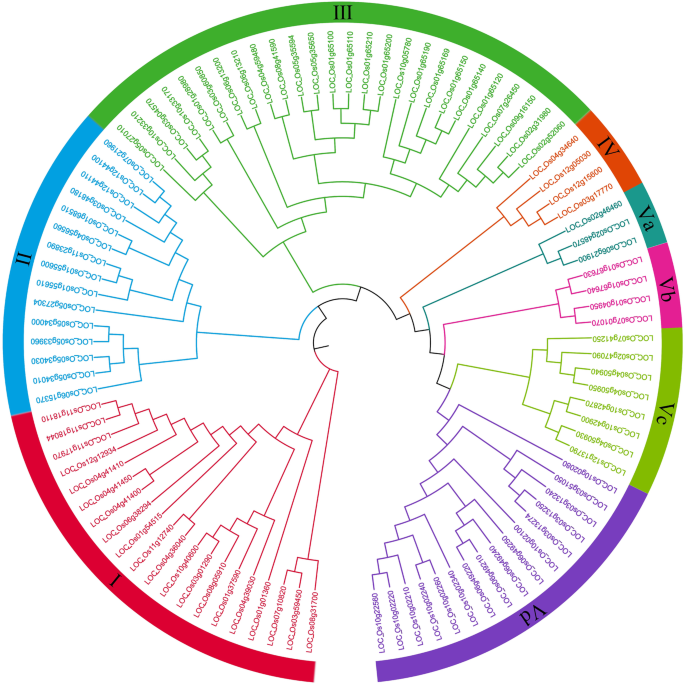
利用MEGA技术对96个水稻PTR基因的蛋白序列进行多序列比对和系统发育分析。根据进化关系,将96个PTR基因分为5类,grouppito V分别包含20、15、26、4和31个基因。V组包含的基因最多,可进一步分为Va(3)、Vb(4)、Vc(8)和Vd(16) 4个亚组(图1)。1).
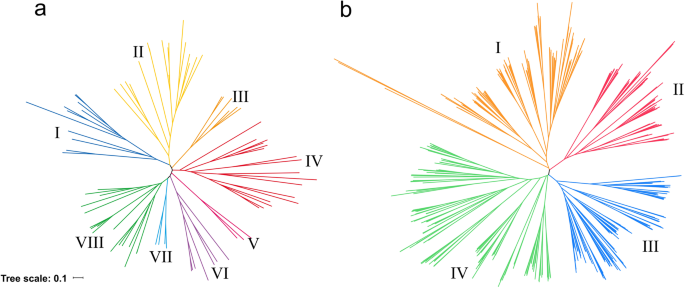
了解PTR基因在不同物种间的进化关系选用利用Nipponbare、R498和Nipponbare的PTR基因进行系统发育分析光稃稻.这些PTR基因可分为8组,其中II组最多,包含73个基因,而VII组只有11个基因。2a). PTR基因在8组中的分布在3组之间是相似的选用物种(表S)5.).Nipponbare Genome用于进一步的分析,如下所示。
豆科植物可与固氮细菌形成共生关系,固氮细菌可转化氮2进入NH3.. 因此,我们分析了豆科植物花生和大豆。我们还分析了双子叶植物拟南芥以及重要的单子叶植物玉米、高粱和水稻。这6种植物的基因组大小为2.5gb[21., 1.1 Gb [22.], 125 Mb[23.],2.3 Gb[24.], 730 Mb[25.]和466 Mb[26.]分别是。虽然玉米基因组很大,但它只包含66个PTR基因,而豆类基因组包含更多的PTR基因:花生125个,大豆117个。这可能与豆科植物固氮后氮转运的需要有关。
基于来自六种物种的543个PTR基因的蛋白质序列构建系统发育树(表S.6.)这些基因可分为I、II、III和IV四组(图。2b) 是的。PTR基因在水稻、玉米和高粱中的分布相似,其中以Ⅲ组单子叶植物PTR基因最多。同样,PTR基因在花生、大豆和大豆中的分布也存在差异拟南芥双子叶PTR基因最多的是IV组。
米PTR基因的染色体位置和分段重复事件
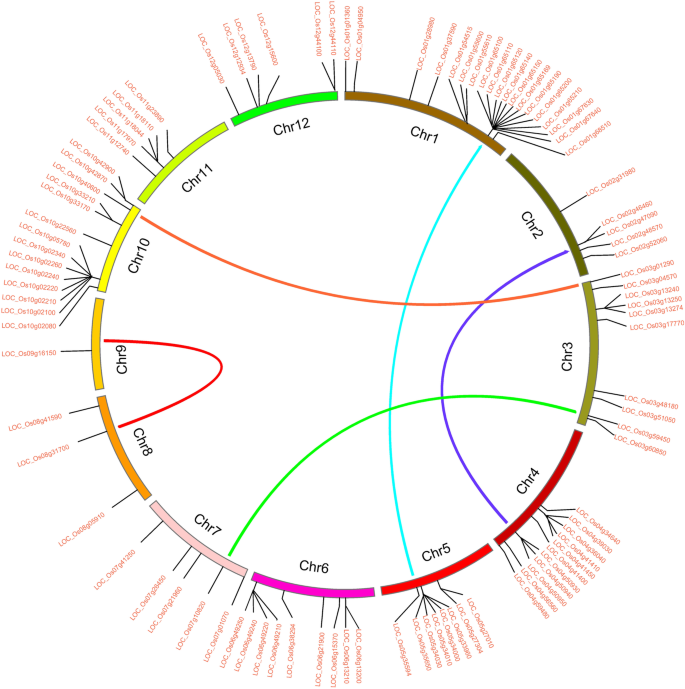
96个PTR基因在水稻中的12条染色体上不均匀分布。染色体1具有最少的PTR基因和染色体9。细节如图2所示。3..59个PTR基因组成17个基因簇,占PTR基因总数的60.4%。除7、8、9号染色体外,其余染色体均含有PTR基因簇,其中1号染色体37.79 ~ 37.83 Mb区域的聚类最大,共含有9个PTR基因。
对96个PTR基因进行了片段重复事件分析。结果表明:1号染色体上有37.76 Mb ~ 38.75 Mb, 5号染色体上有20.31 Mb ~ 21.21 Mb;(ii)第2染色体上的27.74 Mb−28.95 Mb对应第4染色体上的29.11 Mb−30.42 Mb;(iii)第3染色体上的0.15 Mb ~ 0.21 Mb对应第10染色体上的21.76 Mb ~ 21.90 Mb;(iv)第3染色体上33.56 Mb−33.92 Mb,对应第7染色体上5.31 Mb−6.14 Mb;(v) 8号染色体上的19.49 Mb−19.744 Mb对应9号染色体上的12.13 Mb−12.45 Mb1).
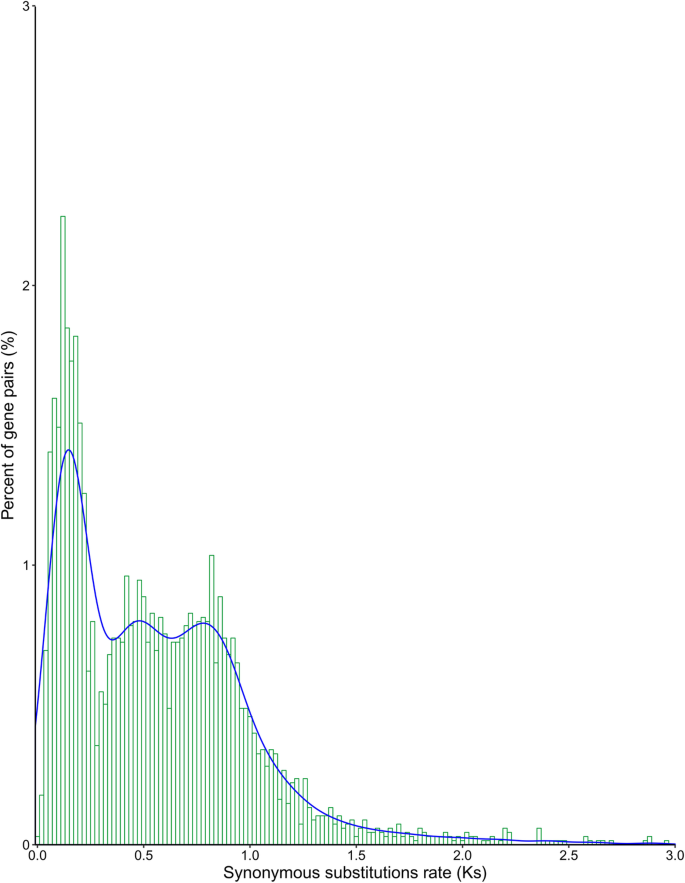
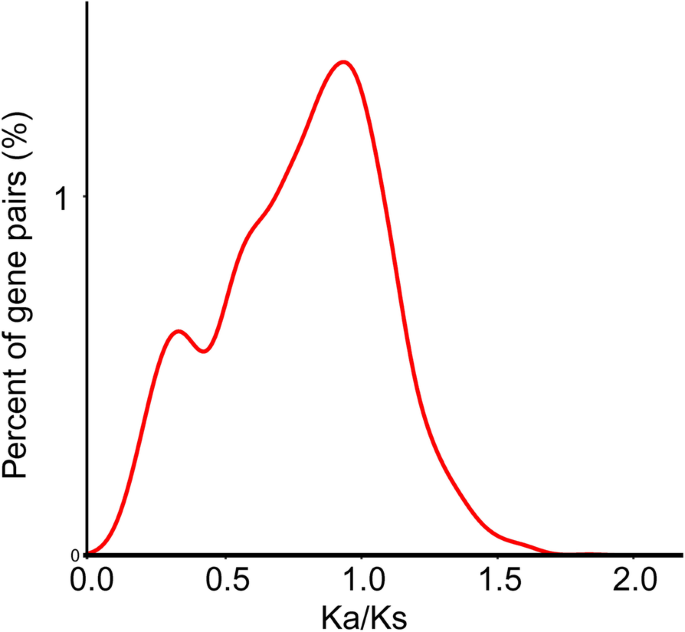
用同义突变率Ks检测全基因组重复。水稻基因组经历了三次基因组复制事件。4.).禾本科植物所共有的全基因组复制事件大约发生在9600万年前[27.]. 之后,水稻又发生了两次独立的基因组复制事件[28.那29.].Ka / Ks比反映了在分化期间基因的所有核苷酸序列的范围。如果Ka / ks大于1,则基因被积极选择;如果Ka / ks等于1,则基因是中性的选择;如果Ka / Ks小于1,则基因经历净化选择。帕拉二pTR基因对的Ka / ks比率为0.08-1.83,693个基因对大于1,表明许多PTR基因接受阳性选择(表S7., 图。5.).
水稻PTR基因的基因结构和基序组成
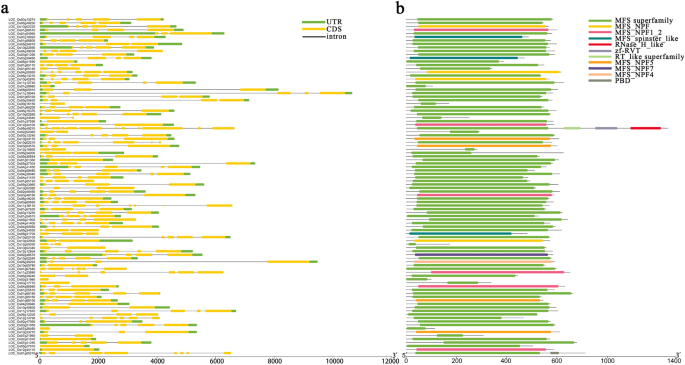
可以从基因结构分析获得与基因家族的演化相关的信息。因此,我们对96个PTR基因进行了基因结构分析(图。6.a).从内含子-外显子组成来看,PTR基因包含0 ~ 11个内含子和1 ~ 12个外显子,其中10个基因只包含1个外显子。通常,结构相似的基因有更密切的进化关系。
以往的研究表明,在大多数PTR基因中有三个高度保守的结构域[30.]. 在本研究中,水稻PTR基因共有5种保守结构域,分别是MFS、RNaseƏHƏu样、zf-RV、RTƏu样超家族和PBD。MFS可进一步分为MFS超家族、MFS\u NPF、MFS\u NPF1\u 2、MFS\u spinster\u like、MFS\u NPF5、MFS\u NPF7和MFS\u NPF4。通过对PTR基因进行保守基因分析,我们发现77个基因具有MFS超家族结构域,16个基因含有NPF结构域(图。6.b)。
PTR基因在近等基因系中的表达谱
基因的表达谱往往与其功能有关。以往的研究表明,PTR基因编码的蛋白质能够转运硝酸盐,在植物生长发育中起着重要的作用。因此,我们利用RNA-seq分析了近等基因水稻系NIL15和NIL19在HN和LN条件下96个PTR基因的表达谱。
转录组测序质量和序列比对结果见表S8..18个样品的cDNA文库测序质量高,基因组覆盖率好,适合进一步分析。根据不同样本的基因表达水平,我们对常见表达基因和差异表达基因(DEGs)进行了相关性分析。同一样本的生物重复间的平均相关系数为r = 0.9357(图S1),表明良好的再现性和可靠的实验结果。
我们使用qRT-PCR方法验证了从RNA-seq中鉴定出的18个表达基因。结果表明,18个DEGs的qRT-PCR表达模式与RNA-seq分析一致(图S)2),表明rna测序结果对进一步分析是可靠的。
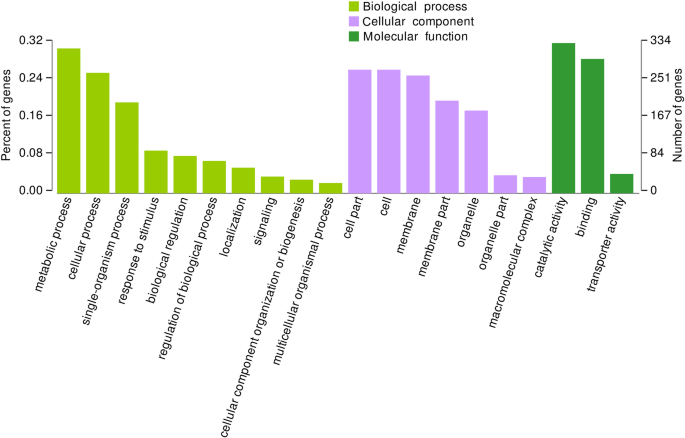
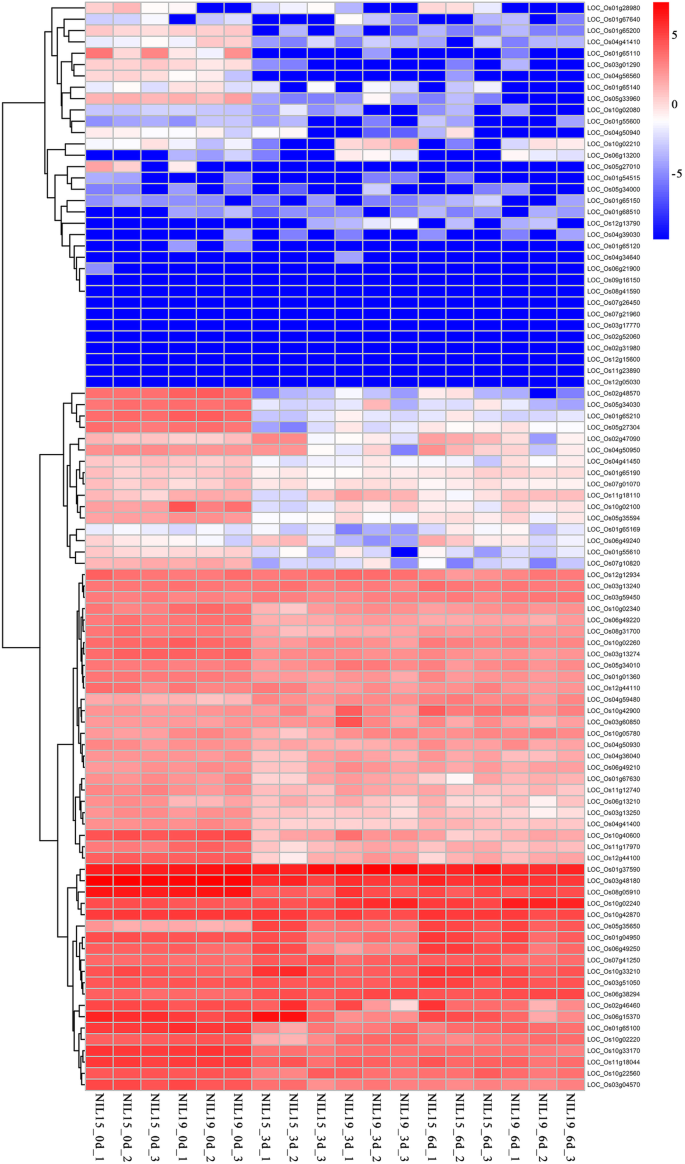
之前的研究表明NIL15和NIL19之间的差异存在于染色体6、8、9和10上[19.].这些区域包含6个PTR基因LOC_Os06g13200那LOC_Os06g13210,L.OC_OS06G15370那地址:Os06g21900那地址:Os06g38294,和LOC_Os10g22560. 转录组测序结果显示,两个品系在三个时期的DEGs数分别为928、1467和1586(图3)3.)分别是。转入LN状态后,DEGs数量先显著增加,然后趋于稳定。我们还使用GO数据库对DEGs执行了函数注释(http://geneontology.org/)分类结果如图所示。7.. 在生物过程中,代谢过程包含的基因最多;在细胞成分中,与膜或膜成分相关的基因较多;在分子功能分类中,出现了与转运体活性相关的基因。
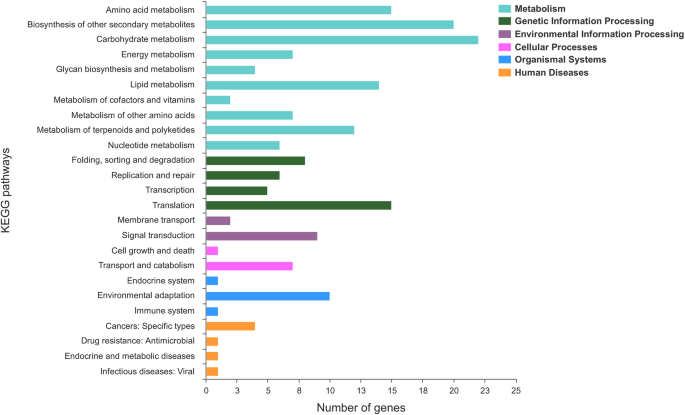
在生物体内,不同的基因相互协作,共同发挥生物功能,不同基因间的相同作用形成一条通路。我们使用KEGG数据库对DEGs进行了分类和注释(https://www.kegg.jp/)分类结果如图所示。8.. 主要参与的途径有:代谢、遗传信息处理、环境信息处理、细胞过程、机体系统和人类疾病,其中代谢途径包含的基因最多。能量代谢组包括氮代谢相关基因。
96个PTR基因的表达谱如图所示。9..在0 d时,两个NILs间差异表达的PTR基因只有2个LOC_Os06g13210和地址:Os10g02100.然而,在3 d和6 d时,差异表达的ptr基因的数量增加到12和7(表S.9.),其中位置:Os04g50950(OSPTR6.)[14.],地址:Os06g49250(OSPTR9.)[15.),LOC_Os11g12740(SP1)[31.]已被报道参与水稻氮素吸收和转运。
稻米NUE的基因调节网络
使用加权基因共表达网络分析(WGCNA)分析表达谱数据以预测基因之间的潜在相互作用。基因表达水平的相关系数计算并采取了n度功率,使得相关系数的数值分布逐渐遵循无缝线分布。筛选58,176基因,标准是:(i)至少50%的样品表达基因;(ii)除去差异变化最小的基因> = 25%。最后,留下了20,282个基因进行进一步分析。
然后对样品进行聚类,发现0 d、3 d和9 d的样品可以很好地分离(图S4.a),表明数据质量足以用于后续分析。然后,选择软阈值以构建基因共存网络(图S4.公元前)。20,282基因用于WGCNA分析,并从基因组随机选择400个基因以绘制表达簇热图(图S5.). 我们将表达模式相似的基因分为一个模块,共鉴定出25个模块。我们分析了每个模块的基因特征,找到了具有生物学意义的模块(图S)6.).随后,使用GO和KEGG数据库进行功能富集分析,发现蓝色、棕色和turquoise模块参与了N代谢。蓝色模块包含6个与nue相关的基因:LOC_OS01G36720[32.],loc_os03g62200.[33.],loc_os04g40410[34.],loc_os04g43070[35.],地址:Os05g39240[36.),地址:Os06g49250[15.].棕色模块包含LOC_Os01g61510[33.],地址:Os01g65000[33.],LOC_Os02g47090[16.),位置:Os04g50950[14.].含有的绿松石模块地址:Os02g02190[34.],地址:Os02g40710[37.],地址:Os02g40730[38.],loc_os03g13274[6.],LOC_Os03g48180[39.],loc_os10g40600.[12.那40],LOC_Os11g12740[31.),位置Os12g44100[13.].
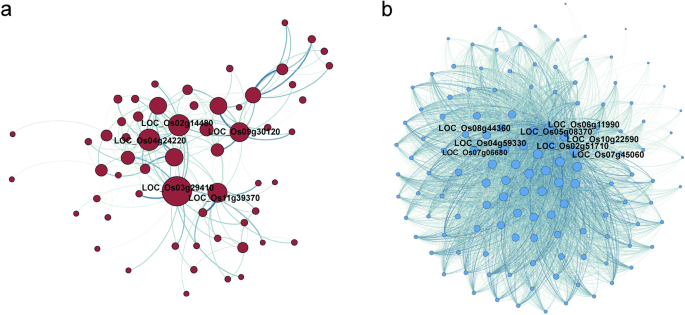
我们在这三种模块化基因上进行了WGCNA。棕色模块中有59次,loc_os03g29410.那loc_os02g14480.那地址:Os04g24220那地址:Os11g39370那位置:Os09g30120是监管网络中的集线器基因(表S10., 图。10.a) 是的。最相关基因的功能loc_os03g29410.那地址:Os04g24220那loc_os02g14480.,和地址:Os11g39370与蛋白激酶有关。基于GO注释,这些基因的功能与活性(GO: 0016301)、应激(GO:0006950)、生物刺激(GO: 0009607)、信号转导(GO: 0007165)和代谢过程(GO: 0008152)有关。绿松石模块里有129个degLOC_Os06g11990那LOC_Os10g22590那loc_os08g44360那位置:Os07g45060那LOC_Os07g06680那loc_os05g08370.那loc_os04g59330.和loc_os02g51710是枢纽基因(表S11., 图。10.b).由于蓝色模块中deg数量较少,我们无法识别hub基因。
讨论
在这项研究中,我们确定了Nipponbare Genome中的96个PTR基因,其与Zhao等人鉴定的84个PTR基因不同[30.]这种差异可能是由于:(1)随着测序技术的发展,日本裸鼠基因组数据库不断被编辑和完善[41.那42.];(ii)两项研究中使用的方法是不同的。赵等人。使用BLASTP和BLASTN根据PTR结构域的保守氨基酸序列来搜索PTR基因。我们的研究使用HMMER3根据PTR基因家族的保守DNA序列搜索PTR基因,然后进行手动验证。
水稻96个PTR基因可分为5组,且每组数量不相同。此外,loc_os03g13250和loc_os03g13274(osnrt1.)[6.],loc_os03g01290.和loc_os10g40600.(OsNRT1.1B)[11.],LOC_Os07g21960和位置Os12g44100(OSPTR2.)[13.],LOC_Os04g50940和位置:Os04g50950(OSPTR6.)[14.],地址:Os04g36040和LOC_Os11g12740(SP1)[31.]他们的关系更密切。因此,这5个基因可能参与了水稻氮素的吸收和转运。
在亚洲栽培米中,japonica.PTR基因数量高于籼稻大米;亚洲栽培稻的PTR基因也比非洲栽培稻多。尽管日本航空、R498和光稃稻含不同数量的PTR基因,其在8个群体中的分布在进化上是一致的。与玉米和高粱相比,水稻基因组最小,但PTR基因最多。豆科植物的根上有根瘤菌,能将空气中的无机氮转化为有机氮。我们发现花生和大豆的PTR基因较多,这可能与固氮后氮转运的需要有关。在系统发育关系上,单子叶植物和双子叶植物的PTR基因分布存在显著差异,这可能是由于这两类植物在进化过程中产生了各自独特的PTR基因[43.].
基因家族可以通过全基因组复制或多倍体化、串联复制和片段复制形成。在本研究中,我们发现许多由串联复制形成的PTR基因往往紧密排列在同一条染色体上。以往的研究也表明,同一基因簇中的基因具有相似的序列和功能[44.].利用PTR基因对片段复制事件进行分析,我们发现水稻基因组中存在5个重复的区块,这与之前的结果一致[28.].在水稻中96个PTR基因中,在节段性重复中含有25个。此外,大米接受了三种全基因组重复事件:所有禾本科植物共享的第一个全基因组重复事件,以及其他两个染色体片段在水稻基因组上独立发生倍增。该结果与之前的研究一致[28.那29.那45.].此外,复制区域中的PTR基因被聚集成相同的组,具有更紧密的系统发育关系。有许多PTR基因对具有大于1的Ka / ks值,表明非同义突变导致PTR基因的功能变化,使其更适合环境变化[46.].
基因的结构与其功能有关。以往的研究表明,水稻PTR基因的蛋白质序列中有三个保守的基序[30.]; 它们都包含在MFS家族中,有12个跨膜结构域[47.].在这项研究中,我们发现大多数PTR基因包含MFS家族基因,以及已知的水稻NUE基因,osnrt1.[6.],OsNRT1.1A[10.],.OsNRT1.1b[8.],OSPTR2.[13.],OSPTR6.[14.],OSPTR9.[15.],osnpf7.2.[20.),SP1[21.]全部包含MFS系列域。此外,96个PTR基因中有16个具有NPF结构域。在植物中,NPF蛋白运输多种底物:硝酸盐、肽、氨基酸等[7.由于内含子长LOC_Os11g18044,序列长度(10.7kb)大于水稻中的其他PTR基因;此外,它含有更长的MFS家族域,表明该基因的功能可能更复杂。
N15和NIL19的转录组分析显示,大部分DEGs与代谢转运激活相关,这与植物N代谢途径一致。在HN条件下,只有2个PTR基因存在差异表达,而在LN条件下,有12个PTR基因的表达水平发生了变化。这些结果表明,PTR基因可能以协同的方式在氮代谢中发挥重要作用。之前的研究已经证明了这一点地址:Os06g49250(OSPTR9.)[15.],位置:Os04g50950(OSPTR6.)[14.],LOC_Os11g12740(第1页)[31.]影响水稻氮素利用效率,这些基因在LN条件下均有差异表达。
在可能与稻米有关的三个模块中,每个模块具有不同数量的n个代谢的基因,但它们含有与铵和硝酸铵的摄取和使用有关的基因,表明铵和硝酸盐基因的协同表达.这一结果表明,N吸收,运输,同化和信号转导涉及大米中的复杂基因监管网络[5.].
在brown模块中,hub基因的功能注释为蛋白激酶。已有研究表明,一些蛋白激酶与氮代谢有关拟南芥. 钙调神经磷酸酶B样(CBL)相互作用蛋白激酶AtCIPK8(CIPK.)发现参与了早期硝酸盐信号传导[48.].CIPK和丝裂剂激活激酶激酶激酶(MEKK)是涉及早期硝酸盐信号传导的调节蛋白[49.].加利福尼亚州2+-传感器蛋白激酶(cpk)是主要的调节器,协调初级硝酸盐反应[50.].此外,芸苔类固醇(BR)信号激酶BSK3在有限的N条件下调节根伸长率[51.]. 在水稻中发现了许多蛋白激酶,主要是钙依赖性蛋白激酶、丝裂原活化蛋白激酶、应激活化蛋白激酶。它们在水稻生长发育、生物胁迫、非生物胁迫和表达调控等方面发挥着重要作用。近年来,研究人员发现多种蛋白质基因与水稻的NUE有关。OsCPK12参与了低氮胁迫下水稻的响应信号途径,OsCPK12的过表达促进了低氮条件下水稻的生长[52.].谢等人[53.]发现氮调节基因,Os02g0120100那OS02G0807000和Os06g0692600编码转录因子,蛋白激酶和蛋白质磷酸酶的等,并且可以参与水稻根部恢复的调节。丝氨酸/苏氨酸/酪氨酸(STY)蛋白激酶,ACTPK1,增强铵吸收和使用,并促进了足够铵下水稻幼苗的生长[54.].钙依赖性蛋白激酶基因,ESL4.,它可以在正新代谢基因的上游作用[55.].过度表达钙调神经磷酸酶b样相互作用蛋白激酶2基因,oscipk2.,可提高水稻氮素利用率[56.].OsSAPK8在激活OsSLAC1时是AtOST1的对应物,OsSLAC1是水稻中的一种硝酸盐选择性阴离子通道[57.].地址:Os11g39370是一种芸苔功能性创新的1-相关接收激酶1前体基因。因此,棕色模块可能与水稻中的N吸收和利用的调节有关。候选基因OsPTR10型在前期研究中发现的褐变模块中也包含有褐变模块,其功能可能与氮代谢有关。
结论
本研究对水稻PTR基因家族进行了综合分析。在Nippobare基因组中鉴定了96个PTR基因,并将其分为5大类。PTR基因在单子叶和双子叶之间的分布不同,豆科植物的PTR基因较多。水稻基因组经历了3次基因组复制事件,25个水稻PTR基因位于5个大片段复制区域。Ka/Ks比值表明许多PTR基因经历了正选择。结构和基元分析表明,水稻PTR基因中存在5种保守结构域。在LN条件下,差异表达的PTR基因显著增加。利用WGCNA,我们发现了3个与NUE相关的基因模块,尤其是黄色模块。这些结果为更好地理解PTR基因的生物学功能提供了信息,为水稻氮素利用效率的遗传改良提供了理论依据。
方法
基因组序列检索及PTR基因鉴定
从以下数据库下载了6个物种的基因组序列:Nipponbare(MSU 7.0,http://rice.plantbiology.msu.edu/index.shtml), R498 [58.] (你可以吗,http://www.mbkbase.org/R498/),光稃稻[59.](野生稻),http://peanutgr.fafu.edu.cn/Genome_Browse.php),拟南芥[23.](塔伊尔10,http://plants.ensembl.org/Arabidopsis_thaliana/Info/Index),落花生hypogaea[21.](PGR,http://peanutgr.fafu.edu.cn/genome browse.php.),Zea Mays.[24.](b73_refgen_v4,http://plants.ensembl.org/Zea_mays/Info/Index),甘氨酸最大[22.](glycine_max_v2.1,http://plants.ensembl.org/Glycine_max/Info/Index), 和高粱双色[25.](高粱,http://plants.ensembl.org/Sorghum_bicolor/Info/Index).PTR基因家族HMM (hidden markov model)文件从pam (http://pfam.xfam.org/).我们使用HMMER3对PTR基因进行了全基因组搜索,e值截断值= 0.001,比对序列大于198(占395的50%)。提取蛋白序列后存入CDD (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),聪明(http://smart.embl.de/)和pfam(http://pfam.xfam.org/).然后手工验证同源家族基因的结构域保守性。如果在其中一个数据库中检测到保守结构域,则认为其为可靠的家族基因。最后,提取其保守结构域和蛋白序列。
种系发生树结构
利用ClustalW对已鉴定的PTR基因进行多序列比对。NJ树是使用MEGA7构建的,引导重复次数为1000,其他参数为默认值。系统发育树用iTOL (https://itol.embl.de/).
染色体分布和基因复制
使用BLAST (basic local alignment search工具)进行all - on -all蛋白序列比对,提取e-value < 0.00001,身份> 90%,查询序列上匹配区域最小覆盖到受试者序列> 75%的蛋白对作为同源蛋白。将同源蛋白文件和蛋白坐标文件输入MCScanX (http://chibba.pgml.uga.edu/mcscan2/)对于共线区域鉴定。最后,使用电池提供了线性结果(http://circos.ca/).
基因结构与保守基序分析
通过GSDS分析了PTR基因的结构,主要包括UTR(untranslated region)、内含子和外显子(http://gsds.cbi.pku.edu.cn/).通过MEME进行主题序列预测(http://meme-suite.org/tools/meme.),并用TBtools绘制了主题图。
植物材料和水栽法
Y11是野生稻(oryza rufipogon.)GH99是精英(栽培稻l . ssp。籼稻),这些材料来源于广西。自2007年秋季以来,我们以Y11为供体,GH998为受体,构建了近等基因系(NILs,BC)4.F6.) . .NIL-13B4(低氮肥利用率)和GH998(高氮肥利用率)的氮肥利用率分别为3.64和39.08% [19.]. 本研究选取NIL15(NIL-13B4)和NIL19(GH998)作为实验材料。选择发芽均匀的种子(NIL15和NIL19)在96孔培养器中培养,培养液浓度为1.4 毫米NH4.不3.(高氮,HN)营养液。营养液按吉田等的方法配制[60.]每3天改变营养溶液,使用MERS-NaOH将pH调节至〜5.5。幼苗在智能人工气候室(顶级仪器,杭州,浙江,中国)在28°C控制,辅助光从6:00至19:00 h(13小时光/ 11小时黑暗)直到3-叶阶段。然后,在3叶阶段,用0.14mm NH改变营养溶液4.不3.(低氮气,LN)溶液,并在0d,3d和6d中收集样品,用于RNA-SEQ和定量实时PCR(QRT-PCR),每个样品具有三种生物学复制。
RNA提取和测序
采用Trizol法提取每个样品的RNA。用琼脂糖凝胶电泳检测每个RNA样本的纯度和完整性。用Nanodrop (Thermo Fisher Scientific, USA)检测DNA纯度(OD值260/280≈2.0)。RNA浓度采用Qubit (Thermo Fisher Scientific, USA)精确定量,最低浓度为50 ng/ul。
用Oligo (dT)包被磁珠从总RNA中分离mRNA。然后用裂解缓冲液将mRNA随机裂解成~ 300bp的片段。在逆转录酶和一个6碱基随机引物(随机六聚体)的作用下,mRNA被逆转录成单链cDNA,通过双链合成形成稳定的双链结构。构建的文库使用Illumina NovaSeq 6000进行测序。
测序质量控制和对准
质控过滤后,将clean reads与参考基因组(http://rice.plantbiology.msu.edu/index.shtml)以获取映射的读取以进行后续分析。序列比对使用TopHat2[61.]. 当参考基因组完全注释且实验无污染时,定位率通常高于70%。
实时定量PCR
采用qRT-PCR检测18个基因的表达水平。引物根据目的基因序列设计(表S1), 和肌动蛋白3用作参考文献[62.].使用QTower 2.2定量实时PCR热循环仪(德国分析仪Jena),在96孔板中进行所有QRT-PCR测定。反应系统包括:10μl2×转型Sybr Green Master混合物(Vazyme,南京,江苏,中国),每种引物的1μl,1μl模板cDNA,由DDH2O递给20μL。产品扩增的循环程序如下:94℃5分钟,然后进行94℃的40℃,30s(变性),55℃,30s(退火),72℃,30 s(延伸).为每个样本设置了海普拉特。当反应完成时,分析熔融曲线,并基于熔化曲线测定产物的特异性。通过参考2计算相对基因表达水平-ΔΔct方法[63.].
差异表达基因分析
根据特征计数的结果对准参考基因组和注释文件,获得每个样品的基因读数,然后进行FPKM(每kiolbase百万的片段)转化以获得标准化的基因表达水平。deseq2 [64.]用于对原始计数进行统计分析。默认的阈值是:p-adjust < 0.05和|日志2fc| > = 1,产生了两组或两个表达倍数大于2倍的样本之间的差异表达基因P.值小于0.05。
加权基因共表达网络分析
对基因表达数据进行背景校正和归一化后,筛选出非标准基因和改变较少的基因,使基因相关强度处于无标度分布。数据预处理后,对基因进行分类:将具有相似表达模式的基因分为一类,称为一个模块。然后分析模块间基因和模块内基因之间的关系。通过与表型数据的关联得到关键模块,通过可视化网络分析得到模块的枢纽基因。
数据和材料的可用性
本研究中分析的所有数据都包含在主稿件和其附加文件中。支持本工作的调查结果的数据可在纸质及其补充信息文件中提供。支持本研究调查结果的RNA-SEQ数据已存入国家生物技术信息中心(NCBI)序列读取档案(SRA),并附有加入代码PRJNA573824 [https://www.ncbi.nlm.nih.gov/bioproject/PRJNA573824].
缩写
- n:
-
氮
- PTR:
-
肽转运蛋白
- 自虐:
-
氮气使用效率
- NPF:
-
NRT1 / PTR家庭
- 度:
-
差异表达基因
- 零:
-
近代线
- 隐马尔可夫模型:
-
隐藏的马尔可夫模型
- NJ:
-
Neighbor-joiningmethod
- UTR:
-
未经翻译的地区
- qRT PCR:
-
定量RT-PCR
- FPKM:
-
每千碱基百万的碎片
- 开始:
-
基因本体论
- 小桶:
-
京都基因和基因组百科全书
- WGCNA:
-
加权基因共表达网络分析
工具书类
- 1。
GodFray HC,Beddington JR,Crute IR,Haddad L,Lawrence D,Muir JF等。粮食安全:喂养90亿人的挑战。科学。2010; 327:812-8。
- 2。
张强绿色超级稻的发展策略。美国国家科学研究院2007;104:11640-6402。
- 3.
李H,胡锦涛,胡锦涛在作物中的氮气利用效率:拟南芥和稻米的课程。J Exp Bot。2017; 68:2477-88。
- 4.
轩辕,北克曼T,徐G.植物氮营养:传感和信令。CurrOp植物BIOL。2017; 39:57-65。
- 5。
王,郑毅,陈克,Tsay yf。硝酸盐运输,信号传播和使用效率。Annu Rev植物Biol。2018; 69:85-122。
- 6。
于淑梅,林天明,刘玉芳,刘玉芳。硝酸盐转运体基因的克隆及功能分析。osnrt1.,来自大米。植物生理学。2000;122:379–88.
- 7。
Lérans,varala k,Boyer Jc,Chiurazzi M,Crawford N,Daniel-Vedele F,等。植物中硝酸盐转运蛋白1 /肽转运蛋白家族成员的统一命名。趋势植物SCI。2014; 19:5-9。
- 8.
风扇X,Feng H,Tan Y,Xu Y,Miao Q,Xu G.一种推定的6 - 跨膜硝酸盐转运蛋白OSNRT1.1b在低氮下在水稻中发挥关键作用。J Intent Plant Biol。2016; 58:590-9。
- 9.
Plett D,Toubia J,Garnett T,测试仪M,Kaiser BN,Baumann U。二分法NRT基因家族的Dicots和草地。Plos一个。2010; 5:E15289。
- 10。
王伟,胡斌,袁丹,刘勇,车瑞,胡勇,等。硝酸盐转运蛋白基因的表达OsNRT1.1A/osnpf6.3.水稻高产早熟。植物细胞。2018;30:638–51.
- 11.
胡b,王w,ou s,唐j,李h,che r等。变异nrt1.1b.有助于米亚种之间的硝酸盐偏差。NAT Genet。2015; 47:834-8。
- 12.
张杰,刘义新,张恩,胡布,金特,徐赫,等。nrt1.1b.与水稻根系微生物组成和氮素利用有关。纳特生物技术公司。2019;37:676–84.
- 13。
李y,欧阳j,王yy,hu r,xia k,duan j等。水稻硝酸盐转运蛋白的破坏OSNPF2.2阻碍了根到硝酸盐的硝酸盐转运和血管发育。SCI REP。2015; 5:9635。
- 14。
acta photonica sinica, 2011, 38(5): 759 - 768 .范晓,谢丹,陈杰,陆辉,徐勇,马超,等。表达的OSPTR6.不同施氮量下,水稻植株生长均有促进作用,但高施氮量下,氮肥利用率降低。植物科学。2014;227:1-11。
- 15.
方Z,夏克,杨X,Grotemeyer Ms,Meier S,Rentsch D等人。改变了表达的PTR./NRT1同系物OSPTR9.影响水稻氮素利用效率、生长发育和产量。植物生物技术杂志。2013;11:446–58.
- 16。
胡瑞,邱德,陈勇,米勒AJ,范X,潘X,等。敲除液泡膜局部低亲和力硝酸盐转运蛋白OsNPF7.2对高硝酸盐供应下水稻生长的影响。前植物科学。2016;7:1529.
- 17。
高Z,王义,陈克,张A,阳,尚L等。这籼稻硝酸还原酶基因OsNR2等位基因提高水稻产量潜力和氮素利用效率。自然公社。2019;10:5207.
- 18.
唐伟,叶杰,姚昕,赵鹏,宣伟,田勇,等。全基因组相关研究鉴定了nac42激活的硝酸盐转运体赋予水稻高氮利用效率。Nat Commun。2019;10:5279。
- 19.
杨晓红,夏学忠,张志强,农博新,曾燕,熊凤琴,等。水稻氮素利用效率的QTL定位及候选基因分析植物科学与技术,2017;
- 20。
Stein JC,Yu Y,Copetti D,Zwickl DJ,Zhang L,Zhang C,等。13个驯养和野生稻豚的基因组突出了矿物质属的遗传保护,周转和创新。NAT Genet。2018; 50:285-96。
- 21。
庄W,陈H,杨米,王j,pandey mk,张c等。栽培花生的基因组提供了熟悉豆科核型,多倍体演化和作物驯化的洞察力。NAT Genet。2019; 51:865-76。
- 22。
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W,等。古多倍体大豆基因组序列。大自然。2010;463:178 - 83。
- 23。
倡议AG。开花植物基因组序列分析拟南芥. 自然。2000;408:796–815.
- 24
等。关键词:Schnable;玉米B73基因组:复杂性、多样性和动态性。科学。2009;326:1112-5。
- 25
Paterson啊,鲍德·杰,Bruggmann r,Dubchak I,Grimwood J,Gundlach H,等。这高粱双色基因组和草的多样性。大自然。2009;457:551-6。
- 26
Goff S,Ricke D,Lan Th,Presting G,Wang R,Dunn M等。水稻基因组的序列(栽培稻l . ssp。japonica.).科学。2002; 296:92-100。
- 27
王X,王j,金d,gu h,lee th,liu t等。基因组对齐跨越主要的Poaceae谱系揭示了异构的进化速率,并改变了关键进化事件的推断历。莫尔植物。2015; 8:885-98。
- 28
王X,史X,郝B,葛S,罗J。水稻基因组中的复制和DNA片段丢失:二倍体的意义。新植物醇。2005;165:937–46.
- 29.
王旭,史晓,李忠,朱强,孔璐,唐伟,等。基于基因共线性的染色体同源性统计推断及其应用拟南芥和米饭。BMC生物信息学。2006; 7:447。
- 30
赵X,黄J,于H,王L,谢W。水稻肽转运蛋白家族的基因组调查、鉴定及表达谱分析(栽培稻L.)。BMC植物生物学。2010;10:92.
- 31
李S,钱Q,傅Z,曾D,孟X,Kyozuka J等。短panicle1.编码一个假定的PTR家族转运体并决定水稻穗的大小。植物J。2009;58:592–605.
- 32
冯H,Yan M,Fan X,Li B,Shen Q,Miller Aj等。氮气地位水稻高亲和力硝酸盐转运蛋白的空间表达及调节。J Exp Bot。2011; 62:2319-32。
- 33
Suenaga A,Moriya K,Sonoda Y,Ikeda A,Von WN,Hayakawa T等.新型铵转运蛋白OsAMT2在水稻中的组成性表达。植物细胞生理学。2003;44:206–11.
- 34。
严M,范X,冯H,米勒AJ,沈Q,徐G。水稻OsNAR2.1与OsNRT2.1、OsNRT2.2和OsNRT2.3a硝酸盐转运蛋白相互作用,在高浓度和低浓度范围内提供吸收。植物细胞环境。2011;34:1360–72.
- 35。
杨S,郝D,丛Y,金M,苏Y。水稻OsAMT1;1是一种非质子依赖性反馈调节的铵转运蛋白。植物细胞代表2015;34:321–30.
- 36。
Gaur vs,Singh US,Gupta Ak,Kumar A.在两种水稻基因型中对铵转运蛋白和谷氨酰胺合成酶基因的影响对氮气具有差异反应的影响。mol biol rep。2012; 39:8035-44。
- 37。
Ferreira Lm,Souza V,Tavares O,Zonta E,Santa-Catarina C,Souza SR等。OsAMT1.3表达改变了水稻铵的摄取动力学和根形态。植物Biotechnol Rep。2015; 9:221-9。
- 38。
Sonoda Y、Ikeda A、Saiki S、Von WN、Yamaya T、Yamaguchi J。三种铵转运蛋白基因的差异表达及功能研究(OsAMT1;1.-1、3)在米饭中。植物细胞生理。2003; 44:726-34。
- 39。
夏X,范X,魏J,冯H,曲H,谢D,等.水稻硝酸盐转运蛋白OsNPF2.4在低亲和力获得和远距离转运中的作用。J实验机器人。2015;66:317–31.
- 40。
胡斌,姜震,王伟,邱勇,张震,刘勇,等。硝酸盐- nrt1.1 b - spx4级联集成了植物的氮磷信号网络。Nat植物。2019;5:401-13。
- 41。
陈鹏,景旭,廖波,朱勇,徐杰,刘锐,等。BioNano基因组地图资源栽培稻单一共享平台。japonica.和籼稻及其在水稻基因组序列校正和空白填充中的应用。摩尔。2017;10:895-8。
- 42。
关键词:水动力学,流体力学,Kanamori H, Mccombie WR,欧阳等。改进的栽培稻Nipponbare参照基因组使用下一代序列和光学地图数据。水稻(ny). 2013;6:4。
- 43。
Cai C,王JY,朱yg,沉QR,李B,童捷,等。基因结构及水稻根系高亲和力硝酸盐输送系统的表达。J Intent Plant Biol。2008; 50:443-51。
- 44。
Ramamoorthy R、Jiang SY、Kumar N、Venkatesh PN、Ramachandran S。基因的全面转录谱分析怀疑不同非生物和植物激素处理下水稻的基因家族。植物细胞生理学。2008;49:865–79.
- 45。
acta photonica sinica, 2011, 38(5): 762 - 768 .郭辉,焦勇,谭鑫,王鑫,黄鑫,金辉,等。谷物基因组的基因复制与基因创新。基因组研究》2019;29:261-9。
- 46。
张梓、李杰、赵旭、王杰、王国科、于杰。KaKsèu计算器:通过模型选择和模型平均计算Ka和Ks。基因组蛋白质组生物信息。2006;4:259–63.
- 47。
闫氮。主要促进剂超家族运输司的结构生物学。annu rev biophys。2015; 44:257-83。
- 48
AtCIPK8是一种cbl相互作用的蛋白激酶,调节初级硝酸盐反应的低亲和期。植物j . 2009; 57:264 - 78。
- 49
马凯,鲁迪埃,铸造L, Bréhaut V, Blondet E, Colot V,等。转录因子NLP7的核保留调控着植物对硝酸盐的早期反应。Nat Commun。2013;4:1713。
- 50.
刘克科,牛勇,Konishi M,吴勇,杜辉,孙春华,等。在中枢营养生长网络中发现硝酸盐- cpk - nlp信号。大自然。2017;545:311-6。
- 51.
Jia Z、Giehl R、Meyer RC、Altmann T、Von WN。BSK3基因的自然变异调节油菜素类固醇信号在低氮条件下的根系觅食。自然公社。2019;10:2378.
- 52.
Asano T,Wakayama M,Aoki N,小松S,Ichikawa H,Hirochika H等人。钙依赖性蛋白激酶基因的过表达增强了低氮条件下水稻的生长。植物生物科技。2010; 27:369-73。
- 53
谢辉,甘聪,吴希杨聪,谢明华。水稻幼苗根系缺氮的早期分子事件。Sci代表,2018年;8:12207.
- 54
Beier MP、Obara M、Taniai A、Sawa Y、Ishizawa J、Yoshida H等。缺乏ACTPK1(一种STY激酶)可促进铵的吸收和利用,并促进水稻幼苗在充足的外部铵胁迫下的生长。植物J。2018;93:992–1006.
- 55
邢莹,郭胜,陈旭,杜丹,刘敏,肖勇,等。缺氮水稻突变体的氮代谢受到影响ESL4.用钙依赖性蛋白激酶基因突变。植物细胞生理。2018; 59:2512-25。
- 56
李平,穆汗,阿姆jad H,汗AQ,阿拉法特Y, Waqas M,等。探索过表达的潜力oscipk2.水稻是一种高效利用氮素的作物及其相关根区微生物群落分析。国际分子科学杂志。2019;20:3636.
- 57
Sun SJ,齐GN,高QF,王HQ,Yao Fy,Hussain J等。蛋白激酶OSSAPK8用作S型阴离子通道OSSLAC1的基本活化剂,其在水稻中是硝酸盐选择性的。Planta。2016; 243:489-500。
- 58。
杜H,YU Y,MA Y,GAO Q,Ca Y,Chen Z等。近完整籼稻基因组测序和DE Novo组装。NAT Communce。2017; 8:15324。
- 59。
王M,Yu Y,Haberer G,Marri Pr,Fan C,Goicoechea JL等。非洲稻的基因组序列(光稃稻)和独立驯化的证据。Nat麝猫。2014;46:982-8。
- 60。
Yoshida S,Forno D,公鸡JH。水稻生理研究实验室手册。菲律宾Irri;1976. p。62。
- 61。
Kim D, Pertea G, Trapnell C, Pimentel H, Kelley R, Salzberg SL. TopHat2:转录组插入、缺失和基因融合的精确对齐。基因组医学杂志。2013;14:R36。
- 62。
孙浩,钱琦,吴凯,罗娟,王松,张超,等。异三聚体G蛋白调控水稻氮素利用效率。Nat麝猫。2014;46:652-6。
- 63。
Livak KJ, Schmittgen TD。使用实时定量PCR和2(−Delta Delta C(T))方法分析相关基因表达数据。方法。2001;25:402-8。
- 64。
爱我,胡伯W,安德斯S。用DESeq2对RNA序列数据的折叠变化和离散度的中度估计。基因组生物学。2014;15:550.
致谢
作者感谢庄杰和严志琼的帮助。
资金
中国科学院广西自然科学基金(2015GXNSFA13139054,2018GXNFAA138124),国家自然科学基金(31860371),中国农业科学院水稻生物重点实验室(160105)(2015JZ16,2019Z08,QN-02),资助广西省自然科学基金(2015GXNSFA131354,2018GXNSFA1313117)。资助机构在研究设计、数据收集、分析和解释以及手稿撰写方面没有任何作用。
作者信息
隶属关系
贡献
XHY和DTL构思了本研究。BXN、ZQZ、YYW和QLT进行数据分析。曾伟英、JG、周伟勇和HFL进行了实验,XZX和YZ撰写了本文。XHY和GFD对该手稿进行了修订。所有作者阅读并批准论文。
相应的作者
伦理宣言
道德认可和参与同意
不适用。
出版许可
不适用。
相互竞争的利益
作者们宣称他们没有相互竞争的利益。
附加信息
出版商说明
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充资料
表S1。
引物序列用于qRT-PCR。
表S2。
日本血吸虫96个PTR基因的鉴定。
附加文件3表S3。
R498中85个PTR基因的鉴定。
附加文件4表S4。
小鼠78个PTR基因的鉴定光稃稻.
附加文件5表S5。
日本血吸虫PTR基因在日本血吸虫、R498和日本血吸虫中的分布光稃稻.
附加文件6表S6。
PTR基因在6种植物中的分布。
表S7。
水稻PTR基因对的Ka/Ks比值。
表S8。
测序数据及日本裸基因组定位。
表S9。
PTR基因的差异表达分三个阶段。
附加文件10表S10。
棕色模块中差异表达的基因。
附加文件11表S11。
绿松石模块中的差异表达基因。
附加文件12图S1。
18个样本的相关系数由基因表达水平决定。彩色框显示相关系数。
附加文件13图S2。
QRT-PCR用于验证从RNA-SEQ鉴定的18个表达基因。X轴表示3个处理的阶段,紫色柱表示QRT-PCR导致NIL19,绿色塔表示QRT-PCR在NIL15中产生QRT-PCR,蓝柱表示RNA-SEQ导致NIL19,黄色柱代表RNA-SEQ结果在NIL15。Y轴表示基因表达的相对水平,QRT-PCR使用2-ΔΔct值,RNA-seq使用FPKM值。误差线表示三次生物重复的标准差。
附加文件14。
图S3。采用DESeq2筛选。A1对A2表示0 d时NIL15和NIL19之间的928度,B1对B2表示3 d时NIL15和NIL19之间的1467度,C1对C2表示6 d时NIL15和NIL19之间的1586度。
附加文件15图S4。
样本聚类树状图和软阈值(β)值。一种基于表达谱的18个样本的样本聚类树状图和临床特征热图。B.分析1 ~ 20各β值的无标度贴合指数。C1 ~ 20 β值的平均连通性分析。后续分析选择β =10,因为当无标度拟合指数达到0.895时,其平均连通性最大。
附加文件16图S5。
随机抽取400个基因的基因网络热图。基因树状图和模块分配也显示在顶部。颜色尺度:黄色为低相关性,红色为高相关性。
附加文件17图S6。
鉴定在两个近代线中与NUE相关的模块。颜色刻度:黄色表示低相关性,红色表示相关性。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非信用额度中另有规定。
关于这篇文章
引用这篇文章
杨,X.,夏,X.,曾,Y。等等。基因组鉴定水稻肽转运蛋白家族及其不同氮气利用效率的两种近代近代近型近代线的分析。BMC植物BIOL.20,193(2020)。https://doi.org/10.1186/s12870-020-02419-Y.
收到:
公认:
出版:
关键词
- 大米
- PTR家庭
- 系统发育分析
- 表达谱
- 基因监管网络