摘要
背景
作为一种独特的硫酸化多糖,Fucoinean是棕色海藻中细胞壁的重要组成部分。其生化特性由硫酸盐基团的位置和量决定。磺基转移酶(STS)催化硫化过程,将硫基转移到碳水化合物骨架上,对岩藻糖生物合成至关重要。然而,结构和功能STS.在棕色海藻中很少被调查。
结果
共有44个 英石我们基因组和转录组分析中鉴定的基因Saccharina粳稻,它位于17个脚手架和11个Contigs中。这S. japonica st.基因具有丰富的内含子和可变的剪接位点,并鉴定了5个串联复制基因簇。一般来说,英石根据系统发育分析,基因可分为5个类群(I ~ V)。因此,由同一组基因编码的ST蛋白含有相似的保守基序。的成员S. japonica st.基因家族在不同的组织和发育阶段表现出不同的表达模式。转录谱表明,转录水平的一半以上英石基因在海带基础叶片中的基因高于远端叶片。除了ST5.和ST28,最多英石基因与海带开发阶段下调。表达水平的九个圣基因通过实时定量PCR检测,这表明它们对低盐度和干旱胁迫作出反应。
结论
各种特征STS.允许的可行性S. japonica用不同的硫酸盐基团合成抗果环糖。这使得海带能够适应昂贵环境并满足需求的可能性S. japonica增长。
背景
Saccharina粳稻是一种在亚洲具有很高商业价值的褐藻。它含有丰富的粗纤维和碳水化合物,被广泛用作提取海藻酸和岩藻聚糖的原料。此外,S. japonica含有许多对化妆品,食品和健康有价值的生物活性物质[1].在所有具有生物活性的代谢物中,岩藻多糖(一种硫酸多糖)被认为在医学领域具有很高的价值。例如,岩藻多糖具有免疫调节、抗炎、抗肿瘤、抗凝和抗血栓作用[2,3.,4,5],对缓解糖尿病肾病和腺嘌呤引起的慢性肾病也有疗效[6,7].
Fucoinean,主要存在于褐藻的棘突和细胞壁中[8],首次被褐藻中的Kylin发现昆布属植物digitata1913年[9].褐藻中岩藻糖苷的生物合成途径直到褐藻基因组序列的释放才被阐明异甲酸硅在2010年 [10].基于大肠siliculosus基因组测序和与糖胺聚糖(GAG)生物合成,Michel等。(2010)推断出岩藻酮可以首先通过岩糖基转移酶聚合成中性多糖,然后通过特异性磺基转移酶硫化[11].他提出了两种GDP - 岩藻糖产生的路线:1)果糖-6-磷酸盐通过甘露糖-6-磷酸异构酶(MPI),磷酰胺酶(PMM)和甘露糖-1-磷酸胍瓜蒽糖酶(MPG)合成GDP-mannose,其次是通过GDP-甘露糖4,6-脱氢酶(GM46D)和双官能酶GDP-L-粘合酶合酶(GFS)催化的GDP - 岩藻糖的产生;2)或者,L-岩藻糖用作基质通过岩藻糖激酶(FK)和GDP - 岩藻糖磷磷酸化酶(GFPP)合成GDP - 岩藻糖。随后使用GDP - 岩藻糖通过岩糖基转移酶(FUT)和磺基转移酶(ST)产生岩藻酮。研究了一些参与岩藻藻生物合成的基因已经研究过S. japonica和Nemacystus decipiens[12,13].Chi等人。(2017)探索了MPI,MPG和PMM的基因来源,表达差异和酶活性S. japonica[14].Nishitsuji等人(2019)证实了这一点颗和GFPP融合在N. Decipiens.基因组(13].Zhang et al.(2018)阐述了PMM/PGM (phosphoglucomutase, PMM/PGM)的表达、纯化、酶活性以及对光照和温度胁迫的响应S. japonica[15].
岩环糖生物合成过程中有许多种类的单糖。硫酸化岩藻烷的主要成分是L-岩藻糖-4-硫酸盐;半乳糖,甘露糖,木糖,葡萄糖,阿拉伯糖和葡糖醛酸存在少量[16,17].认为藻类中岩藻类的含量和结构因藻类种类、组织、年龄、居住环境和季节的不同而不同[18,19].Fucoinean的结构参数,例如单糖和岩藻糖链的类型和多糖的分子量,尤其有助于其生物活性,尤其是硫酸盐基团对大分子骨架上的数量和位置[20.,21,22].例如,2,3 -二硫糖残基是抗凝血活性的常见结构[23,24],而在C-2位置存在2- o -硫酸酯化会降低岩藻糖胶的抗凝活性[25].因此,硫化对岩藻糖糖的功能有影响。据报道,磺基转移酶(ST)将硫基团从通用供体3'-磷甲酸甜(PAPS)转移到碳水化合物骨干上的硫酸盐(PAPS)中[26].因此,作为岩藻多糖生物合成最后一步的关键酶,磺基转移酶决定了岩藻多糖中硫酸根的位置和数量。多个英石序列已经被注释在许多种藻类的基因组中,例如41大肠siliculosus和24 in.Cladosiphon Okamuranus.[10,27].此外,Ye et al.(2015)在14种藻类基因组中重建了碳代谢途径[12, 14个物种中有13个包含英石基因。考虑英石基因家族含有大量推定构件,因此必须全局分析这些的不同特征STS.在褐藻。然而,目前还没有关于英石棕色藻类基因家族到目前为止。
在这项研究中,我们筛查了104个基因(包括三个基因mpi, 二PMMs, 三GM46DS., 22岁FUT., 73年英石和一个颗)参与岩藻多糖的生物合成S. japonica.具体来说,我们描述了英石通过分析基因在低盐和干旱非生物胁迫下的序列特征、支架位置、系统发育关系、组织和时间特异性表达模式和动态转录谱。这是第一次研究英石家庭成员在S. japonica.我们的结果提供了对棕色海藻中硫酸化岩环藻的生物合成的宝贵知识,并具有巨大的STS在岩藻藻合成中的体外应用潜力。
结果
岩藻聚糖生物合成基因的鉴定和表达谱
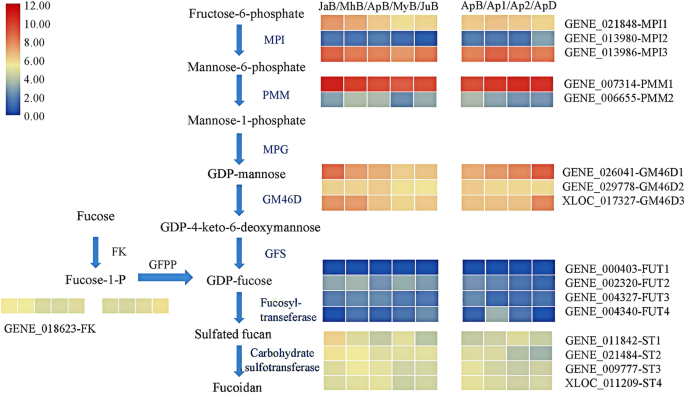
基于我们的基因组和转录组数据库,共注释了104个与岩藻糖胶生物合成相关的基因S. japonica.表S1列出了它们的基因ID、序列长度和FPKM值(附加文件1:表S1)。数字1显示相应基因的表达水平。转录水平MPI3(GENE_013986),PMM1(GENE_007314),GM46D1(GENE_026041)和ST1.(Gene_011842)在每个催化步骤中相对较高,被认为在海带生长和发育期间对岩藻糖糖生物合成至关重要。从表达丰富的角度来看,PMM1显示最高的FPKM值,而所有FUN在非常低的水平上表达。在不同的发育阶段,大多数这些基因被显示出下调趋势。在不同的组织中,MPI2,PMM1,GM46D1,GM46D3和颗叶底向叶底呈上升趋势PMM2,GM46D2,FUT2.,FUT3.被抑制。
表征的鉴定和序列英石基因
S. japonica基因组中有73个基因被自动注释为英石基因。在用Blast,Smart和PFAM数据库进行进一步分析之后,除去具有低置信酶域和重复基因的低置信度的序列。最后,确认了44个序列S. japonica st.至少包含其中一个结构域的基因:gal-3-o_sulfotr域(PF06990),Sulfotransfer_1域名(PF00685),Sulfotransfer-2域(PF03567)或Sulfotransfer_3域(PF13469)。
这些44英石基因命名ST1.来ST44(从高到低的平均fpkm值)。在表中总结了44个基因的名称,基因ID,支架位置,ORF长度,外显子数,氨基酸数,分子量和PI,以及它们的相应蛋白质1.STS的氨基酸的数量范围为82(ST42)至514(ST38),它们的分子量为9.57kDa(ST42)至56.82kDa(ST38)。ST蛋白的预测等电点(PI)值范围为4.66(ST34)至10.16(ST29)。
ST蛋白的定位预测表明,它们具有复杂的细胞区隔化(附加文件2:表S2)。预测显示70.45%的蛋白质是非分泌性的。9种蛋白(ST2、ST3、ST16、ST23、ST35、ST37、ST38、ST39、ST41)具有跨膜螺旋,可能位于质膜或内膜系统。其中4个st (ST3、ST16、ST37、ST38)具有信号肽,2个st (ST2、ST23)具有信号锚。此外,有7个蛋白(ST1、ST4、ST6、ST7、ST10、ST11和ST25)含有无跨膜结构域的信号肽。而ST3、ST7、ST9、ST14、ST16、ST19、ST22、ST25、ST35、ST38、ST43和ST44则定位于线粒体。
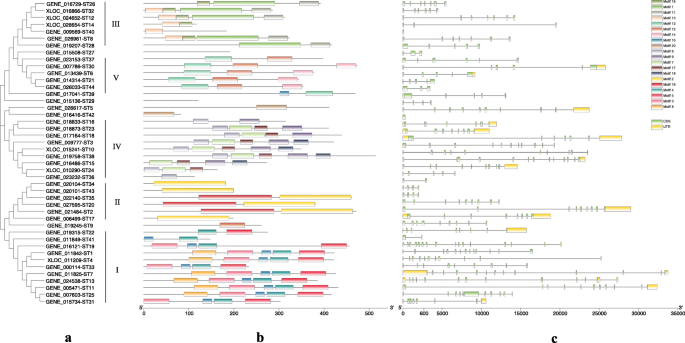
系统发育、基序和基因结构分析英石基因
44个S. japonicaST蛋白主要可分为五组(I - V)(图1)。2a).模因法共鉴定出20个保守基序,其长度为21 ~ 163个氨基酸(图2)。2b和附加文件3.:表S3)。同一组中的ST氨基酸序列含有类似的保守的基序,并表明它们是高度保守的(图。2a和b).包含GroupI (25%)Gal-3-O_sulfotr域(PF06990)、II组(13.6%)、III组(15.9%)和V组(11.36%)Sulfotransfer_1域(PF00685)和Sulfotransfer_3域(PF13469),第四组(20.45%)含有Sulfotransfer-2域(PF03567)。检测到图案1,16和20的两个Sulfotransfer_1域(PF00685)和Sulfotransfer_3域(PF13469)。除了,gal-3-o_sulfotr和Sulfotransfer_3结构域仅存在于藻类中[28].
系统发育关系,保守的基因和基因结构英石基因及其相应的蛋白质S. japonica.一个: 44个物种的系统发育树S. japonica圣蛋白。使用Mega 7.0.26构建最大可能性系统发育树,具有1000个引导复制,WAG + F + G方法,伽马4,部分删除和50%的场地覆盖作为截止。b: 44个ST蛋白中的保守基序。20个假定的主题用不同颜色的盒子表示。关于主题的详细信息列在附加文件中3.:表S3。c:建筑物的结构英石基因。外显子,内含子和UTR分别由绿色盒子,黑线和黄色框表示
基因结构分析显示英石基因家族包含多个内含子。内含子的数量为0 ~ 12个。每一个英石平均含有6.95个内含子。大多数英石基因(77.3%)具有超过三个内含子。只有一个基因(ST42.)没有内含子。内部内含子英石基因大小接近15 kb(图。2c)。
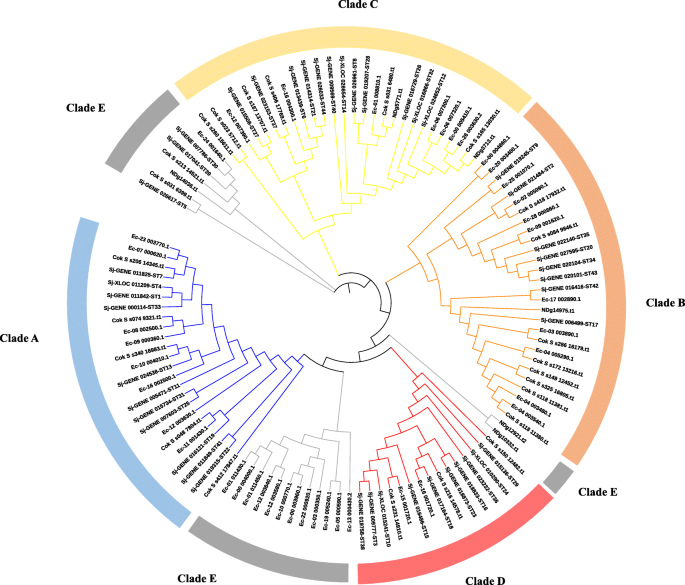
研究它们之间的进化关系STS.带注释的S. japonica和其他褐藻,基于ST氨基酸序列构建最大似然(ML)系统发育树:44S. japonica41,大肠siliculosus, 24C. Okamuranus.和六个N. Decipiens.(无花果。3.和额外的文件4:表S4)。分开五片ST曲折,包括疏水湿的A(24),B(28),C(27),D(15)和E(21)。第三组和第4族 英石基因在S. japonica在无花果。2分为疏枝C并显示比其他群体更近的进化关系。同一落后的STS包含相同的域。ST5,ST30和ST39与其他STS一起进化距离粳稻。每个进化枝都包含来自日本血吸虫,矽肺杆菌和c . okamuranus。有趣的是,12 STs大肠siliculosus在进化枝a和进化枝D之间形成了一个群,并包含了Sulfotransfer-2域(PF03567)。STs从N. Decipiens.仅在Clade B,C和E中找到。
ST蛋白的系统发育树大肠siliculosus,C. Okamuranus.,N. Decipiens.和S. japonica.基于115 sts的全长氨基酸序列构建最大似然性系统,具有1000个自举复制,WAG + F + G型号,γ2,部分缺失和50%位点覆盖为截止。将这些115个蛋白质聚集成五个亚壳。序列列于附加文件中4:表S4
交替剪接分析STS.
我们分析了所有替代拼接(AS)站点的类型和数量S. japonica st.不同组织和发育阶段的基因。该基因家族共鉴定出217个位点。最丰富的网站类型是另一种转录开始网站(TSS)类型(72),其次是外显子跳过(ES, 52岁),其他(31),p3_splice (21), p5_splice(19),可变转录终端站点(TTS, 13)和intergentic(9)。尽管类型和数量的网站没有统一的组织和发展阶段不同,例如,一些基因集中包含某些AS类型,ST1.(p5_splice),ST3.和ST19(p3_splice),ST8.(TTS)和ST9.和ST26.(TSS)。在同一样品中,检测到较多的AS位点英石表达水平较高的基因。此外,基底叶片比远端叶片发现更多的AS位点。这些网站的细节列在附加文件5:表S5。
脚手架地点和基因重复的STS.
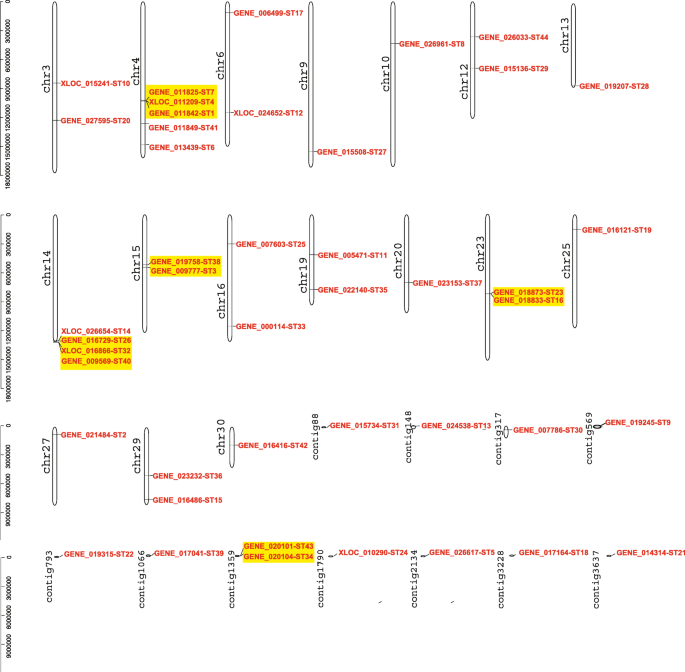
英石基因座随机分散分布于17个支架和11个contigs上S. japonica基因组(图。4).4号和14号脚手架上有5号和4号英石位点,分别。虽然基因家族的产生通常归因于串联复制和节段复制[29], 这英石家族中只存在串联复制,这5对基因占全部基因的27.3%英石基因家庭。同一串联重复对的基因位于同一支架或CONTIG中,并证明了密切的物理距离。通常,在染色体上只有一个序列,或者在同一染色体上显示两个序列。复制英石基因对在脚手架15上发现(ST3.和ST38.)、23日(ST16和ST23.)及contig1359 (ST34.和ST43.).两组串联复制基因,ST1.,ST4.和ST7.在4号断头台上被识别ST14,ST26.和ST32.在14号脚手架上被识别。我们发现了高度相似的基因结构、保守基序和蛋白质二级结构(图。5)在同一对串联复制基因中。没有共线性英石用McScanx观察家庭成员。
ST蛋白的二次结构分析
蛋白质的二级结构主要是指主肽链沿一维方向有规律地与氢键卷曲折叠,形成具有周期性结构的构象。我们使用SOPMA预测STs的蛋白质结构,发现了4个二级结构,它们随机分布在所有肽链中,包括alpha-helix、extended strand、beta-turn和random coil(附加文件6:表S6)。二级结构以α -螺旋和随机螺旋为主,平均分别占41.91和39.87%。延伸链的比例为12.58%。最小的是beta轮,只有5.65%。在五组无花果中。2,III组α-螺旋比例最低,仅为34.19%,而其他四组含有超过40%的α-螺旋。同一串联重复对的基因(图。4)显示出这四种二级结构的相似比例。数字5显示每个组的代表性二次结构。
转录的STS.在不同的组织和发育阶段
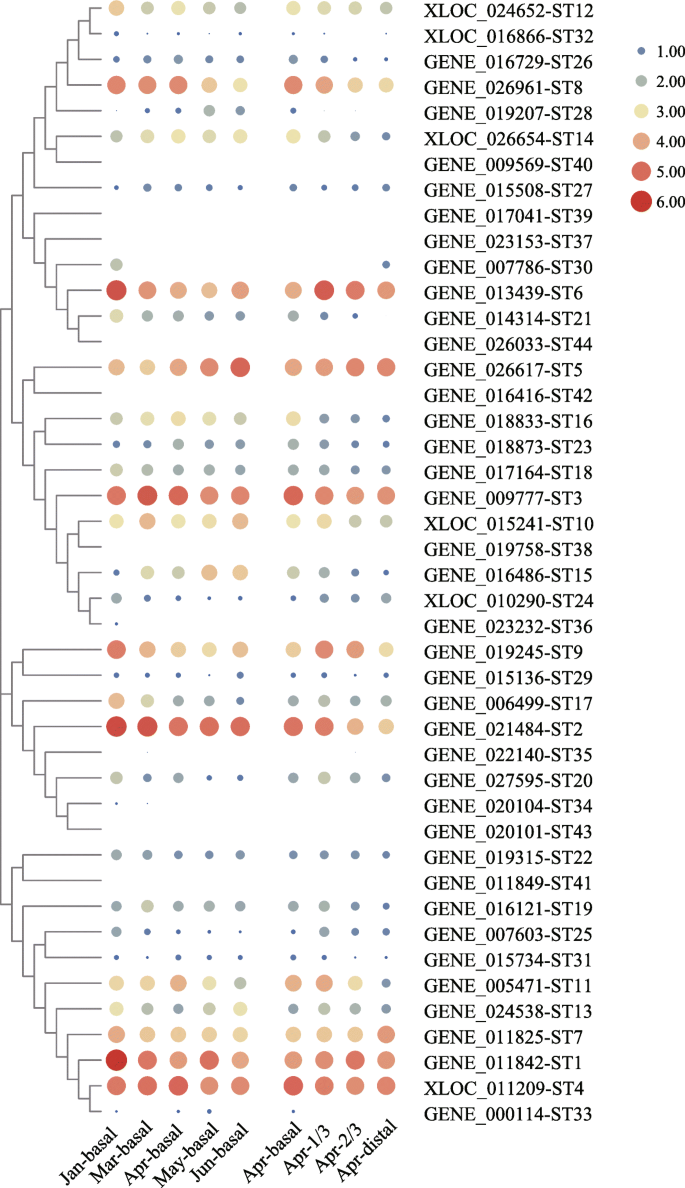
根据RNA-Seq数据,得到了英石通过TBTOOLS说明了各种组织和发育阶段下的基因(图。6).在测试的样本中,ST1.来ST36.,ST38.和ST39.至少在一个组织或发育阶段表达,和ST1.来ST10在所有样品中总是高度表达。相比之下,FPKM值ST37.和ST40来ST44一直都是零。在不同的发展阶段,ST1.,ST17和ST25明显下调,而ST5.,ST15和ST28.明显up-expressed。从基部到末端叶片,ST11.,ST15和ST21.有一个下调趋势,而且ST30.和ST24.呈现上调趋势。超过一半的STS.(54.5%)在基底部表达多于远端部。的表达水平ST13,ST16和ST19没有显著变化。值得注意的是,五对串联复制基因中有三对(ST1.,4,7;ST14,26,32;ST16,23)表现出相似的表达趋势,除了ST34.,ST43.和ST38.待检测到的表达水平太低。
RNA-SEQ资格验证
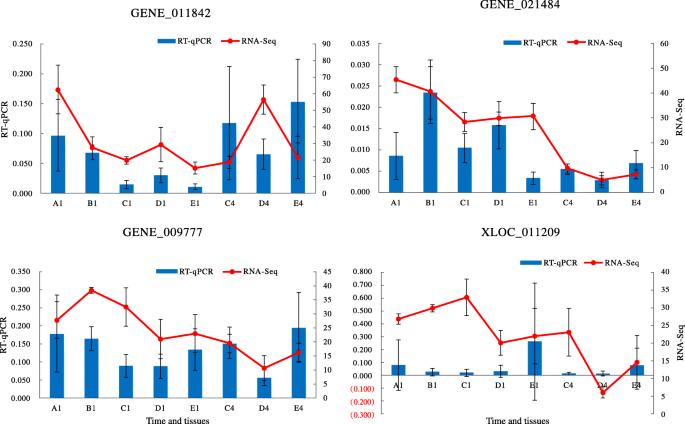
我们随机选择了三个英石基因(GENE_011842、GENE_013439和GENE_014314)进行PCR扩增。这三个的顺序STS.克隆从S. japonicacDNA模板表明,其基因组和RNA-SEQ数据库可靠(附加文件7:表S7)。采用qRT-PCR验证四个靶点的转录谱英石表达水平较高的基因(GENE_011842、GENE_021484、GENE_009777和XLOC_011209)参与岩藻糖胶生物合成。qRT-PCR结果与RNA-Seq结果一致(图2)。7).这两种结果表明我们基因组组件和RNA-SEQ数据的可靠性。
趋势分析和富集的DEGS
转录层面的趋势英石基因在S. japonica1 - 6月的基部叶片见表2.这STS.展示了几种主要的表达模式:profile0 (ST8.,ST17,ST18,ST21.和ST25), profile2 (ST6.,ST24.和ST32.),Profile3(ST1.,ST9.和ST30.), profile6 (ST12和ST20),档案25(ST11.),profile28(ST23.)和profile29(ST5.和ST28.) (桌子2).轮廓0和29是两个代表性概况,前者含有从1月到6月的下调表达模式的基因,而后者具有上调的趋势。概况0中最富集的途径包括光合作用,碳固定和代谢途径。参与核糖体,氮代谢,硫代谢和肌醇磷酸盐代谢的基因在概况29中富集(附加文件8:表S8)。
然后我们分析了其转录水平STS.在不同组织中:远端叶片、1/3、2/3和基部叶片S. japonica于四月收集(表格3.).根据我们的结果STS.展示了五种主要的表达模式:profile0 (ST14,ST15和ST23.),profile1(ST8.和ST26.),简介3(ST28.), profile4 (ST16),Profile9(ST2.,ST11.,ST19和ST21.)和简介17(ST20).最多的转录水平STS.而与基础代谢和光合作用相关的基因在这些图谱中富集(Additional file9:表S9)。
表达概况英石非生物胁迫下的基因
我们使用QRT-PCR探索变化STS.低盐和干旱胁迫下的表达水平。如图所示。8,英石基因在低盐和干旱条件下被观察到上调(图。8a和b)。这些基因在不同的处理时间点达到表达高峰。在低盐度条件下,ST44有0.5小时的最高表达水平;ST1.,ST2.,ST11.,ST12,ST31.,ST32.和ST39.在1.5 h;和ST21.在2.5小时。另外,表达水平ST1.,ST11.,ST12和ST31.在1.5 h上调4倍以上。ST12是最上调的基因,其表达水平高于1.5小时的对照组中的30倍。干旱治疗后,所有选定的表达STS.显著差异。大多数STS.显示出最高表达水平为0.5小时。ST2.和ST44在1.5小时达到峰值。的表达水平ST21.,ST31.,ST39.和ST44在0.5小时增加了4倍以上。作为最显著的上调基因,ST44在1.5 h时为对照组的67倍。在低盐胁迫下,表达量最多英石基因在1.5 h时达到峰值英石正常情况下表达量高的基因表达量较高。然而,在干旱胁迫下,最高STS.表达水平主要出现在0.5小时和STS.正常表达水平的低表达水平显示出更高的表达水平。结果表明英石对干旱的响应比低盐度胁迫更积极。
讨论
在褐藻中,磺基转移酶(sulfotransferase, ST)催化岩藻多糖生物合成中的硫酸酯化反应,决定了硫酸基的数量和位置,从而决定了岩藻多糖的各种生物活性。在这项研究中,我们检索了44个英石通过筛选基因S. japonica基因组和转录组数据库并分析了它们的结构,支架位置,系统,复制模式和表达谱,在不同组织,发育阶段和非生物胁迫下。本研究提供了有价值的信息英石基因家族,促进未来的棕海藻类功能分歧的研究。
多个内含子和可能有助于STS的分集功能
在真核节中,作为许多基因的差异剪切的主要转录物的过程以产生多个MRNA。通过选择性地保留或除去一些外显子,可以转录单个基因以产生各种蛋白质[30.].基本上,替代剪接有助于增加转录组和蛋白质组多样性,并且来自大多数功能类别的基因具有高水平的[31].内含子的选择性剪接被认为在不同的时间和空间调控基因表达[32].和....相比英石家庭拟南芥和奥雅萨苜蓿,几乎不包含内含子[33,34],内含子的数量S. japonica st.基因家庭出乎意料地高。结合在这个家庭中发现,我们推测了内含子和in的发生STS.可以赋予细胞合成多种具有不同功能和定位的ST酶。这一理论在之前对其他蛋白质的研究中已经被发现。例如,已经发现,两个交替剪接的丝氨酸-精氨酸丰富的蛋白质亚型拟南芥,由3'_plice产生,在植物开发中具有不同的生物学功能[35].在植物和动物中,作为类型的频率通过其前mRNA剪接的差异来决定[36].含有大内含子的生物体通常使用外显子定义机制,从而导致外显子跳跃[37].相应地,我们检测到约24%的外显子跳跃性(ES)位点英石基因家族,没有内含子保留(IR)。因此,如在的类型和数量STS.在一定程度上受内含子数量的影响。我们认为,多内含子和AS的产生是在黄芪的进化过程中逐渐形成的一种发育和生理策略S. japonica有效转录。
串列复制在扩展中具有重要意义英石基因家族
串联复制通常发生在染色体重组区域,形成序列和功能相似的同源基因簇,以头尾串联的方式排列在染色体上。结果,一条染色体的数量增加,另一条染色体的数量减少。这种机制在聚集基因的出现中起着不可或缺的作用[38].与这一结论一致,所有检测到的串联重复英石基因是成群出现的,而其他的英石基因往往单独出现。串联复制倾向于放大剂量不敏感基因和代谢途径顶端或末端的基因[39],它与与生物和非生物应激相关的基因的扩增密切相关[40].例如,Teng等人。(2017)发现脂氧酶基因家族的串联复制事件S. japonica[41].考虑到上述理论,串联重复事件可能在S. japonica st.基因家族扩张。
成员英石基因家族可能具有不同的蛋白质结构和功能
另一种观察是类似的基序组织只发生在相同的进化亚群S. japonica st.基因。具有高度相似基序分布的st可能产生相似的三维结构和行使相似的功能。子群间母题分布的不同可以进一步说明母题的不同功能英石基因。
在115个STs的系统发育树中,我们发现每个进化枝中都存在来自不同褐藻的STs。这与它们更紧密的进化关系是一致的。Ye et al.(2015)对vBPO基因家族的研究也有类似的报道[12].进化枝E中含有最少的stS. japonica,这可能表明基因损失的存在S. japonica st.基因家族[42].十二针大肠siliculosus和两个stN. Decipiens.聚集并形成两个独立的子组,这可能是由于它们的特殊功能或更独立的进化关系。
不同的表达模式STS.可能满足生长和发展的需要S. japonica在不同的环境中
的表达水平STS.在不同的发育阶段和组织中差异明显。考虑岩藻聚糖硫酸盐含量每月的变化[43,多种表达模式可以指示具有不同硫酸化程度的岩藻糖糖的合成。作为硫酸化多糖的浓度及其硫酸盐程度与嗜睡剂中的盐度正相关[44,我们认为这一变化可能有意义S. japonica适应沿海环境。
此前,有假说认为磺基转移酶介导的硫酸盐化作用会影响某些化合物的生物活性,从而调节生理过程以适应非生物胁迫[45].滕等。(2017)发现a的表达英石基因的大肠siliculosus在低盐度胁迫条件下上调[46].在我们的研究中,上调英石基因(ST1.,2,11,12,21,31,32,39,44)在非生物胁迫下,表明它们是一种应激抗性基因S. japonica.在褐藻中,含焦硫酸盐多糖不仅作为细胞壁基质激活,还可能在应对渗透胁迫中发挥重要作用[14,47].结合上述理论和RNA-SEQ和QRT-PCR结果,我们推断了一部分英石基因(如。ST1.,ST2.,ST3.的高度表达S. japonica在正常情况下种植,这是维持生长和发展的基本需求所必需的S. japonica.同时,其他委员(ST39.和ST44[响应于非生物应激的高度硫化,合成具有高度硫化的岩环素,虽然它们在正常情况下表现出非常低的表达水平,但显着上调。因此,这些具有强烈反应的这些基因可用作对非生物胁迫性的进一步功能性研究的关键候选基因。STS.在不同处理时间达到峰值表达水平。这种现象意味着不同的STS.有不同的反应和调节机制。各种表达模式是有益的S. japonica在低盐胁迫下维持渗透压稳定,在干旱情况下保持藻类湿润。因此,英石基因家族具有重要的意义S. japonica适应复杂多变的海洋环境。
结论
共44岁 英石通过分析基因组和转录组数据库,鉴定了可分为五个亚组的基因S. japonica.随后,从基因结构、系统发育、支架位置、二级结构、基因复制、选择性剪接和表达模式等方面分析了这些基因在不同组织、时期和非生物胁迫下的表达模式。剪接事件和内含子的选择使更多功能和位置不同的ST的形成成为可能。ST基因表达模式的变化可能与岩藻多糖硫酸化程度的月变化有关S. japonica适应沿海环境。也作为一种应激抗性基因,存在英石基因很重要S. japonica在整个发育期间适应可变的海洋栖息地。我们的报告对未来的功能验证至关重要英石基因和潜在的生物化学操作的岩藻多糖的体外未来。
方法
藻类样品收集和治疗
S. japonica2019年12月8日,在山东荣成市高鹿水产公司养殖的木筏上采集到孢子体。所有大小相似的健壮样本在10°C无光培养箱中过夜处理。低盐胁迫下,孢子体在16‰盐度海水中培养0 h、0.5 h、1.5 h和2.5 h。在干旱胁迫下,孢子体在空气中暴露0 h、0.5 h、1.5 h和2.5 h。每个时间点设置3个健壮个体为生物重复组,用过滤过的海水多次冲洗。每个切片组织标本在液氮中迅速冷冻,在−80°C保存,直到总RNA分离。
岩藻多糖生物合成途径的提取S. japonica
我们已经完成了RNA-SEQS. japonica整个发育时期的孢子体[48].基于此转录组数据(NCBI:PRJNA512328)和我们先前测序的S. japonica结果表明,在岩藻聚糖生物合成途径中鉴定了104个基因。
磺基转移酶家族成员的鉴定S. japonica基因组
我们在转录组序列注释文件中搜索关键词“sulfortransferase”。只要基因注释结果中含有“硫转移酶”,就可以选择该基因作为候选基因进行进一步鉴定。通过这种方法,我们识别了73个被自动注释为磺酸酯酶(英石)催化Fucoidan生物合成的最后一步的基因。首先,将这些73个基因提交给局部爆炸以去除冗余基因。如果两个核苷酸序列对准的结果大于99%的同一性,则我们将这两个视为重复序列,并且只保持较长的序列。其次,其余的基因被提交给智能(http://smart.embl-heidelberg.de/)[49,50]和pfam(http://pfam.xfam.org/search)[51],以斩断e值< 0.05的基因,确认保守结构域的存在磺酸酯酶域被保留。最后,我们根据基因月平均表达量对其余44个基因进行了由高到低的排序和重命名。
序列分析,染色体定位和基因复制
高可信度的开放阅读框架(orf)英石使用ORF finder预测基因(https://www.ncbi.nlm.nih.gov/orffinder/).如果检测到序列超过一个ORF,则默认选择最长的OUT。扩展的protparam工具(https://web.expasy.org/protparam/),分析推断的ST蛋白的理化性质,包括分子量(MW)和氨基酸组成(AA)。SignalP-5.0服务器(http://www.cbs.dtu.dk/services/SignalP/)用于预测信号肽[52]和tmhmm(v2.0;http://www.cbs.dtu.dk/services/tmmm/用于预测蛋白质中的跨膜螺旋。通过靶-P预测叶绿体,线粒体和细胞质的可能定位(http://www.cbs.dtu.dk/services/TargetP/)[53].
结构和保守的主题英石根据Zhao等人分析基因。[42].染色体位置英石基因是通过排列全长获得的英石核酸序列到S. japonica基因组。用TBtools显示染色体位置STS.他们的相对身体距离[54].
我们使用McScanx搜索重复基因英石家庭(55].在海藻基因组中的所有蛋白质序列成对比较。将比较结果用作输入到McScanx软件的文件,以预测重复的基因。该软件选择了默认标准,分为单例,串联,近端和分散复制,并输出结果[56,57].
识别替代拼接事件
Tophat 2.1.1用于分析44中的替代剪接事件 英石来自RNA-Seq数据的基因[58].我们提到了他的分析和分类过程。[59].
序列比对和系统发育分析
所有44 S. japonicaST蛋白由Mafft对齐[60使用默认参数,并在ESPript 3.0中显示二级结构[61].目的:分析该地区44个STs间的进化关系S. japonica,基于MEGA 7.0.26全长氨基酸序列,采用WAG+G + F模型,以1000次bootstrap复制、Gamma 4、部分缺失和50%位点覆盖率为截断值,构建最大似然(ML)系统发育树[62].
STs的氨基酸序列来源于大肠siliculosus(41),C. Okamuranus.(24),N. Decipiens.(6)和S. japonica(44)进行系统发育分析。总共115个序列的细节显示在附加文件中4:表S4。mega7.0.26以115个ST蛋白的全长氨基酸序列(1000次bootstrap复制)、WAG+F + G模型、Gamma 2、部分缺失和50%位点覆盖率为截断值构建了最大似是而非(ML)系统发育树[62].通过运行“查找最佳DNA /蛋白质模型”的Mega 7.0.26,我们分析并获得了最佳的建筑模型和相关参数,即我们用于构建系统发育树的WAG + F + G型号。
成绩单分析英石不同组织和发育阶段的基因
根据Shao等人鉴定样品中的差异表达基因(DEGS)。[48].转录档案S. japonica st.确定,获得,标准化和聚类不同组织和发育阶段的基因[48].热爱含量英石基因表达由TBtools绘制[54].
总RNA提取和cDNA合成
使用SPARKeasy多糖多酚/复合植物RNA试剂盒(SparkJade Science Co., Ltd, China)提取总RNA。使用Nanodrop 2000分光光度计(Thermo Scientific, USA)对提取的RNA进行定量。第一链cDNA合成使用SPARKscript II RT Plus Kit(带有gDNA擦除器)(SparkJade Science Co., Ltd, China),并在−20°C下保存以供后续分析。所有操作都按照制造商的说明进行。
PCR扩增和测序英石基因
随机选取GENE_011842、GENE_013439和GENE_014314 3个基因进行PCR扩增。用于扩增这三个基因全长cDNA序列的引物如表所示4.以合成的cDNA为模板进行PCR扩增。20 μL反应混合物中含有10 μL 2 × Phanta Max Master Mix (Vazyme, China), 3 μL模板,正反引物各1 μL (10 μM)和5 μL ddH2O.将反应混合物短暂离心并置于热循环仪(Takara,Japan)中。PCR的条件如下:95℃,5分钟,接着是95℃的5个循环,65℃,65℃,72℃,90s,30个循环为95℃ s, 60 °C for 15 s, 72 °C for 90 s, and a final extension at 72 °C for 10 min. The PCR products were purified using the gel-cutting recovery kit (Insight, China) and inserted into TOPO cloning vector using a 5 min TA/Blunt-Zero Cloning Kit (Vazyme, China). The 5 μL ligation mixture contained 1 μL 5 × TA/Blunt-Zero Cloning Mix and 4 μL purified PCR products (40 ng/μL). The mixture was briefly centrifuged and incubated at 37 °C for 10 min.
将每个重组质粒的5 μL与50 μL Trans1-T1噬菌体耐药化学感受态细胞(TRANS, China)混合转化。混合物是根据制造商的操作手册进行处理的。利用M13引物进行菌落PCR,质粒进行sanger测序(上海桑贡)。这三个的编码序列STS.是否在附加文件中提供7:S7表。
QRT-PCR验证靶基因
qRT-PCR验证4个基因的RNA-seq (ST1.,ST2.,ST3.和ST4.)通过不同的FPKM值检测到九个靶基因的转录水平(ST1.,ST2.,ST11.,ST12,ST21.,ST31.,ST32.,ST39.和ST44)在低盐度和干旱胁迫下。利用引物- blast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) (桌子4).
QRT-PCR在Takara Thermal Cycle Dice TM实时系统(Takara,Japan)进行。含有10μL的QRT-PCR反应5μL2×Sparkscript II RT加上主混合物(SparkJade科学有限公司,中国),1μL模板,0.2μL正向和反向引物(10μm),和3.6μLDDH.2O.用于QRT-PCR的条件如下:95℃,2分钟30 S,其次为40℃,10s和60℃的40℃,持续30秒;并且为15 s,60℃的一个95℃的一个循环,60℃和72℃。进行三种生物重复和两种技术复制。基因的相对转录水平由2计算——ΔΔCt方法(63),而β肌动蛋白用作内部参考资料[64].
可用性数据和材料
参考基因组S. japonica在全国生物技术信息中心(NCBI)的Genbank发布了Mehq0000000的登录号(https://www.ncbi.nlm.nih.gov/nuccore/MEHQ00000000.1/).RNA-SEQ数据在NCBI序列读取档案(SRA)中存放了Prjna512328的登录号(https://www.ncbi.nlm.nih.gov/bioproject/PRJNA512328).支持本文结果的所有数据集都包含在文章及其附加文件中。英石序列大肠siliculosus从(https://bioinformatics.psb.ugent.be/orcae/overview/ectsi.).英石序列N. decpiens和C. okamuranus从(https://marinegenomics.oist.jp/gallery/).
缩写
- AA:
-
氨基酸
- 度:
-
差异表达基因
- ES:
-
外显子跳跃
- 颗:
-
海藻糖激酶
- FPKM:
-
每千碱基的片段每百万映射读取
- FUT:
-
岩藻糖基转移酶
- GFPP:
-
GDP-focuse焦磷酸化酶
- GFS:
-
GDP-L-fucoidase合酶
- GM46D:
-
GDP-甘露糖4,6-脱氢酶
- 红外:
-
基因内区保留
- ml:
-
最大似然
- 英里/加仑:
-
Mannose-1-phosphate guanylyltransferase
- MPI:
-
甘露糖-6-磷酸异构酶
- MW:
-
分子量
- NCBI:
-
国家生物技术信息中心
- NJ:
-
邻接
- 子:
-
开放阅读框架
- PAPS:
-
3 -phosphosulfate -phosphoadenosine 5
- PMM:
-
磷喃胺酶
- QRT-PCR:
-
实时定量聚合酶链反应
- 英石:
-
磺酸酯酶
- TSS:
-
备选转录起始位点
- TTS:
-
备选转录末端位点
- UTR:
-
翻译区
参考文献
- 1.
Wijesinghea WAJP, Jeona YJ。褐藻中富含硫酸多糖和岩藻多糖的生物活性及其工业应用前景综述。Carbohyd石油。2012;88(1):13-20。
- 2.
Cumashi A, Ushakova NA, Preobrazhenskaya ME, D’incecco A, Piccoli A, Totani L, Tinari N, Morozevich GE, Berman AE, Bilan MI, Usov AI, Ustyuzhanina NE, Grachev AA, Sanderson CJ, Kelly M, Rabinovich GA, Iacobelli S, Nifantiev NE。褐藻中九种不同岩藻聚糖抗炎、抗凝血、抗血管生成和抗粘附活性的比较研究。糖生物学。2007;17(5):541 - 52。
- 3.
Damonte EB, Matulewicz MC, Cerezo AS。硫酸海藻多糖作为抗病毒药物。中华医学杂志。2004;11(18):2399-419。
- 4.
Azevedo TCGD, Bezerra MEB, Santos MGL, Souza LA, Marques CT, Benevides NMB, Leite EL。肝素藻类及其抗凝、出血性和血小板聚集活性。生物医学Pharmacother。2009;63(7):477 - 83。
- 5.
王志强,王志强,王志强,等Cladosiphon Okamuranus.对抗异丙苯酚诱导大鼠心肌梗死。Phytomedicine。2010; 18(1):52-7。
- 6.
王军,王芳,云辉,张辉,张启波。岩藻多糖衍生物的作用及机理Laminaria japonica实验性腺嘌呤诱导的慢性肾病。J属植物。2012; 139(3):807-13。
- 7。
徐永军,张启波,罗东东,王军,段东东。低分子量岩藻糖胶调节p选择素,减轻糖尿病肾病。国际生物医学杂志。2016;91:233-40。
- 8。
Berteau O,Mulloy B.硫酸化岩浆,新的观点:硫酸化糖糖的结构,功能和生物学性质及其对该类多糖的酶概述。糖生物学。2003; 13(6):29R-40R。
- 9。
Kylin H. Zur Biochemie der Meersalgen。Hoppe-Seyler的Zeitschrift毛皮Physoologische Chemie。1913; 83:171-97。
- 10.
旋塞JM, Sterck L Rouze P, Scornet D,艾伦AE, Amoutzias G, Anthouard V, Artiguenave F, Aury JM,獾JH, Beszteri B, Billiau K,阀盖E,博思韦尔JH,圆顶礼帽C, Boyen C, Brownlee C,卡里诺CJ,率直的B,曹GY,科埃略SM, Collen J,收款银行E Silva CD, Delage L, Delaroque N, Dittami SM, Doulbeau年代,伊莱亚斯M,萨利G, CMM G, Gschloessl B,Heesch年代,Jabbari K, Jubin C,卡瓦依H,木村K, Kloareg B, Kupper FC,朗D,保释,勒布朗C, Lerouge P, Lohr M,洛佩兹PJ, Martens C, Maumus F,米歇尔•G Miranda-Saavedra D,莫拉莱斯J,莫罗H来自T, Nagasato C,那不勒斯,纳尔逊博士,Nyvall-Collen P,彼得斯房颤,英国移民C, Potin P,也不会J, Quesneville H,读B,仁瑟SA,里特,Rousvoal S, Samanta M, Samson G, Schroeder DC, Ségurens B, Strittmatter M, Tonon T, Tregear JW, Valentin K, Dassow PV, Yamagishi T, Peer YV, winker p植物网褐藻多细胞生物的基因组与独立进化。大自然。2010;465(7298):617 - 21所示。
- 11.
Michel G,Tonon T,Scornet D,Cock JM,Kloareg B.棕色藻类的细胞壁多糖代谢异甲酸硅.真核生物中细胞外基质多糖进化的见解。新植物。2010; 188(1):82-97。
- 12.
叶乃华,张晓伟,苗敏,范旭,郑勇,徐东,王金峰,周磊,王东升,高毅,王益涛,石文英,纪鹏飞,李德明,管智,邵春旺,庄志明,高志强,齐军,赵凤清。Saccharina.基因组提供对海带生物学的新颖洞察力。NAT Communce。2015; 6:6986。
- 13.
Nishitsuji K,Arimoto A,Higa Y,Mekaru M,Kawamitsu M,Satoh N,Shoguchi E.棕色藻类的基因组草案,Nemacystus decipiens,Onna-1菌株:融合涉及硫酸化FUCAN生物合成途径。SCI批准。2019; 9:4607。
- 14.
志思,刘T,王XM,王R,王SS,王格,刘C.功能基因组学分析揭示了重要细胞成分(藻酸盐和岩藻藻)的生物合成途径Saccharina..咕咕叫麝猫。2018;64(6):259 - 73。
- 15.
张歌,邵血,李升,刘某,姚杰,段DL。棕海海藻中磷酰胺酶/磷酰荧光酶(PMM / PGM)的分子表征及生化特性Saccharina粳稻.J Appl Phycol. 2018; 30:2687-96。
- 16.
Pomin VH, Pereira MS, Valente A, Tollefsen DM, Pavão MSG, Mourão PAS。硫酸岩藻聚糖的选择性裂解和抗凝血活性:通过温和酸水解从多糖中立体特异性去除2-硫酸盐酯,制备寡糖,以及肝素辅助因子ii依赖性的抗凝血活性。糖生物学。2005;15:369 - 81。
- 17。
Fernando IPS, Sanjeewa KKA, Samarakoon KW, Lee WW, Kim HS, Kang N, Ranasinghe P, Lee HS, Jeon YJ。岩藻多糖从Chnoospora最小值;可能是脂多糖诱导的炎症反应的抑制剂。国际生物学杂志。2017;104:1185-93。
- 18。
Skriptsova AV,舍甫琴科NM, Zvyagintseva TN, Imbs TI。褐藻多糖含量及单糖组成的月变化undaria pinnatifida.(层间,phaeophyta)。J Appl Phycol。2010; 22(1):79-86。
- 19。
李波,吕芳,魏旭,赵锐。岩藻胶的结构与生物活性。分子。2008;13(8):1671 - 95。
- 20。
Morya VK,Kim J,Kim E.藻类骨藻藻:结构和大小依赖性生物活跃及其观点。苹果microbiol biotechnol。2012; 93(1):71-82。
- 21。
过硫酸盐岩藻的抗凝血和抗凝血酶活性。Carbohydr研究》1992;229(2):355 - 62。
- 22。
Chevolot L,Foucault A,Chauber F,Kervarec N,Sinquin C,Fisher Am,Boisson-Vidal C.关于棕色海藻岩石结构的进一步数据:与抗凝活动的关系。Carboohydr Res。1999; 319(1-4):154-65。
- 23。
Chevolot L,Mulloy B,Ratiskol J,Foucault A. Colliec-Jouault Sylvia。二糖重复单元是来自两种棕海藻的岩藻葡萄球菌中的主要结构。Carboohydr Res。2001; 330(4):529-35。
- 24。
yoon sj,蟒蛇,汉扬jk,mourãopas。来自棕色藻类的硫酸化生物糖昆布属植物cichorioides主要具有肝素辅助因子ii依赖性的抗凝活性。Carbohydr杂志2007;342(15):2326 - 30。
- 25。
Pereira MS, Melo FR, Mourão PAS。硫酸半乳聚糖和硫酸岩藻聚糖的结构与抗凝作用之间是否存在相关性?糖生物学。2002;12(10):573 - 80。
- 26。
浩编码碳水化合物磺基转移酶,依赖酰甘氨酸硫酸盐酶的藻类序列的系统发育,并推定硫酸酶改性因子。前植物SCI。2015; 6:1057。
- 27。
Nishitsuji K,Arinoto A,Iwai K,Sudo Y,Hisaata K,Fujie M,Arakaki N,Kushiro T,Konishi T,Shinzato C,Satoh N,Shoguchi E.棕色藻类的基因组草案,Cladosiphon Okamuranus.,S-TRACE:“Mozuku”生物学的未来研究平台。DNA Res。2016; 23:561-70。
- 28。
Hirschmann F,Krause F,Papenbrock J.植物中的多蛋白质磺基膦酶:组成,发生,底物特异性和功能。正面。植物SCI。2014; 5:556。
- 29.
Cannon SB,Mitra A,Baumgarten A,Young Nd,May G.节段性和串联基因重复在大基因家族中的演变中的作用拟南芥.植物学报。2004;4(1):10。
- 30.
可选剪接-原则,功能后果和治疗意义。中华医学杂志。2013;139(7):339-42。
- 31.
irima m,Rukov Jl,Penny D,Roy SW。可替代的基因的功能和进化分析与早期真核剪接的早期真核起源一致。BMC EVO BIOL。2007; 7(1):188。
- 32.
谢XZ,吴牛。高等植物基因中的内含子。Chin Sci Bull。2002; 47:1409。
- 33.
Klein M,Papenbrock J.多蛋白质家族拟南芥亚砜转移酶及其亲属在其他植物物种中。J Exp Bot。2004; 55(404):1809-20。
- 34.
陈荣军,蒋云云,董建林,张旭,肖海波,徐志军,高新林。水稻SOT家族基因的全基因组分析及环境响应分析(奥雅萨苜蓿).基因组的基因。2012;34(5):549 - 60。
- 35.
张Xn,Mount Sm。拟南芥SR45蛋白的两个或者拼接同种型在正常植物发育过程中具有明显的作用。植物理性。2009; 150(3):1450-8。
- 36.
Reedy Asn,Marquez Y,Kalyna M,巴塔A.植物中替代拼接景观的Omplexity。植物细胞。2013; 25(10):3657-83。
- 37.
一种包含哺乳动物基因异常剪接突变的新数据库的构建。基因。1994;141(2):171 - 7。
- 38.
关键词:真核生物,重复DNA,进化动力学大自然。1994;317(6494):215 - 20。
- 39.
植物基因含量的偏偏含量不同,不同类型的复制:串联,全基因组,节段性,或通过转置。Annu Rev植物Biol。2009; 60:433。
- 40。
Leister D.植入植物抗病基因演化中的串联和节段性基因复制和重组。趋势类型。2004; 20(3):116-22。
- 41。
滕丽华,韩文婷,范旭,徐东,张新文,白丹明,叶宁华。褐藻中原核生物样脂氧合酶家族的进化和扩展Saccharina粳稻.植物学报2017;8:2018。
- 42。
赵P,王德德,王雷克,孔Nn,张c,杨ch,wu wt,ma hl,chen q.对马铃薯的基因组分析HSP20.基因家族:识别,基因组组织和表达型材响应热应激。BMC基因组学。2018; 19(1):61。
- 43。
Hyna M,森,Anzai M,Araaki Y,Nisizawa K.葡萄糖含量的每月变化,其成分糖和硫酸盐培养Laminaria japonica.水生物学。1999年; 398-399:411-6。
- 44。
Aquino Rs,Grativol C,MourãoPas。从海中升起:硫酸化多糖与植物盐度之间的相关性。Plos一个。2011; 6(4):E18862。
- 45。
Marsolais F,Gidda SK,Boyd J,Varin L.植物可溶性磺当样:与哺乳动物酶的结构和功能相似性。最近的Adv Phytochem。2000; 34:433-56。
- 46。
腾吕,风扇X,徐D,张XW,模拟T,YE NH。阳性选择下基因的鉴定揭示了棕藻种类之间进化适应的差异。前植物SCI。2017; 8:1429。
- 47。
黄志强,王志强,王志强,等。褐藻细胞壁的化学和酶法分离:对褐藻胞外基质结构的深入研究。安Bot-London。2014年,114(6):1203 - 16。
- 48。
邵志荣,张培培,卢超,李绍新,陈振华,王小林,段东东。转录组测序的Saccharina粳稻整个发育期间的孢子体揭示了藻酸盐和甘露醇生物合成的监管网络。BMC基因组学。2019; 20:975。
- 49。
SMART: 2015年的最新更新、新发展和现状。核酸Res. 2015;43(D1): D257-60。
- 50。
Letunic I, Bork P. 20年SMART蛋白域注释资源。核酸Res. 2018;46(D1): D493-6。
- 51。
El-Gebali S, Mistry J, Bateman A, Eddy SR, Luciani A, Potter SC, Qureshi M, Richardson LJ, Salazar GA, Smart A, Sonnhammer ELL, Hirsh L, Paladin L, Piovesan D, Tosatto SCE, Finn RD. 2019年Pfam蛋白家族数据库。核酸Res. 2019;47(D1): D427-32。
- 52.
Armenteros JJA, Tsirigos KD, Sønderby CK, Petersen TN, Winther O, Brunak S, Heijne GV, Nielsen H. SignalP 5.0利用深度神经网络改进信号肽预测。生物科技本质》。2019;37(4):420 - 3。
- 53.
Emanuelsson O,Brunak S,Heijne GV,Nielsen H.使用TargetP,SignalP和相关工具将蛋白质定位。NAT PROTOC。2007; 2(4):953-71。
- 54.
TBtools是一种集成了多种生物数据处理工具和用户友好界面的生物学家工具包。BioRxiv。2018年,48。
- 55.
王yp,唐·赫伯,黛巴里·jd,tan x,李jp,王xy,李th,金赫兹,玛勒b,郭h,kissinger jc,paterson啊。McScanx:一种用于检测和进化分析的工具包,对基因同步和共线性分析。核酸RES。2012; 40(7):E49。
- 56.
段WK,黄泽,宋XM,刘特克,刘HL,侯XL,李玉。综合分析综合分析和果胶甲基酯酶基因芸苔属植物拉伯在不同的进化模式上闪光灯。SCI Rep-UK。2016; 6:25107。
- 57.
黄志仁,段文坤,宋晓明,唐杰,吴鹏,张波,侯小林。生长素/吲哚乙酸和生长素反应因子基因家族的保留、分子进化和表达差异Brassica Rapa.阐明它们在植物中的进化模式。基因组生物学进展。2016;8(2):302-16。
- 58.
Trapnell C,Pachter L,Salzberg SL。Tophat:发现带RNA-SEQ的接头连接点。生物信息学。2009; 25(9):1105-11。
- 59.
他B,胡Zh,Ma L,Li Hr,Ai MQ,韩吉,Zeng B.转录数组分析不同增长阶段Aspergillus oryzae.揭示不同种类的基因在生长过程中的动态变化。BMC Microbiol。2018;十八12。
- 60.
kaoh K, Standley DM. MAFFT多序列比对软件版本7:性能和可用性的改进。生物医学进展。2013;30(4):772-80。
- 61.
Robert X,Gouet P.用新的Andick服务器解密蛋白质结构中的密钥特征。核酸RES。2014; 42(W1):W320-4。
- 62.
MEGA7:用于更大数据集的分子进化遗传学分析7.0版本。生物化学学报。2016;33(7):1870-4。
- 63.
Schmittgen Td,Zakrajsek Ba,Mill Ag,Gorn V,歌手MJ,Reed MW。定量逆转录 - 聚合酶链反应研究mRNA衰减:终点和实时方法的比较。肛门生物化学。2000; 285:194-204。
- 64。
邵志荣,张培培,李启元,王小林,段东东。甘露醇-2-脱氢酶的表征Saccharina粳稻:新的多元醇特异性长链脱氢酶/还原酶的证据。《公共科学图书馆•综合》。2014;9 (5):e97935。
致谢
在此,我们要感谢Gene Denovo Biotechnology Co.的李亚亚和焦腾龙两位技术人员在生物信息学分析方面的帮助。我们感谢TopEdit有限责任公司在本手稿的准备过程中对语言的编辑和校对。
资金
该工作得到了中国天然科学基金(第41806175号),海洋生物学和生物技术实验室青年基金会(YQ2018NO06),山东重点研发计划(2018GHY115023),中国与日本联合研究项目(号2017YFE0130900)。这些资助者提供了购买仪器和试剂进行研究的财政支持。每个融资机构都参与了对数据的研究和收集,分析和解释的设计以及稿件的写作。
作者信息
隶属关系
贡献
CL, ZS和DD构思和设计了研究。CL和ZS进行了实验。PZ对海带样品的培养和采集有一定的作用。CL在Gene Denovo Biotechnology Co的支持下进行了生物信息学分析。CL解释了数据并撰写了手稿。ZS和DD对手稿进行了批判性的修改。所有作者均已阅读并批准本稿件。
相应的作者
道德声明
伦理批准和同意参与
S. japonica从山东荣成高鹿水产公司养殖的木筏中采集到“燕杂”孢子体。所有样品收集活动都得到了公司负责人的批准。段德林教授对我们研究中使用的植物材料进行了正式鉴定。其余样品保存在我们实验室−80°C冰箱中。作者声明符合机构、国家和国际指南。该实验对环境友好,对当地生态系统没有负面影响。
同意出版物
不适用。
利益争夺
提交人声明他们没有竞争利益。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
参与Fucoidan的生物合成的基因粳稻。
附加文件2:表S2。
ST蛋白的亚细胞定位预测。
附加文件3:表S3。
ST蛋白的假定基序。
附加文件4:表S4。
来自所有所选物种的ST氨基酸序列用于系统发育分析。
附加文件5:表S5。
中可选剪接位点的类型S. japonica st.所有样本的基因。
附加文件6:表S6。
st类次生结构的比例S. japonica.
附加文件7:表S7。
克隆和测序基因的测序结果。
附加文件8:表S8。
转录谱富集途径S. japonica不同的发展阶段。
附加文件9:表S9。
转录谱富集途径S. japonica不同的组织。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Lu,C.,Shao,Z.,张,P。et al。全基因组分析Saccharina粳稻全生育期和非生物胁迫下的硫转移酶基因及其转录谱。BMC植物杂志20,271(2020)。https://doi.org/10.1186/s12870-020-02422-3
收到了:
接受:
发表:
关键字
- Saccharina粳稻
- 磺酸酯酶
- 岩藻糖糖
- 转录组
- 基因组