摘要
背景
MicroRNAS是植物免疫系统中的重要基因表达调节剂。曲霉菌曲藤是花生中黄曲霉毒素污染最常见的致病因子,但花生-答:flavus缺乏互动。在该研究中,使用抗性品种(GT-C20)和易感品种(Tifrunner)来研究MiRNA的调节作用答:flavus增长.
结果
共30个miRNA, 447个基因和21对潜在miRNA/mRNA表达显著差异答:flavus.总共62 miRNA,451个基因和44个潜在的miRNA / mRNA对在两个花生品种之间表现出差异表达曲线。基因本体(GO)分析表明,富集了代谢过程相关的GO条款。基因和基因组(Kegg)途径(Kegg)途径的京都百科全书进一步支持了GO结果,其中许多富集的途径与生物合成和代谢有关,例如二次代谢物和代谢途径的生物合成。小RNA的相关分析,转录组和降低组表明,MiR156 / SPL对可能调节抗性和易感基因型中黄酮类化合物的积累。MiR482 / 2118家族可能调节NBS-LRR基因,其在抗性基因型中具有更高的表达水平。这些结果提供了有用的信息,以进一步了解MiR156 / 157 / SPL和MIR482 / 2118 / NBS-LRR对的作用。
结论
本研究对花生抗病品种和敏感品种的转录组、miRNAome和降解组进行了整合分析。获得的知识将有助于理解花生mirna在应答中所起的作用答:flavus.
背景
花生(落花生hypogaea.花生是世界上最重要的油料作物之一。它在亚洲、非洲和北美广泛种植。中国、印度和美国是世界上主要的花生生产国。全球花生总产量约为每年2900万吨(http://www.peanutsusa.com),估值约为350亿元[1].然而,花生生产容易受到黄曲霉毒素污染的威胁,这是由感染曲霉菌曲藤和曲霉属真菌寄生[2].当黄曲霉毒素污染的花生引起英国大量火鸡的死亡时,早于1960年首次揭示了黄曲霉毒素。黄曲霉毒素,可引起宿主细胞中的DNA损伤和染色体不稳定性,具有高毒性和最致癌物质中的一种[3.,4,5].我国花生黄曲霉毒素污染严重,限制了花生的出口。黄曲霉毒素污染可能发生在田间、收获、储存和加工过程中,因此,黄曲霉毒素污染很难预防和控制。
虽然这是一个广泛存在的问题,花生的潜在分子决定因素和机制答:flavus相互作用仍然难以捉摸。分析花生与真菌互作的分子机制,挖掘花生的抗性信息,是培育抗病花生品种的基础。为了更好地了解这一机制,我们构建了抗性品种(GT-C20)和感病品种(Tifrunner)花生种子发育的cDNA文库,共生成了21777条EST序列[6].不久之后,花生寡核苷酸芯片被用于鉴定抗性基因答:flavus和答:寄生共发现抗性品种中62个基因表达上调,22个基因推定表达曲霉属真菌与易感品种相比,抗性栽培品种较高 -1].为了鉴定与黄曲霉毒素污染抗性相关的蛋白,采用二维电泳、质谱和实时RT-PCR方法,鉴定了12个潜在的不同表达蛋白[7].另一组也使用差异蛋白质组学方法,并确定了响应的蛋白质阵列答:flavus感染(8].最近,Wang等人利用Next Generation Sequencing研究花生抗病和易感基因型的基因表达谱答:flavus增长(9,10].这些研究提供了有价值的信息,了解花生响应黄曲霉毒素的生产。然而,由于缺乏花生基因组序列,这些研究很难提供对花生的转录组变化的全面解释答:flavus.最近,二倍体野生花生全基因组序列的公布,A. Duranensis.和答:ipaensis[11,12]和栽培品种,Tifrunner和Shitouqi(https://peanutbase.org/,http://peanutgr.fafu.edu.cn/)提供新的机会,以应对花生抵抗机制答:flavus.
microRNA(miRNA)是一类非编码RNA调节基因表达,并在植物开发中发挥作用,并对应力的反应[13,14,15].到目前为止,已经从近70种植物(http://www.mirbase.org/).广泛探索了生物应力响应中植物miRNA的功能。例如,MiR398通过靶向Cu / Zn超氧化物歧化酶在氧化应激条件下调节植物生产率[16,17].在番茄中,通过干旱胁迫诱导miR169的积累。其靶基因的表达,核因子y亚基基因和多药抗性相关蛋白基因,通过干旱胁迫显着下调。MIR169的过度表达增强了番茄的耐旱性[18,19].许多转录因子,如SPL [20.,21], MYB [22),南京(23,24,25], ARF [26], HD-Zip [27]和ap2 [28,29]是miRNA的目标,暗示在基因调节中位于网络的核心位置的miRNA。
最近,MiRNA在植物疾病抗性中的作用逐渐被发现,并且米兰纳斯被认为在植物天然免疫系统中发挥关键作用[30.,31].in.拟南芥据证明,MiR472 / RDR6沉默途径调制的PAMP和效应器通过转录后控制触发免疫(ETI)[32].在水稻中,多个miRNA参与抗爆炸真菌的抵抗力Magnaporthe oryzae。一组水稻mirna在其上进行了差异表达m . oryzae感染。miR160a和miR398b的过表达增强了抗性m . oryzae在转基因水稻中[33].多种miRNA靶基因与抗病性有关。例如,miR482和miR2118的表达和变化反映了植物NBS-LRR防御系统的平衡的转变[34].在我们以前的研究中,我们使用高通量测序方法识别许多已知和新的miRNA在花生中[35].我们发现,诱导或抑制了一些已知和新的miRNA的表达Ralstonia solanacearum栽培和野生花生的感染。降解组测序结果可能表明部分防御反应基因被miRNA降解[36].这些数据表明,miRNA可能在花生疫病抵抗中发挥重要作用。
在这项研究中,我们旨在识别潜在的答:flavus敏感的mirnas及其在花生的目标,并研究他们在花生的角色 -答:flavus互动。为此目的,我们采用了高通量测序技术,同时分析miRNA和mRNA表达谱及其调节以响应答:flavus使用抗性和易感花生品种的生长。我们的结果为了解花生抗性的分子机制提供了有价值的信息答:flavus.
结果
花生品种GT-C20和Tifrunner的抗性差异答:flavus
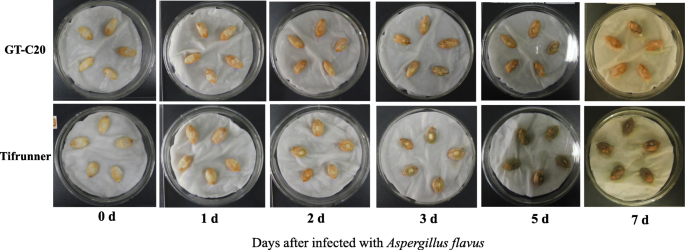
花生品种GT-C20和Tifrunner的抗性答:flavus被验证。因此,在接种后的2天内,只在Tifrunner的种子中观察到菌丝,而在GT-C20中观察不到菌丝。1).从三个戴,在GT-C20和Tifrunner都非常清楚。在Tifrunner的种子中观察到比GT-C20中的种子更多的菌丝(图。1).统计分析表明,GT-C20对血吸虫病高抗性(感染指数为4.7),Tifrunner对血吸虫病高敏感(感染指数为95.3)答:flavus按报告的标准增长[37].收集两种DAI的样品,用于miraome和转录组分析。
miRNA的高通量测序和识别
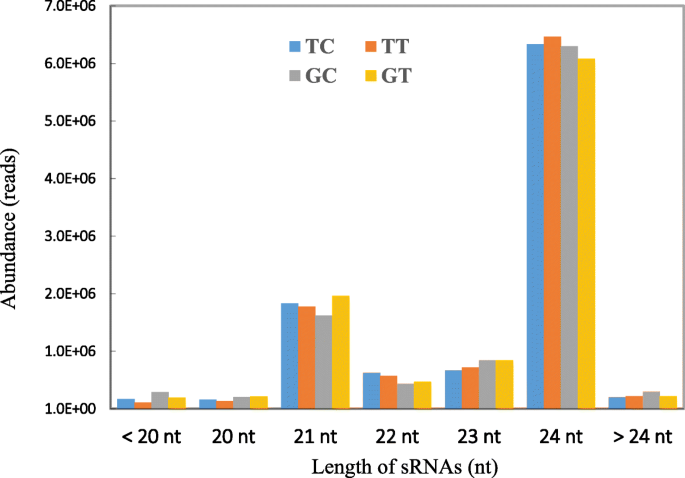
研究花生mirnas的作用答:flavus利用Illumina HiSeq2000平台构建8个小RNA库并测序。这些小RNA库包括两个答:flavus-treated(gt1和gt2)和两个控制(gc1和gc2)耐种品种gt-c20和两个答:flavus接种敏感品种Tifrunner (TT1和TT2)和两个对照(TC1和TC2)文库。测序之后,从8个库中总共获得了111,839,986个原始读和111,324,210个干净读(附加文件:表S1).标准化的清洁读数用于分析小RNA分布。大多数清洁读数的长度为20-24个核苷酸(NT),分别占TC,TT,GC和GT文库中所有清洁读数的96.22,96.67,94.06和95.81%(图。2).24nt小rna是最丰富的一组,其次是21nt小rna。这些结果与之前花生小RNA测序的研究一致[35,36,38].
超过85.5%独特的小RNA和86%的总小RNA被映射到花生基因组(A. Duranensis.和答:ipaensis,https://peanutbase.org/)使用SOAP2软件(附加文件:表格S2).通过对miRNA数据库(miRbase 21.0,http://www.mirbase.org/),我们鉴定出113个已知的miRNA,属于30个miRNA家族(附加文件:表)S3).此外,清洁小RNA也用于爆炸与GenBank和RFAM数据库爆炸,以删除RRNA,TRNA,Snornas和SNRNA(附加文件:图S1).其他小RNA用于根据先前研究描述的标准探索新的MIRNA [35,39,40].总共确定了67个新型miRNA,从20 nt到23 nt,它们的前体发夹结构的负折叠自由能量从184.8到19.1 kcal / mol(附加文件:表格S4).
不同花生品种对mirna表达的响应答:flavus感染
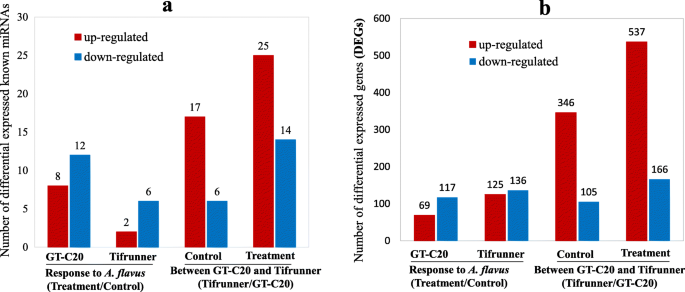
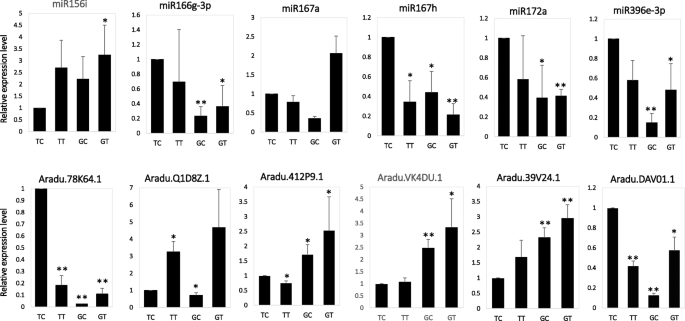
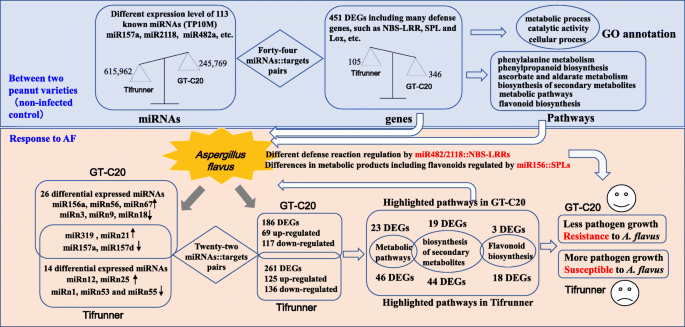
以确定花生中对答:flavus,差异表达的mirna被鉴定在一个显著水平(调整p-value < 0.05, fold change≥1.5),通过TP10M (tag per ten million)比较标准化miRNA表达水平。在花生中鉴定的113个已知mirna中,8个和2个表达上调,12个和6个表达下调答:flavus在GT-C20和Tifrunner中,3.一个,附加文件:表格S3).GT-C20中已知差异miRNAs的总数(20)多于Tifrunner中已知差异miRNAs的总数(8)。在这些差异的已知miRNAs中,miR319的表达在GT-C20和Tifrunner后均增加答:flavus接种。虽然miR157a和mir157d的表达在处理后GT-C20和TIFRUNNER均降低答:flavus.此外,MIR157A在所有已知的miRNA中显示出最高的表达丰度(附加文件:表格S3).也有部分mirna表现出不同的表达趋势答:flavus在不同花生品种中。例如,miR156a在GT-C20中表达增加,以响应答:flavus,但在Tifrunner中保持不变(附加文件:表格S3).此外,鉴定了12个差异表达的新型miRNA以响应于答:flavus,包括6个来自GT-C20的新mirna和6个来自Tifrunner的新mirnaS4).在GT-C20,miRn21,miRn56和miRn67被诱导或特异性响应于表示答:flavus和mirn3,mirn9,mirn18响应于答:flavus.在Tifrunner中,miRn12、miRn21和miRn25被诱导或特异性表达答:flavus,并且令人沮丧地抑制mirn1,mirn53和mirn55答:flavus(附加文件:表格S4).为了确认测序结果,选择六种miRNA(miR156i,mir166g-3p,miR167a,mir167h,miR172a和mir396e-3p)用于茎环qRT-PCR分析(图。4).这些miRNA的表达方式符合深度测序数据(附加文件:图S2).
比较了两个不同品种间的miRNA丰度。接种前,GT-C20的113个已知mirna的丰度均为245,769 (TP10M),低于Tifrunner(615,962)。接种后,GT-C20 (269,075.54 TP10M)中113个已知mirna的丰度也均低于Tifrunner (442,962.68 TP10M)S3).在调整的准则下p值(Padj)<0.05和折叠变化≥1.5,在控制和控制中,共鉴定了两种品种之间的23和39种差异的已知的miRNA。答:flavus(图5)3.a)。许多已知的miRNA在控制和控制中显示出比在GT-C20中的更高表达水平。答:flavus感染样本。例如,在对照样本中,mir - 157a在GT-C20中的表达量为52,711 TP10M,在Tifrunner中的表达量为291,784 TP10M。在治疗样本中,mir - 157a-5p在GT-C20中的表达量为32146 TP10M,在Tifrunner中的表达量为154326 TP10M(附加文件:表)S3).此外,在GT-C20中miR2118和miR482a的表达水平也低于Tifrunner(附加文件:表)S3).
通过高降解组分析鉴定mirna的目标
为了验证花生miRNAs的靶标,我们将降解池与TT、TC、GC和GT的相同样本混合,通过高通量测序,获得了14,338,349个原始reads,包含5,477,891个unique reads。使用SOAP2程序将所有reads与花生基因组和转录组进行比对。结果显示,花生参考基因组和转录组中分别有9,580,557条(66.82%)和6,271,065条(43.73%)reads被成功定位S5).根据每个已占据转录本上的签名数和推定的剪切位置的丰度,这些被切割的转录本可以根据每个已占据转录本上的签名丰度(0、1、2、3和4)分为5类[41,42,43].在我们的数据集中,分别分为0,1,2,3和4类别(附加文件:表格)分类为249,165,554,175和402候选目标S6).共有93和176个候选目标,分别为已知和新的miRNA(附加文件:表格)S7-S8).我们发现许多防务相关的基因可能通过miRNA的切割。例如,两个PPR含有重复蛋白基因,Aradu.7H0DM.1和Aradu.4L72B.1被潜在地由miR3514和miRn10,分别为(附加文件裂解:表S8).
花生的整体mRNA表达谱答:flavus增长
研究靶基因表达的变化响应答:flavus利用与miRNA研究相同的样本构建了8个转录组文库。通过转录组测序,共生成97,021101个原始reads,平均为∼每个库1200万次读取(附加文件:表S9).大约77.83%的读数成功映射到花生参考基因组,映射到多个区域约8.18%的读数(附加文件:表格S9).在花生基因组数据库中,40,636个基因来自A. Duranensis.(一个基因组)和46,984个基因答:ipaensis(B基因组)被沉积。在我们的RNA-SEQ数据中,30,254(74.45%)基因A. Duranensis.34172个(72.73%)答:ipaensis这些基因均在花生种子中表达。
不同表达的基因对答:flavus
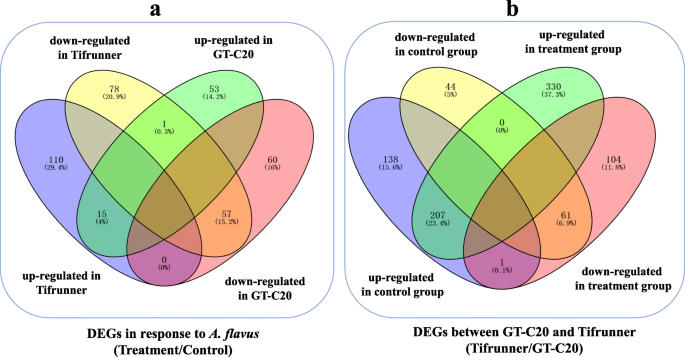
我们进一步研究了花生的整体基因表达谱答:flavus.通过应用概率≥0.8的截止标准和记录2(倍数变化)| ≥ 1, a total of 261 (125 up-regulated and 136 down-regulated) and 186 (69 up-regulated and 117 down-regulated) differentially expressed genes (DEGs) were identified in response to答:flavus分别来自Tifrunner和GT-C20(图。3.b、附加文件S3).在这些deg中答:flavus其中,在GT-C20和Tifrunner中均有15个deg表达上调,在GT-C20和Tifrunner中均有57个deg表达下调(图2)。5a).此外,功能注释结果显示,447个DEGs中有139个(31.09%)是防御相关基因,如几丁质酶、热休克蛋白、WRKY家族转录因子和温度诱导的脂钙素等(表)1).此外,我们发现吲哚-3-乙酸诱导2基因(ARAIP.AN5V8.1)表现出相反的表达趋势,以响应答:flavus.在抗性品种GT-C20中表达上调,而在感病品种Tifrunner中表达下调。5a)。几次QRT-PCR分析(Aradu.78 K64.1,Aradu.Q1D8Z.1,Aradu.412P9.1,Aradu.vk4du.1,Aradu.39 V24.1和Aradu.dav01.1)证实了RNA-SEQ结果(图。4、附加文件图S2).
差异表达基因的Venn图响应于答:flavus(一个)和两种花生品种(b).使用在线软件Venny进行Venny分析2.1(http://bioinfogp.cnb.csic.es/tools/venny/index.html)
花生抗病与感病品种差异表达基因
我们进一步分析了两种花生品种之间的基因表达变化。通过与对照样品(GC和TC)的比较,鉴定了451℃(346℃上调和105个下调)(图。3.b)。对于处理样品(GT和TT),鉴定了703℃(上调和166个下调)(图。3.b).部分疾病相关基因在两个品种中表达水平不同。例如,一个NBS-LRR抗病基因(Araip.WF303.1, log2TC/GC = 9.47, log2TT / gt = 4.66)在Tifrunner中高度表达,但在GT-C20中具有较低的表达水平。同时,另一个NBS-LRR编码基因(Aradu.168 L7.1)显示出相反的表达趋势,高度表达在GT-C20中,但在TiFrunner中具有较低的表达水平(图。3.b)。
DEGs的GO和KEGG通路
进行对DEG的分析,并且可以将DEGS分为三种类别,包括分子功能,细胞组分和生物过程。在细胞组分类别中,“细胞”和“细胞部分”表示顶部术语。对于分子函数的类别,富集“催化活性”的GO期。对于生物过程的类别,最丰富的术语是“代谢过程”和“细胞过程”(图。6a).对照样品与处理样品之间的差异也进行了分析。两个花生品种在对照样品和处理样品中的许多氧化石墨烯项存在差异,如“代谢过程”、“催化活性”和“细胞过程”(图2)。6b)。
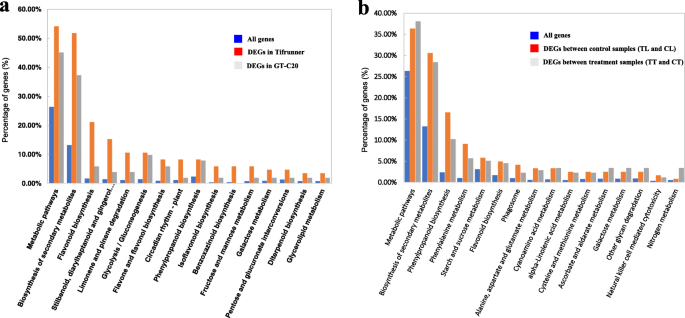
为了更好地了解其功能和基因调控网络,我们进行了KEGG分析。deg的回应是答:flavus分别在Tifrunner和GT-C20中被划分为53和41条KEGG通路(图2)。7,表S10- - - - - -11).最重要的16个富集的途径可能会受到监管答:flavus综述了(图7一个,表格S10).富集途径多与生物合成和代谢有关,如“次生代谢产物的生物合成”、“类黄酮的生物合成”、“二苯乙烯、二芳基庚酸”、“姜酚的生物合成”、“代谢途径”、“柠檬烯和蒎烯的降解”等。有趣的是,我们观察到,在大多数富集途径,包括“次生代谢产物的生物合成”和“代谢途径”中,Tifrunner的DEGs数量都高于GT-C20,这表明两个品种的代谢活性存在差异。有趣的是,“次生代谢产物的生物合成”、“苯丙氨酸的生物合成”、“苯丙氨酸的生物合成”、“代谢途径”、“类黄酮的生物合成”、“抗坏血酸和醛达酸的生物合成”等生物合成和代谢途径也得到了丰富。KEGG结果也支持了两个花生品种之间代谢活性的差异(图。7B,表S11).
mirna表达谱与靶基因的相关性分析
来指定mirna在响应答:flavus生长,分析了miRNA和靶标的表达谱。回应答:flavus感染,鉴定了二十二个潜在的miRNA /靶对(表S12).降解组测序显示,这些靶基因被mirna切割。然而,与这些靶点对应的mirna表达谱并非完全阴性。例如,在Tifrunner中,只有5对mirna /targets表现出一对一的对应关系,其中5对mirna的表达上调/下调,2对mirna的表达下调/上调答:flavus增长。另外,5对mirna /targets对在Tifrunner后表达趋势相反答:flavus感染。对于TIFRUNNER中的其他miRNA /靶对,只有MIRNA或目标才显示出差异表达答:flavus,并且没有观察一对一对应的表达。
在两个花生品种之间,鉴定了44对差异表达的mirna /targets对(见表)向).我们进一步比较了两种花生品种之间的miRNA /靶对的一对一表达对应关系。在控制样本和答:flavus在感染样本(GC和TC)中,只有16对mirna /targets对同时观察到差异表达的mirna和靶标,而这些结果在其他mirna /targets对中未观察到。在这16对miRNA /targets中,miRNA与targets表达呈负相关。其余5个miRNA /targets的表达趋势一致,均为上调或下调。为flavus在14对mirna /targets中同时观察到差异表达的mirna和靶标,其中10对mirna和靶标表达呈负相关。
讨论
揭示花生抗病机制的策略
种植花生(落花生hypogaea.l)是一种异丙醇(2n = 4x = 40,AABB)有机体,其作为两个古代二倍体物种的杂交,可能是A. Duranensis.(基因组)和答:ipaensis(b基因组),然后是自发的全基因组复制[44,45,46].首先使用表达的序列标签(EST)来获得基因序列信息及其对花生中生物和非生物胁迫的反应[47,48,49].基于EST测序和微阵列技术,数十种不同的基因反应答:flavus鉴定感染,提供了理解花生抗性的分子机制的有用信息答:flavus[1,6].最近完成了两个二倍体野生花生品种的全基因组测序(http://peanutbase.org/).这一成就将大大促进这一主要作物的基因发现和遗传改善。在这里,我们使用高通量mRNA和miRNA测序来研究花生的基因表达和调节答:flavus感染。我们的数据涵盖了超过64,426个基因的表达信息。这是花生全基因组基因表达谱研究的首次报道答:flavus.此外,MRNA和MIRNA的综合分析可能会有所了解,了解在对抗抵抗力的调节中的花生中的miRNA监管答:flavus。
花生抗病系与感病系防御相关基因的差异表达
在本研究中,在GT-C20和Tifrunner中鉴定了总共447个差异表达基因(DEGS),响应于答:flavus(无花果。4).功能注释结果显示,139个基因为防御相关基因(见表)1).在花生品种中,大多数防御相关的DEG(139分)差异表达,而只有七只数差异仅在一个花生品种中差异表达答:flavus.
几丁质是真菌细胞壁的重要组成部分,被认为是维持真菌致病性的重要因素。几丁质酶是具有降解几丁质活性的水解酶。越来越多的证据表明,几丁质酶的活性对植物抵抗病原菌侵染具有重要意义[50,51,52],一些几丁酶是致病相关(PR)蛋白[53,54,55].我们观察到几丁质酶基因(Araip.8C3IU.1)在Tifrunner中表达无变化,但在GT-C20中表达。此外,我们的数据显示,两种过氧化物酶(Aradu.BNR06.1和Araip.595JK.1)和两种铜胺氧化酶(Aradu.NCJ0H.)。1和Araip.26B5V。1were up-regulated in GT-C20 but no significant expression difference in Tifrunner (Table1).有趣的是,过氧化物酶和蓝铜蛋白基因也是疾病相关基因[56,57].在植物,过氧化物酶是通过参与杀菌剂和木质素生物合成参与植物抗病性反应。铜胺氧化酶有助于催化氧化脱胺化的聚胺和产生过氧化氢(H2O2).在植物中,H2O2来源于多胺氧化介导的细胞死亡,并与对病原体入侵的应激反应相关[58,59].植物细胞壁被认为是防御的第一道安全屏障系统对细菌和真菌病原体。果胶是植物细胞壁的重要成分。在植物中,果胶酯酶/果胶酯酶抑制剂作用经由果胶的沦落修改细胞壁。在我们的数据集中,二果胶/果胶酯酶抑制剂基因(Aradu.S6DQM.1和Araip.EQZ9W.1)在Tifrunner被显著压下但响应于没有显著表达差异答:flavus(表1).这里,这些基因在抗性和易感花生品种中的差异表达趋势为我们提供了一种有用的线索,以揭示花生响应的分子机制答:flavus.
在这139个防御相关的DEG中,18个基因编码细胞色素P450超家族基因(表1).细胞色素P450作为一种多功能基因,在许多植物中都发现了它在防御反应中的作用[60,61].在大米中,过度表达CYP71Z2增强了对白叶枯病的抵抗力。进一步的研究证实,细胞色素P450基因通过介导二萜类植物抗毒素积累和抑制水稻IAA信号传导来增强水稻抗病能力[62].
NBS-LRR基因代表植物中最大的抗病基因系列中的一种[63].到目前为止,通过图位克隆发现的抗病基因大部分是NBS-LRR基因,如抗稻瘟病[64,抗白粉病基因PM3 / PM8在小麦[65,抗叶锈病基因Lr10在小麦[66].我们发现两种NBS-NBS-LRR型疾病抗性基因(Aradu.168 L7.1,araip.vgw7f.1)在花生品种中诱导。然而,两种基因的表达水平的增加在抗性基因型中比易感基因型花生更显着。例如,对照样本中的两种品种,Aradu.168 L7.1的表达水平几乎相等(日志2TC / GC.= 0.11)。回应答:flavus,这种基因的表达在抗性(对数)中诱导2GT / GC.= 3.28)和易感花生(log2TT / TC.= 0.68)基因型花生。治疗后,答:flavus,这种Aradu.168 L7.1在抗性基因型中的表达显着高于易感基因型(日志)2GT / TT= 2.72)。Araip.VGW7F。1had the same expression patterns with that of Aradu.168 L7.1, which was induced in both peanut varieties but were more dramatically induced in resistant genotype (log2GT / TT = 2.84). Aradu.168 L7.1 and Araip.VGW7F.1 located in chromosome A05 (1880820–1,893,586) and B05 (1716985–1,720,472) of peanut, respectively, and were considered as orthologous genes. Interestingly, sequence alignment results showed that Aradu.168 L7.1 (Expect: 5e-146, Identities: 316/943(34%) and Positives: 506/943(53%)) and Araip.VGW7F.1 (Expect: 2e-119, Identities: 292/931(31%) and Positives: 490/931(52%)) were all homologous with the disease resistance locus,RPM1.,拟南芥。的RPM1.基因座赋予对细菌病原体的抗性两在拟南芥67,68]并且应该在豆,豌豆和其他作物物种中具有功能保护[69].
此外,其他几种防御相关基因也显示出两种不同的花生基因型之间的差异表达曲线,包括两个纤维素合成酶,如E1基因,两种衣物族转录因子基因,两个脂肪酶/脂氧合酶(LOX)基因和一种吲哚-3-乙酸诱导型2基因(表格1).WRKY代表最大的转录因子基因家族之一。一些WRKY基因家族成员已经被报道在调节响应各种生物和非生物胁迫。在这里,我们发现有两个WRKY基因,Aradu.S7YD6和Araip.RC4R7,位于A06染色体和B06,分别与同源WRKY33.其他植物的基因。in.拟南芥,WRKY33是抵抗坏死营养真菌病原体所必需的。在我们的数据集中,上述两个WRKY基因在Tifrunner中上调,而在GT-C20中保持不变,这似乎与其他植物的预期相反。此外,我们还发现了两个纤维素合成酶类E1基因(Aradu。BZH82, Araip.FL29A)在Tifrunner中表达上调,而在GT-C20中表达无显著差异答:flavus。细胞壁是植物对抗真菌的重要屏障,这似乎与其他植物的预期相反.以前的研究表明,脂氧合酶途径与种子抗性有关答:flavus大豆和玉米。例如,使ZmLOX3可以增加玉米对曲霉属真菌.这里,另外两种脂肪酶/脂氧合酶(LOX)基因,ARADU.MAS03和ARAIP.081EX在TIFRUNNER中调节,但在GT-C20中不变。此外,吲哚-3-乙酸可诱导基因响应于其他植物中的生长素刺激和侧向根系形态发生。在这里,我们发现吲哚-3-乙酸诱导的2基因(ARAIP.AN5V8.1)在耐腐蚀性和下调的抗性中升高.
mirna在花生抗寒反应中的调控答:flavus
由于序列相似性,MiR2118被认为是MiR482超家族的成员[34,70].新的证据表明,miR482和miR2118均靶向植物NBS-LRR防御基因的P-loop序列基序,并在植物对疾病的响应中发挥负调控作用[71].在大豆中,miR482的组成型表达导致靶向r基因的降解,导致染色量增加[72].在马铃薯中,miR482e的表达被下调verticillium dahliae.感染导致NBS-LRR靶标上调。miR482e过表达诱导其NBS-LRR靶标表达减少,转基因植株高度敏感诉大丽花感染(73].在我们的数据集中,一个成员的miR2118和三个成员的miR482。我们发现miR2118、miR482a、miR482b和miR482b在抗性基因型中的总体表达量低于易感基因型(表)S3).例如,在抗性基因型中,MiR2118的表达丰度分别为74.10和91.53 Tp10m,分别在模拟和治疗样品中。虽然在易感基因型中,分别在模拟和治疗样品中,MiR2118的表达丰度为170.22和261.62 Tp10m(表S3).有趣的是,花生MiR482 / 2118 MiRNA系列也靶向NBS-LRR防御基因,包括Aradu.168 L7,其在抗性基因型中具有更高的表达水平,而不是易感基因型。
在该研究中,通过组合分析小RNA,降低组序列和mRNA表达分析的数据集来鉴定66个潜在的miRNA /靶对。其中,许多SPL(Squamosa启动子结合蛋白样)可能由miR156 / 157 miRNA家族调节(表S12- - - - - -13).例如,Aradu.0GH1S。1encodes a SPL transcription factor, which is homologous withSPL12大豆(LOC100781289)和SPL2.拟南芥(AT5G43270)。降解组测序显示Aradu.0GH1S。1was potentially cleaved by many miRNA members of miR156/157 family, such as miR156a, miR156e and miR156i, etc. (TableS7).先前的证据表明SPL.对花青素的积累进行了负面调节。并进一步研究证明MIR156 /SPL.调控植物次生代谢和花青素、黄酮醇含量[74].花青素和黄酮醇都是重要的黄酮类化合物。已显示黄酮类化合物对侵袭微生物的抗菌性质进行抗微生物性质,包括答:flavus[75].在本研究中,Aradu.0GH1S的表达水平。1in susceptible genotype was significantly higher than that in resistant genotype, which might be caused by the regulation of miR156. In addition, GO and KEGG results showed that the GO term and pathway related with secondary metabolism of flavonoids was enriched between two peanut varieties. These results provided valuable information for understanding the roles of miR156/SPL in peanut.
结论
本研究报道了花生抗病和感病品种转录组、miRNAome和降解组的整合分析答:flavus.共鉴定了总共30个差异表达的miRNA,447个差异表达基因和21 miRNA /靶对答:flavus.另外,在抗性和易感花生品种之间鉴定了总62个差异表达的miRNA,451差异表达基因和44 miRNA /靶对。此外,讨论了两种miRNA /靶标调节对的功能,包括MiR482 / 2118 / NBS-LRR和MiR156 / 157 / SPL(图。8).我们的研究产生了一个全面的数据集,以进一步了解miRNA在响应答:flavus在花生。
方法
植物材料和治疗状况
两种栽培花生包括GT-C20和TIFRUNNER,从山东作物种质资源,济南,中国,凭证标本沉积在山东作物种质资源中心。GT-C20和Tifrunner的电阻答:flavus曾在以前的研究中报道过[76].的答:flavus株(AF-Wh2014)从花生中分离的好心被Boshou廖教授的实验室提供,油料作物中国农业科学院的研究所。用于响应于评估两个花生品种的电阻的方法答:flavus提到以前的研究,并在本研究中得到改善[9,10,37].简单地说,答:flavus在30℃下在新鲜的土豆葡萄糖琼脂(PDA)培养基中培养7天。然后收集分枝瘤并悬浮进入孢子悬浮液(106 CFU/ml) with sterile distilled water. For inoculation with答:flavus,peanut seeds were surface sterilized with 70% alcohol for 1 min, and 3% sodium hypochlorite (NaClO) for 10 min. Then, the seed coat was wounded with an area of 6 mm × 5 mm. The wounded area was covered with the same size of a piece of cotton, and 20 μl of spores suspension was added on the cotton. For the control sample, 20 μl sterile water was added. To keep the humidity, the infected seeds were placed in the petri dish with two layers of filter paper wet with 5 ml sterilized water. Finally, seeds were placed into incubator at 30 °C in darkness. Evaluation of peanut resistance to答:flavus是按照先前研究的描述进行的[37].基于覆盖率计算了感染指数答:flavus花生种子种皮中的菌丝体。花生与答:flavus使用以下标准进行评估:高抗性(感染指数<5.0),适度抗性(感染率≥5.0和<10.0),中等易感(感染指数≥10.0和<30.0),易感(感染指数≥30.0和<50.0).
图书馆建设和深度测序
接种GT-C20和Tifrunner种子答:flavus对于小型RNA,转录组和降低组文库构建的2 d。未接种种子用作对照。对于每个样品,制备三种单独的生物重复。根据制造商的协议使用Trizol Reagent(Invitrogen)分离出总RNA。使用电泳使用1%琼脂糖凝胶和BioSpector荧光(Eppendorf,Ger)对RNA质量和完整性进行评估。然后,使用总RNA用于构建小RNA,转录组和降低组文库,分别使用先前研究中所述的方法进行[35,36,40,77].简而言之,为了构建转录组文库,纯化总RNA以使用寡核苷酸(DT)磁珠获得mRNA。然后,将mRNA分成短片段(约200bp)并进一步用于合成使用随机六烯底漆的第一链CDNA。接下来,使用缓冲液,DNTPS,RNase H和DNA聚合酶I合成第二链cDNA,然后将其连接到测序适配器上。在CDNA片段之后通过PCR扩增富集。最后,图书馆产品已准备好进行排序。对于构建小RNA文库,首先用于纯化小RNA分子(18-30nt),然后将一对适配器连接至5'和3'末端。在后,通过逆转录和PCR扩增制备测序文库。对于构建降级库,执行以下步骤。首先,使用MRNA作为输入RNA和用生物素化的随机引物退火。 Second, Strapavidin capture of RNA fragments through Biotinylated Random Primers. Third, 5′ adaptor ligation to only those RNAs containing 5-monophosphates. Finally, Reverse transcription and PCR amplification were performed. Before sequencing, the QC step was performed to qualify and quantify of the sample library using Agilent 2100 Bioanaylzer and ABI StepOnePlus Real-Time PCR System. High-throughput sequencing of small RNA, transcriptome and degradome was performed in BGI (Shenzen, China) using Illumina HiSeq2000 platform.
MiRNA鉴定和生物信息学分析
首先,通过去除低质量reads、5 '底漆污染物、没有3 '底漆的reads、poly A的reads和小于18 nt的reads来清理原始reads。使用BLAST对Rfam数据库(11.0版本,http://rfam.janelia.org/) [78,79]和GenBank非编码RNA数据库去除rRNA、scRNA、snoRNA、snRNA和tRNA。然后,其余的reads与miRbase中的成熟mirna(版本21,http://www.mirbase.org/) [80,81]识别已知的miRNA。根据报告的方法鉴定了新的miRNA [39].清洁读数首先映射到全基因组序列A. Duranensis.和答:ipaensis(https://peanutbase.org/).然后,利用Minreap (http://sourceforge.net/projects/mireap)根据以下参数。候选序列可以形成适当的次级发夹,其中成熟的miRNA映射区域具有18-25nt的大小,并且miRNA和miRNA *之间的距离为16-300nt。允许形成发夹结构的最大自由能量是 - 18千卡/摩尔[40].不同sRNA文库的mirna基因表达水平采用TP10M (tags per ten万reads)标准化。使用R package DESeq (http://biocidodder.org/packages/degseq/)根据调整的准则p值(padj)<0.05并折叠变化≥1.5[82].
降解组测序和数据分析
为了识别miRNAs调控的潜在靶点,我们将抗性和敏感基因型对照(TC1, TC2, GC1和GC2)和处理样本(TT1, TT2, GT1和GT2)中等量的rna混合在一起,构建降解组文库并进行深度测序。通过预处理,生成干净的标签。然后与Genbank、Rfam数据库和miRNA数据库进行比对,对干净标签进行分类。接下来,这些reads被映射到的转录组数据A. Duranensis.和答:ipaensis使用SOAP2程序只允许两个不匹配[83].的转录组数据A. Duranensis.和答:ipaensis下载自花生基地(https://peanutbase.org).花生cDNA意义链利用CLeaveLand pipeline预测miRNA剪切位点[84].基于每个占用的转录物的签名数和裂缝位置的丰度,可裂开的转录物可以分为五个类别(0,1,2,3和4)[43].
转录组测序和数据分析
为了分析靶基因的表达谱,测序了八种独立的转录组文库。排序后,首先删除适配器序列,低质量读取和空读取。然后,清洁读数与基因组序列对齐A. Duranensis.和答:ipaensis(https://peanutbase.org/)使用SOAP2程序[83].采用FPKM(每千碱基对测序的转录本序列的预期片段数)法计算基因表达水平。采用对数法计算不同样本间的相对基因表达量2率的方法。以概率≥0.8和|log进行差异表达基因(DEGs)鉴定2(fold change)|≥1 using NOIseq method [85].为了鉴定目标基因和DEGS的推定生物功能和途径,基因本体论(GO)注释和KEGG(京都基因与基因组百科)通路分析如先前所述进行了[77,86,87].
qRT-PCR验证miRNA和mRNA的表达
为验证mirna和目的基因的高通量测序结果,采用ABI 7500 Real-Time PCR System (Applied Biosystems)进行定量RT-PCR (qRT-PCR)。每个样品准备三个生物重复。miRNA表达分析,按照前面介绍的方法采用茎环RT-PCR方法[88].对于靶基因,使用Primer Premier 5.0软件设计引物(www.plemierbiosoft.com.).所有的引物包括茎环的miRNA引物,参考基因和目标基因在其他文件中被列为:表S14系列.使用2计算不同样本之间的相对表达水平-△△CT.方法。进行学生的T检验以分析治疗后基因表达的变化答:flavus(*P< 0.05,* *P< 0.01)。
可用性数据和材料
本研究中的RNA-SEQ数据可在NCBI BIOPROJECT中获得:PRJNA438019(https://www.ncbi.nlm.nih.gov/bioproject/PRJNA438019).
缩写
- GC:
-
GT-C20控制
- GT:
-
GT-C20治疗
- TC:
-
Tifrunner控制
- TT:
-
Tifrunner治疗
- 去:
-
基因本体论
- KEGG:
-
京都基因和基因组百科全书
- NBS-LRR:
-
核苷酸结合位点富含亮氨酸重复
- SPL:
-
Squamosa启动子结合蛋白质
- DEG:
-
差异表达基因
参考文献
- 1.
Guo B,Fedorova Nd,Chen X等人。基因表达分析和抗性基因的鉴定曲霉菌曲藤在花生通过EST和微阵列策略感染。毒素(巴塞尔)。2011; 3:737-53。
- 2.
Guo B,Yu J,Holbrook Cc等。在花生中预防预贲门素污染的策略:黄曲霉毒性生物合成,遗传学和基因组学。花生科学。2009; 36:11-20。
- 3.
Martin CN, Garner RC。黄曲霉毒素B -由黄曲霉毒素B1的化学或酶氧化产生的氧化物,引起核酸中的鸟嘌呤取代。大自然。1977;267:863-5。
- 4.
沈慧敏,王志强,李宝林,等。黄曲霉毒素b1诱导大鼠肝DNA 8-羟基脱氧鸟苷形成。致癌作用。1995;16:419-22。
- 5.
类胡萝卜素抑制黄曲霉毒素b1诱导HepG2细胞毒性:形态学、凋亡和DNA损伤。生物化学杂志。2006;387:87 - 93。
- 6.
Guo B,Chen X,Dang P等人。在不同再生阶段的种子中发展种子的花生基因表达剖析曲霉属真菌寄生感染。BMC DEV BIOL。2008; 8:12。
- 7.
王T,张娥,陈X等。鉴定与抗抵抗性预收获的黄曲霉毒素污染的种子蛋白(落花生hypogaeal)。BMC植物BIOL。2010; 10:267。
- 8.
王志强,严胜,刘超,等。蛋白质组学分析揭示了黄曲霉毒素对花生子叶的免疫反应曲霉菌曲藤.J Proteome Res. 2012; 11:2739-53。
- 9。
王H,莱伊,万L等。抗性和易感花生后收获种子的比较转录物分析响应于黄曲霉毒素的产生曲霉菌曲藤.BMC植物BIOL。2016; 16:54。
- 10.
王浩,雷勇,严磊,等。功能基因组分析曲霉菌曲藤与抗病、易感花生互作。毒素(巴塞尔)。2016年,立法机关。
- 11.
Bertioli DJ, Cannon SB, Froenicke L等。的基因组序列arachis duranensis.和落花生ipaensis是栽培花生的二倍体祖先。Nat麝猫。2016;48:438-46。
- 12.
陈X,李H,Pandey M等人。花生A-基因组祖细胞的基因组草案(arachis duranensis.)提供了对地瓜、石油生物合成和过敏原的深入了解。美国国立科学院学报2016;113:6785-90。
- 13.
温斯坦EG、罗德斯MW等。小分子核糖核酸的植物。基因Dev。2002;16:1616-26。
- 14.
MicroRNAS及其在植物中的调控作用。植物学报2006;57:19-53。
- 15.
Chuck G,Candela H,Hake S.小RNA在植物开发中的影响大。CurrOp植物BIOL。2009; 12:81-6。
- 16.
Sunkar R,Kapoor A,Zhu JK。拟南芥中两种Cu / Zn超氧化物歧化酶基因的前剖视诱导是通过MiR398的下调介导的介导,并且对于氧化应激耐受性重要。植物细胞。2006; 18:2051-65。
- 17.
Jagadeeswaran G,Saini A,Sunkar R.生物和非生物胁迫下调拟南芥中的mir398表达。Planta。2009; 229:1009-14。
- 18.
张旭,邹志,龚鹏,等。过表达microRNA169增强番茄的抗旱性。Biotechnol。2011;33:403-9。
- 19.
倪震,胡震,姜强,等。GmNFYA3是miR169的靶基因,是植物抗旱性的正调控基因。acta botanica yunnanica(云南植物研究). 2013;82:113-29。
- 20。
Wang JW,捷克B,Weigel D.Mir156调节的SPL转录因子定义内源性开花途径拟南芥.细胞。2009;138:738-49。
- 21。
王建伟,王志强,王志强,等。mir156靶向SPL基因和CYP78A5/KLUH对拟南芥叶绿体长度和器官大小的双重影响植物细胞。2008;20:1231-43。
- 22.
蔡恩华。ABA诱导miR159调控拟南芥种子萌发过程中两个MYB因子的转录水平。植物j . 2007; 49:592 - 606。
- 23.
Mallory Ac,Dugas Dv,Bartel DP等。适当的形成和分离相邻的胚胎,营养和花动器官需要NAC结构域靶标的microRNA调节。Curr Biol。2004; 14:1035-46。
- 24.
关键词:水稻,mir164, NAC基因,抗旱性J Exp Bot. 2014; 65:2119-35。
- 25.
冯华,段旭,张强,等。来自NAM亚家族的新NAC转录因子tae-miR164的靶基因负调控小麦对条锈病的抗性。植物学报。2014;15:284-96。
- 26.
miR390,拟南芥的TAS3 tasiRNAs及其生长素反应因子靶标定义了一个定量调控侧根生长的自动调控网络。植物细胞。2010;22:1104-17。
- 27.
鲍曼JL,III级。HD-ZIP基因调节,Argonaute活动的金色羊毛。生物。2004; 26:938-42。
- 28.
水稻AP2家族基因SNB和OsIDS1协同调控花序结构和花分生组织的建立。植物j . 2012; 69:445 - 61。
- 29.
王y,王l,zou y等。大豆MiR172C靶向抑制AP2转录因子NNC1以激活ENOD40表达并调节结节引发。植物细胞。2014; 26:4782-801。
- 30.
小rna是植物天然免疫的重要组成部分。植物学报。2009;12:465-72。
- 31.
李飞,李志刚,李志刚,等。植物固有免疫受体的MicroRNA调控。美国国家科学研究院2012;109:1790-5。
- 32.
Boccara M,Sarazin A,Thiebeauld O,等。拟南芥MIR472-RDR6沉默途径通过抗病性抗病基因的转录后控制调节PAMP和效应触发的免疫。PLOS POAROG。2014; 10:E1003883。
- 33.
李y,lu yg,shi y等。多种稻米微稻草对抗爆炸真菌矿物质的免疫力。植物理性。2014; 164:1077-92。
- 34.
ShivaPrasad PV,Chen HM,Patel K,等人。MicroRNA超细含量调节核苷酸结合位点 - 富氨酸富含的重复和其他MRNA。植物细胞。2012; 24:859-74。
- 35.
赵C,夏H,弗雷泽TP等。深度测序识别花生新颖的和保守的微RNA(落花生hypogaeaL.)。BMC植物BIOL。2010; 10:3。
- 36.
赵C,夏H,Cao T等人。小RNA和降解深度测序响应病原体感染而揭示了花生MicroRNA角色。植物mol biol报告。2015; 33:1013-29。
- 37.
Mehan VK, Mcdonald D, Nigam SN等。种子抗黄曲霉入侵的花生品种。Oleagineux。1981;36:501-7。
- 38.
高C,王平,赵S,等人。小RNA分析和降解组分析揭示花生胚胎发育和早期荚果发育的microRNA调控。BMC基因组学。2017; 18:220。
- 39.
Meyers Bc,Axtell MJ,Bartel B等人。植物微稻草注释标准。植物细胞。2008; 20:3186-90。
- 40。
张Q,Zhao C,Li M,等。基因组识别Thellungiella salsuginea微大稻草,具有盐应激反应的推定作用。BMC植物BIOL。2013; 13:180。
- 41。
徐X,YIN L,Ying Q等。高通量测序和降级分析识别miRNA及其参与果实衰老的目标Fragaria Ananassa..Plos一个。2013; 8:E70959。
- 42。
杨X,王L,袁D等。小RNA和降低测量测序显示棉躯体胚胎发生过程中的复杂miRNA调节。J Exp Bot。2013; 64:1521-36。
- 43。
韩X,尹H,宋X等。小RNA,降解组和超富集植物东南景天转录组测序的集成揭示一个复杂的监管网络,并提供了深入了解植物修复镉。植物生物技术J. 2016; 14:1470至1483年。
- 44。
莫雷兹森,巴博萨,阿尔维斯-弗雷塔斯等。花生(豆科)b基因组的连锁图谱及其与A基因组的同步性。BMC Plant Biol. 2009;9:40。
- 45。
莫雷兹森等。利用内含子序列和微卫星标记研究栽培花生(Arachis hypogaea)与最接近野生种的亲缘关系。安机器人。2013;111:113-26。
- 46。
Bertioli DJ,Vidigal B,Nielen S等人。花生(阿拉氏血吸虫)基因组的重复分量及其在重塑基因序列空间中的作用,因为其从B基因组进化分歧。Ann Bot。2013; 112:545-59。
- 47.
毕勇,刘伟,夏辉,等。栽培花生(Arachis hypogaea L)的EST测序和基因表达谱分析。
- 48.
宋刚,李敏,肖华,等。栽培花生EST测序及SSR标记的开发落花生hypogaeaL.)。电子J Biotechnol。2010; 13:7-8。
- 49.
冯S,王X,张X等。花生(落花生hypogaea)表达序列标签项目:进度和应用。Comp Funct基因组学。2012年。第373768条。https://doi.org/10.1155/2012/373768.
- 50。
姜旭,李志,康志,等。几丁质酶与植物抗病。西北农业学报,2002;11:71-5。
- 51.
范登堡。番茄病原菌黄分枝杆菌(Cladosporium fulvum) AVR4天然二硫键断裂突变体对蛋白水解敏感,绕开了cf -4介导的抗性,但保留了几丁质结合能力。中国生物化学杂志。2003;278:27340-6。
- 52.
Cletus J,Balasubramanian V,Vashisht D等人。植物花序酶的转基因表达以增强抗病性。Biotechnol Lett。2013; 35:1719-32。
- 53.
Legrand M,Kauffmann S,Geoffroy P等人。病因相关蛋白质的生物学功能:四种烟草发病性相关的蛋白质是几丁质酶。Proc Natl Acad Sci U S A. 1987; 84:6750-4。
- 54.
张志强,张志强,张志强,等。截茎紫花苜蓿根在菌根形成、结瘤和病原菌感染过程中8个几丁质酶基因的差异表达。植物与微生物的相互作用。2000;13:763-77。
- 55.
张志强,张志强,张志强,等。葡聚糖酶和几丁质酶作为保护金合欢花外花蜜免受植物病原体侵害的致病因子。植物杂志。2010;152:1705-15。
- 56.
Seevers PM,达利JM,大教堂FF。过氧化物酶同工酶的阻力作用,小麦杆锈病。植物理性。1971; 48:353-60。
- 57.
阮XM,罗,李DD,等人。编码推定蓝铜结合蛋白棉BCP基因在纤维发育的功能性表达,涉及响应于高盐度和重金属的应力。植物生理学。2011; 141:71-83。
- 58.
rea g,metoui o,imantino a等。铜胺氧化酶在防御反应中的表达和ascochyta rabiei入侵。植物理性。2002; 128:865-75。
- 59.
Cona A,Rea G,Angelini R等。胺氧化酶在植物开发和防御中的作用。趋势植物SCI。2006; 11:80-8。
- 60。
张志强,王志强,王志强。p450基因的功能基因组学研究。植物学报2003;54:629-67。
- 61。
王志强,王志强,王志强,等。CYP709B3是一个参与拟南芥耐盐的细胞色素P450单加氧酶基因。BMC Plant Biol. 2013;13:169。
- 62。
李W,王F,王杰,等。过表达CYP71Z2通过抑制水稻中的生长素生物合成来增强对细菌枯萎的抗性。Plos一个。2015; 10:e0119867。
- 63。
鸟居骨。植物中富含亮氨酸重复受体激酶的结构、功能和信号转导途径。Int Rev Cytol. 2004; 234:1-46。
- 64。
qu s,liu g,周b等。宽光谱抗抗抗抗基因PI9编码核苷酸结合位点 - 亮氨酸富氨氨酸的重复蛋白,是水稻中多尾族的成员。遗传学。2006; 172:1901-14。
- 65。
胡尼,布伦纳,布赫曼等。黑麦Pm8和小麦Pm3是同源基因,具有抗白粉病的进化保守功能。植物j . 2013; 76:957 - 69。
- 66。
王志强,王志强,王志强,等。六倍体小麦抗叶锈病基因Lr10的图谱分离Triticum Aestivum.l .)基因组。美国国家科学研究院2003;100:15253-8。
- 67。
Stahl Ea,Dwyer G,Mauricio R等人。拟南芥RPM1位点的抗病性多态性动态。自然。1999年; 400:667-71。
- 68。
Mackey D,Holt Br,Wiig A等。RIN4与拟合蛋白酶III型效应分子相互作用,并且是拟南芥中的RPM1介导的抗性所必需的。细胞。2002; 108:743-54。
- 69。
Dangl JL,Ritter C,长臂猿M等人。拟南芥RPM1疾病抗性基因的功能同源物豆荚和豌豆。植物细胞。1992年; 4:1359-69。
- 70。
朱庆华,范玲,刘勇,等。miR482对棉花NBS-LRR防御基因的调控。《公共科学图书馆•综合》。2013;8:e84390。
- 71。
de Vries S, Kloesges T, Rose LE。在茄科植物中,miR482/2118基因家族针对抗性基因的进化动态,但健壮。基因组生物学进展。2015;7:3307-21。
- 72。
李h,邓y,wu t等。MiR482,miR1512和miR1515的Misexpression增加了大豆瘤。植物理性。2010; 153:1759-70。
- 73。
杨林,穆旭,刘超,等。过表达miR482e增强植物对黄萎病的敏感性。acta botanica yunnanica(云南植物研究). 2015;57:1078-88。
- 74。
郭建勇,刘建杰,等。mir156靶向的SPL转录因子对拟南芥花青素合成的负调控植物细胞。2011;23:1512-22。
- 75。
Medina ML, Kiernan UA, Francisco WA。黄曲霉芦丁诱导分泌蛋白的蛋白质组学分析。真菌生物学报2004;41:327-35。
- 76.
张海涛,张海涛,张海涛,等。黄曲霉素的产生缺乏曲霉菌曲藤与真菌生长减少和黄曲霉毒素途径基因表达延迟有关。世界真菌毒素J. 2014; 8:335-40。
- 77.
王军,张强,崔飞,等。基因表达的全基因组分析为寒冷反应提供了新的见解Thellungiella salsuginea.植物学报2017;8:713。
- 78.
Griffiths-Jones S, Moxon S, Marshall M, Khanna A, Eddy SR, Bateman A. Rfam:注释完整基因组中的非编码rna。核酸学报2005;33(sup1): D121-4。
- 79.
培训SW,Daub J,Eberhardt R,Tate J,Barquist L,Nawrocki EP,Eddy SR,Gardner PP,Bateman A. RFAM 11.0:10年的RNA家庭。核酸RES。2012; 41(D1):D226-32。
- 80.
Griffiths-Jones S. MicroRNA注册表。核酸RES。2004; 32(SUPPLE_1):D109-11。
- 81.
Griffiths-Jones S, Grocock RJ, Van Dongen S, Bateman A, Enright AJ。miRBase: microRNA序列、靶标和基因命名。核酸Res. 2006;34(suppl_1): D140-4。
- 82。
王磊,冯志强,王昕,张晓霞。基于RNA-seq数据的差异表达基因鉴定。生物信息学,2009,26(1):136 - 8。
- 83。
Li R,Yu C,Li,等。SOAP2:改进的超快工具,用于短读对准。生物信息学。2009; 25:1966-7。
- 84。
Addo-Quaye C, Miller W, Axtell MJ。CleaveLand:一个管道,使用降解数据,以发现剪切小RNA靶点。生物信息学。2008;25(1):130 - 1。
- 85。
等。RNA-seq的差异表达:一个深度问题。基因组研究》2011;21:2213-23。
- 86。
ashburner m,ball ca,blake Ja,Botstein D,Butler H,Cherry Jm,Davis Ap,Dolinski K,Dwight Ss,Eppig Jt,Harris Ma。基因本体:生物学统一的工具。NAT Genet。2000; 25(1):25。
- 87。
Kanehisa男,转到S. KEGG:基因和基因组的京都百科全书。核酸RES。2000; 28(1):27-30。
- 88。
Varkonyi-Gasic E,吴R,木M等人。协议:用于检测和定量微RNA一个高度敏感的RT-PCR法。植物的方法。2007; 3:12。
致谢
作者感谢中国农业科学院油料作物研究所廖伯寿教授和雷勇教授提供的答:flavus应变(AF-Wh2014)。
资金
国家自然科学基金项目(no . 31861143009);山东省种质创新利用项目[2016LZGC025],山东省自然科学基金[ZR2017MC005, ZR2015YL061, ZR2015CQ012, ZR2018BC029],山东省重点研发计划项目(2019GNC106026, 2019GSF107008)和山东省泰山学者项目(ts20190964);农业科技创新项目[CXGC2018E13, CXGC2016B02]和山东省农业科学院青年人才培养计划。创始人在本研究或相关数据的设计、分析和解释中没有扮演任何角色。
作者信息
隶属关系
贡献
CZ和XW构思和设计了实验;CZ、TL、YZ1、BZ、JQ、SF、BG分析数据并撰写稿件;TL、AL、SZ、LH、HX、PL和YZ2进行实验。所有作者均已阅读并批准本稿件。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
竞争利益
所有的作者都宣称他们没有相互竞争的利益。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1。
每个图书馆的不同的RNA片段类别。
附加文件2:图S2。
使用热图的测序和QRT-PCR结果的比较。Heatmap由在线软件Morpheus生成(https://software.broadinstitute.org/morpheus.),根据相对表达量与对照样品TC进行比较。
附加文件3:图S3。
火山图显示了不同样本之间的基因表达差异。rpkm归一化的转录本计数数据集通过NOIseq进行分析:x轴表示对数比(挑战后基因表达倍数变化),y轴表示每个基因差异表达的概率。在概率≥0.8和|log2(fold change)|≥1的标准下鉴定上调和下调基因。
附加文件4:表S1。
小型RNA概述从个体图书馆读取。
附加文件5:表S2。
总斯尔纳的统计分析花生基因组映射。
附加文件6:表S3。
已知miRNA的信息有两种花生品种。
附加文件7:表S4。
花生miRNA文库中新发现的miRNA信息。
附加文件8:表S5。
退化库读本的分类分析。
附加文件9:表S6。
候选人的分类裂解位点。
附加文件10:表S7。
通过降解组测序鉴定已知mirna的靶基因。
附加文件11:表S8。
通过降序测序的新型miRNA鉴定的靶基因。
附加文件12:表S9。
RNA-seq从单个文库中读取的摘要。
附加文件13:表S10。
Tifrunner和GT-C20对AF感染反应的前16个KEGG通路。
附加文件14:表S11。
Tifrunner与GT-C20之间的16条KEGG富集通路。
附加文件15:表S12。
mirna表达谱与靶基因的相关性分析答:flavus.
附加文件16:表S13。
花生品种间mirna表达谱及其靶基因的相关性分析
附加文件17:表S14。
本研究中使用的引物。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
赵,C.,李,T.,赵,Y.等等。整合的小RNA和mRNA表达谱揭示了mirna及其靶基因的响应曲霉菌曲藤花生种子的生长。BMC植物杂志20,215(2020)。https://doi.org/10.1186/s12870-020-02426-z
收到了:
接受:
发表:
关键词
- 花生
- 曲霉菌曲藤
- microRNA.
- 转录
- Degradome