抽象的
背景
酸浆属L.是家庭Solanaceae草本植物的属性,具有重要的药用,食用和观赏价值。形态特征酸浆属物种是相似的,仅凭形态特征很难快速准确地区分它们。目前,在物种分类和系统发育方面酸浆属仍然存在争议。在本研究中,四个完整的叶绿体(CP)基因组酸浆属物种(酸浆属angulata,P. Alkekengi.var。franchetii,p .最小值和p .下毛竹)测序,并首次进行了全面的cp基因组分析酸浆属进行了,其中包括先前公布的CP基因组序列酸浆属peruviana.
结果
的酸浆属Cp基因组表现出典型的四联体和环状结构,在结构和基因同源性上相对保守。然而,酸浆属Cp基因组在4个区域边界上表现出明显的变异,尤其是反向重复区域和大的单拷贝区域。cp基因组长度为156,578 bp ~ 157,007 bp。4个新序列共检测到114个不同基因,80个蛋白编码基因,30个tRNA基因和4个rRNA基因酸浆属cp基因。检测到重复序列和简单序列重复的差异酸浆属cp基因。茄科11属36种植物cp基因组序列的系统发育关系酸浆属在系统发育树的中部和上部,具有100%自举值的单晶的进化。
结论
我们的结果丰富了属于属的CP基因组的数据酸浆属.这些cp基因组的获得将为进一步的物种鉴定提供丰富的信息,提高物种的分类和系统发育分辨率酸浆属,并协助调查和利用酸浆属植物。
背景
属酸浆属L.组成的75-120种,是一家着名的Solanaceae,因为其成员的药用,食用和装饰用途1,2,3.].它主要分布在美洲的热带和温带地区,在欧亚大陆和东南亚仅发现少数品种[1,4,5,6].我国约有五种,两种酸浆属植物,作为药材被中国人使用了2000多年。许多酸浆属物种具有各种药理活动,导致抗炎,抗氧化和抗癌益处,并用于治疗许多疾病,包括疟疾,风湿病,肝炎,哮喘,癌症和肝病[2,7,8,9,10,11].包括《中华人民共和国药典》physalis alkekengi.var。franchetii作为一个标准酸浆属2015年医疗植物[7].此外,许多酸浆属物种,如p .下毛竹,p . peruviana,P. Alkekengi.var。franchetii, 和p . philadelphica,在世界许多地区都有种植,其果实可食用或用作观赏植物[4,12].
摘要叶绿体是植物细胞中重要的细胞器,在植物细胞的许多功能中起着重要的作用,如光合作用、固碳作用、胁迫反应等[13,14].在most plants, the cp genome’s structure is very conservative, being circular with a length of 120–170 kb, including four typical areas: two inverted repeats (IRs), large single-copy (LSC) and small single-copy (SSC) regions [15].在CP基因组中,基因含量和基因组合物高度保守,通常含有120-130个基因[16].此外,与核DNA序列相比,CP基因组的进化率通常缓慢[17].然而,在一些被子植物cp基因组的进化过程中,已经观察到一些重大的基因组结构变化,包括基因丢失、大反转和IR区域的收缩或扩张[16,17,18].例如,infA,rpl22,rpl33.,rps16,ycf1,ycf2,ycf4和ACCD.一些植物的基因已经丢失了[16,19,20.,21].此外,一些物种的红外区,如Pisum一[22],大豆[23],Crytomeria粳稻[24],Taxus chinensis.var。mairei[25),而豇豆属辐射动物[26]显示完整或部分损失。这些CP基因组差异可能是植物物种演化过程中差分诱导和替代率的结果[27].由于结构,中等进化率和CP基因组的分娩遗传,序列通常被用作DNA条形码的遗传标志物,以及系统发育和进化研究[17,28,29,30.].
近年来,由于其各种重要的商业价值,对其进行了分类酸浆属已成为关注的焦点,其特性被认为是茄科中最具挑战性的[1,3.,31,32].传统上,属酸浆属通过形态和/或地理特征,如习惯,发型和花萼角度的数量分为物种组[5,31].近年来,随着分子分类的兴起,核糖体内部转录间隔体(ITS) 1和ITS2,即叶绿体ndhF,TRNL-F.和psba-trn.序列,蜡实基因,已被用于物种鉴定和系统发育分析酸浆属,以及确定它们与Solanaceae家族中其他属的关系[1,3.,32,33].此外,简单序列重复序列(simple sequence repeat, SSR)、简单序列间重复序列(inter-simple sequence repeats, sequence characterized amplified region marker)和序列特征扩增区域标记(sequence characterized amplified region marker, sequence characterized amplified region marker)等DNA标记系统也已被应用于植物的遗传研究酸浆属植物 [4,12,34,35].然而,由于这些传统的遗传标记的信息有限,在物种鉴定和分类方面仍存在一些争议酸浆属[3.,28].cp基因组在植物系统发育研究中的应用和发展,为更好地研究植物系统发育分类提供了新的研究思路酸浆属.下一代测序技术的进步促进了CP基因组学领域的快速进展[36,37].截至2019年9月,超过3000个完整的cp基因组序列,包括p . peruviana(GenBank登录号:NC_026570酸浆属在未经进一步分析或研究的情况下,被发布到国家生物技术信息中心(NCBI)的细胞器基因组数据库(https://www.ncbi.nlm.nih.gov/genome/organelle/).
在这里,我们测序了四个的CP基因组酸浆属物种(p . angulata,P. Alkekengi.var。franchetii,p .最小值和p .下毛竹),并对基因组进行了深入分析,首次对cp基因组进行了全面分析酸浆属,包括先前发布的p . peruvianacp基因。本研究的目的是:(1)提供四种植物的cp全基因组序列酸浆属物种;(2)表征和比较可用的全球结构模式酸浆属CP基因组;(3)检查SSRS中的变化并在五个中重复序列酸浆属CP基因组;(4)改善我们对属的进化和系统性职位的理解酸浆属基于它们的cp基因组序列。
结果
全基因组测序和组装
从每6个健康、干净和新鲜的个体叶片池中提取约0.1 g的总基因组DNA酸浆属物种(附加文件1:表S1),用于通过远程PCR生成相应的Illumina MiSeq库(参见方法部分)。illumina测序(配对端,250x)后,读数被QC过滤,映射到p . peruvianacp参考基因组(NC_026570)组装得到4个完整的cp基因组。干净的基地映射到p . peruvianaCP参考基因组,平均覆盖范围从480x到1756x(附加文件1:表S2)。
酸浆属CP基因组特征
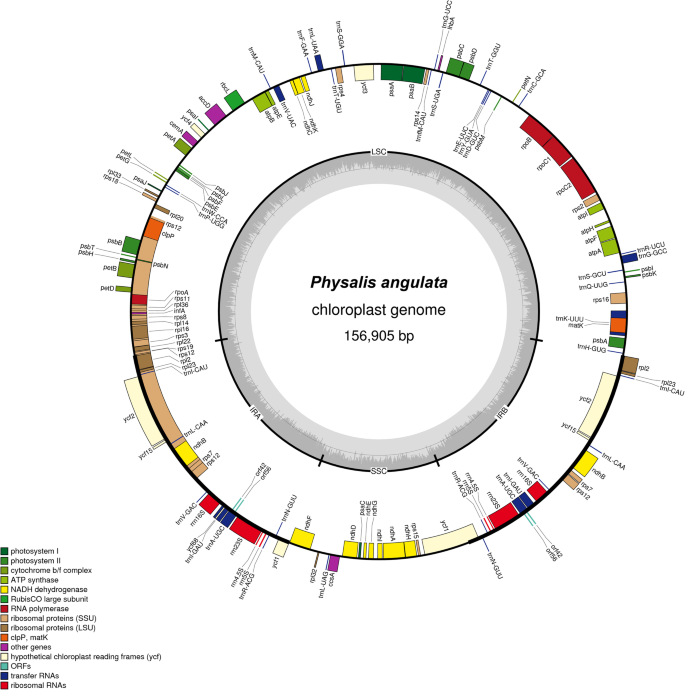
全部长度酸浆属Cp基因组范围为156,578 bp (P. Alkekengi.var。franchetii)至157,007 bp (p .下毛竹)(表1).新测序的基因图谱酸浆属图2中提供了CP基因组。1(p . angulata)及附加文件2:图S1-S3 (P. Alkekengi.var。franchetii,p .最小值和p .下毛竹).像大多数高级植物一样,酸浆属CP基因组还表现出典型的四边形结构,分布在一个LSC区域(86,845bp-88,309bp)中,一个SSC区域(18,363bp-18,503bp)和一对红外区域(a和b; 24,953 bp-25,685 bp).每个CP基因组的总体GC含量可相当,范围为37.52至37.65%。虽然GC含量在每个区域之间差异分布,但在IR区显示比LSC或SSC更大的GC含量(表1).相比之下,p . peruviana,新的CP基因组含有2种更多的基因(总基因132 Vs 130),其中一些其中一些在IR区通常位于IR区(见表)1).当IR区域的复制基因只被计数一次时,四个新的cp基因组(p . angulata,alkekengi变种,p .最小值, 和p .下毛竹)包含相同的114个基因,分布为80个蛋白编码基因、4个rRNA基因和30个tRNA基因。而p . peruvianaCP基因组仅含有113个基因,缺少蛋白质编码基因。这些114/113基因编码自我复制相关的功能,与与其他蛋白质相关的函数,以及未知的蛋白质(表2).在114/113个基因中,17个是内含子基因,15个包含1个内含子(rpl2,rpl16,rpoc1.,rps12,rps16,trna-ugc.,trnG-GCC,TRNI-GAU.,trnK-UUU,trnL-UAA,trnV-UAC,atpF,ndhA,ndhB, 和petB)和两个包含两个内含子(clpP和ycf3).
密码子使用酸浆属cp基因
在五个对齐之后酸浆属在MEGA的cp基因组中,共发现了20个氨基酸编码,并根据不同的使用情况进行了不同的编码trnL密码子。蛋氨酸和色氨酸只有一个trnL每一个。而苯丙氨酸、酪氨酸、组氨酸、谷氨酰胺、天冬酰胺、赖氨酸、天冬氨酸、谷氨酸和半胱氨酸则由两个同义词密码子编码(附加文件)1:表S3和附加文件2:图S4)。
IR扩张和收缩
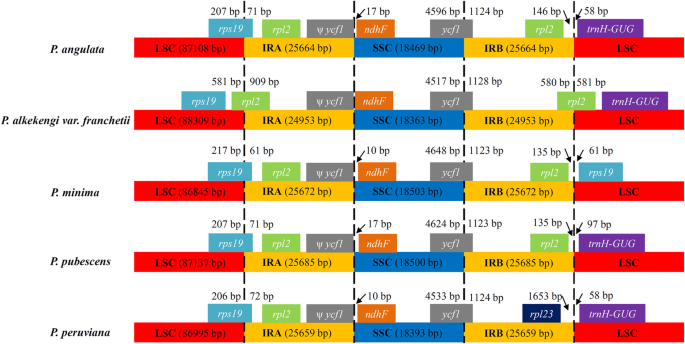
五个IR区域(A和B)酸浆属Cp基因组是最保守的区域,长度为24,953 ~ 25,685 bp。然而,IR边界有潜在的扩张和收缩,这被认为是进化事件和cp基因组长度变化的主要原因。LSC/IR和SSC/IR的边界酸浆属cp基因组比较(图。2).的rps19,rpl2,rpl23.和trnH-GUG基因主要分布在LSC / IR边界附近ycf1和ndhF基因分布在SSC / IR边界附近。基因ycf1通过SSC/IRB区域,和假基因片段ψycf1.位于IR-A地区,靠近南中国海/IR-A边界。与SSC/IR边界相比,LSC/IR边界表现出较大的变异。在P. Alkekengi.var。franchetii,rps19基因完全定位于LSC区域。然而,rps19基因p . angulata,p .最小值,p .下毛竹, 和p . peruviana分别以71、61、71和72 bp延伸至IRA区。有两份rpl2基因p . angulata,p .最小值, 和p .下毛竹,它们位于靠近LSC/IR边界的IR- a和IRB区域。在P. Alkekengi.var。franchetii,一式两份rpl2基因分别跨越LSC / IRA和LSC / IRB边界。一份副本rpl2在LSC/IRB边界缺失p . peruviana;相反,有一个rpl23.在LSC / IRB边界的IRB区域1653 BP处的基因。
基因组序列差异酸浆属物种
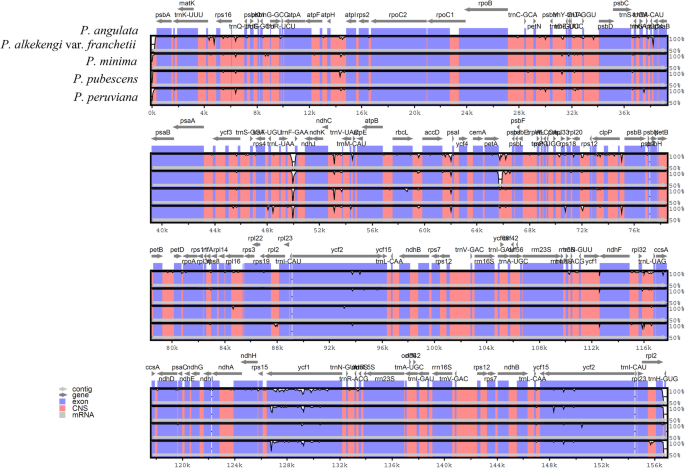
五个的完整CP基因组酸浆属将4个cp基因组与参考基因进行比对,用mVISTA软件进行物种比较和绘图p . angulata,阐明序列分歧的水平(图。3.).LSC和SSC区域的序列差异高于IR区域。编码区中的序列分歧是有限的,并且大多数序列发散在非编码区域中浓缩。在基因组水平,五个中的遗传距离酸浆属物种范围为0.0007至0.0048,平均遗传距离仅为0.0029(附加文件1:表S4)。
重复序列和SSR分析
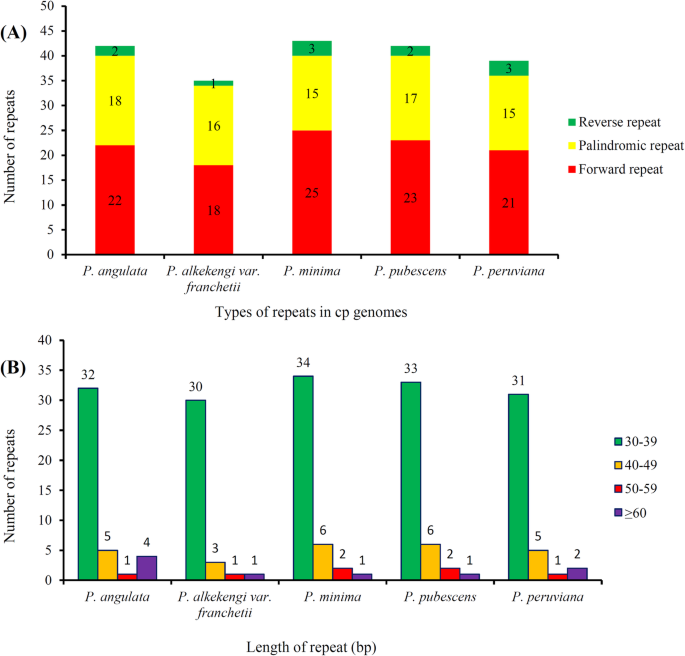
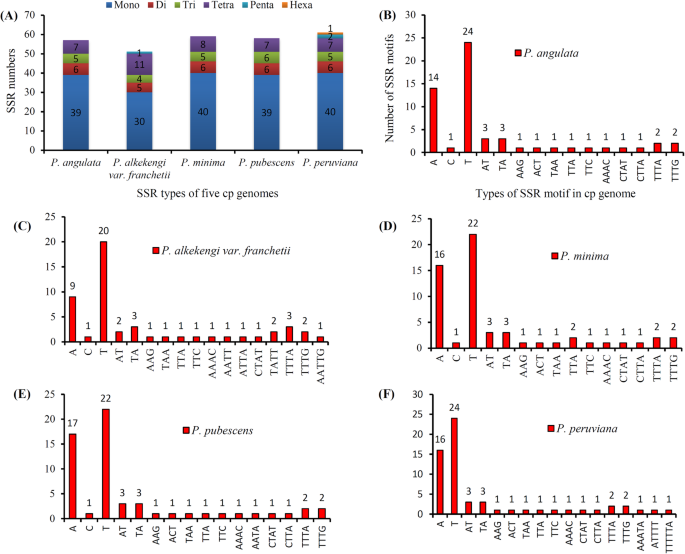
声示者用于分析每个CP基因组中的重复序列。鉴定了总共201重复序列,包括109前进的重复,81例回文重复,并且每重复单元的11个反向重复,序列同一性≥90%(图。4).每种基因组的每种基因组的重复的分布和每种物种的这种重复序列的重复长度和数量的分布如图2所示。4分别是A和b。
SSRs通常由长度为1 - 6bp的一系列重复单位组成(图中标记为单-六聚体)。5A),分布在整个基因组中。共检测到286个长度在10 bp以上的SSRs,每个基因组分布在51 ~ 61个SSRs之间(图1)。5一种)。这些SSR的大部分是单核苷酸(主要是聚 - A或Poly-T),每个CP基因组中有30-40个成员(图。5b-f)。所有种属中仅检出AT和TA二核苷酸,仅检出TTTTTA六核苷酸p . peruviana(图。5f).三核苷酸(AAG, ACT, TAA, TTA,和/或TTC),四核苷酸(AAAC, AATA, CTAT, CTTA, TTTA,和/或TTTG)和五核苷酸SSRs (AATTG和/或AAATA)有特定的分布,这可能用于未来的群体研究(图)。5b, f)。
系统发育分析
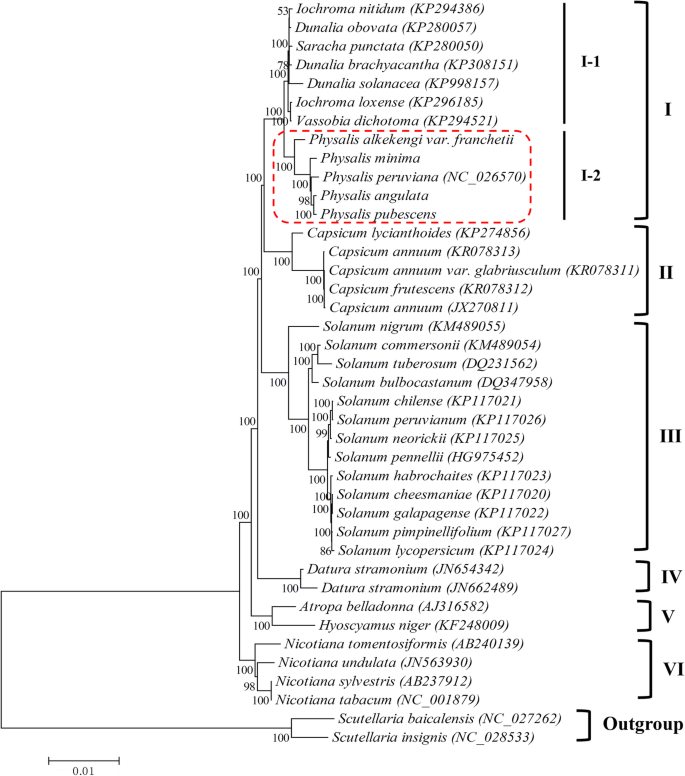
检查五个的系统发育位置酸浆属利用茄科11属36种植物的38个完整cp基因组进行系统发育分析。ML和NJ的系统发育重建(图。6和额外的文件2:图。S5)将所有物种分为六组(I至VI),对每个树拓扑的基于Bootstrap支持(BS)值略有差异。小组是最复杂的,有12种,进一步分为两个亚组。一个包含五种物种的子组酸浆属(I-2, BS = 100%)P. Alkekengi.var。franchetii作为基础物种。和第二个子组(I-1,BS = 100%),具有物种iochroma.,Dunalia,Saracha, 和Vassobia属。第II集团载有四个辣椒物种(c .建立,c .建立var。glabriusculum.,C. Lycianthoides.和C. Frutescens.)在ML和NJ和NJ的系统发育树中,具有100%的引导值。第三组包括13Solanum.(ML和NJ树种的BS = 100%)。曼佗罗聚集成ML和NJ的IV组,并具有100%的引导值。组v包括Atropa颠茄和Hyoscyamus尼日尔.这四个烟草物种,n的抗旱性,n tomentosiformis,n波动, 和N. Tabacum.,与其他茄科植物距离较远,被归为VI组(ML和NJ树的BS = 100%)。
讨论
Cp基因组结构与序列差异
在这项研究中,四个酸浆属利用Illumina MiSeq获得cp基因组,并与已发表的cp基因组进行比较p . peruviana.Illumina MiSeq是集放大、测序和数据分析于一体的下一代测序仪,由Illumina于2011年发布[38].Illumina MiSeq在实用性和工作流便捷性方面非常接近,并且在叶绿体基因组测序方面有很好的应用[17,39].五种方法的比较分析酸浆属CP基因组显示出高度保守的基因和结构。像那些大多数的Anviosperms,五个的CP基因组酸浆属有一个四分结构,通常由一个LSC、一个SSC和两个IR区域组成[15,40].cp基因组的大小p . angulata,P. Alkekengi.var。franchetii,p .最小值,p .下毛竹, 和p . peruviana范围为156,578 bp ~ 157,007 bp酸浆属是高度保守的。被子植物的cp基因组以相对较快的速度进化,在进化过程中发生逆转和基因丢失[16].在基因组成方面,大部分编码基因、tRNAs和rnas为5种酸浆属物种是相同的,但也有细微的差异。例如,cp基因组p .最小值有一个额外的PSBZ.基因,而LHBA.基因缺失,提示基因的缺失和插入发生在进化过程中p .最小值.除此之外PSBB.基因和一份副本rps12基因在CP基因组中缺少基因p . peruviana.事实上,在其他植物cp基因组中,也有许多关于cp缺失的报道LHBA.,infA,rpl22和rps16,以及内含子和复制缺失rpl2,clpP和rps12[20.,41,42,43].
陆地被子植物的cp基因组高度保守,但IR和SC边界的扩张和收缩被认为是cp基因组大小变化的主要原因[40,44].例如,在CP基因组中鉴定了逆转和/或基因损失事件黄芪膜[16,和Taxus chinensis.var。mairei被发现缺乏红外线区副本[25].Tetracentroncp基因组在IR区域显示扩张/收缩事件[45,以及Veroniceae包含rps19红外线区基因复制[46].在比较了五种cp基因组后酸浆属结果发现,SSC与两个IR区域的边界区域相对保守,基因类型在该区域的分布和具体位置高度一致。与其他四个相比酸浆属物种,IR地区P. Alkekengi.var。franchetii显示收缩,其长度最小(24,953 bp),主要是由于rpl2位于LSC / IR边界的基因将LSC区域扩展581 BP。在p . peruviana,没有rpl2LSC / IRB边界的基因,与其他四个不同酸浆属物种。此外,只有rps19基因p .最小值在5种植物的LSC/IRA和LSC/IRB边界上发现,表明它被复制。这种现象也在cp基因组中发现Veroniceae nakaiana[46].因此,LSC/IRB边界的变化似乎是导致区域红外区扩张/收缩的主要原因酸浆属.
密码子使用是正确表达遗传信息的关键因素[47].所有五个酸浆属共有61个氨基酸密码子(起始密码子AUG)和3个终止密码子(UAA、UAG、UGA)。编码20个氨基酸的密码子数量和类型存在差异,存在优先使用密码子的情况。多数首选氨基酸编码密码子的第三核苷酸为A或U。这一现象已经在许多被子植物中被发现,例如Aconitum Barbatum.var。Puberulum[47],烟草otophora[48] 和选用minuta[49].其他cp基因组的密码子使用频率不同,这可能与密码子的亲水性、同义替换率和/或表达水平有关[50].密码子偏好与物种的进化模式密切相关。因此,研究密码子的使用对进一步了解该属的历史演变具有重要价值酸浆属.
在大多数高等植物中,IR区域的变异小于SC区域,这主要是由于两个IR区域之间的基因转换引起的重复修正[40].mVISTA结果表明,cp基因组酸浆属具有较低的序列发散度,IR区域的保守性高于SC区域。此外,编码区域的保守性高于非编码区域,这与大多数高等被子植物cp基因组的保守性一致[40].
重复序列和SSR位点
重复序列在系统发育研究中很有用,并在基因组重组中发挥关键作用[48].此外,对不同cp基因组的比较研究表明,重复序列是导致基因插入、缺失和替换的重要因素[51,52].重复分析这五个酸浆属CP基因组检测到201重复序列,其中大部分是30-39bp的长度。五五酸浆属物种,p .最小值有最多的重复序列。基因组重组和序列变异主要是由滑移链错配和重复序列的不恰当重组引起的[48,51].这些重复序列是群体和系统发育研究的遗传标记基础,由于在其他特征中具有较高的多态率而被广泛使用[53,54,55,56].本研究共检测到286个SSR位点,其中大部分为既往报道的A/T型[56,57].
系统发育分析
由于种类多,形态相似,分布区域广,酸浆属植物被认为是形态学和分子水平的相对复杂的分类组。Whitson和Manos(2005)使用了它的序列和蜡实基因对属进行系统发育研究酸浆属及其亲属[1].植物的许多形态特征酸浆属似乎是同源的,和几个以前定义的属内分类群酸浆属不是单系的[1].Olmstead et al.(2008)对茄科进行了系统发育研究,其中包括5个酸浆属物种p . heterophylla,p . peruviana,p . philadelphica,P. Alkekengi., 和P. Carpenter.,基于CP DNA区域ndhF和TRNL-F.[32].研究表明,该属酸浆属与属有密切关系吗Margaranthus,Chamaesaracha,Quincula, 和Oryctes, 然后P. Alkekengi.和p . carpenteri和其他三种物种相比,一夫一妻制在进化中不是更好吗酸浆属物种。在我们以前的2016年和2018年的研究中[3.,33]、ITS2序列和cpPSBA.-trnH分别用于分子鉴定和系统发育分析酸浆属物种。结论与Whitson和Manos(2005)的结论相似[1]和olmstead等。(2008)[32].系统分类酸浆属物种应该被进一步探索。这些研究为分类鉴定奠定了重要的基础酸浆属物种。然而,核/ cp基因序列段的长度相对较短,其中限制了系统发育研究并导致具有低载体值的系统发育树。基于全CP基因组序列,本研究在11个属(包括属)中进行了36种的系统发育分析酸浆属茄科)。ML和NJ分析结果表明酸浆属物种在茄科的系统发育演进中形成了单线(支持率为100%),与其他金属密切相关,包括iochroma.,Dunalia,Saracha, 和Vassobia.P. Alkekengi.var。franchetii和其他四个人有远亲关系吗酸浆属种(支持率100%);因此,我们推测P. Alkekengi.var。franchetii比其他四更早地分化酸浆属基因进化过程中的物种。在某种程度上,这个结果也支持这个观点P. Alkekengi.var。franchetii应归入小属[1,3.].当然,只有部分cp基因组序列酸浆属以及茄科植物;因此,进行系统的分类酸浆属物种无法完成。我们计划获得更多CP基因组酸浆属在未来使用高通量测序的物种,这将使我们更准确地分析系统发育关系酸浆属物种。
虽然许多研究表明,使用CP基因组在系统发育研究中具有优势,仍存在许多问题[28].例如,不同的物种有不同的进化速度,对于一些进化速度快的群体,单凭整个cp基因组信息并不能完全确定其系统发育进化[58].另外,cp DNA是单性生殖的,其基因组信息只能反映母系或父系的进化过程,不能完全解释物种本身的整个系统进化[59].因此,更好地揭示了系统发育演变酸浆属物种除了对CP基因组的研究外,未来的研究应与核和线粒体基因组的数据分析相结合。
结论
在本研究中,四种cp基因组酸浆属物种,p . angulata,P. Alkekengi.var。franchetii,p .下毛竹, 和p .最小值首先通过高通量测序获得。进行了比较基因组分析,其中包括已发表的cp基因组p . peruviana允许我们用典型的四重形结构确定圆形性质酸浆属cp基因。整个酸浆属cp基因组相对保守,在IR/SC和LSC/IR边界存在差异。目前已鉴定出近290个SSR位点,可作为未来的分子标记酸浆属种内多样性研究。整个cp基因组重建了茄科植物的系统发育树,鉴定了6组种,并发现了茄科植物酸浆属作为Solanaceae Group I中的独立思考。我们的结果丰富了属于属的CP基因组的数据酸浆属为准确的分子鉴定和系统发育重建奠定了重要的基础酸浆属物种。
方法
植物材料,DNA提取和测序
在中国分布广泛的四种p . angulata,P. Alkekengi.var。franchetii,p .最小值和p .下毛竹,被群体收集(四分之一的抽样信息的详细信息酸浆属研究中收集的物种见附加文件1:表S1)。惠忠王(杭州师范大学)开展了植物材料的正式鉴定。所有收集物种的凭证标本占杭州师范大学药用植物遗传改善和质量控制的浙江省重点实验室(附加档案1:表S1)。这些物种未列入国家重点保护植物名录,采集不需要许可。干净,健康,新鲜的绿叶从收集酸浆属植物被取样(每个物种6个样品)。将叶片洗涤,干燥并储存在-80℃,直至DNA提取。
根据改性的CTAB方法从〜0.1g保存的叶子(相等量的6个体混合物)中提取总基因组DNA [60.].改性主要是在CTAB提取缓冲液中,其中含有4%的CTAB而不是2%,~ 0.2%的dl -二硫苏糖醇(DTT)和1%的聚乙烯聚吡咯烷酮(PVP),其余的协议描述[60.].每个物种的cp全基因组是通过总基因组DNA的远程PCR获得的,如前所述[17,61.].简而言之,PCR在含有1×矢粒子GXL缓冲液中进行25μL[10mM Tris-HCl(pH8.2),1mM MgCl2, 20 mM NaCl, 0.02 mM EDTA, 0.02 mM DDT, 0.02% Tween 20, 0.02% NP-40, 10% glycerol], 1.6 mM dNTPs,每个引物对0.5 μM(如Yang et al. [61.])(附加文件1:表S5),1 u Primestar GXL DNA聚合酶(Takara Bio Inc.;大连,中国)和50 ng基因组DNA模板。使用以下PCR程序使用Geneamp PCR系统9700 DNA热循环仪(PerkinElmer,Norwalk,CT,USA)进行PCR:94℃,1分钟,然后进行30次循环68℃,持续15分钟,以及最终延伸在72℃下10分钟。每次进行九种PCR反应酸浆属物种。将上述反应得到的PCR产物大致按比例混合,进行Illumina MiSeq测序。按照制造商的协议(Illumina),这些混合物被破碎并用于短插入(500 bp)文库构建[17].在中国科学院昆明植物研究所中国西南野生物种种质资源库,利用Illumina Miseq机器对不同物种的DNA文库进行配对,读取长度为250 bp。
基因组组装,注释和比较分析
采用从头法和参考导向法组装cp基因组。首先,使用NGS QC Toolkit v2.3.3将Illumina短reads组装成contigs (www.nipgr.res.in ngsqctoolkit.html).其次,使用CLC Genomics Workbench version 8 (CLC Bio, Aarhus, Denmark)和SOAPdenovo (http://soap.genomics.org.cn/soapdenovo.html), k-mer长度为63。第三,利用BLAST (http://blast.ncbi.nlm.nih.gov/)默认参数。将大于1000 bps的输出支架/斑块被映射到参考CP基因组p . peruviana(NC_026570)。最后,我们根据参考基因组确定了支架/contigs的排列顺序,并通过将原始读取映射到组装来解决存在的任何缺口。
双细胞器基因组注释器(DOGMA) (http://dogma.ccbb.utexas.edu/)工具(62.]用来注释四个完整的酸浆属cp基因。基于参考基因组(NC_026570)手工校正蛋白编码基因的起始密码子和终止密码子及内含子位置。和tRNA scan-SE 1.21版本[63.,以获得和鉴定tRNA基因。圆形基因图谱是使用OrganellarGenomeDRAW工具构建的,然后手工修改[64.].注释后的CP基因组被提交给GenBank数据库(GenBank登录号:MH045574,MH045575,MH045576和MH045577)。CP基因组比较五酸浆属使用MVISTA程序进行物种(http://genome.lbl.gov/vista/mvista/about.shtml.).使用mega6软件分析GC含量、密码子使用和系统发育分析,如下图[65.].
重复序列和SSR分析
Perl脚本MISA (http://pgrc.ipk-gatersleben.de/misa//)[66.]用于探测潜在的微卫星(SSRs)酸浆属cp基因。参数设置如下:单核苷酸SSRs 10个重复单元,二核苷酸SSRs 5个重复单元,三核苷酸SSRs 4个重复单元,四、五、六核苷酸重复单元3个重复单元。使用REPuter在cp基因组中识别正向(直接)重复、反向重复和回文重复,最小重复大小为30 bp,序列一致性为90%(汉明距离为3)[67.].
系统发育分析
阐明…的系统发育地位酸浆属利用代表11属的36种茄科植物的cp全基因组序列进行了多次比对1:表S6),包括黄芩(NC_027262)和美国旧址(NC_028533),外围集团。利用MAFFT7.017和ClustalX比对软件对所有物种的cp全基因组序列进行比较分析,必要时进行人工调整[68.].使用Mega 6进行最大似然(ml)和邻接(nj)分析[65.],使用具有不变站点(GTR + G + I)的伽玛分布站点的替换速率的一般时间可逆模型,具有完整的差距消除和1000个引导重复,以确定分支支持,如在Mega中实现。在Mega的模型测试之后选择核苷酸和系统发生推理模型。
数据和材料的可用性
完整的叶绿体基因组p . angulata,P. Alkekengi.var。franchetii,p .最小值和p .下毛竹已提交至NCBI数据库(https://www.ncbi.nlm.nih.gov/)Genbank登录号MH045574(p . angulata),MH045575(P. Alkekengi.var。franchetii),MH045577(p .最小值)及MH045576 (p .下毛竹).所有其他数据和材料产生在本手稿可从通讯作者在合理的要求。
缩写
- Cp:
-
叶绿体
- 其:
-
内部转录的垫片
- 苏维埃社会主义共和国:
-
简单序列重复
- 国税局:
-
倒置重复
- LSC:
-
大单副本
- SSC:
-
小单副本
- ml:
-
最大似然
- NJ:
-
邻接
- BS:
-
分支机构的支持
参考
- 1.
Whitson M, Manos PS:解缠酸浆属(茄科)从Physaloids:一个physalae的双基因系统发育。系统机器人。2005;30(1):216 - 30。
- 2.
张卫宁,童文耀。本属植物的化学成分及生物活性酸浆属.化学Biodivers。2016;13(1):48 - 65。
- 3.
冯绍成,蒋美美,史玉军,焦柯良,沈昌杰,卢俊杰,应QC,王洪志。核糖体DNA ITS2区的应用酸浆属茄科植物DNA条形码与系统发育研究。植物学报。2016;7:1047。
- 4.
Wei JL,Hu XR,杨杰,杨卫生。鉴定单拷贝之间的单拷贝基因酸浆属和Solanum lycopersicum和遗传多样性分析酸浆属使用分子标记。7。2012;(11):e50164。
- 5.
Martinez M.修订酸浆属部分Epeteiorhiza(茄科)。Ann Ins Biol Bot. 1998; 69:71-117。
- 6。
中国科学院。《中国植物志》,第67卷。中国:科学出版社;1978.50页。
- 7。
国家药典委员会。中华人民共和国药典(第一卷)北京:化学工业出版社;2015.p . 360 - 1。
- 8。
姬升,元友,马ZJ,陈泽,甘ls,马xq,黄ds。醌类还原酶(QR)的诱导physalis pubescens.l .(茄科)。类固醇。2013;78(9):860 - 5。
- 9。
丁辉,胡志军,于奕礼,马志军,马晓强,陈智,王东,赵晓峰。醌类还原酶(QR)的诱导酸浆属angulatal . var。摘要Bonati(茄科)。类固醇。2014;86:32-8。
- 10。
徐XM,关益,山SM,罗杰,孔利。Saphysalin-型含油苷空泡最小值.Phytochem。2016;15:1-6。
- 11.
Zhan XR,Liao Xy,Luo Xj,朱yj,冯SG,Yu Cn,Lu Jj,Shen Cj,Wang Hz。比较代谢组和蛋白质组学分析揭示了梅贾诱导的切叶接地中的生物活性化合物积累的调控机制(酸浆属angulatal .)的根源。食品科学,2018;66(25):6336-47。
- 12.
皮番茄的遗传多样性和遗传结构(酸浆属philadelphica在墨西哥西部。植物遗传资源学报。2015;62(1):141-53。
- 13.
雷德兰·吕,笑道,康马尔SV。完整的MD-2菠萝叶绿体基因组序列及其在亚组成的亚群中其他植物中的比较分析。BMC植物BIOL。2015; 15:196。
- 14.
Martin Avila E,Gisby MF,Day A.植物中叶绿体基因组的无缝编辑。BMC植物BIOL。2016; 16(1):168。
- 15.
关键词:多年生黑麦草,叶绿体,全基因组序列多年生黑麦草l .)。DNA研究》2009;16(3):165 - 76。
- 16。
雷文杰,倪丹普,王艳娟,邵建杰,王小春,杨东,王俊生,陈慧敏,刘超黄芪膜.Sci众议员2016;6:21669。
- 17。
罗y,ma pf,李ht,杨杰,王h,李迪兹。体积系统核糖分析分析TOFIELTIACEAE作为早期发散单子宫秩序的根源。基因组Biol Evol。2016; 8(3):932-45。
- 18。
marama叶绿体基因组的一个新反转Tylosema esculentum).J Exp Bot。2017; 68(8):2065-72。
- 19。
Doyle JJ,Doyle JL,Palmer JD。两种基因的多种独立损失和豆类叶绿体基因组的一种内含子。SYST BOT。1995; 20(3):272-94。
- 20.
Millen RS, Olmstead RG, Adams KL, Palmer JD, Lao NT, Heggie L, Kavanagh TA, Hibberd JM, Gray JC, Morden CW等。许多平行损失infA从叶绿体DNA期间从核心转移到细胞核中的高血管培训期间。植物细胞。2001; 13(3):645-58。
- 21.
Guisinger MM, Chumley TW, Kuehl JV, Boore JL, Jansen RK。质体基因组序列的意义香蒲(Typhaceae,Poales)用于了解Poaceae的基因组进化。J Mol Evol。2010; 70(2):149-66。
- 22.
DR . Shapiro, Tewari KK。转移RNA基因的核苷酸序列Pisum一叶绿体DNA。植物mol biol。1986; 6(1):1-12。
- 23.
Saski C, Lee SB, Daniell H, Wood TC, Tomkins J, Kim HG, Jansen RK。完整的叶绿体基因组序列京霉素Max.并与其他豆科植物基因组进行比较分析。植物学报。2005;59(2):309-22。
- 24.
Hirao T, Watanabe A, Kurita M, Kondo T, Takata K.全核苷酸序列Cryptomeria japonica叶绿体基因组学与比较叶绿体基因组学:针叶树种基因组结构的多样性。BMC Plant Biol. 2008;8:70。
- 25。
张YZ,MA J,YANG BX,LI RY,ZHU W,SUN LL,TIAN JK,张L.完全叶绿体基因组序列Taxus chinensis.var。mairei(紫杉科):倒重复区域的丧失及与近缘种的比较分析。基因。2014;540(2):201 - 9。
- 26。
Tangphatsornruang S,Sangsrakru D,Chanprasert J,Uthaipaisanwong P,Yoocha T,Jomchai N,Tragoonrung S. Mungbean的叶绿体基因组序列(豇豆属辐射动物)的高通量焦磷酸测序:结构组织和系统发育关系。DNA研究》2010;时间为17(1):月11日至22日。
- 27。
帕尔默·JD,汤普森WF。绿豆和豌豆叶绿体基因组的重排。中国科学院院刊1981;78(9):5533-7。
- 28。
李XW,杨y,亨利rj,rossetto m,王yt,chen sl。植物DNA条形码:从基因到基因组。Biol Rev Chab Philos SoC。2015; 90(1):157-66。
- 29。
董文平,徐超,吴鹏,程涛,于军,周少林,洪元元。石杉科植物叶绿体基因组数据的分析。分子系统学进展。2018;126:321-30。
- 30。
杨智,赵婷婷,马庆华,梁立生,王光新。比较基因组学和系统发育分析揭示了该植物叶绿体基因组变异和种间关系Corylus.(Betulaceae)物种。前植物SCI。2018; 9:927。
- 31。
AXELIUS B.基于形态学数据的液体酸碱(Solanaceae)的系统发育关系。Amer J Bot。1996年; 83:118-24。
- 32。
Olmstead RG, Bohs L, Migid HA, Santiago-Valentin E, Garcia VF, Collier SM。茄科植物的分子系统发育。分类单元。2008;57(4):1159 - 81。
- 33。
冯SG,娇吉,朱yj,王鹤,江我,王赫兹。物种的分子鉴定酸浆属(Solanaceae)使用候选DNA条形码:叶绿体psba-trn.基因间区域。基因组。2018;61(1):15 - 20。
- 34。
Vargas-Ponce O,Perez-Alvarez LF,Zamora-Tavares P,Rodriguez A.评估墨西哥稻番茄种的遗传多样性。植物mol Biol rep。2011; 29(3):733-8。
- 35.
冯曙光,朱永军,余春林,焦柯良,姜敏,JJ YL,沈昌杰,应QC,王洪志。基于SCoT分析,开发种特异性SCAR标记进行鉴定酸浆属(茄科)的物种。麝猫。2018;9:192。
- 36.
关键词:被子植物,质体基因组,焦磷酸测序BMC Plant Biol. 2006;6:17。
- 37.
王松,杨长平,赵学勇,陈松,曲国珍。完整的叶绿体基因组序列桦木属platyphylla:基因组织,RNA编辑,比较和系统发育分析。BMC基因组学。2018;19(1):950。
- 38.
Quail MA, Smith M, Coupland P, Otto TD, Harris SR, Connor TR, Bertoni A, Swerdlow HP, Gu Y.三个下一代测序平台的故事:离子激流、太平洋生物科学和Illumina MiSeq测序器的比较。BMC基因组学。2012;13:341。
- 39.
张YX,IAFFALDANO BJ,庄XF,Cardina J,Cornish K.叶绿体基因组资源和分子标记从杂草亲属区分橡胶蒲公英物种。BMC植物BIOL。2017; 17(1):34。
- 40.
张玉军,杜丽伟,刘安,陈建军,吴丽,胡文敏,张伟,Kim K, Lee SC,杨天军,等。完整的叶绿体基因组序列淫羊藿物种:进入系统发育和分类分析的光。植物科学学报2016;7:306。
- 41.
罗杰,侯bw,niu zt,liu w,xue qy,ding xy。光合兰花的比较叶绿体基因组:对系统发育应用的植物学演变的见解及分子标志物的发展。Plos一个。2014; 9(6):E99016。
- 42.
Jansen RK, Wojciechowski MF, Sanniyasi E, Lee S-B, Daniell H.鹰嘴豆质体基因组全序列(Cicer Arietinum.的系统发育分布rps12和clpP豆类中的内含子损失(Leguminosae)。mol phylocyet evol。2008; 48(3):1204-17。
- 43.
Zuo LH,Shang AQ,张S,Yu Xy,Ren YC,Yang Ms,王JM。第一个完整的叶绿体基因组序列榆属物种新创测序:基因组比较和分类位置分析。《公共科学图书馆•综合》。2017;12 (2):e0171264。
- 44.
Kim KJ, Lee HL。韩国人参叶绿体全基因组序列(人参schinseng17种维管植物序列演化的比较分析。DNA研究》2004;11(4):247 - 61。
- 45.
Sun YX,Moore MJ,Meng AP,Soltis PS,Soltis de,Li JQ,王HC。TrochoCheDendRaceae的完全塑性基因组测序揭示了倒置重复的显着膨胀,并表明两种现存物种之间的古常态分歧。Plos一个。2013; 8(4):E60429。
- 46.
崔克思,郑明国,朴淑芳Veroniceae种(车前草科):比较分析和高度分化地区。植物科学学报2016;7:355。
- 47.
陈晓春,李启群,李勇,钱娟,韩建平。叶绿体基因组的Aconitum Barbatum.var。puberulum(毛茛科)从CCS读取使用PacBio RS平台。植物学报2015;6:42。
- 48.
Asaf S, Khan AL, Khan AR, Waqas M, Kang SM, Khan MA, Lee SM, Lee IJ。完整的叶绿体基因组烟草otophora及其与相关物种的比较。前植物SCI。2016; 7:843。
- 49。
Asaf S, Waqas M, Khan AL, Khan MA, Kang SM, Imran QM, Shahzad R, Bilal S, Yun BW, Lee IJ。野生稻叶绿体的完整基因组(选用minuta)及其与近缘种的比较。植物学报2017;8:304。
- 50.
黄鹤,施,刘义,毛苏,高lz。十三山茶花通过高通量测序确定的叶绿体基因组序列:基因组结构和系统发育关系。BMC EVOL BIOL。2014; 14:151。
- 51.
yi x,gao l,王b,su yj,王t。完全叶绿体基因组序列Cephalotaxus奥利维头红豆杉科:头红豆杉叶绿体dna的进化比较及裸子植物中反向重复副本的丢失。生物医学进展。2013;5(4):688-98。
- 52.
姚旭,谭玉华,刘云云,宋勇,杨建平,杨建平冬青属植物(冬青科)。Sci众议员2016;6:28559。
- 53。
Praxelis (Praxelis)叶绿体全基因组序列泽兰属植物catariumVeldkamp),这是一个重要的侵入性物种。基因。2014; 549(1):58-69。
- 54。
NIE XJ,LV SZ,Zhang YX,Du Xh,Wang L,BiraDar SS,Tan XF,WAN FH,WAINE S.完整的叶绿体基因组序列的主要侵入性物种,CROFTON WEED(Ageratina adenophora.).《公共科学图书馆•综合》。2012;7 (5):e36869。
- 55。
pawels M, Vekemans X, Gode C, Frerot H, Castric V, Saumitou-Laprade P.核和叶绿体DNA系统地理学揭示了研究金属耐受的模式物种欧洲居群的距离。拟南芥halleri(十字花科)。新植醇。2012;193(4):916 - 28。
- 56。
刘立,王yw,he pz,李p,李俊,solt de,fu cx。脱岩姐妹属的叶绿体基因组分析和基因组资源开发Oresitrophe和Mukdenia(石杉科),利用基因组略读数据。BMC基因组学。2018;19(1):235。
- 57。
陈建辉,郝志东,徐海波,杨利明,刘光新,盛勇,郑超,郑文伟,程立涛,史俊生。残存木本植物叶绿体的完整基因组序列水杉胡锦涛等程。植物科学学报2015;6:447。
- 58。
Moore MJ, Soltis PS, Bell CD, Burleigh JG, Soltis DE. 83个质体基因的系统发育分析进一步解决了eudicots的早期分化。美国国立科学院。2010年,107(10):4623 - 8。
- 59.
张yj,李迪兹。基于完全叶绿体基因组的系统源研究进展。植物多样性和资源。2011; 33(4):365-75。
- 60.
柯南道尔JJ。小数量新鲜叶组织的快速DNA分离程序。Phytochem公牛。1987;19:11-5。
- 61.
杨建斌,李德志,李海涛。九种新的通用引物对对被子植物叶绿体基因组进行高效测序。生态学报。2014;14(5):1024-31。
- 62.
Wyman SK, Jansen RK, Boore JL。用DOGMA自动注释细胞器基因组。生物信息学,2004;20(17):3252 - 5。
- 63.
Schattner P, Brooks AN, Lowe TM。tRNAscan-SE、snoscan和snoGPS网络服务器用于检测tRNAs和snoRNAs。核酸学报2005;33:W686-9。
- 64.
Lohse M, Drechsel O, Kahlau S, Bock R. OrganellarGenomeDRAW——一套用于生成质体和线粒体基因组物理图和可视化表达数据集的工具。核酸学报2013;41:W575-81。
- 65.
Tamura K,SteCher G,Peterson D,Filipski A,Kumar S. Mega6:分子进化遗传学分析6.0版。mol Biol Evol。2013; 30(12):2725-9。
- 66.
MISA-web:微卫星预测的网络服务器。生物信息学。2017;33(16):2583 - 5。
- 67.
引用本文:陈志强,陈志强,陈志强,陈志强:重复分析在基因组规模上的多重应用。核酸学报2001;29(22):4633-42。
- 68.
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30(4):772-80。
致谢
我们要感谢杭州师范大学生命与环境科学学院对这项工作的支持。我们也感谢野生物种种质资源库(KUN)的技术支持。最后,我们感谢国际科学编辑公司的工作人员,感谢他们为本稿件的编辑提供的服务。
资金
国家自然科学基金项目(no . 31970346);浙江省自然科学基金项目(LY20H280012, LY19C160001);杭州市科技计划项目(20191203B02);中央政府重点项目:中药资源可持续利用能力建设(2060302);浙江省重点研发计划资助项目(2018C02030);浙江省大学生科技创新项目(2019R426027)这些资助机构与这份手稿的出版没有任何关系。
作者信息
从属关系
贡献
SF和HW构思了这项研究,设计了实验,对叶绿体基因组进行了测序,起草了手稿,并最终批准了即将发表的版本;SF、KZ、KJ进行了分子研究;SF、KZ、KJ、YC、CC、YM、LW、XZ、QY分析数据;HW获得了资金,并帮助起草了手稿。所有作者阅读并批准了最终的手稿。
通讯作者
道德声明
伦理批准和同意参与
收集到的酸浆属种在中国广泛分布。实验研究酸浆属品种符合杭州师范大学指南(https://hsdsbc.hznu.edu.cn/c/2014-09-15/897442.shtml),并且不包括相同的遗传转化,保留所用物种的遗传背景。它不需要道德批准。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
关于四个方面的信息酸浆属研究中使用的物种。表S2。叶绿素测序的质量控制叶绿体基因组酸浆属物种。表S3。相对同义词密码子用法(RSCU)五酸浆属叶绿体基因组。表S4。进化分歧之间的进化分歧酸浆属基于完全叶绿体基因组序列的物种。表S5。完整叶绿体基因组扩增的通用引物。表S6。研究了茄科11属的36种植物,并给出了相应的叶绿体全基因组GenBank登录号。
附加文件2:图S1。
基因图P. Alkekengi.var。franchetii叶绿体基因组。外圆的基因是顺时针转录的,而内圆的基因是逆时针转录的。属于不同功能群的基因是用颜色编码的。内圈中深灰色表示GC含量,浅灰色表示AT含量。内环还表明叶绿体基因组包含两个反向重复序列(IRA和IRB),一个大的单拷贝区(LSC)和一个小的单拷贝区(SSC)。地图是使用细胞细胞预构建的。图S2。基因图p .最小值叶绿体基因组。外圆的基因是顺时针转录的,而内圆的基因是逆时针转录的。属于不同功能群的基因是用颜色编码的。内圈中深灰色表示GC含量,浅灰色表示AT含量。内环还表明叶绿体基因组包含两个反向重复序列(IRA和IRB),一个大的单拷贝区(LSC)和一个小的单拷贝区(SSC)。地图是使用细胞细胞预构建的。图S3。基因图p .下毛竹叶绿体基因组。外圆的基因是顺时针转录的,而内圆的基因是逆时针转录的。属于不同功能群的基因是用颜色编码的。内圈中深灰色表示GC含量,浅灰色表示AT含量。内环还表明叶绿体基因组包含两个反向重复序列(IRA和IRB),一个大的单拷贝区(LSC)和一个小的单拷贝区(SSC)。地图是使用细胞细胞预构建的。图S4。五种叶绿体基因组中的氨基酸频率酸浆属物种。图S5。基于36种Solanaceae的完整叶绿体基因组序列的邻居加入(NJ)树。在分支上面的数字表示引导支持,并由红色虚线圈出是五个酸浆属物种。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
冯绍峰,郑凯,焦凯。et al。四个完整的叶绿体基因组酸浆属物种(Solanaceae):灯进入基因组结构,对比分析和系统发育关系。BMC植物杂志20.242(2020)。https://doi.org/10.1186/s12870-020-02429-w
已收到:
接受:
发表:
关键字
- 酸浆属
- 叶绿体基因组
- 分子标记
- 物种鉴定
- 系统发育关系