抽象的
背景
水稻黄斑驳病毒(Rice yellow mottle virus, RYMV)是非洲主要的水稻病原体。三种抗性基因,即RYMV1那RYMV2和RYMV3,之前已经描述了。RYMV1编码翻译发起因子EIF(ISO)4G1和最佳候选基因RYMV2和RYMV3编码的同系物拟南芥核孔蛋白(CPR5)和一个核苷酸结合结构域和富含亮氨酸的重复含有结构域(NLR)蛋白,分别。高电阻是亚洲栽培稻的非常少见(奥雅萨苜蓿),目前只鉴定出两种高度抗药的品种,但在非洲栽培水稻中更为常见(oryza glaberrima)。
结果
在这里,我们在参考集合中报告了抵抗调查的结果268o . glaberrima登记入册。总共40份抗性种质被发现,从而确认电阻的该物种的高频RYMV。我们分析了抗性基因或候选基因的变异此集合根据高深度Illumina的数据或Sanger测序英寸等位基因先前示出具有电阻在31个抗性种质但不是在任何敏感菌观察到相关联。五个原等位基因移码或过早终止密码子的候选基因RYMV2是在耐种质也确定。遗传分析表明,这些等位基因,以及在候选基因的T-DNA插入,分别负责RYMV抗性。所有40个抗性种质最终被连接到验证或候选抗性等位基因在三个抗性基因RYMV之一。
结论
这项研究表明RYMV2抗性基因对其同源拟南芥CPR5并在该位点上发现5个新的抗性等位基因。它还证实了耐药与NLR候选基因中富含亮氨酸重复序列中的氨基酸替代之间的密切联系RYMV3.在此基础上,作者还综述了水稻抗RYMV的遗传多样性o . glaberrima物种,同时强调与之间的多样性形成对比模式o . glaberrima和o .漂白亚麻纤维卷此特征。不同的抗性基因和等位基因将在选育到RYMV可持续田间抗性品种的工具。
背景
奥雅萨苜蓿水稻在亚洲被驯化,在世界上几乎所有的水稻种植区都有种植。然而,一个独立的水稻驯化过程发生在非洲,这产生了栽培的物种oryza glaberrima[1那2].更有效率的o .漂白亚麻纤维卷物种在东非在1000多年前和西非在十六世纪介绍,逐步取代o . glaberrima.Breeding initiatives over the last 60 years have essentially concernedo .漂白亚麻纤维卷品种并进一步扩大了两种物种品种之间产生潜力的差距。尽管如此,o . glaberrima有特定的兴趣和适应局部应力的特征,例如干旱,铁毒性,不孕症和杂草竞争[3.那4.].这种丰富的基因多样性来源具有很大的育种兴趣,以增加全球变暖和减少投入的稻米产量。o . glaberrima因此被引入育种计划[5.那6.]导致例如非洲(NERICA)品种的新米饭,这是由此产生的o .漂白亚麻纤维卷Xo . glaberrima种间杂交并在21世纪成功传播[7.那8.].
水稻黄斑驳病毒(RYMV)是非洲的地方病,对灌溉地区或低地地区的水稻作物造成重大损失[9.].高抗性,定义为缺乏症状和病毒检测系统叶片使用血清学方法,似乎是非常罕见的o .漂白亚麻纤维卷,目前只鉴定出两个高抗性品种[10.那11.,而31则是高度耐药的o . glaberrima已接获报告[12.那13.].而且,虽然这两个人o .漂白亚麻纤维卷抗病品种具有相同的等位基因RYMV1编码翻译起始因子的抗性基因[14.]并且似乎限制了细胞对细胞病毒Mouvement [15.],至少三种不同RYMV1抗性等位基因在o . glaberrima[12.].这些结果表明o . glaberrima这一性状的多样性对水稻育种尤其有用。
两个额外的抗性基因,即。RYMV2和RYMV3,已被映射o . glaberrima物种。RYMV2-介导的抗性与1 bp的缺失有关,导致同源基因的一个空等位基因拟南芥的致病性相关蛋白-5的组成型表达(CPR5)双亲生绘制群体和一系列的核偶姻基因o . glaberrima登记入册(13.].在拟南芥,这CPR5.核孔蛋白基因参与防御机制和衰老的调控[16.那17.].考虑序列同源性和与之前的研究一致[13.那18.],候选基因RYMV2在下文中被称为CPR5-1,尽管其核孔蛋白在水稻防御机制中的作用和参与尚未有文献记载。最近,一个核苷酸结合域和富亮氨酸重复基因(NLR)超家族的基因被确定为最佳候选基因RYMV3显性抗性基因[19.].此后,此基因称为NLRRYMV3.抗性与蛋白质的富氨氨氨酸富含重复(LRR)结构域中的单个氨基酸取代相关,已知该酶可参与病原体识别特异性。
在这里,我们描述了ryMV抗性基因或候选人的多样性o . glaberrima涵盖物种的地理分布,包括165个完全序列的加入[20.那21.].我们还验证了候选基因RYMV2使用在o . glaberrima多样性和o .漂白亚麻纤维卷本文分别突变体。
结果
在一份样本中筛选对RYMV的抗性o . glaberrima种质
Thiemele等。[12.]和orjuela等。[13.]筛选的120个种质o . glaberrimaorjuela等人描述的集合。[20.,共发现31份高抗性材料。本研究采用双抗体夹心酶联免疫吸附试验(DAS-ELISA)对4种植物的系统叶片进行了抗性表型分析。与Thiemele等人报告的病毒分离物相同[12.]和orjuela等。[13.使用了)。在之前报道的31份抗性材料中,我们确认了28份抗性材料,3份敏感材料,可能是由于种子种群之间的异质性。148份新供试材料中,大部分4株均明显敏感。然而,在有种子的12份材料中观察到高抗性植株,这些植株的高抗性表型在其他植株中被证实(见表)1;额外的文件1:表S1)。其中8种,高达1 / 3的植物使病毒增殖,表明抗性外显率不完全或可能的抗性打破事件,如以前报道的[18.那19.那22.].然而,易感植物的易感速率与携带的TOG7291的加入率没有显着不同RYMV2主基因(Fisher精确测试,P. > 0.05) and these accessions were thus considered resistant. Finally, the accessions identified as being resistant in this study were: Og26, Og111, Og133, Og183, Og213, Og256, Og406, Og423, Og447, Og452, Og491 and Og498 (Table1;额外的文件1:表S2)。因此总共40个种质总分268是到RYMV的BF1分离高度抗性。
RYMV抗性基因或候选基因的等位基因挖掘
在耐药o . glaberrima以前的研究结果表明,12份材料中有一个抗性等位基因RYMV1基因[12., 7有与之相关的等位基因RYMV2在介导性CPR5-1基因[13.],1有一个抗性等位基因RYMV3,对于那么NLRRYMV3是候选人[19.],虽然Tog5672加入携带阻力等位基因RYMV1和RYMV3[19.].在剩下的19份材料中,导致抗性的基因或等位基因尚不清楚。
多态性的RYMV1吉恩,在RYMV2和RYMV3候选基因中,CPR5-1基因与NLRRYMV3分别在174处进行分析,包括40个耐抗性附加和134个易感值。对于165种附加,可以获得全基因组序列[1那21.那23].对于九种额外的抗性腐蚀,靶基因的部分或完全序列是从硫脲等人获得的。[12.[聚合酶链式反应(PCR)片段的桑克序列。
等位基因矿业RYMV1
共检测到10个单核苷酸多态性(SNPs)或小插入/删除(indels),定义9个不同的单倍型RYMV1基因(附加文件)1:表S3)。在核苷水平的三种最常见的单倍型表示载体的83%,与CG14加入的蛋白质变体相对应,而其他蛋白质变体在不到5%的豁免中检测到其他蛋白质变异。五个突变 - 三个SNP和两个吲哚 - 位于外显子中,所有导致氨基酸变化(图。1a).在易感材料中存在一种引起单一氨基酸取代的氨基酸(P541L)。其他的,之前被描述为描述性的rymv1–3(R322_D324DEL,S576N),rymv1–4(E321K)和rymv1–5(K312_G315delinsN)抗性等位基因[12.那14.,与整个采集过程中的阻力有关。15份抗性材料携带这些抗性等位基因(见表)2;额外的文件1表S2),包括本研究中鉴定为抗性的两份材料,Og208和Og423,它们携带等位基因rymv1–3和rymv1–4, 分别。
蛋白质水平抗性基因或候选物的变体。为RYMV1(一种), Albar等人引用的等位基因名称[14.和Thiemele等[12.使用]使用,但在易感过程中观察到额外的蛋白质变异是给出的名称“Rymv1-1-Og2“而且提高了澄清的等位基因”Rymv1-1-Og“在[12.]被称为“Rymv1-1-Og1”。为了RYMV2(B.) 和RYMV3(C)候选基因,根据其与RYMV抗性的关联(R)或非关联(S)对不同等位基因进行命名。CG14等位基因为内参等位基因。与抗性相关的多态性用红色表示,而与抗性无关的则用绿色表示。重要的保守域用彩色框架表示。图的右侧显示了携带每个等位基因的品种数量,并区分了165份全序列的品种(没有括号)和9份附加抗性品种(括号中)的品种。由于缺失数据,基因间的总加入数有时会有所不同,从而导致未定义的等位基因
等位基因矿业CPR5-1
在CPR5-1基因,均在核苷酸水平基于从屈布里等人的基因组数据中检测到12个多态性。[21.](附加文件1:表S4)。然而,在这项分析中使用的过滤器阻碍了检测密码子17上的1个bp缺失,该缺失特征是耐药Tog7291的等位基因[13.]因为它位于一个人工创建的SNP集群中,可能是由于一个富含GCC的区域[24].尽管如此,所有收集的标本都已经在此位置根据CAPS标记进行了基因分型[13.].此外,通过手动管理Tog7291登录的读对齐数据(BAM文件)来确认删除。13个多态性CPR5-1基因在核苷酸水平上定义了8个单倍型和8个蛋白质变异(图。1b;额外的文件1:表S4)。在种质的89%中观察到CG14参考单倍型。通过移码(A18FS,G75fs,N229fs,A390fs)或过早终止密码子(R222 *,L485 *)六个单倍型进行了表征,导致蛋白的截短形式,而从蛋白序列的3至93%的节约。有趣的是,这些单倍体关注19个种质说都具有很高的耐RYMV(表2;额外的文件1:表S2),与先前在七个种质[描述的最常见的是所述单倍型Tog729113.,而其他的单倍型频率较低,在一到三份遗传中发现。最后,4个rymv易感基因(Og186、Og426、Og459和Og89)具有相同的单倍型,其特征是内含子中有3个SNPs和3个引起氨基酸替换的SNPs (T310K、L396F、N433D)。
等位基因矿业NLRRYMV3
的可变性NLRRYMV3,在核苷酸水平上有66个多态性,远远大于在RYMV1和CPR5-1(附加文件1:表S5)。然而,作为作图参考的Nipponbare序列与CG14序列之间存在显著差异,可能低估了第一个内含子的多态性那这可能阻碍了该地区正确的定位和广泛的SNP呼叫。共检出11个单倍型,其中71.5%为CG14单倍型。49个突变位于外显子,包括35个非同义突变(图。1C)。这些突变定义了十种蛋白质变体,其中三种是特定的抗性探索(表2;额外的文件1:表S5):与参考等位基因相比,两种呈单个氨基酸取代(PIDON等人中描述的K779R。[19.],和A823V),而第三个显示该LRR结构域(S672fs)和11个氨基酸的置换的移码。Tog5307和Tog5672 - - 在这两个种质观察K779R突变已知携带的抗性等位基因RYMV3[19.],还有5种表现出抗性表型,但没有携带抗性特异等位基因RYMV1或者CPR5-1,提示其抗性可能与K779R突变有关。相反,携带A823V突变体(Og20、Og36)的材料也具有已知的抗性等位基因RYMV1(rymv1–4和rymv1–3分别)和携带S672FS突变的录取(OG87,OG103)具有一个CPR5-1等位基因与抗性相关。
最后,所有的抗加入描述在o . glaberrima集合进行与抗性相关的等位基因的三个分析的基因中的至少一个(表2)。
与比较o .漂白亚麻纤维卷
而且,我们寻找多态性RYMV1, CPR5-1和NLRRYMV3在里面o .漂白亚麻纤维卷基于SNP-Seek数据库[25,汇集了3000个水稻基因组计划的基因分型数据[26].鉴定了17个非同义突变RYMV1它们导致氨基酸替换或小缺失,但在真核起始因子4G(MIF4G)的中间域中仅发生三。那在赋予RYMV抵抗的所有突变(附加文件)1:表S6)。其中一个(A303D)只出现在少数o . glaberrima包括3000水稻基因组计划,以及所有来自我们o . glaberrima收集。因此,这种突变被认为是特异性的o . glaberrima并且不与至RYMV抗性相关。另外两个,即K352R和P395S,在四个和10能分别检测到的,。而坐落在MIF4G域,他们并没有在其在抗性等位基因到目前为止所描述的突变的,而是检测到至少28个氨基酸下游的15个氨基酸的区域发生。在检测到二十三个非同义突变CPR5-1基因(附加文件)1:表S6)。然而,它们都没有导致不合时宜的终止密码子或帧移。和我们在o . glaberrima数据集,o .漂白亚麻纤维卷表现出高度的可变性NLRRYMV3位点,具有112个非同义突变(附加文件1:表S6)。在10.4%的样本中检测到8个突变,导致终止密码子或移码。另外104例出现了框架突变,导致蛋白质中氨基酸替换或单个氨基酸插入或删除。有趣的是,三个o .漂白亚麻纤维卷spp。indic材料携带与RYMV抗性相关的K779R突变体o . glaberrima.这些加入共享一个非常特异的单倍型,其中27个额外的罕见非同义突变,将它们与其他不同o .漂白亚麻纤维卷和o . glaberrima登记入册。
功能突变丧失CPR5-1基因赋予rymv的抵抗力
进行遗传分析以检查截断的CPR5-1形式之间的关联o . glaberrima和RYMV阻力。抗性品种Og256、Og213、Og491、Og133和Og183分别代表CPR5-1的不同截尾形式,与易感品种杂交o . glaberrima加入(OG82或OG431)和耐抗的TOG7291加入,其电阻由其控制RYMV2[13.].除了除(OG183 x TOG7291)之外的所有组合获得F2种子,每群抗性抗性,每种群体至少45个F2植物进行了表型。所有与易感OG82或OG431载体开发的群体中注意到的抵抗偏析与1R:3S隔离比例一致(表3.),表明抗性为单基因隐性控制。基因分型的CPR5-1基于Sanger测序的Sanger测序总共281个植物进行基因,并为所有其他群体切割扩增的扩增的多晶序列(帽)或衍生的裂解扩增的多晶序列(DCAPS)标记。大多数用于截断形式的蛋白质纯合的植物是抗性的(76分,79分),而其他其他人(202分中的200分)易感,显示了CPR5-1等位基因状态和RYMV电阻。我们假设,那不适合这种模式的5株误判的可能是因为缺乏接种或电阻击穿。此外,从与耐Tog7291加入开发群体256个F2植株抗性(表3.),虽然一个人易感。这些结果表明,不同截短的形式的CPR5-1是阻力等位基因RYMV2隐性抗性基因。
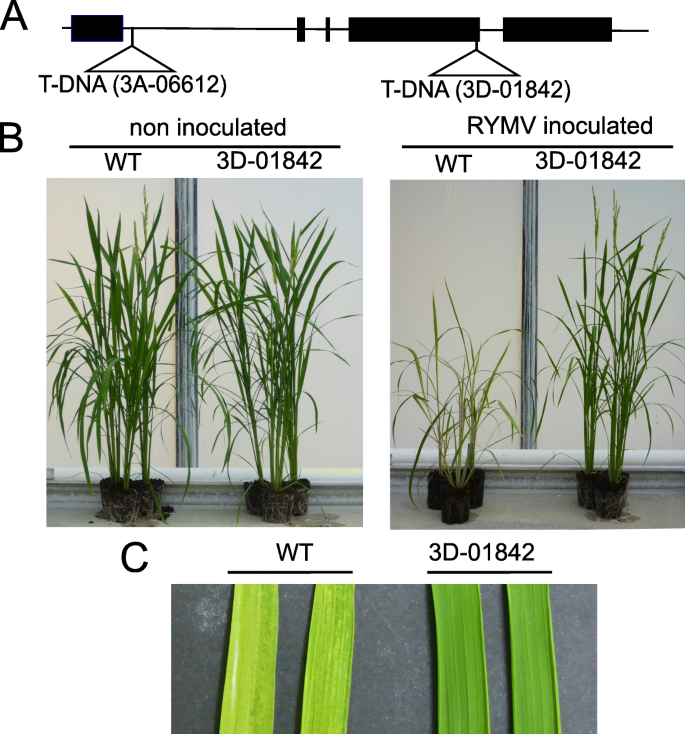
此外o .漂白亚麻纤维卷线在CPR5-1基因进行了表征。T-DNA插入突变体线标记在CPR5-1基因通过搜索侧翼序列数据库进行鉴定[27]由Jeon等人开发的突变库。[28和郑等人[29].两个独立的T-DNA插入CPR5-1通过对T-DNA侧翼区测序,证实了该基因的存在。在3D-01842细胞系中,T-DNA插入ATG下游1975 bp的第4外显子;在3A-06612细胞系中,T-DNA插入ATG下游315bp的第一个内含子(图)。2一种)。这些突变体的表型在最少的12种纯合的纯合中进行。播种后十周,3D-01842和3A-06612与野生型对照相比,非接种突变体未显示出植物形态或开发的任何可见差异(图。2b).与未接种的对照相比,接种RYMV的突变体没有表现出任何症状或生长下降,而野生型植株表现出非常明显的黄变和斑驳症状,明显的生长下降甚至生长停滞(图2)。2(b和c)此外,与野生型植物相反,根据ELISA检测结果,突变体没有积累病毒。共分析了117株假T3或F3植物,它们因一种或另一种T-DNA插入而分离。除一株植物外,在纯合状态下观察到抗性和T-DNA插入之间存在完美的共分离(表4.)。这表明,在两者中o .漂白亚麻纤维卷和o . glaberrimaCPR5–1的改变形式导致RYMV抵抗。
T-DNA突变株3D-01842和3A-06612的特性。一种结构CPR5-1基因与P.ositions of T-DNA, inserted 315 bp downstream of the ATG in the 3A-06612 line and 1975 bp downstream of the ATG in the 3D-0184 line. Exons are represented as black boxes and introns as black lines.B.10周龄未接种植株上野生型对照和3D-0184突变体的表型,以及8 在接种植株上接种RYMV数周后,在整个植株水平上。C野生型对照的叶子和3D-0184突变体与RYMV接种后2周
讨论
Orjuela等人的结果。[13.强烈建议CPR5-1是个RYMV2基因,赋予rymv抗性。在这里,我们使用两个独立的T-DNA突变体验证了这一假设o .漂白亚麻纤维卷和六种不同的等位基因导致截断形式的蛋白质o . glaberrima.虽然拟南芥cpr5已知突变体会影响生物抵抗力[16.,这是该基因首次被描述为作物物种的自然抗性基因。在拟南芥, CPR5是一种跨膜核孔蛋白,参与核孔复合体的膜环[17.].功能突变的缺失可渗透核孔并介导细胞周期转录因子的激活,从而导致防御基因的表达。对几种病原体的构成性抗性是导致的表型之一,但突变体显示出额外的有害发育表型,如变小[16.和种子产量[30.,这与作物的生物抗性育种策略是不相容的。其中的一些RYMV2鉴定的抵抗等位基因o . glaberrima对于发现远程发生的停止或颤抖的突变非常可能是无效的等位基因,而是rymv2-R6等位基因保留了93%的野生型蛋白质序列。蛋白质是否完全失去其蜂窝功能或仍有待研究。但是,基于同源性拟南芥[17.],甚至rymv2-R6等位基因将至少缺少一个跨膜结构域。出乎意料的是,这些突变似乎没有明显的有害表型。此外,对几个独立进化并被维持的无效等位基因的检测很难支持强烈的有害影响CPR5-1昏死。这可以通过两种存在进行解释拟南芥CPR5稻米同源物[13.可能存在部分功能冗余。的功能同系物也有可能拟南芥CPR5是CPR5-2而不是CPR5-1.每个副本的细胞功能将有进一步分析。然而,我们的研究结果表明,使用空的或截短CPR5-1水稻育种程序中的等位基因,或通过从o . glaberrima或通过诱变,将实现RYMV电阻的有效途径。类似的突变可以提供其他pathosystems性和种等位基因挖掘窝藏两个同源拟南芥CPR5这可能有助于发现抵抗病原体的新途径。
与观察到的携带相反RYMV1或者RYMV3抗性等位基因,无携带aRYMV2抗性等位基因在筛选的100%的植物中表现出抗性。这可能是由于抗性的不完全外显率造成的,如之前在其他植物/病毒相互作用中所述[31那32]或抗性事件。Pinel-Galzi等人报道的抵抗率高率。[18.]在Tog7291加入携带rymv2-R1抗性等位基因提示抗性打破事件是最有可能的假设。事实上,他们报告的耐药打破率高达96%,这取决于分离株,而其他结果则报告了RYMV1[33] 和RYMV3[19.]提出对这些基因较不频繁的电阻击穿。
本研究还揭示了新的抗性来源在RYMV2轨迹。这RYMV1遗址近年来一直是广泛分析的重点[12.那14.那34]这项更大规模的研究发现另外两份材料携带已知的抗性等位基因,但没有发现任何新的抗性等位基因NLRRYMV3基因,我们鉴定了五种额外的辅助,显示了LRR区域中的K779R氨基酸取代,提出的是负责高抗性表型的负责[19.].这五个种质显示高的耐RYMV,这进一步加强了的所述候选状态NLRRYMV3基因,特别是K779R突变,但仍需要正式的功能验证来证实这一点NLRRYMV3对抗性遗传具有特异性,但相应的遗传携带等位基因RYMV1和RYMV2,这足以解释它们的高抗性水平NLRRYMV3- 我序列变异的特征是LRR域的截断,表明功能的丧失,这与抗性的增益不一致。我们认为,这两个序列变异很可能并不赋予RYMV抗性,但需要进一步的遗传分析来证实这一点。
相反,我们没有发现任何令人信服的候选抗性等位基因RYMV1和RYMV23000米基因组项目中的加入基因[26],主要包括o .漂白亚麻纤维卷登记入册。在RYMV1在MIF4G区域发现了两个罕见的突变,需要进一步分析。然而,基于预测的MIF4G域三维结构[14.[它们在α-螺旋发夹的下游发生,形成突起,其中已知负责高电阻的突变。因此,我们不认为这些突变可能是抵抗的候选人。观察到高变异性NLRRYMV3基因座和简单的序列分析不足以确定可能与耐药性有关的突变。特别是K779R突变,它与耐药有关o . glaberrima已被检测到三个o .漂白亚麻纤维卷spp。indic然而,很难推测它们的抗性,因为这些材料在LRR结构域中还有14个额外的罕见突变。
可能没有候选的抗性等位基因RYMV1和RYMV23000水稻基因组计划,主要种质内o .漂白亚麻纤维卷,与表型筛选观察到的该物种缺乏抗性相一致。事实上,只有两份对RYMV具有高水平抗性的材料,如本研究中所描述的,已经被报道o .漂白亚麻纤维卷[10.那11.].这两份来自东非的作品,都带有rymv1–2抗性等位基因。这一结果与检测到的相对较多的抗性等位基因形成了对比o . glaberrima.在本研究中使用的集合的268中,检测到40个高度耐抗性探索,其与收集约为15%。然而,这一速度可能超过,因为预先识别出抗性的大约十个申请[12.那35]在建立收藏时故意将其包括在内[20.].抗性侵染的实际速率o . glaberrima可能更接近8%,这是根据本研究新评估的148份样本计算出来的比例,对此我们没有任何先验知识。尽管如此,这一比率还是远远高于o .漂白亚麻纤维卷.
多样性概况RYMV1那RYMV2和NLRRYMV3基因进行了对比。首先,我们观察到大量的基因突变NLRRYMV3基因,其中35个非同义突变检测到o . glaberrima收集。预计这么高的变异性并已被广泛记录NLR基因家族,众所周知是高诱变的,并且由于植物和病原体之间的军备竞赛,经常处于平衡或多样化选择的状态[36那37那38那39那40那41].其次,RYMV1和RYMV2呈下较低的可变性,分别根据其在植物细胞中的中心作用,分别检测到五个非同义突变。的确,RYMV1为起始因子(异)4G-1 [码14.],翻译启动因素是细胞翻译机械的一部分,而且拟南芥基因同源RYMV2核孔复合体的一个组成部分的编码。因此,这两个基因被认为是保守选择的。有趣的是,基因中五分之三的非同义突变RYMV1十分之六的人RYMV2直接参与抗性表型。在类似的基因/病原体相互作用中,Charron等人的结果。[42]提供了eIF4E基因座上多样化选择的证据,至少部分是由马铃薯Y病毒诱导的选择压力驱动的。正如RYMV最近在19世纪中期出现一样[43那44,病毒和病毒之间并没有长期的共同进化o . glaberrima这可能解释了在抗性位点观察到的等位基因多样性。然而,对这些基因座的选择压力可能是由使用这些相同植物因子的其他病毒施加的。
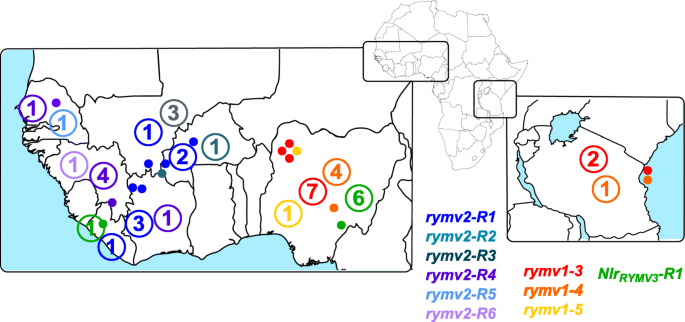
不同的抗性基因和等位基因被定位在Orjuela等人提出的物种遗传多样性树上[20.]对于所有三个抗性基因,携带相同抗性等位基因的材料通常显示出相似的地理来源(图。3.)并在遗传多样性树上聚集(附加文件2:图S1),自o . glaberrima有按地理位置划分的人口结构[45].更令人惊讶的,种质与不同RYMV1或者RYMV2抗性等位基因也似乎是聚集的,尽管特征等位基因的突变是独立的。登记入册,RYMV2抗性等位基因均位于贝宁-尼日尔轴以西RYMV1抵抗等位基因位于该轴的东部。RYMV3显然不符合这种分布模式,但低数量的加入限制了这些发现的范围。几个假设可以解释观察到的结果RYMV1和RYMV2结构化。首先,EIF(ISO)4G和CPR5-1 - 如果确认为核话 - 是大蛋白质复合物的一部分。它们的可变性可能是由相同复合物的其他成员的遗传结构驱动的。此外,环境条件可能导致选择压力差异RYMV1和RYMV2,或他们参加的复杂成员。登记入册,RYMV2抗性等位基因主要源自历史致密水稻种植的区域,而与种质RYMV1根据Portères的说法,抗性等位基因起源于水稻种植较稀疏的地区[46].该轴也与RYMV分离株的两个不同遗传类群的分离兼容[43那44],其中最东端包括能克服大多数已知耐药性来源的高毒性菌株[33].正如前面所强调的,抗性基因通过施加RYMV选择压力下的多元化是非常不可能的,但相反的假设还有待调查,随着对病毒和抗性基因多样性两者的环境条件的影响,一起。此外,像答:芥CPR5[17.], 如果RYMV2是一种效应触发免疫和程序性细胞死亡的调节器,它可能赋予对几种病原体的抗性,并可能在另一种病原体施加的选择压力下进化。由于现有抗病品种数量有限,我们无法进行更详细的群体遗传学分析,但已描述了更多的收集[45那47]现在应该进行调查。
携带抗性等位基因的品种的地理来源RYMV1那RYMV2和NLRRYMV3基因。从屈布里等人获得了种质的地理起源。[21.].有GPS坐标的资料用彩色点表示。在每个国家,携带特定等位基因(有或没有GPS坐标)的入库总数用一个数字表示。背景图来自Freesvg.org并且可在Creative Commons 0许可下提供
结论
我们的研究结果反对RYMV三个已知的抗性基因强调了等位基因多样性。所有40o . glaberrima在本研究中被鉴定为高度抗性的材料至少携带一个已确认或候选抗性等位基因RYMV1那RYMV2和NLRRYMV3(表2).这表明,我们可能已经确定大部分发生在抗性基因o . glaberrima,即使是野生亲缘物种,如oryza barthii,也可能包含原始电阻源。因此,对RYMV抗性基因及其多样性的可靠知识以及对受控条件下抗性分解的频率和分子决定因素的良好评估现已获得[18.那22.那33].这一知识提供了一个机会,以设计策略的抗性基因部署,将优化抗性耐久性。先前的研究结果表明,这三个基因对RYMV的大范围分离株都有效。然而,一些病毒分离物具有进化和克服抗性的高能力,这促使育种人员根据当地病毒的多样性,通过堆积、品种混合或时间旋转来管理抗性基因的使用。到目前为止,只有一个RYMV1起源于水稻的抗性等位基因o .漂白亚麻纤维卷新品种已转化为高产品种[48那49即将被部署到战场上的。为了提高RYMV抗性的可持续性,应促进抗性等位基因或基因组合的使用。
方法
植物材料
这o . glaberrima本研究中使用的集合在orjuela等人中描述。[20.].该系列由法国国家可持续发展研究所(IRD)和非洲稻米中心和所学习的养殖和地理分布所选的稻米中心共同建立。所有入门均在粮食和农业植物遗传资源国际条约中注册。种子的主要原因是在orjuela等人中列出。[20.]种子乘以IRD温室。orjuela等人的登录标识符(ID)。[20.],但CG14参考系加入或以前具有RYMV抗性特征且采用以前使用的名称的品种除外[12.那13.].不同ID之间的通信在附加文件中给出1:表S2。
T-DNA突变系从韩国浦项科技大学获得[28那29].3D-01842系列来自于东京品种的Hwayoung品种和3A-06612系列。对于3D-01842线,植物杂合对于T-DNA插入CPR5-1基因被自授粉,以发育用于插入的伪T2和伪T3后代。由于除了3A-06612系中可获得T-DNA插入的植物的杂合,其纯合的插入常合的植物与Dongjin品种和F1杂交物相交,以依次衍生F2,以及用于T-的F3后代。DNA插入。
阻力评价
植株在温室中生长,播种后约2周机械接种来自布基纳法索的RYMV分离物(BF1)。抗性评价以症状观察为基础,必要时用DAS-ELISA法测定接种后2-3周采集的系统叶片样品中的病毒含量。Pinel-Galzi等人先前描述了接种和ELISA协议的细节[50].样品被认为是阳性时的光密度,减去阴性对照(缓冲液)的值,均优于0,1。
基因数据
基因组数据o . glaberrima从屈布里等得到基于高覆盖基因组测序(平均35X,范围20-55X)种质。[21.].IRGSP 1.0 Nipponbare序列[51]已被用作映射和SNP呼叫的参考。多态性数据库[52那53包括163的基因组数据o . glaberrima和83年O. barthii在第一步中,数据库被筛选出o . glaberrima目的基因的atg终止密码子区域。ORGLA04G0147000.1、ORGLA01G0359000.1和orgla11g017580.1基因模型建立在CG14接入上[1],对应于OS04G42140.1,OS01G68970.1和OS11G43700.1参考Nipponbare序列的基因模型被认为是RYMV1那CPR5-1和NLRRYMV3, 分别。对应于位置24,946,655-24,952,068上用于参考日本晴序列的染色体4的目标区域RYMV1, 40,071,092-40,073,727在日本晴1号染色体上CPR5-1在Nipponbare Composome 11上的26,377,263-26,380,577NLRRYMV3.只有SNPs和indele具有多态性o . glaberrima种质是保守的。在第二步骤中,多态性与GATK 3.7 VariantFiltration [过滤54qal < 200, MQ0 > 4 && MQ0/DP > 0.1, DP < 10, clusterSize 3 in clusterWindowSize 10, DP > 20,000。筛选掉缺失数据超过10%或杂合数据超过10%的snp,以单次阅读确定的基因型为缺失数据。基因组数据o . glaberrimaCG14 [1]和Tog5681种质[23]RYMV易感CG14的单倍型被用作描述在人群中观察到的变异的参考。
来自3000个水稻基因组计划的基因组数据[26]从Rice SNP-Seek数据库检索[25].将数据库过滤在上面的目标区域上,用于来自基本SNP集的Indel和非同义SNP,其包括约1800万SNP。从数据库中检索SNP效果,而手动估计indel效果。
Den Dunnen等人提出的序列变体命名法[55用来描述突变及其对CDS和蛋白质的影响。基于本文或以前描述的结果[12.那14.那19.],主导等位基因在大写字母中用第一个字母表示,具有小写的第一个字母;当没有首选假设时,等位基因名称用大写的所有字母写入。为了RYMV2和RYMV3候选基因,不同的等位基因取决于它们的关联(R)或不(S)与RYMV电阻命名。
桑格测序和分子标记
如orjuela等人所述,在叶提取物或DNA上进行PCR扩增。[13.].使用Primer3设计引物[56]除了用于DCAPS标记的引物,其设计与DCAPS Finder工具的设计[57].引物序列在附加文件中提供2:图S2。部分或全部桑格测序RYMV1那CPR5-1和NLRRYMV3基因经PCR扩增后转包给Genewiz (Takeley, UK)。
上限和DCAPS标记设计在CPR5-1基因型间多态性位点的鉴定o . glaberrima登记入册。标记特征在附加文件中描述2:图S2和表S7。
T-DNA分离分析是基于一个T-DNA特异性片段和一个包含共同引物的基因特异性片段的扩增。引物序列和位置在附加文件中说明2:图S2。
数据和材料的可用性
这o . glaberrima目前研究分析的基因组数据集已在[21.],并可在税务局的吉瓦资料库,https://gigwa.ird.fr/gigwa/.在本研究中通过Sanger获得的序列在GenBank中可用下参考数字种质到MT348348为MT348356RYMV2,MT348357至MT348360RYMV1,以及MT348361至MT348369NLRRYMV3.本研究中生成的所有其他数据都包含在本文及其补充信息文件中。
缩写
- CPR5:
-
发病相关蛋白-5的组成性表达
- Das-Elisa:
-
双抗体夹心酶联免疫吸附试验
- dCAPS:
-
衍生的裂解扩增的多态性序列
- ID:
-
标识符
- Indel:
-
小的插入/删除
- MIF4G:
-
真核发起因子4g的中间结构域
- 远程雷达:
-
富亮氨酸重复
- NLR:
-
核苷酸结合结构域和富含亮氨酸的重复含有域
- PCR:
-
聚合酶链反应
- RYMV:
-
水稻黄斑驳病毒
- SNP:
-
单核苷酸多态性
参考文献
- 1。
王敏,于宇,王玉华,等。非洲水稻基因组序列(oryza glaberrima)和独立驯化的证据。Nat麝猫。2014;46:982-8。
- 2。
陈志强,陈志强,陈志强,等。从非洲水稻的SNP图推断的驯化历史和地理适应。Nat麝猫。2016;48(9):1083 - 8。
- 3.
infares。非洲米(oryza glaberrima):历史和未来的潜力。Proc Natl Acad SCI U S A. 2002; 99(25):16360-5。
- 4.
Sarla N,Mallikarjuna Swamy BP。oryza glaberrima改进…的来源奥雅萨苜蓿.Sci 2005; 89(6): 955 - 63。
- 5.
Jones MP,Dingkuhn M,Aluko GK,Sermon M. Titerspectific奥雅萨苜蓿洛杉矶o . glaberrimaSteud。旱稻改善的后代。Euphytica。1997年; 94(2):237-46。
- 6.
王志强,王志强,王志强,等。水稻种间杂交对水稻生物多样性的影响oryza glaberrima(steud。)x栽培稻籼(l)。Euphytica。2003;132(1):31-40。
- 7.
Kijima Y,Otsuka K,Sserunkuuma D.评估NERICA对乌干达中西部和西部收入和贫困的影响。农业经委会。2008; 38(3):327-37。
- 8。
Dibba L, Zeller M, Diagne A.冈比亚采用非洲新大米(NERICA)对家庭粮食安全和健康的影响。食品安全内核。2017;9(5):929 - 44。
- 9。
Kouassi NKK,N'Guessan P,Albar L,Fauquet Cm,Brugidou C.米黄色斑驳病毒的分布与鉴定:对非洲农民的威胁。植物DIS。2005; 89(2):124-33。
- 10。
关键词:水稻黄斑驳病毒(RYMV),抗性,遗传基础工厂说。1999;83(10):931 - 5。
- 11.
Rakotomalala M, Pinel-Galzi A, Albar L, Ghesquière A, Rabenantoandro Y, Ramavovololona P, et al.;马达加斯加水稻种质对水稻黄斑驳病毒的抗性。acta botanica yunica(云南植物研究). 2008;22(2):277-86。
- 12.
Thiémélé D, Boisnard A, Ndjiondjop M-N, Chéron S, Séré Y, Aké S,等。水稻黄斑驳病毒第二主要抗病基因的鉴定RYMV2在非洲栽培稻品种,o . glaberrima.Al Appl Genet。2010; 121(1):169-79。
- 13.
水稻黄斑驳病毒的隐性抗性与水稻黄斑驳病毒的同源物有关CPR5.基因,主动防御机制的监管机构。Mol植物微生物相互作用。2013; 26(12):1455-63。
- 14.
Albar L,Bangratz-Reyser M,Hebrard E,Ndjiondjop M-N,Jones M,GhesequièreA.EIF(ISO)4G翻译引发因子在米黄色斑点病毒中赋予高耐药性的高阻。工厂J. 2006; 47(3):417-26。
- 15.
Ndjiondjop MN,Brugidou C,藏S,Fargette d,盖斯基埃A,水稻黄斑驳病毒Fauquet C.高电阻两个栽培水稻品种与细胞的失败细胞运动相关。physiol mol植物疗法。2001; 59:309-16。
- 16.
Bowling Sa,Clarke JD,Liu Y,Klessig DF,Dong X.拟南芥的CPR5突变体表达了NPR1依赖性和NPR1无关的阻力。植物细胞。1997年; 9(9):1573-84。
- 17.
Gu Y,Zebell Sg,梁Z,王S,康B-H,董X.核孔渗透是效应引发的免疫力的会聚信号事件。细胞。2016; 166:1-13。
- 18.
皮内尔-Galzi A,迪布勒伊-Tranchant C,HEBRARD E,马里阿克C,盖斯基埃A,阿尔巴L.突变米黄色斑驳病毒蛋白P2A参与RYMV2基因抗性分解。前植物SCI。2016; 7:1-11。
- 19。
Pidon H, Ghesquière A, Chéron S, Issaka S, Hébrard E, Sabot F,等。细的映射RYMV3:来自米黄色斑块病毒的新抗性基因oryza glaberrima.Al Appl Genet。2017; 130(0):807-18。
- 20.
OrjuelaĴ,木鞋楼CHERON S,Vigouroux Y,亚当H,Chrestin H,等人。通过全球基因分型的非洲水稻遗传多样性的深入分析。Al Appl Genet。2014; 127(10):2211-23。
- 21.
等。对246个新基因组的分析揭示了非洲水稻种植的兴衰。咕咕叫医学杂志。2018;28:2274 - 82。
- 22.
Traoré O, Pinel-Galzi A, Issaka S, Poulicard N, Aribi J, Aké S, et al。水稻黄斑驳病毒对eIF(iso) 4g介导的水稻抗性的适应性。病毒学。2010;408(1):103 - 8。
- 23.
Monat C,Pera B,Ndjiondjop Mn,Sow M,Tranchant-DubReuil C,Bastianelli L等人。德诺维三个组件oryza glaberrima加入提供关于非洲群泛基因组的首要见解。基因组Biol Evol。2017; 9(1):1-6。
- 24.
中村,大岛,森本,池田,吉川,Shiwa Y,等。Illumina定序器的特定序列误差剖面。核酸学报2011;39(13):e90。
- 25。
Mansueto L,Fuentes Rr,Borja Fn,Detras J,Abriol-Santos JM,Chebotarov D等人。米SNP-Seek数据库更新:新的SNP,Indels和查询。核酸RES。2017; 45(D1):D1075-81。
- 26。
3000米基因组项目。3,000个水稻基因组项目。GigaScience。2014; 3(1):7。
- 27。
安S,朴s,郑D-H,李D-Y,姜H-G,余J-H,等。水稻T-DNA标记系末端序列数据库的建立与分析。植物杂志。2003;133(4):2040 - 7。
- 28。
Jeon J-S,Lee S,Jung K-H,Jun S-H,Jeong D-H,Lee J,等。水稻功能基因组学的T-DNA插入诱变。工厂J. 2000; 22(6):561-70。
- 29。
Jeong D-H,AN S,Park S,Kang H-G,Park G-G,Kim S-R等人。生成用于激活标记线的侧翼序列标签数据库japonica.大米。植物j . 2006; 45(1): 123 - 32。
- 30
Heidel AJ,Clarke JD,Antonovics J,Dong X.影响系统获得性耐药途径的突变的适应成本拟南芥.遗传学。2004;168(4):2197 - 206。
- 31
王志强,王志强,王志强,等。拟南芥抗西瓜花叶病毒基因rwm1的克隆为自然抗病基因提供了新的功能。植物j . 2014; 79:705-16。
- 32。
Poque S,Pagny G,Ouibrahim L,Chague A,JP Eyquard,Caballero的M,等。在梅痘病毒感染的RPV1轨迹控制部分抗性拟南芥等位基因变异。BMC植物BIOL。2015; 15:159。
- 33。
Hebrard E,Pinel-Galzi A,Oludare A,Poulicard N,Aribi J,Fabre S等人。鉴定水稻黄色斑点病毒的超潜型型:非洲西部遗传抵抗部署的威胁。植物病理学。2018; 108(2):299-307。
- 34。
ag名词Y, Yelome I, Sie M, Albar L, Ghesquière A, Silue D.抗性选择oryza glaberrima地方品种和它们的具体内育种品系到贝宁米黄色斑驳病毒株。作物科。2019; 119:172-6。
- 35。
Thottappilly GG,Rossel HW。橘子物种耐水稻黄色斑块病毒评价。印度j病毒。1993; 9(1):65-73。
- 36。
McDowell, Simon SA。对R基因进化的最新见解。植物学报。2006;7(5):437-48。
- 37.
郭玉林,菲茨J,施尼伯格K,奥索夫斯基S,曹J,魏格尔D.人类基因组中核苷酸结合位点富含亮氨酸重复编码基因的比较拟南芥.植物杂志。2011;157(2):757 - 69。
- 38.
张y,guo m,shen j,song x,dong s,wen y等。草地上的比较基因组学分析揭示了r基因采用的两个不同的进化策略。SCI批准。2019; 9(1):1-10。
- 39.
Karasov TL,Horton MW,Bergelson J.基因组变异作为植物病原体参数的驾驶员?CurrOp植物BIOL。2014; 18:24。
- 40.
Mauricio R,Stahl EA,Korves T,Tian D,Kreitman M,Bergelson J.抗病性基因多态性的自然选择Rps2的拟南芥.遗传学。2003;163(2):735 - 46所示。
- 41.
田东,Araki H ., Stahl E ., Bergelson J ., Kreitman M.拟南芥.中国科学(d辑:地球科学)2002;99(17):11525-30。
- 42.
Charron C, Nicolai M, Gallois J-L, Robaglia C, Moury B, Palloix A, et al.;自然变异和功能分析为植物eIF4E与potyvirus VPg的协同进化提供了证据。植物j . 2008; 54(1): 56 - 68。
- 43.
Pinel-Galzi A,TraoréO,Séréy,HébrardE,Fargette D.病毒苗的生物地理:米黄色斑块病毒作为案例研究。Currination病毒。2015; 10:7-13。
- 44.
Trovãons,baele g,vrancken b,Bielejec F,Ma,Fargette d等。宿主生态决定了植物病毒的分散模式。病毒evol。2015; 1(1):VEV016。
- 45.
Choi JY, Zaidem M, Gutaker R, Dorph K, Singh RK, puruganan MD.非洲水稻驯化的复杂地理oryza glaberrima《公共科学图书馆遗传学》2019;15:1-34。
- 46.
PortèresR. Berceaux AGRICOLES法原色河畔乐洲africain。ĴAFR历史。1962; 3(2):195-210。
- 47.
Ndjiondjop M-N,Semagn K,豪达AC,Kpeki SB,德罗蒂亚d,母猪M等人。遗传变异和群体结构oryza glaberrima以及使用DArTseq开发迷你核心集合。植物学报2017;8:1748。
- 48.
Bouet A,Amancho An,Kouassi N,Anguete K. Comportement de Nouvelles LigniquesIsogéniquesdeRizInriguéDotéesduGèneDeRésistance(rymv1)《西非地区的难民:科特迪瓦局势》,国际生物化学杂志,2013;7(3):1221。
- 49。
Ndjiondjop Mn,Albar L,Sow M,Yao N,Djedatin G,Thieméléd,等。分子标志物在水稻改善中的整合 - 抗抗素米黄色斑块病毒的案例研究。在:Wopereis M,Johnson D,Ahmadi N,Tollens E,Jalloh A,编辑。实现非洲的稻米承诺。瓦莱特福德:Cabi;2013. p。161-72。
- 50
水稻RYMV接种与抗性鉴定。Bio-protocol。2018; 8(11): 1-13。
- 51
Kawahara Y、de la Bastide M、Hamilton JP、Kanamori H、McCombie WR、Ouyang S等。改进奥雅萨苜蓿使用下一代序列和光学图数据的Nipponbare参考基因组。米。2013; 6(1):4。
- 52
Gigwa v2.2发布。https://gigwa.ird.fr/gigwa/.进入2019年9月9日。
- 53
Sempéré G, Pétel A, Rouard M, Frouin J, Hueber Y, De Bellis F, Larmande P. Gigwa v2 -扩展和改进的基因型调查者。GigaScience。2019; 8 (5): giz051。
- 54
McKenna A,Hanna M,Banks E,Sivachenko A,Cibulskis K,Kernytsky A等。基因组分析工具包:分析下一代DNA测序数据的MapReduce框架。基因组研究。2010;20:254–60。
- 55
den Dunnen JT, Dalgleish R, Maglott DR, Hart RK, Greenblatt MS, McGowan-Jordan J,等。关于序列变体描述的HGVS建议:2016年更新。37哼Mutat。2016;(6):564 - 9。
- 56。
Rozen S,Skaletsky H. Primer3关于一般用户和生物学家程序员的WWW。在:Misener S,Krawetz SA,编辑。生物信息学方法和协议。TOTOWA:Humana媒体;1999. p。365-86。
- 57。
Neff MM,Turk E,Kalishman M.基于Web的单核苷酸多态性分析的底漆设计。趋势类型。2002; 18(12):613-5。
致谢
我们感谢Christine DubReil-Tranchant和FrançoisSabot的帮助,在生物信息学分析中,Gatean Maillot用于T-DNA突变体表征和种子乘法的Harold Chrelin。我们也很感谢Denis Fargette,FrançoisAnthony,Mathias Lorieux和Nils Poulicard为了有用的讨论。我们感谢Gynheung AN提供T-DNA突变线。
资金
这项工作得到了国际农业研究磋商小组水稻农业粮食体系研究计划(rice, 2017-2022)的财政支持。法国Ministère de l ' enseignment Supérieur et la Recherche提供一个博士授权到H. Pidon。资助机构并没有参与这项研究,并收集,分析和解释数据的设计,并以书面稿件。
作者信息
从属关系
贡献
计划,HP和La计划和设计了实验,SC开发了群体,SC,HP和La表现表型,基因分型和测序,HP和LA分析了数据并编写了纸张,AG和SC修改了纸张。所有作者都读过并批准了稿件。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商说明
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:
表S1。以DAS-ELISA结果为例o . glaberrima登记入册。DAS-ELISA were performed on the systemic leaves of individual plants harvested 15 days after inoculation as described in [50].OD是405nm处的光学密度;ODCOR表示OD减去负(缓冲区)控件的平均值。当odcor优于0.1时,样品被认为是阳性的。包括耐用控制措施,致电5681和TOG7291;CG14和OG82为易感。该表呈现在单个实验中获取的数据,在每次加入的8个工厂的子集上,除OG423外,所有所识别为耐药的所有申请。表S2。表征的ID,表型和基因型,其特征在于RYMV抗性。在本研究中的BF1分离物或之前的研究中,在机械接种后评估对RYMV的抗性[12.那13.].抗性基因或候选等位基因参考附录1:表S3、表S4、表S5或既往研究结果[12.].表S3。基因型在RYMV1抗性基因。只有多态性被检测到的位置o . glaberrimaCubry等人分析的集合[21.)被包括在内。核苷酸位置是指IRGSP1.0参考序列o .漂白亚麻纤维卷日本加入[51用作映射参考。突变的影响是基于ORGLA04G0147000.1基因模型上建立的o . glaberrimaCG14加入[1].突变根据由传Dunnen等人提出的命名法描述。[55,除了同义突变和发生在内含子中的突变分别被表示为“syn”和“intron”。在蛋白质水平上的不同变异被认为是不同的等位基因。抗性等位基因的名称以前是由Albar等人确定的[14.和Thiemele等[12.]但是在易感过程中观察到额外的蛋白质变异是给出了名称“Rymv1-1-Og2,为了更清楚地了解,在[12.]被称为“Rymv1-1-Og1”。表S4。基因型在CPR5-1基因,候选人RYMV2.被多态性的检测,只有位置o . glaberrimaCubry等人分析的集合[21.)被包括在内。核苷酸位置参考IRGSP1.0的参考序列o .漂白亚麻纤维卷日本加入[51用作映射参考。突变的影响基于orgra01g0359000.1基因模型o . glaberrimaCG14加入[1].突变根据由传Dunnen等人提出的命名法描述。[55,除了同义突变和发生在内含子中的突变分别被标记为“syn”和“intron”。在蛋白质水平上的不同变异被认为是不同的等位基因。选择等位基因名称是为了区分与RYMV抗性相关或不相关的蛋白变异。表S5。基因型在NLRRYMV3基因,候选人RYMV3.在到的样本中仅检测到多态性位置o . glaberrimaCubry等人分析的集合[21.)被包括在内。核苷酸位置是指IRGSP1.0参考序列o .漂白亚麻纤维卷日本加入[51用作映射参考。突变的影响是基于在上建立的orgla11g01758001基因模型o . glaberrimaCG14加入[1].突变根据由传Dunnen等人提出的命名法描述。[55,除了同义突变和发生在内含子中的突变分别被标记为“syn”和“intron”。在蛋白质水平上的不同变异被认为是不同的等位基因。选择等位基因名称是为了区分与RYMV抗性相关或不相关的蛋白变异。表S6。来自3000米基因组项目的ryMV抗性基因或候选者的多样性[26].只有从基础的SNP组非同义的SNP这里报告。SNP效应从SNP-寻找数据库中检索[25],人工评价吲哚效果。突变对CDS和蛋白质的影响基于基于Nipponbare IRGSP1.0序列建立的Os04g42140.1和Os01g68970.1基因模型[51], 为了RYMV1和CPR5-1,分别。为NLRRYMV3那CD基于OS11G43700.1基因模式,不同之处在于ATG密码子从原始起始密码子下游的180个核苷酸移位,以最佳地拟合在CG14参考序列上建立的orgla11g0175800.1基因模型的相应CD。因此调整了对CD和蛋白质的影响。频率是指完整的换档中的替代变体的百分比。表示,位于PFAM结构域MA3,MIF4G和LRR和HMM Panther的突变中,突变显示。
附加文件2:
图S1。与抗性等位基因种质的位置RYMV1那RYMV2和NLRRYMV3遗传多样性树上的基因。敏感品种用深灰色着色,未进行抗性评估的品种用浅灰色着色。改编自Orjuela等人的遗传树[20.].图S2。引物特性及标记或Sanger测序扩增片段。基因被表示为外显子和灰线内含子的灰色框。引物被表示为三角形并且三角形下面的数字指的是相应的序列。(A,B,C)蓝性状表示扩增,然后用在红色着色的引物进行测序的片段。对应于设计在CPR5-1基因的CAPS或dCAPS特异性标记物(c)中的扩增片段被表示为绿色的性状。表S7:在附加文件2设置在这些标记的附加信息。(d)在线3A-06612和3D-01842在CPR5-1基因T-DNA插入物的位置表示。用于测序的T-DNA侧翼位点和基因分型的存在下,T-DNA特异性和基因特异性引物/不存在插入的在棕色和蓝色分别表示。表S7。CAPS和dCAPS标记的特征。标记名称表明是否有CAPS或dCAPS标记,以及它们针对的是CPR5-1基因的哪些等位基因。引物序列前括号内的数字为附加文件2中引物参考:图S2在具有CG14参考单倍型(WT)或alternate单倍型(R)的植物中,除了琼脂糖电泳难以检测到的低于30 bp的片段外,其余片段的大小均显示出来。CAPS-CPR5-1-R1标记已经在Orjuela等人中进行了描述[13.].
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
毕东,H. Chéron, S. Ghesquière, A。et al。等位基因挖掘解锁了非洲栽培水稻中RYMV抗性基因和等位基因的鉴定。BMC植物杂志20.222(2020)。https://doi.org/10.1186/s12870-020-02433-0
已收到:
公认:
发表:
关键字
- 大米
- oryza glaberrima
- rymv.
- 耐药基因
- CPR5.