抽象的
背景
分子研究表明,一些欧洲人Calypogeia根据形态学标准所描述的种是遗传异质性的,实际上是种复合体。DNA条形码技术已广泛应用于难以确定的物种的正确鉴定、隐种的揭示和新分类群的检测。在地苔类植物中,有些DNA片段被推荐为通用的植物DNA条形码,但在扩增方面存在问题。基于基因组数据的超级条形码技术为物种鉴定提供了新的机遇。
结果
以22人为基础,代表10人Calypogeia物种质体基因组作为超级条形码被测试。虽然不是100%有效,但其物种识别成功率(95.45%)仍然显著。不排除上述结果可能是由隐蔽的物种形成c . suecica,因为我们的结果表明。具有欧洲整体塑料的序列Calypogeia物种,我们也发现了ndhB和ndhH基因和trnT-trnL垫片识别物种100%。
结论
这项研究表明,即使超级条形码不是100%有效,这种方法也没有关闭传统的单位点或多位点条形码的大门。此外,它避免了由于需要扩增选定的DNA片段而产生的许多并发症。在质体数据的基础上,为特定的分类群开发所谓的“特定条形码”,似乎是一种很好的物种鉴别方法。
背景
属Calypogeia苔属是地苔科(地苔门)的四属之一。地苔是苔藓植物的三种(除了苔藓和角苔)之一,世界上大约有7000种地苔[1].肝番是在陆地植物演变中发挥关键作用的生物。随着化石证据表明,利物套是第一个土地植物之一,约有475万年前目前的土地上[2那3.那4.].假设,即利物草是基于大分子数据集的系统发育分析支持的最早发散的土地植物之一[5.那6.那7.那8.].家庭裂纹眼灰质构成具有在CA估计的发散时间的叶状肝曲线(亚类Jungermannidae)中最年轻的思考。5000万年[9.].属Calypogeia是欧洲唯一的属。它包括超过90种已描述的物种[10那11].然而,该属物种多样性最高的地区是热带地区[12].在Holarctis,物种丰富度Calypogeia低得多,在不同的部分只有9-13种。在欧洲,只有九种Calypogeia:C. AzureaStotler & Crotzc . integristipula斯蒂芬。,c . neesiana(Massal。&Carestia) M üll. Frib.,c . suecica(阿奈尔& J.Perss。)妳。Frib。c . muelleriana(Schiffn。)MÜLL。Frib。c . sphagnicola(阿奈尔& J.Perss。)Warnst。& LoeskeC. Fissa.(l)Raddi,C. Arguta.需要雇&蒙特。和C. Azorica.Bischl。那which is endemic for Islands of Macaronesia [13那14].一些物种如图所示。C. Azurea那c . suecica那c . sphagnicola那c . integristipula那c . neesiana在北半球分布较广,在北美、欧洲和亚洲均有报道[15那16那17].
在地苔中,生活史的主导阶段是单倍体配子体。简单的配子体形态提供了有限数量的诊断特征,因此地苔在分类上是一个困难的类群。因此,仅根据形态特征进行种、属/亚属分类和系统发育分析是困难的,而且往往模棱两可[18].
快速发展的分子生物学和生物信息学具有广泛使用DNA序列在分类学研究中的可能性。Hebert等人提出的DNA条形码。[19],已经常用于正确识别难以确定物种,揭示隐秘物种,或检测新的分类群[20.那21那22].遗憾的是,不能为植物选择通用的单位点条形码,这足以在近缘分类群中进行区分。因此,双位点组合(: + matK)获CBOL植物工作小组推荐为最佳植物条码[23].然而,没有在所有植物组中,特别是在肝脏中,这种组合由于困难而良好的普遍性引物良好良好matK轨迹(24那25].高通量测序技术为使用各种植物物种的生物多样性研究中使用基因组数据提供了新的机会 - 超级条形码[26那27].因此,当单轨或多轨道条形码失败时,塑性超级条码是使用植物鉴定的全体积基因组序列,特别是在情况下。证明整个塑性基因组和核rRNA簇对区分密切相关的物种非常有用[28那29那30.,品种和个体基因型[31或隐种[32].近年来,全质体和线粒体基因组越来越多地用于不同分类水平的系统发育分析[33那34那35那36那37].完全基因组序列的分析是检测基因组部分的方法,其对个体植物组具有最佳变化,并且可以充当特定条形码[38].虽然第一种叶绿体基因组中的一种由Liverwort测序Marchantia paleaceaBertol。大约30年前[39[与血管植物相比,数据库中可用的血管植物相比,具有完整基因组的Liverwort种类的数量仍然非常稀缺。到目前为止,全体积基因组已被测序为21只Liverwort Genera和23种[40].
只有几种物种Calypogeia对属进行了分子水平的分析。同工酶和分子标记显示一些欧洲Calypogeia基于形态学标准描述的物种是基因上异质的,实际上是由以前未被识别的物种组成的物种复合物,例如。C. Fissa.[41],c . muelleriana[42那43],c . sphagnicola[44].基于同工酶多态性、细胞学和流式细胞术的研究表明,杂交和基因组复制是物种形成的重要过程Calypogeia属(45那46].四种完整线粒体基因组的比较分析Calypogeia物种发现了意外的内含子丢失[47]表明Liverworts的分子资源仍然是未开发的。该属的分子研究涉及所选物种,并基于叶绿体基因组的小片段[44].
在本研究中,我们:一世)建立并表征了欧洲完全叶绿体基因组序列的组织Calypogeia物种;2)鉴定并评估各种DNA条形码Calypogeia物种III)分析了欧洲种之间的系统发育关系Calypogeia.
结果
裂纹石蛋白质大基因组的结构和序列变异
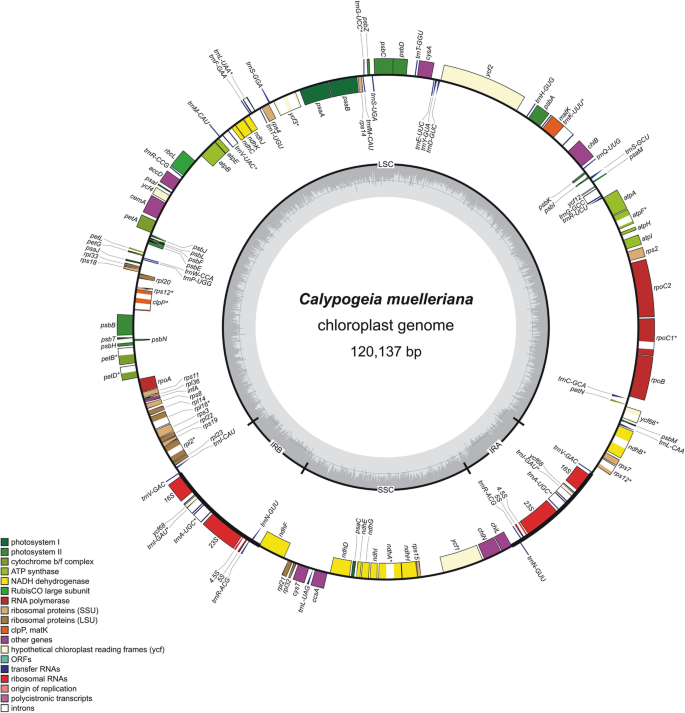
plastid基因组Calypogeia是一个由陆地植物的典型区域组成的环状分子:一个大的单拷贝(LSC),范围为82,377 bpC. Arguta.到83,289 bpc . muelleriana,小单副本(SSC)从19,933 BP到20,016 BP中的范围C. Arguta.和两个反向重复区(IRs)的范围在8,236 bpc . muelleriana至8674 bpC. Azorica.(无花果。1).共鉴定出122个独特的基因(仅考虑到一个反向重复序列区域的拷贝)Calypogeia:81蛋白编码基因,四个核糖体RNA,31转移RNA和六个ycf具有不确定功能的基因。一个基因的ycf家庭 (ycf68)标记为伪基因(图Ѱ)。1).在整个叶绿体基因组中Calypogeia已鉴定出20个内含子。十个蛋白编码基因:rps12那ndhA那ndhB那rpl2.那rpl16那rpoc1.那ycf66那atpF那PETB.那佩德和六次转移RNA:TRNI-GAU.那trna-ugc.那trnV-UAC那trnL-UAA那trnK-UUU那trnG-UCC含有一个内含子,而CLPP.和ycf3有两个内含子。CPDNA(叶绿体DNA)的基础组成如下:(32.3%),C(17.6%),G(17.7%),T(32.3%),总GC含量为34.6%。
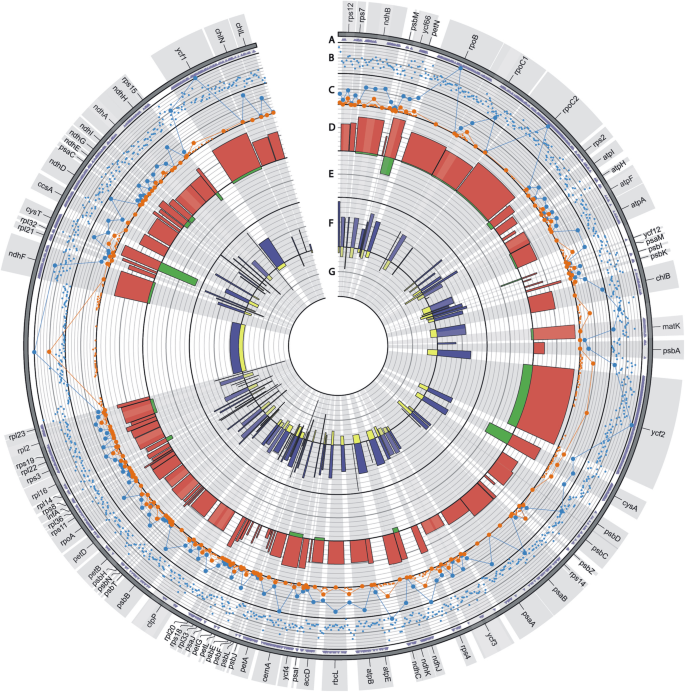
的叶绿体基因组长度Calypogeia属范围为119、628 bpC. Arguta.至120,170bpc . muelleriana(表1).22个代表10Calypogeia物种为122,472bp,其对身份为95.6%。序列变异性是由SNP和诱导的存在引起的,但SNP的数量是主要的。在塑性的编码和非编码区域中,鉴定了666,666,666个SNP,其中36%以上是非纯(图。2,年代2,年代3.).多态位点的百分比(p%)塑料Calypogeia为13.67%,而平均值π为0.035076。
塑料中的SNP和indel变异Calypogeia.跟踪A显示基因内出现非同义SNP。轨迹B和C代表每100 bp窗口大小(最大值为40)中识别的SNPs(小蓝点)和indels(小橙色点)。线形图由B和C轨道组成,代表每个外显子、内含子或基因间隔(snp max.)内的snp(蓝线)和indels(橙线)。值= 400,indel最大。值= 100)。轨迹D代表每个CDS长度的SNPs百分比(最大值= 22),而轨迹E代表每个CDS长度的indels百分比(最大值= 2)。轨迹F代表每个非编码区域长度(最大。Value = 30),而轨道G表示每个非编码区域长度的indels百分比(最大值= 20)
植物质体内遗传变异分析Calypogeia发现长度超过100 bp的最易变的蛋白编码区为囊肿基因(表格1,年代2),与光合作用无关。多态性位点占20.65%%), π = 0.07389。这个867 bp长的区域包含15个indels和164个SNPs,其中92个是非同义的。第二、第四和第五最易变的蛋白质编码区是属于基因ycf具有未定义函数的族:ycf2那ycf66和ycf1分别。这些基因的多态位点比例为17.54 ~ 20.17 (π = 0.04912 ~ 0.07282)。在最易变的蛋白质编码区排名中位居第三matK含218个snp和1个indel (P%π = 19.89, π = 0.056961,表S.2).提到的基因:囊肿那matK那ycf1那ycf2通过将它们分配给适当的物种,正确地区分多达21个序列的基因,而歧视性能力ycf66基因略低,90.9%(表1)。多态位点的百分比(p%)对于蛋白质编码序列,范围为2.5至20.65,而π值从0.00474拉伸至0.07389(表S.2).
在长于100bp的非编码区域中,存在最高变异性rpl14-rpl16垫片(121 bp,p%= 29.75, π = 0.12371)。在该区域共鉴定出30个SNPs和6个indels。第二多态区域,介于rpl32和囊肿多态性位点为28.93%,π = 0.09156。可变的非编码区为atpH-atpF与p的垫片%28.89%和π= 0.11377的值(表S3.).然而,所有的非编码区以及位于前10位的片段并不能区分被测物种。
在不考虑其生物学功能的情况下,人工测定质体中最具多态性的500bp核苷酸片段具有类似于非编码区的可变性。13个易变片段的多态位点百分率在26.4 ~ 30.6%之间,而相同片段的π值在0.07332 ~ 0.09668之间(表S4.).所有这些碎片属于ycf2基因和22个序列中的22个序列中的20-21个,将它们分类为适当的Calypogeia物种(表1).同时考虑到核苷酸多样性(π = 0.1533-0.23231),共发现10个500 bp的多态片段psbA-ycf2正确分配了22个序列中的垫片,以适当的Calypogeia物种。
系统发生
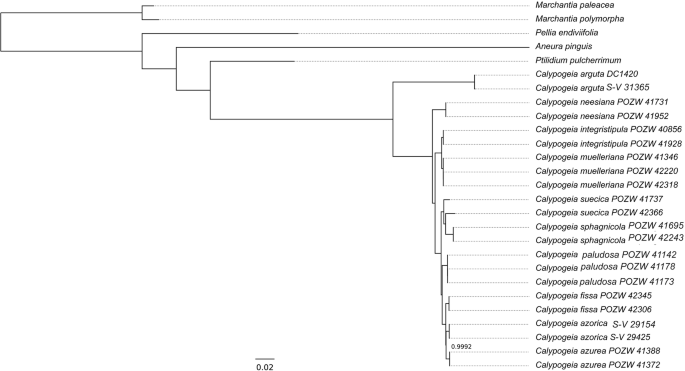
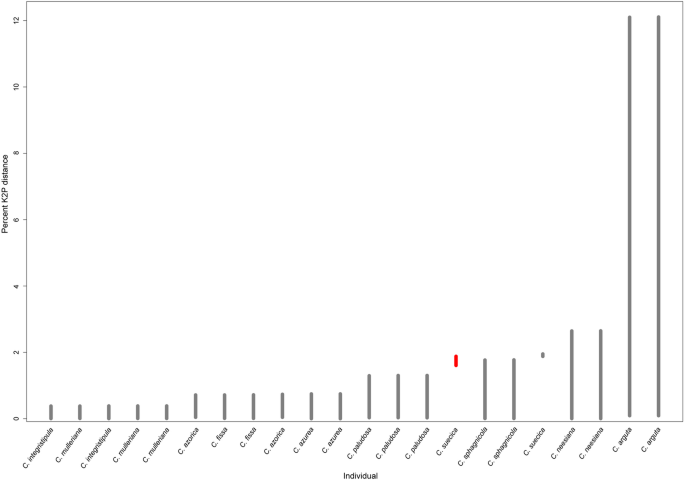
将一组叶绿体CDS序列比对,构建系统发育树(图1)。3.).基于整个质体和氨基酸序列的树与基于cds的系统发育图非常相似(图S1).分析从分区叶绿体CDS数据集推断出几乎所有具有强大分支支持的所有物种的肝曲线。所有副本Calypogeia物种在系统发生树上是正确配对的。与其他物种分离最多的物种Calypogeia系统发生树中的物种是C. Arguta.,正如预期的那样。最有趣的是位置c . suecica分组c . sphagnicola.更重要的是我们之间的距离C. Suecica物种配对是迄今为止最伟大的Calypogeia可能表明两者的对c . suecica物种实际上是两个不同的物种。
物种界定
泊松树进程(PTP)分析分析22Calypogeia个体分为11个物种,这比本研究中使用的物种数量多了一个。PTP分析将两个标本分类为c . suecica作为两个不同的物种。物种划分的支持度较高,范围为0.922 (forC. Arguta.1.00(对于c . suecica) (桌子2).上述结果反映在系统发育树中(图1)。3.,图.. s1),其中每个物种的代表聚在一起属于同一支系,而个体c . suecica彼此分离,并与c . sphagnicola.的两个质体的两面性c . suecica达到97.7%,其他物种为99.6%c . neesiana99.99%(forC. Fissa.).
所有物种的叶绿体基因组彼此不同。发生最强的差异C. Arguta.和别的Calypogeia物种(图。3.,图.. s1),平均间隙距离为0.121079,固定的核苷酸差异的数量从11,329(之间)C. Arguta.和c . suecica)至12,414(之间)C. Arguta.和c . sphagnicola).最大的诱导物种数量为687并发生在C. Arguta.和C. Azorica..结果发现,最相似的物种是c . muelleriana和c . integristipula仅在412个SNP和428 indels中不同。然而,这些差异足以鉴定上述物种,如蜘蛛和物种标识符分析所显示的(图。4.f、表1)。根据这些课程的整个叶绿体基因组Calypogeia该方法可作为一种超条码,成功率为95.45%。蜘蛛分析表明,只有一个质体c . suecica不能用作超级条形码。本种有一个标本不存在条形码间隙(图。5.)。类似地,物种标识符显示出一个标本c . suecica被错误的识别和误认为C. Paludosa..然而,对最易变异的DNA编码片段的分析表明,在所有分析的质体序列中,有一个基因序列可以进行适当的物种鉴定,即ndhB基因(表格1)。在10个最易变的蛋白质编码序列中,有7个基因能够在95%以上的分析序列中正确匹配物种(表1)。的非编码质体序列Calypogeia不太有效。仅有的ndhB内含子在分析的22个序列中正确识别出21个,在90%以上的研究序列中,其他8个非编码片段被正确地分配到物种(表1)1).
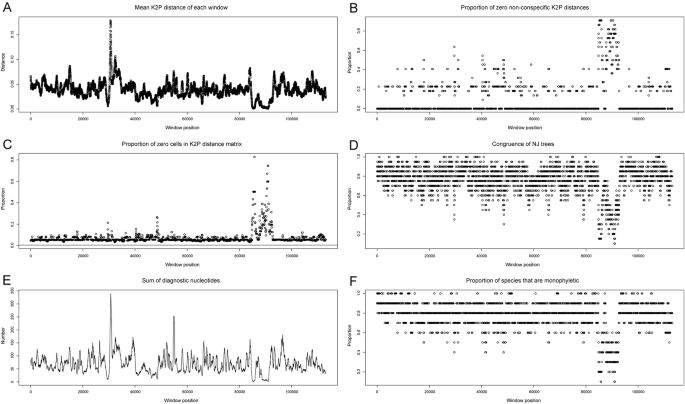
血液序列的几种分析结果Calypogeiasp。使用滑动窗口方法。一种-每个500 bp长窗口的平均木村2参数距离矩阵的曲线图。最大值约为30000 bp,最小值约为90000 bp。B.- 零非特异性距离的比例,发现其最大值约为90,000 bp。C-距离矩阵中零单元的比例。这是最大约90000 bp。与y轴相交于0处的不间断水平线是从完整数据集创建的距离矩阵中零单元格的比例。D.-在Windows和完整数据集中相同的分支的比例。它在大多数窗口相当高,在90000 bp左右的位置明显较低。E.-所有物种的诊断核苷酸位置的总和。这些核苷酸大部分位于约30000 bp位置,最少位于约90000 bp位置。F-单系物种的比例。大约90000 bp的位置明显处于低潮,但大多数位置都能很好地区分物种。
滑动窗口分析也显示了被研究物种集的高分辨能力,尽管低于蛋白质编码序列(表1).在10个最易变的500 bp长的片段中,两个正确地识别了21个序列,而在10个最易变的蛋白质编码区中,多达8个可以正确地区分22个序列中的21个。10个最易变异的500bp长的叶绿体DNA片段均属于该片段ycf2基因。在滑动窗口分析生成的18个最具多态性的片段中,只有两个代表质体DNA的其他区域:一个片段ycf1基因和碎片psbA-ycf2垫片。这些结果也图形化地显示在蜘蛛图(图。4.A-F),表明用于产生条形码的最佳叶绿体区域是约30,000bp位置,由此占据ycf2基因。
对文献中出现的植物条形码的有效性进行了测试,证实了这些DNA片段在植物的鉴定中具有较高的效率Calypogeia物种(81.81-100%之间的歧视权;表S5.).的序列ndhH基因和trnT-trnL间隔层100%正确鉴定分析物种。
讨论
比较塑性基因组学
地苔质体基因组测序正在取得进展。目前,在GenBank数据库中可以找到5种复杂叶状体的完整质体序列,3种简单叶状体的完整质体序列和14种叶状体的完整质体序列。在这里,我们提出了22个新测序的叶绿体基因组10Calypogeia物种(叶苔类)。上述质体的结构是大多数植物的典型结构,被分为两个IRs区,由LSC区和SSC区分开(图1)。1)。叶绿体基因组的长度似乎不仅可变,而且在属级别,而且在物种中甚至在各个层面上都是可变的。塑料Calypogeia824-1, 366 bp是否比已知叶状叶苔叶绿体最长基因组短Gymnomitrion concinnatum[48]和1,880-2,422bp比最长已知的苯体喉叶绿体基因组短Dumortiera物种[49].Calypogeia物种在质体基因组长度上也有类似的变化Aneura pinguis神秘的物种(32].此外,我们观察到一个物种的质体长度不同(表S1).类似的事件也记录在Marchantia polymorpha无性系种群。ruderalis,两个独立的研究团队获得两种不同的叶绿体基因组长度:120,457 bp [50]和120,304 bp [51].
GC含量Calypogeia塑料是34.6%,几乎相同Gymnomitrion concinnatum(34.5%),最近测序完全叶绿体基因组的叶状植物曲谱物种[48].中的GC内容Calypogeia塑性基因组落在其他肝曲线物种所知的值范围内,从28.8%Marchantia paleacea[39至40.6%Aneura mirabilis.[52].
三种已知的叶状疱疹塑料的比较分析(Ptilidium pulcherrimum那Gymnomitrion concinnatum和测序CalypogeiaSP。),揭示了基因含量和顺序的相似性。所有这些都含有122个独特的基因:81蛋白编码,6个未知功能(ycf),31个TRNA和4个RRNA。但是,在Ptilidium pulcherrimum cysa(在LSC地区)和囊肿(在SSC区域)是假基因[53],而在叶绿体基因组中Calypogeia和Gymnomitrion concinnatum以及复杂的Thalloid Livorworts(Marchantia paleacea那Marchantia polymorpha那Dumortiera物种),这些基因是功能性的。这ycf68作为伪基因的主题被注释的主题Calypogeia质体基因组。在其他苔类植物中,也被登记为无功能基因。Aneura mirabilis.那Gymnomitrion concinnatum)或跳过塑动的描述(例如薄层终极玉米糖尿病那Aneura pinguis那Dumortiera物种那Marchantia sp。).另一方面,Forrest等人[53]对假设的功能感到困惑ycf68基因Ptilidium pulcherrimum.在许多维管植物中,上述基序被报道,但作为功能基因仅存在于几个谱系中:大针sp。[28],Lolium Multiflorum那羊茅属pratensis[54].Raubeson等人[55)建议ycf68可以是假基因,但许多植物的叶绿体基因组中的RNA编辑的发生可能导致完全工作的基因恢复。
系统发育关系
研究之间的系统发育关系Calypogeia在全叶绿体基因组分析的基础上获得的种,通常与以往的研究一致Calypogeia[56].整个塑料基因组分析证实了密切关系c . muelleriana和c . integristipula(无花果。3.)。先前的研究[41那46]揭示了这一点c . muelleriana是异源多倍体吗c . integristipula是单倍体物种,因此可以假设c . integristipula是其中之一c . muelleriana作为其叶绿体基因组的捐赠者的父母。c . sphagnicola和C. Paludosa.,最初被认为是c . sphagnicola即。c . sphagnicolaf。Sphagnicola.和c . sphagnicolaf。pal[57那58]属于两种不同的片状,支持它们代表遗传截然不同的物种的假设[44].Calypogeia sphagnicola属于同一支系c . suecica(均为单倍体种),异源倍体种C. Paludosa.形成自己独特的思考,这是一个姐妹C. Azurea那C. Azorica.和C. Fissa..此外,我们的研究显示出高度的变化c . suecica,表示本种内的隐种形成。c . suecica是一种专性木质素,它几乎局限于潮湿去皮的原木,表现出较低的形态变异[57].然而,在欧洲有两种细胞形式c . suecica n= 9,N = 18 are reported by Lorbeer [59]和paton [60]分别可以支持在内部存在未识别的物种的假设c . suecica.我们的结果表明c . suecica需要进一步的分子和形态学研究。
热点和DNA条形码
对整个苔类叶绿体或线粒体基因组变异性的分析在研究中很少出现。迄今为止,这种类型的分析只在隐种之间进行Aneura pinguis[32].因此,我们的结果是对属于一种属的一组物种,难以与复杂物种的结果进行比较答:pinguis.
研究结果:答:pinguis已经表明,在蛋白质编码区基因中ycf1和ycf2是最可变的基因中的一个[32].同样,在我们的研究中Calypogeia, 这ycf基因:ycf1那ycf2那ycf66在最具变量编码区域的前五个中,将它们易于成为潜在的DNA条形码。在过去的几年里,越来越多地报道了关于的有用性ycf1和ycf2鉴定植物物种的基因[29那61那62那63].特别是两个地区的ycf1基因:YCF1A.和YCF1B.是高度可变的,可以作为陆地植物的有效条码。Ycf1B片段被证明比任何matK那:和trnH-psbA单独使用比组合使用稍好matK和:在木质植物中[64].另一方面,应用YCF1A.碎片在歧视巴黎种苗比单纯使用种苗更有效YCF1B.基因片段(65].然而,一种歧视的成功YCF1B.Dong等人的研究片段(约72%)[64)和YCF1A.基因片分别(52.63%)和与之结合YCF1B.(89.47%) (65]比整个应用程序要小ycf1基因序列在我们的研究中Calypogeia物种(超过95%)。但是,想要使用ycf1和ycf2基因作为条形码,一个人应该记住这些序列在扩增中的局限性。上述基因很长(例如ycf1- 3,147 bp和ycf2-6,216 bpCalypogeia)在PCR反应中恢复这些基因的整个序列将是一个挑战。没有理由Dong等人。[64]仅作为木质工厂的条形码应用,只有最具变量,最大的分辨率力量ycf1碎片。我们的结果表明最有希望ycf2500 bp长的片段Calypogeia物种划界。多种作为最具变量片段的列表中的前13个位置Calypogeia质体体的长度为500bpycf2基因(表格1)并且可能是潜在的DNA条形码。在属中10种最可变的蛋白质编码区的歧视力Calypogeia在高水平的95.45%的情况下在大多数情况下(表1).只有决心的力量ndhB那ycf66和rpoC2基因分别具有不同的值:100,90.9和90.9%。虽然这一点ndhB基因而是通常在植物叶绿体基因组中进行,ycf66基因不一定。这个的存在ycf没有报告基因Aneura mirabilis.[52),Aneura pinguis[32].类似地,出现了囊肿基因,最易变异的蛋白质编码序列Calypogeia,在Liverworts中是可变的。例如,上述基因缺乏,例如Ptilidium pulcherrimum[53),Aneura物种(32那52].囊肿上述物种的基因是假基因,因此高变异是合理的[28].然而,文学作品并没有提到囊肿和ycf66基因作为DNA条形码。此外,没有人报告过ndhB基因作为一种有效的植物条形码,但它似乎是物种歧视中的核心序列之一Calypogeia.另一方面,Krawczyk等人[28]指出了该技术的潜力ndh基因家族的物种鉴定,表明ndhH基因作为最佳表演的基因座大针.虽然ndhH是不是列在最可变的编码区域的顶部Calypogeia叶绿体基因组(因此没有在我们的研究中测试),它的辨别能力是100%。但在物种鉴定方面也相当有效的是rpoC2基因 - 据报道属于相对较快的发展rpo基因[66].通过该序列的高多态性在我们的分析中确认了最后一个陈述(表1).最近,报告了rpoC2基因作为条形码更频繁[22那67那68那69].
寿命条形码(CBol)工厂工作组联盟推荐两个基因座::和matK作为植物的核心DNA条形码[23].在我们的研究中matK基因是编码区中变异性最高的第三位,在22个序列中正确识别了21个。不幸的是,matK在苔藓植物和蕨类植物中,基因的扩增是很麻烦的[23那70那71].因此,它是不确定是否使用matK物种鉴定的基因可扩展到苔藓植物[71].与以上相比,PCR的成功:据报道,基因很高[23那70那71].然而,在物种层面上分辨标本的能力却很平庸[23].尽管事实上是这样的:基因不是最可变的编码区之一Calypogeia,歧视的权力:基因(90.9%)几乎与之相同matK基因(表格1,表S.2).高分辨率的力量:在苔藓植物中也有报道[21那70那71[一些研究人员,注意到其作为苔藓植物的条形制标志物的潜力[25那32那72].然而,Stech和Quandt [73]一般来说,对于苔藓植物:基因在家系水平上表现出较低的变异,因此不能用于早期陆生植物的DNA条形码。在我们的测试中,应用了双位点条形码: + matK没有提高歧视性的权力,这是一样的为matK单独(表格1).
Liu等人[70]还提到了rpoc1.和rps4.地区作为苔藓的好潜在条形码。实际上,在这种情况下,这些序列的分辨率成功Calypogeia相当大(95.45%;表S5.).
非编码区域的分辨成功率为100%Calypogeia给了trnT-trnLspacer,之前在部落Stipeae中测试过[28那74那75].然而,作为一个单独的区域,这个间隔器的变量不够大,无法给出令人满意的结果。在文献中trnT-trnL垫片未被列为血症细胞中的潜在条形码(仅作为系统发育标记物[73与以下地区形成鲜明对比:trnh-psba[20.那70],atpF-atpH那PSBK-PSBI.[76),TRNL-TRNF.[20.那73].这trnH-psbA间隔物是CBOL植物工作小组推荐的植物DNA条形码之一[23].但是,在Calypogeia它不是这样的信息一枝黄花属(77]因为它的短长度(只有131 bp)。作为结果,trnH-psbA建议用于两位或三个轨迹条形码,以提供可接受的分辨率[77那78].
同样,序列太短,用于鉴定Calypogeia物种具有剩下的间隔物,提出用于苔藓(71-288 bp)。此外,是否有质疑是垫片的序列:atpF-atpH和PSBK-PSBI.可以在PCR反应中毫无问题地获得。Liu等报道了苔藓中这些区域的低扩增率[70].另一方面TRNL-TRNF.据报道,在一些苔类植物中,间隔区序列较长,且扩增成功[20.那71].在属Calypogeia那TRNL-TRNF.只有71个BP长区域。因此,根据Hebert等人,我们已经测试了至少400 BP长期的非编码区碎片。[19他报告说,标准条码的长度为400-800 bp。理论上,有可能应用较短的序列作为DNA条形码,即所谓的迷你条形码(100 - 250 bp),甚至微条形码(100 bp以内)[79那80].然而,这些类型的条形码是分类特有的,而不是通用的[81.].目前,通过对叶绿体全基因组的搜索来寻找物种鉴定中信息最丰富的片段已成为现实。Mini-barcodes为Calypogeia应该追求ycf2基因或基因之间psbA和ycf2正如演示的滑动窗口分析。
我们的研究表明Calypogeia质体中有许多具有条形码潜力的区域,最佳匹配/最佳接近匹配分析表明整个叶绿体基因组可以作为条形码。根据整个质体数据,我们发现大多数物种之间存在条形码间隙。根据整个质体序列和选定的叶绿体区域,只有一个个体被错误识别,即c . suecica.PTP分析表明了两个代表c . suecica作为两个单独的物种(表2。),这与有关的变异性结果是一致的Calypogeia叶绿体基因组。质体的两个代表c . suecica97.7%是相似的,考虑到该属所有研究质体序列的两两一致性,什么表明相当显著的差异Calypogeia是95.6%。这可能是由于内隐物种形成的原因c . suecica.但是,三个地区:ndhB和ndhH基因和trnT-trnLSpacer对求解物种遗传识别的谜语(100%的权力歧视)的谜语承受得很好。
与传统的条形码方法相比,在研究Liverwort Genus的情况下,Super-Barcoding已经略低于略低效率(占动力歧视的95.45%)(100%的权力歧视)。然而,一些具有100%效率的塑体区很长(ndhB- 1,503 bp;ndhH-1,182bp)和整体的扩增可能是有问题的。另一方面,使用塑性基因组作为标记物解决了低于PCR效率或基因损失的问题[82.].李等人[27]提出了一种新的植物DNA条形码方法(即所谓的“1 + 1模型”),该方法将超位点条形码和单位点条形码结合起来。这种方法包括一种“特定条形码”的发展,它是从目标植物群的叶绿体基因组中提取出来的,因此可以进行物种识别。我们检测了最易变化的DNA片段,发现了最特定的条形码Calypogeia蛋白质编码区中的物种(表1).7个基因正确地分配了22个序列中的21个,2个位点(ycf66那rpoC2) - 22个序列中的20个和一个基因座(ndhB)正确地确定了所有人。与滑动窗法产生的非编码区域和片段相比,蛋白质编码区域是最小的可变的。允许最终的方法获得最多可变的塑性DNA片,长度超过400bp,但遗憾的是其在类似于非编码区的物种歧视中的效率较低。我们的结果证明,良好的条形码可能是一个平均变异性的区域,如ndhH基因服用51.在属中最可变的蛋白质编码区排名的位置Calypogeia.
结论
总之,完全塑性序列作为超级条形码应用Calypogeia虽然不是100%有效,但它们的物种识别成功率(95.45%)仍然很明显。上述结果很可能是中隐种形成的结果c . suecica.需要进一步的研究来清除这个问题。另一方面,用于物种识别的超级条形码方法并不将门关到传统的单个或多轨道条形码。此外,它避免了由于需要扩增选定的DNA片段而产生的许多并发症。具有欧洲整体塑料的序列Calypogeia物种,我们发现ndhB和ndhH基因和trnT-trnL垫片识别物种100%。似乎,物种歧视的良好解决方案是基于塑料数据的给定分类组的所谓的“特定条形码”的发展。
方法
植物材料
本研究中使用的植物材料来自以下标本室:亚当·米凯维茨大学标本室、卡拉汉标本室和标本室Schäfer-Verwimp(表S1).22个标本代表10个类群Calypogeia:整合褐藻、褐藻、褐藻、沙棘褐藻、褐藻、蘑菇褐藻、玉米褐藻、软枣褐藻、褐藻.每个欧洲人的两个样本的总基因组DNACalypogeia采用ZR植物/种子DNA MiniPrep™试剂盒(Zymo Research, Irvine, CA, USA)提取。只有两个物种-C. Paludosa.和c . muelleriana由三个人代表。使用Qubit荧光计和Qubit™DSDNA BR测定套件(Invitrogen,Carbad,NM,USA)估计DNA量。
质体基因组测序、组装与注释
基因组文库由Truseq纳米DNA试剂盒(Illumina,San Diego,Ca,USA)构建,并使用Hiseqx(Illumina)测序,在Macrogen Inc.(首尔,韩国)生成150个BP配对读数,具有350bp插入尺寸在成对末端之间。由于少量可用的植物物料到亚速尔群岛C. Azorica.基因组文库必须使用能够降低输入DNA浓度的替代试剂盒来构建。测序的库C. Azorica.根据制造商协议使用QIAGEN FX库套件制备。测序后,通过去除适配器序列和用Trimmomatic v0.36的低质量读取来清洁读取[83.].过滤的读数是使用佐埃R8软件组装的De Novo [84.].为验证装配结果,将过滤后的reads定位于参比的叶绿体基因组Ptilidium pulcherrimum(序列相似性设置为90%)。映射后派生的下一个contigs被迭代地映射(序列相似性设置为100%),直到后续的迭代没有产生序列扩展。迭代映射方法的结果与从头组装方法的结果一致。以上分析采用genious R8软件进行[84.].
基于最近已知的叶绿体基因组来鉴定和注释基因:Aneura pinguis那Marchantia Paleacea,Pellia Endiviifolia,Ptilidium pulcherrimum.使用genius R8软件进行预测[84.]及BLAST工具[85.].带注释的序列Calypogeia将叶绿体基因组提交到GenBank,登录号见表S1.使用OGDraw软件创建圆形基因组图[86.].
变异分析
22个叶绿体基因组10Calypogeia使用MAFFT基因组对准器[87.].之后基于塑料的对准进行多态性分析,分别针对每种蛋白质编码序列,内含子,代际间隔物和通过滑动窗口分析产生的每个500bp长片段进行。上述区域内的每个变化被鉴定为单核苷酸多态性(SNP)或插入/删除(indel),并使用自定义素剧脚本计数。编码序列中的每个SNP被定义为同义词或非同意替代。使用电池软件可视化变化(SNP和Indels)[88.]结合Python脚本。使用Tassel 5.0计算每个编码和非编码区域的核苷酸多样性(π)[89.),对于每一个500 bp长的质体片段,在Spider [90.].因为核苷酸多样性仅基于取代,多态性位点的百分比(P%)(表S2,年代3.,年代4.).
系统发育分析
对27种植物的叶绿体基因组进行了系统发育分析Calypogeia属和其他5种已知的地苔。从每个质体序列中提取所有物种共有的68个基因的CDS,并翻译成氨基酸序列。MAFFT软件(87.]建立了三个比对:68个基因的27个CDS序列、68个基因的27个氨基酸序列和27个整个质体序列。接下来,使用PartitionFinder2 [91.[每个对准的最佳分区方案和相应的替换模型估计。之后,基于对准和获得的模型,使用MRBAYES 3.2.6进行贝叶斯分析[92.].MCMC算法运行了500万次(每500次采样),有4个增量加热链(从随机树开始)。跟踪器1.7.1 [93.]软件用来确定达到平稳性所需的代数,大约为30万代。因此,最初的600棵树被丢弃作为烧成树,剩下的树被用来发展贝叶斯共识树。Marchantia paleacea和Marchantia polymorpha在三棵获得的系统发育树中,每个都作为外群。
物种界定
采用泊松树过程(PTP)方法划分物种边界[94.].PTP模型使用替换的数量限定物种,而无需校准的困难和误差程序。该分析的基本假设是物种之间的替代数量明显高于物种内的替代数量。PTP模型将一组查询序列中的物种数量放在参考文学的特定分支中。因此,它只需要系统发育输入树,例如raxml的输出 - 分支长度应表示突变的数量。使用生根树进行分析,MCMC算法运行1,000,000代,100种变薄,0.2刻录。
叶绿体基因组的比较分析在DNASP V6.12中进行[95.](固定核苷酸差异的数量),使用自定义PHP脚本(特定物种的诱导塑性数量)和蜘蛛计划[90.]根据核苷酸取代木村2参数模型(K2P)计算的种间和种内距离[96.].
整个条码分析Calypogeia通过滑动窗产生的塑料和500个BP长片段在蜘蛛中进行了[90.],而TAXON-DNA软件包中的Species Identifier 1.8程序进行了最佳匹配/最佳接近匹配分析[97.].阈值设置为95% [97.].后一种分析是对整个叶绿体基因组和最可变的蛋白质编码区和非编码区进行分析Calypogeia根据Hebert等人给出的植物DNA条形码定义,长度至少为400 bp的质体[19].我们还测试了其他研究人员推荐作为条形码的序列的有效性(表S5.).
数据和材料的可用性
补充数据可在支持资料中获得。所有Calypogeia质体提交到GenBank,其登录号见表S1.
缩写
- cbol:
-
联盟为生命的条形码
- cd:
-
编码序列
- cpdna:
-
叶绿体DNA
- 红外:
-
反向重复区域
- LSC:
-
大的单拷贝区域
- SSC:
-
小的单拷贝区域
- P.%:
-
多态站点的百分比
参考
- 1.
Crandal-Stotler B,Stotler Re,Long DG。Marchantiophyta的系统发育和分类。edinb j bot。2009; 66(1):155-98。https://doi.org/10.1017/S0960428609005393.
- 2.
Clarke JT, Warnock RCM, Donoghue PCJ。建立植物进化的时间尺度。新植醇。2010;192:266 - 301。https://doi.org/10.1111/j.1469-8137.2011.03794.x.
- 3.
Rubinstein CV,Gerrienne P,De La Puente GS,Astini Ra,Steveans P. Argentina陆地植物的早期奥陶涅师证据(东吉隆东)。新植物。2010; 188(2):365-9。https://doi.org/10.1111/j.1469-8137.2010.03433.x.
- 4.
Wellman Ch,Osterloff Pl,Mohiuddin U.最早的土地植物的碎片。自然。2003; 425:282-5。https://doi.org/10.1038/nature01884.
- 5.
鲍曼杰。漫步在植物的长枝上进化。植物生物学杂志。2013;16(1):70-7。https://doi.org/10.1016/j.pbi.2012.10.001.
- 6.
Cox CJ,Li B,Foster PG,梅迪卡,奇皮马岛P.(2014)。早期陆地植物的影响突出的系统发育是由代名词中的组成偏差引起的。系统中的生物学。2014; 63:272-9。https://doi.org/10.1093/sysbio/syt109.
- 7.
隐孢子和隐植物揭示了早期陆地植物区系的隐性多样性。新植醇。2014;202:50 - 78。https://doi.org/10.1111/3ph.12645.
- 8。
邱Yl,李L,王B,陈Z,Knoop V,Groth-Malonek M等。从系统托儿核查证据推断出土地植物中最深处的分歧。PROC NATL ACAD SCI。2006; 103(42):15511-6。https://doi.org/10.1073/pnas.0603335103.
- 9。
关键词:叶苔,进化,惩罚可能性,遗传多样性分类单元。2007;56:31-44。https://doi.org/10.2307/25065733.
- 10.
舒斯特尔RM。北美的肝蕨和花蕨科,在子午线一百度以东,第2卷。纽约-伦敦:哥伦比亚大学出版社;1969.
- 11.
Söderström L, Hagborg A, Konrat M, Bartholomew-Began S, Bell D, Briscoe L.世界角苔和地苔清单。PhytoKeys。2016; 59:1 - 829。https://doi.org/10.3897/phytokeys.59.6261http://phytokeys.pensoft.net..
- 12.
Bischler H.该属CalypogeiaRaddi在Central和南美洲。I- III。Canlollea。1963年; 18:19-128。
- 13.
Söderström胡志明,杨永明,Váňa J.欧洲和南美的肝苔类和花苔类的分布。Lindbergia。2002;27(1):3-47。https://doi.org/10.2307/20150088.
- 14.
Söderströml,urmie,váðaj。欧洲和Macaronesia的肝癌和Anthocerotae的分布 - 更新1-427。Cryptogam Bryol。2007; 28(4):299-350。
- 15.
井上二新种Calypogeia来自日本的Raddi。公牛Natl Mus Nat和SCI Ser B Bot。1975; 1:135-40。
- 16.
konstantinova na,bakalin va。俄罗斯的Liverworts(Marchantiophyta)清单。arctoa。2009; 18(1):1-64。https://doi.org/10.15298/arctoa.18.01.
- 17.
辛格美联社,纳特vCalypogeia来自印度的Raddi。台庄。2007; 52(4):320-3。https://doi.org/10.6165/tai.2007.52(4).320.
- 18。
舒斯特尔RM。Jungermanniidae,III的系统发育和分类学研究。calypogeiaceae。Fragmenta floritiva et geobotanica。1995年; 40:825-88。
- 19。
Hebert PD, Cywinska A, Ball SL, de ward JR.,通过DNA条形码进行生物鉴定。生物科学。2003;270:313-21。https://doi.org/10.1098/rspb.2002.2218.
- 20.
BąCZKiewiczA,SzczecińskaM,Savicki J,Stebel A,Buczkowska K. DNA条形码,生态和地理的神秘种类Aneura pinguis以及他们与Aneura最大值和Aneura mirabilis.(Metzgeriales Marchantiophyta)。《公共科学图书馆•综合》。2017;12 (12):e0188837。https://doi.org/10.1371/journal.pone.0188837.
- 21.
刘国平,刘国平,刘国平,等。植物条形码位点的选择:在3个不同的陆生植物类群中,用物种水平取样评价7个候选位点。Mol Ecol Resour. 2009; 9:39 - 57。https://doi.org/10.1111/j.1755-0998.2008.02439.x.
- 22.
Krawczyk K, Szczecińska M, Sawicki J. 11个单位点和7个多位点DNA条形码的评价薄层l .(唇形科)。分子生态资源。2014;14(2):272-85。https://doi.org/10.1111/1755-0998.12175.
- 23.
Cbol植物工作组,Hollingsworth PM,Forrest LL,Spouge JL,Hajibabaei M,Ratnasingham S等人。土地植物的DNA条形码。Proc Natl Acad Sci U S A. 2009; 106(31):12794-979。https://doi.org/10.1073/pnas.0905845106.
- 24.
贝尔D,Long DG,Forrest Ad,Hollingsworth Ml,Blom Hh,Hollingsworth PM。欧洲的DNA条形码赫伯腾(Marchantiopsida,Herbertaceae)以及一个新物种的发现和描述。Mol Ecol Resour。2012; 12(1):36-47。https://doi.org/10.1111/j.1755-0998.2011.03053.x..
- 25.
Hassel K,Segreto R,Ekrem T.植物条形码标记的受限制变化限制了密切相关的白细胞物种的鉴定。Mol Ecol Resour。2013; 13(6):1047-57。https://doi.org/10.1111/1755-0998.12074.
- 26.
用于下一代生物多样性分析的基因组略读。植物科学与技术,2015;https://doi.org/10.1016/j.tplants.2015.06.012.
- 27.
李晓,杨永强,王永强,陈顺。植物DNA条形码:从基因到基因组。生物Rev Camb Philos Soc. 2015;90(1): 157-66。https://doi.org/10.1111/brv.12104.
- 28.
Krawczyk K, Nobis M, Myszczyński K, Klichowska E, Sawicki J. Plastid超级条形码在羽毛草(禾草科)物种鉴别中的应用大针).Sci众议员2018;8(1):1924。https://doi.org/10.1038/s41598-018-20399 -2198-018-20399-W..
- 29.
szczecińskam,Sawicki J.三种基因组资源白头翁物种揭示了毛茛科的进化热点、物种特异性位点和可变质体结构。国际分子科学杂志。2015:22258-79。https://doi.org/10.3390/ijms160922258..
- 30.
杨建波,杨树新,李海涛,杨军,李德志。比较叶绿体基因组茶花物种。《公共科学图书馆•综合》。2013;8 (8):e73053。https://doi.org/10.1371/journal.pone.0073053.
- 31.
杨建勇,张丹,Engels JMM,等。可可的超条码(Theobroma.SPP .;Malvaceae)使用全叶绿体基因组和核核糖体DNA。我是J机器人。2012; 99:320-9。https://doi.org/10.3732/AJB.1100570.
- 32.
Myszczyński K, Bączkiewicz A, buzkowska K, Ślipiko M, Szczecińska M, Sawicki J.Aneura pinguis揭示了早期陆生植物的高级隐种形成。Sci众议员2017;7:9804。https://doi.org/10.1038/s41598-017-10434-7.
- 33.
Sawicki J, Szczecińska M, Bednarek-Ochyra H, Ochyra R.线粒体系统基因组学支持分离传统的属racomitrium.(Bryophyta:Grimmiaceae)。Nova Hedwigia。2015; 100:293-317。https://doi.org/10.1127/nova_hedwigia/2015/0248.
- 34.
Sawicki J,Plášekv,ochyra r,szczecińskam,ślipikom,myszczyńskik等。含有丝育的分析支持最近的属Orthotrichum(Orthotrichaceae,Bryophyta)。SCI批准。2017; 7(1):4408。https://doi.org/10.1038/s41598-017-04833-z.
- 35.
王文昌,陈绍勇,张学忠。猕猴桃科的叶绿体基因组进化:CLPP.损失,异源性分歧和系统育方法。Plos一个。2016; 11(9):E0162324。https://doi.org/10.1371/journal.pone.0162324.
- 36.
张永军,马鹏飞,李德志。6个竹叶绿体基因组的高通量测序:温带木本竹子(竹科:竹亚科)的系统发育意义《公共科学图书馆•综合》。2011;6 (5):e20596。https://doi.org/10.1371/journal.pone.0020596..
- 37.
周t,陈c,魏y,chang y,白g,li z等。两种相关的比较转录组和叶绿体基因组分析Dipteronia物种。植物科学与技术,2016;https://doi.org/10.3389/fpls.2016.01512.
- 38.
Coissac E, Hollingsworth PM, Lavergne S, Taberlet P.从条形码到基因组:延伸DNA条形码的概念。摩尔生态。2016;25:1423-8。https://doi.org/10.1111/mec.13549..
- 39.
Ohyama K,Fukuzawa H,Kohchi T,Shirai H,Sano T,Sano S等人。从Liverwort完整序列中推断出叶绿体基因组织Marchantia polymorpha叶绿体DNA。自然。1986; 322:572-4。
- 40.
- 41.
植物的遗传分化Calypogeia fissa波兰的双耳肝虫(双耳肝虫目)。植物系统演化。2004;247:187-201。https://doi.org/10.1007/s00606-003-0156-9.
- 42.
buckzkowska K, Bączkiewicz A.(2011)。标题属的新分类单元Calypogeia(Jungermanniales,Hepaticae)在波兰。Acta Soc Bot Pol。2011; 80:327-33。https://doi.org/10.5586/asbp.2011.039.
- 43.
Buczkowska K,Dabert M.为划界的特定瘢痕标记的开发Calypogeia物种。J Bryol。2011;33:291-9。https://doi.org/10.2478/v10119-011-0014-x.
- 44.
Buczkowska K,Sawicki J,Szczecińskam,克拉马H,BąCZKiewiczA. AlloplyPloid形态Calypogeia sphagnicola(Jungermanniopsiada,萼藓科)植物生态学报,2012;https://doi.org/10.1007/S00606-011-0565-5.
- 45.
Buczkowska k,Odrzykoski IJ,Chudzińskae.一些欧洲物种的界限Calypogeia基于细胞学特征和多酶表型的Raddi (Jungermnniales, hepatcae)。Nova Hedwigia。2004;78:147 - 63。https://doi.org/10.2478/v10119-010-0004-4.
- 46.
布兹考夫斯卡,霍尼可,Czołpińska M.遗传界限类群的两个倍性水平Calypogeia fissa复杂(Jungermanniopsida Calypogeiaceae)。Biodivers Conserv。2015;39:1-6。https://doi.org/10.1515/biorc-2015-0022.
- 47.
ślipikom,myszczyńskik,buczkowska-chmielewska k,bączkiwicza,szczecińskaism,Sawicki J.四个比较分析Calypogeia物种揭示了进化稳定的Liverwort Mitogomes的意外变化。基因。2017; 8(12):395。https://doi.org/10.3390/genes8120395.
- 48.
Myszczyński K, Górski P, Ślipiko MGymnomitrion concinnatum(Jungermanniales)揭示了进化稳定的Liverworts Mitogomes的结构和基因顺序中的第一个例外。BMC植物BIOL。2018; 18(1):321。https://do.org/10.1186/s12870-018-1558-0..
- 49.
蒲根W,Kim Y,Park J.完整的叶绿体基因组Dumortiera物种(Sw)。需要雇(Marchntiophyta Dumortieraceae)。4(1): 1586-7。https://doi.org/10.1080/23802359.2019.1596767.
- 50。
Kijak H, Rurek M, Nowak W, Zielezinski A, Karlowski WM, Dabert M, Odrzykoski IJ。重新排序Marchantia polymorpha叶绿体基因组。论文发表于EMBO研讨会,早期植物进化的新模型系统,维也纳,奥地利,(2016,6月22-24日)。
- 51。
Bowman JL,Kohchi T,Yamato Kt,Jenkins J,Shu S,Ishizaki K,等。洞察陆地植物的进化Marchantia polymorpha基因组。细胞。2017;171(2):287 - 304。https://doi.org/10.1016/j.cell.2017.09.030.
- 52.
Wickett NJ, Zhang Y, Hansen SK, Roper JM, Kuehl JV, Plock SA等。功能基因的损失发生在寄生地苔质体基因组的最小大小的减少Aneura mirabilis..中国生物医学工程学报。2008;25(2):393-401。https://doi.org/10.1093/molbev/msm267.
- 53.
Forrest LL, Wickett NJ, Cox CJ, Goffinet B.深度测序Ptilidium.Ptilidiaceae在Liverwort Plastid基因组结构中表明进化瘀滞。植物ECOL EVOL。2011; 144(1):29-43。https://doi.org/10.5091/plecevo.2011.535.
- 54.
手ml,spangenberg gc,forster jw,cogan no。塑性序列固定和散发性轻链分析洛利姆-羊茅属草地物种复杂。G3(贝塞斯达)。3(4): 607 - 2013; 16。https://doi.org/10.1534/g3.112.005264.
- 55.
罗布森(Raubeson LA, Peery R, Chumley TW, Dziubek C, Fourcade HM, Boore JL,等。比较叶绿体基因组学:包括被子植物新序列的分析黄睡莲advena和毛茛属macranthus..BMC基因组学。2007;8:174。https://doi.org/10.1186/1471-2164-8-174.
- 56.
巴卡林,Bączkiewicz A,阿奎罗,戈纳拉,Ślipiko M,等。做Calypogeia azurea(Calypogeiaceae,Marchantiophyta)发生在欧洲以外?分子和形态证据。Plos一个。2018; 13(10):E0204561。https://doi.org/10.1371/journal.pone.0204561.
- 57.
Damsholt K.北欧Liverworts和Hornworts的插图植物群。北欧浏览学会:隆德;2002年。
- 58.
波兰苔属植物注释清单。生物多样性的波兰。Kraków:波兰科学院W. Szafer植物研究所;2006.
- 59.
麋鹿Lorbeer G. Zytologie der Lebermoose的研究对象Berücksichtigung allgemeiner Chromosomenfragen。I Teil Jahrb Wiss Bot 1934; 80:567-818。
- 60.
Paton Ja。英国群岛的Liverwort植物群。德文:哈利书籍;1999年。
- 61.
关键词:叶绿体标记,植物系统发育,DNA条形码《公共科学图书馆•综合》。2012;7 (4):e35071。https://doi.org/10.1371/journal.pone.0035071.
- 62.
江盖夫,艾恩辛德,Strijk JS。狼人中三角异性和叶绿体多样性的比较。SCI REP。2016; 6:31473。https://doi.org/10.1038/srep31473.
- 63.
Yi DK,Choi K,Joo M,Yang Jc,Mustafina Fu,Han J-S等人。完全叶绿体基因组序列Andephrolepis.(Pinaceae:Apietoideae)。J Asia Pac Biodivers。2016; 9(2):245-9。https://doi.org/10.1016/j.japb.2016.03.014.
- 64.
董伟,徐超,李超,孙杰,左勇,石松,等。ycf1,最有前途的陆生植物质体DNA条形码。Sci众议员2015;5:8348。https://doi.org/10.1038/srep08348.
- 65.
金歌Y, Shaojun W,陶渊明D X,明FL, Shuifang Z, et al。叶绿体基因组资源巴黎对于物种歧视。Sci众议员2017;7:3427。https://doi.org/10.1038/s41598-017-02083-7.
- 66.
Igloi GL, Meinke A, Döry I, Kössel H.玉米叶绿体的核苷酸序列rpoB / C1/ C2操纵子:比较不同生物衍生的蛋白质一级结构与功能域。中国生物医学工程学报,2001;https://doi.org/10.1007/bf00259403.
- 67.
Ford CS, Ayers KL, Haider N, Toomey N, van Alpen SJ, Kelly L等。陆生植物DNA条形码候选区域的选择。acta photonica sinica, 2009;https://doi.org/10.1111/j.1095-8339.2008.00938.x.
- 68.
匡王,吴H,王YL,高LM,张SZ,LU L.完全叶绿体基因组序列木兰kwangsiensis(木兰科):DNA条形码和群体遗传学的意义。基因组。2011;54(8):663 - 73。https://doi.org/10.1139/G11-026.
- 69.
Logachewa MD, Peva AA, Samigulin TH, Vallejo-Roman CM, Antonov AS。用叶绿体基因组序列研究开花植物的系统发育:寻找“幸运基因”。生物化学(Mosc)。72(12): 1324 - 2007; 30。
- 70.
刘燕,严海峰,曹涛,葛晓军。苔藓类10种植物条形码的鉴定。系统演化。2010;48(1):36-46。https://doi.org/10.1111/j.1759-6831.2009.00063.x.
- 71.
yoodphaka S, Boonpragob K, Lumbsch HT, Kraichak E.评价6个区域作为泰国叶生地苔DNA条形码的潜力。应用植物科学。2018;6(8):e1174。https://doi.org/10.1002/aps3.1174.
- 72.
Forrest LL,Davis EC,Long DG,Crandall-Stotler BJ,Clark A,Hollingsworth ML。解开Liverworts(Marchantioophyta)的进化史:多个分类群,基因组和分析。苔原。2006; 109(3):303-34 10.1639 / 0007-2745(2006)109 [303:Utehot] 2.0.co; 2。
- 73。
施泰克M,围棋D. 20,000种和五个关键标记:分子苔藓植物发育的状态。Phytotaxa。2010; 9:196-228。https://doi.org/10.11646/phytotaxa.9.1.11.
- 74。
Cialdella Am,Salariato DL,Aageen L,Giussani LM,Zuloaga Fo,Morrone O.新世界Phyleogy(Poaceae):对单层的评估Aciachne和Amelichloa..26支序分类学。2010;(6):563 - 78。https://doi.org/10.1111/j.1096-0031.2010.00310.x..
- 75。
Cialdella AM, Sede SM, Romaschenko K, Peterson总理,Soreng RJ, Zuloaga FO等。的发展史Nassella(STIPEAE,Pooideae,Poaceae)基于分析叶绿体和核核糖体DNA和形态学。AUST SYST BOT。2014; 39(3):814-28。https://doi.org/10.1600/036364414x681419..
- 76.
Pennisi E.分类。通缉:植物的条形码。科学。2007; 318(5848):190-1。https://doi.org/10.1126/science.318.5848.190.
- 77.
Kress WJ, Wurdack KJ, Zimmer EA, weight LA, Janzen DH。利用DNA条形码识别开花植物。国家自然科学基金资助项目:国家自然科学基金资助项目。https://doi.org/10.1073/pnas.0503123102.
- 78.
蔡斯MW, Cowan RS, Hollingsworth PM, van den Berg C, Madrinan S, Petersen G等。对所有陆地植物进行条形码的标准化协议的建议。56分类单元。2007;(2):295 - 9。https://doi.org/10.1002/tax.562004.
- 79.
Hajibabaei M, Smith MA, Janzen DH, Rodriguez JJ, Whitfield JB, Hebert PDN。一个最低限度的条形码可以识别DNA被降解的标本。Mol Ecol Notes. 2006;6(4): 959-64。https://doi.org/10.1111/j.1471-8286.2006.01470.x..
- 80。
一种用于生物多样性分析的通用DNA微型条形码。BMC基因组学。2008;9:214。https://doi.org/10.1186/1471-2164-9-214.
- 81。
[15]董伟,刘慧,赵晓,左英,陈卓,周松。植物分类单元特异性DNA微条形码设计的基因组策略:以人参为例。BMC麝猫。2014;15:138。https://doi.org/10.1186/s12863-014-0138-z.
- 82。
黄春英,Grünheit N, Ahmadinejad N, Timmis JN, Martin W.最近叶绿体和线粒体基因组的突变衰变和年龄转移到被子植物核染色体。植物杂志。2005;138:1723-33。https://doi.org/10.1104/pp.105.060327.
- 83.
Bolger AM, Lohse M, Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014;30(15):2114 - 20。https://doi.org/10.1093/bioinformatics/btu170.
- 84.
张国强,张国强,张国强,等。天才基础:一个集成的、可扩展的桌面软件平台,用于组织和分析序列数据。生物信息学。2012;28(12):1647 - 9。https://doi.org/10.1093/bioinformatics/bts199.
- 85.
NCBI BLAST:一个更好的网络界面。核酸杂志2008;36:W5-9。https://doi.org/10.1093/nar/gkn201.
- 86.
Lohse M, Drechsel O, Kahlau S, Bock R. organellargenome edrawa -一套用于生成质体和线粒体基因组物理图和可视化表达数据集的工具。核酸杂志2013;41:W575-81。https://doi.org/10.1093/nar/gkt289.
- 87.
Katoh K,Standley DM。(2013)。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30(4):772-80。https://doi.org/10.1093/molbev/mst010.
- 88.
Krzywinski M,Schein J,Birol I,Connors J,Gascoyne R,Horsman D等人。电讯:对比较基因组学的信息审美。Genome Res。2009; 19(9):1639-45。https://doi.org/10.1101/gr.092759.109.
- 89.
张志明,张志明,张志明。TASSEL:用于在不同样本中进行复杂性状关联映射的软件。生物信息学。2007;23(19):2633 - 5。https://doi.org/10.1093/bioinformatics/btm308.
- 90。
Brown SD, Collins RA, Boyer S, Lefort MC, Malumbres-Olarte J, Vink CJ等。蜘蛛:一个用于分析物种身份和进化的R包,特别涉及到DNA条形码。Mol Ecol Resour. 2012;12(3): 562-5。https://doi.org/10.1111/j.1755-0998.2011.03108.x.
- 91。
LANFEAR R,Frandsen PB,Wright Am,Senfeld T,Capott B. PartitionFinder 2:用于选择分配的分子和形态学系统发育分析的分区模型的新方法。mol Biol Evol。2016; 34(3):772-3。https://doi.org/10.1093/molbev/msw260.
- 92。
Huelsenbeck jp,罗魁福特F.Mrbayes:贝叶斯植物发育丛林的推论。生物信息学。2001; 17(8):754-5。https://doi.org/10.1093/bioinformatics/17.8.754.
- 93.
李国强,李国强,李国强,李国强。使用示踪剂1.7进行贝叶斯系统发育的后验总结。系统医学杂志。2018;67(5):901 - 4。https://doi.org/10.1093/sysbio/syy032.
- 94.
张j,kapli p,pavlidis p,stamatakis aa。一般物种与系统发育展示作用的划分方法。生物信息学。2013; 29(22):2869-76。https://doi.org/10.1093/bioinformatics/btt499.
- 95.
Rozas J, Ferrer-Mata A, Sánchez-DelBarrio JC, Guirao-Rico S, Librado P, Ramos-Onsins SE等。dnasp6:大数据集的DNA序列多态性分析。生物学报。2017;34(12):3299-302。https://doi.org/10.1093/molbev/msx248.
- 96.
Kimura M.通过核苷酸序列的比较研究估算基取代的进化率的简单方法。J Mol Evol。1980; 16(2):111-20。
- 97。
关键词:DNA条形码,分类学,生物信息学双翅目:一个高种内变异性和低鉴定成功率的故事。55系统杂志。2006;(5):715 - 28。https://doi.org/10.1080/10635150600969864..
确认
我们感谢Tatra国家公园和Bieszczady国家公园能够收集样本。也感谢卡拉汉检察官和Alfons Schäfer-Verwimp分享这些样本。
资金
我们的研究在波兰克拉科夫国家科学中心经济支持。由于Grant No.2015 / 19 / B / NZ8 / 03970我们能够支付测序Calypogeia塑料,被委托给商业公司。数据和写作稿件的研究和收集,分析和解释的设计仅由手稿的作者而无需财政支持。
作者信息
从属关系
贡献
NGS测序,数据分析,文本写作,表格和数字制备的样品的分离和制备。km-数据分析,文字写作,桌子准备和数字;KB和AB-植物材料收集,文字写作;MS-数据分析;JS-研究设计,数据分析,纸张审查草案。所有作者阅读并认可的终稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
同意出版
不适用。
利益争夺
作者们宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:表S1。
本研究中使用的物种,采样数据和测序结果。
附加文件2:表S2。
叶绿体基因的SNP和indel变异Calypogeia物种。
附加文件3:表S3。
叶绿体非编码区SNP和indel变异Calypogeia物种。
附加文件4:表S4。
叶绿体500bp片段的SNP和indel变异Calypogeia物种。
附加文件5:表S5。
植物DNA条形码的鉴别能力。
附加文件6:图S1。
基于氨基酸和完整质体基因组的系统图Calypogeia物种。在节点处给出小于1的后验概率值。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览http://creativecommons.org/licenses/by/4.0/.“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Ślipiko, M., Myszczyński, K.,巴克科夫斯卡,K.。et al。欧洲叶状地苔属植物的分子分界Calypogeia基于塑性超级条形码。BMC植物杂志20.243(2020)。https://doi.org/10.1186/s12870-020-02435-Y.
收到了:
公认:
发表:
关键字
- Super-barcoding
- DNA条形码
- Calypogeia
- ndhB
- ndhH
- trnT-trnL