摘要
背景
长链非编码rna (Long noncoding RNAs, lncrna)在调控众多复杂机制的生物过程中发挥着重要作用。然而,对于lncrna的数量、特征、序列以及可能对植物碱胁迫反应的影响,我们知之甚少。
结果
根据对照采集叶片样品甜菜属L.以及在短期和长期碱性处理下的那些对高通量RNA测序进行高通量RNA测序,通过该测量,检测到具有可靠表达的8535克朗。在这些LNCRNA,102和49中,分别在短期和长期碱性应激后改变102和49。此外,7个LNCRNA被认为是前体以前鉴定的miRNA。据估计响应碱性应激的四种LNCRNA为8 miRNA的靶标。此外,计算分析将4318个电位靶基因预测为响应于碱性应力的LNCRNA。功能注释分析表明,上述可能的靶基因涉及各种生物处理,例如激酶活性,核糖体的结构成分,核糖核蛋白复合物和蛋白质代谢过程。关联分析提供了令人信服的令人信服的候选靶基因与LNCRNA的相互作用。
结论
lncrna可能通过与蛋白编码基因的相互作用,在调控植物的碱性胁迫反应和适应过程中发挥重要作用。本研究的发现有助于全面检测lncRNAs甜菜属并进一步阐明lncrna响应碱性胁迫的可能作用和相互调节作用,从而为这些类型的功能分析奠定一定的基础甜菜属L. lncrna。
背景
盐胁迫通过渗透胁迫和离子失衡极大地影响植物的生长发育[1]。由于盐渍化土壤的大面积分布及其对作物生产的负面影响,植物耐盐机制在上个世纪得到了深入的探讨[2]。以往的相关研究主要集中在中性盐胁迫下的耐盐机制上。关于植物对碱盐胁迫的反应的研究较少。碱盐胁迫(NaHCO3.和钠2有限公司3.),简称碱性胁迫,比中性盐更严重地抑制植物生长和生存[3.]。中性盐胁迫和碱性应激之间存在许多常见因素,例如离子毒性以及渗透压[4.]。然而,碱性胁迫表现出其独特性,因为高pH值会引发丙二醛(MDA)和活性氧(ROS)的产生,从而破坏植物的细胞内成分和细胞膜。因此,碱胁迫是一种不同的胁迫形式,植物耐碱的机制相当复杂。植物耐盐碱胁迫的机制涉及多种基因表达谱和基因产物相互作用,而不是单个基因表达[5.那6.]。
基于改进的高通量测序技术,建议90%以上的基因组产生大量的非编码rna (ncrna) [7.那8.]。当基于长度划分时,NCRNA可以分为长的非分量RNA(LNCRNA),小干扰RNA(SIRNA),以及小RNA,如MicroRNA(miRNA)[9.那10]。其中,LNCRNA长度超过200个核苷酸,具有较低的蛋白质能力,它们占据绝大多数NCRNA [11那12]。LNCRNA的表达谱经常细胞或组织特异性,其转录物位于亚细胞室中[13]。此外,根据关于基因组蛋白质编码基因的位置,LNCRNA分为反感,感测,双向,基因克和内文类别[14]。
有大量证据有力证明lncrna在多种植物生物过程中发挥重要的调节作用[12那15]。此外,一些lncrna已被鉴定为在(cis-acting)或遥远的(trans-acting),通过不同的机制,如通过核小体重新定位的启动子活性修饰、DNA甲基化、组蛋白修饰、辅助蛋白激活/聚集/运输、抑制和表观遗传沉默[15那16]。越来越多的研究揭示了lncRNA在植物中的作用。例如,At4和AtIPS1被认为是miR399 -通过隔离和结合的特异性模拟miR399以及减少PHO2乳沟由miR399,在磷酸盐吸收中起着至关重要的作用[17]。此外,Swiezewski等人[18)改变开花轨迹C(FLC.)的表达,并发现lncrna参与调控开花。LDMAR在水稻中调节对光周期敏感的雄性不育[19]。此外,PINOID是调节极性生长素和lncRNA运输的重要因素Apolo.表达可诱导染色质形成的变化上调PINOID表达式[20.]。共有13087个和11641个lncrnam . truncatula分别对盐和渗透胁迫表现出响应;同时,5634个lncrna被认为对盐和渗透胁迫都有响应[21]。在拟南芥中,lncRNAHID1被发现介导红光诱导的光形态建成[22]。PDIL1抑制MtPHO2下调,后者编码mir399调节的泛素缀合的e2酶,而PDIL2和PDIL3表现出对磷酸盐运输的直接转录调控。大量的研究取得了巨大的进展,但与哺乳动物lncRNA相比,植物lncRNA的功能以及相关的调控网络在很大程度上仍不清楚。lncrna在一小部分植物中被系统识别,如拟南芥[23那24)、大米(25)、玉米(26],杨树[27),Medicago truncatula[28]。然而,Chenopodiaceae sugar beet (甜菜属l.)模型在很大程度上仍不为人知。
甜菜(甜菜属l.)是一种重要的经济作物,为全球食糖供应做出了巨大贡献。它不仅用于食品工业,而且用作可再生能源[29]。甜菜可以适应非生物和生物应力,包括盐度,干旱,热和冷,在温带气候下[30.]。甜菜是一种耐盐作物,已被用来研究作物对氯化钠(NaCl)的适应性,作为一个良好的模型。此前对生理盐水应激反应的大量研究已经在生理和分子水平进行,如抗氧化酶、蛋白质组和转录组[31那32那33]。甜菜基因组测序已经完成[34];因此,甜菜已成为研究植物抗逆性和抗逆性的极好模型。然而,lncrna参与甜菜对碱性胁迫的反应从未被报道过。
本研究在全基因组水平上对甜菜叶片中检测到的响应碱性胁迫的lncrna进行了全面检测和表征;此外,预测了与鉴定的lncrna相互作用的候选基因和mirna。总的来说,研究结果更清楚地说明了甜菜属l.响应于碱性应力并奠定基础以研究以研究AS鉴定的LNCRNA功能的LNCRNA。
结果
碱胁迫影响生理和生长特性
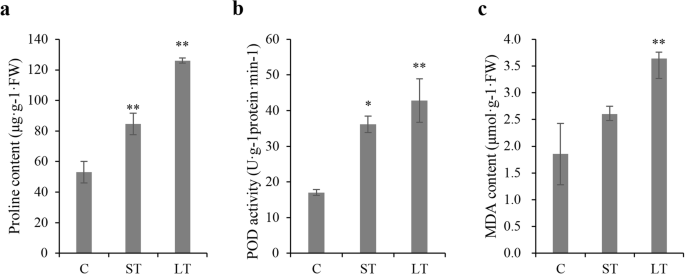
为了研究不同时间长度的碱处理对甜菜生理特性的影响,本研究分别测定了碱处理0、3和7天的甜菜叶片中脯氨酸含量、丙二醛含量和POD活性。如图所示。1A-c、不同时间长度的碱性处理对这三个生理特性均有显著影响。以上生理上的异质性说明甜菜碱处理后基因表达(包括lncrna)存在明显的改变。
为了研究长期碱性处理对甜菜生长的影响,本研究测定了有或无碱性处理7天的水培植物的生长和光合特性。碱性处理后观察形态学变化(附图S1a).碱处理显著抑制植物生长。碱胁迫后,光合特性发生了明显的变化1罪犯)。例如,TrG年代和Pn与对照相比,碱处理植株的叶绿素含量显著降低(图S1罪犯)。而光系统II (Y (II))量子产量在对照和碱胁迫下无显著差异。光合作用和生长特性的显著变化表明碱度对植物生长具有抑制作用。
甜菜lncRNA的特征
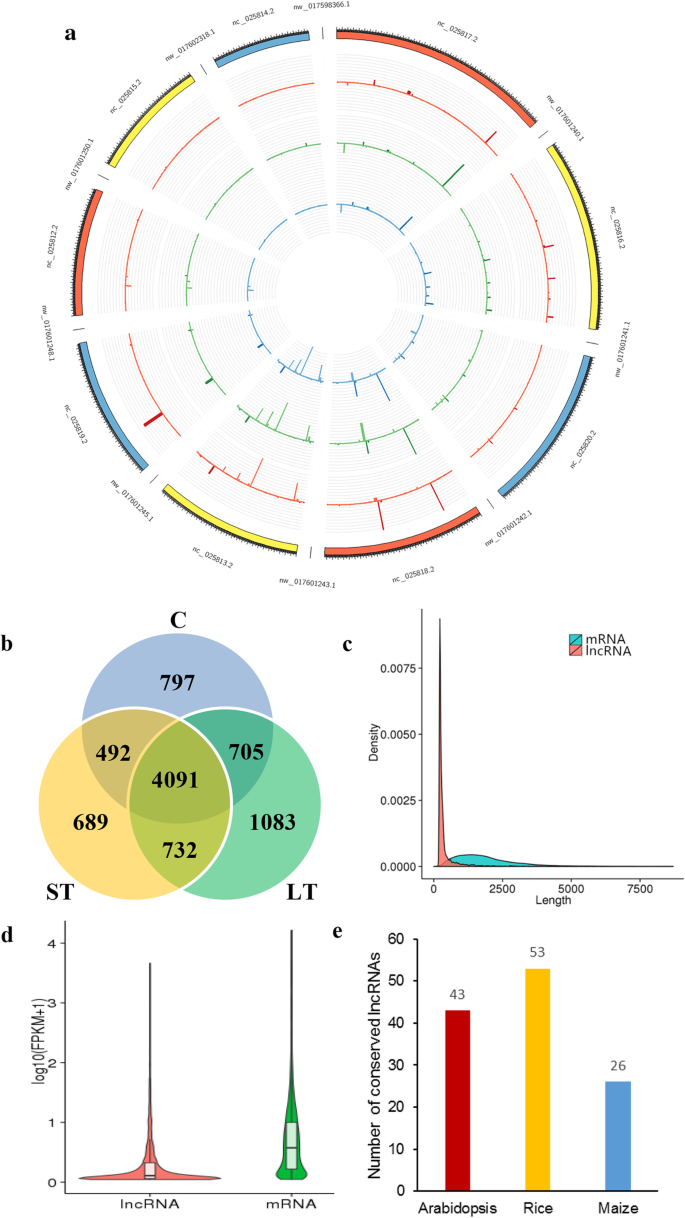
根据上述生理特征的变化,分别采集对照、3天碱性胁迫和7天碱性胁迫植物的叶片样本,进行高通量RNA测序。随后,在全基因组中系统鉴定了lncrna,基于对照、短期和长期碱胁迫植物库分别鉴定出6085、6004和6611个lncrna (FPKM> 0.5)(图2)。2b).共鉴定出8535个可靠表达的lncrna (FPKM> .5对1个或1个以上文库),其中反义2051个,义6034个。
此外,对8535个lncrna进行了基本基因组特征分析。lncRNA的分布甜菜属l.染色体检测显示,每Mb的lncrna平均密度为15.06 lncrna(图1)。2a). lncRNA的转录本长度在201-12,882核苷酸之间,平均为424个核苷酸,与之相比较短甜菜属l.编码蛋白质编码基因(平均,1998个核苷酸)(图。2c). lncRNA表达模式的平均计数(FPKM = 15.83)相对于编码转录本(FPKM = 19.11)较低(图。2d).根据保存分析的结果,只有少数甜菜属L. lncrna在玉米、水稻和拟南芥中表现出保守性。2e).此外,根据BLAST分析甜菜属L.相对于ncRNAs NONCODE数据库,98.6%的检测到的lncRNAs特异性在甜菜属l
此外,研究人员还对lncrna的序列进行了研究,以确定其作为先前鉴定的mirna的靶点或前体的潜力。miRNA前体与8535个lncrna进行比对,3个lncrna被认为是两个已经鉴定的miRNA的前体(见表)1).例如lncRNALNC_003048被估计是gma-miR4995前体。
碱响应型lncrna的鉴定
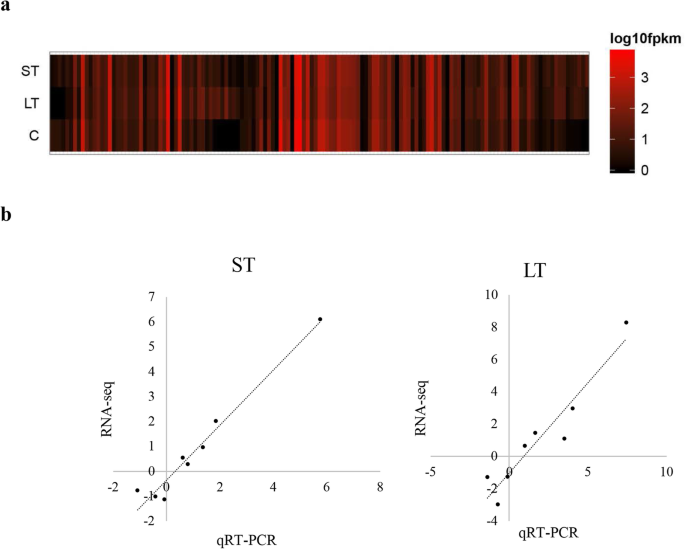
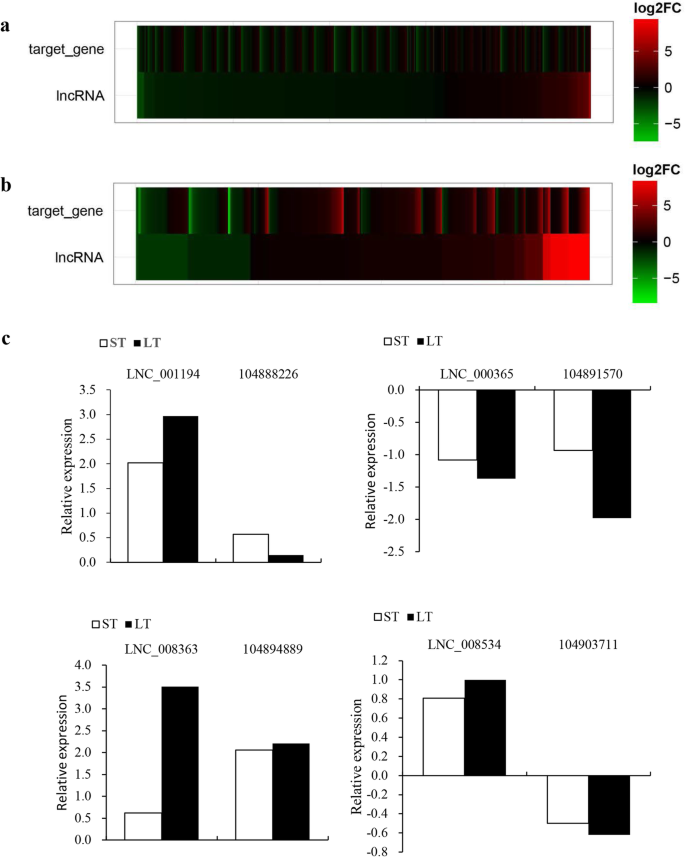
我们鉴定了93种lncrna(附加文件表S1)具有碱性响应表达式模式(P < 0.05), with 24 upregulated and 40 downregulated under short-term alkaline treatment and 26 upregulated and 11 downregulated under long-term alkaline treatment (Fig.3.a).在这些碱性应答lncrna中,有5种lncrna在短期和长期碱性胁迫下均表达上调,3种lncrna在短期和长期碱性胁迫下均表达下调。其中差异表达lncRNAs 10和2 > 4倍的增加以及减少> 4倍,以应对短期碱性压力,分别和9和4显示4倍增加和减小> 4倍碱性长期面临的挑战,分别。
为确定其对碱性胁迫的表达响应,筛选了8个lncrna,并通过qRT-PCR验证其特异性表达谱。根据表2, RNA-seq和qRT-PCR分析显示,表达谱存在相同的变异,而不考虑表达的某些异质性。例如,qRT-PCR和RNA-seq分析显示LNC_008366对长期和短期碱性处理的反应上调。绘制qRT-PCR和RNA-seq数据(图。3.b)显示出很大的相关系数(R2= 0.964,P < 0.05 for short-term alkaline challenged samples and R2= 0.902,P在长期碱性处理样本中< 0.05)。
响应碱性胁迫的lncrna候选靶基因及其功能
lncrna在调控基因表达谱中发挥着重要作用;因此,识别和分析特异性靶基因,研究其相关功能可能会有所帮助。根据计算预测,共筛选出133个响应碱性胁迫的lncrna候选靶基因(Additional file Table S .)2).
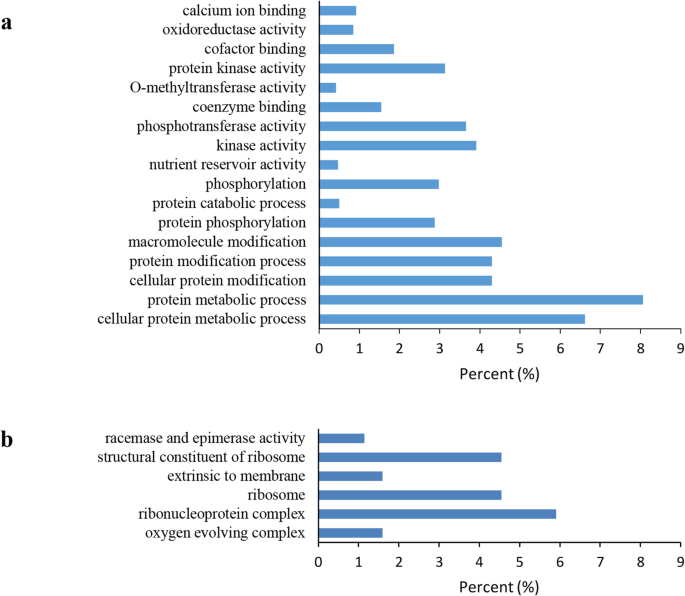
如先前作品所示,LNCRNA更有可能靠近其规定的基因[35那36那37那38]。为了揭示这些被鉴定的lncrna可能的作用,我们对碱反应lncrna靶向基因进行了Gene Ontology (GO)分析。6个和17个GO项显著富集(P< 0.05)。4.).主要的分子功能是激酶活性(GO:0016301)和核糖体结构成分(GO:0003735)。参与核糖核蛋白复合物(GO:0030529)的基因具有较高的细胞组分代表性。在生物过程中,蛋白质的代谢过程(GO:0019538)是最具代表性的GO术语,细胞蛋白的代谢过程(GO:0044267)排名第二。蛋白质代谢过程在各GO项中具有较高的代表性,涉及482个基因。以上结果表明,碱性应答的lncrna可能调节基因参与了响应slat和渗透胁迫的能量合成、信号转导、解毒、分子代谢、翻译和转录等众多生物过程。
在胁迫下,大量氧化石墨烯项显著富集,包括蛋白质(GO:0019538)和核糖核蛋白复合物(GO:0030529)的代谢过程,在短期和长期碱性胁迫下,这些代谢过程在叶片中表现出高度显著性(图2)。4.).2-methyl-6-phytyl-1基因LOC104894575,属于上述2个GO项,在短期碱性胁迫下增加。LOC104894575被估计在lncRNA的监管下LNC_000365, coexpressingLOC104894575.蛋白质代谢是植物调节生长发育和应对环境胁迫的重要生物学过程[39]。我们的结果表明LNC_000365可以通过调节LOC104894575表达式。
在非生物胁迫条件下,信号转导网络将被激发来管理这种胁迫环境。有人认为磷脂代谢途径在应对各种非生物挑战中发挥着重要作用[40]。在本研究中,假设的蛋白质亚型B基因LOC104888232,属于GO:0046488(磷脂酰肌醇代谢过程),在长期碱性胁迫下下调,lncRNAlnc_001194表达于LOC104888232编码序列。根据研究结果,lnc_001194可能会调整LOC104888232表达式。
受到非生物挑战的植物可能会出现氧化损伤,大量活性氧(ROS)的积累证明了这一点,从而破坏膜系统。当活性氧过量积累时,植物会激活保护酶来消除活性氧[41.]。在这项研究中,基因的表达LOC104906740在碱性处理下,过氧化物酶(POD)的编码基因在短期内会增加。我们确定了lncralnc_007731.共同用LOC104906740.这些结果表明lnc_007731.可能通过调节POD的表达参与调控植物对OS的耐受。
碱度对植物生长的抑制作用分为pH胁迫、渗透胁迫和离子毒性[42.]。植物通常表现出类似的耐受机制,包括磷脂信号转导、能量合成和解毒对盐或碱胁迫的改变[43.]。在本研究中,离子转运基因LOC104906281和LOM104892091.通过短期碱性应激均上调。我们确定了lncraLNC_000365coexpressingLOM104892091..这些结果表明LNC_000365可以参与离子舱。肽基 - 脯氨酸改性基因LOC104888024甜菜碱醛脱氢酶基因LOC104894203通过长期和短期碱性应激来上调。lncrna.LNC_004748被预测与LOC104888024和LOC104894203.这些结果表明LNC_004748可能参与渗透调节。
碱性应答lncRNA表达与相关候选靶基因的关联
通过预测对碱胁迫反应的lncrna的候选靶基因,确定碱胁迫后候选靶基因表达的变化。在133个候选共定位靶基因中,4个和4个在转录水平上表现出对短期和长期碱性胁迫的显著响应差异(P< 0.05);在候选共表达靶基因中,3492个基因中有516个和210个在转录水平上表现出显著差异(P< 0.05)。上述候选靶基因表现出碱性反应(P< 0.05)表达水平可能是碱性应答lncRNA的靶点。分析碱度响应型lncRNA表达与碱度响应型候选靶基因的相关性(P< 0.05)表达谱,比较碱胁迫后的表达趋势。在短期碱性处理下,1550(65.8%)和807 (34.2%)lncrna -基因对(65.8%)的表达水平分别呈现相同和相反的趋势(图2)。5.a),而243对(65.9%)和126对(34.1%)lncrna -基因对在长期碱性处理下表现出相同和相反的表达趋势(图3)。5.b).因此,大多数差异表达的靶基因在碱胁迫后与相关lncrna表现出相同的趋势。筛选4对lncrna和目的基因,通过qRT-PCR检测特异性表达水平。lncRNA表达与qRT-PCR识别的候选靶基因的关联与RNA-seq识别的相同(图)。5.例如,c)。LNC_008363及其潜在的靶基因(LOC104894889)在长期和短期碱性处理的反应中上调,其表达趋势相同。此外,与特异性候选靶基因相比,碱性应答lncRNA表达的异质性关系揭示了lncRNA调控机制的多样性。
与光合碳同化相关的碱性应答lncrna
碳同化在植物的生长发育过程中起着至关重要的作用。在本研究中,碱应答lncrna的许多靶基因都与光合碳同化相关(表)3.).例如光系统I亚基O基因的表达LOC104907718葡萄糖-6-磷酸1-脱氢酶基因LOC104897526是由lncRNA调控的LNC_004949,在短期碱性处理下下调了两倍多。三个靶基因lnc_007731.(在长期碱性处理下下调2倍以上)为碳酸酐酶基因LOC104902037,镁原卟啉IX单甲基酯[氧化]环化酶基因LOC104889639葡萄糖-6-磷酸1-脱氢酶基因LOC104897526.总的来说,结果表明,LNCRNA可以通过调节相关基因的表达来参与光合碳固定。
讨论
目前,lncrna仅在几种植物中被系统识别。目前的工作发现了8535个可靠表达的lncrna,揭示了甜菜(甜菜属L.)与其他物种发现的lncrna。首先,基于之前在斑马鱼、人类、水稻、黄瓜和拟南芥上的研究,与蛋白质编码转录本相比,lncrna的长度较短,表达量明显减少[13那25那38那44.那45.]。第二,不同于在各种植物物种中表现出高度保守的mirna [46.],植物中的lncrna表现出较低程度的进化限制。根据我们对玉米、水稻和拟南芥基因组序列的BLAST分析,只有少数甜菜属L. lncrna是保守的(图。2e).此外,对的lncrna进行BLAST分析甜菜属L.相对于NONCODE数据库只识别了少数我们lncrna的保存(1.4%)。其余植物物种的lncrna也显示出类似的观察结果,包括玉米、水稻拟南芥、黄瓜、小麦和杨树[25那26那27那45.那47.那48.]。鉴于这种低保守水平,植物中的这种LNCRNA可以体验快速的进化。三,类似于水稻,拟南芥,人类和其他物种的lncrnas [44.那49.那50.,一些lncrna甜菜属l已被鉴定为靶模拟物或miRNA前体(表1).以上研究结果提示,mirna与lncrna的关联可能在lncrna中发挥重要作用。因此,本研究为探讨其功能提供了丰富的证据甜菜属l . lncRNAs。
lncrna在之前的研究中被认为在各种生物过程中发挥重要作用。然而,lncRNA的碱基响应功能目前还不完全清楚。有报道称lncrna通过与mirna和靶基因的相互作用发挥其功能[51.那52.]。因此,预测和分析与lncRNA相互作用的mirna和靶基因是一种有效的方法,可以检测特定的lncRNA功能。在目前的工作中,我们预测了碱反应lncrna的候选靶基因。此外,我们还鉴定出7个lncrna是17个已经鉴定出的mirna的前体(见表)1)和4个碱基应答lncrna作为4个家族的8个mirna的靶点(表1)2).
植物已进化出多种反应机制来释放非生物胁迫所造成的损害[53.]。许多蛋白质编码基因在植物对非生物胁迫的响应中发挥着重要的作用,包括SOS1和DREB1A / CBF3[54.那55.那56.那57.]。此外,lncrna已经被鉴定为植物增强非生物胁迫耐受性的强大途径[58.]。因此,胁迫应答型lncrna的识别、功能表征和调控网络的构建,为植物环境胁迫耐受性研究提供了新的思路。在最近的一些研究中发现了植物lncrna对非生物/生物胁迫的响应。Liu等[23]发现6484个lincrna,其中1832个对盐度、寒冷、干旱和脱落酸有响应。最近,在杨树中发现了504个对干旱响应的lncrna [49.]。Wang等[21在根和叶样本中鉴定了471个对盐和渗透胁迫响应的lncrna。Wang等[28]共发现10785个lncrnaMedicago truncatula其中根和叶对磷素缺乏反应的分别为224和358种。在本工作中,所有的lncrna,如反义lncrna和义lncrna,都是通过使用最先进的测序技术(链特异性测序)和分析方法(如Cuffcompare分析)发现的。此外,从短期和长期碱处理的叶片样本中发现了特异性和常见的lncRNA,以研究甜菜lncRNA在碱胁迫下可能的功能。据我们所知,目前的工作首次报道了利用高通量测序技术从短期和长期碱处理的甜菜叶片样品中分离出的lncrna的系统检索、表征和分析。
在本研究中,17和6个GO项显著富集(P< 0.05),分别在短期和长期的碱性胁迫下(图。4.).分子功能、细胞成分和生物过程的主要类别分别是激酶活性(GO:0016301)、核糖核蛋白复合物(GO:0030529)和蛋白质代谢过程(GO:0019538),这与Wang等人的研究结果相似[21]。此外,在本研究中,估计了几种lncrna在响应非生物胁迫时调节基因表达。例如lncRNAlnc_007731.与该基因共表达LOC104906740,编码过氧化物酶和肽基-脯氨酸修饰基因LOC104888024甜菜碱醛脱氢酶基因LOC104894203是由lncRNA调控的吗LNC_004748.以上研究结果证实了lncrna在植物应答环境胁迫中的重要作用。
除了可能与特定靶基因相互作用外,响应碱性胁迫的lncrna也与mirna相互作用。在目前的工作中,我们发现了3个lncrna作为4个家族的8个mirna的候选靶点(表)3.).根据研究结果,对碱性胁迫反应的lncrna也可能通过与mirna的相互作用影响许多不同的生物过程。
结论
从3个样本中共发现8537个可靠的lncrna甜菜属L. RNA-seq文库的高通量测序;其中102只对短期碱性胁迫有反应,49只对长期碱性胁迫有反应。在这些碱性应答lncrna中,有6个lncrna在短期和长期碱性胁迫下表达上调,而有5个lncrna在短期和长期碱性胁迫下表达下调。在这些对碱性胁迫反应的lncrna中,4个被确定为来自4个家族的8个mirna的靶点。将miRNA前体与8535个lncrna比对后,发现7个lncrna是17个已鉴定miRNA的前体。此外,计算预测结果显示,有4318个lncrna候选靶基因对碱胁迫反应。在细胞结构、分子功能和生物学过程中,包括激酶活性、核糖体结构成分、核糖核蛋白复合物和蛋白质代谢过程中,发现氧化石墨烯富集。建立了基因组蛋白编码基因与lncrna共配共表达的相互作用网络。根据我们的研究结果,lncrna可能通过与蛋白质编码基因的相互作用,在调节植物的反应以及对碱性处理的适应中发挥重要作用。上述结果为进一步研究植物lncRNA的功能特性提供了参考甜菜属在非生物胁迫下。
方法
植物材料及处理条件
德国KWS公司提供的粒状甜菜品种“KWS0143”种子在含有蒸馏水的蛭石中发芽一周。然后在相对湿度65%、白天/晚上25°C/20°C、光照强度450 μmol·m条件下,用pH为6.85的霍格兰溶液(Hoagland solution)灌溉育苗4周−2·S.−1光-暗周期为16 h - 8 h。霍格兰解决方案的组件在附加文件表S中列出3..最后,用75mM碱性溶液混合物(NaHCO)处理幼苗(NaHCO3.: Na2有限公司3.分别为0天(对照,指定为C)、3天(短期处理,指定为ST)和7天(长期处理,指定为LT)。采集不同处理相同位置的鲜叶,制备3个生物重复。采集的叶片标本采集后立即在液氮中冷冻,在−80℃保存,用于生理参数测定、RNA-seq和qRT-PCR检测。
生理参数测量
过氧化物酶(POD)活性按照Zhang的方法测定[59.]。通常,1单位POD活性被认为是在37℃下1 mg蛋白在1 min内催化1 μg底物所需的酶量。丙二醛(MDA)含量由Li [60]。将植物组织收集到预冷的乙酰caustin中,10000 rpm离心10分钟。然后,收集上清液,与硫代巴比妥酸混合,然后转移到沸水浴中15分钟。在10000 rpm离心10分钟后,在波长为600、532和450 nm处测定MDA含量。根据茚三酮比色法测定脯氨酸水平[61]。
通过使用CI-340便携式光合作用系统(CID,Inc.,USA),基于三个全面延伸的叶片确定蒸腾速率(TR),气孔导率(GS)和净光合速率(PN)。根据Oelze等人的描述,利用迷你PAM荧光计(沃尔兹,德国)测定光照条件下的量子产量。[62]。
RNA提取,文库构建,RNA测序
利用0-d、3-d和7-d碱处理植物的叶片样本(每个处理3个生物重复)进行高通量rna测序(RNA-seq)。然后使用Qiagen RNAeasy试剂盒(Qiagen China, Shanghai, China)按照制造商协议提取总RNA。1%琼脂糖凝胶用于监测RNA的污染和降解。采用纳米光度计®分光光度计(IMPLEN, CA, USA)检测RNA纯度。使用Qubit®2.0 A荧光仪和Qubit®RNA检测试剂盒(Life Technologies, CA, USA)测量RNA含量。采用Bioanalyzer 2100系统和RNA Nano 6000检测试剂盒(美国安捷伦技术公司,CA,美国)进行RNA完整性评估。
从每个样本中采集3微克RNA,作为RNA制备的输入材料。首先,使用Epicentre Ribo-zero™rRNA Removal Kit (Epicentre, USA)去除核糖体RNA (rRNA),通过乙醇沉淀去除rRNA的游离残基。随后,根据制造商协议,利用Illumina®(NEB,美国)的NEBNext®Ultra™定向RNA库Prep Kit生成基于r-RNA-free RNA的测序库。简而言之,使用NEBNext第一链合成反应缓冲液(5X),二价阳离子在升高的温度下被裂解。然后采用M-MuLV逆转录酶(RNaseH-)和随机六聚体引物制备cDNA第一链。用RNase H和DNA聚合酶i合成二链cDNA,用dUTP替代反应缓冲液中含有dTTP的dNTPs。通过酶外切/聚合酶的活性,剩余的悬垂部分变为钝端。DNA片段3 '端腺苷化后,连接具有发夹环结构的NEBNext接头进行杂交。为优先筛选长度为150 ~ 200 bp的cDNA片段,采用AMPure XP系统(Beckman Coulter, Beverly, USA)纯化文库片段。然后用3 μl USER Enzyme (NEB, USA)与根据大小选择的cDNA在37℃下连接15 min,然后在95℃下连接5 min,然后进行PCR。 Later, PCR was carried out using Index (X) Primer, Universal PCR primers, and Phusion High-Fidelity DNA polymerase. Finally, the Agilent Bioanalyzer 2100 system was used to purify products (AMPure XP system) and assess library quality.
利用cBot聚类生成系统,使用TruSeq PE聚类试剂盒v3-cBot-HS (Illumina),按照制造商协议对指标编码的样本进行聚类。在集群生成之后,使用Illumina HiSeq 4000平台(诺福金生物信息技术有限公司,北京,中国)对所有文库进行测序。
LNCRNA的基因组特征
fastq格式的原始读取首先由内部Perl脚本处理,然后通过删除包含适配器、包含poly- n的读取以及原始读取中的低质量读取来获得干净的读取。确定clean reads的GC水平、Q20和Q30。每一个下游分析都是在高质量的清洁数据的基础上进行的。
基因模型和参考基因组的注释文件直接从基因组网站获得。利用Bowtie2 v2.2.8软件构建参考基因组指数,HISAT2 [63应用v2.0.4软件将对端干净数据与参考基因组进行比对。在运行HISAT2时设置'——rna链度RF ',其余参数设置为默认值。
StringTie (v1.3.1) [64]软件在参考文献的基础上,为每个样品组装映射读数。在StringTie中,一种新的网络流算法和一个可选的从头组装过程被用来组装和量化每个基因位点的多个剪接变体的全长转录本。
编码-非编码-索引(CNCI) (v2)描述了邻近的核苷酸三联体,用于有效区分蛋白质编码序列和那些不受已知注释影响的非编码序列[65]。在本研究中,CNCI使用默认参数。此外,编码潜能计算器(CPC) (0.9-r2)评估一个转录本内的ORF质量和程度,并根据已识别的蛋白序列数据库搜索序列,从而明确非编码和编码转录本[66]。本研究采用NCBI真核生物蛋白数据库,e值设为“1e-10”。所有3个潜在框架中的每个转录本都被翻译,并使用Pfam Scan (v1.3)从Pfam数据库中识别每个已知蛋白家族结构域的出现频率(第27版;均使用Pfam A和Pfam B) [67]。在随后的步骤中,有一个Pfam命中的文字记录被删除。在Pfam搜索中使用了默认参数,包括-E 0.001和——domE 0.001 [67]。此外,PhyloCSF(系统发育密码子取代频率)(v20121028)还可以确定保守编码区比对的特定进化特征,如较大的同义密码子取代,以及保守的氨基酸取代频率,以及其他错义或无意义替代频率的降低,从而将蛋白质编码转录本与非编码转录本区分开来[68]。本研究构建了多个物种的基因组序列比对,使用默认参数运行phyloCSF。剔除通过上述四种方法中任意一种估计具有编码能力的转录本,将无编码能力的转录本纳入候选lncrna。
Phast (v1.3)软件包涵盖了许多统计程序,它经常被用于系统发育分析[69];此外,phastCons已被开发为保守元素的保守评分和识别程序。在本研究中,利用系统发育fit计算跨物种的非保守和保守区域的系统发育模型,并将HMM转换和模型参数提供给phyloP,用于计算各种lncrna和蛋白编码基因的保守评分。
使用ASProfile V1.0软件,替代剪接(AS)事件被聚集成12种基本类型。此后,预测每个样本的AS事件编号。
lncRNA预测和碱性应答lncRNA鉴定
本研究采用Cuffdiff (v2.1.1)计算所有样本lncrna的FPKMs(每千碱基外显子模型每百万映射片段的片段数)以及编码基因[70]。通常,基因FPKMs是通过添加每个基因组的所有转录本FPKMs来计算的。根据片段长度和映射读的个数确定FPKM。
Ballgown套件能够交互地探索转录组组装,可视化转录组结构以及每个位点的特征特异性丰度,并将这些组装的特征注释为已注释的特征[71]。经过调整的成绩单P-值< 0.05视为差异表达。在Cuffdif中,使用一个模型来提供统计例程,根据负二项分布确定数字转录本或基因表达谱数据的不同表达模式[70]。经过调整的成绩单P-值< 0.05视为差异表达。
靶基因预测
CIS的作用表明LNCRNA对邻近靶基因作用。在该研究中,在LNCRNA的上游和下游搜索了编码基因10 k / 100k;后来,随后分析了具体功能。相反,跨越作用表明LNCRNA基于表达量识别彼此。给出了具有定制脚本的编码基因的LNCRNA的表达相关性,给出了<25的小样本大小;否则,使用WGCNA聚集基于各种样品获得的基因[72]识别常见表达模块;然后,进行功能性富集分析以检查其功能。
基因本体(GO)分析
对这些deg或lncrna的靶基因进行GO分析,在纠正基因长度偏倚后,使用GO seq R包进行分析[73]。一个被修正过的GO术语P-值< 0.05表示deg显著富集。
实时定量PCR (qRT-PCR)
分别以碱处理0天、3天、7天的植物叶片为样本,提取总RNA,反向转录制备cDNA,用于测定碱胁迫应答lncrna、靶向mirna的碱应答lncrna、qRT-PCR检测候选碱基应答lncRNA靶基因。采用ABI StepOne Plus装置和SG Fast qPCR Master Mix试剂盒进行qRT-PCR。引物设计采用Primer Express 5.0,通过PCR产物测序确定引物对特异性。每次qRT-PCR扩增采用统一反应程序进行3次,通过生成熔融曲线确定扩增片段的特异性。采用Opticon Monitor Analysis Software 3.1对根据2生成的实时数据进行分析-ΔΔct方法(74]。附加文件表S4.显示了qRT-PCR所使用的引物。
数据和材料的可用性
序列数据已提交给NCBI基因表达式综合征(Geo Rescession Number GSE107627)。
缩写
- lncRNAs:
-
长非编码rna
- ncRNAs:
-
非编码RNA.
- microrna:
-
小分子核糖核酸
- sirnas:
-
小干扰rna
- FPKM:
-
每千碱基外显子模型片段每百万映射片段
- 存在:
-
定量实时聚合酶链反应
- ROS:
-
活性氧
- 荚:
-
过氧化物酶
- 走:
-
基因本体论
参考文献
- 1.
Wakeel A,Asif Ar,Pitann B,Schubert S.糖甜菜蛋白酶蛋白酶分析(Beta寻常L.)在盐胁迫的第一阶段阐明了组成型适应。J植物理性。2010; 168(6):519-26。
- 2.
Flowers TJ, Colmer TD。盐生植物的耐盐性*布莱克威尔出版有限公司;2008.
- 3.
王应祥,孙桂荣,王俊波,陈刚,曹文忠,刘爱萍,王超。Na2CO3和NaCl胁迫对细叶Puccinellia tenuiflora幼苗叶绿体抗氧化酶的影响比较。草业学报。2007;16(1):81-6。
- 4.
Gong B,Li X,Bloszies S,Wen D,Sun S,Wei M,Li Y,Yang F,Shi Q,王X.通过一氧化氮和多胺相互作用的王X. Sodic碱性应力减缓涉及溶氧化剂和生理策略在Solanum Lycopersicum中的抗氧化剂和生理策略。自由Radic Biol Med。2014; 71(6):36-48。
- 5。
徐东,土延多。大豆耐盐和耐钠性的遗传研究。Sci繁殖。2012;61(5):559 - 65。
- 6.
龚B,Wen D,Vandenlangenberg K,Wei M,Yang F,Shi Q,Wang X. NaCl和NaHCO 3对番茄叶片光合参数,营养代谢和抗氧化系统的对比作用。SCI Hortic。2013; 157(3):1-12。
- 7.
Chekanova JA, Gregory BD, Reverdatto SV, Chen H, Kumar R, Hooker T, Yazaki J, Li P, Skiba N, Peng Q. Genome-wide高分辨率mapping of exosome substrates reveals hidden features in Arabidopsis转录组。细胞。2007;131(7):1340 - 53年。
- 8.
Kapranov P, Cheng J, Dike S, Nix DA, Duttagupta R, Willingham AT, Stadler PF, Hertel J, Hackermüller JR, Hofacker IL. RNA图谱揭示了新的RNA类和普遍转录的可能功能。科学。2007;316(5830):1484 - 8。
- 9.
非编码rna的长与短。细胞生物学杂志。2009;21(3):416-25。
- 10.
金玉娟,郑波,于勇,袁淑英,莫波,陈晓霞。拟南芥小而长的非编码RNA产生的中介作用。EMBO j . 2011; 30(5): 814 - 22所示。
- 11.
长链非编码rna对基因组的调控。生物化学学报。2012;81(1):145。
- 12.
JA C.长期非编码RNA及其在植物中的功能。CurrOp植物BIOL。2015; 27:207-16。
- 13.
Cabili Mn,Trapnell C,Goff L,Koziol M,Tazon-Vega B,Regev A,Rinn JL。人类大的非基础非编码RNA的整合性注释揭示了全局性质和特定子类。基因开发。2011; 25(18):1915。
- 14。
长链非编码rna的进化与功能。细胞。2009;136(4):629 - 41。
- 15.
李军,黄欢,张南海。植物长链非编码RNA转录组研究。植物生态学报;2015;13(3):319-28 ..
- 16.
Kornienko AE, Guenzl PM, Barlow DP, Pauler FM。长链非编码RNA转录的基因调控。BMC医学杂志。2013;11(1):59。
- 17.
Franco-Zorrilla JM, Valli A, Todesco M, Mateos I, Puga MI, Rubio-Somoza I, Leyva A, Weigel D, García JA, Paz-Ares J.靶标拟态为调控microRNA活性提供了新的机制。Nat麝猫。2007;39(8):1033。
- 18.
刘芳,刘芳,王志刚,等。拟南芥低温诱导沉默的研究进展。大自然。2009;462(7274):799。
- 19.
丁俊,卢强,欧阳云,毛浩,张鹏,姚军,徐超,李昕,肖军,张强。一种长链非编码RNA对杂交水稻光周期敏感雄性不育的调控。中国科学院院刊2012;109(7):2654-9。
- 20.
Ariel F, Teddy J, David L, Barrios R, Aurélie N.非编码转录由替代RNA聚合酶动态调节生长素驱动的染色质环。55摩尔细胞。2014;(3):383 - 96。
- 21.
王tz,刘米,赵mg,陈立,张wh。基因组高通量测序,Medicago Truncatula中渗透渗透和盐胁迫的长编码RNA的鉴定与表征。BMC植物BIOL。2015; 15:131。
- 22.
王Y,风扇X,林F, G, Terzaghi W,朱D,邓小平XW。拟南芥非编码RNA介导红光对光形态建成的调控。中国科学(d辑:地球科学)2014;
- 23.
刘建军,荣超,徐建军,王华,邓胜,Bernad L, Arenashuertero C, Chua NH。全基因组分析揭示了拟南芥中长基因间非编码rna的调控。植物细胞,2012;24(11):4333 - 45。
- 24.
朱庆华,王明波。拟南芥尖孢镰刀菌(Fusarium oxysporum)侵染过程中长链非编码rna的响应。新植醇。2014;201(2):574 - 84。
- 25.
张永超,廖建勇,李志勇,余勇,张建平,李庆峰,曲丽华,舒文伟,陈玉强。通过全基因组筛选和功能分析,鉴定出大量参与水稻有性生殖的长链非编码rna。基因组医学杂志。2014;15(12):512。
- 26.
李磊,Eichten SR, Shimizu R, Petsch K, Yeh CT, Wu W, Chettoor AM, Givan SA, Cole RA, Fowler JE。玉米长链非编码rna的全基因组发现和特征分析。基因组医学杂志。2014;15 (2):R40。
- 27。
田军,宋勇,杜强,杨旭,慈东,陈静,谢军,李斌,张东。杨杨赤霉素响应长链非编码rna的群体基因组分析。J Exp Bot. 2016;67(8):2467。
- 28。
王涛,赵敏,张旭,刘敏,杨超,陈勇,陈锐,文俊,迈苏尔KS,张文辉。豆科模型植物截茎紫花苜蓿中新型磷酸缺乏应答长链非编码rna的研究。J Exp Bot. 2017;68(21-22): 5937-48。
- 29.
Magaña C, Núñez-Sánchez N, Fernández-Cabanás VM, García P, Serrano A, Pérez-Marín D, Pemán JM, Alcalde E.用近红外光谱法直接预测甜菜粕中生物乙醇的得率。Bioresour抛光工艺。2011;102(20):9542。
- 30.
陈志强,王志强,王志强,等。干旱胁迫下甜菜的渗透调节反应及其基因表达分析。寻常的和maritima。植物学报,2013;04(01):11-6。
- 31.
李华,潘勇,张勇,吴超,马超,于波,朱宁,高军,陈胜。甜菜单体添加系M14膜蛋白组对盐胁迫的响应。蛋白质组学杂志。2015;127(Pt A): 18-33。
- 32.
杨丽,马超,王丽,陈树华,李海霞。盐胁迫对甜菜单倍体添加系M14蛋白质组和转录组的影响。植物生理学杂志。2012;169(9):839-50。
- 33.
侯赛因女士,埃尔赛义德AI,摩尔M,迪茨KJ。甜菜耐盐驯化中的氧化还原和活性氧网络。J Exp Bot. 2017;68(5): 1283-98。
- 34.
Dohm JC, Holtgräwe D, Zakrzewski F, Tafer H, Rupp O, Stracke R, Reinhardt R.最近驯化的作物植物甜菜(Beta vulgaris)的基因组。大自然。2014;505(7484):546。
- 35.
Wang JK, Xu X, Brugmann SA, Goodnough LH, Helms JA, Farnham PJ, Segal E. human HOX位点活性和沉默染色质结构域的非编码rna功能划分。细胞。2007;129(7):1311。
- 36.
默瑟TR,丁格ME,马特克JS。长链非编码rna:功能研究。中国农业科学。2009;10(3):155-9。
- 37.
Yu W,Gius D,Onyango P,Muldoon-Jacobs K,Karp J,Feinberg AP,Cui H.其反义RNA对肿瘤抑制基因P15的表观遗传沉默。自然。2008; 451(7175):202-6。
- 38.
Pauli A,Valen E,Lin MF,Garber M,Vastenhouw NL,Levin JZ,风扇L,Sandelin A,Rinn JL,Regev A等。斑马鱼胚胎发生期间表达长的非分量RNA的系统鉴定。Genome Res。2012; 22(3):577-91。
- 39。
郝丽丽,温艳艳,赵艳艳,陆伟,肖凯。小麦丝裂原激活蛋白激酶TaMPK4基因通过调控根生长、活性氧代谢和营养物质获取改善植物对多种胁迫的耐受性。植物学报2015;34(12):2081-97。
- 40.
王雪梅,张伟,李伟,李伟。植物对干旱和盐胁迫响应的磷脂信号通路研究;2007.
- 41.
抗氧化应激、抗氧化剂与应激耐受性。植物科学进展。2002;7(9):405-10。
- 42.
Gao P, Bai X, Yang L, Lv D, Pan X, Li Y, Cai H, Ji W, Chen Q, Zhu Y. Osa-MIR393:盐碱胁迫相关的microRNA基因。中国人民大学学报(自然科学版)2011;38(1):237。
- 43.
熊磊,朱建军。寒冷、干旱和盐胁迫下的细胞信号。植物学报。2002;14(Suppl (Suppl)):S165。
- 44.
王H,忠宝,刘家,jang ic,kean mj,xu j,chua nh。全基因组鉴定长度非划分的天然反义转录物及其对拟南芥光的反应。Genome Res。2014; 24(3):444。
- 45.
郝志强,范超,程涛,苏勇,魏强,李国强。黄瓜基因间长链非编码rna的全基因组鉴定、鉴定及进化分析。《公共科学图书馆•综合》。2015;10 (3):e0121800。
- 46.
Sunkar R,Jagadeeswaran G.在大量多种植物物种中的Silico识别保守MicroRNA。BMC植物BIOL。2008; 8(1):37。
- 47.
麦金塔GC,绿色PJ。拟南芥非编码rna特征序列标签的鉴定与分析。植物杂志。2001;127(3):765 - 76。
- 48.
关键词:小麦,白粉病,热胁迫,长链非蛋白编码rna,微阵列分析,序列分析植物学报。2011;11(1):61。
- 49.
梁东,唐绍平,叶春英,苏勇,夏旭,尹伟。白杨lincrna基因的克隆及功能预测。[J] .机器人。2014;65(17):4975-83 .]
- 50.
关键词:长链非编码RNA,番茄,成熟,功能分析中国生物医学工程学报。2015;66(15):4483-95。
- 51.
吴HJ,王祖,王M,王XJ。广泛的长期非致rNA作为植物中微小RNA的内源目标模拟。植物理性。2013; 161(4):1875。
- 52.
陈建军,全敏,张东东。毛白杨张、对生木和正常木木质部新型长链非编码rna的rna序列分析。足底。2015;241(1):125 - 43。
- 53.
朱镕基j - k。植物的盐和干旱胁迫信号转导。植物学报2002;53:247-73。
- 54.
刘Q,Kasuga M,Sakuma Y,Abe H,Miura S,Yamaguchi-Shinozaki K,Shinozaki K.两种转录因子,DREB1和DREB2,eSBP / AP2 DNA结合结构域分别在干旱和低中分别两种细胞信号转导途径- 在拟南芥中分别进行浓度响应基因表达。植物细胞。1998年; 10(8):1391-406。
- 55.
Jagloottosen KR,Gilmour SJ,Zarka DG,Schabenberger O,Thomashow MF。Arabidopsis CBF1过表达诱导COR基因并增强冷冻耐受性。科学。1998; 280(5360):104-6。
- 56.
刘军,朱家宽。植物耐盐性所需的钙传感器同系物。科学。1998;280(5371):1943 - 5。
- 57.
邱秋生,郭勇,马迪特里希,Schumaker KS,朱建军。SOS2和SOS3对拟南芥质膜Na+/H+交换剂SOS1的调控中国科学院院刊2002;99(12):8436-41。
- 58.
Jian-Kang。非编码rna作为作物改良的有力工具。中国科学(d辑:地球科学)(英文版);
- 59.
张铮。植物生理学实验指导。北京:高等教育出版社;2004.
- 60.
李辉。植物生理生化实验原理与技术。北京:高等教育出版社;2003.
- 61.
林志强,王志强,王志强。一种测定脯氨酸的光度法。[J] .中国生物医学工程学报,2017;
- 62.
Oelze ML, Vogel MO, Alsharafa K, Kahmann U, Viehhauser A, Maurino VG, Dietz KJ。拟南芥叶片叶绿体抗氧化防御响应10或100倍的光增长和可能参与的逆行信号的有效驯化。中国生物医学工程学报。2012;63(3):1297。
- 63.
Langmead B, Salzberg SL.快速间隙读对齐与领结2。Nat方法。2012;9(4):357。
- 64.
Pertea M, Kim D, Pertea GM, Leek JT, Salzberg SL. HISAT、StringTie和Ballgown RNA-seq实验的转录水平表达分析。Nat Protoc。2016;11(9):1650。
- 65.
孙丽,罗华,卜东,赵刚,于凯,张超,刘颖,陈瑞,赵颖。利用序列固有成分对蛋白质编码和长链非编码转录本进行分类。核酸学报2013;41(17):e166。
- 66.
孔玲,张勇,叶志强,刘新强,赵树清,魏玲,高刚。CPC:利用序列特征和支持向量机评价转录本的蛋白编码潜力。核酸Res. 2007;35(Web服务器版):W345。
- 67.
Finn RD, Tate J, Mistry J, Coggill PC, Sammut SJ, Hotz HR, Ceric G, Forslund K, Eddy SR, Sonnhammer EL,等。Pfam蛋白家族数据库。核酸Res. 2008;36(数据库版):D281-8。
- 68.
林MF,Irwin J,Manolis K.Hhylocsf:区分蛋白质编码和非编码区的比较基因组学方法。生物信息学。2011; 27(13):I275。
- 69.
Siepel A, Bejerano G, Pedersen JS, Hinrichs AS, Hou M, Rosenbloom K, Clawson H, Spieth J, Hillier LW, Richards S.脊椎动物、昆虫、蠕虫和酵母基因组的进化保守元素。基因组研究杂志2005;15(8):1034。
- 70.
Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ, Salzberg SL, Wold BJ, Pachter L.通过RNA-Seq转录本组装和定量揭示了细胞分化过程中未注释的转录本和亚型转换。生物科技Nat》。2010;28(5):511 - 5。
- 71.
Frazee AC, Pertea G, Jaffe AE, Langmead B, Salzberg SL, Leek JT。灵活的分析与Ballgown转录组。Biorxiv。2014;8:1-11。
- 72.
Langfelder P,Horvath S.WGCNA:用于加权关联网络分析的R包。BMC生物信息学。2008; 9(1):559。
- 73.
Young MD, Wakefield MJ, Smyth GK, Oshlack A. RNA-seq的基因本体论分析:考虑选择偏差。基因组医学杂志。2010;11 (2):R14。
- 74.
Livak KJ, Schmittgen TD。使用实时定量PCR和2(−Delta Delta C(T))方法分析相对基因表达数据。方法。2012;25(4):402 - 8。
致谢
不适用。
资金
国家自然科学基金项目(no . 31671622);中国糖业技术体系项目(no . CARS-170201)。关键词:糖业;所有资助人都没有参与研究设计、数据提取和讨论、发表决策或手稿撰写。
作者信息
从属关系
贡献
CLZ和CFL负责实验设计;CLZ、BW、DL、ZJG和LL实现了实验;YBW提供了部分实验仪器;CLZ分析了数据;CLZ负责稿件的撰写。每个作者都仔细阅读并批准了最终出版的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
所有作者都声明没有利益冲突。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:表S1。
对短期和长期碱性处理有差异表达的lncrna。
附加文件2:表S2。
碱性应答lncrna的靶基因。
附加文件3:表S3。
Hoagland解决方案的组件。
附加文件4:表S4。
QRT-PCR中使用的引物。
附加文件5:图。S1。
碱性处理对生长特性的影响(本照片比例为1:4)。(a)在无或有75 mM碱性处理7天的植物形态(b-e)蒸腾速率(Tr)、气孔导度(Gs)、净光合速率(Pn)和光系统II (Y(II))量子产量的变化。C代表对照,而A代表碱胁迫的叶子。错误条表示3个生物重复的SDs (n= 3)。星号表示碱激发样本与对照样本的统计学差异(*P< 0.05;Arunachal Pradesh,P< 0.01)。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
邹超,王勇,王博。et al。甜菜碱胁迫反应中的长链非编码rna (甜菜属l.).BMC植物BIOL.20.227(2020)。https://doi.org/10.1186/s12870-020-02437-w
收到了:
公认:
发表:
关键字
- 长非编码rna
- 甜菜属l
- 碱性的压力
- RNA序列
- 计算分析
- 功能注释