抽象的
背景
Web Blotch是花生世界上最重要的叶面疾病之一(arachis hypogaea.l .)。用于花生腹板斑电阻的定量性状基因座(QTLS)代表基因采矿的基础及分子育种技术的应用。
结果
在该研究中,使用了一种基于高通量基因组的测序来映射用于映射β斑块电阻的QTL的花生重组近红细(RIL)。五种环境中疾病级和疾病指数的频率分布表明rils之间的腹板斑点的宽度变化。构建高密度遗传图,含有3634纳箱标记物,分布在20个花生键(LGS)上,平均遗传距离为0.5cm。总共检测到至少两个环境中的花生腹板斑块的八个QTL,从2.8%到15.1%的表型方差下解释。两个主要的QTLqwbra04.和qWBRA14在所有五种环境中检测到并链接到编码核苷酸结合位点的40个候选基因,与疾病抗性有关的富含核苷酸富含亮氨酸的重复(NBS-LRR)或其他蛋白质。
结论
本研究的结果为培育花生品种具有腹带斑岩抗性提供了基础。
背景
栽培花生(arachis hypogaea.L.)是许多国家最重要的石油豆类之一。A. hypogaea.为异源四倍体(AABB, 2n = 4x = 40),基因组大小约为2.7Gb [1].关于许多中国花生品种“福华市”和“石油季”和“石油乐”和一家美国品种“TIFRUNNER”的两个着名父母品种的基因组大会结果出版于2019年[2,3.,4].组装序列为约2.54gb,追踪基因约为60〜8000万2,3.,4].
花生网状斑点,也称为泥质点或净污染[5,6),由araehidieola phomaMarasas,扶手&Boerema [7,8],是在花生中最重要的叶面疾病之一。花生网斑病最早是在得克萨斯州(中美)在20世纪70年代报道[9]然后,在20世纪80年代初,它是在山东和辽宁省的主要花生生长区发现[10.,11.].随后,在山西省发现了它[12.]及河南省[13.].在整个花生生长期间,卷曲斑疾病可能会导致产量损失10%〜20%,但最重的损失超过50%。因此,它被认为是一些花生生长领域的最紧急问题之一。
真菌P. Araehidieola.病原体通常在农作物残留物和所有年龄段的植物中易受影响[6].多云的天气频繁下雨和温度(15-30°C)有利于真菌活动[14.].孢子萌发和角质层渗透发生在花生传单上,症状在接种后出现了7至9天。观察到沿着年轻和老叶的中间叶中的小,不规则,棕色到红棕色病变的初始症状,并且在感染的易感品种的感染领域观察到严重的脱落[15.].在角质层渗透后,真菌菌丝在角质层下长大于角质层,超出了渗透点并停止生长,这表明防御反应P. Araehidieola.感染是过敏型反应[6].
虽然网斑病在花生种植系统中具有重要的经济意义,但与其他主要花生叶面病害相比,对网斑病的研究相对较少。Zhang et al.(2019)提出了aaraehidieola phoma分离为WB2并表明WB2的基因组草案约为34.11 MB,并包含37,330个开放阅读框架(ORF),G + C含量为49.23%[16.].史密斯等人。(1979)报道,弗吉尼亚州和亚军市场型花生品种对网斑比西班牙市场型品种[更耐17.].张等人。(2011)表明,对网状斑点的抗性由三个主要基因和几种次要基因控制,并确定了位于连杆基团(LG)7上的一种定量性状基因座(QT1)[18.].目前,对花生腹板斑点的研究主要关注病原体无性发电,病原体分子生物学,化学控制和疫情规则的分类状态[6,7,8,9,14.,15.].一种类似的疾病叫网斑,由Pyrenophora Teres.也是大麦的主要病害之一[19.].许多研究鉴定了大型染色体6h,负责大麦的净爆破抗性[20.,21.,22.,23.[染色体3H中的一个区域来自NBS-LRR基因家族的两个预测基因[24.].
随着基因组测序技术的进步和二倍体和四倍体基因组组装的可用性arachis.物种 [1,4[高通量基因组宽测序已成为花生的主要策略,以鉴定与抗性基因和QTL有关的单核苷酸多态性(SNP)标记。例如,使用RIL群体和2187个SNP标记在染色体B02上鉴定了四种细菌枯萎性QTL [25.].用于后期叶斑病抗性的主要QTL,染色体B05,以及用于向早期叶斑病电阻两个主要的QTL,在染色体A03和B04,使用高密度遗传学图,其包括2753个SNP标记和A F被映射9192个单独的线条的人口[26.].此外,使用具有2636重组箱标记和F的高密度遗传图谱,在三种环境中检测到14个产量相关性具有62个产量相关性状的QTLS。包括2636个重组箱标记物和F.6RIL人口242行[27.].
本研究利用高通量全基因组测序技术获取SNP标记,构建基于SNP的遗传连锁图谱,鉴定花生网斑抗性qtl。本研究的结果将有助于更好地理解二者相互作用的机制P. Araehidieola.和花生并造成抗性品种。
结果
腹板斑抗性评估
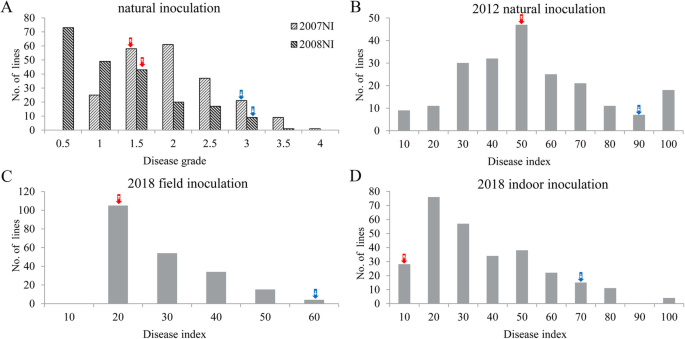
在本研究的所有五种测试环境下,在rils中观察到响应花生腹板斑的宽表型变化,并且群体的两个父母品种也显示出腹斑性耐受性显着差异(图。1).在2007和2008年期间记录的疾病规模的分布如图2所示。1一个,而在图中示出在2012年和2018(无论是在野外和室内)记录病情指数的分布。1B-D。疾病指数的在2012年的频率分布几乎是正常分布(图1b)。在其他条件下的频率分布显示偏态分布(图1a,c和d)对以下绘图结果没有影响,因为只需要表型数据的随机误差项来遵循正常分布[28.].
测序,SNP和BIN标记发现
采用全基因组重测序方法构建亲本及其212个RIL子代的对端文库。文库的DNA片段长度约为350 bp。大约产生了490 Gb的干净数据(Q20 > 96%),由6.285亿次读取产生,每一次读取的长度为150 bp。两个父代各生成6亿个读,而每个ril生成的读从2237万到2483万不等(补充表)S1).在补充表中示出了覆盖率,映射读取率,序列深度和其他结果与参考基因组的对准结果S1.特别是,与双父母Zheng8903和yuhua4相关的覆盖率分别为98.51和99.05%,而群体群体的53%至63.63%(补充表S1).父母的测序深度为35.23×,而且群体的范围从1.31倍到1.46×。最初,使用GATK协议从214个样品调用636,831个SNP。然后,由于缺少值,杂小尿量,深度<10和GQ <20,在两个父母中过滤低质量基因座后,保留了556,615个SNP。最后,使用两种父母之间的纯合的138,039个SNP和两种父母之间的多态性进行进一步分析。
物理重组图的构建和高密度遗传联系地图
为了避免由与RIL测序相关的低覆盖率引起的错误,使用具有15个连续SNP的滑动窗口来寻找更准确的重组断点。基于每个后代的重组图构建212个rils的物理重组图(补充图S1).然后,对212条ril的所有染色体进行比对,每100 kb的最小间隔进行比较。通过该方法共获得212个品系的3634个箱标记,箱的基因型和物理位置详见补充表S2.
利用获得的3634个bin标记,利用JoinMap软件构建遗传连锁图谱®v5.0 [29.].产生20个连杆基团并根据物理位置分配给栽培花生的20染色体。总基因组长度为1817.91cm,20个连杆基团的标记密度范围为0.39至0.66cm,平均为0.50cm(表1).LG16标记数最低(129个),遗传长度最短(54.58 cM), LG3标记数最高(277个),遗传长度最长(135.61 cM)(见表)1和图。2).97.5%以上的标记间距小于3 cM。标记间距离最高(16.06 cM)与LG6相关(表2)1).
花生网斑抗性QTL定位及候选基因预测
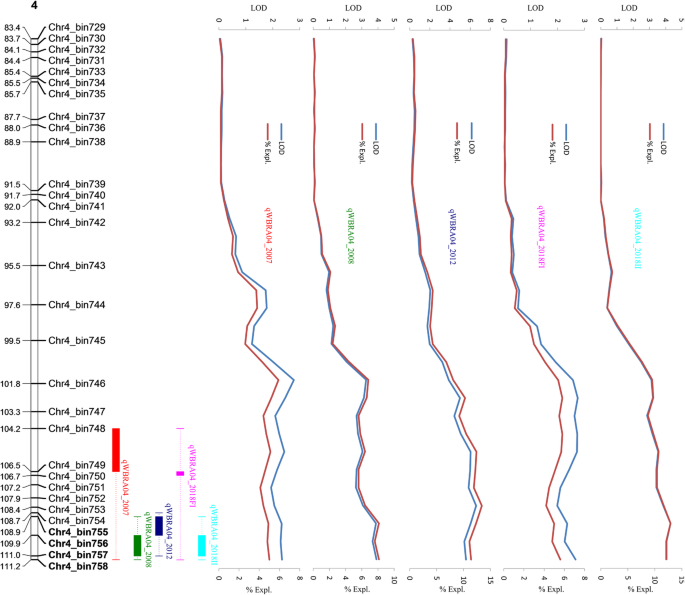
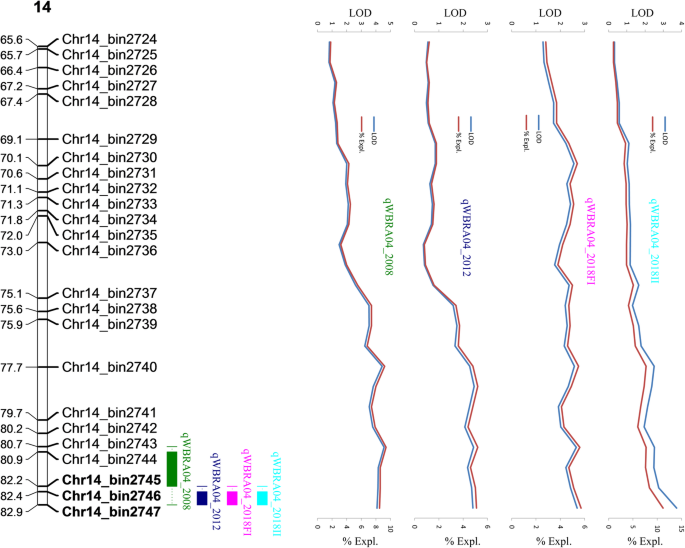
利用MapQTL进行花生网斑抗性的QTL定位®v6.0 [30.],使用在五个环境中收集的表型数据。八个与花生腹板斑块相关的八杆,位于八个不同的LG中,在至少两个环境中确认,从2.8%到15.1%的表型变化中解释,显示从1.32到7.45的睡眠价值(表2).的QTL(qwbra04.和qWBRA14)位于LG04和LG14上,在本研究中所有五种测试环境中具有显着与抵抗力相关(表2, 图。3.和图。4),解释了10%以上的表型变异,表明它们可能是稳定表达的主要qtl。除了qWBRA13和qwbra05.,这是在四个和两个环境分别检测其它四个的QTLqwbra03.,qWBRA16,qWBRA17,qWBRA19在三个环境中检测到(表2).绝对值显示在由加性效应从0.11范围至8.29,并且是用于在LG4,LG5,LG13,LG14,LG19(指示有利的等位基因源自抗性亲本Zheng8903),和正对LG3的QTL的QTL负,LG16和LG17(指示有利等位基因源自易感亲Yuhua4)。
为了鉴定花生腹板斑性抗性的候选基因,与QTL相关的基因组区域中的编码序列qwbra04.和qWBRA14的预测功能检查,根据arachis hypogaea.简历。Tifrunner参考基因组注释数据库[4].在抗病抵抗力的推定作用中鉴定了总共40个候选基因(表3.).详细地,区域qwbra04.的连锁区间为1.10 cM,物理区间为86kb,包含4个核苷酸结合位点-富亮氨酸重复序列(NBS-LRR)基因。的基因Arahy。Q7VTCQ和Arahy.9yx67z.含有卷曲线圈(CC)结构域和基因Arahy。SK6LYR和Arahy.1RZ0PJ含有toll -白介素受体(TIR)结构域(表3.).的区域qWBRA14跨越0.48厘米,物理上对应于〜2.8MB,含有编码抗病蛋白的37个基因。其中,19个基因含有TIR结构域,一个基因含有CC结构域,而剩余的17个基因编码其他蛋白质,具有疾病抵抗力的推定作用,例如aBzip.转录因子和一个WRKY转录因子的蛋白质。
为了进一步验证候选基因,分析位于候选基因中的SNP的突变类型(表4).有位于7个基因双亲,其中八个是在非编码DNA区,其中两个是同义突变之间24个多态单核苷酸多态性,并在A03 A04染色体,剩余14个SNP,和A14是非的代名词。的14个非同义SNP被分布在四个编码序列区域,其中4个是在的区域Arahy.lfe0tk.,一个in.Arahy。Q7VTCQ,三个Arahy.1RZ0PJ和六个Arahy.MR7539。4个基因均为NBS-LRR,除NBS-LRR外,其余3个基因均含有TIR结构域Arahy。Q7VTCQ包含的CC域。
KASP设计和验证
为了实现纤维网状抗性花生品种的分子标记辅助育种,两个父母之间的24个多态性SNP(表4),建立KASP(竞争性等位基因特异性PCR)标记,并对该群体的47个品系(23个抗性品系和24个易感品系)进行验证。结果表明,A04染色体上有3个SNPs位于Arahy。Q7VTCQ可能与花生抗网斑有关(补充表)S3.).大多数所测试的线路(42出来的47线)显示了他们的表型与基因型之间的一致性在不同的测试条件。两个易感基因型(WB6235和WB6360),其表现出感染在一定条件下的水平低,可能是由接种(补充表逃脱S3.).1个易感系(WB6204)和2个抗性系(WB6248和WB6367)的表型不一致(补充表)S3.).
讨论
基于高通量全基因组测序和滑动窗口策略,构建了包含3634个bin标记的高密度遗传连锁图谱。除LG3和LG13之间、LG6和LG16之间有一些易位外,其余标记在遗传图谱上的排列顺序与花生基因组装配的物理顺序基本一致。LG3上共有277个bin标记,后70个标记的物理位置在A13染色体上,LG13的后47个标记的物理位置在A03染色体上。在LG6中插入了43个来自A16染色体的bin标记。出现这种看似易位的原因可能是参考基因组中存在一些装配错误,这在Peanutbase (https://www.peanutbase.org/data/public/arachis_hypogaea/tifrunner.gnm1.kyv3/)[31.].LG1,LG4,LG5,LG10,LG12和LG18上的标记的遗传订单与它们的物理订单完全一致,并且其他LG的一些标记与相邻标记反转。
总共鉴定出在八种染色体上分布的八种新型QTL用于花生腹板斑抗性,在至少两项实验试验中确认。其中三个是主要的QTL,因为它们与表型方差相关(PVE)> 10%相关,而另外五个是张等人的预测结果。(2011)。除了qwbra03.,仅在天然条件下检测到,在天然和人工接种条件下检测到其他七个QTL。两个主要的QTLqwbra04.和qWBRA14在本研究中的所有五种测试环境中稳定地检测到,因此可能具有抗性花生品种的巨大潜力。
在本研究中,我们在8个qtl的靶区中鉴定了55个候选抗病基因(table3.和5),其中40个与2个主效稳定qtl连锁qwbra04.和qWBRA14(表3.),而另一个15与其他六个QTL间隔相关联(表5).通过的区间所涵盖的40个候选基因qwbra04.和qWBRA14包括21个TIR-NBS-LRR和3个CC-NBS-LRR,这两个是众所周知的R基因类型[32.].此外,还有两种候选基因编码的LRR和NB-弧(与APAF-1共享的核苷酸结合结构域,各种R-蛋白和CED-4)结构域,其也编码了抗性基因[33.].在剩余的15个基因中,其中七个在抗性信号传导的下游路径中起作用,其他八个编码蛋白质含有抗病抗抗性相关结构域,但不能分配到众所周知的R-基因类型(表3.).
鉴定了总共26个NBS-LRR基因与本研究中的花生腹板斑抗性有关。NBS-LRR是R基因的最大类别[34.]并已在基因组水平中鉴定arachis.[35.].结果表明,nbs - lrr参与了对晚叶斑病、番茄斑点枯萎病毒和青枯病的响应A. Duranensis.,答:ipaensis,A. hypogaea.[36.].可以得出结论,NBS-LRR也参与了对花生腹板斑的抵抗力,但需要进一步研究抗病性过程中的调节机制。
3个验证的KASP标记的结果表明,该基因具有较高的表达能力Arahy。Q7VTCQ(CC-NBS-LRR)可能是一个很大的可能性耐药基因花生网斑病之一。表型和三种测试线基因型之间的不一致的原因可能是三个KASP标志物没有完全与花生网斑病抗性相关。因此,进一步的研究,将需要在设计辅助育种(MAS)的分子标记要采用更接近连锁的标记。
结论
在这项研究中,检测到八个用于花生腹板斑块的QTL和两个主要QTLqwbra04.和qwbra04.与编码NBS-LRR或与抗病抗性有关的其他蛋白质的40个候选基因有关,这可能揭示了了解Web斑块抗性并促进耐花生品种的发展。
方法
植物材料
一个群体,由212 f组成12.本研究以品系郑8903与品种玉花4号亲本杂交组合衍生的品系为材料。亲本为郑8903,系谱为‘79-266 // 71-31 /Chico’,是一个对花生网斑病表现出高度抗性的选育系[37.].雄性父母Yuhua4是1991年由河南农业科学院发布的品种,我们从我们自己的库存中获得了种子[38.],显示易受网页污迹影响的程度[37.].所有包括rils及其父母在内的所有植物材料都在相应的作者的实验室中开发并保存。
实验试验和表型
从5月到9月,在河南农业科学院研究站进行了评估对花生网爆炸的答复的所有实验试验。在自然感染后,上述植物材料在2007年,2008年和2012年进行的现场试验中进行了评估,并在2018年进行的现场接种,在人工中接种,在田间和室内试验之后。对于田野踪迹,每种基因型的20种种子播种3 m long and 0.4 m wide plots, according to a complete random block design with two replicates for natural infection and three replicates for artificial inoculation. For natural infection in the year 2007, 2008 and 2012, disease evaluation was carried out before harvest according to the 0–4 scale described in Yuan et al. (2004) [39.].对于2018年的田间接种,用1.6×10的接种喷洒图- 3. g/ml at the flowering stage [40],接种后20 d,根据Yu(2011)的0-9量表进行疾病评价[41.].对于室内接种,每条线接种五种植物,在6个叶片阶段有两种复制。接种浓度为2×106Conidia / ml和张(2019)描述的Conidia悬浮液的制备[37.] 被跟从。接种后两周,收集在主茎处的12个接种叶片,叶面积计(Wanshen La-S)扫描各自的病变面积。花生网状斑点的室内分类标准如下:对于尺度0,没有检测到病变;缩放1,0 <损伤区域<6%;2,6%≤病变面积<25%;3,25%≤病变面积<50%;4,50%≤病变面积<75%;规模5,75%≤病变区域。
测序和基因分型
使用植物基因组DNA提取试剂盒(天根)从幼叶组织中提取父母和212个rils的基因组DNA,并通过超声处理随机剪切。通过电泳回收具有300bp的DNA片段。在用适应剂连接DNA片段后,使用Illumina Hi-SEQ平台与读取长度为150bp的配对端进行配对末端。
修剪适配器和低质量读取后,使用清洁数据用于对准参考基因组,允许SNP鉴定和基因分型。详细地,组装arachis hypogaea.简历。Tifrunner被用作参考基因组[4]。软件BWA-0.7.10中的ALN命令用于将清洁数据与参考基因组对齐,并且独特的读取用于软件GATK3.3.0的后续SNP变异检测。将获得的两个父母的SNP组使用过滤使用缺失的值,杂小尿,深度和GQ值,并且纯合和多晶型基因座用于RIL群体。本研究中获得的二进制对齐映射(BAM)文件已在BioProject ID下提交到NCBI的Bioproject数据库:Prjna602098。
联系地图结构和QTL映射
由于单个SNP基因座的呼叫的准确性很低,因此由于针对RIL测序所选择的低覆盖率,施加滑动窗法以评估一组连续的SNP进行基因分型[42.].将212个rils的所有染色体的基因型对齐,并将其与100kb间隔的最小值相比,并且在整个RIL群体中具有相同基因型的相邻的100kb间隔被认为是单一重组箱[42.].从垃圾箱标记,使用JoinMap V5.0构建联动组[29.]选择从2到10中的LOD得分,以识别组和回归算法,以在每个LG内执行排序。使用R包LinkageMapView绘制最终的链接图。使用MAPQTL V6.0执行QTL映射[30.],通过选择多个QTL作图(MQM),以在至少一个环境检测潜在的QTL具有2.5的LOD阈值。
缩写
- QTLS:
-
定量特质基因座
- 瑞来斯:
-
重组自交系
- LG:
-
联动组
- NBS-LRR:
-
核苷酸结合位点富含亮氨酸重复
- 子:
-
开放阅读框
- SNP:
-
单核苷酸多态性
- CC:
-
卷绕线圈
- TIR:
-
Toll-Interleukin受体
- NB-ARC:
-
与APAF-1、各种r蛋白和CED-4共享核苷酸结合结构域
- KASP:
-
Kompetitive等位基因特异性PCR
- BAM:
-
二进制对齐映射
- PVE:
-
表型方差解释了
qWBR
Qtl抗网斑
- 马斯:
-
标记辅助繁殖
参考
- 1。
Bertioli DJ,Cannon Sb,Froenicke L,Huang G,Farmer Ad,Cannon Ek,Liu X,Gao D,Clevenger J,Dash S,Ren L,Moretzsohn Mc,Shirasawa K,Huang W,Vidigal B,Abernathy B,Chu Y,Niederhuth CE, Umale P, Araújo AC, Kozik A, Burrow MD, Varshney RK, Wang X, Zhang X, Barkley N, Guimarães PM, Isobe S, Guo B, Liao B, Stalker HT, Schmitz RJ, Scheffler BE, Leal-Bertioli SC, Xun X, Jackson SA, Michelmore R, Ozias-Akins P. The genome sequences of落花生duranensis和arachis ipaensis.,栽培花生的二倍体祖先。NAT Genet。2016; 48(4):438-46。
- 2。
陈XP,鲁Q,刘H,张JN,香港YB,兰HF,李HF,王JP,刘HY,李SX,潘迪MK,张ZK,周庚寅,喻JG,张广钦,袁JQ,李XY文SJ,孟FB,俞SL,王XY,Siddiquw KHM,刘ZJ,帕特森AH,Varshney RK,梁XQ。种植花生的排序,arachis hypogaea.,产生对基因组进化和石油改进的见解。莫尔植物。2019; 12(7):920-34。
- 3。
庄文军,陈辉,杨明,等。栽培花生的基因组为了解豆科植物的核型、多倍体进化和作物驯化提供了依据。Nat麝猫。2019;51:865 - 76。
- 4.
Bertioli DJ, Jenkins J, Clevenger J, Dudchenko O, Gao D, Seijo G, SCM L-B, Ren L, Farmer AD, Pandey MK, Samoluk SS, Abernathy B, Agarwal G, Ballén-Taborda C, Cameron C, Campbell J, Chavarro C, Chitikineni A, Chu Y, Dash S, MEl B, Guo B, Huang W, Kim KD, Korani W, Lanciano S, Lui CG, Mirouze M, Moretzsohn MC, Pham M, Shin JH, Shirasawa K, Sinharoy S, Sreedasyam A, Weeks NT, Zhang X, Zheng Z, Sun Z, Froenicke L, Aiden EL, Michelmore R, Varshney RK, Holbrook CC, EKS C, Scheffler BE, Grimwood J, Ozias-Akins P, Cannon SB, Jackson SA, Schmutz J. The genome sequence of segmental allotetraploid peanutarachis hypogaea..NAT Genet。2019; 51(5):877-84。
- 5。
Alcorn JL,菲特语e,麦卡锡GJP。花生净污染造成的兼奥米洛西亚·斯拉契尼科拉(Chochryakov)梳子。NOV TRASS BR MYCOR SOC。1976; 66:351-5。
- 6。
王志强,王志强,王志强。花生网斑:ⅱ型病原症状及寄主范围。花生科学。1986;13(1):。
- 7。
Marasas WFO,Pauer Gd,Boerema GH。粉底的严重叶片斑疾病(arachis hypogaea.在非洲南部由Phoma arachidicola.sp。11月植物。1974; 6:195-202。
- 8。
泰伯RA,Petti RE,Philley GL。花生网斑病:一个,文化特征和因果真菌的身份。花生科学。1984; 11(2):109-14。
- 9。
Pettit Re,Taber Ra,Harrison al。ascochyta花生的网状斑点。植物病理学。1973; 63:447。
- 10。
徐MX,Shi Ym。介绍了中国花生网爆炸的真菌。花生SCI技术。1990; 1:19-20(中文)。
- 11.
徐MX,Shi Ym,Xu XJ。花生网状斑点因果真菌的文化特征及其特征及其特征。Acta phytopathologica sinica。1992年; 22:270(中文)。
- 12.
李杰,李捷,娇F,王YC。研究山西省花生网爆炸的真菌。花生SCI技术。1991; 1:1-6(中文)。
- 13。
王征镒,王淑珍,李海林,袁海霞。河南省花生网斑病真菌的研究。河南农业科学。1993;7:23-5。
- 14。
致敬FPC,Chapman J,Young BW。腰果白学Phoma网页底色和Cercospora.在西班牙地生的叶子斑点。植物植物。1977; 9:63-4。
- 15.
花生网斑病初报Phoma arachidicola.在斯里兰卡的干燥区。植物DIS。2007; 81:832。
- 16。
张X,徐ml,吴jx,东WB,陈德克,王L,Chi Yc。草稿基因组序列Phomaarachidicola wb2导致花生网斑点在中国。Curr Microbiol。2019; 76:200-6。
- 17。
史密斯od,史密斯dh,辛普森ce。网斑抗性arachis hypogaea.L.花生SCI。1979; 6(2):99-101。
- 18。
张XY。花生中的映射关系到产量,品质和抗病性的主要特征及其QTL的继承(arachis hypogaea.l .)。杭州:浙江大学;2011.(博士学位论文。
- 19。
Novakazi F,Afanasenko O,Anisimova A,Platz GJ,Snowdon R,Kovaleva O,Zubkovich A,Ordon F.植物遗传分析对净污染疾病净形态的抵抗抗净形态的全球大麦收集(Pyrenophora Teres F.TERES)。Al Appl Genet。2019; 132:2633-50。
- 20.
Adhikari A,Steffenson BJ,Smith MJ,Dill-Macky R. Gearomewide Association在埃塞俄比亚和厄立特里亚尔大麦收藏中的幼苗净污染抗污染抗性。农作物科学。2019; 59:1625-38。
- 21。
Amezrou R,Verma的RPS,晁S,Brueggeman RS,Belqadi L,Arbaoui男,拉赫曼S,在春大麦幼苗收集和成株阶段网斑病抗性的净形式的Gyawali S.全基因组关联分析。mol品种。2018; 38:1-14。
- 22。
理查兹JK,弗里森TL,Brueggeman RS。利用多样大麦系关联映射揭示了净形式网斑病苗电阻/易感基因位点。Al Appl Genet。2017; 130:915-27。
- 23。
Wondeberger R,Ficke A,Lillemo M.在北欧大麦种质集团集中鉴定与净净爆破率的定量特质基因座。Al Appl Genet。2017; 130:2025-43。
- 24。
利用全基因组关联作图法鉴定当代美国大麦种质抗网形网斑病的数量性状位点。Theor Appl Genet. 2020; 133:1019-37。
- 25。
王L,周X,Ren X,Huang L,Luo H,Chen Y,Chen W,Liu N,Liao B,Lei Y,Yan L,Shen J,Jiang H.染色体细菌枯萎性抗性的主要和稳定QTL使用高密度SNP族遗传联系地图鉴定B02,在栽培的花生yuanza 9102衍生人群中。前群体。2018; 9:652。
- 26。
韩元,元M,克利维尔jp,李c,海根A,张X,陈c,他G。基于SNP的联系地图揭示了QTLS抵抗早期和晚期叶斑病患者(arachis hypogaea.l .)。前植物SCI。2018; 9:1012。
- 27。
王Z,淮维,张Z,程克,康y,万立,延l,江鹤,雷y,廖b。基于具体长度扩增片段测序的高密度遗传图谱及其在定量中的应用栽培花生产量相关性状的特质基因座分析。前植物SCI。2018; 9:827。
- 28。
李浩,张丽,王杰克。在定量特质轨迹映射中的常见问题分析和答案。acta。2010; 36(6):918-31。
- 29。
范Ooijen JW。JoinMap®5,用于计算实验群体遗传连锁图的软件,Kyazma BV。瓦赫宁根,https://www.kyazma..NL /指数。PHP / JoinMap /;2006年。
- 30.
范Ooijen JW。MAPQTL®6,在二倍体物种的实验群体中定量特征基因座的映射软件。瓦赫宁根,https://www.kyazma.nl/index.php/mapqtl/:Kyazma BV;2009年。
- 31.
Dash S, Cannon EKS, Kalberer SR, Farmer AD, Cannon SB. PeanutBase和花生的其他生物信息资源(第8章)。见:Stalker HT, Wilson RF,编辑。花生的遗传、加工和利用。圣地亚哥:爱思唯尔公司;2016.241 - 52页。
- 32.
Tarr Dek,Alexander HM。蒂德 - NBS-LRR基因在单像中罕见:来自不同单码下令的证据。BMC RES笔记。2009; 2:197。
- 33。
Chandra S,Kazmi Az,Ahmed Z,Roychowdhury G,Kumari V,Kumar M,Mukhopadhyay K.小麦内Nb弧抗性基因的基因组鉴定和表征(Triticum Aestivum.L.)和叶锈病感染期间它们的表达。植物细胞报道2017; 36(7):1097-112。
- 34。
Jones JD, Dangl JL。植物的免疫系统。大自然。2006;444(7117):323 - 9。
- 35。
宋辉,王鹏,李超,韩松,赵超,夏辉,毕莹,郭斌,张欣,王欣。NBS-LRR基因及其对黄曲霉的响应比较分析arachis..《公共科学图书馆•综合》。2017;12 (2):e0171181。
- 36。
钱歌H,郭ZL,胡锦涛XH, L,苗族跳频,陈张XJ, j .进化平衡远程雷达领域损失和年轻NBS-LRR生产管理抗病基因arachis hypogaea.简历。Tifrunner BMC基因组学。2019; 20:844。
- 37。
张兰研究花生生理和生化抗性研究(arachis hypogaea.L.)对抗Web Blotch。郑州:郑州大学;2019.(硕士论文,中文)。
- 38。
马吉。一种新的花生品种玉华4,具有早期成熟和高产。J河南农业科学。1992; 2:24(中文)。
- 39。
元HX,Sun BJ,Li HL,Xing XP,唐FS,张XY。花生品种抗叶斑鉴定(线)。J河南农业科学。2004; 12:35-8(中文用英文摘要)。
- 40。
王泽,李诗,张XY,高米,崔XW,王n,桑股。一种识别花生纤维网斑的抗性的方法。中国专利。2017; ZL201510708861.x。(用中文(表达)。
- 41。
yu sl。中国花生遗传学和育种。上海:上海科技出版社;2011.(中文)。
- 42.
黄X,冯Q,钱Q,赵Q,王L,王A,关伙,范多,翁Q,黄T,东彤,桑特,汉B.全基因组重新划分的高通量基因分型。Genome Res。2009; 19(6):1068-76。
致谢
作者谨此感谢Stefano Pavan博士(Bari Aldo Moro,意大利大学)讨论QTLS分析和编辑本手稿草案的英文文本。
资金
这项工作得到了中国农业研究体系(Cars-13),河南省农业研究系统,中国(S2012-5),河南农业科学院(2019 Q02)和河南学院的优秀青年学者作者:王莹,农业科学学报(2005年第05号)。此外,我们感谢国家自然科学基金(No.31871663)的部分资金(第31871633号),以及中国河南(第182102110137号)和河南农业科学学院自主创新专项研发项目(2020ZC13)资金机构在研究和收集,分析和解释方面没有作用在数据和撰写稿件的设计中。
作者信息
从属关系
贡献
HL和ZS进行了实验室和现场实验,并写了稿件。LQ,FQ,PD,JX和ZZ1进行了基因型分析和QTLS分析。ZW,SL和MG提供了野外接种的帮助。LZ和YC提供室内接种的帮助。SH,BH,ZZ2和WD提供帮助设计实验并修改纸张的第一稿。XZ构思和设计了实验,促进了项目,并协助稿件准备。所有作者阅读并认可的终稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
排序和比对结果的汇总信息。
附加文件2:图S1。
物理重组的重组自交系212上映射20条染色体。蓝色:抗性亲本Zheng8903的基因型;红色:病亲Yuhua4的基因型;黄:杂合子;重组自交系从顶部布置为底部,染色体从左至右排序。
附加文件3:表S2。
在本研究中获得的花生遗传联系地图中箱的基因型和位置。字母A表明父母线Zheng8903的基因型,字母B表示父母线Yuhua4的基因型。
附加文件4:表S3。
47条线的表型和凯斯族基因分型结果。*代表缺少数据。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
刘华,孙志强,张旭东。等等。花生网斑抗性QTL定位的高通量全基因组测序研究BMC植物BIOL.20.249(2020)。https://doi.org/10.1186/s12870-020-02455-8
收到了:
接受:
发表:
关键词
- QTL作图
- 花生
- 网斑抗性
- 重新排序