抽象的
背景
蔗糖非发酵相关蛋白激酶(SnRK)通过激活蛋白磷酸化途径,在应对生物和非生物胁迫中发挥重要作用。然而,很少的信息SNRK.基因可用芸苔属植物显著,一个重要的石油作物之一。最近,该参考基因组的释放序列甘蓝型提供了进行基因组识别和表征的良好机会BnSnRK油菜的基因家族。
结果
完全114SNRK.在19个基因组中鉴定了分布在19染色体上的基因甘蓝型并根据系统发育分析和域类型分为三个亚属植物。根据基因结构和基序成分分析,BnSnRK序列在三个亚属中表现出明显的分歧。基因复制和草案基因组之间的复制和同时性拟南芥也进行了分析,以提供对BnSnRK家庭基因。C是-元素分析显示BnSnRKs可能反应不同的环境压力。而且,表达模式BnSnRKs在各种组织中,在不同的非生物胁迫下是不同的差异。此外,单核苷酸多态性(SNP)分布分析表明函数差距BnSnRK不同基因型油菜的家族基因。
结论
我们检测了114的基因组结构、进化特征、表达模式和SNP分布BnSnRKs.结果为功能表征提供了有价值的信息BnSnRK未来研究中的基因。
背景
植物开发各种分子防御机制以应对非生物胁迫,包括盐度,干旱和冷应力。基因表达调节和蛋白质改性是植物处理这些压力的两个重要方式[1].蛋白激酶介导的磷酸化和去磷酸化方法在蛋白质改性中起关键作用[2].在报告的蛋白激酶基因中,蔗糖非发酵1(SNF1) - 相关蛋白激酶(SnRKs)参与不同的生理过程[3.].
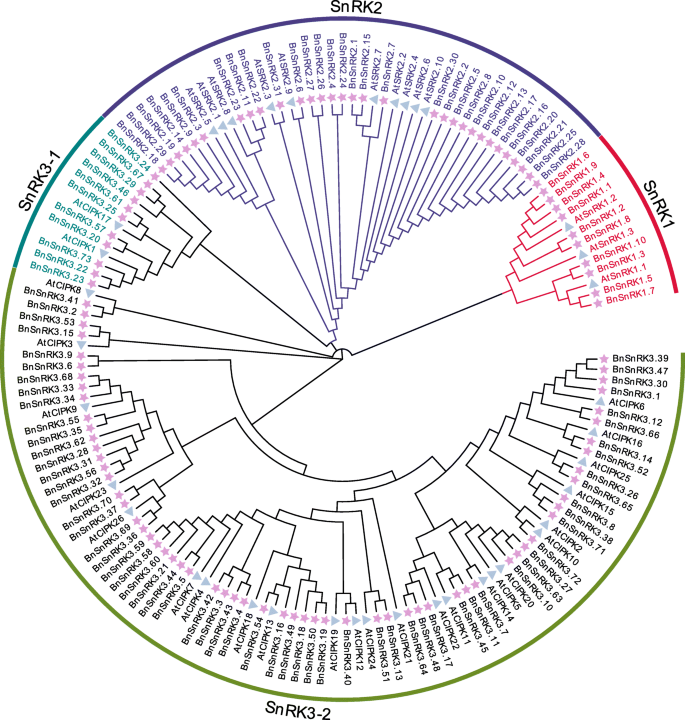
在植物中,SnRKs可以分为三个亚属:SnRK1,SnRK2和SnRK3基于它们的序列相似性和基因结构[3.,4].详细,SnRK1亚家族含有高度保守的N-末端蛋白激酶(Pkinase)结构域,其是同源基因SNF1在酵母和安瓿在哺乳动物5].另一个亚属SnRK2/3在植物中是独一无二的,两者都比植物更多样化SnRK1亚科的成员。的成员SnRK2Harbors保守的Pkinase结构域和C末端变量调整保守域[6].此外,SnRK3被称为CIPK.(cbl相互作用蛋白激酶),也包含保守的蛋白激酶n端结构域和NAF结构域,c端PPI结构域[7,8].
的SnRK1家族基因参与植物细胞对饥饿和代谢应激的反应。SnRK1激酶是异络络合物的催化亚基,其与另外两个亚基相互作用[9].在茄属植物tuberosum, SnRK1被证明参与诱导蔗糖合成酶表达,并在碳水化合物代谢调节中发挥了重要作用[10.].此外,低能量压力(如黑暗和缺氧)可能触发SNRK1α.核易位和进一步诱导SnRK1为植物生长而更新细胞能量的目标基因[11.].此外,相当大的证据表明SnRK1基因是各种信号通路网络中的集线器,包括细胞周期调节,病原体反应和分生发育[12.].
另一方面,SnRK2基因在植物应对非生物胁迫,尤其是渗透胁迫和盐胁迫中发挥着重要作用。例如,SnRK2.10磷酸化几种靶基因,包括脱氢ERD10和ERD14处理渗透压力拟南芥蒂利亚纳[13.].SnRK2.1实际调节耐盐性尼科尼亚塔哈瓦姆[14.,15.].在A. Thaliana.,SnRK2亚家族基因可以分为三组:第1组含有无关的激酶,第2组包括响应干旱胁迫的基因,并通过ABA强烈刺激第3组激酶[6,16.].目前对ABA依赖性组3激酶的研究是最广泛的。例如,atsnrk2..6(OST1),其中之一SNRK2家族基因,在气孔保卫细胞中ABA信号通路中起核心作用OST1可以通过E3-泛素连接酶调节蛋白质稳定性Hos15.减少ABA信号灵敏度拟南芥[17.,18.].
SnRK3激酶被称为CIPKs(CBL(钙传感器钙素B样蛋白) - 交互式蛋白激酶),对植物中的各种应力进行抗痛作用[3.,19.].例如,SOS.(盐过度敏感)系统是第一个发现CBL-CIPK.途径A.thaliana.详细地,SOS3感测了通过SOS 3感测量的钙信号(ATCBL4.)在细胞膜上,然后SOS3结合使用SOS2.(ATCIPK24)形成磷酸化合物SOS1(NA+/H+altiporter)去除多余的na+在根细胞外[20.,21.].除了,MdCIPK13和MdCIPK22通过靶向蔗糖转运体增强耐盐耐旱性MDSUT2.2.用于苹果中的磷酸化[22.,23.].过度表达BnCBL1-BnCIPK6在甘蓝型可以增强高盐度耐受性和低钾耐受性[24.].总之,提高证据强调了重要性SnRKs在营养利用率和应力反应中的功能,最后研究人员可以使用这些基因通过遗传操作来改善植物对应力的抵抗力。
芸苔属植物显著是世界上一个重要的石油作物。最近,Darmor的基因组 -BZH.(冬季生态型),中顺11和NY7(森冬季生态型)成功测序并组装[25.,26.,27.];但是,系统分析BnSnRK基因家族没有得到很好的报道。在这项研究中,114SNRK.基因成员在甘蓝型基因组。我们系统地分析了它们的系统发育关系,蛋白质基序,基因结构,染色体分布和CIS-启动子区域的元素。此外,表达式概况BnSnRKs在不同的组织中以及在非生物胁迫下测定。此外,每一个的SNPsBnSnRK在全球收集中系统地识别基因,具有300个核心种质种质。这些结果将提供有用的信息,以进一步调查分子机制BnSnRK非生物抗逆性基因及其分子育种甘蓝型.
结果
鉴定和系统发育分析SNRK.基因甘蓝型
共有114个具有丝氨酸/苏氨酸激酶结构域的蛋白被鉴定为SnRK家族成员甘蓝型基因组(表S2).选择每种蛋白质的最长氨基酸序列以进一步分析。表格中列出了基因名称,ID,染色体位置,氨基酸数,分子量(MW),等电点(PI)和域的信息S1和S2.氨基酸长度为114BnSnRKs蛋白质从190〜1241的范围内,相应地,新的新型重量范围为22.0至140.6kDa。表中列出了每个基因的编码区和序列S3.
分析进化关系SNRK.基因甘蓝型和A.thaliana,利用氨基酸全长序列构建无根系统发育树SnRKs.完全,39SnRKs从A.thaliana114.SnRKs从甘蓝型鉴定并在研究中使用(图。1).据报道,39atsnrks.可以聚集成三组[3.].本研究基于系统发育分析,114BnSnRKs也分为三组。细节,10个蛋白质BnSnRK1Pkinase (Pfam的PF00069)、UBA (PF00627)和KA1 (PF02149)结构域的亚家族除外BNSNRK1.3,而31种蛋白质含有pkinase域BnSnRK2具有高相似性的亚家族atsnrk2.亚家族和73种蛋白质,具有梭菌和NAF(PF03822)结构域SnRK3分别是亚家族(图。1).
基因结构和蛋白质基序分析BnSnRK基因
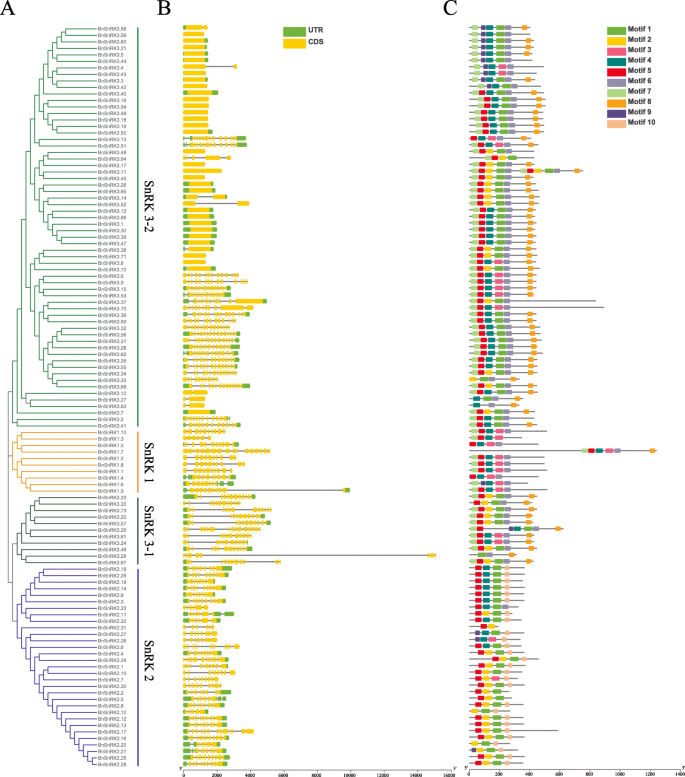
完成Web服务器GSD(基因结构显示服务器)分析以确定基因结构BnSnRKs。如图1所示。2,其中一到十五个外显子BnSnRK基因。的BnSnRK1亚家族基因具有超过6个外显子,BN为6至13个外显子SnRK2亚科的基因。虽然它是各种各样的BnSnRK3亚家族,范围从1到15个外显子。此外,还有两个亚组BnSnRK3家庭。基因SnRK3亚群1的基因包含超过8个外显子,而亚群2的基因包含少于4个外显子。
系统发育关系,保守蛋白质基序的结构和基因结构SNRK.基因甘蓝型.一个104的系统发育树BnSnRK蛋白质。通过104的全长氨基酸序列用MEGA7构建大邻近的系统发育树BnSnRK,自举试验重复设置为1000次。b外显子/内含子组织BnSnRK基因。黄色框代表外显子和带有相同长度的黑线代表内含子。上游/下游区域BnSnRK基因在绿色盒子中表示。外显子的长度可以通过底部的刻度推断出来。c主题组成BnSnRK蛋白质。数字1-10的主题被陈列在不同颜色的盒子里。每个motif的序列信息如表所示S4.可以使用底部的刻度估计蛋白质的长度
所有的结构图BnSnRK使用MEME(MOTIF ELITITITITIONT)基序分析结果构建蛋白质(图。2C)。保守基序的序列和长度信息显示在补充表中S4.在这项研究中,我们发现BnSnRK基因保留含有含有基序1,2,3,4,5的保守的氏藻酶结构域(图。2C)。而且,BnSnRK同一亚家族内的基因分享类似的主题组合物BnSnRK不同亚家族间的基因在基序组成上存在明显差异。在细节,BnSnRK1亚家族基因含有图案3,4,5,6,7;BnSnRK2基因有motif 1,2,5,6,10或motif 1,4,5,6,10;BnSnRK3-1基因含有基序1,2,5,6,7,8或基序1,4,5,6,7,8;BnSnRK3-2基因具有基序1,5,6,7,8或基序1,6,7,8,9(图。2C)。总之,保守的基序组合物和类似的基因结构SNRK.同一亚家族内的基因,通过系统发育分析强烈支持亚家族分类的可靠性。
染色体分布、基因组共同和基因复制BnSnRK基因
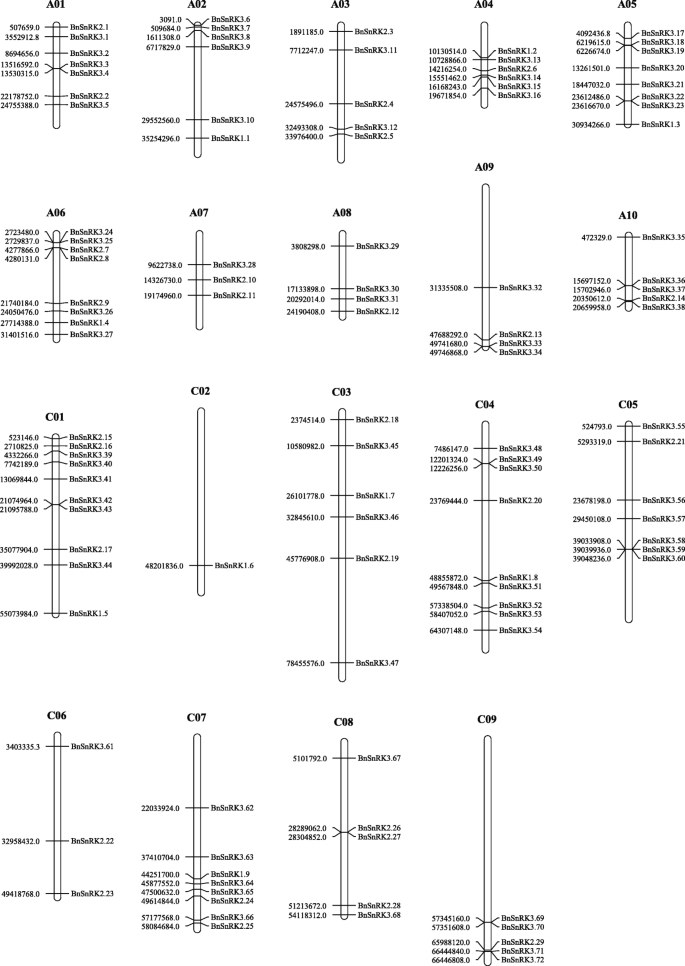
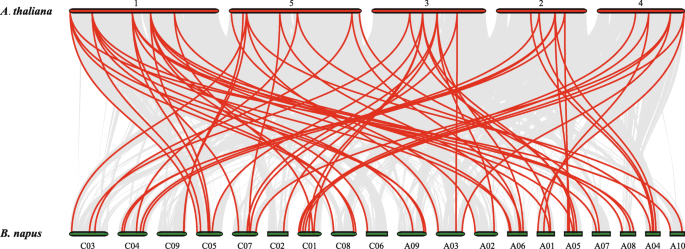
染色体分析表明110BnSnRK基因分布在19个染色体上,除了4基因(BnSnRK1.10, BnSnRK2.30, BnSnRK2.31, BnSnRK3.73)无法映射到油菜基因组的染色体(图。3.).其中,56BnSnRK基因定位于AA亚基因组,包括4个BnSnRK1基因,14BnSnRK2基因,38BnSnRK3基因;虽然54个基因位于CC亚基因中,但含有5个BnSnRK1基因,15BnSnRK2基因,34BnSnRK3基因。另外,一些BnSnRK2和BnSnRK3亚家族基因在不同的染色体上形成簇状,如BNSNRK3.22和3.23(图。3.).这些结果表明BnSnRK基因被随机分布在AA或CC亚组的染色体中。
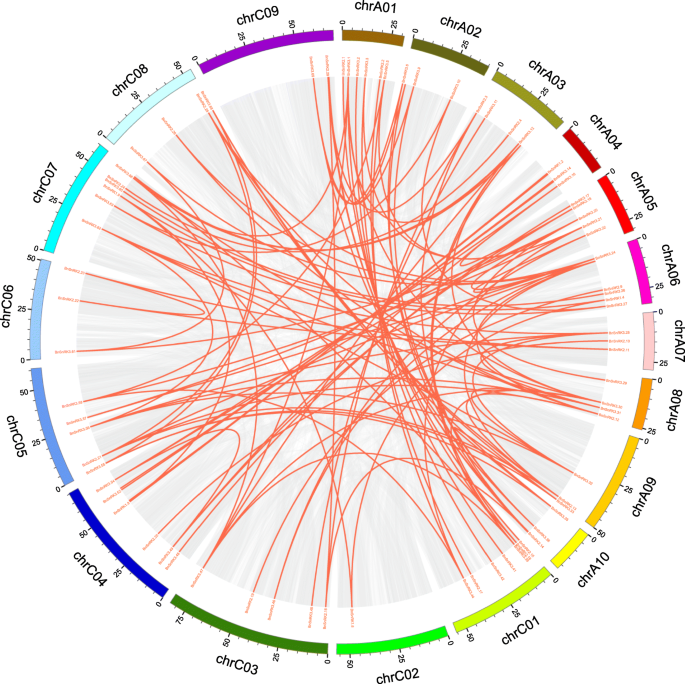
使用BLAST和MCSCANX方法,识别了106个分段重复事件(图。4和表格S5).在这些事件中,在不同的染色体之间发生了104个事件,而在同一染色体中只检测到2个重复事件。结果表明这一部分BnSnRK基因可能是由基因复制产生的,片段复制事件在基因扩增中起着重要作用SNRK.基因的甘蓝型基因组。我们还分析了串联复制事件的发生。在这里,25紧密相连BnSnRK基因(BnSnRK2.7 / 2.8,BnSnRK3.3/3.4,BnSnRK3.18/3.19,BnSnRK3.22 / 3.23,BnSnRK3.24 / 3.25,BnSnRK3.33/3.34,BnSnRK3.36/3.37,BnSnRK3.42 / 3.43,BnSnRK3.49/3.50,BnSnRK3.58/3.59/3.60,BnSnRK3.69/3.70,BnSnRK3发现,发现距离小于200千克。然而,这些基因对的身份小于70%,表明它们不是串联复制事件。
Futhermore,SyntenySNRK.基因之间的对甘蓝型基因组和A.thaliana基因组。结果表明,65SNRK.基因甘蓝型表现出同情关系atsnrk.基因(图。5和表格S6),表明这些基因可能导致进化BnSnRK基因家庭。测试演义的制约因素,ks.值,K a值,Ka / Ks平行和同源的比值和发散时间SNRK.计算家族基因(表格S5和S6).大多数分段重复BnSnRK基因对有Ka / Ks比率小于1,平均值BnSnRK3基因对(Ka / Ks= 0.30)低于BnSnRK1(Ka / Ks = 0.41) andBnSnRK2(Ka / Ks = 0.75). In addition, theKa / Ks最直观的比例SNRK.基因对小于1,平均值SnRK2基因对(Ka / Ks= 0.09)明显低于SnRK1(Ka / Ks = 0.75) andSnRK3(Ka / Ks= 0.23)。这些结果表明BnSnRK基因家族可能在进化期间遭受较强的纯净选择性压力。
与压力有关独联体- 在启动子中的元素BnSnRK基因
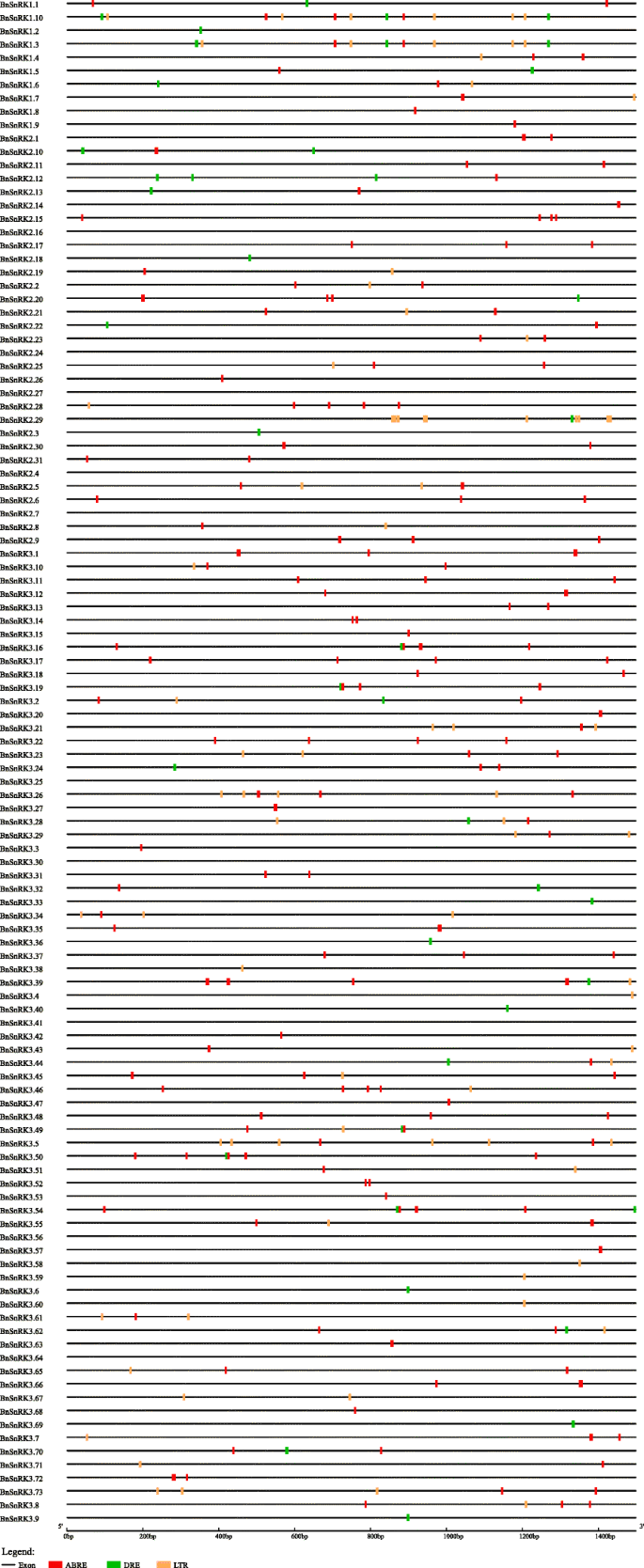
了解潜在的功能和监管机制BnSnRK基因,CIS-通过使用PlantCare分析元素(从ATG上游的1.5 kB)。完全104公里的114BnSnRKs被确定为CIS-包括DRE,ABRE和LTRE的元素,涉及脱水反应,ABA响应和低温响应(图。6、表S2).详细,88BnSnRK77.19%的人有ABRE独联体元素,33岁BnSnRK基因(28.95%)携带DRE独联体- 和44BnSnRK基因(38.60%)所有的LTRE独联体-Elements。还发现大多数基因有多种独联体-element类型。此外,数量独联体-ElementsBnSnRK1(1.80)家庭高于BnSnRK2(1.17)和BnSnRK3(1.21)家庭(表S2).总之,CIS-元素分析表明最多BnSnRK基因可以对不同的环境胁迫作出响应,不同的亚家族基因可能受到不同的调控。
表达概况BnSnRKs在不同的组织中
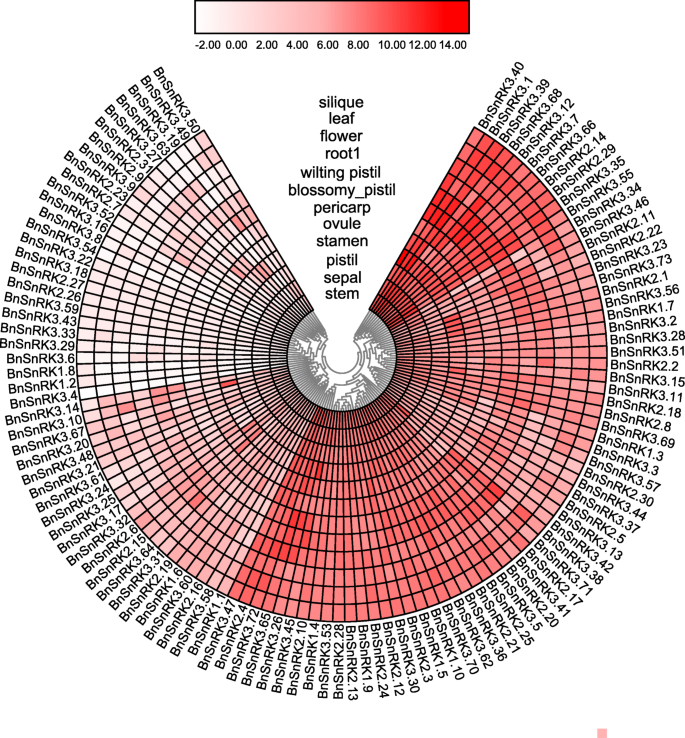
114的表达概况BnSnRK比较了油菜品种ZS11的12个组织间的基因差异。7和表格S7).的表达模式BnSnRKs分为三组。将所有检查组织中具有高表达水平的67个基因(基于LOG2基值> 6)分为组1。例如,BNSNRK3.40在所有营养器官中高度表达,平均基于LOG2的值为10.85。21.BnSnRK属于Group2的基因在检测到的组织中表现出相对低的表达水平(基于LOG2的值> 2)。Group3包括26个BnSnRK基因,在所有组织中具有最低表达水平(基于LOM2的值<2)。此外,属于Group3的6个基因(BnSnRK2.26/2.27和BnSnRK3.18 / 3.22 / 3.43 / 3.59)甚至没有在所有组织中表达。同时,Group1包含6个bnsnrk1,21.BnSnRK2,40BnSnRK3基因;第二组有2个bnsnrk1,4BnSnRK2,15.BnSnRK3基因和3组由2组成bnsnrk1,6BnSnRK2,18.BnSnRK3基因。这些结果表明BnSnRKS显示不同的表达式模式,同一亚家族内的基因也表达不同。
表达概况BnSnRKs在不同的非生物胁迫下
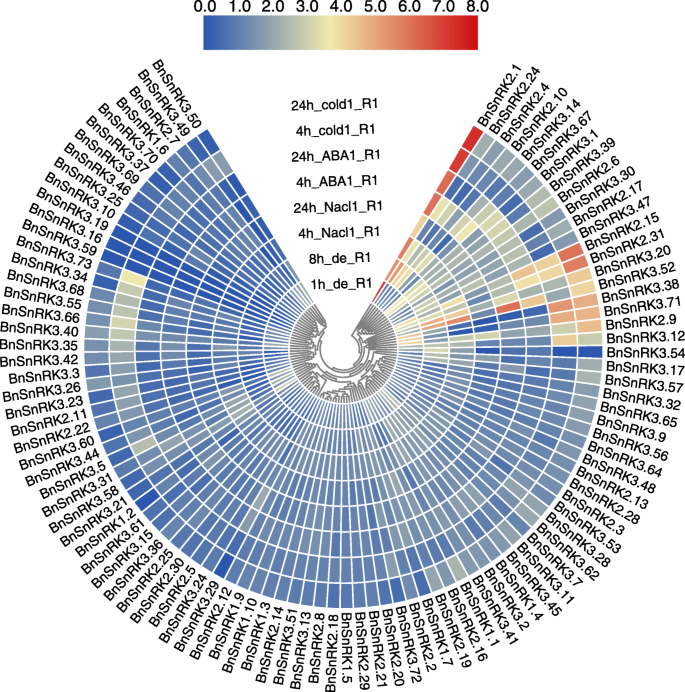
的表达模式BnSnRK利用已发表的转录组数据研究了不同非生物胁迫下的基因甘蓝型在干旱,盐度,ABA诱导和冷应力[28.].全面的,BnSnRK基因在不同的非生物应激下显着改变了表达水平(图。8、表S8).多个BnSnRK通过各种治疗显着诱导基因。例如,bnsnrk2.1.所有治疗都非常诱导。bnsnrk3.39.促进的表达水平响应脱水和ABA治疗。相比之下,几个BnSnRKs没有反应任何非生物的压力。例如,BnSnRK2.12和BnSnRK2.13在所有治疗过程中表明几乎没有表达更改。有趣的是,许多基因在不同的治疗中显示出对立的表达谱。例如,BnSnRK3.54通过脱水高度调节,而受到阿巴和冷治疗的压抑。
SNPS分析BnSnRKs在核心种质中甘蓝型
多态性的BnSnRK基因是利用我们之前对包含300份材料的核心种质资源的重测序数据确定的[29.,30.].MAF大于5%的单核苷酸多态性BnSnRK选择基因(表S9.).结果表明了BnSnRK基因含有平均11.76个SNP。详细,每个BnSnRK1每个亚家族基因包含14.00个SNPsBnSnRK2和BnSnRK3亚家族基因平均含有9.67和12.53个SNP。此外,每个SNP密度BnSnRK同一亚家族内的基因也不同。例如,没有确定的SNPBNSNRK1.1.,而有62只SNPbnsnrk1.7..
此外,详细的SNP分布分析BnSnRK2.10和bnsnrk3.39.被进行,因为两种基因在干旱胁迫下显着改变了它们的表达水平(图。9).有发现,1500bp启动子区中有9个SNP基因座,在外显子/内部区域的19个SNP和3'UTR区域中的1个SNPbnsnrk2.10;虽然我们在启动子区确定了6个SNP基因座,在外显子/内部区域的8个SNP和3'UTR区域中的1个SNPbnsnrk3.39..结合核心种质资源在干旱胁迫下的表型数据(数据未显示),发现在干旱胁迫下,该核心种质资源的SNP16、SNP26、SNP27在干旱胁迫下表现出显著性差异(p < 0.05)BnSnRK2.10,SNP4,SNP7,SNP15 INbnsnrk3.39.与他们的旱耐忍耐明显相关。
讨论
在这项研究中,114名成员BnSnRK基因在甘蓝型基因组,被指定为BNSNRK1.1.到BNSNRK3.73.在他们的亚家族分类的基础上。系统地调查BnSnRK对基因家族进行了研究,包括系统发育关系、基因结构、蛋白质基序、染色体分布、基因复制等CIS-启动子中的元素。此外,对表达模式和SNP测定进行了深度分析BnSnRK使用已发布数据的家庭基因。本研究提供了进一步函数表征的基本信息SNRK.增强植物在非生物胁迫下适应能力的基因。
以前的研究确定了39,48,44和34SNRK.基因拟南芥蒂利亚纳[3.],栽培稻[31.],Broachypodium distachyon.[32.),桉树茅[33.),分别。在甘蓝型基因组,BnSnRK基因数量远高于二倍体植株。114年BnSnRK将基因鉴定并分为三个亚属,其中包括10个BnSnRK1基因,31BnSnRK2基因和73BnSnRK3基因。更具体的描述显示了与每个亚壳之间的相似成员比例甘蓝型和其他物种。甘蓝型起源于自然交叉B.Rapa.(AA)和B.oleracea(CC) [25.].56和54BnSnRK基因定位于AA亚基因组和CC亚基因组,表明SNRK.基因在祖先物种中起着同样重要的功能作用。
各种各样的SNRK.亚家族基因含有不同的保守结构域,但所有基因保留了N-末端蛋白激酶结构域。例如,SnRK3由于NAF结构域允许,报道亚家族基因以钙依赖性方式与CBL相互作用。此外,NAF结构域定义了一组涉及各种信号过程的异源激酶,作为CBL钙传感器蛋白的靶标[7].在这项研究中,也发现了不同的BnSnRKS亚家族基因具有不同类型的保守结构域。这可能表明,有功能多样化BnSnRK基因家族根据他们的域名。
在atsnrk.和BnSnRK基因家族,不同的亚家族基因表现出显着的基因长度和外显子内含子结构分歧。在先前的研究中,具有较少内含子的基因被认为是植物中具有更高表达水平[34.,35.].具有较少内含子的紧凑型基因结构允许快速基因激活,并及时响应不同的环境压力[35.].然而,结合本研究中使用的转录组数据,我们没有发现BnSnRK内含子较少的基因表达量较高BnSnRKs.
越来越多的证据表明,基因活动通常与启动子区域的差异相关[36.].的CIS-位于基因启动子区域的元件在生长和环境变化中调控基因表达中起着关键作用[37.,38.].启动子分析显示BnSnRKs包含各种类型的CIS-元素,如DRE、ABRE和LTRE。大多数基因启动子至少包含其中一种成分,表明许多BnSnRKs能够对不同的非生物压力做出反应。例如,通过结合4 h冷胁迫下的基因表达谱,可以得到BnSnRKs随着荔枝和雪人独联体-元素平均增加了2.71倍,而BnSnRKs没有独联体-Elements仅增加了1.47倍。因此,这是CIS-元素分析提供了基因函数研究的线索,特别是对于不同应力下的基因表达模式。
基因表达型材提供了映射基因官能度的命令线索。首先,我们使用预先发布的转录组数据来调查BnSnRK不同组织和器官的基因表达水平甘蓝型[26.].分析结果表明,将这些基因的表达模式分为三组(图。7).与此同时,我们发现BnSnRKs与组1和组3相比,组2启动子中含有较少的顺式元件。其中,组1每个基因平均ABRE 2.84, DRE 0.40, LTRE 0.93,组3每个基因平均ABRE 2.81, DRE 0.38, LTRE 0.30,而组2每个基因平均ABRE 2.00, DRE 0.19, LTRE 0.57。这些证据表明BnSnRKs活性与启动子区域的差异相关。
一些角色的作用BnSnRKs对各种非生物胁迫的响应也被推导出来。根据干旱胁迫数据(表S8),响应基因BnSnRK2.10是直观的atsnrk2.3,调控ABA的合成和对干旱的响应信号A.thaliana[39.],表明相同的功能BnSnRK2.10在回应干旱压力.bnsnrk2.24,在干旱、盐胁迫和ABA诱导下表现出极显著的表达变化AtSnRK2.2,也可以反应渗透压力和ABA诱导在A.Thaliana,表明这一点BnSnRK2.24在甘蓝型[40,41.].然而,BNSNRK3.3.9基因可以在不同的压力下显着诱导,只存在于此甘蓝型。并结合核心种质资源的重测序数据,分析了各品种的snp位点BnSnRK确定基因。和SNP分布分析BnSnRKs如BnSnRK2.10和3.39,能否进一步提供功能性分子标记和等位基因BnSnRK家庭基因。
本研究提供了全面的知识SNRK.基因家庭甘蓝型.它为进一步理解个体的生物学功能提供了重要的启示BnSnRK基因甘蓝型.然而,该研究仅提供了初步的表征BnSnRK基因和大型功能验证工作需要在进一步的工作中进行,以了解其作用BnSnRK家庭。
结论
SNRK.基因在植物对生物和非生物胁迫的响应等信号通路中发挥着重要作用。在本研究中,全面研究了SNRK.基因家庭甘蓝型正在进行中。共114岁BnSnRK基因的特征和分为三种亚壳,其在同一亚家族内的基因结构和基因组合物中显示出高相似性。系统发育比较与联合培养分析SNRK.基因之间A.thaliana和甘蓝型提供了有价值的线索BnSnRK基因。而且,这是CIS-作用元件、基因表达及单核苷酸多态性分布BnSnRK家庭也得到了决心。这些结果提供了进一步了解生物学功能的重要信息BnSnRK基因甘蓝型.
方法
的识别BnSnRK家族基因甘蓝型基因组
氨基酸序列SnRKs基因家庭A.thaliana从NCBI下载(https://www.ncbi.nlm.nih.gov/).的同源基因atsnrks.在甘蓝型基因组在菜单宁宁7的参考基因组中被喷射(http://ibi.zju.edu.cn/bnedigome/download.php?con=ny7.)[27.].隐藏马尔可夫模型(HMM)和BLASTP计划初步鉴定BnSnRK蛋白质。基于隐马尔可夫模型(HMM)进行局部BLASTP (e-20)搜索SNRK.PFAM数据库的基因域(http://pfam.janelia.org/).所有候选序列SNRK.通过SMART数据库再次确认基因(http://smart.embl-heidelberg.de/)[42.],ncbi保守域数据库[43.]和pfam数据库[44.].氨基酸的数量、分子量(MW)和等电点(pI)SNRK.使用扩展物的工具计算蛋白质(http://www.expasy.ch/tools/pi_tool.html.).
系统发育分析及分类SNRK.基因家庭甘蓝型
具有默认参数的Clustalw用于114的多个序列对齐BnSnRK非冗余氨基酸序列[45.,46.].基于对准,通过邻近邻接(NJ)方法使用Mega 7.0构建系统发育树[46.],具有以下参数:Poisson模型,成对删除和1000个引导复制。所有的NJ树SNRK.蛋白质序列A.thaliana和甘蓝型也是使用MEGA 7.0构建的。
鉴定基序组合物和基因结构
识别保守的主题BnSnRK蛋白,Motif Elicitation (MEME)在线程序的多重期望最大化[47.](http://meme.sdsc.edu/meme/itro.html.)用以下参数进行:重复次数=任何,最大矩阵数= 10;和最佳的主题长度= 6到100个残留物。外显子内部结构BnSnRK使用基因结构显示服务器在线程序(GSDS:http://gsds.cbi.pku.edu.ch)[48.].
染色体位置和基因复制
所有染色体的位置BnSnRK根据使用MapChart版本3.0和Circos从NY7 Genome数据库的物理位置信息映射到油菜基因组的19染色体的19染色体[49.,50.].识别基因重复,所有甘蓝型基因序列使用BLASTP对齐,E值为1E-10。具有默认值的McScanx用于对复制模式进行分类SNRK.分成分段、串联复制[51.].串联复制的定义是在200 kb内包含两个或两个以上基因的染色体区域[52.].显示同系关系显示同系关系SNRK.基因之间甘蓝型和A.thaliana,同步分析图是使用自己编写的Python脚本构建的。非同义(K a)同义词(ks.)替代每重的BnSnRK基因的计算kaks._Calculator 2.0 [53.].发散时间采用公式T =ks./ 2R,其中R为每年每个网站的1.5×10-8同义词[53.].
CIS-推动者区域中的元素BnSnRKs
来自每个开始密码子的上游序列(1500 bp)BnSnRK从NY7的基因组序列中提取基因,然后用于CIS-使用PlantCare软件推动者区域的元素分布(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)[54.].
的表达模式BnSnRKs
转录组数据12不同甘蓝型由Sun等人从先前的研究中获得组织。[26.],项目编号为PRJNA394926。此外,转录组数据甘蓝型在脱水下,Zhang等人描述了盐,ABA诱导和冷应激条件。[28.].数据可用于(https://bigd.big.ac.cn/),项目编号为CRA001775。差异表达分析BnSnRKs使用DSEQ2 R封装进行,并通过TBTOOLS软件创建热量。
SNP分布分析BnSnRKs在甘蓝型加入
编码区域中的SNPBnSnRK从300个snp中提取基因甘蓝型通过我们之前的研究中的基因组重新测序确定的摘要[29.,30.].使用MAF的高质量SNP大于5%,缺失率小于50%用于进一步分析。所有高质量的SNP都被映射到“DARMOR-BZH.“基因组(甘蓝型V4.1基因组,http://www.genoscope.cns.fr/brassicanapus/data/).
可用性数据和材料
本研究期间分析的所有数据均包含在本文及其附加文件中。
缩写
- 阿坝:
-
脱盐酸
- 伊稳定石:
-
ABA回应
- 安培:
-
AMP活化蛋白激酶
- atsnrk.:
-
拟南芥SnRK
- BnSnRK:
-
芸苔属植物显著SnRK
- BLASTP:
-
基本的局部比对搜索工具-蛋白质
- CBLS:
-
钙传感器钙素B样蛋白
- CIPK.:
-
CBL相互作用的蛋白激酶
- Dre:
-
脱水反应元素
- GSDS:
-
基因结构显示服务器
- 唔:
-
隐藏的马尔可夫模式
- LTRE:
-
低温响应元件
- ROS:
-
反应性氧气
- SAPK:
-
应激活化蛋白激酶
- SNRK.:
-
蔗糖非发酵1(SNF1) - 相关蛋白激酶
- SOS:
-
盐过度敏感
参考文献
- 1。
李鹏飞,龚qq,马世生。非生物抗胁迫机制的研究——启动基因组学。植物学报2006;9:180-8。
- 2。
Hunter H.蛋白激酶和磷酸酶:粘性蛋白磷酸化和信号传导。细胞。1995年; 80:225-36。
- 3.
Hrabak Em,Chan CW,Gribskov M,Harper JF,Choi JH,Halford N,Kudla J,Luan S,Nimmo Hg,Sussman Mr,Thomas M,Walker-Simmons K,Zhu Jk,Harmon AC。拟南芥CDPK-SNRK Superfamily蛋白激酶。植物理性。2003; 132:666-80。
- 4.
Halford NG, Hardie DG。snf1相关蛋白激酶:植物碳代谢的全球调节者?植物学报1998;37:735-48。
- 5。
一种酵母基因是葡萄糖抑制释放所必需的,编码蛋白激酶。科学。1986;233:1175 - 80。
- 6。
Kulik A,Wawer I,Krzywińskae,Bucholc M,Dobrowolska G.SnRK2蛋白激酶 - 植物应激胁迫的关键调节因子。omics。2011; 15:859-72。
- 7。
Albrecht V, Ritz O, Linder S, Harter K, Kudla J. NAF结构域定义了一个新的蛋白-蛋白相互作用模块2+- 调节的激酶。embo J.2011; 20:1051-63。
- 8。
Masaru O,Yan Y,Ursula H,Zhu JK。蛋白激酶SOS2中的新型结构域介导与蛋白质磷酸酶2C ABI2的相互作用。Proc Natl Acad Sci U S A. 2003; 100:11771-6。
- 9。
Hardie DG,Carling D,Carlson M.AMP活化/ SNF1蛋白激酶亚家族:真核细胞的代谢传感器?annu rev biochem。1998年; 67:821-55。
- 10。
Purcell PC, Smith AM, Halford NG。在马铃薯中蔗糖非发酵相关蛋白激酶序列的反义表达导致块茎中蔗糖合酶的表达减少,叶片中蔗糖合酶诱导性的丧失。植物j . 1998; 14:195 - 202。
- 11.
植物细胞能量传感器SnRK1的默认激活和核易位调节代谢应激反应和发育。植物细胞。2019;31:1614-32。
- 12.
Halford Ng,嘿SJ。SNF1相关的蛋白激酶(SNRK)在复杂的网络中起作用,其在植物中联系起代谢和应力信号传导。Biochem J. 2009; 419:247-59。
- 13。
Maszkowska J, Dębski J, Kulik A, Kistowski M, Bucholc M, Lichocka M, Klimecka M, Sztatelman O, Szymanska KP, Dadlez M, Dobrowolska G.磷蛋白组学分析表明,snf1相关蛋白激酶2.10在渗透胁迫反应中磷酸化了脱氢酶ERD10和ERD14。植物学报。2019;42:931-46。
- 14。
DiédhiouCJ,Popova OV,Dietz KJ,Goldack D. SNF1型丝氨酸苏氨酸蛋白激酶SAPK4调节水稻中的应力响应基因表达。BMC植物BIOL。2008; 8:49。
- 15.
张清,贾赫夫,刘Gs,杨sn,张s,阳yx,杨pp,崔氏菌克隆与尼古拉塔克烟囱SNRK2亚家族II基因的克隆与鉴定。Mol Biol rep. 2014; 41:5701-9。
- 16。
Kawa D, Meyer AJ, Dekker HL, Abd-El-Haliem AM, Gevaert K, Van De Slijke E, Maszkowska J, Bucholc M, Dobrowolska G, De Jaeger G, Schuurink RC, Haring MA, Testerink C. SnRK2蛋白激酶和mRNA解码机制控制根的发育和盐反应。植物杂志。2020;182:361 - 77。
- 17。
Anna-Chiara M,Sylvain M,Alain V,Francesca F,Giraudesca j.Arazidopsis Ost1蛋白激酶通过脱落酸介导气孔孔的调节,并作用于反应性氧物种生产的上游。植物细胞。2002; 14:3089-99。
- 18。
Ali A,Kim JK,Jan M,Khan Ha,Khan Iu,Shen Mz,Park J,Lim CJ,Hussain S,Bae D,Wang K,Chung Ws,Rubio V,Lee Sy,Gong Zz,Kim Wy,Bressan Ra,Pardo JM,Yun DJ。通过Hos15介导的OST1降解ABA信号的流变控制。莫尔植物。2019; 12:1447-62。
- 19。
Kim KN, Lee JS, Han H, Choi SA, Go SJ, Yoon IS。一种新型水稻钙的分离与鉴定2+- 涉及不同信号的蛋白激酶基因,包括寒冷,光,细胞素,糖和盐。植物mol biol。2003; 52:1191-202。
- 20。
郭y,xiong l,歌曲cp,锣,半,zhu jk。钙传感器及其相互作用的蛋白激酶是拟南芥中脱落酸信号传导的全局调节因子。DEV细胞。2002; 3:233-44。
- 21。
刘杰,伊希提米,半,金cs,朱杰克。Arabidopsis Thaliana SOS2基因编码了耐盐所需的蛋白激酶。Proc Natl Acad SCI U S A. 2000; 97:3730-4。
- 22。
马QJ,Sun MH,康H,Lu J,You CX,Hao YJ。CIPK蛋白激酶靶向SER(254)的蔗糖转运蛋白MDSUT2.2,用于磷酸化以增强耐盐性。植物细胞环境。2019A; 42:918-30。
- 23。
马庆军,孙明辉,卢杰,康辉,尤春霞,郝玉娟。苹果蔗糖转运体MdSUT2.2是响应干旱的蛋白激酶MdCIPK22的磷酸化靶点。Plant Biotechnol J. 2019b; 17:625-37。
- 24。
陈L,仁F,周L,王QQ,Zhong H,Li XB。芸苔钙钙素B样1 / CBL相互作用蛋白激酶6(CBL1 / CIPK6)组分参与植物对非生物应激和ABA信号传导的反应。J Exp Bot。2012; 63:6211-22。
- 25。
Chalhoub B,Denoeud F,Liu S,Parkin IAP,Tang H,Wang X,Chiquet J,Belcram H,Tong C,Samans B等。晚期新石器时代芸苔油菜籽基因组的早期大型国民多倍曲面演变。科学。2014; 345:950-3。
- 26。
Sun FM,粉丝GY,胡Q,周ym,Guan M,Tong Cb,Li Jn。甘蓝型豆腐品种“ZS11”的高质量基因组揭示了半冬季Mor晶型的血阶历史。工厂J. 2017; 92:452-68。
- 27.
邹军,毛丽芳,邱娟,王敏,雷军,吴大伟,何忠生,陈明辉,沈玉芳,沈慧华,黄玉杰,李瑞芳,胡德东,史林,王凯,朱庆华,叶春燕,Ian B, Graham JK,孟JL,范丽娟。亚洲年轻异源四倍体油菜籽的全基因组选择足迹和有害变异。生物技术学报2019;17:1998-2010。
- 28.
张YT,Ali U,张GF,Yu LQ,方氏,IQBAL S,Li Hh,Lu Sp,Guo L.转录组分析揭示了油菜籽中多种非生物胁迫的基因。mol品种。2019; 39:158。
- 29.
吴DZ,梁Z,燕T,徐Y,宣LJ,唐J,周G, Lohwasser U, SJ,王,为什么陈XY,王Q,朱L, Maodzeka,侯赛因N,李ZL,李XM,沙姆西IH,显得优雅G,吴LD,郑港元,张GP, Chalhoub B,沈LS, Yu H,江泽民LX。对世界范围内的一批油菜籽进行全基因组重测序,揭示了生态型分化的遗传基础。摩尔。2019;12:30-43。
- 30.
Xuan Lj,Yan T,Lu Lz,赵Xz,吴迪兹,华SJ,江吕克。基因组 - 范围的协会研究揭示了在多倍体油菜籽中携带叶片毛细血管形成的新基因(芸苔属植物显著L.)。植物细胞环境。2019; 43:675-691。
- 31。
高渗透胁迫和脱落酸对水稻蔗糖非发酵相关蛋白激酶2家族活性的影响。植物细胞。2004;16:1163 - 77。
- 32。
王立忠,胡伟,孙金堂,梁学勇,杨学勇,魏淑英,王新涛,周勇,肖强,杨国祥,何国贵。二穗短柄草SnRK基因家族全基因组分析及BdSnRK2.9功能分析植物科学。2015;237:33-45。
- 33。
王永军,闫慧峰,邱志峰,胡波,曾伯生,钟瑞林,范家杰。巨桉SnRK基因家族及其对盐胁迫的响应。国际分子生物学杂志。2019;20:11。
- 34。
Chung by,Simons C,Firth Ae,Brown Cm,Hellens RP。5'UTR IntroneN基因表达在拟南芥中的影响。BMC基因组学。2006; 7:120。
- 35。
Jeffares DC,Penkett CJ,BählerJ.迅速调节基因是内部的。趋势类型。2008; 24:375。
- 36。
斯蒂芬J,Fei N,Juergen E,张斯蒂芬J,Fei N,Juergen E,张S,Dong W,Xue T,Zheng C,Yuan Z.蛋白质磷酸酶2C在水稻和拟南芥中的基因组宽和表达分析。BMC基因组学。2008; 9:1-21。
- 37。
干旱胁迫下水稻叶鞘的蛋白质组学分析。蛋白质组学杂志2006;5:396-403。
- 38。
Sarda X,Tousch D,Ferrare K,Legrand E,Dupuis J,Casse-Delbart F,Lamaze T.编码水通道蛋白的两种尖端基因在向日葵防护池中表示。工厂J. 1997; 12:1103-11。
- 39。
谭文荣,张德伟,周海平,郑涛,尹永华,林海辉。转录因子HAT1是SnRK2.3激酶的底物,负调控拟南芥对干旱响应的ABA合成和信号转导。公共科学图书馆麝猫。2018;14:e1007336。
- 40。
中岛美嘉K, Fujita Y, Kanamori N,片瞳T, Umezawa T, Kidokoro年代,Maruyama K,吉田T,西山贵子K,小林M, Shinozaki K, Yamaguchi-Shinozaki K三个拟南芥SnRK2蛋白质激酶,SRK2D / SnRK2.2 SRK2E / SnRK2.6 OST1和SRK2I / SnRK2.3,参与了ABA信号对发展和种子休眠的控制至关重要。acta botanica yunnanica(云南植物研究). 2009;
- 41。
Yoshida T, Fujita Y, Maruyama K, Mogami J, Todaka D, Shinozaki K, Yamaguchi-Shinozaki K.四种拟南芥AREB/ABF转录因子主要作用于SnRK2激酶下游的基因表达,以响应渗透胁迫。植物学报。2015;38:35-49。
- 42。
SMART 7:蛋白质结构域注释资源的最新更新。核酸学报2012;40:302-5。
- 43。
Marchler-Bauer A,Lu S,Anderson JB,Chitsaz F,Derbyshire Mk,Deweese-Scott C,Fong Jh,Geer Ly,Geer Rc,Gonzales Nr,Gwadz M,Hurwitz di,Jackson JD,Ke Z,Lanczycki CJ,LuF,Marchler GH,Mullokandov M,Omelchenko MV,Robertson Cl,Song Js,Chrenthi N,Yamashita Ra,Zhang D,Zhang N,Zheng C,Bryant Sh。CDD:蛋白质功能注释的保守域数据库。核酸RES。2011; 39:225-9。
- 44。
Finn Rd,Coggill P,Eberhardt Ry,Eddy SR,Mistry J,Mitchell Al,Potter SC,Punta M,Qureshi M,Sangradorvegas A. PFAM蛋白质家庭数据库:迈向更加可持续的未来。核酸RES。2016; 44:279-85。
- 45。
Saitou N,Nei M.邻居加入方法:一种重建系统发育树的新方法。mol Biol Evol。1987; 4:406-25。
- 46。
Kumar S,SteCher G,Tamura K. Mega7:用于更大数据集的分子进化遗传学分析版本7.0。mol Biol Evol。2016; 33:1870-4。
- 47。
Bailey TL,Boden M,Buske Fa,Frith M,Grant Ce,Clementi L,Ren J,Li WW,Noble WS。MEME SUITE:主题发现和搜索的工具。核酸RES。2009; 37:202-8。
- 48。
胡斌,金军,郭爱华,张华,罗军,高国刚。gss2.0:一种改进的基因特征可视化服务器。生物信息学。2015;31:1296-7。
- 49。
Voorrips RE. MapChart:图形化表达连锁图和qtl的软件。J在这里。2002;93:77-8。
- 50。
Krzywinski M,Schein J,Birol I,Connors J,Gascoyne R,Horsman D,Jones SJ,Marra Ma。电讯:对比较基因组学的信息审美。Genome Res。2009; 19:1639-45。
- 51。
Wang Y, Tang H, DeBarry JD, Tan X, Li J, Wang X, Lee T, Jin H, Marler B, Guo H. MCScanX:一种用于基因共线性和共线性检测的工具箱。核酸学报2012;40:49。
- 52。
Holub EB。在野生植物拟南芥(Arabidopsis)中,军备竞赛早已成为历史。Nat Rev Genet, 2001;2:516。
- 53。
王D,张Z,张Z,朱家,余J.Kaks_Calculator 2.0:一种工具包,包括伽玛系列方法和滑动窗策略。基因组学蛋白质组学生物信息学。2010; 8:77-80。
- 54。
Lescot M,DéhaisP,Thijs G,Marchal K,Moreau Y,Yves Vdp,Pieree R,Stephane R. Plantcare,植物顺式作用调节元件数据库和用于启动子序列的硅分析的工具的门户网站。核酸RES。2002; 30:325-7。
致谢
我们感谢Guoping Zhang博士在手稿修订中的富有洞察力和贡献。
资金
这项工作得到了中国国家自然科学基金(31961143008,31701411),中国浙江省科技计划(LGN20C130007),江苏省近代农作物生产协作创新中心,以及111个外国专家引入项目(b17039)。融资机构都没有在研究或收集,分析和对数据的解释中具有任何作用以及写作稿件。
作者信息
隶属关系
贡献
WZ,DW和LY构思和设计了研究。WZ和LY执行了实验和数据分析。WZ,DW,LJ和LY写了这篇文章;所有作者都阅读并批准了最终文章。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:表S1。
的信息答:芥SnRKs在查询中。表S2。特点SNRK.家庭甘蓝型.表S3。114的清单BnSnRK本研究中确定的基因。表S4。保守的氨基酸基序和注释BnSnRKs.表S5。同时性块Ka / Ks的值BnSnRK基因。表S6。之间的一对一的正交关系甘蓝型和A.thaliana.表S7。表达配置文件(基于LOG2的值)BnSnRKs在不同的组织下。表S8。表达式配置文件(比率与控制值)BnSnRKs在不同的非生物胁迫下。表S9。的列表BnSnRKsnp在本研究中被鉴定。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
朱,W.,Wu,D.,江,L.et al。基因组 - 范围鉴定和表征SNRK.家庭基因芸苔属植物显著.BMC植物BIOL.20,287(2020)。https://doi.org/10.1186/s12870-020-02484-3
已收到:
接受:
发表:
关键字
- 芸苔属植物显著
- SNRK.
- 全基因组
- 非生物压力
- 表达式模式