抽象的
背景
CRISPR / CAS9技术的发展以有效和精确的方式促进了有针对性的诱变。以前,RNAI沉默易感性(S.)基因P.Oowdery.millow.R.eSistance 4.(PMR4.)已被证明能增强番茄对白粉病病原菌的抵抗力Oidium neolypopersici(在).
结果
研究是否完全敲门番茄PMR4.基因会导致比我们生成的RNAi沉默的转基因植物更高水平的抵抗力PMR4.Crispr突变体。我们使用了一个旨在瞄准番茄的四个单引导RNA(SGRNA)的CRISPR / CAS9构造PMR4.增加突变体中大量缺失基因的可能性。通过对转化子的pcr筛选和测序,我们确定了5个不同的突变事件,包括4 - 900-bp的缺失,1-bp的插入和892-bp的倒置。这些突变体都表现出对在基于疾病症状视觉评分及相对真菌生物量的定量。组织学观察揭示了在真菌感染的位置显着较高的过敏反应性细胞死亡PMR4.与野生型植物相比的突变体。在突变体中减少了出血形成和悬鼠生长,但不完全抑制。
结论
CRISPR / CAS-9番茄的靶向诱变PMR4.基因导致突变体对PM病原体的敏感性降低,但不是完全丧失在。我们的研究表明CRISPR / CAS9系统的效率和多功能性作为学习和表征的强大工具S.通过产生不同类型的突变来。
背景
番茄中的粉状霉菌(PM),由Inveriace Biotrophic真菌引起Oidium neolypopersici(在),是一种威胁生产温室和野外西红柿的全球疾病[1那2].在过去的几十年中,重点关注番茄中PM抗性的研究导致鉴定了五种主导抵抗(R.)基因(ol-来自野生番茄种的基因[3.].这些基因含有溶血性番茄品种金钱制造商,并进行了近代源线(尼尔)[4.].白粉病感染后这些NILs的组织学研究允许识别与耐药性相关的两种不同类型的宿主反应:单细胞超敏反应(HR)OL-4和OL-6.,导致对PM完全抵抗;和缓慢的多细胞小时OL-1那ol-3和ol-5,导致阻力不完全[4.那5.那6.].
除了这些占主导地遗传的抗性基因之外,患有隐性基因的命名ol-2被确定(7.].这是粉末状霉菌易感性的功能突变体等位基因(S.)基因,霉菌阻力位于o(MLO)[7.].在纯合状态下ol-2等位基因通过诱导乳头形成和胼胝质沉积,阻止真菌在渗透阶段的发育,从而对不同的PM物种产生广谱抗性[6.].功能丧失等位基因枣疯病是对PMS的隐性遗传性阻力的最佳研究示例之一8.].近年来,许多其他抵抗的例子赋予了损害所赋价S.在不同的危机系统中描述了基因[9.].使用这样的S.植物育种中的基因,由于它们的广谱和潜在的持久的抗性特性,代表了一个有希望的替代品的渐渗R.一直驾驶传统抗性育种的基因[9.那10.那11.].
在拟南芥中,一些突变体对适应的PM病原体表现出抗性Golovinomyces cichoracearum在筛选EMS人群后已被识别[12.].这些突变体被命名粉状霉菌抗性1(PMR1.)PMR4..的PMR4.突变体不仅表现出抗性g . cichoracearum,但也反对G. Orontii.和霜霉病Hyaloperonospora Arabidopsidis..PMR4.被证明是胼inse合酶基因AT4G03550,也称为葡聚糖SYNTHASE-LIKE 5(GSL5)要么胼胝质合成酶12(CALS12)[13.那14.].的GSL./CAL基因家族在拟南芥中包含12个基因[15.那16.那17.].胼callose合成酶编码PMR4.负责响应生物和非生物应激的愈伤组织[14.].这俩PMR4.和一个GSL5.突变体(纯合用于T-DNA插入的纯合PMR4.基因)显示几乎完全缺乏感染叶子的胼unse [12.那13.].组织学分析证明,即使在没有召唤的情况下,这些突变体在PM感染后形成乳头蛇膜[13.那14.].
缺乏在尝试的渗透位点形成的乳头缺乏调用的突变植物似乎奇怪地显示出PM抗性,尤其是因为拟南芥植物过表达PMR4.显示完全渗透性阻力g . cichoracearum[18.那19.].后一种抗性基于在感染部位的增加的胼舌沉积,其用作针对PM分泌的细胞壁水解酶的物理屏障[19.].
然而,PMR4.突变体在PM感染后,使病变让人清醒过敏细胞死亡[14.].这种细胞死亡可能来自激活水杨酸(SA)信号转导途径,因为该途径中的基因在上调PMR4.与野生型控制相比突变体,并阻止了SA途径PMR4.突变体(使用双突变体)足以恢复对PM的完全敏感性[14.].可能,PMR4不仅在尝试真菌渗透后在特定位点愈伤组合,而且还负面控制SA相关的防御途径[20.].在过表达的植物中PMR4.SA通路没有被诱导[18.].因此,通过过表达或敲除抗性涉及到不同的机制PMR4.基因。
为了调查除拟南芥的其他植物种类中是否保守了PMR4功能,最接近的番茄果实PMR4.(solyc07g053980;SLPMR4.)被RNAi沉默了[21.].该基因沉默的转基因植物显示出对番茄PM病原体的增强抗性(在),而与非静音控制相比,工厂规模略有缩小[21.].这些RNAi植物仍然表现出低水平的表达SLPMR4.基因。基于击倒的方法表征S.基因,如RNAi或病毒诱导的基因沉默(VIGS),通常导致目标基因的残留表达,这通常导致部分表型使基因的功能分析复杂化[22.].产生全敲除基因函数的突变有时会产生比RNAi沉默获得的滞下转基因植物更强的表型[23.那24.]。我们出发了生产番茄SLPMR4.突变体调查是否完全阻力在可以以这种方式获得,与RNAI转基因植物中的大量但不完全抗性相比[21.]。因此,靶向诱变是首选的方法。精确的,稳定的和可遗传的敲除等位基因的产生S.现在可以随着集群的发展,定期间隙的短语重复(CRISPR)技术的发展成为可能。该技术已被用来产生抗PM的slmlo1.番茄突变体(25.]和抗pm小麦,三者同时修饰EDR1.同系物(26.].此外,使用CRISPR诱导的靶向突变实现了其他致病系统中的宿主阻力[27.那28.].在这项研究中,我们通过CRISPR / CAS9在番茄原木中举报了五种不同突变事件的成功生成和表征PMR4.基因。此外,我们展示了这些下午的PMPMR4.突变体与PM感染的细胞死亡有关。
结果
CRISPR / CAS-9目标诱变SLPMR4.
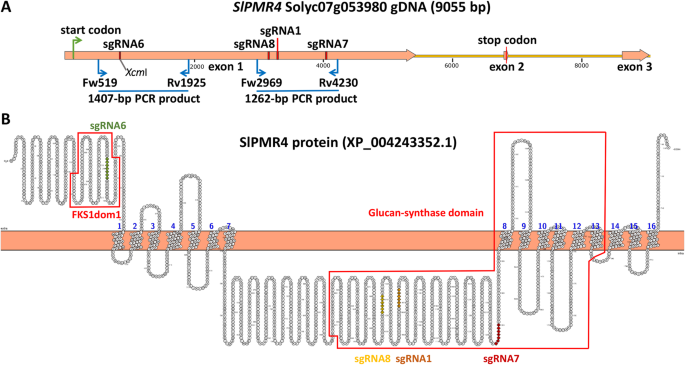
产生番茄的突变体PMR4.Ortholog(solyc07g053980)[21.一个包含4个sgrna (sgRNA6, sgRNA8, sgRNA1和sgRNA7;补充表1)是制作的。我们使用了四个SGRNA,以增加获得大缺失的突变体的机会[29.那30.]。四个sgrnas的位置SLPMR4.基因组序列和预测的SLPMR4蛋白可以在图1中看到。1.PMR4.包含两个已知的NCBI保守结构域(https://www.ncbi.nlm.nih.gov/cdd/): FKS1dom1 [32.]和葡聚糖合酶结构域[33.].四种sgrna中的一种(sgRNA6)靶向于n端区域的FKS1dom1结构域。其余三种sgrna靶向葡聚糖合酶结构域的细胞内部分。的SLPMR4.CRISPR / CAS9构建体用于改变敏感的番茄品种金钱制造商(MM)。
sgrna的靶位点位置SLPMR4..一个。基因组序列的表示SLPMR4.显示旨在产生敲除突变体的四个单一导向RNA(SGRNA)的位置SLPMR4..PCR引物FW519 + RV1925和FW2969 + RV4230用于鉴定突变体。b。普罗特(31.] SLPMR4蛋白的表示。SGRNA6(绿色)位于蛋白质的N末端FKS1Dom1结构域;SGRNA8(黄色),SGRNA1(橙色)和SGRNA7(红色)葡聚糖 - 合成酶结构域内部分的靶位点
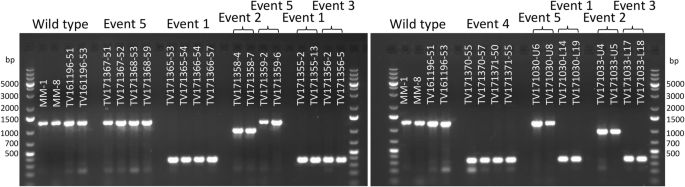
总共获得37种初级转化体(T1)并分析。含有突变的个体T1植物SLPMR4.通过PCR扩增选择基因。使用了两种不同的引物组合:引物FW519 + RV1925侧翼SGRNA6和引物FW2969 + RV4230侧翼SGRNA8,SGRNA1和SGRNA7(图。1).当大缺失发生较小的PCR产物时,比来自野生型(WT)的预期产品将在琼脂糖凝胶上可见。另外,SGRNA6目标站点加PAM包含限制酶的识别序列XCM.我(CCANNNNN↓NNNNTGG)。当一个小indel发生在该靶点时,PCR产物不会被酶切XCM.一世。
PCR具有引物FW519 + RV1925侧翼SGRNA6(图。1)没有在任何一级转化体中明显小于1407-BP WT PCR产物的PCR产物,表明该区域未发生大量缺失。相反,PCR具有引物FW2969 + RV4230的侧翼三个其他SGRNA导致选择17个T1转化体,与WT等位基因的1262-BP PCR产物相比具有大的缺失。随后将它们自拍以产生T2后代。通过PCR分析T2后代,选择突变的单个T2植物,以产生T3后代以促进突变的进一步表征(下一部分所示的结果)。
为了分析任何所选择的T2或T3突变体在SGRNA6靶位点上含有小型吲哚,用引物FW519 + RV1925获得的PCR产物被消化XCM.I.对于所有测试的植物消化完整,表明SGRNA6靶位部位没有发生突变。为了确定突变体中的任何突变体都含有SGRNA6区域中的突变,对PCR产物进行测序。没有观察到WT等位基因差异。这些结果表明,FKS1Dom1结构域中的SGRNA6在产生突变方面无效,而葡聚糖合酶结构域中的三个SGRNA中的一种或多种成功。
CRISPR/ cas -9介导的突变体的特征SLPMR4.
我们以衍生自原始转基因T1植物的T2和T3后代的突变事件(表格)1).根据与WT等位基因相比,从分离T2族常合的潜在突变等位基因纯合的植物均匀差异,所述尺寸在PCR扩增产物中显示出尺寸差异(使用引物FW2969 + RV4230)。另外,获得纯合T3系,通过重复PCR扩增来验证突变等位基因的存在(图。2).
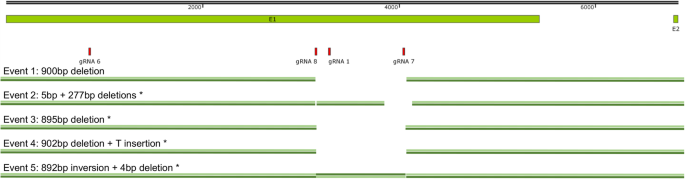
通过PCR片段的测序,我们在突变系中鉴定了5个不同的突变事件(“事件”)(图)。3., 桌子1和补充文件1).事件1包含sgRNA8和sgRNA7之间900-bp的帧内缺失。事件2显示sgRNA8位点缺失5 bp, sgRNA7位点缺失277 bp。事件3在sgRNA8和sgRNA7之间有895 bp的缺失。事件4在sgRNA8和sgRNA7之间有一个902-bp的缺失,并且在突变位点插入了一个T。Event 5是一个特例,在sgRNA8和sgRNA7之间的缺失位点旁边有一个4-bp的缺失位点和一个892-bp的倒置位点。除事件1外,预测所有突变事件均会在转录本中产生提前翻译终止密码子(PTTCs) (Supplementary figure)1).事件1的预测蛋白在葡聚糖合酶结构域缺乏300个氨基酸。
表型,CRISPR / CAS9PMR4.突变体类似于非转化的mm。然而,与MM和WT等位基因的转化体相比,它们略有尺寸略微降低。这是一个例外是线TV171370,显示用矮化表型的植物的分离(八个T3后代中的三个)。为了探讨该矮种表型的原因,进行PCR以检查Cas9序列是否存在和与T-DNA整合位点的可能缔合。所有八种植物都携带Cas9基因;因此,没有发现T-DNA集成的关联。接下来,通过对FW519 + RV1925 PCR产物进行测序来研究SGRNA6靶部位。在侏儒植物中没有观察到额外的突变。
选择sgrna的标准之一是最小的脱靶几率(非预期突变在基因组的其他地方而不是SLPMR4.).使用Cas-OFFinder程序时[34.在SGRNA和(OFF-)靶序列之间的3或更低错配的阈值,对于SGRNA 6,一个用于SGRNA8(在代际区域中),一个用于SGRNA1(在基因溶解的内含子中,1)三个用于SGRNA7(补充表2).这些偏离目标全部含有3个不匹配,其中大部分存在于SGRNA的种子中。来自SGRNA7的三个偏移目标是在基因区域和基因溶液的编码序列中。该基因编码指定的愈合合酶样基因SLPMR4.h2 (21.].因此,我们调查了是否有突变发生在SLPMR4.我们所选的-H2基因SLPMR4.(-H1)突变体。为此,使用引物PMR4_H2_FW1和PMR4_H2_RV1进行PCR,得到含有SGRNA7推定的脱靶位点的765-BP PCR产物。总共36个单独的T2和T3突变植物和6种对照植物(MM和WT-等位基因携带转基因植物)的PCR产物进行测序。在对照和突变体(包括三种矮人)植物之间没有发现序列差异,表明在没有发生偏离目标突变SLPMR4.-H2基因。
抗白粉病SLPMR4.突变体
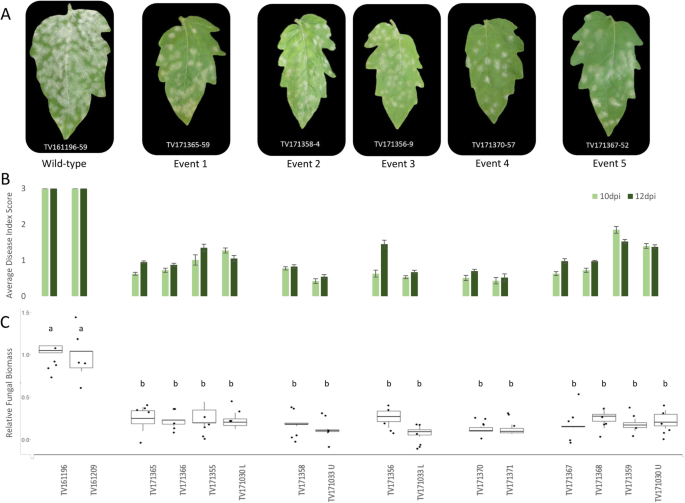
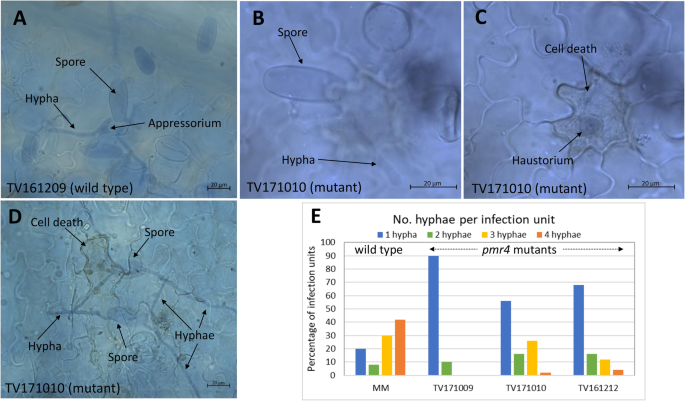
以前的RNAi线路研究表明倒下的SLPMR4.增强粉末霉菌的抗性[21.].评估我们是否SLPMR4.突变线显示出对PM的增加或充分抵抗,我们接种了它们在评估疾病指数(图。4.A和B)。此外,我们通过测量相对来量化疾病严重程度在突变体中的生物质确认表型观察结果(图。4.C)。使用携带WT等位基因的两条不成功的转基因线(TV161196和TV161209)用作对照。在突变体中没有观察到疾病指数或相对真菌生物量的显着差异。然而,与下降疾病指数指示和显着降低真菌生物量的对照相比,所有突变体显示出降低的敏感性(图。4.,补充图形2).
拟南芥感染部位的组织学分析PMR4.突变体揭示了过敏反应(HR)的存在[14.].调查我们是否可以在我们的情况下观察到这种现象SLPMR4.突变体,我们在除了野生型MM植物之外,使用代表所有五种突变事件的21种植物的严重感染叶样品进行了组织学研究。样品在感染后44小时(HPI),并用台盼蓝染色。在突变体和野生型植物中量化真菌结构和植物细胞死亡(表2).在任何样品中没有观察到乳头。HR样细胞死亡以更高的感染遗体可见PMR4.突变体与野生型等位基因携带MM相比(表2和图。5.).同时,与MM相比,突变体的初级吸器百分率和每个感染单位的菌丝数量均有所下降PMR4.突变体显示出降低的易感性在,但不完全抵抗。
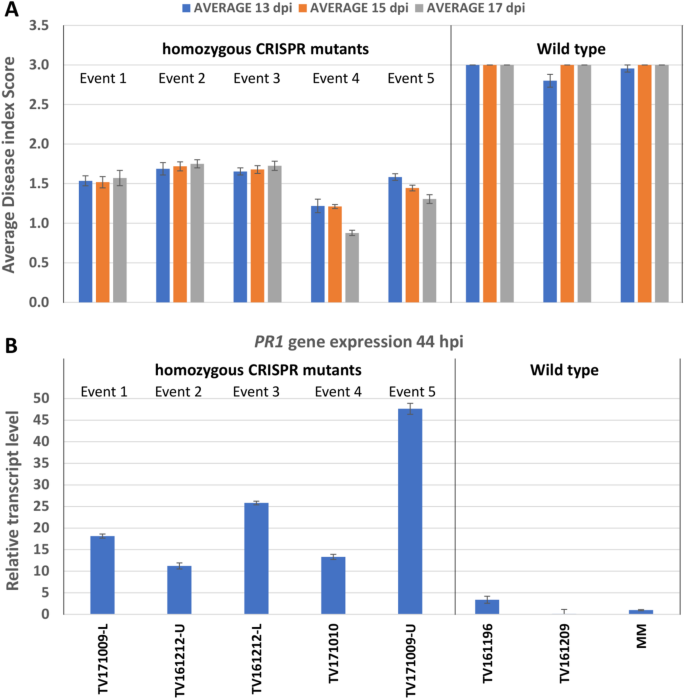
在拟南芥中,它表明PM阻力PMR4.突变体与SA信号转导途径的激活相关[14.].分析SA途径是否也激活SLPMR4.番茄的突变体表达水平PR1PM感染后的基因44 h通过QPCR测定。来自代表所有五种突变事件和对照植物的单个纯合突变体T2植物(MM和不成功的转基因TV161196和TV161209)中取出感染的叶样品。该实验的疾病指数评分如图1所示。6.A和亲戚PR1图2中的基因表达式。6.b。与控制线相比,来自所有五个突变事件的植物显示到PM的易感性(但不完全电阻)(图。6.a).所有突变体植株均表现出显著增加PR1与非突变对照相比,感染后44小时(hpi)的基因表达(图。6.b)。
讨论
多指导RNA获得敲除突变体的有效性
使用功能损失等位基因S.-基因在植物育种中是一种很有前途的替代传统的方法R.- 基于基于型血频繁殖,因为它具有耐用和广谱特性[10.].在这项研究中,我们成功地生产了Crispr / Cas-9介导的易感基因的敲除PMR4.在番茄中对阵PM病原体在.我们的结果表明,使用四个SGRNA用于CRISPR诱导的突变和选择使用PCR扩增对可见(大)缺失的选择是有效的,以获得五个突变事件。不同目标站点的分析表明了四个SGRNA的有效性差异,尽管所有四个SGRNA是基于相同的严格标准选择,如GC含量,二次结构和基本配对分数(补充表1).没有发现突变靠近SGRNA6靶位点,因此在引导CAS9蛋白诱导双链突破是不有效的。然而,所有五个突变事件似乎是SGRNA8和SGRNA7的靶位点的双链断裂的结果,因为在这些位置处或之间发现缺失。因此,SGRNA8和SGRNA7似乎非常有效。我们无法判断SGRNA1是否有效,因为该SGRNA的靶位位点位于SGRNA8和SGRNA7之间,并在五个突变事件中的三个中删除(图。3.).我们专注于选择大缺失的突变体SLPMR4.在原始的初级转化体中,我们尚未检查在SGRNA1,7或8的靶位点处是否存在具有小吲哚(插入或缺失的少量核苷酸的缺失)的任何额外突变体。这种分析将允许更好地比较SGRNA1,7和8的有效性。
我们表征的五种突变事件之一包含892bp反演,显示通过使用CRISPR / CAS9技术发生染色体重新布置。据报道,通过CRISPR / CAS9系统的基因组编辑,据报道,在SGRNA之间产生反转[35.那36.].诱导倒转在植物育种中特别重要,因为它有可能使连接固定或断开[36.].
推定的脂肪效应SLPMR4.突变
类似于在转基因植物上的观察SLPMR4.(Solyc07g053980)被RNAi沉默[21.[Crispr / Cas9中观察到植物大小的略微减少SLPMR4.突变体。这可能是由于突变体中升高的SA水平。组成型高水平的SA可能导致拟南芥的增长障碍[37.那38.].虽然我们没有测量突变系中SA的水平,但表达PR1基因(作为SA水平的指标)在pm侵染的叶片中增加SLPMR4.突变体与含有WT等位基因的对照相比SLPMR4..
T2家族TV171370的八个人中有三个患有矮化表型。我们试图确定这种矮化表型的原因。没有植物在SGRNA6靶位点显示额外的突变SLPMR4.或者在Solyc02G078230中可能的离目标网站(SLPMR4-H2))。此外,所有8种植物对Cas9基因呈阳性。因此,确定了患矮化植物的明显原因。但是,T-DNA集成的网站仍然可能发挥作用。当CRISPR / CAS9构建体的T-DNA集成在影响植物生长的基因内时,仅当T-DNA插入以纯合状态存在时才获得矮化植物。另一种可能性是T-DNA的多种集成在TV171370的父母T1植物中发生,其中一个是植物发育的关键基因。
由于侏儒植物仅在一个T2家族中发现而不是在任何其他家族中,这种表型似乎并不是由突变引起的SLPMR4.基因。已经讨论了易感线丧失损失线的可能的渗透效应作为部署的挫折S.-植物育种中的基因[9.].显然,在标准温室条件下进一步的表型分析和产量估算将决定抗性的增加是否PMR4.番茄植物还携带植物的健身成本。
在淘汰赛中抵抗PMSLPMR4.植物
我们旨在调查是否完全敲门SLPMR4.将导致比RNAi沉默的转基因植物更高的抵抗水平[21.]。在这项研究中,我们已经表明我们的SLPMR4.与含有WT等位基因的对照植物相比,易感品种金融制造商背景的突变体显示出增强的番茄PM抗性SLPMR4.,但不完全抵抗。我们在10-17dpi评估了疾病指数的植物(图。4.和6.)在21 dpi下确定真菌生物量。可以认为突变体中的易感性不会减少但延迟。但是,我们保持接种的PMR4.突变体直至30-35dpi,仍然植物仍显示出比WT对照植物更少的感染。因此,这是PMR4.突变体显示对PM的易感性降低。
与拟南芥以前的观察结果一致PMR4.突变体[14.]我们观察到番茄Crisprpr的感染遗迹更高的HR样细胞死亡PMR4.与野生型植物相比的突变体。这种增加的HR可能是由SA信号传导途径的激活产生的,如PR1基因表达明显增加PMR4.突变体与对照相比。然而,这种细胞死亡并没有完全阻断番茄的真菌生长PMR4.突变体。在大麦,RNAi用于下调基因HVGSL6.,这是最接近的orthologATGSL5.(PMR4.)[39.].正如预期的那样,这导致了较低的乳头状和伤口胼胝的积累。然而,与在拟南芥和番茄中发现的相反,植物的沉默HVGSL6.导致大麦到PM的更高易感性Blumeria Graminis.f . sp。HORDEI.与控制线相比。有人发现沉默HVGSL6.不会激活sa依赖的防御通路。是否降低了对白粉病的敏感性PMR4./GSL5.突变体是植物物种特异性现象仍有待研究。我们还无法检查在Crisprpr突变体中真菌渗透部位附近的细胞壁上的调用胼ise,尽管与野生型植物相比,我们期望缺席或较低的胼une,具体取决于胼sheglease的冗余基因。为了验证番茄中是否观察到PM的电阻PMR4.突变体是肯定的,应在PM攻击后未来的实验中包含胼舌沉积的量化。
在番茄中另一个可能的同系物PMR4.,指定SLPMR4.-H2(solyc02g078230)已被发现[21.].该基因是与拟南芥At4g04970最接近的番茄同源基因[21.],也被称为GSL1/CALS11.solyc07g053980(GSL5/CALS12-like)和solyc02g078230(GSL1/CALS11据报道,克状物在病原体感染期间在胼uy形成中的作用[40].研究这些基因是否显示出与PM电阻/易感性有关的功能冗余,因此是否同时爆震两种基因会导致PM的抗性更高。但是,GSL1和GSL5也在植物开发中发挥冗余角色[15.]。在拟南芥中,一个GSL5.具有一个突变等位基因的突变体GSL1严重发育不足,展示了高度降低的生育率,不能获得双突变体[15.].
角度为SLPMR4.抗抗抗PM抗性的突变体
我们改变了易感番茄品种的钱制造商SLPMR4.CRISPR构建,并观察到获得的与SA信号通路激活相关的突变体的易感性降低。想知道会有什么影响SLPMR4.突变将在不同的遗传背景中,特别是在抗性番茄背景中,目的是金字塔不同的抗性基因。由于抵抗赋予PMR4.突变与升高的SA途径防御响应相关,我们预计PM在番茄线上的抵抗力OL-4要么OL-6.不会受到突变的影响SLPMR4.基因。OL-4和OL-6.编码NB-LRR型电阻基因[41.[含有这些基因的线在PM感染后显示出快速的HR。这种强烈的抵抗力不会变得更强大,因为SA水平进一步提升PMR4.突变。抗性基因OL-1那ol-3和ol-5尚未克隆但含有这些基因的线显示出慢的人力资源响应。可能,将这些基因中的一种与之结合在一起PMR4.突变可能导致更强或更快的人力资源响应。PM阻力由隐性基因赋予ol-2,含有突变SlMlo1基因,与乳头形成的形成和愈合沉积增加。分析双突变体中的PM电阻水平将是有趣的SLMLO1 SLPMR4..阻力赋予ol-2当胼胝质合酶基因SLPMR4.是突变的,并且与另一个胼胝质合酶基因(例如。SLPMR4.h2)。然而,在拟南芥中ATMLO2 ATPMR4.双突变体对PM表现出相同水平的抗性G. Orontii.作为单一ATMLO2突变体(42.].因此,这是ATMLO2抗性与pmr4介导的胼胝质沉积无关[42.].在番茄和番茄中是否相同SLMLO1 SLPMR4.双突变体仍然表现出与单突变体相同的抗性水平slmlo1.(ol-2)突变体仍有待测试。
结论
指某东西的用途S.-基因由于其耐久和广谱特性,在植物育种中是一个很有前途的选择。在本研究中,我们使用CRISPR/Cas-9靶向突变来敲除S.-基因PMR4.在番茄中。我们以五种不同的突变事件为特征,并确认了突变线对抗的降低在。我们的研究表明CRISPR / CAS9系统的效率和多功能性作为学习和表征的强大工具S.基因。
方法
GRNA和转型设计
番茄的全长CDPMR4.从Sol基因组学网络数据库中检索同源物(solyc07g053980)[43.].使用Liang等人描述的指导来选择靶向基因的四个单一指导RNA(SGRNA)。[35.](补充表1).使用CC-Top CRISPR/Cas9靶标预测工具生成了第一个grna列表[44.].使用EndMemo WebTool计算SGRNA的G + C含量(http://www.endmemo.com/bio/gc.php.).使用MFOLD Web服务器预测GRNA的折叠[45.].另外,使用SGRNA分机器预测GRNA的活性[46.].选择4个sgrna (sgRNA1:TTAAAGCAGTCCCATACTCG, sgRNA6: GTACTGCCCCACACTCTGCG, sgRNA7: GCCAAGGTTGCCAGTGGCAA, sgRNA8: ggatcagagaaggatcag)进行转化。利用从Addgene中获得的12个质粒对sgRNAs的位置和预测蛋白的拓扑结构进行分析,并构建用于转化的构建物:pICH86966(作为扩增模板);pICSL01009(作为0级质粒);pICH47751、pICH47761、pICH47772、pICH47781、pICH47732(为1级质粒);pICH41766、pICH41780和pICH41822(作为连接器);pAGM4723(作为二级二进制矢量)(补充图3.).克隆质粒使用大肠杆菌DH5α转化为农杆菌菌株AGL1。根据[中的方法,使用易感番茄品种金钱制造商(来自植物育种种子收集)进行转化。47.根据荷兰法律,转基因许可证01-135。
基于PCR的选择SLPMR4.突变体和突变事件的特征
携带缺失的植物的选择SLPMR4.通过分析SGRNA靶区域的PCR产物来完成。PCR使用引物FW519(5'- TGGTGCTTTTTTCTCGGGGGGGGGGGGGGGGGGGGGGGGGGCGGGCGGGCGGCATCA-3)产生1407-BP产品,其侧翼为WT等位基因的SGRNA6靶,PCR与引物FW2969(5'-GCGAATGCGTAGAGAGAGAAGAAGAAA3')和RV4230(5'-CCCCactaAGTGCCGTAA-3')产生1262-BP PCR产物侧翼SGRNAS8,1和7,用于WT等位基因。与WT等位基因相比,转基因植物中扩增片段的较小尺寸表明靶向区域中的缺失。SGRNA6目标站点与PAM网站一起包含限制酶的识别序列XCM.I. FW519 + RV1925 PCR产物用这种酶(新英格兰生物Biolabs)消化了1069-BP和WT的338-BP片段SLPMR4.等位基因。测序PCR产物以进一步表征突变事件。使用这些方法选择携带突变等位基因的初级转化体(T1)并自拍以产生T2后代。选择来自两条T2双位线(TV171030和TV171033)的纯合植物用于疾病测定。选择衍生自其他单或双等位基因T1转化体的纯合突变体T2植物并自行获得T3后代。其中包括TV161212-U,TV161212-L,TV171009-U,TV171009-L和TV171010(如果您代表琼脂糖凝胶中的PCR产物的上部带,L用于下频)。在疾病测定中还测试了来自个人T2植物和T3突变线(TV171367,TV171367,TV171366,TV17131366,TV171356,TV171370和TV171371)的电视171355,TV17131370和TV171371)也在疾病测定中进行测试。
偏离目标分析
程序CAS-DECORINDER [34.] 在http://www.rgenome.net/cas-offinder/用于检查SLPMR4..不匹配号设置为3或更小。分析基因SolyC02G078230中可能的脱靶突变(SLPMR4.-H2)使用引物PMR4_H2_FW1(5'-AACGTGTTTCTTTCTGATC-3')和PMR4_H2_RV1(5'-CAAAGTGGCTGCGAGAGADA-3')进行PCR,得到765bp的PCR产物SLPMR4.h2等位基因。聚合酶链反应的产物SLPMR4.测序突变体并与WT对照序列进行比较。
疾病测定和相对真菌生物质的定量
10种常合的T2系中的等位基因和来自纯合T3系的十种植物的等位基因接种了Wageningen大学孤立的在通过喷洒4个星期的植物,悬浮从受感染的番茄金钱制造厂植物的叶子获得的Conidiospores,并调整为3.5 * 10的浓度4.以携带野生型等位基因的转基因株系TV161196和TV161209为对照。接种的植株在荷兰瓦赫宁根大学Unifarm的温室中生长,温度为20±2℃,相对湿度为70±15%,日照时间为16小时。接种后10、12 d进行疾病指数评分。白粉病症状采用白等人描述的0 - 3级量表直观评分[7.].为定量测定真菌相对生物量,采用适应的CTAB协议,从21 dpi采集的感染叶片中分离出植物和真菌基因组DNA [48.].10ng DNA用作扩增的模板。通过使用底漆对的基于FW / ON-REV放大内部转录的间隔序列(其)通过实时PCR来定量相对真菌生物量。Oidium neolypopersici[49.]和EF-FW(5'-GGAacttgagaAgGagcCtraAG-3')/ EF-Rev(5'-caacaCaacaacaacagtct-3')扩增番茄参考基因伸长因子1α.(EF1α.)[50.].2-ΔΔct方法 [51.那52.用来计算真菌和番茄GDNA之间比率的折叠变化。
组织学分析
每条线的至少两种植物与上述疾病测定中的植物一起生长,但使用较重的接种3 * 10感染5.每毫升孢子。感染4厘米的叶样品2接种后44小时,在1:3(v / v)乙酸/乙醇溶液中漂白,并通过沸点在萘酚(1:2 v / v)溶液中煮沸在0.005%台盼蓝中染色48h3-5分钟,并在几乎饱和水溶液中清除,如[含量)(5:2W / v)所描述的[53.].将载玻片安装在玻璃载玻片上,含有1:1(v / v)甘油 - 水溶液。使用Zeiss Axiophot明亮场显微镜进行幻灯片的分析。用于定量真菌结构和宿主细胞死亡50感染单位(O. Neolypopersici每种基因型分析Conidia),从每种基因型两种不同植物获得的两种载玻片分析。
分析PR1表达
番茄的表达水平PR1用QPCR感染PM感染后44 HPI测量基因。来自代表所有五种突变事件和对照植物的单个纯合突变体T2植物(MM和不成功的转基因TV161196和TV161209)中取出感染的叶样品。将叶样品在液氮中冷冻,并在使用杵和砂浆研磨成细粉末之前储存在-80℃。使用Magmax TM 96总RNA分离试剂盒(Qiagen,德国)进行总RNA分离。使用IScript TM cDNA合成试剂盒(Bio-rad Laboratories,U.S.)进行cDNA合成。10 ng cDNA用作反应的模板。表达水平PR1使用引物SLPR1A_FW(5'-GTGTCCGAGAGAGGCCAGAGA-3')和SLPR1A_REV(5'-CattGTGCAACGCCCCCC GA-3')测量,并与番茄的表达相比EF1α.使用引物的参考基因EF_FW(5'-AttggaaAcggatatGcccct-3')和EF_REV(5'-TCCTTACCTGAACGCCTGTCA-3')[21.].
可用性数据和材料
本研究中的Sanger测序获得的序列可在GenBank数据库中获得(https://www.ncbi.nlm.nih.gov/genbank/)在参考附加号码MT521499到MT521504。本文生成的数据包含在稿件和附加文件中,并且原始数据可以从相应的作者获得合理的请求。
缩写
- CRISPR:
-
集群定期间隔的短文重复
- CAS:
-
CRISPR相关系统
- DPI:
-
接种后的天数
- HPI:
-
接种后的小时
- 零:
-
近代线
- 在:
-
Oidium neolypopersici
- 帕姆:
-
Protospacer相邻的图案
- 下午:
-
白粉病
- PMR4.:
-
粉状霉菌抵抗4
- PTTC:
-
过早翻译终止密码子
- R.基因:
-
电阻基因
- SA:
-
水杨酸
- SGRNA:
-
单引导RNA
- S.基因:
-
易感性基因
- WT:
-
野生型
参考
- 1。
琼斯H,Whipps JM,Gurr SJ。番茄粉霉素霉菌真菌Oidium neolypopersici.莫尔植物途径。2001; 2:303-9。
- 2。
Lebeda A, Mieslerová B, Petřivalský M, Luhová L, Špundová M, Sedlářová M, Nožková-Hlaváčková V, Pink DAC。野生番茄种质对番茄红素侵染的抗性机制Oidium neolypopersici.EUR J植物途径。2014; 138:569-96。
- 3。
Seifi A,Gao D,Zheng Z,Pavan S,Faino L,Visser RGF,Wolters AMA,Bai Y.促进番茄粉末状霉菌的遗传和分子机制(Solanum lycopersicum.)和它的野生亲戚。EUR J植物途径。2014; 138:641-65。
- 4.
Bai Y,Van der Hulst R,Bonnema G,Marcel Tc,Meijer-Dekens F,尼克斯Re,Lindhout P.番茄防御Oidium neolypopersici: 主导的Ol基因通过不同的机制赋予隔离依赖性电阻而不是隐性ol-2.Mol植物微生物相互作用。2005; 18:354-62。
- 5。
黄建中,叶根华,梅杰-德kens F, Niks RE, Lindhout P.抗白粉病(oidium lycopersicum.) 在Lycopersicon.物种主要与过敏反应有关。EUR J植物途径。1998年; 104:399-407。
- 6。
李C,Bonnema G,Che D,Dong L,Lindhout P,Visser R,Bai Y.对番茄粉状霉菌的单一抗性反应中的生化和分子机制。Mol植物微生物相互作用。2007; 20:1161-72。
- 7。
白益,帕班班S,Zheng Z,Zappel NF,ReinstädlerA,Lotti C,De Giovanni C,Ricciardi L,Lindhout P,Visser R,Theres K,Panstruga R.天然存在于中美洲番茄中的广谱粉末状抗性加入是由损失引起的mlo.函数。植物与微生物互动学报2008;21:30-9。
- 8。
Kusch S,Panstruga R.mlo.基于抵抗力:一种显然普遍的“武器”,以击败白粉病。植物与微生物互动学报2017;30:179-89。
- 9。
van Schie Ccn,Takken Flw。易感性基因101:如何成为一个好主持人。Annu Rev phytopathol。2014; 52:551-81。
- 10。
Pavan S,Jacobsen E,Visser RGF,Bai Y.易受耐用和广谱抗性的新型育种策略。mol品种。2010; 25:1-12。
- 11.
Gawehns F,Cornelissen BJC,Takken Flw。植物生先生免疫育种育种靶基因的潜力。Microb BioTechnol。2013; 6:223-9。
- 12.
陈志强,陈志强,陈志强,等。水稻白粉病抗性的研究进展拟南芥突变体。美国国立科学学院2000;97:1897-902。
- 13。
Jacobs Ak,Lipka V,Burton Ra,Panstruga R,Strizhov N,Schulze-Lefert P,Fincher GB。rabidopsis召唤合成酶GSL5是伤口和乳头状核糖形成所必需的。植物细胞。2003; 15:2503-13。
- 14。
Nishimura Mt,Stein M,Hou Bh,Vogel JP,Edwards H,Somerville SC。呼吸愈合合酶导致水杨酸依赖性抗病性。科学。2003; 301:969-72。
- 15.
两种胼胝质合成酶GSL1和GSL5在植物和花粉发育和育性中起着重要和多余的作用。中国农业科学(英文版)。
- 16。
陈晓燕,金建勇。高等植物中胼胝质的合成。植物信号行为。2009;4:489-92。
- 17。
Schneider R,Hanak T,Persson S,Voigt CA。纤维素和核糖综合综合,什么是新的?Curr Op植物BIOL。2016; 34:9-16。
- 18。
Ellinger D,Naumann M,Falter C,Zwikowics C,Jamrow T,Manissi C,Somerville SC,Voigt Ca.升高的早期胼舌沉积导致拟南芥粉末状霉菌的完全抗渗透性抗性。植物理性。2013; 161:1433-44。
- 19。
Eggert D,Naumann M,Reimer R,Voigt Ca.纳米级葡聚糖聚合物网络导致病原体抗性。SCI REP。2014; 4:4159。
- 20.
Eichmann R,HückelhovenR.完整植物细胞中粉状霉菌的适应。J植物理性。2008; 165:5-18。
- 21.
Huibers Rp,Loonen Aehm,Gao D,Van Den Ackerveken G,Visser RGF,Bai Y.番茄中的粉状抗性造成的损伤SLPMR4.和SlDMR1.《公共科学图书馆•综合》。2013;8:e67467。
- 22.
Langner T,Kamouns,Belhaj K. Crispr作物:植物基因组对抗病性进行编辑。Annu Rev phytopathol。2018; 56:479-512。
- 23.
Boettcher M,McManus MT。为工作选择合适的工具:RNAi,Talen或Crispr。Mol细胞。2015; 58:575-85。
- 24.
基于CRISPR和rnai的筛选技术比较。生物科技Nat》。2016;34:621-3。
- 25.
Nekrasov V,Wang C,Win J,Lanz C,Weigel D,Kamoun S.通过基因组缺失快速产生转基因无粉状霉菌抗性番茄。SCI批准。2017; 7:482。
- 26.
张y,白益,吴g,zou s,chen y,gao c,唐d。同时修饰三个同源性taedr1.通过基因组编辑提高小麦的抗白粉病能力。植物j . 2017; 91:714-24。
- 27.
Peng A,Chen S,Lei T,Xu L,他,吴L,姚L,Zou X.工程溃疡植物通过CRISPR / CAS9靶向编辑易感基因cslob1.柑橘的启动子。植物Biotechnol J. 2017; 15:1509-19。
- 28.
关键词:ERF转录因子,CRISPR/ cas9,基因突变,水稻抗稻瘟病OSERF922..《公共科学图书馆•综合》。2016;11 (4):e0154027。
- 29。
谢凯,杨志强,杨志强。利用内源性trna加工系统增强CRISPR/Cas9多重编辑能力的研究。中国科学院院刊。2015;112(11):3570-5。
- 30.
高效的多重基因组编辑诱导番茄植株的精确和自结扎型突变。植物学报。2018;9:916。
- 31。
Omasits U, Ahrens CH, Müller S, Wollscheid B. Protter:交互蛋白质特征可视化和与实验蛋白质组数据的集成。生物信息学。2014;30:884-6。
- 32。
冈田H,Abe M,Aakawa-Minemura M,Hirata A,Qadota H,Morishita K,Ohnuki S,Nogami S,OHYA Y.酵母1,3-β-葡聚糖合成酶亚单位FKS1p的多功能结构域通过定量表型分析揭示温度敏感突变体。遗传学。2010; 184:1013-24。
- 33。
INOUE SB,Takewaki N,Takasuka T,Mio T,Adachi M,Fujii Y,Miyamoto C,Arisawa M,Furuichi Y,Watanabe T.表征和基因克隆1,3-β-D-葡聚糖合成酶酿酒酵母酿酒酵母.EUR J Biochem。1995年; 231:845-54。
- 34。
裴s, Park J, Kim JS。Cas-OFFinder:一种快速、通用的算法,用于搜索Cas9 rna引导内切酶的潜在脱靶位点。生物信息学。2014;30:1473-5。
- 35。
梁光国,张洪波,娄丹丹,于丹。基于CRISPR/ cas9的植物基因组编辑高效sgrna的筛选。Sci众议员2016;6:21451。
- 36。
利用CRISPR/Cas系统高效诱导植物基因组的遗传转位。植物j . 2019; 98:577 - 89。
- 37。
Heidel AJ,Clarke JD,Antonovics J,Dong X.影响系统所获得的电阻途径的突变的健身成本拟南芥蒂利亚纳.遗传学。2004; 168:2197-206。
- 38。
Šašekv,janda m,delage e,puyaubert j,guivarc'h a,lópezmasedae,dobrev pi,caius j,bókak,volyváo,burketovál,zachowski a,zachowski a,ruelland E.组成型水杨酸积累pi4kiiiβ1β2拟南芥植物特技莲豆,但不是根生长。新植物。2014; 203:805-16。
- 39。
Chowdhury J,Schober Ms,Shirley NJ,Singh Rr,Jacobs AK,Douchkov D,Schweizer P,Fincher GB,Burton Ra,小A.下调葡聚糖synthase-like 6基因(HVGSL6.)导致胼胝质积累减少,细胞壁穿透增加Blumeria Graminis.f . sp。HORDEI..新植醇。2016;212:434-43。
- 40。
adkar-purushothama cr,brorseau c,giguèret,sano t,moffett p,perreault jp。来自马铃薯主轴块茎病原体沉默的毒力调节区域的小RNA调用番茄植物的合成酶基因。植物细胞。2015; 27(8):2178-94。
- 41.
Seifi A, Kaloshian I, Vossen J, Che D, Bhattarai KK, Fan J, Naher Z, Goverse A, Tjallingii WF, Lindhout P, Visser RGF, Bai Y.链接,如果不一样,MI-1同源物赋予番茄粉状霉菌和根结线路酸的抗性。Mol植物微生物相互作用。2011; 24:441-50。
- 42.
辅音C, Bednarek P, Humphry M, Francocci F, Ferrari S, Harzen A. Ver Loren van Themaat E, Panstruga R.色氨酸衍生的代谢物在拟南芥的抗真菌防御中是必需的mlo2.突变体。植物理性。2010; 152:1544-61。
- 43.
Fernandez-Pozo N,Menda N,Edwards JD,Saha S,Tecle Iy,Strickler SR,Bombarely A,Fisher-York T,Pujar A,Foerster H,Yan A,Mueller La。溶胶基因组学网络(SGN) - 从基因型到育种表型。核酸RES。2015; 43:D1036-41。
- 44.
Stemmer M,Thumberger T,Del Sol Km,Wittbrodt J,Mateo JL。CCTOP:直观,灵活可靠的CRISPR / CAS9目标预测工具。Plos一个。2015; 10:E0124633。
- 45.
Zuker M. Mfold Web服务器用于核酸折叠和杂交预测。核酸RES。2003; 31:3406-15。
- 46.
Chari R, Yeo NC, Chavez A, Church GM. SgRNA评分器2.0:预测CRISPR/Cas9活性的物种独立模型。ACS合成器。医学杂志。2017;6:902-4。
- 47.
McCormick S,Niedermeyer J,Fry J,Barnason A,Rossch R,Franey R.栽培番茄的叶片转换(L. Esculentum.)使用根癌土壤杆菌.植物细胞代表1986; 5:81-4。
- 48.
Doyle JJ,Doyle JL。少量新鲜叶组织的快速DNA隔离程序。Phytochem Buart。1987; 19:11-5。
- 49.
郑志刚,李志刚,李志刚,李志刚。番茄全基因组研究进展slmlo.响应白粉病真菌的基因家族及其功能特征Oidium neolypopersici.前植物SCI。2016; 7:380。
- 50。
Lilo C. LILLO C.番茄中定量实时PCR标准化的参考基因选择,对氮气,冷,轻应力进行定量实时PCR标准化。肛门生物化学。2009; 387(2):238-42。
- 51。
Livak KJ,Schmittgen TD。使用实时定量PCR和2分析相对基因表达数据——ΔΔCT.方法。方法。2001; 25:402-8。
- 52。
Pfaffl兆瓦。一种新的实时RT-PCR相对定量数学模型。核酸学报2001;29:2002-7。
- 53。
小麦叶锈菌的预吸器抗性,PUCCINIA TRITICINA, 在小麦属植物monococcum(砂岩)。Euphytica。2001;117:209-15。
确认
在真菌生物量量化期间,我们感谢何萌为实验室和温室的工作。我们感谢Fien Meijer-Dekens为她提供帮助照顾Crispr植物和Katharina Hanika的帮助,让她的帮助表现。4..我们感谢匿名审稿人有用的评论,以改善稿件。
资金
MISM是由墨西哥国家科学技术理事会(National Council for Science and Technology CONACYT)的一项研究资助的。该资助机构没有在研究的设计、数据的收集、分析和解释以及手稿的撰写中发挥作用。
作者信息
从属关系
贡献
MISM参与了实验设计、突变体的鉴定、筛选和测试、结果解释和手稿起草等工作。用VB设计了转换结构、生成、测试并对突变体进行了初步表征。EK和MA参与突变事件的选择和表征、表达分析、组织学分析和疾病检测。EJ、RGFV和YB提出了实验的想法。AMAW参与了对结果的解释。AMAW和RGFV对这篇论文进行了严格的审查。AMAW和YB编辑了手稿。所有作者阅读并批准了最终的手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。YB是BMC植物生物学编辑委员会的成员。
附加信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:补充文件1。
番茄PCR产物序列的对准PMR4.CRISPR突变等位基因。A.通过使用引物FW2969和RV4230用于野生型MoneyMaker(mm)和五个的PCR产物的序列比对PMR4.CRISPR变异事件。表示引物和SGRNA1,7和8。在红色的中,显示了不同形式的核苷酸。删除由破折号表示。事件5包含一个大的反转,以红色表示。B.图中显示了突变事件5中sgRNA8和sgRNA7与MM相比的序列倒置(蓝线)。
附加文件2:补充图1。
番茄预测蛋白的对准PMR4.CRISPR突变等位基因。蛋白质序列是基于引物Fw2969和Rv4230两侧区域扩增的片段的DNA测序数据。
附加文件3:补充图2。
感染时的表型Oidium Neolypopersici。来自野生型等位基因的携带控制和不同的植物SLPMR4.显示突变类。野生型植物的叶片存在重型真菌孢子,而突变植物的叶片会看到较少的感染。
附加文件4:补充图3。
CRISPR/Cas9转化的2级载体图。NPTII、Cas9、四种sgrna和AtU6启动子被重点强调。
附加文件5:补充表1。
四个选定的SGRNA的特征SLPMR4..使用CCTOP程序选择SGRNA。Pam,Protospacer相邻的主题。
附加文件6:补充表2。
SLPMR4.sgRNAs不相干。sgRNA8和sgRNA7与Cas-OFFinder发现最大3个不匹配的脱靶[34.].CRRNA,SGRNA序列。DNA,脱靶序列。不匹配以红色表示。
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
SantillánMartínez,M.I.,Bracuto,V.,Koseoglou,E。等等。CRISPR / CAS9靶向番茄敏感性基因的诱变PMR4.用于对粉末状霉菌的抵抗力。BMC植物BIOL.20.284(2020)。https://doi.org/10.1186/s12870-020-02497-y
已收到:
公认:
发表:
关键词
- CRISPR / CAS9.
- 有针对性的诱变
- PMR4.
- 白粉病
- 易感性基因