摘要
背景
海岛棉(海岛棉)具有明显优良的优质纤维,在纺织工业中发挥重要作用,是陆地棉(g .分子)纤维品质改善。通过对遗传特性的分析和关键基因的鉴定,有助于了解海岛棉纤维的发育机理和育种利用。
结果
本研究对279份不同产地的海岛棉材料进行了纤维品质性状的基因分型和表型分析。利用高密度棉花snp80k序列获得6303个高品质单核苷酸多态性(SNPs)。群体特征表明,海岛棉种质具有广泛的遗传多样性,可分为3个类群,其中类群1与原海岛棉地方品种Menoufi亲缘关系密切,类群2和类群3与从埃及、美国和前苏联广泛引进的品种亲缘关系密切。利用249份材料,在正常和盐环境下对5个品质性状进行了2年多的评价。盐条件下,除纤维均匀性(FU)外,纤维长度(FL)和伸长率(FE)显著降低,纤维强度(FS)和马克隆值(MIC)显著提高。基于6303个单核苷酸多态性和全基因组关联分析(GWAS),共鉴定出5个纤维品质性状的34个稳定数量性状位点(qtl),其中25个在正常和盐环境下同时检测到。基因本体论(GO)分析表明,25个重叠qtl中的候选基因主要富集于“细胞和生物学过程”。此外,还发现了“木质部发育”和“激素反应”途径。单倍型分析发现GB_A03G0335在QTL TM6004中编码E3泛素蛋白连接酶的基因区存在SNP变异(A/C),与FL、FS、FU和FE显著相关,提示其在纤维品质中起关键作用。
结论
本研究为海岛棉棉加工遗传多样性提供了遗传多样性,并有助于纤维质量改善育种实践。
背景
棉(Gossypium是为纺织工业生产天然纤维的最重要作物之一。陆地棉(g .分子)及海岛棉(g .取得)两种主要栽培的四倍体物种占棉花总产量的97%以上[1].陆地棉产量高,纤维质量中等,而海岛棉纤维质量特别高[2].随着纺织工业的快速发展,对纺织产品的需求不断多样化,棉纤维质量的提高越来越重要[3.].染色体片段的渐渗可以有效地将陆地棉的产量优势与海岛棉的品质提升结合起来[4].因此,研究其染色体的优良片段具有重要的意义g .取得用于育种中提高棉纤维品质。
对目标性状的qtl定位和标记辅助选择对棉花品种的选育起到了重要的促进作用。随着分子标记、SNP序列和重测序技术的发展,已探索出一批与陆地棉纤维产量、品质和耐盐性相关的qtl。Shen et al.(2005)利用SSR标记鉴定了陆地棉纤维品质性状相关的39个qtl [5].Sun et al.(2018)通过Illumina CottonSNP63K序列和62个SNP位点对719份陆地棉花种质资源进行基因分型,发现其与产量相关性状显著相关[6].通过特异性基因座扩增的片段测序(SLAF-SEQ),SU等人。(2016)在四种不同环境中检测到与Ulpland棉的棉绒百分比相关的12个SNP [7].在耐盐性状方面,鉴定出8个和23个SNPs与耐盐相关性状显著相关[8,9].这些研究为开发耐盐高产陆地棉品种提供了qtl和基因资源。
相比之下,g .分子在我国,利用自然居群对目标性状的遗传多样性和QTL挖掘研究较少g .取得.此外,新疆是我国海岛棉的主要产区,但新疆耕地多为盐碱地[10].盐度对纤维产量和质量构成严重威胁[11].在陆地棉花中,Na含量较高+土壤中微量元素的含量会降低纤维长度、强度和马克隆值[12].然而,有关海岛棉的报道却不多见。在此基础上,系统研究了279份不同产地海岛棉种质资源在不同土壤环境下2年的群体结构基因分型和纤维品质表型。通过结合两组数据,GWAS分析确定了与纤维品质相关的稳定qtl和候选基因。这一发现将为棉花纤维品质改良提供优质的qtl候选基因。
结果
基于SNPs的遗传变异
279个海岛棉的加入(附加文件1:表S1)使用棉花NP80K阵列进行基因分型。基因型数据透露,这些棉花加入具有96.54%的高平均呼叫率(附加档案2:图S1)。进一步筛选了呼叫频率< 80%、次要等位基因频率(MAF) < 0.01或映射到染色体多个位点的基因座。最终获得6303个SNPs,其中At亚基因组上的SNPs为3656个,Dt亚基因组上的SNPs为2647个3.:图S2)。为评价基因分型的准确性和重现性,对8份不同来源的棉花材料进行生物重复分型分析。基因分型的一致性在86.3 ~ 99.8%之间,表明同一种质在不同来源间存在异质性。其中不同来源的海7124和新海26的一致性最高,分别为99.8和99.3%。279份材料的多态信息含量(PIC)在染色体间的变化范围为0.189 ~ 0.469,At和Dt亚基因组的PIC平均值分别为0.391和0.338(附加文件)4:表S2)。结果表明,利用CottonSNP80K基因序列对海岛棉材料进行基因分型具有较高的效率和准确性。
人口特征分析
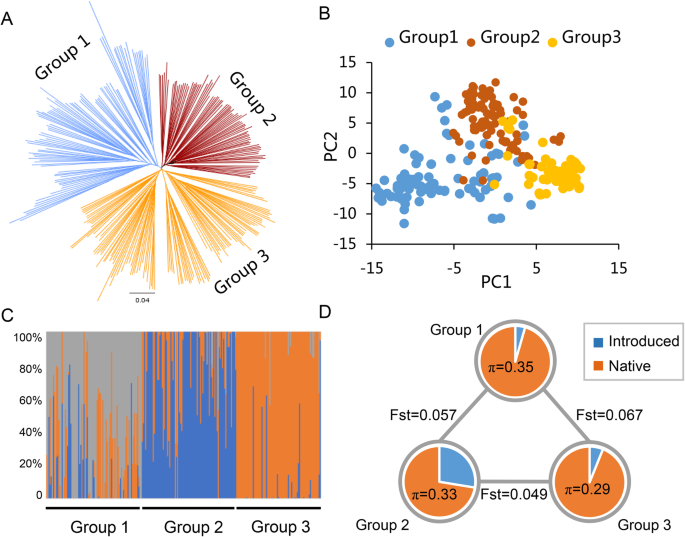
为了探讨海岛棉棉花的人口特征,使用Tassel 5.0进行邻近的树。它表明,279种载体可以聚集成三组,其分别包含87,112和80,分别包含87,112和80个(图。1一种)。早些时候介绍了中国的39个地位,并为中国海岛棉花养殖贡献,广泛分布在三组。Group1载有三个引入的介绍,Giza 36,Giza 81和Menoufi,他们都来自埃及,Menoufi是一个原来的海岛棉花兰德。Group2和Group3集团与埃及,美国和前苏联的广泛引进的院长集中在一起,31次介绍了Group2和Five的第2组介绍。PCA(图。1b)和种群结构分析(图。1C)也支持279个加入可以聚为三组,这与邻居连接树很好。我们通过计算核苷酸多样性(π)值来测量每个组的遗传多样性(图)。1d),发现组1的遗传多样性最高(π = 0.35),组3的遗传多样性最低(π = 0.29)。此外,人口固定统计(FST.)表示FST.组1与组2之间、组1与组3之间的值大于组2与组3之间的值(图3)。1d),暗示Group1与其他两组相比具有最高的分化。
纤维品质性状的表型变异
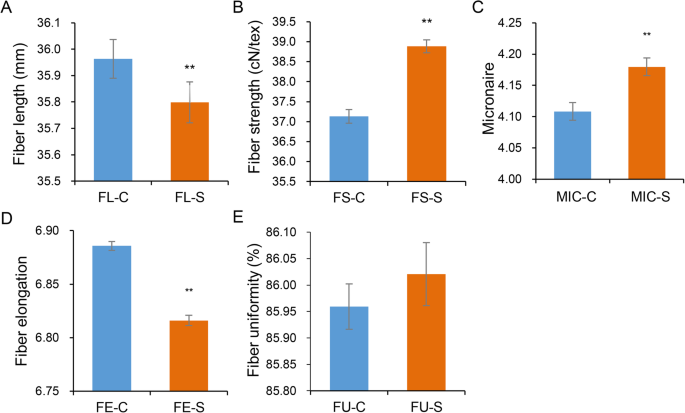
为了评价群体中纤维品质性状的表型变异,我们在2年多的时间里分别对5个性状进行了分析。结果表明,自然种群的变异范围较广,FL为29.35 ~ 39.68 mm, FS为28.29 ~ 48.10 cN/tex, MIC为3.10 ~ 5.23,FU为81.75 ~ 88.65%,FE为6.52 ~ 7.12%1).MIC和FS变异系数最大,分别为6.28 ~ 8.31%和8.53 ~ 10.91%,其次为FL(4.27 ~ 5.35%)。FE和FU的变异系数最小,分别为1.04 ~ 1.48%和0.93 ~ 1.06%。密度分布表明,正常条件下FL和FE的值分布比盐条件下高。其中,正常条件下FL值高于37,FE值高于6.9的分布要大于盐条件下的分布。FS和MIC在盐条件下的值分布高于正常条件,分别大于32和4。对FU而言,盐条件下的分布比正常条件下更集中(附文件)5:图S3)。配对样本t -进一步试验表明,与正常条件相比,盐条件下FL和FE显著降低,分别为0.46和1.01%,FS和MIC分别增加,分别为4.73和1.74%。在两种环境中,FU没有显著差异(图。2).相关分析表明,各性状在正常和盐碱条件下均具有较高的表型相关性。此外,在相同条件下,FL与FS、FE、FU呈显著正相关,FS、FE、FU之间也呈显著正相关。MIC与FL、FS呈显著负相关,与FE呈显著正相关6:图S4)。
全基因组关联研究
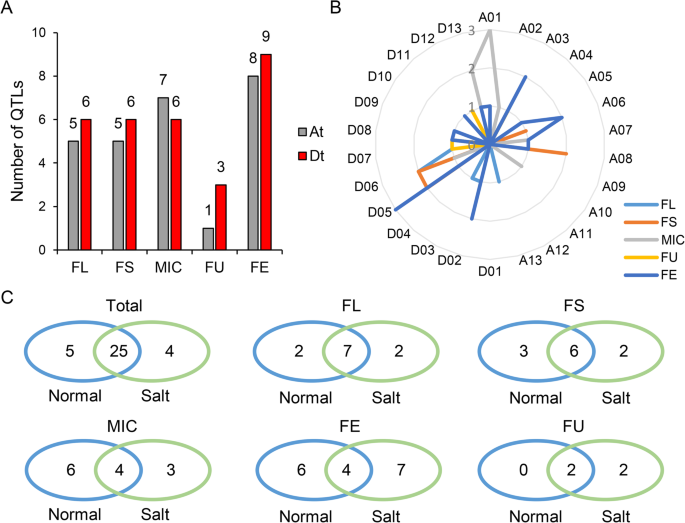
采用多位点混合线性模型对5个纤维品质性状进行GWAS分析[13].在26条染色体上共鉴定出102个数量性状核苷酸(QTNs)与性状显著相关(附加文件)7:表S3)。我们将QTN的侧翼200 KB区域定义为初始QTL,并合并重叠的QTL以获得如前所述的最终QTL [14].总共在不同环境下检测到81个QTL 413次,并通过六个多基因座MLM模型进行最佳线性无偏见的预测器(BLUP)。考虑到QTL在具有较少关联频率的QTL中可能是假的误报,我们选择了一个以上三个关联时间作为稳定QTL的QTL,以进行后续分析(附加文件8:图。S5)。结果,鉴定了34个稳定的QTL,包括九个高频相关QTL,其关联时间大于10(表2和额外的文件9:表S4)。
由于高相关性,还检测到五个特征中的重叠稳定的QTL(附加文件9:表S4)。结果表明,4个性状(FL: 4、FS: 4、MIC: 5和FE: 5)各检测到18个稳定的qtl,在2个或2个以上性状中检测到16个稳定的重叠qtl。其中QTL TM6004与FL、FS、FE、FU同时关联,关联次数最多,达75次。检测到4个与3个性状相关的qtl(附加文件)9:表S4)。这些稳定的qtl广泛分布在At和Dt亚基因组上,而FL、FS、FE和FU在Dt亚基因组上的位点较多(图2)。3.一种)。染色体分布显示MIC和Fe的大多数QTL分别位于A01和D05上(图。3.b).在不同环境下,在正常和盐胁迫条件下也检测到许多重叠qtl(占总qtl的74%),特别是FL和FS的重叠qtl分别为64个和55%(图2)。3.C),表明大部分纤维品质性状遗传力高且稳定。
QTLS中候选基因的鉴定
在这些稳定的QTL区域中提取潜在的候选基因g .取得Hai7124基因组(15].共鉴定出998个候选基因,其中FL 302个,FS 251个,MIC 373个,FU 84个,FE 583个(附加文件)10:表S5)。在两种环境下分别鉴定出118个和95个与纤维品质性状相关的基因,同时鉴定出785个基因。GO分析表明,在盐条件下特别确定的95个基因在“对刺激的反应”(P4.8 × 10−5),而在正常条件下特异识别的118个基因主要参与“细胞过程”。我们发现在正常和盐条件下同时鉴定的785个候选基因主要富集在“细胞过程”和“生物过程”。此外,还检测到“木质部发育”、“对激素的反应”和“胚胎后期发育的调节”11:表S6)。根据组织表达谱,我们进一步鉴定了这些稳定的QTL区域中的572个纤维表达基因,表明它们在光纤开发中发挥作用(附加文件12:表S7)。
纤维品质性状关键基因的挖掘
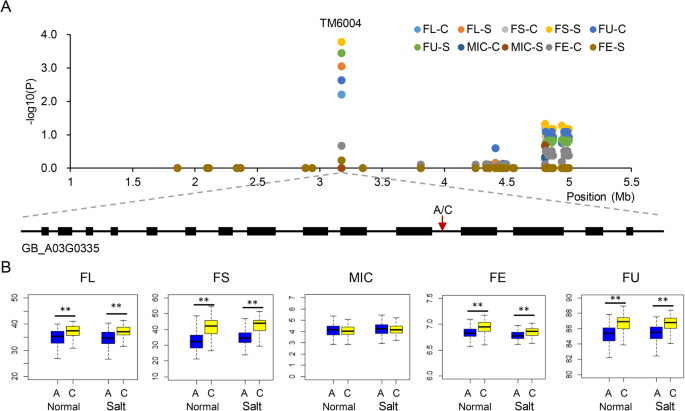
进一步分析9个高频相关qtl,挖掘关键候选基因。QTL TM6004的QTN为4186,816 bp, QTL位于A03: 3,986,816 bp - 4,386,816 bp(图3)。4a),与FE、FL、FS和FU同时存在高频率检测次数,解释了正常和盐环境中19.9%以上的纤维品质性状变异。序列分析表明TM6004的QTN(从A到C)位于该基因的内含子区域GB_A03G0335它编码E3泛素蛋白连接酶。通过学生的t结果表明,与A单倍型相比,C单倍型的FL、FS、FU和FE分别提高了6.9、22.5、1.5和1.2%(图2)。4b),说明QTN TM6004可能在纤维发育中起重要作用。除此之外,另一个候选基因,gb_a03g0342编码漆酶3的基因也被鉴定出来。漆酶在植物的细胞伸长、木质素化和色素沉着中起重要作用,对棉纤维的品质也有重要作用[16].另一个高频相关QTL TM68443位于D08位点:21500,627bp - 21,900,627bp, SNP位点为A到G,在正常和盐环境下与FL、FS和FU同时相关。结果表明,与G单倍型相比,A单倍型的FL、FS和FE分别提高了8.1、26.9和1.5%13:图S6)。在QTL区域,一个基因GB_D08G1034编码一个生长素渠化蛋白,已鉴定。前人研究表明,生长素运输在陆地棉纤维发育中起重要作用[14].此外,我们还发现了5个只与单一性状相关的高频qtl,其中1个与FL相关,2个与MIC相关,2个与FE相关。在这些qtl中,许多基因与纤维发育有关,如GB_D09G2489编码Ca2+- 绕线蛋白1 [17),GB_D09G2484编码氢钠交换器2 [18],是正常或盐环境下纤维开发中的重要基因。
讨论
陆地棉(g .分子)及海岛棉(g .取得)是两种产纤维的四倍体栽培棉花,纤维品质在海岛棉中明显优越。然而,对海岛棉自然群体遗传特性的研究较少。本研究利用6303个优质单核苷酸多态性(SNPs)对279份海岛棉种质资源的遗传多样性和群体特征进行了研究。与以往基于其他分子标记如ISSR的研究相比[19], SRAP [20.]及SSR [21[目前的研究中,SNP标记的数量大大增加。279种载体的平均照片值表明,亚基因组的照片高于DT亚基因组的照片。系统发育树和PCA分析支持,可以将279种载体聚集成三组,类似于基于SSR标记的先前研究[21,22].现代栽培海岛棉主要有埃及型、美洲型和中亚型三种生态型[22].在39个引种的地方品种中,第1组包含3个主要来自埃及的引种,其中原海岛棉地方品种Menoufi也聚在第1组。来自埃及、美国和前苏联的引种材料中,2组和3组有31个,3组有5个,说明原始生态型的1组分化程度最高。目前,现代栽培海岛棉的遗传基础相对狭窄。根据3个群体的遗传多样性,可以将不同群体的材料杂交成与目标性状相关的金字塔型多位点,为今后海岛棉育种提供参考。
盐度被认为是限制作物生长发育的最重要的环境因素之一。在我国,新疆生态区主要种植海岛棉,但该地区大部分耕地为盐碱地。海岛棉在胁迫条件下的研究报道甚少。本研究以海岛棉自然群体为研究对象,选择正常和盐碱地两种不同的田间环境,历时2年对棉纤维品质性状进行系统研究。通过表型分析,我们发现在盐条件下FL和FE显著降低,而FS和MIC增加。此外,在两种环境中没有检测到FU的差异。其中,FS在盐条件下显著增加,且与其他4个性状的变异幅度最大。5个纤维品质性状间的高度相关性进一步支持了上述结果。与先前的研究一致[11,23],在盐胁迫下纤维长度降低,这就要求海岛棉品种在盐碱条件下种植时,纤维品质特别是纤维长度要提高。
通过GWAS分析,共鉴定出34个与5个品质性状相关的稳定qtl,其中包括9个高频相关位点。这些qtl中约有一半与多个性状相关,表型分析表明5个品质性状间的相关性较高。我们发现4个qtl TM27446_TM27440、TM33841_TM33859、TM58446和TM68443与最近报道的海岛棉纤维品质性状相关qtl存在重叠[24].其中TM68443是一个高频关联QTL,其单倍型能显著提高海岛棉的FL、FS和FE。此外,大部分qtl均在正常和盐碱条件下检测到,特别是FL和FS,表明不同环境下纤维品质性状具有较高的遗传力。此外,我们还检测了部分与纤维品质相关的qtl,并发现这些qtl所定位的基因富集于“响应刺激”中,这可能对纤维品质在盐碱条件下的改善起到重要作用。我们还发现在正常和盐条件下同时鉴定的候选基因在“木质部发育”中富集,表明木质部参与了纤维的发育。棉纤维细胞壁中的木质素有效地提供了机械强度,而木质素在海岛棉纤维品质中的作用尚待探讨。
为了进一步挖掘影响纤维品质的关键基因,我们对关联次数超过10次的高频qtl进行了分析。有趣的是,9个高频qtl中有7个在正常和盐条件下都能检测到。QTL TM6004解释了21.6%以上的纤维品质性状变异。在这个QTL区域,该基因GB_A03G0335其编码E3泛素 - 蛋白质连接酶,其内含子区域的QTN变异,表明,与C单倍型相比,FL,FS,FU和Fe显着增加了6.9,22.5,1.5和1.2%分别是单倍型。蛋白质泛素在多种植物发育阶段和几种非生物应激反应中起关键作用[25].据报道,泛素蛋白连接酶也参与了棉花的几个生物学过程。泛素连接酶基因的过度表达,GHHUB2.,增加纤维长度和SCW厚度,而RNAi敲除GHHUB2.导致棉花中缩短的纤维和较薄的细胞壁,涉及泛素-26s蛋白酶体途径[26].泛素E3连接酶RHA2b促进MYB30降解[27],而MYB转录因子被广泛报道与纤维发育有关[28,29].此外,E3泛素连接酶也被报道参与棉花的耐受性,如大丽轮枝菌属[30.],干旱[31]及耐盐性[32].稻瘟病e3连接酶基因的过度表达OsSIZ1在棉花增强的干旱和耐热性,在减少灌溉和雨量养殖条件下,该领域的纤维产量显着提高了纤维产量[33].根据不同营养和纤维发育组织的转录模式g .取得简历。Hai7124,我们还发现了多种类型的转录本GB_A03G0335这意味着该基因GB_A03G0335可能对棉纤维的发育和其他抗逆性有重要作用。但其分子机制尚待进一步研究证实。
结论
探索海岛棉的遗传特性,筛选关键优良位点,对纤维品质改良具有重要意义。本研究系统报道了一套海岛棉自然群体的遗传多样性,并建立了正常和盐环境下5个纤维品质性状的表型和GWAS分析。结果表明,海岛棉种质资源可分为3个类群,遗传多样性较强。5个纤维品质性状在同一环境或不同环境下均存在较高的相关性。基于SNPs和GWAS分析,共鉴定出34个稳定的qtl,其中正常和盐环境下同时检测到25个qtl。单倍型分析发现GB_A03G0335编码E3泛素蛋白连接酶,在纤维品质中起关键作用,并与FL、FS、FU和FE显著相关。这些发现将有助于了解海岛棉的遗传多样性,并有助于在育种实践中提高棉纤维品质。
方法
植物材料
共有279个海岛棉质棉花加入,从中国收集240个品种/线和39线,用于基因分型和人口特征分析,使用249种辅助用于纤维表型分析(附加文件1:表S1)。这些登记入册,11长白猪(吉萨吉萨e24 - 3389, 36岁,45岁,l - 3398 Mi 10,皮马人,皮玛s 3,铁尔梅兹16日Yinzi 6022 504 - n, 9899И)收集国家中期基因棉花在中国,银行和其他收集和保存268到达南京农业大学、新疆农业大学,中国。所有供试材料经南京农业大学、新疆农业大学和国家棉花中期基因库批准种植和调查。
表型调查和数据分析
2017年和2018年,279个海岛棉品种在新疆农业大学实验站2个自然环境下进行种植。对这些植物进行实地评估的所有必要许可均由中国新疆农业大学批准。为了评估田间土壤盐分浓度,在棉花种植前,每个环境在15-30 cm深度采集9个采样点的土壤样品,并使用Singh等人(2017)描述的方法分析电导率(EC)。[34].平均土壤EC分别为0.5和2.7ds / m,分别标记为正常和盐环境。每次加入都在两排中生长,在每个环境中有两个复制地块的植物之间的3米长度和0.13米。我们收集了来自每次加入纤维品质检测的中间分支机构开发的30次开发的开放式铃铛样品。通过棉纤维质量检验和农业部的棉纤维质量检验和测试中心测量五种纤维质量性状,FL,FS,MIC,FU和FE和FE。使用基于R包'LME4'中的混合线性模型,使用Blup的复制,年和环境中还标准化了原始的表型数据。35].通过Excel软件计算表型数据的描述性统计数据。配对样本t-利用SPSS软件对不同环境和性状间的表型数据进行检验和相关分析。由于30份供试材料的幼苗数量不足以进行表型研究,我们利用249份供试材料的纤维品质性状的表型数据进行进一步的GWAS分析。
SNP基因分型
SNP基因分型时,从279份棉花材料(共288份样品,其中8份来自不同来源)中收集幼叶,并使用Paterson et al.(1993)描述的CTAB方法提取基因组DNA [36].利用含有77774个SNPs的CottonSNP80K序列对材料进行基因型分析。合格的DNA在CottonSNP80K阵列上杂交,并按照Cai et al.(2017)所描述的协议进行基因分型。[8].初始SNP数据集以呼叫率< 0.8和MAF < 0.01进行过滤。此外,为了确定SNP在参考基因组上的物理位置,我们将探针序列映射回Hai7124基因组[15],并对映射到多个位点的探针序列进行进一步过滤。最终获得的高质量数据集用于后续的遗传和GWAS分析。
人口特征
通过自编shell脚本调整SNP数据格式,利用PLINK V1.90软件对288份棉花种质进行相似性分析[36].Tassel 5.0采用neighbor-joining方法构建系统发生树[37],并由FigTree软件进行可视化编辑(http://tree.bio.ed.ac.uk/software/figtree/).采用Tassel 5.0软件进行主成分分析(PCA),用R软件包“ggplot2”作图。采用Admixture 1.3软件进行种群结构分析,K = 3 [38].核苷酸多样性(π)与群体分化(F圣),利用Vcftools软件计算海岛棉两两组间的差异[39].
GWAS分析
采用多位点随机snp效应混合线性模型(mrMLM)进行GWAS分析[13在R包“mrMLM”中。对于模型第一步中相关的SNPs过滤,设置如下参数:CriticalP- rMLM中的值:0.001;候选基因搜索半径(Kb): 100;mrMLM的临界LOD评分:3。同时,Q + K模型还设置了一个由外加剂1.3计算的种群结构(Q)矩阵和一个由R包“mrMLM”计算的亲属关系(K)矩阵。利用5个性状在2个环境下的BLUP值,用R软件绘制了相关QTN的曼哈顿图。
候选基因鉴定
我们将QTN的侧翼200 Kb区域定义为相同的QTL,并将重叠的QTL合并,确定QTL的数量。在参考基因组QTL区域中,通过自写shell脚本鉴定了可能的候选基因。为了确定候选基因的功能,使用AgriGO V2.0和SEA方法实现了基因本体(GO)分析[40].从NCBI Sequence Read Archive collection PRJNA490626下载了来自Hai7124的各种组织和器官的转录组谱[15[纤维发育过程中的采矿候选基因分析。当FPKM(每百万次映射读数的外显子模型的片段)大于3时胚轴和纤维在发生开花后的胚珠(DPA)时大于3时,认为基因在纤维发育过程中表达。
数据和材料的可用性
这项研究中的RNA-Seq数据已保存在美国国家生物技术信息中心(NCBI,http://www.ncbi.nlm.nih.gov/)。
缩写
- BLUP:
-
最佳线性无偏预测;
- 简历:
-
变异系数
- 电子商务:
-
导电性
- 菲:
-
纤维伸长
- FL:
-
纤维长度
- 麦克风:
-
纤维微素
- FS:
-
纤维强度
- 傅:
-
纤维的均匀性
- 走:
-
基因本体论
- GWAS:
-
全基因组关联研究
- MAS:
-
分子标记辅助选择
- 传销:
-
多位点混合线性模型
- 图片:
-
多态性信息内容
- 经济价值:
-
数量性状位点
- QTN:
-
定量特征核苷酸
- SLAF-seq:
-
特异性基因座扩增片段测序
- 单核苷酸多态性:
-
单核苷酸多态性
参考文献
- 1.
CAI C,YE W,张T,GUO W.纤维品质性状的关联分析与高地棉花品种/牧业精英等位基因探索(gossypium hirsutuml .)。acta botanica yunnanica(云南植物研究). 2014;56(1):51-62。
- 2.
石勇,李伟,李安,葛瑞,张博,李军,刘刚,李军,刘安,尚洪,等。构建的高密度链接图gossypium hirsutum×海岛棉并鉴定衣分qtl。植物学报,2015;57(5):450-67。
- 3.
余静,王永华,王志强。棉花纤维品质性状的分子定位与表征。Euphytica。2001;121(2):163 - 72。
- 4.
方l,田罗,李X,陈继,王某,王P,张T.纯棉纤维伸长率网络透析较常见的纤维线延伸的较长纤维线的表达分析海岛棉染色体片段。BMC基因组学,2014;15(1):838。
- 5.
沉XL,郭WZ,朱XF,元YL,YU JZ,Kohel RJ,张TZ。使用SSR标记用SSR标记在Upland棉中三种不同线路纤维品质的QTL的分子映射。mol品种。2005; 15(2):169-81。
- 6.
Sun Z,Wang X,Liu Z,GU Q,Zhang Y,Li Z,Ke H,Yang J,Wu J,Wu L等。基因组 - 范围的协会研究揭示了旱地棉中产量相关性状的新型基因组区域和候选基因。Al Appl Genet。2018; 131(11):2413-25。
- 7.
苏军,范胜,李丽,王超,王辉,宋敏,张超,顾丽,赵胜,等。中国陆地棉衣分QTL优良等位基因和候选基因的GWAS检测植物学报2016;7:1576。
- 8.
蔡鹏程,朱国珍,张梓荣,郭伟忠。高密度80k SNP序列是基因分型的有力工具g .分子资源和基因组分析。BMC基因组学。2017;18(1):654。
- 9.
关键词:棉花,苗期耐盐性,SNPs,候选基因gossypium hirsutuml .)。植物科学。2018;9:1011。
- 10。
张泽,阿卜杜瓦威·j,yimit h。西北延奇盆地土壤盐的发生,来源和土壤盐渍化风险评估。PLoS ONE。2014; 9(9):E106079。
- 11.
Sharif I, Aleem S, Farooq J, Rizwan M, Younas A, Sarwar G, Chohan SM。棉花耐盐胁迫的效应、机理及处理策略植物学报。2019;25(4):807-20。
- 12.
Longenecker de。土壤中高钠对两种棉种脱落,棉铃特性,纤维性能和产量的影响。土壤SCI。1974; 118(6):387-96。
- 13.
王书森,冯建勇,任伟林,黄波,周丽,文永杰,张杰,Dunwell JM,徐胜,张玉明。通过多位点混合线性模型方法提高全基因组关联研究的能力和准确性。Sci众议员2016;6:19444。
- 14.
朱刚,高伟,宋旭,孙飞,侯胜,刘宁,黄勇,张东,倪震,陈强,等。全基因组关联揭示了盐田条件下棉花皮棉产量构成因素的遗传变异(gossypium hirsutuml .)。植物学报。2020;20(1):23。
- 15.
胡勇,陈建东,方磊,张志勇,马伟,牛永昌,鞠立忠,邓建强,赵涛,连建民,等。海岛棉和gossypium hirsutum基因组为异源四倍体棉花的起源和进化提供了深刻的见解。51 Nat麝猫。2019;(4):739 - 48。
- 16.
苏建军,沈恩,王强,傅建军,王强,金海瑞,李春华,王强。苯胺原位漆酶聚合导电棉的研究。聚合物。2018;10(9):1023。
- 17.
唐伟,何勇,屠龙,王敏,李勇,阮玉玲,张欣。膜联蛋白基因的下调GhAnn2抑制棉纤维伸长,降低Ca2+细胞顶端的内流。acta botanica sinica(云南植物学报). 2014;40(6):613-25。
- 18.
何超,严军,沈刚,傅兰,Holaday AS, Auld, Blumwald E,张华拟南芥棉花液泡性钠/质子反转运蛋白基因可改善盐条件下的光合性能,提高棉花纤维产量。植物生理学报。2005;46(11):188 - 54。
- 19.
姜伟,朱海波,何建民。基于ISSR标记的不同地区棉花种质资源遗传多样性研究棉花科学。2008;20(5):348 - 53年。
- 20.
李伟,倪伟,林志,张欣。海岛棉品种的SRAP遗传多样性分析。作物学报,2008;34(5):893-8。
- 21.
王秀琴,冯超,林正祥,张晓丽。海岛棉的遗传多样性(海岛棉)。中国海洋大学学报(自然科学版)2011;10(4):3620-31。
- 22。
马强,赵军,林浩,宁旭,刘鹏,邓飞,司安,李军。海岛棉SSR标记与纤维性状的关联海岛棉)种质资源。J麝猫。2017;96 (6):e55 - 63。
- 23。
彭军,张丽,刘军,罗军,赵旭,董辉,马勇,隋宁,周智,孟Y.土壤盐分对棉花纤维蔗糖代谢的影响。PLoS ONE。2016; 11 (5): e0156398。
- 24.
聂晓东,温涛,邵平,唐斌,Nuriman-Guli A,余勇,杜兴,游成,林志强。新疆旱地棉和皮马棉品种种间不对称渗透与农艺性状改良的紧密相关分析。植物j . 2020。https://doi.org/10.1111/tpj.14760.
- 25.
杨伟。泛素连接酶E3在植物发育和非生物胁迫反应中的作用。植物生理学报。2017;58(9):1461-76。
- 26.
冯浩,李旭,陈浩,邓军,张建军,刘杰,王涛,张学勇,董建林。GhHUB2是一种泛素连接酶,通过泛素- 26s蛋白酶体途径参与棉纤维发育。J Exp Bot. 2018;69(21): 5059-75。
- 27.
关键词:泛素E3连接酶,MYB30,脱落酸,信号转导植物杂志。2018;178(1):428 - 40。
- 28.
孙伟,高智,王军,黄颖,陈勇,李军,吕敏,王军,罗敏,左昆。棉花纤维伸长需要转录因子GhMYB212调控蔗糖向膨大纤维的运输。新植醇。2019;222(2):864 - 81。
- 29.
黄建军,郭勇,孙强,曾伟,李军,李旭,徐伟。R2R3-MYB转录因子在棉纤维发育中调控细胞壁增厚的全基因组鉴定。植物生理学杂志。2019;60(3):687-701。
- 30。
秦涛,刘胜,张震,孙丽,何鑫,Lindsey K,朱丽,张欣。GhCyP3对棉花抗病性的影响黄萎病dahliae通过抑制GhPUB17的E3泛素连接酶活性。acta botanica sinica(云南植物学报),2019;
- 31。
关键词:拟南芥E3连接酶HUB2,组蛋白H2B,单倍化,抗旱性植物生态学报;2019;17(3):556-68。
- 32.
刘艳,张旭,朱松,张华,李勇,张涛,孙杰GhSARP1编码棉花E3连接酶降低了转基因植株的耐盐性拟南芥.生物化学学报。2016;478(4):1491-6。
- 33.
孙丽,朱旭,杨旭,罗华,沈光,等。水稻SUMO E3连接酶基因的过表达OsSIZ1在减少灌溉和雨养条件下,棉花提高了抗旱和耐热性,并显著提高了纤维产量。植物生理学报。2017;58(4):735-46。
- 34。
关键词:小扁豆,盐胁迫,形态解剖,生理特性,分子多样性PLoS ONE。2017; 12 (5): e0177465。
- 35。
Bates D, Machler M, Bolker BM, Walker SC.使用lme4拟合线性混合效应模型。J stat software . 2015;67(1): 1 - 48.36。Paterson, AH, Brubaker CL, Wendel JF。棉花快速提取方法(Gossypium适用于RFLP或PCR分析的基因组DNA。植物学报1993;11(2):122-7。
- 36.
Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MA, Bender D, Maller J, Sklar P, de Bakker PI, Daly MJ,等。PLINK:全基因组关联和基于群体的连锁分析的工具集。中国科学(d辑:地球科学)2007;
- 37.
Bradbury PJ,张Z,Kroon de,Casstevens TM,Ramdoss Y,Buckler ES。TASSEL:多样化样本中复杂性状的关联映射软件。生物信息学。2007; 23(19):2633-5。
- 38.
基于快速模型的非亲缘关系个体祖先估计。基因组研究》2009;19(9):1655 - 64。
- 39.
Danecek P, Auton A, Abecasis G, Albers CA, Banks E, DePristo MA, Handsaker RE, Lunter G, Marth GT, Sherry ST,等。不同的调用格式和VCFtools。生物信息学。2011;27(15):2156 - 8。
- 40。
田T,刘y,yan h,你q,yi x,du z,xu w,su z.农业多v2.0:2017年农业社区的GO分析工具包,2017年更新。核酸RES。2017; 45(W1):W122-9。
确认
我们感谢南京农业大学生物信息学中心的高性能计算平台,提供数据分析设施。非常感谢中国中的棉花国家中期基因库,提供11个地形(E24-3389,Giza 36,Giza 45,L-3398,MI 10,PIMA,PIMA S-3,Termez 16,Yinzi 6022,504-N,9899И),南京农业大学和新疆农业大学,提供本研究中使用的其他268张海岛棉棉。
资金
国家作物育种重点研发计划(2018YFD0100400);国家自然科学基金(u190203);中央高校基本科研业务费专项资金(KYYJ201901);资助方没有参与研究的实验设计、数据收集、分析和解释,也没有参与手稿的撰写。
作者信息
从属关系
贡献
实验由WZG设计。实验用XJS、GZZ、XHS、HJX、WXL、XZN和QJC进行。GZZ和WZG起草了稿件,WZG对稿件进行了修改。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1
279份海岛棉种质资源的信息。
附加文件2
利用CottonSNP80K基因分型总snp的调用频率分布。
附加文件3图S2。
SNP数在每条染色体上的分布。
附加文件4表S2。
利用CottonSNP80K序列对高品质snp进行基因分型。
附加文件5图S3。
海岛棉自然群体中5个纤维品质性状的密度分布
附加文件6
不同条件下纤维品质性状的相关性分析。盒子里的数字表示相关系数(R值)。*、**和***表示P值分别在0.05、0.01和0.001水平。
附加文件7表S3。
利用多位点混合线性模型鉴定5个纤维品质性状的稳定qtl。
附加文件8图S5。
5个性状81个相关qtl的检测次数分布。x轴表示检测次数;y轴表示qtl数量。
附加文件9
稳定qtl的相关时间和相关性状信息。
附加文件10
QTL区域内与纤维品质性状相关的候选基因。
附加文件11表S6。
分别在正常和盐环境下具体且同时关联的基因分析。
附加文件12表S7。
与纤维品质相关的QTL基因表达谱。
附加文件13图S6。
QTN TM68443表型值箱线图。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
Su,X.,Zhu,G.,Song,X.et al。全基因组关联分析揭示了海岛棉纤维品质性状相关的基因位点和候选基因海岛棉).BMC植物杂志20.289(2020)。https://doi.org/10.1186/s12870-020-02502-4
收到了:
接受:
发表:
关键字
- 海岛棉
- 遗传多样性
- 全基因组关联研究
- 纤维质量
- 数量性状位点
- 盐胁迫