摘要
背景
光周期信号为植物调节其生长发育以应对可预测的季节变化提供了重要的线索。光敏色素(Phytochromes)是一个红色和远红色光受体家族,在响应光周期变化调节开花时间方面发挥着关键作用。先前的一项研究表明,两者都存在功能缺失突变、或PHYC在诱导长日照条件下生长的小麦植株,其抽穗时间延迟较大,并导致大量基因的差异调控。
结果
我们发现,在非感应短日(SD)光周期下,、零和phyC-null突变体比野生型(WT)植株更高,分蘖数更少,叶片更长更宽,抽穗时间更晚。在WT和之间的航向延迟体育突变体中LD大于SD,证实了、和PHYC在ld中加速航向日期。这两个突变体在SD中都比LD开花早,与WT植株相反。在SD和LD光周期中,、调控的基因比PHYC。我们确定了差异表达和选择性剪接基因的亚群,这些基因是由、和PHYC在SD或LD光周期中,以及在两个光周期中都受到调控的一组较小的基因。我们发现光周期对开花促进基因的转录水平有不同的影响VRN-A1和PPD-B1在、和phyC突变体与WT相比。
结论
我们的研究证实了两者的主要作用、和PHYC在LD条件下促进开花。转录组分析结果显示,小麦的LD植株出人意料地逆转为SD植株、零和phyC-null突变体和已鉴定的开花基因显示光敏色素和光周期之间的显著相互作用可能参与了这一现象。我们的RNA-seq数据提供了对诱导和非诱导光周期的光信号通路的深入了解,并提供了一组候选基因来剖析小麦潜在的发育调控网络。
背景
作为无根生物,植物必须能够对环境的波动做出反应,以最大限度地提高其繁殖成功率。为了实现这一目标,植物进化出了一系列调节机制,以确保它们发育的关键阶段与最佳环境条件相一致。开花时间是繁殖成功的一个重要决定因素,而开花时间受光照周期和温度的季节性变化的强烈影响[1]。在谷类作物中,这些线索是确保植物不会过早开花的基础,以防止敏感的生殖组织暴露在晚春霜冻中,或太晚开花,以尽量减少在灌浆期间受到破坏性高温的影响[2]。生殖成功与粮食生产之间存在直接联系,因此描述开花时间背后的调控网络对于支持开发具有抗逆性的作物品种、帮助满足世界日益增长的粮食需求至关重要[2]。
根据植物所适应的环境,植物对光周期的季节变化有不同的反应。一些植物在短日照期(SD植物)开花速度加快,而另一些植物在长日照期(LD植物)开花速度更快。第三类植物是昼中性的,无论光周期如何都开花。温带谷物,包括普通小麦(小麦L.),是LD植物。这确保了植物在冬季保持在营养阶段,直到春天的白昼变长触发向生殖发育的不可逆转的过渡[1]。另一个需要长时间低温的条件(春化)阻碍了秋季开花,而此时白天仍然相对较长[3.]。
在小麦和其他温带谷物中,夜晚的长度,而不是白天的长度,对感应光周期的感知至关重要。实验证明了这一点,在这些实验中,将小麦植株暴露在夜间(在漫长的夜晚,15米的光照周期)至少12天,足以加速开花[4]。小麦光敏色素基因的功能缺失突变光敏色素B(、)或PHYC,或在PHOTOPERIOD1(PPD1)基因取消了夜间休息加速开花,这表明这些基因对测量夜间持续时间至关重要[4]。
最近的一项研究Brachypodium提出了这些基因在决定光周期反应中的作用机制[5]。光敏色素是一类红光(R, ~ 650 nm)和远红光(FR, ~ 720 nm)光受体,以两种可互换的形式之一存在R和PFR。在黑暗中,无生物活性的PR形式在细胞质中积累,但在吸收R光后,PR转化为生物活性PFR形成并转移到细胞核[6,7,8]。相反,暴露在FR光下会导致P的快速还原FR彻底地R反应在夜间也逐渐发生(黑暗或热还原)。因此,夜间的持续时间影响生物活性P的量FR它被认为是降解时钟蛋白EARLY开花3 (ELF3)的关键,ELF3是一种直接抑制因子PPD1[5]。高ELF3蛋白水平和抑制PPD1已被提出作为晚开花表型的主要原因phyC突变体中Brachypodium[5]。
PPD1编码一种PSUEDO-RESPONSE REGULATOR (PRR)家族蛋白,该蛋白对LD草的开花起到正向调节作用[9,10,11]而是在SD禾本科中作为ld抑制因子[12]和高粱[13],这种基因被称为PRR37。在小麦中,等位基因变异发生在PPD1基因座影响光周期敏感性。而WTPpd-A1b等位基因在夜间的表达水平非常低Ppd-A1a等位基因携带包含ELF3结合位点的启动子缺失,在白天,特别是晚上,转录物水平增加[14]。小麦品种携带Ppd-A1b等位基因被称为光周期敏感(PS)和那些携带Ppd-A1a作为光周期不敏感(PI),因为它们在光周期不敏感(SD)和光周期不敏感(LD)下表现出加速抽穗和缩短抽穗时间的差异。需要指出的是,携带PI等位基因的小麦品种在光周期不敏感(LD)下仍表现出显著的加速抽穗[9,11]。PPD1诱导表达开花位点t1(FT1),它编码一种与磷脂酰乙醇胺结合蛋白(PEBP)家族相似的蛋白质[15]。FT1蛋白通过韧皮部转运到茎尖分生组织,在那里形成六聚体花激活复合体,直接激活分生组织身份基因的表达,包括春化1(VRN1),FRUITFULL 2(FUL2)。这些MADS-box基因在触发生殖发育中起着关键作用[16,17,18]。在谷物中,ft1-null突变体在开花时表现出强烈的延迟[19]。
除了对ELF3的调控作用外,生物活性PFR光敏色素在细胞核内直接与光敏色素相互作用家族蛋白(PHYTOCHROME INTERACTING FAMILY, PIF)相互作用,这是一类bHLH转录因子[20.,21]。在拟南芥中,这些相互作用诱导PIF蛋白的生化变化,导致其泛素化并通过26S蛋白酶体途径降解[22]。在这个物种中,积累的PIF蛋白主要作为光信号转录网络的负调节因子,因此它们在R光下的降解触发了一系列光周期介导的转录反应。尽管PIF蛋白在拟南芥光信号通路中发挥着重要作用,但其在温带谷物光周期响应调控中的作用尚不清楚。
光敏色素还可以通过调节选择性剪接(AS)诱导转录变异[23]。在拟南芥中,在1500多个基因中检测到AS以依赖phyb的方式响应R光[23]。这些靶基因包括PIF3,因此PFRPHYB增加了该基因中内含子保留事件的频率,破坏了翻译蛋白的功能[24]。在青苔中Physcomitrella金属盘,光敏色素蛋白PpPHY4直接与剪接调节因子相互作用,介导AS对光的响应[25]。先前,剪接因子RRC在拟南芥中被发现介导光敏色素反应,这表明该机制可能在被子植物中保守[26]。
单子叶植物基因组包含三个光敏色素基因,巴,、、和PHYC,在六倍体小麦中每个基因有三个同源副本[27]。在小麦和Brachypodium,两个、和PHYC都是在LD条件下及时开花所必需的,携带两种光敏色素无功能拷贝的植物开花时间极度延迟,营养形态也会发生变化[28,29,30.]。使用、零和phyC在四倍体小麦品种“Kronos”中,我们先前描述了由、和PHYC在ld [31]。尽管两种突变体的开花时间延迟相似,但我们发现、调节的基因数量大约是PHYC,并且只有104个基因的一小部分核心在转录水平上受到这两种光敏色素的调控[31]。这些常见的调控基因包括几个很好表征的开花时间基因,如PPD1和FT1,以及分生组织识别基因,包括VRN1和FUL2。
小麦光敏色素在非诱导光周期中的作用仍然是一个悬而未决的问题。之前,我们发现whilephyC-null突变体在SD和LD光周期都比WT植株开花晚,在SD的影响大约小5倍[28]。光周期与PHYC, WT植株在ld中比在sd中抽穗早phyC-null突变体在SDs中比LDs更早起头[28]。在目前的研究中,我们发现、-null Kronos突变体在SD中的开花时间也明显早于LD。
表征与早熟抽穗有关的基因、零和phyC我们比较了SD和LD条件下这些突变体的转录组。我们发现了由、和PHYC在SD和LD光周期中,以及仅在特定光周期下调控的基因。本研究结果有助于我们了解小麦光周期调控开花的复杂调控网络。
结果
的影响、零和phyC-航向时间上的null突变
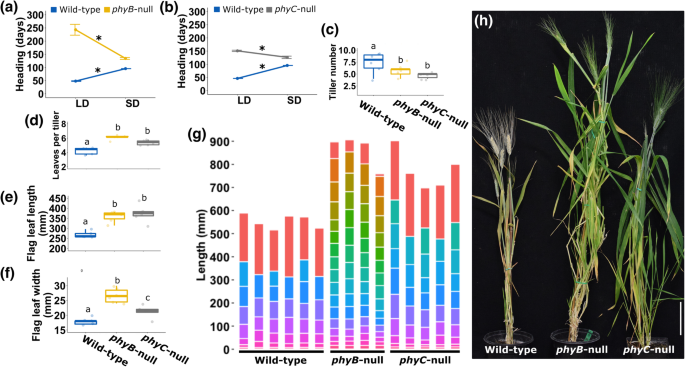
我们首先描述了Kronos-的作用、- - - - - -null和KronosphyC -与光周期不敏感的野生型Kronos (WT)相比,在LD和SD条件下,null突变体在抽穗时间上的差异较大。Ppd-A1a)春小麦(Vrn-A1).WT Kronos在LD期47 d, SD期95 d(延迟48 d)。P< 0.0001),符合LD工厂的预期。结果表明,克罗诺斯植物携带Ppd-A1a等位基因对光周期的变化仍有响应。
在劳工处下,phyC-null突变体比WT晚104 d开花、-null比WT晚194 d(图1)1a-b),证实这两个基因在LD下促进开花的主要作用[28,31]。相比之下,两者、零和phyC-null突变体在SD中比在LD中早头(比在LD中早108天)、空,P< 0.001,图。1A, 24天前phyC空,P< 0.001,图。1b).这种逆转是由于零突变体在LD处理下(104-195 d)比SD处理下(31 d,比WT晚39 d)的抽穗时间延迟得多。P< 0.0001,图。1a - b)。光周期与基因型之间的互作均显著、和PHYC(无花果。1a - b,P< 0.0001)。
的表型特征、零和phyC在SD条件下(光照8 h /黑暗16 h) -null突变体。(一)WT和标题日期、-null植株在SD和LD中表现出显著的交互作用、光周期。点表示至少五个生物重复的平均值,误差条对应于SEM。(b)WT和标题日期phyC-null植株在SD和LD中表现出显著的交互作用PHYC光周期。点表示至少五个生物重复的平均值,误差条对应于SEM。(c)每株分蘖数。(d)每分蘖平均叶数。(e)旗叶长度。(f)旗叶宽度。(g)节间长度和数目。每个条形图代表一个单独的植物,水平线对应节点的位置。每个节间用不同的颜色表示,根据它们在茎中的位置排序。每个个体中最上面的段表示最后一个节点到穗(花序梗)之间的长度。(h)图片中的代表性植物当、-未达到抽穗日期的植株,bar = 10 cm。(c来f箱形图表示至少五个生物重复的值。不同字母表示显著差异(Tukey’s test)P< 0.05)。对于(a)和(b), *表示每个基因型的光周期之间存在显著差异;P< 0.0001。WT与突变型等位基因之间的差异也非常显著(P< 0.0001)
与先前在lcd中对这些材料的表征一致[28,31],克罗诺斯植物在a或B同源上携带单个空等位基因、或PHYC与SDs(附加文件)中的WT相比,在抽头日期上没有明显的延迟1,图S1)。在其他特征上也观察到同样的情况,所以所有随后的结果都描述了在、空,phyC-null突变体和Kronos-PI背景下的WT。
的影响、零和phyC-null突变体对SD下植物表型的影响
接下来,我们扩展了这些突变系在SD条件下的表型特征。两种突变体的分蘖数均显著低于野生型(图2)。1c),而两个突变体的每分蘖平均叶片数显著高于WT植株(图2)。1D),可能是由于茎尖分生组织延迟过渡到生殖阶段。在这两个、零和phyC-null突变体的旗叶明显长于WT(图2)。1e-f)。
茎的发育也受到影响体育突变体。两种突变体均显著高于WT植株(、-空高310毫米;P= 9.72E-06和phyC-null高220毫米;P= 0.00016,图1g)、零和phyC-null突变体总体高度差异不显著,茎部结构差异显著。的、-null突变体的节间数量比任意一种WT都多(比WT多9个节间);P= 7.14E-09)或phyC-null突变体(节间比phyC,P= 3.53E-07),而phyC与WT对照相比,-null植株的节间数略有增加(多2.1个节间;P= 0.00013,图1g).各基因型的代表性植株如图所示。1H,这是在、-null突变体达到标题日期。综合来看,这些结果表明、和PHYC在非诱导SD条件下发挥重要的调节营养和生殖发育的作用。
描述的、- - -PHYC-调控小麦转录组
研究与开花较晚相关的转录变化、零和phyC在Kronos背景下,我们在WT中进行了RNA-seq实验,、零和phyC在SD条件下-null植物。我们在8周龄时从每个基因型的四个生物重复的最后完全展开的叶片上收集组织(附加文件)1,图S2)。为了便于与先前在LD条件下对相同材料进行的RNA-seq研究进行比较[31],我们在光周期的同一点(黎明后4小时)采集样本。在我们的SD实验中,我们从8周龄的植物中采集组织,因此WT植物与LD RNA-seq研究中的WT植物处于相似的发育阶段,后者是在4周龄时采样的。
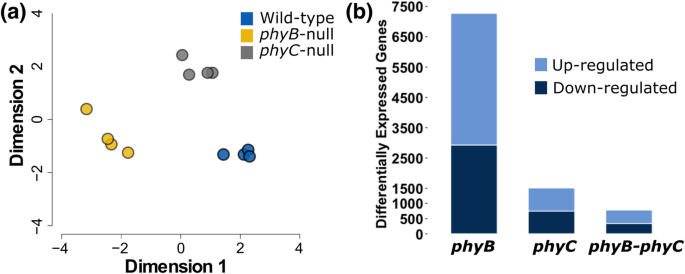
在对原始reads进行质量和适配器污染修剪后,每个样本平均有45.0 M修剪的100 bp单端reads被映射到IWGSC RefSeq v1.0基因组组装中的独特位置(附加文件)1表1)。使用所有归一化的读取计数映射到每个样本的高置信度和低置信度基因模型,我们生成了一个多维缩放(MDS)图(图2)。2a).样本根据基因型分为三个不同的簇,反映了基因型之间总体转录组谱的一致差异和生物重复之间的有限差异(图2)。2一个)。
接下来,我们对WT和两个突变体进行两两比较以确定、- - - - - -和PHYC- SD条件下的差异表达(DE)基因。我们发现有4.8倍的基因被、(7272个DE基因)PHYC(1511个DE基因,图2)2b).在这些DE基因中,受、(59.7%,在WT中的表达高于、-null)比byPHYC(50.6%)。共有815个基因受到两者的调控、和PHYC,其中783个基因在同一方向调控,27个基因在相反方向调控、并且被PHYC反之亦然,图。2b).每个两两比较的表达数据和统计检验的详细信息在附加文件中提供2。
为了确定与这些转录变化相关的假定功能,我们对差异表达基因的每个子集进行了氧化石墨烯富集分析。在7272个由、在SDs中,最显著富集的术语包括“氧化还原过程”和“蛋白质磷酸化”,而在1511个由PHYC,重要的术语包括“防御反应”和“细胞铁稳态”1,表S2)。在由两者共同调控的基因中、和PHYC,丰富的术语包括“防御反应”和“蛋白质磷酸化”(附加文件)1,表S2)。
发育的变化通常与编码转录因子的基因的差异表达有关。与我们数据集中编码转录因子的基因的总体比例(72,120个表达基因的3.2%)相比,观察到的转录因子基因的比例有所增加、-(5.3%)和PHYC-调控的基因(5.4%),两者调控的基因增加幅度更大、和PHYC(6.5%)。更重要的是,参与开花调控的几个关键基因在植物中有差异表达、和phyC的WT转录水平PPD-B1,的同源物FT1和FT2,VRN-B1和FT-B3都明显降低了吗、零和phyC-null突变体与WT植物的比较(附加文件1,图S3,附加文件2).
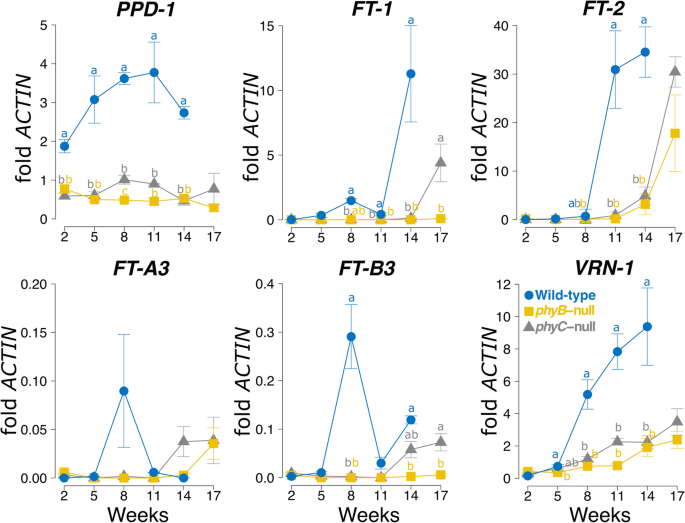
为了验证这些表达数据,并研究这些基因在SD条件下的长期表达趋势,我们使用与RNA-seq分析相同的基因型,对六个时间点上选择的候选基因进行了qRT-PCR分析。在8周时间点,qRT-PCR实验证实了RNA-seq结果,显示VRN1,FT1,FT2,PPD1和发生都明显降低了吗、零和phyC-null突变体与WT相比(图2)。3.).重要的是要注意PPD1,这些值代表的组合转录水平Ppd-A1a和Ppd-B1bhomeologs。
在表达谱上也存在差异FT-like基因型之间的家族。在WT植物中,FT1转录水平是FT2在5w和8w时间点(图2)3.),与先前的研究结果一致[32]。相比之下,两者、和phyC植物,FT2在更早的时间点(14w)上调FT1,在17w时表达量增加phyC突变体,但在整个实验中都很低、突变体(无花果。3.).发生的表达水平远低于FT1和FT2,在WT中两者都有FT-A3和FT-B3在8 w时出现瞬时表达高峰phyC突变体,发生14w时水平开始上升,17w时甚至更高、我们只观察到突变的上调FT-A317 w(图1)3.).VRN1在整个时间过程中,两种突变系的表达逐渐增加,但其转录物水平在5 w以后的所有时间点都明显低于WT系(图2)。3.).
综上所述,光敏色素突变体和野生型之间的这些表达差异与玉米的延迟抽穗日期一致、和phyC这些结果也证实了光敏色素在SD和LD光周期下对关键开花基因的调控中起着重要作用。的早期表达式FT2相对于FT1及其高转录水平(图2)。3.),提示该基因可能在SD条件下对小麦开花时间的延长起重要作用。
光周期对光敏色素调控基因的影响
为了探究光周期对WT和光敏色素突变体之间差异的影响,我们比较了本研究中从8周龄植物中收集的SD中产生的DE基因与之前使用相同植物材料在LD条件下从4周龄植物中收集的DE基因[31]。选择这个SD时间点是为了匹配SD和LD条件下WT植株的发育阶段(Waddington阶段3 [33])。
为了允许数据集之间的直接比较,我们使用相同的定位和定量参数调整了读取长度,将早期LD研究中的RNA-seq reads重新映射到IWGSC RefSeq v1.0基因组组装中。使用这个更新的基因组参考,52.8%的读取被唯一地映射(附加文件1表S3)。该LD数据集包括两个实验重复,每个实验重复4个生物重复。只有在两个实验中都有显著差异的基因才被认为是差异表达。这种方法降低了假阳性率,但意味着SD和LD数据集之间差异表达基因数量的直接比较应该谨慎处理,因为SD数据只代表一个实验重复。
MDS图根据SD和LD样品的整个转录组谱显示,光周期之间的差异比光周期内基因型之间的差异更大1,图S4)。在LD实验中,不同实验重复之间表达谱的差异可能是由于生长室光照条件的变化[31]。我们确定了3668个基因在WT和、-null突变体在两个实验重复和424个基因进行相应的比较phyC零变异。这些基因中只有141个被两者调控、和PHYC在LD条件下。这些结果与我们之前将这些测序数据映射到一个较早版本的小麦基因组的研究结果是一致的。31]。在附加文件中提供了LD光周期中每个两两比较的表达数据和统计测试的详细信息3.。
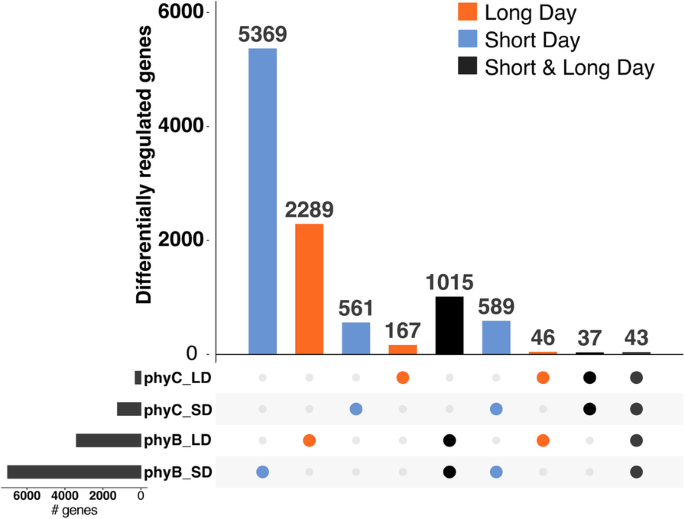
含有SD和LD表达数据的MDS图显示,不同光周期生长的小麦植株的转录组存在明显差异1,图S4)。在无花果。4,我们根据基因在WT和突变等位基因之间的差异表达条件(即受、或PHYC在SD或LD条件下)。为清楚起见,该图排除了一些基因数量较少的两两比较,因此文本中提供的数字之和不等于DE基因的完整数量,后者在附加文件中给出4。在两个光周期中,更多的基因仅由、而不仅仅是PHYC(无花果。4).在SDs中,有9.6倍以上的基因被、(5369个基因)PHYC(561个基因),而在ld中,由、(2,289个基因)比PHYC(167个基因,图。4).
调控的差异表达基因综述、和PHYC在SD或LD光周期。在两两比较中,每个互斥的类别包括WT和各自光敏色素突变体之间差异表达的基因。为清楚起见,并不是所有的两两比较都在附加文件中提供4都显示在这里
WT和突变基因型之间的差异表达基因在SD中(589个基因)多于在LD中(46个基因)。4).此外,单个光周期中差异调控的基因数量大于两个光周期中差异调控的基因数量。例如,有1015个基因由、在SD光周期和LD光周期中,分别有5369个和2289个基因在SD光周期和LD光周期中显著(图2)。4).因为小麦抽穗时间的LD加速需要两者同时存在、和PHYC,我们将重点放在两个突变体的DE基因上。我们仅在SD中检测到589个DE基因,在LD中检测到46个,在SD和LD中同时检测到43个(图2)。4).
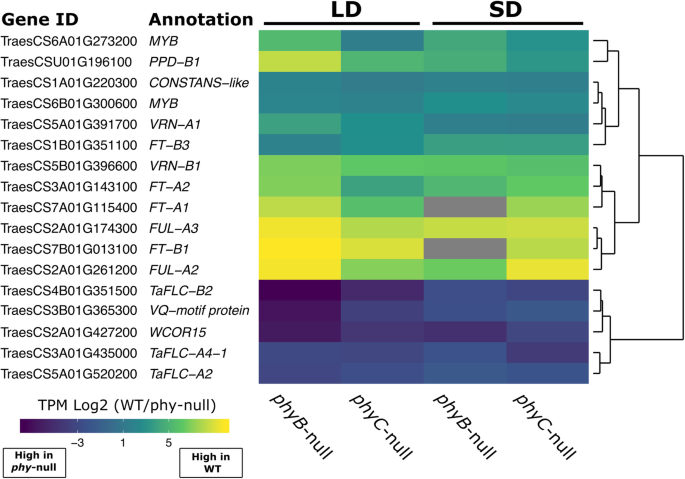
在GO术语分析中,与光敏色素在SD和LD下调控的43个基因相关的功能术语显著丰富,包括“转录调控”和“光周期”1(表S4)。光敏色素正调控的24个基因(即在WT中的表达高于在WT中的表达)体育突变体)包括Ft1, ft2, ft3, ppd-b1, vrn1, ful2和FUL3(无花果。5、附加文件1表5)。虽然对LD的影响更大,但这些结果证实、和PHYC也在Kronos-PI背景下SD中这些基因的激活中发挥了重要作用。这些结果与我们的qRT-PCR分析一致(图2)。3.).与前一组具有相同表达谱的其他基因包括编码a君士坦斯类似的cct结构域蛋白(TraesCS1A01G220300),以及两个同源物编码myb转录因子,具有高度相似性辐TraesCS6A01G273200和TraesCS6B01G300600,图5、附加文件1表5)。其中一个基因(TraesCS1A01G569000LC)表达上调、在SD和LD和byPHYC在SD中,但是被PHYC在ld(附加文件4).
其中17个基因被两者负调控、和PHYC在两个光周期中都有trescs3b01g365300基因,该基因编码转录调控因子VQ motif蛋白家族的一个成员方法MADS-box TFs分支(图2)5、附加文件1表5)。其中两个基因编码同源物FLC2,它是与OsMADS51,是水稻开花的SD促进剂[34]。FLC4的同源编码ODDSOC2它的作用是抑制开花Brachypodium小麦经冷处理后诱发[35]。有趣的是,TraesCS2A01G427200,它编码WCOR15在两个突变系中,光敏色素在抑制环境温度条件下小麦的耐冷途径中发挥了重要作用(图2)。5、附加文件1表5)。其中一个基因(TraesCS2A01G019700LC)被下调PHYC在SD和LD和byPHYC在LD中,但被、在LD条件下(附加文件4).
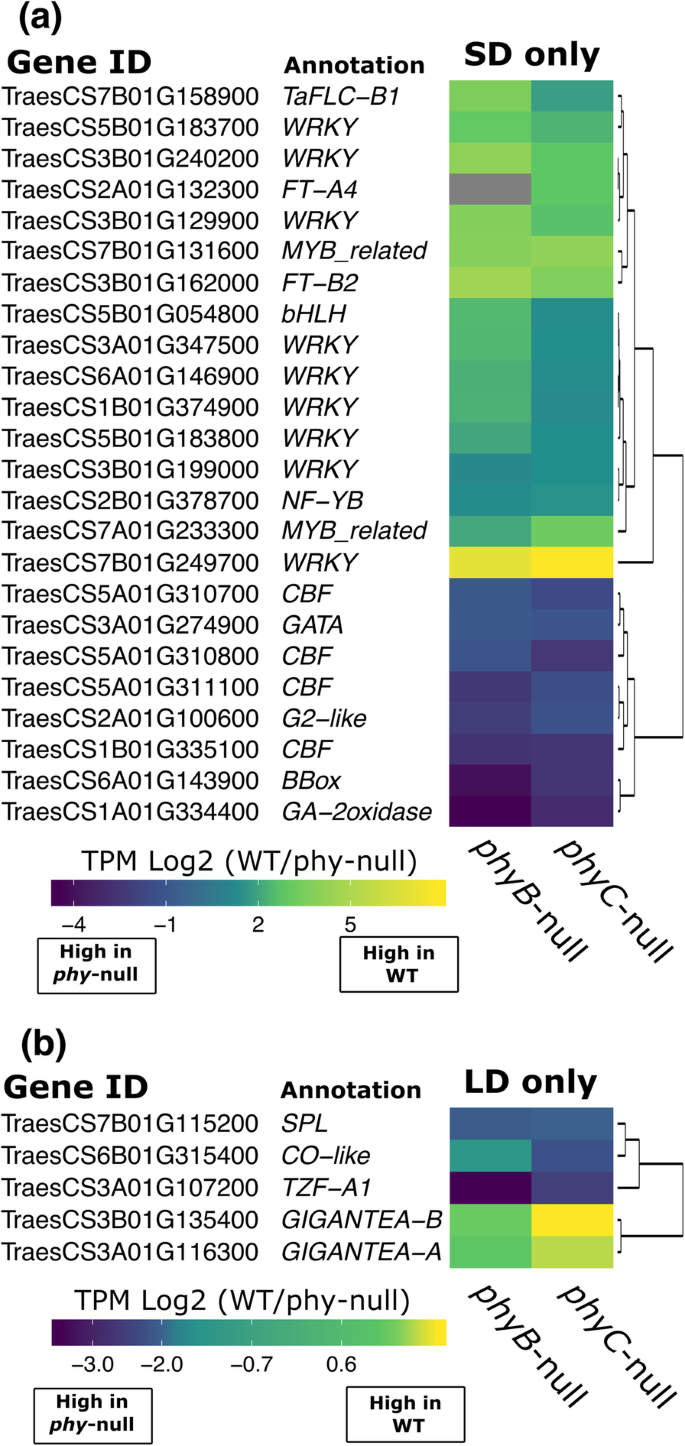
对两者调控的589个基因的GO术语分析、和PHYC仅在SD下,显示了丰富的功能术语“蛋白磷酸化”和“稳态”(附加文件)1(表S4)。在该组367个正调控基因中,我们检测到9个WRKY转录因子,它们都是a辐类似于myb家族的转录因子(TraesCS7A01G233300和TraesCS7B01G131600)和TraesCS5B01G054800,它们编码与PIF亚家族相似的bHLH TF(图2)。6a、附加文件1表6)。我们在这组中也发现FT-A2,FT-A4和FLC-A1(无花果。6a、附加文件1表6)。在受两种光敏色素仅在SD下负调控的213个基因中,我们鉴定出GATA、g2样和B-box转录因子家族成员以及编码GA-2oxidase- a4失活酶的TraesCS1A01G334400(图2)。6a、附加文件1表6)。四名成员的上调CBF两种光敏色素突变体的冷激活转录调控因子家族(图2)。6a,附加文件1,表S6),建议一个类似的角色WCOR15在环境温度下抑制耐冷途径,但在这种情况下仅限于SD条件。其他19个基因要么受到、并且受到PHYC,或反之亦然(附加文件4).
接下来,我们研究了这两种基因调控的46个基因、和PHYC特别是在LD条件下。与这些基因相关的最丰富的功能术语是“茎系统发育”、“长日光周期”和“昼夜节律调节”1(表S4)。有27个基因被两者正调控、和PHYC的同源物GIGANTEA表明该基因可能在小麦开花的LD激活中起作用(图2)。6b、附加文件1(表S7)。在这两种光敏色素负调控的16个基因中,TraesCS6B01G315400编码a君士坦斯类似蛋白质的一种SPL转录因子家族和串联锌指1,在拟南芥中,它与生物钟调节网络的PRR蛋白组分相互作用[36(图。6b、附加文件1(表S7)。另外三个基因受到、但是受到了PHYC(附加文件4).
基因型对光周期调控基因的影响
最后,我们对每种基因型的SD和LD样本进行了直接两两比较(附加文件)5)来鉴定光周期调节基因(PRGs)。两者都有更多的PRGs、-null(19,749)和phyC-null(13,740)突变比WT(12,873),附加文件1,图S5),提示在、零和phyC-null不足以降低不同光周期对小麦转录组产生的巨大影响。
虽然在LD (4 w)和SD (8 w)中选择了不同的采样点,因此在两个实验中WT基因型处于相似的发育阶段,但这些结果应该谨慎解释,因为光周期的影响与实足年龄差异的影响相混淆。这两个、零和phyC-null突变体在SD下比在LD下更早抽头,因此很可能突变系在采样时处于不同的发育阶段。这种特殊的采样策略可能导致WT基因型中PRGs的数量相对较少、零和phyC零突变体。
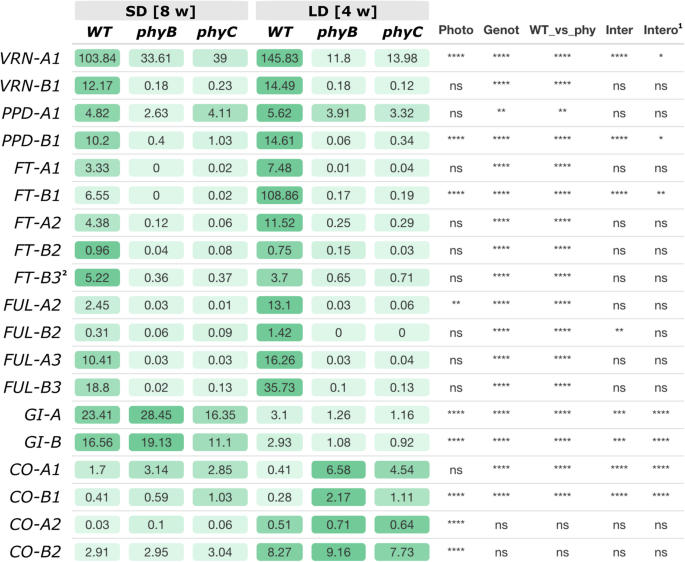
我们利用该数据集探索了19个开花时间基因在不同基因型和光周期中的表达谱及其相互作用(图2)。7).
该分析证实了先前的结果,显示转录水平VRN-A1, PPD-B1,FT-B1, FUL-A2,胃肠道,CO-B1和二氧化碳都受到光周期的显著影响(图2)。7).值得注意的是,转录水平的光周期不敏感Ppd-A1a在该数据集中,等位基因受光周期的影响不显著,而基因的受光周期的影响不显著Ppd-B1b等位基因对光周期(P< 0.0001)。转录水平胃肠道在SD中表达更高,而CO1和二氧化碳在LD中表达更高(图2)。7).
大部分花期基因的表达也受花期因子的影响、零和phyC零突变。三种基因型之间存在显著差异,WT与联合基因型之间也存在显著差异、- - - - - -和phyC-null突变,除了CO-A2和CO-B2。后者的结果与之前的一项研究一致CO1在白天被高度调控phyC-null突变但是二氧化碳转录水平未受影响[28]。的VRN1假字(FUL2和FUL3)和与花素有关的基因(FT1和FT2)都有相似的特征,在WT中的转录水平相对于phy -零突变体和相对于SD的LD(图2)。7).FT-A3未检测到转录本,而FT-B3SD的转录水平高于LD,这与该基因作为抽穗日期SD启动子的已知作用一致[19,37]。
VRN-A1和PPD-B1我们所分析的开花促进基因中是否只有我们观察到的转录水平显著高于phy -SD中的零突变比LD中的多,WT中的相反(LD > SD)。基于这一结果,我们推测,这些基因可能有助于提早开花phy -这些基因的表达受光周期和基因型的显著影响,且三者均表现出显著的基因型与光周期的相互作用(图2)。7).重要的是要指出,SD RNA-seq样品用于phy -无效突变体是在抽穗前70-78天收集的,因此它们可能代表开花诱导的早期阶段。这将是一件有趣的事情,研究更接近头部的时间点,看看基因是否被诱导VRN-A1,例如VRN-B1,FT1,FT2[32,38],在SD中比在LD中更早上调。
小麦的光信号和选择性剪接(AS
除了转录水平的差异外,我们还探讨了、或PHYC利用转录剪接多变量分析(Multivariate Analysis of Transcript Splicing, rMATS)统计方法调控小麦AS事件[39]。我们的RNA-seq数据集显示两者都有、和PHYC调节编码剪接机制成分的基因表达(附加文件)1,表S8)。例如,编码剪接因子U2AF大亚基的TraesCS2A01G122400在、-null突变体在SD和LD条件下和编码富含精氨酸/丝氨酸剪接因子的TraesCS1B01G130200中上调phyC-null SD和LD中的突变(附加文件)1,表S8)。还有一些剪接相关基因在SD条件下被特异性调控。编码剪接因子亚基的三个基因在两者中均上调、零和phyC-null突变体,而编码前mrna剪接因子cwc26的TraesCS1B01G125800在SD条件下在两种突变体中均显著下调(附加文件)1,表S8)。
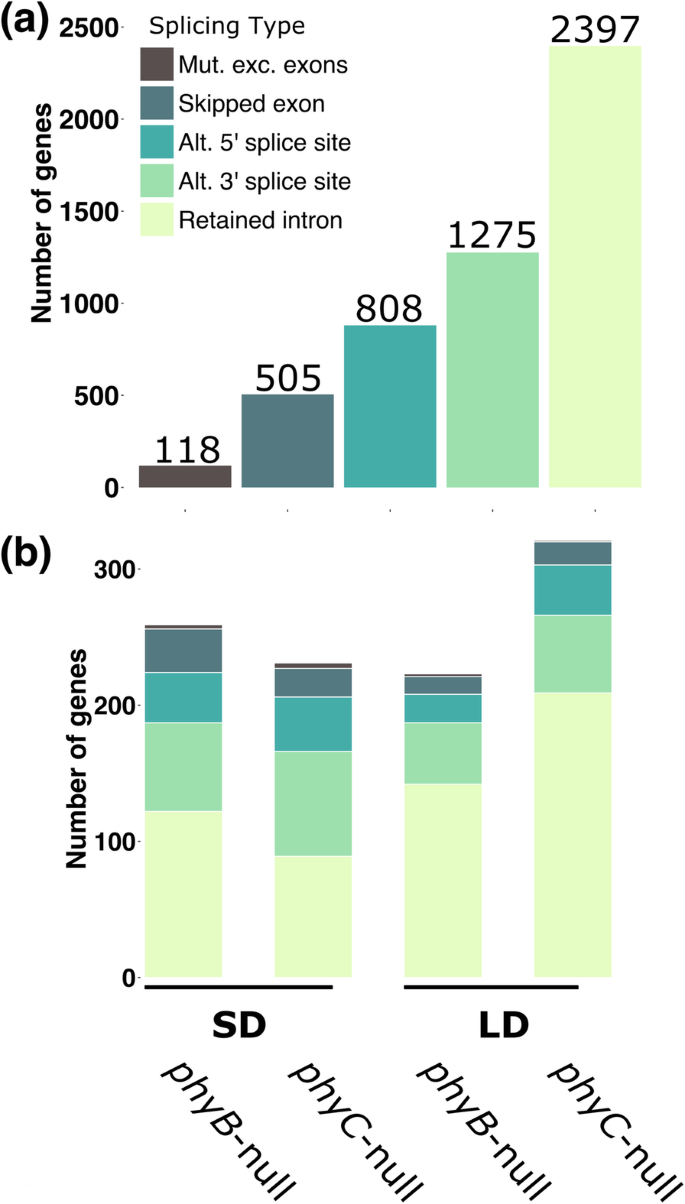
为了量化这些变化对小麦AS的影响,我们首先确定了标记基因中指向外显子-内含子连接的RNA-seq读数,并计算了五种不同类别(保留内含子,跳过外显子,替代5 '或3 '剪接位点和相互排斥的外显子)AS事件的频率。比较不同光周期内WT基因型和突变基因型之间每个事件的频率,我们发现5175个AS事件受到任何一种基因型的显著调控、或PHYC(罗斯福P -adj < 0.05)。最常见的AS事件是内含子保留,其次是替代的3 '剪接位点(图2)。8一个)。
为了对可能对基因功能产生更大影响的事件进行分类,我们研究了基因型之间同种异构体表达水平差异> 30%的注释基因子集。在这些基因中,SD和LD中受AS事件影响的数量相似(图2)。8B),尽管我们发现了稍微多一些的基因保留了由PHYC在LD条件下(图1)8b).附加文件提供了受不同AS事件影响的个体基因的完整信息6。
讨论
光敏色素与PPD1在小麦抽穗时间的调控上
在植物物种中,光敏色素的一个功能是根据光周期的变化调节开花时间。先前的研究表明,大脑的一个主要功能是、和PHYC在低温条件下,加速抽穗时间[28,29,30.,31],这些结果在这里得到了证实(图2)。1a - b)。相比之下,同源基因的功能丧失突变体育SD草、水稻和高粱的基因在LD条件下使抽穗期提前。40,41,42,43]。尽管有相反的效果、零和phyC-null突变体对抽穗时间的影响,这两个基因促进了PPD1 / PRR37在两组草中。他们之间的区别似乎是顺流而下的、和PHYC,因为在LD条件下PPD1 / PRR37在水稻中起抑制开花的作用[12]和高粱[13而是作为温带禾本科植物的开花促进剂[9,10,11]。
一个值得注意的结果,从先前的观察phyC-null mutants [28在目前的研究中,两者都包括在内、- - - - - -零和phyC -在这两种情况下,突变体在SD中比在LD中早熟,这表明这些植物的行为就像它们是SD植物一样。由于WT Kronos在SD中的开花时间比在LD中的晚,因此两者在抽穗时间上的差异体育-null和WT在LD (100-200 d)下比在SD (30-40 d)下大得多。1a - b)。
值得注意的是,这些实验都是在携带PI (PI)的“Kronos”品种中进行的。Ppd-A1a)等位基因。这PPD1等位基因在包含ELF3蛋白抑制因子结合位点的启动子区域有缺失[5],导致的异位表达PPD1在夜里[14]。的夜间表达PPD1对光周期反应至关重要,正如在夜间实验中所证明的那样。感应的PPD1在一个16小时的夜晚(SD)中,15米的光脉冲加速了航向时间,几乎与LD光周期一样多[4]。在Brachypodium,有人提出PHYC激活PPD1是由ELF3 [5],因此消除了一个ELF3结合位点Ppd-A1a小麦中的等位基因可能会限制光敏色素信号的传递PPD1。因此,确定……的效果是很重要的、零和phyC-在PS存在的情况下,标题日期的null突变Ppd-A1b等位基因来测试SD相对于LD的抽穗时间是否加快体育突变体是在这种遗传背景下维持的。我们启动了十字架来做这个实验。
在SD下的航向时间加速度phy -null突变体与sd -春化既有相似之处,也有不同之处。冬小麦和Brachypodium,在室温下暴露于SD 6 - 8w,然后进行LD,取代了春化以加速抽穗日期的需要[44,45,46,47],但这在PI小麦品种中没有观察到[46]。相比之下,我们在Kronos-PI背景下观察到SD加速、零或phyC-null突变,这表明这两种现象可能涉及不同的调节机制。
为了假设一种机制,使抽头日期在SD中相对于在LD中加快phy -研究光周期对开花促进基因调控的对比效应将是一个有趣的研究方向VRN-A1和PPD-B1在phy -空突变体(在SD中上调)与WT(在SD中下调)相比,在南瓜的早期开花中起作用phy -与LD相比,SD中的null突变体在时间上是可逆的FT1和FT2基因在WT和体育突变体也可能有助于提早开花phy -SD中的零突变体。在WT光敏色素等位基因存在的情况下,FT1在Kronos-PI发育早期比FT2SD中的基因(图2)3.)及LD [32]。然而,在phyC -SD下的零突变体,FT2转录本上调的时间早于FT1。17周后,当这些植物开始抽芽时,FT2达到非常高的表达水平(> 10倍)肌动蛋白)。、- - - - - -零和phyC -零突变体。在LD条件下的生长室试验、SD再LD条件下的生长室试验和秋种田间试验中,ft2-null等位基因只使抽头日期有轻微延迟[32]。总而言之,将需要进一步的研究来确定PPD1, VRN1和FT2在a中SD下的航向时间的规定phy -空的背景。
虽然发生转录水平低于其他被测基因,它们也较早上调FT1在这两个、零和phyC-null突变体(图2)3.).在大麦中,同源物的过度表达HvFT3在LD中加速抽穗,促进茎尖分生组织从营养阶段向生殖阶段的过渡[48]。在Brachypodium,BdFTL9的成员发生进化枝在SD条件下促进开花[47]。这种蛋白只有在缺乏BdFT1(即SD条件),描述了PEBP家族的多样性可以根据光周期精细调节开花时间控制的可能机制[49]。我们确定了在LD条件下上调PEBP家族的其他几个成员(附加文件)5),因此,描述它们在小麦抽穗日期中的作用将是有趣的。
除了PEBP基因,GIGANTEA,VRN2 / GHD7和有限公司已被证明在水稻的光周期响应中起重要作用[50,51]。GIGANTEA是拟南芥中FT的直接启动子[52和大米GIGANTEA移植有限公司(Hd1)激活英国《金融时报》[51,53]。在这项研究中,我们展示了小麦GIGANTEA在SD下的表达水平明显高于LD,并受到两者的正向调节、和PHYC特别是在LD下(图2)。7),这表明GIGANTEA也可能在小麦光周期途径中发挥作用。在水稻中,CO能促进SD植株开花GHD7 / VRN2或PRR37 / PPD1等位基因和LDghd7prr37双突变体[50提供了一个例子,说明这些光周期基因的突变如何导致光周期反应的逆转。两种小麦CO1同源物在两者中都高度上调体育变种人,而二氧化碳同源物不受相同突变的影响,这表明这两种同源物受光敏色素基因的不同调控。有趣的是,CO1WT中SD的转录水平较高,LD的转录水平较高体育突变导致基因型和光周期之间的强相互作用(图2)。7).我们还确定了TraesCS7A01G211300,它编码的是BdCONSTANS-Like 1(附加文件6).该基因在WT基因型的LD中表达上调,而在SD基因型中表达上调、零和phyC零突变体。有趣的是,这个基因在Brachypodium elf3-null突变体,提示TraesCS7A01G211300和BdCONSTANS-Like 1同源物可能有相似的调控机制[5]。
我们无法就……的作用得出结论VRN2基因座(重复基因)ZCCT1和ZCCT2),因为功能ZCCT-B2a和ZCCT-B2b基因在我们研究中使用的参考基因组中没有注释,并且没有功能ZCCT-A1(TraesCS5A01G541300)和ZCCT-A2基因(TraesCS5A01G541200)低水平表达(附加文件5).
在分析开花时间基因的表达谱时,重要的是要记住,RNA-seq数据代表了白天和植物发育过程中多个开花基因相互作用的一个非常动态的过程中的单个时间点。因此,如果在不同的时间或发育阶段分析,这些表达谱可能会发生变化。尽管存在这一限制,但这一单一时间点产生的信息为了解小麦发育响应光敏色素信号的复杂网络提供了重要的见解。
光敏色素影响植物的结构和营养发育
除了开花时间,我们还发现、和PHYC都与营养发育的差异有关。在SD和LD光周期中,叶片、零和phyC-null突变体比WT更长、更宽,这表明它们的生长范围更广或更强劲(图2)。1e-f, (28,31])。这与phyC-零突变表型Brachypodium。的前四叶phyC-null植株在SDs条件下比WT短,在LD条件下长度差异不显著[30.]。这种差异可能是由于发育阶段,因为在我们的研究中,我们测量了旗叶和叶片Brachypodium,研究了嫩叶。
减少分蘖数、零和phyC-null突变体相对于WT(图2)。1 c),尽管它们的航向时间较晚(图2)。1A-b)表明、和PHYC可能对分蘖数的促进作用独立于它们对抽穗时间的影响。据报道,分蘖数也有类似的减少、高粱的突变体[54]。在这个突变体中,腋窝分生组织的形成和芽的形成是正常的,但与WT相比,从种植后6天开始,芽的生长受到抑制。这种生长停滞与转录水平的增加有关墨西哥类蜀黍Branched1(SbTB1)和与休眠有关的基因[54,55]。
这些等位基因对株高的影响在不同光周期间存在显著差异。而在LD条件下、零和phyC-null突变系比WT短(分别短51.1 cm和59.6 cm) [31]),在SDs中,两个突变体均显著高于WT (31.0 cm)、-null和22.1 cmphyC零,无花果。1g).有趣的是,尽管总身高为、和phyC如果相似,每个突变体的茎发育是不同的、-null突变体表现出更多的节间数(图2)。1g).有几个基因受、但不是PHYC这可能与这些表型差异有关。在SD和LD中,GA20ox-B2和GA20ox-B4,编码GA生物合成酶,显著高于、-null突变比WT或phyC-null(附加文件4).
光敏色素基因的突变也影响短日草的植株形态。在非诱导性LD条件下,与具有功能光敏色素的姐妹系相比,没有功能光敏色素的水稻抽穗时间较早,穗部较短PHYC在一个巴、背景(42]。在巴、背景下,PhyC基因还影响叶绿素含量、叶片角度和晶粒大小,证实了光敏色素突变体在禾草中的多重多效性。高粱植株携带无功能、等位基因对蓝光的响应表现为下胚轴生长拉长[56]在诱导光周期内茎伸长和节间数增加[57]。类似地,拟南芥,一种ld植物,在、突变体(58]。这些营养表型是遮荫回避反应的特征,这是介导的、在SD和LD物种中均存在。表型上的相似性表明、可能在小麦的避荫途径中发挥作用,而在上述其他植物物种中是保守的。多种基因中的一些在、但不是PHYC可能在避荫反应中起作用。
我们的转录组学结果也与先前的研究一致,这些研究已经建立了光敏色素与冷调节途径之间的联系[59,60]。在大米、、-null突变体表现出更好的耐寒性[61],在拟南芥中,PIF3结合CBF抑制其表达的基因[62]。我们确定了四个CBF基因和二天哪在光敏色素无突变体中高度表达的基因(图2)。5和6).成绩单四级天哪基因在SD中显著高于LD,在WT中也显著高于LD体育突变体,但差异更大体育突变体。这表明,在温暖的环境温度下,两者、和PHYC在白天抑制冷反应途径的激活。在小麦中,光照质量和耐寒性之间的联系已经被发现[63]并表明光敏色素的不稳定性对FR光(通常在黄昏较高水平)或黑暗的响应提高了整体的耐寒性。在生理水平上测试Kronos光敏色素突变体的耐寒性将是一件有趣的事情。
结论
功能丧失突变体的特征、和PHYC在四倍体小麦中发现,这些基因同时调控营养发育和抽穗时间,但不同品种间差异较大体育-null突变体和LD下的WT大于SD下的WT。的主要作用、和PHYC在低温胁迫下,这些基因在抑制开花方面也发挥了一定的作用。本研究的RNA-seq结果鉴定了一些开花基因在光周期和植物之间表现出显著的相互作用、/ PHYC这些等位基因可能导致光敏色素在不同光周期对抽穗时间的相反影响。他们还揭示了由、和PHYC在SD或LD光周期中,或在两个光周期中,提供对小麦光信号通路的见解。
方法
植物材料,生长条件和表型测量
本研究的所有植物材料均来自于四倍体中经ems诱变的TILLING群体小麦属植物turgidumlssp。硬质Desf。品种“Kronos”(基因组AABB),由Jorge Dubcovsky博士的实验室开发,并在之前[64]。的、零和phyC从该群体的外显子组测序系中鉴定出-null突变体[65]和之前的描述[28,31]。简单地说,我们通过标记辅助选择将每个基因A和B同源的零突变结合起来,并进行两次回交以减少背景突变。我们对突变体进行了几代自花授粉,并使用了BC2F4、-null和BC2F5phyC-null突变体用于RNA-seq研究。WT线对应于回交中使用的相同的克洛诺斯父代。在这个实验中所有的植物都携带了Ppd-A1a对光周期敏感性降低的等位基因[11]。所有植物在SD条件下(8 h光照/16 h黑暗)生长(PGR15, Conviron, Manitoba, Canada), 20°C昼/18°C夜,光照强度为~ 260 μM−2年代−1。所有实验室包括本次SD实验和之前的LD实验[31使用类似的卤化物灯配置,并位于同一房间。
抽穗时间被记录为播种后穗从靴部长出一半的天数(Zadoks 55 [66]),每个基因型使用5个生物重复(n)。所有生理测量在成熟时一次完成。我们测量了总高度和单个节间长度(n= 4),总分蘖数、叶数、旗叶宽度和长(n每个基因型= 6)。我们将抽穗时间的SD数据与先前发表的在LD下相同生长室配置下生长的相同突变体的抽穗数据进行了比较[31]。
存在化验
用于收获组织进行qRT-PCR实验的植物在上述条件下生长。从植株两周大开始,我们每隔三周在液氮中收集最后一片完全展开的叶片的组织,直到播种后17周,以覆盖突变体的大部分发育阶段。所有样品在生长室(ZT4)亮灯后4 h采集。我们收集了所有三种基因型(WT,、零和phyC-null)在每个时间点,使用单一野生型基因型作为两个突变体的对照。我们按照制造商的说明使用Spectrum™Plant Total RNA试剂盒(Sigma-Aldrich, St. Louis, MO)提取RNA。使用高容量逆转录试剂盒(Applied Biosystems)从1 μg的总RNA合成cdna,并在7500 Fast Real-Time PCR系统(Applied Biosystems, Foster City, CA)使用SYBR Green进行定量RT-PCR。目标基因的引物PPD1[28],FT1[15],FT2[16],FT-A3,FT-B3[67],VRN1[15],以及控制基因肌动蛋白[31前面已经描述过了。表达式数据以折线表示肌动蛋白水平(靶基因分子/肌动蛋白).
RNA-seq文库构建及测序
用于RNA-seq实验的单株植物与用于qRT-PCR和表型研究的植物相同。在SD RNA-seq实验中,我们从8周龄植物的每个基因型的4个生物重复中提取RNA样本。用一种野生型基因型作为两者的对照、零和phyC零突变体。在这个阶段,WT植株的顶端处于穗发育的早期阶段(Waddington阶段3 [33])和两者的顶端、零和phyC-零植株仍处于营养阶段(Waddington阶段1)[33])。LD RNA-seq实验的数据如前所述[31],由4周龄植物完全伸展的第三叶中提取的RNA产生,此时WT植株的顶端与8周龄sd植株处于相同的发育阶段。该数据有两个实验重复,每个重复包括16个样本(4个生物重复)、空,phyC -每个突变体的Null和各自的野生型姐妹系)。我们使用前面描述的方法组装并确定rna测序文库的质量[31]。文库被条形码以允许多路复用,并在加州大学戴维斯分校基因组中心的HiSeq4000测序仪上使用100 bp单读模块跨两条通道(每种基因型的两个生物复制,或每条通道6个文库)进行测序。
RNA-seq数据处理
使用先前描述的管道和参数对原始读数进行质量和适配器污染处理[31]。处理后的reads被映射到IWGSC RefSeq v1.0基因组组装中[68],使用GSNAPl [69]。我们对100 bp单端读取的SD数据使用参数- m2 -n 1 -A sam -n 1 -t 24,对50 bp单端读取的LD数据使用参数- m2 -n 1 -A sam -n 1 -t 24,为每个样本生成序列比对/地图(sam)文件。我们使用了IWGSC Refseq v1.0注释中的高置信度和低置信度基因模型。为了提供基因功能的额外背景,我们使用每个注释基因对NCBI NR蛋白质数据库进行了BLASTP搜索。我们还为编码不同转录因子家族成员的基因添加了额外的注释信息[70], MADS-box基因家族的MIKC亚类成员[71]和FT-like基因家族[67]。与每个差异表达基因相关的注释的完整信息在附加文件中提供2。
使用htseq-count (https://github.com/simon-anders/htseq)如前所述[31]。在三种基因型的任何复制中,原始计数值不大于或等于3的基因被丢弃,留下72,108个基因。使用DESeq2将这些剩余基因的原始计数归一化,并使用FDR阈值在基因型之间的两两比较中检测差异表达基因Padj在DESeq2和edgeR统计检验中均< 0.01 [72]。对于LD数据,我们分别对两个实验重复进行分析,只分析在两个比较中都显著的基因(在我们早期的研究中称为“高置信度”DE基因)[31]),被纳入本分析。
可变剪接
使用rMATS v4.0.1对选择性剪接事件进行表征[39]。使用Stringtie[]为SD和LD数据集创建了GTF注释文件。73]。该文件的输入是RNA-seq作图过程中生成的排序后的BAM文件和IWGSC RefSeq v1.1中用于指定外显子-内含子边界的高置信度和低置信度基因注释。rMATS使用的基因组索引是从IWGSC RefSeq v1.0汇编中使用STAR(参数—runMode genomeGenerate)创建的[74]。使用自定义perl脚本,将每个样本的Fastq文件分别修剪为100 bp和50 bp,分别用于SD和LD数据集。rMATS在每个数据集上运行两次,比较WT与、-null和WT withphyC- SD和LD数据集中的null样本,使用各自的GTF注释文件[68]。每个备选剪接事件的包含级别差异是根据映射到一个可能的包含事件的每个复制的读取次数来计算的,并通过这些可能事件的长度进行规范化。每种事件类型的值代表了四个WT重复的平均值和各自的两两比较体育零基因型。正的包含水平差异表明在WT中有更多的读取映射到AS事件体育-null样本,反之亦然。采用0.01%剪接差异初始值和FDR < 0.05筛选,确定了保留内含子、跳过外显子、替代5 '剪接位点、替代3 '剪接位点和互斥外显子等重要剪接事件。采用更严格的30%纳入水平差异的临界值来更详细地分析这些事件的一个子集。
功能注释
为IWGSC RefSeq v1.1基因组中的每个高置信度和低置信度基因生成GO术语的功能注释如前所述[31]。
数据和材料的可用性
RNA-seq读数和原始计数数据可在NCBI GEO (https://www.ncbi.nlm.nih.gov/geo/),登记号为GSE141000。先前发表的RNA-seq研究的原始数据,登录号为GSE79049。本研究的遗传材料可按要求提供。小麦耕作系可从Jorge Dubcovsky博士的实验室(https://dubcovskylab.ucdavis.edu/wheat-tilling/)或透过SeedStor (https://www.seedstor.ac.uk/).
缩写
- 方差分析:
-
方差分析
- 为:
-
选择性剪接。
- BAM:
-
二进制对齐图。
- bHLH:
-
基本螺旋环螺旋。
- 爆炸:
-
基本的局部对齐搜索工具。
- 林后:
-
寒冷的响应。
- 德:
-
差异表达。
- EMS:
-
Ethyl-Methane磺酸盐。
- 罗斯福:
-
错误发现率。
- FR:
-
远红。
- 走:
-
基因本体论。
- GTF:
-
基因转移格式。
- IWGSC:
-
国际小麦基因组测序联盟。
- LD:
-
漫长的一天。
- MDS:
-
多维标度。
- 子:
-
打开阅读框。
- PEBP:
-
Phosphatidylethanolamine-Binding蛋白质。
- 体育:
-
光敏色素。
- 论坛:
-
光敏色素相互作用因子。
- PRG:
-
光周期调控基因。
- PRR:
-
Pseudo-Response监管机构。
- PI:
-
光周期不敏感。
- PS:
-
光周期敏感。
- 存在:
-
定量逆转录酶聚合酶链反应。
- 接待员:
-
红色的。
- rMATS:
-
转录物剪接的多变量分析
- 山姆:
-
序列比对图。
- 扫描电镜:
-
均值的标准误差。
- TF:
-
转录因子。
- 耕作:
-
基因组中的靶向诱导局部病变。
- TPM:
-
每百万份成绩单。
- WT:
-
野生型。
- ZT型:
-
Zietgeber时间。
参考文献
- 1.
杨建军,李建军。植物对季节信号的遗传反应。[j] .地理学报。2012;13:627-39。
- 2.
发育途径是设计成功作物的蓝图。植物科学,2018;9:745。
- 3.
李春华,李春华,李春华,等。温带作物开花规律研究。植物学报,2009;12(2):888 - 888。
- 4.
Pearce S, Shaw LM, Lin H, Cotter JD, Li C . Dubcovsky jPhotoperiod1介导开花。植物生理学报,2017;34(4):1139 - 1139。
- 5.
高敏,耿峰,Klose C, Staudt A-M,黄宏,Nguyen D, Lan H, Mockler TC, Nusinow DA, hilthbrunner A, Schäfer E, Wigge PA, Jaeger KE。光敏色素测量短柄花序的光周期。bioRxiv。2019年,697169年。
- 6.
黄晓明,黄晓明,黄晓明,等。光敏色素光感受器的光依赖性核/胞质分配的分子机制。新植物学报,2015;26:965 - 71。
- 7.
罗克韦尔NC,苏苏YS, Lagarias JC。光敏色素的结构和信号传导机制。植物学报,2006;37(5):837 - 858。
- 8.
张志强,张志强。光敏色素的核定位研究进展[j] .植物化学学报,2006;10(3):559 - 568。
- 9.
Beales J, Turner A, Griffiths S, Snape JW, Laurie DA。伪响应调节器在光周期不敏感中被错误表达Ppd-D1a小麦突变体(小麦l .)。理论与应用学报。2007;15(1):721 - 733。
- 10.
Turner A, Beales J, Faure S, Dunford RP, Laurie DA。伪响应调节器Ppd-H1提供大麦对光周期的适应性。科学。2005;310:1031-4。
- 11.
威廉EP,特纳AS,劳瑞DA。光周期不敏感Ppd-A1a四倍体小麦(小麦属植物硬质Desf)。理论与应用,2009;118:285-94。
- 12.
具宝宝,刘世成,朴建伟,权成成,李国栋,安刚,张震,李俊,李忠,白国荣。自然变异OsPRR37调节抽穗日期,有助于广泛纬度的水稻种植。植物学报。2013;6:1877 - 1888。
- 13.
Murphy RL, Klein RR, Morishige DT, Brady JA, Rooney WL, Miller FR, Dugas DV, Klein PE, Mullet JE。光与时钟同步调节假反应调节蛋白(PRR37)控制高粱的光周期开花。科学通报,2011;38(1):1 - 4。
- 14.
肖LM,特纳AS,劳瑞DA。光周期不敏感的影响Ppd-1a六倍体小麦3个基因组光周期通路的突变小麦).植物学报,2012;31(1):1 - 4。
- 15.
闫丽,傅东,李超,Blechl A, Tranquilli G, Bonafede M, Sanchez A, Valarik M, Yasuda S, Dubcovsky J.小麦和大麦春化基因VRN3的同源物是英国《金融时报》。科学通报,2006;33(3):591 - 591。
- 16.
李春华,李春华,李春华,等。小麦FT蛋白的调控VRN1通过与FDL2的相互作用进行转录植物学报。2008;35(5):533 - 536。
- 17.
李超,林海,陈安,刘敏,Jernstedt J, Dubcovsky J. WheatVRN1,FUL2和FUL3在小穗发育和穗决定中起关键和多余的作用。发展。2019;146:dev175398。
- 18.
李超,林慧,Dubcovsky,等。小麦和大麦花原激活复合物的转录因子组合。植物学报,2015;34(4):870 - 882。
- 19.
吕波,Nitcher R,韩旭,王松,倪峰,李凯,Pearce S,吴健,Dubcovsky J,傅东开花位点t1(FT1基因Brachypodium和小麦。科学通报,2014;9:e94171。
- 20.
pif:细胞信号中枢的关键成分。植物学报,2011;16:19-28。
- 21.
范文娟,王晓明,王晓明。光敏色素与光敏色素相互作用的研究进展。植物生理学报。2018;36(6):1025 - 1038。
- 22.
陈晓明,陈晓明,陈晓明,等。光敏色素诱导PIF5快速磷酸化和降解的研究进展。植物生理学报。2007;45(5):563 - 567。
- 23.
Shikata H, Hanada K, Ushijima T, Nakashima M, Suzuki Y, Matsushita T.光敏色素调控拟南芥的选择性剪接。科学通报,2014;31(1):481 - 481。
- 24.
董军,陈华,邓小文,魏宁。光敏色素B诱导内含子保留和光敏色素相互作用因子3的翻译抑制。植物物理学报。2019;42(2):1509 - 1509。
- 25.
林碧比,石俊杰,谢海燕,陈慧慧,涂世林。光敏色素与hnRNP协同作用调控外显子剪接沉默子的选择性剪接。植物生理学报。2020;32(2):444 - 444。
- 26.
Shikata H, Shibata M, Ushijima T, Nakashima M, Kong SG, Matsuoka K, Lin C, Matsushita T.拟南芥剪接因子RRC1 RS结构域在光敏色素B信号转导中的作用。植物科学学报,2012;37(2):379 - 379。
- 27.
裴刚,崔刚。植物光敏色素及其相互作用蛋白的光信号解码。植物学报,2008;39(5):591 - 591。
- 28.
陈安,李超,胡伟,刘美梅,林宏,Rockwell NC, Martin SS, Jernstedt JA, Lagarias JC, Dubcovsky J.光敏色素C在长日光期小麦开花加速中的重要作用。科学通报,2014(1):1 - 4。
- 29.
西田H,石原D,石井M,金子T,川东H,明石Y,赛正D,田中K,韩田H,武田K,加藤K。光敏色素C是控制大麦长日开花的关键因子。植物生理学报,2013;33(3):564 - 564。
- 30.
Woods DP, Ream TS, Minevich G, Hobert O, Amasino RM。光敏色素C是温带禾本科植物光周期开花必不可少的光受体。Brachypodium distachyon。遗传学。2014;198:397 - 408。
- 31.
Pearce S, Kippes N, Chen A, Debernardi JM, Dubcovsky J.小麦rna序列研究光敏色素B和光敏色素C突变体揭示了在开花和避荫途径的调节中共享和特定的功能。植物学报,2016;16(6):1441。
- 32.
肖立明,刘波,特纳瑞,李超,陈飞,韩旭,傅东,杜布科夫斯基。开花位点t2调节温带谷物穗发育和肥力。[J] .中国生物医学工程学报,2019;33(2):391 - 391。
- 33.
Waddington SR, Cartwright PM, Wall PC。大麦和小麦穗初生和雌蕊发育的定量尺度。中华医学会杂志。1983;51(1):119 - 30。
- 34.
金士林,李松,金海军,南洪,安刚。OsMADS51是上游起作用的短日开花启动子吗Ehd1,OsMADS14,Hd3a。植物生理学报。2007;45(5):544 - 544。
- 35.
夏尔玛,李建军,李建军,李建军,李建军,李建军,李建军,李建军Brachypodium在小麦中受低温调控。植物生理学报,2017;33(3):1301 - 1501。
- 36.
李波,王勇,张勇,田伟,庄坤,张建超,王磊。PRR5、7和9通过抑制TOR信号正调控根细胞增殖串联锌指1在拟南芥。核酸学报,2019;47(5):591 - 591。
- 37.
福尔S,希金斯J,特纳A,劳瑞DA。的开花地点大麦(大麦芽).遗传学。2007;176:599 - 609。
- 38.
刘建军,刘建军,刘建军,等VRN-1正常和转基因多倍体小麦的春化基因。植物生理学报,2005;38(2):564 - 564。
- 39.
沈松,朴建伟,卢志新,林林,Henry MD,吴永宁,周强,邢宇。rMATS:基于复制RNA-Seq数据的差分选择性剪接检测。中国科学:自然科学版,2014;33(2):591 - 591。
- 40.
Childs KL, Miller FR, Cordonnier-Pratt MM, Pratt LH, Morgan PW, Mullet JE。高粱光周期敏感基因;马Plant Phyiol. 1997;113:611 - 69。
- 41.
杨建军,李建军,李建军,李建军,李建军。光敏色素B通过激活蛋白的表达抑制长日开花SbPRR37和SbGHD7,抑制因子SbEHD1,SbCN8和SbCN12。科学通报,2014;9:e105352。
- 42.
李艳,郑超,张志,周健,张宏,谢霞。光敏色素C在水稻去黄化及农艺性状中的调控作用。植物生理学报。2019;42(2):117 - 24。
- 43.
高野M,稻垣N,谢新,Yuzurihara N, Hihara F, Ishizuka T, Yano M, Nishimura M, Miyao A, Hirochika H, Shinomura T.光敏色素A、B和C在水稻脱腐和开花中的不同协同作用。植物学报。2005;17(3):391 - 391。
- 44.
Dubcovsky J, Loukoianov A, Fu D, Valarik M, Sanchez A, Yan L.光周期对小麦春化基因调控的影响VRN1和VRN2。植物化学学报。2006;26(2):469 - 498。
- 45.
一些冬小麦品种花序形成的短日诱导。[J] .植物生理学报。1987;14(2):277 - 86。
- 46.
张勇,李建军,李建军。大麦和小麦日中性突变对光周期与春化互作的影响。理论与应用,2013;26(6):1267 - 1277。
- 47.
Woods D, Dong Y, Bouche F, Bednarek R, Rowe M, Ream T, Amasino R。eLife。2019; 8: e42153。
- 48.
Mulki MA, Bi X, von Korff M.开花位点T3控制小穗形成,但不控制花发育。植物生理学报,2018;18(2):1170 - 1186。
- 49.
秦志,白燕,Muhammad S,吴霞,邓萍,吴军,安华,吴林。不同日照长度下FT-like 9在开花转变中的不同作用Brachypodium distachyon。自然科学学报。2019;10:812。
- 50.
藤野K,山内U,野上Y, Obara M, Yano M.开花时间基因转换的遗传效应即在LD条件下Ghd7和OsPRR37在大米。畜牧学报,2019;69:127-32。
- 51.
张震,胡伟,沈刚,刘海,胡艳,周霞,刘涛,邢宇。长日照条件下Hd1在抑制或促进抽穗中的作用取决于Ghd7的状态。科学通报,2017;7:5388。
- 52.
Sawa M, Kay SA。GIGANTEA直接激活开花位点T在拟南芥。科学通报,2011;38(1):1 - 9。
- 53.
Hayama R, Yokoi S, Tamaki S, Yano M, Shimamoto K.水稻短日开花的光周期调控途径的适应。大自然。2003;422:719-22。
- 54.
Kebrom TH, Burson BL, Finlayson SA。光敏色素B抑制墨西哥类蜀黍Branched1对光信号的表达及诱导高粱腋芽生长。植物生理学报。2006;14(4):1109 - 1107。
- 55.
Kebrom TH, Mullet JE。分蘖芽转录组分析为研究PhyB对高粱分蘖和不确定生长的调控提供了新的思路。植物生理学报,2016;17(2):1232 - 1250。
- 56.
Childs KL, Cordonnier-Pratt M-M, Pratt LH, Morgan PW。发育的遗传调控高粱二色的:七世。妈3.R开花突变体缺乏在绿色组织中占主导地位的光敏色素。植物物理。1992;99:765-70。
- 57.
[3]李春华,李春华。植物成熟与身高的生理与遗传关系。高粱:起源、历史、技术和生产。编辑史密斯CW,弗雷德里克森RA。威利;2000:227 - 326。
- 58.
光敏色素在拟南芥发育中的作用。[J] .中国生物医学工程学报,2010;31(1):1 - 4。
- 59.
富兰克林KA,怀特拉姆GC。耐冻性的光质调控拟南芥。[j] .生物医学工程学报。2007;39(9):1410 - 3。
- 60.
Lee CM, Thomashow MF。植物c -重复结合因子(CBF)冷驯化途径的光周期调控与抗冻性拟南芥。科学通报,2012;39(4):391 - 391。
- 61.
何艳,李艳,崔玲,谢玲,郑超,周刚,周健,谢霞。光敏色素B通过调控对耐寒性产生负性影响OsDREB1水稻光敏色素相互作用因子样蛋白OsPIL16的基因表达植物科学,2016;7:1963。
- 62.
蒋波,石燕,张欣,辛霞,齐丽,郭华,李军,杨松。PIF3是水稻生长发育的负调控因子CBF途径与抗冻性拟南芥。中国科学:自然科学版,2017;31(2):591 - 591。
- 63.
Novák A, Boldizsár A, Ádám E, Kozma-Bognár L, Majláth I, bamatga M, Tóth B, Chibbar R, Galiba G.光质量与温度的关系CBF14基因表达调节谷物的抗冻性。[J] .中国生物医学工程学报,2016;22(1):389 - 391。
- 64.
王晓明,张晓明,张晓明,张晓明,张晓明,张晓明,等。小麦四倍体和六倍体诱导突变的遗传分析。植物学报,2009;9(1):115。
- 65.
Krasileva KV, Vasquez-Gross HA, Howell T, Bailey P, Paraiso F, Clissold L, Simmonds J, Ramirez-Gonzalez RH, Wang X, Borrill P, Fosker C, Ayling S, Phillips AL, Uauy C, Dubcovsky J。科学通报,2017;35(4):913 - 921。
- 66.
赵建军,张涛,孔泽克,等。谷物生长阶段的十进制编码。医学杂志。1974;14:15 - 21。
- 67.
韩伟杰,高德华,高德华,高德华,高德华,高德华,高德华,高德华,高德华,高德华,高德华,高德华开花地点-like Poaceae基因家族鉴定短日表达开花途径基因;TaFT3在小麦(小麦l .)。植物科学,2016;7:857。
- 68.
国际小麦基因组测序联盟(IWGSC)。利用全注释参考基因组改变小麦研究和育种的限制。科学。2018;361:6403。
- 69.
Wu TD, Watanabe CK。GMAP: mRNA和EST序列的基因组定位和比对程序。生物信息学。2005;21:1859 - 75。
- 70.
Ramírez-González RH, Borrill P, Lang D, Harrington SA, Brinton J, Venturini L, Davey M, Jacobs J, van Ex F, Pasha A等。多倍体小麦的转录景观。科学。2018;361:6403。
- 71.
李建军,李建军,李建军,等。小麦MADS-box基因的全基因组分析。新植物学报,2019;25(5):511 - 29。
- 72.
刘建军,刘建军。控制错误发现率:一种实用且强大的多重测试方法。[J] .社会科学与技术。2007;22(1):1 - 3。
- 73.
Pertea M, Pertea GM, Antonescu CM, Chang TC, Mendell JT, Salzberg SL. StringTie可以改进RNA-seq读取的转录组重建。生物工程学报。2015;33:290-5。
- 74.
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,等。超快速通用rna序列比对技术。生物信息学。2013;29:15-21。
致谢
不适用。
资金
JD承认来自霍华德休斯医学研究所的研究人员工资和实验室消耗品和试剂的财政支持,以及来自美国农业部国家粮食和农业研究所的农业和食品研究倡议竞争补助金2017-67007-25939 (WheatCAP)。HB和AA承认TUBITAK资助的实验室耗材和测序费用。AA奖学金由Tubitak-BİDEB奖学金提供。
作者信息
从属关系
贡献
NK开发植物材料,进行所有表型分析和分子实验,进行数据分析,并参与撰写稿件。CVG、JH、AA、HB进行表达数据分析。JD参与了项目的初步协调、数据分析和手稿的撰写。SP进行数据分析并撰写稿件。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非在数据的信用额度中另有说明。
关于本文
引用本文
基普斯,N.,范塞尔,C.,汉密尔顿,J.。et al。的影响、和phyC短日照和长日照条件下小麦转录组的功能缺失突变。BMC Plant Biol20.297(2020)。https://doi.org/10.1186/s12870-020-02506-0
收到了:
接受:
发表:
关键字
- 小麦
- 标题日期
- 光敏色素
- FT1
- FT2
- 发生
- PPD1
- VRN1