抽象的
背景
远缘杂交是创造水稻种间遗传变异和培育新品种的重要途径。为解决水稻科学问题,已构建了大量回交渐渗系。然而,对栽培稻、野生稻及其BIL后代中关键调控因子lncRNA的研究报道甚少。
结果
这里,使用高通量RNA测序技术探讨LNCRNA的功能特征和差异o .漂白亚麻纤维卷,O. longistaminata.和公元前3年2F12.后代。共筛选出1254个lncrna,以及子代与子代之间差异表达的lncrna数量o .漂白亚麻纤维卷明显少于后代和O. longistaminata..一些LNCRNA调节多于一个mRNA,89.5%的LNCRNA通过靶基因的表达调节通过CIS.代理。共有78个lncrna和271个mrna被280个mirna靶向,22个lncrna被预测为20个microrna的前体。一些mirna被发现以自身潜在的前体lncrna为靶点。在三个子代中,超过50%的lncrna表现出亲本表达水平优势(ELD),大部分lncrna表现出ELD- o。漂白亚麻纤维卷而不是eld-O. longistaminata..进一步分析表明,LNCRNA可能调节植物激素相关基因的表达和适应性o .漂白亚麻纤维卷,O. longistaminata.和他们的后代。
结论
综上所述,这些结果为阐明不同lncrna的功能特征和表达差异提供了有价值的线索o .漂白亚麻纤维卷,O. longistaminata.及其Bil Pronsies,并扩大了我们对稻米中LNCRNA的生物功能的理解。
背景
米 (栽培稻L.)是世界人口的主食之一,其产量对于全球粮食生产至关重要。随着基因组大小和高质量参考基因组的优点,大米被广泛被认为是典型的模型植物,以研究单子叶植物的遗传结构和功能。Genus.选用已进化成24种,包括2种栽培种(o .漂白亚麻纤维卷和o . glaberrima)及22种野生物种[1,2].经过长时间的人工选择,栽培水稻失去了许多重要的有用基因。然而,野生稻经历了恶劣的自然环境,含有大量有价值的基因,是水稻育种的宝贵资源[3.].具有优良基因和远缘遗传关系的不同物种之间的种间远缘杂交是创造遗传变异和培育新品种的重要途径,也是基因组进化和物种形成的重要驱动因素[4].从野生稻中鉴定和利用具有农艺性状的有价值等位基因,通过杂交回交将其引入栽培稻,是扩大栽培稻遗传多样性的一种普遍接受的有效策略[5,6].在过去的几十年里,人们构建了大量的回交渐渗系(backcross -渐渗系,BILs)来研究水稻的抗旱性等科学问题[7,8基因组结构[3.,杂种不育性[5基因、miRNA和代谢分析[2,9].O. longistaminata.是一种多年生野生稻,广泛分布在热带非洲,对生物和非生物胁迫,强根茎,长花药和自我不相容的强烈抵抗力[10.,11.,12.].
长链非编码RNA (Long non-coding RNA, lncRNA)是指长度超过200bp且无蛋白编码能力的RNA。lncrna参与多种分子和遗传机制,包括转录水平、转录后水平和表观遗传水平[13.,14.].LNCRNA参与许多生物学过程,包括人肿瘤细胞的生长,植物形态发生,生物胁迫和非生物威胁[15.,16.,17.].lncrna的功能分为信号、诱饵、引导和支架四类[18.].具体地,当它们充当信号分子时,LNCRNA可以调节基因的空间/时间表达[19.].lncrna可作为mirna的诱饵或靶拟态物参与维持基因表达的稳定性[20.,21.,22.].lncrna还可以引导核糖核蛋白复合物定位到特定位点,发挥其引导作用[23.].此外,lncrna可以作为支架与转录因子形成骨架复合物,调控上下行效应元件,进一步激活或抑制基因的转录[24.].此外,lncrna可以通过在转录水平调控蛋白编码基因的表达CIS.- 或者反式监管。当lncRNACIS.-调控靶基因,它是从其靶基因所在的核酸链中编码的。相反,lncRNA编码的核酸序列与目的基因编码序列不在同一个核酸链上反式-调控靶基因。例如,一个lncRNA (COLDAIR)CIS.- 解释这一点开花基因座C.(方法)基因,在拟南芥中的开花时间的调节中非常重要;寒冷也反式- 解释这一点方法基因与蛋白质复合物PCG结合[25.,26.].此外,lncrna可以作为microrna (mirna)的前体,部分lncrna还可以直接与mirna结合调节其功能[27.].虽然在以往的研究中已经鉴定出了大量的lncrna,但对其生物学功能的研究仍处于起步阶段,尤其是在植物中。
近年来,高通量测序技术常被用于检测低水平表达的转录本,识别大量在生物过程中发挥重要作用的mrna、小rna和lncrna [15.,16.,28.,29.].在我们以前的研究中,基因和miRNA的表达模式o .漂白亚麻纤维卷,O. longistaminata.和他们的3个BIL子代进行了实验,并探索了mirna对基因的调控[2].本研究采用高通量链特异性RNA测序(ss-RNAseq)技术,研究lncrna及其靶基因的表达差异及特征o .漂白亚麻纤维卷,O. longistaminata.和他们的三个BILs (BC2F12.)的后代(L1710、L1817和L1730)。我们还研究了这些物种中作为mirna前体或靶模拟物的lncrna。进一步分析表明,亲本表达水平显性(ELD)现象在3个子代中最为常见。这为揭示野生稻杂交回交基因渗入的分子机制提供了有价值的线索。
结果
测序数据概述
探讨lncrna的表达特点及其在肿瘤中的作用o .漂白亚麻纤维卷,O. longistaminata.本研究采用链特异性RNA-seq (ssRNA-seq)技术。总体而言,每个样本平均获得13.70 Gb数据,3个生物重复之间的基因表达相关性较高,平均系数(R2)的0.98(补充图S1).平均为101,808,785 (L1710)、91,859,276 (L1817)、88,566,772 (L1730)、98,879,278 (o .漂白亚麻纤维卷)和103,545,393(O. longistaminata.)生成的原始读数分别产生,其中超过94%的读数是干净的读取(补充表S1).15个ssRNA文库测序获得的所有干净reads上传到NCBI 's Sequence Read Archive (SRA)数据库(https://trace.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=announcement)使用加入号SRR9822767-SRR9822781。重新组装和映射后,约为56%的清洁阅读O. longistaminata.在三个子代中,70%的读数是干净的o .漂白亚麻纤维卷被唯一定位到水稻参考基因组(Supplementary Table S1)和66,338个转录本被鉴定为已知mrna。此外,将已知信息无法识别的mrna和转录本剔除,其余转录本(下文称为新转录本)进一步确定为lncrna的候选转录本。结果共组装了16038个新转录本,其中大部分的转录本长度在4500 nt以内,包含10个以上外显子的转录本比例较高,而近一半的基因只有一个转录本(Supplementary Table S)2).
LNCRNA的识别与序列特征
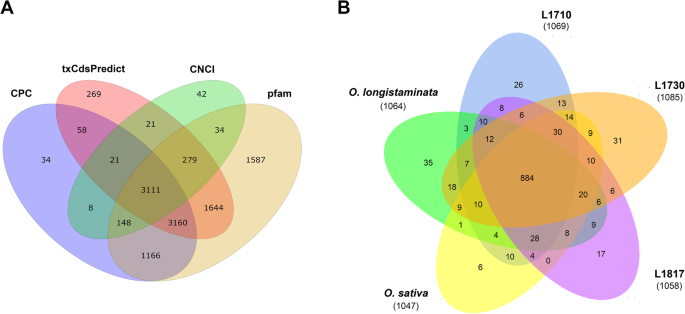
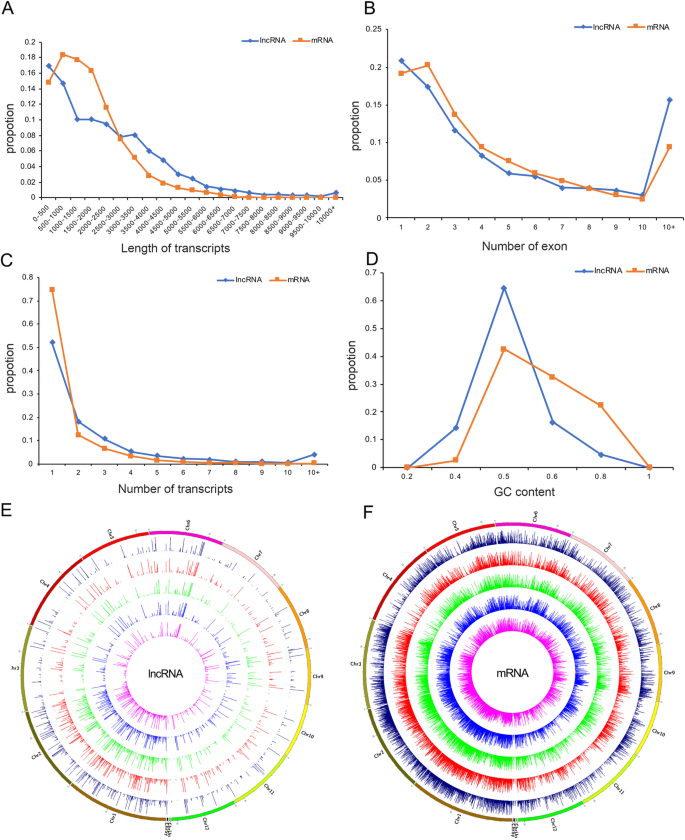
来识别o .漂白亚麻纤维卷,O. longistaminata.及其三个BIL后代,使用三种软件(CPC,CNCI和TXCDSPREDICT)和PFAM蛋白质数据库预测了16,038个新转录物的编码能力。通过预测其编码能力,共鉴定了总共6719个新的LNCRNA(图。1a),通过RSEM软件定量分析筛选出1254个新的lncrna(图1)。1b,表1及补充表S3.).另外,通过与mrna的序列比较,对所鉴定的lncrna进行序列特征分析。lncrna的长度在200 ~ 18313 bp之间,平均2348 bp,比已知mrna(平均1708 bp)长(图2)。2一种)。大约50%的LNCRNA长度超过2000磅,其中42克尔中的42磅长于10,000bp。编码LNCRNA的基因的外显子数与已知的mRNA编码基因的基本上一致,49.8%的LNCRNA编码基因和53.2%分别含有1-3个外显子(图。2b)。最着名的MRNA(87.1%)和LNCRNA(70.1%)衍生自具有一个或两个转录物的基因(图。2c). lncrna编码基因的GC含量在23.45 ~ 78.93%之间变化,平均为46%,其中大部分(79%)GC含量小于50%(图2)。2d),而已知mrna编码基因的GC含量在28.73 ~ 84.16%之间,平均为52.88,其中GC含量超过50%的占55%。综上所述,lncRNA和mRNA序列特征多样化,lncRNA长度大于已知mRNA,但编码lncRNA的基因外显子数小于编码mRNA的基因,lncrna编码基因的GC含量也低于已知mrna编码基因。
采用RSEM软件计算lncrna和mrna的表达水平。表达lncrna和mrna的数量o .漂白亚麻纤维卷,O. longistaminata.他们的三个生物后代显示在表格中1,并且它们的特定FPKM值显示在补充表S中3..如图所示。1B,70.5%(884个中的1254个)的LNCRNA在所有五条线中表达。仅在一行中表达的LNCRNA数量最多(35)O. longistaminata.最少(6)o .漂白亚麻纤维卷(无花果。1b)。此外,使用电池可视化表达的LNCRNA和MRNAs的分布(图。2E&F)。结果表明,在染色体1和2上表达了超过50%的LNCRNA和超过40%的MRNA,表达的LNCRNA的百分比高于表达的MRNA在染色体1,2和11上的百分比。
五行中的差异表达的LNCRNA
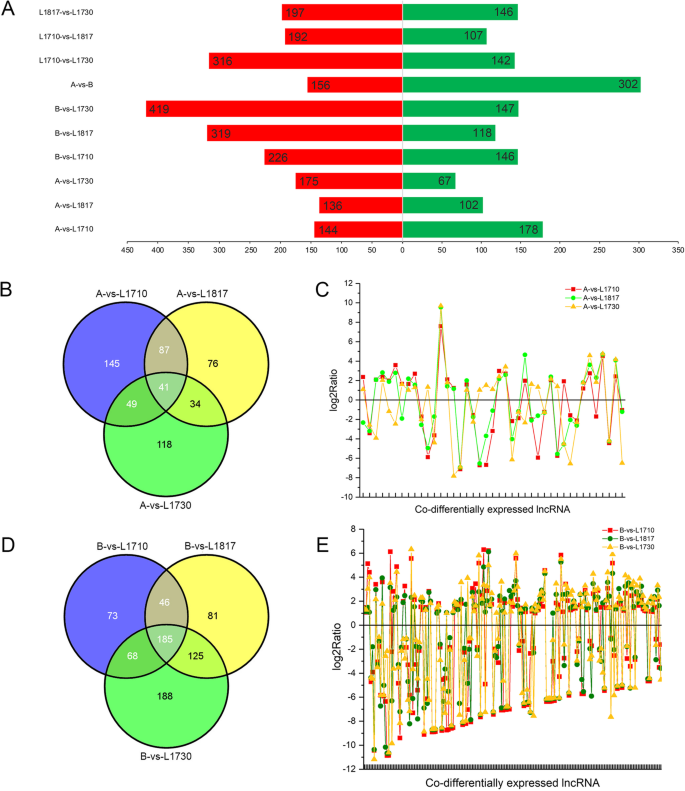
lncrna和mrna的FPKM值(Supplementary Table S .3.)分析各组间lncrna / mrna的差异表达o .漂白亚麻纤维卷,O. longistaminata.和他们的三个有|日志的BIL后代2FC | ≥ 1 and FDR ≤ 0.001 by DEseq software. The analysis of the differentially expressed lncRNAs (DE-lncRNAs) was shown in Fig.3..与亲本相比,de - lncrna在后代中大多上调(图2)。3.a).此外,子代与子代之间的DE-lncRNAsO. longistaminata.(平均458个,其中69.2%在子代中上调)显著高于子代。o .漂白亚麻纤维卷(平均267个基因,58.1%的基因在后代中表达上调)。同时,与两个亲本相比,三个子代分别发现了41个和185个常见的de - lncrna(图)。3.在这些已鉴定的常见de - lncrna中,子代中上调lncrna的数量也高于下调lncrna的数量(图2)。3.C&E)。上述结果表明,三个BIL后代和父母之间存在更大的差异O. longistaminata.,而在BIL子代中上调的de - lncrna可能发挥了关键作用。在三个子代的差异分析中,L1710对L1817中发现了299个DE-lncRNAs, L1710对L1730中发现了458个DE-lncRNAs(图)。3.a).这一现象与L1710和L1730株高差异最大是一致的。为了更详细地分析,我们统计了3个子代与亲本相比具有不同fold变化(FC > 2、FC > 10、FC > 50、FC > 100、FC > 200)的DE-lncRNAs(补充图S)2).随着de - lncrna FC的增加,比较组的lncrna数量增多,后代与亲本株高差异较大。例如,在三个子代和o .漂白亚麻纤维卷,L1710中具有FC> 2的DE-LNCRNA的数量最多,而具有FC> 50/100/200的DE-LNCRNA的数量是L1730中最大的。
LNCRNA靶蛋白编码基因的预测及其GO分析
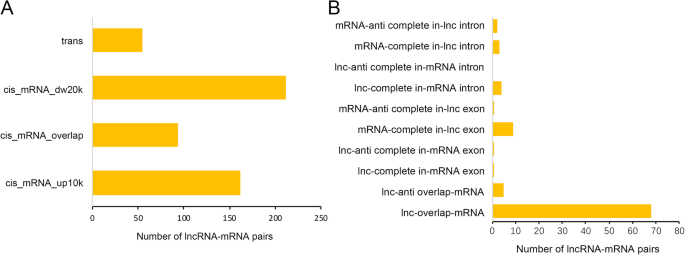
lncrna发挥生物学功能的一种方式是通过调节蛋白编码基因的表达CIS.要么反式互动。lncrnas可以通过一个调节靶基因的表达反式- 三和三CIS.-Reguted方法(CIS_MRNA_UP10K,CIS_MRNA_OVERLAP和CIS_MRNA_DW20K)(图。4a). cis_mRNA_overlap类可以进一步划分为10个子类(图1)。4b).目前的数据显示,523个lncrna中有89.5%(468个)通过调控靶基因的表达CIS.- 为此,其中45.3%(212例468)属于CIS_MRNA_DW20K监管类。这些结果表明CIS.监管而不是反式- 是主要的调节类型,和CIS.-调控位于靶基因下游20 kb处的lncrna是常见的CIS.- 在本研究中预测的LNCRNA-mRNA调节对中的类型。如补充表S所示4,共检测到431对lncRNA-mRNA调控对o .漂白亚麻纤维卷,O. longistaminata.及其三个BIL后代,其中373 mRNA是潜在的297lncRNA的靶标,表明一些LNCRNA可以同时调节多个MRNA。
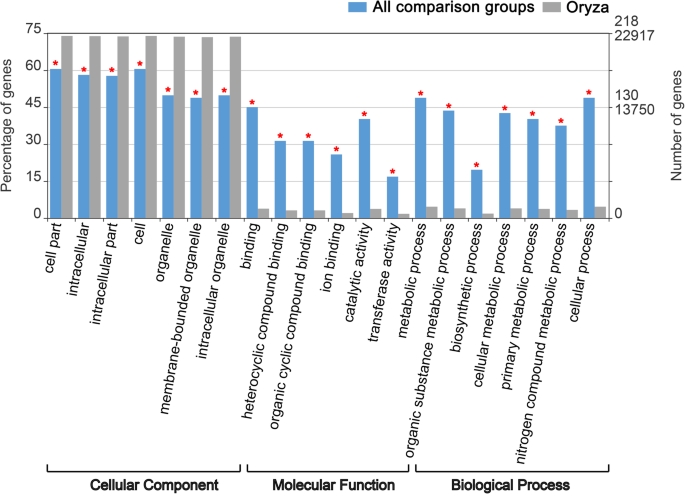
为了进一步了解de - lncrna在其中的作用o .漂白亚麻纤维卷,O. longistaminata.以水稻基因组为参考,对所有对照组的de - lncrna的靶基因进行GO富集分析(图)。5).结果表明,DE-LNCRNA的潜在靶基因显着富集(P< 0.001)。5).此外,在细胞组分类显著富集GO项中,de - lncrna的潜在靶基因百分比低于水稻基因组(背景),而在分子功能和生物过程类显著富集GO项中,de - lncrna的潜在靶基因百分比高于水稻基因组(背景)。这一现象表明,在分子功能和生物学过程范畴上显著富集的de - lncrna靶基因可能在调控的生长发育中发挥重要作用o .漂白亚麻纤维卷,O. longistaminata.和他们的三个BIL后代。
LNCRNA作用作为miRNA的前体
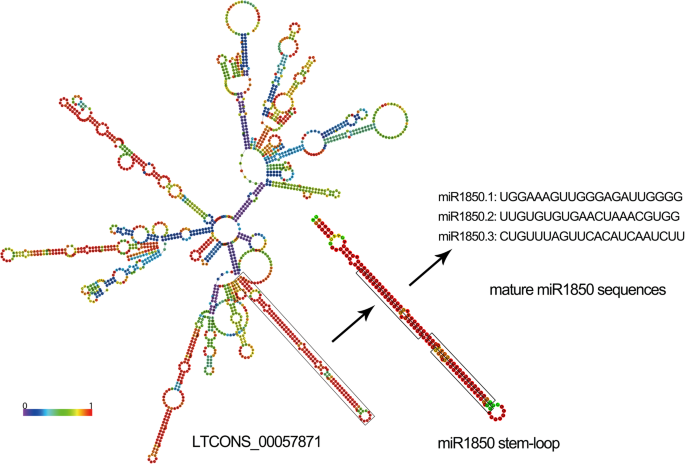
lncrna可以作为microrna (mirna)的前体。为了筛选这5个序列中的mirna前体,我们使用BLAST将lncrna的序列与miRbase数据库进行比对。如表所示2,共预测22个表达的lncrna为20个mirna的前体,其中18个lncrna为5个系谱的de - lncrna。大多数lncrna作为一个miRNA的前体,而其中两个(ltcon_00035053, ltcon_00007959)作为多个miRNA的前体(表)2).此外,几个lncrna也可能是同一个miRNA的前体。例如,ltcon_00034708和ltcon_00034707都是miR396c的前体(表2).另外,超过50%的预测为miRNA前体的lncrna被发现转录自2号染色体。使用RNAfold web服务器预测lncrna和miRNA前体的二级结构,可视化它们之间的关系。预测ltcon_00057871的二级结构包含多个茎环结构,其中一个是miR1850的潜在前体(图)。6).前体经酶处理后最终形成成熟mirna (miR1850.1, miR1850.2和miR1850.3)。
lncRNA作为mirna的诱饵或靶模拟物的分析
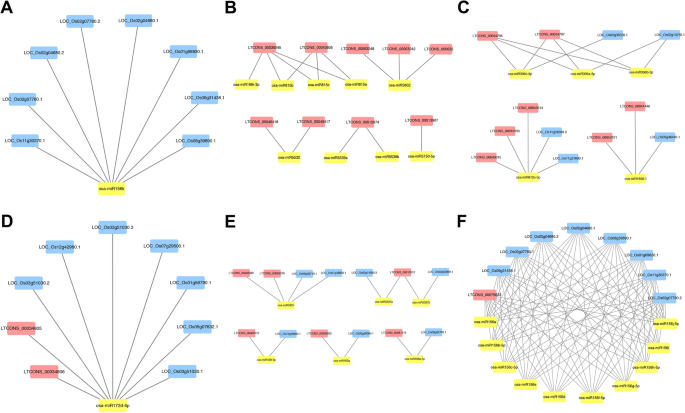
LNCRNA可以用作直接或间接调节靶基因表达的诱饵[30.].进一步探讨三种rna (lncRNA, miRNA, mRNA)在肿瘤中的作用o .漂白亚麻纤维卷,O. longistaminata.和他们的三个BIL后代,相互作用网络是基于之前的miRNA测序研究构建的[2].网络中280个mirna共靶向78个lncrna和271个mrna (Supplementary Table S)5).统计显示,72.2%(202 / 280)的mirna仅靶向mrna (Supplementary Table S5).例如,OSA-MIR156K靶向8 mRNA(图。7A),其中三个在水稻生长中被发现是重要的。具体来说,两个mrna (LOC_Os02g04680.1和LOC_Os02g04680.2)是两个转录本OsSPL3 (SQUAMOSA启动子结合蛋白like3),水稻中的强调根冠发育[31.].另一个mRNA (LOC_Os08g39890.1)为osspl14 / ipa1.,它可以监管DEP1(密集和直立胰腺1),是影响株高和穗长的关键基因[32.].此外,只有少量的miRNA(3.2%,9个,共280只)仅靶向的LNCRNA(补充表S.5, 图。7b).有24.7%(280个mirna中有69个)mirna同时靶向mrna和lncrna(补充表S)5).进一步分析发现,只有5种mirna靶向lncrna多于mrna(图)。7c),且当mirna同时靶向mrna和lncrna时,大多数mirna靶向mrna多于lncrna(84.1%, 58 / 69),这表明在大多数情况下lncrna与mirna的结合能力弱于mrna。例如,osa-miR172d-5p靶向2个lncrna和7个mrna(图)。7d),其中3个mrna (LOC_Os03g51030.1、LOC_Os03g51030.2和LOC_Os03g51030.3)为3个转录本凤凰(光敏色素的),在控制水稻节间伸长中起多重作用[33.].此外,有6个miRNA靶向等量的MRNA和LNCRNA(图。7e)。不同的miRNA也可以同时调节多个相同的目标(图。7F)。有趣的是,发现七个miRNA瞄准自己的潜在前体LNCRNA(表3.),表明这些LNCRNA不仅作为miRNA的前体,而且也可以与它们结合以参与靶基因表达的调节。
亲本表达水平显性分析
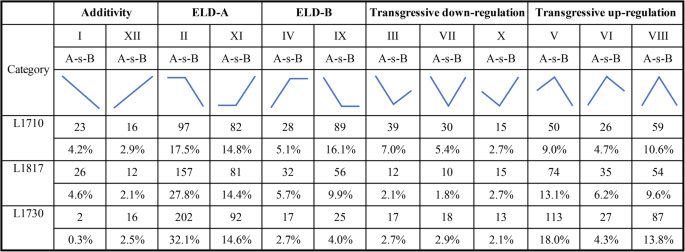
表达水平显性(Expression level dominance, ELD)是指某些基因在后代中的表达水平接近亲本一方,但不同于亲本另一方。最近,许多关于杂交种及其亲本的研究发现,mrna的表达在后代中表现出亲本ELD [2,29.,34.].根据Yoo等人定义的标准。[34.,将基因表达模式分为12类,如图所示。8.超过50%的lncrna表现出亲本ELD (II、XI、IV和IX类),约40%的lncrna在所有三个子代中均表现出越位上调/下调(III、VII、X、V、VI和VIII类)。此外,lncrna的数量显示亲本ELD-A (A代表o .漂白亚麻纤维卷)高于亲本ELD-B (B代表O. longistaminata.)。为了进一步了解ELD表达模式lncrna可能的生物学功能,我们将其潜在的靶mrna用于GO富集分析。如补充表S所示6在美国,大约30%的lncrna在每个子代中都有潜在的靶mrna。在L1817和L1730 GO的细胞组分、分子功能和生物学过程三个分类中,lncrna的靶蛋白数量显示ELD-A高于ELD-B (Supplementary Fig. S .)3.).然而,在L1710的三个GO类别中,lncrna显示ELD-A的靶标数量都小于ELD-B。此外,在所有三个子代中,ELD-A lncRNAs靶标均在“细胞”、“细胞部分”和“细胞器”区域富集,而ELD-B lncRNAs靶标均在“膜”区域富集(Supplementary Fig. S .)3.).此外,ELD-A lncRNAs靶点在L1710的‘binding’位点富集,而ELD-B lncRNAs靶点在L1817和L1730的‘binding’位点富集(补充图S)3.).在L1710和L1730中,ELD-A lncRNAs靶点富集在‘cellular process’区域,而在L1817中,ELD-B lncRNAs靶点富集在这一区域(Supplementary Fig S)3.).ELD-A lncRNAs靶标在L1730的“代谢过程”项中富集,而ELD-B lncRNAs靶标在L1710和L1817的“代谢过程”项中富集(补充图S)3.).总之,非上瘾基因在比尔后代和父母之间的比较中占了大多数,而且,所有三个后代(L1710,L1817和L1730)偏向o .漂白亚麻纤维卷.
QRT-PCR验证数据
为验证测序数据的准确性,随机选取9个lncrna及其可能的靶基因进行qRT-PCR分析。结果显示,测序数据与qRT-PCR结果基本一致(图1)。9),表明测序数据是可靠的。用于QRT-PCR的引物在补充表S7中列出。
讨论
近年来,lncrna作为一种调节性rna已成为研究热点。随着测序技术的发展,lncrna已经被发现在植物生长和有性生殖中发挥关键作用[14.,35.].具体地,研究表明,LNCRNA参与植物的雌性/雄性不育[36.,37.,38.,39.[植物应激响应过程[40,41].到目前为止,没有关于LNCRNA表达模式的报告及其在其父母的BIL后代的功能。基于我们之前对mRNA和miRNA表达模式的研究o .漂白亚麻纤维卷,O. longistaminata.他们三个生物的后代[2],对lncRNA表达模式的研究将有助于我们进一步了解BIL后代基因相对于亲本差异表达的调控因素。
lncrna可能参与调控植物激素相关基因的表达o .漂白亚麻纤维卷,O. longistaminata.和他们的BIL后代
植物激素可以独立地或以各种协调方式调节植物生长,开发和分化的生理反应。植物激素含量受相关基因表达的影响,而后续受到各种因素的转录和转录后水平的调节调节[29.,42,43].许多下调多聚腺苷酸化(DPA)的lncrna参与了水稻ABA的生物合成、运输和代谢,从而激活了一系列应激反应基因的表达[44].如下所述,在本研究中,一些LNCRNA靶向嗜酸甘油蛋白(GA),乙烯和生长素相关基因,其调节效应可能会对水稻的生长和发育产生影响。LTCons_00063919目标D35 / OsKO2(LOC_Os06g37364,补充表S4),编码一个ent-kaurene氧化酶(KO)的催化赤霉素生物合成,而没有该基因的水稻表现出严重的矮化表型[45].SLRL1(loc_os01g45860),成员肝基因家族,ltcon_00002962靶向(补充表S4), GA诱导表达SLRL1,过表达也会导致水稻的矮化表型[46].GID2.(LOC_Os02g36974)为ltcon_00033182的靶基因(Supplementary Table S4),在GA信号转导中调控一个抑制因子(SLR1)的降解,当水稻出现严重矮化表型时GID2.基因突变[47].此外,预测ltcon_00032876为目标OSCTR2.(LOC_Os02g32610,补充表S4),并且RAF样蛋白质组成型三重反应1(CTR1)参与乙烯受体信号转导,以调节大米中的多种生长和发育过程[48].此外,osarf24.是ltcon_00025454的靶基因(Supplementary Table S4),低表情Osarf23.和osarf24.会降低水稻对生长素的反应,从而影响水稻的生长和形态发生[49].因此,针对植物激素相关基因的lncrna可能调控植物的生长发育,影响植物的株高o .漂白亚麻纤维卷,O. longistaminata.和他们的BIL后代。
lncrnas可能会调节适应性o .漂白亚麻纤维卷,O. longistaminata.和他们的BIL后代
研究表明,LNCRNA参与植物对各种生物和非生物应激的反应[40,44,50].Jain等人。[41]从24个水稻抗稻瘟病系中鉴定了多个lncRNA候选基因,揭示了它们在水稻抗稻瘟病中的调控作用。许多含有DPA的lncrna可能在水稻在各种非生物胁迫(如热、冷、干旱和盐胁迫)下的生长中发挥重要作用[44].如补充表S所示4, LTCONS_00033755目标trehalose-6-phosphate磷酸酶(OsTPP1,loc_os02g44230),这是一个成员TPS /泛太平洋伙伴关系基因家族。的过表达OsTPP1能增强水稻对冷、盐胁迫的耐受性,同时激活多种胁迫响应基因的表达[51].编码的蛋白质GF14e(LOC_Os02g36974, ltcon_00033182的靶向基因,补充表S4)影响防御反应基因,细胞死亡和对细菌枯萎病和鞘疱疹的表达[52].同时,编码的蛋白质OsGF14e,正调控水稻穗瘟病抗性[53].此外,WRKY13(LOC_Os01g54600)是ltcon_00010291的目标(补充表S4),已知WRKY13可直接抑制WRKY42,而WRKY42可负调控水稻对稻瘟病菌(magnaporthe oryzae.),通过抑制JA信号相关基因[54].而且,OsPCF5由LTCons_00010204定位(补充表S4),作为TCP转录因子家族的成员,OsPCF5在水稻应对低温胁迫中起负作用[55].因此,上述四种LNCRNA可以调节抗病相关基因的表达o .漂白亚麻纤维卷,O. longistaminata.和它们的BIL后代,从而影响它们的适应性。
lncrnas可能会规范增长o .漂白亚麻纤维卷,O. longistaminata.通过竞争性地将miRNA与MRNA联系起来
一些lncrna作为诱饵与mirna结合,使这些mirna不能正常与靶基因结合,从而影响其对靶基因的调控[20.].在水稻花药、雌蕊和种子中发现了2种lncrna分别吸附miR160和miR164 [35.].本研究预测ltcon_00001063能竞争性结合miR169f。1与miR169o配合OSHAP2G.(LOC_Os07g41720)和OSHAP2H.(LOC_Os03g44540),为HAP复合体HAP2亚基的两个编码基因[56].由此推测,ltcon_00001063可能参与了水稻生长发育过程中HAP2亚基基因表达的调控。此外,预测ltcon_00034806和ltcon_00034805与miR172d-5p竞争结合光敏色素的(凤凰;LOC_Os03g51030),调控水稻节节伸长,在水稻营养生长期起关键作用[33.,57].因此,LTCONS_00001063,LTCONS_00034806和LTCONS_00034805可以通过具有特异性mRNA的竞争性结合miRNA来调节miRNA靶基因的表达水平,然后参与水稻的连接阶段的茎的生长过程。
两组间lncRNA表达的差异o .漂白亚麻纤维卷比BIL后代之间的小O. longistaminata.
我们以前的研究主要探讨了基因表达和miRNA条例o .漂白亚麻纤维卷,O. longistaminata.他们三个生物的后代[2].本研究对这些物种中lncrna及其潜在靶基因的表达特征进行了研究。这两项研究发现了一些共同之处。例如,之前的研究表明,后代和后代之间的deg的数量O. longistaminata.比后代和o .漂白亚麻纤维卷.类似地,子代与子代之间的de - lncrna的数量O. longistaminata.也高于后代和o .漂白亚麻纤维卷在这项研究中。此外,前期研究表明,多数基因表现出亲本的ELD,且有多个ELD-A (A代表o .漂白亚麻纤维卷)基因比Eld-B(B代表O. longistaminata.)基因在三个后代中被观察到。同样,在本研究的三个子代中,超过50%的lncrna显示亲本ELD,且显示亲本ELD- a的lncrna数量高于显示亲本ELD- b的lncrna。以上结果表明,在BIL子代和亲代之间lncRNA表达的差异o .漂白亚麻纤维卷比BIL后代之间的小O. longistaminata..以往的研究表明,大多数BIL后代的染色体补体是遗传自轮回亲本o .漂白亚麻纤维卷[2].因此,LNCRNA表达差异的最可能原因是BIL后代和每个父母之间的遗传背景差异。
结论
本研究采用高通量ss-RNA-seq技术,探讨lncrna的功能特征及差异o .漂白亚麻纤维卷,O. longistaminata.和他们的三个BIL后代。lncrna在5个细胞系中均有表达。对de - lncrna的分析表明,3个子代之间lncrna的差异o .漂白亚麻纤维卷,包括de - lncrna的数量和倍数变化,均大于3个子代之间的O. longistaminata..此外,与亲本相比,大多数de - lncrna在后代中表达上调。部分lncrna能调控mrna的表达,其中大部分为CIS.监管。当mrna和lncrna同时被靶向时,大多数mirna靶向更多的mrna而不是lncrna。一些mirna被发现以自身潜在的前体lncrna为靶点。三个子代中有超过一半的lncrna表现出亲本ELD,且有大量的lncrna表现出亲本ELD- o。漂白亚麻纤维卷高于父母的父母 -O. longistaminata.,这表明三个后代都倾向于o .漂白亚麻纤维卷.进一步分析表明,lncrna可能参与调控五种植物激素相关基因的表达,并调控其适应性。综上所述,这些结果为阐明不同lncrna的功能特征和表达差异提供了有价值的线索o .漂白亚麻纤维卷,O. longistaminata.及其自生物的后代,并扩大了我们对水稻中LNCRNA的生物功能的理解。
方法
植物材料
栽培稻ssp。籼稻简历。9311年,O. longistaminata.和他们的三个BILs (BC2F12.)后代(L1710、L1817和L1730)为材料。这些材料均来自武汉大学生命科学学院李少青博士的实验室,中国武汉。BC的建设2F12.由4个步骤组成:1)o .漂白亚麻纤维卷(母系)与O. longistaminata.(父系)产生F1杂种;2)与亲本杂交一代进行回交o .漂白亚麻纤维卷(母性)产生BC1F1;3)公元前1F1个人(父亲)被回交o .漂白亚麻纤维卷(母性)产生BC2F1;4)BC2F1最后产生于公元前2F12.通过11代单粒下降法自花受精。3个子代的基因组组成基本相同,大部分遗传自栽培稻(o .漂白亚麻纤维卷),而其中只有10%到15%是遗传自O. longistaminata.[2].如Cao等[2, 3个赤霉素后代的株高不同。其中5个株系成熟期株高排序为L1710 <o .漂白亚麻纤维卷< L1817 <O. longistaminata. < L1730 [2].种子萌发的后代和o .漂白亚麻纤维卷播种于土壤中,30天后移栽于武汉大学温室内。的根状茎O. longistaminata.也分块种植。在拔节-孕穗期采得5个株系3个生物重复的茎,立即用液氮保存,用于后续RNA提取。
RNA文库的构建和测序
采用TRIzol试剂按照制造商(Invitrogen, Carlsbad, CA, USA)的方案提取5个品系茎的总rna。纯度、浓度及OD值260./ od.280采用Agilent 2100 Bioanalyzer (Agilent RNA 6000 Nano Kit)检测各样品中总RNA的比例。使用Ribo-Zero™rRNA去除试剂盒从总rna中去除核糖体rna (rRNA),然后将rna随机碎片化。以片段RNA为模板,以随机的六碱基序列为引物,逆转录合成第一链cDNA,然后用dUTP替代dTTP合成第二链cDNA。然后通过cDNA末端修复、添加poly a -尾端和adapter、Uracil-N-Glycosylase酶切、多轮PCR扩增构建测序文库。对所有文库进行了质量控制和定量分析。最后,利用Illumina HiSeq 4000平台对15个文库进行测序,生成150 bp的配对端reads。每个株系都有3个生物重复进行测序。
数据过滤、序列比对及汇编
为了进一步确保rRNA未在原始reads中出现,使用短reads比对工具SOAPnuke (v1.5.2)将reads与核糖体数据库进行比对[58],并删除对齐的读取(最多允许5个不匹配)。去除带有适配器的reads、N比大于10%且质量较差的reads后,将过滤后的reads与水稻参考基因组(https://www.ncbi.nlm.nih.gov/assembly/GCF_001433935.1)使用HISAT2软件(v2.0.4) [59]并使用Stringtie重新组装(v1.0.4)[60].为了获得重新组装的转录物的位置关系,将它们与使用沟槽(v2.2.1)的工具之一与已知的MRNA和LNCRNA进行比较,而是将其与已知的MRNA和LNCRNA进行比较(V2.2.1)[61[然后使用绒毛(袖扣的一个工具,v2.2.1)合并最终转录物[61].
鉴定MRNA和LNCRNA
参考Liu等人的研究鉴定了LNCRNA。[39.].利用蛋白数据库Pfam预测组合转录本(FPKM≥0.5,Coverage > 1, Length > 200)的编码能力。http://pfam.xfam.org/)[62]和三种软件,包括编码潜能计算器(CPC, v0.9-r2, http://CPC.cbi.pku.edu.cn)[63,编码-非编码索引(CNCI,https://github.com/www-bioinfo-org/CNCI)[64]和txcdspredict(http://hgdownload.soe.ucsc.edu/admin/jksrc.zip.).四种判断方法具体阐述如下:1)如果转录本映射到Pfam数据库,则识别为mrna,否则识别为lncrna;2) CPC_threshold = 0,值大于0的转录本为mrna,否则为lncrna;3) CNCI_threshold = 0,值大于0的转录本为mrna,否则为lncrna;4) txCdsPredict_threshold = 500,值大于500的转录本为mrna,否则为lncrna。当上述四种方法中至少有三种一致时,转录本最终被鉴定为mrna或lncrna。
lncrna的差异表达分析
使用Bowtie2软件(v2.2.5)将Clean reads与参考基因组进行比对[65],然后用RSEM (v1.2.12)计算转录本的表达水平[66].RSEM软件采用的归一化方法为FPKM,公式为:FPKM =\(\ frac {10 ^ 6c} {nl / {10} ^ 3} \).In this formula, ‘C’ is the number of unique fragments for the target gene, ‘N’ is the total number of fragments which were uniquely matched the reference genome, and ‘L’ is the total number of bases in the coding region of the target gene. The calculated FPKM values, representing the gene expression levels, can be directly used to compare the gene expression differences among different samples. Correlations for three biological replications were calculated based on FPKM values using cor function in R (v3.3.0,https://www.r-project.org/).软件DEGseq [67[用于分析比较组的差异。在本研究中,表现出折叠变化(Fc)≥2的转录物(|2FC | ≥ 1) and the false discovery rate (FDR) ≤ 0.001 were regarded as significantly differentially expressed transcripts. The differentially expressed lncRNAs were screened from the differentially expressed transcripts according to the ID of lncRNAs.
lncrna靶基因鉴定及GO分析
lncrna通过两种方式调控靶基因,包括CIS.- 和反式监管。当lncRNA扮演CIS.-调控作用,lncRNA在染色体上的位置与靶基因接近,故选择lncRNA邻近的mRNA作为其靶基因;当lncRNA扮演反式-调控作用,不依赖于与靶基因的位置关系,其靶基因可以通过计算结合能来预测。具体来说,根据Liu等人的研究[39.],分析靶基因三个步骤:1)统计上分析所有识别的LNCRNA和MRNA之间的相关性(Spearman值≥0.6和Pearson值≥0.6);2)确定LNCRNA播放CIS.-当它们位于目标基因上游10 k或下游20 k内时的调控作用;3)当lncrna不在该范围内时,使用RNAplex分析lncrna与mrna的结合能。如果结合能小于- 30,则判定为具有反式监管的效果。此外,如果lncRNA与目的基因存在重叠,将其进一步划分为10个亚类(如lnc-overlap-mRNA和lnc-anti -overlap-mRNA),这将有助于加深我们对lncRNA的理解CIS.- lncRNA的规管详情[68,69].此外,所有比较组中发现的差异lncrna (de - lncrna)的潜在靶基因均通过WEGO网站(http://wego.genomics.org.cn),以水稻基因组为背景。
预测lncRNA作为miRNA前体
为了预测可能是microRNAs (miRNAs)前体的lncRNAs,使用BLAST工具将所有lncRNAs与miRbase (http://www.mirbase.org.)[70].当MiRNA前体序列的LNCRNA序列的覆盖率超过90%时,LNCRNA被认为是miRNA前体,并且miRNA的前体序列来自先前的数据[2].lncRNA和miRNA前体的二级结构由RNAfold web服务器绘制(http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi).通过Cytoscape软件显示lncRNA、miRNA和mRNA的交互网络关系(v3.7.1,http://www.cytoscape.org).
QRT-PCR验证数据
为了验证本研究中的LNCRNA测序数据的准确性,从所有表达的LNCRNA随机选择9LNCRNA和它们的预测潜在靶基因并通过定量实时PCR(QRT-PCR)验证。使用Trizol试剂(Invitrogen)提取来自五条线的茎的总RNA,并用随机引物逆转录。使用Primer 5.0软件设计LNCRNA / mRNA特异性引物(http://www.premierbiosoft.com/index.html.).使用ABI步骤一加上实时PCR系统(Applied Biosystems,USA)中的SYBR @ QPCR混合物(Toyobo)进行PCR扩增。如前所述用三种生物学重复和三种技术重复处理QRT-PCR反应[29.].此外,OsActin1[71]作为内控基因,对每个lncRNA/mRNA阈值循环反应进行归一化。
数据和材料的可用性
在当前研究期间分析的数据集可在NCBI序列读取存档(SRA)存储库中(https://trace.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=announcement), 15次运行的登录号为SRR9822767-SRR9822781。水稻参考基因组的网络连接为https://www.ncbi.nlm.nih.gov/assembly/GCF_001433935.1.蛋白质数据库pfam的网页是http://pfam.xfam.org/.miRbase的网络链接为http://www.mirbase.org..本研究中产生或分析的所有其他数据均包含在本文和附加文件中。
缩写
- 阿坝:
-
脱盐酸
- BILS:
-
回交基因渗入行
- CNCI:
-
Coding-Non-Coding指数
- 中国共产党:
-
编码潜在的计算器
- CTR1:
-
组成型三重响应1
- DE-lncRNAs:
-
差异表达lncRNAs
- DEP1:
-
密集和直立胰腺1
- 分区:
-
聚腺苷酸化
- 古人说:
-
表达水平的主导地位
- FC:
-
折叠变化
- 罗斯福:
-
错误发现率
- 方法:
-
开花基因座C.
- 遗传算法:
-
吉布林素
- 柯:
-
ent-Kaurene氧化酶
- lncrana:
-
长非编码RNA
- mirnas:
-
microRNA.
- OsTPP1:
-
Trehalose-6-phosphate磷酸酶
- 光敏色素的:
-
凤凰
- 存在:
-
定量实时PCR
- rrnas:
-
核糖体rna
- OsSPL3:
-
SQUAMOSA PROMOTER-BINDING PROTEIN-LIKE3
- SRA:
-
顺序读取存档
- SS-RNASEQ:
-
股线特异性RNA测序。
参考
- 1。
葛松,桑涛,吕博良,洪大勇。水稻基因组系统发育研究——以异源四倍体物种起源为重点。美国国立科学学院1999;96:14400-5。
- 2。
曹安,金娟,李树华,王军。水稻回交后代mRNA和miRNA表达谱的综合分析2F12.),不同株高。《公共科学图书馆•综合》。2017;12:e0184106。
- 3。
马旭,傅莹,赵旭,姜玲,朱鹏,顾鹏,徐伟,苏智,孙超,谭磊oryza nivara利用全基因组重测序鉴定产量相关qtl。Sci众议员2016;6:27425。
- 4.
李X,guo w,王b,李x,陈h,魏l,王y,wu j,长h.杂交和杂氮染色体甲基化变异的不稳定性萝卜L和芸苔属植物alboglabra贝利。BMC Plant Biol. 2010;10:207。
- 5.
陈泽,胡福,徐P,李杰,邓X,周j,李F,陈S,陶氏菌杂交无菌和植物高度探索野生稻的三种植物高度,选用longistaminata.Sci繁殖。2009;59:441-5。
- 6.
Yugander A, Sundaram RM, Singh K, Ladhalakshmi D, Subba Rao LV, Madhav MS, Badri J, Prasad MS, Laha GS。新的抗白叶枯病基因的加入Xa38转化为优良水稻品种桑巴·马赫苏里的遗传背景。《公共科学图书馆•综合》。2018;13:e0198260。
- 7.
Saikumar S, Gouda PK, Saiharini A, Varma CMK, Vineesha O, Padmavathi G, Shenoy VV。研究了水稻在旱作生殖胁迫下增产的主要QTLo .漂白亚麻纤维卷/o . glaberrima渐进线。现场作物RES。2014; 163:119-31。
- 8。
Moumeni A,Satoh K,VenupraSad R,Serraj R,Kumar A,Kikuchi S.近同位素稻米叶片的转录分析,对着卵巢缺陷的生殖阶段对比耐旱性耐旱性。BMC基因组学。2015; 16:1110。
- 9。
Brog YM, Osorio S, Yichie Y, Alseekh S, Bensal E, Kochevenko A, Zamir D, Fernie AR. A茄属植物neorickii迟滞的人口为广泛的特征提供了强大的补充Solanum Pennellii.人口。植物j . 2019; 97:391 - 403。
- 10。
歌曲WY,Wang GL,Chen Ll,Kim HS,Pi Ly,Holsten T,Gardner J,Wang B,Zhai Wx,Zhu LH,Fauquet C,Ronald P.稻瘟病抗性基因编码的受体激酶样蛋白,Xa21.科学。1995年; 270:1804-6。
- 11.
胡凤英,陶大勇,Sacks E,傅BY,徐鹏,李俊,杨勇,McNally K, Khush GS, Paterson AH,李志康。水稻和高粱多年生性的趋同进化。美国国家科学研究院2003;100:4050-4。
- 12.
徐鹏,董磊,周军,李军,张勇,胡芳,刘松,王强,邓伟,邓欣。一株抗稻瘟病新基因的鉴定与定位PI.57(t)在选用longistaminata.Euphytica。2015; 205:95-102。
- 13。
Ulitsky I, Bartel DP。LincRNAs:基因组学、进化和机制。细胞。2013;154:26-46。
- 14。
Chekanova JA。长期非编码RNA及其在植物中的功能。CurrOp植物BIOL。2015; 27:207-16。
- 15.
秦涛,赵海涛,崔鹏,熊丽丽。一种核定位的长链非编码RNA增强对干旱和盐胁迫的耐受性。植物杂志。2017;175:1321-36。
- 16.
Shumayla SS, Taneja M, Tyagi S, Singh K, Upadhyay SK.高通量RNA-seq数据调查揭示了lncrna在面包小麦发育和应激反应中的潜在作用。植物学报2017;8:1019。
- 17.
王志,刘勇,李丽,李东,张强,郭勇,王胜,钟超,黄辉假单胞菌含油PV.actinidiae- 培养的猕猴桃植物揭示了长期非编码RNA和编码基因之间的物质特异性相互作用。SCI REP。2017; 7:4910。
- 18.
关键词:长链非编码rna;摩尔细胞。2011;43:904-14。
- 19。
迪C,袁j,吴y,李杰,林h,胡l,张t,qi y,gerstein mb,guo y,lu zj。应力响应LNCRNA的表征拟南芥综合表达、表观遗传和结构特征。植物j . 2014; 80:848 - 61。
- 20.
吴海军,王志明,王明,王新军。广泛分布的长非编码rna作为植物microrna的内源性靶模拟物。植物杂志。2013;161:1875 - 84。
- 21.
关键词:玉米,lincrna, miRNA靶标,诱骗基因,功能分析BMC基因组学。2015;16。
- 22。
长链非编码rna:植物发育反应的微调。J Biosci。2019;44。
- 23。
Campalans A,Kondorosi A,Crespi M. Enod40,一种含有短的读取型框架的mRNA,诱导核RNA结合蛋白的细胞质定位Medicago truncatula.植物细胞。2004;16:1047-59。
- 24。
关键词:表观基因组长链非编码RNA, RNA模板,分子支架表观遗传学。2011;6:539-43。
- 25。
关键词:冷诱导沉默,长反义转录本,冷诱导沉默拟南芥Polycomb目标。自然。2009; 462:799-U122。
- 26。
春化介导的非编码RNA的表观遗传沉默。科学。2011;331:76-9。
- 27.
弗朗科·扎罗拉·杰姆,瓦莉A,托斯德士米,马特索斯I,Puga Mi,Rubio-Somoza I,Leyva A,Weigel D,Garcia Ja。PAZ-ARES J;目标模仿提供了一种用于调节MicroRNA活性的新机制。NAT Genet。2007; 39:1033-7。
- 28.
李爱华,刘丹,吴建军,赵旭,郝敏,严军,姜兴,张磊,吴杰,尹磊,张锐,吴磊,郑勇,毛磊。六倍体小麦异源多倍体杂种优势的动态同源调控。植物细胞。2014;26:1878 - 900。
- 29.
沈艳艳,赵强,邹娟,王伟林,高艳,孟JL,王jb。合成小rna的特性和表达模式芸苔属植物六倍体。植物学报。2014;85:287-99。
- 30.
Vrbsky J, Akimcheva S, Watson JM, Turner TL, Daxinger L, Vyskot B, Aufsatz W, Riha K. sirna介导的甲基化拟南芥端粒。Plos Genet。2010; 6:E1000986。
- 31.
邵友,周赫兹,吴越,张h,林俊,江xy,他qj,zhu js,李y,yu h,毛cz。OSSPL3,SBP域蛋白质调节水稻的皇冠根系发育。植物细胞。2019; 31:1257-75。
- 32.
引用本文:吕志峰,余辉,熊广生,王健,焦永强,刘广发,景玉红,孟XB,胡晓明,钱强,付晓东,王艳红,李建勇。水稻转录激活因子IDEAL PLANT ARCHITECTURE1的全基因组结合分析揭示了调控水稻植株结构的复杂网络。植物细胞。2013;25:3743-59。
- 33.
Iwamoto M,Kiyota S,Hanada A,Yamaguchi S,Takano M.植物植物对水稻水稻伸长率控制的多贡献。植物理性。2011; 157:1187-95。
- 34。
刘MJ, Szadkowski E, Wendel JF异源多倍体棉花同源基因表达偏倚和表达水平优势。遗传。2013;110:171 - 80。
- 35。
张永超,廖建勇,李志勇,余勇,张建平,李庆峰,曲丽华,舒文伟,陈玉强。通过全基因组筛选和功能分析,鉴定出大量参与水稻有性生殖的长链非编码rna。基因组医学杂志。2014;15:512。
- 36。
丁俊,卢强,欧阳云,毛浩,张鹏,姚军,徐超,李昕,肖军,张强。一种长链非编码RNA对杂交水稻光周期敏感雄性不育的调控。美国国家科学学院学报2012;109:2654-9。
- 37。
宋建华,曹建生,王刚。BcMF11,一种新的非编码RNA基因来自芸苔CameStris.,是花粉发育和雄性不育所必需的。植物细胞代表2013;32:21-30。
- 38。
Komiya R, Ohyanagi H, Niihama M, Watanabe T, Nakano M, Kurata N, nomura K. Rice germline specific argonaute MEL1 protein binding to phasiRNAs generated from more 700 lincRNAs。植物j . 2014; 78:385 - 97。
- 39.
关键词:水稻,胚珠发育,配子体败育,lncrna,功能分析BMC基因组学。2019;20:90。
- 40.
关键词:小麦,白粉病,热胁迫,长链非蛋白编码rna,微阵列分析,序列分析BMC Plant Biol. 2011;11:61。
- 41.
jain p,sharma v,dubey h,singh pk,kapoor r,kumari m,singh j,pawar dv,bisht d,solanke au,mondal tk,sharma tr。鉴定耐水稻毒药稻水稻线长的非编码RNAMaganaporthe oryzae..生物信息。2017; 13:249-55。
- 42.
江B,娄Q,吴Z,张W,王D,Mbira kg,Weng Y,Chen J. Retrotransposon-和微卫星序列相关的新合成的同种异体四倍体早期几代内序列相关的基因组变化Cucumis×休养温陈和科克布莱德。植物学报。2011;77:225-33。
- 43.
卢X,陈X,穆米,王j,王x,王d,yin z,风扇w,王s,guo l,ye w。长度非划分RNA的基因组分析及其对棉花干旱胁迫的反应(陆地棉l .)。Plos一个。2016; 11:e0156723。
- 44.
袁j,李杰,杨y,棕褐色,朱y,胡l,qi y,lu zj。长非编码RNA多腺苷酸化的应力响应调节栽培稻.植物j . 2018; 93:814-27。
- 45.
Itoh H, Tatsumi T, Sakamoto T, Otomo K, Toyomasu T, Kitano H, Ashikari M, Ichihara S, Matsuoka M.水稻半矮化基因,坦加博祖(D35),编码Gibberellin生物合成酶,ent-kaurene氧化酶。acta botanica sinica(云南植物学报);2004;54:533-47。
- 46。
在水稻中,缺乏DELLA结构域的GRAS蛋白的过度表达改变了赤霉素的反应。植物j . 2005; 44:669 - 79。
- 47。
佐佐木A, Itoh H, goomi K, Ueguchi-Tanaka M, Ishiyama K, Kobayashi M, Jeong DH, An G, Kitano H, Ashikari M, Matsuoka M. F-box突变体中磷酸化抑制因子的积累。科学。2003;299:1896-8。
- 48。
王Q,张W,Yin Z,Wen CK。大米本构三 - 反应2参与乙烯 - 受体信号传导和水稻生长和发育的各个方面的调节。J Exp Bot。2013; 64:4863-75。
- 49。
Li G, Liang W, Zhang X, Ren H, Hu J, Bennett MJ, Zhang D. Rice actin binding protein RMD是生长素-肌动蛋白调控环路中调控细胞生长的关键环节。中国科学技术大学学报(自然科学版)2014;111:10377-82。
- 50.
徐晓伟,周晓华,王瑞荣,彭伟林,安勇,陈丽丽。利用竞争性内源RNA网络分析缺磷水稻基因间长链非编码RNA的功能。Sci众议员2016;6:20715。
- 51.
葛丽芬,超达元,史敏,朱明中,高建平,林海霞。海藻糖-6-磷酸磷酸酶基因的过表达OsTPP1增强水稻的抗逆性,并激活抗逆性基因。足底。2008;228:191 - 201。
- 52.
Manosalva PM,Bruce M,Leach Je。水稻14-3-3蛋白(GF14E)对细胞死亡和抗病性产生负面影响。工厂J1111; 68:777-87。
- 53.
Liu Q, Yang J, Zhang S, Zhao J, Feng A, Yang T, Wang X, Mao X, Dong J, Zhu X, Leung H, Leach JE, Liu B. OsGF14e对水稻穗部稻瘟病抗性的正向调控。生物化学学报。2016;471:247-52。
- 54.
关键词:水稻,WRKY45-2, WRKY13, WRKY42,转录调控植物杂志。2015;167:1087 - 99。
- 55.
Li D, Yang C, Li D, Mao D, Liu X, Ji C, Li X, Zhao X, Cheng Z, Chen C, Zhu L. micrororna319过表达对水稻叶片形态发生和抗寒性的影响(英文)栽培稻l .)。植物学报。2013;36(4):497 - 497。
- 56.
陈志强,陈志强,陈志强,等偶然发生水稻家族基因。基因组学杂志。2008;279:279-89。
- 57.
Takano M,Inagaki N,Xie X,Kiyota S,Baba-Kasai A,Tanabata T,Shinomura T.PhytoChromes是植物瘢痕瘤是在水稻中感知红色/远红光的唯一光感受器。Proc Natl Acad Sci U S A. 2009; 106:14705-10。
- 58。
李荣强,李玉玲,王杰。SOAP:短寡核苷酸比对程序。生物信息学。2008;24:713-4。
- 59。
HISAT:低内存要求的快速拼接对准器。Nat方法。2015;12:357-U121。
- 60。
Pertea M, Pertea GM, Antonescu CM, Chang T-C, Mendell JT, Salzberg SL. StringTie能够从RNA-seq读取改进的转录组重建。生物科技Nat》。2015;33:290-5。
- 61。
Trapnell C,Williams Ba,Pertea G,Mortazavi A,Kwan G,Van Baren MJ,Salzberg SL,Wold BJ,RNA-SEQ的成绩单组件和定量揭示了细胞分化期间未经发布的转录物和同种型切换。NAT BIOTECHNOL。2010; 28:511-U174。
- 62。
Finn RD, Coggill P, Eberhardt RY, Eddy SR, Mistry J, Mitchell AL, Potter SC, Punta M, Qureshi M, Sangrador-Vegas A, Salazar GA, Tate J, Bateman A. Pfam蛋白家族数据库:朝着更可持续的未来发展。核酸Res. 2016;44: D279-85。
- 63.
孔玲,张勇,叶志强,刘新强,赵树清,魏玲,高刚。CPC:利用序列特征和支持向量机评价转录本的蛋白编码潜力。核酸学报2007;35:W345-9。
- 64.
孙丽,罗华,卜东,赵刚,于凯,张超,刘颖,陈瑞,赵颖。利用序列固有成分对蛋白质编码和长链非编码转录本进行分类。核酸学报2013;41。
- 65.
Langmead B, Salzberg SL.快速间隙读对齐与领结2。Nat方法。2012;9:357-U354。
- 66.
Li B,杜威CN。RSEM:具有或不具有参考基因组的RNA-SEQ数据的准确转录物定量。BMC生物信息学。2011; 12:323。
- 67.
王磊,冯志强,王昕,张晓霞。基于RNA-seq数据的差异表达基因鉴定。生物信息学。2010;26:136-8。
- 68.
孙涛。长链非编码rna在脊椎动物中枢神经系统发育和功能中的调控机制。神经科学。2013;235:200-14。
- 69.
Kornienko AE,Guenzl PM,Barlow DP,Pauler FM。长期非编码RNA转录作用的基因调节。BMC BIOL。2013; 11:59。
- 70.
miRBase:从microRNA序列到功能。核酸Res. 2019;47: D155-62。
- 71.
高桥平,田志刚,田志刚,等。水稻胚珠发育的激光显微解剖转录组分析。acta botanica yunnanica(云南植物研究). 2013;54:750-65。
致谢
不适用。
资金
国家重点基础研究发展计划(no . 2013CB126900)。资助机构在实验设计、数据收集、分析和解释以及手稿准备中没有发挥作用。
作者信息
从属关系
贡献
JW和AC构思和设计了这项研究。ML和AC进行了大多数生物信息学分析并分析了大多数数据。ZL进行了一些生物信息学分析。RW执行了QRT-PCR实验。SL创建了BC2F12.线条并提供了实验材料,提出了对数据分析的一些关键建议并修改了稿件。AC负责种植材料。ML为来自所有作者的贡献编写了手稿。JW修改了稿件。所有作者均阅读并批准最终手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
补充信息
附加文件1:图S1。
基因表达数据三种生物学重复的相关系数
附加文件2:图S2。
与亲本相比,三个子代的lncrna表达差异。FC为de - lncrna的折叠变化。红色表示上调的de - lncrna数量,蓝色表示下调的de - lncrna数量
其他文件3:图S3。
LNCRNA潜在靶靶MRNA与LNCRNA潜在靶靶MRNA的致富集分析,L1710一个),L1817(B)及L1730 (C).红色标记‘*’表示氧化石墨烯显著富集,其中P-值小于0.05
附加文件4:表S1。
十五个图书馆测序综述
附加文件5:表S2。
组装16,038个新型转录物的统计分析
附加文件6:表S3。
所有样品中所有LNCRNA和MRNA的标准化FPKM值
附加文件7:表S4。
lncRNA-mRNA对的调控关系类型
附加文件8:表S5。
靶向mirna和lncrna / mrna之间的关系
附加文件9:表S6。
3个子代中亲代表达水平显性的lncrna及其靶mrna
附加文件10:表S7。
引物用于实时荧光定量PCR
权利和权限
开放访问本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信贷额度中另有说明。
关于这篇文章
引用这篇文章
李,M.,Cao,A.,Wang,R.等等。水稻回交渐渗系(BC)中lncrna的全基因组鉴定与整合分析2F12.).BMC植物杂志20.300(2020)。https://doi.org/10.1186/s12870-020-02508-Y.
收到了:
接受:
发表:
关键词
- 长非编码RNA
- 回交基因渗入行
- 多种多样
- 转录组测序
- 大米