- 研究文章gydF4y2Ba
- 开放访问gydF4y2Ba
- 发表:gydF4y2Ba
中长链非编码rna的发现、鉴定和功能表征gydF4y2Ba落花生hypogaeagydF4y2BalgydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba20.gydF4y2Ba, 文章编号:gydF4y2Ba308.gydF4y2Ba(gydF4y2Ba2020.gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
长度的非致rnas(lncrna),其通常长度为200nt,涉及许多生物过程。栽培花生中LNCRNA的研究(gydF4y2Ba落花生hypogaeagydF4y2Bal)很大程度上仍然未知。gydF4y2Ba
结果gydF4y2Ba
对花生的基因组扫描(gydF4y2Ba落花生hypogaeagydF4y2BaL.)转录组鉴定了1442个LNCRNA,其通过分布在每种染色体上的基因座编码。长期性非数性RNA占这些LNCRNA的85.58%。另外,189克尔中的根部,叶或种子差异丰富。通常,LNCRNA显示出较低的表达水平,细胞特异性表达,比mRNA少拼接。可替代地拼接约44.17%的外显子/内部结构的LNCRNA;该速率略低于mRNA的剪接率。开始网站事件的转录是花生LNCRNA中最高频率(28.05%)的替代剪接(AS)事件,而内含子保留事件的发生率(30.19%)是MRNA中最高的。随着LNCRNA的目标基因谱改变,增加了LNCRNA的多样性和灵活性,这对于执行其功能可能是重要的。另外,大量的花生作为从蛋白质编码基因产生的同种型的花生似乎是非沉积的,因为它们被截短了转录物;这种同种型可以合法地被视为一类LNCRNA。 The predicted target genes of the lncRNAs were involved in a wide range of biological processes. Furthermore, expression pattern of several selected lncRNAs and their target genes were examined under salt stress, results showed that all of them could respond to salt stress in different manners.
结论gydF4y2Ba
本研究提供了候选lncrna的资源和组织间的表达模式,这些lncrna是否具有功能性将在我们后续的实验中进一步研究。gydF4y2Ba
背景gydF4y2Ba
分子生物学的中心信条,提出了从DNA到RNA再到蛋白质的信息流动[gydF4y2Ba1gydF4y2Ba)的理论不再适用于不断增加的非编码蛋白质的rna。这些非编码rna (ncRNAs)根据其位置、长度和生物学功能以不同的方式分类[gydF4y2Ba2gydF4y2Ba].广泛地说,NCRNA分为两类:保留和监管[gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba].后者可以分为短NCRNA(<200nt),包括短干扰RNA(20-31NT),小NCRNA和长的非编码RNA(> 200nt,lncrnas)[gydF4y2Ba4gydF4y2Ba].短干扰RNA包括小干扰RNA,MicroRNAS(MiRNA)和PiWi相互作用的RNA;这些序列在发现它们的不同真核序列中不同[gydF4y2Ba5gydF4y2Ba].基于其基因组来源和/或其相对于其相邻的蛋白质编码转录物的基于基因组来分类为反义LNCRNA,长期性NCRNA(LincrNA)和内肠菌(Incrnas)分类为反义LNCRNA,和inscronaCrNA(Incrnas)[gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba].gydF4y2Ba
毫无疑问,基因组学和生物信息学的进步,特别是下一代测序的广泛应用,促进了已经确定参与一系列监管角色而不是简单地代表转录噪声的NCRNA的识别和注释[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba].尽管lncrna可能是这些ncrna中研究最少的,但越来越多的证据表明,在真菌、植物和动物中,它们在转录、转录后和表观遗传水平上发挥其功能[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10.gydF4y2Ba,gydF4y2Ba11.gydF4y2Ba,gydF4y2Ba12.gydF4y2Ba,gydF4y2Ba13.gydF4y2Ba,gydF4y2Ba14.gydF4y2Ba,gydF4y2Ba15.gydF4y2Ba].特别是,动物lncRNAs与衰老、造血、pri-miRNA加工、肌肉分化、神经发育和免疫反应相关[gydF4y2Ba16.gydF4y2Ba,gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba18.gydF4y2Ba,gydF4y2Ba19.gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21.gydF4y2Ba].Xist是研究得最好的lncrna之一,在雌性哺乳动物发育过程中,通过直接与SHARP相互作用、招募SMRT、激活HDAC3和去乙酰化组蛋白来排除X染色体上的Pol II来沉默转录[gydF4y2Ba12.gydF4y2Ba].尽管与人类和动物的研究相比,植物上的研究有限,但通过RNA-seq和生物信息学分析,已经在一些植物中识别出了数以万计的lncrna,如拟南芥[gydF4y2Ba22.gydF4y2Ba], Medicago [gydF4y2Ba23.gydF4y2Ba)大豆(gydF4y2Ba9gydF4y2Ba)、大米(gydF4y2Ba24.gydF4y2Ba], 小麦 [gydF4y2Ba25.gydF4y2Ba],玉米[gydF4y2Ba26.gydF4y2Ba,番茄gydF4y2Ba27.gydF4y2Ba],桑树[gydF4y2Ba28.gydF4y2Ba],poplar [gydF4y2Ba29.gydF4y2Ba]和海鼠[gydF4y2Ba11.gydF4y2Ba].此外,已有研究表明,有少量的lncrna在植物中发挥着与动物相似的调节功能[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba].例如,lncrnagydF4y2BaCOOLAIRgydF4y2Ba这是一种长链内含子非编码RNA,通过与PRC2的相互作用在FLC上建立稳定的抑制性染色质是必需的[gydF4y2Ba31gydF4y2Ba].在水稻中,Ding等人发现长日照特定的雄性育性相关的lncRNA RNA对于长日照条件下植物的正常花粉发育是必不可少的[gydF4y2Ba30.gydF4y2Ba].而且,越来越多的研究表明lncrna在各种应激反应的基因表达调控中发挥着重要的作用[gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba].拟南芥LncrNa,干旱诱导的LNCRNA(DRIR)是植物对干旱和盐胁迫的新阳性调节剂[gydF4y2Ba37gydF4y2Ba].在对照条件下,DRIR表达量较低,但在干旱、盐胁迫和ABA处理下显著增加。而且,gydF4y2Ba酒后德gydF4y2BaDgydF4y2BaRNA-seq结果表明,在拟南芥中突变或过表达drr可以提高转基因植物对干旱和盐胁迫的耐受性gydF4y2Ba酒后德gydF4y2Ba可以调节一系列参与ABA信号转导、水转运和其他胁迫缓解过程的基因的表达。尽管越来越多的证据支持lncrna在植物中的多种潜在作用,但lncrna在栽培花生(gydF4y2Ba落花生hypogaeagydF4y2Bal)很大程度上仍然未知。最近,Zhao等人从公开发表的大规模RNA测序数据中,鉴定出花生50873个lncrna,属于124个样本,涉及15个不同组织,并预测了目标基因与386个hub lncrna的共表达[gydF4y2Ba39gydF4y2Ba].gydF4y2Ba
花生是源于野生二倍体之间的自然杂交的同种异体四倍体物种gydF4y2BaA. Duranensis.gydF4y2Ba和gydF4y2BaA. iPaensis.gydF4y2Ba[gydF4y2Ba40gydF4y2Ba].花生在种子生产方面排名第六;约有三分之二的收获花生种子用于石油生产,残留的种子饼用作富蛋白膳食的牲畜[gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba].现在已经获得了花生祖种类的基因组序列[gydF4y2Ba43gydF4y2Ba],从而为分析lncrna对植物转录和最终表型的贡献提供了机会。本研究通过扫描花生的链特异性序列数据来检测lncRNA含量,并结合生物信息学分析和实验来说明可能涉及lncRNA活性的生物过程的范围。提出了一些新的观点。gydF4y2Ba
结果gydF4y2Ba
花生转录组分析gydF4y2Ba
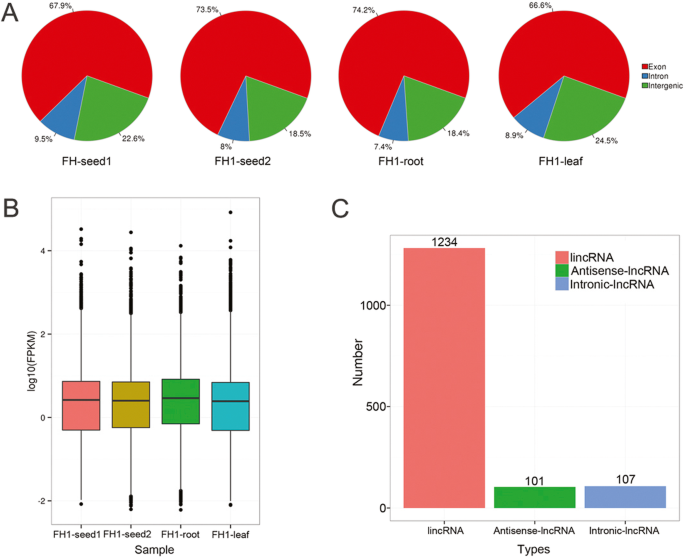
在对适配器序列和低质量reads进行裁剪后,从FH1-seed1、FH1-seed2、FH1-root和FH1-leaf四个库中各获得超过11.14 Gb的干净数据,用于进一步分析。拆分为2038亿条长度为125 bp的末端对。各个图书馆的QgydF4y2Ba30.gydF4y2Ba价值范围从88.90到89.79%,他们的GC含量从44.48到50.48%(附加档案gydF4y2Ba1gydF4y2Ba:表S1)。79.91和84.16%的读数与花生参考基因组序列成功对齐(附加文件gydF4y2Ba2gydF4y2Ba:表S2)。另外,关于在注释的蛋白质编码基因(外源和内肠道)和非基因区域中,关于RNA-SEQ读数的分布的整体映射结果应该在图2中呈现。gydF4y2Ba1gydF4y2Ba一种。大多数读取(66.60-74.25%)被映射到外源序列,映射到内肾序列<10%映射(图。gydF4y2Ba1gydF4y2Ba一个)。gydF4y2Ba
识别LncRNAsgydF4y2Ba
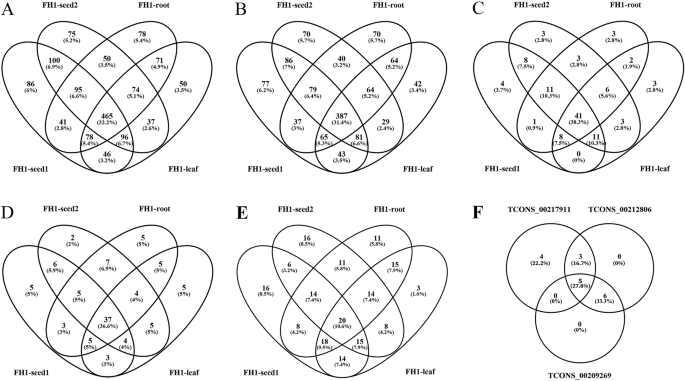
在施加旨在识别推定的LNCRNA的各种标准之后,总共剩下1442个序列(附加文件gydF4y2Ba3.gydF4y2Ba:表S3);每个库的FPKM值表明4个库的lncRNA丰度分布大致相似,但也有证据表明fh1根库的序列分布和丰度略有不同(略高)(图1)。gydF4y2Ba1gydF4y2Bab).鉴定出三种类型的lncRNA序列:lincRNAs、incRNAs和反义lncrnas,其中lincRNAs占85.60%(图1)。gydF4y2Ba1gydF4y2BaC);在这些序列中,1007在FH1-SEED2,952中的FH1-SEED1,992中以FH1-根部的FH1-SEED2,952和FH1-叶中的917表示(图。gydF4y2Ba2gydF4y2Ba一个)。在所有四个文库中表示的序列数为465,而文库特异性序列的数量为86,75,78和50;LNCRNA的261是特异于显影种子(特异于FH1-Seef1或FH1-Seef2,或者在两个文库中存在,但不存在于FH1根和FH1-叶中)。在所有四个库中表示的LincrNA数量为387,而图书馆特定的Lincrnas的数量为77,70,70和42(图。gydF4y2Ba2gydF4y2Bab)。还识别了大量的器官特异性内肠RNA和反义-LNCRNA(图。gydF4y2Ba2gydF4y2Bac, d)。共有189个lncrna被归类为差异丰富的lncrna (DALs;无花果。gydF4y2Ba3.gydF4y2Ba;额外的文件gydF4y2Ba4gydF4y2Ba:表S4)。4个文库中DALs的数量均为20个,而库特异性序列的数量分别为16、16、11和3个(图1)。gydF4y2Ba2gydF4y2Bae)。gydF4y2Ba
lncrna的基因组分布gydF4y2Ba
这组已鉴定的lncrna是由分布在花生基因组所有20条染色体上的序列转录而来的,尽管与B亚基因组相关的数量高于与a亚基因组相关的数量(863 vs 579,附加文件gydF4y2Ba5gydF4y2Ba:表S5)。染色体Araip。与Aradu.A07染色体转录的31个lncrna相比,B02转录了112个。A亚基因组中每100个基因的平均lncrna数量为2.57,而B亚基因组中相应的统计量为3.37。各染色体上转录的lncrna数量与转录的基因数量(FPKM阈值为0.1)之间的相关性为+ 0.61(显著于gydF4y2BaPgydF4y2Ba< 0.01)。gydF4y2Ba
mrna和lncrna的比较gydF4y2Ba
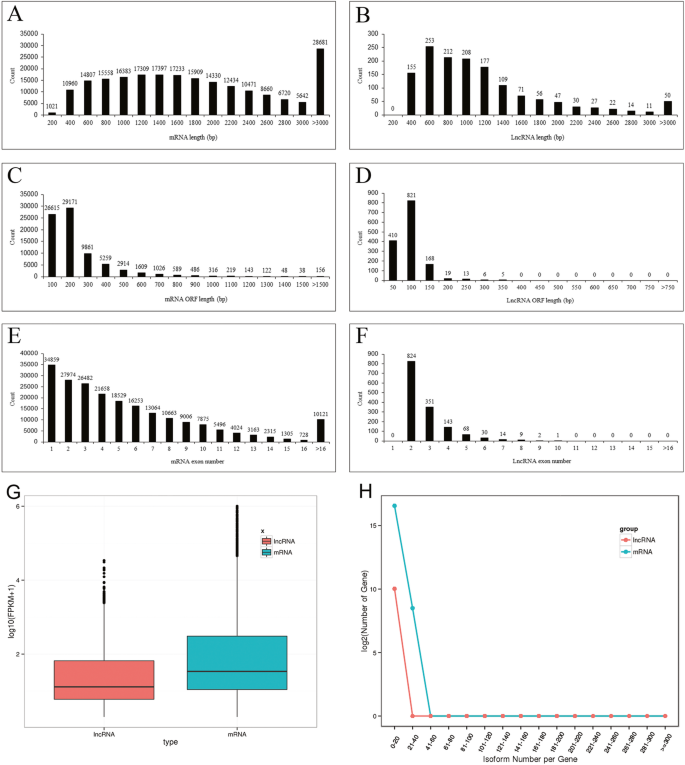
获得从55,621个基因产生的213,515转录物的序列:这组序列包括作为同种型,在此统称到“MRNA”。MRNA的平均长度为2017年,其大部分长度落在400-2600nt的范围内(图。gydF4y2Ba4gydF4y2Baa),而lncrna的等效长度分别为1074 nt和400-1400 nt。(无花果。gydF4y2Ba4gydF4y2Bab)。MRNA的开放阅读框架(ORF)的平均长度为240nt,大多数落在100-300nt的范围内,而LNCRNA的范围为96nt,长度为50nt至150 nt(无花果。gydF4y2Ba4gydF4y2BaC,D)。包括至少一个内含子的所有LNCRNA具有比mRNA的平均外显子数为2.77的外显子,其平均外显子数为5.48(图。gydF4y2Ba4gydF4y2Bae, f)。mrna的丰度普遍高于lncrna(图5)。gydF4y2Ba4gydF4y2BaG)。mRNAS产生比LNCRNA多数量的同种型(图。gydF4y2Ba4gydF4y2Bah)。gydF4y2Ba
预测lncrna的靶基因gydF4y2Ba
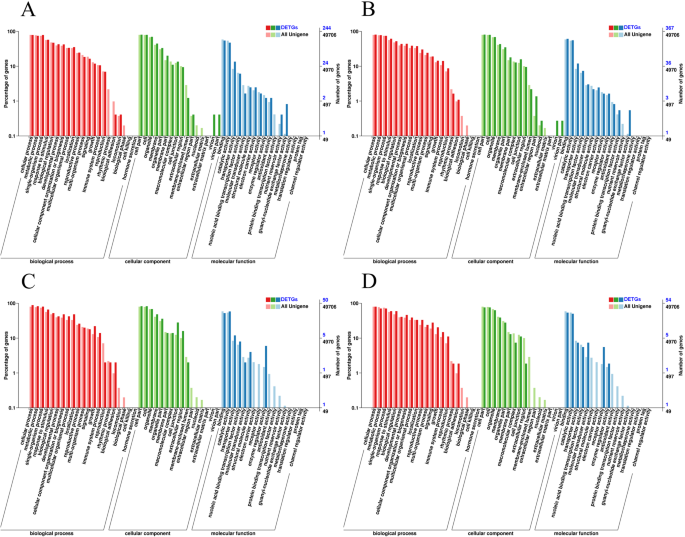
为了研究LNCRNA的潜在功能,检查其靶基因gydF4y2Ba独联体gydF4y2Ba(附加文件gydF4y2Ba6gydF4y2Ba:表S6)和在gydF4y2Ba反式gydF4y2Ba(附加文件gydF4y2Ba7gydF4y2Ba:表S7)。基于5个蛋白数据库对所有目的基因进行了整合功能注释。共获得11765个目的基因gydF4y2Ba8gydF4y2Ba:表S8),在FH1-SEED1,9415的FH1-SEED2,8742中的9326,FH1根,8497中的FH1-叶。在这些注释的靶基因中,6550名在COG数据库中注释,在GO Database的4209中,1922年在Kegg数据库中的4422中,NR中的10,019。分析了这四种文库之间的差异表达的靶基因(DETGS),并使用GO术语作为示例以分析DETGS的功能分类。总共分配了49,706个unigenes术语,富集的术语在不同的组织中不同(图。gydF4y2Ba5gydF4y2Ba).在FH1-seed1和FH1-seed2之间富集的DETGs数量为244个。gydF4y2Ba5gydF4y2Baa),367之间的FH1根和FH1-叶(图。gydF4y2Ba5gydF4y2Ba(b),而fh1根与fh1 -籽1& fh1 -籽2、fh1 -叶与fh1 -籽1& fh1 -籽2之间富集的DETGs分别为50和54(图5)。gydF4y2Ba5gydF4y2Bac, d).所有的GO术语都被分为三大类(生物过程、细胞成分和分子功能),这意味着lncrna可能在许多生物过程中发挥重要的调控作用。gydF4y2Ba
lncrna的实验验证gydF4y2Ba
为了验证预测的lncrna的真实性,我们对其中7个lncrna (lncRNA1-7)进行了PCR验证(Additional file)gydF4y2Ba9gydF4y2Ba:表S9)。Sanger测序的结果表明,除了两种(LNCRNA1和LNCRNA2,附加文件外,5个序列同一性非常高(99.33-99.86%)gydF4y2Ba11.gydF4y2Ba:图。S1)。当PCR测序的LNCRNA1-7与A和B子基因组的相应基因组序列对齐时,其中六个(LNCRNA1除外)良好匹配相应染色体上的预测位置(附加文件gydF4y2Ba11.gydF4y2Ba:图S1)LNCRNA1仅与TCONS_00179561共享85.68%的身份;在段130,713,152-130,713,435中的Aradu.a03上定位的基因组序列的基因组序列进行基因组序列,而Tcons_00179561位于araip.b03 131,673,533-131,674,406中;因此,LNCRNA1不能是Tcons_00179561的产物,也许PCR扩增其同源基因。LNCRNA2与TCons_00109592共享96.84%的身份。Blastn搜索位于预期染色体位置(Tcons_00109592)中的该序列,但在基因组序列和LNCrNA之间突出了9 nt indel和几种核苷酸多态性。我们推测产品可能是LNCRNA2的同种型。LNCRNA3-7和相应的基因组序列之间的差异分别为1,1,2,1,6个核苷酸。对于LNCRNA4,核苷酸在克隆序列的609 nT中以及A01亚基组和RNA-SEQ中的核苷酸是C.因此,差异可能是由于RNA-SEQ中的测序误差。对于其他方式,差异应该是测序误差PCR扩增的结果,因为RNA-SEQ中的序列与其子组织相同(图。gydF4y2BaS1gydF4y2Ba).简单地说,这些差异可能来自测序错误,或PCR扩增。7个lncrna中有6个的成功验证表明,大多数lncrna的预测是可信的。gydF4y2Ba
lncrna的AS事件gydF4y2Ba
lncRNA和基因组DNA序列的比较表明AS可能参与了它们的转录。根据RNA-seq数据,5个AS事件(转录起始位点,TSS;转录末端位点,TTS;外显子跳跃,ES;基因内区保留,红外;选择性外显子,AE)的研究(表gydF4y2Ba1gydF4y2Ba).总的来说,637个lncrna涉及1811个AS事件,约占1442个lncrna的44.17%。同时,约47.82%的mrna产生了替代转录本;这一比率高于lncrna。TSS是花生lncRNAs中AS事件的最高发生率(28.05%),而IR在mrna中最高发生率(30.19%)。此外,在lncrna和mrna中ES事件发生频率最低。gydF4y2Ba
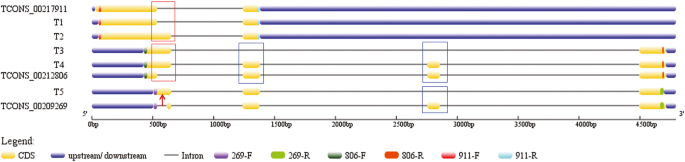
利用染色体Araip上的基因组片段94,398,232-94,402,918。以B05为例,3个lncrna (tcon_00217911, tcon_00212806, tcon_00209269)(附加文件gydF4y2Ba3.gydF4y2Ba:表S3)推测为一组as亚型,通过PCR检测进行验证。五个转录本,即T1-T5,被测序(图。gydF4y2Ba6gydF4y2Ba额外的文件gydF4y2Ba11.gydF4y2Ba:图S2)。T1与tcon_00217911相同,T2为tcon_00217911亚型,发生TSS事件。T3和T4为tcon_00212806的AS亚型,发生TSS和ES事件。T5为tcon_00209269的AS亚型,发生IR和ES事件。这些比对表明AS是lncRNA中普遍存在的现象,并可能在其功能调控中发挥重要作用。gydF4y2Ba
另一个与lncrna AS事件相关的现象被发现;即一个lncRNA的不同AS亚型具有不同的靶基因。以3个lncrna (tcon_00217911, tcon_00212806, tcon_00209269)的靶基因为例进行比较。他们都没有gydF4y2Ba独联体gydF4y2Ba-target基因(附加文件gydF4y2Ba6gydF4y2Ba:表S6)但有很多gydF4y2Ba反式gydF4y2Ba-target基因(附加文件gydF4y2Ba7gydF4y2Ba:表S7)。每一个都有12 14和11gydF4y2Ba反式gydF4y2Ba—target基因,分别。有五名gydF4y2Ba反式gydF4y2Ba-目标基因属于其中的三个。八个类似gydF4y2Ba反式gydF4y2Ba-靶基因分别属于tcon_00217911和tcon_00212806;五个相似gydF4y2Ba反式gydF4y2Ba-Target基因属于TCons_00217911和TCons_00209269;11类似.gydF4y2Ba反式gydF4y2Ba-target基因属于TCons_00209269和TCons_00212806。只有Tcons_00217911只有四个特定的靶基因(图。gydF4y2Ba2gydF4y2Baf).其他例子包括tcon_00011553、tcon_00013006和tcon_00013007,它们都位于Aradu基因组上。A01(99975619-99,981,506,附加文件gydF4y2Ba3.gydF4y2Ba:表S3)。基因结构分析结果显示,它们具有不同的AS亚型(附加文件gydF4y2Ba12.gydF4y2Ba:图S3A),共发现29个靶基因(附加文件gydF4y2Ba6gydF4y2Ba:表S6,附加文件gydF4y2Ba7gydF4y2Ba:表S7)。在这些靶基因中,有26个在3个基因中有代表性,而只有tcon_00013006没有特异性靶基因,其他两个只有一个(附加文件gydF4y2Ba12.gydF4y2Ba:图S3B)。AS可以改变lncRNA的靶基因谱,增加lncRNA的多样性和灵活性。这可能是lncrna执行其功能的一种重要方式。gydF4y2Ba
蛋白编码基因作为lncrna的来源gydF4y2Ba
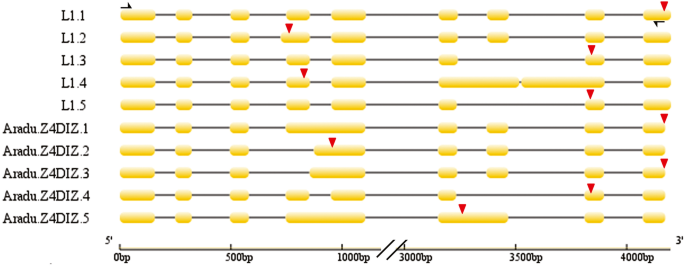
并不是所有的AS亚型都编码蛋白质并且在一定程度上是非编码的。花生AS蛋白编码基因产生的大量亚型似乎是非编码的,因为它们是截短的转录本;这些异构体可以被合理地视为一类lncRNA。几篇论文报告说,大约三分之一的替代文本可能是非编码的[gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba].这里,一种蛋白质编码基因gydF4y2BaAradu。Z4DIZ,gydF4y2Ba选择五个转录物,被选择为验证该点的示例(图。gydF4y2Ba7gydF4y2Ba额外的文件gydF4y2Ba13.gydF4y2Ba:图S4)。只有两款AS产品(gydF4y2BaAradu.z4diz.1.gydF4y2Ba和gydF4y2BaAradu.z4diz.3.gydF4y2Ba)可以编码完整的蛋白质。对该基因引物扩增产物的Sanger测序结果显示存在5种亚型:L1.5匹配gydF4y2BaAradu.z4diz.4gydF4y2Ba考虑到两个转录本都缺少第七外显子。L1.1编码232个氨基酸(aa),比预测的短18个残基gydF4y2BaAradu.z4diz.1.gydF4y2Ba产品。L1.2编码的118 AA,L1.4编码177 AA,L1.3编码240AA;三种编码的蛋白质不完整。预测的产品gydF4y2BaAradu。Z4DIZgydF4y2Ba是一种二酰基甘油o酰基转移酶,在其c端携带一个保守的LPLAT结构域;被截断的转录本都缺少一个完整的LPLAT结构域的决定因素。这一特点损害了翻译产品的功能。gydF4y2Ba
通过qRT-PCR验证lncrna的表达水平gydF4y2Ba
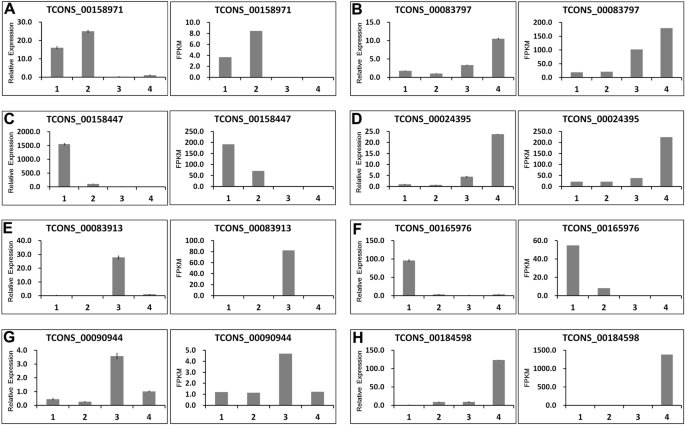
为了验证LNCRNA的丰度,通过QRT-PCR随机选择并分析八个LNCRNA(LNCRNA8-15)(附加文件gydF4y2Ba9gydF4y2Ba:表S9)。如图1所示。gydF4y2Ba8gydF4y2Ba,这些lncrna的表达模式与RNA-seq结果相对一致;这表明基于RNA-seq数据的lncRNA表达模式是可靠的。gydF4y2Ba
lncrna及其靶基因的RT-PCR共表达gydF4y2Ba
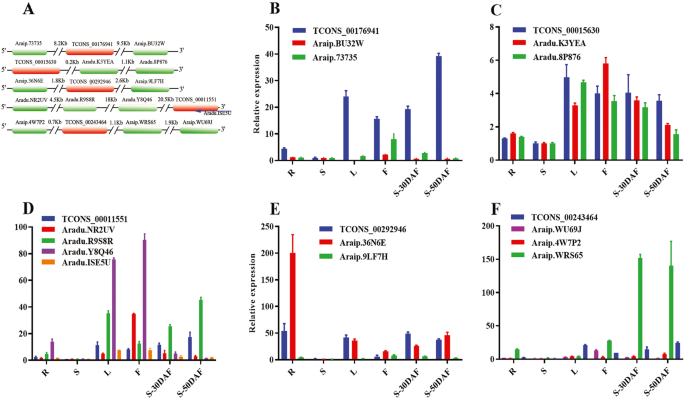
一个lncRNA可以与多个基因靶标相互作用,反之亦然(附加文件gydF4y2Ba14.gydF4y2Ba:图。S5)。五个lncrnas(lncrna16-20)及其目标基因(附加文件gydF4y2Ba9gydF4y2Ba: Table S9)来验证lncRNA与mRNA的关系,5个lncRNA与其靶基因的相对位置如图所示。gydF4y2Ba9gydF4y2Baa. tcon_00176941与两个靶基因分别位于上游8.2 Kb和下游9.5 Kb,距离较远。在所有六个器官中,tcon_00176941的表达水平均高于其两个靶基因的表达水平(图1)。gydF4y2Ba9gydF4y2Bab)。Tcons_00015630下游接近其两个靶基因(图。gydF4y2Ba9gydF4y2Baa),并在六个器官中显示出与Aradu.8p876类似的表达模式(图。gydF4y2Ba9gydF4y2BaC)。Tcons_00011551有三个gydF4y2Ba顺式gydF4y2Ba目标基因和一个gydF4y2Ba反式gydF4y2Ba-靶基因在6个器官中表现出不同的表达模式(图。gydF4y2Ba9gydF4y2Baa, d). tcon_00292946及其两个靶基因在花后30天(DAF)和花后50天(DAF)的茎、叶、花和种子中均表现出相似的表达模式。gydF4y2Ba9gydF4y2Bae)。Tcons_00243464及其三个靶基因在六个器官中显示出不同的表达模式(图。gydF4y2Ba9gydF4y2BaF)。gydF4y2Ba
盐胁迫下lncrna及其靶基因的表达谱分析gydF4y2Ba
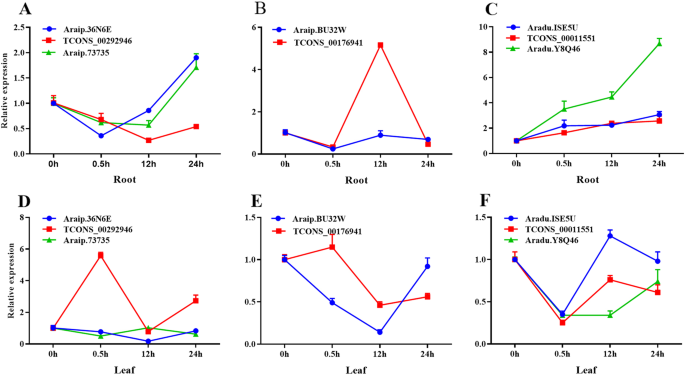
LNCRNA在应力阻力中起重要作用。这里,三个lncrnas及其目标基因(附加文件gydF4y2Ba9gydF4y2Ba:表S9),利用qRT-PCR检测盐胁迫下不同器官的表达模式(图。gydF4y2Ba10.gydF4y2Ba).这些靶基因与盐胁迫下的胁迫相关和不同的表达水平,因此选择相应的LNCRNA用于盐胁迫分析。结果表明,这三种LNCRNA及其靶基因显示出不同的盐处理表达模式。在根中,Tcons_00292946的表达水平在12小时内降低,然后在24小时内增加(图。gydF4y2Ba10.gydF4y2Baa).在叶片中,表达水平波动(图5)。gydF4y2Ba10.gydF4y2Bad)。Tcons_00176941在根和叶中显示出对立的表达水平(图。gydF4y2Ba10.gydF4y2Ba是)。Tcons_00011551的表达水平与盐渍一起缓慢增加(图。gydF4y2Ba10.gydF4y2Bac),但表达水平在叶子中波动(图。gydF4y2Ba10.gydF4y2BaF)。gydF4y2Ba
LNCRNA及其靶基因响应于盐胁迫而显示出不同的表达模式。Tcons_00292946及其两种靶基因在根中显示出类似的表达模式(图。gydF4y2Ba10.gydF4y2Baa)叶中的相反表达模式(图。gydF4y2Ba10.gydF4y2Bad). tcon_00176941和Araip。BU32Wshowed similar expression patterns in the roots (Fig.10.gydF4y2Bab)叶中的不同表达模式(图。gydF4y2Ba10.gydF4y2Bae). tcon_00011551和Aradu。ISE5Ushowed similar expression patterns in the roots and leaves, whereas TCONS_00011551 and Aradu.Y8Q46 showed different expression patterns in the leaves (Fig.10.gydF4y2BaC,F)。将进一步研究这些LNCRNA的动作模式,将进一步研究响应盐胁迫。gydF4y2Ba
讨论gydF4y2Ba
核苷酸测序技术的进步揭示了lncrna的存在,其中一些作为一系列真核细胞过程的调节因子[gydF4y2Ba15.gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba].通过转录组分析鉴定的LNCRNA的数量反映了所施加的测序方法的深度。一组> 13,000乌拉索乌米多克斯lncrnas [gydF4y2Ba2gydF4y2Ba, > 20,000玉米lncrna [gydF4y2Ba26.gydF4y2Ba, > 2000水稻lncrna已被报道[gydF4y2Ba49gydF4y2Ba].这里,对花生转录组的调查显示1442毫克。通过将我们的结果与Zhao的已发表的花生LNCRNA进行比较[gydF4y2Ba39gydF4y2Ba],其中从124个样本中鉴定出花生的50873个lncrna,涉及15个不同的组织,我们发现39个相同的lncrna,约占2.7%(数据见表S10)。识别出的序列数量少可能仅仅反映了实验规模小,实验只涉及4个文库,而赵的[gydF4y2Ba39gydF4y2Ba].此外,这里应用的选择标准比常用的选择标准更严格。例如,通过去除放置在外显子数量的限制,将预测> 10,000LNCRNA(数据未示出),其中大量将是误报。当对基于PCR的验证的小样本(七序列)进行PCR的验证时,> 70%证明在花生转录组中真正表示。gydF4y2Ba
如普遍采用的转录后基因调节所采用[gydF4y2Ba50gydF4y2Ba,gydF4y2Ba51gydF4y2Ba].在植物中,它被用于控制生长、发育、信号转导、开花、昼夜时钟功能和环境线索反应[gydF4y2Ba49gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba].类似于蛋白质编码基因,作为动物和植物的变体开发的许多LNCRNA的转录序列[gydF4y2Ba56gydF4y2Ba,gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba].例如,lncRNA-PXN-AS1缺乏外显子4,它与PXN mRNA的编码序列结合,抑制PXN mRNA的翻译。而包含外显子4的lncRNA-PXN-AS1则优先结合到PXN mRNA的3 '非翻译区,保护PXN mRNA不被降解,从而增加PXN的表达[gydF4y2Ba58gydF4y2Ba].这里,从花生RNA-SEQ数据预测了许多同种型,并且通过PCR进行实验验证了这些同种型是真实的(图。gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba).与蛋白质编码基因一样,在LNCRNA中发现各种作为蛋白质编码基因的类型(表gydF4y2Ba1gydF4y2Ba),但也存在显着的差异。我们发现IR最常见于mRNA中的事件,TSS是LNCRNA中的最高频率。由于LNCRNA的事件也可以增加LNCRNA的多样性。它可以改变LNCRNA的靶基因(图。gydF4y2Ba2gydF4y2Baf,额外的文件gydF4y2Ba12.gydF4y2Ba:图S3),从而增加了lncRNA的灵活性。这可能是lncrna的重要调控。gydF4y2Ba
另外,许多蛋白质编码基因转录各种变体;这些转录物中的几种不能编码功能蛋白,因为它们代表了截断形式的完全官能mRNA(图。gydF4y2Ba7gydF4y2Ba).这些序列在某种意义上可以看作是另一类lncrna或伪基因衍生的lncrna [gydF4y2Ba59gydF4y2Ba].编码蛋白质的mRNA转录本可以通过竞争常见的microrna与其他mRNA转录本进行串扰[gydF4y2Ba59gydF4y2Ba].据推测,假基因PTENP1在前列腺癌细胞中具有生物学活性,其与miR-17、miR-19、miR-21和miR-26家族竞争性结合,调节PTEN的细胞水平并发挥生长抑制作用[gydF4y2Ba60gydF4y2Ba].被认为不编码蛋白质的lncRNA也可以翻译小多肽来发挥它们的功能,但不是通过lncRNA本身[gydF4y2Ba61gydF4y2Ba,gydF4y2Ba62gydF4y2Ba].例如,Anderson等人发现了一个由骨骼肌特异性RNA编码的保守微肽,它被假定为lncRNA,在肌肉表现中起着重要作用[gydF4y2Ba16.gydF4y2Ba].类似地,作为从蛋白质编码基因衍生的变体的官能截短的多肽的生产存在实验证据。例如,gydF4y2Ba答:盐沼CCA1gydF4y2Ba生成两个亚型,其中一个是全尺寸转录本(gydF4y2BaCCA1αgydF4y2Ba),而另一种是删节形式(gydF4y2BaCCA1βgydF4y2Ba)缺乏功能性n端MYB dna结合域[gydF4y2Ba63gydF4y2Ba].后一转录物通过形成非官能异二二二二二二二二二聚物并通过低温调节来抑制CCA1α和后细长的胚轴转录因子。人类gydF4y2BaUSP2.gydF4y2Ba基因产生7个AS变异,其中2个没有翻译成功能蛋白[gydF4y2Ba64gydF4y2Ba].斑马鱼基因gydF4y2BaLGP2gydF4y2Ba形成三个作为转录物,其中只有全长版本可以赋予病毒感染保护[gydF4y2Ba65gydF4y2Ba].编码RNA和非编码RNA之间的区别并不像以前认为的那么明显。一些编码蛋白的基因产生非编码AS变异,可以被识别为lncRNAs;这些序列可能与基因调控有关。随着技术的进步和不懈的努力,我们将进一步明确lncrna可能的调控机制。gydF4y2Ba
越来越多的研究表明,lncRNAs在植物抗逆中发挥着重要作用[gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba47gydF4y2Ba].在这项研究中,在盐胁迫下测试一些具有靶基因的花生LNCRNA,结果表明,它们的所有表达水平改变了(图。gydF4y2Ba10.gydF4y2Ba).在杨树中,鉴定了超过10,000个LNCRNA,其中大约40%的盐胁迫表达组织特异性表达模式[gydF4y2Ba34gydF4y2Ba].在棉花中,lncRNA973定位于细胞核中,并通过盐处理而增加。lncRNA973在拟南芥中过表达可以提高耐盐性,而在棉花中敲低lncRNA973则降低耐盐性[gydF4y2Ba36gydF4y2Ba].此外,许多研究报道lncRNAs在植物抵御病原体方面发挥重要作用[gydF4y2Ba66gydF4y2Ba,gydF4y2Ba67gydF4y2Ba,gydF4y2Ba68gydF4y2Ba].在番茄中,发现了tylcv诱导疾病和宿主抗病毒免疫的可信模型;也就是说,lncRNAs与ir衍生的vsRNAs相互作用,在TYLCV感染过程中控制疾病的发展,这为控制植物病毒病原体提供了一个有效的策略[gydF4y2Ba67gydF4y2Ba].在水稻中,证实了lncrna与调控白叶枯病的JA途径之间的联系,这为植物抗病提供了新的思路[gydF4y2Ba68gydF4y2Ba].LNCRNA具有三种突出特征:低表达,物种之间缺乏保护和组织特异性表达模式[gydF4y2Ba23.gydF4y2Ba].这些特征表明,可以根据特定的物种和lncrna,针对特定的疾病开发出特定的防治方法。这将是今后植物抗病研究的发展方向。gydF4y2Ba
结论gydF4y2Ba
我们利用链特异性RNA-Seq技术在花生转录组中鉴定出1442个lncrna。其中,189个lncrna在根、叶或种子中差异丰富。具有外显子/内含子结构的lncrna中,约44.17%的lncrna选择性剪接。此速率略低于mRNA的剪接速率。AS改变了lncrna的靶基因谱,增加了lncrna的多样性和灵活性。这可能是lncrna执行其功能的一种重要方式。此外,蛋白质编码基因通过AS产生了许多截断的转录本,这可能是lncrna的另一个来源。选择部分lncrna及其靶基因进行盐响应实验。结果表明,三种植物对盐胁迫的响应方式各不相同。花生lncrna的鉴定将对花生的发育、生长和抗逆性育种产生重要影响。gydF4y2Ba
材料和方法gydF4y2Ba
材料gydF4y2Ba
花生品种‘奉化-1’在26℃下光照16小时,24℃下黑暗8小时的生长室内生长。从12日龄的幼苗(命名为fh1 -根和fh1 -叶)中采集根和叶,从开花后30天(DAF)(命名为fh1 -籽1)和50天(DAF)(命名为fh1 -籽2)的植株中采集发育中的种子。gydF4y2Ba
图书馆建设和排序gydF4y2Ba
总rna来源于fh1根、fh1叶、fh1籽1和fh1籽2。全转录组文库制备和深度测序如前所述[gydF4y2Ba69gydF4y2Ba].简而言之,在制造商的建议之后使用Nebnext®Ultra™定向RNA库预备套件(新英格兰Biolabs,MA,MA)制备整个转录组文库。最终,通过在Hiseq2500平台(Illumina,San Diego,CA,USA)上施加配对端125循环来执行测序。原始数据被存入NCBI序列读取存档(gydF4y2Bahttp://www.ncbi.nlm.nih.gov/sragydF4y2Ba),项目为PRJNA354652。gydF4y2Ba
转录组的组装gydF4y2Ba
序列数据被剥离适配器序列和低质量读取。每个文库获得的序列数据分别与花生基因组序列进行比对[gydF4y2Ba43gydF4y2Ba)(gydF4y2Bahttps://www.peanutbase.org/gydF4y2Ba)使用TopHat2软件[gydF4y2Ba70gydF4y2Ba].通过Cufflinks v2.2.1程序将对齐后的reads组装成一个完整的转录组[gydF4y2Ba71gydF4y2Ba].gydF4y2Ba
花生中lncrna的全基因组识别和可变剪接事件gydF4y2Ba
组装的转录物使用袖口组织设施(gydF4y2Bahttp://cufflinks.cbcb.umd.edu/gydF4y2Ba).选择长度超过200nt和至少两个外显子的转录物作为LNCRNA候选物。随后,使用编码潜在计算器(得分<0)分析转录物[gydF4y2Ba72gydF4y2Ba],编码 - 非划分指数(得分<0)[gydF4y2Ba73gydF4y2Ba],编码潜在评估工具[gydF4y2Ba74gydF4y2Ba]和pfam(e-value <0.001)[gydF4y2Ba75gydF4y2Ba移除所有可能残留的蛋白质编码基因。只有通过这四种扫描的序列被认为是可能的lncRNA候选序列。选择性剪接(AS)事件使用ASTALAVISTA程序(gydF4y2Bahttp://genome.crg.es/astalavista/gydF4y2Ba).作为事件的不同类别(TSS,TTS,ES,IR和AE)[gydF4y2Ba76gydF4y2Ba使用房屋中开发的Perl脚本来识别。gydF4y2Ba
目标基因预测和功能注释gydF4y2Ba
lncrna是否起作用gydF4y2Ba独联体gydF4y2Ba或在gydF4y2Ba反式gydF4y2Ba预测。还预测了靶基因的推定功能。在每个LNCRNA下游的5'上游或3'或3'中展示在100kb内的编码基因被鉴定为潜力gydF4y2Ba独联体gydF4y2Ba目标[gydF4y2Ba77gydF4y2Ba基于这些基因的表达水平之间的Pearson和Spearman相关系数是否≥0.6或≤-0.6,和gydF4y2BaPgydF4y2Ba < 0.05, whereas potential反式gydF4y2BaPearson相关系数r > 0.9, P < 0.05;利用LncTar程序构建基因共表达网络[gydF4y2Ba78gydF4y2Ba],寻找mrna和lncrna之间的序列互补性。随后,利用以下数据库对目的基因进行功能注释分析:NCBI非冗余蛋白序列[gydF4y2Ba79gydF4y2Ba],Kog / Cog [gydF4y2Ba80gydF4y2Ba],瑞士人[gydF4y2Ba81gydF4y2Ba], KEGG [gydF4y2Ba82gydF4y2Ba,和GO [gydF4y2Ba83gydF4y2Ba].具有默认参数的Kobas软件用于在Mao等人描述的方法后测试Kegg途径中的差异表达基因的统计富集。[gydF4y2Ba84gydF4y2Ba].gydF4y2Ba
lncrna的差异丰度gydF4y2Ba
使用Cufflinks v2.2.1软件计算各文库中与lncrna及编码基因相关的FPKM值。使用EBseq (2010) R包进行分析[gydF4y2Ba85gydF4y2Ba].gydF4y2BaPgydF4y2Ba-Values调整为Q值[gydF4y2Ba86gydF4y2Ba].只有符合q-value < 0.01和log2 (fold change) > 1标准的lncrna被认为是DALs。gydF4y2Ba
PCR验证LNCRNAgydF4y2Ba
从RNA-seq数据中鉴定出的7个假定的lncrna (lncRNA1-7)通过PCR检测进行了验证(附加文件gydF4y2Ba9gydF4y2Ba:表S9)。按照制造商的建议,使用RevertAid第一链cDNA合成试剂盒(Thermo Scientific™,Waltham, MA)对用于构建文库的总RNA进行逆转录。随后,将反应产物稀释20倍作为PCR模板,由补充表S9中的引物驱动。每50μL反应由5μL(10×TransTaq®HiFi缓冲II (Transgen生物技术,北京),4μL 2.5Μ核苷酸(每10μm), 1μL的正向引物和1μL反向引物(每10μm), 4μL的cDNA模板(50 ng /μL), 1μL TransTaq®HiFi DNA聚合酶(Transgen生物技术)和35 ddH的μLgydF4y2Ba2gydF4y2BaO.扩增产物经纯化(TIANgel Midi纯化试剂盒DP209,天根生物技术,北京,中国)并亚克隆到pEASY-T1克隆载体(Transgen生物技术)中进行Sanger测序。gydF4y2Ba
利用实时荧光定量PCR定量lncrna和目的基因的丰度gydF4y2Ba
利用实时定量PCR (RT-qPCR)平台验证lncrna (lncRNA8-20, Supplementary Table S1)和邻近靶基因(gydF4y2BaARAIP.73735.gydF4y2Ba,gydF4y2BaAraipgydF4y2Ba.gydF4y2BaBU32WgydF4y2Ba,gydF4y2BaAradugydF4y2Ba.gydF4y2BaK3YEAgydF4y2Ba,gydF4y2BaAradugydF4y2Ba.gydF4y2Ba8 p876gydF4y2Ba,gydF4y2BaAraipgydF4y2Ba.gydF4y2Ba36n6e.gydF4y2Ba,gydF4y2BaAraipgydF4y2Ba.gydF4y2Ba9 lf7hgydF4y2Ba,gydF4y2BaAradugydF4y2Ba.gydF4y2Banr2uv.gydF4y2Ba,gydF4y2BaAradugydF4y2Ba.gydF4y2BaR9S8R.gydF4y2Ba,gydF4y2BaAradugydF4y2Ba.gydF4y2BaY8Q46.gydF4y2Ba,gydF4y2BaAradugydF4y2Ba.gydF4y2BaISE5UgydF4y2Ba,gydF4y2BaAraipgydF4y2Ba.gydF4y2Ba4 w7p2gydF4y2Ba,gydF4y2BaAraipgydF4y2Ba.gydF4y2BaWRS65gydF4y2Ba,gydF4y2BaAraipgydF4y2Ba.gydF4y2BaWU69JgydF4y2Ba,补充表S9)。通过使用MMLV逆转录酶根据制造商的方案,通过使用MMLV逆转录酶合成具有随机六聚体引物的组织的QRT-PCR组织的组织的总RNA。所有RT-QPCR反应在ABI 7500快速实时PCR平台上以SYBR®Green实时PCR主混合物(Toyobo,Osaka,Japan)在每次cDNA样品中以三态酸盐进行。通过熔化曲线分析验证了PCR产物的特异性,并通过使用2来定量LNCRNA和基因表达gydF4y2Ba——ΔΔCtgydF4y2Ba方法,具有基因的丰富转录物gydF4y2BaActin-1gydF4y2Ba(XP_015966232.1)用于规范化。引物序列见补充表S1。gydF4y2Ba
作为同种型通过PCR验证LNCRNAgydF4y2Ba
设计了3对特异性引物(911-F/R, 269-F/R和806-F/R,附加文件:表S9),以检测具有相同基因组位置的3个lncRNA (tcon_00217911, Araip)的AS亚型的存在。B05 94398232 - 94402710;TCONS_00209269 Araip。B05 94398710 - 94402916;TCONS_00212806 Araip。B05 94398630 - 94402918)。一个基于KOD-Plus-neo酶的反应(TOYOBO,大阪,日本)根据制造商的建议进行。纯化后,将扩增产物如上所示转移至测序。gydF4y2Ba
来自蛋白质编码基因的LNCRNAgydF4y2Ba
一对引物(Aradu.z4diz-F / R,附加文件gydF4y2Ba9gydF4y2Ba表S9)用于鉴定该基因产生的AS亚型gydF4y2BaAradu。Z4DIZgydF4y2Ba.该PCR基于制造商指令后的50μL反应体积的Kod-Plus-Neo酶(Toyobo,Osaka,日本)。纯化扩增子并如上所述转移并测序。gydF4y2Ba
盐处理和qRT-PCR验证gydF4y2Ba
在盐处理条件下,花生幼苗生长状态均匀(2周龄,约8 cm高),1% NaCl处理。在处理后0、0.5、12和24 h收获根和叶。收获的材料在液氮中快速冷冻,并在−80°C保存,直到需要提取RNA。总RNA使用DP441 RNAprep Pure Plant试剂盒(Tiangen, Beijing)制备,所得到的RNA使用RevertAid First Strand cDNA Synthesis kit (K1621, Thermo Scientific™)转化成cDNA。RT-PCR分析在ABI 7500 FAST实时PCR平台上使用SYBR®Green Realtime PCR Master Mix (TOYOBO,大阪,日本)进行三次重复。gydF4y2Ba
三组LNCRNA及其靶基因(额外文件中的引物gydF4y2Ba9gydF4y2Ba:表S9)。每20 μL反应包括TaqMan Fast qPCR Master Mix 10 μL,非标记引物各0.4 μL (10 μM),荧光标记引物各0.4 μL (10 μM), cDNA 2 μL (100 ng/μL), ddH 6.8 μLgydF4y2Ba2gydF4y2Ba相对转录本丰度用2gydF4y2Ba——ΔΔCTgydF4y2Ba方法 [gydF4y2Ba87gydF4y2Ba].每个反应为每个cDNA样本运行三次。gydF4y2Ba
可用性数据和材料gydF4y2Ba
转录组原始数据gydF4y2BaA. hypogaea.gydF4y2Ba可在NCBI Project Prjna354652获取登录号SRR5053815,SRR5054076,SRR5054058和SRR5054059。gydF4y2Ba
缩写gydF4y2Ba
- AE:gydF4y2Ba
-
替代外显子结束gydF4y2Ba
- 作为:gydF4y2Ba
-
替代拼接gydF4y2Ba
- DAF:gydF4y2Ba
-
天前,花gydF4y2Ba
- DETGs:gydF4y2Ba
-
差异表达靶基因gydF4y2Ba
- es:gydF4y2Ba
-
外显子跳跃gydF4y2Ba
- FPKM:gydF4y2Ba
-
每千个碱基的片段每百万个片段的外显子gydF4y2Ba
- 走:gydF4y2Ba
-
基因本体论gydF4y2Ba
- incRNAs:gydF4y2Ba
-
内泌系统LNCRNA.gydF4y2Ba
- 红外光谱:gydF4y2Ba
-
基因内区保留gydF4y2Ba
- KEGG:gydF4y2Ba
-
序列京都基因和基因组百科全书gydF4y2Ba
- KOG /齿轮:gydF4y2Ba
-
直际蛋白质群体gydF4y2Ba
- Lincrnas:gydF4y2Ba
-
长基因间ncRNAsgydF4y2Ba
- lncRNAs:gydF4y2Ba
-
长非编码rnagydF4y2Ba
- microrna:gydF4y2Ba
-
microRNA.gydF4y2Ba
- ncRNAs:gydF4y2Ba
-
非编码RNA.gydF4y2Ba
- Nr:gydF4y2Ba
-
冗余蛋白质序列gydF4y2Ba
- 子:gydF4y2Ba
-
开放阅读框gydF4y2Ba
- 聚合酶链反应:gydF4y2Ba
-
聚合酶链反应gydF4y2Ba
- RT-PCR:gydF4y2Ba
-
逆转录 - 聚合酶链反应gydF4y2Ba
- TSS:gydF4y2Ba
-
转录起始点gydF4y2Ba
- TTS:gydF4y2Ba
-
转录终端网站gydF4y2Ba
参考gydF4y2Ba
- 1。gydF4y2Ba
Simms CL, Zaher HS。化学损伤RNA的质量控制。细胞和分子生命科学:cml。2016; 73(19): 3639 - 53。gydF4y2Ba
- 2。gydF4y2Ba
刘杰,荣昌昌,徐杰,王华,邓森,阿雷纳斯-胡尔特罗,蔡nh。全基因组分析揭示了拟南芥长基因间非编码rna的调控。植物细胞,2012;24(11):4333 - 45。gydF4y2Ba
- 3.gydF4y2Ba
陈亚,Aravin AA。转录调控中的非编码rna:当前分子生物学报道综述。2015;1(1): 10-8。gydF4y2Ba
- 4.gydF4y2Ba
PONTING CP,OLIVER PL,REIK W.长的NOODING RNA的进化和功能。细胞。2009; 136(4):629-41。gydF4y2Ba
- 5.gydF4y2Ba
Donaldson Me,Saville Bj。真菌的天然反义转录物。mol microbiol。2012; 85(3):405-17。gydF4y2Ba
- 6.gydF4y2Ba
Quan M,Chen J,张D.探索长码RNA的秘密。int j mol sci。2015; 16(3):5467-96。gydF4y2Ba
- 7.gydF4y2Ba
刘X,Hao L,Li D,Zhu L,Hu S.长期非编码RNA及其在植物中的生物学作用。基因组学,蛋白质组学与生物信息学。2015; 13(3):137-47。gydF4y2Ba
- 8.gydF4y2Ba
Chekanova农协。长链非编码rna及其在植物中的功能。植物生物学杂志。2015;27:207-16。gydF4y2Ba
- 9.gydF4y2Ba
大豆基因组的长基因间非编码RNA (lincRNA)图谱。植物杂志。2017;176(3):2133 - 47岁。gydF4y2Ba
- 10。gydF4y2Ba
Lukiw WJ, Handley P, Wong L, Crapper McLachlan DR.在正常人类新皮质、非阿尔茨海默病(NAD)和阿尔茨海默病(AD)的老年性痴呆中发现BC200 RNA。Neurochem杂志1992;17(6):591 - 7。gydF4y2Ba
- 11.gydF4y2Ba
吕勇,梁智,葛明,齐伟,张涛,林芳,彭智,赵宏:玉米氮响应基因间rna和内含子长非编码rna的全基因组鉴定和功能预测(gydF4y2Ba玉米gydF4y2BaL.)。gydF4y2BaBMC基因组学gydF4y2Ba2016,17(1):350。gydF4y2Ba
- 12.gydF4y2Ba
研究表明,Xist lncRNA通过HDAC3与SHARP直接相互作用来抑制转录。大自然。2015;521(7551):232 - 6。gydF4y2Ba
- 13。gydF4y2Ba
Stojic L,Niemczyk M,Orjalo A,ITO Y,Ruijter AEM,Uribe-Lewis S,Joseph N,Weston S,Menon S,Odom DT。长度非数性RNA GNG12-AS1的转录沉默不稳定其转录和产品相关的功能。NAT Communce。2016; 7:10406。gydF4y2Ba
- 14.gydF4y2Ba
沙棘成熟期长链非编码rna的全基因组分析(gydF4y2Bahippophae rhamnoides.gydF4y2BaLinn)水果。基因。2016;596:130-6。gydF4y2Ba
- 15.gydF4y2Ba
转录组分析揭示长链非编码rna参与棉花纤维发育(英文)gydF4y2BaGossypium Arboreum.gydF4y2Ba).中国科学:生命科学,2016;59(2):164-71。gydF4y2Ba
- 16.gydF4y2Ba
安德森DM,安德森公里,昌Cl,Makarewich Ca,Nelson Br,Mcanally Jr,Kasaragod P,Shelton JM,Liou J,Bassel-Duby R等。通过推定的长性非编码RNA编码的微肽调节肌肉性能。细胞。2015; 160(4):595-606。gydF4y2Ba
- 17.gydF4y2Ba
非编码RNA调控适应性免疫反应的动态特性。中国生物医学工程学报,2013;gydF4y2Ba
- 18.gydF4y2Ba
essers pb,nonnekens j,Goos yj,Betist Mc,Viester Md,Mossink B,Lansu N,Korswagen HC,Jelier R,Brenkman Ab,等。终点延伸需要核糖体上的长非编码RNA。细胞批准。2015; 10(3):339-45。gydF4y2Ba
- 19.gydF4y2Ba
Kadakkuzha BM,刘XA,McCrate J,Shankar G,Rizzo V,Afinogenova A,Young B,Reallahi M,Carvalloza Ac,Raveenra B等人。成人小鼠脑的转录组分析显示在特定脑区和神经元群体中LNCRNA的富集。前细胞Neurosci。2015; 9:63。gydF4y2Ba
- 20.gydF4y2Ba
li J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J, liu J。摩尔细胞。2014;55(1):138 - 47。gydF4y2Ba
- 21.gydF4y2Ba
Paralkar VR,Weiss MJ。生物学和血液缺血中的长度不编码RNA。血液。2013; 121(24):4842-6。gydF4y2Ba
- 22。gydF4y2Ba
王T-Z,刘米,赵M-G,陈岚,张W-H。基因组高通量测序,Medicago Truncatula中渗透渗透和盐胁迫的长编码RNA的鉴定与表征。BMC植物BIOL。2015; 15(1):131。gydF4y2Ba
- 23。gydF4y2Ba
王辉,钟培军,刘杰,张智勇,张金明,徐杰,蔡惠慧。拟南芥长链非编码自然反义转录本的全基因组鉴定及其对光的响应。基因组研究》2014;24(3):444 - 53年。gydF4y2Ba
- 24。gydF4y2Ba
关键词:水稻,甲基化,光周期,光敏感,雄性不育摩尔。2012;5(6):1210 - 6。gydF4y2Ba
- 25。gydF4y2Ba
小麦条锈病胁迫应答的非编码基因gydF4y2BaTriticum Aestivum.gydF4y2BaL.)。J分子生物学报告。2013; 40(11):6245-53。gydF4y2Ba
- 26。gydF4y2Ba
Li L, Eichten SR, Shimizu R, Petsch K, Yeh CT, Wu W, Chettoor AM, Givan SA, Cole RA, Fowler JE等。玉米长链非编码rna的全基因组发现与鉴定。基因组医学杂志。2014;15 (2):R40。gydF4y2Ba
- 27。gydF4y2Ba
1 .王静,于伟,杨勇,陈涛,刘涛,刘瑞:番茄长链非编码rna全基因组分析及microRNA内源靶模拟物的鉴定。Sci Rep 2015, 5(16946)。gydF4y2Ba
- 28.gydF4y2Ba
宋X,Sun L,Luo H,MA Q,Zhao Y,Pei D.基因组 - 范围内鉴定和桑树长期非编码RNA的特征(gydF4y2BaMorus notabilis.gydF4y2Ba)RNA-SEQ数据。基因(巴塞尔)。2016; 7(3):13。gydF4y2Ba
- 29.gydF4y2Ba
帅P,梁D,唐S,张Z,叶凯,苏y,x x,尹w。基因组鉴定和杨树育植物中新型和干旱响应Lincrnas的功能预测。J Exp Bot。2014; 65(17):4975-83。gydF4y2Ba
- 30.gydF4y2Ba
关键词:杂交水稻,长链非编码RNA,光敏感,雄性不育abstract:中国科学院学报,2012;gydF4y2Ba
- 31。gydF4y2Ba
HEO JB,SUNG S. vernalization介导的介导的外膜遗传沉默,长内肾内含RNA。科学。2011; 331(6013):76-9。gydF4y2Ba
- 32。gydF4y2Ba
Sun Q,Dean C. R环稳定抑制拟南芥FLC基因座的反义转录。科学。2013; 340(6132):619-21。gydF4y2Ba
- 33。gydF4y2Ba
艾哈迈德W,夏y,李河,白克,盾牌khm,郭P.非编码RNA:芸苔作物应力反应调节中的功能作用。基因组学。2019; S0888-7543(18):30626-8。gydF4y2Ba
- 34。gydF4y2Ba
Ma J,Bai X,Luo W,Feng Y,Shao X,Bai Q,Sun S,Long Q,WAN D.全基因组鉴定长的非划分RNA及其对两个密切相关杨树的盐胁迫的反应。前群体。2019; 10:777。gydF4y2Ba
- 35。gydF4y2Ba
关键词:热应激,长链非编码rna,全基因组识别,功能预测gydF4y2Ba绿僵菌属robertsiigydF4y2Ba.Microbiol前面。2019;10:2336。gydF4y2Ba
- 36。gydF4y2Ba
张X,东杰,邓福,王文,程义,宋L,胡m,shen j,xu q,沉f.长期非编码RNA lncrna973参与棉花胁迫的棉花应激。BMC植物BIOL。2019; 19(1):459。gydF4y2Ba
- 37。gydF4y2Ba
Qin T,Zhao H,Cui P,Albesher N,Xiong L.核局部长的非编码RNA增强干旱和盐胁迫耐受性。植物理性。2017年; 175(3):1321-36。gydF4y2Ba
- 38.gydF4y2Ba
关键词:长链非编码RNA,植物,逆境响应,调控机制生物化学与生物物理学报。2018;503(2):402-7。gydF4y2Ba
- 39.gydF4y2Ba
赵X,GaN L,Yan C,Li C,Sun Q,Wang J,Yuan C,张H,Shan S,Liu Jn。基因组 - 花生长期非编码RNA的识别与表征。基因。2019; 10(7):536。gydF4y2Ba
- 40.gydF4y2Ba
Hammons Ro:地生的起源和历史。在:gydF4y2Ba花生作物。gydF4y2Ba编辑J S, Smartt J edn。多德雷赫特:施普林格;1994: 24-42。gydF4y2Ba
- 41.gydF4y2Ba
Mallikarjuna N,Varshney R. K .:遗传学,基因组学和花生育种。Boca Raton,FL,USA:CRC压力机;2014年。gydF4y2Ba
- 42.gydF4y2Ba
瓦尔斯特·瑞克,Pandey Mk。Puppala n:[植物基因组纲要]花生基因组||用于映射基因和量化性状在花生中的古典和分子方法;2017年。gydF4y2Ba
- 43.gydF4y2Ba
Huang G, Farmer AD, Cannon EK, Liu X, Gao D, Clevenger J, Dash S,等。基因组序列gydF4y2Ba落花生duranensisgydF4y2Ba和gydF4y2Baarachis ipaensis.gydF4y2Ba花生的二倍体祖先。Nat麝猫。2016;48(4):438 - 46。gydF4y2Ba
- 44.gydF4y2Ba
关键词:木薯,转录组,冷胁迫,选择性剪接,转录组机器人学报。2010;30(1):1 - 7。gydF4y2Ba
- 45.gydF4y2Ba
Lewis BP, Green RE, Brenner SE。人类中选择性剪接和无意义介导的mRNA衰减广泛耦合的证据。《美国国家科学院学报》上。2003, 100(1): 189 - 92。gydF4y2Ba
- 46。gydF4y2Ba
二穗短柄草非编码rna的全基因组鉴定与表达分析。Mol Biol. 2018; 1667:31-42。gydF4y2Ba
- 47。gydF4y2Ba
关键词:棉花,盐胁迫,长链非编码rna (lncRNAs)植物生物学学报。2018;18(1):23。gydF4y2Ba
- 48。gydF4y2Ba
关键词:香蕉,‘Cavendish’香蕉,镰刀菌,非编码rnagydF4y2Ba穆萨acuminatagydF4y2Ba).Sci众议员2017;7(1):16939。gydF4y2Ba
- 49。gydF4y2Ba
张永超,廖建勇,李志勇,于勇,张建平,李秋芳,曲丽华,舒伟生,陈永强。全基因组筛选和功能分析发现了大量参与水稻有性生殖的长链非编码rna。基因组医学杂志。2014;15(12):512。gydF4y2Ba
- 50。gydF4y2Ba
Reddy AS, Marquez Y, Kalyna M, Barta A.植物交错景观的复杂性植物细胞。2013;25(10):3657 - 83。gydF4y2Ba
- 51。gydF4y2Ba
Stamm S, Ben-Ari S, Rafalska I, Tang Y, Zhang Z, Toiber D, Thanaraj TA, Soreq H. Function of alternative splicing。基因。2005;344:1-20。gydF4y2Ba
- 52。gydF4y2Ba
Emmy E,Cabrito Tr,Batista Ra,Hussein Ma,Teixeira MC,Athanasiadis A,Sa-Correia I,Duque P. Intron Intron在新型ZIF2转运蛋白的5'UTR中留下来改变促进拟南芥锌耐受性。Plos Genet。2014; 10(5):E1004375。gydF4y2Ba
- 53。gydF4y2Ba
唐,郑义,东俊,玉俊,悦吉,刘福,郭X,黄S,wisniewski m,sun j等。综合转录组分析显示果实发育期间长的非沉积RNA表达和替代剪接调节,并在Kiwifruit成熟(gydF4y2BaActinidia chinensis.gydF4y2Ba).植物生态学报,2016;gydF4y2Ba
- 54。gydF4y2Ba
杨某,唐F,朱H.植物免疫替代剪接。int j mol sci。2014; 15(6):10424-45。gydF4y2Ba
- 55。gydF4y2Ba
张Q,张X,王S,Tan C,周G,Li C.替代剪接在大麦种子萌发中的参与。Plos一个。2016; 11(3):E0152824。gydF4y2Ba
- 56。gydF4y2Ba
关键词:长链非编码基因,小干扰RNA,选择性剪接,多聚腺苷酸,耦合新植醇。2018;217(4):1535 - 50。gydF4y2Ba
- 57。gydF4y2Ba
杨t,周h,刘p,yan l,姚w,陈k,zeng j,李h,胡锦涛gydF4y2Ba等gydF4y2Ba: lncRNA PVT1及其剪接变体作为内源性竞争性RNA调控透明细胞肾细胞癌的进展。Oncotarget。2017;8(49):85353 - 67。gydF4y2Ba
- 58。gydF4y2Ba
袁杰,刘Xn,王tt,潘w,陶qf,周wp,王f,sun sh。通过通过LNCRNA-PXN-AS1的替代剪接增加PXN表达,MBNL3剪接因子通过替代剪接提高肝细胞癌。NAT细胞BIOL。2017; 19(7):820-32。gydF4y2Ba
- 59。gydF4y2Ba
假基因来源的lncrna:基因表达的新兴调控因子。麝猫。2014;5:476。gydF4y2Ba
- 60.gydF4y2Ba
Tay Y, Lev K, Leonardo S, Dror W, Shen Mynn T, Ugo A, Florian K, Laura P, Paolo P, Ferdinando DCgydF4y2Ba等gydF4y2Ba:通过竞争性内源性mrna对肿瘤抑制因子PTEN的编码独立调控。单元2011,147(2):0 - 357。gydF4y2Ba
- 61.gydF4y2Ba
关键词:长链lncRNA, HOXB-AS3,结肠癌,抑制摩尔细胞。2017;68(1):171 - 84。gydF4y2Ba
- 62.gydF4y2Ba
Tajbakhsh s . lncrna编码的多肽SPAR(s)与mTORC1调节骨骼肌再生。细胞干细胞。2017;20(4):428-30。gydF4y2Ba
- 63.gydF4y2Ba
Park MJ, Seo PJ, Park CM。拟南芥的昼夜节律时钟- associated1的自我调节回路是温度响应的昼夜节律时钟调节的基础。植物细胞。2012;7(9):1194 - 6。gydF4y2Ba
- 64.gydF4y2Ba
朱汉平,高富华。USP2选择性剪接调控的分子机制及其产物的意义。中国生物医学工程学报。2017;13(12):1489-96。gydF4y2Ba
- 65.gydF4y2Ba
张QM,赵X,李Z,吴M,GUI JF,Zhang YB。斑马鱼LGP2基因的替代剪接转录物差异有助于IFN抗病毒反应。J免疫酚。2018; 200(2):688-703。gydF4y2Ba
- 66.gydF4y2Ba
Zaynab M, Fatima M, Abbas S, Umair M, Sharif Y, Raza MA。长链非编码rna作为植物抵御病原体的分子机制。活细胞Pathog。2018;121:277 - 82。gydF4y2Ba
- 67.gydF4y2Ba
杨勇,刘涛,沈丹,王军,凌翔,胡忠,陈涛,胡军,黄军,于伟,等。番茄黄曲叶病毒基因间sirna靶向宿主长链非编码RNA调节疾病症状。公共科学图书馆Pathog。2019;15 (1):e1007534。gydF4y2Ba
- 68.gydF4y2Ba
俞y,周你,冯yz,何H,连杰,杨yw,雷米克,张yc,陈yq:稻米病原体响应Lncrnas的转录景观揭示了alex1在茉莉酮族途径中的作用和疾病性。gydF4y2Ba生物科技植物》JgydF4y2Ba2019:gydF4y2Bahttps://doi.org/10.1111/pbi.13234gydF4y2Ba.gydF4y2Ba
- 69.gydF4y2Ba
阮j,guo f,王y,李x,万s,山l,彭z:花生替代剪接转录组分析(gydF4y2Ba落花生hypogaeagydF4y2BaL.)。BMC植物BIOL 2018,18(139)。gydF4y2Ba
- 70.gydF4y2Ba
Kim D,Pertea G,Trapnell C,Pimentel H,Kelley R,Salzberg SL。TOPHAT2:在存在插入,缺失和基因融合的情况下,转录om的精确对准。基因组Biol。2013; 14(4):R36。gydF4y2Ba
- 71。gydF4y2Ba
Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ, Salzberg SL, Wold BJ, Pachter L.转录本组装和定量的RNA-Seq揭示了细胞分化过程中未注释的转录本和亚型转换。生物科技Nat》。2010;28(5):511 - 5。gydF4y2Ba
- 72。gydF4y2Ba
关键词:转录本,蛋白质编码,序列特征,支持向量机gydF4y2Ba核酸研究gydF4y2Ba2007、69 (suppl_2): 1-13。gydF4y2Ba
- 73。gydF4y2Ba
关键词:蛋白质编码,长链非编码转录本,序列内在组成核酸杂志2013;41(17):e166。gydF4y2Ba
- 74。gydF4y2Ba
关键词:编码潜力评估,无对齐logistic回归模型,CPAT2013;41(6):e74。gydF4y2Ba
- 75。gydF4y2Ba
本文介绍了一种新的研究方法,该方法可用于临床应用。pam:蛋白家族数据库。核酸研究2014;42(数据库版本):D222-30。gydF4y2Ba
- 76。gydF4y2Ba
Florea L, Song L, Salzberg SL:数千个外显子跳跃事件区分了16个人体组织的剪接模式。gydF4y2BaF1000ResearchgydF4y2Ba2013,2:188。gydF4y2Ba
- 77。gydF4y2Ba
4 .李建勤,姜晓明,张丽丽,李建勤:灵芝长基因间非编码rna的全基因组鉴定与鉴定。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba2014年,9 (6):e99442。gydF4y2Ba
- 78。gydF4y2Ba
李家,MA W,Zeng P,Wang J,Geng B,Yang J,Cui Q.LNCTAR:一种用于预测长rNA的RNA靶的工具。简短生物形式。2015; 16(5):806-12。gydF4y2Ba
- 79。gydF4y2Ba
邓益,李杰克,吴SF,朱yp,陈耶,他fc。蛋白质注释系统中的集成NR数据库及其本地化。计算ENG。2006; 32(5):71-2。gydF4y2Ba
- 80.gydF4y2Ba
Tatusov RL, Galperin MY, Natale DA, Koonin EV。COG数据库:用于蛋白质功能和进化基因组规模分析的工具。《核酸研究》2000;28(1):33-6。gydF4y2Ba
- 81.gydF4y2Ba
Apweiler R,Bairoch A,Wu Ch,Barker Wc,Boeckmann B,Ferro S,Gasteiger E,Huang H,Lopez R,Magrane M等。UNIPROT:通用蛋白质知识库。核酸RES。2004; 45(D1):D158-69。gydF4y2Ba
- 82.gydF4y2Ba
关键词:KEGG,基因组,KEGG资源,生物信息学核酸研究2004;32(数据库版):D277-80。gydF4y2Ba
- 83.gydF4y2Ba
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT等。基因本体论:生物学统一的工具。Nat麝猫。2000;得分上以25 - 9胜过25(1):。gydF4y2Ba
- 84.gydF4y2Ba
关键词:KEGG Orthology (KEGG Orthology, KO),自动基因组注释,路径识别生物信息学。2005;21(19):3787 - 93。gydF4y2Ba
- 85.gydF4y2Ba
Leng N, Dawson JA, Thomson JA, Ruotti V, Rissman AI, Smits BMG, Haag JD, Gould MN, Stewart RM, Kendziorski C. EBSeq:一个用于RNA-seq实验推理的经验贝叶斯层次模型。生物信息学。2013;29(8):1035 - 43。gydF4y2Ba
- 86.gydF4y2Ba
楼层jd。正面错误发现RAE:贝叶斯解释和Q值。安统计。2003; 31(6):2013-35。gydF4y2Ba
- 87.gydF4y2Ba
Livak KJ,Schmittgen TD。使用实时定量PCR和2分析相对基因表达数据gydF4y2Ba——ΔΔCTgydF4y2Ba方法。方法。2001;25:402-8。gydF4y2Ba
致谢gydF4y2Ba
我们感谢SS博士(山东花生研究所,中国青岛。gydF4y2Bashansh_spri@163.comgydF4y2Ba),以获得花生lncrna的数据。gydF4y2Ba
资金gydF4y2Ba
国家重点研发计划项目(no . 2018YFD1000900);山东省自然科学基金重大基础研究项目(no . 2018GHZ007);山东省重大科技创新项目(no . 2018YFJH0601);山东省农业科学院科技创新项目(CXGC2018B05, CXGC2018D04, CXGC2018E13)。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
HT和FG获得数据,JM和HD分析解释数据,ZZ获得和分析数据,XL、SW和ZP参与了稿件的设计和起草,修改稿件并最终批准出版版本。所有作者阅读并批准最终稿件。gydF4y2Ba
相应的作者gydF4y2Ba
伦理宣言gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
花生品种“凤凰瓦”是由山东农业大学永山湾教授提供的。'Fenghua1'是一个很好的花生品种,广泛种植在华北,可以随时购买和销售。gydF4y2Ba
同意出版gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者们宣称他们没有相互竞争的利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意事项gydF4y2Ba
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:表S1。gydF4y2Ba
四个总RNA-seq数据集的概述。gydF4y2Ba
附加文件2:表S2。gydF4y2Ba
RNA-SEQ数据产生和四个样品的对准结果。gydF4y2Ba
附加文件3:表S3。gydF4y2Ba
1442 Lncrnas的详细信息。gydF4y2Ba
附加文件4:表S4。gydF4y2Ba
189个差异表达lncrna。gydF4y2Ba
附加文件5:表S5。gydF4y2Ba
花生染色体中LNCRNA的分布。gydF4y2Ba
附加文件6:表S6。gydF4y2Ba
独联体gydF4y2Ba-LNCRNA的-Target基因。gydF4y2Ba
附加文件7:表S7。gydF4y2Ba
反式gydF4y2Ba-LNCRNA的-Target基因。gydF4y2Ba
附加文件8:表S8。gydF4y2Ba
靶基因的集成功能注释。gydF4y2Ba
附加文件9:表S9。gydF4y2Ba
本文所用的引物。gydF4y2Ba
附加文件10:表S10。gydF4y2Ba
与已发表的花生LNCRNA的比较gydF4y2Ba
附加文件11:图S1。gydF4y2Ba
7个lncrna (lncRNA1-7)与相应的rna序列比对。gydF4y2Ba
附加文件12图S2。gydF4y2Ba
5个lncrna的Sanger测序(T1-5)。gydF4y2Ba
附加文件13:图S3。gydF4y2Ba
lncrna和不同AS亚型的AS事件具有不同的靶基因。gydF4y2Ba
附加文件14:图S4。gydF4y2Ba
Sanger测序五个lncrnas(l1.1-1.5)。gydF4y2Ba
附加文件15:图S5。gydF4y2Ba
lncrna -蛋白相互作用网络。gydF4y2Ba
权利和权限gydF4y2Ba
开放访问gydF4y2Ba本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料包括在文章的创作共用许可中,除非在材料的信用线中另有说明。如果材料没有包含在文章的创作共用许可证中,而您的预期使用不被法律法规允许或超过允许的使用,您将需要直接获得版权持有人的许可。如欲浏览本许可证的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.Creative Commons公共领域奉献豁免(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非另有用入数据的信用额度。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
田浩,郭峰,张志明。gydF4y2Baet al。gydF4y2Ba中长链非编码rna的发现、鉴定和功能表征gydF4y2Ba落花生hypogaeagydF4y2BalgydF4y2BaBMC植物杂志gydF4y2Ba20.gydF4y2Ba308(2020)。https://doi.org/10.1186/s12870-020-02510-4gydF4y2Ba
已收到gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-020-02510-4gydF4y2Ba
关键字gydF4y2Ba
- 落花生hypogaeagydF4y2BalgydF4y2Ba
- 转录组分析gydF4y2Ba
- lncRNAsgydF4y2Ba
- 替代拼接gydF4y2Ba
- 器官特异性表达gydF4y2Ba
- 逆境条件gydF4y2Ba