摘要
背景
冷应激引起基因表达的动态变化,部分原因是由小的非编码rna引起的,因为它们调节蛋白质编码转录物并在表观遗传基因沉默途径中起作用。因此,详细分析属于所有已知sRNA类别的小RNA (sRNAs),如microrna (miRNA)和小干扰RNA (sirna)在低温响应中的转录变化有助于理解与寒冷相关的转录组变化。
结果
我们接受答:芥我们发现了93个低温响应差异表达的mirna,其中只有14个之前被证明是低温响应的。我们对所有差异表达的miRNA进行了miRNA靶预测,GO分析揭示了miRNA靶转录本的过度表达,这些转录本编码蛋白质在转录调控中起作用。我们还发现了大量的差异表达顺式而且反式-nat-siRNAs,以及由长非编码rna衍生的sRNAs。通过将sRNA和mRNA谱分析结果与miRNA靶预测和公开的转录因子信息相结合,我们重建了一个冷特异性的、miRNA和转录因子依赖的基因调控网络。我们通过测试网络在冷驯化下预测目标基因表达的能力,验证了网络中链接的有效性。
结论
在答:芥,衍生的mirna和sRNAs顺式而且反式-NAT基因对和lncrna衍生的sRNAs在冷驯化条件下的基因表达调控中起着重要作用。本研究为加深我们对冷驯化调控网络的认识和理解提供了基础数据库。
背景
植物受到动态和极端气候条件的严重影响。温度变化是植物茁壮生长的最关键因素之一,低温胁迫在全球范围内影响植物的发育并限制其空间分布,影响农业总产量[1].虽然大多数植物物种都进化出了一定程度的耐寒性,但偏离最佳条件会导致基因水平的重组,使植物能够应对环境的波动[2].
植物细胞通过检测细胞膜流动性的降低来感知冷胁迫,从而触发特定的信号级联[3.]以诱导冷反应基因的表达[4].目前,最具特征的通路是c -重复序列结合因子(CBF)依赖的信号通路,其中2C型蛋白磷酸酶(pp2c)响应ABA升高释放OPEN气孔1 (OST1)/ snf1相关蛋白激酶2 (SnRK2.6/SnRK2E) [5]水平,通过磷酸化激活CBF表达的上游转录因子(TF)诱导物(ICE1) [6].ICE1进一步诱导了几种CBF/脱水响应元件结合因子(DREB) tf的表达,这些tf结合到冷响应的冷响应敏感tf /脱水响应元件(CRT/DRE)启动子元件上。天哪)基因,以适应低温条件[7,8].另一种依赖aba的途径控制天哪被称为abre结合因子(ABFs)的bZIP tf与aba响应启动子元件的结合介导了基因表达[9,10].此外,有研究表明DREB/CBF可与abf发生物理相互作用,表达ABA响应基因[11].CRT/DRE和ABRE区域存在于许多冷诱导基因中,表明aba依赖通路和ICE-CBF-COR通路之间存在紧密联系[10].
除了TF介导的转录控制外,表观遗传修饰主要通过染色质重塑改变转录机制染色质的可及性来控制冷胁迫下的基因表达[12,13].除了转录控制,基因调控还涉及转录后和翻译后水平的调控过程[14].基因表达的一个重要的转录后控制是由不能翻译成功能蛋白的非编码rna (ncrna)介导的。ncrna被分为长链非编码rna (lncrna),有助于控制基因表达,包括转录和转录后途径[15]以及sRNAs与反向互补靶RNA结合,从而产生靶RNA裂解或翻译抑制[16]或通过rna定向DNA甲基化(RdDM)等表观遗传机制干扰转录[17].
lncrna长度大于200 nt,具有类似于mrna的5 '帽和3 '多聚腺苷酸[18,19,20.].lncrna通过不同的作用方式发挥其功能,例如,lncrna通过充当诱饵抑制调控蛋白对核酸的可及性[21].另一种机制是由磷酸饥饿诱导的lncRNA (IPS1),这是一个不可分割的竞争对手磷酸2(PHO2)miR399靶向降解的mRNA [22].lncrna还会引起表观遗传改变,如春化过程中发现的组蛋白修饰,在春化过程中,长时间的冷胁迫导致开花位点C的表观遗传沉默(方法控制开花时间[23,24].此处,lncRNA冷诱导的长反义基因内RNA (COOLAIR)与多梳抑制复合体(PRC2)相互作用,随后引起组蛋白甲基化和蛋白沉默方法轨迹。lncrna还协助DNA胞嘧啶残基的从头甲基化,并通过RdDM引起基因的转录沉默[25,26].
小rna (sRNA)大小为21-24 nt,可有效调节mRNA转录水平、翻译并介导表观遗传沉默[27].两种主要的sRNA类别是由单链前体形成部分双链发夹结构的microRNA (mirna)和由双链RNA前体产生的小干扰RNA (siRNAs)。miRNA的生物发生以多步骤的方式发生,从核编码的转录开始米尔通过RNA聚合酶II产生5 '帽和多尾的初级miRNA转录本(pri-miRNA) [28].含有Dicer-like1 (DCL1)及其附属蛋白Hyponastic Leaves 1 (HYL1)和Serrate (SE)的切丁复合体从双链发夹结构中分离出一个miRNA双工,由出口蛋白匆匆(HST)转移到细胞质中。成熟的miRNA被装载到rna诱导沉默复合体中的argonaute蛋白中,通过miRNA的反向互补结合介导靶mrna的裂解[29].
植物miRNAs在包括发育和胁迫适应在内的广泛生物过程中发挥着重要作用[30.].为了揭示受胁迫调控的miRNA库,从受到不同胁迫条件的植物中生成sRNA文库,并通过RNA测序方法进行分析[31,32,33,34].以前的研究答:芥确定了miR171家族的成员会被低温和高温上调[35)目标SCARECROW-LIKE6-III(SCL6-III),SCL6-IV属于tgf的GRAS家族[36,37].MiR408被认为是由寒冷和其他非生物胁迫诱导的。它调节编码植物青蛋白家族蛋白质的转录本(铜还蛋白、plantacyanin和uclacyanin),这些转录本充当蛋白质之间的电子转移穿梭器[38]和称为漆酶的植物酚氧化酶转录物[39]它们在种子发育和环境胁迫过程中氧化黄酮类化合物[40].这些对维持细胞壁功能至关重要,对调节非生物应激反应所必需的生物途径也很重要[41].最近的研究证实了miR394及其靶标卷叶响应性(lcr)以调节叶片的发育[42,43]并以依赖aba的方式参与对寒冷、盐和干旱胁迫的响应[44,45].在答:芥, miR397通过cbf依赖的信号通路和过表达调控冷耐受性MIR397a导致增加CBF转录水平导致诱导冷反应天哪基因(46].
与mirna相反,siRNAs由dsRNA分子生成,并根据其特定的生物发生途径进行子分类。反式-作用siRNAs (ta-siRNAs)是内源性植物特异性小rna,能够作用于反式并有可能抑制不同的mRNA转录。ta-siRNAs的产生是由mirna介导的原代分裂触发的助教转录本以分阶段的方式生成21 nt ta-siRNAs [47,48].ta - sirna已被证明可调节植物发育[49].最近的研究表明,它们在环境压力适应中的作用,例如,14个缺氧响应的ta-siRNAs已在答:芥都是从TAS1a, b, c,TAS2而且TAS3a前体(50].a的表达式TAS1-衍生的ta-siRNA及其靶转录本热诱导的TAS1靶标(HTT4)均受到温度变化的调节[51].此外,的生成TAS4-衍生的ta-siRNAs被证明在磷酸盐缺乏下由miR828触发[52].
另一类siRNAs是天然反义转录本衍生的短干扰RNA (nat-siRNAs),由RNA聚合酶II衍生的反义转录本的重叠区域产生[53].根据重叠转录本的基因组位置,NATs可以分为两种类型。两个转录本要么在同一基因组区域内的相反DNA链上编码,以产生重叠的转录本(独联体-NATs)或两种转录本均来自不同的基因组区域(反式-NATs),但能够相互配对。一种高盐度响应的nat-siRNA首次在答:芥组成型表达基因转录本在哪里delta-pyrroline-5-carboxylate脱氢酶(P5CDH)和盐诱导的基因转录类似于胚根诱导细胞死亡(SRO5)编码在重叠基因组区域的相反链上形成dsRNA, DCL2处理来自dsRNA区域的不同的24 nt nat-siRNA。生成的nat-siRNA裂解P5CDH转录并抑制脯氨酸降解,从而诱导耐盐性[54].除了产生的nat-siRNAs独联体nats,反式-当来自非重叠基因的反义介导的转录本配对发生时,可以产生nats [55].这些dsRNAs的形成发生在不同的反式-组合,即长链非编码rna、蛋白质编码转录本、同源假基因和转座元件(TE)之间的组合[56,57].例如,的类反式-由假基因产生的nats可调节其同源蛋白编码转录本水平[58].
中观察到大量te衍生的siRNAsDNA甲基化1降低(DDM1)突变体的答:芥并被称为表观遗传激活的sirna (ea- sinas)。这些siRNAs是由转座子编码的转录本产生的,这些转录本以mirna依赖的方式被切割,并转化为dsRNAs, dsRNAs再被DCL4加工成21 nt ea-siRNAs。研究表明,这些ea- sirna主要是通过靶向TE的内在转录物来沉默TE所必需的,而这些sirna的一个子集也靶向蛋白质编码mrna以降低其表达水平[59].此外,类似于米尔一些te衍生的转录本可以形成茎环结构,从中可以加工siRNAs [60].TE也编码lncrna,越来越多的证据表明,环境因素导致染色质组织和lncrna表达的改变,这些改变可能在适应改变的环境条件方面起作用,甚至可以遗传。一项研究答:芥有报道称te衍生的TE-lincRNA1195参与了ABA反应,并有助于非生物胁迫适应[61].
在我们的研究中,我们使用RNA测序来揭示冷响应非编码RNA库答:芥并研究它们在各种靶rna调控中的作用。我们测序了来自答:芥将植物置于冷驯化条件下3小时、6小时和2天,并分析差异表达的sRNAs与其蛋白质编码目标之间的假定相关性。为了进一步深入了解mirna调控的直接靶点和由tf调控的间接靶点之间的冷响应互连,我们使用mirna -靶点的信息和公开的tf相关数据库生成了一个基因调控网络(GRN),生成的网络可以识别mirna在冷驯化下的连通性和调控影响.
结果
低温驯化过程中sRNAs表达的改变答:芥
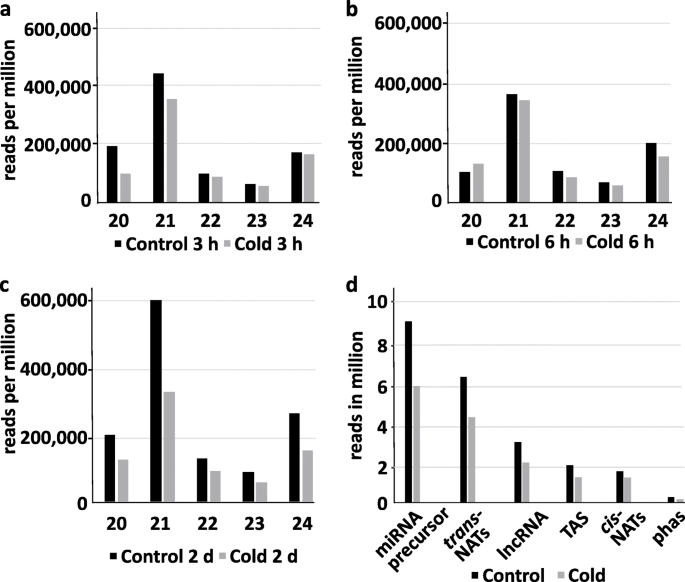
为了分析我们所研究的sRNA库中的冷响应变化答:芥幼苗以4°C冷处理3 h、6 h和2 d的时间点。以前有关冷驯化的研究发现,当温度从正常温度转移到4℃时,光合机制会迅速受到抑制[62].此外,研究还发现,丰富的冷反应基因在早期时间点(3 h和6 h)和后期时间点(48 h)表达差异很大[33,34,62].因此,为了研究可能调控这些冷改变基因的sRNAs,我们选择3 h、6 h和2 d时间点进行RNA测序分析。处理过的和对照样品的RNA被用于进行转录组分析,每个文库至少有700万次读取。日志含义sRNA读取信息映射到答:芥在所有样本中,平均约10%的reads映射到miRNA位点,10%映射到反式-2%独联体-nat-siRNA位点,4%的reads映射到lncRNAs, 3%映射到ta-siRNA产生区域,0.3%映射到pha- sirna(附加文件)1:表S2)。只有约1%的总reads映射到编码最丰富的RNA(如核糖体RNA、snoRNA、tRNA和snRNA)的位点,这表明sRNA文库质量良好。其余部分的reads大多映射到其他RNA类,如TE和已知参与表观遗传途径的重复相关区域。
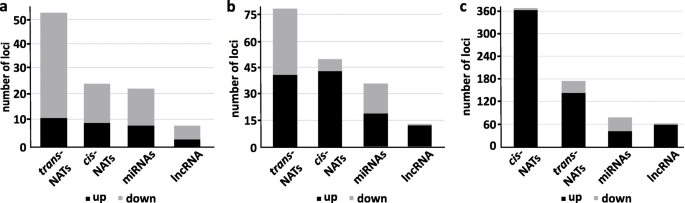
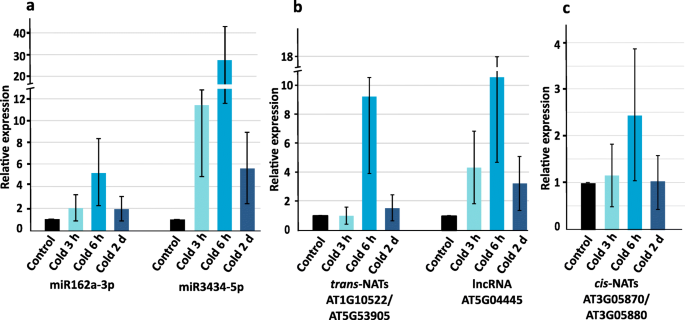
从21到24 nt的sRNAs的大小分布显示了两个不同的峰值,在21 nt表明miRNAs、nat-siRNAs和ta-siRNA的富集,在24 nt对应的是来自重复/基因间rna、倒置重复序列和TE的sRNAs(图2)。1a, b, c,附加文件1:表S3)。我们观察到,与对照组相比,冷驯化后sRNAs的总体减少。sRNA reads的分布映射到不同的sRNA产生位点,包括miRNAs、nat-siRNAs、ta-siRNAs、phasiRNAs和lncRNAs产生的sRNAs反式-nat-siRNAs是在我们的数据集中检测到的两种主要sRNA类别(图。1d)在经过冷处理的样品和相应的未处理对照之间鉴定差异表达(DE) sRNAs(折叠变化≥2 &≤- 2,Benjamini-Hochberg校正)p-value≤0.05),根据归一化reads数计算成熟miRNAs和siRNAs的相对表达量。在分析的时间过程中,冷胁迫主要影响从反式- - -独联体- nts -对,然后是由lncRNA衍生的mirna和srna(图2)。2a, b, c)。此外,我们观察到所有sRNA类别的上调和下调sRNA的数量在时间过程中不断增加,在冷处理2 d后达到最高(图。2c).为了评估sRNA测序结果的可靠性,我们对所有分析sRNA类别的sRNA进行了茎环qrt - pcr,以验证和确认它们在冷处理时间过程中的表达变化(图2)。3.).miR162a-3p miR3434-5p,独联体产生的-nat-siRNAAT3G05870-AT3G05880成绩单,反式生成的-nat-siRNAAT1G10522-AT5G53905在冷处理过程中发现了lncRNA AT5G04445转录本和来源于lncRNA AT5G04445的sRNA,证实了我们的sRNA测序结果。
冷驯化应答mirna的表达谱分析
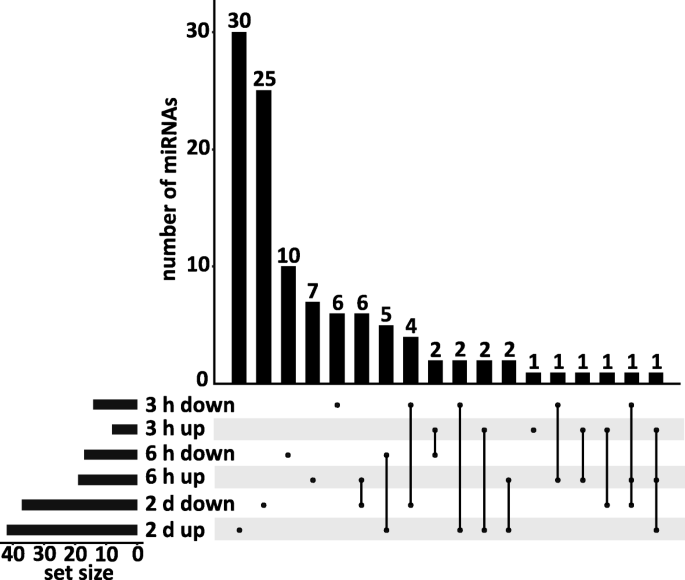
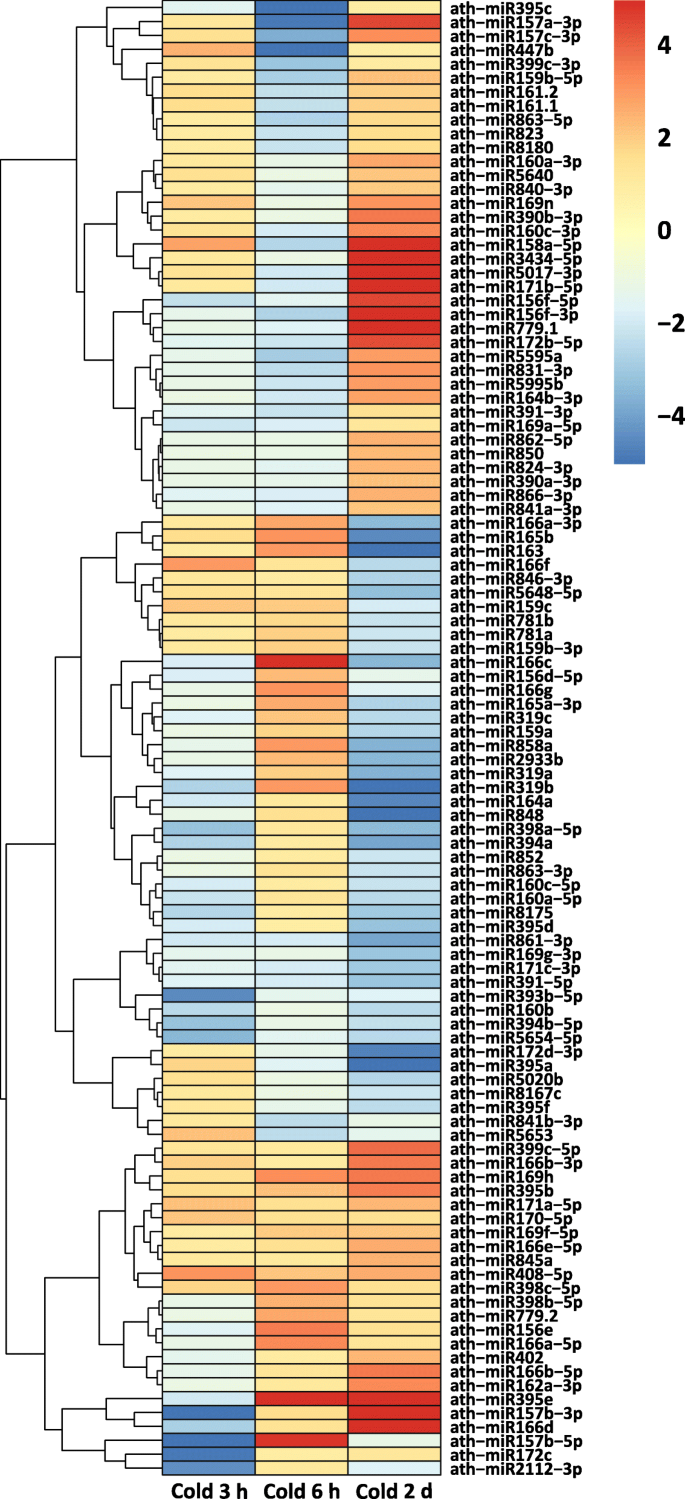
sRNA测序方法允许区分单个mirna,即使是单个核苷酸差异。在精确的读取映射后,分析序列读取以识别差异调控的miRNAs (FC≥2 &≤- 2,Benjamini-Hochberg校正p-value≤0.052,附加文件2:表S4)。我们在所有样本中观察到一个总体趋势,大约10%的检测到的mirna具有非常高的归一化读取计数(每个样本> 1000个读取),约50%的mirna表现为中等表达(< 1000和> 20个归一化读取),11%的mirna表现为减少的读计数(< 20和> 5个归一化读取),27%的mirna表现为非常低的表达(< 5个归一化读取)(附加文件)2:表S5)。在低温处理的反应中,我们观察到22个mirna(8个上升和14个下降)在3小时后为DE, 36个成熟的DE mirna(19个上升和17个下降)在6小时后,79个DE成熟的mirna(42个上升和37个下降)在2天后。我们发现mirna在特定时间点表现出差异表达,以及在两个或所有三个时间点表现出差异表达。在整个冷处理过程中发现了2个DE miRNAs,在6 h和2 d检测到13个DE miRNAs,在3 h和2 d检测到8个DE miRNAs,在3 h和6 h时间点检测到4个DE miRNAs。我们还观察到7、17和55个DE mirna在3 h、6 h和2 d时间点被特异性调控(图2)。4).我们在冷处理过程中检测到DE个体miRNA数量的增加,这表明miRNA水平的改变似乎是冷驯化过程中的一个重要步骤。
近年来,有22个miRNA家族被鉴定为保守的答:芥,水稻和白杨[63,64,65]其中一些被证明在非生物应激适应中具有重要作用,因为它们主要调节编码tf或促进应激耐受性的酶的靶标[66,67,68].在这22个miRNA家族中,我们检测到16个家族的个体成员在3小时、6小时和2天分别对应15、20和43个DE成熟miRNA表达差异(图2)。5,附加文件2:表S6)。总的来说,我们发现107个非冗余的成熟miRNA在整个冷处理过程中表达差异,其中36个成熟miRNA属于9个保守miRNA家族,已知在其他植物物种中受冷调控(附加文件)2:表S6) [35,69,70].在107个mirna中,有14个是已知的冷反应答:芥我们的研究表明,与其他冷应激相关研究相比,这些mirna的诱导或抑制模式具有相似性[35,71,72].其余93个DE成熟miRNA属于55个miRNA家族,未见冷调节的报道答:芥(附加文件2:表S6)。我们发现了几种表达模式不同的mirna,即在不同时间点上调和下调。如miR156f-5p和miR157b-3p在3 h下调,在2 d上调,miR166f在3 h上调,在2 d下调,miR447b和miR5653在3 h上调,但在6 h下调,miR157b-5p在3 h下调,在6 h上调。同样,12种mirna在6 h和2 d表现出不一致的调控,而我们观察到miR408-5p、miR395e、miR159c、miR169h在至少两个时间点上一致上调,miR160a-5p、miR160b、miR398a-5p、miR8175、miR319b下调。这表明低温处理不同时间点miRNA的调控模式不同,成熟miRNA的稳态水平取决于植物在胁迫条件下的生理需求。
差异表达的miRNA靶标
由于miRNA和mRNA/lncRNA来自相同的RNA样本,我们能够将miRNA表达的变化与其同源靶标的变化进行比较。为了识别在冷处理过程中发现差异表达的miRNA的靶点,我们使用psRNAtarget预测工具,其严格的期望截断值为2.5,并允许miRNA以最大能量接近其mRNA靶点,以25 [73].应用这些严格的参数,预测工具揭示了107个DE mirna中的93个假定目标。93个非冗余DE mirna的目标预测确定了338个mrna和14个非编码rna作为假定的目标(附加文件)3.:表S7, S8)。在3小时内,18个DE mirna(5个上调,13个下调)可以靶向96个非冗余mrna和3个非编码转录本。33个DE mirna在6小时(18个上调和15个下调)可以靶向173个非冗余mrna和3个非编码RNA靶点,69个DE mirna在2天后(34个上调和35个下调)能够靶向267个非冗余mrna和12个非编码RNA靶点(附加文件)3.:表S7, S8)。为了分析这些靶点的调控如何与mirna的表达相关,我们使用从相同RNA池中生成的mRNA和lncRNA转录组测序数据作为3 h、6 h和2 d冷处理及其各自未处理对照的sRNA数据集(附加文件)4:表S9, S10)。我们使用mRNA/lncRNA转录组数据来检测93种差异调控mirna靶向的所有338个转录本的表达水平,以便将目标转录本的表达与其同源mirna的表达相关联(附加文件)3.:表S7)。在常见的情况下,我们观察到一个转录本可以被一个miRNA家族的各种亚型靶向,但在少数情况下,目标转录本也可以被序列不相关的不同miRNA切割。总的来说,我们将所有单独的DE miRNAs及其同源蛋白编码靶标(mRNA)视为miRNA:mRNA对,并在低温处理3小时、6小时和2天的时间点分别鉴定出111对、246对和376对3.:表S7)。对于每个时间点,我们根据其表达与其同源miRNA表达的相关性将miRNA:mRNA目标对分为不同的亚组。这些miRNA:mRNA靶对亚群在mRNA和miRNA表达呈反相关时被归类为反相关,当miRNA和靶蛋白表达上调或下调,miRNA被调控,靶蛋白保持不变或未检测到时,miRNA:mRNA靶对亚群的表达趋势相同(表2)1).我们分别在3小时、6小时和2天观察到2个、12个和27个反相关miRNA:mRNA靶对,共有39个非冗余反相关miRNA:mRNA靶对,表明这些miRNA在冷处理后控制转录组中的作用(附加文件)3.:表S7)。除了mRNA靶标外,靶标预测工具还鉴定出14个DE mirna的假定非编码RNA靶标,但ncRNA靶标转录物的表达水平低于5 reads或无差异表达。
基于Araport (Version 11;https://araport.org/)注释,我们观察到来自所有四个亚组的54个DE miRNAs靶点在所有时间点一致存在(附加文件5:表S11)。主要编码含MYB结构域蛋白、核因子Y亚基基因、热休克结构域蛋白(HsFs)、TCP结构域蛋白和Squamosa启动子结合(SPLs)蛋白。我们还检查了每个时间点特定的miRNA靶点的功能。具体而言,在6小时时间点,我们发现了几种已知对各种细胞器中RNA成熟很重要的PPR蛋白,编码植物信号通路和细胞器导入所需基因的TPR蛋白,以及编码膜多重抗菌挤压的基因[22在外源性化合物运输中起作用的外排蛋白。在第2天的时间点,我们发现了大量的转录本编码参与转录调节和蛋白质磷酸化的因子,这些因子控制细胞内信号通路以应对应激。综上所述,我们发现编码转录因子的基因、与转录调控相关的蛋白质以及参与RNA加工和翻译控制的蛋白质的显著过度表达。
我们发现39个mirna和它们的假定靶标呈负相关,例如,在冷处理3小时后,我们注意到miR172c (FC =−4.86)的强烈下调,其预测靶标target of EARLY ACTIVATION TAGGED (EAT, FC = 2.18)的上调,已知在低温处理中降低A. thaliana冰突变体(33].此外,吃在4°C时,根和叶的表达水平也有所增加答:芥[74].经过6小时冷处理后,我们观察到miR395c (FC =−19.27)下调,其编码镁螯合酶亚单位h的靶转录本随之上调GUN5该基因(FC = 2.18)是质体向细胞核信号转导的重要组成部分。另一个miRNA miR5595a表达水平降低(FC =−2.88),而其编码甲基酯酶9的靶基因表达上调(FC = 3.58),被认为是植物核心环境胁迫响应基因(PCESR) [75].此外,在冷处理2 d后,我们观察到miR319的三种亚型被下调,其中一个编码TCP2 TF的靶转录本(FC = 2.56)上调。之前的一项研究表明TCP2转换后的成绩单答:芥在100 μE光照条件下,植株适应低温条件,而在黑暗条件下不适应,推测低温条件下来自叶绿体的光依赖信号对增加叶绿体生长有重要作用TCP2对光合作用相关基因的控制可能很重要的水平[76,77].冷处理2 d后,我们还检测到miR159亚型下调(FC =−2.53),导致其一个靶转录物水平升高转位酶内膜亚基44 (TIM44) -相关编码线粒体内膜转位酶复合体亚基(FC = 2.80)。
预测miRNA靶点的基因本体分析
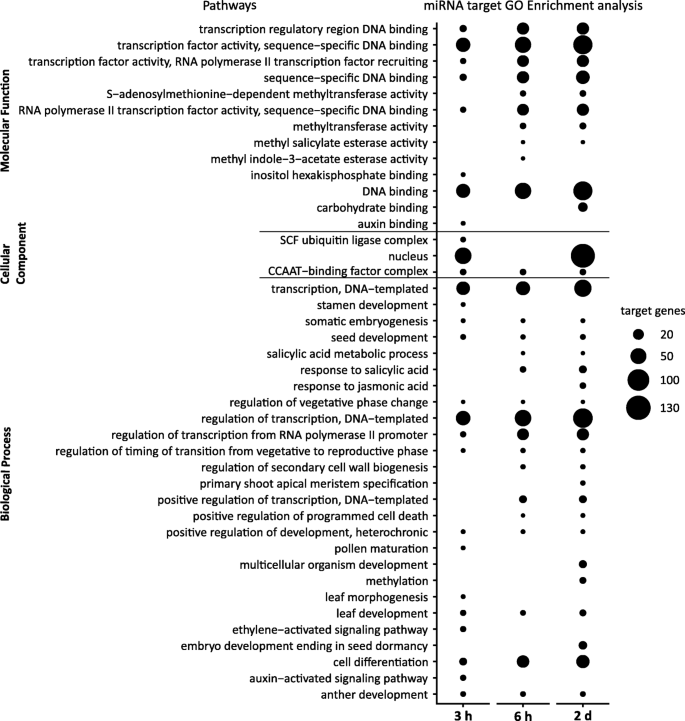
为了获得DE冷响应mirna及其靶标的可能作用,我们使用David生物信息学工具对所有假定靶标进行了基因本体(GO)分析[78].基于三类;生物过程,细胞成分和分子功能,我们观察到所有三个时间点的GO项都有丰富的Benjamini-Hochberg校正p-从Fisher检验得到的值(图;6,附加文件6:表S12)。在3小时时间点,重要的生物过程包括转录的调节(47)、转录(41)、细胞分化(12)、乙烯激活信号通路(7)和生长素激活信号通路(7),这表明miRNAs对编码主要作用于信号和基因转录的蛋白质的基因的早期反应有重要影响。关于细胞成分,我们确定了与细胞核相关的靶数最多(63个),这与之前tf的过度代表很好地相关。此外,在分子功能类别中,TF活性、序列特异性DNA结合(46)、DNA结合(44)和生长素结合功能最显著,也表明编码基因转录的调控蛋白和参与基因转录的因子的转录本的过度代表。在6 h时间点,基因数量最多的显著生物学过程包括转录调控(62个靶基因)、对水杨酸的反应(8)、次生细胞壁生物发生的调控(5)和程序性细胞死亡的正向调控。我们还发现s -腺苷甲硫氨酸依赖的甲基转移酶活性(7)在分子功能类别中显著富集。与3小时时间点相似,我们在6小时时间点观察到转录相关基因的富集。与此同时,甲基转移酶活性相关基因的过度表达表明与非生物胁迫相关的表观遗传修饰,可能在次生细胞壁生物发生中起作用的基因可能导致胁迫条件下细胞壁的增强和孔径的减小。在第2天时间点,重要的生物过程包括转录调控(89)、胚胎发育结束于种子休眠(15)、多细胞生物发育(13)、甲基化(9)和对茉莉酸的反应(8)。在所有三个时间点,我们都观察到编码tf的基因的富集,这表明这些是冷驯化过程中转录重编程的一组基因的关键调节因子。关于细胞成分类别,我们观察到与细胞核相关的靶基因数量最多(136个靶基因),这与前面概述的类别一致,并强调了应对冷驯化的大量转录调节过程(图2)。6).
基因调控网络(GRN)的构建
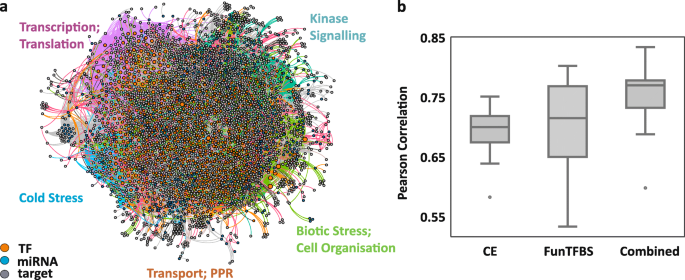
为了了解主要基因调控类可能的相互作用和贡献,我们重建了miRNA和TF调控网络(附加文件)7,数据S1)。该网络包括直接miRNA介导的靶控,调控转录产物编码的转录因子调控下游靶(间接靶)的miRNA,以及不受miRNA控制但调控miRNA调控下游靶(直接靶)的转录因子。为了构建最终的网络,我们考虑了生成的miRNA和mRNA表达数据,并分析了所有使用psRNATarget工具预测的miRNA靶点,以及TF结合位点(TFBS)和下游靶点的公开信息。我们从拟南芥转录调控图谱中纳入了实验验证的调控连接[79和Agris [80].此外,我们从PlantRegMap中纳入了高置信度的TF目标相互作用[81],只考虑结合地点保育标准不同的tf。第一个标准包括结合位点位于不同植物物种保守元素(CE)内的tf及其靶标,第二个标准包括TFBS守恒扫描时发现结合位点在不同植物物种中保守的tf及其靶标(FunTFBS)。
网络中连接的有效性是通过预测miRNA和TF控制的靶mRNA表达水平来测试的,该表达水平基于给定时间点的miRNA或TF表达水平。在这里,预测力被用作网络中调控环节可靠性的指标,并通过预测的mRNA表达水平与测量的mRNA表达水平之间的Pearson相关性计算(图2)。7b).我们测试了三种不同网络版本的预测能力,以确保模型中的信息最大。在这里,合并版本平均能够解释77%的靶基因表达变化(0.77 Person相关系数),并被考虑进行进一步研究。
(一个)通过推断miRNA和tf介导的基因表达控制生成冷响应基因调控网络。TF和靶标连接从公开的数据库中获得,并与psRNATarget预测的miRNA靶标相结合。顶点颜色表示各自的调节活动,边缘颜色标记与计算模块的关联。最大的模块被标记为它们最突出的功能组,这些功能组是使用本体丰富来标识的。(b)网络的预测能力。考虑到不同来源的调控连接,不同网络版本的预测和测量表达水平的Pearson相关性。所有版本都包含经过实验验证的TFBS。此外,CE包括仅在保守区域出现的TFBS预测,而FunTFBS仅在结合位点功能保守时包含基于TF的预测连接。Combined合并所有资源。与GRN相关的数据可以通过GEPHI Software(附加文件)访问7:数据S1)
该网络模型包含350个miRNAs,分为166个家族,由38个家族的657个tf组成,这些tf激活或抑制2420个下游靶基因。总的来说,有36,523种调控关系,其中3846种是基于miRNA的,而其余32,677种是基于tf的。7a,附加文件7:数据S1,附加文件8:图S1)。
在对网络重构进行验证后,对网络的模块化进行了分析。模块是与网络中的其他节点相比彼此紧密连接的节点集群。在生物系统中,一个模块的节点往往相互调控,在功能上密切相关。因此,模块可以解释为细胞的功能单元[82].通过使用社区检测方法[83],我们找到了17个模块。使用GO和MapMan本体的功能富集显示,信号、运输、冷和生物应激成分、RNA和蛋白质合成以及细胞组织在五个主要网络模块中被过度代表。
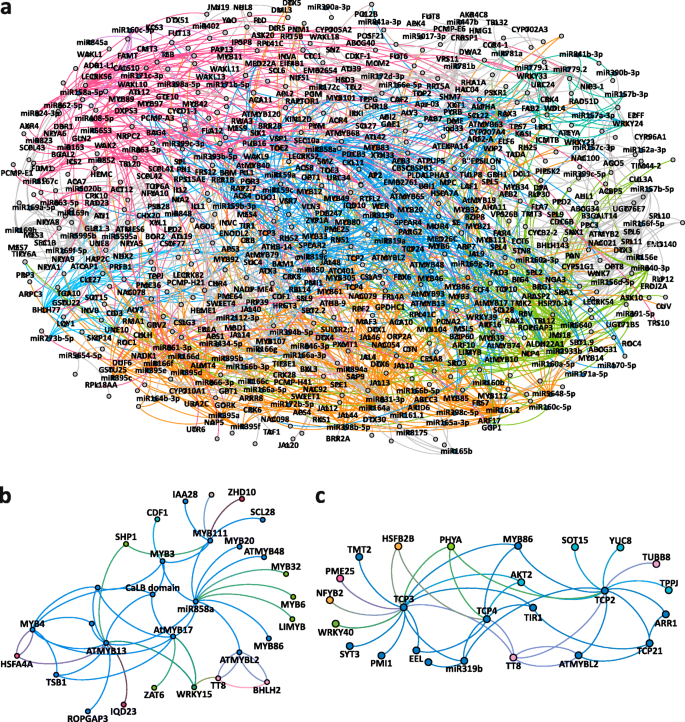
冷响应子网络(图。8a,附加文件7: Data S2,附加文件9:图S2)由差异表达miRNA的靶标和编码tf的靶标及其下游靶标从GRN中提取。所描述的目标在至少一个时间点上差异表示,所提取的网络由830个节点和1332条边组成。我们观察到103个成熟的mirna和58个tf参与了669个直接和间接靶标的调控。功能富集揭示了与冷驯化、转录/翻译、生物应激/细胞组织、信号/蛋白质降解和细胞壁/木质素合成相关的基因的主要调控。
提取的冷响应GRN包括DE miRNAs的直接和间接靶标.(一)在任何一个分析时间点上差异表达的miRNAs和靶标网络(FC≥2&≤- 2,Benjamini-Hochberg校正p值≤0.05)。与冷驯化、激酶信号、转录相关的功能模块;转换和传输分别用蓝色、深绿色、粉红色和橙色表示。(蓝色节点= miRNAs,橙色节点= tf,灰色节点=靶标)(b)miR858的子网。(c)miR319子网.在(b)而且(c)miRNAs的直接和间接靶标在至少一个分析时间点上有差异表达(FC≥2&≤- 2,Benjamini-Hochberg校正p值≤0.05)。弯曲的边缘表示一个调节器和它的目标的调节连接。节点颜色描述了基于GO富集分析的推断函数。绿色:生物压力,细胞组织;蓝色:冷应激;粉色:转录、翻译;橙色:运输,PPR;深蓝色:细胞壁,木质素合成;红色:信号,蛋白质降解。与GRN相关的数据可以通过GEPHI Software(附加文件)访问7:数据S2)
我们选择了两个子网,miR319在所有三个时间点都是DE, miR858在6 h和2 d都是DE。从整个网络中提取这两个mirna的miRNA-TF子网(图2)。8b, c)和网络中所描述的目标在至少一个分析时间点是DE(附加文件10:图S3和附加文件11:图S4)。miR858子网由30个节点和51条边组成。在其靶标中,miR858控制蛋白的表达色氨酸合成酶(TSB1, AT5G54810)催化色氨酸的合成,色氨酸是生长素吲哚-3-乙酸的前体[84].MiR858还控制编码TF MYB111 (AT5G49330)的转录本,该转录本通过调节类黄酮生物合成来调节盐胁迫反应[85]和热休克因素HSFA4A(AT4G18880)参与热应激反应。我们发现了25个节点和43条边与miR319相连,miR319介导转录本的调节,如透明测试8(TT8, AT4G09820)编码的一个TF,通过控制花青素的生物合成dihydroflavonol 4-reductase[86].MiR319还调节与耐热相关的热休克因子HSFB-2b (AT4G11660)的mRNA和可能的果胶酯酶/果胶酯酶抑制剂25 (PME25, AT3G10720)的转录本编码,这些转录本可以促进冷应激下细胞壁的修饰。
差异表达的sRNAs来源于不同种类的RNA
我们利用我们的sRNA测序数据不仅分析了miRNA对低温的调控,而且还鉴定了来自其他RNA类别的sRNA,这些RNA类别可以为它们在冷驯化中的作用提供联系。我们将sRNA reads映射到公开的lncrna参考数据库,反式-而且顺式NATs对,助教而且pha[57,87,88,89]并且我们能够将大量的DE srna与这些RNA类别联系起来。
来源于非重叠lncRNAs的sRNAs
在这里,我们将非重叠的lncrna定义为大小大于200 nt的单链RNA转录物,且不与蛋白质编码转录物或其他非编码转录物重叠。在我们的sRNA数据中,我们观察到15个非冗余非重叠的lncRNA位点产生DE sRNA,其中13个sRNA产量上调,而其余两个sRNA在低温响应下下调(附加文件)12:表S13)。然而,即使这些lncRNAs生成DE sRNAs,整个分析样本的lncRNAs转录水平保持不变。我们发现在冷处理3小时有1个lncRNA, 6小时有1个lncRNA, 2天时间点有7个lncRNA产生DE srna。此外,我们发现两种lncrna在3小时和6小时内产生sRNA的差异,其中AT5G07745减少了sRNA的产生,而另一种(AT5G04445)在两个时间点都上调了sRNA的产生。在6小时和2天,我们检测到四种常见的lncRNAs产生表达水平升高的sRNAs。lncRNA AT5G05455是唯一一个在第2天时间点产生数量减少的srna,而其他的都是上调的。单链转录本能够产生折叠结构,形成dsRNA, dsRNA可以加工成小rna,但我们观察到这些lncRNA转录本的正反意义链产生srna。由于这些lncRNA不与任何其他转录本重叠,并且在其他基因组位点上也没有任何配对伙伴,这可能表明RNA依赖的RNA聚合酶以独立于引物的方式参与了这些lncRNA的dsRNA的形成,这些dsRNA随后被转化为sRNAs [9,90].
来源于nat的sRNAs
NATs是非编码(nc)或蛋白编码(pc)基因的一对转录本,由于序列互补而重叠并形成dsrna。nc-nc、nc-pc和pc-pc转录本之间的配对是可能的,所得到的配对转录本可作为dcl介导的转化为sRNAs的靶标。我们找到了大部分顺式而且反式-从pc:pc或pc:nc生成的nat对。在pc:nc的情况下,nc配对伙伴主要代表来自TE的pre-tRNAs或转录本,它们也有单独产生sRNAs的能力[91,92,93].已知pre-tRNA和te衍生的srna具有通过序列互补调节其他转录本的能力,这可能表明它们在调节冷驯化相关网络中的贡献[92,94].我们的数据集显示,在寒冷反应中产生高水平sRNAs的转录本对可能具有不同的表达模式。它们可以表现为反相关(一个转录本上调而另一个转录本下调)、相同的表达倾向(两个转录本上调或下调)或不相关(一个转录本上调而另一个保持不变)。在胁迫条件下,胁迫诱导基因和组成型表达基因对的互补转录反向测序,产生24 nt和21 nt siRNAs。产生的siRNAs有能力切割构成性表达的转录本,导致其下调,以促进应激适应。该机制代表了NATs的经典表达模式[54].其特征是诱导的nat-siRNAs的差异表达以及正义和反义转录本的反相关表达模式。我们观察到大量受到调控的sRNAs,但其转录水平保持不变。第二个最丰富的情况是一个转录的上调或下调,而另一个转录保持不变(表2).
大部分的反式-NATs基因对在冷处理2 d后产生大量sRNAs,从mRNA数据推测基因转录水平下降或没有变化。这表明两种转录本可能配对,它们被进一步加工成nat-siRNA,而在冷驯化中,这些nat-siRNA的更高产量可能需要将至少一种转录本保持在稳定的水平。
独联体-nat-siRNAs
我们找到了5个,20个和100个独联体- nats位点(104个非冗余对)分别在3小时,6小时和2天,从两个重叠的转录本中产生DE sRNAs,其中一个转录本上调或下调,而另一个转录本保持不变(表2)(附加文件12:表S14)。此外,我们还检测到24、34和278独联体-NATs(308对非冗余对)在3小时,6小时和2天的时间点产生DE sRNAs,但同源重叠转录本保持不变或无法检测到。通常,我们观察到大多数对产生独联体-nat-siRNA为pc:pc转录对。我们发现了一对在6小时和9对在2天的NATs,类似于nat-siRNAs反义转录调控的经典机制(表2)3.) [54].我们检测到一个基因对,该基因对增加了nat- sirna的产量,包括一个冷诱导的转录本,编码ras相关的gtp结合核蛋白(RAN2AT5G20020)及其配对转录本编码植物都铎样rna结合蛋白(AT5G20030)的下调。到目前为止,缺乏对植物都铎样RNA结合蛋白的功能研究,但已知RAN2是蛋白质核易位和RNA输出所必需的[95].盐胁迫应答基因编码一种油酸蛋白b3样蛋白(AT1G13930)的另一个转录本[96]这是由aba诱导的[97]也被低温诱导,其转录本可与T-box TF (AT1G13940)转录本配对诱导产生独联体nat-siRNAs。除了上述差异调节siRNA产生的转录本表达模式外,我们发现sRNA产生位点表现出相同的转录本表达趋势,表现为两个配对转录本的上调(16个非冗余对;分别在3 h、6 h和2 d时添加1、1和15),从而诱导sRNA的生物生成。在这一类中,我们观察到pc:pc和pc:nc转录本对的富集。我们的结果中突出的例子包括应激诱导的pc:pc转录本RARE-COLD-INDUCIBLE 2(AT3G05880)和后期促进复合物/环体(AT3G05870)导致增加顺式nat-siRNA生产。我们还发现了一个冷诱导的pc:nc转录对编码一个叶绿体β淀粉酶和一个lncRNA,并上调独联体-nat-siRNAs的生成在所有三个时间点一致。-淀粉酶促进淀粉降解为糖,糖可作为渗透剂在低温胁迫条件下维持渗透平衡[98].
反式-nat-siRNAs
我们发现了38个非冗余的反式-生成DE的nat对(分别在3 h、6 h和2 d时为5、14和26)反式-nat-来自每个转录本的sirnas。这38对基因的转录水平显示,其中一对转录物要么上调(5对),要么保持不变(33对)。在这38个中,我们发现了4个反式-NATs基因对产生DE反式-nat-siRNA在3 h后(基因转录物均不变)和6 h后(一个转录物上调,另一个转录物不变)均为常见。我们观察到41,39和88(95非冗余)反式-NATs基因对分别在3 h、6 h和2 d产生DE反式-nat-来自两个转录本重叠区域的sirnas具有不变或未检测到的转录本水平(表2).我们观察到2、5和23对由重叠的pc:pc转录本组成的trans-NAT对产生DE反式-nat-siRNAs。我们发现一个pc:pc NAT对在3小时产生减少的NAT - sirnas,但在6小时和2天时间点产生增加的NAT - sirnas。两种转录本都编码ZED相关激酶(ZRK 1, AT3G57710和ZRK 7, AT3G57770),已知这些激酶在高温下被诱导,并在没有植物病原体的情况下抑制免疫反应[99].在我们的数据中,这两个基因的转录水平没有变化,但是生成反式-nat-来自两个重叠转录本的sirnas可能对将转录本保持在稳定水平很重要。经过2 d的冷处理,我们找到了pc:pc反式导致nat对增加反式-nat-siRNA由编码Plastid Redox Insensitive (PRIN2, AT1G10522)和prolamin like protein (AT5G53905)的转录本产生,但这两个基因的转录本水平保持不变。众所周知,PRIN2是一种参与氧化还原介导的逆行信号转导的质体蛋白,是光激活的pep依赖转录所必需的。另一个类似的例子包括一个ncRNA (AT1G70185)和一个假设蛋白质(AT5G53740)的转录本,它们产生大量的氨基酸反式-nat-siRNAs,但其转录水平没有变化。除了pc:pc对外,我们还检测到能够与不同的pre-tRNA配对的pc转录本。其中7%的转录本与36个pre-tRNA转录本配对产生DE反式-nat-siRNAs在3小时,10份转录本在6小时配对46份pre-tRNAs, 15份冷藏2天后配对82份pre-tRNAs。大多数的反式-NAT基因对包括一个nc转录配对,编码一个来自TE的前trna或RNA。我们发现大量pc:nc对产生的DE sRNAs(分别在3 h、6 h和2 d时分别为41、37和65个位点)转录本水平未检测到或不变。这些pc:nc NATs对有可能是从两个完全或部分重叠转录本的双链区域产生sRNA,可称为反式-nat-siRNAs或这些可以来源于两个部分重叠的tRNA或TE转录本的单链区域(附加文件)12:表S15)。特别地,我们观察到1、8和17个pc:nc反式-NATs分别在3 h, 6 h和2 d从TE转录物中产生DE sRNAs。te衍生siRNA的一个广为人知的例子是siRNA854,它显示出与其目标的3 ' UTR部分互补,该3 ' UTR编码一种参与胁迫颗粒形成的rna结合蛋白,称为UBP1b转录本[One hundred.].我们还检测到te衍生的sRNAs能够靶向mRNA转录本以促进冷处理适应。有关反式-nat-siRNA产生位点6 h后发现13对,2 d后发现34对反式-nat- sirnas,每对中的一个转录本要么上调要么下调,配对伙伴保持不变。时间剖面显示DE数量最高反式-nat-siRNAs在2天后鉴定反式-nat-siRNA介导的基因表达调控似乎对冷驯化的晚期反应最为重要。
Pha-siRNA
在6小时的时间点,我们鉴定出来自线粒体PPR蛋白(AT1G63070)转录本编码的上调的sRNAs,这已经被证明可以产生pha-siRNAs [101](附加文件12:表S16)。尽管pha- sirna的丰度不断增加,但我们无法在mRNA数据中检测到相应的PPR转录。最多的sRNAs大小为21 nt,其次是由此产生的22 nt sRNAsPPR成绩单。已知21 nt pha- sirna被加载到argonaute和rna诱导的沉默复合体中,以介导mrna靶的切割。我们使用psRNATarget应用严格的参数对21 nt pha-siRNA进行了靶点预测,并鉴定了编码其他PPR和TPR蛋白、光系统II亚基QA (AT4G21280)、RNA处理因子2 (AT1G62670)和HVA22同源物a (AT1G74520)的假定靶点转录本。RNA处理因子2也属于一类PPR蛋白,可促进线粒体中的RNA处理[102].光系统II亚基QA是电子传递链的一个组成部分,具有未知功能的HVA22同源物a蛋白先前被证明是ABA和应激诱导蛋白[103].与观察到的pha-siRNA上调一致,我们发现编码PPR蛋白的一个假定靶标(AT1G18485)的转录水平显著下调。
讨论
我们的研究旨在深入了解不同类型sRNA的冷响应调控,以及它们对sRNA生成基础转录本的控制或sRNA靶向转录本控制的影响。我们将sRNA测序与mRNA和lncRNA测序结合起来,将mRNA/lncRNA稳态水平的变化与sRNA表达的变化联系起来。我们观察到经典的冷应激相关标记基因在mRNA测序数据中被上调,在之前的研究中发现这些基因表达差异(Lee et al. 2005)(附加文件)13:表S17)。在低温处理的时间过程中,我们观察到从miRNAs等RNA类别产生的sRNAs总体减少,反式- - -独联体- nat -对和lncrna。为了排除这些变化不是由参与sRNA生物发生的主要成分水平的改变引起的,我们分析了编码sRNA生物发生相关蛋白质的转录本水平,如Hua增强子1 (HEN1)、RNA依赖RNA聚合酶(ATRDR1-6)、DCL1-4、HST1、HYL1、Serrate和基因沉默抑制3 (SGS3)。它们的水平在冷处理过程中未受影响,我们推测sRNA产量的减少可能是由于sRNA前体转录本在冷驯化过程中的转录减少。
分析mirna及其推测的靶标
我们分析了DE mirna,因为它们是基因表达的强大调节因子,参与了几乎所有细胞通路的控制[104].我们在治疗过程中发现了107个DE mirna,并将我们的结果与之前报道的in中的冷响应mirna进行了比较答:芥[32,35,71].Baev et al.(2014)在4°C下处理植物24小时,对莲座丛叶片的RNA进行测序,并检测到44个DE mirna。我们发现了7个表达模式相同的mirna重叠,其中大多数是冷处理2 d后的DE。同样,Liu et al.(2008)将植物置于4°C环境中,从整个植物组织中分离出RNA,并通过微阵列实验检测出11种DE miRNAs。我们检测到这11个miRNAs中的5个遵循相同的表达模式。Sunkar et al.(2004)研究了0°C处理24小时的整株植物的DE mirna,在我们的研究中,两个mirna也被鉴定为DE mirna。我们发现107个DE mirna中有14个之前在答:芥在冷胁迫中,这些比较表明不同研究之间有有限的重叠,这可能是由于应用温度,处理的持续时间或研究中使用的植物组织类型。在其他研究中,miR167c、miR168、miR397、miR389、miR400、miR837-5p、miR838和miR857等mirna被报道对冷胁迫有反应,但在本研究中未发现差异表达[32,35,71].
我们分析了psRNATarget工具预测的DE miRNA的推测靶点,分别在3 h、6 h和2 d时间点发现了96、173和267个miRNA靶点对,这反映了miRNA在长时间低温处理下调节转录组的重要性。通常,miRNA表达的改变通过互补配对后靶转录本的裂解影响靶基因的丰度。几种非生物胁迫的反应是由促进多种信号通路相互作用的共同介质调节的[105].为了维持应激相关基因的时空表达,由tf和sRNAs组成的调控因子是至关重要的。在DE miRNAs的预测靶点中,我们发现了编码tf的mrna,如NFY, MYB, TCP和HSFs。所有预测miRNA靶点的氧化石墨烯富集结果表明,与细胞核相关的靶点数量最多(136个mrna),其中85个编码tf。一些miRNAs与抗相关靶点不相关,但它们的表达模式支持了先前与寒冷相关的研究结果,如miR161.1和miR159b,这些研究发现miR161.1和miR159b在6小时时下调。研究SNRK1过表达系miR161和miR159b启动子活性降低,转录水平降低米尔可能导致miR161和miR159b水平降低的前体[106].植物有大量生长和胁迫反应所必需的tf,我们预测了85个编码tf的DE miRNA靶点。我们预测TCP2 (AT4G18390)和TCP4 (AT3G15030)会被miR319靶向,这与之前的研究一致答:芥还有甘蔗[107].所有miR319亚型在低温处理2 d后都被下调,这与水稻中miR319及其靶标被下调的研究一致TCP21经冷处理后上调[108].我们观察到类似的miR319下调和伴随的目标上调TCP2而且TCP4冷处理2 d后。
已知MYB tf可促进细胞增殖并控制苯丙类代谢和激素反应[109].我们观察到miR858的上调和其假定靶标的相应下调MYB48,MYB34而且MYB20.除了tf, miRNAs的靶标还包括表观遗传调控因子如甲基转移酶的转录本。冷藏6 h后miR163上调,冷藏2 d后miR163下调。其编码s -腺苷- l-蛋氨酸依赖甲基转移酶超家族蛋白的一个靶点(AT1G15125)在冷处理6 h后下调,编码N2, N2-二甲基鸟苷tRNA甲基转移酶的另一个靶点(AT5G15810)在冷处理2 d后上调。tRNA甲基转移酶(AT5G15810)被证明会引起应激相关的N2, N2-二甲基鸟苷(m22G)的tRNAs的修饰答:芥[110].通常,tRNA的核苷酸修饰发生在tRNA的成熟和加工过程中,这些修饰是特定应激的生物标志物,并被观察到在氧化剂的响应中被诱导[111].众所周知,应激诱导的外转录组变化调节tRNA稳定性、翻译起始和基于microrna的转录本调控[111].
miR159改变线粒体蛋白导入和乙烯生物合成
同样,miR159异构体在冷处理3 h时表达上调,但在冷处理2 d后表达下调。编码线粒体转位酶TIM-44相关蛋白(AT5G27395)的miR159的假设靶转录与3小时1.4倍的下调和2天后2.8倍的上调呈反相关。由于线粒体蛋白在细胞质中翻译并需要导入线粒体,我们的结果表明mirna介导的TIM-44调控可能导致冷处理期间线粒体蛋白导入的改变。众所周知,环境压力会抑制和刺激蛋白质的输入[112].TIM44招募线粒体HSP70,并促进含有转运肽的蛋白质从内膜导入线粒体基质[113].miR159也已知靶向编码MYB tf的rna,一种氨基环丙烷-1-羧酸合酶(ACC合酶)和蛋白质小的生长素RNA(阿富汗二月)家庭[114].与之前的研究结果一致,miR159的上调伴随着13的下调阿富汗二月mrna和ACC合成酶(AT4G37770)的转录本,该合成酶是乙烯生物合成所必需的,已知是耐寒性的负调节因子[115].因此,mir159介导的下调ACC合酶在我们的研究中观察到乙烯生物合成减少和转录增加CBF基因。
miR395c靶向mg螯合酶的mRNA,促进冷驯化中的产热
miR395c在冷处理6 h后被下调,其推测的Mg螯合酶h亚基编码基因也随之上调。镁螯合酶是一种多功能蛋白质,参与叶绿素合成,催化镁的插入2 +离子转化为原卟啉IX,生成镁原卟啉IX (Mg- proto -IX) [116].最近一项研究证实mg - proto - ix衍生信号在诱导基因Alternative氧化酶1a (AOX1a) [117].AOX1a还原O2到H2而不需要将质子从基质抽到膜间空间,从而以热的形式耗散多余的能量。产生的热量在冷胁迫条件下发挥产热作用,并促进应力耐受性。此外,Mg-Proto-IX信号还导致抗氧化酶活性的增加,从而增加了冷应激下氧化还原平衡的维持[118].
编码半乳糖氧化酶/海草重复蛋白的miR408可能以aba依赖的方式诱导驯化
有趣的是,miR408-5p在所有分析时间点均上调。一个鹰嘴豆MIR408干旱胁迫下过表达系的质体青苷编码水平降低。叶绿体色素苷的缺乏导致了铜的积累,铜水平的升高导致干旱响应基因如DREB因子的上调,并诱导其下游基因COR47 / RD17低温诱导78/对干燥敏感29A (LTI78 / RD29A) [119].同样,我们观察到miR408-5p、dreb转录本及其下游转录本的上调COR47/RD17和LTI78/RD29A[120,121].此外,MIR408过表达系显示光系统II效率提高,电解质泄漏和脂质过氧化减少,叶绿素荧光增加,导致ROS水平降低,耐寒性增强[122].我们预测了一个编码半乳糖氧化酶/海草重复超家族蛋白(AT1G67480)的miR408-5p靶点,发现其在6小时和2天的时间点下调,表明mRNA转录物发生裂解。Song等人(2013)研究了miR394及其编码半乳糖氧化酶kelch家族蛋白LCR (Leaf Curling Responsiveness)的靶标之一A. thaliana MIR394超表达和电感电容电阻测量突变体线。他们证实了miR394的上调和miR394的下调电感电容电阻测量表明它们在盐胁迫和干旱胁迫中的调节作用。其他半乳糖氧化酶kelch家族蛋白,如ZEITLUPE (AT5G57360)已被观察到在低温下会减少[123]和KISS ME DEADLY基因(AT1G80440)的表达下调,以诱导耐受紫外线[124].假设的目标半乳糖氧化酶/海草重复超家族蛋白(AT1G67480)也可能通过miR408-5p的下调,以aba依赖的方式介导耐寒性[45].
mirna介导的叶绿素生物合成和低温开花的抑制
miR171-3p在第2天时间点下调,其同源mRNA靶基因编码GRAS结构域TF稻草人样27 (AT2G45160)上调。已知SCL27结合到蛋白的启动子上猪肉基因(原叶绿素氧化还原酶)通过GT独联体-element重复并抑制其表达,导致叶绿素合成减少[125].的上调SCL27由于miR171水平的降低可以促进低温处理对叶绿素生物合成的抑制。
我们检测到miR156/157亚型在第2天的时间点上调,同时其靶蛋白下调SPL3(Squamosa Promoter Binding Protein-Like 3)MIR156a维持低水平的SPL3导致开花延迟的转录本答:芥[126].相反,miR172c下调,其假定的靶编码RAP2.7,也称为早期激活标记靶1 (target of Early Activation Tagged 1, TOE1)上调。A. thaliana过表达系也显示花期延迟[127], miR156和miR172c可能调控转录水平SPL3而且TOE1低温处理下抑制开花。
冷反应基因调控网络表明miRNA-TF-mRNA相互作用的重要性
通过将暂时的miRNA和mRNA表达数据与公开的miRNA、tf及其下游靶基因的调控结合行为的知识相结合,我们能够构建冷相关的GRN答:芥.在得到的GRN中,我们观察到mirna和TF的不同靶调控模式,既可以调控直接靶,也可以调控TF转录本,从而以间接方式控制其他靶。在mirna和它们的直接靶标之间观察到大量的连接,但当mirna靶向的tf被纳入网络时,受影响的靶标数量增加。这表明tf作为从miRNAs传递信息到几个tf影响目标的中心节点。提取的冷响应GRN显示了冷胁迫、生物胁迫和细胞组织、转录和翻译、运输和PPR、细胞壁和木质素合成、信号和蛋白质降解等不同功能模块的过度表达。这表明mirna调控似乎对控制已知参与冷适应的主要细胞通路很重要。完整的GRN以及特定的子网络可用于研究miRNA、tf及其直接和间接靶标的调控关系,以探索可能促进冷驯化的新型相互作用调控成分。
来自其他RNA类别的差异表达sRNAs
我们进一步研究了来自其他RNA类别的sRNAs,如lncRNA,独联体- - -反式nats,助教而且pha.我们发现15个非冗余、非重叠的lncrna在冷处理过程中产生DE srna。由于其中12个lncRNA转录本未被RNAseq检测到,3个未被DE检测到,我们推测lncRNA转录本被有效地加工成srna以抑制其转录本水平。这种自我调节机制已在水稻中得到证实,其中lncRNA长日特异性男性生育力相关RNA(LDMAR)能够生产Psi-LDMAR能够抑制亲本的sirnaLDMARrna依赖的DNA甲基化(RdDM)转录[128].
除了不重叠的lncrna外,我们发现了429个非冗余的lncrna独联体-NATs和179非冗余反式-产生pc:nc和pc:pc转录对比例较高的DE sirna的nats对。衍生的DE sRNAs独联体-NATs已在答:芥经过干旱、寒冷和盐胁迫处理[87].Zhang et al.(2012)在23°C条件下培养了29天的幼苗,并将其转移到5°C条件下24小时,我们检测到3个顺式本研究中报道的NATs对可引起冷诱导的nat-siRNAs。其中一对转录本AT5G15845 (ncRNA)和AT5G15850 (CONSTANS-like 1)在低温和盐胁迫下显示出相同的nat-siRNA产生模式,这两个基因的转录本水平以及nat-siRNA水平都上调了[87].另一对转录本AT5G19220 (adp -葡萄糖焦磷酸化酶)和AT5G19221 (ncRNA)转录本水平没有变化,但nat-siRNA产量升高。在Zhang等人(2012)的研究中,与寒冷、盐和干旱胁迫相比,第二对在未经处理的样品中显示更少的归一化读数。另一对由AT3G22120(细胞壁-质膜连接蛋白同源物)和AT3G22121 (ncRNA)组成的NATs导致了nat-siRNA产量的增加。在之前的研究中发现,相同的基因对在低温下产生减少的nat-siRNA,但在盐胁迫下产生升高的nat-siRNA [87].
我们观察到pc:nc基因对以pre-tRNA或TE作为非编码转录本搭档的优势。我们发现大量的pre-tRNA转录本与蛋白质编码转录本配对,并从一个或两个配对转录本中产生siRNAs。一些pre-tRNA转录本能够与编码Gly-Asp-Ser-Leu (GDSL)样脂肪酶/酰基水解酶超家族蛋白(AT5G55050)的mRNA配对,并且辣椒中的GDSL型脂肪酶基因已被证明与耐旱性、aba诱导基因的表达和氧化应激信号传导有关[129].编码含有F-Box蛋白的转录本(AT2G33655, AT1G11270, AT2G16365)已知与几种非生物应激相关基因共表达[130]或激活应激反应基因[131]显示与pre-tRNA转录本配对产生反式-nat-siRNAs。关于配对转录本和由此产生的nat-siRNA的表达模式,sirna可能是由pre-tRNA单独产生的,也可能是由pre-tRNA和蛋白质编码转录本配对形成的dsRNA加工而成的。trna衍生的小rna (tsRNAs)最初被认为是内切酶的降解产物,但最近的研究进展表明其在维持基因组稳定性、表观遗传、应激反应和细胞增殖等方面的功能作用[132].在其他生物中的研究表明,这些被称为转移rna衍生片段(5'tRF和3'tRF)的sRNAs的表达可能与蛋白质合成的质量控制有关[133,134].之前的实验A.thaliana和人类研究表明,trna衍生的sRNA的生物发生依赖于miRNA通路[135]和tRFs靶向TE转录本促进基因组稳定性[91,136].最近的一项研究证实了19-25 nt tRFs加载到AGO蛋白中,这表明tRNA产生的sRNAs在转录后基因沉默中的作用[94,137,138,139,140].German et al.(2017)从成熟tRNA转录物的5 '端观察到19 nt tRNA衍生的sRNAs的积累答:芥花粉。结论是后机匣的处理与mirna相似,因为在a中后机匣的积累减少了ddm1 / dcl1双突变体。在有磷和无磷的情况下,观察到trf和te衍生的sRNAs在大麦中是DE [141]和对磷酸盐缺乏的反应A.thaliana[142].而且,最近有一个新的类DCL-被称为sidRNAs的独立siRNAs被鉴定为纳入AGO4并在中触发从头甲基化答:芥[143表明与后机匣相似。除了tRNAs,我们还检测到差异调控tran从含有Ty3 Gypsy、CACTA和Ty1 Copia元素的转座子中获得的s-nat- sirna。TE来源的siRNAs可引起DNA甲基化或诱导抑制性组蛋白尾修饰以抑制TE位点[144].此外,在答:芥te衍生的siRNAs也可以靶向蛋白质编码基因。例如,te衍生的siRNA854被发现可以控制UBP1植物胁迫颗粒上游结合蛋白1a组分的转录水平[One hundred.].我们分别在3 h、6 h和2 d时间点发现了4、6和26个假设的蛋白质编码转录本与TE编码转录本、假基因RNA、mRNA和非编码RNA转录本配对,这表明几种RNA类别参与了对低温处理的适应。
除了来自pc:nc配对转录本的nat-siRNAs外,我们还鉴定出pc:pc独联体- - -反式-NATs对产生siRNAs,我们观察到在治疗过程中,nat-siRNAs的数量不断增加。我们检测到9个国家的nat-siRNAs表达升高独联体-NAT对对低温的反应,其中nat-siRNA产生的重叠转录本遵循nat-siRNA调控的经典表达模式[54].其特征是对刺激的反应中nat-siRNAs的表达增加,这是由于配对伴侣之一的转录升高导致同源伴侣转录下调。我们观察到寒冷诱导的一个转录本上调,同时其同源配对转录本被抑制,这些基因对组成了转录本RAN2 GTPase(AT5G20020)和植物都铎样rna结合蛋白(AT5G20030),TCP-1伴侣蛋白家族蛋白(AT3G11830)和电站u型箱24(AT3G11840)和PPR(AT1G03100)配对线粒体/3-甲基crotonyl- coa羧化酶(AT1G03090)。这些配对转录本的反向表达模式伴随着诱导独联体-nat-siRNAs冷处理。一个理想的例子是编码MATE外排蛋白(AT5G52450)的mRNA的冷响应上调,该蛋白参与外生解毒、抗病和植物激素及其配对伙伴的控制高叶绿素荧光106(HCF106(AT5G52440),显示出相应的下调。迄今为止,对MATE外排蛋白的功能研究尚缺乏,而编码HCF106蛋白的重叠转录本已被很好地表征。HCF106是一种叶绿体类囊体蛋白,可将蛋白质导入类囊体腔内。的hcf106敲除突变体是白化突变体,对幼苗是致命的,而较弱的T-DNA等位基因颜色较浅,气孔孔径减小,失水减少,因此引起较高的脱水耐受性[145].从这两个转录本中产生的nat-siRNAs导致的蛋白水平升高伴侣转录和下调HCF106转录结果提示,在冷驯化过程中存在一种冷反应调控机制。
基于我们的研究结果,我们得出结论,冷处理导致sRNA水平发生了相当大的变化,这可能导致在冷驯化的基础上基因表达的变化答:芥.多水平高通量测序和生物信息学分析的结合被证明是一个强大的工具,可以创建一个响应冷胁迫的sRNAs和mrna的调控网络。大量的miRNAs是DE,它们的预测靶点包括大量编码tf、PPR和TPR蛋白的mrna,这些蛋白分别调控基因表达和蛋白质生物合成,以及编码在冷驯化中起作用的重要酶的转录物。与mirna一起,大量的srna由lncrna和转录本产生顺式而且反式-NATs对表明所有sRNA类在冷适应中的强烈影响。
结论
根据这项研究答:芥,衍生的mirna和sRNAs顺式-而且反式-NAT基因对和来自lncrna的基因对在冷驯化中起着重要的调节作用。构建的基因调控网络提供了与miRNA及其相关的直接和间接靶标相互作用相关的大量信息。总体而言,本研究为加深我们对冷驯化调控网络的认识和理解提供了一个基础数据库。
方法
植物材料和胁迫处理
的种子答:芥生态型哥伦比亚(Col-0)与土壤基质高密度播种(约50粒,9 × 9 cm花盆),4℃黑暗下分层2 d。分层后,将花盆转移到LED-41 HIL2柜(Percival, Perry, USA),并在控制条件下培养,光照强度为16 h (80 μmol光子m)−2年代−1;对照植物在连续4°C光照开始4 h后转移,光照强度为35 μmol光子m,光照强度为35 μmol光子m−2年代−1.冷处理在三个独立的试验重复中进行,使用相同的生长室和相同的设置。分别在3 h、6 h和48 h (2 d)后,从3个冷处理的实验重复和对照样品中收获肺组织。
RNA分离和sRNA测序
根据制造商的说明,使用tri试剂(Sigma)从每个样品的生物三拷贝中分离总RNA。对于每个mRNA和包括多尾lncRNA在内的lncRNA文库,10 μg总RNA采用稳定RNA (Sigma-Aldrich)真空干燥。这些文库由Novogene(中国)公司使用Next Ultra RNA文库准备试剂盒(NEB)制备。在HiSeq-2500平台上,每个库至少有1500万个读对,以150 bp成对的方式进行链测序。
每个sRNA文库提取50 μg总RNA,用15%的天然聚丙烯酰胺凝胶分离。ZR small-RNA Ladder (Zymo Research)作为RNA大小标记,从凝胶中切除17-29 nt对应的sRNAs。凝胶片被转移到LoBind Eppendorf管中,并使用一次性聚丙烯杵进行粉碎。加入0.3 M NaCl浸泡凝胶片,试管在−80°C冷冻15分钟,随后在4°C洗脱RNA过夜。将缓冲液转移到Spin-X离心管过滤器(COSTAR)中,在4°C下离心1分钟,以去除凝胶片。加入2.5体积的100% (v/v)乙醇、1/10体积的3m NaOAc (pH 5)和1 μl糖原(10 mg/ml),在−80℃下孵育4 h。样品以17.000 x g在4℃离心30 min, 80%乙醇洗涤2次,室温干燥,7 μl无核酸酶水重悬。采用分光光度法测定RNA浓度,并使用NEBNext多重小RNA文库准备试剂盒Illumina进行文库制备,遵循制造商的方案,进行了微小的修改。将3 ' SR适配器在16°C下结扎过夜,并将SR逆转录引物与过量的3 ' SR适配器杂交以防止适配器二聚体的形成。连接3 ' SR适配器后,将5 ' SR适配器连接到RNA上,在25°C下孵育1.5 h。 PCR amplification of the libraries was performed using specific index primers for 12 cycles and the cDNA amplicons were separated on a 6% native acrylamide gel at 120 V. The gel was stained with SYBR gold and RNAs with a size between 138 and 150 nt corresponding to adapter-ligated sRNAs with a size between 18 and 30 nt were excised. Gel elution of the DNA was performed as described above except the addition of 1 μl linear acrylamide (5 mg/ml) prior to precipitation to increase the DNA pellet mass. The cDNA library with concentration of at least 8 ng/μl was considered optimum for sequencing. The sRNA libraries were sequenced with an Illumina deep sequencing platform (Illumina HiSeq 1500) with a read length of 50 nt and a minimum of 7 million reads per library.
转录组生物信息学分析
冷驯化3 h、6 h和2 d的三份样品以及相应对照的mRNA/lncRNA测序数据使用开放网络平台GALAXY (https://usegalaxy.org/) [146].使用FASTQ Trimmomatic工具使用默认参数对适配器序列进行修剪。来映射原始读取答:芥参考基因组(https://www.arabidopsis.org, release: TAIR10),使用Tophat工具,最大内含子长度参数为3000 nt。arapport11注释[147]对转录本进行注释,长度超过200 bp的ncRNA转录本被认为是lncrna。我们使用featurerts工具来计算映射到参考基因组的读取数(附加文件1:表S1)。使用计数文件作为GALAXY的DeSeq2工具的输入,我们获得了最终的基因列表。所有基因基于arapport11参考注释进行分类(https://araport.org/).
sRNA原始读数被映射到TAIR10 (https://www.arabidopsis.org,发布:TAIR10)参考基因组使用Shortstack软件[148].大约80%获得的读取有效地映射到它(附加文件1:表S2)。我们为miRNA (miRBase version 22.1)、lncRNA (arapport11)、反式-而且顺式nat-siRNA [57,87,88,89], ta-siRNA和phasiRNA [101],用于生成从这些RNA类别中获得的srna的读计数。使用DeSeq2工具对GALAXY和FC≥2&≤- 2的sRNAs进行差异表达分析,Benjamini-Hochberg校正p-value≤0.05为DE。使用UpSetR包生成DE mirna的全局比较(https://CRAN.R-project.org/package=UpSetR).
茎环qRT-PCR cDNA的合成
使用300 ng RNA从处理过和未处理过的样品中三个生物重复合成cDNA [149].RNA用DNAse I (2 U, NEB)在37°C下处理30分钟以消除基因组DNA污染,酶在65°C下热灭活10分钟,RNA用M-MuLV逆转录酶(200 U, NEB)在42°C下逆转录30分钟。特异性茎环引物和通用反向引物用于cDNA合成(附加文件14:表S18)。在cDNA合成过程中,我们添加UBI1(AT4G36800)特异性反向引物,通过PCR检测cDNA的成功合成UBI1特异性基因引物。
茎袢qRT-PCR
使用EvaGreen和srna特异性引物进行Real-time PCR(附加文件)14:表S18)。每个样品进行3个技术重复,每个反应包含相当于20 ng/μl初始RNA的cDNA量。将qRT-PCR程序调整为95°C初始变性2 min,然后在95°C 12 s,退火30 s, 72°C 15 s下扩增40个循环。在每个循环后测量SYBR绿色信号,并监测熔化曲线以确认引物的特异性。Ct值通过ΔΔC计算表达水平t方法(150].表达水平归一化使用UBI1管家基因(AT4G36800)。
miRNA靶点预测
使用psRNATarget预测工具预测MiRNA靶点(2017年更新)[73].使用DE mirna作为查询进行搜索答:芥蛋白质编码和arapport11的非编码转录本保持默认参数,并允许计算目标可达性(最大能量解对目标位点= 25)。我们使用严格的截断值2.5作为选择潜在目标的最大期望分数。
miRNA靶点的基因本体
GO分析使用DAVID生物信息学工具[78].输入提供miRNA靶基因列表,输出列表包含生物过程、细胞间隔和分子功能分类的基因。我们用Benjamini-Hochberg校正过滤显著的GO项p-value≤0.05,由所有类别的Fisher检验得出。可视化GO术语的点图是使用ggplot2包(https://CRAN.R-project.org/package=ggplot2).
监管网络模型的构建与验证
基因调控网络(GRN)是利用ATRM的高置信度实验验证的调控连接构建的[79和Agris [80].我们没有包括PlantRegMap中所有可用的连接[151]而是那些符合约束母题保存标准的。第一个标准包括结合位点位于不同植物物种的保守元件上的TF连接(motif_CE),第二个标准包括扫描TFBSs守恒时发现结合位点在不同植物物种中保守的TF连接(FunTFBS) [81].遵循这两个标准的基于TF的调控连接与psRNATarget工具预测的miRNA靶标合并,以获得完整的网络模型。目标基因表达预测使用来自Dotnet的快速树回归学习器进行。ML 0.8版[152].结果变量为目标基因在不同时间点(3 h、6 h、2 d)的表达FPKM。作为输入变量,我们使用时间点、各调节因子家族在相应时间点的表达水平和目标基因结合位点的计数。每个TF的家族分配和每个目标的绑定位点信息均取自AtTFDB数据库[153].与GRN相关的数据可通过免费可视化软件GEPHI访问,该软件可在https://gephi.org/(附加文件7:数据S1, S2)。
热图集群
雉图函数(https://cran.r-project.org/web/packages/pheatmap/index.html)的“Pheatmap”用于创建热图,显示在冷处理的三个时间点差异表达的mirna的分层聚类。
数据和材料的可用性
Illumina sRNA和mRNA测序原始数据存入NCBI SRA数据库,ID为PRJNA592037。本研究中用于分析的所有原始数据均可在https://dataview.ncbi.nlm.nih.gov/object/PRJNA592037?reviewer=lhkljqn6c6qp67vp6p70ra9l59.
缩写
- srna / siRNAs:
-
小rna /小干扰rna
- microrna的:
-
微
- ncRNA:
-
非编码RNA
- lncRNA:
-
长链非编码RNA
- ta-siRNA:
-
Trans-acting核
- cis /反式nat:
-
Cis /反式-自然反义转录本
- 奈特:
-
自然反义转录本
- 度:
-
差异表达基因
- TE:
-
转座因子
- TF:
-
转录因子
- pc:
-
蛋白质编码
- nc:
-
非编码
- 走:
-
基因本体论
- OST1:
-
开放气孔1
- 阿坝:
-
脱落酸
- ICE1:
-
CBF表达诱导因子
- CBF:
-
c -重复序列结合因子
- 含有DREB:
-
脱水响应元件结合因子
- CRT / DRE:
-
冷反应敏感转录因子/脱水反应元件
- 天哪:
-
Cold-responsive
- 沛富:
-
ABRE-binding因素
- RdDM:
-
rna定向DNA甲基化
- 方法:
-
开花位点C
- DCL1:
-
DICER-LIKE1
- 电感电容电阻测量:
-
卷叶响应性
- P5CDH:
-
Delta-pyrroline-5-carboxylate脱氢酶
- SRO5:
-
类似于胚根诱导细胞死亡
- DDM1:
-
DNA甲基化降低1
- ea-siRNA:
-
表观遗传激活的siRNA
- TCP:
-
大刍草分枝1,Cycloidea和Pcf转录因子2
- 蒂姆:
-
转位酶内膜亚基
- RAN2:
-
ras相关的gtp结合核蛋白
- PPR:
-
五肽重复超家族蛋白
- NFY:
-
核Factor-Y
- HSF:
-
热休克因素
- ACC合酶:
-
Amino-cyclopropane-1-carboxylate合酶
- 配偶:
-
多抗菌挤压蛋白
- 扶轮基金会:
-
trna衍生的RNA片段
参考文献
- 1.
Gornall J, Betts R, Burke E, Clark R, Camp J, Willett K,等。21世纪初气候变化对农业生产力的影响。中国生物医学工程学报(英文版);2010;29(3):344 - 344。
- 2.
朱镕基JK。植物的非生物胁迫信号及其响应。细胞。2016;167(2):313 - 24。
- 3.
索兰克AU,夏尔马AK。低温胁迫下植物信号转导的研究。植物学报。2008;14(1-2):69-79。
- 4.
托马斯休曼氏金融。低温反应基因在植物抗冻性中的作用。植物科学进展。1998;18(1):1 - 8。
- 5.
阿布拉梅,孙华,李震,魏超,高飞,周勇,等。黄芪miRNAs的鉴定及其对冷胁迫的响应。生物分子。2019;9(5):182。
- 6.
丁艳。OST1激酶通过提高ICE1的稳定性调节抗冻性拟南芥.生物工程学报。2015;32(3):278-89。
- 7.
朱建军,李志强,李志强,等。植物冷胁迫驯化过程中的基因调控。方法Mol生物学,2010;639:39-55。
- 8.
Cuevas-Velazquez CL, renton - luna DF, Covarrubias AA。解剖脱氢酶的冷冻保护机制。植物科学进展(英文版);
- 9.
Devert A, Fabre N, Floris M, Canard B, Robaglia C, Crete P.通过纯化的拟南芥RNA依赖RNA聚合酶RDR2和RDR6启动双链RNA合成的引物依赖和引物独立。PLoS One. 2015;10(3):e0120100。
- 10.
王德忠,金燕,丁晓华,王文军,翟世生,白丽萍,等。低温胁迫下植物ICE-CBF-COR信号通路的基因调控和信号转导生物化学,2017;82(10):1103 - 17所示。
- 11.
李世杰,姜志勇,朴海杰,金MD,裴ms,崔熙,等。DREB2C与ABF2相互作用,ABF2是调节脱落酸反应基因表达的bZIP蛋白,其过表达影响脱落酸敏感性。植物科学进展。2010;32(2):344 - 344。
- 12.
李志强,李志强,李志强,等。植物胁迫适应和胁迫记忆的表观遗传和染色质机制。中国生物医学工程学报,2017;18(1):124。
- 13.
王志强,王志强,王志强,等。植物低温响应的表观遗传控制。中国植物科学,2017;8:1643。
- 14.
帕鲁萨SG,阿里GS,雷迪AS。的前mrna的可选剪接拟南芥富含丝氨酸/精氨酸的蛋白质:由激素和压力调节。中国生物医学工程学报,2007;49(6):1091-107。
- 15.
Chekanova农协。长链非编码rna及其在植物中的功能。植物学报,2015;27:207-16。
- 16.
李松,张晓霞,张晓霞,张晓霞。植物小rna在植物发育和胁迫响应中的作用。中国生物医学工程学报,2017;29(4):344 - 344。
- 17.
顾玉生,黄建文,梅志,刘旭,惠建华,陈廷峰,等。植物对非生物胁迫反应中的小rna:调控作用和研究方法。中华分子化学杂志,2015;16(10):24532-54。
- 18.
Ransohoff JD, Wei Y, Khavari PA。长基因间非编码RNA的功能和特点。中国生物医学工程学报,2018;19(3):143-57。
- 19.
王晓强,王晓强,王晓强。基于非编码rna的基因组构建。中国生物医学工程学报。2011;12(5):307-21。
- 20.
狄超,袁杰,吴勇,李军,林华,胡林,等。胁迫响应型lncrna的特征拟南芥通过整合表达、表观遗传和结构特征。中国生物医学工程学报,2014;30(5):344 - 344。
- 21.
王kc,常海海。长链非编码rna的分子机制。生物化学学报,2011;43(6):904-14。
- 22.
Franco-Zorrilla JM, Valli A, Todesco M, Mateos I, Puga MI, Rubio-Somoza I,等。目标拟态为调控microRNA活性提供了一种新的机制。植物学报。2007;39(8):1033-7。
- 23.
刘芳,李志刚,李志刚。冷致沉默基因的克隆与表达研究拟南芥Polycomb目标。自然。2009;462(7274):799 - 802。
- 24.
Csorba T, Questa JI,孙Q, Dean C.反义COOLAIR介导春化过程中FLC染色质状态的协调切换。中国科学(自然科学版),2014;29(4):344 - 344。
- 25.
Matzke MA, Mosher RA。rna导向的DNA甲基化:一种日益复杂的表观遗传途径。植物学报。2014;15(6):394-408。
- 26.
郑志康先生。Argonaute 4相关长链非编码RNA的分析拟南芥通过rna定向的DNA甲基化为基因调控提供了新的见解。基因。2017;8(8):198。
- 27.
马洛丽,李志强,李志强,等。植物中microRNAs及相关小rna的功能。中国科学,2006;38(增刊):331 - 6。
- 28.
古斯塔,崔查克R,威瑟CJ。植物低温驯化:脱落酸的作用。植物生长学报,2005;24(4):308-18。
- 29.
陈霞。MicroRNA在植物中的生物发生和功能。中国农业科学,2005;26(2):523 - 31。
- 30.
de Lima JC, Loss-Morais G, Margis R. MicroRNAs在植物发育和响应非生物胁迫过程中发挥关键作用。中国生物医学杂志,2012;35(4):1069-77。
- 31.
宋刚,张荣,张松,李勇,高洁,韩旭,等。普通小麦幼穗中microRNAs对低温处理的响应。BMC基因组学杂志,2017;18(1):212。
- 32.
巴耶夫V,米列夫I,纳杰诺夫M,瓦切夫T, Apostolova E,梅特罗夫N,等。深入研究小RNA在高温和低温胁迫反应中的丰度和表达拟南芥.植物化学学报。2014;34(4):344 - 344。
- 33.
李国宝,戴恒胜,朱建科。的拟南芥冷响应转录组及其ICE1的调控。植物学报。2005;17(11):3155-75。
- 34.
李华,熊林,石谷M,史蒂文森B,朱建科。拟南芥突变体冷调节基因表达及耐寒性研究。植物细胞分子生物学杂志,1999;17(3):301-8。
- 35.
刘海辉,田霞,李玉杰,吴昌昌,郑成成。基于微阵列的胁迫调控microRNAs分析拟南芥.Rna。2008;14(5):836 - 43。
- 36.
马哈尔B, Fakrudin B,高希S, Krishnaraj PU。LNA介导的miR171和miR397a在叶片和环境根组织中的原位杂交结果显示,在响应笋热休克时,miR171和miR397a的表达具有同质性拟南芥.植物生物化学学报。2013;23(1):93-103。
- 37.
张勇,朱旭,陈旭,宋超,邹智,王勇,等。茶树低温响应microrna的鉴定与鉴定(茶树)和它们的目标,使用高通量测序和降解分析。中国生物医学工程学报。2014;
- 38.
De Rienzo F, Gabdoulline RR, Menziani MC, Wade RC。蓝铜蛋白的分子相互作用特性的比较分析。蛋白质科学,2000;9(8):1439-54。
- 39.
Pilon SEA-GaM。microrna介导的铜蛋白表达的系统性下调,以应对低铜可用性拟南芥.中国生物医学工程学报,2008;29(3):344 - 344。
- 40.
Pourcel L, Routaboul J-M, Cheynier V, Lepiniec L, Debeaujon I.植物类黄酮氧化:从生化性质到生理功能。植物科学进展,2007;12(1):29-36。
- 41.
梁敏,蔡霞,吴勇。漆酶基因ZmLAC1在胁迫下玉米初生根中的表达。植物细胞环境。2006;29(5):746-53。
- 42.
宋杰比,黄淑琪,达尔梅特,杨志明。microRNA394对叶片形态的调控及其卷曲效应。中国生物医学工程学报,2012;29(7):344 - 344。
- 43.
李志强,李志强,李志强,等。原真皮miR394信号定义了干细胞能力的区域拟南芥分生组织。科学通报。2013;24(2):125-32。
- 44.
宋俊杰,高阿生,王勇,李宝文,张玉玲,杨志明。miR394及其靶基因LCR参与低温胁迫应答拟南芥.植物基因。2016;5:56-64。
- 45.
宋俊杰,高松,孙东,李华,舒XX,杨志明。miR394和LCR参与拟南芥盐胁迫和干旱胁迫以脱落酸依赖的方式响应。BMC植物生物学2013;13:210。
- 46.
董春华,裴华。miR397的过表达提高了植物对低温胁迫的耐受性拟南芥.中国生物工程学报,2014;29(4):344 - 344。
- 47.
Peragine A, Yoshikawa M, Wu G, Albrecht HL, Poethig RS. SGS3和SGS2/SDE1/RDR6在幼年发育和反式作用sirna的产生中是必需的拟南芥.基因开发,2004;18(19):2368-79。
- 48.
Vazquez F, Vaucheret H, Rajagopalan R, Lepers C, Gasciolli V, Mallory AC,等。内源性反式作用siRNAs调节的积累拟南芥信使rna。生物化学学报,2004;16(1):69-79。
- 49.
关超,吴波,于涛,王强,Krogan NT,刘旭,等。空间生长素信号通路控制叶片扁平化拟南芥.中国生物医学杂志,2017;27(19):2940-50。
- 50.
Moldovan D, Spriggs A, Yang J, Pogson BJ, Dennis ES, Wilson IW。缺氧反应microRNAs和反式作用小干扰rna拟南芥.中国科学(d辑),2010;26(1):1 - 5。
- 51.
Kohei K. TAS1反式作用siRNA靶标在低温下被差异调控,TAS1反式作用siRNA介导温度控制下At1g51670的表达。中国生物工程学报。2010;42(7):457 - 457。
- 52.
谢长廷L-C。拟南芥小rna介导的缺磷应答的深度测序研究2009.
- 53.
陈晓明,陈晓明。反义RNA在高等真核生物细胞中的作用与命运。中国生物医学工程学报,1998;29(4):344 - 344。
- 54.
鲍珊,朱杰,朱建科,孙嘉荣。来自一对天然顺反义转录本的内源性siRNAs调节植物的耐盐性拟南芥.细胞。2005;123(7):1279 - 91。
- 55.
怀特,韦纳。自然反义转录的功能。生物化学学报,2013;
- 56.
王晓军,Gaasterland T,蔡新华。植物顺天然反义转录本的全基因组预测与鉴定拟南芥.中国生物医学工程学报,2005;6(4):R30。
- 57.
袁超,王娟,陈敏,陈东,陈敏。中国自然反义转录物的全基因组研究拟南芥.DNA res 2015;22(3): 233-43。
- 58.
张旭,李勇,吴震,Polishko A,张宏,Chinnusamy V,等。应答环境和发育线索的顺式核酸生成小RNA的机制。植物学报。2013;6(3):704-15。
- 59.
Creasey KM, Zhai J. miRNAs从转座子引发广泛的表观遗传激活sirna拟南芥.大自然。2014;508:411-5。
- 60.
Piriyapongsa J, Jordan IK。植物转座元件对siRNAs和miRNAs的双重编码。Rna。2008;14(5):814 - 21所示。
- 61.
王东,曲忠,杨林,张强,刘志华,杜涛,等。转座元件(TEs)是植物胁迫相关长基因间非编码rna的重要组成部分。中国生物医学工程学报,2017;29(1):344 - 344。
- 62.
Fowler S, Thomashow MF。拟南芥转录组分析表明,在冷驯化过程中,除了CBF冷反应通路外,还有多条调控通路被激活。植物学报。2002;14(8):1675-90。
- 63.
张b, Pan X, Cannon CH, Cobb GP, Anderson TA。植物microRNA基因的保存与分化。植物学报,2006;46(2):243-59。
- 64.
Pelaez P, Trejo MS, Iniguez LP, Estrada-Navarrete G, Covarrubias AA, Reyes JL,等。中microrna的鉴定和特征菜豆通过高通量测序。BMC Genomics. 2012;13:83。
- 65.
潘文杰,李志强,李志强,等。植物小分子rna的研究进展拟南芥而且栽培稻识别重要的靶基因。中国科学(d辑),2004;26(4):531 - 531。
- 66.
Khaksefidi RE, Mirlohi S, Khalaji F, Fakhari Z, Shiran B, Ebrahimie E. 7种保守microrna在非生物胁迫下的差异表达及其调控网络向日葵.植物科学进展(英文版);
- 67.
于勇,倪震,王勇,万华,胡忠,姜青,等。大豆miR169c的过表达增加了转基因拟南芥对干旱胁迫的敏感性。植物科学通报2019;
- 68.
秦志,李超,毛玲,吴玲。植物非保守microrna的新发现。植物科学进展(英文版);
- 69.
Megha S, Basu U, Kav NNV。microRNAs对植物低温胁迫的调控。植物细胞环境学报,2018;41(1):1 - 15。
- 70.
吕冬冬,白旭,李勇,丁小东,葛勇,蔡宏,等。水稻冷胁迫响应mirna的微阵列分析。基因。2010;459(1 - 2):39-47。
- 71.
孙卡尔,朱建科。来自新型和压力调节的microRNAs和其他小rna拟南芥.植物学报。2004;16(8):2001-19。
- 72.
朱建军,朱军。mirna和sirna在植物生物和非生物胁迫响应中的作用。生物化学学报,2012;1819(2):137-48。
- 73.
戴X,庄Z,赵PX。psRNATarget:植物小RNA靶标分析服务器(2017年发布)。核酸研究,2018;46(W1): W49-54。
- 74.
王晓霞,张海生,朱涛,王晓霞,李建峰。转录组变化拟南芥对盐、渗透和冷胁迫的反应。植物科学进展。2002;30(4):339 - 339。
- 75.
韩恩,Kilian J, Mohrholz A, Ladwig F, Peschke F, Dautel R,等。植物核心环境胁迫响应基因在非生物胁迫下是系统协调的。中华分子化学杂志,2013;14(4):7617-41。
- 76.
王晓明,王晓明,王晓明,王晓明,等。mir319调控TCP转录因子在叶片发育过程中表达的空间调控。中国生物医学工程学报,2018;26(2):344 - 344。
- 77.
Soitamo AJ, Piippo M, Allahverdiyeva Y, Battchikova N, Aro EM.光具有特定的调制作用拟南芥低温下的基因表达。BMC植物生物学,2008;8:13。
- 78.
焦X, Sherman BT,黄达W, Stephens R, Baseler MW, Lane HC,等。DAVID-WS:促进基因/蛋白质列表分析的有状态web服务。生物信息学,2012,28(13):1805 - 6。
- 79.
金J, K,唐X, Z, Lv L,赵Y, et al。拟南芥转录调控图谱揭示了新转录因子的独特功能和进化特征。中国生物医学杂志,2015;32(7):1767-73。
- 80.
Palaniswamy SK, James S, Sun H, Lamb RS, Davuluri RV, Grotewold E. AGRIS和AtRegNet。将顺式调控元件和转录因子连接到调控网络的平台。植物学报。2006;40(3):818-29。
- 81.
田F,杨,孟YQ、金J,高g . PlantRegMap:地图制图功能监管的植物。核酸研究进展2019;48(D1): D1104-13。
- 82.
哈特韦尔LH,霍普菲尔德JJ,莱伯乐S,默里AW。从分子到模块细胞生物学。自然科学。1999;402(6761增刊):C47-52。
- 83.
布隆德尔,纪尧姆J-L,兰贝特R,列斐伏尔E.大型网络中社区的快速展开。机械工程学报。2008;10:P10008。
- 84.
布兹洛德,古尼亚,胡宁,伯纳达克,米拉,本达乌,等。生长素反应因子(ARFs)是番茄对生物和非生物胁迫反应中生长素作用的潜在调节因子。PLoS One. 2018;13(2):e0193517。
- 85.
郭珊珊,王萍,朱霞。拟南芥MYB转录因子MYB111通过调控类黄酮生物合成调控盐反应。环境与实验植物学。103807.https://doi.org/10.1016/j.envexpbot.2019.103807.
- 86.
Petridis A, Doll S, Nichelmann L, Bilger W, Mock HP。拟南芥G2-LIKE类黄酮调控因子和油菜素类固醇增强表达1是类黄酮积累的低温调控因子。植物学报。2016;211(3):912-25。
- 87.
张旭,夏杰,李烨。植物nat-siRNAs的全基因组分析揭示了它们的分布、生物成因和功能。基因组生物学,2012;13:R20。
- 88.
王慧,钟普杰,刘杰,张义昌,Kean MJ,徐洁,等。长非编码自然反义转录本的全基因组鉴定及其对光的反应拟南芥.基因组学报,2014;24(3):444-53。
- 89.
金华,瓦契奇V,吉克T,洛纳尔迪S,朱建科。小rna与顺自然反义转录的调控拟南芥.BMC Mol biology, 2008;9:6。
- 90.
唐国强,莱因哈特BJ,巴特尔DP,扎莫尔PD。植物中RNA沉默的生化框架。基因工程学报,2003;17(1):49-63。
- 91.
马丁内斯G,乔杜里SG,斯洛特金RK。trna衍生的小rna靶向转座因子转录本。中国生物医学工程学报,2017;45(9):5142-52。
- 92.
转座子来源的非编码rna及其在植物中的功能。植物科学,2018;9:600。
- 93.
Creasey KM, Zhai J, Borges F, Van Ex F, Regulski M, Meyers BC等。拟南芥转座子中mirna引发广泛的表观遗传激活sirna。大自然。2014;508(7496):411 - 5。
- 94.
Loss-Morais G, Waterhouse PM, Margis R.与argonaute相关的植物trna衍生RNA片段(tRFs)的描述及其假定靶点的鉴定。《生物指南》2013;8:6。
- 95.
马玲,洪志,张震。拟南芥核周和核膜蛋白定位。植物细胞学报,2007;26(8):1373-82。
- 96.
杜军,黄亚平,席杰,曹明梅,倪卫生,陈旭,等。利用拟南芥的耐盐基因功能挖掘。中国生物医学工程学报。2008;26(4):344 - 344。
- 97.
Leonhardt N, Kwak JM, Robert N, Waner D, Leonhardt G, Schroeder JI。拟南芥保护细胞的芯片表达分析及隐性脱落酸超敏蛋白磷酸酶2C突变体的分离。植物学报。2004;16(3):596-615。
- 98.
卡普兰F,盖伊CL。麦芽糖在温度休克中的β -淀粉酶诱导及保护作用。植物科学进展。2004;35(3):344 - 344。
- 99.
王志,崔东,刘娟,赵娟,程玲,辛伟,等。拟南芥zed1相关激酶介导免疫反应和生长稳态的温度敏感交集。新植物,2017;215:711-24。
- One hundred.
McCue AD, Slotkin RK。转座因子小rna作为基因表达的调节因子。植物学报,2012;28(12):616-23。
- 101.
Howell MD, Fahlgren N, Chapman EJ, Cumbie JS, Sullivan CM, Givan SA,等。RNA依赖RNA聚合体6/DICER-LIKE4通路的全基因组分析拟南芥揭示了对miRNA和tasirna定向靶向的依赖性。植物细胞学报。2007;19(3):926-42。
- 102.
Binder S, Stoll K, Stoll B. p类pentatricopeptide重复蛋白是植物线粒体转录本5 '端高效形成所必需的。中国生物工程学报。2013;10(9):1511-9。
- 103.
郭文杰,何铁清。脱落酸诱导蛋白HVA22抑制赤霉素介导的谷物糊粉细胞程序性死亡。植物科学进展。2008;37(4):344 - 344。
- 104.
Samad AFA, Sajad M, Nazaruddin N, Fauzi IA, Murad AMA, Zainal Z,等。MicroRNA和转录因子:植物调控网络中的关键角色。中国植物科学,2017;8:565。
- 105.
Atkinson NJ, Urwin PE。植物生物和非生物胁迫的相互作用:从基因到田间。应用物理学报,2012;29(10):1043 - 1043。
- 106.
Confraria A, Martinho C, Elias A, Rubio-Somoza I, Baena-Gonzalez E.拟南芥中mirna介导snrk1依赖的能量信号。植物科学进展(英文版);
- 107.
Thiebaut F, Rojas CA, Almeida KL, Grativol C, Domiciano GC, Lamb CR,等。低温胁迫下甘蔗miR319的调控。植物细胞环境学报。2012;35(3):502-12。
- 108.
王st,孙晓林,星野勇,于勇,贾波,孙志伟,等。MicroRNA319通过靶向OsPCF6和OsTCP21正向调控水稻的耐寒性(栽培稻l .)。PLoS One. 2014;(3)。https://doi.org/10.1371/journal.pone.0091357.
- 109.
Ambawat S, Sharma P, Yadav NR, Yadav RC。MYB转录因子基因作为植物反应的调节因子:综述。植物学报。2013;19(3):307-21。
- 110.
王勇,庞超,李霞,胡震,吕震,郑波,等。鉴定水稻和拟南芥胁迫响应和发育的关键tRNA核苷修饰基因。中国生物医学工程学报,2017;17(1):261。
- 111.
Huber SM, Leonardi A, Dedon PC, Begley TJ。在细胞对毒性暴露和环境应激的反应过程中,tRNA表名组的多功能作用。毒物。2019;7(1):17。
- 112.
李志强,李志强,李志强,等。环境胁迫抑制和刺激植物线粒体中不同的蛋白质输入途径。中国生物医学工程学报,2003;29(3):344 - 344。
- 113.
Krimmer T, Rassow J, Kunau WH, Voos W, Pfanner N.线粒体蛋白输入发动机:基质Hsp70的atp酶结构域对Tim44的结合至关重要,而肽结合结构域和carboxy末端段起刺激作用。分子生物学杂志,2000;20(16):5879-87。
- 114.
任宏,王志强,王志强,等。植物生长过程中SAUR蛋白在激素和环境信号中的作用。植物学报,2015;8(8):1153-64。
- 115.
石勇,田松,侯林,黄霞,张霞,郭辉,等。乙烯信号通路通过抑制CBF和a型ARR基因表达负向调控拟南芥抗冻性。植物学报。2012;24(6):2578-95。
- 116.
增田T.四吡咯生物合成导致叶绿素的mg分支的最新综述。光子学报,2008;36(2):344 - 344。
- 117.
张忠文,袁松,徐峰,杨华,陈晔,袁敏,等。mg -原卟啉,血红素和糖信号双细胞总RNA对抗除草剂和高光氧化应激。植物细胞环境学报。2011;34(6):1031-42。
- 118.
张志伟,吴志林,冯丽英,董丽华,宋亚杰,袁敏,等。mg -原卟啉IX信号增强了植物对冷胁迫的耐受性。前沿植物学报2016;7:1545。
- 119.
佐久间Y,丸山K,大阪部Y,秦F,关M,筱崎K,等。拟南芥转录因子DREB2A参与干旱响应基因表达的功能分析植物学报。2006;18(5):1292-309。
- 120.
Hajyzadeh M, Turktas M, Khawar KM。miR408过表达导致鹰嘴豆抗旱性增强。基因。2015;555(2):186 - 93。
- 121.
蒋强,孙霞,牛峰,胡志,陈锐,张慧。GmDREB1过表达对转基因小麦种子中microrna表达的影响。PLoS One. 2017;(5)。https://doi.org/10.1371/journal.pone.0175924.
- 122.
Ma C, Burd S. Lers a: miR408参与植物非生物胁迫响应拟南芥.中国植物学报,2015;26(1):344 - 344。
- 123.
权玉杰,朴MJ,金SG, Baldwin IT,朴CM。环境胁迫条件下拟南芥生物钟基因的选择性剪接和无意义介导的衰退。BMC植物生物学。2014;14:136。
- 124.
张旭,勾明,郭超,杨华,刘佳杰。下调拟南芥含Kelch结构域的F-box蛋白可增强(多)酚的产生和对紫外线辐射的耐受性。中国生物医学工程学报。2015;26(2):337 - 348。
- 125.
马震,胡旭,蔡伟,黄伟,周旭,罗强,等。拟南芥mir171靶向的稻草人样蛋白与GT顺式元件结合,在光照条件下介导赤霉素调控的叶绿素生物合成。PLoS Genet, 2014。https://doi.org/10.1371/journal.pgen.1004519 (8).
- 126.
吴国强,王晓明,王晓明,等。拟南芥miR156及其靶基因SPL3对幼苗发育的调控。发展。2006;133(18):3539 - 47。
- 127.
张志刚,张志刚,张志刚,等。MicroRNA及其APETALA2-like靶基因对花期和花器官特性的调控。植物学报。2003;15(11):2730-41。
- 128.
丁娟,沈娟,毛红,谢伟,李霞,张强。rna介导的DNA甲基化参与调控水稻光周期敏感雄性不育。植物学报。2012;5(6):1210-6。
- 129.
洪景杰,崔华伟,黄伟,金世德,金新华,崔世德,等。新型gdsl型辣椒脂肪酶基因CaGLIP1在疾病易感性和非生物胁迫耐受性中的作用足底。2008;227(3):539 - 58。
- 130.
Gonzalez LE, Keller K, Chan KX, Gessel MM, Thines BC。转录组分析揭示了拟南芥F-BOX胁迫诱导1作为茉莉酸和脱落酸胁迫基因表达的调节因子。BMC基因组学,2017;18:533。
- 131.
李强,王伟,王伟,张刚,杨林,王勇,等。小麦F-box蛋白基因TaFBA1与植物耐热性有关。植物科学,2018;9:521。
- 132.
朱玲,欧德伟,董哲。植物中转移rna衍生小rna的研究。中国科学:生命科学,2018;36(2):344 - 344。
- 133.
王强,李涛,徐坤,张伟,王霞,权健,等。trna衍生的小rna通过触发卵菌病原体大豆疫霉菌(Phytophthora sojae)靶转录本的序列特异性降解来调节基因表达。前沿植物科学2016;7:1938。
- 134.
秦超,徐培平,张旭,张超,刘长胜,杨德东,等。trna衍生小rna在神经系统疾病中的病理意义。神经再生科学,2020;15(2):212-21。
- 135.
Cole C, Sobala A, Lu C, Thatcher SR, Bowman A, Brown JW,等。对深度测序数据进行筛选,发现存在大量源自tRNAs的dicer依赖小rna。Rna。2009;15(12):2147 - 60。
- 136.
豪赛克D,黄Y,刘A,副手P,火AZ,马凯。人类trna衍生的小RNA在RNA沉默全局调控中的作用。Rna。2010;16(4):673 - 95。
- 137.
Alves CS, Vicentini R, Duarte GT, Pinoti VF, Vincentz M, Nogueira FT.陆生植物trna来源RNA片段的全基因组鉴定和表征。中国生物医学工程学报,2017;29(3):344 - 344。
- 138.
杨明明,Bennasser Y, Watashi K, Le SY, Houzet L, Jeang KT。在HIV-1感染细胞中小非编码RNA的焦磷酸测序:病毒-细胞双链RNA杂交处理的证据。中国生物医学工程学报,2009;37(19):6575-86。
- 139.
Garcia-Silva MR, Cabrera-Cabrera F, Guida MC, Cayota A. trna衍生小RNA在RNA沉默机制中的作用。基因(巴塞尔)。3(4): 603 - 2012; 14。
- 140.
Kuscu C, Kumar P, Kiran M, Su Z, Malik A, Dutta A. tRFs在转录后以dicer独立的方式调控基因表达。Rna。2018;24(8):1093 - 105。
- 141.
Hackenberg M,黄普杰,黄春春,史宝杰,Gustafson P, Langridge P.大麦缺磷和充足磷条件下microrna和其他非编码小rna的综合表达谱。DNA res 2013;20(2): 109-25。
- 142.
谢立龙,林思,施爱平,陈建伟,林文文,曾春春,等。揭示小rna介导的磷酸盐缺乏反应拟南芥通过深度测序。植物科学进展。2009;32(4):344 - 344。
- 143.
叶荣,陈震,毕林,Jordan Rowley M,夏娜,柴静,等。拟南芥中直接DNA甲基化的siRNAs生物发生的dicer独立途径。中国生物医学工程学报,2016;36(2):344 - 344。
- 144.
谢敏,于斌。sirna介导的植物DNA甲基化。中国生物医学工程学报。2015;16(1):23-31。
- 145.
王忠,王峰,洪艳,黄娟,石辉,朱建科。两种叶绿体蛋白通过影响保护细胞中ROS的产生来抑制抗旱性。中国生物医学工程学报,2016;29(4):344 - 344。
- 146.
Afgan E, Baker D, van den Beek M, Blankenberg D, Bouvier D,切赫M,等。用于可访问、可重复和协作的生物医学分析的银河平台:2016年更新。核酸研究,2016;44(W1): W3-W10。
- 147.
程春春,陈ap, Thibaud-Nissen F, Schobel S, Town CD.拟南芥参考基因组的完整重标注。中国生物医学工程学报,2017;29(4):369 - 369。
- 148.
Axtell乔丹。ShortStack:小RNA基因的综合注释和定量。Rna。2013;19(6):740 - 51。
- 149.
克莱默曼氏金融。miRNAs的茎环RT-qPCR。:分子生物学的最新协议;2011.第十五章:第十五单元
- 150.
Livak KJ, Schmittgen TD。使用实时定量PCR和2(−Delta Delta C(T))方法分析相关基因表达数据。方法。2001;25(4):402 - 8。
- 151.
金J,田F,杨,孟YQ,香港L,罗J,等。PlantTFDB 4.0:朝着植物转录因子和调控相互作用的中心枢纽。核酸研究,2017;45(D1): D1040-5。
- 152.
Rashmi KV, Gilad-Bachrach R. DART: dropout满足多个加性回归树。2015.可于http://www.arxivorg/pdf/150501866v1.
- 153.
Yilmaz A, Mejia-Guerra MK, Kurz K, Liang X, Welch L, Grotewold E. AGRIS:拟南芥基因调控信息服务器,最新进展。核酸决议2011;39(数据库issue): D1118-22。
确认
我们感谢Martin Simon对sRNA图书馆准备工作的技术支持和建议,以及Oguz Top对手稿的有益评论。
资金
研究经费由德国研究基金会提供(SFB-TRR 175,资助W.F.项目C03, T.M.项目D02和T.K.项目C01)。资助机构没有参与研究的设计、数据的分析或解释,也没有参与手稿的撰写。
作者信息
从属关系
贡献
WF和MAA设计了研究;BT在MAA和KH的帮助下进行了研究;BT、MAA、KH和WF对数据进行分析;AGM和TK提供了3 h和2 d mRNA/lncRNA mRNA测序原始数据;用HLW和TM构建miRNA-TF网络;BT、MAA、HLW和WF撰写了论文。作者们阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1表S1
:总mRNA测序读数映射到答:芥对照和冷处理样品(生物三拷贝)经转接头修整后的参考基因组。表S2:总sRNA测序读取映射到不同sRNA产生RNA类别的对照和冷处理样品。表S3: sRNA的大小分布,单位为reads / million。转接头修整后,对照和冷处理样品中总sRNAs的尺寸分布。
附加文件2表S4:
冷驯化过程中差异表达的miRNAs。这三个子表分别描述了在3 h、6 h和2 d时的DE mirna。橙色突出显示的miRNA属于进化保守的miRNA家族。表S5:冷驯化过程中所有mirna的归一化读计数和折叠变化。这三个子表分别描述了在3 h、6 h和2 d时的所有mirna。表S6:差异表达的冷响应mirna答:芥.低温处理3 h、6 h和2 d后miRNAs的折叠变化,当log2FC≥1&≤−1时,认为是DE, Benjamini-Hochberg校正p-value≤0.05时,保守的mirna以粗体显示。
附加文件3表S7:
使用psRNATarget预测的mirna靶向mrna列表。子表描述了在三个时间点上所有预测的DE mirna靶点。使用严格的期望值2.5来筛选目标。N/A =没有明显的折叠变化表S8:使用psRNATarget预测的mirna靶向ncrna列表。子表描述了DE mirna在三个时间点的所有预测ncRNA靶点。使用严格的期望值2.5来筛选目标。N/A =没有明显的折叠变化。
附加文件4表S9:
从mRNA测序数据生成的所有mRNA的列表。这三个子表描述了对照和冷处理样品在3小时、6小时和2 d时的归一化读计数(三次)。表S10:从mRNA测序数据生成的所有重要DE mRNA的列表。子表显示了3 h、6 h和2 d后的对照和冷处理样品的所有细节。
附加文件5表S11:
来自所有四个亚群的54个差异表达mirna靶点的列表,这些靶点在所有三个时间点都一致存在。维恩图描述了在3小时、6小时和2天后观察到的差异表达miRNAs的所有靶标。
附加文件6表S12:
差异表达miRNAs预测靶点的基因本体术语富集分析。子表描述了冷驯化3 h、6 h和2 d后的GO项。
附加文件7。
可以使用免费软件GEPHI访问的数据文件https://gephi.org/包括数据S1:冷驯化中的基因调控网络,数据S2:差异表达miRNAs的冷响应网络。
附加文件8图S1:
冷驯化完整基因调节网络(GRN)。用于冷驯化的GRN概述。选择所有预测的低温miRNA靶点,并推断调控这些靶点的tf。顶点颜色表示各自的调节活动,边缘颜色标记与计算模块的关联。最大的模块被标记为它们最突出的功能组,这些功能组是使用本体丰富来标识的。
图S2:
由DE miRNAs的直接和间接靶点组成的冷响应基因调控网络。miRNAs和靶标在任何一个分析时间点上都有差异表达(FC≥2&≤- 2,Benjamini-Hochberg校正p值≤0.05)。与冷应力相关的功能模块;激酶信号;转录、翻译和转运分别用蓝色、深绿色、粉色和橙色表示。
图S3:
从完整网络中提取miR858a的子网。miRNAs的直接和间接靶标在至少一个分析时间点上有差异表达(FC≥2&≤- 2,Benjamini-Hochberg校正p值≤0.05)。
附加文件11
从完整网络中提取miR319b的子网。miRNAs的直接和间接靶标在至少一个分析时间点上有差异表达(FC≥2&≤- 2,Benjamini-Hochberg校正p值≤0.05)。
附加文件12表S13:
由非重叠lncRNAs产生的差异表达sRNAs。子表描述了详细的sRNA和lncRNA转录物在3 h、6 h和2 d的测序数据。表S14系列:产生的差异表达sRNAs独联体nat对。子表描述了详细的sRNA和独联体- 3 h, 6 h和2 d的nat测序数据。表S15:产生的差异表达sRNAs反式nat对。子表描述了详细的sRNA和反式- 3 h, 6 h和2 d的nat测序数据。表S16:产生的差异表达sRNAspha对。子表描述了详细的sRNA和pha3 h、6 h和2 d的转录本测序数据。
附加文件13表S17:
在Lee et al. 2005中发现的经典冷反应基因的差异表达列表,在我们的研究中也是差异表达。
附加文件14表S18:
本研究中使用的寡核苷酸序列进行茎环qRT-PCR。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
蒂瓦里,B.,哈伯曼,K.,阿里夫,硕士et al。低温驯化过程中小rna的鉴定拟南芥.BMC植物生物学20.298(2020)。https://doi.org/10.1186/s12870-020-02511-3
收到了:
接受:
发表:
关键字
- 拟南芥
- 低温驯化
- 小的非编码RNA
- 基因调控
- RNA序列
- mirna转录因子网络