摘要
背景
红薯(番薯甘薯(左.蓝莓(Lam.)是一种高度杂合的自六倍体作物,产量高,花青素含量高。紫甘薯是花青素的主要来源,贮藏根中花青素的生物合成机制尚未完全阐明。
结果
为了揭示甘薯贮藏根中花青素生物合成的机制,并鉴定新的参与花青素生物合成的同源基因,我们以宁子舒1号和吉子舒2号为亲本,构建了甘薯贮藏根中花青素生物合成的F1混合人口。选择7个含有花青素的品系和3个不含花青素的品系进行全长和第二代转录组分析。从全长转录组测序中共鉴定出598,375个循环一致测序reads。经二代转录组数据分析校正,共获得41356个转录本和18176个unigenes。通过对含花青素组和不含花青素组的比较分析,发现有2329个unigenes表达显著差异,其中1235个显著上调,1094个显著下调。GO富集分析表明,差异表达的ungenes在分子功能和生物学过程中均显著富集。KEGG富集分析表明,上调的unigenes在黄酮类生物合成途径和苯丙类生物合成途径中显著富集,下调的unigenes在植物激素信号转导途径中显著富集。差异表达单基因加权共表达网络分析显示,花青素生物合成基因在转录水平上与转录因子MYB、bHLH和WRKY共表达。
结论
本研究将在转录组水平上揭示甘薯贮藏根系花青素生物合成的调控机制。
背景
甘薯是一种高度杂合的六倍体,是一种重要的粮食作物,在世界各地种植面积达920万公顷(粮农组织,2017年)。甘薯具有产量高、耐瘠薄、适用性广的特点[1].紫色红薯含有花青素。花青素是植物类黄酮二级代谢途径的产物,已被应用为天然水溶性色素[2].研究表明,花青素具有抗氧化活性,对人类多种疾病有预防作用[3.].紫甘薯花青素具有较高的光稳定性和热稳定性[4],使其成为提取花青素的优良原料。
植物花青素的生物合成机制已被广泛研究[5,6].花青素生物合成以苯丙氨酸为底物,采用苯丙氨酸解氨酶(PAL)、肉桂酸4-羟化酶(C4H)、肉桂酸4-羟化酶(C4L)、查尔酮合成酶(CHS)、查尔酮异构酶(CHI)、黄酮3-羟化酶(F3H)、类黄酮3 ' -羟化酶(F3 ' h)、类黄酮3 ' - 5 ' -羟化酶(F3 ' 5 ' -h)、二氢黄酮醇还原酶(DFR)、白花青素双加氧酶(LDOX)、花青素合成酶(ANS)、和葡萄糖黄酮3-o-糖基转移酶(UF3GT) [7].编码这些酶的基因主要受MYB-bHLH- wdr (MBW)复合物的转录调控,该复合物由MYB、bHLH和WD40组成[8].参与调控花青素生物合成基因的转录因子还包括WRKY [9], bZIP [10],及NAC [11)等。研究表明,miRNAs还参与植物花青素生物合成的调控[12,13].调控花青素生物合成中结构基因表达的转录因子受到激素、糖和光等生物和环境因素的不同调控[14].这些研究表明,植物花青素的生物合成受到多种因素的调控。
在甘薯贮藏根中花青素的转录组分析方面进行了三项研究。谢(15]比较了京疏6号和广疏87号的两组rna序列数据,发现udp -葡萄糖-黄酮3-O-糖基转移酶是花青素生物合成途径中的关键酶之一。马(16]和赵[17]通过对甘薯突变体的转录组分析,发现了许多与花青素生物合成相关的结构基因和转录因子。
本研究以花青素含量低的宁子树1号为母本,以花青素含量高的吉子树2号为父本构建F1混合种群。从杂交群体中筛选出7个含花青素系和3个不含花青素系进行转录组测序。本研究旨在通过对甘薯贮藏根全长转录组数据的比较分析,揭示甘薯贮藏根花青素生物合成的机制,并鉴定新的参与甘薯贮藏根花青素生物合成的同源基因。
结果
花青素含量测定
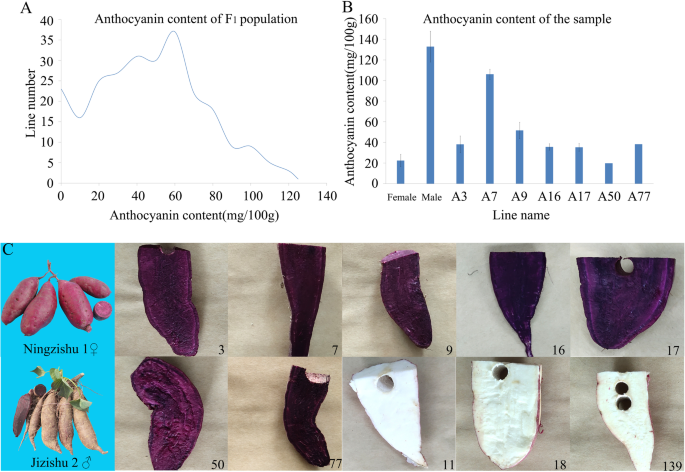
我们连续三年每年从两个地点收集种群线,测定花青素含量(图。1a).在F中1群体中,花青素含量最高的品系为125 mg/100 g鲜重,部分品系不含花青素。
甘薯贮藏根的mRNA全长测序
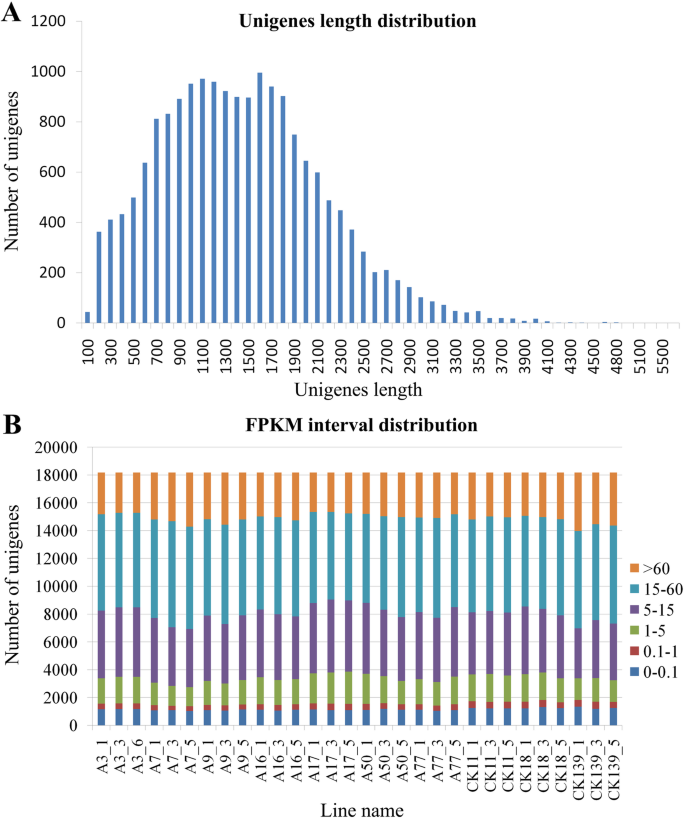
从甘薯发育贮藏根中筛选出7个含花青素品系和3个不含花青素品系1杂交群体(图;1从每一株系中选取9个独立个体,平均分成3个样本进行总RNA提取和第二代转录组测序。每个样品获得的洁净数据不少于8G。同时,从每个样本中混合等量的总RNA,进行第三代转录组文库构建(single-molecule real-time, SMRT)和测序分析。共获得598,375条循环一致性测序(CCS) reads,长度从50 bp到14,913 bp,平均长度为1234 bp,包括459,960条全长非嵌合(FLNC)序列和121,847条非全长非嵌合(NFL)序列。全长非嵌合序列长度为50 bp ~ 13622 bp。经二代转录组数据校正后,得到41356个转录本,长度为54 ~ 5567 bp,平均长度为1200 bp,其中优质转录本41027个,低质转录本329个。重复数据删除得到18176个unigenes,长度为55 ~ 5567 bp,平均长度为1393 bp。unigenes长度分布如图所示。2a. unigenes的FPKM区间分布如图所示。2b.对转录组数据进行Pearson分析,发现每个系的3个重复具有良好的一致性,满足后续分析的要求(附加文件1:表S1)。
甘薯全长unigenes的功能注释
我们根据七个数据库对得到的unigenes进行了标注,结果如表所示1.95.64%的ungenes至少与一个数据库进行了比对,这为后续差异表达基因的分析提供了基础。GO (Gene Ontology)富集分析通过国际标准化的分类系统解密基因功能[18].GO富集分析发现,10483个unigenes在生物过程、细胞成分和分子功能上均显著富集。其中,生物过程是GO术语中最多的。在生物过程中,大多数GO术语被分配到代谢过程(5037个unigenes),细胞过程(4925个unigenes)和单生物过程(3507个unigenes)。在细胞成分中,大多数GO术语被分配给细胞(2305个unigenes),细胞部分(2305个unigenes)和细胞器(1706个unigenes)。在分子功能中,大多数GO术语是结合(5836个unigenes)和催化活性(4600个unigenes)(附加文件)2:图S1)。KOG (euKaryotic Ortholog Groups)富集分析揭示了真核生物基因的保守核心和与真核生物基因组进化相关的多样化[19].KOG分析表明,10407个unigenes分布在25个功能簇上。如附加文件所示3.:图S2中,4个最大的类别分别是仅一般功能预测(1626个)、翻译后修饰、蛋白质翻转、伙伴(1398个)、信号转导机制(1010个)和翻译、核糖体结构和生物发生(916个)。KEGG(京都基因和基因组百科全书)富集分析可用于识别功能基因,了解基因在生物合成途径中的功能和相互作用[20.].在KEGG注释中,在KEGG数据库中注释了16,026个unigenes(附加文件4:图S3)。最大的途径是翻译途径,包含881个unigenes。第二和第三通路分别为信号转导通路和碳酸代谢通路,分别含有762个和684个unigenes。
含花青素系与不含花青素系差异表达单基因的分析
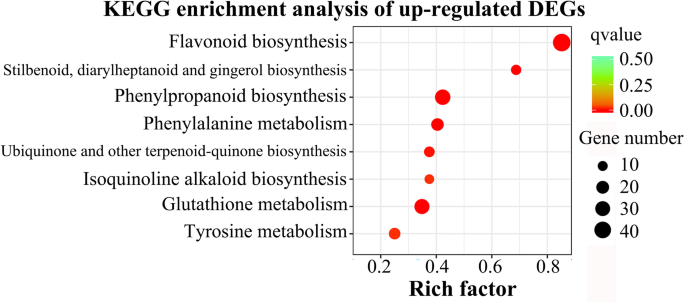
基因控制表型,基因的差异表达使得个体之间的表型不同[21].我们将含有花青素的品系作为实验组(Anth)和不含花青素的品系作为对照组(CK)的转录组数据进行比较,发现2329个unigenes在这两组之间表达有显著差异。其中上调基因1235个,下调基因1094个(附加文件)5:图S4)。GO富集分析发现,差异表达的ungenes在分子功能和生物学过程中均显著富集。在分子功能类别中,大多数GO术语被分配到催化活性(735个unigenes)和氧化还原酶活性(194个unigenes)。对于生物过程类别,分配的是单一生物代谢过程(314个unigenes),氧化还原过程(183个unigenes)和碳水化合物代谢过程(104个unigenes)(附加文件)6:图S5)。KEGG富集分析发现,上调的unigenes在黄酮类生物合成途径(46个)、苯丙类生物合成途径(33个)和谷胱甘肽代谢途径(31个)中显著富集(图)。3.),下调的unigenes在植物激素信号转导通路中显著富集(24个unigenes)(附加文件)7:图S6)。
与花青素生物合成相关的差异表达单基因
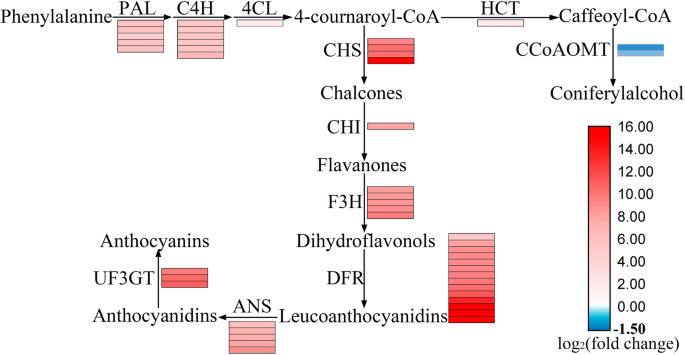
我们从转录组数据中鉴定出54个富集在类黄酮生物合成途径中的unigenes和81个富集在苯丙类生物合成途径中的unigenes。对两组(含花青素系和不含花青素系)差异表达的unigenes进行分析发现,黄酮生物合成途径中有49个差异表达的unigenes,其中46个上调,3个下调。苯丙烷生物合成途径中有38个ungenes差异表达,其中33个上调表达,5个下调表达(附加文件)8:表S2)。对差异表达的unigenes进行分析,发现54个unigenes表达上调(log2(fold change) > 2, qvalue < 0.01)参与花青素的生物合成。这些基因包括苯丙氨酸解氨酶(朋友)、反式肉桂酸4-单加氧酶(C4H), 4-香豆酸酯:辅酶a连接酶(4 cl)、查尔酮合酶(CHS)、查尔酮-黄酮异构酶(孩子)、黄酮3-羟化酶(F3H)、二氢黄酮醇-4还原酶(DFR)、白花色素双加氧酶(LDOX /答)、花青素3- o -葡萄糖苷2- o -木糖基转移酶(UF3GT)、udp -糖基转移酶78D2 (UGT78D2)(图。4和附加文件8:表S2)。其中包括大部分参与苯丙氨酸生物合成花青素的基因。
差异表达的转录因子
转录因子(Transcription factors, TFs)是一种反式作用因子,可与基因启动子区结合,调节基因的时空表达[22].我们使用iTAK软件预测植物转录因子[23].共发现29种828种转录因子(附加文件9:表S3)。其中,C3H(63个unigenes)、bZIP(55个unigenes)和bHLH(53个unigenes)是最大的3个类型。bHLH转录组因子参与调控花青素生物合成[8].此外,还发现了20个可能参与花青素生物合成的MYB转录因子。转录因子基因的表达水平决定了其对照基因的表达水平。通过两组(Anth和CK)比较,共发现165个转录因子基因存在差异表达,其中55个显著上调,110个显著下调(附加文件)10:图S7和附加文件11:表S4)。
激素信号转导途径中差异表达的unigenes
植物激素调节植物生长发育和次生生物量合成[14].通过两组(Anth和CK)比较,共发现33个激素信号转导通路unigenes存在差异表达,其中9个表达显著上调,24个表达显著下调(附加文件)12:表S5)。下调的unigenes在生长素、细胞分裂素、油菜素内酯、脱落酸(ABA)、赤霉素酸(GA)和茉莉酸(JA)等信号转导途径中显著富集(图)。3.b).上调基因经KEGG分析无显著富集。
差异表达的lncRNAs
植物长链非编码rna (Plant long noncoding RNAs, lncRNAs)参与植物发育、胁迫响应和次生代谢等生物过程的调控[24].通过CPC、CNCI、plek和pfam软件共预测了2054个lncrna,长度为200 ~ 3356 bp。通过两组(Anth和CK)比较,共发现228个差异表达,其中110个显著上调,118个显著下调(附加文件)13:表S6)。
差异表达ungenes加权共表达网络分析(WGCNA)
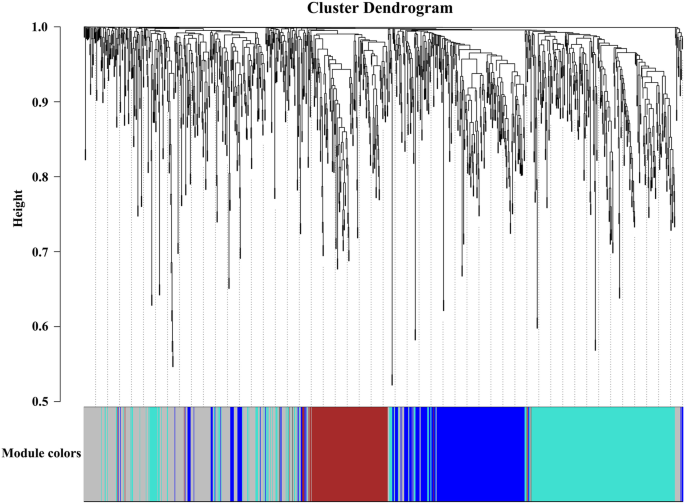
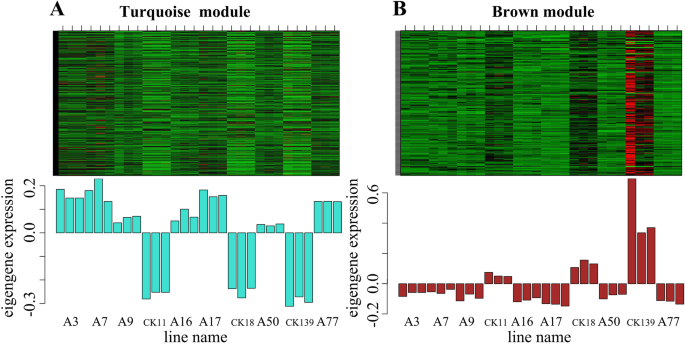
功能相似的基因具有相似的表达模式。WGCNA可用于发现参与特定过程的新基因[25].在此基础上,我们对两组(Anth和CK)的差异表达单基因进行了无向网络WGCNA。样品的聚类分析显示,含花青素组(Anth)和无花青素组(CK)明显分为两类(附加文件14:图S8)。差异表达的unigenes聚集成4个模块,分别用蓝绿色、灰色、棕色和蓝色表示(图2)。5).模块与性状的相关分析表明,绿松石模块与花青素组(Anth)显著相关(图2)。6a和附加文件15:图S9),棕色模块与无花青素组(CK)显著相关(图S9)。6b和附加文件15:图S9)。在绿松石模块中,GO富集分析显示,在分子功能类中,unigenes主要参与催化活性和氧化还原酶活性,在生物过程类中,unigenes主要参与氧化还原过程(附加文件16:图S10)。KEGG富集分析表明,unigenes在黄酮类生物合成、苯丙氨酸代谢、苯丙氨酸生物合成和谷胱甘肽代谢中均显著富集(附加文件17:图S11)。在brown模块中,GO和KEGG富集分析没有发现显著的基因富集(附加文件18:图S12)。
qRT-PCR验证差异表达的unigenes
根据转录组数据,我们在含花青素品系和不含花青素品系中随机选取12个unigenes进行qRT-PCR验证。qRT-PCR引物在附加文件中列出19:表S7。12个unigenes的qRT-PCR结果和FPKM见附加文件20.:图S13,与转录组数据一致(r2= 0.9060),说明转录组数据可靠。
讨论
甘薯是一种高度杂合的六倍体(2n = 6x = 90)作物,具有复杂的遗传背景。在此条件下,通过转录组分析研究甘薯贮藏根花青素生物合成的机制,可以用背景差异表达基因掩盖花青素相关差异表达基因。本研究以宁子舒1号和吉子舒2号为亲本进行有性杂交,在F . F .中筛选出7个含花青素系和3个无花青素系1杂交群体为实验材料。用DEseq方法对两组(Anth和CK)进行全长转录组分析,qvalue < 0.05。这样可以有效去除实验组(Anth)和对照组(CK)之间的背景差异表达基因,从而找到真正的花青素相关差异表达基因。在植物中,花青素是通过类黄酮生物合成途径生物合成的,上游途径是苯丙类生物合成。KEGG分析显示,上调的差异表达unigenes在这两条生物合成途径中显著富集(图2)。3.A),这也是富集的两个最丰富的信号通路。这表明我们的实验结果可以揭示甘薯贮藏根中花青素的生物合成机制。
与第二代测序相比,全长转录组测序在识别同源基因方面更加准确。我们利用PacBio后续平台单分子实时进行了第三代全长转录组测序,获得了18176个高质量unigenes。到目前为止,已有3篇关于甘薯贮藏根花青素生物合成的转录组文章。谢(15]比较了京薯6号(27.02 mg/100 g FW)和黄甘薯广薯87的两组rna序列数据,发现udp -葡萄糖-类黄酮3-O-糖基转移酶是花青素生物合成的关键酶之一。马(16]和赵[17]通过对甘薯突变体的转录组分析,发现了许多与花青素生物合成相关的结构基因和转录因子。马所用甘薯为宁子舒1号(22.41 mg/100 g FW)及其白色果肉突变体。赵所用的甘薯为京薯6号(27.02 mg/100 g FW)及其突变体JS6-5 (79.80 mg/100 g FW),花青素含量高。在本研究中,我们选择了含花青素系和不含花青素系1宁子树1号和吉子树2号进行转录组比较分析,发现花青素生物合成基因表达上调。参与苯丙氨酸向4- cournaryl - coa转化过程的PAL和C4H上分别有5个和6个ungenes表达上调。LDOX/ANS是一种依赖于2-氧谷氨酸的加氧酶,可将白花青素转化为花青素,是花青素生物合成中有色代谢物形成的重要步骤[26].在本研究中,我们发现5个经LDOX/ANS标记的unigenes表达上调。UF3GT作为花青素产生的主要控制点,将葡萄糖转移到花青素的C-3羟基上[27],从而产生花青素- 3- o -葡萄糖苷[28].这是植物花青素稳定性和水溶性的关键步骤[29].在本研究中,我们发现3个经UG3GT标记的unigenes表达上调。将我们的结果与宁子树1号突变体的结果进行比较[16],我们发现了更多与花青素生物合成相关的同源单基因。我们推测,与第二代测序相比,全长转录组测序可以识别更多的同源基因序列。
植物花青素的生物合成受一个由MYB-bHLH-WD40三元复合物组成的调控网络[30.].调控基因包括MYB75(IbMYB1transcript18862),MYB114(transcript13511),bHLH2/TT8(transcript2642)。AtMYB75作为一种转录激活剂,通过调控花青素和原花青素的表达,促进花青素和原花青素的生物合成CHS,DFR而且LDOX[31,32].IbMYB1控制甘薯贮藏根果肉花青素的生物合成[33].AtMYB114作为MYB转录激活剂,影响花青素生物合成后期步骤中涉及的酶的表达[34].AtTT8作为一种helix-loop-helix (bHLH2)转录激活因子,通过调控二氢黄酮醇-4-还原酶(DFR) [34].这些编码上述基因的unigenes在含花青素的品系中表达上调。在这项研究中,我们发现了12个差异表达的ungenes,编码8个MYB基因和17个差异表达的ungenes编码9bHLH基因。更少的MYB而且bHLH与宁子树1号突变体中花青素生物合成相关基因的鉴定比较16].我们推测在群体中进行多系比较可以有效去除背景差异表达基因。这些结果表明,我们的实验可以更准确地反映甘薯贮藏根中花青素的生物合成机制。
此外,WRKY的一些成员[9], MADs [35], nac [11], ap2 / erf [36], bZIP [10据报道,这些家族参与了花青素生物合成的调节。在本研究中,我们发现在含花青素组(Anth)和无花青素组(CK)中,有55个转录因子上调,110个转录因子下调(附加文件)11:表S4)。WGCNA分析发现,在绿松石模块中,共有10个上调的unigenes编码7个转录因子,9个下调的unigenes编码9个转录因子(附加文件)11:表S4)。有2个编码WRKY转录因子的unigenes (transcript7517和11522)在含花青素的品系中上调表达。其中2个编码NF-YA10转录因子的单基因(transcript9883和transcript11323), 1个编码B-box型锌指蛋白的单基因(transcript31941), 1个编码GRAS家族转录因子的单基因(transcript4453), 1个编码锌指的单基因(transcript12965), 1个编码MADS-box蛋白的单基因(transcript22607)在含花青素品系中下调表达。在brown模块中,共有12个下调的unigenes,编码11个转录因子,没有发现上调的unigenes编码转录因子(附加文件11:表S4)。含花青素组和不含花青素组转录因子表达差异表明,甘薯贮藏根中花青素的生物合成在转录水平上受到调控。本研究结果为发现新的调控花青素生物合成的转录因子提供了基础。
为了研究激素在花青素生物合成中的潜在作用,我们对植物激素信号转导相关基因的表达进行了分析。在GA信号转导途径中,GA与其受体GID1结合,通过泛素-蛋白酶体途径导致DELLAs降解[37].DELLA蛋白通过隔离MYB/bHLH/WD40复合体的JAZ抑制子和随后的PAP1和TT8的释放促进花青素的生物合成拟南芥[38].转录组分析显示,该基因编码一个单基因(transcript14390)GID1表达明显下调。而单基因(transcript16804)编码的表达JAZ没有明显变化。这些unigenes (transcript2642和transcript2950)编码TT8在含花青素的品系中表达均显著上调。基于这些结果,我们推测通过降低的表达GID1在甘薯中,GA解除了对花青素合成的抑制,促进了花青素合成。然而,负调控JAZ甘薯花青素合成基因可能不在转录组水平。
研究表明,许多lncrna具有保守的二级结构和剪接形式[39],并在特定的组织、细胞和发育阶段受到调节[40].功能分析表明lncRNAs在转录调控基因表达[41],后转录[42],以及表观遗传水平[43].全长转录组分析共鉴定出2054个lncrna。通过组间比较,我们发现110个上调的lncrna和118个下调的lncrna(附加文件)13:表S6)。WGCNA分析显示,绿松石模块中有24个lncrna表达上调,11个lncrna表达下调。基于这些结果,我们推测lncRNAs在甘薯贮藏根花青素生物合成中起调控作用。
结论
通过对甘薯F .的转录组测序分析,共鉴定出18176个甘薯单基因1混合人口。在我们的研究中,与之前的转录组研究相比,我们发现了更多参与甘薯花青素生物合成的同源基因。通过比较7个含花青素株系和3个不含花青素株系的全长转录组1结合WGCNA分析,我们发现除了MBW调控复合物(MYB、bHLH、WD40)外,WRKY、NF-YA10、GRAS、MADS-box等转录因子也参与了花青素生物合成的调控。在转录组水平上,激素和lncrna可能参与了花青素生物合成的调控。
方法
植物材料
以江苏省农业科学院选育甘薯品种宁子舒1号(22.41 mg/100 g FW)和山东省农业科学院作物研究所选育甘薯品种吉子舒2号(132.95 mg/100 g FW)分别为母本和父本。在暗室中对这两个亲本进行短时间光照(光照8小时/暗16小时)诱导开花,然后进行人工授粉和采集种子。共获得256个分离性状系。
7个含花青素品系和3个无花青素品系移栽后第90天收获贮藏根。收集中等贮藏根中部直径0.5 cm的圆柱体,快速冷冻于液氮中进行转录组测序。剩余物用来测定花青素含量。
花青素的测定
采用酶联免疫吸附法(ELISA)测定甘薯贮藏根中花青素含量。44],每系不少于6个独立的生物重复。
RNA分离及全cDNA文库构建
总rna用RNAprep Pure Plant Plus Kit(多糖多酚富)(天根生物科技(北京)有限公司)提取。将提取的总RNA进行琼脂糖凝胶电泳,分析RNA降解情况和污染程度。RNA纯度用Nanodrop分析。RNA浓度用Qubit测量。RNA完整性用Agilent 2100进行分析。
全文cDNA文库构建方法如下:(1)利用oligo (dT)富集含polyA的mRNA;(2)利用smart PCR cDNA Synthesis Kit将mRNA逆转录为cDNA;(3) PCR扩增富集cDNA;(4)利用磁珠筛选片段进行大规模PCR,获得足够的总cdna;(5)损伤修复、末端修复全长cDNA,连接SMRT哑铃形适配器,构建全长转录组文库;(6)用外切酶消化,去除cDNA两端未连接的连接子序列;(7)最终,引物与DNA聚合酶结合形成一个完整的SMRT钟形文库。按照秦[[]描述的方法进行第二代转录组RNA分离和文库构建。45].
测序,鉴定,标注
在一个细胞中使用PacBio Sequel平台对合格的完整cDNA文库进行测序。使用Hiseq 2000测序平台(Illumina)和PE150对第二代文库进行测序。使用SMRTlink 7.0软件处理完整的cDNA序列,并使用LoRDEC软件对第二代转录数据进行校正,以获得一致的unigenes。用CD-HIT软件去除冗余unigenes。用DESeq R包(1.10.1)进行含花青素品系与不含花青素品系的差异表达基因分析,qvalue < 0.05。对于花青素生物合成基因,我们选择的标准是对数2(折叠变化)> 2,qvalue < 0.01。通过检索NCBI核苷酸序列(Nt)、NCBI非冗余蛋白(NR)、基因本论(GO)、京都基因与基因组百科全书(KEGG)、真核同源群(KOG)、蛋白家族(Pfam)和瑞士- prot数据库(e-value = 1e-5)对unigenes进行注释。
LncRNA预测
我们使用PLEK [46], cnci [47], cpc2 [48],及Pfam [49]来预测unigenes的编码潜力。首先,利用PLEK和CNCI软件根据unigenes的序列特征对其编码潜能进行预测。其次,利用CPC2软件,根据转录本各编码帧的生物序列特征,评估unigenes的编码潜力。第三,将上述软件预测的unigenes序列通过hmmscan在Pfam数据库中进行同源搜索,最终得到lncRNA序列。
转录组数据的qRT-PCR验证
选择12个unigenen在含花青素品系和不含花青素品系中的表达。采用引物5设计unigenes qRT-PCR引物对。qRT-PCR使用Roche LightCycler®480II系统和TaKaRa SYBR Premix Ex Taq™(Tli RNaseH Plus)在以下条件下进行:95°C持续10秒,然后在95°C持续15秒,55°C持续15秒和72°C持续15秒的40个循环。将各基因的表达水平归一化至构成水平Ibactin表达式。qRT-PCR结果用2−△△CT方法(50].
数据和材料的可用性
在本研究中产生的所有数据都包含在论文和支持信息文件中。转录组数据已提交给NCBI SRA (PRJNA608140),https://www.ncbi.nlm.nih.gov/sra/?term=PRJNA608140.宁子书1号由江苏省农业科学院育种所保存。“鸡子树2号”及用于转录组测序的株系由通讯作者所在的山东省农业科学院作物研究所育种所保存。
缩写
- CCS技术:
-
循环共识排序
- FLNC:
-
全身non-chimeric
- NFL:
-
Non-full-length non-chimeric
- SMRT:
-
单分子实时
- Nt:
-
核苷酸序列
- NR:
-
NCBI非冗余蛋白
- 走:
-
基因本体论
- KEGG:
-
京都基因与基因组百科全书
- KOG:
-
真核正正交基
- 包含了:
-
蛋白家族
- ELISA:
-
酶联免疫吸附试验
- CK:
-
对照组
- 尖刺外壳:
-
实验组
- 弗兰克-威廉姆斯:
-
鲜重
- TFs:
-
转录因子
- 阿坝:
-
脱落酸
- 遗传算法:
-
赤霉酸
- 是:
-
茉莉酸
- lncRNAs:
-
长非编码rna
- WGCNA:
-
加权基因共表达网络分析
- 朋友:
-
苯丙氨酸ammonialyase
- C4H:
-
Trans-cinnamate 4-monooxygenase
- 4 cl:
-
4-Coumarate:辅酶a连接酶
- CHS:
-
查耳酮合酶
- 孩子:
-
Chalcone-flavanone异构酶
- F3H:
-
黄烷酮3-hydroxylase
- DFR:
-
Dihydroflavonol-4-reductase
- LDOX /答:
-
Leucoanthocyanidin加双氧酶
- UF3GT:
-
花青素3- o -葡萄糖苷2- o -木糖基转移酶
- UGT78D2:
-
78年UDP-glucosyltransferase d2
参考文献
红薯:回顾它的过去,现在和未来在人类营养中的作用。Adv Food Nutr res 2007; 52:1-59。
霍尔顿TA,康沃尔EC花青素生物合成的遗传学与生物化学。植物科学进展。1995;7(7):1071-83。
松井T, Ebuchi S,小林M,福井K,杉田K,寺原N,等。从番薯品种山药中提取的二酰化花青素可以通过抑制-葡萄糖苷酶来达到抗高血糖的作用。食品化学学报。2002;50(25):7244-8。
安第斯紫玉米和红肉甘薯花青素基水浸提物与合成和天然着色剂的稳定性。食品化学,2004;86(1):69-77。
Dooner HK, Robbins TP, Jorgensen RA。花青素生物合成的遗传与发育控制。《热内年鉴》1991;25:173-99。
寇敏,刘玉军,李志颖,张永刚,唐伟,闫华,等。甘薯中一种新的谷胱甘肽s转移酶基因IbGSTF4参与了花青素的分离。植物物理与生物化学。2019;135:395-403。
水果花青素生物合成调控的新认识。植物科学进展,2013;18(9):477-83。
Ramsay NA, Glover BJ。MYB-bHLH-WD40蛋白复合物与细胞多样性的进化。植物科学进展,2005;10(2):63-70。
Lloyd A, Brockman A, Aguirre L, Campbell A, Bean A, Cantero A,等。MYB - bhlh - wd重复(MBW)色素调控模型的研究进展:添加WRKY因子和增选花青素MYB调控甜菜素。中国生物医学工程学报,2017;29(4):344 - 344。
安建平,姚建峰,徐瑞仁,游彩霞,王晓峰,郝玉军。苹果bZIP转录因子MdbZIP44调控脱落酸促进花青素积累。植物细胞环境学报,2018;41(11):2678-92。
周华,柯林旺,王华,顾晨,Dare AP, Espley RV,等。血红桃的分子遗传学揭示了NAC转录因子对花青素生物合成的激活作用。中国植物学报,2015;29(1):344 - 344。
何玲,唐睿,石旭,王伟,曹强,刘旭,等。通过甘薯块茎根小RNA和降解组测序,揭示花青素生物合成相关microRNAs及其靶基因。BMC植物生物学,2019;19(1):232。
郭建勇,Felippes FF,刘佳杰,Weigel D,王继伟。mir156靶向SPL转录因子对拟南芥花青素生物合成的负调控植物学报。2011;23(4):1512-22。
Das PK, Shin DH, Choi SB, Park YI。花青素生物合成中的糖-激素串扰。生物化学学报。2012;34(6):501-7。
谢峰,Burklew CE,杨勇,刘敏,肖鹏,张波,等。紫甘薯(Impomoea batatas L.)转录组从头测序和综合分析。足底。2012;236(1):101 - 13所示。
马萍,边旭,贾震,郭霞,谢艳。紫甘薯(Ipomoea batatas L.)突变体转录组的从头测序与综合分析。基因。2016;575(2 Pt 3): 641-9。
赵华,张松,王峰,赵楠,何松,刘强,等。紫肉甘薯的转录组比较分析为花青素生物合成的分子机制提供了新的思路。农学前缘,2018;5(2):214-25。
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM,等。基因本体论:生物学统一的工具。中国科学,2000;25(1):25 - 9。
Koonin EV, Fedorova ND, Jackson JD, Jacobs AR, Krylov DM, Makarova KS,等。完整真核生物基因组中编码的蛋白质的全面进化分类。中国生物医学工程学报,2004;5(2):R7。
KEGG:京都基因和基因组百科全书。核酸研究,2000;28(1):27-30。
王磊,冯志,王霞,张霞。一种从RNA-seq数据中识别差异表达基因的R包。生物信息学,2010,26(1):136 - 8。
Dubos C, Stracke R, Grotewold E, Weisshaar B, Martin C, Lepiniec L.拟南芥MYB转录因子研究。植物科学进展,2010;15(10):573-81。
郑勇,焦超,孙华,Rosli HG, Pombo MA,张萍,等。植物转录因子、转录调节因子和蛋白激酶全基因组预测和分类的iTAK程序。植物学报。2016;9(12):1667-70。
张永春,陈永强。长链非编码rna:植物发育中的新调控因子。生物化学学报。2013;36(2):111-4。
张斌,张志强,张志强,等。加权基因共表达网络分析的总体框架。中华生物医学杂志2005;4:1。
中岛J,田中Y,山崎M,齐藤K.花青素生物合成中着色的关键反应——白花青素合成花青素3-葡萄糖苷的反应机理。中国生物医学工程学报,2001;26(4):344 - 344。
王华,王超,范伟,杨杰,Appelhagen I,吴勇,等。一种新的糖基转移酶催化紫甘薯中葡萄糖向糖基化花青素的转移。应用物理学报,2018;69(22):5444-59。
赵志才,胡国光,胡光峰,王华辉,杨志智,赖斌。UDP葡萄糖:黄酮-3- o -葡萄糖基转移酶(UFGT)基因调控荔枝(litchi chinesis Sonn.)果实着色过程中花青素生物合成。Mol生物学杂志2012;39(6):6409-15。
吉原N, Imayama T, Fukuchi-Mizutani M, Okuhara H, Tanaka Y, Ino I,等。荷兰鸢尾花色素苷3- o -葡萄糖转移酶cDNA的克隆与鉴定。植物科学进展,2005;26(3):496-501。
Schaart JG, Dubos C, Romero De La Fuente I, van Houwelingen AM, De Vos RC, Jonker HH,等。草莓果实原花青素合成调控复合物MYB-bHLH-WD40的鉴定与鉴定中国农业科学,2013;27(2):454-67。
赵丽娟,杨晓明,杨晓明,杨晓明。MYB基因在苯丙类生物合成中的调控作用。植物科学进展。2000;12(12):2383-94。
滕斌,李志强,李志强,李志强,等。拟南芥MYB75/PAP1基因对拟南芥花青素合成的影响。植物科学进展。2005;39(4):344 - 344。
马野浩,小浅原F,佐藤K,高高华,米诺贝。紫肉甘薯块根花青素合成调控基因的分离。植物科学进展。2007;43(3):344 - 344。
Zimmermann IM, Heim MA, Weisshaar B, Uhrig JF。拟南芥MYB转录因子与R/ b样BHLH蛋白相互作用的综合鉴定植物学报,2004;40(1):22-34。
Lalusin AG, Nishita K, Kim SH, Ohta M, Fujimura T.,甘薯(Ipomoea batatas (L.) lam)中一个新的MADS-box基因(IbMADS10)参与了花青素的积累。中国生物医学工程学报。2006;36(1):344 - 344。
于志祥,李建新,杨长青,胡伟力,王丽娟,陈晓宇。黄花蒿(Artemisia annua L. Mol)茉莉酸响应型AP2/ERF转录因子AaERF1和AaERF2正向调控青蒿素生物合成。2012;5(2):353-65。
孙春平,李志强。植物赤霉素信号转导的分子机制研究。植物学报2004;55:197-223。
谢艳,谭红,马忠,黄娟。DELLA蛋白通过隔离MYB/bHLH/WD40复合体MYBL2和JAZ抑制子促进拟南芥花青素合成。植物学报。2016;9(5):711-21。
Dinger ME, Amaral PP, Mercer TR, Pang KC, Bruce SJ, Gardiner BB,等。长链非编码rna在小鼠胚胎干细胞多能性和分化中的作用。基因组学报,2008;18(9):1433-45。
Mercer TR, Dinger ME, Sunkin SM, Mehler MF, Mattick JS。长链非编码rna在小鼠大脑中的特异性表达。中国科学(d辑),2008;26(2):344 - 344。
张娟,Mujahid H,侯勇,Nallamilli BR,彭哲。植物长链ncRNAs:基因调控控制的新前沿。中国植物科学,2013;4(5):1038-45。
金德,Gerbasi VR, Sontheimer EJ。sirna的内部工作。生物化学学报。2008;31(3):309-12。
朱庆华,王明民。植物长链非编码rna的分子功能。基因(巴塞尔)。杂志2012;3(1):176 - 90。
李娟,任磊,高志,姜敏,刘勇,周林,等。结合转录组学和蛋白质组学分析,构建了光诱导茄子花青素生物合成的新模型。植物生态学报。2017;40(12):3069-87。
秦忠,李安,侯峰,王强,董松,张磊。两种甘薯基因型RNA-seq基因鉴定及类胡萝卜素生物合成分析。中国生物医学工程学报。2017;39(2):379 - 379。
李安,张娟,周哲。基于改进k-mer方案的长链非编码rna和信使rna预测工具。生物医学工程学报。2014;15(1):311。
孙林,罗华,卜东,赵刚,于凯,张超,等。利用序列内在组成对蛋白质编码和长非编码转录本进行分类。中国生物医学工程学报,2013;41(17):e166。
康永杰,杨德强,孔林,侯敏,孟YQ,魏林,等。CPC2:基于序列内在特征的快速准确编码势计算器。中国生物医学工程学报,2017;45(W1): W12-6。
芬恩·RD,科吉尔·P,埃伯哈特·RY,艾迪·SR,米斯特里·J,米切尔·AL,等。Pfam蛋白家族数据库:迈向更可持续的未来。核酸研究,2016;44(D1): D279-85。
Livak KJ, Schmittgen TD。使用实时定量PCR和2−ΔΔCT方法分析相关基因表达数据。方法。2001;25(4):402 - 8。
确认
不适用。
资金
国家重点研发计划项目(2018YFD1000700/2018YFD1000705-8)、国家重点研发计划项目(2019yfd1001300 / 2019yfd1001304 - 4)、中国甘薯农业科研体系(car -10- b07)、山东省马铃薯产业技术体系(SDAIT-16-04)、山东省农业科学院农业科技创新项目(CXGC2018E01)资助。资助机构只提供资金支持,不参与实验的设计、数据的收集、解释和分析以及手稿的起草。
作者信息
从属关系
贡献
ZQ, WQM和LMZ设计了研究。ZQ撰写了这篇论文,并与所有合著者进行了讨论。FYH, AXL SXD和CXH进行了研究。所有作者均已阅读并批准稿件。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:表S1。
样本人分析。
附加文件2:图S1。
unigenes的GO分类。
附加文件3:图S2。
unigenes的KOG分类。
附加文件4:图S3。
unigenes的KEGG分类。
附加文件5:图S4。
差异表达unigenes的火山图。
附加文件6:图S5。
富集GO的差异表达unigenes。
附加文件7:图S6。
下调DEGs的KEGG富集分析。
附加文件8:表S2。
与花青素生物合成相关的DEGs。
附加文件9:表S3。
转录因子统计。
附加文件10:图S7。
差异表达的转录因子。
附加文件11:表S4。
差异表达转录因子概述。
附加文件12:表S5。
差异表达激素unigenes概述。
附加文件13:表S6。
差异表达lncrna总结。
附加文件14:图S8。
WGCNA样本聚类。
附加文件15:图S9。
模块和样本的关系。
附加文件16:图S10。
绿松石模块中最丰富的GO术语。
附加文件17:图S11。
绿松石模块的KEGG富集分析。
附加文件18:图S12。
棕色模块的KEGG富集分析。
附加文件19:表S7。
qRT-PCR引物。
附加文件20:图S13。
差异表达基因qRT-PCR分析。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
秦,Z,侯,F,李,A。et al。基于杂交群体的全长转录组比较分析揭示了甘薯花青素生物合成的调控机制(番薯甘薯(l)Lam)。BMC植物生物学20.299(2020)。https://doi.org/10.1186/s12870-020-02513-1
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-020-02513-1
关键字
- 红薯
- 完整的转录组
- 花青素生物合成
- 转录因子
- 加权基因共表达网络分析