- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发布:gydF4y2Ba
Gentiaeae(龙叶酸)的塑料系统母孕学研究:广泛的基因树不间间断及其与体积基因进化率异质性的关系gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba20.gydF4y2Ba, 文章编号:gydF4y2Ba340gydF4y2Ba(gydF4y2Ba2020.gydF4y2Ba)gydF4y2Ba
抽象的gydF4y2Ba
背景gydF4y2Ba
在重建植物生命树的过程中,质体尺度的数据一直很普遍。然而,目前基于质体的系统发育学研究主要依赖于质体基因串联序列的最大似然推断,因此单个质体基因产生的系统发育不一致通常被忽视。此外,质体的结构和功能特征表明质体基因可能不是单一的位点进化,并具有不同的进化力量,但一个谱系内质体基因的遗传特征仍然缺乏研究。gydF4y2Ba
结果gydF4y2Ba
我们测序,并注明Gentianeae的10个质体序列。Phylogenomic分析产生内Gentianeae属间强大的关系。我们发现基因树的拓扑结构变化较大并显示,超过一半的基因,其中一个(gydF4y2BaATPB.gydF4y2Ba),是三种广泛使用的质体标记(gydF4y2Ba:gydF4y2Ba,gydF4y2BaATPB.gydF4y2Ba和gydF4y2BamatKgydF4y2Ba在龙胆科系统发育推断中,可能导致龙胆科系统发育的歧义。核苷酸取代率的估计显示,不同质体基因之间和不同功能基因组之间存在广泛的比率异质性。比较分析表明,龙胆科质体基因中核糖体蛋白(RPL和RPS)基因和RNA聚合酶(RPO)基因具有较高的替代率和遗传变异。我们的研究表明,只有一个(gydF4y2BamatKgydF4y2Ba)三(gydF4y2BamatK基因,ndhB位gydF4y2Ba和gydF4y2Ba:gydF4y2Ba)广泛使用的标记具有较高的系统发育信息值。由于高PI和最低的基因树不一致,gydF4y2BarpoC2gydF4y2Ba提倡作为有力的质体DNA条形码的Gentianeae的分类学研究。此外,我们的分析揭示了与质体基因的遗传变异进化率的正相关关系,但与基因树不一致下净化选择呈负相关关系。gydF4y2Ba
结论gydF4y2Ba
总体而言,我们的研究结果显示了质体基因间核苷酸替代率和遗传特征的异质性,为质体进化提供了新的视角,同时强调了基于质体尺度数据的系统基因组研究中考虑基因树不一致性的必要性。gydF4y2Ba
背景gydF4y2Ba
随着下一代测序(NGS)技术的爆炸性发展,整个塑料变得更加易于易于访问[gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba].由于独特的遗传方式,基因内容和秩序中的保守性,以及每个细胞的高拷贝数[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba],质体已被广泛用于重建植物的生命之树(例如[gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba])。此外,与标准片段DNA条形码相比,塑性尺度数据可以提供丰富的系统发育分析的信息位点[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba].尽管如此,塑料级数据的有效性最终反映了它们揭示给定谱系的“真实”系统发育关系的程度gydF4y2Ba14gydF4y2Ba].gydF4y2Ba
尽管质体由于其单亲本遗传和缺乏有性重组而被常规地认为是一个连接的单位点[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba15gydF4y2Ba],对塑料的结构和功能特征的实证研究表明,体液基因可能不会作为单个基因座而发展,并且可能会经历不同的进化力[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba].此外,尽管进化的速度比原子核低[gydF4y2Ba19gydF4y2Ba研究发现,质体中核苷酸替换率在被子植物谱系中存在差异,在反向重复区域和单拷贝区域之间也存在差异,在不同功能基因组之间也存在差异(例如[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba])。促进率的变化以及影响不同质体基因的进化的因素包括在单拷贝区跨越家庭和编码/非编码区之间的突变率的变化,以及变化,由于反转的两种配置[存在gydF4y2Ba14gydF4y2Ba].然而,在使用全plastomes最近许多phylogenomic的研究,只有从全连结数据集的结果呈现(例如[gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba23gydF4y2Ba])。在这些情况下,由于个体基因的进化速率变化而导致的基因树不一致仍然知之甚少。除了连接方法外,多物种结合(MSC)方法还考虑了基因树的异质性,通过使用基因树的合合数来估计物种树,从而可以评估古代杂交、渐渗和不完全谱系分类(ILS) [gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba].近日,phylogenomic研究认为,串联和成膜助剂相结合的方法能产生精确的系统发育,有利于调查的基因树和物种树[之间的不一致性gydF4y2Ba18gydF4y2Ba,gydF4y2Ba26gydF4y2Ba].gydF4y2Ba
基于质体的比较基因组研究主要集中于结构变化,如反向重复序列(IR)的收缩或扩张(例如[gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba])和基因组重排(例如[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba]),又一个谱系中质体基因,如遗传变异和系统发育信息量的遗传特征,仍然研究很少。这些特性可以不同的基因或基因的功能组之间各不相同,在我们的质体进化和系统发育推断的理解是非常重要的。此外,进化速率和基因特征之间的相关性可被调用原质体系演进的主要动力的说明[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba].gydF4y2Ba
龙胆科包括龙胆亚科和獐牙菜亚科两个亚族,共有21属约950种,是龙胆科中物种多样性最高的[gydF4y2Ba36gydF4y2Ba].龙胆亚科的成员与獐牙菜亚科的区别很容易,因为在花冠裂片之间存在萼内膜和花冠裂片之间的褶,这两种特征在獐牙菜亚科中都没有[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba].虽然一些系统发育研究已经证实了这两个亚族的单系[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba],内Gentianeae通用划界仍然不明确,特别是内Swertiinae,具有一些属(例如,gydF4y2Ba斯伯里亚gydF4y2BaL.,gydF4y2BaGentianellagydF4y2Ba烟囱,gydF4y2BaComastomagydF4y2Ba(Wettst)。Toyok。gydF4y2Ba侧蕊属gydF4y2BaA.Braun)是并系[gydF4y2Ba38gydF4y2Ba].牙蛋白的目前的系统发育基于几个DNA标记,通常包括其,gydF4y2BaATPB,RBCL.gydF4y2Ba,gydF4y2BamatKgydF4y2Ba,gydF4y2BatrnL-trnFgydF4y2Ba[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba,因此,对这些类群的全面的分类学和进化理解受到不令人满意的系统发育解析的阻碍。gydF4y2Ba
为了提高塑料的进化的新见解,并改善龙胆炎属中属的系统发育亲界描绘,我们构建了塑性序列的数据集,包括29种牙棕榈树和三个小组。我们通过级联和结束方法生成76个蛋白质编码基因(PCG)序列以推断系统发育,并表征塑性基因的遗传特征。我们的具体目标是(a)测试塑料尺寸数据是否有效地解决耿氏模组中的神秘关系;(b)调查龙钙素塑植基因的特征多样性;(c)探讨进化率异质性与基因特征的相关性以及基因树的不等。gydF4y2Ba
结果gydF4y2Ba
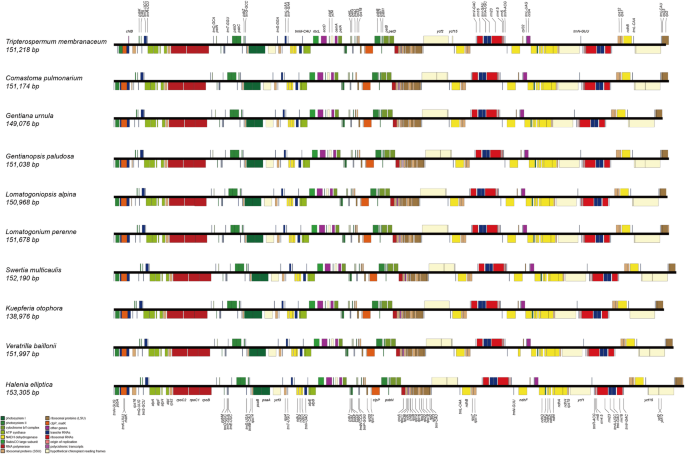
Gentianeae plastomes特点gydF4y2Ba
共测序总共10种(附加文件gydF4y2Ba1gydF4y2Ba:表S1)代表GentiALEA的10个属(新报告)。在De Novo组件之后,我们为每个新测序的塑料产生了单个contig。平均测序覆盖范围从646×(gydF4y2Ba秦艽urnulagydF4y2Ba)至3538×(gydF4y2BaGentianopsis paludosagydF4y2Ba).所有10个塑料显示由大型单拷贝(LSC),小单拷贝(SSC)和两个反转重复(IRA和IRB)组成的典型的四重结构结构。10个塑料的长度范围为139,976 bpgydF4y2BaKuepferia otophoragydF4y2Bato 153,305 bp inhalenia ellipticagydF4y2Ba(表gydF4y2Ba1gydF4y2Ba).所有塑料均具有四个rRNA和30个TrNA,并且具有相同的基因级(图。gydF4y2Ba1gydF4y2Ba).基因丧失涉及gydF4y2BaNDH.gydF4y2Ba基因属gydF4y2Ba绅士gydF4y2Ba被检测到。而且,这是gydF4y2Barpl33.gydF4y2Ba吉恩被发现迷失在gydF4y2BaComastoma pulmonariumgydF4y2Ba和gydF4y2Bashertia hispidicalyx.gydF4y2Ba(图。gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba和附加文件gydF4y2Ba1gydF4y2Ba:图S1)。龙胆科的胞浆高度保守,只发生过一次IR扩张gydF4y2Ba花锚,gydF4y2Ba其中红外区域扩展到gydF4y2Barpl22.gydF4y2Ba基因。gydF4y2Ba
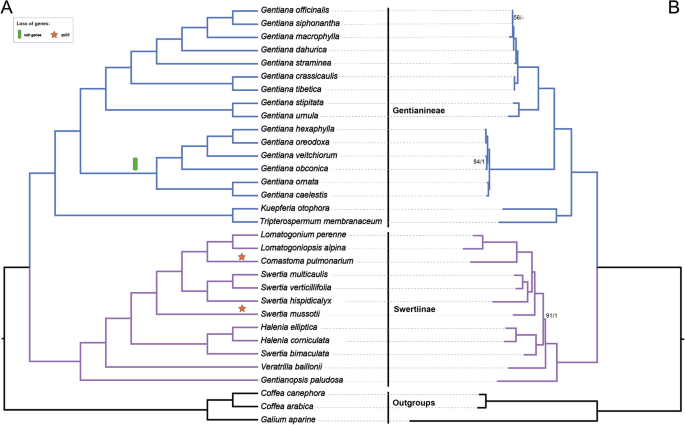
龙胆炎细胞发育关系gydF4y2Ba
76个基因、32个类群的串联序列长度为69579 bp,包含9228个简约信息位点。我们的系统基因组分析提高了龙胆科属间亲缘关系的分辨率和稳健性,大多数分支显示出较高的支持值。对于连接数据集,进行分区最大似然(ML)和贝叶斯推理(BI)分析(图。gydF4y2Ba2gydF4y2Ba)生成具有未分区数据集的相同树拓扑(附加文件gydF4y2Ba1gydF4y2Ba:图S2)。通过合并分析也推断出了相同的树拓扑(图。gydF4y2Ba2gydF4y2Ba).在所有分析中都支持两种体外的两种体,即Gentianina和Swertiinae。在Gentianina,一个由一个组成的思工gydF4y2BaTripterospermumgydF4y2Ba和gydF4y2BaKuepferiagydF4y2Ba是姐姐gydF4y2Ba龙胆。gydF4y2Ba之内gydF4y2Ba龙胆,gydF4y2Ba物种表现出gydF4y2BaNDH.gydF4y2Ba基因损失事件形成了一个独特的思考,并且是其他成员的姐妹gydF4y2Ba绅士gydF4y2Ba.在Swertiinae,gydF4y2Ba斯伯里亚gydF4y2Ba被揭示为非单系,因为gydF4y2Ba斯伯里亚双重镰状gydF4y2Ba用单胞嘧啶属gydF4y2BaHaleniagydF4y2Ba.gydF4y2Ba
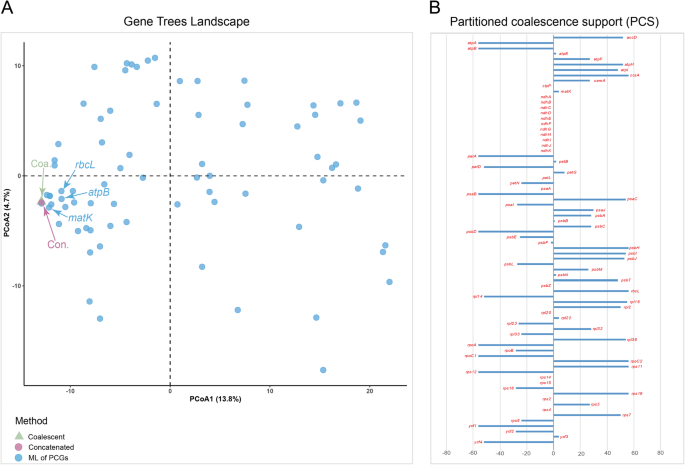
基因树的风景gydF4y2Ba
我们采用主坐标分析(后交通动脉)利用ML推断基因树和物种树从级联和成膜助剂分析估计探讨基因树不一致[gydF4y2Ba42gydF4y2Ba].结果表明,两种方法推断的物种树高度一致,而个体基因树表现出更大的变异。PCoA的第一和第二轴分别解释了树拓扑结构中13.8和4.7%的变异。我们计算了基因树与合并种树之间的距离,以表示每个基因的基因树不一致性(GD)(均值:14.789;值:14.560)。在测试的75个基因中,gydF4y2BarpoC2gydF4y2BaGD值最低(GD = 0),gydF4y2BapetLgydF4y2Ba有最高的(gd = 35.626)。来自三种传统上使用的塑性基因的基因树(gydF4y2Ba:gydF4y2Ba,gydF4y2BaATPB.gydF4y2Ba和gydF4y2BamatKgydF4y2Ba;Gd = 2.213,2.029和0.928)接近物种树(图。gydF4y2Ba3.gydF4y2Ba一个,附加文件gydF4y2Ba1gydF4y2Ba:表S2)。gydF4y2Ba
我们还计算了76个PCG的分区聚结符(PCS)。PC可以分别为正,负或零,指示支持,冲突或歧义[gydF4y2Ba43gydF4y2Ba].结果显示,33个pcg的PCS得分为正,23个pcg的PCS得分为负,20个pcg的PCS得分为零。gydF4y2Ba3.gydF4y2Bab,额外的文件gydF4y2Ba1gydF4y2Ba:表S2)。六个pcg (gydF4y2BaccsA, psbH, rbcL, rpoC2gydF4y2Ba,gydF4y2Barps11gydF4y2Ba和gydF4y2Barps19gydF4y2Ba)以最高的PC得分(PCS = 56)估计。在牙果藻前期系统发育研究中的三种广泛使用的塑性标记中,gydF4y2Ba:gydF4y2Ba和gydF4y2BamatKgydF4y2Ba有积极的PCS分数,而gydF4y2BaATPB.gydF4y2Ba有负面的PC得分。gydF4y2Ba
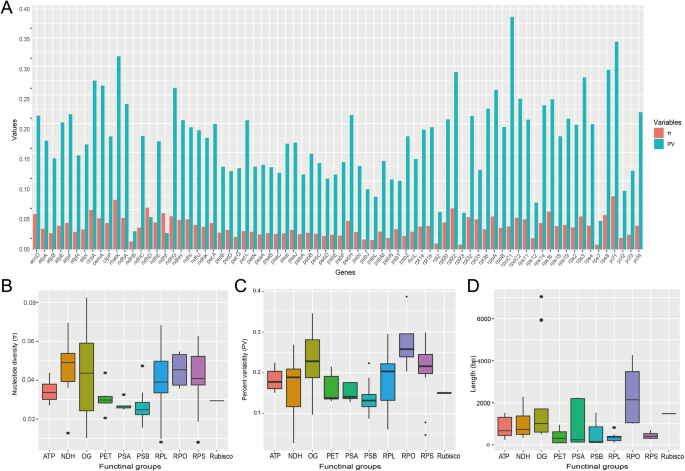
核苷酸替代率gydF4y2Ba
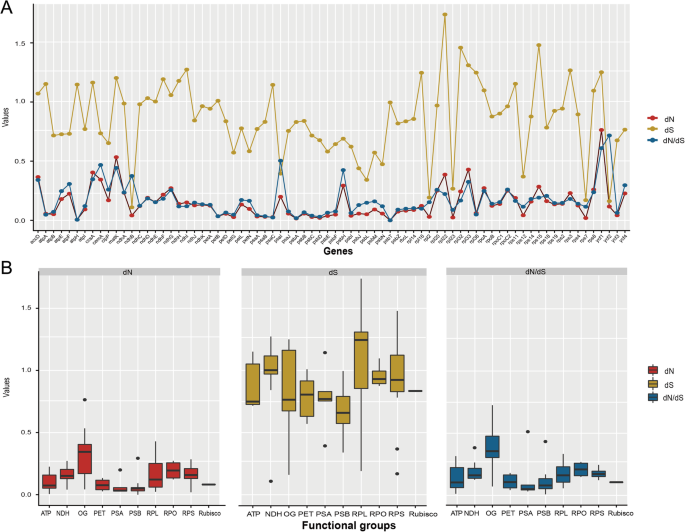
同义(dgydF4y2Ba年代gydF4y2Ba)和非同义词(dgydF4y2BaNgydF4y2Ba)替代率,与d沿gydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba估计为76个PCG来检测进化速率异质性和来表示作用于个PCG(表S2)不同的选择方案。间的76个基因,gydF4y2Barps22gydF4y2Ba,gydF4y2Barps15gydF4y2Ba和gydF4y2Barpl32.gydF4y2Bad值相对较高gydF4y2Ba年代gydF4y2Ba值,gydF4y2BaYCF1,MATK.gydF4y2Ba和gydF4y2Barpl33.gydF4y2Ba有较高的dgydF4y2BaNgydF4y2Ba值(图gydF4y2Ba4gydF4y2Ba一个,附加文件gydF4y2Ba1gydF4y2Ba:表S2)。所有76个pcg的d值都相当低gydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba,表明它们一直在净化选择中。我们还比较了9个功能组和一组其他基因(OG, Table)的进化速率gydF4y2Ba2gydF4y2Ba).OG中值d最高gydF4y2BaNgydF4y2Ba和D.gydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba但是一个温和的dgydF4y2Ba年代gydF4y2Ba中位数的值。编码光合作用亚基的基因,如光系统I和II (PSA和PSB), ATP合酶(ATP)和细胞色素b6f复合物(PET),核苷酸取代率低于其他功能基团。RNA聚合酶(RPO)基因表达量显著增加gydF4y2BaNgydF4y2Ba和D.gydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba值最高,而编码核糖体大亚基(RPL)蛋白的基因d值最高gydF4y2Ba年代gydF4y2Ba值(无花果。gydF4y2Ba4gydF4y2Bab).我们还连接了位于LSC、SSC和IR的基因,以研究IR与SC区域的替代率差异。红外区d值最低gydF4y2BaNgydF4y2Ba和D.gydF4y2Ba年代gydF4y2Ba值分别为0.095和0.200,其中SSC区最高(d .gydF4y2BaNgydF4y2Ba= 0.464;dgydF4y2Ba年代gydF4y2Ba= 0.958),其次是LSC区域(d .gydF4y2BaNgydF4y2Ba = 0.134; d年代gydF4y2Ba = 0.878).
非同义的估计(dgydF4y2BaNgydF4y2Ba)、同义(dgydF4y2Ba年代gydF4y2Ba)和dgydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba的gydF4y2Ba一个gydF4y2Ba塑性蛋白编码基因(PCG)和gydF4y2BabgydF4y2Ba基因的官能团。官能团的详细的信息提供在表gydF4y2Ba2gydF4y2Ba
质体基因的遗传特征gydF4y2Ba
我们计算了核苷酸多样性(π)和百分比变异性(PV)来代表pcg的遗传变异(附加文件)gydF4y2Ba1gydF4y2Ba:表S2)。π的值范围为0.0077(gydF4y2BaRPS7gydF4y2Ba)至0.0884 (gydF4y2Baycf1gydF4y2Ba),以及PV的值从0.0271范围(gydF4y2BaNDHF.gydF4y2Ba)至0.3872(gydF4y2Barpoc1.gydF4y2Ba,无花果。gydF4y2Ba5gydF4y2BaA,表S2)。在官能团中,RPO,RPS,RPL和NDH显示出高核苷酸多样性和变异性百分比(图。gydF4y2Ba5gydF4y2Bab和c)中,RPO组中特别基因。此外,RPO有基因长度的最高中间值(图gydF4y2Ba5gydF4y2Bad).使用PhyDesign测量用于系统发育分析的76个pcg的净系统发育信息(PI)(附加文件)gydF4y2Ba1gydF4y2Ba中:图S3和表S2)。这gydF4y2Baycf1gydF4y2Ba基因的净PI值最高,其次为gydF4y2BarpoC2gydF4y2Ba,gydF4y2BaNDHF.gydF4y2Ba和gydF4y2BamatK。gydF4y2Ba具有较长长度的基因通常表现出较高的PI值(附加文件gydF4y2Ba1gydF4y2Ba表S2),说明基因长度对系统发育信息的贡献很大。gydF4y2Ba
相关分析gydF4y2Ba
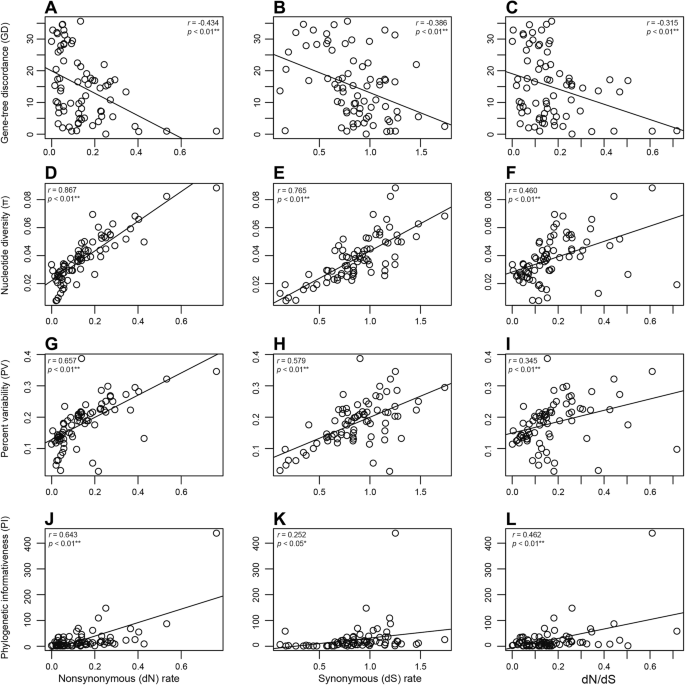
相关分析表明所有测试的相关性是用0.05截止(图显著。gydF4y2Ba6gydF4y2Ba).具体地,核苷酸多样性(π)(图gydF4y2Ba6gydF4y2BaD,E和F),百分比变异性(PV)(图。gydF4y2Ba6gydF4y2Bag,h和i)和系统发育信息(pi)(图。gydF4y2Ba6gydF4y2Baj, k和l)均与核苷酸替换率呈正相关,而基因树不一致性(GD)(图。gydF4y2Ba6gydF4y2BaA,B和C)与核苷酸取代的速率负相关。gydF4y2Ba
进化率异质性的相关性,包括非同义(dgydF4y2BaNgydF4y2Ba)、同义(dgydF4y2Ba年代gydF4y2Ba)和dgydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba,gydF4y2Ba一个gydF4y2Ba-gydF4y2BacgydF4y2Ba基因树不调和,gydF4y2BadgydF4y2Ba-gydF4y2BafgydF4y2Ba核苷酸多样性(π)gydF4y2BaggydF4y2Ba-gydF4y2Ba我gydF4y2Ba%的可变性(PV)和gydF4y2BajgydF4y2Ba-gydF4y2BalgydF4y2Ba系统信息量。p值旁边的星星被用来评估显著性水平gydF4y2Ba
讨论gydF4y2Ba
塑料级数据集的系统发育意义gydF4y2Ba
据我们所知,此处呈现的结果是第一个利用系统托儿科数据集的结果来研究Gentiaeae属的文学性和血症发育亲症。与前一个系统发育重建相比,我们的研究表明树分辨率的大量改进[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,为龙胆科提供了强健的脊梁。我们的系统基因组主干显示了龙胆亚科清晰的细分为龙胆亚科和獐牙菜亚科,这与之前的形态学一致[gydF4y2Ba45gydF4y2Ba]及分子系统发育[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba,gydF4y2Ba44gydF4y2Ba].龙胆科一直被认为包括四个属gydF4y2Ba绅士gydF4y2BaL.,gydF4y2BaTripterospermumgydF4y2Ba布鲁姆,gydF4y2Bametagentiana.gydF4y2Bat.n.ho&s.w.liu,和gydF4y2BaCrawfurdiagydF4y2Ba墙。Favre等人[gydF4y2Ba41gydF4y2Ba)被排除在外gydF4y2Ba绅士gydF4y2Ba教派。gydF4y2Baotophora.gydF4y2Ba从gydF4y2Ba绅士gydF4y2Ba和高gydF4y2BaKuepferiagydF4y2Baadr.favre和描述gydF4y2Basinogentiana.gydF4y2BaADR。Favre&Y.M.从两种物种中排除了袁gydF4y2BaMetagentiana。gydF4y2Ba我们的结果解决了gydF4y2Ba绅士gydF4y2Ba为单系,与系间表现出密切的关系gydF4y2BaKuepferiagydF4y2Ba和gydF4y2BaTripterospermumgydF4y2Ba,支撑的高度gydF4y2BaKuepferia。gydF4y2Ba与牙龈相比,Swertiinae由于令人醒目而言更加复杂gydF4y2Ba斯伯里亚gydF4y2Ba[gydF4y2Ba38gydF4y2Ba].我们phylogenomic框架推断使用与Swertiinae的系统发育全等gydF4y2BaTRNL.gydF4y2Ba-intron +gydF4y2BamatKgydF4y2Ba[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba44gydF4y2Ba],gydF4y2BaatpB-rbcLgydF4y2Ba间隔[gydF4y2Ba38gydF4y2Ba],八个质标志物的超级矩阵(gydF4y2BaRBCL + MATK + ATPB + NDHF + RPL16 + RPS16 + TRNL-TRNFgydF4y2Ba + atpB-rbcLgydF4y2Ba) [gydF4y2Ba40gydF4y2Ba]及ITS [gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba44gydF4y2Ba].综上所述,本研究将龙胆纳入系统基因组框架,为深入了解龙胆的进化史迈出了第一步。在生物地理学和多样性研究方面,需要更密集的采样和更先进的方法。gydF4y2Ba
大多数已在过去几十年中被调查主要依靠质的基因串联比对ML推断被子植物的主要群体的亲缘关系(例如[gydF4y2Ba7gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba])。然而,由于塑性在结束理论中塑性的紧密连接单位的基本假设,通过各种塑性基因产生的系统发育不足通常被忽略。goncalves等。[gydF4y2Ba18gydF4y2Ba表明,连接的矩阵可能产生高度支持的系统发育,与单个基因树不一致。Walker等[gydF4y2Ba14gydF4y2Ba]证明在被子植物系统发育的各级质体中的基因横行树冲突,突出了未来研究的必要性入考虑质体系的冲突。这两项研究都强调了考虑整个质的基因进化信号变化的重要性,并主张用多品种成膜助剂(MSC)的方法,在phylogenomic调查质体矩阵。在我们的分析,尽管与使用MSC方法级联矩阵生成的树形拓扑结构的一致性,基因树拓扑显示与所串接的数据推断出该品种的树木变化很大。此外,我们的PCS的计算后的76个基因质与负分和20个基因与暧昧估计23,表示超过了基因的一半促进Gentianeae的系统发育不确定性。通过这两种方法产生一致的拓扑结构的一个可能的解释是,个体的基因家谱效果太小,模糊系统发育推断的准确性当所有的基因连接成一个“超矩阵”。在方法上,被用作在ASTRAL-III MSC方法的输入,各个基因树木及其引导重复使用ML在RAxML [推断gydF4y2Ba46gydF4y2Ba].在这种情况下,用作为单个轨迹的级联基因进行系统发育推理将代表MSC的特殊情况[gydF4y2Ba25gydF4y2Ba].尽管如此,这类基因树异质性不应该被忽视,因为它可能会影响到分歧时间估计或更高分类级别发育重建。gydF4y2Ba
我们对PCS分数的估计显示,gydF4y2BaATPB.gydF4y2Ba),是三种广泛使用的质体标记(gydF4y2Ba:gydF4y2Ba,gydF4y2BaATPB.gydF4y2Ba和gydF4y2BamatKgydF4y2Ba)在龙胆科的系统发育推断中是一个异常基因,它可能对有争议关系的解决产生不成比例的影响[gydF4y2Ba43gydF4y2Ba].然而,利用质体超基质对龙胆科系统发育进行分析gydF4y2BaATPB.gydF4y2Ba基因[gydF4y2Ba40gydF4y2Ba]获得与其他标记的拓扑结构大致一致的树状拓扑结构[gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba].对观察到的一致性的一个可能的解释是gydF4y2BaATPB.gydF4y2Ba与其他塑性基因可能抵消潜在的偏差gydF4y2BaATPB.gydF4y2Ba在Gentianeae关系的重建。此外,相对较低的PCS得分gydF4y2BamatKgydF4y2Ba(PCS = 4)是XI等人类研究中存在的广泛平行片的可能原因。[gydF4y2Ba44gydF4y2Ba].gydF4y2Ba
龙胆塑性塑性基因特征多样性gydF4y2Ba
在基因组结构方面高度保守牙淋血糖,只检测到一个琐碎的IR扩张(图。gydF4y2Ba1gydF4y2Ba和gydF4y2Ba2gydF4y2Ba).基因丢失事件包括gydF4y2BaNDH.gydF4y2Ba基因和gydF4y2Barpl33.gydF4y2Ba在一些物种中进行检测。损失gydF4y2BaNDH.gydF4y2Ba塑料中的基因gydF4y2Ba绅士gydF4y2BaFu等的研究报道[gydF4y2Ba47gydF4y2Ba]和Sun等人。[gydF4y2Ba48gydF4y2Ba].在绿色植物,gydF4y2BaNDH.gydF4y2Ba基因编码参与光合电子运输的类囊体ndh复合物的组成部分[gydF4y2Ba49gydF4y2Ba].近日,plastomes的基因丢失和进化的全面调查显示,gydF4y2BaNDH.gydF4y2Ba基因是最常见的基因丢失,这表明并非所有的gydF4y2BaNDH.gydF4y2Ba基因参与或功能性的电子传输必不可少[gydF4y2Ba50gydF4y2Ba].值得注意的是gydF4y2BaNDH.gydF4y2Ba基因发生在属内gydF4y2Ba绅士gydF4y2Ba并形成了一个独特的分支,这表明这种损失可能与特定的适应有关gydF4y2Ba绅士gydF4y2Ba物种。由于很少的质体gydF4y2Ba绅士gydF4y2Ba与物种总数(约360-400种)相比,尚需进一步探索质体测序。相比gydF4y2BaNDH.gydF4y2Ba基因的缺失gydF4y2Barpl33.gydF4y2Ba可能更随机。编码区域中有一个停止密码子gydF4y2Barpl33.gydF4y2Ba在gydF4y2BaComastoma pulmonariumgydF4y2Ba由于胞嘧啶(C)(T)在碱基对22的变化为胸腺嘧啶我们映射的所有的测序读取gydF4y2Bac . pulmonariumgydF4y2Ba使用亵渎验证的组装序列。映射的读取显示,几乎所有读数都支持突变。在塑料中gydF4y2Bashertia hispidicalyx.gydF4y2Ba,有一个小的删除,包含的编码区域gydF4y2Barpl33.gydF4y2Ba之间的gydF4y2BapsaJgydF4y2Ba和gydF4y2Barps18gydF4y2Ba基因。之前的一项研究表明gydF4y2Barpl22,rpl32gydF4y2Ba,gydF4y2Barpl33.gydF4y2Ba基因的缺失比其他基因的缺失更为突出gydF4y2BarplgydF4y2Ba基因[gydF4y2Ba50gydF4y2Ba].此外,反向遗传学研究中发现的基因的编码核糖体蛋白rpl33不影响标准条件下植物存活和生长敲除[gydF4y2Ba51gydF4y2Ba].因此,rpl33可以是植物光合作用中的非必需质体核糖体蛋白,和损失gydF4y2Barpl33.gydF4y2Ba基因可用于通过其它被补偿gydF4y2BarplgydF4y2Ba基因或核编码基因。gydF4y2Ba
估计不同基因和不同官能团之间的核苷酸取代率为洞察洞察于对塑料进化作用的不同选择制度(例如[[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba22gydF4y2Ba])。在禾本科(gydF4y2Ba34gydF4y2Ba]和天竺葵科[gydF4y2Ba17gydF4y2Ba据报道,据报道,编码参与光合作用过程的亚基的基因,例如NAD(P)H脱氢酶(NDH),ATP合酶(ATP),光系统I和II(PSA和PSB)和细胞色素B6F复合物(PET),表现出比其他官能团的核苷酸取代率相对较低。在牙龙(图)中观察到类似的图案(图。gydF4y2Ba4gydF4y2Bab)。在光合官能团中,NDH具有相对较高的核苷酸取代率,其可能与基因损失事件相关联gydF4y2Ba绅士gydF4y2Ba.我们在龙胆科中发现了一些具有加速取代率的功能基因,主要是核糖体蛋白(RPL)和RPS基因以及RNA聚合酶(RPO)基因。类似的模式以前也被记录过,例如RPL和RPS基因在天竺葵科中被证明是高度加速的[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba28gydF4y2Ba[Annonaceae,Passifloraceae和Geraniacee的RPO基因[gydF4y2Ba52gydF4y2Ba].除了gydF4y2Ba,ACCD,CLPP,YCF1gydF4y2Ba,gydF4y2Baycf2gydF4y2Ba有最快的加速速度[gydF4y2Ba22gydF4y2Ba的质体中检测到的gydF4y2Ba米瓦gydF4y2Ba(石竹科),和gydF4y2BaclpPgydF4y2Ba和gydF4y2Baycf1gydF4y2Ba被发现具有最高dgydF4y2BaNgydF4y2Ba豆类中基因之间的价值[gydF4y2Ba16gydF4y2Ba].在目前的研究中,gydF4y2Baycf1gydF4y2Ba和gydF4y2BamatKgydF4y2Ba有最高的dgydF4y2BaNgydF4y2Ba值,和gydF4y2Baycf2gydF4y2Ba有最高的dgydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba价值。尽管个体基因或基因官能团中的核苷酸替代率分解,但没有阳性选择作用于龙钙血糖塑性的塑性基因。另外,鉴于未检测到明显的结构变化,牙果果树中的取代率变化模式可归因于基因组突变率的异质性。此外,塑料中的不同的取代速率以及基因树的不驾驶率支持该视图,即塑性基因不如前所述紧密地连接,并且正在经历不同的进化力[gydF4y2Ba18gydF4y2Ba].gydF4y2Ba
质体IR区域的进化动力学以前已经有文献记载[gydF4y2Ba19gydF4y2Ba].最近的一项研究表明,IR基因的同义替代率平均比使用69个血管素,裸子植物和蕨类植物的69个塑料在SC基因中慢3.7倍[gydF4y2Ba27gydF4y2Ba].但是,一项研究gydF4y2Ba天竺葵属植物gydF4y2Ba(Geraniaceae)观察到d的相反模式gydF4y2Ba年代gydF4y2Ba与SC区域相比,IR区域的基因值更高[gydF4y2Ba29gydF4y2Ba].我们的结果显示dgydF4y2Ba年代gydF4y2Ba使用每个区域的基因串联数据集,LSC和SSC区域的发病率大约是IR区域的4到5倍。观测到的高dgydF4y2Ba年代gydF4y2BaSSC区域中的值可能来自参与基因损失的六个NDH基因。反过来,高同义替代率可能表示放松的选择性约束对基因损失事件负责。IR区域中的低替换速率可以通过为在其中一个副本中发生突变时提供用于纠错模板的两个相同副本来解释[gydF4y2Ba29gydF4y2Ba].gydF4y2Ba
进化率异质性与基因特征和GD的相关性gydF4y2Ba
质体基因的特性对我们理解质体的进化和系统发育具有重要意义。我们发现质体基因和功能基因组之间存在广泛的差异。pcg的变异率(PV)与光合功能基团(ATP、NDH、PET、PSA和PSB)的核苷酸替换率(PV)相似,均低于核糖体蛋白(RPL、RPS)和RNA聚合酶(RPO)组,而NDH组核苷酸替换率(π)较高。π值由两个序列间每个位点的平均核苷酸差异数及其抽样方差估算[gydF4y2Ba53gydF4y2Ba],因此它的估计可以通过的损失影响gydF4y2BaNDH.gydF4y2Ba基因gydF4y2Ba绅士gydF4y2Ba.我们的研究结果揭示了与核苷酸替代率遗传变异的显著正相关,这表明多样化的选择压力,发挥着重要作用的原质的演变。gydF4y2Ba
质体基因的净系统发育信息量(PI)反映了其在解决复杂系统发育关系中的表现。只有一个(gydF4y2BamatKgydF4y2Ba)三(gydF4y2BamatK基因,ndhB位gydF4y2Ba和gydF4y2Ba:gydF4y2Ba)在龙胆科系统发育研究中广泛使用的标记显示出高的净PI值,这解释了之前分析的有限分辨率,并突出了质体规模数据集的实用性。在测试的基因中,gydF4y2Baycf1gydF4y2Ba和gydF4y2BarpoC2gydF4y2Ba具有较高的净PI值,且基因长度相对较长,将是未来龙胆科系统发育推断的最佳标记。的确,系统发育的效用gydF4y2Baycf1gydF4y2Ba已在兰花中得到证实[gydF4y2Ba54gydF4y2Ba]以及在辐射谱系中[gydF4y2Ba55gydF4y2Ba],并作为陆生植物的核心条码[gydF4y2Ba56gydF4y2Ba].我们的分析显示出良好的表现gydF4y2BarpoC2gydF4y2Ba,具有较高的PI,最低的基因树不一致和正高个得分。因此,我们提倡gydF4y2BarpoC2gydF4y2Ba作为一种有前景的质体DNA条形码,用于龙胆科的分类研究gydF4y2BarpoC2gydF4y2Ba在Agiosperm phylogy的系统发育重建中[gydF4y2Ba14gydF4y2Ba].gydF4y2Ba
我们发现PI与核苷酸替换率显著正相关,表明质体基因的核苷酸替换只是轻微饱和。当序列经过多次替换,由于使用表观距离低估了实际遗传距离,从而减少了序列中包含的系统发育信息时,该序列被认为是饱和的[gydF4y2Ba57gydF4y2Ba].我们的分析揭示了基因树不一致(GD)和核苷酸替代率之间的负相关关系。以前的研究已经引起重视与插入缺失和基因组重排,如牻牛儿[数核苷酸替代率之间的相关性gydF4y2Ba30.gydF4y2Ba],豆类[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba21gydF4y2Ba]和狸藻科[gydF4y2Ba58gydF4y2Ba],而GD仍然差不多。一般来说,D的变化gydF4y2Ba年代gydF4y2Ba都有可能通过促进突变率,如DNA修复的变化的潜在因素受到影响。然而,改变dgydF4y2BaNgydF4y2Ba和D.gydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba通常不仅通过各种突变率而导致,而且是通过选择性约束。Geniaeeae的塑性基因中没有阳性选择的迹象,GD与D之间的相关性gydF4y2BaNgydF4y2Ba和D.gydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba提示GD可能受纯化选择强度或对有害突变的选择性去除控制。GD与d呈负相关gydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba表明在净化选择的较高强度下,基因树不道议更加猖獗。对有害突变的选择可能导致连接中性位点的遗传变异量的减少[gydF4y2Ba59gydF4y2Ba],因此快速去除的突变可能会模糊谱系的进化足迹。gydF4y2Ba
结论gydF4y2Ba
我们的研究结果呈现在这里是第一个利用phylogenomic数据集进行调查Gentianeae的属间亲缘关系。该phylogenomic框架奠定了这个不同部落的进化历史的深刻理解的基础。比较基因组分析揭示了质体基因中都广泛进化速率的异质性和遗传变异,支撑认为质体基因没有紧密连锁如以前认为与正在经历不同的进化力。在Gentianeae的系统发育推理常用的指标中,只有gydF4y2BamatKgydF4y2Ba有高系统信息,同时gydF4y2BaATPB.gydF4y2Ba可能会对解决有争议的关系产生不成比例的影响。很少使用的基因gydF4y2BarpoC2gydF4y2Ba是表现最好的基因,类似于在被子植物系统发育重建中的有用性,因此被认为是一种有前景的质体DNA条形码,用于龙胆科的分类研究。值得注意的是,基因树的系统发育不一致现象十分严重,强调了在未来的系统基因组研究中考虑基因树异质性的必要性。gydF4y2Ba
方法gydF4y2Ba
分类种类和测序gydF4y2Ba
我们采样10种表示从青藏高原(QTP)和相邻区域Gentianeae的10个属。鲜叶收集在日提交的并用硅胶购买DNA分离物干燥。我们的现场采集遵循道德和当地政府的合法性,并经政府批准。植物材料的正式鉴定通过昆明植物研究所(KUN)的标本室进行,和凭证标本在KUN(附加文件沉积gydF4y2Ba1gydF4y2Ba:表S1)。使用植物基因组DNA试剂盒(DP305)从中国天根生物技术(北京)有限公司(北京)有限公司(北京)有限公司提供全基因组DNA。在制造商手册之后,用Truseq DNA样品制备套件(Illumina,Inc.,Unitional)构建了捷卡(500bp)配对终端库。然后在中国昆明,云南,云南省昆明有限公司的Illumina Hiseq 4000平台上测序文库。gydF4y2Ba
塑料组装和注释gydF4y2Ba
使用Trimmomatic v.0.36对原始reads进行质量评估[gydF4y2Ba60gydF4y2Ba]通过去除低质量和适配器污染的读数。随后,使用Novoplacty V.2.7.2将剩余的高质量读数组装成Contigs [gydF4y2Ba61gydF4y2Ba].在Shen等人的描述之后。[gydF4y2Ba62gydF4y2Ba],种子和 - 与延伸的原质体系序列采用算法gydF4y2Ba獐牙菜属mussotiigydF4y2Ba(Genbank accession: NC_031155.1)作为种子输入,其他参数保持默认设置(参见NOVOPlasty手册)。然后使用Plastid Genome Annotator (PGA)对组装好的质体进行注释[gydF4y2Ba63gydF4y2Ba].参照已发表的龙胆科细胞体,手工检查起始/终止密码子和内含子/外显子边界。tRNA基因通过tRNAscan-SE鉴定[gydF4y2Ba64gydF4y2Ba].为了比较,我们使用OGDRAW可视化了所有测序质体的线性图形图[gydF4y2Ba65gydF4y2Ba].gydF4y2Ba
系统发育分析gydF4y2Ba
龙胆亚科29个分类群(17个龙胆亚科+ 12个獐牙菜亚科)和3个外群(gydF4y2Ba咖啡阿拉伯里卡gydF4y2Ba,gydF4y2BaCoffea Canephora.gydF4y2Ba和gydF4y2Ba猪殃殃gydF4y2Ba)被纳入系统基因组分析(附加文件gydF4y2Ba1gydF4y2Ba:表S1)。进行了连接和聚结分析。对于连接方法,使用系统套件v.1.1.15提取常见的76个蛋白编码基因(PCGs) [gydF4y2Ba66gydF4y2Ba]随后的手动修改。76个PCG的序列分批进行比使用“G-INS-i”的策略和密码子取向模式用MAFFT v.7.313,然后在PhyloSuite串联。进行分区和未分区分析。对于数据分区,PartitionFinder v.1.0.1 [gydF4y2Ba67gydF4y2Ba],在贝叶斯信息准则(BIC)下确定最优划分方案和进化模型选择。在RAxML v.8.2.10中进行最大似然(ML)分析[gydF4y2Ba68gydF4y2Ba]下用“快速自举”算法(1000次重复)的“GTRGAMMA”的模式。使用MrBayes v.3.2.3 [进行贝叶斯推理(BI)gydF4y2Ba69gydF4y2Ba]具有四个马尔可夫链(一个冷和三个加热),用于从随机起始树5000000个世代和运行每5000个世代采样。树的第一个25%的烧机丢弃,剩余的树木被用于构建多数规则一致树。gydF4y2Ba
对于合并方法,在“GTRGAMMA”模型下,在RAxML中分别推断个体基因树。每次运行都使用500个重复的引导重采样。由此产生的无根基因树被输入到ASTRAL-III v.5.6.2中[gydF4y2Ba46gydF4y2Ba]来估计物种树与使用局部后验概率计算节点支持。gydF4y2Ba
质体基因树景观的探索gydF4y2Ba
探讨基因树的变化,我们绘制的树木罗宾逊-Foulds算法的统计分布[gydF4y2Ba70gydF4y2Ba],通过使用R包Treespace v.1.0.0计算大型树木之间的距离[gydF4y2Ba42gydF4y2Ba],遵循Goncalves等人的工作流程[gydF4y2Ba18gydF4y2Ba],并用GGPLOT2 V.2.2.1进行可视化[gydF4y2Ba71gydF4y2Ba].由于TREESPACE只接受包含相同的刀片树群,我们去掉了gydF4y2Barpl33.gydF4y2Ba在某些物种中缺失的基因座。此外,我们移除了六种gydF4y2Ba绅士gydF4y2Ba由于损失而分析gydF4y2BaNDH.gydF4y2Ba基因:gydF4y2BaG.木瓜,G oreodoxa,蓝玉簪G.,G华丽gydF4y2Ba和gydF4y2BaG. Caelestis.gydF4y2Ba.我们还包括从连接和膨胀分析中推断出两个物种树。总共,数据集由来自26个分类群和两个物种树的75棵基因树组成。我们利用基因树与膨胀物种树之间的距离来估计塑性基因的基因树不间断(GD)。使用前两种PCOAS计算的距离进行计算。gydF4y2Ba
此外,使用由Gatesy等人提供的脚本计算每个PCG的分区共聚载支持(PC)。[gydF4y2Ba43gydF4y2Ba)(gydF4y2Bahttps://github.com/dbsloangydF4y2Ba).PCS量化每个基因树的阳性或负面影响在摘要聚结方法支持的蛹中的系统组中[gydF4y2Ba43gydF4y2Ba,gydF4y2Ba72gydF4y2Ba].我们使用ASTRAL-III生成的系统进化树作为最优种树拓扑,使用RAxML推断的树作为备选种树。gydF4y2Ba
核苷酸取代率分析gydF4y2Ba
估算核苷酸替代的速率,非同义词(dgydF4y2BaNgydF4y2Ba)、同义(dgydF4y2Ba年代gydF4y2Ba)和非同义词与同义率的比率(dgydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba)在PAML v.4.9中计算[gydF4y2Ba73.gydF4y2Ba使用CODEML选项,使用F3 × 4模型估计密码子频率。用串联法生成的系统发育关系作为约束树。使用“cleandata = 1”选项去除间隙区域。“模型= 0”选项用于允许单个dgydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba值到分支之间变化。在CODEML控制文件中的其他参数的默认设置被留下。For the comparisons between different functional groups of PCGs, we consolidated the 76 PCGs into nine groups, i.e. photosystem I (PSA), photosystem II (PSB), cytochrome B6f complex (PET), ATP synthase (ATP), rubisco large subunit (Rubisco), RNA polymerase (RPO), ribosomal proteins large subunit (RPL), ribosomal proteins small subunit (RPS) and NADH dehydrogenase (NDH), and other genes (OG, includingycf1, ycf2, ycf3, ycf4, accD, clpP, ccsA, cemAgydF4y2Ba和gydF4y2BamatKgydF4y2Ba).官能团的详细的信息提供在表gydF4y2Ba2gydF4y2Ba.gydF4y2Ba
遗传变异和PCG中的系统发育信息量gydF4y2Ba
我们使用核苷酸多样性(π)和百分比变异性(PV)来表示的PCG的遗传变异。每个PCG的百分比变异性除以分离位点(S,即变量位置的数目)由基因的长度来估计。两个π和S是在该程序DnaSP v.6.0.7计算[gydF4y2Ba74.gydF4y2Ba]使用由MAFFT对齐的76个PCG的序列分开。在Phydesign Web应用程序中估计了76pcgs的净系统血易信息(PI)概况(gydF4y2Bahttp://phydesign.townsend.yale.edu/gydF4y2Ba) [gydF4y2Ba75.gydF4y2Ba]利用HyPhy替代率算法对DNA序列进行分析[gydF4y2Ba76.gydF4y2Ba].在此基础上重建了一棵相对时间超度量树gydF4y2BadnamlkgydF4y2BaPhylip中的程序[gydF4y2Ba77.gydF4y2Ba使用]级联ML树由RAxML推断。与级联矩阵分区由76个PCG沿转换的相对时间超度量树被用作在PhyDesign输入文件来计算与默认设置的系统发育信息量。gydF4y2Ba
相关分析gydF4y2Ba
核苷酸取代率的相关性(包括dgydF4y2BaNgydF4y2Ba,D.gydF4y2Ba年代gydF4y2Ba和D.gydF4y2BaNgydF4y2Ba/ dgydF4y2Ba年代gydF4y2Ba用基因树不一致性(GD)、核苷酸多样性(π)、百分数变异性(PV)和系统发育信息(PI)进行检测gydF4y2BacorgydF4y2BaR包中的函数gydF4y2Ba统计数据gydF4y2Bav3.6.1 (gydF4y2Bahttps://www.rdocumentation.org/packages/statsgydF4y2Ba).功能gydF4y2Bacor.testgydF4y2Ba用于计算gydF4y2BapgydF4y2Ba-值与皮尔逊检验。gydF4y2Ba
可用性数据和材料gydF4y2Ba
本研究中使用的所有序列均来自美国国家生物技术信息中心(NCBI)(登录号:MT228723- MT228732;见附加文件gydF4y2Ba1gydF4y2Ba:表S1)。gydF4y2Ba
缩写gydF4y2Ba
- BI:gydF4y2Ba
-
贝叶斯推理gydF4y2Ba
- BIC:gydF4y2Ba
-
贝叶斯信息准则gydF4y2Ba
- CTAB:gydF4y2Ba
-
溴化十六烷基trimethylammoniumgydF4y2Ba
- dgydF4y2BaNgydF4y2Ba:gydF4y2Ba
-
不幸的是gydF4y2Ba
- 达克斯:gydF4y2Ba
-
DNA序列多态性gydF4y2Ba
- dgydF4y2Ba年代gydF4y2Ba:gydF4y2Ba
-
代名词gydF4y2Ba
- GD:gydF4y2Ba
-
基因树不间断gydF4y2Ba
- GTR:gydF4y2Ba
-
一般时间可逆gydF4y2Ba
- 红外光谱:gydF4y2Ba
-
反向重复gydF4y2Ba
- 图书馆:gydF4y2Ba
-
不完整的谱系排序gydF4y2Ba
- 它的:gydF4y2Ba
-
核糖体DNA的内部转录间隔gydF4y2Ba
- LSC:gydF4y2Ba
-
大单拷贝gydF4y2Ba
- ml:gydF4y2Ba
-
最大似然gydF4y2Ba
- 硕士:gydF4y2Ba
-
Multispecies合并gydF4y2Ba
- NCBI:gydF4y2Ba
-
国家生物技术信息中心gydF4y2Ba
- 门店:gydF4y2Ba
-
下一代测序gydF4y2Ba
- PCOA:gydF4y2Ba
-
主坐标分析gydF4y2Ba
- PCG:gydF4y2Ba
-
蛋白质编码基因gydF4y2Ba
- 件:gydF4y2Ba
-
分区合并支持gydF4y2Ba
- 职业:gydF4y2Ba
-
质体基因组注释器gydF4y2Ba
- PI:gydF4y2Ba
-
系统信息量gydF4y2Ba
- PV:gydF4y2Ba
-
百分比变异性gydF4y2Ba
- QTP:gydF4y2Ba
-
青藏高原gydF4y2Ba
- rrna:gydF4y2Ba
-
核糖体核糖核酸gydF4y2Ba
- SSC:gydF4y2Ba
-
小单副本gydF4y2Ba
- TRNA:gydF4y2Ba
-
转移核糖核酸gydF4y2Ba
参考gydF4y2Ba
- 1.gydF4y2Ba
关键词:被子植物,质体基因组,焦磷酸测序BMC Plant Biol. 2006;6:17。gydF4y2Ba
- 2.gydF4y2Ba
Stull GW, Moore MJ, Mandala VS, Douglas NA, Kates HR, Qi X, Brockington SF, Soltis PS, Soltis DE, Gitzendanner MA。被子植物质体基因组大规模平行测序的靶向富集策略。应用植物学报,2013;gydF4y2Ba
- 3.gydF4y2Ba
Cronn R,Liston A,Parks M,Gernandt DS,Shen R,Mockler T.使用SOLEXA测序技术的植物叶绿体基因组的多重测序。核酸RES。2008; 36:E122。gydF4y2Ba
- 4.gydF4y2Ba
Wicke S,Schneeweiss GM,dePamphilis CW,穆勒KF,在陆生植物质染色体的匡特D.进化:基因内容,基因顺序,基因功能。植物mol biol。2011; 76:273-97。gydF4y2Ba
- 5.gydF4y2Ba
Ruhlman Ta,Jansen RK。开花植物的塑性基因组。在:Maliga P,编辑。叶绿体生物技术:方法和协议。TOWA,NJ:Humana媒体;2014. p。3-38。gydF4y2Ba
- 6.gydF4y2Ba
Jansen RK, Cai Z, Raubeson LA, Daniell H, Depamphilis CW, Leebens-Mack J, Muller KF, Guisinger-Bellian M, Haberle RC, Hansen AK,等。通过对64个质体基因组中的81个基因的分析,揭示了被子植物的亲缘关系,并确定了基因组规模的进化模式。美国国家科学研究院2007;104:19369-74。gydF4y2Ba
- 7.gydF4y2Ba
利用质体基因组规模数据分析基部被子植物之间的神秘关系。中国科学院院刊2007;104:19363-8。gydF4y2Ba
- 8。gydF4y2Ba
Ruhfel Br,Gitzendanner Ma,Soltis PS,Soltis de,Burleigh JG。从藻类到缓解植物 - 推断绿色植物(Viridiplantae)的系统发育(viridiplantae)从360塑性基因组中。BMC EVOL BIOL。2014; 14:23。gydF4y2Ba
- 9。gydF4y2Ba
王志强,王志强,王志强。植物叶绿体系统发育研究进展[j]。Am J Bot. 2018; 105:291-301。gydF4y2Ba
- 10.gydF4y2Ba
李海涛,易天生,高利明,马鹏飞,张涛,杨建平,马吉赞,Fritsch PW,蔡俊,罗勇,等。被子植物的起源与侏罗纪裂谷之谜。Nat植物。2019;5:461 - 70。gydF4y2Ba
- 11.gydF4y2Ba
公园男,Cronn R,利斯顿A.在低分类级别使用叶绿体基因组的大规模平行测序增加系统发育的分辨率。BMC BIOL。2009; 7:84。gydF4y2Ba
- 12.gydF4y2Ba
闫米,弗里奇PW,摩尔MJ,冯T,孟安,杨杰,邓T,赵开,姚X,太阳,et al。塑体系统核科学学决定了突发术的侵犯关系,并在肉体的骨干关系上脱光。mol phylocyet evol。2018; 121:198-211。gydF4y2Ba
- 13.gydF4y2Ba
Sun Y, Moore MJ, Zhang S, Soltis PS, Soltis DE, Zhao T,孟A, Li X, Li J, Wang H. early- derived eudiicots几乎所有科18个完整质体的系统基因组学和结构分析,包括被子植物IR基因含量的进化分析。分子系统学进展。2016;96:93-101。gydF4y2Ba
- 14.gydF4y2Ba
沃克JF,沃克 - 黑尔N,Vargas的OM,拉尔森DA,Stull GW。表征基因树冲突在质体推断的系统发育。peerj。2019; 7:e7747。gydF4y2Ba
- 15.gydF4y2Ba
绿色品牌。光合真核生物叶绿体基因组。植物j . 2011; 66:34-44。gydF4y2Ba
- 16.gydF4y2Ba
Schwarz EN, Ruhlman TA, Weng ML, Khiyami MA, Sabir JSM, Hajarah NH, Alharbi NS, Rabah SO, Jansen RK。整个质体的核苷酸替换率揭示了凤蝶亚科的加速速率和与豆类亚科基因组特征的相关性。中国科学(d辑:地球科学)2017;gydF4y2Ba
- 17.gydF4y2Ba
Guisinger MM, Kuehl JV, Boore JL, Jansen RK。天竺葵科质体DNA的全基因组分析揭示了前所未有的核苷酸替换增加模式。美国国家科学学院学报2008;105:18424-9。gydF4y2Ba
- 18。gydF4y2Ba
Goncalves DJP, Simpson BB, Ortiz EM, Shimizu GH, Jansen RK。质体基因树与种树不一致及系统发育信号变异。分子系统学进展2019;138:219-32。gydF4y2Ba
- 19。gydF4y2Ba
KH,李万辉,夏普总理。在植物线粒体、叶绿体和核dna中,核苷酸替代率差异很大。美国国家科学研究院1987;84:9054-8。gydF4y2Ba
- 20。gydF4y2Ba
翁曼丽,李文杰,杨文杰。天竺葵科质体核相互作用和质体核糖体基因的加速共同进化。基因组生物学进展。2016;8:1824-38。gydF4y2Ba
- 21。gydF4y2Ba
Dugas DV, Hernandez D, Koenen EJ, Schwarz E, Straub S, Hughes CE, Jansen RK, Nageswara-Rao M, Staats M, Trujillo JT,等。含羞草类豆科植物的质体进化:IR扩展,串联重复扩展,加速的进化速度gydF4y2BaclpPgydF4y2Ba.SCI代表2015年; 5:16958。gydF4y2Ba
- 22.gydF4y2Ba
斯隆DB,阿尔弗森AJ,吴男,帕尔默JD,泰勒DR。质序列,并与被子植物蝇子草属极端线粒体发散结构演变一致的近期加速。基因组Biol Evol。2012; 4:294-306。gydF4y2Ba
- 23.gydF4y2Ba
穆尔MJ,SOLTIS PS,贝尔CD,伯利JG,SOLTIS DE。质基因的系统发育分析进一步解决真双子叶植物的早期多样化。国家科学院院刊美A. 2010; 107:4623-8。gydF4y2Ba
- 24.gydF4y2Ba
Degnan JH, Rosenberg NA。基因树不一致,系统发育推断和多物种合并。趋势生态演变。2009;24:332-40。gydF4y2Ba
- 25。gydF4y2Ba
Edwards SV, Xi Z, Janke A, Faircloth BC, McCormack JE, Glenn TC, Zhong B, Wu S, lemon EM, lemon AR, et al.,等。多物种合并模型的实施和测试:系统基因组学的一个有价值的范例。分子系统学进展。2016;94:447-62。gydF4y2Ba
- 26。gydF4y2Ba
Simmons MP,Gatesy J. Coalesceary与倾斜:复杂分析与生根服药的第一个原则。mol phylocyet evol。2015; 91:98-122。gydF4y2Ba
- 27。gydF4y2Ba
猪啊,郭W,古普塔S,范W,割草机JP。质反向重复的进化动态:扩张,收缩和流失的替代率的影响。新植物。2016; 209:1747至1756年。gydF4y2Ba
- 28。gydF4y2Ba
Guisinger MM, Kuehl JV, Boore JL, Jansen RK。重排,重复和密码子使用:在被子植物家庭牛儿苗质体基因组的极端的重新配置。mol Biol Evol。2011; 28:583-600。gydF4y2Ba
- 29。gydF4y2Ba
翁曼丽,李文杰,杨文杰。扩大倒重复并不会降低替代率gydF4y2Ba天竺葵属植物gydF4y2Ba质体基因组。新植醇。2017;214:842-51。gydF4y2Ba
- 30.gydF4y2Ba
Weng ML,Blazier JC,Govindu M,Jansen RK。Geraniaceae中祖先塑性基因组的重建揭示了基因组重排,重复和核苷酸取代率之间的相关性。mol Biol Evol。2014; 31:645-59。gydF4y2Ba
- 31.gydF4y2Ba
Sun Y, Moore MJ, Lin N, Adelalu KF,孟A, Jian S, Yang L, Li J, Wang hgydF4y2BaNDH.gydF4y2Ba基因家庭。BMC基因组学。2017; 18:592。gydF4y2Ba
- 32.gydF4y2Ba
诺克斯EB。在被子植物中,桔梗科质体基因组的动态历史是独一无二的。中国科学技术大学学报(自然科学版)2014;111:11097-102。gydF4y2Ba
- 33.gydF4y2Ba
拉巴SO,什雷斯塔B,Hajrah NH,萨比尔MJ,Alharby HF,萨比尔MJ,Alhebshi AM,萨比尔JSM,吉尔伯特LE,Ruhlman TA,等人。gydF4y2Ba西番莲gydF4y2Ba质体测序揭示了广泛的基因组重排。[J] .地球科学进展,2019;gydF4y2Ba
- 34.gydF4y2Ba
Guisinger MM, Chumley TW, Kuehl JV, Boore JL, Jansen RK。质体基因组序列的意义gydF4y2Ba香蒲gydF4y2Ba(香蒲科,禾本目)为禾本科理解基因组进化。J Mol Evol。2010; 70:149-66。gydF4y2Ba
- 35.gydF4y2Ba
Shrestha B,Weng Ml,Theriot Ec,Gilbert Le,Ruhlman Ta,Krosnick Se,Jansen RK。质量基因组中的基因组重排和核苷酸取代的高度加速率gydF4y2Ba西番莲gydF4y2Ba亚因子gydF4y2BaDecalobagydF4y2Ba.分子系统学进展2019;138:53-64gydF4y2Ba
- 36.gydF4y2Ba
王志刚,王志刚,王志刚。龙胆科植物的分类、进化和生物地理学。中国科学:地球科学,2018,36(4):591 - 598。在:Struwe L, Albert VA,编辑。龙胆科:系统学和自然史。剑桥:剑桥大学出版社;2002.21 - 309页。gydF4y2Ba
- 37.gydF4y2Ba
KBv H, Kadereit JW。獐牙菜科(龙胆科-龙胆科)的系统发育和花的进化:同源性和可变比例原则。Syst Bot. 2002; 27:548-72 525。gydF4y2Ba
- 38.gydF4y2Ba
Favre A,Yuan Ym,Kupfer P,Alvarez N.Subtribe Gentianina(龙氏曲霉)的系统发育:尽管时间校准点的局限性,但生物地图推断。分类。2010; 59:1701-11。gydF4y2Ba
- 39.gydF4y2Ba
元ym,kupfer p.核核糖瘤DNA内部转录间隔件(Therianceae)的子系统龙氏蛋白酶(龙叶酸)的分子系统发育。植物系统中的Evol。1995年; 196:207-26。gydF4y2Ba
- 40.gydF4y2Ba
杨LL,李呼伦,魏L,杨T,匡DY,李MH,廖YY,陈ZD,吴H,张SZ。一个超级矩阵方法提供了龙胆全面的属级系统发育。J Syst Evol。2016; 54:400-15。gydF4y2Ba
- 41。gydF4y2Ba
Favre A,Matuszak S,Sun H,Liu Ed,元YM,Muellner-Riehl An。两个新的Gentianina(隆尼松群):gydF4y2Basinogentiana.gydF4y2Ba和gydF4y2BaKuepferiagydF4y2Ba由分子系统发育证据支持。分类。2014; 63:342-54。gydF4y2Ba
- 42。gydF4y2Ba
Jombart T,Kendall M,Almagro-Garcia J,Colijn C. Treespace:系统发育树景观的统计探索。Mol Ecol Resour。2017; 17:1385-92。gydF4y2Ba
- 43。gydF4y2Ba
Partitioned coalescence support揭示了物种树方法的偏差,并检测决定系统基因组冲突的基因树。分子系统学进展2019;139:106539。gydF4y2Ba
- 44。gydF4y2Ba
熙HC,太阳Y,薛CY。基于ITS序列数据和Swertiinae(龙胆科-Gentianeae)的分子系统发生gydF4y2BamatKgydF4y2Ba.植物多样性资源。2014; 36:145-56。gydF4y2Ba
- 45。gydF4y2Ba
何天宁,刘诗文。属的亚属分类gydF4y2Ba绅士gydF4y2Ba(龙胆科)。公牛英国人亩纳特嘘,机器人。1990; 20:169-92。gydF4y2Ba
- 46.gydF4y2Ba
张超,张志强。基于部分解析基因树的多项式时间物种树重建。BMC生物信息学。2018;19:153。gydF4y2Ba
- 47.gydF4y2Ba
傅培培,张永忠,耿宏明,陈少林.植物叶绿体全基因组序列gydF4y2Ba秦艽lawrenceigydF4y2Bavar。gydF4y2Ba扇贝gydF4y2Ba(龙叶酸)和对比例分析其对照物种。peerj。2016; 4:E2540。gydF4y2Ba
- 48.gydF4y2Ba
孙少森,付培培,周晓军,程永文,张福强,陈少林,高启波。七种植物的完整质体序列gydF4y2Ba绅士gydF4y2Ba教派。gydF4y2BaKudoagydF4y2Ba(龙胆科):洞察质基因丢失和分子进化。正面。植物SCI。2018; 9:493。gydF4y2Ba
- 49。gydF4y2Ba
Martin M,Sabater B.植物演化中的塑体NDH基因。植物理性生物化学。2010; 48:636-45。gydF4y2Ba
- 50。gydF4y2Ba
Mohanta TK, Khan A, Khan A, Abd Allah EF, Al-Harrasi A。bioRxiv。2019;gydF4y2Bahttps://doi.org/10.1101/676304gydF4y2Ba.gydF4y2Ba
- 51。gydF4y2Ba
Rogalski男,Schottler MA,蒂勒W,舒尔茨WX,博克R. Rpl33,在烟草的非必需的质体编码的核糖体蛋白,被冷胁迫条件下必需的。植物细胞。2008; 20:2221-37。gydF4y2Ba
- 52。gydF4y2Ba
Blazier JC, Ruhlman TA, Weng ML, Rehman SK, Sabir JS, Jansen RK。RNA聚合酶亚基在被子植物质体基因组中的分化是由基因组重排介导的。Sci众议员2016;6:24595。gydF4y2Ba
- 53。gydF4y2Ba
nei m,李夫。用于研究限制性内切核酸酶遗传变异的数学模型。Proc Natl Acad Sci U S A. 1979; 76:5269-73。gydF4y2Ba
- 54。gydF4y2Ba
Neubig KM,惠顿的WM,Carlsward BS,兰科MA,Endara L,威廉姆斯NH,的摩尔M.系统发育效用gydF4y2Baycf1gydF4y2Ba在兰花:质体基因比matK序列更多的变量。植物系统中的Evol。2009; 277:75-84。gydF4y2Ba
- 55.gydF4y2Ba
张旭,邓涛,Moore MJ,季勇,林宁,张辉,孟A,王辉,孙勇,孙辉gydF4y2Ba光学gydF4y2Ba(Asteraceae:Cardueae)。BMC植物BIOL。2019; 19:290。gydF4y2Ba
- 56.gydF4y2Ba
董伟,徐超,李超,孙军,左勇,石胜,程涛,郭军,周胜。gydF4y2Baycf1gydF4y2Ba是陆生植物质体DNA条形码研究中最有前途的一种。Sci众议员2015;5:8348。gydF4y2Ba
- 57.gydF4y2Ba
Philippe H,Brinkmann H,Lavrov Dv,Littlewood Dtj,Manuel M,Worheide G,Baurain D.解决困难的系统发育问题:为什么更多序列不够。Plos Biol。2011; 9:E1000602。gydF4y2Ba
- 58.gydF4y2Ba
Wicke S,Schaferhoff B,Depamphilis CW,Muller Kf。肉食锂致素叶片基因的替代速率和放松纯化选择的分类塑性血液的增加。mol Biol Evol。2014; 31:529-45。gydF4y2Ba
- 59.gydF4y2Ba
D.有害突变对中性分子变异的影响。遗传学。1993;134:1289 - 303。gydF4y2Ba
- 60.gydF4y2Ba
Trimmomatic:用于Illumina序列数据的灵活微调器。生物信息学。2014;30:2114-20。gydF4y2Ba
- 61.gydF4y2Ba
Dierckxsens N,Mardulyn P,Smits G. Novoplasty:来自整个基因组数据的细胞器基因组的De Novo组装。核酸RES。2017; 45:E18。gydF4y2Ba
- 62.gydF4y2Ba
沈建军,张旭,兰迪丝·杰比,张华,邓涛,孙辉,王洪涛。白云石(菊科,山葵科)的胞浆体进化及其系统基因组学分析。植物学报。2020;11:376。gydF4y2Ba
- 63.gydF4y2Ba
曲XJ,Moore MJ,Li Dz,Yi Ts。PGA:一种用于快速,准确,灵活的塑料批量注释的软件包。植物方法。2019; 15:50。gydF4y2Ba
- 64.gydF4y2Ba
Lowe TM,EDDY SR。Trnascan-SE:一种改进基因组序列转移RNA基因检测的程序。核酸RES。1997年; 25:955-64。gydF4y2Ba
- 65。gydF4y2Ba
Lohse M, Drechsel O, Kahlau S, Bock R. OrganellarGenomeDRAW——一套用于生成质体和线粒体基因组物理图和可视化表达数据集的工具。核酸学报2013;41:W575-81。gydF4y2Ba
- 66。gydF4y2Ba
张D,高F,Jakovlic I,Zou H,张J,李WX,王GT。文学:一种集成和可伸缩的桌面平台,用于简化的分子序列数据管理和进化系统研究。Mol Ecol Resour。2020; 20:348-55。gydF4y2Ba
- 67。gydF4y2Ba
李志强,李志强,李志强,等。基于遗传算法的系统发育分析。生物科学进展。2012;29:1695-701。gydF4y2Ba
- 68。gydF4y2Ba
Stamatakis a . RAxML版本8:系统发育分析和大系统发育后期分析的工具。生物信息学。2014;30:1312-3。gydF4y2Ba
- 69。gydF4y2Ba
胡森贝克JP,Ronquist F. MRBAYES:进化树的贝叶斯推理。生物信息学。2001; 17:754-5。gydF4y2Ba
- 70。gydF4y2Ba
罗宾逊DF,Foulds LR。进化树的比较。数学BIOSCI。1981; 53:131-47。gydF4y2Ba
- 71。gydF4y2Ba
Wickham H. Ggplot2:用于数据分析的优雅图形。第二版。纽约:斯普林格出版社;2016.gydF4y2Ba
- 72。gydF4y2Ba
Gatesy J,Meredith RW,Janecka Je,Simmons MP,Murphy WJ,Springer MS。分辨率/聚结kerfuffle:分区聚结符和哺乳动物的强大家庭级树。克莱斯。2017; 33:295-332。gydF4y2Ba
- 73。gydF4y2Ba
杨志。PAML 4:最大似然法的系统发育分析。生物化学学报2007;24:1586-91。gydF4y2Ba
- 74。gydF4y2Ba
Rozas J,Ferrer-Mata A,Sanchez-Delbarrio JC,Guirao-Rico S,Librado P,Ramos-Onsins Se,Sanchez-Gracia A.DNASP 6:大数据集的DNA序列多态性分析。mol Biol Evol。2017; 34:3299-302。gydF4y2Ba
- 75。gydF4y2Ba
Lopez-Giraldez F,Townsend JP。phydesign:用于分析系统发育信息的在线申请。BMC EVOL BIOL。2011; 11:152。gydF4y2Ba
- 76。gydF4y2Ba
池塘,弗罗斯特SD,缪斯SV。螺旋:使用系统发育的假设测试。生物信息学。2005; 21:676-9。gydF4y2Ba
- 77。gydF4y2Ba
Felsenstein J. PHYLIP -系统发育推断包(3.2版)。支序分类学。1989;5:164-6。gydF4y2Ba
致谢gydF4y2Ba
我们要感谢三位匿名审稿人,感谢他们对改进我们的手稿提出的深思熟虑的意见和建设性的建议。gydF4y2Ba
资金gydF4y2Ba
国家自然科学基金联合基金重点项目(no . U1802232);第二次青藏高原科学考察与研究(STEP)项目(no . 2019QZKK0502);国家重点研发计划项目(no . 2017YFC0505200);中国科学院战略性先导研究计划项目(XDA20050203);国家自然科学基金重大项目(31590823)。资助方在研究设计、数据收集和分析、决定发表或手稿准备方面没有作用。gydF4y2Ba
作者信息gydF4y2Ba
隶属关系gydF4y2Ba
贡献gydF4y2Ba
HW和HS构思和设计了这项研究。XZ和YS进行了从头组装、基因组注释和系统发育分析。LZ、JS1、NL和TD进行了其他分析。XZ, YS, JBL, HW, HS起草了手稿。JS1、HZ、LL和JS2进行DNA提取实验。XZ、ZL、NL和TD收集叶片材料。所有的作者讨论结果,并帮助形成研究,分析和最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准和同意参与gydF4y2Ba
不适用。gydF4y2Ba
同意出版gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
两位作者宣称他们没有相互竞争的利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意事项gydF4y2Ba
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。gydF4y2Ba
补充信息gydF4y2Ba
附加文件1:图S1。gydF4y2Ba
损失gydF4y2Barpl33.gydF4y2Ba塑料中的编码区域gydF4y2BaComastoma pulmonariumgydF4y2Ba和gydF4y2Bashertia hispidicalyx.gydF4y2Ba.有一个在编码区终止密码子gydF4y2Barpl33.gydF4y2Ba在gydF4y2BaComastoma pulmonariumgydF4y2Ba由于胞嘧啶(C)在22 bp处转变为胸腺嘧啶(T),且含有gydF4y2Barpl33.gydF4y2Ba之间的gydF4y2BapsaJgydF4y2Ba和gydF4y2Barps18gydF4y2Ba基因的质体gydF4y2Bashertia hispidicalyx.gydF4y2Ba.gydF4y2Ba图S2。gydF4y2Ba从使用RAxML 76质蛋白编码基因的未分区的级联矩阵Gentianeae的最大似然系统发生。最大似然自举(BS)的值和从MrBayes计算的PP在节点被示出,除了用100%BS和1.0 PP节点。gydF4y2Ba图S3。gydF4y2BaPhyDesign中估计的系统发育信息谱。(A)龙胆科超微树。(B) 76个质体蛋白编码基因的系统发育信息网。10个信息量最大的基因被用颜色编码并显示在右边。X轴和y轴分别代表相对时间和净系统发育信息。gydF4y2Ba表S1。gydF4y2Ba分类群包括在本研究中。gydF4y2Ba表S2。gydF4y2Ba76个蛋白编码基因的遗传特征。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.创作共用及公共领域专用豁免书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本文中提供的数据,除非另有用入数据的信用额度。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
张旭,孙勇,兰迪斯,J.B.gydF4y2Baet al。gydF4y2Ba龙胆科质体系统基因组学研究:广泛分布的基因树不一致性及其与质体基因进化速率异质性的关系。gydF4y2BaBMC植物杂志gydF4y2Ba20,gydF4y2Ba340(2020)。https://doi.org/10.1186/s12870-020-02518-00.gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-020-02518-00.gydF4y2Ba
关键字gydF4y2Ba
- 质体系gydF4y2Ba
- 系统不一致gydF4y2Ba
- GentianeaegydF4y2Ba
- 合并gydF4y2Ba
- 基因树木gydF4y2Ba
- 核苷酸替代率gydF4y2Ba
- 基因特征gydF4y2Ba
- 系统信息量gydF4y2Ba