摘要
背景
高温和干旱是作物生长发育的严重威胁。作为世界第六大谷物作物,珍珠小米不仅可以作为食物和饲料,还可以作为生物能源的来源。珍珠小米具有很强的耐热耐旱性。鉴于此,它被认为是研究植物抗逆性的理想作物,并可用于鉴定耐热基因。
结果
在本研究中,我们以Pacbio测序数据为参考序列,分析了经过高温和干旱胁迫48 h的珍珠小米的Illumina数据。总结前人研究,发现新基因26299个,新转录本63090个,基因注释数量增长20.18%。我们鉴定了2792个转录因子和1223个转录调控因子。热胁迫下差异表达的TFs有318个,TRs有149个;干旱胁迫下差异表达的TFs有315个,TRs有128个。我们通过RNA测序分别鉴定了在热胁迫和干旱胁迫下差异表达的6920个基因和6484个基因。
结论
通过Pacbio测序,我们发现了更多的新基因和新的转录本。另一方面,通过比较耐热胁迫下的差异表达基因和干旱胁迫下的差异表达基因,我们发现即使在同一途径下,珍珠粟的反应蛋白也不同。
背景
目前,剧烈的气候变化,包括酷热和干旱,正威胁着农业生产[1].根据政府间气候变化专门委员会(IPCC)的报告,全球气温预计每十年上升0.2°C,到2100年,将比目前的水平高出1.8至4.0°C。以前的一项研究表明,全球平均气温每升高1°C,主要农作物的产量就会减少以下数量:小麦6%,水稻3.2%,玉米7.4%,大豆3.1% [2].许多中纬度和亚热带干旱地区的平均降水量预计将减少,并严重影响作物产量。联合国环境规划署的一份报告显示,半干旱和干旱地区的作物产量减少了约36亿公顷(占世界高地面积的25%)。
珍珠小米(狼尾草glaucum(l)R. Br.)在世界上最重要的谷物作物如小麦、水稻、玉米、高粱和大麦之后排名第六[3.],全球种植面积为3100万公顷(ICRISAT 2016)。珍珠谷子是一种理想的生物乙醇生产植物,由于其高浓度的易提取的可发酵糖,可以用作可持续和替代能源。
珍珠谷子具有很强的耐热性,因为在一定的高温下,它的根枝和开花速度会随着新叶的生长而增加[4].高温(38/27℃)下,珍珠粟的相对生长率和净同化率(NAR)显著升高,玉米的相对生长率和净同化率略有降低[5].先前的报道认为珍珠粟是一种高度抗旱的作物。在一定程度的干旱胁迫下,珍珠谷子的抗旱性高于其他谷子(指谷子、薏米、谷子、谷子、谷子、谷子),且形态保持不变,尤其是叶面积和笋鲜干重[6,7].因此,珍珠谷子是研究谷类作物抗热抗旱机理的理想材料。根活性研究[4],种子发芽率[8]和热胁迫下珍珠谷子的幼苗生长速率[9]之前也有过报道。一些研究人员克隆并研究了HSP (sHSP [10], hsp70 [11]和HSP90 [12])基因,但仅有少量报道的存在并不能提供足够的关于热胁迫下珍珠小米的测序信息。此外,珍珠谷子在干旱胁迫下的测序研究仍然有限[13,14].
珍珠小米基因组于2017年报道[15],但由于基因组注释不完整,短读无法映射到基因。单分子实时测序技术也被称为下一代测序,如PacBio测序,允许直接产生全长转录本,使其成为转录本恢复和良好测序和/或不完整基因组序列的异构体检测的理想选择[16,17],但缺点是吞吐量低[18].使用Illumina平台的第二代测序是量化基因表达和高质量reads的有效方法。然而,由于第二代测序产生的reads长度较短,需要进行计算装配[18,19].为了避免这些问题,我们结合两种测序技术,研究了珍珠谷子在热胁迫和干旱胁迫下响应分子机制的异同。首先,我们使用Illumina测序数据对全长转录组的原始数据进行校正,其次,我们将校正后的全长转录组数据作为参考序列对短测序数据进行分析。
在本研究中,我们获得了以下结果:a.我们鉴定了63090个新转录本和26299个新基因;b.与以往研究相比,珍珠粟基因的注释率提高了20.18%;c.热休克蛋白在热应激48 h后仍有功能;d.在48 h干旱胁迫下,植株仍可通过ABA途径调控;e.即使反应机制相同,植物面对不同胁迫时也倾向于选择不同的蛋白质种类。本研究在分子水平上展示了珍珠粟在48 h热胁迫和干旱胁迫下的变化,为珍珠粟的耐受性研究提供了新思路。此外,新发现的基因为育种项目提供了更多的资源。
结果
通过全域转录组建模共鉴定出132,488个非冗余转录本和64,878个基因
全长转录组数据的获取基于PacBio Sequel第三代测序平台。通过过滤原始数据,我们去除长度小于50 bp的连接器和原始脱机数据,基于17.89 G得到6,842,837 Subreads(表1)。Subreads的平均长度为2615 bp。通过条件筛选(满分为1,质量为0.80),共获得354139个循环共识(circular consensus, CCS)。最终,使用完整的5 ' -引物、3 ' -引物和poly-A尾获得306,369条全长非嵌合读(Flnc),其中Flnc平均读长为2897条。
以PacBio为代表的第三代测序技术具有读取长度长的优点,但该技术单碱基错误率较高。为了降低高错误率,使用Illumina数据进行校正。修正数据后,共得到132,588个共识,3302个N50, 2006个N90。CD-HIT软件删除了冗余和相似的序列,结果是132,488个非冗余转录本和64,878个基因。
共注释基因62436个(96.24%)
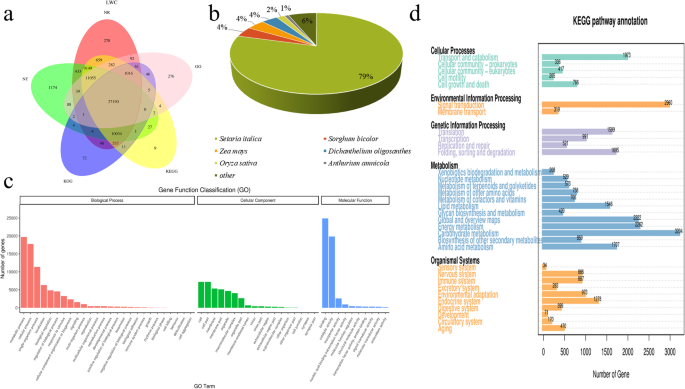
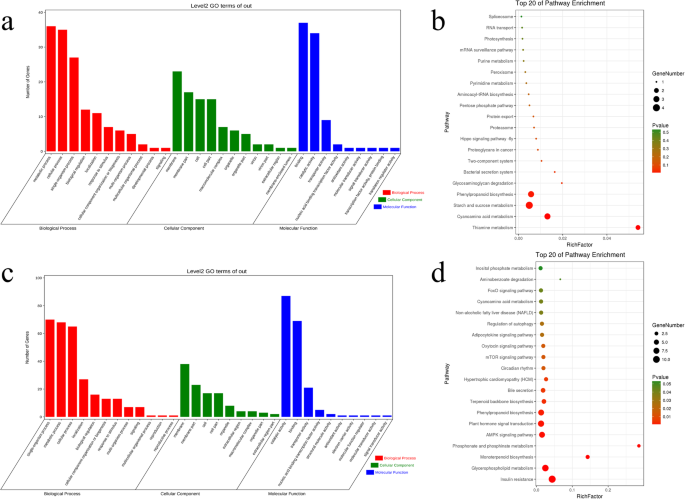
为了获得全面的遗传注释,我们使用BLASTN (E-value 1)分析了共64,878个基因的NCBI核苷酸序列(Nt)平台以及)、NCBI非冗余蛋白序列(Nr)、蛋白家族(Pfam)、真核同源群(KOG)、Swiss-prot、京都基因与基因组百科全书(KEGG)、基因本体论(GO)和BLASTx (E-value 1)平台以及)数据库。Venn图显示,所有数据库(Nr、Nt、Pfam、GO、KEGG、KOG和Swiss-prot)中同时标注了27190个基因(图2)。1a).至少有一个数据库标注了62,436个基因,其中NR数据库标注了60,870个(93.82%)基因。序列同源性比对结果显示,共发现48226条(74.33%)序列与之相反Setaria italica;有2471个序列(3.81%)有显著命中高粱二色的,然后是玉米(2422 3.73%),Dichanthelium oligosanthes(2218, 3.42%)栽培稻(1100年1.70%)。共13.01%的序列与其他物种同源(图;1b)。
共40159个(61.90%)基因标注为GO术语。GO富集分析结果表明,基因主要富集于代谢过程、细胞过程、单生物过程、细胞、细胞部分、膜、结合、催化活性、生物过程转运蛋白活性(BP)、分子功能(MF)和细胞成分(CC)(图。1c)。
在KEGG数据库中共注释了59981个基因(92.45%)。1d). KEGG通路分析显示,碳水化合物代谢途径中聚集了3204个(4.94%)基因,信号转导途径中聚集了2960个(4.56%)基因,能量代谢途径中聚集了2262个(3.49%)基因。
我们在KOG数据库中注释了39024个(60.15%)基因。仅在一般功能预测方面注释的基因有7332个(11.30%),在信号转导机制方面注释的基因有5256个(8.10%),在翻译后修饰、蛋白翻转和伴侣方面注释的基因有3997个(6.16%)。
在珍珠小米中共鉴定出2792个tf、1223个TRs、694个lncRNA和1124个替代剪接事件
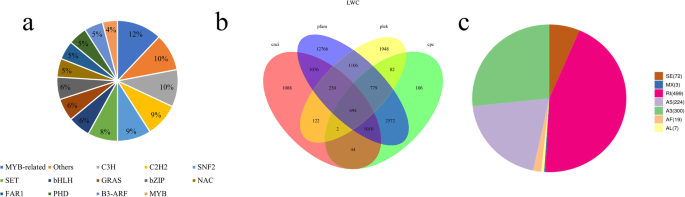
通过iTAK软件对132488个非冗余转录本进行预测,2792个基因预测为tf, 1223个基因预测为TRs(图2)。2a).共272个转录本预测为myb相关的tf。myb相关转录因子是重要的端粒结合蛋白,具有维持染色体结构完整性和调节基因转录的作用,预测有235个转录物与其他转录因子一样。共有196个转录因子被认为是C3H转录因子,它参与植物的非生物胁迫反应[20.].有190个转录本被锚定为C2H2转录因子,主要与植物生长发育和对环境胁迫的响应有关[21].
此外,使用CNCI、CPC、Pfam-scan和PLEK工具预测长链非编码RNA (LncRNA)(图。2b).基于已知注释,通过CNCI检测到4266个转录本为长非编码蛋白。与NCBI数据库一致,CPC (e-value 1)预测5389个转录本为长非编码蛋白平台以及)。通过Pfam-scan(默认参数为-E 0.001—domE 0.001)对转录本进行翻译和鉴定,结果显示20,233个转录本为长链非编码蛋白。通过PLEK(默认参数为-minlength 200)筛选得到4963个属于长蛋白的非编码转录本,通过CNCI、CPC、Pfam-scan和PLEK同时筛选得到694个转录本。
SUPPA共鉴定出1124个可选剪接事件(图2)。2c).有499个基因属于保留内含子型,300个基因为Alternative 3’Splice Sites, 224个基因为Alternative 5’Splice Sites。
光合作用蛋白可能参与抵抗热应激
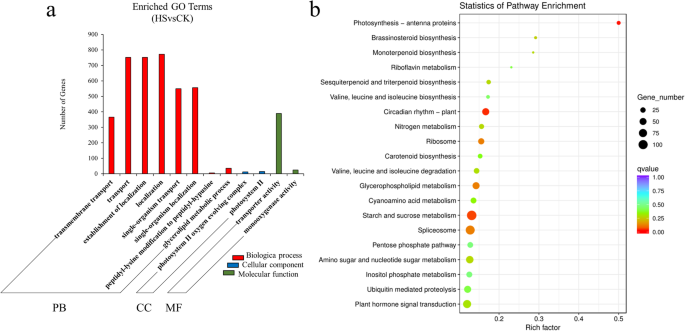
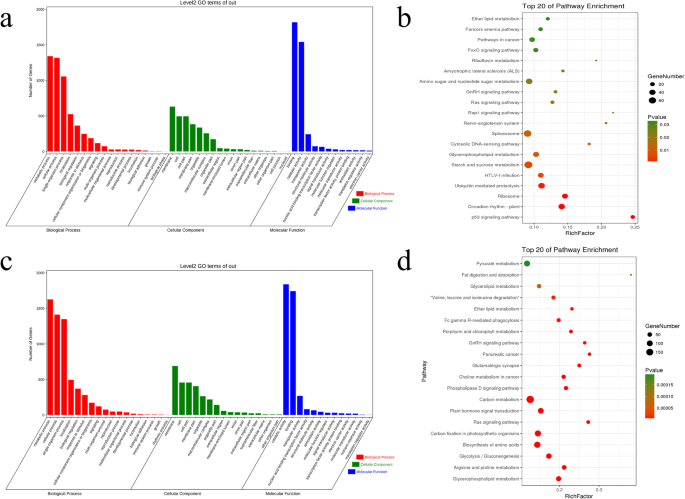
共检测到6920个差异表达基因,其中上调表达3555个,下调表达3365个。
GO富集分析显示,“跨膜转运”(上调114个,下调252个)、“转运”(上调308个,下调444个)和“定位建立”(上调308个,下调444个)是生物过程中富集最多的术语。“光系统II氧进化复合体”(5上调和7下调)和“光系统II”(5上调和10下调)是细胞成分中最丰富的术语(附加文件)2)。“转运蛋白活性”(141个上调,248个下调)和“单加氧酶活性”(104个上调,10个下调)是细胞成分中含量最多的术语。我们发现“RNA剪接”、“解剖结构形态发生”、“翻译调控”、“剪接体复合体”、“翻译调控活性”、“锌离子结合”和“核酸结合”是差异表达上调基因中GO表达最丰富的术语。“单生物定位”、“单生物转运”、“跨膜转运”、“膜固有成分”、“膜整体成分”、“膜部分”、“跨膜转运蛋白活性”、“转运蛋白活性”和“二酰基甘油O -酰基转移酶活性”是差异表达下调基因中氧化石墨烯含量最高的术语(图。3.一个)。
6920个DEGs的KEGG富集分析表明,“光合作用-天线蛋白”、“昼夜节律-植物”和“淀粉和蔗糖代谢”通路富集程度最高。对3555个上调表达的DEGs进行KEGG富集分析,发现“剪接体”、“缬氨酸、亮氨酸和异亮氨酸降解”和“缬氨酸、亮氨酸和异亮氨酸降解”是富集最多的词语,对3365个下调差异表达的基因进行KEGG富集分析,发现“光合作用-天线蛋白”、“甘油磷脂代谢”和“昼夜节律-植物”是富集最多的通路(图)。3.b)。
甘油磷脂代谢途径可能在珍珠谷子抗旱性中起重要作用
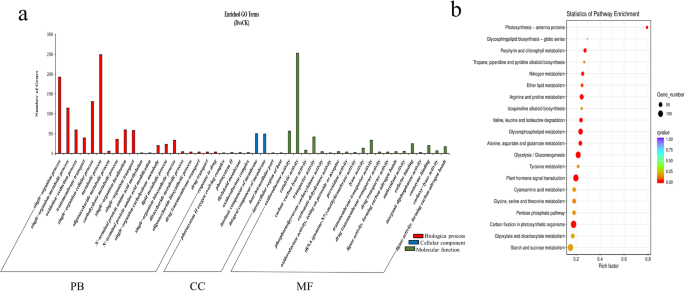
通过比较干旱处理与对照条件下的基因表达量,共得到6484个DEGs (P< 0.05)在48 h时呈上调或下调的趋势。表达上调的有3041个,下调的有3443个。
GO富集分析显示,“单生物过程”(964上调、959下调)、“单生物代谢过程”(593上调、552下调)、“氧化还原过程”(289上调、303下调)、“光系统II氧进化复合体”(0上调、3下调)、“光系统II”(0上调、21下调)、“类囊体膜”(0上调、18下调)、“氧化还原酶活性”(上调284个,下调276个)、“催化活性”(上调1254个,下调3165个)和“碳−碳裂解酶活性”(上调81个,下调17个)的含量最高。结果表明,上调的DEGs主要富集在“单生物过程”、“单生物代谢过程”、“低聚糖代谢过程”、“tRNA(鸟嘌呤- N7−)-甲基转移酶活性”、“催化活性”和“tRNA(鸟嘌呤)甲基转移酶活性”等词语中。而下调的DEGs主要富集在“单-有机体过程”、“代谢过程”、“蛋白质输入到细胞核”、“光系统II”、“光系统II氧进化复合物”、“类囊体膜”、“碳-碳裂解酶活性”、“磷酸烯醇丙酮酸羧激酶活性”和“裂解酶活性”(图)。4一个)。
KEGG富集分析结果表明,DEGs主要富集在“光合作用-天线蛋白”,“甘油磷脂代谢”(附加文件)3.)和“糖酵解/糖异生”途径。上调的DEGs主要富集在“甘油磷脂代谢”、“缬氨酸、亮氨酸和异亮氨酸降解”和“精氨酸和脯氨酸代谢”途径。下调的DEGs主要富集在“碳固定在光合生物”,“光合作用-天线蛋白”和“卟啉和叶绿素代谢”途径(图。5b)。
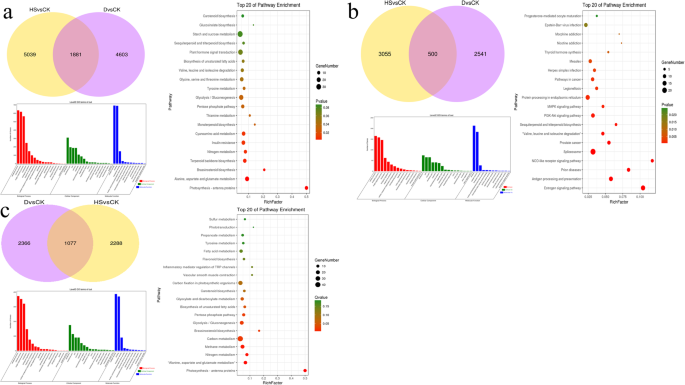
共有1881个基因参与了热胁迫和干旱胁迫反应
在热胁迫和干旱胁迫下,共发现1881个deg。5a). GO富集分析结果显示,富集程度最高的GO术语为“代谢过程”、“细胞过程”、“膜”、“膜部分”、“催化活性”和“结合”(图)。5a). KEGG富集分析显示,“光合作用-天线蛋白”、“丙氨酸、天冬氨酸和谷氨酸代谢”、“油菜素类固醇生物合成”和“萜类主干生物合成”是富集最多的通路(图)。5a). 1881个基因中,有500个基因在高温胁迫和干旱胁迫下表达上调(图2)。5b).通过氧化石墨烯富集分析,发现500个deg主要富集在“细胞过程”、“代谢过程”、“膜”、“细胞”、“结合”、“催化活性”等氧化石墨烯术语(图。5b). KEGG富集分析发现,主要富集的是“雌激素信号通路”、“抗原加工和呈递”和“nod样受体信号通路”通路(图。5b).在热胁迫和干旱胁迫下,共有1077个DEGs同时下调(图。5c).氧化石墨烯富集分析显示,主要富集在“代谢过程”、“细胞过程”、“膜”、“膜部分”、“催化活性”、“结合”等方面(图。5c). KEGG富集分析发现,主要富集在“光合作用-天线蛋白”、“丙氨酸、天冬氨酸和谷氨酸代谢”和“氮代谢”(Fid 5c)。共122个DEGs在高温胁迫下表达上调,而在干旱胁迫下表达下调。对122个deg的氧化石墨烯富集分析显示,“代谢过程”、“细胞过程”、“膜”、“膜部分”、“结合”、“催化活性”是氧化石墨烯富集最多的术语(图。6a). KEGG富集分析结果显示,122个差异表达基因主要富集在“硫胺素代谢”、“氰基氨基酸代谢”、“淀粉和蔗糖代谢”和“苯丙类生物合成”通路(图2)。6b). 182个DEGs在高温胁迫下下调,而在干旱胁迫下上调。对182个deg的氧化石墨烯富集分析显示,主要富集在“单生物过程”、“代谢过程”、“膜”、“膜部分”、“催化活性”和“结合”等氧化石墨烯方面(图2)。6c). KEGG富集分析结果表明,主要富集在“甘油磷脂代谢”、“单萜类生物合成”和“磷酸盐和磷酸盐代谢”途径(图。6d)。
分别有5039个和4603个针对热胁迫和干旱胁迫的deg。5a). GO富集分析了5039个deg,发现这些基因主要富集在“代谢过程”、“细胞过程”、“膜”、“细胞”、“结合”和“催化活性”(图。7a). KEGG富集分析显示,5039个基因主要富集在“p53信号通路”、“昼夜节律-植物”、“核糖体”和“泛素介导的蛋白水解”通路(图。7b). 4603个DEGs仅在干旱胁迫中发现,富集了“代谢过程”、“细胞过程”、“膜”、“细胞”、“催化活性”和“结合”等GO术语(图)。7c). KEGG富集分析显示,4603个DEGs主要富集在“甘油磷脂代谢”、“精氨酸和脯氨酸代谢”、“糖酵解/糖异生”和“氨基酸生物合成”通路(图2)。7d)。
讨论
共鉴定出63090个新转录本,26299个新基因,注释增加20.18%
珍珠小米是一种多用途的谷物,可以用作谷物、干草和绿色饲料。分布在干旱和半干旱地区,如印度和撒哈拉以南非洲[22].近年来,有很多关于玉米测序的报道[23,24],大米[25]和小麦[26],但只有少数研究了珍珠粟。一些研究人员利用GBS测序(测序分型)构建了640 cM的遗传连锁图谱,以鉴定与Striga和其他农艺性状相关的基因[27].2016年4月,研究人员对种植在两种气候梯度下的珍珠谷子进行了RNA-Seq分析,以探索其对气候的适应性[28].2016年5月,研究人员利用GBS测序构建了最高密度的遗传连锁图。图中SNP标记的总平均密度为23.23 SNP/cM,总长度为716.7 cM [29].2016年6月,研究人员对珍珠粟进行从头测序,鉴定霜霉病相关基因,获得684.97 Mb数据和1,295,196个高质量reads [30.].2017年,Varshney等人[15]利用全基因组霰弹枪和细菌人工染色体测序获得了珍珠粟基因组的草图。Concetta et al.(2018)通过对包括野生型和传统品种在内的221种珍珠粟的基因组测序,发现了珍珠粟的起源[31].据我们所知,在以前的研究中没有关于珍珠粟全长转录组的报道。在本研究中,我们对珍珠粟3叶期、5叶期、开花期和抽穗期4个阶段12份样品的总RNA进行了单分子长读测序。在本研究中,我们在2017年同一作物中鉴定出132488个转录本(平均长度3102 bp, N50 3302 bp),比之前报道的69998个转录本(平均长度725 bp, N501014 bp)多出63090个转录本(表2)1)。共鉴定出64878个基因,其中26299个是新发现的。在本研究中,27,190个基因(41.91%)同时在7个数据库(Nt、Nr、Pfam、KOG、Swiss-prot、KEGG和GO)中被注释,62,436个基因(96.24%)在至少一个数据库中被注释。我们用KEGG注释了59,981个基因,比之前报道的多38,526个基因(21,455个基因)。我们用SwissProt标记了51764个基因,比之前鉴定的21455个基因多30238个。这些信息为珍珠粟重要基因的鉴定和发现提供了更多的资源。我们发现1507个DEGs在高温胁迫和干旱胁迫下均有上调或下调。我们建议研究人员更详细地研究这些基因,它们不仅与耐热性有关,而且与抗旱性有关。
珍珠小米经典热相关基因的表达
先前发表的文献显示,珍珠小米具有很强的耐热性,但没有关于RNA测序的信息。我们对热应激珍珠小米进行了RNA测序,发现了与暴露于热应激产生的活性氧(ROS)相关的deg。ROS作为植物的启动信号,促使局部组织响应局部非生物胁迫刺激,但ROS基因的表达是暂时的[32,33].我们鉴定了9种与ROS产生相关的DEGs, 2种编码胺氧化酶(AOC),其余7种编码多胺氧化酶(PAO)。除了两个基因上调,其他基因下调(附加文件)4)。这些结果表明,热应激48 h后,多种参与ROS生成的基因被抑制。这与之前关于ROS产生相关基因表达的研究一致,这些基因在应激下没有连续表达[14].ROS清除酶在保护植物免受温度胁迫方面发挥着重要作用,对于在胁迫条件下产生的ROS解毒至关重要[34].我们发现10个deg编码ROS清除酶,其中5个上调(附加文件)4),而其他5个基因则下调。这些数据表明,当ROS减少时,ROS清除酶也会减少,而且这种影响是不持续的。在热应激转录因子(HSFS)的控制下,热休克蛋白(HSP)的积累在热应激反应(HSR)中发挥重要作用,进而在植物和其他生物中产生耐热性[35,36].在这项研究中,我们发现30种deg与HSPs相关;3个DEGs编码HSP100;14个DEGs编码HSP90;12个DEG编码HSP70, 1个DEG编码sHSP,均为上调DEG(附加文件4)。我们假设即使植物遭受持续的热胁迫,热休克蛋白仍能发挥作用。
珍珠谷子典型干旱相关基因的表达
干旱胁迫下,植物ABA含量增加,调节气孔的开闭,减少水分流失,维持植物水分含量[37,38].在干旱胁迫下,我们发现了5个编码9-顺式环氧类胡萝卜素双加氧酶(NCED)的DEG, 1个编码玉米黄质环氧化酶(ABA1)的DEG, 2个编码短链脱氢酶/还原酶(ABA2)的DEG,其中4个是上调的DEG,另外4个是下调的DEG(附加文件)5)。我们还发现了一个编码PYL8的DEG,它是ABA的受体。干旱胁迫下基因下调(附加文件)5)。这说明在48 h的干旱胁迫下,ABA通路仍然在干旱胁迫响应中发挥作用。前期研究发现,缓慢阴离子通道相关(SLAC)调节气孔的开闭,这是气孔关闭的必要条件[39].在干旱胁迫48 h后,发现了一个编码slac的DEG,这表明即使在长期干旱胁迫下,珍珠粟也通过调节气孔关闭来减少水分蒸发。(附加文件5)。的Asr基因家族被归类为一组新的晚胚源性丰富(LEA)基因,参与干旱胁迫的适应[40].在压力条件下,的表达Asr基因上调。我们找到了两个Asr不同表达水平的基因,在干旱胁迫下表达上调,与以往报道一致。(附加文件5)。
在相同的分子机制下,珍珠粟选择不同的蛋白质来应对不同的胁迫
结果表明,在热胁迫和干旱胁迫下,分别有5039个DEGs和4603个DEGs受到调控,表明植物对热胁迫和干旱胁迫具有不同的分子机制[41].此外,即使在两种胁迫下存在相同的机制,但所涉及的蛋白质类型、蛋白质数量以及相关基因的选择是不同的,如ROS清除机制。在干旱胁迫下,我们发现了14个与ROS清除酶相关的DEG, 2个编码SOD, 11个编码APX, 1个编码GPX,其中6个表达上调,8个表达下调。编码CAT的基因在热胁迫48 h后显著表达,而在干旱胁迫下表达不显著。干旱胁迫下编码APX蛋白的deg数量高于热胁迫。干旱胁迫下活性氧清除酶的表达与热胁迫下活性氧清除酶的表达不同,表明活性氧清除酶是特定于胁迫类型的(表2)2)。在高温胁迫和干旱胁迫下共表达了1881个deg,表明这1881个deg在高温胁迫和干旱胁迫下均发挥了重要作用。在1881个表达模式相同的基因中,有1577个表达模式相同(在热胁迫和干旱胁迫下上调或下调)的基因是对抗热胁迫和干旱胁迫所必需的,如HSPs。HSPs是真核细胞中必不可少的分子伴侣,在参与信号转导和细胞周期调节的蛋白质的折叠和激活中发挥重要作用。在干旱胁迫下,我们发现有26个DEGs编码HSP,其中11个DEGs编码HSP70, 14个DEGs编码HSP90, 1个DEGs编码sHSP,这些DEGs均上调了DEGs的表达(表2)3.)。其中12个编码HSP的deg在热胁迫和干旱胁迫下均存在。在高温胁迫和干旱胁迫下,共有304个表达模式不同的基因,说明不同胁迫对同一基因的调控水平不同。
结论
我们结合两种测序技术,研究了谷子在高温和干旱胁迫下分子机制的异同。本研究共鉴定出63090个新转录本,26299个新基因,基因功能注释提高了20.18%。在高温胁迫和干旱胁迫下,差异表达基因分别为6920个和6484个,两种胁迫下差异表达基因均为1881个。这些信息为鉴定和发掘珍珠粟的重要基因提供了额外的资源。此外,我们还发现,在相同的反应机制下,植物在面对不同类型的胁迫时会有不同的蛋白质选择。这为研究珍珠粟的抗热抗旱机理奠定了基础。
方法
植物材料、栽培和处理
本研究使用的珍珠谷子品种Tifleaf 3由四川农业大学提供,保存于中国四川省雅安四川农业大学动物科技学院草地科学系。珍珠小米种子在含有石英砂的罐子(10*15厘米)中种植,并放置在生长室中。将花盆暴露在26°C光照14小时和22°C黑暗10小时下13天,使用50% Hoagland的营养液(1 mM MgSO)4, 1mm KH2阿宝4, 1mm NH4没有3., 0.5 mM CaCl2, 0.1 mM FeNa-EDTA, 25 mM NaCl, 0.1 mM H3.薄3., 0.1 mM Na2SiO3., 1.5 μM CuSO4, 50 μ KCl, 10 μMMnSO4, 0.075 μMNa2MoO42 μM ZnSO4)。将13日龄植株分为3组(对照组、热处理组和干旱处理组)。对照组的培养条件为26℃光照14 h, 22℃暗10 h,添加50%的霍格兰营养液。热处理组植物分别暴露在40°C光照14 h和35°C黑暗10 h,并添加50%的Hoagland营养液。干旱处理组在26℃光照14 h, 22℃黑暗10 h, 50% Hoagland营养液中溶解20% PEG(聚乙二醇6000)。所有治疗时间均为48 h(2天)。
Iso-Seq的RNA制备
分别在三叶期和五叶期采集处理植株的叶片和根系样品。在珍珠粟抽穗期采集小穗、叶、茎、根。在开花阶段,收集小穗、叶、茎和根,并立即将这些样品保存在−80°C。RNA提取使用RNeasy Plant Mini Kit [42],采用RNA凝胶电泳分析RNA质量。从12个样品中提取等量的RNA(每个样品1 μg),聚合成总RNA,然后用3 μg总RNA制备SMRT文库。
PacBio Iso-Seq文库的制备和测序
Iso-Seq文库使用Isoform测序协议(Iso-Seq)、Clontech SMARTer PCR cDNA Synthesis Kit和BluePippin Size Selection System协议(太平洋生物科学公司描述的P/ N100-377-100-05和P/ N100-377-100-04)进行了一些修改。首先,将3 μl总RNA溶液加入含有单一引物的去电离水中,在72°C下孵育3分钟(3 ' SMART引物IIA来自Clontech SMARTer kit 5 ' -AAGCAGTGGTATCAACGCAGAGTACTNN-3 ')。接下来,将SMARTER II A寡核苷酸(来自Clontech SMARTER kit 5 ' -AAGCAGTGGTATCAACGCAGAGTACXXXXX-3 ')、5X第一链缓冲液、DTT、dNTP混合物、RNase抑制剂和SMARTScribe逆转录酶添加到混合物中,并在72°C下孵育1小时。最后,在70℃时终止反应。经过23个PCR循环后,用BluePippin Size Selection System对PCR产物进行长度筛选,分为1-2 Kb、2-3 Kb和3-10 Kb片段。对cDNA进行尺寸筛选后,进行12循环PCR反应。PCR扩增产物采用Iso-Seq协议构建SMRTbell Template文库。通过对测序引物(SMRTbell Template Prep Kit 1.0的组成部分)进行退火,并将聚合酶结合到引物退火的模板上,制备这些文库进行测序。
Iso-Seq数据分析
原始数据处理采用SMRTlink 5.1软件(https://www.pacb.com/videos/tutorial-minor-variant-analysis-smrt-link-v5-0-0/.参数设置为min_length 50, max_drop_fraction 0.8, no_polish TRUE, min_zscore−9999.0,min_passes 2, min_predicted_accuracy 0.8, max_length 15,000,从sub read BAM文件中生成循环一致序列。使用pbclassification .py, ignorepolyA false, minSeqLength 200将样本分为全长和非全长读取。生成非全长和全长FASTA文件,送入聚类步骤,执行isoform-level聚类(ICE),然后进行最终的Arrow打磨,hq_quiver_min_accuracy 0.99, bin_by_primer false, bin_size_kb 1, qv_trim_5p 100, qv_trim_3p 30。由于Iso-Seq测序中核苷酸缺失和错配的频率远高于较短的高通量测序,因此使用LoRDEC软件来纠正基于Illumina RNA-Seq数据的一致测序中的额外核苷酸错误。使用CD-HIT软件(−c 0.95 -T 6 -G 0 -aL 0.00 -aS 0.99)获得校正读数中的冗余,以获得后续分析的最终转录本。
基因功能注释
我们使用以下5个数据库对所有预测的蛋白编码序列进行比对,NCBI非冗余蛋白(NR, cutoff E value≤1e-5), NCBI非冗余核苷酸(NT, E值≤1e-5)、Swiss-Prot蛋白(http://www.expasy.org/sprot/,截止E值≤1e-5), kog (http://www.ncbi.nlm.nih.gov/COG/,截止E值≤1e - 3) [43],蛋白质家族(Pfam:http://pfam.sanger.ac.uk/,截止e值≤0.01),KEGG (http://www.genome.jp/kegg,截止E值≤1e-5)路径[44].利用Blast2GO (http://www.blast2go.com) GO注释程序(http://www.geneontology.org)(截止e值≤1平台以及)。
cd预测
来自cDNA的蛋白质编码序列由ANGEL管道(ANGLE的长读实现)识别。我们使用密切相关的物种来确保蛋白质序列经过ANGEL训练,然后对给定的序列进行ANGEL预测。
TF分析和lnrna分析
用iTAK软件预测转录因子。
我们使用四种软件预测lncRNA: a) CNCI(编码-非编码-索引,参数为默认参数)是一种有效的软件,可以在不依赖已知注释的情况下,通过相邻核苷酸三联体的谱来区分蛋白编码序列和非编码序列。b)使用CPC (Coding Potential Calculator)评估转录本的ORF范围和质量,并从NCBI中检索测序的基础真核生物蛋白数据库(e值为“1”)平台以及),以识别编码文本和非编码文本。c)翻译所有转录本,然后使用Pfam扫描(−E 0.001—domE 0.001)来确定它们是否具有已知蛋白质家族的结构域。d)使用PLEK SVM分类器预测Lnc-RNA(−minlength 200)。PLEK SVM分类器通过优化K-mer方法来识别编码蛋白质的转录本,该方法消除了参考基因组和注释的需要。上面描述的四个软件程序都识别出了非编码转录物,这些转录物被鉴定为lnrna。
RNA- seq的RNA制备
3个处理同时进行。处理48小时后,我们随机选择16棵幼苗的叶子,收集在低温小瓶中,并立即保存在−80°C(附加文件)1)。每个处理设置3个生物重复。使用RNeasy Plant Mini Kit从样品中提取RNA,并使用RNA凝胶电泳检测RNA质量。
RNA-Seq文库制备和测序
RNA纯度由NanoDrop分光光度计检测(美国加州),RNA浓度由Qubit RNA检测试剂盒在Qubit 2.0荧光计系统中测定(美国加州)。该文库使用NEBNext®UltraTM定向RNA文库Prep Kit for Illumina®(California, USA)构建。使用NEBNext®Poly (A) mRNA磁分离模块富集mRNA,并添加片段缓冲液将mRNA分解成短片段。用随机六聚体引物合成了一条cDNA。通过添加缓冲液、dNTPs和DNA聚合酶i合成cDNA第二链。双链cDNA用AMPure XP珠纯化。最后对纯化后的cDNA进行修复;添加一条尾巴并测序,用AMPure XP珠子选择片段大小。最后通过PCR富集得到最终的cDNA文库。量子比特2.0用于初步量化;使用Agilent 2100检测文库插入片段,最后使用Illumina Hi-Seq 2000进行测序。 A total of 9 RNA-Seq libraries were established.
基因表达水平的量化
通过RSEM鉴定每个样本的基因表达水平[45].将Illumina测序生成的干净数据映射到SMRT测序数据中,从映射结果中获得每个基因的读计数[46].将每个基因的读计数值转换为FPKM值(Fragments per Kilobase Million),选取FPKM> 0.3的基因进行分析。
DEGs的鉴定与功能分析
差异表达分析采用DESeq R包(1.10.1)[47]以识别热胁迫和对照样品之间以及干旱胁迫和对照样品之间的deg。DESeq是一个统计程序,它使用基于模型的负二项分布来确定数字基因表达数据中的差异表达。P率由P .adjust函数调节,以控制错误率。基因有一个调整PDESeq软件求得的-value <被认为是差异表达,以log2 (Group1/Group2)≥1的绝对值为阈值确定的差异表达的显著性。
我们使用GOseq R包对DEGs进行GO富集分析。该软件包基于Wallenius非中心超几何分布,估计和调整deg长度的偏好。最后,利用KOBAS软件对DEGs进行KEGG富集分析[48].
数据和材料的可用性
支持本文结论的数据集包含在本文(及其附加文件)中。珍珠小米测序数据库可从NCBI下载,登录号为SRR11816223,数据将根据通讯作者的合理要求进行共享。
缩写
- 度:
-
差异表达基因
- 走:
-
基因本体论
- KEGG:
-
京都基因与基因组百科全书
- HSP:
-
热休克蛋白
- ROS:
-
活性氧
- AOC:
-
胺氧化酶类
- PAO:
-
聚胺氧化酶类
- hsf:
-
热应激转录因子
- nc:
-
9-cis-epoxycarotenoid加双氧酶
- ABA1:
-
玉米黄质环氧酶
- ABA2:
-
短链脱氢酶/还原酶
- 线性:
-
慢负离子通道相关
- SOD:
-
超氧化物Dismutas
- 猫:
-
过氧化氢酶
- apx型:
-
aseorbateperoxidase
- 李:
-
食用胚性丰富
参考文献
- 1.
植物常见和独特胁迫响应基因的鉴定拟南芥通过RNA-Seq元分析。弗吉尼亚理工大学;2018.
- 2.
赵超,刘波,朴松,王旭,Lobell DB,黄艳,黄敏,姚勇,Bassu S, Ciais P.气温升高降低全球主要作物产量的四个独立估算。中国生物医学工程学报,2017;29(3):344 - 344。
- 3.
杨晓明,杨晓明,杨晓明,等。半干旱热带环境下固氮菌、农家肥和氮肥对珍珠谷子杂交种产量的影响。农学通报,2003;49(1):21-4。
- 4.
皮尔森CJ。狼尾草的热适应:幼苗发育。植物生态学报,2007;2(3):413-24。
- 5.
杨晓明,陈晓明,陈晓明,等。水稻籽粒和玉米籽粒生长发育与营养关系的研究。生物工程学报。2004;48(1):81-6。
- 6.
黄志刚,李志刚,李志刚。珍珠谷子抗旱性中无机组分在渗透调节和叶片折叠中的作用。中国生物医学工程学报。2010;29(4):344 - 344。
- 7.
陈志伟,陈志伟,陈志伟。珍珠谷子根系深层吸水能力和水分利用效率的比较研究。植物科学学报,2005;8(4):454-60。
- 8.
Ong CK, Monteith JL。珍珠粟对光照和温度的响应。作物学报1985;11(2-3):141-60。
- 9.
废人TE。新南威尔士州西北平原早季土壤温度对夏季作物出现的影响。德国农业科学。1971;11(48):39-44。
- 10.
Nitnavare RB, Yeshvekar RK, Sharma KK, Vadez V, Reddy MK, Reddy PS.半干旱热带C4谷物植物狼尾草(Pennisetum glaucum, L.)热休克蛋白10 (Hsp10)的分子克隆、特征表征和表达分析。Mol生物学杂志2016;43(8):861-70。
- 11.
李文敏,李文敏,李文敏,李文敏,李文敏。白狼草细胞质Hsc70基因的克隆和鉴定及其对非生物胁迫的保护作用。中国生物医学工程学报,2010;29(3):344 - 344。
- 12.
王志刚,李志刚,王志刚,王志刚。白狼草细胞质Hsp90基因的分子特征、表达及其在非生物胁迫适应中的作用。基因。2011;474(1):第29 ~。
- 13.
Bidinger F, Mahalakshmi V, Rao GDP。珍珠粟(Pennisetum americum (L.))抗旱性评价Leeke)。2基因型对应激反应的估计。德国农业学报,1987;38(1):49-59。
- 14.
Choudhary M, Jayanand PJC。珍珠粟(Pennisetum glaucum L.R. Br.)的转录谱分析鉴别差异表达的干旱响应基因。植物学报。2015;21(2):187。
- 15.
Varshney RK, Shi C, Thudi M, Mariac C, Wallace J, Peng Q, He Z, Zhao Y, Wang X, Rathore a .干旱环境下珍珠粟基因组序列为改良农艺性状提供了资源。中国生物工程学报,2017;35(10):969-76。
- 16.
Abdelghany SE, Hamilton M, Jacobi JL, Ngam P, Devitt N, Schilkey F, Benhur A, Reddy ASN。高粱转录组的单分子长读研究。Nat Commun, 2016;7:11706。
- 17.
王敏,王鹏,梁峰,叶震,李娟,沈超,裴林,王峰,胡娟,涂林。异源多倍体棉花选择性剪接的格局、复杂性与调控。中国生物工程学报,2017;29(1):344 - 344。
- 18.
Rhoads A, Au KF。PacBio测序及其应用。生物信息学。2015;13(5):278-89。
- 19.
古德温,麦克弗森,JD,麦康比WR。成年:下一代测序技术的十年。植物学报,2016;17(6):333-51。
- 20.
蒋爱安,徐志生,赵光元,崔晓宇,陈明,李立龙,马一智。陶氏蛇C3H锌指转录因子家族及成员干旱应答的全基因组分析植物分子生物学研究。2014;32(6):1241-56。
- 21.
植物c2h2型锌指转录因子在胁迫响应中的参与。中国植物科学,2012;185-186(4):78-85。
- 22.
Anuradha N, Satyavathi CT, Chellapilla B, Thirunavukkarasu N, Sankar SM, Singh SP, Meena MC, Singhal T, Srivastava RK。利用关联作图破解珍珠谷子高粒铁锌含量的基因组区域。中国植物科学,2017;8:412。
- 23.
孙松,周勇,陈娟,史军,赵红,赵红,宋伟,张明,崔勇,董霞。Mo17与其他玉米基因组的广泛种内基因顺序及基因结构变异。植物学报。2018;50(9):1289。
- 24.
王波,曾娥,Regulski M, Clark TA,韩涛,焦杨,陆震,Olson A, Stein JC, Ware D.利用单分子长读测序揭示玉米转录组的复杂性。Nat Commun, 2016;7:11708。
- 25.
王伟,Mauleon R, Hu Z, Chebotarov D, Tai S S, Wu Z, Li ming,郑涛,Fuentes RR,张峰。亚洲栽培水稻3010个不同种质的基因组变异。自然。2018;557(7703)。
- 26.
王旭,陈松,石霞,刘东,马超。面包小麦热敏感信号的杂交测序研究。植物J. 2019。
- 27.
Moumouni KH, Kountche BA, Jean M, Hash CT, Vigouroux Y, BIG H, Belzile F.珍珠粟遗传图谱的构建,狼尾草glaucum(l)r . Br。,using a genotyping-by-sequencing (GBS) approach. Mol Breed. 2015;35(1):5.
- 28.
Berthouly-Salazar C, Thuillet AC, Rhoné B, Mariac C, Ousseini IS, Couderc M, Tenaillon MI, Vigouroux Y.基因组扫描显示选择作用于与野生珍珠粟胁迫反应相关的基因。分子生物学杂志,2016;25(21):5500-12。
- 29.
Punnuri SM, Wallace JG, Knoll JE, Hyma KE, Mitchell SE, Buckler ES, Varshney RK, Singh BP。珍珠谷子叶斑病高密度连锁图谱的建立及基因分型测序标记。植物基因组,9(2)。
- 30.
Kulkarni KS, Zala HN, Bosamia TC, Shukla YM, Kumar S, Fougat RS, Patel MS, Narayanan S, Joshi CG。新转录组测序分析珍珠粟霜霉病(Sclerospora graminicolaSacc.)相关候选基因交互。前沿植物科学,2016;7(52):847。
- 31.
Burgarella C, Cubry P, Kane NA, Varshney RK, Mariac C, Liu X, Shi C, Thudi M, Couderc M, Xu X.从珍珠粟基因组推断西撒哈拉驯化中心。《自然生态》,2018。
- 32.
亚伦B, Ron M, Nobuhiro S. ROS在植物胁迫信号传递中的关键作用。中国机械工程学报,2014;26(5):529 - 529。
- 33.
王玲,马克波,卢志刚,任世祥,金斌。拟南芥叶片在长时间升温和热休克下的差异生理、转录组和代谢组反应。BMC植物生物学2020;20:86。
- 34.
大滨N,佐藤H, Shinozaki K, Yamaguchi-Shinozaki K.植物热胁迫应答的转录调控网络。植物科学进展,2017;22(1):53-65。
- 35.
杨晓明,李志刚,李志刚,等。植物热胁迫响应和耐热性的研究进展。中国植物科学,2013;4(3):329。
- 36.
杨晓明,杨晓明,杨晓明,等。植物热感知和信号传导的研究进展。植物学报。2011;19(3):556-65。
- 37.
McAdam SA, Brodribb TJ, Banks JA, Hedrich R, Atallah NM, Cai C, Geringer MA, Lind C, Nichols DS, Stachowski K.脱落酸对维管植物蒸煮前性的控制。中国生物医学工程学报(自然科学版),2016;
- 38.
Mcadam SAM, Brodribb TJ。蕨类和石松的保护细胞对内源性脱落酸没有反应。植物学报。2012;24(4):1510-21。
- 39.
Triin V, Hannes K,永飞W, Noriyuki N, Wai-Yin C, Gabriel V, Airi LK, Mikael B, Heino M, Radhika D. SLAC1是植物保护细胞s型阴离子通道在气孔信号传导中的作用。自然。2008;452(7186):487 - 91。
- 40.
马旺加·罗,卢鹏,金基伦古,陆红,王旭,蔡旭,周震,张震,Salih H,王凯。陆地棉晚期胚胎发生丰富蛋白家族的特征及其在抗旱性中的作用。BMC基因工程,2018;19(1):6。
- 41.
Zandalinas SI, Mittler R, Balfagón D, Arbona V. Gómez-Cadenas A:植物对干旱和高温组合的适应性。中国生物医学工程学报。2017;42(1)。
- 42.
黄丽龙,闫海东,赵XX,张晓强,王杰,Frazier T,尹刚,黄霞,闫东峰,臧文杰,等。通过转录组分析鉴定热胁迫下果园草(Dactylis glomerataL.)的差异表达基因并开发分子标记。分子生物学杂志,2015;15(6):1497-509。
- 43.
黄玲,冯刚,颜宏,张震,Bushman BS,王杰,Bombarely A,李敏,杨忠,聂刚。基因组组装为研究园草基因组进化和开花调控提供了新的思路。植物生物技术,2019。
- 44.
周松,陈娟,赖勇,殷刚,陈鹏,Pennerman KK,闫红,吴波,张宏,易欣。草种狼尾草花青素合成调控的代谢组和转录组综合分析。作物学报。2019;138:111470。
- 45.
Bo L, Victor R, Stewart RM, Thomson JA, Dewey CN。带有读作图不确定性的RNA-Seq基因表达估计。生物信息学,2010,26(4):493 - 500。
- 46.
李B,杜威CN。RSEM:从RNA-Seq数据中精确的转录物定量,有或没有参考基因组。生物信息学杂志,2011;12(1):1 - 16。
- 47.
王丽坤,冯志兴,王曦,王晓武,郑雪功。一种利用RNA-seq数据识别差异表达基因的R包。生物信息学,2010,26(1):136 - 8。
- 48.
丁明峰,吴桂杰。基于转录组序列的水稻抗旱性相关功能基因SSR位点分析马尾松在干旱胁迫下。2018年决议。
确认
不适用。
资金
这项工作得到了t重庆市绩效激励引导专项(cstc2019jxjl80010)、四川省科研基金(2016NYZ0036)、现代农工技术研究体系(CARS-34)和现代农业产业体系四川牧草创新团队(SCCXTD-2020-16)。
作者信息
从属关系
贡献
MS、DH和AZ参与了材料处理和转录组数据分析。IK、HY、XW和XZ提供生物信息学支持。XZ对论文进行了修改。JZ, LH设计了研究,提供了监督,并贡献了手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们与这项工作没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
孙M,黄D,张A。et al。基于Pacbio全长转录组测序的珍珠谷子热胁迫和干旱胁迫转录组分析。BMC植物生物学20.323(2020)。https://doi.org/10.1186/s12870-020-02530-0
收到了:
接受:
发表:
关键字
- 珍珠粟
- Pacbio测序
- Illumina公司测序
- 热应力
- 干旱胁迫