摘要
背景
抽穗期是小麦最重要的农艺性状之一,它在很大程度上影响着小麦对不同农业生态条件的适应性和产量潜力。小麦抽穗期调控基因的鉴定和诊断标记的开发有助于我们了解这一过程的遗传控制。
结果
在这项研究中,我们通过杂交γ射线诱导的早期抽穗突变体(eh1晚熟品种伦选987。通过对由不同RILs组成的RNA和DNA库的大量分离分析(Bulked isolation Analysis, BSA),在5B染色体上发现了一个抽穗日期的数量性状位点(QTL),进一步的遗传连锁分析将QTL限制在3.31 cM的区域内。然后我们在春化基因的第一个内含子中发现了一个大的缺失VRN-B1在eh1,并表明它与RIL群体的抽穗表型相关。然而,与野生型相比,突变位点并不是导致突变体早熟的原因。RNA-seq分析表明Vrn-B3还有一些新发现的基因,包括-淀粉酶1 (BMY1)和花药特异蛋白(即时战略游戏)在突变体和早熟池中均有高表达Vrn-B1与伦选987和晚抽穗池的基因型进行了比较。差异表达基因(DEGs)富集分析确定了几个先前报道的与开花有关的关键途径,包括脂肪酸伸长、淀粉和蔗糖代谢以及类黄酮生物合成。
结论
新标志物的开发Vrn-B1本研究为优化小麦抽穗期的标记辅助育种提供了另一种解决方案,而DEGs分析为小麦抽穗期优化提供了基础信息VRN-B1监管的研究。
背景
小麦是世界上种植最广泛的粮食作物之一,这种重要的粮食作物能否在不同的农业生态条件下成功适应,主要取决于开花和成熟时间。抽穗期与作物成熟期和产量密切相关。抽穗期变异相关基因的鉴定和分离将有助于更好地理解控制植物开花时间的遗传途径,并为培育适应各种条件的高产品种提供有效的遗传资源[1].
植物已经进化出多种基因途径来调节开花途径,包括春化要求和光周期敏感性,整合外部和内部信号以适应气候条件的变化。春化是指许多适应温带气候的植物必须长时间暴露在低温下才能开花[2].在小麦中,春化诱导的开花途径主要是由调控春化处理基因系列:VRN1[3.),VRN2[4),VRN3[5),VRN-D4[6].这些基因的鉴定和特征进一步加深了我们对春化介导的谷物开花调控网络的理解。MADS-box转录因子VRN1基因,首次被发现于小麦属植物monococcum在春化后加速开花的作用[3.].VRN2编码锌指- cct结构域转录因子并抑制谷物开花[4,7,8].CCT域在VRN2是小麦和大麦春化的关键元素,CCT结构域的突变消除了春化的要求[9,10].VRN3是同源的拟南芥光周期的基因开花位点t (ft)其基因产物作为一种移动信号蛋白,从叶片移动到茎尖分生组织,以加速开花[5].VRN-D4起源于染色体臂5AL向近端插入的约290 kb区域,包含的一个拷贝VRN-A1[6].
VRN1在小麦中有很好的报道,并且与拟南芥花分生组织特征基因花椰菜(cal),无瓣(ap1),FURITFULL(富尔语)[3.,11,12,13,14].在六倍体小麦中,小麦的同源拷贝VRN1该基因存在于染色体5A、5B和5D的近端区域,并已被指定VRN-A1,VRN-B1而且VRN-D1,分别。等位变异在VRN1基因座是小麦春化需要量遗传变异的主要来源之一。三种同种异体中任一种的单一显性等位基因足以赋予春季生长习性[15,16,17].之前的一项研究表明Vrn1春季生长习性在第一个内含子处有较大的缺失,在隐性缺失区有一个2.8 kb的保守片段vrn1对小麦春化需求有重要影响[16].此外,研究了核糖核酸免疫沉淀片段3 (RIP3)的多态性VRN-A1第一个内含子提示RIP3中单核苷酸多态性的影响VRN-A1转录本水平,与不同地理区域冬小麦品种抽穗期的差异有关[17].显性和隐性等位基因之间的差异与VRN-B1基因座位于第一个内含子附近,主要包括5 ' -UTR区域的大缺失、snp、小缺失和大插入[16,18,19,20.,21,22].
的VRN1- - - - - -VRN2- - - - - -VRN3调节反馈网络集成春化和光周期信号,精确调控小麦开花时间。大麦中含有大量的VRN2压抑的表达VRN3抑制开花。在小麦方面,上调VRN1转录水平导致开花抑制因子表达水平下降VRN2春化后,加速了诱导VRN3表达水平,从而诱导春季开花[23,24].上位论分析表明VRN2与VRN1它们有着共同的遗传途径[25].VRN3的表达上调VRN1通过与基本亮氨酸拉链(bZIP)转录因子FDL2相互作用[26].FDL2与bZIP转录因子FD同源拟南芥的启动子中结合ACGT元素VRN1[26,27].的表达式VRN3还能受竞争影响吗有限公司而且VRN2与核因子- y (NF-Y)复合物相互作用,在春化和光周期季节信号的整合中发挥重要作用[28].
在小麦抽穗期的控制上,已经发现了许多QTL。迄今为止,已在小麦中检测到240多个与抽穗期相关的QTL [29,30.],对小麦育种中的标记辅助选择具有重要意义。然而,这些基因座下的大多数基因仍然未知。本研究通过Bulked isolation Analysis (BSA)和遗传连锁作图,在5B染色体上鉴定了一个有关抽穗时间的QTLVRN-B1在RIL群体中参与抽穗时间变化的基因,但它不是突变体中提早抽穗表型的原因(eh1)与野生型(WT,中元9号)比较。为了鉴定受VRN-B1,我们鉴定了早抽穗和晚抽穗间差异表达的基因,并研究了其相关途径。本研究为小麦抽穗期的标记辅助育种提供了重要线索,拓宽了我们对小麦抽穗期遗传调控的认识VRN-B1基因。
结果
RIL群体的构建与抽穗时间的变化
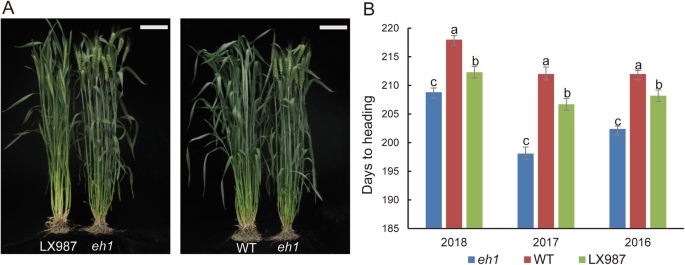
早期抽穗突变体eh1经284 Gy γ射线处理,对中原9号小麦种子进行诱变。为了定位导致抽穗期变异的QTL,本研究采用杂交方法选育了400个重组自交系eh1选育优良品种伦选987 (LX987),该品种抽穗较晚,有轻微缺陷。LX987与eh1, WT和eh1如图所示。1a.的高清eh1在野外条件下,3年内比WT早10-14天(图;1b,表S1).LX987的HD比的晚4-8天eh1,差异有统计学意义(图;1b,表S1).在3年的时间内测量RILs的HD表明,HD的最大值为215天,最小值为197天(表S1,年代2).此外,RIL人群的HD呈连续分布(图S1),说明开花时间在RIL中是一个数量性状。
利用BSR-seq方法定位早期抽穗基因
Bulked secant RNA-Seq (BSR-Seq)是一种高效的基因定位方法[31],特别是在具有大基因组的小麦植物中。我们通过BSR-Seq来定位RIL群体中控制HD的QTL。基于3年期间收集的HD数据,在早熟株系开始抽穗时对21个早熟株系和22个晚早熟株系的幼穗进行了采样。分离单个样品的RNA,构建早、晚航向RNA体。同时,对两个亲本系的6个个体进行了RNA-Seq分析。通过RNA-Seq共从两个亲本和两个bulk中获得357,941个snp。共筛选出160个多等位基因snp和180,469个reads < 4的位点。692222个具有相同基因型的SNPs和来自与相应亲本不同的bulk的61288个SNPs也被丢弃。最后,在所有染色体上鉴定出了46,802个可靠的高质量snp,并用于进一步分析。
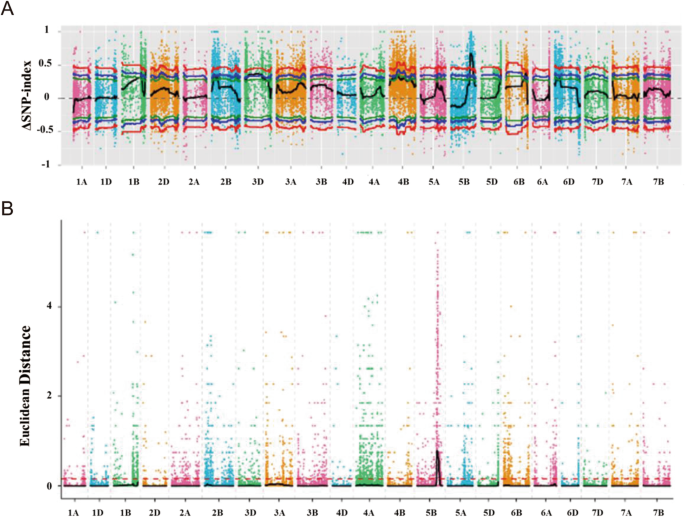
根据reads和亲本的基因型计算Δ (SNP-index)。结果表明,在RIL群体中,与HD相关的QTL主要集中在5B染色体546.9 Mb - 636.6 Mb区域,该区域存在SNP-index峰(图2)。2a).通过计算欧几里得距离(ED)值来鉴定HD的控制位点,根据中国春季参考序列的基因信息(http://www.wheatgenome.org/)(图。2b). SNP-index和ED分析均在5B染色体上发现了一个共同的区间,表明5B染色体89.57 Mb的区域含有导致RILs抽穗变异的QTL。
5B染色体上与HD相关的相同区域被BSA检测到,由各种RILs组成
根据2年的表型数据,分别使用3个极早抽穗期大群(每个大群有18-22个早抽穗期株系)和3个极晚抽穗期大群(每个大群有16个晚抽穗期株系)进行BSA分析。采用660 K SNP阵列对6个bulk和2个亲本进行基因分型。选择一对早、晚抽穗体中表现出不同基因型的SNP,以及表型相同的体与亲本间表现出相同基因型的SNP,统计21条染色体间的SNP数。结果显示,在头穗期早期和头穗期晚期1号染色体2A和5B上检测到大量SNP(图S .2A),在染色体5B、2B和3A上的早期和晚期头体2(图S2B)在染色体3B、5B、2B和6A上(图S . 3)2C),表明这些染色体上存在控制HD的qtl。分析了5B染色体上SNP频率的分布。观察到5B染色体540 ~ 600 Mb的高峰值区域在三对体块中重叠(图S2D),与BSR-Seq分析一致。
通过基因作图确定区域
为了验证基于bsa的映射区域,在5B染色体上选择87个rna - seq衍生的snp进行KASP标记的开发。利用PolyMarker对3个小麦基因组上的特异性snp进行了筛选[32],在亲本系和种群系上进行了确认,最终开发出20个特异性KASP标记(表S3.).鉴定了RIL群体中400个系的基因型。我们利用QTL IciMapping技术在5B染色体上生成了一个跨度为112.83 cM的连锁图,平均遗传距离为5.6 cM。3.).结合3年多的遗传和表型数据,在两个标记之间的区域检测到一个LOD评分大于22.8的稳定QTL,ch14而且ch8,遗传距离为3.31 cM,对应物理位置为560.4 ~ 580.1 Mb(图;3.).
的Vrn-B1是导致RILs中HD变化的原因吗
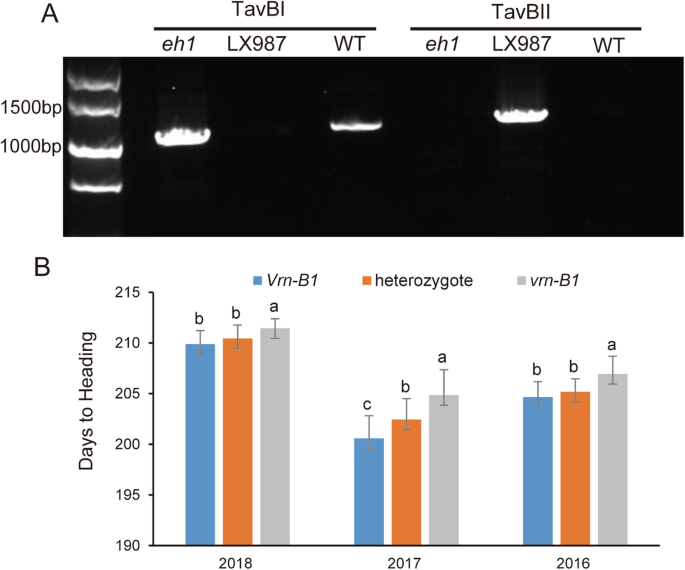
春化基因VRN-B1位于染色体5B上,是与小麦抽穗和开花有关的主要位点[3.].在自然变异中,占优势Vrn-B1具有春季生长习性的等位基因不同于隐性等位基因vrn-B1具有冬季生长习性的,由于第一个内含子的大量缺失Vrn-B1[16].我们发现5B染色体上的映射区域含有VRN-B1基因。目的检测5B染色体上控制HD的主要QTL是否与HD相关Vrn-B1基因,我们使用了以前研究开发的标记[16],但未能在WT中得到PCR产物eh1.因此,具体标记为Vrn-B1(图S3.A、表S3.).为VRN-B1基因标记,两对基因组特异性引物设计,以确定是否在第一个内含子的大缺失Vrn-B1(图S3.一).中均检测到扩增产物eh1和WT,但没有使用TavBI引物对从LX987分离的DNA产生。然而,用TavBII引物对从LX987 DNA中扩增出PCR产物(图。4A),表示存在较大的删除eh1和WT,但在LX987中没有。基因型VRN-B1在eh1、WT和LX987的基因型差异提示VRN-B1在RIL群体中,WT与LX987之间的遗传背景差异是主要原因,而γ射线照射与遗传背景差异无关。然后我们鉴定了基因型VRN-B1结果表明,199个RIL群体中均存在Vrn-B1等位基因显示显著(P< 0.001)比185个具有vrn-B1等位基因。的Vrn-B1大约提前2.7天开始排队vrn-B1平均线(图;4b).此外,杂合子的抽穗时间为206天,优势度为0.28,说明Vrn-B1有一个小的主导效应。进一步测序VRN-B1在WT,eh1和LX987发现引物Intrl/B/F的3 '端A变为G,中间C变为A [16]在WT和eh1(无花果。3.A, B).除了WT和第一个内含子的大量缺失外eh1,仅在中检测到位于大缺失下游的37 bp缺失eh1(无花果。3.A)。然后我们开发了针对37 bp缺失的特异性标记物(图S3.C),在RIL群体中进行基因型鉴定,发现37 bp缺失与大缺失共分离。进一步分析使用F2由杂交获得的1060个个体eh1WT表明37bp的缺失与HD的变化无关(图S4,P= 0.757)。
基因标记的发展VRN-B1和抽穗时间的变化VRN-B1等位基因。一个中删除的识别VRN-B1通过两个互补的引物。产品在eh1WT和TavBI,而TavBII没有,表明这一区域在两种实验中都不存在eh1全长凝胶见补充图9.b与不同的RILs的航向天数VRN-B12016年、2017年和2018年的等位基因。数值为平均值±标准差。不同字母表示各组间差异有统计学意义P< 0.05
以确定是否自然变异VRN-A1存在于eh1和LX987对抽穗时间的影响,我们使用先前开发的标记来区分显性Vrn-A1和隐性vrn-A1[16].结果表明,两者eh1LX987为隐性vrn-A1等位基因(图S5一个)。Ppd-A1是一个重要的抽穗时间调节因子,位于染色体2A上。确定…的序列变化Ppd-A1基因之间的eh1和LX987,我们对Ppd-A1基因及其上游和下游区域。序列比较显示,在编码区没有变化Ppd-A1基因之间的eh1, WT和LX987(图S .5C).在的3 '区域上有131 bp的缺失Ppd-A1是在eh1这种变异在中国春季也存在(图S5我们还比较了RIL群体中该位点携带不同变异的株系的抽穗时间,发现131 bp的缺失不影响抽穗时间(图S . C .)5D)。
之间的航向时间之差eh1, WT和LX987在春化或非春化条件下
研究突变的早穗基因是否与春化、早穗时间有关eh1WT和LX987分别从春化处理和未春化处理种子开始进行比较。在秋季播种(冬季春化),在3年的调查中,对种子进行了研究eh1比WT和LX987早10-14天和4-8天,差异有显著统计学意义[22].LX987种子在春季播种,未进行春化处理,不能开花eh1比WT早约13天(表1,无花果。5).之间的航向日期也有类似的变化eh1和WT,无论有无春化,都表明观察到的早期抽穗表型eh1不依赖于春化。
差异表达基因(DEGs)的RNA-seq鉴定
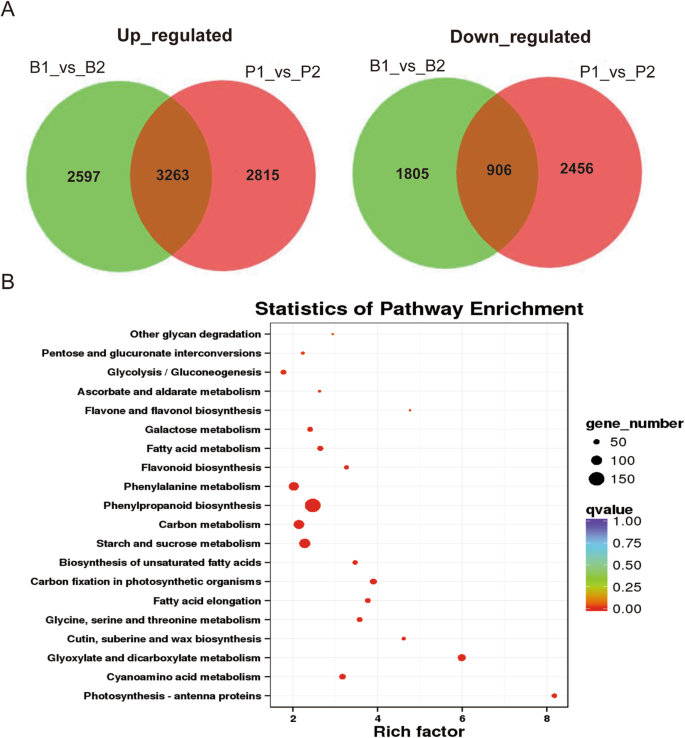
基因型Vrn-B1表明所有用于早期抽穗池的ril均含有一个显性等位基因(Vrn-B1), 22个用于后期抽穗池的RILs中有21个是隐性的(vrn-B1)(表4).RNA-Seq数据在一定程度上可以可靠地用于研究由于存在不同的转录组变化VRN-B1等位基因。根据我们的标准(fold change≥2,FDR < 0.05),两亲本间共鉴定出9440个deg,其中上调6078个,下调3362个(表S .)5).在早穗期和晚穗期基因库中,共鉴定出8571个deg,包括5860个上调基因和2711个下调基因(表S5).在所有的deg中,有4169个基因在两个亲本组和两个散装组的比较中重叠,其中上调基因3263个,下调基因906个(图2)。6a,表S5).其中上调基因103个,包括-淀粉酶1 (BMY1,3-oxoacyl-[酰基载体蛋白]合成酶I (KAS12,TraesCS2B01G156800)和花药特异性蛋白(RTS,TraesCS3D01G436700),具有折叠变化> 100和P< 0.0001被识别(表S5).此外,我们还发现了春化基因VRN-B3(TraesCS7B01G013100)上调(P< 0.01)eh1与LX987和LX987相比,早期抽穗散装样品的折射率分别为4.63和3.36。
RT-qPCR比较所选DEGs和春化基因的表达
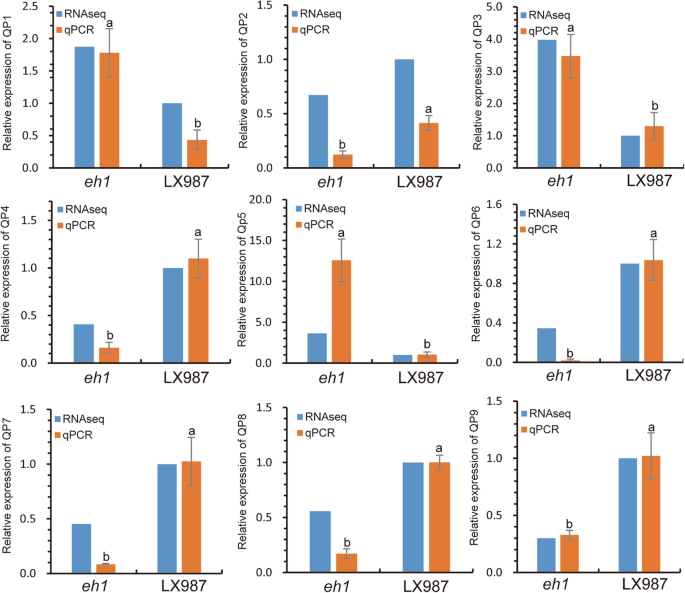
为了验证RNA-seq生成的基因表达水平的可靠性,使用了9个基因,包括Vrn-B3采用实时定量PCR方法进行验证。如图所示。7, RT-qPCR分析所选基因的相对表达量与RNA-seq数据中观察到的表达趋势相似,且Pearson相关系数较高(R2= 0.79)。
实时qPCR检测eh1和LX987。QP1 - QP9标记基因的详细信息见表S7.RNA-seq值(蓝色)表示表达折叠变化的比例eh1相对于LX987。qPCR生成的数据(橙色)为三个生物重复的均值±SD,不同字母表示基因表达水平之间存在显著差异eh1qPCR检测LX987P< 0.05
比较春化基因的表达水平eh1和LX987, RT-qPCR定量表达VRN-A1,VRN-B1,VRN-D1,VRN2基因。当eh1开始抽穗,幼嫩的穗子和叶子eh1分别从10个重复中选取LX987和LX987。表达水平VRN-A1,VRN-B1而且VRN-D1的年轻尖峰显著高于eh1与LX987相比(图S6).在叶片中,表达量为VRN-B1而且VRN-D1也明显高于eh1比LX987(图S6).相比之下,表达水平VRN2在幼穗和叶片中都有基因eh1显著低于LX987(图S6).分析这些基因的表达水平eh1LX987和LX987在同一发育阶段,幼穗和叶片eh1也比LX987早8天采样,表现出相似的发育水平。同一发育阶段的样品与同一天的样品相比,得到了完全相反的结果(图S7).
GO和KEGG通路富集分析
通过基因本体(GO)富集分析,研究两个亲本和两个bulk之间重叠的DEGs的功能。共富集了1537个GO类,经校正后显著富集的有42个P-value < 0.05(表S6).结果表明,光合系统I中的光合电子传递、还原戊糖-磷酸循环、碳水化合物代谢过程和脂肪酸生物合成过程等生物过程都受到了氧化石墨烯含量显著增加的前20个术语的影响VRN-B1变化(表S)6).重叠DEGs的KEGG通路富集表明光合作用相关通路显著富集,包括天线蛋白、脂肪酸延伸、光合生物固碳、淀粉和蔗糖代谢、碳代谢和脂肪酸代谢等参与了打穗过程(图2)。6b,表S7).
讨论
HD变异对小麦品种适应当地环境具有重要意义,其优化有助于小麦育种过程中的产量提高[33].诱导突变是种质资源创造的有效途径[34].γ射线诱导小麦早抽穗突变体,eh1,为HD提供了新的遗传变异。通过使用eh1以晚抽穗期品种LX987为亲本,构建一个RIL群体进行BSA分析和遗传作图分析。通过结合BSA和遗传作图方法,检测到一个稳定的HD QTL, LOD评分超过22.8,侧边标记位于20 Mb的间隔内,其中包括HDVRN-B1轨迹(3.].使用两个基因组特异性引物的进一步分析表明,在基因的第一个内含子中存在或不存在一个大的缺失VRN-B1与RIL群体的抽穗时间变化有关。与这些结果一致的是,之前的一项研究表明,基因的第一个内含子内的大量缺失VRN1导致春季生长习性,并影响开花时间[16].的第一个内含子中有一个2.8 kb的区域vrn-B1在不同的隐性等位基因中表现出高度的序列保守性[16,35].假设这一关键区域包含一个由春化下调的假定抑制因子的结合位点;的第一个内含子存在较大的缺失Vrn-B1消除抑制功能VRN-B1[16,22,35].由于基因型的不同VRN-B1在本研究中,在RIL群体的两个亲本之间,确定主要QTL是合理的(图2)。2,3.).通过使用这两对Vrn-B1基因标记,我们发现用于构建BSR-Seq早期和晚期抽穗体的株系几乎平分Vrn-B1而且vrn-B1,(表S .4),表明BSR-Seq的结果是可靠的。第一个内含子大缺失的相同基因型VRN-B1在WT和eh1(无花果。4a),结合了WT和之间相似的HD变化eh1有无春化作用(图。5、表1),表明早穗表型在eh1是不是由于突变中的大量缺失VRN-B1.此外,由于的第一个内含子的缺失VRN-B1,eh1而WT不经春化也能开花(表1,无花果。5).因为WT和eh1底火Intrl/B/F位置[16]导致扩增失败(图S3.A, B),本研究开发的新标记将是小麦标记辅助育种的替代解决方案。排序VRN-B1基因提示位于大缺失下游37 bp的缺失发生了突变eh1(无花果。3.A, C),这个37 bp的缺失与RIL群体中的大缺失共分离。37bp缺失区与先前报道的36bp缺失区相似Vrn-B1b,出现在春小麦品种中[18].据报道,含有Vrn-B1b等位基因表现出比携带等位基因的人更长的抽穗期Vrn-B1a等位基因(36].相比之下,通过表型分析F2个体由杂交而来eh1和WT,我们发现37bp的缺失不会导致HD的变化(图S4).自始仅有2项参赛携带Vrn-B1bCho等人使用等位基因进行比较。[36],可能是不同种质间影响抽穗时间的其他位点的背景差异导致了HD变异的检测。
由于用于BSR-Seq的株系有限,使用这种方法仅检测到位于5B染色体上的一个QTL(图2)。2).当我们使用不同的早标题和晚标题线构建三对bulk进行BSA分析时,检测到位于染色体5B、2A、2B、3A、3B和6A上的qtl(图S .)2a - c)。在5B染色体上3对bulk检测到的QTL也包括VRN-B1基因(图S2D),这与BSR-Seq和遗传作图的结果一致(图2)。2,3.).之前的研究也通过基因定位在染色体5B、2A、2B、3A和3B上发现了HD相关qtl [37,38,39和开花调节器光周期基因Ppd-A1而且Ppd-B1分别位于染色体2A和2B上[40].虽然在3 '区有131 bp的缺失Ppd-A1基因被检测到eh1与LX987相比(图S5C),在2A染色体上检测到的位点不存在Ppd-A1由于基因缺失和不缺失的RILs的抽穗日期没有差异(图S5D).此外,全基因组关联分析也在6A染色体上检测到HD的QTL [41,42].WT和LX987之间HD的遗传背景多样性可能导致RIL群体中检测到几个qtl。由于不同的RILs包含不同的HD基因,使用不同的RILs进行BSA分析是一种可行的、全面的QTL鉴定方法。然而,由于HD的自然变异存在于eh1和LX987,很难从这些qtl中区分突变位点。这一问题可以通过进一步的精细定位或利用突变体与小波杂交得到的群体来解决。
总之,几项研究已经阐明了不同变异的遗传和表型效应VRN1在过去的几十年里。然而,影响VRN1全局基因表达缺失尚未被广泛报道[42].使用BSR-Seq数据,在年轻刺突中的差异表达基因(DEGs)在两个池中被识别Vrn-B1或vrn-B1基因(表S)4).虽然在RNAseq分析中检测到的deg可能不仅仅是由于基因中的大量缺失VRN-B1,通过表达模式分析新发现的基因和通路可能部分受到基因的调控VRN1或受开花时间不同的影响。一个限制是RNA-seq的不可复制数据,这将通过分析亲本和大体之间的重叠DEGs部分抵消。据报道,Vrn1蛋白与蛋白的启动子结合FT1/Vrn3染色质免疫沉淀测序(ChIP-seq),以及转录水平FT1/Vrn3的高表达上调Vrn1在大麦中[43].VRN1也会下调开花抑制因子VRN2基因(24].一致地,更高的表达水平VRN-A1,VRN-B1而且VRN-D1在尖峰eh1与LX987相比,检测出较低的含量VRN2观察到eh1(无花果。6).同时,表达增加Vrn-B3是在eh1,或早期头部散装,有Vrn-B1基因型相对于vrn-B1组,表明Vrn-B1可能在调节中发挥作用Vrn-B3在小麦。在我们的分析中,发现了可能参与花发育的基因,包括constans样基因、MADS-box基因和MYB转录因子(表S5).这些基因也被邓等人鉴定为vrn1结合靶点,可能通过RNA-Seq和ChIP-seq调节开花。[43].此外,高度上调的基因> 100,包括β -淀粉酶(BMY1)和花药特异蛋白(即时战略游戏),亦可能由Vrn-B1并在抽穗和开花之间的关系中起关键作用。此外,功能富集分析也表明,在DEGs之间存在多条显著富集的通路,如碳代谢、淀粉和蔗糖代谢、糖酵解、糖异生、脂肪酸生物合成和代谢(表S .)7).邓等人确定了的直接目标VRN1包括参与茉莉酸、脱落酸和赤霉素生物合成或分解的基因[43].同样,除了赤霉素外,这些通路在我们的氧化石墨烯分析中都得到了丰富(表S6),表明这些途径在调节谷物开花方面是保守的。碳水化合物在植物开花转变的调控中起着重要作用。蔗糖是叶片和根尖分泌物中的主要糖类,在分生组织中的积累先于能量消耗过程的激活,如有丝分裂激活[44,45].的拟南芥晚开花突变体碳水化合物accumulation1(cam1)中淀粉含量高于WT,但晚开花表型并非由淀粉含量增加引起[46].有趣的是,这项研究表明,流动时间对直接或间接参与碳水化合物代谢的基因表达模式有影响。脂肪酸代谢对花粉发育有重要作用。花粉的细胞壁含有由花药绒毡层产生的脂肪物质[47].此外,脂肪酸酰胺水解酶的过表达引起了早花表型拟南芥也证实了脂肪酸在花诱导中起重要作用的观察结果[48].因此,我们观察到在花期变异的品系中,参与脂肪酸生物合成和代谢的DEGs含量较高。结果表明,脂肪酸代谢途径与小麦花发育途径相互作用,并可能受到基因变异的影响VRN-B1基因的表达。类黄酮生物合成途径已被充分证明,并且已知与植物开花时间的变化有关[49,50,51].类黄酮是一类强抗氧化化合物,其合成由苯丙烷途径启动,随后进入9个不同的代谢分支,包括产生黄酮和黄酮醇的代谢分支[52].有趣的是,我们的转录组分析表明,与苯丙类生物合成、苯丙氨酸代谢、类黄酮生物合成、黄酮生物合成和黄酮醇生物合成相关的通路在我们的数据集中显著富集(表S .)7),进一步详细分析苯丙烷生物合成的deg,发现98个上调基因和16个下调基因参与了该途径的大部分步骤(图S8),间接表明这些途径参与了小麦的早期抽穗过程或受开花时间的影响。
结论
在本研究中,利用BSA和遗传作图相结合的方法,在小麦RIL群体中确定了一个主要的抽穗期QTL。分子标记的发展Vrn-B1表明第一个内含子的大量缺失是RIL群体中朝向时间变化的部分原因。此外,我们的研究结果还揭示了与开花有关的几个重要基因和途径。分子标记VRN-B1本研究开发的标记辅助育种项目将用于优化抽穗时间,以及与之相关的候选DEGsVrn-B1基因调控有助于我们更好地理解小麦抽穗相关通路的分子功能。
方法
植物材料及表型分析
早期抽穗突变体(eh1)是用284 Gy γ射线诱变春小麦中源9号干种子得到的。的eh1将该突变体与晚熟的中国优良品种伦选987 (LX987)杂交,得到F6:7单籽下降重组自交系。中远9号小麦品系和eh1LX987的种子由刘炳华(中国农业科学院作物科学研究所)提供。RIL群体(400个系)和亲本系在中国农业科学院作物科学研究所(北京)中浦场站播种,在管理良好的田间条件下种植。每一行1米长,种植15株植物。在2016年至2018年的3年试验中,记录每个RIL的抽穗时间,一行中有2 / 3的植株抽穗,超过一半的穗已出现。
BSR-Seq分析
在早抽穗穗轴开始抽穗时,将极早抽穗穗轴和极晚抽穗穗轴及2个亲本在液氮中闪冻,- 80℃保存。总共使用了21条早标题线和22条晚标题线。用trnzel - a从这些细胞系中提取RNA+试剂(天根生物科技,中国)。RNA样本用DNase I (Takara)处理,并使用RNA纯化试剂盒(Tiangen Biotech)清洗。从每一株系中提取等量的RNA进行混合,以构建早穗轴和晚穗轴体。RNA质量使用NanoDrop™One (Thermo Scientific)和Bioanalyzer 2100系统(Agilent Technologies)进行评估。使用NEBNext®Ultra™RNA Library Prep Kit for Illumina®(NEB)成功构建了4个cDNA文库,包括两个亲本和两个bulk。RNA测序在Illumina HiSeq平台上根据制造商的协议进行,并生成150 bp的配对末端reads (Biomarker Technologies Corporation, Beijing)。从原始数据中去除低质量reads、带有适配器的reads或含有超过10%的未识别核苷酸的reads后,总共获得了54.71 Gb的干净reads。每个库的read number、Q30和GC content的详细信息见表S8.然后将干净的reads与国际小麦基因组测序联盟(IWGSC)发布的参考基因组中国春小麦v1.0 (http://www.wheatgenome.org/)使用预设参数的STAR软件[53].根据重复在参考基因组中的位置,使用Picard (https://sourceforge.net/projects/picard/).使用GATK软件进行局部重新校准、碱基重新校准和SNP呼叫[54].根据以下过滤标准获得高质量的snp: (i)如果存在多个等位基因,则删除位点;(ii)筛选reads支持度< 4的SNP位点;(iii)排除大量具有相同基因型的snp;(iv)来自较晚抽穗批次的与较晚抽穗亲本不同的snp被丢弃。本研究生成的测序数据集保存在美国国家生物技术信息中心(NCBI),编号为PRJNA517367,序列读取实现(SRA)接入号为SRP182626。
ED与SNP-index的相关性分析
欧几里得距离(ED)算法基于标记与批量测序数据的显著差异来评估目标区域与感兴趣性状之间的相关性[55].理论上,在两个极端表型体中,靠近目标区域的标记是不同的,而区域外的标记是一致的,其ED值接近于0。ED值越高,两个散装的差异越大。ED的公式如下:
X无足轻重的人, XWT分别为突变型和野生型体中X碱基的出现频率。在分析中,计算了大块中每个不同SNP的测序深度和ED值。为了消除背景噪声,将原始ED值的五次方作为相关值,然后使用SNPNUM算法对ED值进行拟合。snp指数定义为大量突变型等位基因频率与野生型等位基因频率的比值[56,57].当所有SNP读都与野生型等位基因不同时,该指数等于1,当所有SNP读都与野生型等位基因相同时,该指数等于0。为了排除假阳性,根据位于同一染色体上的snp在基因组中的位置进行了拟合。在关联阈值以上的区域被认为是与早抽穗表型相关的候选区间。根据参考基因组对应区域的预测基因数,计算候选区域的基因数。
基因组DNA提取
基因组DNA从苗期的新鲜叶片样本中提取,如前所述[58].简单地说,用MM301振动磨机(Restsch GmbH, Germany)将冷冻叶片研磨成均匀的粉末后,加入600 μL DNA提取缓冲液并完全混合。然后将样品在65°C下孵育1小时。加入200 μL 5 mol∙L−1离心后取上清液KAc 300 μL。DNA样品用异丙醇沉淀,用70%乙醇洗涤。使用NanoDrop ND-2000分光光度计(Thermo Scientific)测定DNA的质量和数量,最终浓度归一化为60 ng∙μL−1.
基于小麦660 K SNP阵列的堆积分离分析(BSA
根据2年表型数据,选择极早抽穗或极晚抽穗的RILs。通过从每个RILs中混合等量的DNA构建三个早抽穗或晚抽穗体。其中,早抽穗1号和早抽穗2号分别与18个早抽穗ril混合。早抽穗散装3与22个株系混用,3个晚抽穗散装分别与16个株系混用。使用Axiom®小麦660 K SNP阵列(Thermo)对6个散装小麦和2个亲本进行基因分型。采用DQC (Dish QC) > 0.82和CR (Call-Rate) > 94的阈值,获得高质量的基因分型数据。选择一对早抽穗和晚抽穗大穗中表现出不同基因型的snp,大穗和亲本之间表现出相同表型的基因型。最后,计算每条染色体上所选SNP的数量。选择SNP数目较多的染色体被认为是HD相关QTL。染色体上的SNP频率由10mb区域内SNP的数量除以染色体上SNP的总数决定。
竞争性等位基因特异PCR (KASP)标记的开发
基于RNA-seq数据,候选染色体上的SNP被选择,并使用在线引物设计管道PolyMarker (http://polymarker.tgac.ac.uk/).用两个亲本系检测KASP标记的特异性。PCR检测使用CFX 96实时系统(Bio Rad, Hercules, CA, USA)进行。5 μL反应,包括2.5 μL KASP主混合物(LGC Genomics, Middlesex, UK), 2.4 μL 60 ng∙μL−1DNA, 0.04 μL 50 mM Mg2 +0.06 μL引物混合物(引物A (100 μM):引物B (100 μM):引物R (100 μM): ddH2O = 12:12:30: 46),按以下步骤进行PCR: 95°C 15 min,然后触摸10个周期(95°C 20 s;最初在65°C触地,每周期下降0.5°C,持续30秒),然后再进行30个额外的退火周期(95°C, 10秒;57°C, 60秒)。
遗传连锁图谱的构建
利用QTL IciMapping version 4.1构建了候选染色体的连锁图谱[59,60].利用Kosambi map函数将重组频率转换为厘摩根(cM)距离[61].QTL作图采用包涵复合区间作图(ICIM)方法,采用3个年份的到穗天数进行。表型变异的解释值(通过采用ICIM法测定单个QTL。显著qtl的对数比值(LOD)阈值为3.0。
的识别VRN-B1
设计了两对引物来鉴定基因中是否存在较大的缺失VRN-B1.被称为TavBI的正向和反向引物对被设计为缺失区域的侧面,因此产物只能在显性区域被检测到Vrn-B1.对于被称为TavBII的引物对,正向引物被设计为退火到缺失区域,反向引物被设计为退火到缺失之外的基因组区域,因此只能在隐性基因中检测到产物vrn-B1.PCR反应用T5超级PCR mix (Tsingke, Beijing),程序如下:98°C 3 min,然后退火32个循环(98°C 10 s, 58°C 10 s;72°C 1分钟)。显性效应代表两个亲本杂合子与中点的抽穗时间之差,加性效应代表两个亲本差异的一半值,被认为是显性程度。对测序Vrn-B1,设计了11对引物对整个基因进行扩增并进行测序。测序的引物VRN-B1而且Ppd-A1和标记引物检测37 bp的缺失VRN-B1还有一个131 bp的缺失Ppd-A1表S9.
基因表达水平的定量和DEGs分析
基于BSR-Seq数据,使用Cuffinks软件中的Cuffquant和Cuffnorm模块计算映射到每个基因的干净读取数。利用每百万片段转录率(Per Kilobase of transcript Per Million fragments mapped, FPKM)计算来自两个亲本和两个bulk的预期基因表达水平。FPKM算法规范化转录本长度和映射读数,是估计基因表达水平的常用方法[62].差异表达基因(DEGs)分析使用EBSeq R包进行,该包提供了基于经验贝叶斯层次模型的统计方法。由此产生的P-value采用Benjamini-Hochberg方法进行调整,以控制错误发现率。如果调整,则认为基因在两组之间有差异表达PEBSeq检验-values < 0.05 (FDR < 0.05), Fold Change≥2.0。
GO项和KEGG富集分析
基因本体(GO;http://www.geneontology.org)使用GOseq R包进行DEGs的富集分析(修正P-value < 0.05),其中校正了基因长度偏倚。GO术语P-值小于0.05被认为显著富集。基于DEGs的KEGG通路检索(http://www.genome.jp/kegg/),利用KOBAS软件分析各KEGG通路中DEGs富集的统计学意义。KEGG制图仪用于分析最显著富集通路(https://www.kegg.jp/kegg/mapper.html).
实时荧光定量PCR分析
与RNAseq分析中使用的组织相同(eh1和LX987尖刺样品)进行实时荧光定量PCR验证。用于定量测定春化基因、幼穗和叶片的相对表达量eh1和LX987取样时eh1开始航向。为分析同一发育阶段的基因表达情况,以小刺和叶片为研究对象eh1也比LX987早8天采样,表现出相似的发育水平。RNA提取和qRT-PCR均按先前报道进行[63].通常,从穗或叶样本中分离总RNA使用RNeasy Plant Mini Kit (Qiagen)。使用DNase I (Takara)和RNA纯化试剂盒(Tiangen)去除提取RNA中的DNA污染。利用TransScript first - strand cDNA Synthesis SuperMix (TransGen)试剂盒合成了第一链cDNA。qRT-PCR使用SsoFast EvaGreen Supermix Kit (Bio-Rad,美国)和CFX 96 real - time系统(Bio-Rad,美国)进行。本试验采用2个技术重复和3个独立的生物重复。为了验证deg,使用Oligo 7软件设计了基因特异性引物。引物的详细情况见表S10.表达分析VRN1而且VRN2基因,先前研究中描述的引物[7,64]被使用。肌动蛋白作为一种内部控制来规范表达式数据。相对表达水平用2——ΔΔCT方法(65].
数据和材料的可用性
支持本研究结论的数据在论文及其附加文件中。所有测序数据集都保存在国家生物技术信息中心(NCBI),编号为PRJNA517367,序列读取实现(SRA)接入号为SRP182626。
缩写
- 瑞来斯:
-
重组自交系
- eh1:
-
早抽穗突变体1
- BSA:
-
散装隔离分析
- QTL:
-
数量性状位点
- BMY1:
-
Beta-amylase 1
- RTS:
-
Anther-specific蛋白质
- 度:
-
差异表达基因
- 英国《金融时报》:
-
开花位点t
- 卡尔:
-
菜花
- AP1:
-
APETALA1
- 富尔语:
-
FURITFULL
- RIP3:
-
RNA免疫沉淀片段3
- bZIP:
-
基本leucine-zipper
- NF-Y:
-
核factor-Y
- 高清:
-
标题日期
- BSR-Seq:
-
散装分离RNA-Seq
- 艾德:
-
欧氏距离
- 走:
-
基因本体论
- KEGG:
-
京都基因与基因组百科全书
参考文献
- 1.
荣格C,穆勒AE。花期控制及其在植物育种中的应用。植物科学进展,2009;14(10):563-73。
- 2.
春化及其与休眠的关系。植物物理学报,1960;11:191-238。
- 3.
闫玲,卢koianov A, Tranquilli G, Helguera M, Fahima T, Dubcovsky J.小麦春化基因的位置克隆VRN1.中国科学(d辑:自然科学),2003;29(3):344 - 344。
- 4.
严L, Loukoianov A, Blechl A, Tranquilli G, Ramakrishna W, SanMiguel P, Bennetzen J, Echenique V, Dubcovsky JVRN2基因是一种因春化而下调的开花抑制因子。科学。2004;303:1640-4。
- 5.
Yan L, Fu D, Li C, Blechl A, Tranquilli G, Bonafede M, Sanchez A, Valarik M, Yasuda S, Dubcovsky J.小麦和大麦春化基因VRN3是FT. Proc Natl Acad Sci U S A. 2006;103(51): 19581-6的同源。
- 6.
Kippes N, Debernardi JM, Vasquez-Gross HA, Akpinar BA, Budak H, Kato K, Chao S, Akhunov E, Dubcovsky j春化4基因揭示了南亚古代小麦春季生长习性的起源。中国生物医学工程学报(自然科学版),2015;29(3):344 - 344。
- 7.
杜bcovsky J, Loukoianov A, Fu D, Valarik M, Sanchez A,闫玲。光周期对小麦春化基因调控的影响VRN1而且VRN2.植物分子生物学杂志,2006;40(4):469-80。
- 8.
李志刚,李志刚,李志刚,等。水稻花期调控研究进展。植物学报。2009;12(2):178-84。
- 9.
陈超,闫玲,陈超,等。中国水稻品种等位基因变异的分子特征VRN-H2大麦春化位点。Mol Breed. 2005; 15:395-407。
- 10.
李勇,徐敏。谷类作物CCT家族基因研究进展。Crop J. 2017; 5:449-58。
- 11.
Ferrándiz C, Gu Q, Martienssen R, Yanofsky MF。分生组织特征和植物结构的冗余调控FRUITFULL,APETALA1而且菜花.发展。2000;127:725-34。
- 12.
Dhillon T, Pearce SP, Stockinger EJ, Distelfeld A, Li C, Knox AK, Vashegyi I, Vagujfalvi A, Galiba G, Dubcovsky J.温带谷物抗冻性和开花的调控VRN-1连接。中国生物医学工程学报。2010;29(4):344 - 344。
- 13.
李志强,李志强,李志强,等。草本植物APETALA1/FRUITFULL-like基因的研究进展。遗传学。2006;174(1):421 - 37。
- 14.
Golovnina KA, Kondratenko EY, Blinov AG, Goncharov NP。春化位点的分子特征VRN1野生和栽培小麦。植物学报。2010;10:168。
- 15.
阎L, Helguera M, Kato K, fuyama S, Sherman J, Dubcovsky JVRN-1多倍体小麦启动子区。应用理论,2004;109:1677-86。
- 16.
傅东,schzucs P, Yan L, Helguera M, Skinner JS, von Zitzewitz J, Hayes PM, Dubcovsky JVRN-1与大麦和小麦的春季生长习性有关。中国生物医学工程学报,2005;29(1):344 - 344。
- 17.
王晓明,王晓明,王晓明,王晓明,等。植物基因多态性在植物基因调控位点上的研究进展VRN-A1第一,内含子与冬小麦春化需求的差异有关。Mol Gen Genomics, 2018;
- 18.
Santra DK, Santra M, Allan RE, Campbell KG, Kidwell KK。春化基因的遗传和分子特征Vrn-A1,Vrn-B1,Vrn-D1来自美国西北太平洋地区的春小麦种质资源。植物营养学报。2009;29(6):344 - 344。
- 19.
褚春春,谭婷婷,于国涛,钟山,徐世生,闫磊。一种插入显性基因的新型逆转录转座子Vrn-B1等位基因赋予四倍体小麦春季生长习性(粗粒小麦。).G3(贝塞斯达)。2011; 1(7): 637 - 45。
- 20.
李志强,李志强,李志强,等。一种新的多重PCR检测方法Vrn-B1面包小麦中的等位基因(小麦。).动物营养学报。2012;30(1):317-23。
- 21.
黄志强,黄志强,黄志强。一种新植物的鉴定Vrn-B1两个抽穗期不同的近等基因小麦品系的等位基因分析。Mol Breed. 2012;29(3): 675-85。
- 22.
张波,王旭,王旭,马玲,王智,张旭。一个春化等位基因的分子特征Vrn-B1d及其对中国小麦抽穗期的影响(小麦。)红春麦。生物育种,2018;38(10):127。
- 23.
Hemming MN, Peacock WJ, Dennis ES, Trevaskis B.低温和白昼长度线索集成调节开花位点t在大麦。植物科学进展。2008;37(1):359 - 366。
- 24.
陈a, Dubcovsky J.小麦TILLING突变体显示春化基因VRN1下调开花抑制因子VRN2在叶片中,但不是开花所必需的。公共科学学报,2012;8(12):e1003134。
- 25.
李春霞,李春霞,李春霞,杨丽娟,等VRN2四倍体小麦中的基因座。植物科学进展。2009;29(1):344 - 344。
- 26.
李春华,李春华,王晓明,等。小麦FT蛋白调控VRN1通过与FDL2相互作用进行转录。植物学报,2008;36(4):344 - 344。
- 27.
Abe M, Kobayashi Y, Yamamoto S, Daimon Y, Yamaguchi A, Ikeda Y, Ichinoki H, Notaguchi M, Goto K, Araki T. FD,一种bZIP蛋白介导茎尖花途径集成器FT的信号。科学。2005;309:1052-6。
- 28.
李春,李春梅,李春梅,李春梅,李春梅。小麦开花抑制因子VRN2和启动子CO2与核因子- y配合物的相互作用。植物学报,2011;36(5):344 - 344。
- 29.
Milec Z, Valarik M, Bartos J, Safar J.大器晚成能成为早起鸟吗?开花时间调整工具。生物技术,2014;32(1):200-14。
- 30.
小麦抽穗期的遗传调控。王晓明。2018;54(4):375-88。
- 31.
刘珊珊,叶婷婷,唐慧敏,Nettleton D, Schnable PS.基于BSR-Seq的基因定位。公共科学学报,2012;7(5):e36406。
- 32.
Ramirez-Gonzalez RH, Uauy C, Caccamo M. PolyMarker:快速多倍体引物设计管道。生物信息学,2015,31(12):2038 - 9。
- 33.
兰格,朗吉纳,吴晓明,等。欧洲冬小麦花期控制研究。植物科学进展(英文版);
- 34.
许琪。植物化学和物理诱导突变体的分子性质研究进展。植物植物资源特性。2014;12:S74-8。
- 35.
冯泽维茨J, schzucs P, Dubcovsky J, Yan L, Francia E, Pecchioni N, Casas A, Chen TH, Hayes PM, Skinner JS。大麦春化基因的分子结构特征。植物分子生物学杂志,2005;59(3):449-67。
- 36.
赵娥,姜超,郑杰,尹勇,朴c。韩国小麦Rht-1、Vrn-1和Ppd-1等位基因变异及其对农艺性状的影响。植物育种技术,2015;3(2):129-38。
- 37.
罗伟,马军,周晓辉,孙敏,孔晓春,魏亚明,蒋永峰,齐普芬,蒋广涛,刘玉霞,等。控制农艺性状的数量性状位点的鉴定显示了西藏半野生小麦的育种潜力(小麦。tibetanum).作物科学,2016;56(5):2410-20。
- 38.
李峰,文伟,何忠,刘健,金辉,曹松,耿辉,闫娟,张鹏,万勇,等。利用高密度SNP标记对三个中国面包小麦群体产量相关性状的全基因组连锁定位。理论应用,2018;131:1903-24。
- 39.
Enid PL, Semagn K, Chen H, Iqbal M, N’diaye A, Kamran A, Navabi A, Pozniak C, Spaner D.利用单核苷酸多态性标记定位cutler x AC Barrie春小麦群体农艺性状相关qtl。公共科学学报,2016;11(8):e0160623。
- 40.
斯内普JW,巴特沃斯K,怀特丘奇E,沃兰德AJ。等待好时光:小麦开花时间的遗传。Euphytica。2001;119:185 - 90。
- 41.
Le Gouis J, Bordes J, Ravel C, Heumez E, Faure S, Praud S, Galic N, Remoue C, Balfourier F, Allard V,等。全基因组关联分析,以确定决定小麦早熟性成分的染色体区域。应用理论与实践。2012;26(3):597-611。
- 42.
Diallo AO, Agharbaoui Z, Badawi MA, Ali-Benali MA, Moheb A, Houde M, Sarhan f最有价值球员突变体揭示了全局基因表达的重要变化和茉莉酸甲酯在小麦春化和开花中的作用。中国机械工程学报,2014;26(3):344 - 344。
- 43.
邓w, Casao MC,王萍,Sato K, Hayes PM, Finnegan EJ, Trevaskis B.谷类作物春化响应与其他关键性状的直接联系。Nat Commun. 2015;6:5882。
- 44.
乔治B,安德烈H,克劳德H,安妮P,皮埃尔L.诱导开花的生理信号。植物科学。1993;5:1147-55。
- 45.
李志刚,李志刚,李志刚。碳水化合物在拟南芥开花诱导中的作用:野生型和无淀粉突变体的比较。足底。1998;206:131-7。
- 46.
王明敏,陈娟。拟南芥花60岁晚期花起始和淀粉过量积累的单基因隐性突变。《植物科学》1995;7:1703-12。
- 47.
李文杰,李文杰,李文杰。开花植物花粉壁发育的研究进展。中国农业科学,2007;26(3):344 - 344。
- 48.
Teaster ND, Keereetaweep J, Kilaru A, Wang YS, Tang Y, Tran CN, Ayre BG, Chapman KD, Blancaflor EB。过量表达脂肪酸酰胺水解酶可诱导花期提前拟南芥.中国植物科学,2012;3:32。
- 49.
库马尔V, Nadda G,库马尔S, Yadav SK.过表达茶叶二氢黄酮醇4-还原酶和花青素还原酶cDNA的转基因烟草诱导提前开花并提供生物耐受性。公共科学学报。2013;8(6):e65535。
- 50.
姚启勇,黄华,童勇,夏恩华,高丽珍。转录组分析鉴定了与三酰甘油和色素生物合成以及观赏和产油植物光周期开花相关的候选基因,山茶花试(山茶花)。前沿植物科学,2016;7:163。
- 51.
岳娟,朱超,周勇,牛霞,苗敏,唐霞,陈峰,赵伟,刘艳。楚菊花发育过程中黄酮类化合物合成相关基因的转录组分析。科学通报2018;8(1):13414。
- 52.
类黄酮生物合成。遗传学、生物化学、细胞生物学和生物技术的彩色模型。植物科学学报,2001;26(1):1 - 4。
- 53.
Dobin A, Davis CA, Schlesinger F, Drenkow J, Zaleski C, Jha S, Batut P, Chaisson M, Gingeras TR. STAR:超快速通用RNA-seq校准器。生物信息学。2013;29(1):15至21。
- 54.
McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M,等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组学报2010;20(9):1297-303。
- 55.
Hill JT, Demarest BL, Bisgrove BW, Gorsi B, Su YC, Yost HJ。MMAPPR:聚合RNA-seq的突变映射分析管道。中国生物工程学报,2013;23(4):687-97。
- 56.
阿部A,小杉S,吉田K,夏目漱石S,高木H, Kanzaki H,松村H,吉田K,光冈C,田鲁M,等。利用MutMap测序水稻的重要农艺位点。中国生物工程学报。2012;30(2):174-8。
- 57.
高木H,安部A,吉田K,小杉S,夏目石S,光冈C,植村A,宇史H,田鲁M,高野S,等。QTL-seq:通过对两个大群体的DNA进行全基因组重测序,快速定位水稻数量性状位点。植物学报,2013;26(1):344 - 344。
- 58.
李伟,郭红,王艳,谢艳,赵玲,顾娟,赵松,赵波,王刚,刘玲。小麦籽粒硬度和淀粉合成关键基因ems诱变新等位基因的TILLING鉴定。基因工程学报,2017;39(4):387-95。
- 59.
李华,叶刚,王娟。一种改进的复合区间映射算法。遗传学。2007;175(1):361 - 74。
- 60.
李华,张玲,王娟。QTL IciMapping:双亲本群体遗传连锁图谱构建与定量性状位点定位的集成软件。作物学报,2015;3(3):269-83。
- 61.
Kosambi DD.从重组值估计映射距离。优生学。1943;12(1):175 - 5。
- 62.
Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ, Salzberg SL, Wold BJ, Pachter L. RNA-Seq转录本组装和定量揭示了细胞分化过程中未注释的转录本和异型转换。生物技术学报。2010;28(5):511-5。
- 63.
熊红,郭红,谢艳,赵玲,顾娟,赵松,李娟,刘玲。小麦航天诱导突变体耐盐性的RNAseq分析揭示了耐盐相关通路和候选基因。科学通报2017;7(1):2731。
- 64.
卢科亚诺夫A,严L, Blechl A,桑切斯A, Dubcovsky jVRN-1正常和转基因多倍体小麦春化基因的研究。植物科学进展。2005;38(4):344 - 344。
- 65.
Livak KJ, Schmittgen TD。相关基因表达数据的分析采用实时定量PCR和2——ΔΔCT方法。方法。2001;25(4):402 - 8。
致谢
不适用。
资金
国家重点研究与发展计划(No. 2016YFD0102100, No. 2016YFD0101802)、国家自然科学基金(No. 31801346)和中国农业科学研究系统(No. 2016YFD0101802)资助。CARS-03)。这些资助机构在研究设计、数据收集、分析和解释以及撰写手稿方面没有任何作用。
作者信息
从属关系
贡献
LL指导了研究并设计了研究。YL和HX完成了大部分的实验和数据分析。HG分析了eh1.CZ和YX进行DEGs分析。LZ和JG协助进行富集分析。SZ和YD参与数据分析。YL撰写论文,LL修改稿件。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
附加文件1:图S1。
2016年、2017年和2018年RIL种群抽穗期分布。
附加文件2:图S2。
通过BSA分析检测RIL人群HD的qtl。(A-C)与HD相关的SNP在抽穗早期和晚期的1号簇(A)、2号簇(B)和3号簇(C)之间的每条染色体上的数量。QTL存在于具有较高数量选择SNP的染色体上。(D) HD相关SNP的频率分布在5B染色体上。蓝色、红色和绿色线分别代表散装1、散装2和散装3。
附加文件3:图S3。
序列比较VRN-B1在LX987, WT和eh1.(A)的示意图表示VRN-B1在LX987, WT和eh1.(B) LX987、WT、和引物Intrl/B/F的序列分析eh1.(C)聚合酶链反应扩增37 bp的缺失eh1通过使用特定的引物。全长凝胶见补充图10.
附加文件4:图S4。
离F方向还有几天2有或没有37bp缺失的个体VRN-B1.WT表示没有37bp的缺失eh1表示删除37bp。
附加文件6:图S6。
相对表达量VRN-A1,VRN-B1,VRN-D1,VRN2小刺和叶子中的基因eh1和LX987在同一天采样。使用学生t检验来评估显著性。*P< 0.05。
附加文件7:图S7。
相对表达量VRN-A1,VRN-B1,VRN-D1,VRN2小刺和叶子中的基因eh1和LX987在同一发育阶段取样。使用学生t检验来评估显著性。**P< 0.01和*P< 0.05。
附加文件8:图S8。
苯丙类生物合成中的DEGs。蓝色填充的红框表示抽穗前期体和亲本中上调的基因eh1,用蓝色填充的黑盒子表示下调基因,没有颜色的盒子表示没有表达差异的基因。
附加文件9:图S9。
图的原始凝胶图像。4一个。
附加文件10:图S10。
图S原始凝胶图像3.C。
附加文件11:图S11。
图S原始凝胶图像5一个。
附加文件12:图S12。
图S原始凝胶图像5B。
附加文件13:表S1。
RILs、WT和亲本的HD变异。
附加文件14:表S2。
2016年、2017年、2018年RIL线开航日期
附加文件15:表S3。
引物用于测绘和变异鉴定。
附加文件16:表S4。
用于RNA池化的RILs的基因型和离穗天数。
附加文件17:表S5。
亲本和穗池差异表达基因。
附加文件18:表S6。
富集的GO表示重叠的deg。
附加文件19:表S7。
重叠的DEGs富集KEGG通路。
附加文件20:表S8。
本研究中产生的测序数据摘要。
附加文件21:表S9。
测序引物VRN-B1和标记引物检测37 bp的缺失VRN-B1.
附加文件22:表S10。
RT-qPCR所用引物。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条所提供的资料,除非在资料的信用额度中另有说明。
关于本文
引用本文
李勇,熊宏,郭宏,郭宏。et al。春化基因的鉴定VRN-B1利用小麦RIL群体进行抽穗期QTL定位。BMC植物生物学20.331(2020)。https://doi.org/10.1186/s12870-020-02539-5
收到了:
接受:
发表:
关键字
- 标题的时间
- 小麦
- BSR-Seq
- Vrn-B1
- 基因映射
- 代谢途径