摘要
背景
日本龙胆花的蓝色色素是由于聚酰基化花青素,龙胆蝶呤,和所有相关的生物合成基因和几个调控基因已经克隆和特征。然而,花青素在花瓣液泡中积累的最后步骤尚不清楚。我们克隆并分析了谷胱甘肽S.-转移酶(GSTs)在日本龙胆草已知参与花青素在其他植物的运输。
结果
我们克隆了GST1.,它在龙胆花瓣中表达。另外,该基因属于与花青素生物合成相关的PHI型GST型。我们使用CRISPR / CAS9介导的基因组编辑系统来产生功能损失GST1.等位基因。Sanger和下一代测序分析证实了编辑的等位基因。这GST1.基因组编辑的株系表现出两种突变花表型,严重(几乎是白色)和温和(淡蓝色)。表型与花瓣中花青素积累减少有关。在GST1.综合的线,糖诱导的应力条件抑制了茎和叶中花青素的积累,建议GST1.是与胁迫相关的花青素在非花器官中积累所必需的。这些观察清楚地证明了这一点GST1.是日本龙胆草中负责花青素转运的基因,是龙胆草中花青素积累的必需基因。
结论
在这项研究中,与花青素相关的销售税对日本龙胆草基因进行了功能分析。不同于其他生物合成基因,其功能销售税在体外研究中难以检查基因。因此,本文所述的基因组编辑策略对于在龙族植物中的传输相关基因的作用体内研究。
背景
耕种的日本绅士的蓝色花是由于聚酰化的花青素的积累,永埃塞氏蛋白[玻璃蛋白3-O.-β-D-glucosyl-5 -O.——(6-O-caffeoylβ-D-glucoside) 3 ' -O.- (6 -O.-Caffeoyl-β-D-葡糖苷)],这是一个主要颜料[1].此外,分子内共色素作用负责这种蓝色着色和色素稳定[2那3.].为了澄清介导蓝龙胆花的发育的分子机制,涉及相关代谢途径的基因已被分离并表征。这些包括编码Chalcone合酶的生物合成基因(CHS.),chalcone异构酶(ch),二氢烷醇4-还原酶(DFR.),黄烷酮3-羟基化酶(F3h.)、类黄酮3 ' -羟化酶(F3'H.)、类黄酮3 '、5 ' -羟化酶(F3'5'H.)、黄酮合酶II (FNSII),以及花青素合酶(答),以及修饰基因,如编码花青素5-葡萄糖基转移酶的基因(5GT.), 3 '葡糖基转移酶(3 'gt)和5,3 ' -芳香酰基转移酶(5/3'AT)和编码MYB和BHLH转录因子的调节基因。在涉及天然突变体珍珠品种/线的先前调查中分析了花瓣色素沉着的转录调节[4.],通过稳定变换过表达嵌合抑制剂(例如,SRDX-GTMYB3)[5.和病毒载体[6.].通过这些方法确定的珍贵蜥蜴的转录规定已经详细审查了[7.].这些信息还被用于开发基于pcr的分子标记,用于区分开出白色或粉色花朵的植物和开出蓝色花朵的植物[8.那9.].基于这些分子标记的后代早期选择可能有助于促进日本龙胆育种[10].然而,据我们所知,与花青素运输有关的龙胆基因尚未表征。
在高等植物中,通过与转运蛋白(如ATP结合盒(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(Ab)和谷胱甘肽(ABC)和谷胱甘肽(ABC)和谷胱甘肽(ABC)转移到真空溶解中的花青素。S.- 转移酶(GST)。开拓研究揭示了玉米Bronze2(BZ2.)和佩妮花青素9.(An9)编码GST的原因,负责花青素的真空积累[11].直向同源销售税与花青素积累相关的基因已经在几种观赏开花植物中被鉴定出来,如DCGSTF2在康乃馨[12] 和CkmGST3在仙客来[13,但其他物种相对较少。虽然在许多植物物种中已经检测到负责花青素转运的假定GST和ABC蛋白的编码基因,并在公共数据库中可用,但只有少数的基因得到了功能特征。其中一个原因是花青素在体内的转运分析比较困难。除非有突变系,否则很难确认基因功能,花青素色素到液泡的运输主要是在人工膜泡的体外摄取试验中进行的。一些研究涉及拟南芥的遗传互补TT19突变系显示GSTs的潜在功能[13那14那15那16]但仍然目前还不清楚GST在原植物物种中是否实际上是功能性的。病毒诱导的基因沉默也可用于分析基因功能,但通常难以将该系统应用于园艺作物和一系列高度同源基因。
聚类定期间隙的短语重复(CRISPR)/ CAS9系统是高等工厂中最有前途的基因组编辑工具[17那18]而且预计新颖的Crispro技术将为植物科学研究作出重要贡献[19].CRISPR/Cas9系统已被用于研究几种观赏花卉的特定基因,如DFR.在日本牵牛花(Ipomoea nil.)[20.] 和F3h.在托雷尼娅[21].此外,我们还首次建立了以phytoene脱氢酶为靶点的CRISPR/Cas9系统(PDS)检查绅士[10].此外,我们完成了对花青素改性基因的体内功能分析,包括5GT.那3'GT.,5/3'AT[22].因此,CRISPR / CAS9系统现在可以用于加速绅士基因的功能分析,这是非模型植物。
本研究的目的是鉴定一种与花青素相关的植物销售税并通过CRISPR/Cas9系统进行基因组编辑,分析其功能。一个龙胆销售税基因(GST1.)被克隆并证实是花青素积累的主要来源。我们证明了该基因影响龙胆草花和叶中花青素的积累。据我们所知,这是首个利用CRISPR/Cas9系统研究高等植物花青素转运相关基因的报道。此外,我们的研究结果证实,本研究应用的基因组编辑策略有助于鉴定未特征的龙胆草基因。
结果
a的分离与鉴定销售税与花青素色素沉着有关的基因
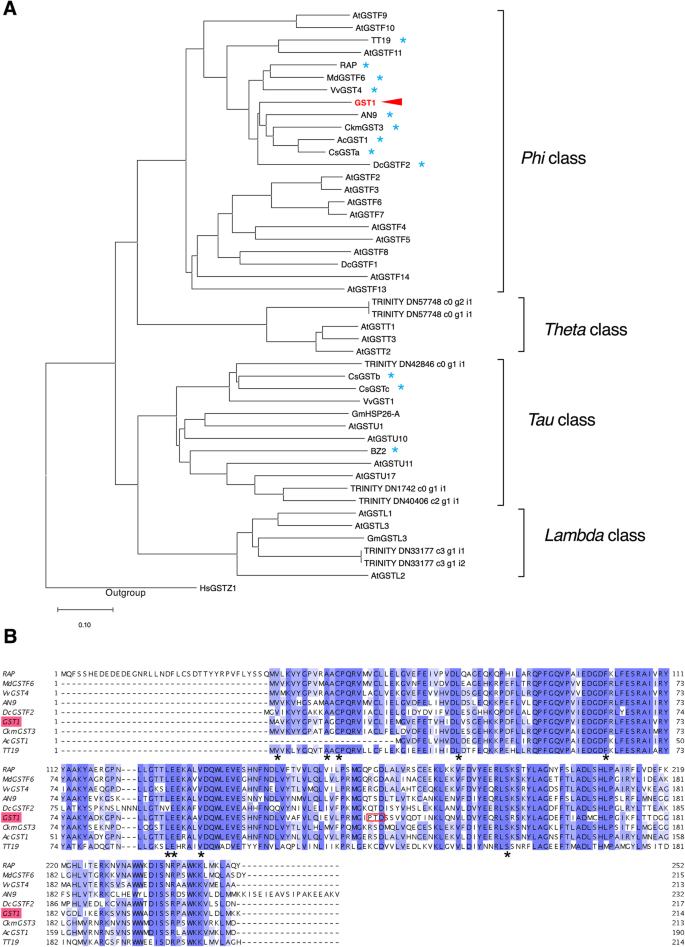
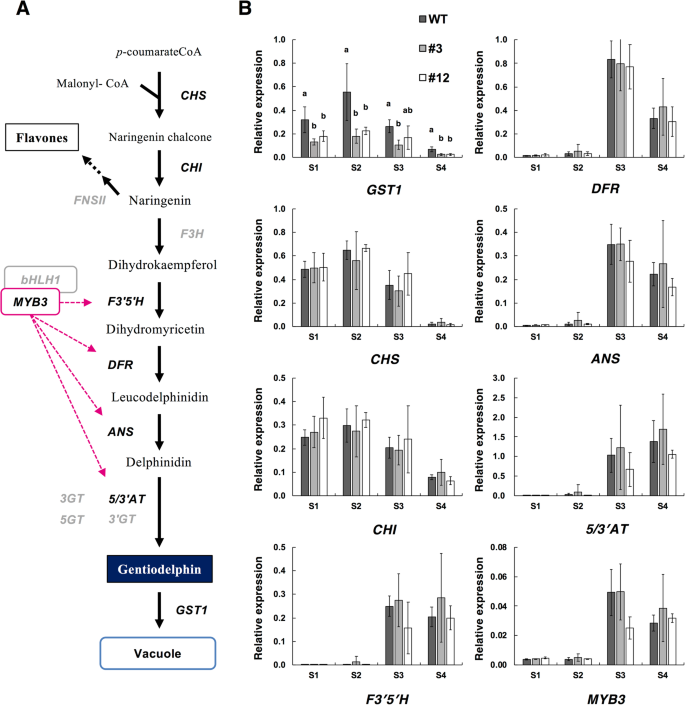
推导的两位等位基因的全长序列销售税对龙胆草品种‘Albireo’花瓣的cDNA样品进行PCR扩增,并对扩增产物进行测序。所推导出的编码区域序列销售税全长642 bp,两个等位基因之间存在3个单核苷酸多态性(SNPs),均编码214个氨基酸残基,114位有1个氨基酸差异(GST1-1,苯丙氨酸;GST1-2亮氨酸)。我们给这些等位基因命名GST1-1(登录号LC536038)和GST1-2(登录号LC536039),此后将它们作为GST1..原始测序数据可在DDBJ序列读取归档(DRA)数据库中获得(登录号为。DRA010021)。分析GST1-1和GST1-2基因组序列(登录号LC536040和LC536041)揭示了这两种等位基因分别包括1623和1667个碱基分别从开始到阻止密码子。系统发育分析表明GST1属于PHI型GST组(图。1a).推断的与花青素生物合成相关的GST1和phi型GSTs序列同源性为53 ~ 66%:RAP(66%)、VvGST4(66%)、AN9(65%)、DcGSTF2(62%)、MdGSTF6(66%)、CkmGST3(68%)和TT19(53%)。此外,在GST1中检测到与花青素相关的gst特异的保守氨基酸残基(图。1b)。
GSTs的系统发育树和比对。一种根据具有Clustalw(Thompson等,1994)和Mega计划的邻近接合方法(1000靴子复制),基于氨基酸序列构建包含GSTS的系统发育树。蓝色星号表示与青少年素相关的GST。B.花青素相关gst与CLUSTALW和Jalview(版本2.10.3)的比对。星号表示与花青素相关的gst所特有的保守氨基酸残基的位置。红框表示的是由于等位基因1的缺失而导致的三个氨基酸残基的缺失GST1.基因组编辑的线#3。用于构建系统发育树和序列对齐的GST的登录号在附加文件中列出了1:表S4
的生产GST1.genome-edited龙胆行
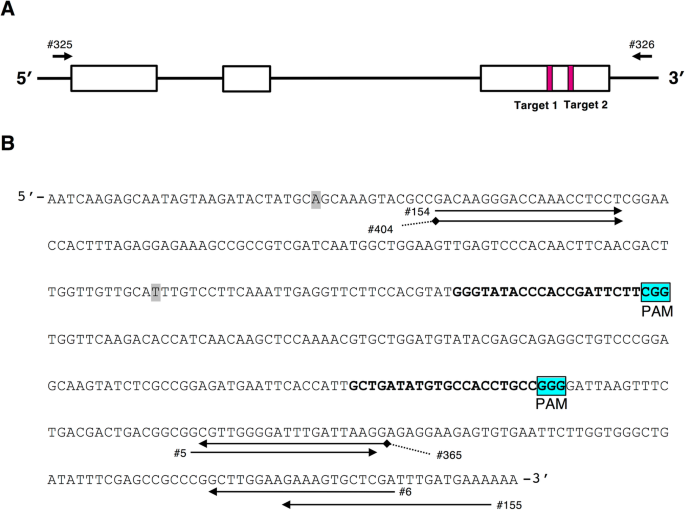
这GST1.基因组序列包括三个外显子和两个内含子。我们瞄准了第三外显子的两个地点GST1.(图。2a)用于CRISPR/Cas9系统的基因组编辑。通过应用根癌土壤杆菌- 介导的转化,我们获得了54个Bialaphos抗性转基因植物,并确认它们被局部基因组PCR扩增正确转化Cas9顺序。通过分析包括靶位位点的PCR扩增子的序列来预先筛选这些线。因此,选择了八条线,并在亚克隆后进行桑珀序列分析。结果表明,七条线是基因组编辑线,其中目标GST1.网站被突变(附加文件1:表S1)。所有的七个GST1.基因组编辑的线条适应并在封闭的温室中生长,直到开花。
原理图的GtGST1.一种电路图GST1.基因组结构。白盒子表示外显子。箭头表示引物位置。有关引物的详细信息列于附加文件1:表S3。红色阴影框表示CRISPR/Cas9系统基因组编辑的目标位点1和2。B.含有目标位点1和2的第三外显子序列用于基因组编辑。阴影单核苷酸是两种等位基因之间的SNP。粗体核苷酸代表靶位部位1和2的序列,并且相邻的蓝色阴影盒(PAM)表示相邻的基序(NGG)的原始旋流器。箭头表示底漆位置
下一代测序(NGS)分析GST1.基因组编辑龙胆植物
在第一次适应时,植物在大约4个月内绽放。在七中GST1.对5个株系(#3、#12、#13、#23和#29)进行基因组编辑,观察到花色表型的变化。第1行产生的花在颜色上与野生型(WT)花相似,第8行没有开花。因此,我们利用NGS平台分析了这5个品系的花瓣转录本和基因组DNA中靶位点的扩增序列。扩增片段序列基于高质量过滤的对端读取序列之间的重复序列区域进行合并。我们从WT和基因组DNA的转录本和文库中获得了70.5-99.9%的阅读对构建的片段GST1.genome-edited线。去除被认为是PCR和/或测序错误产生的非特异性或低拷贝片段后,对每个等位基因的主要片段进行计数。各文库中主要片段所占比例为86.9 ~ 90.2%。这两个等位基因是基于序列扩增区的SNPs进行区分的(图。2b).基因组DNA中靶位点1、2和转录本的测序结果GST1.表中总结了基因组编辑的第3和第12行1,其他行数据显示在附加文件中1:表S2和附加文件3..等位基因之间的DNA片段的数量几乎相等和/或小于2倍。关于转录分析,还有更多的等位基因1片段,而不是所有植物中的等位基因2片段。在线#3和#29中,检测到对应于等位基因1的DNA和转录片段。突变几乎与其他文件中呈现的Sanger测序分析结果相对应1:表S1。扩增子测序分析GST1.基因组编辑的线揭示了若干特征突变,例如靶位位点1和/或2的2,7,9和19个碱基的缺失,或者在靶位点1和2之间的碱基(表1额外的文件1:表S2)。确认线#12,#13和#23具有杂合的双曲髓突变。在#3中,删除靶位点1中的九个核苷酸,可能导致编码蛋白中的三种氨基酸缺失。我们在所有情况下检测到基因组DNA和转录物之间的一致突变模式GST1.genome-edited线。
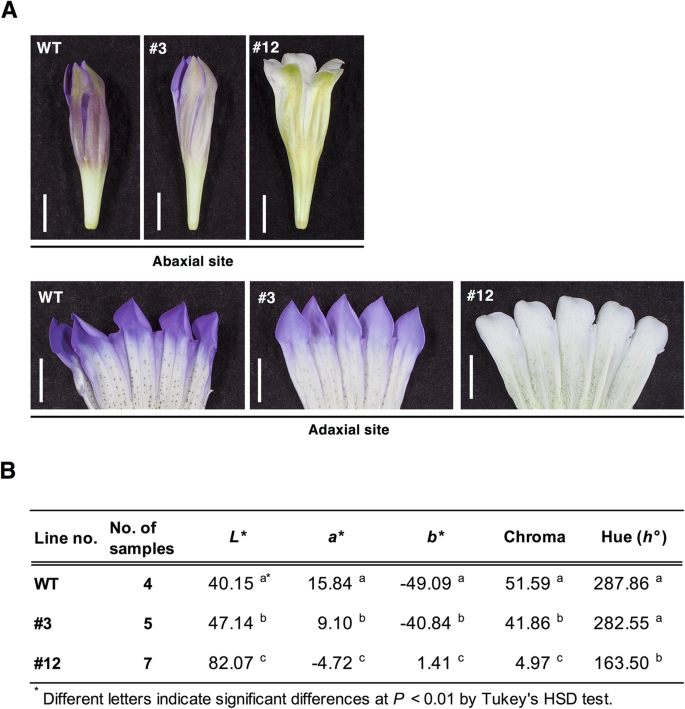
基因组编辑龙胆植物的花瓣表型
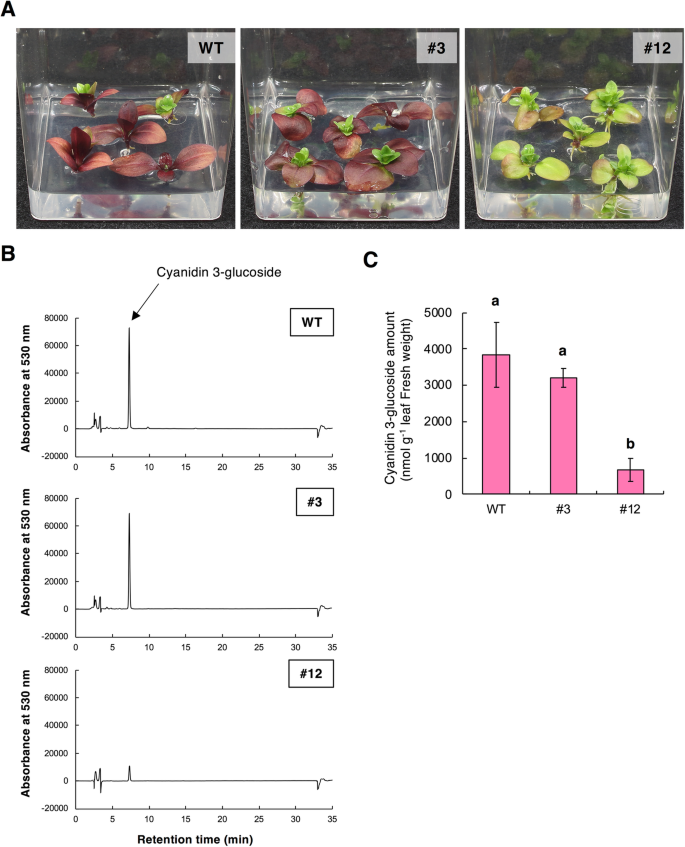
典型的花GST1.基因组编辑的线在图1中示出。3.a和附加文件2:图S1A。第12行、第13行、第23行和第29行的花几乎全是白色的。相比之下,第3行花与WT花相似,但它们更苍白。花翼正面的比色值为GST1.基因组编辑的线条#3和#12总结在图1中。3.湾用分光光度计分析花瓣的曲面表面的比色性状,并基于CIE评估l * a * b *.为了描述颜色色调,根据记录的数据计算色相角和色度(一种*和B *).这L.*,一种*,B.*、色度和色相角值显著高于或低于野生型植物。这L.*,一种*,B.* 3线的色度值也与WT植物的色度显着不同,但色调角度没有显着差异。这些结果证实了WT控制之间的可见差异GST1.genome-edited线。其他行对应的值汇总在附加文件中2:图印地。第13、23、29行比色值与第12行相似,但与第3行不同。
龙胆草基因组编辑植物花青素组成分析
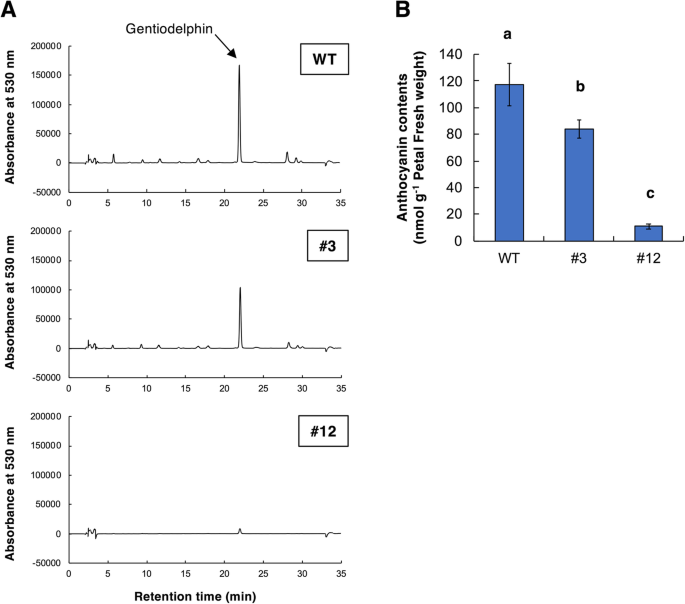
来研究玫瑰花瓣中积累的色素是否GST1.基因组编辑株系发生变化后,花青素提取液在530 nm的监测波长下进行高效液相色谱分析。的色谱图GST1.基因组编辑的品系3和12和WT对照如图所示。4.一种。WT植物的色谱图包括代表通埃塞氏蛋白的单峰,而色谱图GST1.基因组编辑的第12和第3行分别有极小的峰值(约6.1%的WT)和中等大小的峰值(约63.4%的WT)。此外,在WT色谱图中没有检测到的#3和#12线没有明显的峰。用分光光度法测定了基因编辑后的龙胆草和野生龙胆草的花瓣中总花青素的含量(图。4.b)。与HPLC分析一致,含有的半粘蛋白含量较低GST1.基因组映射线小于WT对照,分别具有线#3和#12的三孔蛋白水平分别为WT植物的约71和<10%。
类黄酮生物合成相关基因表达分析
定量逆转录PCR (qRT-PCR)检测表达GST1.以及花瓣发育过程中类黄酮生物合成相关基因。我们选择了两个生物合成基因(CHS.和ch)为早期基因及四个生物合成基因(F3'5'H.那DFR.那答,5/3'AT)作为晚期基因。这MYB3调控晚期生物合成基因表达的转录因子基因也被包括在表达分析中。四个发育阶段(S1-S4)的结果数据(图4)。5.的表达模式GST1.在WT植物中类似于早期基因的植物,CHS.和ch.此外,GST1.表达水平在显着降低GST1.基因编辑的品系#3和#12(约为WT的32.2-63.5%)比对照的WT增加。早期基因的表达水平和模式无显著差异(CHS.和ch)、晚期基因(F3'5'H.那DFR.那答,5/3'AT), 和MYB3在。。之间GST1.基因组编辑的品系和WT植物。
编辑的互补GST1.通过粒子轰炸
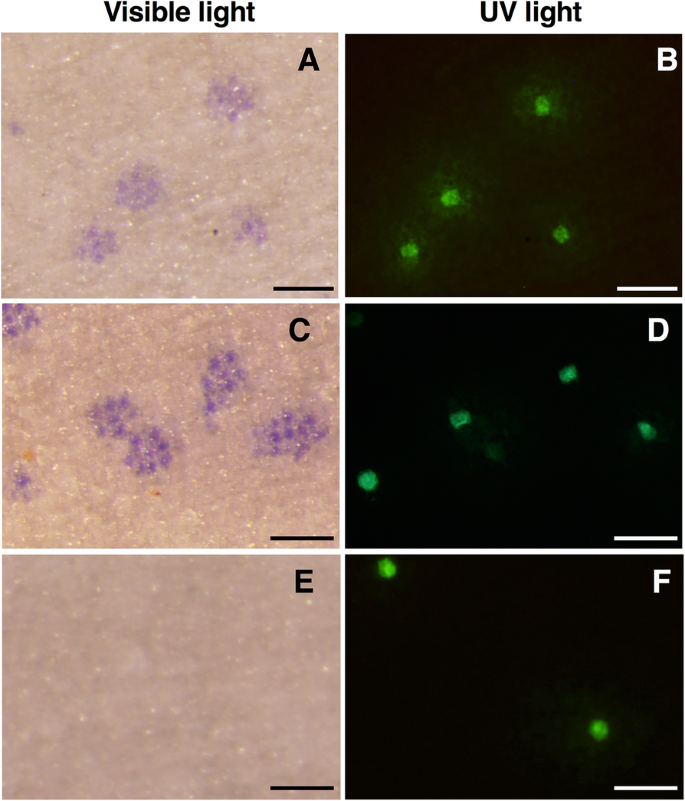
为了确认白色着色是由于GST1因基因组编辑引起的GST1缺乏,我们进行了涉及瞬态的互补测定GST1.通过粒子轰击表达。花瓣的正面GST1.基因编辑的第12系在开花前用p35S-GST1-1(或p35S-GST1-2)和p35S-sGFP重组质粒轰击。在轰击后24小时左右,在光镜下,一些表皮样品在白色背景下观察到反映花青素积累恢复的蓝色细胞(见图)。6.a和c)。在UV光下的同一细胞中也观察到GFP荧光(图。6.B和D)。轰炸P35S-GUS和P35S-SGFP没有产生蓝细胞,并且仅检测到GFP荧光荧光(图。6.e和f)。这些结果表明,花青素的积累GST1.通过瞬态表达恢复基因组编辑的线#12GST1..
叶片诱导叶片中的花青素色素沉着
花青素产生的若干因素诱导,包括低温,高强度光,渗透胁迫和病原体感染。调查是否GST1.基因组编辑的品系可以在应激反应中积累花青素,我们将它们培养在通常诱导花青素积累的富糖培养基中。WT龙胆草变红是因为花青素3-葡萄糖苷作为主要花色苷的积累(图。7.A和B)。然而,GST1.基因组编辑的第12行植物没有变红,积累的花青素3-葡萄糖苷相对较少。3号株系的花呈淡蓝色,呈与WT株系相似的红色,并且在花青素3-葡萄糖苷的积累上与WT株系无显著差异(图3)。7.c)。
讨论
花青素积聚在各种植物器官,如花瓣,水果,种子涂层,茎,叶和根。这些化合物是黄酮类化合物,它们对花色的贡献已经彻底审查了[23].此外,也研究了它们对人类健康的有益影响[24].类黄酮的生物合成途径已经在高等植物中得到了很好的表征,包括生物合成酶(如查尔酮合酶到花青素合酶)以及催化糖基化和酰化的花青素修饰酶(图。5.a).最近的研究表明,一种被称为代谢子的结构功能复合物是由内质网(ER)的类黄酮生物合成相关酶构成的,用于类黄酮化合物的高效生物合成[25].通过转录因子,包括MYB,BHLH和WD40的这些生物合成基因对这些生物合成基因的转录调节已在许多植物物种中得到阐明[26].如上所述,在日本龙胆草中已经发现了与龙胆素生物合成相关的生物合成基因和转录因子基因。
由于它们的水溶性,花青素积聚在真空中,这是所有植物中存在的膜结合的细胞细胞器。已经提出,通过多药抗性相关的ABC转运仪将花青素传输到液泡中[27在一种途径中,需要通过GST将谷胱甘肽(GSH)缀合到花青素。然而,基于GST在花青素转运中的涉及的机制仍然未知,因为在大多数情况下没有明确证实GSH缀合的花青素。PHI思潮GSTS,如AN9(Petonia)[28, AcGST1(猕猴桃)[14]和CsGSTa(茶)[15,据说能与几种类黄酮结合。因此,这些gst可能作为细胞质类黄酮载体蛋白,将花青素从内质网转移到液泡膜。
在这项研究中,我们选择了一个候选的Phi-型与嗜酸性蛋白相关的销售税基于RNA-seq数据的从头组装构建的转录组草案龙胆triflora花瓣和叶子(DRA010021;BioProject PRJDB9616)。基于这个连续序列,GST1.是从' Albireo '品种(龙胆triflora×作用研究),并进行功能分析。具体来说,我们有针对性的GST1.使用CRISPR / CAS9基因组编辑系统,该系统已被用于功能性地表征龙胆基因[22].双胞胎GST1.根据Sanger测序和NGS扩增子测序数据计算出突变效率为13.0%(7/54个株系)1:表S1和S2)。这一效率略高于前人对龙胆草花青素修饰基因的研究GT5GT.(5.7%),Gt3'GT(4.9%),和gt5 / 3'at.(8.6%) (22].效率的差异取决于目标序列。在与WT花颜色不同的5条线中,我们重点关注两个典型的GST1.基于花色变化的程度,敲除线,#3和#12。
第3行的表型变化相对温和,花朵比蓝色WT龙胆的花朵略微苍白。在基因组DNA和转录物中的靶位点区域的NGS扩增子期间,由于插入衍生自叶绿体基因组的特定的长序列,因此没有用FASEQ-Join脚本构建线#3的等位基因1片段。目标站点1和2之间的序列删除(数据未示出)。因此,我们认为该突变消除了来自等位基因1的功能性蛋白的翻译。另外,我们推测,线#3的等位基因2中的缺失不是至关重要的突变,因为它导致仅缺失三个没有a的氨基酸架子。如图1中所示。3.a,我们在第3行观察到轻微的花颜色变化,花青素含量与WT水平相比没有显著下降(图3)。4.b).此外,直线3和WT植物在花瓣表面的色调角度没有差异(图3)。3.b).第3行和WT植株花瓣提取物的HPLC图谱上的峰型相同,检测到的小峰也相同(图3)。4.一种)。这些结果表明,在目标网站1的删除GST1.3号外显子消除了3个氨基酸,产生了一种功能蛋白,活性略低于正常水平。这种活性下降的结果是轻微抑制龙胆蝶呤的积累,导致轻微的花颜色变化(即,GST1.可拆卸的)。
相比之下,在GST1.基因组编辑的第12行,这些花是淡蓝色到近乎白色的。第12行总花青素含量小于WT对照的10%。靶位点1和2的双等位基因基因组编辑均得到证实(表)1).这些突变被认为会导致框架移位和异常终止密码子。其他GST1.基因组编辑的线(即,#13,#23和#29)也被证实在目标站点处具有双倍裂突变(附加文件1:表S1),产生的花朵颜色类似于第12行(附加文件2:图。S1)。因此,我们总结了基因组编辑GST1.结果显示,第12、13、23和29行基因敲除表型与第3行基因敲除表型相反。
因为目前尚不清楚观察到的表型是否是由于GST1缺乏,所以我们进行了互补测定。花瓣的蓝色着色恢复了GST1.基因组编辑第12行,由任一瞬时表达GST1-1或GST1-2(图。6.),表明白花表型确实是由aGST1.突变。GST1的重要性也表现出来。具体而言,即使花青素生物合成途径是活性的,也是GST1介导的最终运输对于龙胆植物中的花青素的积累是必要的。
花发育的qRT-PCR结果显示,两种植物的表达模式相似GST1.那CHS.,ch,早期表达在阶段2.在这个阶段,GST1.两者的表达水平均显著降低GST1.基因组编辑的线,而不是WT植物。这可能与废话介导的mRNA衰减(即真核mRNA质量控制)有关[29那30.].调节患有花青素相关表达的转录因子销售税基因已在Kiwifruit中鉴定(ACMYBF110ACGST1.)[14]和苹果(mdmyb1 formdgstf6.)[16].以龙胆为基础GST1.表达模式(无花果。5.b),GTMYB3不太可能调节GST1.表达。下调表达GST1.没有显着影响检查的类黄酮生物合成基因的表达水平,表明通过GST的反馈调节没有影响花青素积累。不幸的是,我们没有识别转录因子调节GST1.在龙胆植物中的表达。因此,需要进一步的研究来澄清GST1.转录调控。
通过外源糖处理诱导花青素积累,并且已经表征了相关蔗糖信号传导中涉及的调节基因31那32].在本研究中,我们观察到糖诱导的花青素色素沉着在离体培养的植物中被抑制GST1.基因组编辑线(图。7.).这种抑制在敲除株12中表现严重,与花色表型一致。这些结果表明GST1.虽然更详细的分析,但还有助于龙胆植物中的应激诱导的花青素积累销售税其他器官的表达是必要的。从实用的角度来看,叶色的形成通常是观赏花卉的不良特征。因此,研究花青素转运对胁迫诱导的花青素积累的调控机制是十分必要的。
我们的GST1.基因组编辑的龙胆线没有产生完全白花,暗示其他蛋白质也参与了龙胆植物的花青素运输。我们在本研究中选择并分析了PHI(F)类GST。但是,编码TAU(U)类GST的几个基因,如BZ2.在Zea Mays.[33] 和VviGST1在葡萄[34,也与花青素有关。最近,CsGSTb和CsGSTc从茶树[15被证实为Tau分支销售税与花青素运输有关的基因。我们的系统发育分析(图。1a)表示a销售税基因,基于De Novo Gentria转录物检测的Trinity_DN42846_C0_G1_I1属于TAU疏水板,分别推导的氨基酸序列同一性分别与VVIGST1,CSGSTB和CSGSTC的42,54和55%。因此,未来的研究应该检查TAU型GST以及ABC运输扣,以阐明龙胆植物中的花青素积累的潜在机制。
结论
在这项研究中,我们分析并分析了一种与花青素相关的销售税的基因,GST1.,种植日本龙胆草。CRISPR/Cas9基因组编辑系统证实了其对花色素的贡献。我们生产一些GST1.基因组编辑的线条分析了突变对花色的影响。本文呈现的数据揭示了两种不同的表型,即敲除(几乎白色)和敲低(淡蓝色)表型。这些结果表明了通过CRISPR / CAS9系统的基因组编辑可以应用于敲出靶基因以及略微修饰感兴趣的基因的等位基因。我们的研究结果可能与澄清高等植物中的花青素运输相关。此外,我们的突变线可能对龙胆育种有用,因为在天然突变体中未检测到这种突变。我们的结果表明GST1.尽管仍有待鉴定额外的相关基因,但有助于介导龙胆植物中的花青素积累。基因组编辑技术的使用将增强龙胆植物中的花青素运输的未来研究。
方法
植物材料与转化体的选择
蓝花龙胆品种' Albireo ' (龙胆triflora×作用研究)(即WT)用于CRISPR/Cas9系统的靶向基因组编辑。该品种由西原正弘博士鉴定,是在岩手县培育的,已在日本农林水产省植物品种保护数据库中登记(注册号:028928986)。2553)。这是由岩手农业研究中心好心提供的。在我们之前的研究中,WT植株使用添加3%蔗糖的半强度MS培养基作为基础培养基,在相同的培养条件下保持离体培养[22].
龙胆草属植物按一种农-介导的过程,然后如前所述选择基因组编辑的转化子[22那35].简单地说,在含0.75 mg l的培养基上筛选转化苗−1Bialaphos。基于PCOCAS9片段的PCR扩增用Mightyamp DNA聚合酶(Takara,Shiga,Japan)来选择候选转基因植物。对PCOCAS9特异的引物#61和#62用于根据制造商的指示作为模板的原油提取物的PCR分析。通过通过与靶位点和目标位点区域特异的引物#154和#155的PCR产物的直接和亚克隆序列分析检测靶位位点中的插入或缺失来选择基因组编辑的植物。(图。2一种)。关于所有引物的详细信息列于其他文件中1:表S3。再生GST1.基因组编辑的线适应于闭合的温室或在发光二极管灯下培养,如前所述直到它们绽放[36].收获花瓣和叶,立即在液氮中冷冻,并在-80℃下储存直至分析。
孤立销售税与花青素积累相关的cDNA和基因组序列
从WT植物的花瓣中提取的总RNA合成单链CDNA,并如前所述用于PCR扩增[22].用于设计龙台的引物的序列销售税基因来源于DDBJ DRA数据库。DRA010021)。在13万多个contigs中,有30个被确定为销售税基于BLASTX(版本2.2.26)的基因匹配,包括一个GO术语“含花青素化合物代谢过程”的基因。我们根据测序数据设计了引物276和277销售税随后3'-快速扩增cDNA的CONTIG与Generacer Kit(Invitrogen,USA)结束。全长GST1.用引物#325和#326通过PCR扩增序列,之后将扩增子用大染料终止子循环测序试剂盒(版本1.1)和ABI棱镜3130xL或3500遗传分析仪(Applied Biosystems,Ca,Ca,美国)。
以隔离整个长度GST1.基因组序列,基因组DNA与基因特基因组DNA分离系统(Sigma-Aldrich,St Lois,Mo,USA)的WT龙胆植物的幼叶中分离出来。如上所述进行具有引物#325和#326和扩增子的测序的PCR扩增。用于隔离的底漆GST1.列于其他文件1:表S3。这销售税将编码区翻译成具有扩展的相应推导的氨基酸序列(https://web.expasy.org/translate/).生成系统发育树,并将GST氨基酸序列与CLUSTALW (Thompson et al. 1994)、MEGA (version 10.1.7)和Jalview (version 2.10.3)程序进行比对。用于构建系统发育树的GST蛋白序列列于附加文件1:表S4。
编辑二元载体的构建GST1.在龙胆草基因组中
针对绅士的二进制Crispr / cas9矢量GST1.构建了两个单引导RNA表达盒,使用与我们之前研究相同的程序[22].两个目标网站GST1.外显子3如图所示。2一种。得到的二元载体psbar-pcocas9-atu6-26p-gst1target-1grna-atu6-26p-gst1target2grna(图。2b)转化为农菌株EHA101通过电穿孔并用于龙胆转换。
下一代基因组编辑的靶位点区域测序分析GST1.
花瓣中的基因组DNA和转录本GST1.利用Illumina MiSeq下一代测序仪对基因组编辑的品系和WT植物进行分析,确定目标位点序列。基因组dna和cdna的制备如上所述。提取的RNA样本用PrimeScript II (TaKaRa)进行逆转录。如前所述,在Illumina系统上进行扩增子测序[21].简而言之,使用FASTX Toolkit预处理原始读取,之后使用FASTQ-Join脚本构建包含目标站点区域的单个连续序列(片段)[37].最后,所有独特的碎片都被计算在内。用于NGS分析的引物在附加文件中列出1:表S1。
龙胆花瓣的测量
采用CIE法测定色差l * a * b *,其中L *亮度,一种*红色/绿色的坐标是和吗B *是黄色/蓝色坐标。量化花色表型GST1.基因组编辑的品系和WT植物,比色值L.*,一种*, 和B.*以及色度(C*)值和色调角,它们是根据L.*,一种*, 和B.*值,测量鲜瓣瓣翼面近轴面面积。使用台式CM-3600A分光光度计(日本东京柯尼卡美能达)进行分析。
花瓣中的花青素组合物分析GST1.基因组编辑龙胆植物
通过HPLC系统(PU-4180泵,MD-4010光电二极管阵列检测器和Chromnav(2.03.05版)软件分析了花瓣提取物的花瓣素组合物;Jasco,东京,日本]配备UnifinePak C18柱(内径4.6毫米×250 mm; jasco)。从新鲜花瓣中提取花青素,并如前所述进行HPLC分析[22].在30分钟内,我们在1%磷酸水溶液中使用了14-22%乙腈的线性梯度洗脱(1.1mL min -1)。V-730BiO分光光度计(JASCO)用于估计基于Delphinidin氯化物的摩尔吸收率的花青素浓度[εmol= 506,783,在530nm处,评价在含有0.1%(v / v)三氟乙酸的80%甲醇溶液中。
表达分析GST1.以及类黄酮生物合成相关基因
从如上所述的四个发育阶段收集的花瓣中提取总RNA,然后根据制造商的说明,用PrimeScript™RT试剂试剂盒和gDNA Eraser (Perfect Real Time) (TaKaRa)合成cDNA。使用TB Green™Premix Ex Taq™II (Tli RNaseH Plus) (TaKaRa)和QuantStudio 5 Real-Time PCR系统(Applied Biosystems Japan)完成qRT-PCR检测。总体积为20 μl的反应混合物由主混合10 μl、引物各0.8 μM和cDNA模板1 μl组成。qRT-PCR引物为GST1.那CHS.那ch那F3'5'H.那DFR.那答那5/3'AT,my列于其他文件1:表S3。PCR条件为:50℃,2 min;95°C 10分钟;40个循环,95°C 15 s, 60°C 1 min。采用熔融曲线分析验证qRT-PCR产物的特异性和特异性。每个基因的表达水平被归一化UBQ表达水平并基于最大表达级别作为相对值记录。
编辑的互补GST1.通过粒子轰炸
在本研究中,p35S-sGFP含有sGFP在花椰菜花叶病毒35S启动子的控制下,以p35S-GUS为阴性对照质粒,作为转化对照质粒。这sGFP片段被修改过的片段所取代GST1-1(要么GST1-2)(目标网站中的六个基替代;其他文件中的详细信息2图S2),旨在破坏整合的CRISPR/Cas9结构的编辑,导致p35S-GST1-1和p35S-GST1-2。重组质粒共沉淀到金颗粒上。采用PDS-1000/He生物粒子输送系统(Bio-Rad, CA)对其进行了轰击GST1.如前所述的基因组编辑龙胆花瓣与颗粒[38].24小时后,在可见的和紫外光下显微镜地分析花瓣。
叶片诱导叶片中的花青素色素沉着
wt和GST1.将体外培养的基因组编辑龙胆草植株转移到含10%蔗糖的半强度MS培养基中。植物在22°C、16小时光照/8小时暗光周期的培养室内生长,由白光二极管提供光照。用高效液相色谱法进行了上述分析。花青素的含量是根据花青素-葡萄糖苷的六种连续稀释的HPLC峰面积产生的标准曲线估计的。
数据和材料的可用性
本研究产生的数据包括在这篇发表的文章及其附加文件中。在当前的研究中使用和/或分析的主要数据集可从通信作者在合理的要求。
缩写
- GST:
-
谷胱甘肽S.转移酶
- CRISPR:
-
集群定期间隔的短文重复
- 门店:
-
下一代测序
- 聚合酶链反应:
-
聚合酶链反应
- 高效液相色谱法:
-
高效液相色谱
参考
- 1.
Goto T, Kondo T, Tamura H, Imagawa H, Iino A, Takeda K. structural of gentiodelphin, an酰化花青素龙胆maikinoi,在稀水溶液中稳定。四面体。1982;23:3695-8。
- 2.
Yoshida K,Toyama Y,Kameda K,Kondo T. Gentiodelphin颜料分子的每种咖啡酰基对蓝色发育的贡献。植物化学。2000; 54:85-92。
- 3.
从化学结构到细胞生理学的研究。Nat Prod众议员2009;26:884-915。
- 4.
Nakatsuka T,Haruta Ks,Pitaksutheepong C,Abe Y,Kakizaki Y,Yamamoto K,Shimada N,Yamamura S,Nishihara M. R2R3-MyB和BHLH转录因子在龙胆花中调节花青素生物合成的R2R3-MYB和BHLH转录因子的鉴定及表征。植物细胞生理。2008; 49:1818-29。
- 5.
中图卡,西原。利用CRES-T技术生产龙胆草皮果型花的研究。生物科技植物》。2011;28:173 - 80。
- 6。
Tasaki K,Atsumi G,Nishihara M,Sekine Kt。发展A.蚕豆枯萎病毒2基于龙胆草的表达载体。Sci长的矮。2016;201:279 - 86。
- 7。
王志强,王志强,王志强,等。龙胆草中黄酮类化合物合成的转录调控因子及其在花色修饰中的应用。生物科技植物》。2014;31:389 - 99。
- 8.
Nakatsuka T, Saito M, Sato-Ushiku Y, Yamada E, Nakasato T, Hoshi N, Fujiwara K, Hikage T, Nishihara M.在日本龙胆草植物中区分白色和蓝色花的DNA标记的开发。Euphytica。2011;184:335-44。
- 9.
Kakizaki Y, Nakatsuka T, Kawamura H, Abe J, Abe Y, Yamamura S, Nishihara m作用研究.繁殖研究》2009;11:9-14。
- 10.
西原M,田崎K,佐佐木N,高桥h。日本龙胆草育种和栽培改良基础技术的开发。Sci繁殖。2018;68:14-24。
- 11.
黄酮类化合物的转运机制:如何去,和谁一起去。植物学报。2015;20:576-85。
- 12.
Sasaki N, Nishizaki Y, Uchida Y, Wakamatsu E, ummoto N, Momose M, Okamura M, Yoshida H, Yamaguchi M, Nakayama M,等。识别的谷胱甘肽S-转移酶康乃馨花色强度的基因。生物科技植物》。2012;29:223-7。
- 13.
一种与花青素相关的谷胱甘肽的分子特性S.- 仙客来的 - 转移酶基因。J植物理性。2012; 169:636-42。
- 14.
刘勇,齐勇,张安,吴华,刘震,任欣ACGST1.,一种与花青素相关的谷胱甘肽S.- 猕猴桃中的转移酶基因(猕猴桃对).acta botanica sinica(云南植物学报)2019;
- 15.
刘y,姜h,赵y,李x,戴x,庄j,朱m,江x,王p,gao l等。三茶树谷胱甘肽S.- 转移酶参与花青素,黄酮醇和原花青素的储存。Planta。2019; 250:1163-75。
- 16。
蒋某,陈某,他,陈X,王n,孙q,张t,徐h,方h,王y等。MDMYB1激活的MDGSTF6在Apple中的花青素积累中起重要作用。Hortic Res。2019; 6:40。
- 17。
Soda N,Verma L,Giri J. Crispr-Cas9基础植物基因组编辑:意义,机会和最近的进步。植物理性生物化学。2018; 131:2-11。
- 18。
马X,张q,朱q,刘文,陈y,秋r,王b,杨z,李h,林y等。一种强大的CRISPR / CAS9系统,用于单子床和DICOT植物中的方便,高效多重基因组编辑。莫尔植物。2015; 8:1274-84。
- 19.
张Y,Malzahn Aa,Sretenovic S,齐Y.植物科学中CRISPR技术的新兴和未开发潜力。Nat植物。2019; 5:778-94。
- 20.
Watanabe K,Kobayashi A,Endo M,Sage-Ono K,Toki S,Ono M. Crispr / Cas9介导的诱变dihydroflavonol-4-reductase-B (DFR-B)日本吉罗斯的遗迹IPOMOEA(Pharbitis)零.SCI批准。2017; 7:10028。
- 21.
Nishihara M, Higuchi,渡边,Tasaki k CRISPR / Cas9系统的应用程序修改花的颜色Torenia Fournieri..BMC Plant Biol. 2018;18:331。
- 22.
Tasaki K,Higuchi A,Watanabe A,Sasaki N,Nishihara M.敲除三种花青素修饰基因对龙胆花的蓝色色素沉着的影响。SCI REP。2019; 9:15831。
- 23.
黄酮类化合物(包括花青素)对花色的研究进展。Nat Prod Commun. 2015; 10:529-44。
- 24.
何乔,阿志兰,邓街,林思敏。花青素和花青素:色素作为食品、医药成分的潜在健康益处。食品营养学报2017;61:1361779。
- 25.
Nakayama T,Takahashi S,Waki T.形成类黄酮代谢粘土:蛋白质 - 蛋白质相互作用的功能意义和对黄酮化化学大学的影响。前植物SCI。2019; 10:821。
- 26.
徐W,Dubos C,Lepiniec L. myB-BHLH-WDR复合物对类黄酮生物合成的转录控制。趋势植物SCI。2015; 20:176-85。
- 27.
Behrens CE, Smith KE, Iancu CV, Choe JY, Dean JV。拟南芥atp结合盒转运体AtABCC2运输花青素和其他类黄酮。Sci众议员2019;9:437。
- 28。
Mueller La,Goodman Cd,Silady Ra,Walbot V.An9,矮牵牛谷胱甘肽S.-转移酶,是一种类黄酮结合蛋白。植物杂志。2000;123:1561 - 70。
- 29。
无意义介导的mRNA衰减对遗传疾病、基因编辑和癌症免疫治疗的影响。Nat麝猫。2019;51:1645-51。
- 30.
Tuladhar R, Yeu Y, Tyler Piazza J, Tan Z, Rene Clemenceau J, Wu X, Barrett Q, Herbert J, Mathews DH, Kim J,等。基于crispr - cas9的突变经常引起靶上mRNA的错误调控。Nat Commun。2019;10:4056。
- 31.
Ai TN, Naing AH, Arun M, Lim SH, Kim CK。蔗糖诱导花青素在植物营养组织中的积累矮牵牛植物需要花青素调控转录因子。植物科学。2016;252:144-50。
- 32.
李亚平,王志强,王志强,等。蔗糖诱导花色素苷合成的研究进展。摩尔。2014;7:570-2。
- 33.
Marrs KA, Alfenito MR, Lloyd AM, Walbot v.a谷胱甘肽S.-转移酶参与玉米基因编码的液泡转移Bronze-2.大自然。1995;375:397 - 400。
- 34.
张伟。谷胱甘肽的分离纯化、分子克隆及性质研究S.-转移酶(GSTs)葡萄作为假定的花青素转运蛋白的L.细胞悬浮培养。J Exp Bot. 2008; 59:3621-34。
- 35.
关键词:龙胆草,35s基因,转录沉默,基因表达植物j . 2005; 44:541-56。
- 36.
关键词:龙胆草,悬浮培养细胞,粒子轰击,转基因Plant Cell rep 2000; 19:454-8。
- 37.
Aronesty E.测序实用程序的比较。Open Bioinform J. 2013; 7:1-8。
- 38.
Nakatsuka T,Nishihara M,Mishiba K,Yamamura S.两种不同的突变参与了白花龙胆植物的形成。植物SCI。2005; 169:949-58。
致谢
我们感谢Holger Puchta博士(德国Karlsruhe理工学院Karlsruhe TechnitiTute of Germany)提供PCOCAS9。一些计算分析与Rois国家遗传学研究所的NIG超级计算机完成。我们还感谢Drs。Nobuhiro Sasaki,Toyo大学,日本和Keiichirou Nemoto,岩手生物技术研究中心,为他们有用的讨论。作者感谢MSES。R. Washiadshi和Y.Kurokawa,岩手生物技术研究中心,为其技术支持。我们感谢Edanz Group(https://en-author-services.edanzgroup.com/)为编辑本手稿的初稿。
资金
这项工作得到了岩手县的财政支持,同时也得到了日本科学促进协会的科学研究补助金(16 K18654和18 K14462)的部分支持。这项工作还得到了日本农林水产省委托项目“适合高实际需求的花卉植物育种和低成本栽培技术开发”和“通过基因组编辑开发作物新品种和育种材料”的部分支持。资助机构没有参与实验设计或数据分析。
作者信息
从属关系
贡献
KT和MaN构思并设计了实验。AH和AW对试验材料进行了制备和处理。KT、MY、MiN分析了数据。KT对NGS分析做出了贡献。MaN指导了这项研究。卡帕头写的手稿。他批判性地阅读并回顾了手稿。所有作者都认可了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
提交人声明他们没有竞争利益。
附加信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:表S1。
Sanger测序DNA PCR扩增子的分析GST1.基因组编辑的品系和WT对照。表S2。DNA和RNA的PCR扩增子的下一代测序分析GST1.基因组编辑的品系和WT对照。表S3。本研究中使用的引物。表S4。谷胱甘肽S.- 用于构建图1中的系统发育树的转移酶蛋白。1一种。
附加文件2:图S1。
花的颜色特征GST1.基因组编辑龙胆草品系#13,#23和#29。(A)花的颜色表型GST1.基因组编辑的线#13,#23和#29。(b)L.*,一种*, 和B.*用CM-3600A分光光度计(东京,日本Konica Minolta,日本)测量新鲜花瓣表面的颜色值。还计算了色度值和色调角。图S2第三外显子的序列GST1.对于互补测定。突出显示的核苷酸(即阴影,粗体或盒装)与图1中的相同。2b.瞬时表达试验的取代核苷酸用红色表示。这些取代并没有改变编码的氨基酸。
额外的文件3
WT和WT的下一代扩增子测序结果GST1.genome-edited龙胆线。Excel工作表中序列名称后面的数字是每个片段的连续序列号和读取计数。
权利和权限
开放获取本文根据创意公约归因于4.0国际许可证,这允许在任何中或格式中使用,共享,适应,分发和复制,只要您向原始作者和来源提供适当的信贷,提供了一个链接到Creative Commons许可证,并指出是否进行了更改。除非信用额度另有说明,否则本文中的图像或其他第三方材料包含在文章的创造性公共许可证中,除非信用额度另有说明。如果物品不包含在物品的创造性的公共许可证中,法定规定不允许您的预期用途或超过允许使用,您需要直接从版权所有者获得许可。要查看本许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
多崎,吉田,中岛,中岛。et al。花青素相关谷胱甘肽的分子特征S.利用CRISPR/Cas9系统分析日本龙胆草-转移酶基因。BMC植物杂志20.370(2020)。https://doi.org/10.1186/s12870-020-02565-3
收到了:
公认:
发表:
关键字
- 花青素
- CRISPR / CAS9.
- 花卉色素沉着
- 基因组编辑
- 龙胆
- 销售税