摘要
背景
在真核生物中,选择性剪接是一个重要的过程,可以从一个前体信使RNA产生多个转录本,极大地增加了转录组和蛋白质组的多样性。这个过程是由一种被定义为剪接体的超蛋白复合体完成的。具体来说,剪接因子1/分枝点结合蛋白(SF1/BBP)是一种能够与内含子分枝点序列(BPS)结合的单一蛋白,在早期剪接体组装过程中连接5 '和3 '剪接位点结合复合体。该蛋白在酵母、后生动物和哺乳动物中的分子功能已被广泛研究。然而,它在植物中的对应物却很少报道。
结果
为此,我们进行了系统的表征SF1跨植物谱系的基因家族。在这项工作中,共鉴定了59种植物的92个序列。构建了这些序列的系统发育关系,随后的生物信息学分析表明,该家族可能起源于一个古老的基因转位复制事件。大多数植物物种都保持着这种基因的单一拷贝。此外,与动物和酵母基因相比,该基因家族的大多数成员中都存在一个额外的RNA结合基序(RRM),这表明它们的潜在作用在植物谱系中被保留了下来。
结论
我们的分析展示了该剪接因子家族的基因和蛋白质结构的一般特征,为进一步的植物功能研究提供了基础信息。
背景
在真核生物中,规范剪接去除非编码内含子序列并将编码元件组装成成熟的mrna,而选择性剪接(alternative splicing, AS)通过不同的外显子或剪接位点的使用产生不同的多重转录本,对具有不同结构和功能的蛋白质进行编码[58,70].AS产生的转录本对真核生物的转录后调控、生物复杂性和蛋白质组多样性有很大贡献[20.,50,74].考虑到人类转录组中每个转录本平均约有8个外显子,以及相应剪接位点的退行性[20.], pre-mRNA的剪接是由剪接体复杂地催化的。剪接体是一种多兆吨级的蛋白质复合体,由5个(U1、U2、U4、U5和U6)小核核糖核蛋白粒子(snRNPs)和100多个剪接体蛋白质组成[74].此外,剪接体复合体E或承诺复合体的早期组装是一个atp无关的过程,包含U1 snRNP、SF1和U2 snRNP辅助因子(U2AF大亚基和U2AF小亚基)[48,51].随后,U2 snRNPs的SF3b155/SAP155取代SF1,形成前剪接体复合体A [19,67,77].以下剪接体在剪接反应中的逐步组装也有报道[44,63];然而,剪接位点的识别是剪接体早期组装的关键步骤。目前的模型描述了U1 snRNP和U1 snRNA在5 '剪接位点与6个核苷酸的短链结合,在剪接位点与剪接因子1 (SF1)/哺乳动物分支点结合蛋白(mBBP)结合,在3 '剪接位点与U2 snRNP辅助因子结合[46].这三个独联体-元素是必要的,但通常不足以定义一个特定的外显子-内含子边界。因此,位于外显子和内含子的附加剪接增强子或沉默子可能允许在早期剪接体组装过程中识别真正的剪接位点[29].
重要的是,SF1优先结合U2AF大亚基(哺乳动物U2AF65和裂变酵母U2AF59)结合位点(多嘧啶束,Py)附近的内含子分支点序列(BPS),连接U1和U2AF形成中间套索结构[58,81].特别地,SF1的特征是在n端存在两种类型的RNA结合基元,一个K同源/震动2 (KH/QUA2)结构域起源于人类异质核糖核蛋白(hnRNP) K蛋白[17,66和一个或两个锌关节motif(s) (CX2残雪4HX4C, X代表任意氨基酸)。SF1在c端也含有一个富含脯氨酸的区域[2,3.].有趣的是,酵母KH结构域特异性地与带有Gly-Pro-Arg-Gly基基和KH结构域可变环的pre- mrna的BPS结合[39并且是剪接体组装所必需的[57].酵母中的第一个而不是第二个锌节结构域已被证明与RNA具有高亲和力[16].此外,SF1的几个丝氨酸残基(Ser20、Ser80和Ser82)在体外的磷酸化状态进一步影响了SF1 - u2af65 - rna复合体的稳定性[45].酵母和人类中SF1的富脯氨酸区与U1 snRNP Prp40/FBP11相互作用[2,38].对于其相互作用伙伴U2AF大亚基,SF1的n端与其非规范RNA识别基序(RRM)或U2AF同源基序(UHM)相互作用[57,62],而U2AF大亚基的另外两个rrm则绑定在Py区域[65].
先前对裂变酵母的研究(粟酒裂殖酵母)表明,分支位点和3 '剪接位点的初始共识别对于目标pre- mrna的正确剪接至关重要[60].由于剪接位点识别对基因表达和蛋白质多样性的重要性,SF1已被证明在许多真核生物物种中发挥重要作用,包括人类(智人),老鼠(亩骶)、芽孢酵母(酿酒酵母)、普通果蝇(黑腹果蝇)及蛔虫(秀丽隐杆线虫) [2,27,47,64,68].例如,在人类中,负责剪接位点识别的剪接因子(如SF1)的错义突变与肿瘤的发生有关[33].同样,杂合子SF1(+/−)敲除小鼠易受有机营养致癌物偶氮甲烷诱导的结肠肿瘤发生[64], SF1已被发现与-连环蛋白/TCF4复合体相关,表明其在致癌中的作用[49].相比之下,敲低SF1抑制了小鼠生殖细胞肿瘤的发展[83],表明其在癌症研究中的组织依赖性。此外,SF1在酵母中的分子功能已被广泛研究。例如,sf1突变菌株在裂变酵母中引起频繁的外显子跳跃[52].此外,SF1被认为可以识别特定内含子中的次优序列,并导致具有异常剪接的pre-mRNA的核积累[73].然而,越来越多的证据表明,该蛋白是剪接位点识别的调节因子,并不减少一般剪接,特别是在针对基因子集的选择性剪接过程中[46,52,68].这一假设得到了酵母和人类提取物中敲除SF1对剪接结果的轻微影响的事实的支持[22].该基因的RNAi靶向性已被证明不会影响测试的几个拼接标记基因的拼接模式[68].
与人类和酵母的研究相比,关于植物的报道很少SF1基因。类似的功能拟南芥SF1基因在2014年的一项早期研究中被提出[30.].据报道,这种植物SF1的同源物负责一组转录本的剪接。功能缺失突变体(atsf1-2该基因的异常发育(早花和侏儒症)和ABA或热应激敏感性拟南芥[30.,36].随后,畴结构及其功能关系得到了深入研究[36RRM结构域被认为是维持其在植物中的功能的关键。此外,SF1在植物中可能具有不同的3’剪接位点识别机制,因为与真菌和后生动物的对应物相比,植物SF1同源物包含不同的RRM结构域[53,78].另一方面,一项研究发现,AtSF1可能在细胞质中发挥功能性作用,因为它被发现在细胞核和细胞质之间穿梭[54].然而,在植物的系统发育分析方面还没有相关的研究SF1基因及其调控机制。虽然它是一个高度保守的家族,在真核生物中具有保守的功能SF1与哺乳动物基因相比,基因可能具有重叠和不同的作用。因此,研究植物的系统发育关系和调控机制SF1基因可以帮助我们了解该基因家族的进化历史、特征和表达谱,并预测植物的特定功能。这为进一步研究绿堇属植物的功能奠定了基础。为此,我们系统地确定了92个SF159种植物的序列,从藻类到高等植物。同时,进一步研究了这些基因的基因和蛋白结构、启动子区域的潜在调控作用和表达模式。在本研究中,我们假设植物SF1在结构上与动物和酵母中的同类蛋白不同,但在低等植物和高等植物中是保守的,表明其在分枝点识别中选择性剪接的特定作用。
方法
植物序列的获取与鉴定SF1基因
的拟南芥SF1蛋白序列(AT5G51300)从Phytozome v12.1数据库(https://phytozome.jgi.doe.gov/pz/portal.html) [18通过运行一个电子值截止= 1e-的blasp程序10(其他参数为默认设置)[7].然后,使用HMMER评分(默认设置)对检索到的蛋白质序列进行检查和过滤[31],包含PF16275(剪接因子1螺旋-发夹结构域,SF1-HH)、PF00013 (K同源结构域,KH_1)和PF00076 (RNA识别基序,RRM_1)。最后,92年的SF1鉴定了59种植物的序列。用于后续分析的每种植物的类群、植物种类、常见名称和SF1同源物数量等详细信息列于表中S1.利用WoLF PSORT (https://wolfpsort.hgc.jp/) [25].
植物分子系统发育树的构建SF1基因
上述植物的蛋白质序列SF1从Phytozome v12.1数据库中提取基因进行系统发育关系分析。对具有多个不同剪接异构体的基因选择编码序列最长的序列。然后,多SF1蛋白序列与默认设置的Muscle v3.8软件对齐[13].植物的分子系统发育树SF1采用最大似然法(ML, JTT + G + I模型)通过PhyML v3.0程序进行基因构建,参数如下:初始树:BioNJ;离散伽马模型:是;类别数目:4类;Gamma形状参数:0.709;不变量比例:0.021子树模式混叠:no [21].利用FigTree v1.4.3对系统发育树进行可视化和编辑。
基因结构、蛋白质结构域及多Em的模因分析
需要基因组,cDNA和肽序列和所有SF1基因结构从Phytozome v12.1数据库下载。使用在线程序基因结构显示服务器2.0 (GSDS2.0)生成相应的内含子相(http://gsds.cbi.pku.edu.cn) [26].的相关分析SF1外显子通过piece2 webserver (http://www.bioinfogenome.net/piece/search.php?tdsourcetag = s_pctim_aiomsg) [76].SF1蛋白质序列用于在HMMER网站(https://www.ebi.ac.uk/Tools/hmmer/) [14].然后利用TBtools软件绘制蛋白质结构域图[8根据完整的Pfam合成表。保存的植物图案SF1cDNA序列和蛋白质序列在MEME在线程序(http://meme-suite.org/tools/meme) [5]考虑每个序列预测最多保留10个图案,其他设置保持默认参数。
植物启动子区Motif预测SF1基因
植物1.5 kb 5 '侧翼序列SF1从Phytozome数据库的基因组数据中提取基因。植物假定顺式元素的预测是通过在线服务器PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/) [37].选择与组织特异性表达、内部激素和外部环境应激反应相关的基序进行进一步分析和讨论。
基于微阵列数据集和基因表达实验的表达分析
表达数据的拟南芥,美国tuberosum,番茄赤霉病菌,番茄赤霉病菌而且b . distachyon,包括组织特异性和胁迫响应,提取自植物生物学生物分析资源(http://bar.utoronto.ca/) [34].所选植物的表达值SF1利用BAR HeatMapper Tool程序(http://bar.utoronto.ca/ntools/cgi-bin/ntools_heatmapper.cgi).
基因表达实验
用RNeasy Mini试剂盒(QIAGEN, USA)提取不同植物组织样品的总RNA,然后由FastKing gDNA diselling RT SuperMix FastKing (TIANGEN, China)根据制造商说明反转录为cDNA。RT-PCR扩增程序如下:95°C, 3 min;95°C, 30 s;52°C, 15 s;72°C, 45 s;26/30周期;72°C 5分钟。精密生物技术有限公司采用StepOne Plus实时PCR系统进行实时RT-PCR定量分析,优化程序为:95℃,30 s;95°C, 5 s; 60 °C, 30 s; 40 cycles. The data were normalized to the expression of internal reference genes (TableS6),用比较CT值法测定转录本丰度[61].
蛋白质-蛋白质相互作用网络及结构守恒分析
蛋白质-蛋白质相互作用网络由STRING网站(https://string-db.org) [12的代表性蛋白序列拟南芥.采用以下基本设置:网络边的含义、证据(线颜色表示交互证据的类型);主动交互源,实验。
有三个域拟南芥SF1蛋白质。在3 '剪接位点识别过程中剪接因子1的n端结构域的磷酸化和U2AF65的结合智人(PDBID: 2M0G,标识度:36%,E-value: 7E-17)与K同源结构域相似。内含子分支位点RNA被剪接因子1识别的结构智人(PDBID: 1K1G,标识度:47%,E-value: 9E-27)可作为剪接因子1螺旋-发夹域的模板。因此,采用建模器[43基于两种晶体结构。氨基酸保存评分使用基于ML方法的ConSurf Web服务器计算[4].输入属性为三维模型和多序列对齐(图S4).基于Pymol默认设置创建相关图形[79].
分析基因结构演化与同源组SF1基因
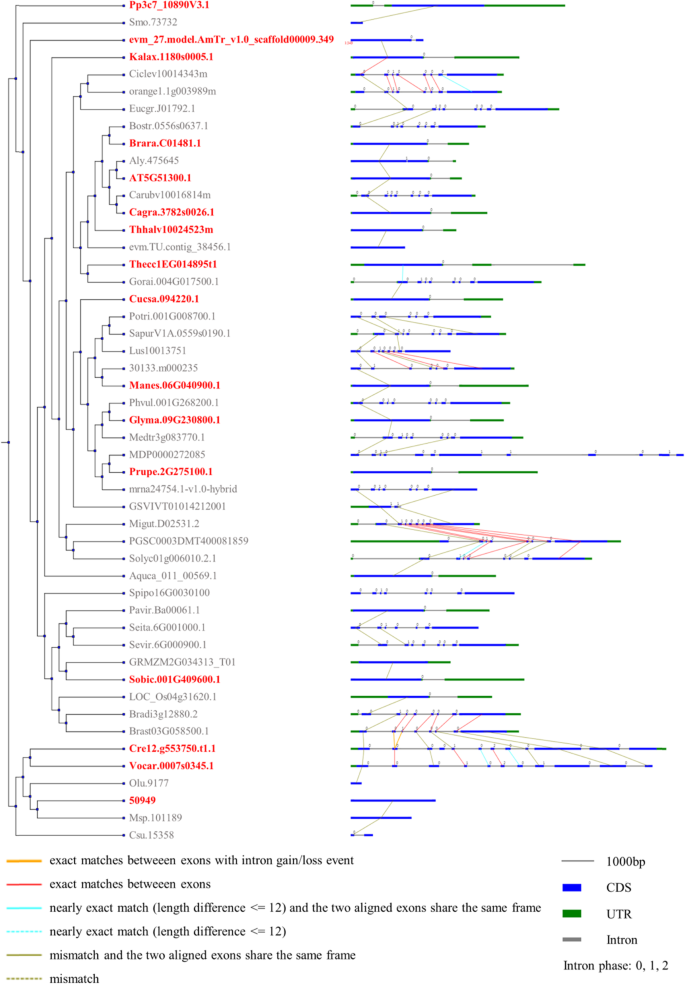
重建植物结构的进化史SF1同源基因家族通过在PIECE 2服务器中搜索AT5G51300.1 (http://www.bioinfogenome.net/piece/index.php).这为与系统发生树相关的基因结构数据集中的同源基因提供了外显子-内含子显示。
结果
植物的序列鉴定与系统发育分析SF1基因家族
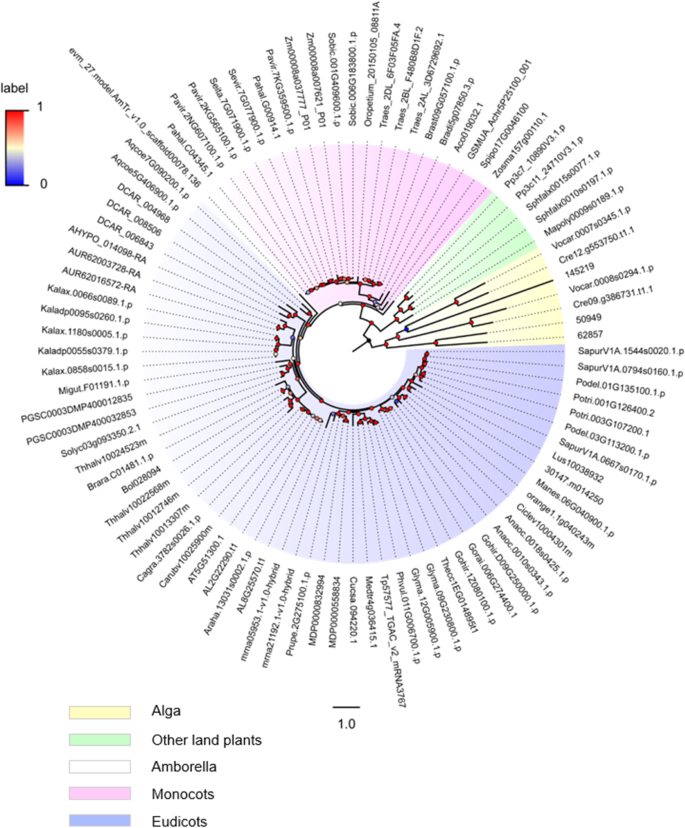
识别SF1基因家族成员,我们进行BLASTp搜索使用拟南芥AtSF1(AT5G51300)氨基酸序列对照Phytozome数据库(v12.1)。过滤后的序列没有SF1共检索到59种植物92条序列,其中藻类7种,苔藓植物5种,基本被子植物1种,单子叶植物21种,优生植物58种(表1)S1).具体来说,唯一具有四个植物SF1s拷贝的物种是Eutrema salsugineum(盐沟)(表S1).特别是三份SF1在五个物种中观察到基因,包括黍virgatum(柳枝稷),小麦(常见的小麦),胡萝卜胡萝卜(胡萝卜),高凉菜属laxiflora(乳白色的寡妇的颤抖)和柳树紫竹(紫色柳树柳树)。另外,20种植物包含2个拷贝,包括模式植物在内的33种植物拟南芥他只有一份植物的拷贝SF1s,分别。的相对较大的数量SF1基因和较高的植物物种数量在这项工作中证明了遗传的普遍性和复杂性SF1基因家族。检索到的59种植物的序列为我们分析植物的系统发育关系提供了更完整的信息SF1基因家族。随后,基于上述92个蛋白质序列,利用极大似然法构建了一棵有根的系统发生树。树的自举(阈值:0-1)由颜色渐变表示(图1)。1).一般来说,所有SF1蛋白质序列聚集成四个主要分支,包括藻类(黄色)、其他陆生植物(绿色)、单子叶植物(粉色)和多子叶植物(蓝色),以及一个物种(Amborella trichopoda)属于基本被子植物(显示为无色)。SF1s的系统发育树(图1)。1而且2(左图)具有清晰的拓扑结构和整体较高的自举值,与其他研究中报道的从较低植物到较高植物的进化趋势相似。例如,黄色分支中的藻类基因是在陆生植物进化之前分化的谱系的代表成员,这是系统发育的基础部分。在蓝色分支中,五个序列来自高凉菜属BS值越高,形成一个子枝,表明它们的进化关系越密切。此外,Cagra.3782 s0026.1。p从危害大花蔷薇和Carubv10025900mc .风疹形成了一个子分支拟南芥序列,因为它们都属于芸苔科,这与APG IV系统一致(图4)。1和表S1).通常情况下,来自同一物种的一些同源SF1序列彼此相邻聚集在同一个小分支中;这些物种包括腰果、大豆、苹果、林地草莓、藜麦、胡萝卜、科罗拉多蓝耧斗菜、玉米、普通小麦、谷类草、苔藓和沼泽苔藓。1和表S1).相比之下,来自同一物种的其他同源SF1成员则聚集在不同的亚分支中,如紫柳柳、杨树、东部杨木、盐芥、马铃薯二倍体甘蓝、乳寡妇草、hall’s panicgrass、柳枝稷、绿藻和团藻(图1)。1和表S1).
循环系统发育树SF1植物可利用的基因家族。的系统发育树SF1基于最大似然JTT + G模型构建植物基因,使用PhyML v3.037。从59种植物中选取92个蛋白质序列,计算系统发育关系,用于建树。在每个主要分支上标记引导值。每个转录本的对应信息如物种名称、共同名称、已识别的转录本数量和转录本ID(命名法)见表S1(基于APG-IV系统的分类法)
基因结构与保守基序分析
有必要对植物的外显子-内含子结构和保守基序进行比较SF1阐明它们的进化过程和潜在功能。基因结构模型SF1基因被附加到系统发育树(图。2),并显示各自的内含子相(图。2、表S2).数字2(中,面板)显示的基因长度和结构的每个成员SF1家庭表现出显著的差异。例如,92个基因中23个成员的基因结构SF1家族基因不含内含子序列;这个子集占全体成员人数的15.7%。48序列SF1基因有2个外显子-1个内含子组织,占所有基因的52.2%。特别是一些来自藻类的基因具有多个外显子,包括Vocar.0008 s0294.1。p (Volvox carteri)其中外显子最多(19个)。此外,在同一分支上还观察到不同的基因结构。例如,来自的两个序列玉米玉米(Zm00008a037777_P01, 3个外显子和Zm00008a007621_P01, 4个外显子)具有独特的基因结构。虽然SF1s的每个成员的基因结构发生了实质性的异化,但我们发现CDSs的长度没有显著变化(图1)。2).因此,是否影响其基因功能的分化还有待进一步研究。利用多Em Motif Elicitation (MEME)搜索工具对保守母题的进一步研究表明,大多数SF179个基因序列特征相似,序列顺序相同,均包含10个分析基序,其中一个序列除外Micromonas pusilla(50949)的位置不同。2右面板)。虽然在基被子植物、单子叶植物和高子叶植物之间的保守基序没有明显差异,但同一种植物的序列却存在不同的基序(图1)。2).例如,Aqcoe5G406900.1。p和Aqcoe7G039300.1。来自欧迪科特的P耧斗菜coerulea分别有10个和9个图案。同样的情况也出现在d .胡萝卜;DCAR_006843、DCAR_008506和DCAR_004968分别有10个、9个和10个基序。有趣的是,DCAR_008506的cd长度是最长的。值得注意的是,一些来自藻类和苔藓的序列具有较少的保守基序。例如,在苔藓植物中Physcomitrella金属盘(Pp3c7_10890V3.1。p和Pp3c11_24710V3.1.p),泥炭藓fallax(Sphfalx0015s0077.1。p和Sphfalx0010s0197.1.p)Marchantia polymorpha(Mapoly0009s0189.1.p)有九个主题。在藻类植物中,来自微单胞菌的145,219和62,857序列分别只有7个和6个基序。此外,虽然序列Volvox carteri(Vocar.0007 s0345.1。p和Vocar.0008 s0294.1.p)衣藻reinhardtii(Cre12.g553750.t1.1和Cre09.g386731.t1.1)包含多个外显子,共有9个基序,说明其序列变化对功能类影响不大。进一步对SF1外显子区域进行相关性分析,以阐明内含子的增益/损失。植物SF1s转录本之间的相关性如图所示。3.,为系统发育分析提供了额外的信息。例如PGSC0003DMT400081859与migat . d02531.2之间的外显子存在多个精确匹配,因此两者之间的相似性更大。
多肽中蛋白质结构域和保守基序的分析
利用上述筛选的59种植物92个肽序列对蛋白质结构域进行分析;肽段注释与剪接因子相关,根据模因分析检索的肽段序列预测保守蛋白基序(图1)。4).因此,我们发现所有的SF1s在多肽的n端都有SF1_HH n端结构域,然后是KH结构域和c端结构域,即RNA识别基序(RRM)(图1)。4中间面板)。有趣的是,在藻类中,3个多肽来自m . pusilla(145219),诉carteri(Vocar.0008 s0294.1.p)和c . reinhardtii(Cre09.g386731.t1.1)有两个RRM域。SF1蛋白的氨基酸长度在499 - 1583 aa之间,大部分含有700 - 800个氨基酸(表1)S3).它们中的大多数都是大约700到800个氨基酸的长度。亚细胞定位预测显示大多数SF1蛋白具有核定位(86,93.4%)(表S3).此外,蛋白质30147。m014250 (萝藦)和Migut.F01191.1。p (Mimulus guttatus)位于液泡内;蛋白质的Traes_2DL_6F03F05FA.4(t . aestivum)及145,219 (m . pusilla)被预测为细胞质;GSMUA_Achr5P25100_001 (穆萨acuminata)和Cre09.g386731.t1.1 (c . reinhardtii)分别位于叶绿体和内质网。
对SF1肽序列进行模因分析,共预测了10个保守基序,这些基序以彩色框表示,并覆盖了大部分蛋白质(图1)。4右面板)。进一步分析表明,77个多肽具有全部10个基序,约占研究中所分析的所有SF1蛋白序列的83.7%。有趣的是,所有来自苔藓的序列在分析中都有10个保守的基序,这表明苔藓植物中SF1蛋白是保守的。此外,几乎所有的eudicots都有10个保守的母题——除了Anacardium occidentale(Anaoc.0018 s0425.1.p)和c .开大花的(Cagra.3782 s0026.1.p)缺少motif 2和马吕斯有明显(MDP0000558834),草莓属vesca(mrna21192.1-v1.0-hybrid)和芸苔属植物拉伯(Brara.C01481.1.p),缺少基序10,而大多数单花有8个保守基序。相比之下,藻类植物由于其具有完整的蛋白结构域的多肽,只拥有大约一半的预测10个基序,这意味着藻类植物SF1蛋白的保守和分化程度最低。所有藻类共有的T基序为基序3、基序4、基序5和基序9。
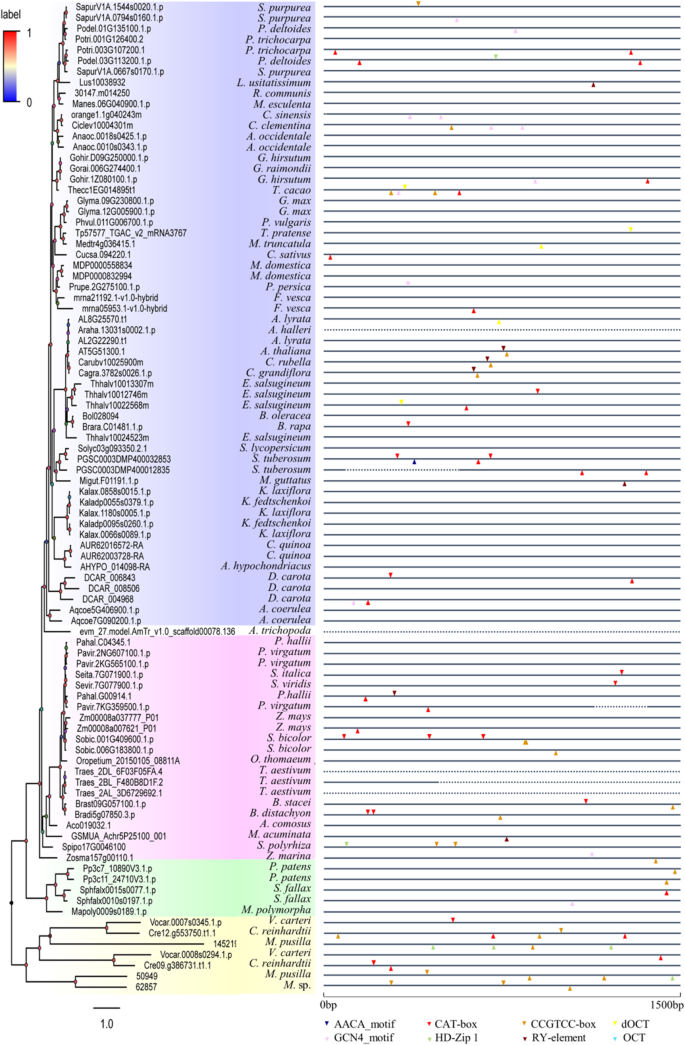
启动子分析及组织特异性表达SF1基因
进一步分析植物的调控作用SF1转录水平的基因,植物上游1.5 kb序列SF1从Phytozome数据库中获取基因,然后进行独联体使用PlantCARE程序识别每个启动子的元素(表S4) [37].结果表明,共预测出108个图案。一般来说,八独联体-选择其中与组织特异性表达相关的元素(图。5和表S4),包括用于栅栏叶肉细胞分化的HD-Zip1,调控种子特异性表达的RY-element,参与胚乳表达的AACA_motif和GCN4_motif,以及用于分生组织表达的CAT-box、CCGTCC-box、dOCT和OCT。进一步分析表明,只有50个启动子SF1具有组织特异性调控的基因独联体元素。其中CAT-box和CCGTCC-box在基因启动子中出现频率最高、丰度最高SF1基因。它们都调控分生组织特异性表达,在植物的发育和生长过程中发挥关键作用。一致,紫色假雀梅(Brachypodium distachyon)不仅具有CAT-box和CCGTCC-box,而且在幼叶、节间、不定根和根中均有高表达(图5)。5和图S2).然而,没有发现连接两者高表达的基序SF1年代的大豆(大豆)在SAM和根尖(图S1).此外,AACA_motif仅在茄属植物tuberosum(PGSC0003DMP400032853),提示其在调控胚乳特异性阴性表达中具有特异作用。此外,HD-Zip 1存在于Podel.03G113200.1中。p (摘要(东部杨木)和Spipo17G0046100的Spirodela polyrhiza(更大的浮萍)。在双子代模型植株的启动子中检测到ry元素拟南芥在干种子中也有低表达拟南芥(无花果。6),表明ry -元素参与了种子特异性的表达拟南芥。同一组织类型在不同生长阶段表达量差异显著;例如,花第15期雄蕊的表达水平拟南芥明显高于其他花发育阶段。但不同生长阶段的表达水平茄属植物lycopersicum在番茄的启动子中未发现基序(图5)。5而且S1).此外,检测到不同的表达模式SF1具有多个拷贝的基因(图;6,S1而且S6).例如,在两个细胞中检测到相似的组织表达谱SF1双字谜中的同义词杨树trichocarpa(potri . 001g12640.1和Potri.003G107200.1)和单子叶玉米(Zm00008a007621_P01和Zm00008a037777_P01S1而且S5).相比之下,两个SF1基因的美国tuberosum表现出不同的表达模式,类似于g·马克斯(无花果。6而且S1).
植物组织特异性相关基序分析SF1启动子区域。八个顺式作用的图案用不同颜色的三角形表示。这些被识别的基序的位置被标记在每个基序的1.5 kb 5 '侧面区域SF1基因。实线和虚线分别表示具有碱基对的区域和无序列的区域或附加的碱基N的区域。线上面的符号表示正链上的图案,而线下面的符号表示负链上的图案。基序的功能:aaca基序,参与胚乳特异性阴性表达;CAT-box,顺式作用的分生组织表达调控元件;CCGTCC-box,顺式作用的分生组织特异性激活调控元件;与分生组织特异性激活相关的顺式作用调控元件dOCT;GCN4_motif,顺式调控元件参与胚乳表达;参与栅栏叶肉细胞分化的蛋白HD-Zip1;ry -元件,顺式作用的调控元件,参与种子特异性调控。 The black vertical lines represent break at that particular branch; OCT, cis-acting regulatory element related to meristem specific activation
启动子及内外激素表达分析SF1基因
植物在长期的进化和发展中,逐渐形成了适应和抵抗逆境的机制,以维持生命和持续生长。了解内外刺激对植物的调控机制SF1s在PlantCARE数据库中,研究了激素和应激相关的顺式作用元件(图1)。7、表S4).从92个植物启动子序列中筛选出19个与激素和胁迫相关的基序SF1s.包括脱落酸(ABRE)、生长素(AuxRE、AuxRE-core、TGA-box、TGA-element)、乙烯(ERE)、赤霉素(gar -motif、P-box、TATC-box)、MeJA (CGTCA-motif、TGACG-motif)和水杨酸(TCA-element)等12个激素相关的基序,以及低温(LTR)、干旱(MBS)、损伤(WUN-motif)和缺氧(are、GC-motif)等5个应激相关的基序。几乎每一个SF1序列有很大的多样性独联体除了一些序列如arha .13031 s0002.1和Traes_2AL_3D6729692.1由于序列中含有' N '或不含启动子而不包含单个基序外,表明多种激素介导的信号通路密切相关SF1植物抗性。分析显示,超过一半的SF1启动子分别为ABRE、CGTCA-motif、TGACG-motif和ARE。此外,外界激素信号也影响SF1成绩单(图S3).例如,在拟南芥(at5g5130.1), MJ(茉莉酸甲酯)抑制其表达(图。7)和其他激素如ACC(乙烯的前体)、IAA(生长素)、ABA和GA(赤霉素)处理调节at5g5130.1的表达。
植物基序相关激素及胁迫分析SF1启动子区域。19个顺式作用元件用不同的颜色符号表示。这些被识别的基序的位置被标记在每个基序的1.5 kb 5 '侧面区域SF1基因。实线和虚线分别表示具有碱基对的区域和无序列的区域或附加的碱基N的区域。线上面的符号表示正链上的图案,而线下面的符号表示负链上的图案。基序功能:ABRE,参与脱落酸反应的顺式作用元件;ARE,顺式作用的厌氧诱导必要的调节元件;AT-rich序列,最大引物介导激活元件(2拷贝);AuxRE,生长素响应元件的一部分;auxrr核心,参与生长素反应的顺式作用调节元件;cgtca motif,参与meja响应性的顺式作用调控元件;之前,ethylene-responsive元素; GARE-motif, gibberellin-responsive element; GC-motif, enhancer-like element involved in anoxic specific inducibility; LTR, cis-acting element involved in low-temperature responsiveness; TATC-box, cis-acting element involved in gibberellin-responsiveness; TCA-element, cis-acting element involved in salicylic acid responsiveness; MBS, MYB binding site involved in drought-inducibility; P-box, gibberellin-responsive element; TC-rich repeats, cis-acting element involved in defence and stress responsiveness; TGA-box, part of an auxin-responsive element; TGACG-motif, cis-acting regulatory element involved in the MeJA-responsiveness; TGA-element, auxin-responsive element; WUN-motif, wound-responsive element
蛋白质-蛋白质相互作用网络及结构守恒分析
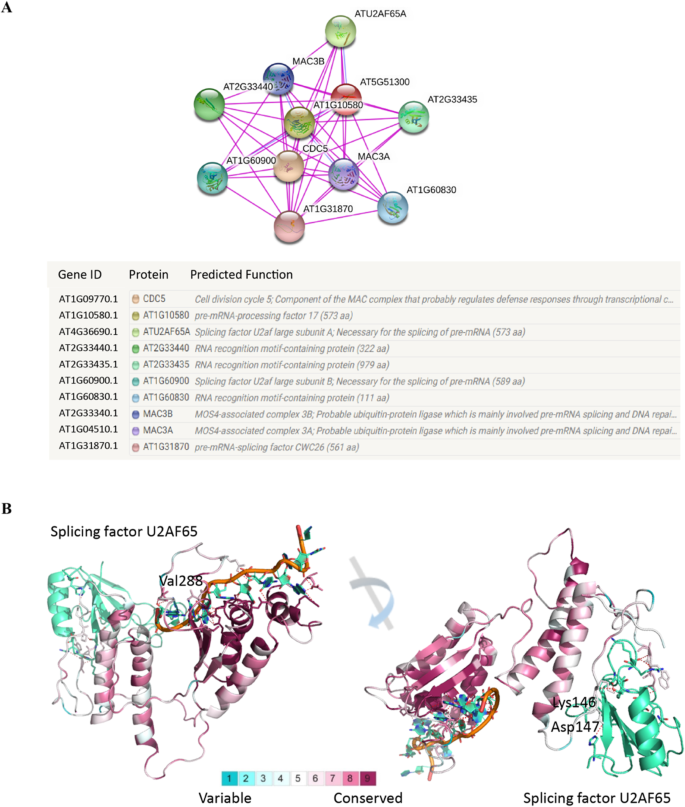
蛋白质-蛋白质相互作用(Protein-protein interaction, PPI)网络分析可以系统地揭示蛋白质在生物系统中的工作原理、生物信号和能量代谢的分子机制以及蛋白质之间的功能关系。在本研究中,我们根据具有代表性的蛋白质序列生成了SF1蛋白的蛋白-蛋白相互作用网络拟南芥(AT5G51300),使用基于实验的STRING数据库(图5。8a).最后得到SF1蛋白的10个预测功能伙伴,包括CDC5 (AT1G09770.1)、AT1G10580 (AT1G10580.1)、ATU2AF65A (AT4G36690.1)、AT2G33440 (AT2G33440.1)、AT2G33435 (at2g3345.1)、AT1G60900 (at1g609001)、AT1G60830 (AT1G60830.1)、MAC3B (AT2G33340.1)、MAC3A (AT1G04510.1)、AT1G31870 (AT1G31870.1)(图1)。8a). CDC5、MAC3A和MAC3B是MAC复合体的组成部分,可能通过转录控制调节防御反应,因此对植物固有免疫至关重要。它们都可能参与了mrna前剪接和DNA修复。AT1G10580是pre- mrna处理因子17,AT1G31870是剪接因子CWC26。这两种蛋白质都参与RNA剪接和pre-mRNA加工。AT2G33440、AT2G33435和AT1G60830是含有RNA识别基元的蛋白,其主要分子功能涉及pre-mRNA剪接位点结合。ATU2AF65A和AT1G60900分别是剪接因子U2af大亚基A和B,是pre-mRNA剪接所必需的。AT5G51300(剪接因子样蛋白1)已经被证明是pre-mRNA的剪接、发育和脱落酸(ABA)反应所必需的。一般来说,SF1蛋白及其功能伙伴通常参与RNA剪接和pre-mRNA加工,其中一些也具有对细菌防御反应的功能(图1)。8一个)。
的答:芥SF1蛋白包括三个结构域:剪接因子1螺旋发夹结构域(残基:126-237)、KH结构域(残基:244-330)和RNA识别结构域(残基:482-552)。多序列比对显示,这些结构域的保守性相对较高S4),表明这些基因的功能相似。此外,根据两种晶体结构,利用同源建模方法重构了剪接因子1螺旋发夹域和KH域的三维模型(图5)。8b).第一结构域(螺旋-发夹结构域)与U2AF65 (UHM)形成二级疏水界面[80].第二个结构域(KH结构域)存在于多种核酸结合蛋白中[15].因此,我们将U2AF65 (2M0G)和RNA (1K1G)的晶体结构叠加在同源建模的结构上,观察其相互作用。ConSurf等级越高的残留物越保守。198个(74.4%)残留的ConSurf Grade超过7级,111个(41.7%)残留的ConSurf Grade超过9级。更重要的是,RNA的结合区域高度保守。8b).除Val288外,所有进口残留物的ConSurf等级均高于7。288位残基具有相似的理化性质,如Val和Ile。另一个结构域没有像剪接因子1螺旋发夹结构域那样保存下来,其中有一个环与U2AF65相互作用。然而,重要残基的ConSurf Grade相对较高,只有两个残基(Lys146和Asp147)的ConSurf Grade小于7。在低等植物中,这两个残基被Ile, Gly, Tyr, Thr, Ala和Gly, Ser或His所取代。与此同时,它们在许多物种中消失了。因此,这些域的函数是守恒的。RNA结合域比U2AF65结合域保守得多,尤其是在低等植物中。
讨论
众所周知,成熟的mRNA是通过顺序结扎外显子来形成的,以维持蛋白质翻译的特定阅读框[60].在人类中,几乎所有注释的蛋白质编码基因都经历了另一种剪接[56,75].在植物中,超过80%的含内含子基因表现为剪接异构体[11,82].此外,剪接的过程受到早期剪接体组装过程中剪接位点的初步识别的严格控制。因此,负责这种识别的蛋白质对于真核生物中剪接的基因控制研究和提供有价值的靶点是非常重要的[35,71].为此,连接5 '和3 '剪接位点确定复合体的分支点结合蛋白SF1成为剪接位点选择的关键成分。
植物结构与功能保护的比较SF1基因
在本研究中,我们系统地对92种植物进行了特征分析SF1来自59个不同物种的基因。虽然超过50%(34/59)的这些物种保持了一个拷贝SF1基因中,26种植物中含有多种SF1成员(表S1),显示它们的功能冗余。有趣的是,大多数SF1除了几个藻类序列外,其他基因只有一个外显子编码目标蛋白产物。2),表明一种古老的基因转位复制事件可能影响了该基因家族在植物谱系中的进化[24].然而,还需要进一步的证据来证实这一假设。在分子水平上,SF1是介导早期剪接体组装和剪接位点识别的重要成分。因此,人们对其在动物和植物中的分子功能进行了大量的研究。例如,据报道,SF1蛋白的初级氨基酸序列和结构域在真核生物中是保守的,如酵母、人类、后生动物和植物[2,6,30.,47].SF1蛋白通常有三个结构域:KH/QUA2、锌指和RRM [36].然而,植物SF1蛋白已被证实含有额外的RRM结构域,但缺乏uhm特异性特征[36].之前的一项研究表明,在ABA处理下,截断的没有RRM结构域的植物SF1蛋白仍然具有足够的pre-mRNA剪接活性[36].因此,这一额外结构域在植物中的潜在作用有待进一步研究。此外,据报道,翻译后修饰如KIS激酶丝氨酸磷酸化可增强SF1-U2AF65-RNA三复合体的组装[45,80]或在拼接位点识别过程中引入其他拼接因子[2,28].
植物功能多样化SF1基因的表达模式
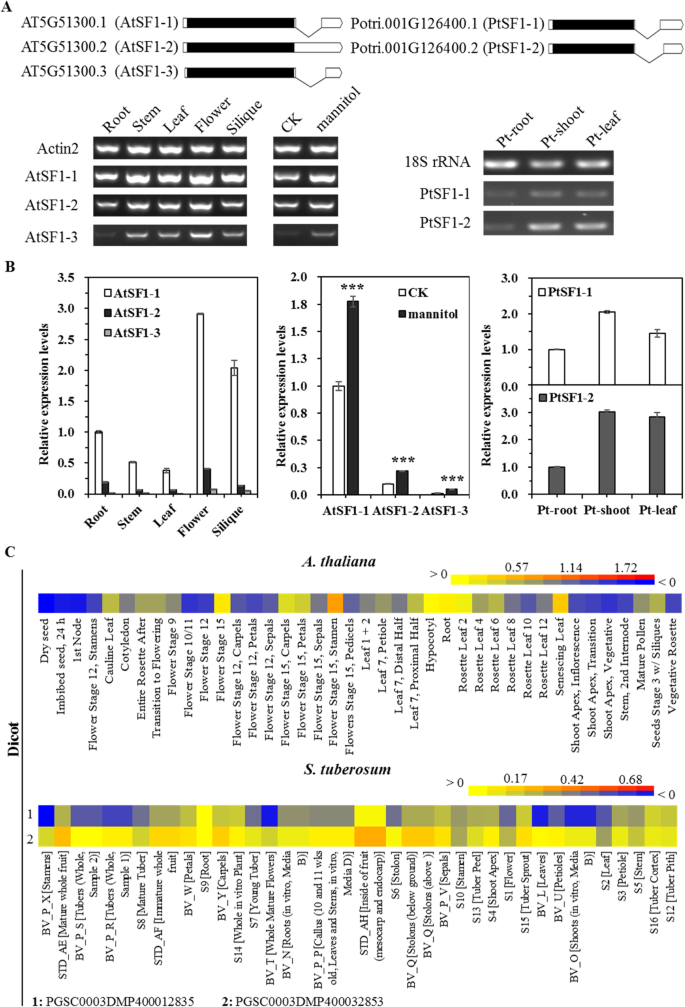
SF1被认为是连接5 '和3 '剪接位点定义复合体的关键成分。此外,大量证据表明SF1在多种真核生物的剪接位点识别过程中发挥着关键作用[46,52,68].然而,它在细胞活力中的作用仍有争议。越来越多的证据表明,SF1可能不是生存能力所必需的,可能只控制植物和动物的基因子集[22,83],表明除了sf1介导的剪接位点识别外,可能还存在另一种机制[23,46,72].此外,SF1的功能可进一步受到细胞、组织或器官特异性的影响。例如,鼠标SF1在大脑和心脏中已经检测到转录本,这意味着它们在转录水平上的组织特异性调控[83].此外,SF1在分化的绒毛细胞中高度表达,但在肠上皮的腺瘤或未分化的肠隐窝细胞中未观察到[49].有趣的是,在植物中,SF1被发现参与了许多植物的发育过程和应激反应[30.,36].特别是,SF1已被观察到影响开花时间和叶片大小拟南芥而且杨树,与其在花部位和叶片中的相对高表达相一致(图。6a).重要的是,通过我们的qRT-PCR和RT-PCR表达分析,SF1剪接异构体也表现出与SF1相似的表达模式,这意味着SF1的表达和剪接差异在花和叶发育过程中存在相互调控(图1)。6a和b)。同时,甘露醇处理强烈诱导与它们亚型相关的SF1表达,表明其与干旱胁迫有关。此外,SF1转录本在几个单子叶和优生子叶中分布不均(图5)。6c,S1而且S2),表明它们在这些物种的植物发育过程中具有潜在的作用。
与组织特异性相比,更多独联体在植物的启动子区域内观察到参与激素和胁迫反应的-元素SF1基因(图。7和表S5),表明它们在应对内外刺激时的假定作用。的拟南芥SF1已被证实参与ABA信号传导[30.,36],与ABRE基序在其自身5 '侧区的存在相吻合(图。7).此外,拟南芥SF1治疗1 h后IAA诱导,MeJA (MJ)抑制。在其启动子区域观察到的auxrr -核和cgtca基序可能负责这一调控(图。7).然而,为了在未来的功能研究中进一步强化这一假设,还需要进一步利用生物信息学和实验数据进行综合研究[9,10].
剪接位点确定复合体的组成揭示了真核生物外显子-内含子边界的分化机制
一般来说,真核生物sf1在介导早期剪接位点识别方面具有类似的分子功能。具体地说,拟南芥SF1被认为具有类似于酵母或后生动物的功能[30.,36].然而,不同的真核生物可能在早期剪接体组装过程中通过SF1进化出自己的识别机制。首先,与动物和植物的序列相比,SF1的靶BPS在酵母中是不同的。特别是,酵母内含子BPS是一个保守的7核苷酸序列(UACUAAC),而哺乳动物SF1已报道结合更多的简并序列(YNCURAY;N,任何核苷酸;R, A或G;Y, C或U) [32].在此阶段,在线虫和植物中未观察到保守的BPS [40,42].这就提出了SF1如何识别这些生物中的BPS以及植物中额外的RRM是否有助于这种识别的问题[30.].第二,不同的生物存在不同的协调机制。例如,哺乳动物U2AF65作为SF1协调3’剪接位点识别的相互作用伙伴,与U2AF小亚基(U2AF35)相互作用。在裂变酵母中发现了类似的相互作用复合体,美国非洲酒,除了小的U2AF亚基命名为U2AF23 [69].相比之下,出芽酵母缺乏一个类似u2af35的小U2AF因子,另外两个蛋白(BBP/SF1和Mud2p/U2AF65)被认为在拼接过程中形成稳定的复合体[55].此外,动物的剪接反应需要U2AF65与BPS下游的Py序列结合,而这两种成分对酵母剪接都不是必需的[1,59].有趣的是,与动物相比,植物表现出一种独特的剪接模式。例如,在植物中观察到内含子保留事件的高比例,而在动物中外显子跳过是主要的AS类型[55].SF1被认为可以提高含有弱保守的3 '剪接位点的内含子的剪接效率秀丽隐杆线虫[41].因此,我们很容易推测这种差异可能是由于动物和植物对sf1中心剪接位点的识别不同。
结论
在这项工作中,我们全面确定了92SF159种植物的序列,从藻类到杜鹃花。随后进行了系统发育和表达分析,以阐明该基因家族的保守性和功能调控。通过考虑SF1在剪接位点识别中的连接作用,我们假设植物SF1可能与动物的SF1重叠,但也具有不同的功能。了解植物中该蛋白家族的分子机制,为通过植物剪接的基因控制来操纵作物性状提供了有趣的可能性。
数据和材料的可用性
数据包含在本文及其支持文件中。
缩写
- SF1 / BBP:
-
剪接因子1/支点结合蛋白
- 个基点:
-
分歧点序列
- RRM:
-
RNA结合主题
- 为:
-
可变剪接
- snRNPs:
-
小型核核糖核蛋白
- U2AF:
-
U2 snRNP辅助因子
- KH / QUA2:
-
/颤2 K的同源性
- hnRNP:
-
异构核糖核蛋白
- 嗯:
-
U2AF同源性主题
- 阿坝:
-
脱落酸
- SF1-HH:
-
剪接因子1螺旋发夹结构域
- KH_1:
-
同源域
- RRM_1:
-
RNA识别主题
- 遗传算法:
-
赤霉素
- 国际宇航科学院:
-
生长素
- 答:
-
甲基jasmonate
- PPI:
-
蛋白质相互作用
- ML:
-
最大似然
- MEME:
-
多Em用于母题诱导
参考文献
- 1.
Abovich N, Liao XC, Rosbash M.酵母MUD2蛋白:与PRP11的相互作用在承诺复合体和U2 snRNP添加之间定义了桥梁。基因Dev。1994;8:843-54 . .
- 2.
哺乳动物酵母承诺复合体中的跨内含子桥接作用是保守的。细胞。1997;89:403-12 . .
- 3.
哺乳动物剪接因子SF1由变异的cdna编码,并与RNA结合。Rna-a出版Rna Soc. 1996; 2:794-810 ..
- 4.
Ashkenazy H, Abadi S, Martz E, Chay O, Mayrose I, Pupko T, Bental N. ConSurf 2016:估算和可视化大分子进化守恒的改进方法。核酸Res. 2016;44: W344-50 ..
- 5.
Bailey TL, Boden M, Buske FA, Frith M, Grant CE, Clementi L, Ren J, Li WW, Noble WS。模因套件:发现和搜索模因的工具。核酸Res. 2009;37: W202-8 ..
- 6.
Berglund JA, Chua K, Abovich N, Reed R, Rosbash M.剪接因子BBP与pre-mRNA分支点序列UACUAAC特异性相互作用。细胞。1997;89:781 . .
- 7.
Camacho C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K, Madden TL. BLAST plus:架构和应用。BMC生物信息学。2009;10:421 . .
- 8.
陈超,瑞鑫,郝超,何颖。TBtools,一个集成了多种hts数据处理工具的界面友好的生物学家工具包;2018 . .
- 9.
引用本文陈MX,孙超,张坤龙,宋永春,田艳,陈旭,刘永刚,叶宁华,张建华,曲淑琴,朱飞*。swth - ms促进了富士苹果和红皮芽运动突变体之间果皮的蛋白质组学分析。BMC Plant Bio. 2019; 19:45 - 55..
- 10.
陈明霞,朱永飞,高斌,马坤龙,叶乃华,张玉军,Fernie AR,陈霞,胡庆军,田勇,刘泰,张建华,刘永刚。基于单分子长读序列的蛋白质基因组学识别水稻基因的复杂基因组组织及其编码能力植物杂志。2020;182:1-17 . .
- 11.
陈MX,朱永飞,王芳珍,叶乃华,高斌,陈曦,赵胜生,范涛,曹云云,刘泰,苏喆,谢立军,胡庆军,吴海军,肖申,张俊,刘永刚。选择性剪接和转译在水稻低氧萌发中起着重要作用。J Exp Bot. 2019; 70:817-33 ..
- 12.
达米安S,安德烈F,斯蒂芬W,克里斯托弗F,达维德H,杰米HC,米兰S,亚历山大R,阿尔贝托S,察福KP。STRING v10:蛋白质-蛋白质相互作用网络,集成在生命树中。核酸学报2015;43:D447..
- 13.
埃德加钢筋混凝土。MUSCLE:多序列对齐,高精度,高吞吐量。核酸学报,2004;32:1792-7 .
- 14.
Finn RD, Jody C, William A, Miller BL, Wheeler TJ, Fabian S, Alex B, Eddy SR. HMMER web服务器:2015年更新。核酸文献2015;43:30-8 ..
- 15.
García-Mayoral MF, Hollingworth D, Masino L, Díaz-Moreno I, Kelly G, Gherzi R, Chou CF, Chen CY, Ramos a。KSRP的c端KH结构域的结构揭示了一个对mRNA降解很重要的非规范基序。结构。2007;15:485 - 98 . .
- 16.
Garrey SM, Rodger V, Andrew J, B.酵母分支点结合蛋白的扩展RNA结合位点及其锌节结构域在RNA结合中的作用。生物化学杂志2006;281:27443-53 ..
- 17.
KH结构域出现在一系列不同的rna结合蛋白中,包括抗阻断剂NusA,可能与核酸结合有关。2月。1993;324:361-6 . .
- 18.
Goodstein DM,盛强S, Russell H, Rochak N, Hayes RD, Joni F, Therese M, William D, Uffe H, Nicholas P. Phytozome:绿色植物基因组学比较平台。核酸学报2012;40:D1178-86 ..
- 19.
Gozani O, Potashkin J, Reed R. U2AF-SAP 155交互在招募U2 snRNP到分支站点中的潜在作用。Mol Cell biology . 1998; 18:4752-60 ..
- 20.
Graveley BR。替代剪接:增加蛋白质组学世界的多样性。趋势麝猫。2001;17:100-7 . .
- 21.
Guindon S, Dufayard JF, Lefort V, Anisimova M, Hordijk W, Gascuel O.估计最大似然系统发育的新算法和方法:评估PhyML 3.0的性能。系统医学杂志。2010;59:307-21 . .
- 22.
U2AF识别多嘧啶束后,哺乳动物SF1/BBP在剪接体组装和功能中的动力学作用。生物化学杂志2000;275:38059-66 .
- 23.
海虹S, Green MR. RS通过人体在酵母中通过一种常见机制接触剪接信号并促进剪接。基因Dev。2006;20:1755 - 65 . .
- 24.
Hofberger JA, Nsibo DL, Govers F, Bouwmeester K, Schranz ME。在十字花科植物中,串联和全基因组复制的复杂相互作用驱动了l型凝集素受体激酶基因家族的扩增。基因组生物学进展2015;7:20 20 - 34..
- 25.
Horton P, Park K-J, Obayashi T, Fujita N, Harada H, Adams-Collier CJ, Nakai K. WoLF PSORT:蛋白质定位预测器。核酸Res. 2007;35(Web Server): W585-7 ..
- 26.
胡斌,金杰,郭爱英,张宏,罗杰,高刚。GSDS 2.0:升级版的基因特征可视化服务器。生物信息学。2014;31:1296 . .
- 27.
胡志生,孙勇,陈建军,赵逸尔,乔红,陈瑞,温晓辉,邓玉强,温建军。脱氧雪腐菌醇通过抑制剪接因子U2AF1和SF1在全球范围内影响人类细胞中3’剪接位点的选择。RNA杂志。2020;17 (4):584 - 95 . .
- 28.
Ingham RJ, Karen C, Caley H, Sabine D, Lim CSH, Joanna Y, Kadija H, Judith R, Gerald G, Geraldine M. WW结构域为多蛋白网络的组装提供了平台。Mol Cell biology . 2005; 25:7092-106 ..
- 29.
Jana K, Sophie HM, Angela KM, Igor V.控制替代剪接的分支位点单倍型。Hum Mol Genet. 2004; 13:3189-202 ..
- 30.
张英华、朴慧英、李金c、Thu议员、金淑坤、徐mc、姜浩、金金坤。剪接因子SF1的同源物对拟南芥的发育至关重要,并参与了pre-mRNA的替代剪接。植物j . 2014; 78:591 - 603 . .
- 31.
隐马尔可夫模型速度启发式迭代HMM搜索过程。Bmc生物信息学。2010;11:431 . .
- 32.
Keller EB, Noon WA。内含子剪接:动物前信使rna内含子中保守的内部信号。美国国家科学研究院1984;81:7417-20。
- 33.
Kenichi Y, Masashi S, Yuichi S, Daniel N, Yasunobu N, Ryo Y, Yusuke S, Aiko SO, Ayana K, Masao N.骨髓异常发育中剪接机制的频繁通路突变。大自然。2011;478:64 . .
- 34.
Kiana T, Brady SM, Ryan A, Eugene L, Provart NJ。植物学阵列资源:e- northern,表达角度和启动子分析。植物j . 2005; 43:153 - 63 . .
- 35.
Kotake Y, Sagane K, Owa T, Mimorikiyosue Y, Shimizu H, Uesugi M, Ishihama Y, Iwata M, Mizui Y.剪接因子SF3b作为抗肿瘤天然产物pladienolide的靶点。Nat Chem Biol. 2012;3:570..
- 36.
李建昌、尹浩杰、金sk、朴海、Thu MP、李志华、金jk。拟南芥剪接因子SF1的RRM结构域对特定基因的pre-mRNA剪接非常重要。植物细胞学报2017;36:1-13 ..
- 37.
Lescot M, Dehais P, Thijs G, Marchal K, Moreau Y, Van De Peer Y, Rouze P, Rombauts S. PlantCARE,植物顺式作用调控元件数据库和启动子序列分析工具门户。核酸Res. 2002a; 30:325-7…
- 38.
林凯,陆锐,谭文威。含有WW结构域的蛋白与早期剪接体相互作用,在体内参与pre-mRNA剪接。分子细胞生物学。2004;24:9176-85 .
- 39.
刘卓,Luyten, I., Bottomley, m.j., Messias, a.c., Houngninou-Molango, S., Sprangers, R., Zanier, K., Krämer, A., Sattler, M.(2001)。剪接因子1识别内含子分支位点RNA的结构基础。科学294,1098 - 1102 . .
- 40.
Long M, Horvitz HR。在体内,秀丽隐杆线虫U2AF大亚基UAF-1的突变改变了3 '剪接位点的选择。公共科学图书馆麝猫。2009;5:e1000708 . .
- 41.
M龙,T志平,T燕玲,Sebastian H, Horvitz HR。在体内U2AF大亚基和SF1/BBP对线虫体内内含子保留和外显子跳过的影响。Rna-a - Publication Rna Soc. 2011;17: 221 - 11..
- 42.
高等植物中Pre-mRNA的剪接。植物科学,2000;5:160-7。
- 43.
Madhusudhan MS, Marti-Renom MA, Eswar N, John B, Pieper U, Karchin R, Shen MY, Sali A.比较蛋白质结构建模。Curr Protoc Bioinform. 2014;47:5.6.1编辑委员会,Andreas D. Baxevanis…(等). .
- 44.
Makarova OV, Makarov EM, Lührmann R. U4/U6的65和110 kDa sr相关蛋白。U5三snrnp对成熟剪接体的组装至关重要。EMBO j . 2014; 20:2553 - 63 . .
- 45.
Manceau V, Swenson M, Caer JL, Sobel A, Kielkopf C, Maucuer A.相邻Ser-pro基序上SF1的大量磷酸化增强了与U2AF的相互作用(65)。2月j . 2010; 273:577 - 87 . .
- 46.
Margherita C, Nicolas A, Goranka T, Mihaela Z, Angela KM。对人剪接因子SF1的原位pre-mRNA靶点的分析揭示了其在选择性剪接中的作用。核酸决议2011;39:1868..
- 47.
Mazroui R, puti A, Krämer A.来自果蝇和线虫的剪接因子SF1: n端RS结构域的存在和生存需求。Rna-a - Publication Rna Soc. 1999; 5:1615-31 ..
- 48.
一个atp独立复合体将pre-mRNA提交到哺乳动物剪接体组装途径。基因Dev。1991;5:2534 . .
- 49.
Miki S, Yasuyoshi N, Masashi I, Kazufumi H, Masaya O, Setsuo H, Tesshi Y.剪接因子-1在-连环蛋白/ t细胞因子4介导的基因转激活和pre-mRNA剪接中的作用。胃肠病学。2007;132:1039-54 . .
- 50.
莫C,曼利JL。选择性剪接调控机制:来自分子和基因组学方法的见解。细胞生物学。2009;10:741-54 ..
- 51.
Mount SM, Pettersson I, Hinterberger M, Karmas A, Steitz JA。U1小核rna -蛋白复合体在体外选择性结合5 '剪接位点。细胞。1983;33:509-18 . .
- 52.
Noriko H, Tomoko A, David F, Tokio T. SF1-U2AF59-U2AF23复合体的突变导致裂糖酵母的外显子跳变。生物化学杂志2007;282:2221-8 ..
- 53.
Park HY, Lee HT, Lee JH, Kim JK。拟南芥U2AF65调控花期和花粉管生长。植物科学进展2019;10:569..
- 54.
Park HY, Lee KC, Jang YH, Kim SK, Thu MP, Lee JH, Kim JK。拟南芥的剪接因子AtU2AF65、AtU2AF35和AtSF1在细胞核和细胞质之间穿梭。植物细胞学报2017;36:1113-23 ..
- 55.
强伟,李振,伯特L,雷蒙德BC。一个BBP-Mud2p异质二聚体在萌芽酵母中调节分支识别并影响剪接底物的丰度。核酸Res. 2008; 36:2787-98 ..
- 56.
Qun P, Ofer S, Lee LJ, Frey BJ, blencwe BJ。通过高通量测序对人类转录组中可选剪接复杂性的深度调查。Nat麝猫。2008;40:1413-5 . .
- 57.
Rain JC, Rafi Z, Rhani Z, Legrain P, Krämer A.人与酿酒酵母pre-mRNA拼接因子SF1中RNA结合和蛋白-蛋白相互作用的功能域的守恒。Rna-a - Publication Rna Soc. 1998; 4:551-65 ..
- 58.
mrna前剪接保真度的机制。Curr Opin Cell Biol. 2000;12:34 - 5.
- 59.
Rutz B, Seraphin B. BBP/ScSF1和Mud2与剪接机制的瞬时相互作用影响剪接体组装动力学。RNA。1999; 5:819-31 . .
- 60.
Sasaki-Haraguchi N, Ikuyama T, Yoshii S, Takeuchi-Andoh T, Frendewey D, Tani T.与19复合体结合的Cwf16p确保了裂变酵母中有秩序的外显子连接到构成的pre-mRNA剪接中。《公共科学图书馆•综合》。2015;10:e0136336 . .
- 61.
Schmittgen TD, Livak KJ。比较CT法分析实时PCR数据。Nat Protoc。2008;3:1101-8 . .
- 62.
Selenko P, Gregorovic G, Sprangers R, Stier G, Rhani Z, Krämer A, Sattler M.人剪接因子U2AF65与SF1/mBBP分子识别的结构基础。摩尔细胞。2003;11:965 - 76 . .
- 63.
陈世鹏,陈秀珍。prp19p相关复合体在剪接体激活中的作用。科学。2003;302:282-1 . .
- 64.
Shitashige M, Satow R, Honda K, Ono M, Hirohashi S, Yamada t。增加Sf1(+/−)小鼠对偶氮甲烷诱导的结肠肿瘤的易感性。癌症科学。2010;99:1862-7 . .
- 65.
Sickmier EA, Frato KE, Shen H, Paranawithana SR, Green MR, Kielkopf CL。关键前mrna剪接因子U2AF65识别多嘧啶束的结构基础。摩尔细胞。2006;23:49-59 . .
- 66.
Siomi H, Matunis MJ, Michael WM, Dreyfuss G. pre-mRNA结合K蛋白包含一个新的进化保守基序。核酸Res. 1993;21:1193..
- 67.
斯特利JP,格思里C.剪接体的机械装置:马达,时钟,弹簧和其他东西。细胞。1998;92:315-26 . .
- 68.
人的剪接因子SF3a在体内对pre-mRNA剪接是必需的,而不是SF1。Mol Biol Cell. 2005; 16:1366-77 ..
- 69.
Tao H, Josep V, Query CC. s.p pombe中前剪接体的形成需要SF1-U2AF的稳定络合物(59)-U2AF(23)。EMBO j . 2014; 21:5516-26 . .
- 70.
黄晓明,王晓明。后生动物中pre-mRNA的选择性剪接和蛋白质组扩展。大自然。2002;418:236-43 . .
- 71.
上原T, Minoshima Y, Sagane K, Sugi NH, Mitsuhashi KO, Yamamoto N, Kamiyama H, Takahashi K, Kotake Y, Uesugi M.抗癌磺酰胺选择性降解剪接因子CAPERα。Nat Chem Biol. 2017;13:675..
- 72.
Valcárcel J, Gaur RK, Singh R, Green MR. U2AF65 RS区与pre-mRNA分支点的相互作用及与U2 snRNA碱基配对的促进作用[更正]。科学。1996;273:1706-9 . .
- 73.
Vincent G, Olivier G, Micheline FR, Alper R, Alain J, Ulf N.酵母中未拼接mrna的核保留是由核周Mlp1介导的。细胞。2004;116:63 - 73 . .
- 74.
剪接体:动态RNP机的设计原理。细胞。2009;136:701-18 . .
- 75.
王et, Rickard S, Shujun L, Irina K, Lu Z, Christine M, Kingsmore SF, Schroth GP, Burge CB。人类组织转录组中的替代异构体调控。大自然。2008;456:470-6 . .
- 76.
王赟,徐磊,Thilmony R,尤fm,顾永强,Coleman-Derr D. PIECE 2.0:植物基因结构比较和进化数据库的更新。核酸Res. 2016;45:1015..
- 77.
威尔,c.l.,施耐德,a.m.,麦克米伦,卡托波迪斯,n.f.,纽鲍尔,G.,威尔姆,M., Lührmann, R.,查询,C.C.(2014)。一种与pre-mRNA分支位点相关的新型U2和U11/U12 snRNP蛋白。EMBO J 20,4536 - 4546 . .
- 78.
沃登AZ、李JH、莫克T、鲁泽P、西蒙斯MP、阿尔茨AL、艾伦AE、库维利埃ML、德雷埃E、埃弗雷特MV、富伦E、格里姆伍德J、刚拉克H、亨里萨特B、纳波利C、麦克唐纳SM、帕克MS、隆鲍茨S、萨拉莫夫A、冯DP、巴杰JH、库蒂尼奥PM、德米尔E、杜布切克I、绅士C、艾克雷姆W、格雷迪JE、约翰U、拉尼尔W、林德奎斯特EA、卢卡斯S、梅尔。海洋皮真核生物微单胞菌基因组揭示的绿色进化和动态适应。科学。2009;324:268 - 72 . .
- 79.
袁松,陈洪春,胡铮。基于PyMOL的计算药物设计平台。Wiley interdiscirev计算分子科学。2017;7:e1298..
- 80.
Yun Z, Tobias M, Ivona B, Thomas K, Hyun-Seo K, Peijian Z, Nina MU, Sieber SA, Angela KM, Michael S. 3 ' -剪接位点识别过程中剪接因子1 n端域的磷酸化和U2AF65结合。核酸Res. 2013; 41:1343-54 ..
- 81.
Zamore PD, Patton JG, Green MR.哺乳动物剪接因子U2AF的克隆及结构域。大自然。1992;355:609-14 . .
- 82.
朱永飞,陈MX,叶宁华,石磊,马克亮,杨建峰,曹云云,张颖,Yoshida T, Fernie AR.拟南芥幼苗脱落酸反应中选择性剪接和转译的蛋白质基因组学分析。植物j . 2017; 91:518-33 . .
- 83.
朱瑞,Heaney J, Nadeau JH, Ali S, Matin A.剪接因子1缺乏抑制睾丸生殖细胞肿瘤的发生。实用癌症杂志2010;70:7264 - 72 . .
确认
不适用。
资金
本研究得到国家自然科学基金项目(NSFC31701341)、江苏省自然科学基金项目(SBK2020042924)、南京理工大学项目资助项目(GXL2018005)的资助,主要负责研究设计和数据收集。广东省自然科学基金(2018A030313030)和湖南省自然科学基金(2019JJ50263)在数据分析和解释方面发挥作用。香港中文大学深圳研究院深圳虚拟大学园资助计划(YFJGJS1.0)及香港研究资助局(AoE/M-05/12, AoE/M-403/16, GRF14160516, 14177617, 12100318)负责修改稿件。
作者信息
从属关系
贡献
fy.z、M.X.C和c.w设计了实验。K.L.Z, Z.F和J.F.Y.进行了实验。K.L.Z, J.F.Y, Y.T和m.x.c分析了数据。kl.z和m.x.c写了手稿。G.F.H、Y.M.F和j.h.z对手稿进行了批判性的评论和修改。所有作者均已阅读并认可该手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者没有需要声明的利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
补充信息
额外的文件1:
:图S1。的表达模式大豆(大豆),茄属植物lycopersicum(番茄)和杨树trichocarpa(杨)SF1年代。图S2。的表达模式Brachypodium distachyon(紫色false布罗姆)SF1.图S3。的表达拟南芥SF1基因受多种植物激素处理的影响。
额外的文件2:
图S4。植物SF1蛋白序列的多重比对。
额外的文件3:
图S5。的表达模式玉米(玉米)和高凉菜属fedtschenkoi(二倍体高凉菜属)SF1年代。
额外的文件4:
图S6。RT-PCR未裁剪的凝胶照片。
额外的文件5:
表S1。SF1从59种植物中鉴定出的基因。表S2。植物的特点SF1基因结构。表S3。预测了植物SF1蛋白的亚细胞定位。表S4。的信息独联体-植物中确定的元素SF1s.
额外的文件6:
表S5。在植物5 '侧面区域识别的基序列表SF1s.
额外的文件7:
表S6。RT-PCR和qPCR分析用引物。
权利和权限
开放获取本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的用途,您将需要直接从版权所有者那里获得许可。欲查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非在数据的信用额度中另有说明。
关于这篇文章
引用这篇文章
张坤龙,冯志明,杨建峰,冯志明。et al。分枝点结合蛋白、剪接因子1、基因家族在植物发育和胁迫响应中的系统表征。BMC植物杂志20.379(2020)。https://doi.org/10.1186/s12870-020-02570-6
收到了:
接受:
发表:
关键字
- 可变剪接
- 表达谱
- 系统发生学
- 植物
- 启动子
- 剪接因子